Method Article
절대 단백질 정량 질량 분석을 모니터링 선정 반응
요약
This protocol describes how to perform absolute quantification assays of target proteins within complex biological samples using selected reaction monitoring. It was used to accurately quantify proteins of the mouse macrophage chemotaxis signaling pathway. Target peptide selection, assay development, and qualitative and quantitative assays are described in detail.
초록
Absolute quantification of target proteins within complex biological samples is critical to a wide range of research and clinical applications. This protocol provides step-by-step instructions for the development and application of quantitative assays using selected reaction monitoring (SRM) mass spectrometry (MS). First, likely quantotypic target peptides are identified based on numerous criteria. This includes identifying proteotypic peptides, avoiding sites of posttranslational modification, and analyzing the uniqueness of the target peptide to the target protein. Next, crude external peptide standards are synthesized and used to develop SRM assays, and the resulting assays are used to perform qualitative analyses of the biological samples. Finally, purified, quantified, heavy isotope labeled internal peptide standards are prepared and used to perform isotope dilution series SRM assays. Analysis of all of the resulting MS data is presented. This protocol was used to accurately assay the absolute abundance of proteins of the chemotaxis signaling pathway within RAW 264.7 cells (a mouse monocyte/macrophage cell line). The quantification of Gi2 (a heterotrimeric G-protein α-subunit) is described in detail.
서문
질량 분석기 (MS)를 사용하여 실험을 프로테옴 비 타겟 (산탄) 또는 타겟팅 방법을 사용하도록 설계 될 수있다. 디스커버리는 일반적 프로테오믹스 전통적인 데이터 의존적 획득 모드를 사용하여, 또는 (예를 들어, MS E, SWATH) 최근에 개발 된 데이터 독립 기술 중 하나를 사용하여 1,2- 어느 상향식 산탄 MS에 의존한다. 샷건 단백질 체학은 높은 처리량 펩타이드 식별 및 상대 정량을위한 강력한 도구이지만, 절대 정량 또는 단백질의 작은 정의 세트 (~ 수십) 대상에 대한 일반적으로 적합하지 않다. 가장 자주 사용되는 표적 단백질 체학 MS 방법이 높기 때문에 감도, 속도 및 동적 범위 3-5 반응 모니터링 (SRM)을 선택한다. SRM 대안 고해상도 풀 스캔 MS 6 활용 병렬 반응 모니터링을 포함한다.
SRM은 대개 나노 유동 되돌리기를 사용하여 수행SED 상계 고속 액체 크로마토 그래피 (RP-나노 LC) 나노 - 전기 분사 이온화 결합 악기 삼중 사중 극 질량 분석 장치 (QQQ-MS)에 부착 (나노 ESI) 이온 소스. 전형적인 실험에서, 단백질 시료는 단백질 분 해적으로 분해하고, 생성 된 펩티드는 크로마토 그래피 분리하고, 탈착 및 이온화된다. 얻어진 전구체 이온 m / 제 중극 (Q1)에 의해 필터링 Z와 충돌 가스로 충돌에 의해 제 2 중극 (Q2)에서 조각난. 생성 된 단편 이온은 m / z 필터링 셋째 중극 (Q3) 및 다이 노드에 의해 정량화. 각각의 전구체 및 단편 이온 쌍 천이로 지칭되고, 각각의 전이는 지정된 기간 (전환 시간 전형적으로 2-50 밀리 초)에 대해 모니터링한다. LC-SRM 동안 전환을 통해 미리 정의 된 목록 QQQ-MS 사이클 (듀티 사이클은 통상적 ≤3 초이다), 각 전환의 크로마토 그램이 생성된다.
대체 전략에서단백질 정량 이거는 일반적으로 도트 말, 서양 오 점, ELISA를, 항체 마이크로 어레이, 역상 단백질 마이크로 어레이, 미세 유체 면역, 디지털 ELISA를, 그리고 마이크로 스피어 기반의 면역 7로 면역을 사용합니다. 최상의 면역은 LC-SRM보다 훨씬 더 민감 할 수 있으며, 면역 샘플 처리량 LC-SRM 5보다 상당히 높을 수있다. 그러나, 현상 면역 비용이 될 수 및 / 또는 시간 소모적, 생성 분석법은 세포 / 조직 용해 / 균질화 방법과 호환, 교차 반응성 및 / 또는 간섭에 취약 할 수 있으며, 및 / 또는 의무하지 5,8-를 멀티플렉싱. 이러한 문제의 일부는 항체와 MS 기반 기술을 결합함으로써 해결 될 수있다. 예를 들면, 표적 단백질 및 단백질 분해 전에 LC-9-12에 SRM을 사용하여 면역 침강 농축 될 수있다. 대안 적으로, 기술은 SISCAPA 펩티드 LEVE에서 단백질 분해에 후속 면역 침전을 채용L (13, 14). 또한 전략 immunoenrichment하는 높은 풍부 단백질은 분석 immunodepletion 15,16 coeluting하여 간섭을 감소함으로써 LC-SRM 감도를 증가시키기 위해 사용될 수있다.
MS 기반 단백질 정량은 라벨이없는 안정 동위 원소 라벨 (예를 들어, 신진 대사 라벨, 화학 라벨, 무거운 표지 단백질과 펩티드 내부 기준)으로도 상대 및 절대 정량으로 구분 될 수있다. 라벨이없는 기술은 상대적으로 단백질 정량에 유용 할 수 있지만, 정확한 절대 정량에 적합하다 할 수 있습니다. 이와 비교하여, 라벨링 기술 샘플 준비 및 MS 차이와 연관된 에러를 감소하고, 상대적으로 자주 단백질 정량화 (17)에 사용된다. 예를 들어, 안정 동위 원소 표지 프로테옴 (SILAP) 표준은 인간 혈청 (18)의 LC-SRM을 통해 잠재적 인 바이오 마커의 상대 정량을 활성화 배양 된 인간 세포 라인을 사용하여 제조. MS에 의해 정확한 절대 단백질 정량은 정제, 정량화, 동위 원소 표지 단백질 또는 펩티드 내부 표준 아군-로하는 것이 MS에 앞서 생체 시료이 필요합니다. LC-SRM 워크 플로우에 무거운 동위 원소 표지 내부 표준의 통합은 높은 재현성과 실험실 16, 19 사이의 양도로 나타났다 절대 정량 할 수 있습니다.
MS에 의해 절대 단백질 정량 안정 동위 원소 표지 된 내부 표준 펩타이드 표준 고체상 합성 (20) 연접 프로테아제 절단 펩티드 표준 21 이루어지는 단백질, 및 전장 단백질 22 표준을 사용하여 제조 포함한다. 대상 단백질 공유 수정 및 불완전한 샘플 준비 (즉, 불완전한 샘플 용해 및 균질화, 불완전 단백질의 가용화, 변성, 알킬화, 및 단백질 분해는) 정확한 정량을 훼손 할 수 있습니다. 내부 Protein 표준은 이러한 잠재적 인 문제의 대부분에 의해 영향을받을 가능성이있는 이상, 그러나 일반적으로 제조하기가 가장 어렵다. 대안은 아미노 - 및 카르복시 말단 측면 네이티브 잔기를 포함하도록 설계된 여러 펩티드 내부 표준을 사용하여 각각의 표적 단백질을 분석하는 것이다. 에 관계없이 내부 표준의 유형이 사용되는의, 그것은 아군-로한다 생물학적 시료 이른 시점에서 가능한 한 샘플 준비 중. 또한, 다수의 시료 전처리 기법 (예를 들어, 다른 변성 조건) 시험해야한다. 다수의 직교 실험 기술 (실험 교차 유효성 검사)의 사용은 23 ~ 25 도전 가장 가능성 정량을 극복하기위한 실행 가능한 전략이다.
단백질의 LC-SRM 정량은 광범위한 응용 분야에서 사용 된 매우 유연한 방법이다. 특히, 이는 내 펩티드 및 단백질 바이오 마커를 연구하는 데 사용 된혈청, 핵심 조직 검사, 미세 침 흡인 (5) 임상 샘플. LC-SRM은 또한, 보툴리눔 신경 독소 (27)를 검출하는 신호 전달 경로 (5) 내에 단백질 인산화 역학 정량화, 단백질 입체 구조 (28)의 변화를 정량화하기 위해, 단백질 복합체 5,26의 화학량 론을 측정하는 데 사용되어왔다.
본 연구실은 화성 경로 시뮬레이션의 개발을 지원하는 대 식세포 주 화성을 매개하는 신호 전달 단백질을 정량화하는 LC-SRM을 사용한다. 프로토콜의 전체 계획 (그림 1) 임시 목표 펩티드를 순위로 시작합니다. 그 후, 원유 외부 펩타이드 기준은 합성 및 생체 시료의 정성 분석을위한 LC-SRM 분석을 개발하는 데 사용됩니다. 생물학적 샘플 유래의 대상 펩티드가 검출되는 경우, 정제 된 무거운 표지 된 펩티드 내부 표준 정량적 LC-SRM을 위해 준비된다. 이 프로토콜은 AC에 사용될 수있다curately 생물학적 시료의 다양한 단백질 정량화, 단백질 표적의 다양한 연구를 지원한다.
프로토콜
참고 :이 방법은 이전에 (56)을 설명하고있다.
1. 펩타이드 대상 선택
- 표적 단백질의리스트를 컴파일하고, 생물학적 시료에 걸쳐 정규화 가사 단백질의 적은 수를 포함하고, 또한 내부 표준 단백질 (예를 들어, 개똥 벌레 루시퍼 라제)를 포함한다. 단백질 분해 29,30 시뮬레이터 등의 소프트웨어 툴을 사용하여 실리에서 트립신 펩티드로 표적 단백질 다이제스트.
- 트립신 펩티드가 완전히 소실되고 트립신 절단 부위를 포함하지 않는 것을 요구한다. 잠재적 불완전 트립신 소화 (31)을 방지하기 위해 이웃 트립신 절단 부위와 펩티드를 피하십시오.
- 각 펩티드의 길이는 5 내지 20 잔기 인 것을 요구한다. 이상 펩티드를 고려하지만, 일반적으로 합성하는 것이 더 비용이 있습니다.
- 그것은 자연적인 유전자 변형 (예를 들어, 단일 nucleotid에 해당하는 경우 펩타이드를 피전자 다형성). 또한, 트립신 사이트 (1.2 단계)의 영향을받지 않습니다 있는지 확인합니다.
- 이 번역 후 변형 (PTM)의 사이트에 해당하는 경우 펩타이드을 피하십시오. 이 LC-MS 샘플 준비하는 동안 화학 수정하는 경향이 될 경우 유사 펩타이드을 피하십시오. 특히, 시스테인과 메티오닌 산화, 아스파라긴과 글루타민 탈 아미드 화 및 피로 글루타민산의 아미노 - 말단 글루타민 형성하는 경향이 펩티드를 피할 수 있습니다. 트립신 사이트 (1.2 단계)의 영향을받지 않습니다 있는지 확인합니다.
- 또한, 단백질의 말단은 번역 후 변형 (아미노 - 말단 메티오닌 손실과 아세틸, 및 카르복시 말단 아미드)하는 경향이 있기 때문에 단백질 아미노 및 카르복시 말단에서 유래 펩티드를 피할 수 있습니다.
- 각 대상 펩타이드는 각 대상 단백질에 고유해야합니다. 이것이 가능하지 않다면, 펩티드 이성체 / 동족체의 세트에 고유 한 것을 필요로한다. 만 류신 / 이소류신 대체하여 다른 펩타이드 치료마치 이러한 LC-SRM 동안 거의 동일하게 수행하기 때문에 펩타이드 고유성을 결정할 때 그들은 동일했다.
- 생물학적 시료의 전체의 포괄적 인 transcriptomic 분석 (예, RNA-SEQ)를 수행 한 경우, 전 사체의 번역에 실리 사용 고유성 펩티드를 결정하는 대신에 32 종의 전체 프로테옴을 사용 시퀀스.
- 대상 펩티드 proteotypic 것을 요구합니다. 생물학적 시료의 산탄 총 질량 분석 1, 2를 사용하여 proteotypic 펩타이드를 확인하는 연구되고있다. 대안 적으로, NIST의 질량 분석 온라인 프로테오믹스 데이터베이스에서 proteotypic 펩티드의 펩티드 식별을하는 라이브러리 검색 33 (http://peptide.nist.gov/), 글로벌 프로테옴 기계 (34) (X HTTP에서 헌터 스펙트럼 라이브러리를! // WWW http://gpmdb.thegpm.org/에서 .thegpm.org / 헌터 / index.html을하고 GPMDB 펩타이드 식별), PeptideAtlas (35) 까지 (http://www.peptideatlas.org/)과 프라이드 데이터베이스 36 (http://www.ebi.ac.uk/pride/archive/).
- 정량적 사체 수준이 수행 된 생체 시료 분석하는 경우 (예를 들어, RNA-SEQ, qPCR의 예), 비교적 낮은 사체 풍부 펩티드에 대응하는 타겟팅 피한다.
- 표적 단백질의 orthologs LC-SRM 분석에 사용될 수 선택 대상 펩티드를 필요한 경우 (예를 들어, 인간과 목적 단백질의 마우스 형태 모두를 분석하기 위해).
- (가능한 경우) 표적 단백질 당 두 개 이상의 대상 펩티드를 선택합니다.
펩타이드 표준 2. 준비
주 : 프로토콜이 섹션 하류 분석 용 스물 동결 건조 펩티드 표준 (양의 각 인 1 nmol의) 세트의 제조를 설명한다. 펩티드의 상이한 수를 들어 다른 펩티드 또는 수량에 대해 그에 따라 조정될 필요가있을 것이다.
- 이러한 SPOT 합성 20,37 같은 펩티드 합성 기술을 사용하여 동결 건조 된 펩티드 수준 (nmol의 ≥1)을 준비한다.
- carbamidomethylated되는 외부 표준 펩티드의 시스테인 잔기 (요오도 아세트 아미드의 알킬화에 의해 형성되는 화학 구조를) 확인. 반대로, 내부 표준 펩타이드의 시스테인 잔기가 수정되지 않은되도록 (이는 후술 샘플 준비 동안에 알킬화 될 것이다).
- 그들은 아미노 및 카르복시 말단 기본 잔류 측면을 포함 할 수 있도록 내부 펩타이드 표준 디자인 (트립신 소화 효율을 제어하는 각은 일반적으로 3-6 잔류 길이).
- 그들은 안정 동위 원소 표지 있도록 내부 펩타이드 표준을 디자인합니다. 펩티드 표지 전략을 선택할 때 타겟 펩타이드 천연 동위 원소 프로파일 및 MS의 질량 측정의 정확도를 고려한다. 중수 소화 표준 펩티드를 사용하지 않는다 펩티드 중수 원인 역상 LC 보유 시간 때문에S (38)을 이동합니다.
참고 : 일반적으로, 트립신 내부 펩타이드 표준이 순수한 ~ 98 %를 사용하여 합성된다 [13 C 6 15 N 4]의 Arg와 [13 C 6, 15 N 2] 카르복시 말단에 통합리스. - HPLC (39) 사용하여 내부 펩타이드 기준을 정화, 정확하게 그 (40)를 정량화.
- V / V 포름산은, 동결 건조 된 펩타이드로 10 μM의 펩타이드 농도 (사용 관리를 생산하는 각 1 nmol의 동결 건조 된 펩타이드 표준에 20 % v / V를 아세토 니트릴 (ACN)는 매우 낮은 밀도를 가질 수있다 0.1 %의 100 μl를 추가 등)을 용이하게 손실 될 수있다. 2 분 및 목욕 샘플은 5 분을 초음파 처리 소용돌이는 펩타이드 분해가 완료 확인합니다.
- 용해 펩티드 풀, 80 μL의 최종 부피로 진공 농축기에서 생성 된 혼합물을 농축시킨다. 어떤 침전 된 펩타이드를 용해 ACN의 20 μl를 추가합니다.
참고 : 샘플을 지금 공동ntains 각각의 펩타이드 20 % V / V의 ACN의 10 μm의. - 내부 펩타이드 표준에 대한 정량적 인 LC-SRM에 대한 희석 시리즈를 준비 :
- 1 μm의 희석 100 μl를 준비 : 20 % V / V의 ACN의 90 μL와 10 μm의 펩타이드 혼합물의 10 μl를 결합합니다.
- 100 nm의 희석 100 μl를 준비 : 20 % V / V의 ACN의 90 μL은 1 μm의 펩타이드 혼합물의 10 μl를 결합합니다.
- 외부 펩타이드 표준, 0.1 % V / V 포름산의 90 μL와 10 μm의 펩타이드 혼합물의 10 μl를 결합하여 LC-MS에 대한 1 μm의 희석 100 μl를 준비합니다.
참고 : 샘플은 현재 각 1 펩티드의 μM, V / V 포름산 0.1 %, 2 %의 V / V의 ACN을 포함하고, LC-SRM 분석 개발에 사용 될 준비가되어 있습니다.
3. LC-SRM 분석 개발
- 나노 유동 HPL을 사용하여 MS를 산탄 외부 펩타이드 표준 (분사 당 펩타이드의 약 1-10 pmol의)의 혼합물을 분석삼중 사중 극자 질량 분석기 (LC-QQQ-MS (/ MS))에 결합 된 C 시스템.
- C-18 미디어 (≤5 μm의 직경 ~ 200 Å 기공 길이 ≥10 cm, ID = 50 ~ 100 μm의)을 충전 한 모세관 컬럼이 장착 된 LC-MS 시스템 및 나노 ESI 팁 (전형적으로 사용하여 제조를 사용 레이저 풀러). 각 LC-MS 런이 ~ 60 분 선형 구배 (일반적 0~40% 용매 B), 열 재생 단계 (~ 80 % 용매 B), 및 열 재 평형화 단계 (0 ~ % 용매 B를 포함하는지 확인 ) (용매 = 0.1 % V / V H에서 포름산 2 O, 용매 B = 0.1 % v / V를 ACN에 포름산은, 유량 = 200-800 NL / 분, ESI 전압 = 1800 V, 3 분기 격리 폭 = 0.7 M / Z, 2 분기 아르곤 압력 = 1.5 mTorr로).
- 각각의 전구체 이온 스캔, 동적 상단을 선택 ~ 탠덤 질량 분석 (MS / MS)에 대한 10 개의 가장 강렬한 전구체 이온. 최적 +2 이온 전구체를 프래그먼트하기위한 하나와 다른 두 가지 충돌 에너지 램프가 사용되도록 중복 각 샘플을 실행+3위한 전구체 (41)를 이온.
- LC-QQQ-MS (/ MS)은 (Q2에서 저에너지 아르곤 충돌에 의한 프리 커서 이온 피크의 테일링을 일으킬 수 Q1 전구체 이온 스캔) Q3 전구체 이온 스캔을 사용하여 분석을 수행한다.
- LC-MS 시스템은 품질 관리 샘플 및 기술 복제를 분석하여 적절하게 수행하고 있음을 확인합니다. 갓 만든 LC 컬럼을 사용하여 샘플 사이에 여러 공백을 실행하여 샘플 이월을 방지합니다.
- 외부 펩티드 (42)의 기준 서열에 대해 데이터베이스를 검색하여 얻어진 샷건 LC-QQQ-MS (/ MS)의 데이터를 분석한다. 해당되는 경우, 펩타이드 식별 신뢰 점수 (예를 들어, 기대 값) 또는 통계 모델링 (42)를 사용하여를 사용하여 모호한 펩티드 식별을 폐기합니다. 에 관계없이, 수동으로 그들은 모두 모호하지 못하도록하기 위해 펩타이드 식별 (43)을 모두 검토합니다.
- 사기에 샷건 MS 펩티드 식별을 사용하여이러한 스카이 같은 소프트웨어 프로그램을 사용하여 스펙트럼 라이브러리 구조체.
- 전구체 이온 (당 3-10 가장 강렬한 천이를 이용한 LC-SRM 전이리스트를 준비 +2와 +3 전구체 이온; +1과 +2 단편 이온, Y-, B-, 및 ≥2 잔기 인 이온 긴).
- 전구체 및 단편 이온 m / z 값 QQQ-MS의 질량 측정의 정확도 내에서 겹치는 경우 전구체 이온 분열이 불완전 때때로 (전이를 버리고, 모노 이소 토픽과 무거운 천연 동위 원소 형태뿐 아니라 비 표지를 고려하는 기억 ) 형태의 무거운 표지.
- 단편 이온의 m / z가 동일한 전구체 이온으로부터 다른 단편 이온의 겹치면 마찬가지로 천이를 버린다.
- LC-SRM 외부 펩타이드 표준의 혼합물로 분석 수행 결과 전환 목록을 사용하여 (단계 3.1에 설명 된대로 질량 분석을 수행, 1 분기와 2 분기 격리 폭 = 0.7 M / Z; 드웰 시간 = 2-50 밀리 초).
- 엄마nually 어떤 실적이 저조한 분석 (LC-SRM 데이터의 분석 제 5 장에서 설명)를 결과 LC-SRM 데이터를 검토하고 삭제합니다.
생물학적 시료 4. LC-SRM 분석 실험
- 배양 된 세포 (44)의 요리를 준비합니다. 여러 실험 조건은, 블록을 비교하여 모든 가능한 45 체계적인 편향을 감소 샘플을 랜덤 화되는 경우.
- 셀들의 각각의 접시를 들어, 무 혈청 버퍼 (44)에 세포를 낭비하고 중단하는 세포 배양 배지를 흡인. 필요하다면, 남아있는 세포 외 단백질을 제거하는 무 혈청 완충액으로 세포를 세척 하였다.
- 각 샘플에 대해, 생존력 얼룩 (예를 들면, 트립 판 블루)과 혈구 또는 자동 세포 계수기 (44)를 이용하여 실행 가능한 총 셀의 개수를 카운트.
- 원심 분리하여 세포를 펠렛 44 낭비 상층 액을 흡인 (전형적인 세포 펠렛 부피는 30 μL ~하다). 각각 (각 세포 펠렛 (100 mM의 HEPES ∙ NaOH로 pH를 8 염산염 bestatin 10 μm의, 펩 스타틴 10 μm의 및 중 8 M 요소 0.1 % W / V 계면 활성제에 우레아 용해 버퍼 또는 계면 활성제 용해 버퍼 400 μl를 추가 ) 갓 제조 된 그러한 RapiGest SF 또는 PPS 사일런트 계면 활성제로서 산해리 MS 호환 계면 활성제를 사용). 다른 단백질 분해 효소 억제제를 사용하는 경우, 그들은 트립신을 억제하지 않도록.
- 비즈 누출 스냅 캡 튜브를 일으킬 수 있습니다 (~ 포함 0.1 mm의 지르코니아 / 실리카 비드의 100 μl를 부드러운 피펫을 사용하여 각 샘플을 혼합하고, (O 링과 나사 캡) 2 ML 튜브에 각각 전송 ). 를 Lyse 최고 속도로 5 분 동안 샘플을 볼 텍싱에 의한 세포 (이것은 비드 쳐서 세포 lyses).
- 대안 적으로, (프렌치 프레스 또는 균질화 팁을 사용하여, 예) 다른 기계적인 방법을 사용하여 세포를 용균.
참고 : 세포 용 해물을 원심 분리하지 마십시오 오티 수있는이 침전 된 단백질을 펠릿 수 있기 때문에herwise tryptically 분해 및 LC-MS에 의해 검출된다.
- 대안 적으로, (프렌치 프레스 또는 균질화 팁을 사용하여, 예) 다른 기계적인 방법을 사용하여 세포를 용균.
- 계면 활성제 변성 된 샘플의 경우, 샘플 균질화 및 단백질 변성을 지원하기 위해 10 분 동안 90 ° C에서 그들을 부화.
- 목욕 균질화 및 단백질 변성을 돕기 위해 실온에서 10 분 동안 샘플을 초음파 처리.
주 : 해물 및 세포 용해 완충액은 -80 ° C에서 저장 될 수있다 (용해 버퍼는 단계 4.9 및 4.13에 대해 필요한 것). - 용 해물의 단백질 농도 분석 (46) (예를 들면, 산 bicinchoninic 분석)을 수행 (그리고 대조 실험으로서 용해 완충액).
- 신선한 1.5 ml의 microcentrifuge 관에 질적 분석 (즉, 어떤 내부 펩타이드 표준 아군하지-으로됩니다 샘플) 세포 용 해물의, 피펫 200 μg의 (단백질 질량)를 참조하십시오. 정량 분석의 경우, 안정 동위 원소 희석 시리즈 네 같은 샘플을 준비합니다.
- 각 샘플에 대해, 내부 표준 단백질 (예, <추가/ EM>) 98 % 순수한 반딧불의 루시 페라 ~의 ~ 5 pmol의를 추가합니다.
- 정량 분석을 위해, 내부 표준 펩타이드의 등몰 혼합물의 안정 동위체 희석 계열을 추가 (예를 들면, 0, 0.2, 2, 각 20 pmol의 펩티드) 샘플.
- 이와 병행하여, 단독으로 내부 표준 펩타이드를 이용한 대조 시료를 준비 (즉, 세포 용 해물 없음) (예를 들면, 0, 0.2, 2, 각 20 pmol의 펩티드).
- 또한 외부 및 내부 표준 펩타이드하여 대조 시료 준비 (예 : 2 각 외부 펩티드 표준 pmol의, 0, 0.2, 2 및 각 내부 표준 펩타이드를 20 pmol의).
주 : 트립신 소화를 방해 할 수 있으므로 펩타이드 기준은 포름산이 없는지 확인합니다.
- 모든 샘플은 동일한 양이되도록 용해 완충액을 추가한다. 주의 : 전형적인 샘플 부피는 70 μL, 그래서이 볼륨이 프로토콜을 사용한다.
- 0을 추가하여 단백질 시스테인 잔기를 줄입니다.각 샘플 (DTT 최종 농도가 10 mM의 경우) 및 30 분 동안 60 ° C에서 샘플을 인큐베이션 (새로 제조) 1 M DTT 7 μL.
- 킬레이트 단백질 버퍼링 요오도 아세트 아미드로 7 μl를 첨가하여 시스테인 (500 mM의 요오도 아세트 아미드, 1 M HEPES ∙ NaOH로 pH를 8; 새로 제조) 각 샘플 (최종 요오도 아세트 아미드 농도가 50 ㎜이다)과 어둠 속에서 실온에서 20 분 동안 샘플을 인큐베이션 (요오도 아세트 아미드 민감한 빛). 필요한 경우, 50 mM의 최종 농도로 DTT를 첨가하여 남아있는 요오도 아세트 아미드를 급냉.
- 각 샘플은 우레아를 사용하여 변성 들어 (트립신 크게> 1 M 우레아 (47)에서 금지된다) 우레아 최종 농도가 1 M이되도록 100 mM의 HEPES ∙ NaOH로 pH를 8 482 μL를 추가한다.
- Tryptically 펩티드로 단백질을 소화.
- 세포 용 해물을 포함하는 각 샘플의 경우, 0.5 μg의 / μL 시퀀싱 등급 수정 트립신의 8 μl를 추가 할 수 있도록 최종 트립신 정광트립신 : 샘플, 18 시간 동안 37 ° C에서 샘플을 품어 : 이온 1:50 (W 단백질 W)입니다.
- 세포 용 해물을 포함하지 않는 각 샘플 0.5 μg의 / μL 시퀀싱 등급 변성 트립신 0.5 μL를 추가하고, 2 시간 (이들 샘플을 각각 제어 실험이며, 일반적으로 10 NG를 포함하여 37 ° C에서 샘플을 배양 용 펩티드 + 단백질 -10 μg의 질량).
- 우레아 변성 된 샘플들 각각에 대해 2 % V / V 포름산 440 μL를 추가 (포름산의 최종 농도는 1 %이다 V / V). 이러한 샘플의 pH ~ 3 있음을 확인합니다.
- 계면 활성제 변성 샘플들의 각각은, 0.5 % V의 914 μl를 들어 추가 / TFA V (TFA의 최종 농도가 0.5 % 인 V / V). 이러한 샘플의 pH ~ 1.5 있는지 확인합니다. 60 분은, 계면 활성제의 가수 분해를 위해 37 ℃에서 이러한 샘플을 인큐베이션.
- 마이크로 원심 모든 샘플 계면 활성제 꼬리 기 및 기타 PREC 펠렛을 실온에서 20 분 동안 21,000 × g에서C-18 SPE 카트리지를 막히게 ipitates.
- 고체상는 일회용 C-18 SPE 카트리지를 사용하여 상층 액을 각각 추출 (버퍼 = 0.1 % V / V 포름산, 버퍼 B = 0.1 % v / V를 포름산, 80 % V / V의 ACN; ~ 100 mg의 C- 카트리지 당 18 수지). 또는 5 분 동안 10 × g에서 15 mL의 원심 분리 튜브에 카트리지를 원심 분리하여 추출 매니 폴드를 이용하여, 평탄한 표면이 고무 전구를 이용한 C-18 SPE 카트리지를 통해 이동상 강제.
- B 완충액 1 ㎖를 가하여 열 젖은, 회 완충액 1 ㎖를 적용하여 평형 샘플을 적용하고, 회 완충액 1 ㎖를 가하여 카트리지를 세척한다. 천천히 완충액 B 1㎖를 적용 (~ 2 분)에 의해 펩티드를 용출.
- ACN을 멀리 증발 100 μL의 최종 볼륨에 진공 농축기의 각 용출액을 집중한다. 각 샘플에 H 2 O의 200 μL를 추가하고 가능한 재를 멀리 증발 98 μL의 최종 볼륨에 진공 농축기의 각을 집중maining ACN.
- 각 샘플에 ACN 2 μL 5 %의 V / V 포름산을 추가 (최종 샘플의 농도는 0.1 % v / V를 포름산, 2 % V / V의 ACN입니다).
주의 : 샘플을 -80 ° C에서 저장 될 수있다. - (단계 3.4에서와 같이) LC-SRM을 사용하여 샘플을 분석한다. LC-SRM 정성 분석의 경우, 생물학적 시료와 외부 펩티드 표준을 실행하고, 동시에 생성 된 모든 데이터를 분석한다. 정량적 LC-SRM 분석은 각 생물학적 샘플의 동위체 희석 시리즈를 실행하고, 또한 단독 펩티드 표준 이루어지는 샘플을 실행 한 결과를 함께 모든 데이터를 분석한다.
5. LC-SRM 데이터 분석
참고 : 펩타이드 식별 및 정량은 매우 간단하고 부분적 스카이 라인 같은 소프트웨어를 사용하여 자동화,하지만 여전히 강력하게 모든 데이터 주석을 수동으로 검토 할 것을 권장 할 수있다. 또한, 수동 ANNOT 동안 단백질 수준의 정보를 제외하는 것이 가장 좋다LC-SRM 데이터의 ATION는 편견을 방지합니다.
- 각 펩티드 식별을 위해, 각각의 전이 용출 프로파일의 형상은 대략 가우시안인지 확인. 각각의 전이의 경우, 용출 프로파일은 MS 검출기에 의해 측정 된 다중 신호의 제품인지 확인 (즉, MS 잡음의 하나 또는 두 개의 임의의 스파이크의 생성물이 아님).
- 각각의 펩타이드 식별을 위해, 전체 펩타이드 용출 프로파일이 선택되어 있는지 확인합니다. 펩타이드 식별 (무거운 표지 펩타이드 형태, 레이블이없는 형태로 개인의 전구체 이온, 개별 전환)의 하위 구성 요소의 용출 프로파일 경계를 조정 수동으로하지 마십시오.
- 각 전이 용출 프로파일은 신호 대 잡음비 (S / N)를 ≥3 있는지 확인한다. 참고 : "노이즈"coeluting 분석에서 신호를 재현하지, 임의 MS 잡음을 의미한다. 크로마토 그램 평활화 함수 크게 잡음 데이터의 분석을 지원할 수 있습니다.
- 각 펩티드 식별,모든 전환이 거의 동일한 용출 프로파일이 있는지 확인 (다만 동일한 피크 정점 번, 용출 프로파일이 서로 다른 진폭을 가질 수있다).
- LC-SRM는 펩티드 표준 (각 펩타이드의 통상적 ≥100 fmol)의 대량 분석, 해당 펩티드 용출 프로파일 펩티드 동정가 명확한, 그리고 (통상적 ≥100 N / S)이 매우 심한 것으로 확인. 참고 :이 매우 확신 식별이 대응하는 생물학적 샘플에서 유래 펩타이드의 식별을 위해 중요하다.
- 각각의 펩타이드 식별을 위해, 상대 전환 강도가 자신있게 확인 된 펩타이드 표준과 일치하는지 확인합니다. 필요한 경우, 소음, 상대적으로 낮은 강도의 전환 용출 프로파일을 삭제하지만, 상대적으로 강렬한 전환의 아무도가 누락되지 않습니다 확인합니다. 크게하고 명확하게 공동으로 영향을 상대적으로 강한 전이 용출 프로파일을 무시오염 물질을 용출.
- 각 펩티드 식별을 위해, 그것은 (coeluting MS 분석에서 랜덤 잡음 플러스 재현성 신호) 배경 신호에 의해 랜덤하게 생성 된 확률을 추정한다.
- 전체 LC-SRM 크로마토 그램을 검사하고 각 펩티드 식별의 고유성을 추정하여 수동으로 확률을 추정한다. 5 %의 추정 오류 발견 레이트 (FDR)가 확신 펩티드 동정의 세트를 생성. 엄격한 기준을 식별 세트 높은 신뢰가 필요한 경우 (느슨한 기준을 사용하는 것은 권장하지 않습니다) (예를 들어, 루즈 벨트 ≤1 %)를 사용합니다.
- 또한, mProphet 알고리즘 (48) 또는 감사 알고리즘 49를 사용하지만, 수동으로 결과를 검토.
- 각 펩티드 식별을 위해, 관측 용출 시간 (스카이가이 계산을 수행하도록 설계된다) 펩티드의 소수성과 일치하는지 확인한다.
- 각각의 펩타이드를 들어, 확인그 용출 시간은 LC 실행 일관성이다.
참고 : LC-SRM 때로는 체계적으로 실행 사이에 왜곡되어 용출 프로파일을 생성 분석. 체계적인 변화는 이러한 열을 교체하는 기기 변경 후 예상하여야한다. 또한, 초기 용출 펩티드 (열이 용량에 가까운로드 특히) 이동하는 경향이있을 수 있고, 늦게 용출 펩티드는 또한 이동하는 경향이 될 수 있습니다. 펩타이드 식별이 완료되었습니다. - 펩티드 정량을 향상시키기 위해, 각각의 펩티드 식별을 재검사하고 펩티드 식별 다른 전이 용출 프로파일과 비교하여, 특히 잡음이 경우 전이 용출 프로파일을 버린다. 상대적인 전이 강도 (펩티드 표준 대비) 잘못되면 전이 용출 프로파일을 폐기.
- 각각의 프리 커서 이온과 전이 용출 프로파일의 경계를 검사한다. 필요한 경우,주의 깊게 배경 신호를 멀리자를 용출 프로파일 경계를 조정 (또는 I에mprove 배경 신호 추정).
- 이 빛 펩타이드의 검출 양으로 오염되어있는 경우 각각의 내부 펩타이드 표준 (LC-SRM에 의해 결정 전술, 혼자 내부 펩타이드 기준 분석)을 확인. 그 후, 크게 내부 펩타이드 기준 내에서 빛 펩타이드의 오염에 의해 손상 된 모든 빛 펩타이드 용출 프로파일을 폐기합니다.
- 그 LC 피크 면적을 계산함으로써 각각의 전이 용출 프로파일을 정량화. 적당한 경우, 추정 된 배경 신호를 공제. 해당 전이 정량 값을 합산하여 각 펩타이드의 용출 프로파일을 정량화 (이하, 이것을 "Sum_Peak_Area"값이라고한다). 내부 또는 외부 펩티드 각 표준 생물학적 샘플 펩티드 Sum_Peak_Area 값, 데이터를 사용하여 생물학적 샘플 풍부 펩티드의 몰을 계산 LC-SRM (사용 성공적 모두 생물학적 샘플 펩티드에 의해 정량화 하였다 만 전환LC-SRM과 펩티드의 표준).
- 외부 펩타이드 기준 (권장하지 않음)를 사용하여 정량 :
- 외부 펩타이드 표준 몰 풍부 값 대 외부 펩타이드 표준 Sum_Peak_Area 값을 플롯. 데이터에 대한 선형 회귀 피팅하여 표준 곡선을 제작 (전형적으로, 동적 범위의 선형 성분이 표기를 임의의 비선형 성분을 매우주의 표기). 각각의 생물학적 샘플 펩티드 Sum_Peak_Area 값, 몰 풍부 펩티드를 계산하는 표준 곡선을 사용한다.
주 : 생물학적 샘플 및 외부 펩타이드 기준은 별도로 LC-SRM으로 제조하고 분석하기 때문에 샘플 준비 및 LC-SRM이 demonstrability 강력한 것이 필요하기 때문에이 정량 방법은 사용하지 않는 것이 좋습니다. 또한, 매트릭스 효과 (검체 이외의 시료의 성분에 의한 영향)을 고려하지 않는다.
- 외부 펩타이드 표준 몰 풍부 값 대 외부 펩타이드 표준 Sum_Peak_Area 값을 플롯. 데이터에 대한 선형 회귀 피팅하여 표준 곡선을 제작 (전형적으로, 동적 범위의 선형 성분이 표기를 임의의 비선형 성분을 매우주의 표기). 각각의 생물학적 샘플 펩티드 Sum_Peak_Area 값, 몰 풍부 펩티드를 계산하는 표준 곡선을 사용한다.
- 내부를 사용하여 정량펩타이드 표준 (권장) :
- 각각의 생물학적 샘플 펩티드 Sum_Peak_Area 값 및 대응 무거운 표지 된 내부 표준 펩타이드 Sum_Peak_Area 값, 광 / 중량 비율을 계산한다. 생물학적 샘플 펩티드의 몰 풍부 계산하도록 해당 광 / 무거운 펩타이드 몰 존재비의 측정으로이 비율을 사용한다. 특히, 생물학적 샘플 펩타이드 몰 풍부한 빛 / 무거운 Sum_Peak_Area 비율을 곱한 내부 펩타이드 표준 몰 풍부 같습니다.
- 각 복제하고 생물학적 표적 펩타이드를 들어, 선형 회귀를 이용하여 표준 곡선을 작성하여 LC-SRM 정량의 선형 범위를 식별한다. 특히, 무거운 / 라이트 Sum_Peak_Area 비율 값에 비해 내부 펩타이드 표준 몰 풍부 값을 플롯 (생물학적 샘플 질량이 데이터 세트에 걸쳐 일정 참고). 각각의 생물학적 샘플 펩티드 Sum_Peak_Area 값이 선형 범위 내에 있는지 (typic 필요아군, 동적 범위의 선형 성분이 사용되어야한다; 어떤 비선형 구성 요소)은 극도의주의를 요합니다. 마찬가지로, 각 대상 펩타이드 전환에 대해이 단계를 수행합니다.
참고 : LC-QQQ-SRM으로 분석 정량 넓은 동적 범위 (~ 10,000)을 통해 선형이어야한다. 로우 엔드에서 배경 신호 (랜덤 MS 잡음 플러스 coeluting 분석에서 재생 가능한 신호) 정량의 정확성과 정밀도를 줄일 수 있습니다. 하이 엔드 검출기 포화도 (결국,이 용출 프로파일은 특정 신호 강도 평평한 시작되게 할 수) 비선형 성을 일으킬 것이다.
- 외부 펩타이드 기준 (권장하지 않음)를 사용하여 정량 :
- 해당되는 경우, 이전에 내부 펩타이드 표준의 스파이크 기능에 대한 샘플 양에서 약간의 차이를 보정하기 위해, 이러한 중앙 경향 정상화 50 샘플에서 글로벌 표준화를 수행합니다. 필요한 경우 단계 1.1 만 하우스 키핑 단백질을 사용합니다.
- 필요한 경우, quantificat 누락 전가이온은 51, 52 값.
그것은 이렇게하면 인위적으로 정량 분산을 줄일 수 있기 때문에 단순히 정량 (LLOQ) 또는 검출 (LOD)의 절반 한도의 절반 하한에없는 값을 대체하기 부적절하고, (통계적 테스트에서 예를 들어, 분산 분석을 초래할 수 참고 : ) 위양성 결과의 제조 (I 형 에러). 그러나, 누락 가치를 무시하는 것도 문제가 될 수 있습니다. 반 진정한 풍부 값 LOD 이하이고, 나머지 절반은 LLOQ 이상일 경우, 예를 들어, 평균은 해당 풍부 의미 과대 것이다 풍부 관찰했다. - 분석은 LC-SRM 23 ~ 25에 대해 광범위하게 허용 기준을 만족하는지 확인하십시오. 특히, 정량 값의 변동 계수는 일반적으로 임상 분석을위한 ≤25 %, 그리고 비 임상 시험 법 25 ≤35 % 인 것을 요구한다. 규제 응용 프로그램을 위해 고려되어야하는 의료 또는 동물 제품이나 서비스와 관련된 LC-SRM 분석을 위해roval는, 분석은 분석을위한보다 엄격한 정밀도가 요구 사항을 만족해야 "각 농도에서 측정 정밀도는 20 %를 초과하지 않아야 LLOQ, 제외 변동 계수 (CV)의 15 %를 초과하지 않아야 이력서 "(23, 24).
결과
신호 전달 경로의 예측 계산 모델의 개발은 시스템 생물학 (53)의 기본적인 목적 중 하나이다. 불행히도, 심지어는 광범위하게 연구하고 높은 임상 적 의의를 가지고있다 신호 전달 경로를 들어, 정량적 섭동에 응답 통로 거동을 예측하는 것은 일반적으로 여전히 불가능하다 (예를 들면,이 MAPK / ERK 경로 (54)도 마찬가지이다). 최근 조사 경로 (56) 신호 마우스 대 식세포 화성을 연구 대상으로 프로테오믹스, transcriptomics, 전산 모델링 및 시뮬레이션을 고용했다. 조사의 초점은 RAW 264.7 세포 (마우스 단핵구 / 대식 세포주)의 스핑 고신 -1- 인산 매개 주 화성이었다. 통로 모델링을 용이하게하기 위해, LC-SRM 분석법 개발 및 RAW 264.7 세포 내의 화성 경로 단백질의 절대 풍부을 측정하기 위해 수행 하였다. 얻어진 풍부 값을 사용했다경로 모델의 매개 변수로 D.
전체 실험 방식 (도 1)의 G, I2, 이종삼 G 단백질 α 서브 유닛을 포함 표적 단백질의 목록을 시작했다. 전체 프로토콜의 성공은 YDEAASYIQSK으로 proteotypic 및 quantotypic있는 펩타이드 대상의 선택에 크게 의존한다. 외부 표준 펩티드의 혼합물이 준비되고 산탄 LC-QQQ-MS (/ MS)로 분석 하였다. 얻어진 YDEAASYIQSK 탠덤 질량 스펙트럼은 여러 조각으로 이루어지는 이온, 그리고 낮은 배경 (도 2)이었다. 스펙트럼은 전구체 이온 당 최고 10 개의 가장 강렬한 조각 이온을 함유 LC-SRM 대상 목록을 작성하는 데 사용되었다.
409 펩티드 외부 표준 ( "JPT409")의 혼합물을 LC-질적 의해 SRM 중으로 분석 하였다 (데이터는 보이지 않음). 이 샘플은 세 개의 G의 I2 펩티드를 포함하고, 세 가지의 식별이었다자신감을 판단. 그 후, RAW 264.7 샘플 (생물학적 복제 "RAW1"와 "RAW2")와 JPT409 샘플은 각각의 질적 LC-SRM에 의해 이중으로 (두 LC-SRM 기술 복제)를 분석 하였다 (이 여섯 LC-SRM 유사 한 분석을 수행 하였다 가능). (- C도 3A) YDEAASYIQSK 펩타이드는 자신있게 여섯 분석에서 확인되었다. 여섯 LC-SRM 분석 걸쳐 YDEAASYIQSK 전이 강도 패턴 일치 하였다, 이들은 산탄 LC-MS (/ MS)의 패턴 ( "라이브러리"의 복제도 3c)과 거의 비슷했다.
전이 강도의 패턴을 단독으로 항상 펩티드의 신분을 확인하기에 충분하지 않다. 펩타이드의 소수성 및 측정 LC 유지 시간은 일치해야합니다. 또한, 외부 펩티드 표준과 대응 생물학적 샘플 펩타이드는 약이 있어야동일 유지 시간. YDEAASYIQSK의 관찰과 체류 시간 (SSRCalc 버전 3.0 100 Å 알고리즘 (55)을 이용하여 추정) 소수성 일관성 (도 4A) 인 것으로 밝혀졌다. 또한 YDEAASYIQSK 체류 시간은 RAW 264.7 분석과 외부 펩티드 표준 분석 모두를 사용하여 측정하고, 체류 시간 값은 모두 (도 4B)와 거의 동일했다 하였다.
질적 LC-SRM의 대부분은 생물학적 시료의 분석은, 매우 무거운 표지 된 정제 대응 성공한 내부 표준 펩타이드를 제조 및 아군으로-RAW 264.7 세포 용 해물을 정량 하였다. 동위 원소 희석 시리즈 LC-SRM 단독 내부 펩타이드 표준 (샘플 "R0"), 내부 및 외부 펩타이드 표준 (샘플 "R1"), 6 RAW 264.7 생물학적 복제 (샘플 "R2"로 구성된 시료를 분석하는 데 사용 된 - "R7"). 두 differeNT를 단백질 변성제가 정확한 표적 단백질 정량을 훼손 할 수있는 가능한 단백질의 가용화, 변성, 알킬화, 및 / 또는 소화 문제를 테스트하는 데 사용되었다 (샘플 R2-R4는 요소를 사용하고, 샘플 R5-R7은 RapiGest SF를 사용). YDEAASYIQSK의 빛과 무거운 형태는 거의 동일한 전환 강도 패턴과 용출 프로파일 (- C도 5a)를 포함. LC-SRM은 피크 면적 비율이 상대적으로 풍부 펩티드의 척도로서 사용하고, YDEAASYIQSK 비가 RAW 264.7 세포 당 매수 단위 G I2 풍부 값을 계산하는데 사용되었다 합산. 이와 병행하여, 제 G 내부 펩타이드 표준 (IAQSDYIPTQQDVLR)이 동일한 RAW 264.7 샘플의 정량적 G의 I2의 LC-SRM 분석을 수행하는 데 사용하고, 두 분석은 생물학적 복제 모두에 걸쳐 매우 유사 G의 I2 풍부 측정치를 생성 I2 (도 5D). 두 G의 계약 I2 분석법 둘 펩티드 quantotypic 있다는 강력한 증거이며, G의 양적인 I2 LC-SRM 분석의 모든 것을 정확하고 정밀한이었다.
전반적으로, 35 단백질 펩티드 58 내부 기준 (표 1)를 사용하여 정량 하였다. 특히, 내부 단백질 표준 LC-SRM 분석 (개똥 벌레 루시페라아제 공정 4.11)은 정확하고 정밀한이었다. 이 조사에서 스카이 라인 데이터의 포괄적 인 세트 (https://panoramaweb.org/labkey/project/NIH_NitaLazar/begin.view에서 Manes_RAW_Chemotaxis 폴더) 파노라마 온라인 LC-SRM 데이터베이스에서 확인할 수있다.
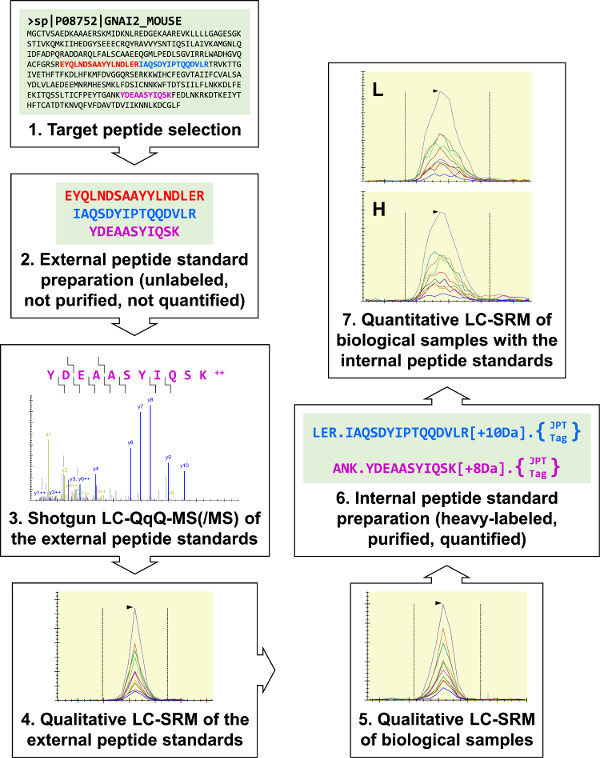
그림 1 :. G의 I2에 대한 프로토콜 세 임시 목표 펩티드의 개요 (이종삼 G 단백질 α-서브 유닛) 외부 펩타이드 표준 합성에 선정되었다. 다음은 analy했다세 G의 I2 LC-SRM 분석법을 개발하는 샷건 LC-MS (/ MS)에 의해 평탄화하고, 이러한 분석은 생물학적 시료의 정성 분석을 수행하기 위해 사용되었다. 세 G I2 대상 펩티드를 확인하고, 두 개의 내부 표준 펩타이드 제조를 위해 선택되었다. 트립신 분해성 "JPT-태그"UV 분광 광도법을 사용하여 내부 표준 펩타이드를 정량화하기 위해 사용되었다. 내부 펩타이드 표준이 RAW 264.7 시료의 정량 G의 I2 LC-SRM 분석을 수행하는 데 사용되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
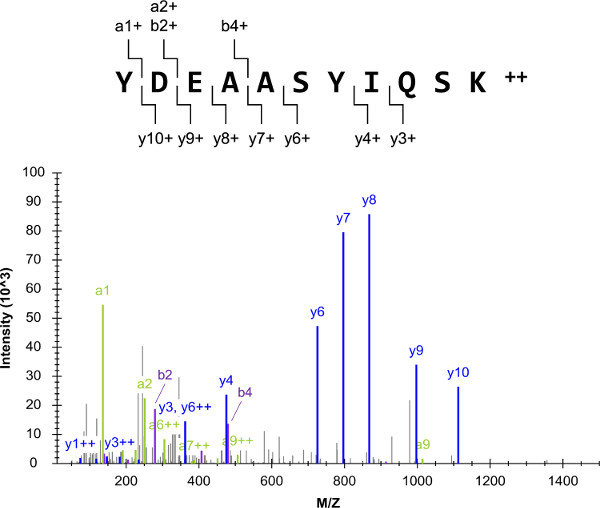
그림 2. 산탄 LC-MS (/ MS)의 질량 스펙트럼을 도시 G I2 외부 펩타이드 표준 (YDEAASYIQSK) 중 하나의 분석에 유래. 이 전구체를 들어10 대 가장 강렬한 전환이 (인해 짧은 길이로 A1 1+ 조각 이온을 제외) LC-SRM으로 선택했다 이온,. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
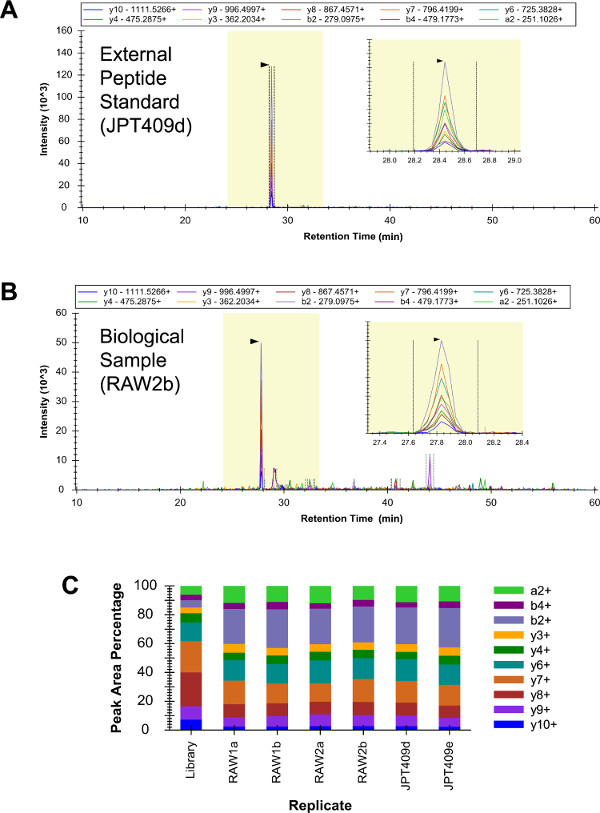
그림 3 :. G의 I2의 질적 LC-SRM JPT409에서 YDEAASYIQSK 크로마토 그램은 매우 낮은 배경을 포함 분석 및 펩타이드 자신있게 (A)를 확인 하였다. 대응 RAW 264.7 분석은 더 백그라운드 신호 초래하지만 펩티드 식별은 여전히 모호 (B)이었다. 상대 강도 전이 패턴 여섯 LC-SRM은 (3 개의 독립적 샘플의 두 복제 기술)을 일관된 분석했고, 이들은 대략 시미 있었다해당 샷건 LC-MS (/ MS) 패턴 ( "라이브러리"복제) (C) 고맙다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

런 걸쳐 펩타이드 보유 시간 예측 및 변화의 선형 회귀 추정 소수성을 이용하여 계산하고 목표 모든 펩타이드의 질적 LC-SRM 용출 시간을 측정하고, YDEAASYIQSK의 예측되고 측정 용출 시간이 일치했다 하였다 (A)도 4. . 또한, LC-SRM 걸쳐 각 펩티드의 관찰 용출 시간의 일관성을 측정 하였다 분석한다. YDEAASYIQSK의 용출 시간은 사용 된 LC-SRM 기기의 정밀도와 일치 하였다 ~ 40 초간의 범위 (스팬 값피크 정점 시간 + 반 최대의 전체 폭, 또한 피크의베이스), (B)의 전체 폭이다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
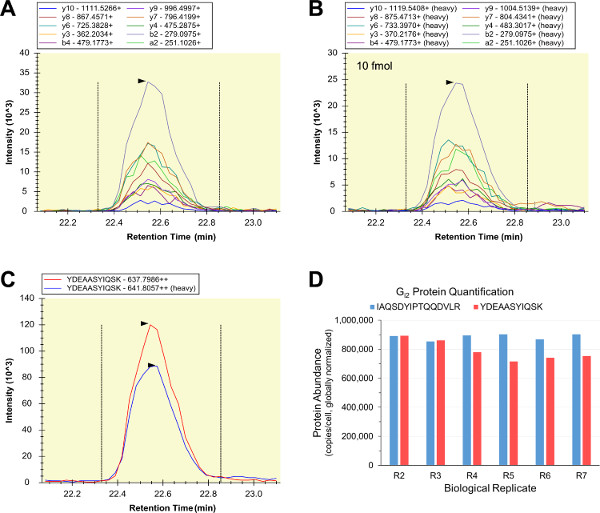
도 5 :. 도시 G의 I2의 정량 LC-SRM 생물학적 시료 펩티드 (A)과 일치 상대적인 전이 강도를 가지고 내부 펩타이드 표준 (B)의 전이 크로마토 그램, 그리고 합산 된 (C)는 ( "R2"분석이다 YDEAASYIQSK에 대한) 10 fmol 내부 펩타이드 표준을 사용하여. 빛과 무거운 용출 프로파일 (C) 일치 하였다, 이들 곡선 아래의 면적의 비율은 LIG의 척도로 사용 하였다HT / 무거운 펩타이드 풍부. 제 G I2 내부 펩타이드 표준 (IAQSDYIPTQQDVLR)는 병렬로 정량적 LC-SRM에 사용하고, 생성 된 G의 I2 풍부 값 여섯 생물학적 복제하고 전체적으로 두 대상 펩티드 (모두에 걸쳐 일치 하였다, N = 12, CV = 8.38 %) (D). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| UniProt의 기탁 | 표적 단백질 | 대상 펩타이드 | 풍요 (fmol / μg의) | 풍요 (복사 / 셀, 정규화) | 이력서 |
| P08659 | 루시 페라 제 | (280 nm에서 UV) | 23.824 | N / A | N / A |
| P08659 | 루시 페라 제 | VVDLDTGK | 24.103 | N / A | 7 % |
| P08659 | 루시 페라 제 | VVPFFEAK | 24.717 | N / A | 4 % |
| P60710 | 액틴, 세포질 1 | IWHHTFYNELR | 760.448 | 61762598 | 3 % |
| P16858 | GAPDH | LISWYDNEYGYSNR | 357.803 | 28906524 | 14 % |
| P06151 | 젖산 탈수소 효소 | LLIVSNPVDILTYVAWK | 129.623 | 10633145 | 30 % |
| P99024 | 튜 불린 β (5) | ALTVPELTQQVFDAK | 78.765 | 6398971 | 3 % |
| P20152 | 멘틴 | SLYSSSPGGAYVTR | 121.100 | 9807488 | 10 % |
| P60766 | CDC42 | DDPSTIEK | 86.647 | 6957317 | 27 % |
| P60766 | CDC42 | QKPITPETAEK | 26.475 | 2153669 | 6 % |
| Q8C3J5 | DOCK2 | ETLYETIIGYFDK | 1.459 | 118539 | 15 % |
| Q8C3J5 | DOCK2 | ISSSPTHSLYVFVR | 1.795 | 143440 | 33 % |
| Q8BPU7 | ELMO1 | ALTTKPSSLDQFK | 2.302 | 186754 | 7 % |
| Q8BPU7 | ELMO1 | SAIDISILQR | 1.244 | 99728 | 25 % |
| Q8BGM0 | FGR | GAYSLSIR | 1.099 | 89473 | 17 % |
| Q8BGM0 | FGR | WTAPEAALFGR | 0.319 | 24766 | 6 % |
| P27601 | Gα (13) | GIHEYDFEIK | 0.843 | 68467 | 15 % |
| P27601 | Gα (13) | VFLQYLPAIR | 1.295 | 106176 | 23 % |
| P08752 | Gα (I) (2) | IAQSDYIPTQQDVLR | 10.900 | 885701 | 2 % |
| P08752 | Gα (I) (2) | YDEAASYIQSK | 9.774 | 790616 | 9 % |
| Q9DC51 | Gα (K) | EYQLNDSASYYLNDLDR | 6.574 | 537862 | 15 % |
| Q9DC51 | Gα (K) | ISQTNYIPTQQDVLR | 3.504 | 285784 | 10 % |
| P62874 | Gβ 1 | AGVLAGHDNR | 17.078 | 1385578 | 4 % |
| Q9CXP8 | Gγ (10) | DALLLGVPAGSNPFR | 2.167 | 179178 | 36 % |
| P63213 | Gγ 2 | EDPLLTPVPASENPFR | 3.284 | 266360 | 8 % |
| Q80SZ7 | Gγ 5 | VSQAAADLK | 6.087 | 493512 | 6 % |
| P08103 | HCK | GPVYVPDPTSSSK | 1.143 | 93044 | 12 % |
| P08103 | HCK | IIEDNEYTAR | 0.944 | 77147 | 19 % |
| P43406 | 인테그린 α V | AGTQLLAGLR | 0.276 | 22264 | 32 % |
| P43406 | 인테그린 α V | SHQWFGASVR | 0.443 | 34235 | 5 % |
| P25911 | LYN | VIEDNEYTAR | 1.461 | 119377 | 13 % |
| Q5SW28 | PI3K 규제 (5) | AGFPGILDTASPGK | 0.301 | 24331 | 11 % |
| Q8K3B3 | PI3K 규제 α | LYEEYTR | 0.472 | 38592 | 16 % |
| Q8K3B3 | PI3K 규제 α | TWNVGSSNR | 0.524 | 42697 | 12 % |
| Q8K3B3 | PI3K 규제 α | VLSEIFSPVLFR | 0.440 | 35902 | 20 % |
| Q5U3K7 | PI3K 규제 β | DTPDGTFLVR | 0.188 | 15262 | 30 % |
| Q5U3K7 | PI3K 규제 β | IAEIHESR | 0.282 | 22561 | 13 % |
| Q0VGQ5 | PI3K (α) | LINLTDILK | 0.102 | 8494 | 38 % |
| Q8CI98 | PI3K의 δ | HEVQEHFPEALAR | 0.178 | 14566 | 22 % |
| Q8CI98 | PI3K의 δ | ITEEEQLQLR | 0.481 | 38709 | 24 % |
| Q9ES52 | PIP3 5 포스 1 | IVVLAKPEHENR | 0.486 | 39194 | 19 % |
| Q9ES52 | PIP3 5 포스 1 | LSQLTSLLSSIEDK | 2.056 | 167123 | 7 % |
| Q69ZK0 | PIP3에 의존하는 라 세미 GEF 1 | DSVLSYTSVR | 0.647 | 52712 | 32 % |
| Q69ZK0 | PIP3에 의존하는 라 세미 GEF 1 | NQLLLALLK | 0.354 | 27425 | 5 % |
| Q9CQE5 | RGS (10) | ASSQVNVEGQSR | 2.460 | 199782 | 4 % |
| Q9CQE5 | RGS (10) | WASSLENLLEDPEGVQR | (2)0.647 | 206005 | 11 % |
| Q9CX84 | RGS (19) | AEANQHVVDEK | 0.495 | 39753 | 21 % |
| Q9CX84 | RGS (19) | LIYEDYVSILSPK | 0.846 | 68481 | 22 % |
| B9EKC3 | 의 Rho GAP (5) | DGLAQELANEIR | 0.448 | 34653 | 10 % |
| Q99PT1 | RHO GDI 1 | SIQEIQELDK | 3.156 | 267063 | 16 % |
| Q99PT1 | RHO GDI 1 | VAVSADPNVPNVIVTR | 37.077 | 3006168 | 5 % |
| Q61599 | RHO GDI 2 | LNYKPPPQK | 37.975 | 3086596 | 3 % |
| Q61599 | RHO GDI 2 | YVQHTYR | 21.436 | 1711908 | 47 % |
| Q61210 | RHO GEF 1 | FDGAEGSWFQK | 2.149 | 176139 | 47 % |
| Q61210 | RHO GEF 1 | SGLELEPEEPPGWR | 2.911 | 236483 | 8 % |
| Q9QUI0 | 에서 RhoA | QVELALWDTAGQEDYDR | 43.500 | 3532438 | 8 % |
| P70336 | ROCK2 | GAFGEVQLVR | 0.527 | 42800 | 23 % |
| P70336 | ROCK2 | IYESIEEAK | 1.011 | 83794 | 33 % |
| P70336 | ROCK2 | LEGWLSLPVR | 0.573 | 48259 | 22 % |
| Q8R0X7 | S1P 분해 효소 (1) | AGYPLEKPFDFR | 1.787 | 147043 | 27 % |
| Q8R0X7 | S1P 분해 효소 (1) | TPEIVAPESAHAAFDK | 3.562 | 291791 | 18 % |
표 1 : 양적 LC-SRM의 RAW 264.7 세포 단백질 서른 다섯 RAW 264.7 세포 단백질이 쉰여덟 내부 펩타이드 표준과 육 생물 복제를 사용하여 정량화 하였다.. 표적 단백질 중 5 (액틴, GAPDH, 락 테이트 탈수소 효소, 튜 불린, 및 멘틴) 단백질 하우스 키핑하고, 생물학적 시료 (단계 1.1)에 걸쳐 정규화 있도록 정량 하였다. 또한, 내부 단백질 표준 (4.765 pmol의 당 반딧불이 루시퍼 라제 200 μg의 샘플, SDS-PAGE에 의해 98 % 순수, 280 nm에서 분광 광도계로 정량; 4.11 공정) LC-SRM 각 세포 용 해물로 아군 및 정량 하였다. CV 값이 전역 정규화 값 풍부하여 여섯 생물학적 복제에 걸쳐 계산 하였다 (루시퍼 라제를 제외한 공정 5.15).
토론
절대 정량 단백질은 바이오 마커 확인 및 신호 전달 경로와 같은 생의학 모델링 애플리케이션 매우 다양한 필수적이다. 최근 LC-SRM을 사용하여 타겟 단백질 체학은 펩타이드 표준 준비, HPLC, QQQ-MS 및 LC-SRM 데이터 분석 등 다양한 기술에 개선의 혜택을했다. 결과적으로, 면역에 강력한 대안이되고있다. 면역 분석 및 / 또는 의무하지 다중화 매우 민감하고 높은 처리량이 될 수 있지만, 면역 세포 / 조직 용해 / 균질화 방법과 호환, 교차 반응성 및 / 또는 간섭에 취약 할 수 있기 때문에 강력한 면역 측정법을 개발하는 것은 매우 어려울 수있다 5,8. 예를 들어, 교차 - 반응성에 대한 포괄적 인 시험 제조 어려울 수 녹아웃 유전자로부터 유래 된 샘플을 사용하여 면역 측정법을 수행하는 것이다.
이 프로토콜은 대상 pept 설명IDE 선택, LC-SRM 분석 개발, 정량적 LC-SRM 및 LC-SRM 데이터 분석. 그것은 RAW 264.7 세포 (56) 내의 통로 (36)의 화학 주성 단백질의 절대 풍부을 측정하는 데 사용하지만, 그 적용은 특정한 응용이 훨씬 넘어 확장 하였다. 이 세포 내 단백질 펠렛을 정량화하도록 설계되었지만, 그것은 다른 생물학적 시료 (예, biofluids) 및 다른 SRM 대상 (예, 포스 포)의 분석을 위해 조정될 수있다. 예를 들어, 균질화 및 / 또는 단백질 분해 프로토콜을 변경하면 훨씬 특히 어려운 표적 단백질 (예를 들면, 막 단백질)의 가용화, 변성, 알킬화, 소화, 및 정량을 개선되거나, 특히 도전 샘플 (예를 들어, 샘플의 분석을 가능하게 할 수 함유 <100 μg의 단백질 질량).
대상 펩티드의 초기 선택은 중요하지만 시간이 소요될 수 있습니다. hundre 들어표적 단백질의 DS는 애드혹 점수는 각 기준에 사용될 수 있으며, (56) 이전에 수행 된 바와 같이 임시 목표 펩티드는, 스코어의 합에 기초하여 순위가 매겨 질 수있다. 대안 적으로, 이러한 분석은 PeptidePicker 크게 대상 펩티드 선정 30 간소화 웹 인터페이스를 이용하여 자동화 될 수있다 (http://mrmpeptidepicker.proteincentre.com/).
대상 펩티드가 선택되었으며, LC-SRM 분석법이 개발되어왔다 한 결과, 단백질이 발현되는 경우에도 매우 proteotypic 및 quantotypic 펩티드가 검출되지 않기 때문에 생물학적 시료의 정성 LC-SRM 분석을 수행하는 것이 중요 악기의 감도 임계 값 이하 수준, 또는 경우 백그라운드 간섭은 특히 문제가된다. 분리 제 치수 단백체 깊이를 증가시킬 수있다 (예를 들면, 강한 양이온 교환 HPLC, 높은 pH는 HPLC, 겔 전기 영동없는 위상 반전), 그러나 필요인스트루먼트 시간과 데이터 분석 훨씬 더. 대안 적으로, 농축 전략 (예를 들어, 펩티드 - 단백질 수준 immunoenrichment 및 세포 분별) 단백체 심도를 향상시킬 수있는, 매우 풍부한 단백질 immunodepletion은 분석 물을 coeluting하여 간섭을 감소시키기 위해 사용될 수있다.
전환 ~ 수백 ~ 수천 개의 샘플의 수십 일반적으로 광범위한 악기 시간을 필요로 ~의 LC-SRM. 이 연구를 위해 사용되지 않지만, LC-SRM는 (미리 - 특정 용출 시간 윈도우 천이 동안 측정) 스케줄링 실행 당 전이의 분석을 가능하게한다. 또한, 스케줄링 LC 구배 비교적 미사용 기간 동안 SRM 듀티 사이클을 감소시키고, 이는 향상된 펩티드 식별 및 정량화가 발생할 수있다. 그러나 일정은 펩타이드 용출 시간이 자신있게 질적 LC-SRM 분석에서 결정해야합니다. 샘플 준비에 미묘한 변화, LC-MS 기기,또는 LC-MS 방법은 스케줄링 자르거나 심지어 완전히 대상 펩티드를 그리워하는 원인이 될 수 있습니다. 예를 들어, 생물학적 샘플 YDEAASYIQSK 용출 프로파일은 약간 외부 펩티드 표준들 (도 4b)에 대하여, 때문인 매트릭스 효과로 시프트 하였다.
요약하면, 단계별 프로토콜은 절대 단백질 정량 LC-SRM의 개발 및 사용을 위해 제공된다. LC-SRM에 의한 단백질의 절대 정량은 이미 실험실 16, 19 사이의 재현으로 증명되었다. 샘플 (예를 들어, 자동화) 제제, 액체 크로마토 그래피, 질량 분석 및 데이터 분석을 포함 프로테오믹스 기술은 급속히 향상되고, 대규모 연구 및 임상 적 적용을위한 실용 될 LC-SRM있게된다. 양적 LC-SRM의 특이성, 감도, 정확성, 재현성 및 높은 처리량은 기초 연구 및 생물 의학을위한 강력한 도구합니다.
공개
The authors have nothing to disclose.
감사의 말
This research was supported by the Intramural Research Program of the NIH, National Institute of Allergy and Infectious Diseases.
자료
| Name | Company | Catalog Number | Comments |
| Acetonitrile (ACN), LC-MS grade | Fisher | A955-1 | |
| BCA (bicinchoninic acid) protein assay kit | Fisher | 23235 | |
| Beads for bead beating, zirconia-silica, 0.1 mm | BioSpec Products | 11079101z | |
| Bestatin hydrochloride | Sigma | B8385-10MG | |
| Cell culture DMEM (with glucose, without L-glutamine) | Lonza | 12-614F | |
| Cell culture EDTA, 500 mM, pH8 | Gibco | 15575 | |
| Cell culture fetal bovine serum (FBS) | Atlanta Biologicals | S11550 | |
| Cell culture L-glutamine | Sigma | G8540-25G | |
| Cell culture phosphate buffered saline (PBS) pH 7.4 | Gibco | 10010-049 | |
| Cell culture Trypan Blue viability stain, 0.4% w/v | Lonza | 17-942E | |
| Cellometer Auto T4 cell counter | Nexcelom Bioscience | Cellometer Auto T4 | |
| Cellometer Auto T4 disposable counting chambers | Nexcelom Bioscience | CHT4-SD100-014 | |
| Dithiothreitol (DTT) | Sigma | D5545-5G | |
| Formic acid, LC-MS grade, ampules | Fisher | A117-10X1AMP | |
| Hemocytometer, Neubauer-improved, 0.1 mm deep | Marienfeld-Superior | 0640030 | |
| HEPES, 1 M, pH 7.2 | Mediatech | 25-060-CI | |
| Hydrochloric acid, 37% w/w | VWR | BDH3028-2.5LG | |
| Iodoacetamide | Sigma | I1149-5G | |
| Laser Based Micropipette Puller | Sutter Instrument Co. | P-2000 | |
| LC coated silica capillary, 50 µm id | Polymicro Technologies | 1068150017 | |
| LC vial, autosampler, 12 mm x 32 mm polypropylene | SUN SRI | 200-268 | |
| LC vial screw cap, autosampler, pre-slit PTFE/silicone | SUN SRI | 500-061 | |
| Luciferase, from Photinus pyralis | Sigma | L9506-1MG | |
| Pepstatin A | EMD Millipore | 516481-25MG | |
| pH strips colorpHast (pH 0.0-6.0) | EMD Chemicals | 9586-1 | |
| PhosStop phosphatase inhibitor cocktail | Roche | 04906837001 | |
| RapiGest SF | Waters | 186001861 | |
| Sep-Pak SPE, C18 1 ml 100 mg cartridge | Waters | WAT023590 | |
| Sep-Pak SPE, extraction manifold, 20 position | Waters | WAT200609 | |
| Sep-Pak SPE, flat-surfaced rubber bulb | Fisher | 03-448-25 | |
| Sodium hydroxide (NaOH) | Fisher | S318-500 | |
| SpeedVac vacuum concentrator | Fisher | SPD111V | |
| Trifluoroacetic acid (TFA), LC-MS grade | Fisher | A116-50 | |
| Trypsin, sequencing grade, modified | Promega | V5113 | |
| Tube decapper for Micronic tubes | USA Scientific | 1765-4000 | |
| Tubes, 2 ml microcentrifuge, o-ring screw-cap, sterile | Sarstedt | 72.694.006 | |
| Urea | Sigma | U0631-500g | |
| Water, LC-MS grade | Fisher | W6-1 |
참고문헌
- Cox, J., Mann, M. Quantitative high-resolution proteomics for data-driven systems biology. Annu Rev Biochem. 80, 273-299 (2011).
- Zhang, Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. 3rd Protein analysis by shotgun/bottom-up proteomics. Chem Rev. 113, 2343-2394 (2013).
- Boja, E. S., Rodriguez, H. Mass spectrometry-based targeted quantitative proteomics: achieving sensitive and reproducible detection of proteins. Proteomics. 12, 1093-1110 (2012).
- Gillette, M. A., Carr, S. A. Quantitative analysis of peptides and proteins in biomedicine by targeted mass spectrometry. Nat Methods. 10, 28-34 (2013).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nat Methods. 9, 555-566 (2012).
- Lesur, A., Domon, B. Advances in high-resolution accurate mass spectrometry application to targeted proteomics. Proteomics. , (2015).
- Wild, D. The immunoassay handbook : theory and applications of ligand binding ELISA., and related techniques. , 4th edn, Elsevier. (2013).
- Sturgeon, C. M., Viljoen, A. Analytical error and interference in immunoassay: minimizing risk. Ann Clin Biochem. 48, 418-432 (2011).
- Adrait, A., et al. Development of a Protein Standard Absolute Quantification (PSAQ) assay for the quantification of Staphylococcus aureus enterotoxin A in serum. J Proteomics. 75, 3041-3049 (2012).
- Lin, D., Alborn, W. E., Slebos, R. J., Liebler, D. C. Comparison of protein immunoprecipitation-multiple reaction monitoring with ELISA for assay of biomarker candidates in plasma. J Proteome Res. 12, 5996-6003 (2013).
- Weiss, F., et al. Catch and measure-mass spectrometry-based immunoassays in biomarker research. Biochim Biophys Acta. 1844, 927-932 (2014).
- Yassine, H., et al. Mass spectrometric immunoassay and MRM as targeted MS-based quantitative approaches in biomarker development: potential applications to cardiovascular disease and diabetes. Proteomics Clin Appl. 7, 528-540 (2013).
- Zhao, L., et al. Quantification of proteins using peptide immunoaffinity enrichment coupled with mass spectrometry. J Vis Exp. , (2011).
- Becker, J. O., Hoofnagle, A. N. Replacing immunoassays with tryptic digestion-peptide immunoaffinity enrichment and LC-MS/MS. 4, 281-290 (2012).
- Wasinger, V. C., Zeng, M., Yau, Y. Current status and advances in quantitative proteomic mass spectrometry. Int J Proteomics. 2013, 180605(2013).
- Abbatiello, S. E., et al. Large-scale inter-laboratory study to develop, analytically validate and apply highly multiplexed, quantitative peptide assays to measure cancer-relevant proteins in plasma. Mol Cell Proteomics. , (2015).
- Rodriguez-Suarez, E., Whetton, A. D. The application of quantification techniques in proteomics for biomedical research. Mass Spectrom Rev. 32, 1-26 (2013).
- Wehr, A. Y., Hwang, W. T., Blair, I. A., Yu, K. H. Relative quantification of serum proteins from pancreatic ductal adenocarcinoma patients by stable isotope dilution liquid chromatography-mass spectrometry. J Proteome Res. 11, 1749-1758 (2012).
- Kennedy, J. J., et al. Demonstrating the feasibility of large-scale development of standardized assays to quantify human proteins. Nat Methods. 11, 149-155 (2014).
- Jensen, K. J., Shelton, P. T., Pedersen, S. L. Peptide synthesis and applications. , 2nd edn, Humana Press Springer. (2013).
- Pratt, J. M., et al. Multiplexed absolute quantification for proteomics using concatenated signature peptides encoded by QconCAT genes. Nat Protoc. 1, 1029-1043 (2006).
- Brun, V., et al. Isotope-labeled protein standards: toward absolute quantitative proteomics. Mol Cell Proteomics. 6, 2139-2149 (2007).
- Guidance for Industry: Bioanalytical Method Validation. , U.S. Food and Drug Administration. Available from: http://www.fda.gov/ucm/groups/fdagov-public/@fdagov-drugs-gen/documents/document/ucm070107.pdf (2001).
- Guidance for Industry: Bioanalytical Method Validation. , U.S. Food and Drug Administration; Revision 1 (draft). Available from: http://www.fda.gov/ucm/groups/fdagov-public/@fdagov-drugs-gen/documents/document/ucm368107.pdf (2013).
- Carr, S. A., et al. Targeted peptide measurements in biology and medicine: best practices for mass spectrometry-based assay development using a fit-for-purpose approach. Mol Cell Proteomics. 13, 907-917 (2014).
- Ori, A., Andres-Pons, A., Beck, M. The use of targeted proteomics to determine the stoichiometry of large macromolecular assemblies. Methods Cell Biol. 122, 117-146 (2014).
- Rosen, O., Feldberg, L., Gura, S., Zichel, R. A new peptide substrate for enhanced botulinum neurotoxin type B detection by endopeptidase-liquid chromatography-tandem mass spectrometry/multiple reaction monitoring assay. Anal Biochem. , (2015).
- Feng, Y., et al. Global analysis of protein structural changes in complex proteomes. Nat Biotechnol. 32, 1036-1044 (2014).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26, 966-968 (2010).
- Mohammed, Y., et al. PeptidePicker: a scientific workflow with web interface for selecting appropriate peptides for targeted proteomics experiments. J Proteomics. 106, 151-161 (2014).
- Rodriguez, J., Gupta, N., Smith, R. D., Pevzner, P. A. Does trypsin cut before proline. J Proteome Res. 7, 300-305 (2008).
- Min, X. J., Butler, G., Storms, R., Tsang, A. OrfPredictor: predicting protein-coding regions in EST-derived sequences. Nucleic Acids Res. 33, W677-W680 (2005).
- Lam, H., et al. Building consensus spectral libraries for peptide identification in proteomics. Nat Methods. 5, 873-875 (2008).
- Craig, R., Cortens, J. P., Beavis, R. C. Open source system for analyzing, validating, and storing protein identification data. J Proteome Res. 3, 1234-1242 (2004).
- Desiere, F., et al. The PeptideAtlas project. Nucleic Acids Res. 34, D655-D658 (2006).
- Vizcaino, J. A., et al. The PRoteomics IDEntifications (PRIDE) database and associated tools: status in 2013. Nucleic Acids Res. 41, D1063-D1069 (2013).
- Frank, R. The SPOT-synthesis technique. Synthetic peptide arrays on membrane supports--principles and applications. J Immunol Methods. 267, 13-26 (2002).
- Ong, S. E., Kratchmarova, I., Mann, M. Properties of 13C-substituted arginine in stable isotope labeling by amino acids in cell culture (SILAC). J Proteome Res. 2, 173-181 (2003).
- Mant, C. T., et al. HPLC analysis and purification of peptides. Methods Mol Biol. 386, 3-55 (2007).
- Alterman, M. A., Hunziker, P. Amino acid analysis : methods and protocols. , Humana Press. (2012).
- Maclean, B., et al. Effect of collision energy optimization on the measurement of peptides by selected reaction monitoring (SRM) mass spectrometry. Anal Chem. 82, 10116-10124 (2010).
- Nesvizhskii, A. I. A survey of computational methods and error rate estimation procedures for peptide and protein identification in shotgun proteomics. J Proteomics. 73, 2092-2123 (2010).
- Tabb, D. L., Friedman, D. B., Ham, A. J. Verification of automated peptide identifications from proteomic tandem mass spectra. Nat Protoc. 1, 2213-2222 (2006).
- Freshney, R. I. Culture of animal cells : a manual of basic technique and specialized applications. , 6th edn, Wiley-Blackwell. (2010).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. J Proteome Res. 8, 2144-2156 (2009).
- Noble, J. E., Bailey, M. J. Quantitation of protein. Methods Enzymol. 463, 73-95 (2009).
- Kiser, J. Z., Post, M., Wang, B., Miyagi, M. Streptomyces erythraeus trypsin for proteomics applications. J Proteome Res. 8, 1810-1817 (2009).
- Reiter, L., et al. mProphet: automated data processing and statistical validation for large-scale SRM experiments. Nat Methods. 8, 430-435 (2011).
- Abbatiello, S. E., Mani, D. R., Keshishian, H., Carr, S. A. Automated detection of inaccurate and imprecise transitions in peptide quantification by multiple reaction monitoring mass spectrometry. Clin Chem. 56, 291-305 (2010).
- Callister, S. J., et al. Normalization approaches for removing systematic biases associated with mass spectrometry and label-free proteomics. J Proteome Res. 5, 277-286 (2006).
- Karpievitch, Y. V., Dabney, A. R., Smith, R. D. Normalization and missing value imputation for label-free LC-MS analysis. BMC Bioinformatics. 13, Suppl 16. S5(2012).
- Oh, S., Kang, D. D., Brock, G. N., Tseng, G. C. Biological impact of missing-value imputation on downstream analyses of gene expression profiles. Bioinformatics. 27, 78-86 (2011).
- Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A., Fraser, I. D. Systems biology in immunology: a computational modeling perspective. Annu Rev Immunol. 29, 527-585 (2011).
- Futran, A. S., Link, A. J., Seger, R., Shvartsman, S. Y. ERK as a model for systems biology of enzyme kinetics in cells. Curr Biol. 23, R972-R979 (2013).
- Krokhin, O. V. Sequence-specific retention calculator. Algorithm for peptide retention prediction in ion-pair RP-HPLC: application to 300- and 100-A pore size C18 sorbents. Anal Chem. 78, 7785-7795 (2006).
- Manes, N. P., Angermann, B. R., Koppenol-Raab, M., An, E., Sjoelund, V. H., Sun, J., Ishii, M., Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A. Targeted Proteomics-Driven Computational Modeling of Macrophage S1P Chemosensing. Mol Cell Proteomics. , Forthcoming.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유