Method Article
Selected Reaction Monitoring spectrométrie de masse pour Absolute quantification des protéines
Dans cet article
Résumé
This protocol describes how to perform absolute quantification assays of target proteins within complex biological samples using selected reaction monitoring. It was used to accurately quantify proteins of the mouse macrophage chemotaxis signaling pathway. Target peptide selection, assay development, and qualitative and quantitative assays are described in detail.
Résumé
Absolute quantification of target proteins within complex biological samples is critical to a wide range of research and clinical applications. This protocol provides step-by-step instructions for the development and application of quantitative assays using selected reaction monitoring (SRM) mass spectrometry (MS). First, likely quantotypic target peptides are identified based on numerous criteria. This includes identifying proteotypic peptides, avoiding sites of posttranslational modification, and analyzing the uniqueness of the target peptide to the target protein. Next, crude external peptide standards are synthesized and used to develop SRM assays, and the resulting assays are used to perform qualitative analyses of the biological samples. Finally, purified, quantified, heavy isotope labeled internal peptide standards are prepared and used to perform isotope dilution series SRM assays. Analysis of all of the resulting MS data is presented. This protocol was used to accurately assay the absolute abundance of proteins of the chemotaxis signaling pathway within RAW 264.7 cells (a mouse monocyte/macrophage cell line). The quantification of Gi2 (a heterotrimeric G-protein α-subunit) is described in detail.
Introduction
Expériences protéomiques qui utilisent la spectrométrie de masse (MS) peuvent être conçus pour utiliser soit non ciblée (fusil de chasse) ou des méthodes ciblées. protéomique de découverte repose généralement sur shotgun bottom-up MS, soit en utilisant un mode d'acquisition traditionnelle dépendant des données, ou en utilisant l'une des techniques de données indépendante récemment développés (par exemple, MS E, COULOIR) 1,2. Protéomique fusil de chasse est un outil puissant pour l'identification à haut débit de peptide et la quantification relative, mais il est généralement impropre à la quantification absolue ou pour cibler les petits ensembles définis (~ RTE) de protéines. La méthode de MS le plus souvent utilisé pour la protéomique ciblées est sélectionné surveillance de réaction (SRM) en raison de sa haute sensibilité, la vitesse, et la gamme dynamique 3-5. Alternatives à SRM comprennent la surveillance de réaction parallèle, qui tire parti de haute résolution, MS balayage complet 6.
SRM est habituellement effectuée en utilisant un rever nano-débitChromatographie sed phase liquide à haute performance (nano-RP-LC) couplée à un instrument ionisation nano-électrospray (nano-ESI) source d'ions attaché à un spectromètre de masse triple quadripôle (QQQ-MS). Dans une expérience typique, les protéines de l'échantillon sont digérés par protéolyse, et les peptides obtenus sont séparés par chromatographie, désorbés, et ionisés. Les ions précurseurs qui en résultent sont m / z filtré par le premier quadrupôle (Q1) et fragmentée dans le deuxième quadrupôle (Q2) par les collisions avec un gaz de collision. Les ions fragments résultant sont m / z-filtré dans le troisième quadrupôle (Q3) et quantifié par une dynode. Chaque paire de précurseur et ion de fragment est considéré comme une transition, et chaque transition est surveillée pour une période déterminée de temps (le temps de séjour; typiquement 2-50 msec). Pendant LC-SRM, les cycles QQQ-MS à travers une liste prédéfinie de transitions (le cycle de service est généralement ≤3 sec), et un chromatogramme de chaque transition est produite.
Alternative stratégs pour la quantification des protéines utilisent généralement des dosages immunologiques tels que des transferts de points, des transferts de Western, ELISA, anticorps, puces inverse des puces à protéines de phase, immunoessais microfluidiques, ELISA numériques et immunoessais base microsphères-7. Les meilleurs dosages immunologiques peuvent être beaucoup plus sensibles que les LC-SRM, et le débit de l'échantillon des immunodosages peuvent être significativement supérieur à celui de LC-SRM 5. Cependant, immunoessais en développement peuvent être coûteux et / ou de temps, et les dosages en résultent peuvent être vulnérables à la réactivité croisée et / ou des interférences, incompatible avec la cellule / lyse des tissus / méthodes d'homogénéisation et / ou ne se prêtent pas au multiplexage 5,8. Certains de ces problèmes peuvent être résolus par couplage techniques anticorps-et MS. Par exemple, les protéines cibles peuvent être enrichies en utilisant une immunoprécipitation préalable à la protéolyse et LC-SRM 12/09. En variante, la technique d'immunoprécipitation SISCAPA emploie ultérieure à la protéolyse à la leve peptidiquel 13,14. En plus des stratégies immunoenrichment, immunodéplétion de protéines de grande abondance peut être utilisé pour augmenter la sensibilité LC-SRM en réduisant les interférences par coélution analytes 15,16.
Quantification des protéines MS peut être divisé en quantification relative et absolue, et aussi dans et stable sans marqueur isotopique étiquetage (par exemple, l'étiquetage métabolique, l'étiquetage des produits chimiques, et de protéines lourde marqué et peptidiques normes internes). Techniques sans étiquette peut être utile pour la quantification des protéines rapport, mais ne conviennent pas pour la quantification absolue précision. Par comparaison, les techniques de marquage ont réduit erreur associée à la préparation de l'échantillon et la variance MS, et sont souvent utilisés pour la protéine par rapport quantification 17. Par exemple, les normes (Silap) des isotopes stables du protéome marqué préparés en utilisant une lignée cellulaire humaine cultivées permis quantification relative de biomarqueurs potentiels via LC-SRM de sérum humain 18. Absolue précise la quantification des protéines par MS exige que les normes internes purifiée, quantifiés, protéines ou peptides marqués par des isotopes être dopés-en échantillons biologiques avant MS. L'incorporation des normes internes marqués d'isotopes lourd dans un flux de travail LC-SRM permet une quantification absolue qui a été montré pour être hautement reproductible et transférable entre les laboratoires 16,19.
Isotopes stables étiquetés normes internes pour la protéine absolue quantification par MS comprennent des normes peptidiques préparés en utilisant la synthèse en phase solide 20, composées de protéines normes peptidiques de la protéase clivable concaténés 21, et 22 normes protéines pleine longueur. Cible de modification covalente de protéines et de préparation de l'échantillon incomplet (c.-à-lyse incomplète de l'échantillon et l'homogénéisation, et incomplète solubilisation de la protéine, dénaturation, alkylation, et la protéolyse) peut nuire à une quantification précise. P internerotein normes sont les moins susceptibles d'être affectés par la plupart de ces problèmes potentiels, mais ils sont généralement les plus difficiles à préparer. Une alternative consiste à analyser chaque protéine cible à l'aide de multiples normes de peptides internes qui sont conçues pour inclure des résidus flanquant natifs amino et du carboxy terminal. Indépendamment du type de l'étalon interne est utilisé, il doit être enrichi dans les échantillons-biologiques au plus tôt un point lors de la préparation de l'échantillon que possible. En outre, les techniques de préparation d'échantillons multiples (par exemple, différentes conditions de dénaturation) doivent être testés. L'utilisation de plusieurs techniques expérimentales orthogonales (cross-validation expérimentale) est une stratégie viable pour surmonter les plus quantification potentiel défis 23-25.
LC-SRM quantification de protéines est une technique très souple qui a été utilisée dans une grande variété d'applications. Notamment, il a été utilisé pour étudier peptides et de protéines à l'intérieur de biomarqueurséchantillons cliniques tels que le sérum, les biopsies de base, et des aspirations à l'aiguille fine 5. LC-SRM a également été utilisée pour mesurer la stoechiométrie des complexes protéiques 5,26, pour détecter des neurotoxines botuliniques 27, pour quantifier la dynamique de phosphorylation de protéine à l'intérieur des voies de signalisation 5, et à quantifier des changements dans la conformation des protéines 28.
Notre laboratoire utilise LC-SRM pour quantifier les protéines de signalisation qui interviennent dans le chimiotactisme des macrophages pour soutenir le développement de simulations de la voie de la chimiotaxie. Le schéma global du protocole (Figure 1) commence avec le classement des peptides cibles provisoires. Par la suite, les normes peptidiques externes bruts sont synthétisés et utilisés pour développer des tests LC-SRM pour des analyses qualitatives des échantillons biologiques. Si le peptide cible biologique de l'échantillon dérivé est détectée, les normes lourds marqué purifiés peptidiques internes sont préparés pour quantitative LC-SRM. Ce protocole peut être utilisé pour acquantifier curately protéines à partir d'une grande variété d'échantillons biologiques, et d'appuyer les enquêtes d'une grande variété de protéines cibles.
Protocole
NOTE: Cette méthode a été décrite précédemment 56.
1. Sélection peptide cible
- Dresser une liste des protéines cibles, et inclure un petit nombre de protéines de ménage pour la normalisation à travers des échantillons biologiques, et comprennent également une norme de protéine interne (par exemple, la luciférase de luciole). Digérer les protéines cibles en peptides tryptiques in silico en utilisant un outil logiciel tels que la digestion des protéines Simulator 29,30.
- Exiger que les peptides sont entièrement tryptique et ne contiennent aucun des sites de clivage de la trypsine manquantes. Évitez les peptides avec des sites de clivage de la trypsine voisins pour éviter potentiellement incomplètes digestion de la trypsine 31.
- Exiger que la longueur de chaque peptide est de 5 à 20 résidus. Envisager des peptides plus longs, mais il faut noter qu'ils sont généralement plus coûteux à synthétiser.
- Évitez un peptide si elle correspond à une variante génétique naturelle (par exemple, un seul nucléotide-e polymorphisme). Confirment également que les emplacements ne sont pas affectés de la trypsine (étape 1.2).
- Éviter un peptide si elle correspond à un site de modification post-traductionnelle (PTM). De même, d'éviter un peptide si elle serait sujette à des modifications chimiques au cours de LC-MS préparation de l'échantillon. En particulier, d'éviter les peptides sujettes à l'oxydation de la cystéine et la méthionine, l'asparagine et la glutamine désamidation, la glutamine et la formation d'amino-terminale de pyroglutamate. Confirmer que les sites de la trypsine ne sont pas affectés (étape 1.2).
- En outre, d'éviter les peptides qui proviennent d'amino- et carboxy de la protéine car extrémités terminales des protéines sont sujettes à la modification post-traductionnelle (la perte de methionine amino-terminal et une acétylation, et l'amidation carboxy-terminale).
- Exiger que chaque peptide cible est unique à chaque protéine cible. Si cela est impossible, exiger que le peptide est unique à un ensemble d'isoformes / homologues. Traiter les peptides ne diffèrent que par la leucine / isoleucine de substitutioncomme si elles étaient identiques lors de la détermination peptide unique parce que ceux-ci effectuent presque identique pendant LC-SRM.
- Si une analyse du transcriptome complet de l'ensemble des échantillons biologiques a été effectuée (par exemple, l'ARN-seq), déterminer peptide unique en utilisant des traductions de la transcription des séquences in silico 32 au lieu d'utiliser l'ensemble du protéome de l'espèce.
- Exiger que les peptides cibles sont protéotypique. Identifier des peptides protéotypiques moyen d'un fusil de spectrométrie de masse 1,2 des échantillons biologiques à l'étude. Alternativement, récupérer identifications peptidiques de peptides protéotypiques des bases de données de protéomique en ligne tels que la spectrométrie de masse NIST bibliothèques 33 (http://peptide.nist.gov/), le protéome machine mondial 34 (X HUNTER bibliothèques spectrales de http: // www .thegpm.org / HUNTER / index.html, et GPMDB peptidiques identifications de http://gpmdb.thegpm.org/), PeptideAtlas 35 (http://www.peptideatlas.org/), et la base de données de PRIDE 36 (http://www.ebi.ac.uk/pride/archive/).
- Si au niveau du compte rendu des analyses quantitatives des échantillons biologiques ont été effectués (par exemple, l'ARN-Seq, qPCR), puis éviter de cibler les peptides qui correspondent aux transcriptions d'abondance relativement faibles.
- Si nécessaire, sélectionner des peptides cibles qui peuvent être utilisées pour des dosages LC-SRM des orthologues des protéines cibles (par exemple, pour analyser à la fois les formes de la souris d'une protéine cible et humain).
- Sélectionner au moins deux peptides cibles par protéine cible (si possible).
2. Préparation d'un peptide normes
NOTE: Cette section du protocole décrit la préparation d'un ensemble de vingt normes de peptide lyophilisé (chacun étant 1 nmol en quantité) pour des analyses en aval. Pour un nombre différent de peptides, ou pour des quantités de peptides différents, il devra être ajustée en conséquence.
- Préparer normes lyophilisées peptidiques (≥1 d'nmol) en utilisant une technologie de synthèse peptidique telle que la synthèse SPOT 20,37.
- Veiller à ce que les résidus cystéine de normes peptidiques externes sont carbamidomethylated (la structure chimique formé par alkylation iodoacétamide). En revanche, en sorte que les résidus de cysteine des normes de peptides internes sont non modifié (ceux-ci seront alkylé pendant la préparation de l'échantillon, décrite ci-dessous).
- Normes de conception de peptides internes de manière qu'elles contiennent native amino- et carboxy-terminale des résidus flanquant (pour contrôler l'efficacité de digestion de la trypsine; chacun d'eux est généralement de trois à six résidus de long).
- Concevoir des normes peptidiques internes afin qu'ils soient isotope stable marqué. Considérons le profil isotopique naturelle du peptide cible et la précision de la mesure de masse de la station mobile lors de la sélection d'une stratégie de marquage peptidique. Ne pas utiliser les normes peptidiques deutérées car les causes peptide de deutération en phase inverse temps de rétention de LCs à 38 déplacer.
Remarque: En règle générale, les normes de peptides tryptiques internes sont synthétisés en utilisant ~ 98% [13 C 6, 15 N 4] Arg et [13 C 6, 15 N 2] Lys incorporé à l'extrémité carboxy-terminales. - Purifier normes peptidiques internes par HPLC 39, et de quantifier avec précision les 40.
- Ajouter 100 ul de 0,1% v / v d'acide formique, 20% v / v d'acétonitrile (ACN) pour chaque 1 nmol lyophilisée Indice de peptide pour produire une concentration de peptide de 10 uM (utilisation de soin que le peptide lyophilisé peut avoir une très basse densité et peut être facilement perdu). Vortex les échantillons pendant 2 minutes et le bain sonication eux pendant 5 minutes pour assurer que le peptide dissolution complète.
- Regrouper les peptides en solution, et on concentre le mélange résultant dans un concentrateur sous vide à un volume final de 80 ul. Ajouter 20 ul d'ACN pour dissoudre tout peptide précipité.
Remarque: L'échantillon désormais contains 10 uM de chaque peptide et 20% v / v d'ACN. - Pour les normes peptidiques internes, préparer une série de dilution pour quantitative LC-SRM:
- Préparer 100 ul d'une dilution 1 uM: combiner 90 ul de 20% v / v d'ACN et 10 ul du mélange de peptides de 10 uM.
- Préparer 100 ul d'une dilution 100 nM: combiner 90 ul de 20% v / v d'ACN et 10 ul du mélange de peptides de 1 uM.
- Pour peptidiques standards externes, préparer 100 ul d'une dilution à 1 uM pour LC-MS en combinant 90 ul de 0,1% v / v d'acide formique et 10 ul du mélange de peptides de 10 uM.
Note: L'échantillon contient maintenant une uM de chaque peptide, v / v d'acide formique à 0,1% et 2% v / v ACN, et est prêt à être utilisé pour le développement de tests LC-SRM.
3. LC-SRM Assay développement
- Analyser les mélanges des normes peptidiques externes (environ 1-10 pmol de chaque peptide par injection) par MS en utilisant un fusil de chasse HPL nano-débitC système couplé à un spectromètre de masse triple quadripôle (LC-MS-QQQ (/ MS)).
- Utilisez un système LC-MS équipé d'une colonne capillaire emballé avec C-18 médias (≤5 de diamètre, um ~ 200 pores de A, longueur ≥10 cm, id = 50-100 um) et une pointe nano-ESI (généralement produites à l'aide un laser-extracteur). Assurez-vous que chaque série LC-MS comprend un gradient min ~ 60 linéaire (typiquement, 0-40% de solvant B), une étape colonne de régénération (~ 80% de solvant B), et une étape colonne ré-équilibrage (~ 0% de solvant B ) (solvant A = 0,1% v / v d'acide formique dans H 2 O, solvant B = 0,1% v / v d'acide formique dans de l'ACN, débit = 200 à 800 nl / min, tension ESI = 1,800 V, la largeur d'isolation de Q3 = 0,7 m / z, pression d'argon de Q2 = 1,5 mTorr).
- Pour chaque balayage précurseur d'ions, sélectionner dynamiquement le haut ~ 10 ions précurseurs les plus intenses pour la spectrométrie de masse en tandem (SM / SM). Exécuter chaque échantillon en double exemplaire, de telle sorte que deux rampes de l'énergie de collision différents sont utilisés: l'un destiné à fragmenter de façon optimale deux ions précurseurs, et l'autrepour trois précurseur ions 41.
- Effectuez la LC-MS-QQQ (/ MS) analyses par analyse ion précurseur de Q3 (de balayage précurseur d'ions Q1 peut causer des résidus des pics précurseur d'ions résultant de collisions d'argon à faible énergie au T2).
- Assurez-vous que le système LC-MS-ci fonctionne correctement en analysant des échantillons de CQ et répétitions techniques. Éviter le report d'échantillon à l'aide de colonnes CL fraîchement préparés et en exécutant plusieurs ébauches entre les échantillons.
- Analyser les fusil de chasse LC-MS-QQQ (/ MS) à l'aide de données issues base de données de recherche contre les séquences des normes peptidiques externes 42. Le cas échéant, jetez identifications peptidiques ambiguës utilisant les scores d'identification de peptide de confiance (par exemple, les valeurs moyennes) ou en utilisant la modélisation statistique 42. Peu importe, examiner manuellement tous les identifications peptidiques 43 pour assurer qu'ils sont tous sans ambiguïté.
- Utilisez le fusil de chasse MS identifications peptidiques de construct une bibliothèque de spectre en utilisant un logiciel tel que Skyline.
- Préparer une liste de transition LC-SRM en utilisant les 3-10 transitions les plus intenses par ion précurseur (2 et 3 ions précurseurs; +1 et +2 fragments ions; y-, b-, et a-ions qui sont ≥2 résidus long).
- Jeter une transition si le précurseur et le fragment ion m / z valeurs se chevauchent au sein de la précision de mesure de masse de la QQQ-MS (fragmentation précurseur d'ions est parfois incomplète, souvenez-vous de considérer la monoisotopique et les formes isotopiques naturelles lourds, ainsi que la non marqué et formes lourde marqué).
- De même, jetez une transition si le fragment ions m / z chevauche celle d'un autre ion fragment de la même ion précurseur.
- Utilisez les listes de transition résultant d'effectuer LC-SRM analyse des mélanges des normes peptidiques externes (effectuer spectrométrie de masse comme décrit dans l'étape 3.1; Q1 et Q3 largeur d'isolement = 0,7 m / z; le temps de séjour = 2-50 msec).
- Mamannuellement examiner les données LC-SRM résultant et de suppression des dosages peu performants (l'analyse de données de LC-SRM est décrit à la section 5).
4. LC-SRM dosages d'échantillons biologiques
- Préparer les plats de cellules cultivées 44. Si plusieurs conditions expérimentales sont comparées, bloc et randomisent les échantillons pour réduire les biais systématiques possibles 45.
- Pour chaque boîte de cellules, aspirer le milieu de culture cellulaire aux déchets et les cellules en suspension dans un tampon sans sérum 44. Le cas échéant, laver les cellules dans un tampon sans sérum pour éliminer toute protéine restante extracellulaire.
- Pour chaque échantillon, compter le nombre de cellules viables et le total en utilisant une tache de viabilité (par exemple, bleu trypan) et un hémocytomètre ou un compteur de cellules automatisé 44.
- Pellet les cellules par centrifugation, et à aspirer les déchets surnageants 44 (un volume typique de culot de cellules est d'environ 30 ul).
- Ajouter 400 pi de l'urée tampon de lyse ou tensioactif tampon de lyse à chaque culot cellulaire (100 mM HEPES ∙ NaOH pH 8, 10 uM bestatine chlorhydrate, 10 pm pepstatine A, et soit 8 M urée ou 0,1% p / v tensioactif (respectivement ); fraîchement préparée; utiliser un tensioactif MS-compatible acidolabile comme RapiGest SF ou PPS silencieux tensioactif). Si l'on utilise d'autres inhibiteurs de protéase, en sorte qu'ils ne sont pas inhiber la trypsine.
- Mélanger chaque échantillon en utilisant pipetage doux, et transférer chacun à un tube de 2 ml (avec un bouchon à vis avec un joint torique) contenant ~ 100 pi de 0,1 mm zircone / billes de silice (à noter que les perles peuvent causer des tubes à bouchon à fuir ). Lyse des cellules par vortex les échantillons pendant 5 min à pleine vitesse (ce lyse des cellules par perle coups).
- Alternativement, la lyse des cellules en utilisant une méthode mécanique différent (par exemple, en utilisant une presse ou d'homogénéisation des conseils français).
Remarque: Evitez de centrifugation lysats cellulaires parce que cela pourrait sédimenter protéine précipitée qui pourrait otherwise tryptically être digéré et détectée par LC-MS.
- Alternativement, la lyse des cellules en utilisant une méthode mécanique différent (par exemple, en utilisant une presse ou d'homogénéisation des conseils français).
- Pour les échantillons dénaturés tensioactifs, les incuber à 90 ° C pendant 10 min pour faciliter l'homogénéisation de l'échantillon et la dénaturation des protéines.
- Bain de traitement par ultrasons des échantillons pendant 10 min à la température ambiante pour aider l'homogénéisation et la dénaturation des protéines.
Remarque: Le tampon de lyse et les lysats peuvent être conservés à -80 ° C (le tampon de lyse sera nécessaire pour les étapes 4.9 et 4.13). - Effectuer une analyse de la concentration de protéine 46 (par exemple, un dosage de l'acide bicinchoninique) des lysats (et du tampon de lyse aussi une expérience témoin).
- Pour les analyses qualitatives (c.-à-pas de normes peptidiques internes seront dopés dans les échantillons), pipette 200 ug (masse de protéines) de lysat cellulaire dans un tube de 1,5 ml frais de microcentrifugeuse. Pour les analyses quantitatives, préparer quatre de ces échantillons pour une série de dilution des isotopes stables.
- Pour chaque échantillon, ajouter un étalon interne de la protéine (par exemple </ Em>, ajouter ~ 5 pmol de ~ 98% pur luciole luciférase).
- Pour les analyses quantitatives, ajouter la série de dilution isotopique stable du mélange équimolaire des normes peptidiques internes (par exemple, 0, 0,2, 2, 20 pmol de chaque peptide) aux échantillons.
- En parallèle, préparer des échantillons témoins en utilisant les normes de peptide interne seul (pas de lysat de cellules) (par exemple, 0, 0,2, 2, 20 pmol de chaque peptide).
- Préparer également des échantillons témoins en utilisant les normes de peptides internes et externes (par exemple: 2 pmoles de chaque norme de peptide externe, et 0, 0,2, 2, et 20 pmol de chaque étalon interne peptidique).
Remarque: Assurez-vous que les normes peptidiques sont libres d'acide formique car elle peut interférer avec la digestion de la trypsine.
- Ajouter du tampon de lyse de telle sorte que tous les échantillons sont des volumes identiques. Remarque: Un volume d'échantillon typique est de 70 pi, donc ce volume sera utilisé pour ce protocole.
- Réduire les résidus de cysteine des protéines par addition de 0.7 pl de DTT 1 M (fraîchement préparé) à chaque échantillon (la concentration finale de DTT est de 10 mM) et l'incubation des échantillons à 60 ° C pendant 30 min.
- Alkyler cystines de la protéine en ajoutant 7 ul de l'iodoacétamide tamponnée (500 mM d'iodoacétamide, 1 M de HEPES ∙ NaOH pH 8, fraîchement préparées) à chaque échantillon (la concentration de l'iodoacétamide finale est de 50 mM) et l'incubation des échantillons à la température ambiante pendant 20 min dans l'obscurité (iodoacétamide est sensible à la lumière). Si nécessaire, arrêter la iodoacétamide restant en ajoutant du DTT à une concentration finale de 50 mM.
- Pour chacun des échantillons dénaturés en utilisant l'urée, ajouter 482 ul de 100 mM d'HEPES ∙ NaOH pH 8, de sorte que la concentration finale de l'urée est de 1 M (trypsine est inhibée de manière significative à> 1 M d'urée 47).
- Digérer Tryptically les protéines en peptides.
- Pour chaque échantillon qui contient un lysat de cellules, ajouter 8 pl de 0,5 pg / pl de qualité de séquençage trypsine modifiée de sorte que le concentrat de la trypsine finaleion est de 1:50 (protéine p: p) trypsine: échantillon, et incuber l'échantillon à 37 ° C pendant 18 heures.
- Pour chaque échantillon ne contenant pas de lysat cellulaire, ajouter 0,5 ul de 0,5 ug / ul teneur modifiée de la trypsine de séquençage, et incuber l'échantillon à 37 ° C pendant 2 heures (chacun de ces échantillons sont pour des expériences de contrôle, et généralement ne contiennent que 10 ng -10 pg de peptide + masse protéique).
- Pour chacun des échantillons dénaturés urée, ajouter 440 ul de 2% v / v d'acide formique (concentration finale de l'acide formique est de 1% v / v). Confirment que ces échantillons sont pH ~ 3.
- Pour chacun des échantillons dénaturés tensioactifs, ajouter 914 ul de 0,5% v / v de TFA (la concentration finale est de 0,5% de TFA v / v). Confirment que ces échantillons sont pH ~ 1,5. Incuber les échantillons à 37 ° C pendant 60 min pour hydrolyser l'agent tensio-actif.
- Microcentrifugeuse tous les échantillons à 21 000 xg pendant 20 min à la température ambiante pour obtenir un culot agent tensio-actif du groupe de queue et tout autre precipitates qui obstruer une cartouche C-18 SPE.
- Phase solide extraire chacun des surnageants en utilisant un C-18 cartouche SPE jetable (tampon A = 0,1% v / v d'acide formique; tampon B = 0,1% v / v d'acide formique, 80% v / v ACN; ~ C- 100 mg 18 résine par cartouche). Forcer les phases mobiles grâce à la cartouche C-18 SPE en utilisant une poire en caoutchouc à surface plane, en utilisant un collecteur d'extraction, ou par centrifugation la cartouche dans un tube de 15 ml de centrifugeuse à 10 g pendant 5 min.
- Mouiller la colonne en appliquant 1 ml de tampon B, équilibrer en appliquant 1 ml de tampon A deux reprises, appliquer l'échantillon, et laver la cartouche en appliquant 1 ml de tampon A deux fois. Éluer les peptides en appliquant 1 ml de tampon B lentement (~ 2 min).
- Concentré chaque éluat dans un concentrateur sous vide à un volume final de 100 ul à évaporer à une distance de ACN. Ajouter 200 ul de H 2 O pour chaque échantillon, et on concentre chacune dans un concentrateur sous vide à un volume final de 98 ul à évaporer à une distance de tout re possiblerestant ACN.
- Ajouter 2 ul de 5% v / v d'acide formique dans de l'ACN à chaque échantillon (les concentrations de l'échantillon final est de 0,1% v / v d'acide formique, de 2% v / v ACN).
Remarque: Les échantillons peuvent être conservés à -80 ° C. - Analyser les échantillons en utilisant LC-MRS (comme dans l'étape 3.4). Pour les analyses qualitative LC-SRM, exécutez les échantillons biologiques et les normes peptidiques externes, et d'analyser toutes les données résultant ensemble. Pour les analyses quantitative LC-SRM, exécutez la série de dilution isotopique de chaque échantillon biologique, et aussi exécuter les échantillons constitués des seules normes peptidiques, et d'analyser toutes les données résultant ensemble.
5. Analyse LC-SRM données
NOTE: identification et la quantification de peptides peuvent être très simplifiées et partiellement automatisés à l'aide de logiciels tels que Skyline, mais il est toujours fortement recommandé que tous annotation des données soit revu manuellement. Aussi, il est préférable d'exclure les informations de niveau de protéines pendant Annot manuelation des données LC-SRM pour empêcher partialité.
- Pour chaque identification de peptide, de confirmer que la forme de chaque profil transition d'élution est à peu près gaussienne. Pour chaque transition, confirment que le profil d'élution est le produit de plusieurs signaux mesurés par le détecteur de MS (c.-à-pas est le produit d'une ou deux des pointes aléatoires de bruit MS).
- Pour chaque identification de peptide, veiller à ce que l'ensemble du profil d'élution peptide est sélectionné. Évitez ajuster le profil d'élution limites de sous-composantes de l'identification de peptides (sous forme de peptide lourde marqué, la forme non marquée, les ions précurseurs individuelles, et les transitions individuelles) manuellement.
- Vérifiez que chaque profil d'élution transition a un rapport signal-sur-bruit (S / N) ≥3. Remarque: "bruit" fait référence au bruit de MS aléatoire, pour ne pas reproductibles signal du co-éluées analytes. Fonctions de lissage CHROMATOGRAMME peuvent grandement aider l'analyse de données bruitées.
- Pour chaque identification d'un peptide,confirmer que toutes les transitions ont des profils d'élution presque identiques (pas seulement les fois apex pointe égales; profils d'élution peuvent avoir des amplitudes différentes).
- Pour LC-SRM analyse d'une grande quantité des normes peptidiques (typiquement ≥100 de fmoles de chaque peptide), confirmer que les profils peptide d'élution correspondants sont très intenses (s généralement / n ≥100), et que les identifications peptidiques sont sans ambiguïté. Remarque: Ces identifications très confiants seront cruciales pour l'identification des peptides d'échantillons biologiques dérivés correspondants.
- Pour chaque identification d'un peptide, confirment que les intensités relatives de transition correspondent à celles de l'étalon de peptide identifié en toute confiance. Si nécessaire, jetez profils bruyants, d'intensité relativement faible transition élution, mais confirmer qu'aucun des transitions relativement intenses sont manquants. Ne tenez pas compte des profils de transition élution relativement intenses qui sont considérablement et sans ambiguïté touchés par un coéluant contaminant.
- Pour chaque identification de peptide, estimer la probabilité qu'il a été produit de façon aléatoire par un signal de fond (bruit de MS aléatoire signal plus reproductible d'analytes co-éluées).
- Estimer cette probabilité manuellement en examinant l'ensemble du chromatogramme LC-SRM et l'estimation de l'unicité de chaque identification de peptide. Produire une série d'identifications peptidiques confiant qui a un taux estimé de fausse découverte (FDR) de 5%. Utilisez des critères plus stricts (par exemple, FDR ≤1%) si une plus grande confiance définir des identifications est nécessaire (utilisation de critères plus souples est pas recommandé).
- Vous pouvez également utiliser l'algorithme mProphet 48 ou l'algorithme de vérification 49, mais examiner manuellement les résultats.
- Pour chaque identification d'un peptide, confirment que le temps d'élution observé est cohérent avec l'hydrophobicité du peptide (Skyline est conçu pour effectuer ce calcul).
- Pour chaque peptide, confirmer queson temps d'élution est cohérent à travers les pistes LC.
NOTE: LC-SRM analyses parfois produire des profils d'élution qui sont systématiquement biaisée entre les courses. Changements systématiques sont à prévoir après les modifications d'instruments tels que le remplacement d'une colonne. En outre, les peptides à élution précoce peut être sujette à l'évolution (en particulier si la colonne est chargé à pleine capacité), et à la fin peptides élution peuvent également être sujettes à se déplacer. l'identification des peptides est maintenant terminée. - Pour améliorer peptide quantification, réinspecter chaque identification de peptides et jeter un profil d'élution transition si elle est particulièrement bruyant par rapport aux autres profils d'élution de transition à l'identification des peptides. Jeter un profil d'élution transition si son intensité relative de transition est faux (par rapport à la norme de peptide).
- Inspecter les limites de chaque profil d'ion précurseur et la transition élution. Si nécessaire, ajuster soigneusement les limites de profil d'élution à rogner signal de fond (ou improve fond d'estimation de signal).
- Pour chaque norme de peptide interne, vérifier si elle est contaminée par une quantité détectable de peptide lumière (déterminé par le LC-SRM analyse des normes peptidiques internes seul, décrit ci-dessus). Par la suite, jeter tous les profils d'élution peptide légers qui ont été considérablement compromises par la contamination lumière peptide dans les normes peptidiques internes.
- Quantifier chaque profil d'élution de transition en calculant surface de son pic de LC. Le cas échéant, déduire le signal de fond estimé. Quantifier chaque profil d'élution des peptides en additionnant les valeurs de quantification transition correspondantes (ci-après, ceci est considéré comme la valeur "Sum_Peak_Area»). Pour chaque valeur de Sum_Peak_Area échantillon de peptide biologique, calculer l'abondance biologique molaire de peptide de l'échantillon en utilisant les données de LC-SRM de normes peptidiques internes ou externes (utilisation seulement les transitions qui ont été quantifiés avec succès à la fois par le peptide échantillon biologiqueLC-SRM et celle de la norme de peptide).
- Quantification en utilisant des normes peptidiques externes (non recommandé):
- Tracer les peptides externe valeurs Sum_Peak_Area standards par rapport aux peptides externe valeurs d'abondance molaires standard. Produire une courbe standard en ajustant une régression linéaire pour les données (généralement, seule la composante linéaire de la plage dynamique doit être utilisé; tout composant non linéaire doit être utilisé avec une extrême prudence). Pour chaque valeur de Sum_Peak_Area échantillon de peptide biologique, utiliser la courbe standard pour calculer l'abondance molaire de peptide.
Note: Cette méthode de quantification est déconseillée car elle exige que la préparation de l'échantillon et LC-SRM être démontrabilité robuste parce que les échantillons biologiques et des normes peptidiques externes sont préparés et analysés par LC-SRM séparément. En outre, il ne tient pas compte des effets de matrice (effets dus à des composants d'un échantillon autres que l'analyte).
- Tracer les peptides externe valeurs Sum_Peak_Area standards par rapport aux peptides externe valeurs d'abondance molaires standard. Produire une courbe standard en ajustant une régression linéaire pour les données (généralement, seule la composante linéaire de la plage dynamique doit être utilisé; tout composant non linéaire doit être utilisé avec une extrême prudence). Pour chaque valeur de Sum_Peak_Area échantillon de peptide biologique, utiliser la courbe standard pour calculer l'abondance molaire de peptide.
- Quantification utilisant internenormes peptidiques (recommandé):
- Pour chaque valeur de Sum_Peak_Area échantillon de peptide biologique et peptide interne valeur Sum_Peak_Area norme lourde marqué correspondant, calculer le rapport clair / lourd. Utilisez ce rapport comme une mesure de la lumière / peptide lourd rapport d'abondance molaire correspondant à calculer l'abondance molaire du peptide de l'échantillon biologique. Plus précisément, l'abondance biologique molaire de peptide de l'échantillon est égale à l'abondance molaire peptide standard interne multiplié par le rapport clair / de Sum_Peak_Area lourde.
- Pour chaque réplicat et peptide cible biologique, d'identifier la plage linéaire de la quantification LC-SRM en créant une courbe d'étalonnage en utilisant une régression linéaire. Plus précisément, tracer les peptides interne valeurs d'abondance molaire standards par rapport à la lumière des valeurs lourds / de Sum_Peak_Area ratio (à noter que la masse de l'échantillon biologique est constante à travers l'ensemble de données). Exiger que chaque échantillon valeur peptide Sum_Peak_Area biologique est dans la gamme linéaire (typicallié, seule la composante linéaire de la gamme dynamique doit être utilisé; tout composant non linéaire doit être utilisé avec une extrême prudence). De même, effectuer cette étape pour chaque transition de peptide cible.
Remarque: Analyte quantification par LC-QQQ-SRM devrait être linéaire sur une large plage dynamique (~ 10 000). À l'extrémité inférieure, signal de fond (bruit de MS aléatoire signal plus reproductible d'analytes co-éluées) permettront de réduire la précision de quantification et de précision. À l'extrémité supérieure, la saturation du détecteur provoquera la non-linéarité (en fin de compte, cela peut entraîner des profils d'élution d'être au départ plat une intensité de signal spécifique).
- Quantification en utilisant des normes peptidiques externes (non recommandé):
- Le cas échéant, procéder à une normalisation internationale pour les échantillons, par exemple une normalisation tendance centrale 50, pour corriger les légères différences dans la quantité de l'échantillon avant l'ensemencement dans des normes de peptides internes. Si nécessaire, utilisez uniquement les protéines d'entretien ménager de l'étape 1.1.
- Si nécessaire, imputer quantificat manquantValeurs ion 51,52.
Remarque: Il est inapproprié de simplement remplacer les valeurs manquantes par la moitié de la limite inférieure de quantification (LIQ) ou la moitié de la limite de détection (LOD) parce que cela permettrait de réduire artificiellement la quantification variance et pourrait entraîner un test statistique (par exemple, une analyse de la variance ) la production d'un résultat faussement positif (une erreur de type I). Cependant, en ignorant les valeurs manquantes peut également être problématique. Par exemple, si la moitié des vraies valeurs d'abondance sont en dessous de la limite de détection, et l'autre moitié sont au dessus de la LIQ, alors la moyenne observée abondance serait surestimer la véritable abondance signifie. - Veiller à ce que les essais satisfont aux lignes directrices largement acceptées pour LC-SRM 23-25. En particulier, exigent que le coefficient de variation des valeurs de quantification est typiquement ≤25% pour des essais cliniques, et ≤35% pour les essais non cliniques 25. Pour les analyses LC-MRS liées à la santé ou vétérinaires produits ou services qui doivent être pris en considération pour l'application réglementaireroval, exigent que les tests répondent aux exigences de précision plus strictes pour ces essais: "La précision déterminée à chaque niveau de concentration ne doit pas dépasser 15% du coefficient de variation (CV) à l'exception de la LIQ, où il ne devrait pas dépasser 20% de la CV "23,24.
Résultats
Le développement de modèles informatiques de prévision de voies de signalisation est l'un des objectifs fondamentaux de la biologie des systèmes 53. Malheureusement, même pour les voies qui ont été largement étudiés et ont une haute signification clinique de signalisation, il est encore généralement pas possible de prédire quantitativement le comportement voie en réponse à des perturbations (par exemple, cela est vrai pour le / ERK MAPK 54). Récemment, une enquête employée protéomique, de la transcriptomique ciblées, et la modélisation computationnelle et simulation pour étudier les chimiotactisme des macrophages de souris voie 56 de signalisation. L'objectif de l'enquête était la sphingosine-1-phosphate chimiotaxie médiation de cellules RAW 264.7 (une lignée de cellules monocytes de souris / macrophages). Pour faciliter la modélisation de la voie, des analyses LC-SRM ont été développés et réalisés pour mesurer l'abondance absolue des protéines de la voie de chimiotactisme dans les cellules RAW 264.7. Les valeurs d'abondance résultant étaient utilisationd en tant que paramètres du modèle de voie.
Le schéma expérimental global (Figure 1) a commencé avec une liste de protéines cibles, qui comprenait G i2, une protéine G α-sous-unité hétérotrimérique. Le succès du protocole global est très dépendante de la sélection des cibles peptidiques qui sont protéotypique et quantotypic, comme YDEAASYIQSK. Mélanges de normes peptidiques externes ont été préparés et analysés par shotgun LC-MS-QQQ (/ MS). Le tandem YDEAASYIQSK spectre de masse résultant a été composée de plusieurs ions fragments, et il y avait un fond de faible (Figure 2). Les spectres ont été utilisés pour composer des listes de cibles LC-MRS contenant les dix premiers ions fragments les plus intenses par ion précurseur.
Un mélange de 409 normes peptidiques externes ("JPT409») a été analysé en triple exemplaire par LC-SRM qualitative (données non présentées). Cet échantillon comprenait trois G peptides i2, et l'identification de tous les trois étéjugée confiant. Par la suite, RAW 264.7 échantillons (réplicats biologiques "raw1" et "raw2") et l'échantillon de JPT409 ont chacun été analysés en double (deux répétitions techniques LC-MRS) par qualitative LC-SRM (ces six LC-SRM a réalisé des analyses de la même façon que possible). Le peptide YDEAASYIQSK a été identifié en toute confiance dans les six analyses (figures 3A - C). Les diagrammes d'intensité de transition YDEAASYIQSK étaient les mêmes dans les six analyses LC-SRM, et ceux-ci étaient à peu près similaire à la carabine LC-MS (/ MS) motif (la "Bibliothèque" répliquer de la figure 3C).
Le motif des intensités de transition est seul ne suffit pas toujours pour l'identification confiante d'un peptide. Le caractère hydrophobe des peptides et mesuré le temps de rétention LC doivent être cohérents. En outre, les normes peptidiques externes et les peptides d'échantillons biologiques correspondants doivent avoir environtemps de rétention égales. Le caractère hydrophobe (estimée en utilisant la version 3.0 SSRCalc 100 algorithme de 55 Å) et un temps de rétention observé de YDEAASYIQSK ont été trouvés être conformes (figure 4A). En outre, le temps de rétention de YDEAASYIQSK été mesurée en utilisant à la fois les analyses RAW 264.7 et les analyses des normes peptidiques externes, et toutes les valeurs de temps de rétention était égal à environ (figure 4B).
La plupart du qualitative LC-SRM analyses des échantillons biologiques ont réussi, de manière correspondante lourde marqué, purifiée, quantifiée normes peptidiques internes ont été préparés et enrichis-en RAW 264.7 lysats cellulaires. Isotope série de dilution LC-SRM a été utilisé pour analyser des échantillons constitués des normes peptidiques interne seules (échantillon "R0"), l'interne et les normes peptidiques externes (échantillon "R1"), et six RAW 264.7 répétitions biologiques (échantillons "R2" - «R7»). Deux différent dénaturants protéiques ont été utilisés pour tester tous les problèmes possibles protéine solubilisation, dénaturation, alkylation, et / ou qui pourrait nuire à la digestion quantification précise de la protéine cible (R2-R4 échantillons utilisé urée, et des échantillons R5-R7 utilisé RapiGest SF). Les formes de YDEAASYIQSK légers et lourds contenaient des motifs d'intensité de transition presque identique et profils d'élution (figures 5A - C). LC-SRM résumé des rapports de surface de pointe ont été utilisés comme une mesure de l'abondance relative de peptide, et les ratios de YDEAASYIQSK ont été utilisés pour calculer les valeurs G i2 d'abondance en unités de copies par RAW 264.7 cellules. En parallèle, une seconde G i2 interne standard de peptide (IAQSDYIPTQQDVLR) a été utilisé pour effectuer quantitatives essais G i2 LC-MRS des mêmes RAW 264.7 échantillons, et les deux dosages produite mesures G i2 d'abondance sont similaires dans toutes les répétitions biologiques (Figure 5D). L'accord des deux G Les tests de i2 est une preuve solide que les deux peptides sont quantotypic, et que tous les tests quantitatifs G i2 LC-SRM étaient exactes et précises.
Dans l'ensemble, 35 les protéines ont été 58 quantifiées en utilisant les normes de peptides internes (Tableau 1). Notamment, les analyses LC-MRS de la protéine de référence interne (luciférase de luciole; Étape 4.11) étaient exactes et précises. L'ensemble complet de données d'horizon de cette enquête est disponible en ligne à la base de données LC-SRM Panorama (dans le dossier Manes_RAW_Chemotaxis au https://panoramaweb.org/labkey/project/NIH_NitaLazar/begin.view).
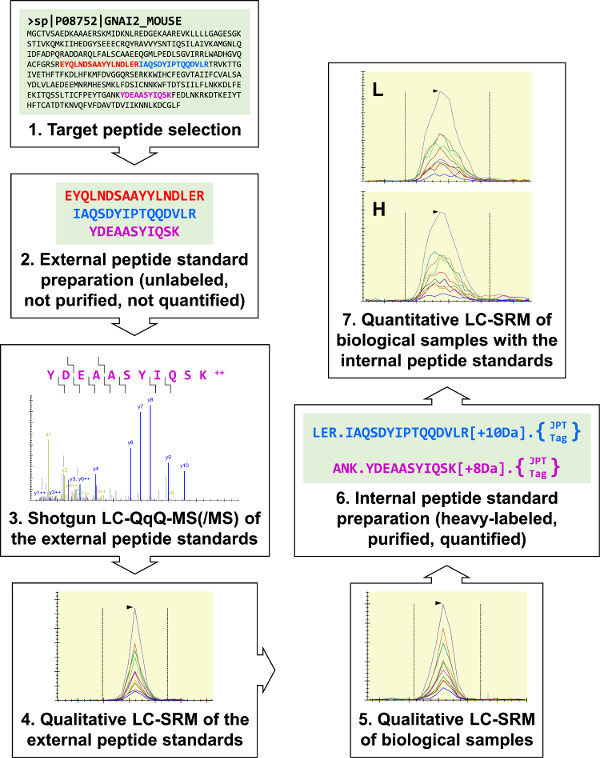
Figure 1:. Présentation du protocole Trois peptides cibles indicatives pour G i2 (une protéine G α-sous-unité hétérotrimère) ont été choisis pour la synthèse peptidique externe standard. Ce sont analyzed par shotgun LC-MS (/ MS) pour développer trois G i2 analyses LC-SRM, et ces essais ont été utilisés pour effectuer des analyses qualitatives des échantillons biologiques. Tous les trois G peptides cibles i2 ont été identifiés, et deux ont été sélectionnés pour le peptide interne préparation standard. Trypsine clivable "JPT-tags" ont été utilisés pour quantifier les normes peptidiques internes utilisant spectrophotométrie UV. Les normes peptidiques internes ont été utilisés pour effectuer quantitatives G LC-i2 SRM dosages de RAW 264.7 échantillons. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
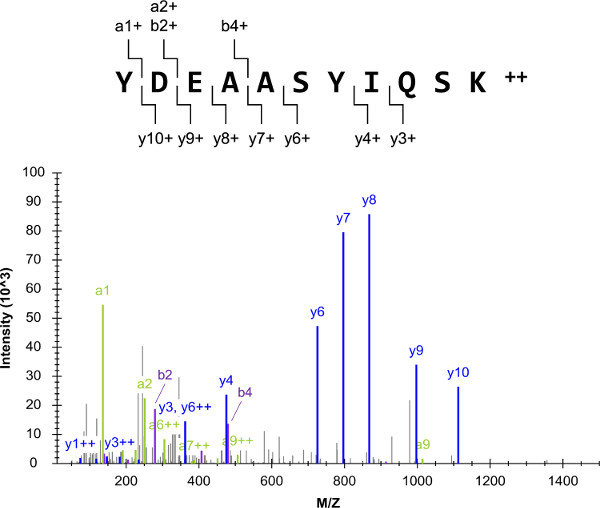
Figure 2:. Shotgun LC-MS (/ MS) Le spectre de masse représenté provenaient des analyses de l'un des i2 normes peptidiques externes G (YDEAASYIQSK). Pour ce précurseurion, les dix premières transitions les plus intenses ont été sélectionnés pour LC-SRM (excluant l'ion a1 1+ fragment raison de sa courte longueur). S'il vous plaît, cliquez ici pour voir une version plus grande de cette figure.
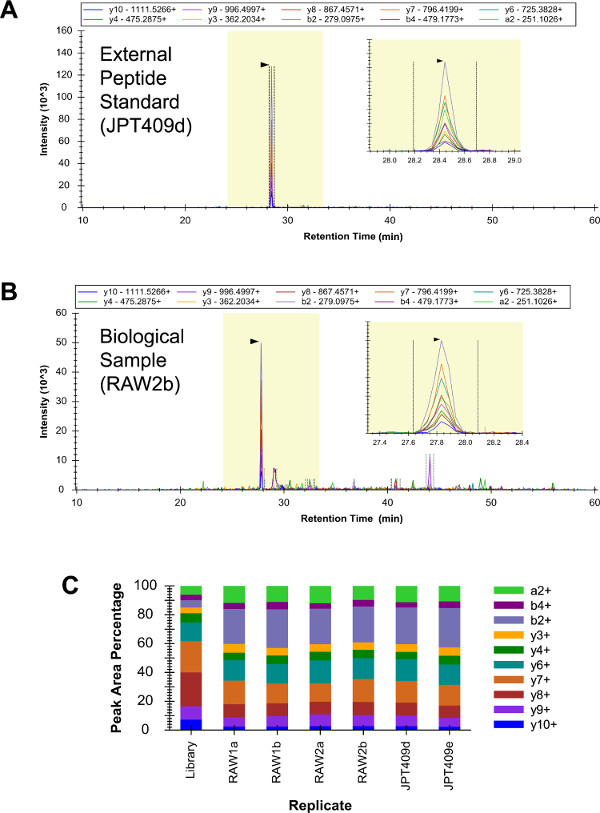
Figure 3:. LC-SRM qualitative de G i2 Le chromatogramme de la YDEAASYIQSK JPT409 analyses contenait une très faible bruit de fond et le peptide a été identifié en toute confiance (A). Les analyses 264.7 RAW correspondant abouti à plus de signal de fond, mais l'identification de peptide était toujours sans ambiguïté (B). Les motifs d'intensité relative de transition étaient les mêmes dans tous les six LC-SRM analyse (deux répétitions techniques de trois échantillons indépendants), et ceux-ci étaient à peu près Similar pour le fusil de chasse correspondant LC-MS (/ MS) motif (la "Bibliothèque" répétition) (C). S'il vous plaît, cliquez ici pour voir une version plus grande de cette figure.

Figure 4: prévision Peptide de temps de rétention et de la variation de l'autre côté exécute une régression linéaire a été calculé en utilisant l'hydrophobie estimée et mesurée qualitative LC-SRM temps d'élution de l'ensemble des peptides cibles, et les temps d'élution prédites et mesurées de YDEAASYIQSK étaient en accord (A). . En outre, la consistance de la durée d'élution observés de chaque peptide pour les analyses LC-SRM a été déterminée. Les temps d'élution de YDEAASYIQSK couvraient une gamme de ~ 40 sec, ce qui était conforme à la précision de l'instrument LC-SRM qui a été utilisé (les valeurssont l'heure de pointe sommet +/- la largeur totale à mi-max, et aussi la pleine largeur à la base du pic) (B). S'il vous plaît, cliquez ici pour voir une version plus grande de cette figure.
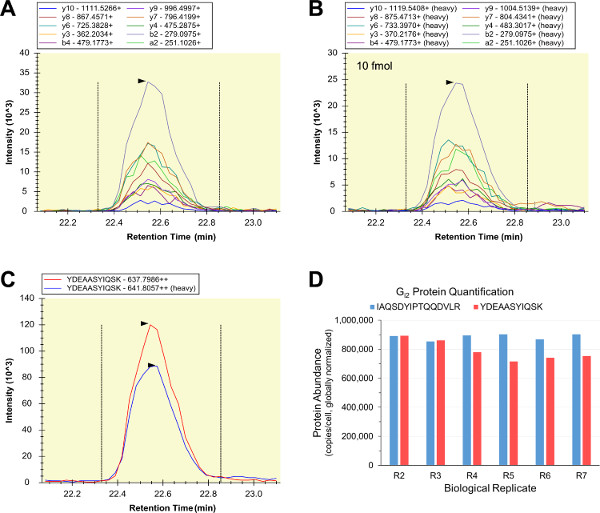
Figure 5:. Quantitative LC-SRM de G i2 Les chromatogrammes de transition de l'échantillon biologique peptide (A) et la norme de peptide interne (B) ont des intensités de transition relativement uniformes, et ont été additionnées (C) (représenté est l'analyse "R2" pour YDEAASYIQSK utilisant 10 fmol peptide standard interne). Les profils d'élution légers et lourds étaient en accord (C), et le rapport de l'aire sous les courbes a été utilisé comme une mesure de la light / lourd abondance de peptide. Un second G i2 peptide standard interne (IAQSDYIPTQQDVLR) a été utilisé pour quantitative LC-SRM en parallèle, et les valeurs d'abondance G i2 résultant étaient conformes dans les deux les six réplicats biologiques et les deux peptides cibles (globaux, n = 12 et CV = 8,38 %) (D). S'il vous plaît, cliquez ici pour voir une version plus grande de cette figure.
| UniProt adhésion | Protein cible | Cible Peptide | Abondance (fmol / ig) | Abondance (copies / cellule, normalisée) | CV |
| P08659 | Luciférase | (UV à 280 nm) | 23,824 | n / A | n / A |
| P08659 | Luciférase | VVDLDTGK | 24.103 | n / A | 7% |
| P08659 | Luciférase | VVPFFEAK | 24,717 | n / A | 4% |
| P60710 | Actin, cytoplasmique 1 | IWHHTFYNELR | 760,448 | 61762598 | 3% |
| P16858 | GAPDH | LISWYDNEYGYSNR | 357,803 | 28906524 | 14% |
| P06151 | Lactate déshydrogénase | LLIVSNPVDILTYVAWK | 129,623 | 10633145 | 30% |
| P99024 | Tubuline β 5 | ALTVPELTQQVFDAK | 78,765 | 6398971 | 3% |
| P20152 | Vimentin | SLYSSSPGGAYVTR | 121,100 | 9807488 | 10% |
| P60766 | CDC42 | DDPSTIEK | 86,647 | 6957317 | 27% |
| P60766 | CDC42 | QKPITPETAEK | 26,475 | 2153669 | 6% |
| Q8C3J5 | DOCK2 | ETLYETIIGYFDK | 1.459 | 118539 | 15% |
| Q8C3J5 | DOCK2 | ISSSPTHSLYVFVR | 1.795 | 143440 | 33% |
| Q8BPU7 | ELMO1 | ALTTKPSSLDQFK | 2.302 | 186754 | 7% |
| Q8BPU7 | ELMO1 | SAIDISILQR | 1.244 | 99728 | 25% |
| Q8BGM0 | FGR | GAYSLSIR | 1.099 | 89473 | 17% |
| Q8BGM0 | FGR | WTAPEAALFGR | 0,319 | 24766 | 6% |
| P27601 | Ga 13 | GIHEYDFEIK | 0,843 | 68467 | 15% |
| P27601 | Ga 13 | VFLQYLPAIR | 1.295 | 106176 | 23% |
| P08752 | Ga (i) 2 | IAQSDYIPTQQDVLR | 10.900 | 885701 | 2% |
| P08752 | Ga (i) 2 | YDEAASYIQSK | 9.774 | 790616 | 9% |
| Q9DC51 | Ga (k) | EYQLNDSASYYLNDLDR | 6.574 | 537862 | 15% |
| Q9DC51 | Ga (k) | ISQTNYIPTQQDVLR | 3.504 | 285784 | 10% |
| P62874 | Gß 1 | AGVLAGHDNR | 17,078 | 1385578 | 4% |
| Q9CXP8 | Gy 10 | DALLLGVPAGSNPFR | 2.167 | 179178 | 36% |
| P63213 | 2 Gy | EDPLLTPVPASENPFR | 3.284 | 266360 | 8% |
| Q80SZ7 | Gy 5 | VSQAAADLK | 6,087 | 493512 | 6% |
| P08103 | HCK | GPVYVPDPTSSSK | 1.143 | 93044 | 12% |
| P08103 | HCK | IIEDNEYTAR | 0,944 | 77147 | 19% |
| P43406 | Intégrine α V | AGTQLLAGLR | 0,276 | 22264 | 32% |
| P43406 | Intégrine α V | SHQWFGASVR | 0,443 | 34235 | 5% |
| P25911 | LYN | VIEDNEYTAR | 1.461 | 119377 | 13% |
| Q5SW28 | 5 PI3K réglementaire | AGFPGILDTASPGK | 0,301 | 24331 | 11% |
| Q8K3B3 | PI3K α réglementaire | LYEEYTR | 0,472 | 38592 | 16% |
| Q8K3B3 | PI3K α réglementaire | TWNVGSSNR | 0,524 | 42697 | 12% |
| Q8K3B3 | PI3K α réglementaire | VLSEIFSPVLFR | 0,440 | 35902 | 20% |
| Q5U3K7 | PI3K β réglementaire | DTPDGTFLVR | 0,188 | 15262 | 30% |
| Q5U3K7 | PI3K β réglementaire | IAEIHESR | 0,282 | 22561 | 13% |
| Q0VGQ5 | PI3K α | LINLTDILK | 0,102 | 8494 | 38% |
| Q8CI98 | PI3K δ | HEVQEHFPEALAR | 0,178 | 14566 | 22% |
| Q8CI98 | PI3K δ | ITEEEQLQLR | 0,481 | 38709 | 24% |
| Q9ES52 | PIP3 5-phosphatase 1 | IVVLAKPEHENR | 0,486 | 39194 | 19% |
| Q9ES52 | PIP3 5-phosphatase 1 | LSQLTSLLSSIEDK | 2.056 | 167123 | 7% |
| Q69ZK0 | PIP3 dépendant Rac FEM 1 | DSVLSYTSVR | 0,647 | 52712 | 32% |
| Q69ZK0 | PIP3 dépendant Rac FEM 1 | NQLLLALLK | 0,354 | 27425 | 5% |
| Q9CQE5 | RGS 10 | ASSQVNVEGQSR | 2.460 | 199782 | 4% |
| Q9CQE5 | RGS 10 | WASSLENLLEDPEGVQR | 2.647 | 206005 | 11% |
| Q9CX84 | RGS 19 | AEANQHVVDEK | 0,495 | 39753 | 21% |
| Q9CX84 | RGS 19 | LIYEDYVSILSPK | 0,846 | 68481 | 22% |
| B9EKC3 | Rho GAP 5 | DGLAQELANEIR | 0,448 | 34653 | 10% |
| Q99PT1 | Rho GDI 1 | SIQEIQELDK | 3.156 | 267063 | 16% |
| Q99PT1 | Rho GDI 1 | VAVSADPNVPNVIVTR | 37,077 | 3006168 | 5% |
| Q61599 | Rho GDI 2 | LNYKPPPQK | 37,975 | 3086596 | 3% |
| Q61599 | Rho GDI 2 | YVQHTYR | 21,436 | 1711908 | 47% |
| Q61210 | Rho FEM 1 | FDGAEGSWFQK | 2.149 | 176139 | 47% |
| Q61210 | Rho FEM 1 | SGLELEPEEPPGWR | 2.911 | 236483 | 8% |
| Q9QUI0 | RhoA | QVELALWDTAGQEDYDR | 43.500 | 3532438 | 8% |
| P70336 | ROCK2 | GAFGEVQLVR | 0,527 | 42800 | 23% |
| P70336 | ROCK2 | IYESIEEAK | 1.011 | 83794 | 33% |
| P70336 | ROCK2 | LEGWLSLPVR | 0,573 | 48259 | 22% |
| Q8R0X7 | S1P lyase 1 | AGYPLEKPFDFR | 1.787 | 147043 | 27% |
| Q8R0X7 | S1P lyase 1 | TPEIVAPESAHAAFDK | 3.562 | 291791 | 18% |
Tableau 1: 264,7 protéines cellulaires quantitatives LC-SRM de RAW Trente-cinq 264.7 protéines cellulaires RAW ont été quantifiés en utilisant cinquante-huit normes peptidiques internes et six réplicats biologiques.. Cinq des protéines cibles sont d'ordre administratif protéines (actine, la GAPDH, la lactate déshydrogénase, la tubuline, et la vimentine), et ont été quantifiés pour permettre une normalisation à travers des échantillons biologiques (étape 1.1). En outre, un niveau de protéine interne a été enrichis-dans chaque lysat cellulaire et quantifié par LC-SRM (4,765 pmol de la luciférase de luciole par 200 ug échantillon; pur à 98% par SDS-PAGE; quantifiée par spectrophotométrie à 280 nm; l'étape 4.11). Les valeurs de CV ont été calculés pour les six réplicats biologiques en utilisant les valeurs d'abondance normalisés à l'échelle mondiale (à l'exception de la luciférase; Étape 5.15).
Discussion
Absolute quantification de protéine est essentielle pour une gamme très diversifiée d'applications biomédicales telles que la validation de biomarqueurs et la transduction du signal de modélisation de la voie. Récemment, la protéomique ciblées utilisant LC-SRM a bénéficié des améliorations apportées à de nombreuses technologies, y compris peptide préparation standard, HPLC, QQQ-MS et LC-SRM analyse des données. Par conséquent, il est devenu une alternative puissante aux immunoessais. Immunoessais peuvent être extrêmement sensibles et à haut débit, mais de développer un dosage immunologique robuste peut être extrêmement difficile parce immunoessais peuvent être vulnérables à la réactivité croisée et / ou des interférences, incompatible avec cellules / tissus méthodes de lyse / d'homogénéisation et / ou ne se prêtent pas au multiplexage 5,8. Par exemple, le test le plus complet pour la réactivité croisée est d'effectuer le dosage immunologique en utilisant des échantillons qui proviennent de KO de gènes, ce qui peut être difficile à préparer.
Ce protocole décrit cible Peptsélection de ide, le développement de tests LC-SRM, qualitative et quantitative LC-SRM, et LC-SRM analyse des données. Il a été utilisé pour mesurer l'abondance absolue de protéines de la voie 36 de chimiotactisme dans les cellules RAW 264.7 56, mais son applicabilité dépasse de loin cette application spécifique. Bien qu'il ait été conçu pour quantifier les protéines au sein de culots cellulaires, il pourrait être ajusté pour l'analyse d'autres échantillons biologiques (par exemple des fluides biologiques) et d'autres objectifs de SRM phosphopeptides (par exemple). Par exemple, en changeant le protocole d'homogénéisation et / ou la digestion des protéines peut améliorer significativement la solubilisation, la dénaturation, l'alkylation, la digestion et la quantification des protéines cibles particulièrement difficiles (par exemple, des protéines de membrane), ou peut permettre l'analyse des échantillons particulièrement difficiles (par exemple, des échantillons contenant <100 ug masse protéique).
La sélection initiale de peptides cibles est critique, mais peut prendre beaucoup de temps. Pour hundreds de protéines cibles, un score ad hoc pourraient être utilisés pour chacun des critères, et les peptides cibles indicatives peuvent être classés en fonction de la somme des scores, comme cela a été fait précédemment 56. Alternativement, cette analyse peut être automatisé en utilisant PeptidePicker, une interface web qui simplifie grandement la sélection cible de peptide 30 (http://mrmpeptidepicker.proteincentre.com/).
Après les peptides cibles ont été sélectionnés et les analyses LC-SRM ont été développés, il est essentiel que les analyses qualitatives LC-MRS des échantillons biologiques seront effectués parce que même un peptide hautement protéotypique et quantotypic ne sera pas détectable si la protéine est exprimée à des niveaux inférieurs au seuil de sensibilité de l'instrument, ou si l'interférence de fond est particulièrement problématique. Une deuxième dimension de séparation (par exemple, forte HPLC d'échange de cations, pH élevé HPLC en phase inverse, et sans gel d'électrophorèse) peut augmenter la profondeur protéomique, mais il faudrabeaucoup plus de temps de l'instrument et l'analyse des données. Alternativement, une stratégie d'enrichissement (par exemple, immunoenrichment peptide et niveau de protéines et de fractionnement cellulaire) peut améliorer la profondeur protéomique, et immunodéplétion des protéines très abondantes peut être utilisé pour réduire les interférences par coélution analytes.
LC-MRS des dizaines de ~ ~ échantillons pour des centaines ou des milliers de ~ transitions nécessite généralement beaucoup de temps de l'instrument. Bien qu'il n'a pas été utilisé pour cette étude, LC-SRM planification (mesure transitions pendant les fenêtres pré-spécifiée élution temps) permet l'analyse de plus de transitions par cycle. En outre, la programmation permet de réduire le cycle de service des MRS pendant des périodes relativement vacants du gradient de LC, ce qui peut entraîner une amélioration de l'identification et la quantification des peptides. Cependant, la programmation nécessite que le temps d'élution du peptide est déterminée à partir de confiance à une analyse LC-SRM qualitative. Des changements subtils à la préparation de l'échantillon, l'instrument LC-MS,ou la méthode LC-MS peut causer ordonnancement pour recadrer ou même entièrement manquez peptides cibles. Par exemple, les profils biologiques d'élution de l'échantillon YDEAASYIQSK sont légèrement décalés par rapport à ceux de type peptidique externe (figure 4B), probablement en raison des effets de matrice.
En résumé, un protocole étape par étape est présentée pour le développement et l'application de LC-SRM pour la quantification absolue de protéines. Quantification absolue de protéines par LC-SRM a déjà été démontrée pour être reproductible entre les laboratoires 16,19. technologies de protéomique, y compris la préparation des échantillons (par exemple, l'automatisation), la chromatographie liquide, spectrométrie de masse, et l'analyse de données, améliorent rapidement, et consiste à permettre à LC-SRM pour devenir pratique pour la recherche à grande échelle et les applications cliniques. La spécificité, la sensibilité, la précision, reproductible, et à haut débit de quantitative LC-SRM, il est un outil puissant pour la recherche fondamentale et de la biomédecine.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
This research was supported by the Intramural Research Program of the NIH, National Institute of Allergy and Infectious Diseases.
matériels
| Name | Company | Catalog Number | Comments |
| Acetonitrile (ACN), LC-MS grade | Fisher | A955-1 | |
| BCA (bicinchoninic acid) protein assay kit | Fisher | 23235 | |
| Beads for bead beating, zirconia-silica, 0.1 mm | BioSpec Products | 11079101z | |
| Bestatin hydrochloride | Sigma | B8385-10MG | |
| Cell culture DMEM (with glucose, without L-glutamine) | Lonza | 12-614F | |
| Cell culture EDTA, 500 mM, pH8 | Gibco | 15575 | |
| Cell culture fetal bovine serum (FBS) | Atlanta Biologicals | S11550 | |
| Cell culture L-glutamine | Sigma | G8540-25G | |
| Cell culture phosphate buffered saline (PBS) pH 7.4 | Gibco | 10010-049 | |
| Cell culture Trypan Blue viability stain, 0.4% w/v | Lonza | 17-942E | |
| Cellometer Auto T4 cell counter | Nexcelom Bioscience | Cellometer Auto T4 | |
| Cellometer Auto T4 disposable counting chambers | Nexcelom Bioscience | CHT4-SD100-014 | |
| Dithiothreitol (DTT) | Sigma | D5545-5G | |
| Formic acid, LC-MS grade, ampules | Fisher | A117-10X1AMP | |
| Hemocytometer, Neubauer-improved, 0.1 mm deep | Marienfeld-Superior | 0640030 | |
| HEPES, 1 M, pH 7.2 | Mediatech | 25-060-CI | |
| Hydrochloric acid, 37% w/w | VWR | BDH3028-2.5LG | |
| Iodoacetamide | Sigma | I1149-5G | |
| Laser Based Micropipette Puller | Sutter Instrument Co. | P-2000 | |
| LC coated silica capillary, 50 µm id | Polymicro Technologies | 1068150017 | |
| LC vial, autosampler, 12 mm x 32 mm polypropylene | SUN SRI | 200-268 | |
| LC vial screw cap, autosampler, pre-slit PTFE/silicone | SUN SRI | 500-061 | |
| Luciferase, from Photinus pyralis | Sigma | L9506-1MG | |
| Pepstatin A | EMD Millipore | 516481-25MG | |
| pH strips colorpHast (pH 0.0-6.0) | EMD Chemicals | 9586-1 | |
| PhosStop phosphatase inhibitor cocktail | Roche | 04906837001 | |
| RapiGest SF | Waters | 186001861 | |
| Sep-Pak SPE, C18 1 ml 100 mg cartridge | Waters | WAT023590 | |
| Sep-Pak SPE, extraction manifold, 20 position | Waters | WAT200609 | |
| Sep-Pak SPE, flat-surfaced rubber bulb | Fisher | 03-448-25 | |
| Sodium hydroxide (NaOH) | Fisher | S318-500 | |
| SpeedVac vacuum concentrator | Fisher | SPD111V | |
| Trifluoroacetic acid (TFA), LC-MS grade | Fisher | A116-50 | |
| Trypsin, sequencing grade, modified | Promega | V5113 | |
| Tube decapper for Micronic tubes | USA Scientific | 1765-4000 | |
| Tubes, 2 ml microcentrifuge, o-ring screw-cap, sterile | Sarstedt | 72.694.006 | |
| Urea | Sigma | U0631-500g | |
| Water, LC-MS grade | Fisher | W6-1 |
Références
- Cox, J., Mann, M. Quantitative high-resolution proteomics for data-driven systems biology. Annu Rev Biochem. 80, 273-299 (2011).
- Zhang, Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chem Rev. 113, 2343-2394 (2013).
- Boja, E. S., Rodriguez, H. Mass spectrometry-based targeted quantitative proteomics: achieving sensitive and reproducible detection of proteins. Proteomics. 12, 1093-1110 (2012).
- Gillette, M. A., Carr, S. A. Quantitative analysis of peptides and proteins in biomedicine by targeted mass spectrometry. Nat Methods. 10, 28-34 (2013).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nat Methods. 9, 555-566 (2012).
- Lesur, A., Domon, B. Advances in high-resolution accurate mass spectrometry application to targeted proteomics. Proteomics. , (2015).
- Wild, D. . The immunoassay handbook : theory and applications of ligand binding ELISA., and related techniques. , (2013).
- Sturgeon, C. M., Viljoen, A. Analytical error and interference in immunoassay: minimizing risk. Ann Clin Biochem. 48, 418-432 (2011).
- Adrait, A., et al. Development of a Protein Standard Absolute Quantification (PSAQ) assay for the quantification of Staphylococcus aureus enterotoxin A in serum. J Proteomics. 75, 3041-3049 (2012).
- Lin, D., Alborn, W. E., Slebos, R. J., Liebler, D. C. Comparison of protein immunoprecipitation-multiple reaction monitoring with ELISA for assay of biomarker candidates in plasma. J Proteome Res. 12, 5996-6003 (2013).
- Weiss, F., et al. Catch and measure-mass spectrometry-based immunoassays in biomarker research. Biochim Biophys Acta. 1844, 927-932 (2014).
- Yassine, H., et al. Mass spectrometric immunoassay and MRM as targeted MS-based quantitative approaches in biomarker development: potential applications to cardiovascular disease and diabetes. Proteomics Clin Appl. 7, 528-540 (2013).
- Zhao, L., et al. Quantification of proteins using peptide immunoaffinity enrichment coupled with mass spectrometry. J Vis Exp. , (2011).
- Becker, J. O., Hoofnagle, A. N. Replacing immunoassays with tryptic digestion-peptide immunoaffinity enrichment and LC-MS/MS. 4, 281-290 (2012).
- Wasinger, V. C., Zeng, M., Yau, Y. Current status and advances in quantitative proteomic mass spectrometry. Int J Proteomics. 2013, 180605 (2013).
- Abbatiello, S. E., et al. Large-scale inter-laboratory study to develop, analytically validate and apply highly multiplexed, quantitative peptide assays to measure cancer-relevant proteins in plasma. Mol Cell Proteomics. , (2015).
- Rodriguez-Suarez, E., Whetton, A. D. The application of quantification techniques in proteomics for biomedical research. Mass Spectrom Rev. 32, 1-26 (2013).
- Wehr, A. Y., Hwang, W. T., Blair, I. A., Yu, K. H. Relative quantification of serum proteins from pancreatic ductal adenocarcinoma patients by stable isotope dilution liquid chromatography-mass spectrometry. J Proteome Res. 11, 1749-1758 (2012).
- Kennedy, J. J., et al. Demonstrating the feasibility of large-scale development of standardized assays to quantify human proteins. Nat Methods. 11, 149-155 (2014).
- Jensen, K. J., Shelton, P. T., Pedersen, S. L. . Peptide synthesis and applications. , (2013).
- Pratt, J. M., et al. Multiplexed absolute quantification for proteomics using concatenated signature peptides encoded by QconCAT genes. Nat Protoc. 1, 1029-1043 (2006).
- Brun, V., et al. Isotope-labeled protein standards: toward absolute quantitative proteomics. Mol Cell Proteomics. 6, 2139-2149 (2007).
- . . Guidance for Industry: Bioanalytical Method Validation. , (2001).
- . . Guidance for Industry: Bioanalytical Method Validation. , (2013).
- Carr, S. A., et al. Targeted peptide measurements in biology and medicine: best practices for mass spectrometry-based assay development using a fit-for-purpose approach. Mol Cell Proteomics. 13, 907-917 (2014).
- Ori, A., Andres-Pons, A., Beck, M. The use of targeted proteomics to determine the stoichiometry of large macromolecular assemblies. Methods Cell Biol. 122, 117-146 (2014).
- Rosen, O., Feldberg, L., Gura, S., Zichel, R. A new peptide substrate for enhanced botulinum neurotoxin type B detection by endopeptidase-liquid chromatography-tandem mass spectrometry/multiple reaction monitoring assay. Anal Biochem. , (2015).
- Feng, Y., et al. Global analysis of protein structural changes in complex proteomes. Nat Biotechnol. 32, 1036-1044 (2014).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26, 966-968 (2010).
- Mohammed, Y., et al. PeptidePicker: a scientific workflow with web interface for selecting appropriate peptides for targeted proteomics experiments. J Proteomics. 106, 151-161 (2014).
- Rodriguez, J., Gupta, N., Smith, R. D., Pevzner, P. A. Does trypsin cut before proline. J Proteome Res. 7, 300-305 (2008).
- Min, X. J., Butler, G., Storms, R., Tsang, A. OrfPredictor: predicting protein-coding regions in EST-derived sequences. Nucleic Acids Res. 33, W677-W680 (2005).
- Lam, H., et al. Building consensus spectral libraries for peptide identification in proteomics. Nat Methods. 5, 873-875 (2008).
- Craig, R., Cortens, J. P., Beavis, R. C. Open source system for analyzing, validating, and storing protein identification data. J Proteome Res. 3, 1234-1242 (2004).
- Desiere, F., et al. The PeptideAtlas project. Nucleic Acids Res. 34, D655-D658 (2006).
- Vizcaino, J. A., et al. The PRoteomics IDEntifications (PRIDE) database and associated tools: status in 2013. Nucleic Acids Res. 41, D1063-D1069 (2013).
- Frank, R. The SPOT-synthesis technique. Synthetic peptide arrays on membrane supports--principles and applications. J Immunol Methods. 267, 13-26 (2002).
- Ong, S. E., Kratchmarova, I., Mann, M. Properties of 13C-substituted arginine in stable isotope labeling by amino acids in cell culture (SILAC). J Proteome Res. 2, 173-181 (2003).
- Mant, C. T., et al. HPLC analysis and purification of peptides. Methods Mol Biol. 386, 3-55 (2007).
- Alterman, M. A., Hunziker, P. . Amino acid analysis : methods and protocols. , (2012).
- Maclean, B., et al. Effect of collision energy optimization on the measurement of peptides by selected reaction monitoring (SRM) mass spectrometry. Anal Chem. 82, 10116-10124 (2010).
- Nesvizhskii, A. I. A survey of computational methods and error rate estimation procedures for peptide and protein identification in shotgun proteomics. J Proteomics. 73, 2092-2123 (2010).
- Tabb, D. L., Friedman, D. B., Ham, A. J. Verification of automated peptide identifications from proteomic tandem mass spectra. Nat Protoc. 1, 2213-2222 (2006).
- Freshney, R. I. . Culture of animal cells : a manual of basic technique and specialized applications. , (2010).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. J Proteome Res. 8, 2144-2156 (2009).
- Noble, J. E., Bailey, M. J. Quantitation of protein. Methods Enzymol. 463, 73-95 (2009).
- Kiser, J. Z., Post, M., Wang, B., Miyagi, M. Streptomyces erythraeus trypsin for proteomics applications. J Proteome Res. 8, 1810-1817 (2009).
- Reiter, L., et al. mProphet: automated data processing and statistical validation for large-scale SRM experiments. Nat Methods. 8, 430-435 (2011).
- Abbatiello, S. E., Mani, D. R., Keshishian, H., Carr, S. A. Automated detection of inaccurate and imprecise transitions in peptide quantification by multiple reaction monitoring mass spectrometry. Clin Chem. 56, 291-305 (2010).
- Callister, S. J., et al. Normalization approaches for removing systematic biases associated with mass spectrometry and label-free proteomics. J Proteome Res. 5, 277-286 (2006).
- Karpievitch, Y. V., Dabney, A. R., Smith, R. D. Normalization and missing value imputation for label-free LC-MS analysis. BMC Bioinformatics. 13, S5 (2012).
- Oh, S., Kang, D. D., Brock, G. N., Tseng, G. C. Biological impact of missing-value imputation on downstream analyses of gene expression profiles. Bioinformatics. 27, 78-86 (2011).
- Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A., Fraser, I. D. Systems biology in immunology: a computational modeling perspective. Annu Rev Immunol. 29, 527-585 (2011).
- Futran, A. S., Link, A. J., Seger, R., Shvartsman, S. Y. ERK as a model for systems biology of enzyme kinetics in cells. Curr Biol. 23, R972-R979 (2013).
- Krokhin, O. V. Sequence-specific retention calculator. Algorithm for peptide retention prediction in ion-pair RP-HPLC: application to 300- and 100-A pore size C18 sorbents. Anal Chem. 78, 7785-7795 (2006).
- Manes, N. P., Angermann, B. R., Koppenol-Raab, M., An, E., Sjoelund, V. H., Sun, J., Ishii, M., Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A. Targeted Proteomics-Driven Computational Modeling of Macrophage S1P Chemosensing. . Mol Cell Proteomics. , .
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon