Method Article
Reacción seleccionada Monitoreo espectrometría de masas para Absolute Proteína cuantificación
En este artículo
Resumen
This protocol describes how to perform absolute quantification assays of target proteins within complex biological samples using selected reaction monitoring. It was used to accurately quantify proteins of the mouse macrophage chemotaxis signaling pathway. Target peptide selection, assay development, and qualitative and quantitative assays are described in detail.
Resumen
Absolute quantification of target proteins within complex biological samples is critical to a wide range of research and clinical applications. This protocol provides step-by-step instructions for the development and application of quantitative assays using selected reaction monitoring (SRM) mass spectrometry (MS). First, likely quantotypic target peptides are identified based on numerous criteria. This includes identifying proteotypic peptides, avoiding sites of posttranslational modification, and analyzing the uniqueness of the target peptide to the target protein. Next, crude external peptide standards are synthesized and used to develop SRM assays, and the resulting assays are used to perform qualitative analyses of the biological samples. Finally, purified, quantified, heavy isotope labeled internal peptide standards are prepared and used to perform isotope dilution series SRM assays. Analysis of all of the resulting MS data is presented. This protocol was used to accurately assay the absolute abundance of proteins of the chemotaxis signaling pathway within RAW 264.7 cells (a mouse monocyte/macrophage cell line). The quantification of Gi2 (a heterotrimeric G-protein α-subunit) is described in detail.
Introducción
Experimentos proteómicos que utilizan espectrometría de masas (MS) se pueden diseñar para utilizar ya sea no específica (shotgun) o métodos específicos. Proteómica Descubrimiento generalmente se basa en la escopeta de abajo hacia arriba MS, ya sea mediante el uso de un modo de adquisición de datos dependiente tradicional, o utilizando una de las técnicas de datos independiente de reciente desarrollo (por ejemplo, MS E, SWATH) 1,2. Proteómica de escopeta es una poderosa herramienta para la identificación de péptidos de alto rendimiento y la cuantificación relativa, pero es generalmente inadecuado para la cuantificación absoluta o para dirigir pequeños conjuntos, definidos (~ decenas) de las proteínas. El método MS más utilizado para la proteómica dirigidos se selecciona reacción monitoreo (SRM) debido a su alta sensibilidad, velocidad y rango dinámico 3-5. Alternativas a SRM incluyen el monitoreo de reacción en paralelo, que se aprovecha de alta resolución, escaneo de MS total de 6.
SRM se realiza generalmente mediante un rever nano-flujocromatografía SED-fase líquida de alto rendimiento (nano-RP-LC) acoplada a un instrumento de ionización por electrospray nano-(nano-ESI) fuente de iones unido a un espectrómetro de masas de triple cuadrupolo (QqQ-MS). En un experimento típico, las proteínas de la muestra se digiere proteolíticamente, y los péptidos resultantes se separaron cromatográficamente, desorbidos, y ionizados. Los iones precursores resultantes son m / z filtrada por el primer cuadrupolo (Q1) y fragmentado en el segundo cuadrupolo (q2) al chocar con un gas de colisión. Los iones de fragmentos resultantes son m / z-filtrada en la tercera cuadrupolo (Q3) y se cuantifica por un dynode. Cada par precursor y el ion fragmento se conoce como una transición, y cada transición se controla por un período determinado de tiempo (el tiempo de permanencia; típicamente 2-50 ms). Durante LC-SRM, los ciclos QqQ-MS a través de una lista predefinida de transiciones (el ciclo de trabajo es típicamente ≤3 seg), y se produce un cromatograma de cada transición.
Strateg Alternativas para la cuantificación de proteínas suelen utilizar inmunoensayos tales como transferencias de puntos, transferencias de Western, ELISA, microarrays de anticuerpos, microarrays de proteínas de fase inversa, inmunoensayos de microfluidos, ELISA digitales, e inmunoensayos basados en microesferas 7. Los mejores inmunoensayos pueden ser significativamente más sensible que la LC-SRM, y rendimiento de la muestra de inmunoensayos pueden ser significativamente mayor que la de LC-SRM 5. Sin embargo, los inmunoensayos en desarrollo pueden ser costosos y / o requiere mucho tiempo, y los ensayos resultantes pueden ser vulnerables a la reactividad cruzada y / o interferencia, incompatible con la célula / tejido de lisis / métodos de homogeneización, y / o no susceptible de multiplexación 5,8. Algunos de estos problemas pueden abordarse mediante el acoplamiento de técnicas en anticuerpos y basados en MS. Por ejemplo, las proteínas diana pueden enriquecerse utilizando inmunoprecipitación antes de la proteólisis y LC-SRM 9-12. Alternativamente, la técnica emplea SISCAPA inmunoprecipitación posterior a la proteólisis en el péptido level 13,14. Además de immunoenrichment estrategias, immunodepletion de las proteínas de alta abundancia puede ser empleado para aumentar la sensibilidad LC-SRM mediante la reducción de la interferencia por coeluyentes analitos 15,16.
La cuantificación de proteínas basado en MS se puede dividir en la cuantificación relativa y absoluta, y también en el etiquetado de isótopos de etiqueta libre y estable (por ejemplo, etiquetado metabólico, etiquetado química, y de la proteína marcada con pesado y de péptidos estándares internos). Técnicas de la etiqueta libre pueden ser útiles para la cuantificación de proteínas relativo, pero no son adecuados para la cuantificación absoluta precisión. En comparación, las técnicas de etiquetado han reducido de error asociado con la preparación de muestras y MS varianza, y se utilizan a menudo para la cuantificación relativa de proteínas 17. Por ejemplo, isótopo estable marcado proteoma normas (SILAP) preparado utilizando una línea celular humana se cultivaron permitido la cuantificación relativa de biomarcadores potenciales a través de LC-SRM de suero humano 18. Precisa cuantificación de proteínas absoluta por la EM requiere que las normas internas, cuantificado, proteína o péptido marcado isotópicamente purificada y se enriquecerán-en muestras biológicas antes de la EM. La incorporación de isótopo pesado estándares internos marcados en un flujo de trabajo LC-SRM permite la cuantificación absoluta que se ha demostrado ser altamente reproducible y transferible entre los laboratorios 16,19.
Isótopos estables etiquetados normas internas para la cuantificación absoluta de proteínas por MS incluyen normas de péptidos preparados mediante síntesis en fase sólida 20, proteínas compuestas de normas péptido proteasa escindible concatenados 21, y las normas de la proteína de longitud completa 22. Target modificación covalente de proteínas y preparación de la muestra incompleta (es decir, la lisis incompleta de la muestra y de la homogeneización, y la solubilización incompleta de proteínas, la desnaturalización, la alquilación, y la proteolisis) puede socavar la cuantificación exacta. P Internanormas rotein son los menos propensos a ser afectados por la mayor parte de estos problemas potenciales, pero por lo general son los más difíciles de preparar. Una alternativa es analizar cada proteína diana utilizando múltiples estándares de péptidos internos que están diseñados para incluir residuos flanqueantes nativas amino- y carboxi-terminal. Independientemente de que se emplea el tipo de patrón interno, se debe claveteado-en las muestras biológicas en un punto temprano como durante la preparación de la muestra como sea posible. Además, las técnicas de preparación de muestras múltiples (por ejemplo, diferentes condiciones de desnaturalización) deben ser probados. El uso de múltiples técnicas experimentales ortogonales (experimental de validación cruzada) es una estrategia viable para superar la mayor cuantificación potencial desafía 23-25.
LC-SRM cuantificación de proteínas es una técnica altamente flexible que se ha utilizado en una amplia variedad de aplicaciones. En particular, se ha utilizado para estudiar péptidos y proteínas dentro de biomarcadoresmuestras clínicas tales como suero, biopsias de núcleo, y aspirados con aguja fina 5. LC-SRM también se ha utilizado para medir la estequiometría de los complejos de proteínas 5,26, para detectar neurotoxinas botulínicas 27, para cuantificar la dinámica de fosforilación de proteínas dentro de las vías de señalización 5, y para cuantificar los cambios en la conformación de la proteína 28.
Nuestro laboratorio está utilizando LC-SRM para cuantificar las proteínas de señalización que median la quimiotaxis de macrófagos para apoyar el desarrollo de simulaciones vía quimiotaxis. El esquema general del protocolo (Figura 1) comienza con la clasificación de los péptidos diana tentativas. Posteriormente, los estándares de péptidos externos crudo se sintetizan y se utilizan para desarrollar ensayos LC-SRM para los análisis cualitativos de muestras biológicas. Si se detecta el péptido diana derivado de muestra biológica, los estándares de péptido interno marcado con pesados purificados se preparan para cuantitativo LC-SRM. Este protocolo se puede utilizar para accurately cuantificar proteínas a partir de una amplia variedad de muestras biológicas, y para apoyar las investigaciones de una amplia variedad de dianas proteicas.
Protocolo
NOTA: Este método ha sido descrito previamente 56.
1. Péptido selección de objetivos
- Compilar una lista de las proteínas diana, e incluyen un pequeño número de proteínas de limpieza para la normalización a través de muestras biológicas, y también incluyen un nivel de proteína interna (por ejemplo, luciferasa de luciérnaga). Digerir las proteínas diana en péptidos trípticos in silico usando una herramienta de software, tales como la proteína de digestión Simulador 29,30.
- Exigir que los péptidos son plenamente tríptico y no contienen los sitios de escisión de tripsina que faltan. Evitar péptidos con sitios de escisión de tripsina vecinos para evitar potencialmente incompleta digestión con tripsina 31.
- Requerir que la longitud de cada péptido es de 5-20 residuos. Considere péptidos más largos, pero en cuenta que generalmente son más caros de sintetizar.
- Evitar un péptido si corresponde a una variante genética natural (por ejemplo, un solo nucleótido-e polimorfismo). También confirman que los sitios de tripsina no se ven afectados (Paso 1.2).
- Evitar un péptido si corresponde a un sitio de la modificación postraduccional (PTM). Del mismo modo, evitar un péptido si sería propenso a la modificación química durante la preparación de la muestra LC-MS. Específicamente, evitar péptidos propensas a la cisteína y la oxidación de metionina, asparagina y la desamidación de glutamina, y la formación de glutamina amino-terminal de piroglutamato. Confirme que los sitios de tripsina se ven afectados (Paso 1.2).
- Además, evitar péptidos que se originan de proteína amino- y carboxi-terminales porque termini de proteínas son propensas a la modificación postraduccional (pérdida de metionina amino-terminal y la acetilación y la amidación carboxi-terminal).
- Requieren que cada péptido diana es única para cada proteína diana. Si esto no es posible, requerir que el péptido es único para un conjunto de isoformas / homólogos. Tratar péptidos difieren sólo por leucina / isoleucina sustitucióncomo si fueran idénticos al determinar singularidad péptido debido a que estos realizan casi idéntica durante LC-SRM.
- Si un análisis transcriptómica completa de todas las muestras biológicas se ha realizado (por ejemplo, RNA-Seq), determinar péptido singularidad usando en traducciones in silico de la transcripción de secuencias de 32 en vez de usar todo el proteoma de la especie.
- Exigir que los péptidos diana son proteotípicos. Identificar péptidos proteotípicos utilizando espectrometría de masas escopeta 1,2 de las muestras biológicas se está estudiando. Alternativamente, recuperar péptido identificaciones de péptidos proteotípicos de proteómica en línea de bases de datos tales como la espectrometría de masas NIST bibliotecas 33 (http://peptide.nist.gov/), El Proteoma Máquina Global 34 (X HUNTER bibliotecas espectrales de http: // www .thegpm.org / HUNTER / index.html y GPMDB péptido identificaciones de http://gpmdb.thegpm.org/), PeptideAtlas 35 (http://www.peptideatlas.org/), y la base de datos PRIDE 36 (http://www.ebi.ac.uk/pride/archive/).
- Si cuantitativa-nivel de transcripción análisis de las muestras biológicas se han realizado (por ejemplo, ARN-ss, qPCR), entonces evitar la orientación péptidos que corresponden a las transcripciones relativamente baja abundancia.
- Si es necesario, seleccione péptidos diana que podrían ser utilizados para los ensayos de LC-SRM de ortólogos proteínas diana (por ejemplo, para analizar tanto las formas del ratón de una proteína diana humana y).
- Seleccione al menos dos péptidos diana por proteína diana (si es posible).
2. Preparación del péptido Normas
NOTA: Esta sección del protocolo describe la preparación de un conjunto de veinte estándares de péptidos liofilizados (siendo cada 1 nmol en cantidad) para análisis posteriores. Para un número diferente de péptidos, o para diferentes cantidades de péptidos, tendrá que ajustarse en consecuencia.
- Preparar estándares liofilizados péptidos (≥1 nmol) utilizando una tecnología de síntesis de péptidos tales como la síntesis SPOT 20,37.
- Asegúrese de que los residuos de cisteína de las normas de péptidos externos están carbamidometilado (la estructura química formada por alquilación yodoacetamida). Por el contrario, garantizar que los residuos de cisteína de las normas de péptidos internos son sin modificar (estos serán alquilados durante la preparación de la muestra, que se describe a continuación).
- Estándares de Diseño de péptidos internos de manera que contienen nativo amino- y carboxi-terminal de residuos flanqueantes (para controlar la eficiencia de la digestión con tripsina; cada uno es típicamente cinco y cincuenta y siete residuos de largo).
- Estándares de Diseño de péptidos internos de manera que sean isótopo estable marcado. Considere el perfil isotópica natural del péptido diana y la precisión de la medición de masa de la MS cuando se selecciona una estrategia de etiquetado péptido. No utilice estándares de péptidos deuterados porque causas deuteración péptido de fase inversa tiempo de retención LCs para cambiar 38.
Nota: Por lo general, las normas internas de péptidos trípticos se sintetizan utilizando ~ 98% [13 C 6, 15 N 4] Arg y [13 C 6, 15 N 2] Lys incorporado en el extremo carboxi-terminal. - Purificar estándares de péptidos internos utilizando HPLC 39, y cuantificar con precisión los 40.
- Añadir 100 l de 0,1% v / v de ácido fórmico, 20% v / v de acetonitrilo (ACN) para cada estándar péptido liofilizado 1 nmol para producir una concentración de péptido de 10 micras (uso de la atención como el péptido liofilizado puede tener una muy baja densidad y se puede perder fácilmente). Vortex las muestras durante 2 min y baño de sonicar durante 5 min para asegurar que la disolución es completa péptido.
- Agrupar los péptidos disueltos, y se concentra la mezcla resultante en un concentrador de vacío a un volumen final de 80 l. Añadir 20 l de ACN para disolver cualquier péptido precipitado.
Nota: La muestra ahora contains 10 M de cada péptido y 20% v / v ACN. - Para los estándares de péptidos internos, preparar una serie de diluciones para cuantitativa LC-SRM:
- Preparar 100 l de una dilución 1 M: combinar 90 l de 20% v / v ACN y 10 l de la mezcla de péptido 10 mM.
- Preparar 100 l de una dilución de 100 nM: combinar 90 l de 20% v / v ACN y 10 l de la mezcla de péptido 1 mM.
- Para los estándares de péptidos externos, preparar 100 l de una dilución 1 M para LC-MS mediante la combinación de 90 l de 0,1% v / v de ácido fórmico y 10 l de la mezcla de péptido 10 mM.
Nota: La muestra ahora contiene 1 mM de cada péptido, 0,1% v / v de ácido fórmico, y 2% v / v ACN, y está listo para ser utilizado para el desarrollo del ensayo LC-SRM.
3. LC-SRM Ensayo Desarrollo
- Analizar las mezclas de los estándares de péptidos externos (aproximadamente 1-10 pmol de cada péptido por inyección) por escopeta MS utilizando un HPL nano-flowSistema C acoplado a un espectrómetro de masas de triple cuadrupolo (LC-MS-QqQ (/ MS)).
- Utilice un sistema LC-MS equipado con una columna capilar lleno de C-18 medios de comunicación (≤5 diámetro micras, ~ 200 Å poros, longitud ≥10 cm, id = 50-100 micras) y una punta nano-ESI (producido típicamente usando un láser-extractor). Asegúrese de que cada corrida LC-MS incluye un gradiente de ~ 60 min lineal (típicamente, 0-40% de Disolvente B), una etapa de regeneración de columna (~ 80% de Disolvente B), y una etapa de la columna re-equilibración (~ 0% de Disolvente B ) (disolvente A = 0,1% v / v de ácido fórmico en H 2 O, disolvente B = 0,1% v / v tasa de ácido fórmico en ACN, caudal = 200-800 nl / min, tensión de ESI = 1800 V, anchura aislamiento Q3 = 0,7 m / z, presión de argón q2 = 1,5 mTorr).
- Para cada exploración ión precursor, seleccionar dinámicamente la parte superior ~ 10 iones precursores más intensos de la espectrometría de masas en tándem (MS / MS). Ejecutar cada muestra por duplicado de modo que se utilizan dos rampas energía de colisión diferentes: uno diseñado para fragmentar de manera óptima 2 iones precursores, y el otropara 3 precursor iones 41.
- Realice la LC-QqQ-MS (/ MS) análisis utilizando el escaneo de iones precursores Q3 (Q1 escaneo ión precursor puede causar colas de los picos de iones precursores resultantes de las colisiones de argón de baja energía en Q2).
- Asegúrese de que el sistema LC-MS está funcionando adecuadamente mediante el análisis de muestras de control de calidad y técnicos repeticiones. Prevenir la muestra remanente utilizando columnas LC recién hechos y mediante la ejecución de múltiples espacios en blanco entre las muestras.
- Analizar los datos resultantes escopeta LC-QqQ-MS (MS / MS) utilizando la base de datos buscando en contra de las secuencias de los péptidos estándares externos 42. En su caso, desechar péptido identificaciones ambiguas utilizando las puntuaciones de identificación de péptidos de confianza (por ejemplo, valores de expectativas) o mediante el uso de modelos estadísticos 42. Independientemente, revisar manualmente todas las identificaciones peptídicas 43 para asegurar que todos ellos son sin ambigüedades.
- Utilice las escopeta MS péptido identificaciones para construct una biblioteca espectro utilizando un programa de software como Skyline.
- Prepare una lista de transición LC-SRM usando las 03.10 transiciones más intensos por ión precursor (2 y 3 iones precursores; 1 y 2 fragmentos de iones; y-, b- y a-iones que son ≥2 residuos de largo).
- Descartar una transición si los valores de m / z precursor y el fragmento de iones se superponen dentro de la precisión de la medición de la masa QqQ-MS (fragmentación ion precursor es a veces incompleta; recuerde considerar la monoisotopic y las formas naturales de isótopos pesados, así como el no marcado y formas marcado pesado).
- Del mismo modo, descarta una transición si el ion fragmento de m / z que se solapa de un ion fragmento diferente de la misma ion precursor.
- Utilice las listas resultantes de transición para realizar LC-SRM análisis de las mezclas de los estándares de péptidos externos (realizar la espectrometría de masas como se describe en el paso 3.1; Q1 y Q3 ancho de aislamiento = 0,7 m / z; tiempo de permanencia = 2-50 ms).
- Mamánually revisar los datos LC-SRM resultantes y elimine los ensayos de bajo rendimiento (el análisis de datos LC-SRM se describe en la Sección 5).
4. LC-SRM ensayos de muestras biológicas
- Preparar los platos de células cultivadas 44. Si varias condiciones experimentales se están comparando, bloquear y aleatoriamente las muestras para reducir los posibles sesgos sistemáticos 45.
- Para cada placa de células, aspirar el medio de cultivo celular que perder y suspender las células en un tampón libre de suero 44. Si es necesario, se lavan las células en un tampón libre de suero para eliminar cualquier proteína extracelular restante.
- Para cada muestra, contar el número de células viables y totales mediante el uso de una tinción de viabilidad (por ejemplo, azul de tripano) y un hemocitómetro o un contador de células automatizado 44.
- Sedimentar las células por centrifugación, y aspirar los sobrenadantes a los residuos 44 (un volumen típico sedimento celular es de ~ 30 l).
- añadir 400 l de tampón de lisis de urea o tensioactivo tampón de lisis a cada sedimento celular (100 mM HEPES ∙ NaOH pH 8, 10 mM de hidrocloruro de bestatina, 10 mu M pepstatina A, y, o bien 8 M urea o 0,1% w / v de tensioactivo (respectivamente ); recién preparada, el uso de un tensioactivo compatible con MS ácido-lábil como RapiGest SF o PPS Silent surfactante). Si el uso de otros inhibidores de la proteasa, asegúrese de que no inhiben la tripsina.
- Mezclar cada muestra utilizando pipeteo suave, y transferir a un tubo de cada 2 ml (con un tapón de rosca con una junta tórica) que contiene ~ 100 l de 0,1 mm de zirconia / perlas de sílice (tenga en cuenta que las perlas pueden causar tubos de tapa a presión a gotear ). Lyse las células por vórtex las muestras durante 5 minutos a velocidad máxima (este lisa las células mediante golpes de talón).
- Alternativamente, lisar las células utilizando un método mecánico diferente (por ejemplo, usando una prensa o de homogeneización francés consejos).
Nota: Evite centrifugación lisados de células ya que esto podría precipitar la proteína precipitada que podría otherwise ser tryptically digerido y detectado por LC-MS.
- Alternativamente, lisar las células utilizando un método mecánico diferente (por ejemplo, usando una prensa o de homogeneización francés consejos).
- Para las muestras desnaturalizadas tensioactivos, incubarlos a 90 ° C durante 10 min para ayudar a la homogeneización de la muestra y la desnaturalización de proteínas.
- Bath sonicar las muestras durante 10 min a temperatura ambiente para ayudar a la homogeneización y la desnaturalización de las proteínas.
Nota: El buffer de lisis lisados y se puede almacenar a -80 ° C (se necesitará el tampón de lisis para los pasos 4.9 y 4.13). - Realizar un ensayo de concentración de proteína 46 (por ejemplo, un ensayo de ácido bicinconínico) de los lisados (y del tampón de lisis como un experimento de control).
- Para los análisis cualitativos (es decir, no hay normas de péptidos internos serán de pinchos-en las muestras), pipetas 200 mg (masa de proteínas) de lisado de células en un tubo de microcentrífuga de 1,5 ml fresco. Para los análisis cuantitativos, preparar cuatro de estas muestras para una serie de dilución de isótopos estables.
- Para cada muestra, añadir un estándar de proteína interna (por ejemplo </ Em>, agregue ~ 5 pmol de ~ 98% de la luciferasa de luciérnaga puro).
- Para los análisis cuantitativos, añadir la serie de dilución de isótopos estables de la mezcla equimolar de los estándares de péptido interno (por ejemplo, 0, 0,2, 2, 20 pmol de cada péptido) para las muestras.
- En paralelo, se preparan muestras de control utilizando los estándares del péptido interno solo (es decir, sin lisado celular) (por ejemplo, 0, 0,2, 2, 20 pmol de cada péptido).
- También hay que preparar muestras de control utilizando los estándares de péptidos internos y externos (por ejemplo: 2 pmol de cada péptido estándar externo, y 0, 0,2, 2, y 20 pmol de cada péptido estándar interno).
Nota: Asegúrese de que las normas de péptidos son libres de ácido fórmico, ya que puede interferir con la digestión con tripsina.
- Añadir tampón de lisis de modo que todas las muestras son volúmenes idénticos. Nota: Un volumen de muestra típico es de 70 l, por lo que este volumen será utilizado para este protocolo.
- Reducir los residuos de cisteína de proteínas mediante la adición de 0.7 l de 1 M DTT (recién preparado) a cada muestra (la concentración final de DTT es 10 mM) e incubando las muestras a 60 ° C durante 30 min.
- Cistinas alquilato de proteínas mediante la adición de 7 l de yodoacetamida tamponada (500 mM yodoacetamida, 1 M HEPES ∙ NaOH pH 8; recién preparados) a cada muestra (la concentración final es yodoacetamida 50 mM) e incubando las muestras a temperatura ambiente durante 20 min en la oscuridad (yodoacetamida es sensible a la luz). Si es necesario, enfriar rápidamente la yodoacetamida restante mediante la adición de DTT a una concentración final de 50 mM.
- Para cada una de las muestras desnaturalizaron usando urea, añadir 482 l de HEPES 100 mM pH 8 ∙ NaOH manera que la concentración final es urea 1 M (tripsina se inhibió significativamente a> 1 M urea 47).
- Tryptically digerir las proteínas en péptidos.
- Para cada muestra que contiene lisado celular, añadir 8 l de grado secuenciación / l modificado tripsina 0,5 g de modo que el concentrat tripsina definitivaion es 01:50 (proteína w: w) tripsina: muestra, e incubar la muestra a 37 ° C durante 18 horas.
- Para cada muestra que no contiene lisado de células, añadir 0,5 l de 0,5 g / grado modificado tripsina secuenciación l, y se incuba la muestra a 37 ° C durante 2 h (cada una de estas muestras son para los experimentos de control, y típicamente contienen sólo el 10 ng -10 g de péptido + masa proteica).
- Para cada una de las muestras desnaturalizadas de urea, añadir 440 l de 2% v / v de ácido fórmico (la concentración final de ácido fórmico es del 1% v / v). Confirme que estas muestras son pH ~ 3.
- Para cada una de las muestras desnaturalizadas de tensioactivo, añadir 914 l de 0,5% v / v TFA (la concentración final es TFA 0,5% v / v). Confirme que estas muestras son pH ~ 1,5. Incubar las muestras a 37 ° C durante 60 min para hidrolizar el agente tensioactivo.
- Microcentrífuga todas las muestras a 21.000 xg durante 20 min a temperatura ambiente para sedimentar el grupo de cola tensioactivo y cualquier otra precipitates que obstruir un cartucho SPE C-18.
- En fase sólida extraer cada uno de los sobrenadantes usando un cartucho SPE C-18 desechable (Tampón A = 0,1% v / v de ácido fórmico; Tampón B = v / v de ácido fórmico, 80% v / v ACN 0,1%; ~ 100 mg C- 18 de resina por cartucho). Vigor las fases móviles a través del cartucho SPE C-18 usando un bulbo de goma de superficie plana, utilizando un colector de extracción, o por centrifugación del cartucho en un tubo de centrífuga de 15 ml a 10 × g durante 5 min.
- Mojar la columna mediante la aplicación de 1 ml de Tampón B, se equilibre mediante la aplicación de 1 ml de Tampón A dos veces, aplicar la muestra y lavar el cartucho mediante la aplicación de 1 ml de Tampón A dos veces. La elución de los péptidos mediante la aplicación de 1 ml de Tampón B lentamente (~ 2 min).
- Concéntrese cada eluato en un concentrador de vacío a un volumen final de 100 l a evaporarse ACN. Añadir 200 l de H 2 O a cada muestra, y concentrar cada uno en un concentrador de vacío a un volumen final de 98 l para evaporar la basura cualquier posible rerestante ACN.
- Añadir 2 l de 5% v / v de ácido fórmico en ACN a cada muestra (las concentraciones de las muestras finales son 0,1% v / v de ácido fórmico, 2% v / v ACN).
Nota: Las muestras pueden ser almacenadas a -80 ° C. - Analizar las muestras usando LC-SRM (como en el paso 3.4). Para los análisis cualitativos LC-SRM, ejecute las muestras biológicas y los estándares de péptidos externos, y analizar todos los datos resultantes juntos. Para los análisis cuantitativo LC-SRM, ejecute la serie de dilución de isótopos de cada muestra biológica, y también ejecutar los ejemplos que consisten en las normas péptido solo, y analizar todos los datos resultantes juntos.
5. Análisis LC-SRM Datos
NOTA: la identificación y cuantificación de péptidos pueden ser altamente simplificadas y parcialmente automatizados usando software como horizonte, pero aún así se recomienda encarecidamente que todos los datos de anotación revisada manualmente. Además, lo mejor es excluir información del nivel de proteínas durante Annot Manualación de los datos de LC-SRM para evitar sesgo.
- Para cada identificación de péptidos, confirme que la forma de cada perfil de elución de transición es más o menos de Gauss. Para cada transición, confirme que el perfil de elución es el producto de múltiples señales medidas por el detector de MS (es decir, no es el producto de uno o dos picos aleatorios de ruido MS).
- Para cada identificación de péptidos, asegúrese de que se selecciona todo el perfil de elución de péptidos. Evite manualmente el ajuste de los límites del perfil de elución de subcomponentes de la identificación de péptidos (la forma péptido marcado pesada, la forma no marcada, iones precursores individuales, y las transiciones individuales).
- Confirme que cada perfil de elución de transición tiene una relación señal-ruido (s / n) ≥3. Nota: "ruido" se refiere al ruido MS azar, no reproducibles señal de analitos coeluyentes. Cromatograma funciones de suavizado pueden ayudar en gran medida el análisis de los datos ruidosos.
- Para cada identificación de péptidos,confirmar que todas las transiciones tienen perfiles de elución casi idénticos (no sólo tiempos iguales vértice del pico; los perfiles de elución pueden tener diferentes amplitudes).
- Para el LC-SRM análisis de una gran cantidad de los estándares de péptidos (típicamente ≥100 fmol de cada péptido), confirmar que los perfiles de elución de péptidos correspondientes son muy intensos (típicamente s / n ≥100), y que las identificaciones de péptidos son inequívocos. Nota: Estas identificaciones altamente seguros serán críticos para la identificación de los correspondientes péptidos derivado de muestra biológicos.
- Para cada identificación de péptidos, confirman que las intensidades de transición relativos coinciden con los de la norma péptido identificado con confianza. Si es necesario, descarte, relativamente perfiles de elución de transición de baja intensidad ruidosos, pero confirman que ninguno de los relativamente intensas transiciones se echa en falta. Haga caso omiso de perfiles de elución transición relativamente intensas que están significativamente y sin ambigüedad afectados por un compañeroeluyendo contaminante.
- Para cada identificación de péptidos, estimar la probabilidad de que se haya producido al azar por la señal de fondo (ruido MS aleatorio más señal reproducible de analitos coeluyentes).
- Estimar esta probabilidad manualmente mediante el examen de todo el cromatograma LC-SRM y la estimación de la singularidad de cada identificación de péptidos. Producir un conjunto de péptido identificaciones confianza que tiene una tasa estimada falso descubrimiento (FDR) de 5%. Usar criterios más estrictos (por ejemplo, FDR ≤1%) si se requiere una confianza mayor conjunto de identificaciones (No se recomienda el uso de criterios más flexibles).
- También puede utilizar el algoritmo mProphet 48 o el algoritmo de auditoría 49, pero revisar manualmente los resultados.
- Para cada identificación de péptidos, confirme que el tiempo de elución observado es coherente con la hidrofobicidad del péptido (Skyline está diseñado para realizar este cálculo).
- Para cada péptido, confirmar quesu tiempo de elución es consistente a través de las carreras de LC.
NOTA: LC-SRM análisis a veces producir perfiles de elución que están sesgadas sistemática entre las corridas. Cambios sistemáticos son de esperar después de las alteraciones de instrumentos tales como la sustitución de una columna. Además, los péptidos que eluyen temprano pueden ser propensos a los cambios (especialmente si la columna se carga cerca de su capacidad), y finales de los péptidos que eluyen también pueden ser propensos al desplazamiento. Identificación de péptidos se ha completado. - Para mejorar la cuantificación de péptidos, vuelva a inspeccionar cada identificación de péptidos y desechar un perfil de elución de transición si es especialmente ruidoso en comparación con los otros perfiles de elución de transición de la identificación de péptidos. Descartar un perfil de elución de transición si su intensidad relativa de transición está mal (en comparación con el estándar de péptido).
- Inspeccione los límites de cada perfil ión precursor y la elución de transición. Si es necesario, ajuste cuidadosamente los límites del perfil de elución para recortar distancia señal de fondo (o para ifondo ejorar la estimación de la señal).
- Para cada estándar péptido interno, compruebe si está contaminado con una cantidad detectable de péptido luz (determinado por la LC-SRM análisis de las normas de péptidos internos solo, descrito anteriormente). Posteriormente, descartar los perfiles de elución de péptidos de luz que fueron comprometidos significativamente por la contaminación-péptido luz dentro de los estándares de péptidos internos.
- Cuantificar cada perfil de elución de transición mediante el cálculo de su área de pico LC. En su caso, deducir la señal de fondo estimado. Cuantificar cada perfil de elución del péptido sumando los valores de cuantificación de transición correspondientes (en lo sucesivo, esto se conoce como el valor "Sum_Peak_Area"). Para cada valor Sum_Peak_Area péptido muestra biológica, calcular la abundancia molar péptido muestra biológica utilizando datos de LC-SRM de normas de péptidos internos o externos (uso sólo las transiciones que fueron cuantificados con éxito tanto por el péptido muestra biológicaLC-SRM y la del patrón de péptido).
- La cuantificación utilizando estándares de péptidos externos (no recomendado):
- Trazar los valores Sum_Peak_Area estándar de péptidos externa frente a los valores de abundancia molares estándar de péptidos externo. Producir una curva estándar mediante el ajuste de una regresión lineal a los datos (típicamente, sólo el componente lineal de la gama dinámica debe ser utilizado; cualquier componente no lineal debe ser utilizado con extrema precaución). Para cada valor Sum_Peak_Area péptido muestra biológica, utilizar la curva estándar para calcular la abundancia molar péptido.
Nota: Este método de cuantificación no se recomienda debido a que requiere que la preparación de la muestra y LC-SRM ser demostrabilidad robusta porque las muestras biológicas y los estándares de péptidos externos se prepararon y analizaron por LC-SRM por separado. Además, no tiene en cuenta los efectos de matriz (efectos debido a los componentes de una muestra que no sea el analito).
- Trazar los valores Sum_Peak_Area estándar de péptidos externa frente a los valores de abundancia molares estándar de péptidos externo. Producir una curva estándar mediante el ajuste de una regresión lineal a los datos (típicamente, sólo el componente lineal de la gama dinámica debe ser utilizado; cualquier componente no lineal debe ser utilizado con extrema precaución). Para cada valor Sum_Peak_Area péptido muestra biológica, utilizar la curva estándar para calcular la abundancia molar péptido.
- Cuantificación usando internanormas de péptidos (recomendado):
- Para cada valor Sum_Peak_Area péptido muestra biológica y el correspondiente péptido interno valor Sum_Peak_Area estándar marcado pesado, calcular la relación luz / pesado. Utilice esta relación como una medida de la / péptido pesado razón de abundancia molar luz correspondiente para calcular la abundancia molar del péptido muestra biológica. En concreto, la abundancia molar péptido muestra biológica es igual a la del péptido interno abundancia molar estándar multiplicada por la relación Sum_Peak_Area ligera / pesada.
- Para cada réplica y péptido diana biológica, identificar el intervalo lineal de la cuantificación LC-SRM mediante la creación de una curva estándar utilizando una regresión lineal. Específicamente, trazar los valores de abundancia molar estándar de péptidos internos frente a los valores pesados / ligeros Sum_Peak_Area relación (tenga en cuenta que la masa de la muestra biológica es constante a través del conjunto de datos). Requerir que cada muestra el valor Sum_Peak_Area péptido biológica está dentro del rango lineal (typicaliado, sólo el componente lineal de la gama dinámica se debe utilizar; cualquier componente no lineal debe utilizarse con extrema precaución). Del mismo modo, realice este paso para cada transición péptido diana.
Nota: Analito cuantificación por LC-QqQ-SRM debe ser lineal en un amplio rango dinámico (~ 10.000). En el extremo inferior, la señal de fondo (ruido MS aleatorio más señal reproducible de analitos coeluyentes) reducirán la precisión de cuantificación y precisión. En el extremo superior, la saturación del detector provocará la no linealidad (en última instancia, esto puede causar perfiles de elución sean partida plana a una intensidad de señal específica).
- La cuantificación utilizando estándares de péptidos externos (no recomendado):
- Si es apropiado, lleve a cabo una normalización global a través de las muestras, tales como una normalización tendencia central 50, para corregir las pequeñas diferencias en la cantidad de la muestra antes de la spiking-in de los estándares de péptidos internos. Si es necesario, utilice sólo las proteínas de limpieza desde el paso 1.1.
- Si es necesario, imputar falta quantificatión valora 51,52.
Nota: Es impropio simplemente reemplazar los valores perdidos con la mitad del límite inferior de cuantificación (LLOQ) o la mitad del límite de detección (LOD), ya que hacerlo sería reducir artificialmente varianza cuantificación, y podría resultar en una prueba estadística (por ejemplo, un ANOVA ) la producción de un resultado falso positivo (un error de tipo I). Sin embargo, haciendo caso omiso de los valores que faltan también puede ser problemático. Por ejemplo, si la mitad de los valores de abundancia verdaderas están por debajo del límite de detección, y la otra mitad están por encima del LLOQ, entonces la media observada abundancia sería sobreestimar la verdadera abundancia significa. - Asegúrese de que los ensayos cumplen las directrices ampliamente aceptadas para LC-SRM 23-25. En particular, requiere que el coeficiente de variación de los valores de cuantificación es típicamente ≤25% para los ensayos clínicos, y ≤35% para los ensayos no clínicos 25. Para los ensayos de LC-SRM relacionadas con la atención sanitaria o veterinaria productos o servicios que se van a tener en cuenta para la aplicación de reglamentaciónRoval, requiere que los ensayos cumplen los requisitos de precisión más estrictas para tales ensayos: "La precisión determinada en cada nivel de concentración no debe superar el 15% del coeficiente de variación (CV) a excepción de la LLOQ, donde no debe superar el 20% de la CV "23,24.
Resultados
El desarrollo de modelos computacionales de predicción de las vías de transducción de señales es uno de los objetivos fundamentales de la biología de sistemas 53. Desafortunadamente, incluso para las vías de señalización que han sido estudiados extensamente y tienen una alta significación clínica, todavía no es generalmente posible predecir cuantitativamente el comportamiento de vía en respuesta a las perturbaciones (por ejemplo, esto es cierto para la MAPK / ERK vía 54). Recientemente, una investigación empleó la proteómica, transcriptómica, dirigidos y modelado computacional y simulación para estudiar la quimiotaxis de macrófagos de ratón vía de señalización 56. El enfoque de la investigación fue la esfingosina-1-fosfato quimiotaxis mediada de células RAW 264.7 (una línea celular de monocitos ratón / macrófagos). Para facilitar el modelado vía, ensayos LC-SRM se desarrollaron y llevaron a cabo para medir la abundancia absoluta de las proteínas de la vía quimiotaxis en células RAW 264.7. Los valores de abundancia resultantes eran usod como parámetros del modelo de vía.
El esquema experimental general (Figura 1) se inició con una lista de proteínas diana, que incluían G i2, un heterotrimeric G-proteína α-subunidad. El éxito del protocolo general es altamente dependiente de la selección de objetivos de péptidos que son proteotípicos y quantotypic, tales como YDEAASYIQSK. Las mezclas de péptidos estándares externos se prepararon y analizaron por LC-QqQ escopeta-MS (MS / MS). El espectro de masas en tándem YDEAASYIQSK resultante se compone de varios fragmentos de iones, y tenía un fondo bajo (Figura 2). Los espectros se utilizan para componer LC-SRM listas de objetivos que contienen los diez primeros fragmentos de iones más intensos por ión precursor.
Una mezcla de 409 estándares de péptidos externos ("JPT409") se analizó por triplicado mediante cualitativa LC-SRM (datos no mostrados). Esta muestra incluyó tres péptidos i2 G, y la identificación de los tres erajuzgados para estar seguros. Posteriormente, RAW 264.7 muestras (repeticiones biológica "raw1" y "raw2") y la muestra se JPT409 cada analizaron por duplicado (dos repeticiones técnica LC-SRM) por cualitativa LC-SRM (estos seis LC-SRM se realizaron análisis tan similar como es posible). El péptido YDEAASYIQSK fue identificado con confianza en los seis análisis (Figuras 3A - C). Los patrones de intensidad de transición YDEAASYIQSK fueron consistentes entre los análisis de los seis LC-SRM, y éstos eran más o menos similar a la escopeta LC-MS (/ MS) patrón (la "Biblioteca" replicar de la Figura 3C).
El patrón de intensidades de transición por sí sola no siempre es suficiente para la identificación confianza de un péptido. La hidrofobicidad del péptido y se mide el tiempo de retención LC deben ser consistentes. Además, los estándares de péptidos externos y los correspondientes péptidos muestra biológica deben tener aproximadamenteveces igual de retención. La hidrofobicidad (estimado utilizando la versión 3.0 SSRCalc 100 algoritmo Å 55) y el tiempo de retención observado de YDEAASYIQSK se encontraron para ser coherente (Figura 4A). También, el tiempo de retención YDEAASYIQSK se midió utilizando tanto los RAW 264.7 análisis y los análisis de los estándares de péptidos externos, y todos los valores de tiempo de retención fueron aproximadamente iguales (Figura 4B).
La mayoría de las cualitativa LC-SRM análisis de las muestras biológicas tuvieron éxito, por lo que corresponde-pesado marcado, purificado, cuantificó estándares de péptidos internos se prepararon y se dispararon-en RAW 264.7 lisados celulares. Serie de dilución isotópica LC-SRM se utilizó para analizar muestras consistentes de las normas internas de péptidos solos (muestra "R0"), el interno y las normas de péptidos externos (muestra "R1"), y seis RAW 264.7 réplicas biológicas (muestras "R2" - "R7"). Dos diferedesnaturalizantes de proteínas nt se utilizaron para probar los posibles problemas de solubilización de proteínas, desnaturalización, alquilación, y / o de digestión que podrían socavar precisa cuantificación de proteínas diana (muestras R2-R4 utiliza urea, y las muestras de R5-R7 utiliza RapiGest SF). Las formas ligeras y pesadas de YDEAASYIQSK contenían patrones de intensidad de transición casi idénticos y perfiles de elución (Figuras 5A - C). LC-SRM resumió relaciones de área de pico se usaron como una medida de la abundancia relativa de péptido, y los ratios YDEAASYIQSK se utilizaron para calcular los valores de abundancia i2 G en unidades de copias por célula RAW 264.7. En paralelo, un segundo G i2 estándar péptido interno (IAQSDYIPTQQDVLR) se utilizó para realizar ensayos de G i2 LC-SRM cuantitativos de los mismos RAW 264.7 muestras, y los dos ensayos produce mediciones de la abundancia G i2 altamente similares a través de todas las réplicas biológicas (Figura 5D). El acuerdo de las dos G ensayos i2 es fuerte evidencia de que ambos péptidos son quantotypic, y que todos los G i2 ensayos cuantitativos LC-SRM eran exactos y precisos.
En general, 35 proteínas se cuantificaron utilizando 58 estándares de péptido interno (Tabla 1). En particular, los ensayos LC-SRM de la proteína estándar interno (luciferasa de luciérnaga; Paso 4,11) fueron exacta y precisa. El conjunto completo de datos de horizonte de esta investigación está disponible en la base de datos en línea LC-SRM Panorama (en la carpeta Manes_RAW_Chemotaxis en https://panoramaweb.org/labkey/project/NIH_NitaLazar/begin.view).
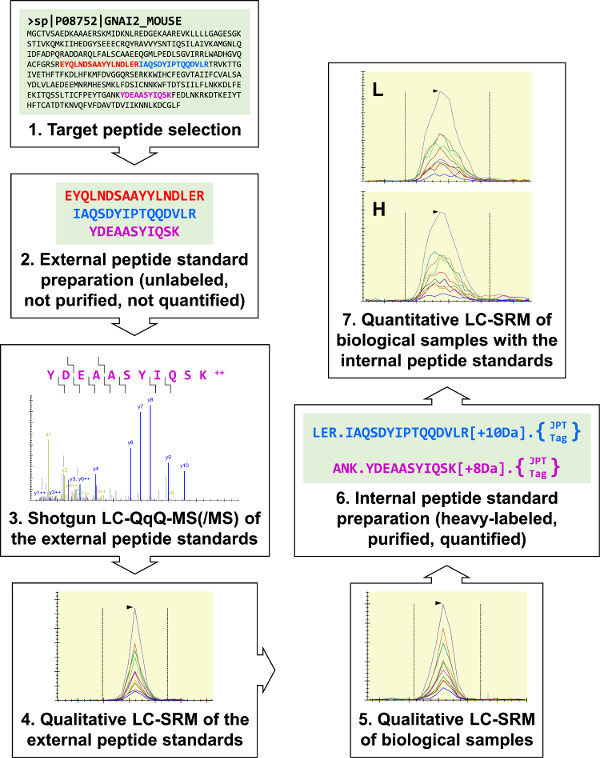
Figura 1:. Descripción general del Protocolo Tres péptidos diana tentativas para G i2 (a heterotrimeric G-proteína α-subunidad) fueron seleccionados para la síntesis de péptido estándar externo. Estos fueron AnalyZED por escopeta LC-MS (MS / MS) para desarrollar tres ensayos LC-SRM G I2, y estos ensayos se utilizaron para realizar análisis cualitativos de muestras biológicas. Se identificaron todos los tres péptidos diana i2 G, y dos fueron seleccionados para la preparación estándar de péptido interno. Se utilizaron-tripsina escindible "JPT-Tags" para cuantificar las normas de péptidos internos utilizando espectrofotometría UV. Las normas internas de péptidos se utilizaron para llevar a cabo ensayos de i2 G LC-SRM cuantitativos de RAW 264.7 muestras. Por favor, haga clic aquí para ver una versión más grande de esta figura.
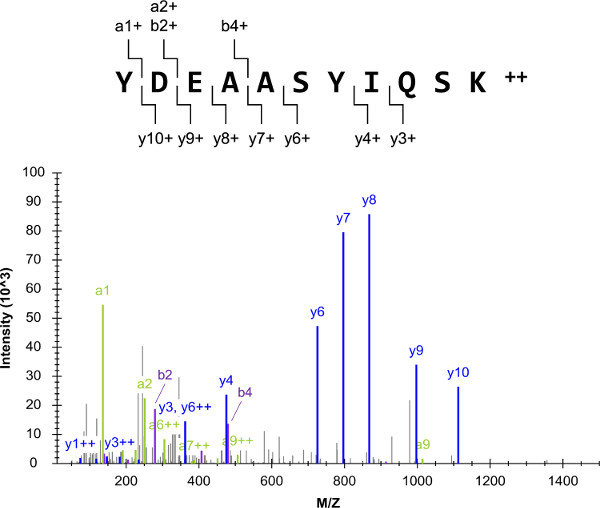
Figura 2:. Shotgun LC-MS (MS / MS) El espectro de masas se muestra originó a partir de los análisis de uno de los estándares G i2 externos de péptidos (YDEAASYIQSK). Para este precursoriones, los diez principales transiciones más intensas fueron seleccionados para LC-SRM (excluyendo el ion fragmento a1 1+ debido a su corta longitud). Haga clic aquí para ver una versión más grande de esta figura.
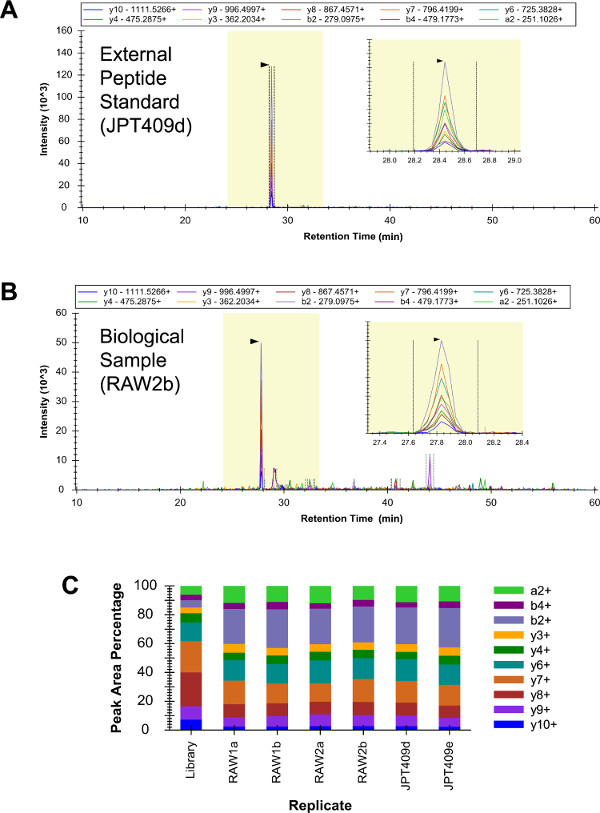
Figura 3:. Cualitativa LC-SRM de G i2 El cromatograma YDEAASYIQSK de la JPT409 análisis contenía un fondo muy bajo y el péptido se identificó con confianza (A). Los correspondientes 264,7 análisis RAW resultaron en más señal de fondo, pero la identificación de péptidos era todavía inequívoca (B). Los patrones relativos de intensidad de transición fueron consistentes entre los seis LC-SRM analiza (dos técnicos repeticiones de tres muestras independientes), y éstos eran más o menos similar a la escopeta correspondiente LC-MS (/ MS) patrón (la "Biblioteca" réplica) (C). Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: predicción tiempo de retención del péptido y la variación entre carreras Una regresión lineal se calculó utilizando la hidrofobicidad estimado y medido cualitativa LC-SRM tiempo de elución de todos los péptidos diana, y los tiempos de elución predichos y medidos de YDEAASYIQSK fueron consistentes (A). . Además, la consistencia de la tiempo de elución observado de cada péptido a través de la LC-SRM análisis se determinó. Los tiempos de elución de YDEAASYIQSK abarcaron un rango de ~ 40 seg, que era consistente con la precisión del instrumento LC-SRM que se utilizó (valoresson el tiempo máximo ápice +/- el ancho total a media-max, y también el ancho en la base del pico) (B). Haga clic aquí para ver una versión más grande de esta figura.
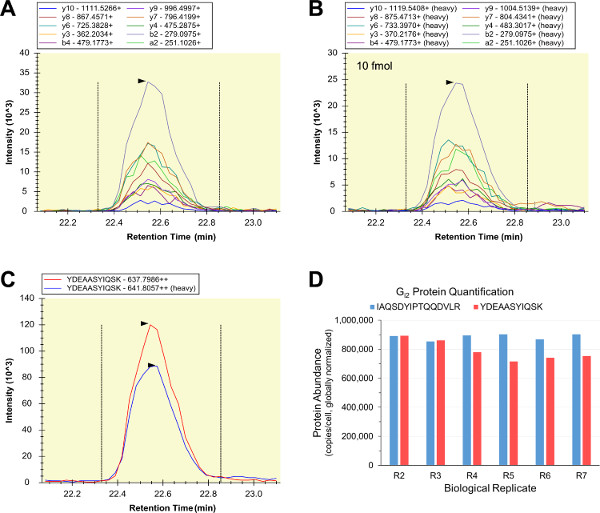
Figura 5:. Cuantitativo LC-SRM de G i2 Los cromatogramas de transición del péptido muestra biológica (A) y el estándar de péptido interno (B) tenía intensidades de transición relativas consistentes, y se resumió (C) (representado es el análisis "R2" para YDEAASYIQSK usando 10 fmol estándar péptido interno). Los perfiles de elución ligeras y pesadas fueron consistentes (C), y la relación del área bajo estas curvas se utilizó como una medida de la light / abundancia péptido pesado. Una segunda norma péptido interno i2 G (IAQSDYIPTQQDVLR) se utilizó para cuantitativa LC-SRM en paralelo, y los valores de abundancia G i2 resultantes fueron consistentes a través de ambos los seis repeticiones biológicos y los dos péptidos diana (en general, n = 12 y CV = 8,38 %) (D). Haga clic aquí para ver una versión más grande de esta figura.
| UniProt adhesión | Proteína diana | Target Péptido | Abundancia (fmol / mg) | Abundancia (copias / célula, normalizado) | CV |
| P08659 | Luciferasa | (UV a 280 nm) | 23,824 | n / A | n / A |
| P08659 | Luciferasa | VVDLDTGK | 24,103 | n / A | 7% |
| P08659 | Luciferasa | VVPFFEAK | 24,717 | n / A | 4% |
| P60710 | Actina, citoplásmica 1 | IWHHTFYNELR | 760.448 | 61762598 | 3% |
| P16858 | GAPDH | LISWYDNEYGYSNR | 357.803 | 28906524 | 14% |
| P06151 | Lactato deshidrogenasa | LLIVSNPVDILTYVAWK | 129.623 | 10633145 | 30% |
| P99024 | Tubulina β 5 | ALTVPELTQQVFDAK | 78,765 | 6398971 | 3% |
| P20152 | Vimentina | SLYSSSPGGAYVTR | 121.100 | 9807488 | 10% |
| P60766 | CDC42 | DDPSTIEK | 86,647 | 6957317 | 27% |
| P60766 | CDC42 | QKPITPETAEK | 26,475 | 2153669 | 6% |
| Q8C3J5 | DOCK2 | ETLYETIIGYFDK | 1,459 | 118539 | 15% |
| Q8C3J5 | DOCK2 | ISSSPTHSLYVFVR | 1,795 | 143440 | 33% |
| Q8BPU7 | ELMO1 | ALTTKPSSLDQFK | 2,302 | 186754 | 7% |
| Q8BPU7 | ELMO1 | SAIDISILQR | 1,244 | 99728 | 25% |
| Q8BGM0 | FGR | GAYSLSIR | 1,099 | 89473 | 17% |
| Q8BGM0 | FGR | WTAPEAALFGR | 0,319 | 24766 | 6% |
| P27601 | Gα 13 | GIHEYDFEIK | 0,843 | 68467 | 15% |
| P27601 | Gα 13 | VFLQYLPAIR | 1,295 | 106176 | 23% |
| P08752 | Gα (i) 2 | IAQSDYIPTQQDVLR | 10,900 | 885701 | 2% |
| P08752 | Gα (i) 2 | YDEAASYIQSK | 9,774 | 790616 | 9% |
| Q9DC51 | Gα (k) | EYQLNDSASYYLNDLDR | 6,574 | 537862 | 15% |
| Q9DC51 | Gα (k) | ISQTNYIPTQQDVLR | 3,504 | 285784 | 10% |
| P62874 | Gβ 1 | AGVLAGHDNR | 17,078 | 1385578 | 4% |
| Q9CXP8 | Gγ 10 | DALLLGVPAGSNPFR | 2,167 | 179178 | 36% |
| P63213 | Gγ 2 | EDPLLTPVPASENPFR | 3,284 | 266360 | 8% |
| Q80SZ7 | Gγ 5 | VSQAAADLK | 6,087 | 493512 | 6% |
| P08103 | HCK | GPVYVPDPTSSSK | 1,143 | 93044 | 12% |
| P08103 | HCK | IIEDNEYTAR | 0,944 | 77147 | 19% |
| P43406 | Integrina α V | AGTQLLAGLR | 0,276 | 22264 | 32% |
| P43406 | Integrina α V | SHQWFGASVR | 0,443 | 34235 | 5% |
| P25911 | LYN | VIEDNEYTAR | 1,461 | 119377 | 13% |
| Q5SW28 | PI3K 5 reglamentario | AGFPGILDTASPGK | 0,301 | 24331 | 11% |
| Q8K3B3 | PI3K α reglamentario | LYEEYTR | 0,472 | 38592 | 16% |
| Q8K3B3 | PI3K α reglamentario | TWNVGSSNR | 0,524 | 42697 | 12% |
| Q8K3B3 | PI3K α reglamentario | VLSEIFSPVLFR | 0,440 | 35902 | 20% |
| Q5U3K7 | PI3K β reglamentario | DTPDGTFLVR | 0,188 | 15262 | 30% |
| Q5U3K7 | PI3K β reglamentario | IAEIHESR | 0,282 | 22561 | 13% |
| Q0VGQ5 | PI3K α | LINLTDILK | 0,102 | 8494 | 38% |
| Q8CI98 | PI3K δ | HEVQEHFPEALAR | 0,178 | 14566 | 22% |
| Q8CI98 | PI3K δ | ITEEEQLQLR | 0,481 | 38709 | 24% |
| Q9ES52 | PIP3 5-fosfatasa 1 | IVVLAKPEHENR | 0,486 | 39194 | 19% |
| Q9ES52 | PIP3 5-fosfatasa 1 | LSQLTSLLSSIEDK | 2,056 | 167123 | 7% |
| Q69ZK0 | PIP3 dependiente de Rac 1 FMAM | DSVLSYTSVR | 0,647 | 52712 | 32% |
| Q69ZK0 | PIP3 dependiente de Rac 1 FMAM | NQLLLALLK | 0,354 | 27425 | 5% |
| Q9CQE5 | RGS 10 | ASSQVNVEGQSR | 2,460 | 199782 | 4% |
| Q9CQE5 | RGS 10 | WASSLENLLEDPEGVQR | 20,647 | 206005 | 11% |
| Q9CX84 | RGS 19 | AEANQHVVDEK | 0,495 | 39753 | 21% |
| Q9CX84 | RGS 19 | LIYEDYVSILSPK | 0,846 | 68481 | 22% |
| B9EKC3 | Rho GAP 5 | DGLAQELANEIR | 0,448 | 34653 | 10% |
| Q99PT1 | Rho GDI 1 | SIQEIQELDK | 3,156 | 267063 | 16% |
| Q99PT1 | Rho GDI 1 | VAVSADPNVPNVIVTR | 37,077 | 3006168 | 5% |
| Q61599 | Rho GDI 2 | LNYKPPPQK | 37,975 | 3086596 | 3% |
| Q61599 | Rho GDI 2 | YVQHTYR | 21,436 | 1711908 | 47% |
| Q61210 | Rho FMAM 1 | FDGAEGSWFQK | 2,149 | 176139 | 47% |
| Q61210 | Rho FMAM 1 | SGLELEPEEPPGWR | 2,911 | 236483 | 8% |
| Q9QUI0 | RhoA | QVELALWDTAGQEDYDR | 43,500 | 3532438 | 8% |
| P70336 | ROCK2 | GAFGEVQLVR | 0,527 | 42800 | 23% |
| P70336 | ROCK2 | IYESIEEAK | 1,011 | 83794 | 33% |
| P70336 | ROCK2 | LEGWLSLPVR | 0,573 | 48259 | 22% |
| Q8R0X7 | S1P liasa 1 | AGYPLEKPFDFR | 1,787 | 147043 | 27% |
| Q8R0X7 | S1P liasa 1 | TPEIVAPESAHAAFDK | 3,562 | 291791 | 18% |
Tabla 1: proteínas celulares cuantitativos 264.7 LC-SRM de RAW Treinta y cinco proteínas de las células RAW 264.7 se cuantificaron utilizando cincuenta y ocho estándares de péptidos internos y seis repeticiones biológicos.. Cinco de las proteínas diana fueron el servicio de limpieza proteínas (actina, GAPDH, lactato deshidrogenasa, tubulina, y vimentina), y se cuantificaron para permitir la normalización a través de muestras biológicas (paso 1.1). Además, un estándar de proteína interna, se añadieron-en cada lisado de células y se cuantificó mediante LC-SRM (4.765 pmol de luciferasa de luciérnaga por 200 g de muestra; 98% puro por SDS-PAGE; cuantificado espectrofotométricamente a 280 nm; Paso 4,11). Se calcularon los valores de CV a través de las seis réplicas biológicas utilizando los valores de abundancia globalmente normalizados (excepto para la luciferasa; Paso 5,15).
Discusión
Cuantificación de proteínas absoluta es esencial para una gama muy diversa de aplicaciones biomédicas como la validación de biomarcadores y el modelado vía de transducción de señales. Recientemente, la proteómica dirigidos utilizando LC-SRM se ha beneficiado de las mejoras a las numerosas tecnologías, incluyendo la preparación de péptidos estándar, HPLC, QqQ-MS, y el análisis de datos LC-SRM. En consecuencia, se ha convertido en una alternativa poderosa para inmunoensayos. Los inmunoensayos pueden ser extremadamente sensible y de alto rendimiento, pero el desarrollo de un inmunoensayo robusto puede ser extremadamente difícil debido a inmunoensayos pueden ser vulnerables a la reactividad cruzada y / o interferencia, incompatible con los métodos de lisis / homogeneización de células / tejidos, y / o no susceptible de multiplexación 5,8. Por ejemplo, la prueba más completa para la reactividad cruzada es llevar a cabo el inmunoensayo utilizando muestras que se originaron de knockouts de genes, lo que puede ser difícil de preparar.
Este protocolo describe Pept objetivoselección ide, desarrollo del ensayo LC-SRM, cualitativa y cuantitativa LC-SRM, y el análisis de datos LC-SRM. Se utiliza para medir la abundancia absoluta de proteínas de la vía 36 de quimiotaxis dentro de células RAW 264.7 56, pero su aplicabilidad se extiende mucho más allá de esta aplicación específica. A pesar de que fue diseñado para cuantificar las proteínas dentro de los sedimentos celulares, podría ser ajustado para el análisis de otras muestras biológicas (por ejemplo, Biofluids) y otros objetivos SRM (por ejemplo, fosfopéptidos). Por ejemplo, cambiar el protocolo de homogeneización y / o proteína de digestión puede mejorar significativamente la solubilización, la desnaturalización, la alquilación, la digestión, y la cuantificación de proteínas diana especialmente difíciles (por ejemplo, proteínas de membrana), o podría permitir el análisis de muestras especialmente difíciles (por ejemplo, muestras que contiene
La selección inicial de péptidos diana es crítica, pero puede llevar mucho tiempo. Para Hundreds de proteínas diana, una puntuación ad hoc se podrían utilizar para cada criterio, y los péptidos diana tentativas pueden ser clasificados en base a la suma de los puntos, como se ha hecho con anterioridad 56. Por otra parte, este análisis se puede automatizar utilizando PeptidePicker, una interfaz web que simplifica enormemente la selección péptido diana 30 (http://mrmpeptidepicker.proteincentre.com/).
Después de que los péptidos diana han sido seleccionados y los ensayos LC-SRM se han desarrollado, es fundamental que los ensayos LC-SRM cualitativos de las muestras biológicas pueden realizar porque incluso un péptido altamente proteotípicos y quantotypic no será detectable si la proteína se expresa en niveles por debajo del umbral de sensibilidad del instrumento, o si la interferencia de fondo es especialmente problemático. Una segunda dimensión de la separación (por ejemplo, fuerte HPLC de intercambio catiónico, de alto pH HPLC de fase inversa, y libre de gel electroforesis) puede aumentar la profundidad proteómica, pero requerirásignificativamente más tiempo de instrumentos y análisis de datos. Alternativamente, una estrategia de enriquecimiento (por ejemplo, immunoenrichment péptido y a nivel de proteínas y fraccionamiento de la célula) puede mejorar la profundidad proteómico, y immunodepletion de proteínas muy abundantes puede ser utilizado para reducir la interferencia por coeluyentes analitos.
LC-SRM de ~ decenas de muestras para ~ ~ cientos o miles de transiciones normalmente requiere tiempo extenso instrumento. A pesar de que no se utilizó para este estudio, LC-SRM programación (que mide las transiciones durante ventanas de tiempo pre-especificado de elución) permite el análisis de más transiciones por corrida. También, la programación reduce el ciclo de trabajo SRM durante períodos relativamente vacantes del gradiente LC, y esto puede resultar en la identificación de péptidos mejorada y cuantificación. Sin embargo, la programación requiere que el tiempo de elución del péptido determinarse con confianza a partir de un análisis LC-SRM cualitativa. Los cambios sutiles a la preparación de la muestra, el instrumento LC-MS,o el método LC-MS puede causar programación para recortar o incluso totalmente pierdas péptidos diana. Por ejemplo, los perfiles de elución de la muestra YDEAASYIQSK biológicos se desplazan ligeramente con relación a los de péptido estándar externo (Figura 4B), posiblemente debido a los efectos de matriz.
En resumen, un protocolo paso a paso se presenta para el desarrollo y aplicación de la LC-SRM para la cuantificación de proteínas absoluta. Cuantificación absoluta de proteínas por LC-SRM ya ha sido demostrado ser reproducible entre laboratorios 16,19. Tecnologías de Proteómica, incluyendo la preparación de muestras (por ejemplo, la automatización), cromatografía líquida, espectrometría de masas y análisis de datos, están mejorando rápidamente, y está permitiendo LC-SRM para convertirse en práctica para la investigación a gran escala y aplicaciones clínicas. La especificidad, sensibilidad, exactitud, reproducible y de alto rendimiento de cuantitativa LC-SRM hace que sea una herramienta de gran alcance para la investigación básica y la biomedicina.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
This research was supported by the Intramural Research Program of the NIH, National Institute of Allergy and Infectious Diseases.
Materiales
| Name | Company | Catalog Number | Comments |
| Acetonitrile (ACN), LC-MS grade | Fisher | A955-1 | |
| BCA (bicinchoninic acid) protein assay kit | Fisher | 23235 | |
| Beads for bead beating, zirconia-silica, 0.1 mm | BioSpec Products | 11079101z | |
| Bestatin hydrochloride | Sigma | B8385-10MG | |
| Cell culture DMEM (with glucose, without L-glutamine) | Lonza | 12-614F | |
| Cell culture EDTA, 500 mM, pH8 | Gibco | 15575 | |
| Cell culture fetal bovine serum (FBS) | Atlanta Biologicals | S11550 | |
| Cell culture L-glutamine | Sigma | G8540-25G | |
| Cell culture phosphate buffered saline (PBS) pH 7.4 | Gibco | 10010-049 | |
| Cell culture Trypan Blue viability stain, 0.4% w/v | Lonza | 17-942E | |
| Cellometer Auto T4 cell counter | Nexcelom Bioscience | Cellometer Auto T4 | |
| Cellometer Auto T4 disposable counting chambers | Nexcelom Bioscience | CHT4-SD100-014 | |
| Dithiothreitol (DTT) | Sigma | D5545-5G | |
| Formic acid, LC-MS grade, ampules | Fisher | A117-10X1AMP | |
| Hemocytometer, Neubauer-improved, 0.1 mm deep | Marienfeld-Superior | 0640030 | |
| HEPES, 1 M, pH 7.2 | Mediatech | 25-060-CI | |
| Hydrochloric acid, 37% w/w | VWR | BDH3028-2.5LG | |
| Iodoacetamide | Sigma | I1149-5G | |
| Laser Based Micropipette Puller | Sutter Instrument Co. | P-2000 | |
| LC coated silica capillary, 50 µm id | Polymicro Technologies | 1068150017 | |
| LC vial, autosampler, 12 mm x 32 mm polypropylene | SUN SRI | 200-268 | |
| LC vial screw cap, autosampler, pre-slit PTFE/silicone | SUN SRI | 500-061 | |
| Luciferase, from Photinus pyralis | Sigma | L9506-1MG | |
| Pepstatin A | EMD Millipore | 516481-25MG | |
| pH strips colorpHast (pH 0.0-6.0) | EMD Chemicals | 9586-1 | |
| PhosStop phosphatase inhibitor cocktail | Roche | 04906837001 | |
| RapiGest SF | Waters | 186001861 | |
| Sep-Pak SPE, C18 1 ml 100 mg cartridge | Waters | WAT023590 | |
| Sep-Pak SPE, extraction manifold, 20 position | Waters | WAT200609 | |
| Sep-Pak SPE, flat-surfaced rubber bulb | Fisher | 03-448-25 | |
| Sodium hydroxide (NaOH) | Fisher | S318-500 | |
| SpeedVac vacuum concentrator | Fisher | SPD111V | |
| Trifluoroacetic acid (TFA), LC-MS grade | Fisher | A116-50 | |
| Trypsin, sequencing grade, modified | Promega | V5113 | |
| Tube decapper for Micronic tubes | USA Scientific | 1765-4000 | |
| Tubes, 2 ml microcentrifuge, o-ring screw-cap, sterile | Sarstedt | 72.694.006 | |
| Urea | Sigma | U0631-500g | |
| Water, LC-MS grade | Fisher | W6-1 |
Referencias
- Cox, J., Mann, M. Quantitative high-resolution proteomics for data-driven systems biology. Annu Rev Biochem. 80, 273-299 (2011).
- Zhang, Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chem Rev. 113, 2343-2394 (2013).
- Boja, E. S., Rodriguez, H. Mass spectrometry-based targeted quantitative proteomics: achieving sensitive and reproducible detection of proteins. Proteomics. 12, 1093-1110 (2012).
- Gillette, M. A., Carr, S. A. Quantitative analysis of peptides and proteins in biomedicine by targeted mass spectrometry. Nat Methods. 10, 28-34 (2013).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nat Methods. 9, 555-566 (2012).
- Lesur, A., Domon, B. Advances in high-resolution accurate mass spectrometry application to targeted proteomics. Proteomics. , (2015).
- Wild, D. . The immunoassay handbook : theory and applications of ligand binding ELISA., and related techniques. , (2013).
- Sturgeon, C. M., Viljoen, A. Analytical error and interference in immunoassay: minimizing risk. Ann Clin Biochem. 48, 418-432 (2011).
- Adrait, A., et al. Development of a Protein Standard Absolute Quantification (PSAQ) assay for the quantification of Staphylococcus aureus enterotoxin A in serum. J Proteomics. 75, 3041-3049 (2012).
- Lin, D., Alborn, W. E., Slebos, R. J., Liebler, D. C. Comparison of protein immunoprecipitation-multiple reaction monitoring with ELISA for assay of biomarker candidates in plasma. J Proteome Res. 12, 5996-6003 (2013).
- Weiss, F., et al. Catch and measure-mass spectrometry-based immunoassays in biomarker research. Biochim Biophys Acta. 1844, 927-932 (2014).
- Yassine, H., et al. Mass spectrometric immunoassay and MRM as targeted MS-based quantitative approaches in biomarker development: potential applications to cardiovascular disease and diabetes. Proteomics Clin Appl. 7, 528-540 (2013).
- Zhao, L., et al. Quantification of proteins using peptide immunoaffinity enrichment coupled with mass spectrometry. J Vis Exp. , (2011).
- Becker, J. O., Hoofnagle, A. N. Replacing immunoassays with tryptic digestion-peptide immunoaffinity enrichment and LC-MS/MS. 4, 281-290 (2012).
- Wasinger, V. C., Zeng, M., Yau, Y. Current status and advances in quantitative proteomic mass spectrometry. Int J Proteomics. 2013, 180605 (2013).
- Abbatiello, S. E., et al. Large-scale inter-laboratory study to develop, analytically validate and apply highly multiplexed, quantitative peptide assays to measure cancer-relevant proteins in plasma. Mol Cell Proteomics. , (2015).
- Rodriguez-Suarez, E., Whetton, A. D. The application of quantification techniques in proteomics for biomedical research. Mass Spectrom Rev. 32, 1-26 (2013).
- Wehr, A. Y., Hwang, W. T., Blair, I. A., Yu, K. H. Relative quantification of serum proteins from pancreatic ductal adenocarcinoma patients by stable isotope dilution liquid chromatography-mass spectrometry. J Proteome Res. 11, 1749-1758 (2012).
- Kennedy, J. J., et al. Demonstrating the feasibility of large-scale development of standardized assays to quantify human proteins. Nat Methods. 11, 149-155 (2014).
- Jensen, K. J., Shelton, P. T., Pedersen, S. L. . Peptide synthesis and applications. , (2013).
- Pratt, J. M., et al. Multiplexed absolute quantification for proteomics using concatenated signature peptides encoded by QconCAT genes. Nat Protoc. 1, 1029-1043 (2006).
- Brun, V., et al. Isotope-labeled protein standards: toward absolute quantitative proteomics. Mol Cell Proteomics. 6, 2139-2149 (2007).
- . . Guidance for Industry: Bioanalytical Method Validation. , (2001).
- . . Guidance for Industry: Bioanalytical Method Validation. , (2013).
- Carr, S. A., et al. Targeted peptide measurements in biology and medicine: best practices for mass spectrometry-based assay development using a fit-for-purpose approach. Mol Cell Proteomics. 13, 907-917 (2014).
- Ori, A., Andres-Pons, A., Beck, M. The use of targeted proteomics to determine the stoichiometry of large macromolecular assemblies. Methods Cell Biol. 122, 117-146 (2014).
- Rosen, O., Feldberg, L., Gura, S., Zichel, R. A new peptide substrate for enhanced botulinum neurotoxin type B detection by endopeptidase-liquid chromatography-tandem mass spectrometry/multiple reaction monitoring assay. Anal Biochem. , (2015).
- Feng, Y., et al. Global analysis of protein structural changes in complex proteomes. Nat Biotechnol. 32, 1036-1044 (2014).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26, 966-968 (2010).
- Mohammed, Y., et al. PeptidePicker: a scientific workflow with web interface for selecting appropriate peptides for targeted proteomics experiments. J Proteomics. 106, 151-161 (2014).
- Rodriguez, J., Gupta, N., Smith, R. D., Pevzner, P. A. Does trypsin cut before proline. J Proteome Res. 7, 300-305 (2008).
- Min, X. J., Butler, G., Storms, R., Tsang, A. OrfPredictor: predicting protein-coding regions in EST-derived sequences. Nucleic Acids Res. 33, W677-W680 (2005).
- Lam, H., et al. Building consensus spectral libraries for peptide identification in proteomics. Nat Methods. 5, 873-875 (2008).
- Craig, R., Cortens, J. P., Beavis, R. C. Open source system for analyzing, validating, and storing protein identification data. J Proteome Res. 3, 1234-1242 (2004).
- Desiere, F., et al. The PeptideAtlas project. Nucleic Acids Res. 34, D655-D658 (2006).
- Vizcaino, J. A., et al. The PRoteomics IDEntifications (PRIDE) database and associated tools: status in 2013. Nucleic Acids Res. 41, D1063-D1069 (2013).
- Frank, R. The SPOT-synthesis technique. Synthetic peptide arrays on membrane supports--principles and applications. J Immunol Methods. 267, 13-26 (2002).
- Ong, S. E., Kratchmarova, I., Mann, M. Properties of 13C-substituted arginine in stable isotope labeling by amino acids in cell culture (SILAC). J Proteome Res. 2, 173-181 (2003).
- Mant, C. T., et al. HPLC analysis and purification of peptides. Methods Mol Biol. 386, 3-55 (2007).
- Alterman, M. A., Hunziker, P. . Amino acid analysis : methods and protocols. , (2012).
- Maclean, B., et al. Effect of collision energy optimization on the measurement of peptides by selected reaction monitoring (SRM) mass spectrometry. Anal Chem. 82, 10116-10124 (2010).
- Nesvizhskii, A. I. A survey of computational methods and error rate estimation procedures for peptide and protein identification in shotgun proteomics. J Proteomics. 73, 2092-2123 (2010).
- Tabb, D. L., Friedman, D. B., Ham, A. J. Verification of automated peptide identifications from proteomic tandem mass spectra. Nat Protoc. 1, 2213-2222 (2006).
- Freshney, R. I. . Culture of animal cells : a manual of basic technique and specialized applications. , (2010).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. J Proteome Res. 8, 2144-2156 (2009).
- Noble, J. E., Bailey, M. J. Quantitation of protein. Methods Enzymol. 463, 73-95 (2009).
- Kiser, J. Z., Post, M., Wang, B., Miyagi, M. Streptomyces erythraeus trypsin for proteomics applications. J Proteome Res. 8, 1810-1817 (2009).
- Reiter, L., et al. mProphet: automated data processing and statistical validation for large-scale SRM experiments. Nat Methods. 8, 430-435 (2011).
- Abbatiello, S. E., Mani, D. R., Keshishian, H., Carr, S. A. Automated detection of inaccurate and imprecise transitions in peptide quantification by multiple reaction monitoring mass spectrometry. Clin Chem. 56, 291-305 (2010).
- Callister, S. J., et al. Normalization approaches for removing systematic biases associated with mass spectrometry and label-free proteomics. J Proteome Res. 5, 277-286 (2006).
- Karpievitch, Y. V., Dabney, A. R., Smith, R. D. Normalization and missing value imputation for label-free LC-MS analysis. BMC Bioinformatics. 13, S5 (2012).
- Oh, S., Kang, D. D., Brock, G. N., Tseng, G. C. Biological impact of missing-value imputation on downstream analyses of gene expression profiles. Bioinformatics. 27, 78-86 (2011).
- Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A., Fraser, I. D. Systems biology in immunology: a computational modeling perspective. Annu Rev Immunol. 29, 527-585 (2011).
- Futran, A. S., Link, A. J., Seger, R., Shvartsman, S. Y. ERK as a model for systems biology of enzyme kinetics in cells. Curr Biol. 23, R972-R979 (2013).
- Krokhin, O. V. Sequence-specific retention calculator. Algorithm for peptide retention prediction in ion-pair RP-HPLC: application to 300- and 100-A pore size C18 sorbents. Anal Chem. 78, 7785-7795 (2006).
- Manes, N. P., Angermann, B. R., Koppenol-Raab, M., An, E., Sjoelund, V. H., Sun, J., Ishii, M., Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A. Targeted Proteomics-Driven Computational Modeling of Macrophage S1P Chemosensing. . Mol Cell Proteomics. , .
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados