Method Article
Coleta e Extração de DNA de saliva para Next Generation Sequencing
Neste Artigo
Resumo
DNA extraction from saliva can provide a readily available source of high molecular weight DNA, with little to no degradation/fragmentation. This protocol provides optimized parameters for saliva collection/storage and DNA extraction to be of sufficient quality and quantity for downstream DNA assays with high quality requirements.
Resumo
The preferred source of DNA in human genetics research is blood, or cell lines derived from blood, as these sources yield large quantities of high quality DNA. However, DNA extraction from saliva can yield high quality DNA with little to no degradation/fragmentation that is suitable for a variety of DNA assays without the expense of a phlebotomist and can even be acquired through the mail. However, at present, no saliva DNA collection/extraction protocols for next generation sequencing have been presented in the literature. This protocol optimizes parameters of saliva collection/storage and DNA extraction to be of sufficient quality and quantity for DNA assays with the highest standards, including microarray genotyping and next generation sequencing.
Introdução
A obtenção de DNA de alta qualidade para estudos genéticos humanos, é essencial no processo de descoberta do gene da doença. Sangue, embora exigindo um procedimento invasivo e também a ser mais caro do que a coleta da saliva, é favorecido para a criação de linhas de células imortalizadas como uma fonte infinita de DNA, ou iPSCs para estudos funcionais e, às vezes DNA do sangue é usado quando as linhas celulares não estão disponíveis. No entanto, a obtenção de sangue requer um flebotomista treinado e sangue tem uma meia-vida mais curta do que a saliva 1. DNA de saliva é mais barata e mais fácil de obter, uma vez que podem ser coletadas e enviadas pelo correio, sem a necessidade de um phlebotomist, aumentando, assim, potenciais piscinas sujeitas bem além da área de abrangência de hospitais e laboratórios 2. Inclusão no estudo pode ser melhorada quando os indivíduos têm a opção de dar uma amostra de saliva em vez de sangue 3, 4. Preocupações sobre a quantidade e qualidade de DNA de saliva pode ter limitado a sua generalizada nose apesar de diversos estudos recentes estudos que comprovem a adequação de toda saliva, com uma média de 4,3 x 10 5 células por mililitro, para testes de DNA ao longo dos mais antigos métodos de cotonetes bucais que não obtiveram uma quantidade significativa de saliva 2, 3, 4, 5, 6. Embora exista uma literatura modesta mostrando a adequação de DNA derivado saliva inteiro para aplicações de genotipagem incluindo métodos baseados em microarrays 8, 9, 10, nenhum estudo examinou sequenciamento de próxima geração (NGS). O objetivo de otimização deste protocolo toda a extração de DNA de saliva era maximizar quantidade e qualidade para aplicações de genética de uma forma rentável que pode ser facilmente implementado em laboratórios com reagentes e consumíveis comuns.
Extracção de DNA a partir da saliva requer vários procedimentos: 1) a recolha e armazenamento, 2) lise celular, 3) tratamento com RNase, 4) precipitação de proteínas, 5) precipitação com etanol, 6) de re-hidratação de ADN. A solução de DNA Estabilização tampão, descrito2 anteriormente, funciona adequadamente sem alteração. Nenhuma tentativa foi feita para otimizar o tratamento RNase e etapas de reidratação de DNA. Para cada etapa restante, foram identificadas diversas variáveis que podem afetar o rendimento. Cada variável foi manipulado individualmente e melhoria na produtividade e qualidade foi avaliada estatisticamente. Para as variáveis que foram mostrados para melhorar a produtividade e / ou qualidade do DNA, os valores ótimos foram incluídos no protocolo final.
Protocolo
NOTA: Antes de fornecer amostras de saliva todos os sujeitos deram consentimento informado em conformidade com as diretrizes para o tratamento de seres humanos do Hospital Nacional Infantil.
1. Saliva coleta e armazenamento
- Antes da coleta de saliva, garantir que a boca do tema é livre de alimentos ou outras substâncias estranhas, tendo o assunto lavar a boca com água e evitar comer ou beber por 30 minutos antes de coletar a amostra.
- Abrir um tubo de centrífuga de 15 ml com 2,5 ml de tampão de estabilização de DNA 2 certificando-se de evitar tocar no interior da tampa ou do tubo, e o sujeito tem cuspir 2,5 ml de saliva na solução-tampão. Nota: Recolha de mais de 2,5 ml de saliva pode levar à degradação da amostra a partir de uma proporção suficiente de amostra para tampão de estabilização do ADN. Coleta de muito pouca saliva irá reduzir os rendimentos esperados do protocolo. Para avaliar volumes da coleção, use os gradientes numeradas no lado de tele tubo.
- Recoloque a tampa e misturar por inversão até que a mistura é homogeneizada. A agitação vigorosa não é necessário. Armazenar as amostras a temperatura ambiente durante o armazenamento a curto prazo ou a 4 ° C para armazenamento a longo prazo (> 3 meses).
2. Preparativos Iniciais e lise celular
- Antes de iniciar a extração, aqueça um banho de água a 37 ° C, e preparar um balde de gelo. Três tubos de 15 ml de centrífuga cônicos serão necessários para cada amostra extraída. Os três tubos será utilizado para conter o sedimento de células e de proteínas, a final ADNg extraída, e os sobrenadantes de isopropanol e etanol.
- Recuperar amostras de armazenamento e amostras inverta várias vezes em seguida, vortex em velocidade média por 15 segundos.
- Dispense 2,5 ml da amostra num tubo de centrífuga de 15 ml limpo, e adicionar 5 ml de solução de lise celular. Misturar a amostra 50 vezes por inversão, e incubar à temperatura ambiente durante 30 min.
Remoção 3 RNA
- Adicionar 40 ul de ARNase A Solbuição de 100 mg / ml, e incubar a 37 ° C durante 15 min.
- Retirar a amostra a partir da 37 ° C e banho de água fria no gelo por 3 min.
- Após a incubação com RNase A, aumentar a temperatura do banho de água a 65 ° C durante o passo de rehidratação ADN do protocolo.
4. Protein e remoção de lipídeos
- Adicionam-se 50 ul de proteinase K a solução de 20 mg / mL, misturar várias vezes por inversão, e incubar à temperatura ambiente durante um mínimo de 30 min. Nota: Este é um possível ponto de pausa para o protocolo. Após a adição da solução de proteinase K, a amostra pode ser armazenada a 4 ° C até que a extracção pode ser concluído. O armazenamento a 4 ° C durante até 24 h não foi demonstrado ter um efeito significativo sobre os rendimentos de extracção ou a qualidade do DNA. Armazenamento de longo prazo nesta fase não foi avaliado.
- Adicionar 1,7 ml de solução de precipitação de proteína, vortex vigorosamente durante 20 segundos a alta velocidade, e colocar em gelo durante 10 min.
- Uma vez que as amostras foram arrefecidas em gelo durante 10 minutos, centrifuga-se durante 10 min a 3000 xg e 4 ° C. As proteínas precipitadas deve formar um sedimento apertado para continuar. Se a pastilha não é apertado ou a solução está ainda turva, as amostras podem ser arrefecida em gelo durante 5 minutos e mais de centrifugação repetida. As amostras devem ser mantidas em gelo para garantir uma bolinha apertado.
5 Isolamento e purificação de ADNg
- Num tubo de centrífuga de 15 ml limpo, pipeta de 5 ml de isopropanol e 8 ul de solução do glicogénio puro a 20 mg / ml.
- Verter o sobrenadante contendo o ADNg do passo 4.3 para dentro do tubo que contém a solução de isopropanol e do glicogénio, deixando para trás a proteína precipitada. Uma vez que tenha sido adicionado ao sobrenadante, misturar delicadamente a amostra 50 vezes por inversão e centrifuga-se durante 30 min a 3000 xg e 4 ° C.
- Verter o sobrenadante lentamente para um tubo limpo de 15 ml. Após a remoção do sobrenadante, adicionar 1 ml de etanol a 70% paralavar o sedimento por balanço e lentamente movendo-se delicadamente sobre o etanol o sedimento precipitado várias vezes. Reter o etanol no tubo.
- Após a lavagem inicial, centrifugar a amostra durante 1 min a 2000 xg e 20 ° C. Este passo de centrifugação pode ser feito em cada 4 ° C ou 20 ° C. Não houve efeito significativo da temperatura tem sido demonstrado para esta etapa.
- Após a lavagem e centrifugação inicial da pastilha, verter lentamente a lavagem com etanol a partir do tubo e descarte, em seguida, executar uma segunda lavagem, repetindo os passos 5.3 e 5.4.
- Após a remoção do sobrenadante a partir da segunda lavagem, o sedimento de permitir secar ao ar durante 15 min.
- Se a amostra não estiver completamente seca, o ar seco durante mais 15 min.
6. Reidratação de gDNA
- Uma vez que a amostra tenha secado, adicionar 300 ul de Tris-EDTA para re-hidratar a pastilha seca ADNg.
- Vortex da amostra durante 5 segundos em velocidade média e coloque em um65 ° C num banho de água quente durante 1 hora.
- Retirar as amostras do banho-maria e incubar O / N à temperatura ambiente.
NOTA: Todos os produtos e reagentes utilizados estão listados na tabela de materiais, bem como a Tabela 4.
Resultados
Para determinar parâmetros ótimos para Extração de DNA de uma série de extrações de DNA pareadas foi realizada. Uma única amostra de saliva foi dividido e cada porção ensaiada por um dos dois valores possíveis para uma determinada variável. Pelo menos oito repetições de cada teste emparelhado foram realizadas (por exemplo, uma única amostra de saliva foi dividido em alíquotas para testar a extracção com e sem inicial de incubação de 50 ° C). Otimização foi baseada em quatro métricas padrão: produção total de DNA, o valor 260/280, o valor de 260/230, e de inspecção visual do DNA electroforese para avaliar a fragmentação. Nem todas as combinações possíveis das variáveis foram avaliadas interações significativas (N = 169 combinações), optando para avaliar o efeito marginal de cada variável individualmente. Os efeitos foram testados usando um multi-way de medidas repetidas ANOVA e efeitos estimados foram obtidos a partir da equação de regressão equivalente. Todos os efeitos significativos encontram-se resumidos na Tabela 1, como mostrado avermudança de idade no rendimento (ng / mL por ml de saliva de entrada) ou a qualidade do DNA (260/280 e 260/230).
A lise celular (passo 2) foi optimizada através da avaliação: 1) a presença / ausência de uma incubação de 50 ° C (1 hora), antes do rompimento das células para assegurar que a proteinase K degradação e lise celular mediada por o tampão de armazenamento ficou completa, 2 ) a presença / ausência de um passo de homogeneização em vortex (velocidade média, 15 seg), e 3) o tempo de incubação solução de lise (5 vs 30 min). A 30 minutos de incubação a lise celular rendimento aumentado por uma média de 3,5% (p <0,01), mas nenhuma outra variável a lise celular teve um efeito significativo sobre o rendimento. Vórtex diminuiu a proporção 260/280 por um estatisticamente significativa (p <0,001), mas praticamente pequeno 0,03.
Precipitação de proteína (passo 4) é precedido por proteinase K de digestão para interromper as cadeias de aminoácidos, a melhoria da eficiência de precipitação de proteína e que libertem de ADN capturado. A quantidade de Proteinase K foi variado de dez vezes.Temperatura de centrifugação foi reduzido de 20 ° C a 4 ° C. Aumentar a quantidade de Proteinase K causou uma diminuição estatisticamente significativa na produtividade (8,7%) e também um pouco melhorou tanto as razões 260/280 e 260/230.
Precipitação com etanol (passo 5) foi a última etapa do protocolo de análise. A quantidade de transportador de glicogénio (0, 8 mL) foi variada, como foi o tempo total de centrifugação (5 min 11 vs 30). Só o tempo de centrifugação afetou significativamente o rendimento, com um aumento médio de 290%. O tempo de centrifugação também diminuiu a proporção 260/280 ligeiramente (0,05). Nenhum efeito significativo sobre a produção de glicogênio foi observado durante os experimentos; embora a quantidade total de ADN nestas extracções foi suficientemente grande para que não seria glicogénio tipicamente utilizado. Apesar da falta de efeito nestas amostras, ainda é recomendável usar o glicogênio para minimizar o risco de uma diminuição do rendimento, sempre que o volume de entrada de saliva é menor do que dado aqui ou se there qualquer outra razão para acreditar que o rendimento será baixo.
A inspecção visual das amostras representativas de ADN (Figura 1) indica que o ADN extraído não foi grandemente fragmentada por qualquer processo de extracção de ADN de saliva, mas mostrou uma banda de elevado peso molecular apropriado sem manchas indicativo de ADN degradado. Após a digestão com ARNase A, o protocolo produziu uma média de 260/280 1.74.
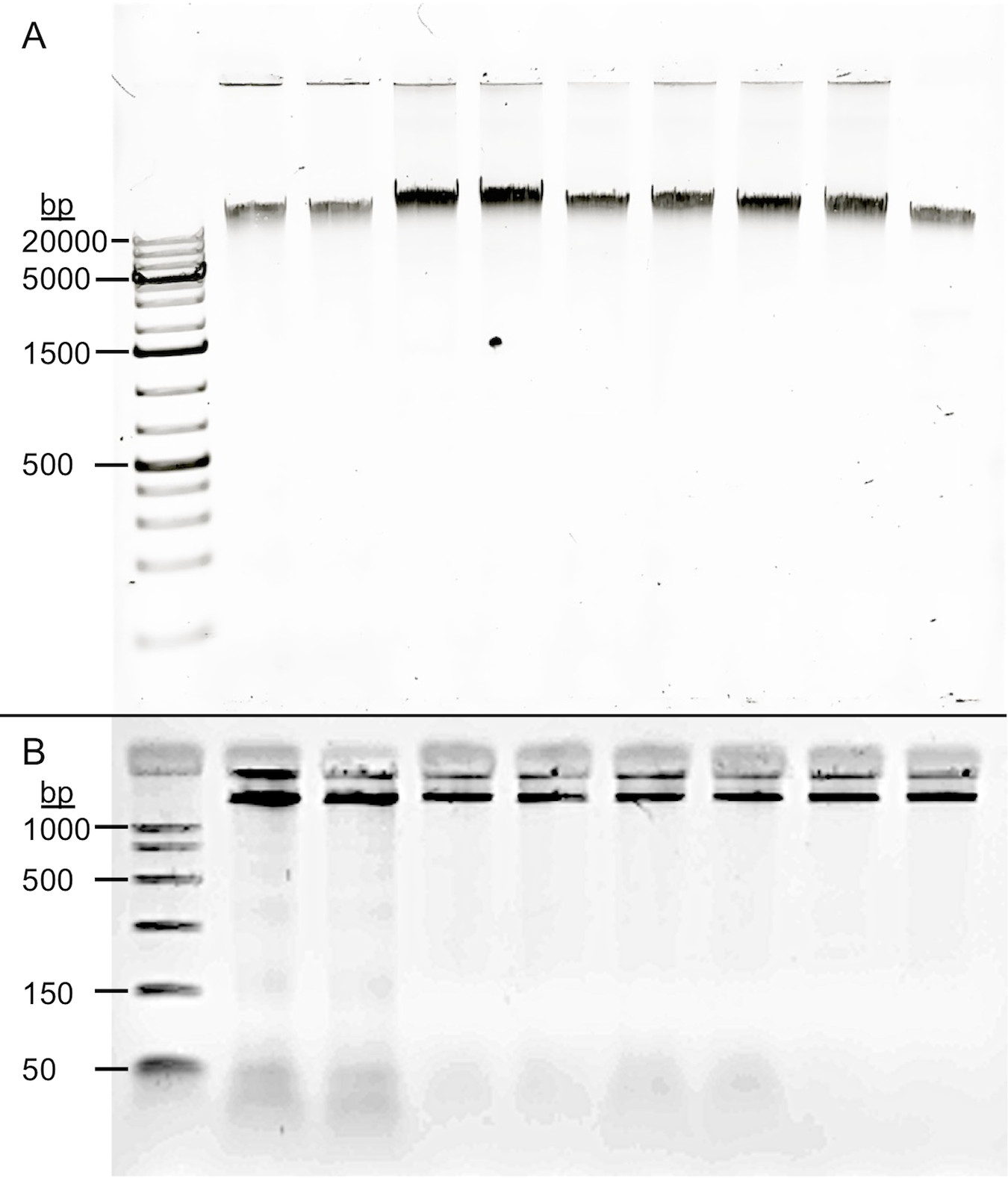
Figura 1 Qualidade de saliva DNA derivado. Quatro procedimentos de extração foram aplicados à mesma coleta de saliva. (A) As amostras foram sujeitas a electroforese num gel de agarose a 0,8% (250 ng de ADN). Todas as variações dos protocolos de extracção de ADN de saliva resulta em elevado peso molecular (> 20 kb) de ADN, com nenhuma evidência de degradação. Lane: 1 DNA ladder, 2 e 3 amostras Oragene prepIT L2P protocolo, 4 e 5 Gentra Puregene Líquidos Corporais Protocolo, 6 e 7 do protocolo otimizado sem etapa de remoção de RNA, 8 e 9 do protocolo otimizado com a etapa de RNA de remoção. As pistas 2-7 são directamente protocolos análogos nas mesmas amostras de saliva. As pistas 8 e 9 mostram que o passo de remoção de ARN não introduz degradação de ADN. (B) As amostras foram sujeitas a electroforese num gel de agarose a 2% (150 ng de ADN). Um ligeiro pico de ARN é observável perto da parte inferior do gel nas pistas 2 a 7 (convenções como acima). As pistas 8 e 9 mostram a eficácia da etapa de remoção de ARN.
O passo de remoção de ARN (passo 3, com RNase A) é crítica para a quantificação exacta de ADN. Durante os testes, o conteúdo de ARN foi observada consistentemente elevada, tal como determinado pela proporção de ADN de cadeia dupla com o ARN medido por um Qubit 2,0 Fluorometer. Por, o teor de ácido nucleico média de amostras sem RNase Um tratamento consistiu em 46,6% (± 0,4) RNA. As amostras que foram submetidos a remoção de RNA Passo lido como "<20 ng / ml", que é a leitura mais baixa possível para detecção do RNA do Qubit.
O DNA obtido através deste protocolo otimizado era de qualidade suficiente para alto rendimento de sequenciamento quando a RNase adicional Um passo foi aplicado. Para obter dados Resequencing alvo, um costume kit Agilent SureSelect Alvo enriquecimento foi aplicado a 24 amostras, visando 2.6 Mb de sequência. Alta taxa de transferência de sequenciamento foi realizado em 12 (indexados) amostras com código de barras por pista. Sequência lê foram BWA alinhado ao genoma de referência hg19 12, então a aplicação de recalibração qualidade GATK 13 base de pontuação, indel realinhamento, remoção duplicado, descoberta SNP e genotipagem simultaneamente em todas as 24 amostras foi realizada utilizando o melhor parâmetro de filtragem duro prática valoriza 14. Todas as 24 amostras produziram alta qualidade NGS dados (Tabela 2). De leituras que passou Illuda mina filtros padrão e teve Q> 20, 91,4% alinhados com as regiões-alvo seqüência de enriquecimento, proporcionando uma profundidade média on-meta de cobertura de> 30x cobertura a Q> 100, bem dentro dos limites necessários para a rara descoberta SNP em cada amostra. O balanço de cadeia média foi de 49,9%. Comparando as chamadas variantes com genótipos de microarray Illumina rendeu uma concordância de 98,9%.
| Variável Candidate | ng / mL | 260/280 | 260/230 |
| Vortex | ns | -0,03 *** | ns |
| x30 min Lise Celular Incubação | 3,5% ** | ns | ns |
| Proteinase K x10 | -8,7% * | -0,05 *** | -0,03 *** |
| 30 min de centrifugação | 290,2%*** | -0,05 *** | ns |
| Glicogênio | ns | ns | -0,37 *** |
Tabela 1 Efeito Tamanho Variável Optimizado na quantidade Metrics / Qualidade. Todos os tamanhos de efeito estão nas unidades listadas no cabeçalho da coluna. p-valores de ANOVA: * p <0,05; ** P <0,01; *** P <0,001; NS não significativo
| Métrica de Qualidade | Valor |
| Comprimento de destino | 1.708 kb |
| Alvo Coberto> 30x | 85,22% |
| SNPs em dbSNP | 92,55% |
| Acordo Matriz | 98,99% |
Tabela 2 Qualidade de High Throughput Sequência da Target Enriquecimento.
| Novo protocolo otimizado | Item | Distribuidor | Catalog # | Unidade de Compras | Custo / Unit | Custo / Colecção |
| 15 ml de tubos de centrífuga | Fisher | 12-565-268 | 500 Tubos | $ 233,50 | $ 3,2690 | |
| Solução de Lise Celular | Qiagen | 158908 | 1 L | $ 401,00 | $ 3,2080 | |
| Proteinase K | Sigma | P6556 | 1 g de | $ 713,00 | $ 1,5686 | |
| Solução Protein Precipitation | Qiagen | 158912 | 350 ml | $ 350,00 | $ 2,7200 | |
| Isopropanol | Fisher | A416-4 | Caso de 4 x 4 L | $ 486,71 | 0,2434 dólares | |
| Solução de glicogénio (20 mg / ml) | EZ-BioResearch | S1003 | 1 ml | $ 51,00 | $ 0,8160 | |
| 70% de Etanol | Fisher | 04-355-305 | Caso de 4 x 1 gal. | $ 123,03 | 0,0325 dólares | |
| Tris-EDTA (TE) | Fisher | BP2473-1 | 1 L | $ 68,67 | $ 0,0412 | |
| NaCl | Fisher | AC194090010 | 1 kg | $ 34,65 | $ 0.000004 | |
| Tris HCl | Fisher | BP1757-100 | 100 ml | $ 57,84 | $ 0,0116 | |
| EDTA (0,5 M) Solução | Fisher | 03-500-506 | 100 ml | $ 33,60 | $ 0,0134 | |
| Dod sódioecyl Sulfato | Fisher | BP166-100 | 100 g | $ 59,95 | $ 0,0060 | |
| Custo total | $ 11,93 | |||||
| Oragene | Item | Distribuidor | Catalog # | Unidade de Compras | Custo / Unit | Custo / Colecção |
| 15 ml de tubos de centrífuga | Fisher | 12-565-268 | 500 Tubos | $ 233,50 | $ 0,9340 | |
| 100% de etanol | Fisher | BP2818-100 | 100 ml | $ 34,29 | $ 1,6459 | |
| 70% de Etanol | Fisher | 04-355-305 | Caso de 4 x 1 gal. | $ 123,03 | 0,0081 dólares | |
| Tris-EDTA (TE) | Fisher | BP2473-1 | 1 L | $ 68,67 | 0,0687 dólar | |
| Tubo de 1,5 ml | Genesee | 22-281A | 500 Tubos | $ 22,85 | $ 0,0457 | |
| Oragene Coleção KIT | Oragene | OG-500 | 1 | $ 25,00 | $ 25,00 | |
| Custo total | $ 27,70 | |||||
| Puregene | Item | Distribuidor | Catalog # | Unidade de Compras | Custo / Unit | Custo / Colecção |
| 15 ml de tubos de centrífuga | Fisher | 12-565-268 | 500 Tubos | $ 233,50 | $ 0,9340 | |
| Solução de Lise Celular | Qiagen | 158908 | 1 L | $ 401,00 | $ 4,0100 | |
| Proteinase K | Qiagen | 158918 | 650 ml | $ 73,10 | $ 7,8723 | |
| Solução Protein Precipitation | Qiagen | 158912 | 350 ml | $ 350,00 | $ 4,0000 | |
| Isopropanol | Fisher | A4164 | Caso de 4 x 4 L | $ 486,71 | $ 0,3650 | |
| Solução de glicogênio | Qiagen | 158930 | 500 ml | $ 64,30 | $ 2,5720 | |
| 70% de Etanol | Fisher | 04-355-305 | Caso de 4 x 1 gal. | $ 123,03 | 0,0975 dólares | |
| Solução ADN Hidratação | Qiagen | 158914 | 100 ml | $ 69,80 | 0,1396 dólar | |
| NaCl | Fisher | AC19409-0010 | 1 kg | $ 34,65 | $ 0.000004 | |
| Tris HCl | Fisher | BP1757-100 | 100 ml | $ 57,84 | $ 0,0116 | |
| EDTA (0,5 M) Solução | Fisher | 03-500-506 | 100 ml | $ 33,60 | $ 0,0134 | |
| Dodecilsulfato de sódio | Fisher | BP166-100 | 100 g | $ 59,95 | $ 0,0060 | |
| Custo total | $ 19,99 |
Tabela 3 Comparação dos custos do protocolo optimizado para extrair o ADN a partir de 2 ml de saliva com outros protocolos comercialmente disponíveis. UMll reagentes e consumíveis necessários para a extração de DNA foram avaliados utilizando os preços de tabela padrão disponíveis na internet a partir de 30 de setembro de 2013 Note-se que este protocolo é escrito para extrair DNA de 1,25 ml de saliva total (ou seja, 2,5 ml de saliva e tampão , veja o passo 2.3) quanto esse valor representa metade do volume total do tubo de coleta de saliva. Para a comparação de custos, de 2 ml foi escolhido porque este é o valor associado com um kit disponível comercialmente comum (Oragene).
| ADN Estabilização tampão (250 ml) | ||
| Componente | Volume (ml) | [Final] |
| NaCl 1 M | 1.461 g | 0,1 M |
| Tris HCl | 2,5 | 0,01 M |
| EDTA 0,5 M | 5 | 0,01 M |
| 10% de SDS | 12,5 | 0,014 M |
| Solução de Proteinase K (20 mg / ml) | 2,5 | 6.92x10 -6 M |
| DDH 2 O | 227,5 | |
| Solução de Proteinase K (20 mg / ml) | ||
| Componente | Volume (ml) | [Final] |
| Proteinase K pó | 500 mg | 6.92x10 -4 M |
| DDH 2 O | 25 | |
| 70% de etanol (500 ml) | ||
| Componente | Volume (ml) | [Final] |
| Etanol 95% | 368,5 | 12,63 M |
| DDH 2 O | 131,5 | |
Tabela 4 Receitas para Reagentes.
Discussão
O presente procedimento é um protocolo de extração de DNA otimizado, que melhorou consideravelmente o rendimento de DNA de alto peso molecular em comparação com os métodos convencionais, sem comprometer a qualidade do DNA. O passo crítico com o maior efeito sobre o rendimento mais foi o passo 5.2, que inclui uma etapa de centrifugação durante a precipitação mais etanol do que qualquer protocolo publicado aqui analisados, com exceção de um que não foi amplamente distribuído 11. Nenhuma alteração na qualidade do ADN associada a este longo centrifugação foram detectados, o que indica que a maior parte do ADN disponível a partir de recolha de saliva total não é degradado e de elevado peso molecular.
Coleta de saliva todo tem limitações na qualidade da amostra, tais como o potencial de contaminantes externos, que precisa ser minimizado na fase de cobrança, bem como a presença de excesso de proteína na amostra que pode ser um sinal de uma infecção subjacente. Grandes quantidades de proteína ou estrangeiras contaminantes podempermanecem no final de ADN extraído tornando impreciso quantificação. Se existem proteínas ou de contaminantes residuais após re-hidratação (passo 6) são suspeitos, uma limpeza da amostra pode ser realizada, iniciando no passo de precipitação de proteína com os volumes de reagente dimensionadas de modo a reflectir o volume de entrada de amostra. Outra limitação de que o protocolo é o período de tempo necessário para realizar os passos. Várias amostras podem ser executados em paralelo; no entanto, recomenda-se que não mais de 24 extrações paralelas ser executados simultaneamente. Isto é particularmente importante para o passo de precipitação de proteínas, em que a amostra deve permanecer frio para assegurar um sedimento apertado e rodando mais de 24 amostras pode permitir peletes tempo de re-dissolver.
O protocolo aqui apresentado é o custo mais eficaz método considerado (ver Tabela 3). Embora este protocolo faz uso de reagentes do kit de extração Puregene, um volume menor do que o recomendado no protocolo Puregene é usadosem comprometer o rendimento de extração e é esta redução reagentes que impulsiona a economia de custos relativos ao protocolo. Note-se que o custo calculado para a extração usa o preço de tabela para cada item e não reflete quaisquer descontos. O custo por extração com o protocolo otimizado pode ser reduzido ainda mais com grandes encomendas ou descontos através de representantes de vendas da empresa.
A evidência também tem sido, desde que este protocolo é adequado para utilização com a próxima geração de sequenciação. Os dados obtidos proporciona uma evidência adicional da utilidade das amostras de saliva para a investigação genética humana em muitas doenças em que o sangue não é rotineiramente disponíveis. Enquanto o sangue e células linha DNA derivado continua a ser a fonte preferida de material genético para o teste, a coleta da saliva todo é uma alternativa viável quando essas fontes não estão disponíveis, quando paciente inscrição é afetada por sua coleção ou flebotomia não está disponível ou impraticáveis.
Divulgações
The authors have nothing to disclose.
Agradecimentos
This work was funded by a National Institutes of Health R01 (DC009453 support to CWB).
Materiais
| Name | Company | Catalog Number | Comments |
| 15 ml Centrifuge Tubes | Fisher | 12-565-268 | |
| Cell Lysis Solution | Qiagen | 158908 | |
| Proteinase K | Sigma | P6556 | |
| Protein Precipitation Solution | Qiagen | 158912 | |
| Isopropanol | Fisher | A416-4 | |
| Glycogen | EZ-BioResearch | S1003 | |
| 70% Ethanol | Fisher | 04-355-305 | |
| Tris-EDTA (TE) | Fisher | BP2473-1 | |
| NaCl | Fisher | AC194090010 | |
| Tris HCl | Fisher | BP1757-100 | |
| EDTA (0.5 M) Solution | Fisher | 03-500-506 | |
| Sodium Dodecyl Sulfate | Fisher | BP166-100 | |
| Analog Vortex Mixer | Fisher | 02-215-365 | |
| Centrifuge 5810R | Eppendorf | 5811 000.010 |
Referências
- Quinque, D., Kittler, R., Kayser, M., Stoneking, M., Nasidze, I. Evaluation of saliva as a source of human DNA for population and association studies. Analytical Biochemistry. 353, 272-277 (2006).
- Min, J. L., et al. High mircosatellite and SNP genotyping success rates established in a large number of genomic DNA samples extracted from mouth swabs and genotypes. Twin Research and Human Genetics. 9, 501-506 (2006).
- Dlugos, D. J., Scattergood, T. M., Ferraro, T. N., Berrettinni, W. H., Buono, R. J. Recruitment rates and fear of phlebotomy in pediatric patients in a genetic study of epilepsy. Epilepsy & Behavior. 6, 444-446 (2005).
- Etter, J. F., Neidhart, E., Bertand, S., Malafosse, A., Bertrand, D. Collecting saliva by mail for genetic and cotinine analyses in participants recruited through the internet. European Journal of Epidemiology. 20, 833-838 (2005).
- Hansen, T. V., Simonsen, M. K., Nielsen, F. C., Hundrup, Y. A. Collection of blood, saliva, and buccal cell samples in a pilot study on the danish nurse cohort: Comparison of the response rate and quality of genomic DNA. Cancer Epidemiol Biomarkers Prev. 16, 2072-2076 (2007).
- Van Schie, R. C. A. A., Wilson, M. E. Saliva: A convenient source of DNA for analysis of bi-allelic polymorphisms of fcγ receptor iia (cd32) and fcγ receptor iiib (cd16). Journal of Immunological Methods. 208, 91-101 (1997).
- Dawes, C. Estimates, from salivary analyses, of the turnover time of oral mucosal epithelium in humans and the number of bacteria in an edentulous mouth. Archives of Oral Biology. 48, 329-336 (2003).
- Bahlo, M., et al. Saliva-derived DNA performs well in large-scale, high-density single-nucleotide polymorphism microarray studies. Cancer Epidemiol Biomarkers Prev. 19, 794-798 (2010).
- Hu, Y., et al. Genotyping performance between saliva and blood-derived genomic dnas on the dmet array: A comparison. PLoS ONE. 7 (3), e33968(2012).
- Simmons, T. R., et al. Increasing genotype-phenotype model determinism: Application to bivariate reading/language traits and epistatic interactions in language-impaired families. Human Heredity. 70, 232-244 (2010).
- Zeugin, J. A., Hartley, J. L. Ethanol precipitation of DNA. Focus. 7, 1-2 (1985).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler Transform. Bioinformatics. 25, 1754-1760 (2009).
- McKenna, A., et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res. 20, 1297-1303 (2010).
- DePristo, M., et al. A framework for variation discovery and genotyping using next-generation DNA sequencing data. Nature Genetics. 43, 491-498 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados