Method Article
Recolección y extracción de ADN de la saliva de Next Generation Sequencing
En este artículo
Resumen
DNA extraction from saliva can provide a readily available source of high molecular weight DNA, with little to no degradation/fragmentation. This protocol provides optimized parameters for saliva collection/storage and DNA extraction to be of sufficient quality and quantity for downstream DNA assays with high quality requirements.
Resumen
The preferred source of DNA in human genetics research is blood, or cell lines derived from blood, as these sources yield large quantities of high quality DNA. However, DNA extraction from saliva can yield high quality DNA with little to no degradation/fragmentation that is suitable for a variety of DNA assays without the expense of a phlebotomist and can even be acquired through the mail. However, at present, no saliva DNA collection/extraction protocols for next generation sequencing have been presented in the literature. This protocol optimizes parameters of saliva collection/storage and DNA extraction to be of sufficient quality and quantity for DNA assays with the highest standards, including microarray genotyping and next generation sequencing.
Introducción
La obtención de ADN de alta calidad para los estudios genéticos humanos es esencial en el proceso de descubrimiento de genes de enfermedades. Blood, aunque requiere un procedimiento invasivo y también de ser más caro que la recolección de saliva, se ve favorecida por la creación de líneas celulares inmortalizadas como una fuente infinita de ADN, o iPSCs para estudios funcionales, y algunas veces de ADN de sangre se utiliza cuando las líneas celulares no están disponibles. Sin embargo, la obtención de la sangre requiere un flebotomista capacitado y la sangre tiene una vida media más corta que la saliva 1. ADN de la saliva es menos costoso y más fácil de obtener, ya que puede ser recogida y enviada a través del correo electrónico sin la necesidad de un flebotomista, aumentando de este modo posibles piscinas temáticas mucho más allá de la zona de influencia de los hospitales y los laboratorios 2. Estudio de inscripción puede ser mejorado cuando los sujetos tienen la opción de dar una muestra de saliva en lugar de sangre 3, 4. Las preocupaciones sobre la cantidad y calidad del ADN de la saliva pueden haber limitado su nos generalizadae pesar de numerosos estudios estudios recientes que demuestran la idoneidad de la saliva total, con un promedio de 4,3 x 10 5 células por mililitro, para pruebas de ADN a través de los métodos de hisopos bucales mayores que no obtuvieron cantidades significativas de saliva 2, 3, 4, 5, 6. Mientras que existe una literatura modesta que muestra la idoneidad de ADN derivado de la saliva entera para aplicaciones de genotipificación incluyendo métodos basados en microarrays 8, 9, la secuenciación de próxima generación 10, ningún estudio ha examinado (NGS). El objetivo de la optimización de este protocolo de extracción de ADN de la saliva entera era maximizar la cantidad y calidad de las aplicaciones de la genética en una manera rentable que se implementa fácilmente en laboratorios con reactivos y consumibles comunes.
Extracción de ADN de la saliva requiere de varios procedimientos: 1) la recolección y almacenamiento, 2) la lisis celular, 3) tratamiento con RNasa, 4) la precipitación de proteínas, 5) la precipitación con etanol, 6) rehidratación ADN. La solución de ADN de Estabilización de Buffer, se describe2 anteriormente, las funciones adecuadamente sin alteración. No se hizo ningún intento de optimizar el tratamiento con RNasa y medidas de rehidratación de ADN. Para cada paso restante, se identificaron diversas variables que pueden afectar el rendimiento. Cada variable fue manipulado de forma individual y la mejora en el rendimiento y la calidad se evaluó estadísticamente. Para las variables que se mostraron para mejorar el rendimiento y / o calidad del ADN, se incluyeron los valores óptimos en el protocolo final.
Protocolo
NOTA: Antes de proporcionar muestras de saliva todos los sujetos dieron su consentimiento informado se ajuste a las directrices para el tratamiento de los sujetos humanos en Hospital Nacional de Niños.
1. Saliva Collection y Almacenamiento
- Antes de la toma de saliva, asegúrese de que la boca del sujeto es libre de alimentos u otras sustancias extrañas al tener el tema enjuague su boca con agua y evitando comer o beber durante 30 minutos antes de recoger la muestra.
- Abrir un tubo de centrífuga de 15 ml con 2,5 ml de tampón de ADN estabilización 2 asegurándose de no tocar la parte interior de la tapa o tubo, y haga que el sujeto escupió 2,5 ml de saliva en la solución tampón. Nota: Recogida de más de 2,5 ml de saliva puede conducir a la degradación de la muestra a partir de una relación insuficiente de la muestra a un tampón de estabilización de ADN. Recopilación demasiado poco de saliva reducirá los rendimientos esperados de protocolo. Para evaluar los volúmenes de recogida, utilice los gradientes numerados en el lado de tél tubo.
- Reemplace la tapa y mezclar por inversión hasta que se homogeneiza la mezcla. Agitación vigorosa no es necesario. Almacenar las muestras a temperatura ambiente para el almacenamiento a corto plazo o 4 ° C para el almacenamiento a largo plazo (> 3 meses).
2. Preparativos iniciales y lisis celular
- Antes de iniciar la extracción, calentar un baño de agua a 37 ° C, y preparar un cubo de hielo. Se necesitan tres tubos de 15 ml de centrífuga cónicos para cada muestra extraída. Los tres tubos se utilizan para mantener la célula y la proteína de pellets, la final gDNA extraídos, y los sobrenadantes isopropanol y etanol.
- Recuperar las muestras de almacenamiento, e invertir las muestras varias veces luego vórtice a velocidad media durante 15 segundos.
- Dispense 2.5 ml de muestra en un tubo de centrífuga de 15 ml limpio, y añadir 5 ml de solución de lisis celular. Mezclar la muestra 50 veces por inversión, e incubar a TA durante 30 min.
Remoción 3. ARN
- Añadir 40 l de RNasa A de Solution a 100 mg / ml, y se incuba a 37 ° C durante 15 min.
- Sacar la muestra del baño a 37 ° C y enfriar en hielo durante 3 min.
- Después de la incubación RNasa A, aumentar la temperatura del baño de agua a 65 ° C para la etapa de rehidratación ADN del protocolo.
4. Proteína y extracción de lípidos
- Añadir 50 l de solución de proteinasa K a 20 mg / ml, mezclar varias veces por inversión, y se incuba a temperatura ambiente durante un mínimo de 30 min. Nota: Este es un posible punto de pausa para el protocolo. Después de la adición de la solución de proteinasa K, la muestra puede ser almacenada a 4 ° C hasta la extracción puede ser completada. El almacenamiento a 4 ° C durante un máximo de 24 horas no ha demostrado tener un efecto significativo en los rendimientos de extracción o la calidad del ADN. El almacenamiento a largo plazo en esta etapa no ha sido evaluada.
- Añadir 1,7 ml de solución de Precipitación de Proteínas y agitar vigorosamente durante 20 segundos a alta velocidad, y el lugar en hielo durante 10 min.
- Una vez que las muestras se han enfriado en hielo durante 10 min, se centrifuga durante 10 min a 3.000 xg y 4 ° C. Las proteínas precipitadas deben formar una bolita apretada para continuar. Si la pastilla no es apretado o la solución es aún turbia, las muestras se pueden enfriar en hielo durante 5 min más y centrifugación repetida. Las muestras deben mantenerse en hielo para asegurar una bolita apretada.
5. Aislamiento y purificación de ADNg
- En un tubo de centrífuga de 15 ml limpio, pipeta de 5 ml de isopropanol y 8 l de solución de glucógeno puro a 20 mg / ml.
- Verter el sobrenadante que contiene el ADNg de la etapa 4.3 en el tubo que contiene la solución de isopropanol y el glucógeno, dejando atrás el sedimento de proteína precipitada. Una vez que el sobrenadante se ha añadido, mezclar suavemente la muestra 50 veces por inversión y centrifugar durante 30 minutos a 3.000 xg y 4 ° C.
- Verter lentamente el sobrenadante en un tubo de 15 ml limpio. Después de la eliminación del sobrenadante, añadir 1 ml de etanol al 70% alavar el pellet oscilando lentamente y moviendo suavemente el etanol sobre el sedimento precipitado varias veces. Conservar el etanol en el tubo.
- Después del lavado inicial, centrifugar la muestra durante 1 min a 2.000 xg y 20 ° C. Esta etapa de centrifugación se puede hacer en cualquiera de 4 ° C o 20 ° C. Ningún efecto significativo de la temperatura se ha demostrado para este paso.
- Después del lavado y la centrifugación inicial de la pastilla, verter lentamente el lavado con etanol del tubo y desechar, a continuación, realizar un segundo lavado repitiendo los pasos 5.3 y 5.4.
- Después de la eliminación del sobrenadante del segundo lavado, permitir que el pellet se seque al aire durante 15 min.
- Si la muestra no se ha secado completamente, secar al aire durante otros 15 min.
6. rehidratación de gDNA
- Una vez que la muestra se ha secado, añadir 300 l de Tris-EDTA para rehidratar el sedimento gDNA seca.
- Vortex la muestra durante 5 segundos a velocidad media y el lugar en un65 ° C baño de agua caliente durante 1 hora.
- Retire las muestras del baño de agua y se incuban O / N a temperatura ambiente.
NOTA: Todos los productos y reactivos utilizados se enumeran en la Tabla de materiales, así como la Tabla 4.
Resultados
Para determinar los parámetros óptimos para la extracción de ADN se realizó una serie de extracciones de ADN emparejados. Una sola muestra de saliva se dividió y cada porción probado con uno de dos valores posibles para una variable dada. Se realizaron al menos ocho repeticiones de cada prueba de pares (por ejemplo, una sola muestra de saliva se dividió en alícuotas para probar la extracción con y sin inicial 50 ° C de incubación). Optimización se basó en cuatro métricas estándar: rendimiento total del ADN, el valor 260/280, 260/230 el valor, y la inspección visual de ADN a electroforesis para evaluar la fragmentación. No todas las combinaciones posibles de las variables se evaluaron las interacciones estadísticos (N = 169 combinaciones), optando en su lugar para evaluar el efecto marginal de cada variable individualmente. Efectos se ensayaron usando un multi-modo de medidas repetidas ANOVA y efectos estimados se derivaron de la ecuación de regresión equivalente. Todos los efectos significativos se resumen en la Tabla 1, mostrada como avercambio de edad en el rendimiento (ng / l por ml de saliva de entrada) o la calidad del ADN (260/280 y 260/230).
La lisis celular (paso 2) se optimizó mediante la evaluación de: 1) la presencia / ausencia de una incubación de 50 ° C (1 hora) antes de la lisis celular para asegurar que la degradación de la proteinasa K y la lisis celular mediada por el tampón de almacenamiento se completó, 2 ) la presencia / ausencia de una homogeneización por agitación paso (velocidad media, 15 sec), y 3) tiempo de incubación de la solución de lisis (5 frente a 30 min). El 30 min de incubación lisis celular incrementó el rendimiento en un promedio de 3,5% (p <0,01), pero ninguna otra variable de la lisis celular tuvo un efecto significativo en el rendimiento. Vórtex disminuyó la proporción 260/280 por una estadísticamente significativa (p <0,001), pero prácticamente pequeño 0.03.
Proteína precipitación (paso 4) está precedida por Digestión con proteinasa K para interrumpir las cadenas de aminoácidos, mejorar la eficiencia de la precipitación de proteínas y la liberación de ADN capturado. La cantidad de proteinasa K era variado diez veces.Temperatura La centrifugación se redujo de 20 ° C a 4 ° C. Aumentar la cantidad de proteinasa K causó una disminución estadísticamente significativa en el rendimiento (8,7%) y también ligeramente mejoradas tanto los 260/280 y 260/230 ratios.
Precipitación con etanol (etapa 5) fue la última etapa del protocolo examinado. La cantidad de vehículo glucógeno (0, 8 l) se varió, como lo fue el tiempo total de centrifugación (5 frente a 30 min 11). Sólo el tiempo de centrifugación afectó significativamente el rendimiento, con un incremento medio del 290%. El giro ya también disminuyó la proporción ligeramente (0,05) 260/280. No se observó ningún efecto significativo de glucógeno en el rendimiento durante los experimentos; aunque la cantidad total de ADN en estas extracciones era suficientemente grande para que el glucógeno no se utilizaría normalmente. A pesar de la falta de efecto en estas muestras, todavía se recomienda utilizar el glucógeno para minimizar el riesgo de una disminución del rendimiento cuando el volumen de entrada de la saliva es más baja que se da aquí o si there es cualquier otra razón para creer rendimiento será bajo.
La inspección visual de las muestras de ADN representativos (Figura 1) indicó que el ADN extraído no se fragmentó en gran medida por cualquier procedimiento de extracción de ADN de saliva, sino que mostró una banda de alto peso molecular apropiado sin la mancha indicativa de ADN degradado. Después de digestión con RNasa A, el protocolo produjo un promedio de 260/280 1.74.
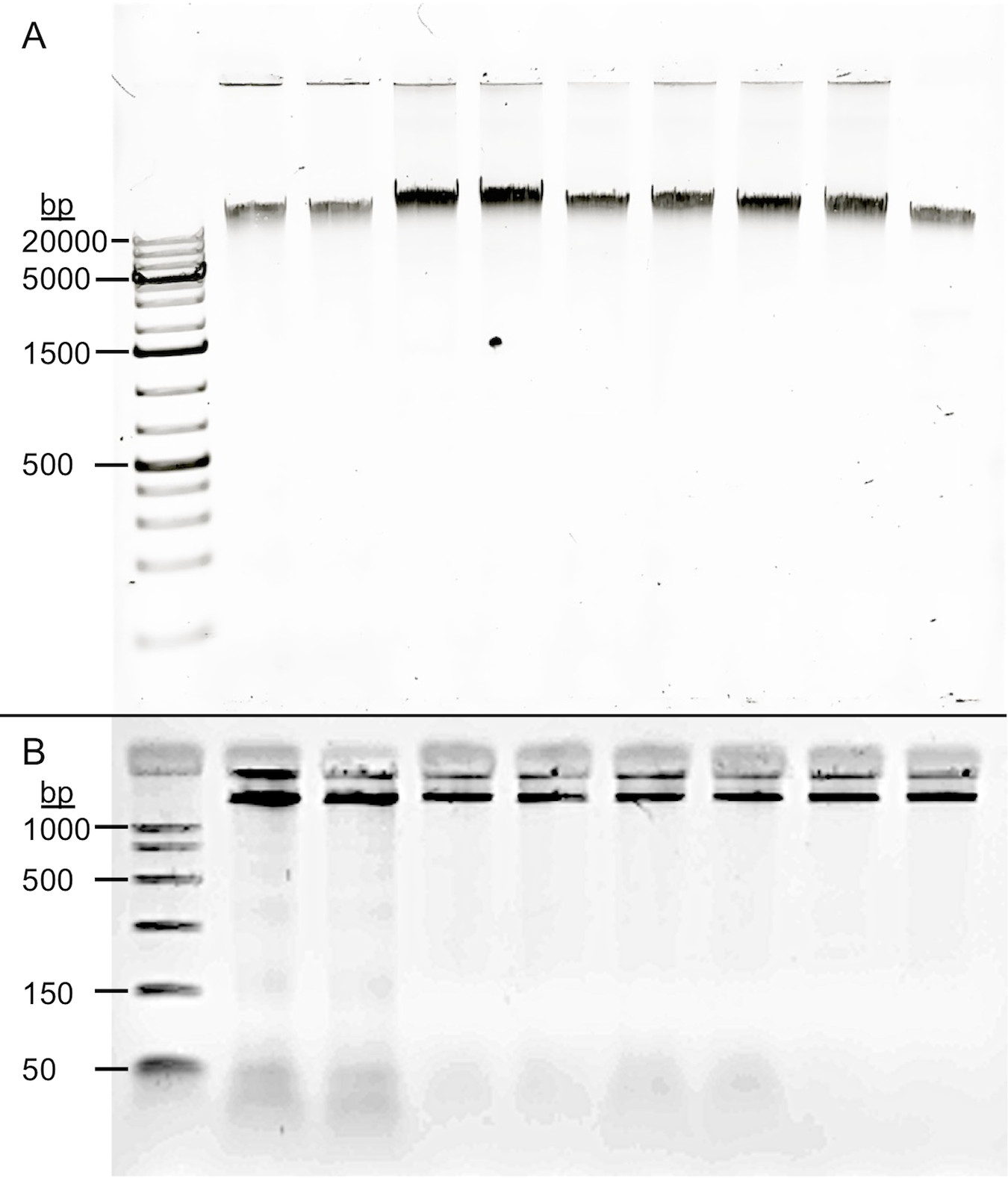
Figura 1. Calidad de ADN derivado de la saliva. Cuatro procedimientos de extracción se aplicaron a la misma colección saliva. Las muestras (A) se sometieron a electroforesis en un gel de agarosa al 0,8% (250 ng de ADN). Todas las variaciones de los protocolos de extracción de ADN resultan en la saliva de alto peso molecular (> 20 kb) de ADN, sin evidencia de degradación. Lane: 1 DNA ladder, 2 y 3 muestras de pozos previos Oragene Protocolo L2P, 4 y 5 Gentra Puregene Cuerpo Protocolo de Fluidos, 6 y 7 del protocolo optimizado y sin etapa de eliminación de ARN, 8 y 9 del protocolo optimizado con el paso de ARN remove. Los carriles 2-7 son directamente protocolos análogos en las mismas muestras de saliva. Los carriles 8 y 9 muestran que la etapa de eliminación de ARN no introduce degradación del ADN. (B) Las muestras se sometieron a electroforesis en un gel de agarosa al 2% (150 ng de ADN). Un pico ligero ARN es observable cerca de la parte inferior del gel en los carriles 2 a través de 7 (convenciones como arriba). Los carriles 8 y 9 muestran la eficacia de la etapa de eliminación de ARN.
La etapa de eliminación de ARN (paso 3 con RNasa A) es crítico para la cuantificación exacta de ADN. Durante las pruebas, se observó consistentemente alto contenido de ARN, tal como se determina por la relación de ADN de doble cadena de ARN a medida por un qubit 2,0 fluorómetro. El contenido de ácido promedio, nucleicos a partir de muestras sin RNasa A de tratamiento consistió en 46,6% (± 0,4) RNA. Las muestras que se sometieron a la etapa de eliminación de ARN leer como "<20 ng / ml", que es la lectura más baja posible para la detección de ARN del qubit.
El ADN obtenido a través de este protocolo optimizado era de calidad suficiente para la secuenciación de alto rendimiento cuando la RNasa adicional Se aplicó un paso. Para obtener los datos de resecuenciación dirigidos, un kit Agilent SureSelect Target Enrichment costumbre se aplicó a 24 muestras, apuntando a 2,6 Mb de secuencia. Alto rendimiento de secuenciación se llevó a cabo en 12 códigos de barras (indexados) muestras por carril. Secuencia lee fueron BWA-alineado con el genoma de referencia hg19 12, se llevó a cabo a continuación, la aplicación de recalibración calidad GATK 13 base de la puntuación, la realineación indel, eliminación de duplicados, el descubrimiento de SNP y genotipado de forma simultánea en todas las 24 muestras utilizando el mejor parámetro de filtrado dura práctica valora 14. Todas las 24 muestras dieron de alta calidad NGS datos (Tabla 2). De lecturas que pasó Illumina de los filtros estándar y tenía Q> 20, el 91,4% alineado a las regiones objetivo de enriquecimiento de secuencias, que proporciona una profundidad media en meta de cobertura de> 30 veces la cobertura en Q> 100, dentro de los límites necesarios para el descubrimiento de SNP raro en cada muestra. El equilibrio cadena media fue de 49,9%. La comparación de las llamadas variantes con Illumina microarrays genotipos produjeron una concordancia de 98,9%.
| Variable Candidato | ng / l | 260/280 | 260/230 |
| Vortex | ns | -0.03 *** | ns |
| x30 min de incubación de Lisis Celular | 3,5% ** | ns | ns |
| Proteinasa K x10 | -8,7% * | -0.05 *** | -0.03 *** |
| 30 min de centrifugado | 290.2%*** | -0.05 *** | ns |
| El glucógeno | ns | ns | -0.37 *** |
TABLA 1 Efecto de Tamaño Variable Optimizada en cantidad / calidad de métricas. Todos los tamaños del efecto se encuentran en las unidades que figuran en el encabezado de la columna. los valores de p de ANOVA: * p <0,05; ** P <0,01; *** P <0,001; ns no significativo
| Métricas de Calidad | Valor |
| Longitud designada | 1708 kb |
| Objetivo cubierto> 30x | 85.22% |
| SNPs en dbSNP | 92.55% |
| Acuerdo de matriz | 98,99% |
Tabla 2. Calidad de alto rendimiento secuencia de Target Enrichment.
| Protocolo optimizado Nueva | Artículo | Distribuidora | Catálogo # | Unidad de Compras | Costo / Unidad | Costo / Colección |
| 15 ml Tubos de centrífuga | Fisher | 12-565-268 | 500 Tubos | $ 233.50 | 3,2690 dólares | |
| Solución de lisis celular | Qiagen | 158908 | 1 L | $ 401.00 | 3,2080 dólares | |
| Proteinasa K | Sigma | P6556 | 1 g | $ 713.00 | 1,5686 dólares | |
| Solución Precipitación de Proteínas | Qiagen | 158912 | 350 ml | $ 350.00 | 2,7200 dólares | |
| Isopropanol | Fisher | A416-4 | Caja de 4 x 4 L | $ 486.71 | 0,2434 dólares | |
| Solución de glucógeno (20 mg / ml) | EZ-BioResearch | S1003 | 1 ml | $ 51.00 | 0,8160 dólares | |
| 70% de etanol | Fisher | 04-355-305 | Caja de 4 x 1 gal. | $ 123.03 | 0,0325 dólares | |
| Tris-EDTA (TE) | Fisher | BP2473-1 | 1 L | $ 68.67 | $ 0.0412 | |
| NaCl | Fisher | AC194090010 | 1 kg | $ 34.65 | $ 0.000004 | |
| Tris HCl | Fisher | BP1757-100 | 100 ml | $ 57.84 | 0,0116 dólares | |
| Solución de EDTA (0,5 M) | Fisher | 03-500-506 | 100 ml | $ 33.60 | 0,0134 dólares | |
| Dod sodioEcyl Sulfato | Fisher | BP166-100 | 100 g | $ 59.95 | 0,0060 dólares | |
| Coste total | $ 11.93 | |||||
| Oragene | Artículo | Distribuidora | Catálogo # | Unidad de Compras | Costo / Unidad | Costo / Colección |
| 15 ml Tubos de centrífuga | Fisher | 12-565-268 | 500 Tubos | $ 233.50 | 0,9340 dólares | |
| 100% de etanol | Fisher | BP2818-100 | 100 ml | $ 34.29 | 1,6459 dólares | |
| 70% de etanol | Fisher | 04-355-305 | Caja de 4 x 1 gal. | $ 123.03 | 0,0081 dólares | |
| Tris-EDTA (TE) | Fisher | BP2473-1 | 1 L | $ 68.67 | $ 0.0687 | |
| Tubo de 1,5 ml | Genesee | 22-281A | 500 Tubos | $ 22.85 | 0,0457 dólares | |
| Oragene Colección KIT | Oragene | OG-500 | 1 | $ 25.00 | $ 25.00 | |
| Coste total | $ 27.70 | |||||
| Puregene | Artículo | Distribuidora | Catálogo # | Unidad de Compras | Costo / Unidad | Costo / Colección |
| 15 ml Tubos de centrífuga | Fisher | 12-565-268 | 500 Tubos | $ 233.50 | 0,9340 dólares | |
| Solución de lisis celular | Qiagen | 158908 | 1 L | $ 401.00 | 4,0100 dólares | |
| Proteinasa K | Qiagen | 158918 | 650 ml | $ 73.10 | 7,8723 dólares | |
| Solución Precipitación de Proteínas | Qiagen | 158912 | 350 ml | $ 350.00 | 4,0000 dólares | |
| Isopropanol | Fisher | A4164 | Caja de 4 x 4 L | $ 486.71 | 0,3650 dólares | |
| Solución de glucógeno | Qiagen | 158930 | 500 ml | $ 64.30 | 2,5720 dólares | |
| 70% de etanol | Fisher | 04-355-305 | Caja de 4 x 1 gal. | $ 123.03 | 0,0975 dólares | |
| Solución de hidratación de ADN | Qiagen | 158914 | 100 ml | $ 69.80 | $ 0.1396 | |
| NaCl | Fisher | AC19409-0010 | 1 kg | $ 34.65 | $ 0.000004 | |
| Tris HCl | Fisher | BP1757-100 | 100 ml | $ 57.84 | 0,0116 dólares | |
| Solución de EDTA (0,5 M) | Fisher | 03-500-506 | 100 ml | $ 33.60 | 0,0134 dólares | |
| Dodecil sulfato de sodio | Fisher | BP166-100 | 100 g | $ 59.95 | 0,0060 dólares | |
| Coste total | $ 19.99 |
Tabla 3 Comparación de costes del protocolo optimizado para extraer el ADN de 2 ml de saliva total con otros protocolos disponibles comercialmente. Unll reactivos y consumibles necesarios para la extracción de ADN se han evaluado utilizando los precios de lista estándar disponibles en Internet a partir del 30 de septiembre de 2013 Tenga en cuenta que este protocolo está escrito para extraer ADN de 1,25 ml de saliva total (es decir, 2,5 ml de saliva y tampón , véase el paso 2.3) ya que este valor representa la mitad del volumen total en el tubo de recogida de saliva. Para la comparación de costos, 2 ml fue elegido como esta es la cantidad asociada con un kit disponible comercialmente común (Oragene).
| ADN Estabilización Buffer (250 ml) | ||
| Componente | Volumen (ml) | [Final] |
| 1 M NaCl | 1.461 g | 0,1 M |
| Tris HCl | 2.5 | 0,01 M |
| EDTA 0,5 M | 5 | 0,01 M |
| 10% de SDS | 12,5 | 0.014 M |
| Solución proteinasa K (20 mg / ml) | 2.5 | 6.92x10 -6 M |
| ddH2O | 227.5 | |
| Solución proteinasa K (20 mg / ml) | ||
| Componente | Volumen (ml) | [Final] |
| Proteinasa K en polvo | 500 mg | 6.92x10 -4 M |
| ddH2O | 25 | |
| 70% de etanol (500 ml) | ||
| Componente | Volumen (ml) | [Final] |
| Etanol 95% | 368.5 | 12.63 M |
| ddH2O | 131.5 | |
Cuadro 4 Recetas para reactivos.
Discusión
El presente procedimiento es un protocolo de extracción de ADN optimizada que ha mejorado considerablemente el rendimiento de ADN de alto peso molecular en comparación con métodos estándar, sin comprometer la calidad del ADN. El paso crítico con el mayor efecto sobre el rendimiento más fue paso 5.2, que incluye una etapa de centrifugación ya durante la precipitación de etanol que cualquier protocolo publicado revisado aquí, excepto que no estaba ampliamente distribuido 11. No se detectaron cambios en la calidad de ADN asociados con esta centrifugación más tiempo, lo que indica que la mayor parte del ADN disponible de la colección de saliva entera no es el peso molecular degradado y alta.
Recogida de saliva entera tiene limitaciones en la calidad de la muestra, tales como el potencial de contaminantes externos, que debe ser reducido al mínimo en la etapa de recolección, y la presencia de exceso de proteína en la muestra que puede ser un signo de una infección subyacente. Grandes cantidades de proteína o extranjeras contaminantes puedepermanecer en el final de ADN se extrajo con lo que la cuantificación inexacta. Si hay proteínas o contaminantes residuales después se sospecha de rehidratación (paso 6), una limpieza de la muestra puede realizarse comenzando en la etapa de precipitación de proteína con volúmenes de reactivos a escala para reflejar el volumen de entrada de la muestra. Otra limitación del protocolo es la longitud de tiempo requerido para realizar los pasos. Múltiples muestras se pueden ejecutar en paralelo; Sin embargo, se recomienda que no más de 24 extracciones paralelas pueden ejecutar simultáneamente. Esto es particularmente importante para la etapa de precipitación de proteínas, donde la muestra debe permanecer frío para garantizar un pellet apretado y ejecución de más de 24 muestras puede permitir pellets de tiempo para volver a disolver.
El protocolo que se presenta aquí es el método más rentable considerado (véase el cuadro 3). Si bien este protocolo hace uso de los reactivos del kit de extracción Puregene, se utiliza un volumen más pequeño de lo recomendado en el protocolo Puregenesin comprometer rendimiento de extracción y es esta reducción en los reactivos que impulsa el ahorro de costes en relación con el protocolo. Tenga en cuenta que el costo calculado para la extracción utiliza la lista de precios para cada artículo y no refleja ningún descuento. El coste por extracción con el protocolo optimizado puede reducirse aún más con las órdenes a granel o descuentos a través de representantes de ventas de la empresa.
La evidencia también ha previsto que este protocolo es adecuado para su uso con la secuenciación de próxima generación. Los datos obtenidos proporcionan una prueba más de la utilidad de las muestras de saliva para la investigación genética humana en muchas enfermedades en las que la sangre no está disponible habitualmente. Mientras que el ADN derivado de células de sangre y la línea continúa siendo la fuente preferida de material genético para la prueba, la recogida de saliva entera es una alternativa viable cuando tales fuentes no están disponibles, cuando la inscripción del paciente se ve afectada por su colección de flebotomía o no está disponible o poco práctico.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
This work was funded by a National Institutes of Health R01 (DC009453 support to CWB).
Materiales
| Name | Company | Catalog Number | Comments |
| 15 ml Centrifuge Tubes | Fisher | 12-565-268 | |
| Cell Lysis Solution | Qiagen | 158908 | |
| Proteinase K | Sigma | P6556 | |
| Protein Precipitation Solution | Qiagen | 158912 | |
| Isopropanol | Fisher | A416-4 | |
| Glycogen | EZ-BioResearch | S1003 | |
| 70% Ethanol | Fisher | 04-355-305 | |
| Tris-EDTA (TE) | Fisher | BP2473-1 | |
| NaCl | Fisher | AC194090010 | |
| Tris HCl | Fisher | BP1757-100 | |
| EDTA (0.5 M) Solution | Fisher | 03-500-506 | |
| Sodium Dodecyl Sulfate | Fisher | BP166-100 | |
| Analog Vortex Mixer | Fisher | 02-215-365 | |
| Centrifuge 5810R | Eppendorf | 5811 000.010 |
Referencias
- Quinque, D., Kittler, R., Kayser, M., Stoneking, M., Nasidze, I. Evaluation of saliva as a source of human DNA for population and association studies. Analytical Biochemistry. 353, 272-277 (2006).
- Min, J. L., et al. High mircosatellite and SNP genotyping success rates established in a large number of genomic DNA samples extracted from mouth swabs and genotypes. Twin Research and Human Genetics. 9, 501-506 (2006).
- Dlugos, D. J., Scattergood, T. M., Ferraro, T. N., Berrettinni, W. H., Buono, R. J. Recruitment rates and fear of phlebotomy in pediatric patients in a genetic study of epilepsy. Epilepsy & Behavior. 6, 444-446 (2005).
- Etter, J. F., Neidhart, E., Bertand, S., Malafosse, A., Bertrand, D. Collecting saliva by mail for genetic and cotinine analyses in participants recruited through the internet. European Journal of Epidemiology. 20, 833-838 (2005).
- Hansen, T. V., Simonsen, M. K., Nielsen, F. C., Hundrup, Y. A. Collection of blood, saliva, and buccal cell samples in a pilot study on the danish nurse cohort: Comparison of the response rate and quality of genomic DNA. Cancer Epidemiol Biomarkers Prev. 16, 2072-2076 (2007).
- Van Schie, R. C. A. A., Wilson, M. E. Saliva: A convenient source of DNA for analysis of bi-allelic polymorphisms of fcγ receptor iia (cd32) and fcγ receptor iiib (cd16). Journal of Immunological Methods. 208, 91-101 (1997).
- Dawes, C. Estimates, from salivary analyses, of the turnover time of oral mucosal epithelium in humans and the number of bacteria in an edentulous mouth. Archives of Oral Biology. 48, 329-336 (2003).
- Bahlo, M., et al. Saliva-derived DNA performs well in large-scale, high-density single-nucleotide polymorphism microarray studies. Cancer Epidemiol Biomarkers Prev. 19, 794-798 (2010).
- Hu, Y., et al. Genotyping performance between saliva and blood-derived genomic dnas on the dmet array: A comparison. PLoS ONE. 7 (3), e33968(2012).
- Simmons, T. R., et al. Increasing genotype-phenotype model determinism: Application to bivariate reading/language traits and epistatic interactions in language-impaired families. Human Heredity. 70, 232-244 (2010).
- Zeugin, J. A., Hartley, J. L. Ethanol precipitation of DNA. Focus. 7, 1-2 (1985).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler Transform. Bioinformatics. 25, 1754-1760 (2009).
- McKenna, A., et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res. 20, 1297-1303 (2010).
- DePristo, M., et al. A framework for variation discovery and genotyping using next-generation DNA sequencing data. Nature Genetics. 43, 491-498 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados
Utilizamos cookies para mejorar su experiencia en nuestra página web.
Al continuar usando nuestro sitio web o al hacer clic en 'Continuar', está aceptando nuestras cookies.