Method Article
Sammlung und Extraktion von DNA-Speichel für Next Generation Sequencing
In diesem Artikel
Zusammenfassung
DNA extraction from saliva can provide a readily available source of high molecular weight DNA, with little to no degradation/fragmentation. This protocol provides optimized parameters for saliva collection/storage and DNA extraction to be of sufficient quality and quantity for downstream DNA assays with high quality requirements.
Zusammenfassung
The preferred source of DNA in human genetics research is blood, or cell lines derived from blood, as these sources yield large quantities of high quality DNA. However, DNA extraction from saliva can yield high quality DNA with little to no degradation/fragmentation that is suitable for a variety of DNA assays without the expense of a phlebotomist and can even be acquired through the mail. However, at present, no saliva DNA collection/extraction protocols for next generation sequencing have been presented in the literature. This protocol optimizes parameters of saliva collection/storage and DNA extraction to be of sufficient quality and quantity for DNA assays with the highest standards, including microarray genotyping and next generation sequencing.
Einleitung
Erzielen hoher Qualität für die menschliche DNA genetische Studien ist wichtig, in der Krankheit Gen Entdeckungsprozess. Blut, obwohl erfordern ein invasives Verfahren und auch teurer als Speichelsammlung wird zum Erzeugen immortalisierter Zelllinien als eine unendliche Quelle von DNA oder iPSCs für funktionelle Studien bevorzugt, und manchmal Blut-DNA wird verwendet, wenn Zelllinien nicht vorhanden sind. , Blutgewinnung erfordert jedoch eine ausgebildete phlebotomist und Blut eine kürzere Halbwertszeit als Speichel 1 hat. DNA aus Speichel ist weniger teuer und leichter zu erhalten, da sie gesammelt und durch die Mail ohne die Notwendigkeit für eine Blutentnahme, wodurch die Versuchsperson Bäder weit über den Einzugsbereich der Krankenhäuser und Laboratorien 2. Studienaufnahme kann verbessert werden, wenn die Probanden die Möglichkeit zu geben eine Speichelprobe statt Blut 3, 4. Können Bedenken hinsichtlich der Quantität und Qualität der DNA aus dem Speichel seine weit verbreitete uns eingeschränkt habenTrotz zahlreicher Studien neueren Studien, die die Eignung der gesamte Speichel, mit einem Durchschnitt von 4,3 x 10 5 Zellen pro Milliliter, zum DNA-Test gegenüber den älteren Tupfern Verfahren, die signifikante Mengen an Speichel 2, 3, 4, 5 nicht erhalten hat e, 6. Während eine bescheidene Literatur existiert, die die Eignung der Gesamtspeichel abgeleiteten DNA für die Genotypisierung Anwendungen wie Microarray-basierte Methoden 8, 9, 10, keine Studien haben untersucht, Next Generation Sequencing (NGS). Das Ziel für die Optimierung dieses ganze Speichel DNA Extraktionsprotokoll war es, Menge und Qualität für Anwendungen in der Genetik einer kosteneffektiven Weise, die leicht in Labore mit gemeinsamen Reagenzien und Verbrauchsmaterialien umgesetzt zu maximieren.
DNA-Extraktion aus Speichel erfordert mehrere Verfahren: 1) Sammlung und Lagerung, 2) Zell-Lyse, 3) RNase-Behandlung, 4) Proteinfällung, 5) Ethanol-Fällung, 6) DNA Rehydrierung. Die DNA-Stabilisierungspuffer-Lösung, beschriebenvorher 2, Funktionen angemessen unverändert. Kein Versuch zur Optimierung der RNase-Behandlung und DNA Rehydratisierung Schritte wurde. Für jeden weiteren Schritt wurden mehrere Variablen, die Rendite auswirken könnten identifiziert. Jede Variable wurde einzeln manipuliert und Verbesserung der Ausbeute und Qualität wurde statistisch ausgewertet. Für Variablen, die gezeigt wurden, um die Ausbeute und / oder DNA-Qualität zu verbessern, wurden die optimalen Werte in der letzten Protokoll enthalten.
Protokoll
HINWEIS: Vor der Bereitstellung Speichelproben alle Probanden gaben Einwilligung gemäß den Richtlinien für die Behandlung von Menschen bei Nationwide Kinderkrankenhaus.
1. Speichelentnahme und Lagerung
- Vor der Speichelentnahme, sicherzustellen, dass der Mund des Subjekts ist frei von Nahrung oder anderen Fremdstoffen, indem er das Thema spülen den Mund mit Wasser und die Vermeidung von Essen oder Trinken für 30 Minuten vor dem Sammeln der Probe.
- Öffnen Sie ein 15 ml Zentrifugenröhrchen mit 2,5 ml DNA-Stabilisierungspuffer 2 achten Sie darauf, berühren Sie nicht die Innenseite der Kappe oder ein Rohr, und haben das Thema spucken 2,5 ml Speichel in die Pufferlösung. Hinweis: Das Sammeln von mehr als 2,5 ml Speichel können Probenabbau von einem unzureichenden Verhältnis von DNA-Probe, um Stabilisierungspuffer führen. Sammeln zu wenig Speichel erwarteten Erträge aus dem Protokoll zu reduzieren. Um Sammelmengen auswerten, die Ziffern Steigungen auf der Seite von Ter Rohr.
- Die Schutzkappe und mischen durch Umdrehen, bis die Mischung homogenisiert. Heftiges Schütteln ist nicht notwendig. Lagern Sie die Proben bei Raumtemperatur für die kurzzeitige Lagerung oder 4 ° C für die Langzeitlagerung (> 3 Monate).
2. Erste Vorbereitungen und Zell-Lyse
- Vor Beginn der Extraktion, Hitze ein Wasserbad auf 37 ° C, und bereiten einem Eimer Eis. Drei 15 ml konischen Zentrifugenröhrchen wird für jeden extrahierten Probe benötigt. Die drei Röhren wird verwendet, um die Zelle und Proteinpellet, die endgültige extrahiert gDNA und das Isopropanol und Ethanol-Überstände zu halten.
- Abrufen Proben von Lagerung und Invertzucker Proben mehrmals dann Wirbel bei mittlerer Geschwindigkeit für 15 Sekunden.
- Verzichten 2,5 ml Probe in ein sauberes 15 ml Zentrifugenröhrchen und 5 ml Zell-Lyse-Lösung. Die Probe 50-mal durch Inversion, und Inkubation bei RT für 30 min.
3. Entfernen RNA
- Dann werden 40 ul RNase A Solution bei 100 mg / ml, und bei 37 ° C für 15 min.
- Entfernen Sie die Probe aus dem 37 ° C warmen Wasserbad und kühlen auf Eis für 3 min.
- Nach der RNase A Inkubation wird die Temperatur des Wasserbades auf 65 ° C für die DNA Rehydratisierung Schritt des Protokolls.
4. Protein und Lipidentfernung
- Dann werden 50 ul Proteinase K-Lösung bei 20 mg / ml, Mischung mehrfach durch Umdrehen und inkubiert bei Raumtemperatur für mindestens 30 min. Hinweis: Dies ist eine mögliche Pause Punkt für das Protokoll. Nach der Zugabe der Proteinase-K-Lösung, kann die Probe bei 4 ° C gelagert, bis die Extraktion abgeschlossen werden kann. Lagerung bei 4 ° C für bis zu 24 Stunden wurde nicht gezeigt, um eine signifikante Wirkung auf die Extraktionsausbeuten oder DNA-Qualität haben. Langfristige Lagerung in diesem Stadium noch nicht bewertet worden.
- Fügen Sie 1,7 ml Protein Precipitation Lösung, Vortex kräftig für 20 Sekunden bei hoher Geschwindigkeit und auf Eis für 10 min.
- Nachdem die Proben auf Eis für 10 min, Zentrifuge für 10 min bei 3.000 × g und 4 ° C abgekühlt. Die gefällten Proteine bilden muss eine enge Pellet, um fortzufahren. Wenn das Pellet ist nicht dicht oder die Lösung ist immer noch bewölkt, können die Proben auf Eis für 5 min mehr gekühlt werden und Zentrifugation wiederholt. Die Proben sind auf Eis gehalten werden, um eine enge Pellet gewährleisten.
5. Isolierung und Aufreinigung von gDNA
- In einen sauberen 15 ml-Zentrifugenröhrchen, Pipetten 5 ml Isopropanol und 8 ul Glycogen reinen Lösung bei 20 mg / ml.
- Gießen der Überstand, der gDNA aus Schritt 4.3 in das Rohr mit dem Isopropanol und Glykogen Lösung, wobei hinter dem ausgefällten Proteinpellet. Nachdem der Überstand wurde hinzugefügt, vorsichtig mischen die Probe 50-mal durch Umdrehen und Zentrifuge für 30 min bei 3.000 × g und 4 ° C liegt.
- Gießen Sie den Überstand langsam in einen sauberen 15-ml-Tube. Nach Entfernen des Überstands, Zugabe von 1 ml 70% EthanolWaschen des Pellets langsam und sanft schaukel Bewegen des Ethanol über dem ausgefällten Pellet mehrmals. Bewahren Sie das Ethanol in die Röhre.
- Nach der ersten Wäsche, Zentrifugieren Sie die Probe für 1 min bei 2.000 xg und 20 ° C. Dieser Zentrifugationsschritt kann entweder bei 4 ° C oder 20 ° C durchgeführt werden. Keine signifikante Wirkung der Temperatur für diesen Schritt hat sich gezeigt.
- Nach der ersten Wäsche und Zentrifugation des Pellets, gießen Sie langsam den Ethanolwasch aus der Tube und entsorgen, dann eine zweite Wasch durch Wiederholen der Schritte 5.3 und 5.4.
- Nach dem Entfernen des Überstands aus der zweiten Wasch Pellet an der Luft trocknen lassen für 15 Minuten.
- Wenn die Probe nicht vollständig getrocknet ist, an der Luft trocknen für weitere 15 min.
6. Rehydrierung von gDNA
- Nachdem die Probe getrocknet werden 300 ul Tris-EDTA, das getrocknete Pellet gDNA rehydrieren.
- Vortex die Probe für 5 Sekunden bei mittlerer Geschwindigkeit und in einen65 ° C heißen Wasserbad für 1 Stunde.
- Entfernen Sie die Proben aus dem Wasserbad inkubiert und O / N bei RT.
HINWEIS: Alle Produkte und Reagenzien sind in der Tabelle aufgeführten Materialien, sowie Tabelle 4.
Ergebnisse
Um die optimalen Parameter zu bestimmen für die DNA-Extraktion eine Reihe von gepaarten DNA-Extraktionen durchgeführt wurde. Eine einzelne Speichelprobe wurde aufgeteilt, und jede Portion mit einem von zwei möglichen Werten für eine bestimmte Variable getestet. Mindestens acht Wiederholungen jeder gepaarten Test durchgeführt (beispielsweise wurde ein einzelner Speichelprobe aliquotiert, um die Extraktion mit oder ohne anfängliche 50 ° C Inkubation Test). Gesamt-DNA-Ausbeute, die 260/280 Wert, desto 260/230 Wert und Sichtprüfung der Elektrophorese DNA, um die Fragmentierung zu bewerten: Optimierung wurde auf vier Standard-Metriken. Nicht alle möglichen Kombinationen der Variablen wurden statistische Interaktionen bewertet (N = 169 Kombinationen), sich stattdessen um den marginalen Effekt der einzelnen Variablen einzeln zu bewerten. Effekte wurden mit einer Mehrwege-wiederholte Messungen ANOVA getestet und geschätzten Effekte wurden aus dem Ersatzregressionsgleichung abgeleitet. Alle wichtigen Effekte sind in Tabelle 1 zusammengefasst, wie aver gezeigtAlter Renditeänderung (ng / ul pro ml Speichel-Eingang) oder DNA-Qualität (260/280 260/230 und).
Zell-Lyse (Schritt 2) wurde durch Beurteilung optimiert: 1) die Anwesenheit / Abwesenheit einer 50 ° C inkubiert (1 h) vor der Lyse der Zelle, um sicherzustellen, daß Proteinase K Abbau und Zelllyse durch den Speicherpuffer vermittelte vollständig abgelaufen war, 2 ) Gegenwart / Abwesenheit einer Homogenisierung durch Vortex-Schritt (mittlere Geschwindigkeit, 15 sec), und 3) Lyselösung Inkubationszeit (5 gegenüber 30 Minuten). Die 30 min Inkubation Zelllyse erhöhten Ertrag von durchschnittlich 3,5% (p <.01), aber keine anderen Zelllyse variable hatte einen signifikanten Effekt auf den Ertrag. Vortexen verringerte die 260/280 Verhältnis durch eine statistisch signifikante (p <.001), jedoch praktisch kleiner 0,03.
Proteinfällung (Schritt 4) wird durch Proteinase K Verdauung voraus, um Aminosäureketten stören, die Verbesserung der Effizienz der Proteinfällung und Loslassen gefangen DNA. Die Höhe der Proteinase K variiert wurde das Zehnfache.Zentrifugation Temperatur von 20 ° C bis 4 ° C reduziert. Erhöhung der Menge der Proteinase K verursachte eine statistisch signifikante Abnahme in der Ausbeute (8,7%) und auch leicht verbessert sowohl die 260/280 und 260/230 Verhältnissen.
Ethanol-Fällung (Schritt 5) die letzte Stufe des Protokolls untersucht. Die Menge an Glykogen Träger (0, 8 ul) wurde variiert, ebenso wie die Gesamtzeit Zentrifugation (5 min 11 gegenüber 30). Nur Zentrifugation Zeit wesentlich beeinflusst Ertrag, mit einer durchschnittlichen Zunahme von 290%. Je länger Spin reduzierte auch die 260/280 Quote leicht (0,05). In den Versuchen wurde keine signifikante Wirkung auf die Ausbeute von Glykogen beobachtet; Obwohl die Gesamtmenge an DNA in diesen Extraktionen war so groß, dass Glykogen nicht typischerweise verwendet werden. Trotz der fehlenden Wirkung in diesen Proben, ist es immer noch empfehlen, Glykogen zu verwenden, um das Risiko von Ertragseinbußen zu minimieren, wenn Speichel Eingangslautstärke niedriger als hier, oder wenn ther gegeben iste ist aus einem anderen Grund zu glauben, Ertrag gering.
Sichtkontrolle der repräsentativen DNA-Proben (Figur 1) angedeutet, dass die extrahierte DNA wurde nicht wesentlich für jede Speichel DNA-Extraktionsverfahren fragmentiert, sondern zeigten einen geeigneten hochmolekularen Bande ohne Verschmieren anzeigt abgebaute DNA. Nach RNase A Verdauung, erzeugt das Protokoll eine durchschnittliche 260/280 von 1,74.
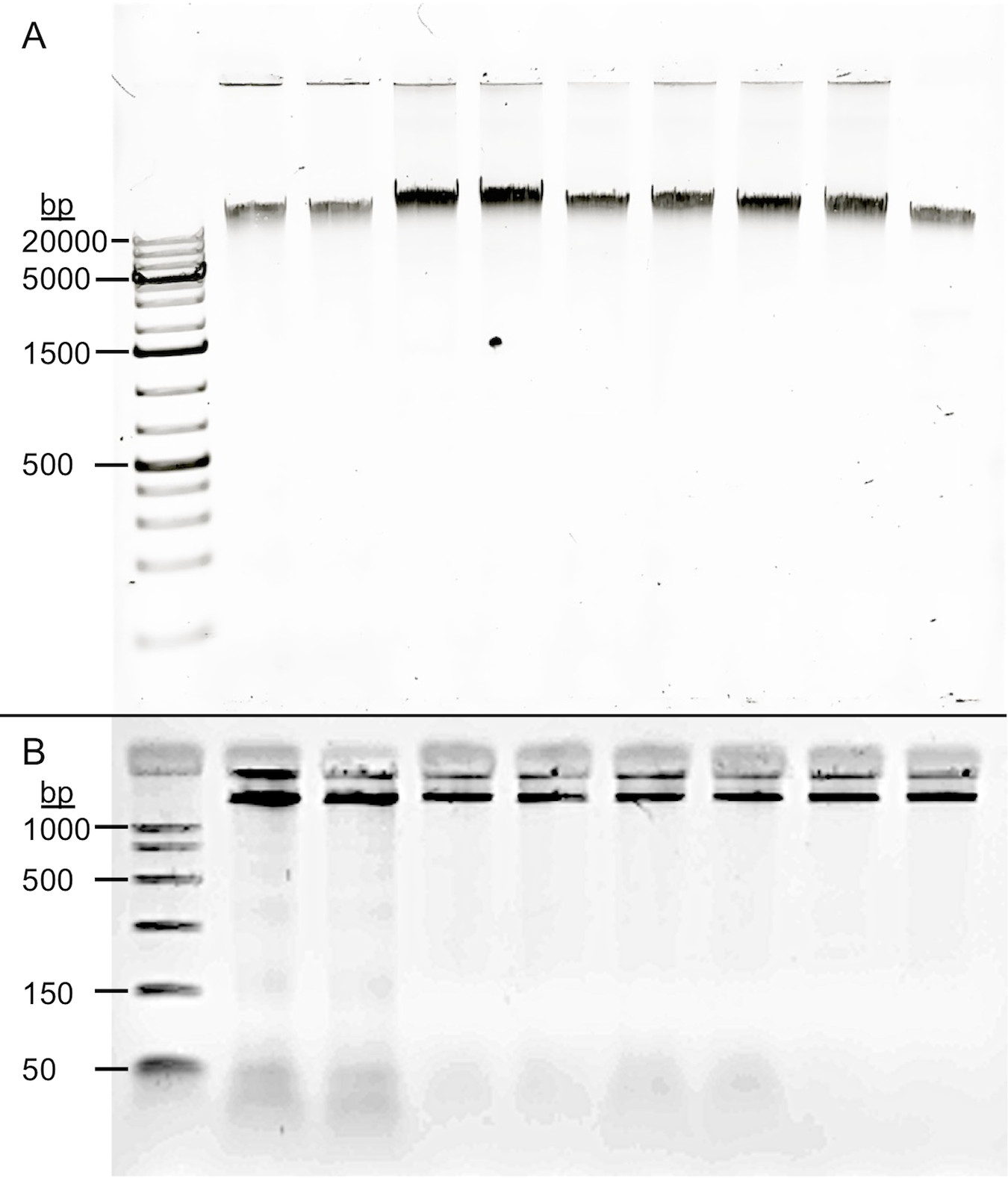
Abbildung 1. Qualität der Speichel stammende DNA. Vier Extraktionsverfahren wurden der gleichen Speichelentnahme angewandt. (A) Die Proben wurden auf einem 0,8% igen Agarose-Gel (250 ng DNA) elektrophoresiert. Alle Variationen der Speichel DNA Extraktionsprotokolle führen zu hohen Molekulargewichts (> 20 kb) DNA, ohne Anzeichen von Zersetzung. Lane: 1 DNA laddeR, 2 und 3 Oragene Prepit L2P Protokoll Proben 4 und 5 Gentra Puregene Körperflüssigkeiten Protocol, 6 und 7 die optimierte Protokoll ohne RNA-Entfernungsschritt, 8 und 9 der optimierten Protokolls mit der Entfernung von RNA Schritt. Spuren 2-7 sind direkt analog Protokolle auf den gleichen Speichelproben. Spuren 8 und 9 zeigen, dass die RNA-Entfernungsstufe nicht DNA-Abbau vorstellen. (B) Die Proben wurden auf einem 2% Agarosegel (150 ng DNA) elektrophoresiert. Eine leichte RNA Spitzen beobachtbar ist in der Nähe der Unterseite des Gels in den Spuren 2 bis 7 (Konventionen wie oben). Spuren 8 und 9 zeigen die Wirksamkeit der RNA-Entfernungsstufe.
Die RNA-Entfernungsschritt (Schritt 3 mit RNase A) ist kritisch für eine genaue Quantifizierung der DNA. Während des Tests wurde konstant hohen RNA-Gehalt beobachtet wurde, wie durch das Verhältnis von doppelsträngiger DNA von RNA durch ein Qubit 2,0 Fluorometer bestimmt. Im Durchschnitt Nukleinsäuregehalt von Proben ohne RNase A Behandlung bestand aus 46,6% (± 0,4) RNA. Proben, die die RNA-Entfernungsstufe als "<20 ng / ml", die die niedrigste mögliche Lesart für die RNA-Erkennung das Qubit ist zu lesen unterzog.
Die durch diese optimierten Protokoll erhaltenen DNA war von ausreichender Qualität für Hochdurchsatz-Sequenzierung, wenn die zusätzliche RNase A Schritt aufgebracht wurde. Gezielte Resequenzierung Daten zu erreichen, wurde eine individuelle Agilent Sureselect Ziel Enrichment Kit zu 24 Proben angewandt, Targeting 2.6 Mb von Sequenz. Hochdurchsatz-Sequenzierung wurde am 12. mit Strichcode (indexiert) Proben pro Bahn durchgeführt. Folge liest wurden dem Referenzgenom HG19 12-BWA ausgerichtet ist, dann Anwendung GATK 13 Basisqualitätsfaktor Rekalibrierung Indel Neuausrichtung, Duplikate entfernen, SNP Genotypisierung Entdeckung und gleichzeitig in allen 24 Proben wurde mit Hilfe des Best-Practice-Festfilterparameterwerte 14. Alle 24 Proben ergaben hohe Qualität NGS-Daten (Tabelle 2). Der liest, dass geben IlluMina Standardfiltern und hatte Q> 20, 91,4% auf die Sequenzanreicherung Zielregionen ausgerichtet und bietet eine durchschnittliche on-Zielabdeckung Tiefe von> 30x Abdeckung bei Q> 100, auch innerhalb der notwendigen Grenzen für seltene SNP Entdeckung in jeder Probe. Die durchschnittliche Strang Bilanz war 49,9%. Vergleich Variante Anrufe mit Illumina-Microarray-Genotypen ergab eine Übereinstimmung von 98,9%.
| Kandidat Variable | ng / ul | 260/280 | 260/230 |
| Wirbel | ns | -0.03 *** | ns |
| x30 min Cell Lysis Incubation | 3,5% ** | ns | ns |
| Proteinase K x10 | -8.7% * | -0.05 *** | -0.03 *** |
| 30 min Schleuder | 290,2%*** | -0.05 *** | ns |
| Glykogen | ns | ns | -0.37 *** |
Tabelle 1. Wirkung Größe der optimierten Variable auf Menge / Quality Metrics. Alle Effektstärken sind in den in der Spaltenüberschrift aufgeführten Einheiten. p-Werte aus ANOVA: * p <.05; ** P <.01; *** P <.001; ns nicht signifikant
| Qualitätsmetrik | Wert |
| Ziellänge | 1.708 kb |
| Zieldachte> 30x | 85.22% |
| SNPs in dbSNP | 92.55% |
| Array Vereinbarung | 98.99% |
Tabelle 2. Qualität der Hochdurchsatz-Sequenz von Target Enrichment.
| Neue optimierte Protokoll | Artikel | Verteiler | Katalog # | Kauf Einheit | Kosten / Einheit | Kosten / Sammlung |
| 15 ml Zentrifugenröhrchen | Fischer | 12-565-268 | 500 Tubes | 233,50 $ | 3,2690 $ | |
| Zell-Lyse-Lösung | Qiagen | 158.908 | 1 L | 401,00 $ | 3,2080 $ | |
| Proteinase K | Sigma | P6556 | 1 g | 713,00 $ | 1,5686 $ | |
| Proteinfällung Lösung | Qiagen | 158.912 | 350 ml | $ 350,00 | 2,7200 $ | |
| Isopropanol | Fischer | A416-4 | Bei 4 x 4 L | 486,71 $ | 0,2434 $ | |
| Glycogen-Lösung (20 mg / ml) | EZ-BioResearch | S1003 | 1 ml | 51,00 $ | 0,8160 $ | |
| 70% Ethanol | Fischer | 04-355-305 | Bei 4 x 1 Gallone. | 123,03 $ | 0,0325 $ | |
| Tris-EDTA (TE) | Fischer | BP2473-1 | 1 L | 68,67 $ | 0,0412 $ | |
| NaCl | Fischer | AC194090010 | 1 kg | 34,65 $ | $ 0.000004 | |
| Tris-HCl | Fischer | BP1757-100 | 100 ml | 57,84 $ | 0,0116 $ | |
| EDTA (0,5 M) Lösung | Fischer | 03-500-506 | 100 ml | 33,60 $ | 0,0134 $ | |
| Natrium DodAcyl-Sulfat | Fischer | BP166-100 | 100 g | 59,95 $ | 0,0060 $ | |
| Gesamtkosten | 11,93 $ | |||||
| Oragene | Artikel | Verteiler | Katalog # | Kauf Einheit | Kosten / Einheit | Kosten / Sammlung |
| 15 ml Zentrifugenröhrchen | Fischer | 12-565-268 | 500 Tubes | 233,50 $ | 0,9340 $ | |
| 100% Ethanol | Fischer | BP2818-100 | 100 ml | 34,29 $ | 1,6459 $ | |
| 70% Ethanol | Fischer | 04-355-305 | Bei 4 x 1 Gallone. | 123,03 $ | 0,0081 $ | |
| Tris-EDTA (TE) | Fischer | BP2473-1 | 1 L | 68,67 $ | 0,0687 $ | |
| 1,5-ml-Röhrchen | Genesee | 22-281A | 500 Tubes | 22,85 $ | 0,0457 $ | |
| Oragene Collection Kit | Oragene | OG-500 | 1 | 25,00 $ | 25,00 $ | |
| Gesamtkosten | 27,70 $ | |||||
| Puregene | Artikel | Verteiler | Katalog # | Kauf Einheit | Kosten / Einheit | Kosten / Sammlung |
| 15 ml Zentrifugenröhrchen | Fischer | 12-565-268 | 500 Tubes | 233,50 $ | 0,9340 $ | |
| Zell-Lyse-Lösung | Qiagen | 158.908 | 1 L | 401,00 $ | 4,0100 $ | |
| Proteinase K | Qiagen | 158918 | 650 ml | 73,10 $ | 7,8723 $ | |
| Proteinfällung Lösung | Qiagen | 158.912 | 350 ml | $ 350,00 | 4,0000 $ | |
| Isopropanol | Fischer | A4164 | Bei 4 x 4 L | 486,71 $ | 0,3650 $ | |
| Glykogen-Lösung | Qiagen | 158930 | 500 ml | 64,30 $ | 2,5720 $ | |
| 70% Ethanol | Fischer | 04-355-305 | Bei 4 x 1 Gallone. | 123,03 $ | 0,0975 $ | |
| DNA-Trinklösung | Qiagen | 158.914 | 100 ml | 69,80 $ | 0,1396 $ | |
| NaCl | Fischer | AC19409-0010 | 1 kg | 34,65 $ | $ 0.000004 | |
| Tris-HCl | Fischer | BP1757-100 | 100 ml | 57,84 $ | 0,0116 $ | |
| EDTA (0,5 M) Lösung | Fischer | 03-500-506 | 100 ml | 33,60 $ | 0,0134 $ | |
| Natriumdodecylsulfat | Fischer | BP166-100 | 100 g | 59,95 $ | 0,0060 $ | |
| Gesamtkosten | 19,99 $ |
Tabelle 3. Kostenvergleich der optimierten Protokoll, um DNA aus 2 ml Voll Speichel mit anderen kommerziell verfügbaren Protokolle zu extrahieren. All Reagenzien und Verbrauchsmaterialien für die DNA-Extraktion erforderlich wurden mit Standard-Listenpreise im Internet zum 30. September verfügbar beurteilt, 2013 Hinweis, dass dieses Protokoll wird geschrieben, um DNA von 1,25 ml Voll Speichel (extrahieren dh 2,5 ml Speichel und Puffer , siehe Schritt 2.3), da dieser Wert entspricht der Hälfte des gesamten Volumens im Speichel Sammelröhrchen. Für die Kostenvergleich wurden 2 ml gewählt, da dies die Menge mit einem gemeinsamen im Handel erhältlichen Kits (Oragene) zugeordnet ist.
| DNA Stabilisierungspuffer (250 ml) | ||
| Komponente | Volumen (ml) | [Finale] |
| 1 M NaCl | 1.461 g | 0,1 M |
| Tris-HCl | 2.5 | 0,01 M |
| 0,5 M EDTA | 5 | 0,01 M |
| 10% SDS | 12,5 | 0,014 M |
| Proteinase-K-Lösung (20 mg / ml) | 2.5 | 6.92x10 -6 M |
| ddH2O | 227,5 | |
| Proteinase-K-Lösung (20 mg / ml) | ||
| Komponente | Volumen (ml) | [Finale] |
| Proteinase K Pulver | 500 mg | 6.92x10 -4 M |
| ddH2O | 25 | |
| 70% Ethanol (500 ml) | ||
| Komponente | Volumen (ml) | [Finale] |
| Ethanol 95% | 368,5 | 12.63 M |
| ddH2O | 131,5 | |
Tabelle 4. Rezepte für Reagenzien.
Diskussion
Das vorliegende Verfahren ist eine optimierte DNA-Extraktionsprotokoll, die deutlich verbesserte Ausbeute von hochmolekularer DNA im Vergleich zu Standardmethoden, ohne dabei DNA-Qualität. Der entscheidende Schritt mit der größten Wirkung auf den Ertrag die meisten war Schritt 5.2, die eine längere Zentrifugation Schritt während Ethanol-Fällung als jeder veröffentlichten Protokoll hier, überprüft mit einer Ausnahme, die nicht weit verbreitet war 11 umfasst. Keine Änderungen in der DNA-Qualität mit dieser längeren Zentrifugation zugeordnet wurden nachgewiesen, was anzeigt, dass die meisten der verfügbaren DNA aus Gesamtspeichelsammlung nicht verschlechtert und hohem Molekulargewicht.
Gesamtspeichelsammlung hat Einschränkungen in der Probenqualität wie das Potenzial für ausländische Verunreinigungen, die an der Sammelphase minimiert werden muss, und die Anwesenheit von übermäßigen Protein in der Probe, die ein Zeichen einer zugrunde liegenden Infektion sein kann. Große Mengen an Protein oder ausländische Verunreinigungenbleiben in der letzten extrahierten DNA, wodurch die Quantifizierung ungenau machen. Wenn es Restproteine oder Verunreinigungen nach Rehydrierung (Schritt 6) stehen im Verdacht, eine Probe aufräumen kann, indem man bei der Proteinfällung Schritt mit Reagenzienvolumina skaliert, um die Eingangslautstärke widerspiegeln Probe durchgeführt werden. Eine weitere Beschränkung des Protokolls ist die Länge der Zeit, die erforderlich ist, die Schritte durchzuführen. Mehrere Proben können parallel ausgeführt werden; Allerdings empfiehlt es sich, nicht mehr als 24 parallel Extraktionen gleichzeitig ausgeführt werden. Dies ist besonders wichtig für das Protein-Fällungsschritt, in dem die Probe kalt, um einen festen Pellets und laufen mehr als 24 Proben können Pellets Zeit wieder in Lösung zu ermöglichen sicherzustellen bleiben.
Die hier vorgestellte Protokoll ist die kostengünstigste Methode betrachtet (siehe Tabelle 3). Während dieses Protokolls macht Verwendung von Reagenzien aus dem Puregene Extraction Kit, ein kleineres Volumen als in dem Puregene-Protokoll verwendet wird empfohlenohne Beeinträchtigung Extraktionsausbeute und es ist diese Verringerung Reagenzien, die Kosteneinsparungen in Bezug auf die Protokoll antreibt. Beachten Sie, dass die berechneten Kosten für die Extraktion verwendet den Listenpreis für jeden Artikel und nicht eventuelle Rabatte wider. Die Kosten pro Extraktion mit dem optimierten Protokoll kann weiter mit Großaufträgen oder Rabatte durch Unternehmen Vertriebsmitarbeiter reduziert werden.
Beweis ist ebenfalls enthalten, dass dieses Protokoll ist für die Verwendung mit der nächsten Generation Sequenzierung. Die erhaltenen Daten liefert einen weiteren Beweis für die Nützlichkeit von Speichelproben für menschliche genetische Forschung in vielen Krankheiten, bei denen Blut nicht routinemäßig verfügbar. Während Blut-und DNA-Zelllinie ist weiterhin die bevorzugte Quelle von genetischen Ressourcen für die Prüfung zu sein, ist ganze Speichelentnahme eine Alternative, wenn solche Quellen nicht verfügbar, nicht verfügbar ist oder unpraktisch, wenn die Aufnahme von Patienten wird durch ihre Sammlung oder Aderlass betroffen.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
This work was funded by a National Institutes of Health R01 (DC009453 support to CWB).
Materialien
| Name | Company | Catalog Number | Comments |
| 15 ml Centrifuge Tubes | Fisher | 12-565-268 | |
| Cell Lysis Solution | Qiagen | 158908 | |
| Proteinase K | Sigma | P6556 | |
| Protein Precipitation Solution | Qiagen | 158912 | |
| Isopropanol | Fisher | A416-4 | |
| Glycogen | EZ-BioResearch | S1003 | |
| 70% Ethanol | Fisher | 04-355-305 | |
| Tris-EDTA (TE) | Fisher | BP2473-1 | |
| NaCl | Fisher | AC194090010 | |
| Tris HCl | Fisher | BP1757-100 | |
| EDTA (0.5 M) Solution | Fisher | 03-500-506 | |
| Sodium Dodecyl Sulfate | Fisher | BP166-100 | |
| Analog Vortex Mixer | Fisher | 02-215-365 | |
| Centrifuge 5810R | Eppendorf | 5811 000.010 |
Referenzen
- Quinque, D., Kittler, R., Kayser, M., Stoneking, M., Nasidze, I. Evaluation of saliva as a source of human DNA for population and association studies. Analytical Biochemistry. 353, 272-277 (2006).
- Min, J. L., et al. High mircosatellite and SNP genotyping success rates established in a large number of genomic DNA samples extracted from mouth swabs and genotypes. Twin Research and Human Genetics. 9, 501-506 (2006).
- Dlugos, D. J., Scattergood, T. M., Ferraro, T. N., Berrettinni, W. H., Buono, R. J. Recruitment rates and fear of phlebotomy in pediatric patients in a genetic study of epilepsy. Epilepsy & Behavior. 6, 444-446 (2005).
- Etter, J. F., Neidhart, E., Bertand, S., Malafosse, A., Bertrand, D. Collecting saliva by mail for genetic and cotinine analyses in participants recruited through the internet. European Journal of Epidemiology. 20, 833-838 (2005).
- Hansen, T. V., Simonsen, M. K., Nielsen, F. C., Hundrup, Y. A. Collection of blood, saliva, and buccal cell samples in a pilot study on the danish nurse cohort: Comparison of the response rate and quality of genomic DNA. Cancer Epidemiol Biomarkers Prev. 16, 2072-2076 (2007).
- Van Schie, R. C. A. A., Wilson, M. E. Saliva: A convenient source of DNA for analysis of bi-allelic polymorphisms of fcγ receptor iia (cd32) and fcγ receptor iiib (cd16). Journal of Immunological Methods. 208, 91-101 (1997).
- Dawes, C. Estimates, from salivary analyses, of the turnover time of oral mucosal epithelium in humans and the number of bacteria in an edentulous mouth. Archives of Oral Biology. 48, 329-336 (2003).
- Bahlo, M., et al. Saliva-derived DNA performs well in large-scale, high-density single-nucleotide polymorphism microarray studies. Cancer Epidemiol Biomarkers Prev. 19, 794-798 (2010).
- Hu, Y., et al. Genotyping performance between saliva and blood-derived genomic dnas on the dmet array: A comparison. PLoS ONE. 7 (3), e33968(2012).
- Simmons, T. R., et al. Increasing genotype-phenotype model determinism: Application to bivariate reading/language traits and epistatic interactions in language-impaired families. Human Heredity. 70, 232-244 (2010).
- Zeugin, J. A., Hartley, J. L. Ethanol precipitation of DNA. Focus. 7, 1-2 (1985).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler Transform. Bioinformatics. 25, 1754-1760 (2009).
- McKenna, A., et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res. 20, 1297-1303 (2010).
- DePristo, M., et al. A framework for variation discovery and genotyping using next-generation DNA sequencing data. Nature Genetics. 43, 491-498 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten