Method Article
확장 현미경 검사법을 가진 Podocytic 단백질 Nephrin, 액틴 및 포도신의 화상 진찰
요약
제시된 방법은 확장 현미경으로 형광 표지된 세포 단백질의 시각화를 가능하게 하고 전통적인 현미경에 70 nm의 해상도로 이끌어 냅니다.
초록
구체 내피, 구체 지하 막 및 podocytes로 구성된 구체 필터의 중단은 알부무뇨증을 초래한다. Podocyte 발 프로세스는 podocin와 같은 세포켈레탈 어댑터 단백질에 결합하는 액틴 번들을 포함합니다. 포도신과 같은 어댑터 단백질은 네프린과 같은 구슬빛 횡격막의 중추를 액틴 세포 골격에 연결합니다. 이들과 그밖 podocytic 단백질의 현지화 그리고 기능을 공부하는 것은 건강과 질병에서 구체 필터의 역할의 이해에 필수적입니다. 제시된 프로토콜은 사용자가 기존의 현미경에 슈퍼 해상도 이미징을 사용하여 세포에서 액틴, 포도신 및 nephrin을 시각화할 수 있게 합니다. 첫째, 세포는 종래의 면역 형광 기술로 염색된다. 시료 내의 모든 단백질은 팽창 가능한 하이드로겔에 공유적으로 고정됩니다. 단백질제 K를 함유한 소화를 통해 구조 단백질은 마지막 단계에서 겔의 이소열대 부종을 허용합니다. 물에서 샘플의 투석은 샘플의 4-4.5 배 팽창을 초래하고 샘플은 기존의 형광 현미경을 통해 이미지 될 수있다, 70 nm의 잠재적 인 해상도를 렌더링.
서문
알부미뇨증은 심혈관 위험의 대리 매개 변수이며 사구형 필터1의중단으로 인한 결과입니다. 구체 필터는 포도세포에 의해 형성된 회향성 내피, 구체 지하 막 및 슬릿 다이어프램으로 구성됩니다. 포도세포의 기본 및 이차 발 공정은구체2의 모세관 벽을 감싸는다. 발 과정의 섬세한 구조는 또한 다중 슬릿 다이어프램 단백질 및 기타 어댑터 단백질2에대한 앵커 역할을 하는 피질 액틴 번들에 의해 유지된다. 슬릿 다이어프램의 백본 단백질은 신장이라고 불리며 반대 되는 podocytes의 네프린 분자와 동종 방식으로 상호 작용합니다. 다양한 어댑터 단백질을 통해, 네프린은 액틴 세포골격2,3에연결된다. 신장 인코딩 유전자 NPHS1의 돌연변이는 핀란드 형4의신 증후군으로 이어집니다.
네프린의 상호 작용 단백질 중 하나는 스토마틴 가족3의헤어핀 과 같은 단백질인 포도신입니다. 포도신은 지질 뗏목에 nephrin을 모집하고 액틴 세포 골격5에연결합니다. 포도신은 NPHS2 유전자에 의해 인코딩된다. NPHS2의 돌연변이는 스테로이드 저항하는 신증후군6로이끌어 낸다.
액틴 어댑터 단백질을 시각화하고 공동 국소화하기 위해 면역 형광 기술이 사용될 수 있다. 불행히도, 빛의 회절 장벽은 종래의 형광 현미경의 해상도를 200-350 nm7로제한한다. 새로운 현미경 기술, 예를 들어, 자극 방출 고갈 (STED)8,사진 활성화 국소 현미경 (PALM)9,금세 광학 재건 현미경 검사법 (STORM 또는 dSTORM) 또는 지상 상태 삭제 현미경 검사법은 개별 분자 반환 (GSDIM)9,10,11,약 10 nm까지 해상도를 가능하게한다. 그러나 이러한 슈퍼 해상도 기술은 고가의 현미경, 잘 훈련된 인력을 필요로 하므로 많은 실험실에서 사용할 수 없습니다.
확장 현미경 검사법(ExM)은 종래의 현미경으로 초분해능 이미징을 가능하게 하고 대형 연구커뮤니티(12)에서잠재적으로 사용할 수 있는 신작이고 간단한 기술이다. 단백질 보존 팽창 현미경 검사법(proExM)에서 관심 있는 샘플(세포 또는 조직)은형광구(13)로고정및 염색된다. 시료 내의 단백질은 그 때 작은 분자에 의해 공유적으로 고정됩니다 (6-((아크릴)아미노산, succinimidyl 에스테르, AcX) 팽창 하이드로겔(13)로. 단백질 효소 K(ProK)를 통한 효소 소화를 통해 단백질과 형광은 팽창 후 겔 내에서 상대적인 위치를유지한다 13. 젤의 붓기 후에, 견본은 대략 60-70 nm (300 nm/4.5)의 효과적인 측량 분해로 이끌어 내는 4.5 배 (90 배 체피 확장)까지 확장합니다. 이 기술의 수정은 10배 의 팽창(1,000배 의 체적 확장)을 허용할 수 있으며, 종래의 현미경14,15,16에대해 20-30 nm의 해상도를 렌더링할 수 있다.
마우스와 인간의 신장의 구체 구조는 ExM17을통해 시각화되었습니다. 본 논문에서, 우리는 기존의 형광 현미경을 사용하여 세포 내F 액틴및 액틴 어댑터 단백질 podocin의 슈퍼 해상도 이미지를 시각화하는 상세한 proExM 프로토콜을 제시한다.
프로토콜
1. 세포의 분할 및 파종
- 멸균 덜벡코의 변형 독수리 매체(DMEM)를 포함하여 10% 태아 종아리 혈청(FCS), 멸균 인산염 완충식식염수(PBS) 및 멸균 트립신을 37°C로 드실 수 있습니다. 깨끗한 벤치를 활성화합니다.
- 멸균 집게를 사용하여 각 웰에 멸균 유리 커버 슬립 (10mm)을 추가하여 6 웰 플레이트를 준비하십시오.
- 깨끗한 벤치 아래에 Cos7 세포가있는 10cm 세포 배양 접시를 넣습니다. 클린 벤치 아래에서 진공 장치를 사용하여 셀의 매체를 흡인합니다.
- 한 손에 는 멸균 된 세포 배양 접시를 잡고 세포 배양 접시의 측면에 멸균 PBS 10 mL을 추가하여 세포에서 헹구지 않도록하십시오. PBS가 전체 세포 배양 접시를 헹구기 위해 세포 배양 접시를 내려 놓습니다.
- PBS를 제거하고 세포 배양 접시의 중간에 트립신 1mL을 추가하고 37 °C에서 5 분 동안 배양하십시오.
- 10% FCS를 포함한 DMEM의 10mL를 추가하여 피펫기로 셀 용액을 위아래로 피펫하여 수동으로 세포를 분리하여 트립시화 반응을 중지합니다.
- 카운팅 챔버를 사용하여 밀리리터당 셀 수를 분석합니다.
- 6-웰 플레이트에서 유리 커버 슬립에 잘 2 mL 배지에 68,000 세포를 종자. 조심스럽게 수평 및 수직으로 판을 흔들어 6 웰 플레이트 내에서 세포를 배포합니다.
참고: 셀은 흩어져 있어야 합니다. - 세포를 37°C 인큐베이터에서 5% CO2로하룻밤 동안 배양한다.
2. 세포의 전환
- 멸균 1.5mL 튜브 2개(희석된 DNA(A)용 튜브와 양이온 지질 경질(B)용 희석 시약용 튜브 2개를 준비합니다. 피펫 0.75 μg 의 네프린 과 0.75 μg 의 podocin cDNA 발현 플라스미드 당 잘 1.5 mL 튜브로 희석 하 고 잘 당 감소 된 혈 청 배지의 100 μL로 희석. 두 번째 1.5mL 튜브에 잘 당 3 μL의 양이온 지질 경질 시약을 추가하고 감소 된 혈청 배지의 100 μL로 희석하십시오. 실온에서 5분 동안 두 반응을 모두 배양합니다.
- DNA 지질 복합체에 반응(A와 B)을 모두 결합하고 실온에서 20분 동안 배양합니다. 각 우물에 DNA 지질 복합체의 200 μL을 조심스럽게 추가하십시오.
- 37°C에서 37°C에서 배지를 변경하지 않고 48h에 대해 5%CO2로 배양한다.
3. 세포 구조의 면역 라벨링
- PBS에서 고정 솔루션(4%(w/v) 파라포멀데히드, 1mL/웰), 퍼메아빌리화 솔루션(PBS0000,1mL/웰) 및 블로킹 솔루션(5% (v/v) PBS, 1mL/웰)을 준비한다.
- 진공 장치로 매체를 제거합니다. 추가 매체를 제거하기 위해 각 웰에 PBS 2 mL을 추가합니다. PBS를 완전히 흡인합니다.
참고: PBS로 셀을 씻어내지 않으려면 유리 커버에 직접 미끄러지지 않도록 PBS를 피펫하지 않도록 하십시오. - 4% (w/v) 파라포름데히드(PFA)를 실온에서 10분 동안 PBS에 용해시키는 세포를 수정합니다.
참고 : 또는, 3 % (v / v) 글리옥살 - 에탄올18에서세포를 수정합니다. - PFA를 폐기하고 유리 커버 전표에 직접 파이펫을 피함으로써 PBS (각 2 mL)에서 두 번 셀을 세척하십시오. PBS에서 Triton X-100 0.5% 0.5%(w/v)로 고정 된 세포를 실온에서 10 분 동안 과음하게합니다.
- 투과성 용액을 제거하고 3.2 단계에서 표시된 대로 PBS로 두 번 세척하십시오.
- 실온에서 1 시간 동안 PBS에 5 % (v /v) BSA의 1 mL을 추가하여 세포를 차단합니다. 1차 항체의 200 μL(PBS에서 1%(v/v) BSA에서 1:200으로 세포를 4°C에서 배양한다.
참고: 또는 실온에서 1시간 동안 1차 항체를 배양한다. - 1 차적인 항체를 제거하고 단계 3.2에서와 같이 PBS로 세 번 세척하십시오. 실온에서 1시간 동안 이차 항체(염소 안티래빗 알렉사 488 1:1000에서 PBS에서 1% (v/v) BSA를 추가합니다. 상자를 사용하여 세포를 어둠 속에서 유지합니다.
- 이차 항체를 버리고 PBS로 세 번 세척하십시오.
- PBS를 제거하고 실온에서 1 시간 동안 PBS에서 1 % (v /v) BSA에서 1 :100에서 안티 네프린 항체의 200 μL으로 세포를 배양하십시오. PBS로 세 번 씻으시면 됩니다. 상자를 사용하여 세포를 어둠 속에서 유지합니다.
- PBS를 제거하고 실온에서 1 시간 동안 이차 항체 당나귀 항기니 피그 633, 1:200으로 인큐베이션하십시오. PBS로 세 번 씻으시면 됩니다. 상자를 사용하여 세포를 어둠 속에서 유지합니다.
4. 확장 현미경 검사법
- 준비
- 젤레이션 챔버용 스페이서를 형성하기 위해 다이아몬드 나이프를 사용하여 유리 커버가 #1.0과 #1.5을 5mm 줄무늬(유리 슬라이드당 4개)로 잘라냅니다. #1.5mm 커버 슬립 스트라이프를 배치하여 유리 슬라이드에 길이 2.5cm의 사각형을 형성하고 염색챔버(그림 2A1 -C1)에배치합니다. 피펫은 ddH2O의 액적을 사각형의 모서리에 넣고 유리 커버 슬립 스트라이프를 서로 부착하고 유리 슬라이드(그림2A2 -C2)에부착합니다.
참고: 접착력이 손실될 수 있기 때문에 ddH2O의 완전한 건조를 피하십시오. 필요한 경우 4.1.2 단계로 시작하기 전에 #1.5mm 커버 슬립을 안정적으로 부착할 수 있도록 ddH2O. Wait의 더 많은 방울을 약 20분 동안 적용하십시오. - 커버 당 #1.0 글래스 커버 슬립 스트라이프 4개(#1.5mm 커버 슬립에 놓습니다. 각 #1.5mm 커버 슬립스트라이프(그림 2A3 -C3)에액적을 피펫팅하여 줄무늬를 부착합니다.
참고: 프로토콜의 진행 중인 처리를 용이하게 하기 위해 젤의 두께는 0.15mm 이상이어야 합니다. 그러나, 세포의 상단에 과도한 젤을 피하기 위해, 세포 기판에 가까운 스페이서 높이를 유지. #1.5 및 #1.0 커버 슬립 스트라이프를 스페이서로 사용하면 스페이서 높이가 약 0.3mm가 됩니다. 셀은 커버 글래스(높이 0.12mm)를 부착합니다. 따라서, 겔은 충분히 두껍지만 셀 위에 과도한 젤은 스페이서로서 #1.0 및 #1.5 커버 유리 줄무늬를 사용하여 피할 수 있다. - 겔화 챔버 뚜껑의 경우, 파라핀 필름으로 커버 글래스(#1.5)를 감쌉니다. 파라핀필름(그림 2A5-C5)에서주름이나 먼지를 피하십시오.
- 젤레이션 챔버용 스페이서를 형성하기 위해 다이아몬드 나이프를 사용하여 유리 커버가 #1.0과 #1.5을 5mm 줄무늬(유리 슬라이드당 4개)로 잘라냅니다. #1.5mm 커버 슬립 스트라이프를 배치하여 유리 슬라이드에 길이 2.5cm의 사각형을 형성하고 염색챔버(그림 2A1 -C1)에배치합니다. 피펫은 ddH2O의 액적을 사각형의 모서리에 넣고 유리 커버 슬립 스트라이프를 서로 부착하고 유리 슬라이드(그림2A2 -C2)에부착합니다.
- 앵커링 및 중합 (겔화)
- 앵커링 버퍼를 준비합니다(표 1참조). 앵커링 처리를 위해 PBS를 유리 커버 슬립에 직접 고정 버퍼 250 μL로 교체하고 실온에서 3시간 동안 배양합니다. 상자를 사용하여 기판을 어둠 속에서 유지합니다.
참고: 또는 실온에서 하룻밤 동안 배양하십시오. 모든 실험에 대한 신선한 앵커링 버퍼를 준비하고 제대로 용해 될 때까지 10-15 분 기다립니다. 6-((아크릴)아미노)헥사노산, 수치니미딜 에스테르(AcX)는 적절한 앵커링을 보장하기 위해 4-5개월마다 교체해야 한다. - 앵커링 버퍼를 제거하고 1.5mL PBS로 한 번 씻어 내다.
- 액틴 섬유를 더러우려면 ExM 호환 프할로이딘 용액을 해동하고 실온에서 45분 동안 1%(v/v) BSA의 195μL에서 희석된 프할로이드민(195μL의 5μL)을 배양한다. 상자를 사용하여 샘플을 어둠 속에서 유지합니다.
- 한편, 교반 장치를 사용하여 ddH2O에서 아크릴나트륨을 용해한다. 얼음에서 단조로운 용액을 준비합니다(표 1참조).
참고: 용존 나트륨 아크릴은 명확하고 무색 솔루션이어야 합니다. 용액이 노란색인 경우 새 아크릴레이트나트륨으로 대체하십시오. - 얼음에 겔화 용액을 준비합니다(표 1참조). 겔화 용액이 겔화 챔버에 적용되기 직전에 파이펫 암모늄peroxidsulfate(APS)가 겔화 용액으로 들어간다.
- 세포에서 남근을 제거하고 실온에서 두 번 PBS의 1.5 mL로 세척. 유리 커버 슬립에 시료를 쉽게 제거할 수 있도록 우물 내에 1.5 mL PBS를 둡니다.
- 커버 글래스에 세포를 집게와 캐뉼라를 사용하여 겔링 챔버에 넣고 6웰 플레이트에서 덮개 유리 미끄러짐을 들어 올립니다.
참고: 셀은 유리 덮개 슬립의 상단에 있어야 합니다. 유리 커버 슬립은 스페이서를 만지지 않아야합니다. - 곧 겔화 용액과 소용돌이에 APS를 추가합니다. 피펫 200 μL 의 겔화 용액샘플(도 2A4-C4). 젤 내의 기포를 피함으로써 조심스럽게 겔화 챔버를 닫습니다(그림 2A5-C5).
- 겔챔버를 37°C에서 적어도 1시간 동안 배양하여 젖은 염색 챔버에서 젤을 중합한다.
- 앵커링 버퍼를 준비합니다(표 1참조). 앵커링 처리를 위해 PBS를 유리 커버 슬립에 직접 고정 버퍼 250 μL로 교체하고 실온에서 3시간 동안 배양합니다. 상자를 사용하여 기판을 어둠 속에서 유지합니다.
- 균질화 (소화)
- 인큐베이터에서 염색 챔버를 꺼내십시오. 겔화 챔버 뚜껑을 열려면 뚜껑과 스페이서 사이에 면도날을 소개합니다. 뚜껑을 조심스럽게 제거합니다. 면도날로 스페이서를 제거하고 면도날로 절단하여 모든 여분의 젤을 제거합니다.
- 젤과 함께 슬라이드를 넣고 PBS로 채워진 접시에 유리를 덮습니다. 부드럽게 흔들어서, 젤에서 분리 된 커버 유리를 제거합니다. 접시에서 젤을 쉽게 제거하려면 젤 아래에 슬라이드를 넣어 젤을 슬라이드에 부착하십시오.
- 젤을 슬라이드에 넣고, 면도날을 사용하여 작은 조각(젤의 분기가 2~3개로 나뉘음)으로 젤을 나눕니다. 젤 한 조각을 유리 바닥으로 6웰 플레이트의 우물에 부드럽게 밀어 붓으로 감싸십시오. 젤의 탈수를 피하기 위해 페인트 브러시를 사용하여 약간의 PBS로 젤을 보습하십시오.
참고: 세포는 아래쪽으로 향합니다. - 반전된 현미경을 사용하여 숫자가 낮은 이미지로 개요 이미지를 사용하여 확장 후 확장 계수를 결정합니다.
- 소화 버퍼를 준비합니다(표 1참조). 단백질을 분해하여 소화 버퍼에 4 U/mL로 희석하여 소화 용액을 받습니다.
참고: Proteinase K가 없는 소화 완충제는 1-2주 동안 4°C로 저장할 수 있습니다. - 소화 용액 500 μL을 각 웰에 넣고 용액 내에 젤을 담급합니다. 실온에서 밤새 소화하고 어두운 샘플을 유지 뚜껑을 닫습니다.
참고 : 또는 1 시간 동안 37 °C에서 소화하십시오.
- 확장
- 파이펫으로 소화 용액을 제거하고 폐기하십시오. 실온에서 10분 동안 담근 젤을 1mLdH2O. 인큐베이션으로 추가합니다.
- 물을 제거하고 신선한 ddH2O. 10 분 동안 기다린 후 확장의 고원에 도달 할 때까지 10 분마다 물을 교환 계속합니다.
참고: 최대 4.5배의 샘플 확장을 달성할 수 있습니다. 젤은 광학적으로 명확해집니다.
- 이미징
- 젤에서 물을 제거하고 현미경 검사를 직접 시작합니다. 반전된 현미경을 사용하여, 주로 사전 확장 상태(Step 4.3.4)에서 이미지 된 세포를 찾기 위해 공기 목표 (낮은 배율)를 사용합니다.
- 더 나은 해상도를 위해 40배(오일/물) 및 63배 의 목표로 전환하십시오. 관심의 파장에 흥분하고 카메라를 통해 이미지를 가져 가라.
- 유효성 검사
- 샘플의 개요 이미지를 가져 가라. 4.3.4 단계에서 이미지된 샘플 내에서 동일한 구조를 찾아 일치합니다. 유효성 검사를 위해 최적의 신호 대 잡음 비율로 채널을사용합니다(그림 3 A-B).
참고: 이미징 매개 변수를 조정하여 확장되지 않은 상태에서 획득한 이미지와 유사한 밝기를 달성합니다(단계 4.3.4). - ImageJ로 회전하고 이동하여 확장 전 및 후 확장 이미지를 오버레이합니다. ImageJ의 거리 측정 도구를 사용하여 명확하게 식별 가능한 구조 사이의 거리를 측정합니다. 적어도 10개의 다른 구조를 측정합니다.
참고: 또는 파이썬 스크립트를 사용하여14에설명된 대로 거리를 측정합니다. - 확장 후/사전 확장 측정값을 나누어 확장 계수를 계산합니다.
- 왜곡을 확인하려면 숫자가 높은이미지(그림 4A-B)를사용하여 이미지를 가져 가라. 이러한 이미지를 오버레이하고 ImageJ 또는15에설명된 대로 분석합니다.
참고: 왜곡의 측정은 정기적으로 수행되어야 하지만 모든 샘플에서 반드시 수행되어야 하는 것은 아닙니다.
- 샘플의 개요 이미지를 가져 가라. 4.3.4 단계에서 이미지된 샘플 내에서 동일한 구조를 찾아 일치합니다. 유효성 검사를 위해 최적의 신호 대 잡음 비율로 채널을사용합니다(그림 3 A-B).
결과
이 proExM 프로토콜의 개념과 타이밍은 그림 1에묘사됩니다. 5일째, 전감염된 세포는 관심 있는 단백질을 표적으로 하는 형광 항체로 고정되고 염색된다(도1A,B). 6일째, AcX를 가진 처리는 모든 단백질에 아민 단의 형성으로 이끌어 냅니다 (형광을 포함)(도 1A,B)12. 하이드로겔의 중합시, 이들 아민 군은 하이드로겔(6일째)에 일연게 결합한다. 겔의 중합 후, 균질화(소화)는 단백질효소 K로 수행되어 세포의 구조적 단백질이 파괴된다(6일째, 도 1A,B). 형광으로 표지된 항체는 소화 후에 주로 보존된 남아 있습니다. 구조 단백질의 붕괴로 인해 하이드로겔의 수중 투석은 7일째(도 1A,B)에하이드로겔 내의 세포가 팽창하는 결과를 낳는다. 시료의 이미징은 종래의 형광현미경(도 1A)으로수행된다. 확장 계수를 결정하고 왜곡을 제외하기 위한 데이터 유효성 검사가 수행되어야합니다(그림 1A).
세포 동위 식물의 확장을 수행하기 위해, 겔화 단계는 필수적이다. 도 2는 겔화 챔버의 측면 및 상단 뷰를 나타낸다. 유리 커버 슬립은 겔화 챔버의 스페이서를 구축(도 2A1-3 / C1-3). 고정 및 스테인드 셀이 있는 커버 글래스는 세포가 유리슬라이드(도 2A4-C4)에위쪽으로 배치된다. 겔화 챔버의 뚜껑은 파라필름으로 감싸고 버블프리(도2A5-C5)로닫혀 있다.
이 ExM 프로토콜을 사용하면 최대 4배까지 확장할 수 있습니다. 확장 계수를 결정하려면 확장 전후의 이미지셀(그림 3A + B)에필수적입니다. 부족한 앵커링과 균질화는 세포의 왜곡과 파열로 이어질 수 있습니다. 도 4A + B는 상이한 배율 이미지에서 파열된 세포의 대표적인 예를 나타낸다.
이러한 방법은 F-액틴 및 액틴 어댑터 단백질, 예를 들어, 포도신 및 네프린(도5)의공동 국소화를 조사하는 데 사용될 수 있다. 포도신은 녹색으로 묘사되며 액틴은 파란색으로 표시되어있습니다(그림 5). 니프린은 녹색으로 표시됩니다. 흰색 영역은 공동 지역화를 나타냅니다.
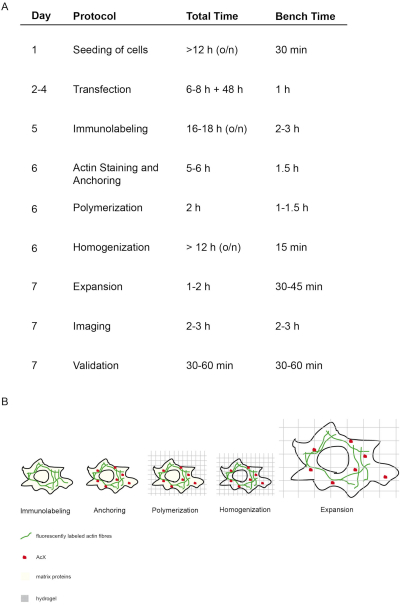
그림 1: 이 ExM 프로토콜의 개념 및 타이밍. (A)"프로토콜" 열에서 프로토콜의 각 단계가 설명되어 있습니다. (A+ B) 세포를 파종하고 변형한 후 면역형성 라벨링(Immunolabeling)이 수행됩니다. (A+ B) 작은 분자 AcX (빨간 점)는 모든 단백질에 결합하고 하이드로겔 (앵커링)에 고정합니다. (A+ B) 중합을 통해 형광을 포함한 모든 단백질은 AcX를 통해 하이드로겔(Polymerization)에 동적으로 결합된다. (A+ B) 균질화는 구조적 매트릭스 단백질의 소화로 이어집니다. (A+ B) 확장은 물 에서 투석에 의해 달성된다. (A)이미징의 이미징 및 검증은 실험을 마무리합니다. (A)전체 프로토콜은 많은 인큐베이션 단계 (하루 열 "총 시간")와 7 일 (열 "일")이 필요하지만 실제 벤치 시간은 각 열 "벤치 시간"에 표시된 것과 같이 훨씬 적습니다. 14에서수정 . 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
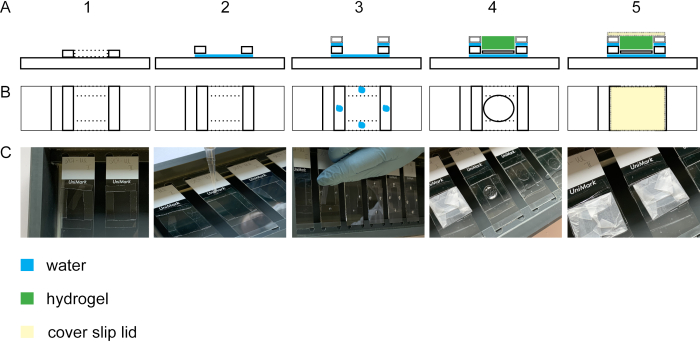
그림 2: 겔화 챔버 를 구축합니다. 사이드뷰(A1)및 상단뷰(B1 + C1)의유리 슬라이드와 4개의 #1.5 커버 스트라이프. 유리 슬라이드와 커버 슬립 스트라이프 사이에 물방울을 추가하면 줄무늬가 유리 슬라이드(측면 보기 A2, 상단 보기 B2, C2)에부착됩니다. #1.5 커버 스트라이프의 물방울은 #1.5 커버 스트라이프 위에 놓인 #1.0 커버 스트라이프의 접착으로이어진다(측면 보기 A3, 상단 뷰 B3, C3). 커버 슬립의 샘플은 집게를 사용하여 사각형 의 중간에 배치됩니다. 겔은 상단에 파이펫(측면 보기 A4, 상단 보기 B4, C4). (A5) 파라필름에 싸인 커버 슬립으로 지어진 닫힌 뚜껑을 포함하여 조립된 겔화 챔버의 측면 뷰 및 상단뷰(B5 및 C5). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
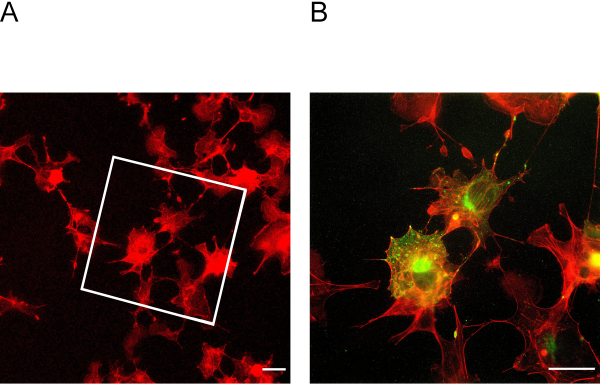
그림 3: 확장 전후의 세포. (A)팽창 전 세포가 액틴에 얼룩진 다. 상자는 도 3B거짓말의 확장된 셀이 있는 영역을 나타냅니다. (B)팽창 후 세포는 녹색의 빨간색과 포도신에 액틴에 대해 염색하였다. Podocin은 세포 주변에서 액틴과 공동 국소화합니다. 스케일 바 = 5 μm, 확장 계수 = 2. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

도 4: 세포의 왜곡 및 파열. (A + B)cos7 세포의 대표적인 현미경 이미지는 액틴(red)을 위해 면역 염색된다. 세포는 고정, 염색, 고정, 소화 및 확장되었습니다. (A)세포의 파열. 화살표는 파열 된 영역을 나타냈다. 스케일 바 = 5 μm, 확장 계수 = 4. (B)세포의 파열 및 왜곡. 흰색 화살표는 파열 된 영역을 나타냈다. 스케일 바 = 5 μm, 확장 계수 = 4. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
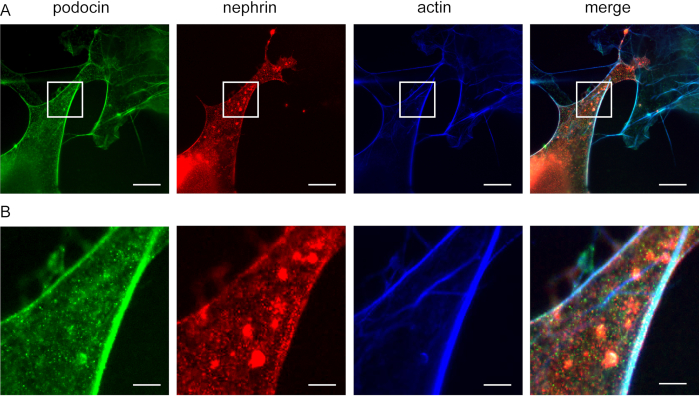
그림 5: 포도닌은 네프린과 액틴과 공동 국한합니다. Cos7 세포 면역형광은 포도신, 액틴 및 네프린을 위해 표기됩니다. (A)Cos7 세포는 ExM을 가진 포도신(녹색), 액틴(blue), 네프린(red)을 위해 염색하였다. 포도신은 액틴과 네프린과 공동 지역화한다. 스케일 바 = 200 nm, 확장 계수 = 4. (B)표시된 영역의 배율(A), 스케일 바 = 40 nm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 버퍼 고정 | 최종 농도 | ||
| 나코3 | 150mM | ||
| 아크릴-X, SE (AcX) | 0.1 mg/ml | ||
| 모노머 솔루션 | 스톡 용액 농도 g/100 ml | 금액(ml) | 최종 농도(g/100 ml) |
| 아크릴산 나트륨 | 38 | 2.25 | 8.6 |
| 아크릴아미드 | 50 | 0.5 | 2.5 |
| N,N'-메틸레네비사크라이엘라미드 | 2 | 0.75 | 0.15 |
| 소금 | 29.2 | 4 | 11.7 |
| PBS | 10x | 1 | 1x |
| 물 | 0.75 | ||
| 합계 | 9.4 | ||
| 겔링 솔루션 | 스톡 솔루션 농도 | 양(μl) | 최종 농도(mg/ml) |
| 단조로운 솔루션 | NA | 190 | NA |
| APS | 10% | 4 | 0.1 |
| 테메드 () | >99% | 4 | 0.1 |
| 물 | NA | 2 | NA |
| 합계 | 200 | ||
| 소화 용액 | 최종 농도 | ||
| 트리스 Cl, pH 8.0 | 1 M | ||
| EDTA pH 8.0 | 0.5M | ||
| 트리톤 X-100 | 0.005% | ||
| 구니딘 HCL | 8 M | ||
| 물 | |||
| 단백질 AK | U/ml 4개 |
표 1: ExM용 솔루션입니다.
토론
제시된 방법은 조사자가 세포 단백질, 예를 들면, podocin, nephrin 및 cytoskeletal 분대, 예를 들면, F-actin를 구상하는 것을 가능하게 합니다. 이 프로토콜 내에서, 전감염된 cos7 세포는 F-actin와 슬릿 다이어프램 단백질의 상호 작용을 연구하는 모델로 이용됩니다. 불행하게도, 불멸의 podocyte 세포주 슬릿 다이어프램 단백질의 충분한 내인성 양을 표현하지않는다(19).
이 방법을 통해 세포 단백질은 기존의 형광 현미경을 사용하여 나노 스케일 해상도로 시각화 될 수 있습니다. 프로토콜 내에서 가장 중요한 단계는 다음과 같습니다: 1) AcX와 하이드로겔에 단백질 아민 그룹의 충분한 고정, 2) 하이드로겔의 적절한 중합, 3) 소화를위한 최적의 타이밍 및 4) 호환 형광의 선택.
하이드로겔에 세포 단백질을 고정하는 것은 확장 시 하이드로겔 내의 단백질 위치를 보존하기 위해 이 방법에 필수적이다. AcX는 세포와 조직 내의 단백질의 아민 단에 결합하는 작은 분자입니다. AcX는 단백질을 가진 탄소 탄소 이중 밴드를 생성하여 중합단계 20에서하이드로겔에 단백질을 통합할 수 있게 한다. AcX는 또한 면역 형광 항체로 라벨링이 AcX 처리 전에 수행 될 수 있도록 항체를 통합한다. 앵커링이 부족하면 세포의 파열과 왜곡이 발생할 수 있습니다. 고정제에 의해 아민 그룹의 수정으로 인해 고정 또는 고정 시간을 최적화해야합니다. 또한 저장소가 부족하거나 최적화되지 않은 앵커링 조건이 파열및 왜곡될 수 있습니다. 우리의 경험을 바탕으로, AcX는 3-4 개월 이상 사용할 때 최적의 효과를 잃게됩니다.
겔의 중합은 온도에 따라 달라집니다. 따라서 우리는 겔화 챔버로 파이프팅하기 전에 중합 솔루션을 얼음에 유지하는 것이 좋습니다. 또한, 겔화 단계의 처리 시간은 조기 겔 형성을 피하기 위해 짧은(5분 미만)을 유지해야 한다. 중합용액의 철저한 혼합은 고르지 않은 중합화를 방지합니다. 기포는 시료를 만질 때 확장 공정에 영향을 미치며 더 많은 중합 용액을 추가하여 방지할 수 있습니다.
하이드로겔 내에 세포 단백질을 통합한 후, 팽창을 보장하기 위해 기계적 균질화 단계(또는 소화)가 필요하다. 상이한 방법, 예를 들어, 열 및 세제 또는 효소 소화, 존재하며 조사된샘플(12,14,20)에사용자 정의할 필요가 있다. 이 프로토콜 내에서 프로테아제 프로테아제 K는 효소 소화에 사용된다. Proteinase K는 형광항체(12)를포함한 대부분의 다른 단백질을 보존하면서 구조적 단백질을 파괴하기에 충분한 투여량에 적용된다. 소화가 불완전하면 샘플 확장이 충분하지 않습니다. 또한, 시료는 팽창 과정에서 찢어수있습니다(도 3). 샘플 확장이 부적절한 경우 물 교체를 권장합니다. 또는 효소 소화의 시간을 조절하거나 Proteinase K의 새로운 알리쿼트열림이 열릴 수 있다.
시료가 과도하게 소화되면 형광 신호가 줄어듭니다. 이 경우 소화 시간을 줄여야 합니다. ExM에서 일반적으로,샘플(14)의부피 팽창에 의한 부피 단위당 형광 신호 강도가 감소된다. 따라서 이미징 중 노출 시간이 길어질 필요가 있습니다.
ExM 호환 형광을 선택하는 것이 필수적입니다. 시아닌 염료는 중합단계(13)동안 분해된다. 박테리오피토크롬에 기초한 형광 단백질도13로크게 파괴된다. 그러나, 대부분의 GFP 와 같은 단백질은13로보존될 것이다. 또한, 스트렙타비딘은 또한 작은 분자태그(13)를통해 S-nitrolysation과 같은 번역 후 변형을 표지하여 사전 확장을 적용할 수 있다.
판로이드인, 액틴 세포골격을 표적으로 하는 작은 라벨 분자는 ExM21과호환되지 않는다. phalloidin의 부족한 앵커링을 극복하기 위해 삼각 앵커링 (TRITON)이21을도입했습니다. 이 접근법은 생체 분자21의동시 표적화, 라벨링 및 접목을 제공합니다.
이 방법은 RNA 분자(ExFish)(22)로염색할 수 있다. 반복 팽창 현미경(iExM) 또는 X10 현미경 검사법에서, 60-70 nm의 분해능은 제1 확장 된 하이드로겔 내에 제2 팽창성 젤을 적용하거나 다른하이드로겔(15,16)을사용하여 단일 팽창 단계를 수행함으로써 약 25 nm로 확장 될 수 있다. 초구조 팽창 현미경 검사법(U-ExM)은 초구조 원소(예: 미토크론드리아, 마이크로투블러)에 대한 저작자를 보존하는 단백질의 슈퍼 분해를 가능하게한다( 23. ExFish (RNA 및 DNA) 및 proExM 방법의 조합은 이전에도22,24뿐만아니라 수행되었다. 제시된 프로토콜은 슬릿 다이어프램 단백질을 조사하기 위하여 모형으로 전염된 cos7 세포를 이용합니다. 우리는 다른 상주 배양 신장 세포, 예를 들어, HEK293T 세포, 유사하게이 프로토콜에 사용할 수 있다고 기대. 세포주에 따라 상이한 배양 및 경질 조건에 맞게 조정이 필요할 수 있습니다.
ExM은 70 nm13의측면 공간 해상도에 약 4배 도달하여 면역 염색 된 샘플의 해상도를 향상시킵니다. 다른 초해상도 기술에 비해 ExM은 종래의 형광 현미경13,14에서수행됩니다. 따라서 ExM방법(14)을수행하기 위해 고가의 장비 나 특별히 훈련된 인력이 필요하지 않습니다. 모든 형광이 ExM과 호환되는 것은 아니지만, 일반적으로 슈퍼 해상도 현미경검사법 14에필요한 사진 물리적 특성을 가진 최적화 된 형광과 함께 많은 사용 가능한 항체가 있습니다. 이 방법의 주요 단점은 ExM이 라이브샘플(12,14)과호환되지 않는다는 것입니다.
미래에, 하이드로겔의 화학 성분을 개선하면 더욱 높은 공간분해능(12)으로이어질 수 있다. 상이한 프로토콜의 조합은 또한 고해상도12를가진 동일 견본 내의 복합체에 있는 단백질, RNA, DNA, 또는 지질의 시각화를 가능하게 할 수 있습니다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
저자는 블랭카 듀브낙과 니콜라 쿠어에게 훌륭한 기술 지원을 해 준 것에 대해 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Acrylamide >99% | Sigma-Aldrich | A3553-100G | |
| 6-((Acryloyl)amino)hexanoic acid, succinimidyl ester, Acryloyl-X, SE | invitrogen | A-20770 | store up to 4 months |
| APS | Sigma-Aldrich | A3678-25G | |
| Deckgläser (cover glasses) | Engelbrecht | K12432 | 24x32mm #1.0 |
| Diamont cutter | VWR | 201-0392 | for cutting the cover slips |
| Guanidine HCl | Sigma-Aldrich | G3272-100G | 8M Stock can be kept at RT |
| Marten hair paintbrush | Leon Hardy | 3 (770) | |
| "Menzel" Deckgläser (cover glasses) | Thermo Fischer | 15654786 | 24x24mm #1.5 |
| N,N`-Methylenbisacrylamide | Sigma-Aldrich | M7256-25G | |
| Objektträger UniMark | Marienfeld | 703010 | |
| Proteinase K | New England Biolabs | P8107S | |
| Sodium Acrylate | Sigma-Aldrich | 408220 | check purity |
| Sodium Bicarbonate | Sigma-Aldrich | S5761 | |
| Staining chamber | produced at the university's workshop | ||
| TEMED | ROTH | 2367.1 | |
| 6-Well glass bottom plates | Cellvis | P06-1.5H-N | |
| Antibodies | |||
| Actin-ExM 546 | chrometra | non-available | 1:40 |
| Anti Podocin produced in rabbit | Sigma | P-0372-200UL | 1:200 |
| Donkey anti guinea-pig CF633 | Sigma | SAB4600129-50UL | 1:200 |
| Goat anti rabbit 488 | Life Technologies | A11034 | 1:1000 |
| Guinea pig anti nephrin | Origene | BP5030 | 1:100 |
| Software | |||
| FIJI | |||
| Visiview | |||
| microscope | |||
| AXIO Observer Z1 | Zeiss | non-available |
참고문헌
- Matsushita, K., et al. Association of estimated glomerular filtration rate and albuminuria with all-cause and cardiovascular mortality in general population cohorts: a collaborative meta-analysis. Lancet. 375 (9731), 2073-2081 (2010).
- Faul, C., Asanuma, K., Yanagida-Asanuma, E., Kim, K., Mundel, P. Actin up: regulation of podocyte structure and function by components of the actin cytoskeleton. Trends in Cell Biology. 17 (9), 428-437 (2007).
- Saleem, M. A., et al. Co-localization of nephrin, podocin, and the actin cytoskeleton - Evidence for a role in podocyte foot process formation. American Journal of Pathology. 161 (4), 1459-1466 (2002).
- Kestila, M., et al. Positionally cloned gene for a novel glomerular protein - nephrin - is mutated in congenital nephrotic syndrome. Molecular Cell. 1 (4), 575-582 (1998).
- Huber, T. B., et al. Podocin-mediated recruitment of nephrin into lipid rafts is required for efficient nephrin signaling. Journal of the American Society of Nephrology. 14, 8(2003).
- Boute, N., et al. NPHS2, encoding the glomerular protein podocin, is mutated in autosomal recessive steroid-resistant nephrotic syndrome. Nature Genetics. 24 (4), 349-354 (2000).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. Journal of Cell Science. 124 (10), 1607-1611 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440 (7086), 935-939 (2006).
- van de Linde, S., et al. Direct stochastic optical reconstruction microscopy with standard fluorescent probes. Nature Protocols. 6 (7), 991-1009 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Testa, I., et al. Multicolor fluorescence nanoscopy in fixed and living cells by exciting conventional fluorophores with a single wavelength. Biophysical Journal. 99 (8), 2686-2694 (2010).
- Wassie, A. T., Zhao, Y., Boyden, E. S. Expansion microscopy: principles and uses in biological research. Nature Methods. 16 (1), 33-41 (2019).
- Tillberg, P. W., et al. Protein-retention expansion microscopy of cells and tissues labeled using standard fluorescent proteins and antibodies. Nature Biotechnology. 34 (9), 987-992 (2016).
- Truckenbrodt, S., Sommer, C., Rizzoli, S. O., Danzl, J. G. A practical guide to optimization in X10 expansion microscopy. Nature Protocols. 14 (3), 832-863 (2019).
- Truckenbrodt, S., et al. X10 expansion microscopy enables 25-nm resolution on conventional microscopes. Embo Reports. 19 (9), (2018).
- Chang, J. B., et al. Iterative expansion microscopy. Nature Methods. 14 (6), 593-599 (2017).
- Chozinski, T. J., et al. nanoscale optical imaging of mouse and human kidney via expansion microscopy. Scientific Reports. 8 (1), 10396(2018).
- Richter, K. N., et al. Glyoxal as an alternative fixative to formaldehyde in immunostaining and super-resolution microscopy. The EMBO Journal. 37 (1), 139-159 (2018).
- Rinschen, M. M., et al. Quantitative deep mapping of the cultured podocyte proteome uncovers shifts in proteostatic mechanisms during differentiation. American Journal of Physiology-Cell Physiology. 311 (3), 404-417 (2016).
- Asano, S. M., et al. Expansion microscopy: protocols for imaging proteins and RNA in cells and tissues. Current Protocols in Cell Biology. 80 (1), 56(2018).
- Wen, G., et al. Evaluation of direct grafting strategies via trivalent anchoring for enabling lipid membrane and cytoskeleton staining in expansion microscopy. ACS Nano. 14 (7), 7860-7867 (2020).
- Chen, F., et al. Nanoscale imaging of RNA with expansion microscopy. Nature Methods. 13 (8), 679-684 (2016).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nature Methods. 16 (1), 71-74 (2019).
- Zhao, Y., et al. Nanoscale imaging of clinical specimens using pathology-optimized expansion microscopy. Nature Biotechnology. 35 (8), 757-764 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유