Method Article
Bildgebung der podozytischen Proteine Nephrin, Actin und Podocin mit Erweiterungsmikroskopie
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die vorgestellte Methode ermöglicht die Visualisierung fluoreszierend markierter zellulärer Proteine mit Erweiterungsmikroskopie, die zu einer Auflösung von 70 nm auf einem herkömmlichen Mikroskop führt.
Zusammenfassung
Die Störung des glomerulären Filters, der aus dem glomerulären Endothel, der glomerulären Kellermembran und den Podozyten besteht, führt zu Albuminurie. Podozyten-Fußprozesse enthalten Aktinbündel, die an zytoskelettale Adapterproteine wie Podocin binden. Diese Adapterproteine, wie Podocin, verbinden das Rückgrat des glomerulären Schlitzzwerchfells, wie Nephrin, mit dem Aktin-Zytoskelett. Die Untersuchung der Lokalisation und Funktion dieser und anderer podozytischer Proteine ist für das Verständnis der Rolle des Glomerularfilters bei Gesundheit und Krankheit von wesentlicher Bedeutung. Das vorgestellte Protokoll ermöglicht es dem Benutzer, Actin, Podocin und Nephrin in Zellen mit Superauflösung simtogebungsbildn auf einem herkömmlichen Mikroskop zu visualisieren. Erstens werden Zellen mit einer herkömmlichen Immunfluoreszenztechnik gefärbt. Alle Proteine innerhalb der Probe werden dann kovalent an einem anschwellenden Hydrogel verankert. Durch die Verdauung mit Proteinase K werden strukturelle Proteine spaltet, was eine isotropische Schwellung des Gels im letzten Schritt ermöglicht. Die Dialyse der Probe in Wasser führt zu einer 4-4,5-fachen Ausdehnung der Probe und die Probe kann über ein herkömmliches Fluoreszenzmikroskop abgebildet werden, was eine mögliche Auflösung von 70 nm ergibt.
Einleitung
Albuminurie ist ein Ersatzparameter des kardiovaskulären Risikos und resultiert aus einer Störung des Glomerularfilters1. Der glomeruläre Filter besteht aus dem fenestrated Endothel, der glomerulären Kellermembran und dem durch Podozyten gebildeten Schlitzzwerchfell. Primäre und sekundäre Fußprozesse von Podozyten wickeln sich um die Kapillarwand des Glomerulums2. Die empfindliche Struktur der Fußprozesse wird durch kortikale Aktinbündel aufrechterhalten, die auch als Anker für mehrere Spaltmembranproteine und andere Adapterproteine dienen2. Das Rückgratprotein des Spaltzeswirdn stutztisch heißt Nephrin und interagiert auf homophile Weise mit Nephrinmolekülen gegensätzlicher Podozyten. Über verschiedene Adapterproteine wird Nephrin mit dem Aktin-Zytoskelett2,3verbunden. Mutationen im Nephrin-kodierenden Gen NPHS1 führen zu einem nephrotischen Syndrom des finnischenTyps 4.
Eines der interagierenden Proteine von Nephrin ist Podocin, ein haarförmiges Protein der Stomatin-Familie3. Podocin rekrutiert Nephrin zu Lipidflößen und verbindet es mit dem Aktin-Zytoskelett5. Podocin wird durch das NPHS2-Gen kodiert. Mutationen in NPHS2 führen zu steroidresistentem nephrotischem Syndrom6.
Um Aktin-Adapterproteine zu visualisieren und mitzulokalisieren, können Immunfluoreszenztechniken eingesetzt werden. Leider begrenzt die Beugungsbarriere des Lichts die Auflösung herkömmlicher Fluoreszenzmikroskope auf 200-350 nm7. Neuartige Mikroskopietechniken, z.B. stimulierte Emissionserschöpfung (STED)8, photoaktivierte Lokalisationsmikroskopie (PALM)9, stochastische optische Rekonstruktionsmikroskopie (STORM oder dSTORM) oder Bodenzustandslöschmikroskopie gefolgt von individueller Molekülrückführung (GSDIM)9,10,11, ermöglichen eine Auflösung bis ca. 10 nm. Diese Superauflösungstechniken erfordern jedoch sehr teure Mikroskope, gut ausgebildetes Personal und sind daher in vielen Laboratorien nicht verfügbar.
Die Erweiterungsmikroskopie (ExM) ist eine neuartige und einfache Technik, die eine hochauflösende Bildgebung mit herkömmlichen Mikroskopen ermöglicht und potenziell einer großen Forschungsgemeinschaft zur Verfügung steht12. In der Proteinretentionsexpansionsmikroskopie (proExM) ist die Vonsamkeitsprobe (Zellen oder Gewebe) fixiert und mit Fluorophoren13gefärbt. Proteine innerhalb der Probe werden dann kovalent durch ein kleines Molekül (6-((Acryloyl)amino)Hexanosäure, Succinimidylester, AcX) in ein anschwellendes Hydrogel13verankert. Durch enzymatische Verdauung mit Proteinase K (ProK) behalten Proteine und Fluorophore ihre relative Position innerhalb des Gels nach der Ausdehnung13bei. Nach Schwellung des Gels dehnt sich die Probe bis zum 4,5-fachen (90-fache volumetrische Ausdehnung) aus, was zu einer effektiven lateralen Auflösung von ca. 60-70 nm (300 nm/4,5) führt. Modifikationen dieser Technik können sogar eine 10-fache Erweiterung ermöglichen (1.000-fache volumetrische Expansion), wodurch eine Auflösung von 20-30 nm bei herkömmlichen Mikroskopen14,15,16.
Glomeruläre Strukturen von Maus und menschlichen Nieren wurden über ExM17visualisiert. In diesem Beitrag stellen wir ein detailliertes proExM-Protokoll vor, um Superauflösungsbilder von F-Actin und dem Actin-Adapter-Proteinpodocin in Zellen mit einem herkömmlichen Fluoreszenzmikroskop zu visualisieren.
Protokoll
1. Spaltung und Aussaat von Zellen
- Warmup sterile Dulbecco Modified Eagle es Medium (DMEM) einschließlich 10% fetales Kalbsserum (FCS), sterile Phosphat gepufferte Saline (PBS) und steriles Trypsin auf 37 °C. Aktivieren Sie die saubere Bank.
- Bereiten Sie eine 6-Well-Platte vor, indem Sie jedem Brunnen mit sterilen Zangen einen sterilen Glasdeckelschlupf (10 mm) hinzufügen.
- Legen Sie eine 10 cm Zellkulturschale mit Cos7-Zellen unter die saubere Bank. Unter der sauberen Bank, aspirieren Sie das Medium der Zellen mit einem Vakuumgerät.
- Halten Sie die gewinkelte Zellkulturschale in einer Hand und fügen Sie 10 ml sterile PBS an der Seite der Zellkulturschale hinzu, um das Abspülen von Zellen zu vermeiden. Legen Sie die Zellkultur auf, damit PBS das komplette Zellkulturgericht spült.
- Entfernen Sie die PBS und fügen Sie 1 ml Trypsin in die Mitte der Zellkulturschale und inkubieren für 5 min bei 37 °C.
- Beenden Sie die Trypsinisierungsreaktion, indem Sie 10 ml DMEM einschließlich 10% FCS und Pipette die Zelllösung nach oben und unten mit einem Pipettier hinzufügen, um die Zellen manuell zu trennen.
- Analysieren Sie die Zellenzahl pro Milliliter mit einer Zählkammer.
- Säen 68.000 Zellen in 2 ml Medium pro Brunnen auf die Glasabdeckung rutscht in die 6-Well-Platte. Verteilen Sie die Zellen innerhalb der 6-Well-Platte, indem Sie die Platte vorsichtig horizontal und vertikal schütteln.
HINWEIS: Zellen sollten verstreut liegen. - Inkubieren Sie die Zellen über Nacht in einem 37 °C Inkubator mit 5%CO2.
2. Transfektion von Zellen
- Bereiten Sie zwei sterile 1,5 ml-Röhrchen vor (eine für verdünnte DNA (A) und eine für verdünnte Reagenz für kationische Lipidtransfektion (B)). Pipette 0,75 g Nephrin und 0,75 g Podocin-cDNA-Expressionsplasmid pro Bohrung in ein 1,5 ml-Rohr und verdünnen Sie es in 100 l reduziertes Serummedium pro Brunnen. Fügen Sie dem zweiten 1,5 ml-Rohr 3 l kationisches Lipidtransfektionsreagenz pro Brunnen hinzu und verdünnen Sie es mit 100 l reduziertem Serummedium. Inkubieren Sie beide Reaktionen für 5 min bei Raumtemperatur.
- Kombinieren Sie beide Reaktionen (A und B) zu einem DNA-Lipid-Komplex und inkubieren Sie 20 min bei Raumtemperatur. Fügen Sie vorsichtig 200 l des DNA-Lipid-Komplexes zu jedem Brunnen hinzu.
- Inkubieren Sie die transfizierten Zellen bei 37 °C mit 5%CO2 für 48 h, ohne das Medium zu verändern.
3. Immunolabeling von Zellstrukturen
- Bereiten Sie eine Befestigungslösung (4% (w/v) Paraformaldehyd in PBS, 1 ml/well), die Permeabilisationslösung (0,5% (w/v) Triton X-100 in PBS, 1 ml/well) und eine Blockierendelösung (5% (v/v) Rinderserumalbumin in PBS, 1 ml/well) vor.
- Entfernen Sie das Medium mit einem Vakuumgerät. Fügen Sie 2 ml PBS zu jedem Brunnen hinzu, um zusätzliches Medium zu entfernen. Aspirieren Sie die PBS vollständig.
HINWEIS: Um das Abwaschen von Zellen mit PBS zu vermeiden, achten Sie darauf, PBS nicht direkt auf die Glasabdeckung zu kinpetten. - Fixzellen mit 4% (w/v) Paraformaldehyd (PFA) in PBS für 10 min bei Raumtemperatur gelöst.
HINWEIS: Alternativ können Sie Zellen in 3% (v/v) Glyoxal-Ethanol18fixieren. - Entsorgen Sie PFA und Waschzellen zweimal in PBS (jeweils 2 ml), indem Sie eine direkte Pipettierung auf die Glasabdeckung vermeiden. Permeabilisieren Sie die festen Zellen mit Triton X-100 0.5% (w/v) in PBS für 10 min bei Raumtemperatur.
- Entfernen Sie die Permeabilisationslösung und waschen Sie zweimal mit PBS, wie in Schritt 3.2 angegeben.
- Blockieren Sie die Zellen, indem Sie 1 ml 5% (v/v) BSA in PBS für 1 h bei Raumtemperatur hinzufügen. Inkubieren Sie die Zellen mit 200 l des primären Antikörpers (Antipodocin-Antikörper 1:200 in 1% (v/v) BSA in PBS) über Nacht bei 4 °C.
HINWEIS: Alternativ können Sie den primären Antikörper für 1 h bei Raumtemperatur inkubieren. - Entfernen Sie den primären Antikörper und waschen Sie dreimal mit PBS wie in Schritt 3.2. Fügen Sie den sekundären Antikörper (Ziegenanti-Kaninchen Alexa 488 1:1000 in 1% (v/v) BSA in PBS) für 1 h bei Raumtemperatur hinzu. Halten Sie die Zellen mit einer Box im Dunkeln.
- Den sekundären Antikörper entsorgen und dreimal mit PBS waschen.
- Entfernen Sie PBS und inkubieren Sie die Zellen mit 200 l Anti-Nephrin-Antikörper 1:100 in 1% (v/v) BSA in PBS für 1 h bei Raumtemperatur. Dreimal mit PBS waschen. Halten Sie die Zellen mit einer Box im Dunkeln.
- PBS entfernen und mit dem sekundären Antikörper Esel Anti-Meerschweinchen 633, 1:200, für 1 h bei Raumtemperatur inkubieren. Dreimal mit PBS waschen. Halten Sie die Zellen mit einer Box im Dunkeln.
4. Erweiterungsmikroskopie
- Vorbereitung
- Um die Abstandshalter für die Gelationskammer zu bilden, schlumpft die Glasabdeckung mit einem Diamantmesser #1,0 und #1,5 in 5 mm Streifen (jeweils vier pro Glasschlitten). Positionieren Sie die #1,5 mm Bezugsstreifen so, dass sie ein Quadrat von 2,5 cm Länge auf einer Glasrutsche bilden, und legen Sie sie in eine Färbekammer(Abbildung 2A1-C1). Pipette ein Tröpfchen von ddH2O in die Ecken des Quadrats, um die Glasabdeckung Slip Streifen zueinander und auf die Glasrutsche(Abbildung 2A2-C2).
HINWEIS: Vermeiden Sie eine vollständige Trocknung von ddH2O, da die Haftkraft verloren geht. Bei Bedarf weitere Tröpfchen von ddH2O auftragen. Warten Sie ca. 20 min, damit #1,5 mm Deckslips stabil befestigt werden, bevor Sie mit Schritt 4.1.2 beginnen. - Legen Sie vier #1,0 Glasabdeckungsstreifen pro Deckelschlupf auf die #1,5 mm Bezugsscheine. Befestigen Sie die Streifen, indem Sie ein Tröpfchen auf jedem #1,5 mm Abdeckstreifen verkleben (Abbildung 2A3-C3).
HINWEIS: Die Dicke des Gels sollte mindestens 0,15 mm betragen, um die Handhabung im Verlauf des Protokolls zu erleichtern. Um jedoch übermäßiges Gel auf der Oberseite der Zellen zu vermeiden, halten Sie die Abstandshalterhöhe in der Nähe des Zellsubstrats. Durch die Verwendung von #1.5- und #1.0-Abdeckungsstreifen als Abstandshalter beträgt die Abstandshöhe ca. 0,3 mm. Die Zellen haften am Deckglas (Höhe 0,12 mm). Daher wird das Gel dick genug sein, um zu handhaben, aber übermäßiges Gel auf der Oberseite der Zellen wird vermieden, mit #1.0 und #1.5 Glasstreifen als Abstandshalter zu bedecken. - Für den Gelationskammerdeckel ein Deckglas (#1.5) mit Paraffinfolie einwickeln. Vermeiden Sie Falten oder Schmutz auf der Paraffinfolie(Abbildung 2A5-C5).
- Um die Abstandshalter für die Gelationskammer zu bilden, schlumpft die Glasabdeckung mit einem Diamantmesser #1,0 und #1,5 in 5 mm Streifen (jeweils vier pro Glasschlitten). Positionieren Sie die #1,5 mm Bezugsstreifen so, dass sie ein Quadrat von 2,5 cm Länge auf einer Glasrutsche bilden, und legen Sie sie in eine Färbekammer(Abbildung 2A1-C1). Pipette ein Tröpfchen von ddH2O in die Ecken des Quadrats, um die Glasabdeckung Slip Streifen zueinander und auf die Glasrutsche(Abbildung 2A2-C2).
- Verankerung und Polymerisation (Gelation)
- Bereiten Sie den Verankerungspuffer vor (siehe Tabelle 1). Für die Verankerungsbehandlung PBS durch 250 l Verankerungspuffer pro Brunnen direkt auf den Glasdeckelschlupf ersetzen und bei Raumtemperatur 3 h inkubieren. Halten Sie Substrat im Dunkeln mit einer Box.
HINWEIS: Alternativ können Sie über Nacht bei Raumtemperatur inkubieren. Bereiten Sie frischen Ankerpuffer für jedes Experiment vor und warten Sie 10-15 min, bis er richtig aufgelöst ist. 6-(Acryloyl)amino)hexanosäure, succinimidylester (AcX) sollte alle 4-5 Monate ersetzt werden, um eine ausreichende Verankerung zu gewährleisten. - Entfernen Sie den Verankerungspuffer und waschen Sie einmal mit 1,5 ml PBS pro Brunnen.
- Um Aktinfasern zu färben, tauen Sie eine ExM-kompatible Phalloidin-Lösung auf und inkubieren Sie das Phalloidin (5 l Phalloidin in 195 l von 1% (v/v) BSA in PBS/Well) für 45 min bei Raumtemperatur. Halten Sie Proben im Dunkeln mit einer Box.
- In der Zwischenzeit Natriumacrylat inddH2O mit einem Rührgerät auflösen. Auf Eis die Monomerlösung vorbereiten (siehe Tabelle 1).
HINWEIS: Gelöstes Natriumacrylat sollte eine klare und farblose Lösung sein. Wenn die Lösung gelb ist, durch neues Natriumacrylat ersetzen. - Bereiten Sie die Gelierlösung auf Eis vor (siehe Tabelle 1). Pipette Ammoniumperoxidsulfat (APS) in die Gelierlösung direkt vor dem Auftragen der Gelierlösung auf die Gelierkammer.
- Phalloidin aus den Zellen nehmen und bei Raumtemperatur zweimal mit 1,5 ml PBS waschen. Lassen Sie 1,5 ml PBS im Brunnen, um die Entnahme der Probe auf dem Glasdeckelzuschlag zu erleichtern.
- Legen Sie die Zellen auf dem Deckglas mit Zangen und einer Kanüle in die Gelierkammer, um den Deckelglasschlupf von der 6-Well-Platte zu heben.
HINWEIS: Die Zellen sollten sich auf der Oberseite des Glasdeckels befinden. Der Glasdeckel-Slip sollte die Abstandshalter nicht berühren. - Fügen Sie APS der Gelierlösung hinzu und wirbeln Sie in Kürze. Pipette 200 L Gelierlösung auf der Probe (Abbildung 2A4-C4). Schließen Sie vorsichtig die Gelationskammer, indem Sie Luftblasen innerhalb des Gels vermeiden (Abbildung 2A5-C5).
- Inkubieren Sie die Gelierkammer für mindestens 1 h bei 37 °C, um das Gel in der nassen Färbekammer zu polymerisieren.
- Bereiten Sie den Verankerungspuffer vor (siehe Tabelle 1). Für die Verankerungsbehandlung PBS durch 250 l Verankerungspuffer pro Brunnen direkt auf den Glasdeckelschlupf ersetzen und bei Raumtemperatur 3 h inkubieren. Halten Sie Substrat im Dunkeln mit einer Box.
- Homogenisierung (Verdauung)
- Nehmen Sie die Färbekammer aus dem Inkubator. Um den Gelationskammerdeckel zu öffnen, führen Sie eine Rasierklinge zwischen deckel und Abstandhalter ein. Entfernen Sie den Deckel vorsichtig. Entfernen Sie die Abstandshalter mit der Rasierklinge und beseitigen Sie alle zusätzlichen Gel, indem Sie es mit der Rasierklinge schneiden.
- Setzen Sie die Folie mit dem Gel und Deckenglas in eine Schale mit PBS gefüllt. Durch sanftes Schütteln entfernen Sie das abgetrennte Deckglas aus dem Gel. Um das Gel leicht von der Schale zu entfernen, legen Sie die Folie unter das Gel, um das Gel an der Folie zu befestigen.
- Mit dem Gel auf der Folie, teilen Sie das Gel in kleine Stücke (Viertel des Gels ist in zwei bis drei Stücke unterteilt) mit der Rasierklinge. Drücken Sie vorsichtig ein Stück Gel in einen Brunnen einer 6-Well-Platte mit Glasboden und umschließen Sie es mit einem Pinsel. Halten Sie das Gel mit einer kleinen Menge PBS mit dem Pinsel, um eine Austrocknung des Gels zu vermeiden.
HINWEIS: Die Zellen zeigen nach unten. - Nehmen Sie mit einem invertierten Mikroskop Übersichtsbilder mit niedriger numerischer Blende, um den Expansionsfaktor nach der Expansion zu bestimmen.
- Bereiten Sie den Verdauungspuffer vor (siehe Tabelle 1). Proteinase K bis 4 U/ml im Verdauungspuffer verdünnen, um die Verdauungslösung zu erhalten.
HINWEIS: Der Verdauungspuffer ohne Proteinase K kann 1-2 Wochen bei 4 °C gelagert werden. - Fügen Sie jedem Brunnen 500 l der Verdauungslösung hinzu und tauchen Sie das Gel in die Lösung ein. Lassen Sie es über Nacht bei Raumtemperatur verdauen und schließen Sie den Deckel, um die Proben im Dunkeln zu halten.
HINWEIS: Alternativ lassen Sie es bei 37 °C für 1 h verdauen.
- Expansion
- Entfernen Sie die Verdauungslösung mit einer Pipette und entsorgen Sie sie. 1 ml ddH2O hinzufügen. Das eingetauchte Gel 10 min bei Raumtemperatur inkubieren.
- Entfernen Sie das Wasser und fügen Sie 1 ml frische ddH2O. Warten Sie auf 10 min und fahren Sie Wasser alle 10 min, bis ein Plateau der Expansion erreicht ist.
HINWEIS: Eine Probenerweiterung bis zum 4,5-fachen ist erreichbar. Das Gel wird optisch klar.
- Imaging
- Entfernen Sie das Wasser aus dem Gel und starten Sie direkt mit der Mikroskopie. Verwenden Sie mit hilfe eines invertierten Mikroskops in erster Linie ein Luftobjektiv (niedrige Vergrößerung), um abgebildete Zellen im Zustand vor der Ausdehnung zu finden (Schritt 4.3.4).
- Wechseln Sie zu einem 40x (Öl/Wasser) und 63x Objektiv für eine bessere Auflösung. Erregen Sie mit der Wellenlänge des Interesses und nehmen Sie das Bild über die Kamera.
- Validierung
- Sehen Sie sich ein Übersichtsbild des Beispiels an. Suchen und anpassen Sie dieselben Strukturen innerhalb des Beispiels, die in Schritt 4.3.4 abgebildet wurden. Verwenden Sie den Kanal mit dem besten Signal-Rausch-Verhältnis für die Validierung (Abbildung 3 A-B).
HINWEIS: Passen Sie die Bildparameter an, um eine ähnliche Helligkeit zu erzielen wie in dem Bild, das im nicht erweiterten Zustand aufgenommen wurde (Schritt 4.3.4). - Überlagern Sie Vor- und Nacherweiterungsbilder, indem Sie sie mit ImageJ drehen und verschieben. Verwenden Sie das Entfernungsmesswerkzeug in ImageJ, um die Abstände zwischen klar identifizierbaren Strukturen zu messen. Messen Sie mindestens 10 verschiedene Strukturen.
HINWEIS: Alternativ können Sie ein Python-Skript verwenden, um Entfernungen zu messen, wie in14beschrieben. - Berechnen Sie den Expansionsfaktor, indem Sie Die Messungen nach/vor der Ausdehnung dividieren.
- Um Verzerrungen zu bestimmen, nehmen Sie Bilder mit einer höheren numerischen Blende auf (Abbildung 4A-B). Überlagern Sie diese Bilder und analysieren Sie sie mit ImageJ oder wie in15beschrieben.
HINWEIS: Die Bestimmung von Verzerrungen sollte routinemäßig, aber nicht unbedingt für jede Probe durchgeführt werden.
- Sehen Sie sich ein Übersichtsbild des Beispiels an. Suchen und anpassen Sie dieselben Strukturen innerhalb des Beispiels, die in Schritt 4.3.4 abgebildet wurden. Verwenden Sie den Kanal mit dem besten Signal-Rausch-Verhältnis für die Validierung (Abbildung 3 A-B).
Ergebnisse
Das Konzept und das Timing dieses proExM-Protokolls sind in Abbildung 1dargestellt. Am 5. Tag werden transfizierte Zellen fixiert und mit fluoreszierenden Antikörpern gefärbt, die auf das Protein von Interesse abzielen (Abbildung 1A,B). Am 6. Tag führt die Behandlung mit AcX zur Bildung von Amingruppen auf allen Proteinen (einschließlich Fluorophoren) (Abbildung 1A,B)12. Bei der Polymerisation des Hydrogels binden diese Amingruppen kovalent an das Hydrogel (Tag 6). Nach der Polymerisation des Gels wird die Homogenisierung (Verdauung) mit Proteinase K durchgeführt, was zur Zerstörung struktureller Proteine der Zelle führt (Tag 6, Abbildung 1A,B). Fluoreszierend markierte Antikörper bleiben nach der Verdauung weitgehend erhalten. Aufgrund der Störung struktureller Proteine führt die Wasserdialyse des Hydrogels zu einer isotropen Ausdehnung der Zelle innerhalb des Hydrogels an Tag 7 (Abbildung 1A,B). Die Bildgebung der Probe erfolgt mit einem herkömmlichen Fluoreszenzmikroskop (Abbildung 1A). Es sollte eine Datenvalidierung durchgeführt werden, um den Erweiterungsfaktor zu bestimmen und Verzerrungen auszuschließen (Abbildung 1A).
Um die Zelldehnung isotrop durchführen zu können, ist der Gelationsschritt unerlässlich. Abbildung 2 zeigt die Seiten- und Oberansicht einer Gelationskammer. Glasabdeckungsschlüpfer bilden die Abstandshalter der Gelationskammer (Abbildung 2A1-3/C1-3). Das Deckglas mit den festen und gebeizten Zellen wird mit den Zellen nach oben auf einem Glasschlitten positioniert (Abbildung 2A4-C4). Der Deckel der Gelationskammer ist mit Parafilm umwickelt und blasenfrei geschlossen (Abbildung 2A5-C5).
Dieses ExM-Protokoll ermöglicht eine Erweiterung des bis zu vierfachen Protokolls. Um den Expansionsfaktor zu bestimmen, ist es wichtig, Zellen vor und nach der Expansion abzubilden (Abbildung 3A + B). Unzureichende Verankerung und Homogenisierung können zu Verzerrungen und Brüchen der Zellen führen. Abbildung 4A + B zeigt repräsentative Beispiele von gebrochenen Zellen in verschiedenen Vergrößerungsbildern.
Diese Methode kann verwendet werden, um die Kolokalisierung von F-Actin- und Aktin-Adapterproteinen, z. B. Podocin und Nephrin (Abbildung 5), zu untersuchen. Podocin ist grün dargestellt, während Actin blau beschriftet ist (Abbildung 5). Nephrin ist grün markiert. Weiße Bereiche weisen auf die Kolokalisierung hin.
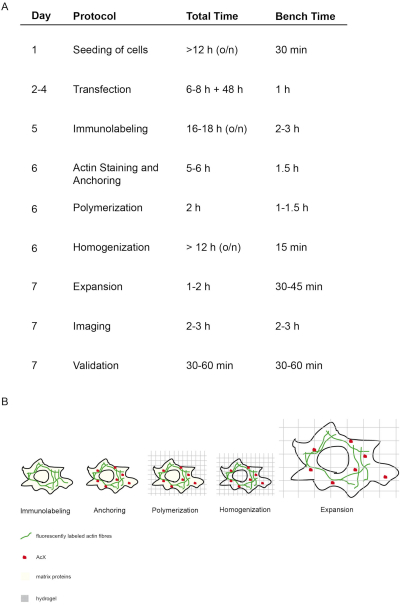
Abbildung 1: Konzept und Zeitpunkt dieses ExM-Protokolls. (A) In der Spalte "Protokoll" wird jeder Schritt des Protokolls beschrieben. (A + B) Nach der Aussaat und Transfizierung von Zellen wird eine immunfluoreszierende Etikettierung durchgeführt (Immunolabeling). (A + B) Das kleine Molekül AcX (roter Punkt) bindet an alle Proteine und verankert sie am Hydrogel (Anchoring). (A + B) Durch Polymerisation werden alle Proteine einschließlich Fluorophoren kovalent über AcX an das Hydrogel (Polymerisation) gebunden. (A + B) Homogenisierung führt zur Verdauung von strukturellen Matrixproteinen. (A + B) Die Expansion erfolgt durch Dialyse im Wasser. (A) Die Bildgebung und Validierung der Bildgebung schließt das Experiment ab. (A) Das gesamte Protokoll benötigt 7 Tage (Spalte "Tag") mit vielen Inkubationsschritten (Gesamtzeit pro Tag Spalte "Gesamtzeit"), aber die tatsächliche Bankzeit ist viel weniger, wie in der jeweiligen Spalte "Bench Time" angegeben. Geändert von14. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
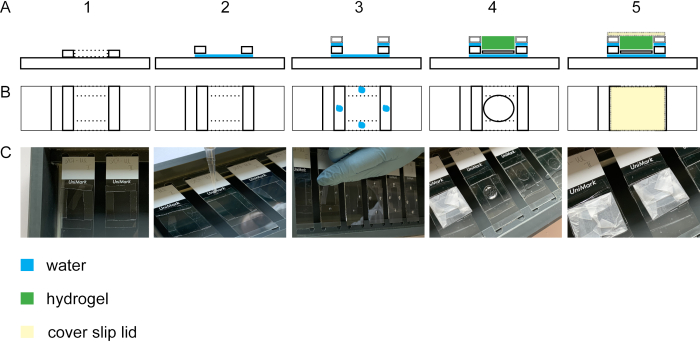
Abbildung 2:Bau der Gelationskammer. Seitenansicht (A1) und Ansicht von oben (B1 + C1) einer Glasrutsche mit vier #1,5 Abdeckstreifen. Durch Hinzufügen eines Tröpfchens Wasser zwischen der Glasrutsche und der Abdeckung Slip Streifen, die Streifen werden auf der Glasrutsche haften(Seitenansicht A2, Ansicht von oben B2, C2). Wassertröpfchen auf den #1,5 Abdeckstreifen führen zur Haftung von #1,0-Abdeckstreifen, die auf den #1,5 Abdeckstreifen gelegt werden(Seitenansicht A3, Ansicht von oben B3, C3). Die Probe auf dem Deckbeleg wird mit Zangen in der Mitte des Rechtecks platziert. Das Gel ist oben pipetiert (Seitenansicht A4, Obere Ansicht B4, C4). (A5) Seitenansicht und Ansicht von Oberseite (B5 und C5) der montierten Gelationskammer einschließlich des geschlossenen Deckels, der aus einem in Parafilm gewickelten Deckschauch besteht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
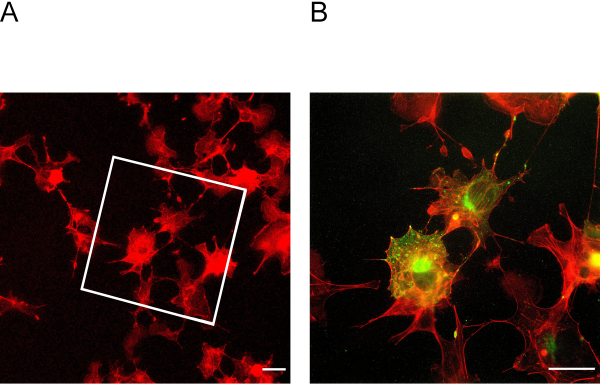
Abbildung 3: Zellen vor und nach der Expansion. (A) Zellen vor der Expansion für Actin gebeizt. Das Feld gibt an, in welchem Bereich die erweiterten Zellen in Abbildung 3B liegen. (B) Zellen nach Derexpansion für Actin in Rot und Podocin in Grün gefärbt. Podocin lokalisiert mit Actin in der Zellperipherie. Skala bar = 5 'm, Expansionsfaktor = 2. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Verzerrungen und Brüche von Zellen. (A + B) Repräsentative mikroskopische Bilder von cos7-Zellen immun-stainiert für Actin (rot). Die Zellen wurden fixiert, gebeizt, verankert, verdaut und erweitert. (A) Brüche von Zellen. Pfeile zeigten gebrochene Bereiche an. Skala bar = 5 'm, Expansionsfaktor = 4. (B) Rupturen und Verzerrung endender Zellen. Weiße Pfeile zeigten gebrochene Bereiche an. Skala bar = 5 'm, Expansionsfaktor = 4. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
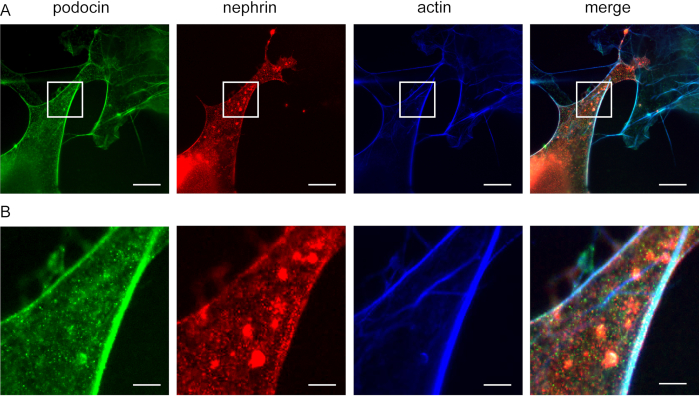
Abbildung 5:Podocin lokalisiert mit Nephrin und Actin. Cos7-Zellen immunfluoreszierend für Podocin, Actin und Nephrin gekennzeichnet. (A) Cos7-Zellen für Podocin (grün), Actin (blau) und Nephrin (rot) mit ExM gebeizt. Podocin lokalisiert mit Actin und Nephrin. Skalenbalken = 200 nm, Expansionsfaktor = 4. (B) Vergrößerung des angegebenen Bereichs in (A), Skalenleiste = 40 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Verankerungspuffer | Endkonzentration | ||
| NaHCO3 | 150 mM | ||
| Acryloyl-X, SE (AcX) | 0,1 mg/ml | ||
| Monomer-Lösung | Lagerlösungskonzentration g/100 ml | Menge (ml) | Endkonzentration (g/100 ml) |
| Natriumacrylat | 38 | 2.25 | 8.6 |
| Acrylamid | 50 | 0.5 | 2.5 |
| N,N'-Methylenbisacrylamid | 2 | 0.75 | 0.15 |
| Natriumchlorid | 29.2 | 4 | 11.7 |
| Pbs | 10x | 1 | 1x |
| Wasser | 0.75 | ||
| gesamt | 9.4 | ||
| Gelling-Lösung | Lagerlösungskonzentration | Menge (l) | Endkonzentration (mg/ml) |
| Monomerlösung | Na | 190 | Na |
| Aps | 10% | 4 | 0.1 |
| TEMED | >99% | 4 | 0.1 |
| Wasser | Na | 2 | Na |
| gesamt | 200 | ||
| Verdauungslösung | Endkonzentration | ||
| Tris Cl, pH 8,0 | 1 M | ||
| EDTA pH 8,0 | 0,5 M | ||
| Triton X-100 | 0.005% | ||
| Guanidin HCL | 8 M | ||
| Wasser | |||
| Proteinase K | 4 U/ml |
Tabelle 1: Lösungen für ExM.
Diskussion
Die vorgestellte Methode ermöglicht es dem Forscher, zelluläre Proteine, z.B. Podocin, Nephrin und zytoskelettale Komponenten, z.B. F-Actin, zu visualisieren. Innerhalb dieses Protokolls werden transfizierte cos7-Zellen als Modell verwendet, um die Wechselwirkung von Spaltmembranproteinen mit F-Actin zu untersuchen. Leider drücken verewigte Podozytenzelllinien nicht genügend endogene Mengen an Spaltmembranproteinenaus 19.
Mit dieser Methode können zelluläre Proteine mit nanoskaliger Auflösung mit einem herkömmlichen Fluoreszenzmikroskop visualisiert werden. Die wichtigsten Schritte innerhalb des Protokolls sind: 1) ausreichende Verankerung von Proteinamingruppen am Hydrogel mit AcX, 2) ausreichende Polymerisation des Hydrogels, 3) optimales Timing für die Verdauung und 4) Auswahl kompatibler Fluorophore.
Die Verankerung zellulärer Proteine am Hydrogel ist für diese Methode unerlässlich, um die Position des Proteins innerhalb des Hydrogels während der Expansion zu erhalten. AcX ist ein kleines Molekül, das an Amingruppen von Proteinen in Zellen und Geweben bindet. AcX schafft ein Carbon-Carbon-Doppelband mit Proteinen, das die Aufnahme der Proteine in das Hydrogel im Polymerisationsschritt20ermöglicht. AcX integriert auch Antikörper, so dass die Etikettierung mit Immunfluoreszenz-Antikörpern vor der AcX-Behandlung durchgeführt werden kann. Unzureichende Verankerung kann zu Brüchen und Verzerrungen der Zellen führen. Aufgrund der Modifikation von Amingruppen durch Fixative muss man die Fixierung oder die Zeit der Fixierung optimieren. Darüber hinaus kann eine unzureichende Lagerung oder nicht optimierte Verankerungsbedingungen zu Brüchen und Verzerrungen führen. Basierend auf unserer Erfahrung verliert AcX seine optimale Wirkung, wenn es mehr als 3-4 Monate verwendet wird.
Die Polymerisation des Gels ist temperaturabhängig. Wir empfehlen daher, die Polymerisationslösungen auf Eis zu halten, bevor sie in die Gelierkammer geleitet werden. Darüber hinaus sollte die Bearbeitungszeit des Gelationsschritts kurz gehalten werden (weniger als 5 min), um eine vorzeitige Gelbildung zu vermeiden. Durch das gründliche Mischen der Polymerisationslösung wird eine ungleichmäßige Polymerisation verhindert. Luftblasen beeinflussen den Expansionsprozess beim Berühren der Probe und können durch Hinzufügen weiterer Polymerisationslösungen verhindert werden.
Nach der Einbindung der zellulären Proteine in das Hydrogel ist der mechanische Homogenisierungsschritt (oder die Verdauung) erforderlich, um die Ausdehnung zu gewährleisten. Es gibt verschiedene Methoden, z.B. Wärme- und Waschmittel oder enzymatische Verdauung, und müssen an die untersuchte Probe12,14,20angepasst werden. Innerhalb dieses Protokolls wird die Protease Proteinase K zur enzymatischen Verdauung eingesetzt. Proteinase K wird in einer Dosierung angewendet, die ausreicht, um strukturelle Proteine zu zerstören und gleichzeitig die meisten anderen Proteine, einschließlich fluoreszierender Antikörper, zu erhalten12. Wenn die Verdauung unvollständig ist, ist die Probenerweiterung unzureichend. Darüber hinaus kann die Probe während des Expansionsprozesses reißen (Abbildung 3). Wenn eine unzureichende Probenerweiterung aufgetreten ist, wird ein Wasseraustausch empfohlen. Alternativ kann die Zeit für die enzymatische Verdauung angepasst oder ein neues Aliquot der Proteinase K geöffnet werden.
Wenn die Probe überverdaut ist, werden fluoreszenzsignale verringert. In diesem Fall sollte die Verdauungszeit verkürzt werden. In ExM im Allgemeinen wird die Fluoreszenzsignalintensität pro Volumeneinheit durch die volumetrische Ausdehnung der Probe14reduziert. Daher müssen längere Belichtungszeiten während der Bildgebung berücksichtigt werden.
Es ist wichtig, ExM-kompatible Fluorophore auszuwählen. Cyaninfarbstoffe werden während des Polymerisationsschritts13abgebaut. Fluoreszenzproteine auf Basis von Bakteriophytochromen sind ebenfalls weitgehend zerstört13. Die meisten GFP-ähnlichen Proteine bleiben jedocherhalten 13. Darüber hinaus kann Streptavidin auch vor der Erweiterung angewendet werden, wodurch posttranslationale Modifikationen wie S-Nitrolysation über ein kleines Molekül-Tag13gekennzeichnet werden.
Phalloidin, ein kleines Etikettierungsmolekül, das auf das Aktin-Zytoskelett ausgerichtet ist, ist nicht kompatibel mit ExM21. Um eine unzureichende Verankerung von Phalloidin zu überwinden, wurde die dreiwertige Verankerung (TRITON) eingeführt21. Dieser Ansatz bietet gleichzeitiges Targeting, Etikettieren und Verpflanzen von Biomolekülen21.
Diese Methode kann modifiziert werden, um RNA-Moleküle (ExFish)22zu färben. In der iterativen Erweiterungsmikroskopie (iExM) oder X10-Mikroskopie kann die Auflösung von 60-70 nm durch Auftragen eines zweiten anschwellenden Gels innerhalb des ersten expandierten Hydrogels oder durch Durchführung eines einzigen Expansionsschritts mit einem anderen Hydrogel15,16auf ca. 25 nm erweitert werden. Ultrastruktur-Erweiterungsmikroskopie (U-ExM) ermöglicht eine Superauflösung von Proteinen, die ihre Zuordnung zu einem ultrastrukturellen Element (z. B. Mitochrondrien, Mikrotubuli) erhalten23. Eine Kombination aus ExFish (RNA und DNA) und proExM Methoden wurden zuvor auchdurchgeführt 22,24. Das vorgestellte Protokoll verwendet transfizierte cos7-Zellen als Modell, um Spaltmembranproteine zu untersuchen. Wir erwarten, dass andere ansässige kultivierte Nierenzellen, z.B. HEK293T-Zellen, für dieses Protokoll in ähnlicher Weise verwendet werden können. Je nach Zelllinie müssen möglicherweise Anpassungen für die unterschiedlichen Kultivierungs- und Transfektionsbedingungen vorgenommen werden.
ExM verbessert die Auflösung von immunbefleckten Proben um etwa das Vierfache und erreicht eine seitliche räumliche Auflösung von 70 nm13. Im Vergleich zu anderen Super-Resolution-Techniken wird ExM auf einem herkömmlichen Fluoreszenzmikroskop13,14durchgeführt. Daher ist für die Durchführung der ExM-Methode14keine teure Ausrüstung oder speziell geschultes Personal erforderlich. Auch wenn nicht alle Fluorophore mit ExM kompatibel sind, gibt es in der Regel viele verfügbare Antikörper mit optimierten Fluorophoren mit fotophysikalischen Eigenschaften, die für die Superauflösungsmikroskopie14benötigt werden. Der Hauptnachteil dieser Methode ist, dass ExM mit Live-Samples12,14inkompatibel ist.
In Zukunft kann die Verbesserung der chemischen Zusammensetzung des Hydrogels zu einer noch höheren räumlichen Auflösung12führen. Die Kombination verschiedener Protokolle kann auch die Visualisierung von Proteinen, RNA, DNA oder Lipiden in Komplexen innerhalb derselben Probe mit einer derart hohen Auflösung12ermöglichen.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Die Autoren danken Blanka Duvnjak und Nikola Kuhr für ihre hervorragende technische Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| Acrylamide >99% | Sigma-Aldrich | A3553-100G | |
| 6-((Acryloyl)amino)hexanoic acid, succinimidyl ester, Acryloyl-X, SE | invitrogen | A-20770 | store up to 4 months |
| APS | Sigma-Aldrich | A3678-25G | |
| Deckgläser (cover glasses) | Engelbrecht | K12432 | 24x32mm #1.0 |
| Diamont cutter | VWR | 201-0392 | for cutting the cover slips |
| Guanidine HCl | Sigma-Aldrich | G3272-100G | 8M Stock can be kept at RT |
| Marten hair paintbrush | Leon Hardy | 3 (770) | |
| "Menzel" Deckgläser (cover glasses) | Thermo Fischer | 15654786 | 24x24mm #1.5 |
| N,N`-Methylenbisacrylamide | Sigma-Aldrich | M7256-25G | |
| Objektträger UniMark | Marienfeld | 703010 | |
| Proteinase K | New England Biolabs | P8107S | |
| Sodium Acrylate | Sigma-Aldrich | 408220 | check purity |
| Sodium Bicarbonate | Sigma-Aldrich | S5761 | |
| Staining chamber | produced at the university's workshop | ||
| TEMED | ROTH | 2367.1 | |
| 6-Well glass bottom plates | Cellvis | P06-1.5H-N | |
| Antibodies | |||
| Actin-ExM 546 | chrometra | non-available | 1:40 |
| Anti Podocin produced in rabbit | Sigma | P-0372-200UL | 1:200 |
| Donkey anti guinea-pig CF633 | Sigma | SAB4600129-50UL | 1:200 |
| Goat anti rabbit 488 | Life Technologies | A11034 | 1:1000 |
| Guinea pig anti nephrin | Origene | BP5030 | 1:100 |
| Software | |||
| FIJI | |||
| Visiview | |||
| microscope | |||
| AXIO Observer Z1 | Zeiss | non-available |
Referenzen
- Matsushita, K., et al. Association of estimated glomerular filtration rate and albuminuria with all-cause and cardiovascular mortality in general population cohorts: a collaborative meta-analysis. Lancet. 375 (9731), 2073-2081 (2010).
- Faul, C., Asanuma, K., Yanagida-Asanuma, E., Kim, K., Mundel, P. Actin up: regulation of podocyte structure and function by components of the actin cytoskeleton. Trends in Cell Biology. 17 (9), 428-437 (2007).
- Saleem, M. A., et al. Co-localization of nephrin, podocin, and the actin cytoskeleton - Evidence for a role in podocyte foot process formation. American Journal of Pathology. 161 (4), 1459-1466 (2002).
- Kestila, M., et al. Positionally cloned gene for a novel glomerular protein - nephrin - is mutated in congenital nephrotic syndrome. Molecular Cell. 1 (4), 575-582 (1998).
- Huber, T. B., et al. Podocin-mediated recruitment of nephrin into lipid rafts is required for efficient nephrin signaling. Journal of the American Society of Nephrology. 14, 8(2003).
- Boute, N., et al. NPHS2, encoding the glomerular protein podocin, is mutated in autosomal recessive steroid-resistant nephrotic syndrome. Nature Genetics. 24 (4), 349-354 (2000).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. Journal of Cell Science. 124 (10), 1607-1611 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440 (7086), 935-939 (2006).
- van de Linde, S., et al. Direct stochastic optical reconstruction microscopy with standard fluorescent probes. Nature Protocols. 6 (7), 991-1009 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Testa, I., et al. Multicolor fluorescence nanoscopy in fixed and living cells by exciting conventional fluorophores with a single wavelength. Biophysical Journal. 99 (8), 2686-2694 (2010).
- Wassie, A. T., Zhao, Y., Boyden, E. S. Expansion microscopy: principles and uses in biological research. Nature Methods. 16 (1), 33-41 (2019).
- Tillberg, P. W., et al. Protein-retention expansion microscopy of cells and tissues labeled using standard fluorescent proteins and antibodies. Nature Biotechnology. 34 (9), 987-992 (2016).
- Truckenbrodt, S., Sommer, C., Rizzoli, S. O., Danzl, J. G. A practical guide to optimization in X10 expansion microscopy. Nature Protocols. 14 (3), 832-863 (2019).
- Truckenbrodt, S., et al. X10 expansion microscopy enables 25-nm resolution on conventional microscopes. Embo Reports. 19 (9), (2018).
- Chang, J. B., et al. Iterative expansion microscopy. Nature Methods. 14 (6), 593-599 (2017).
- Chozinski, T. J., et al. nanoscale optical imaging of mouse and human kidney via expansion microscopy. Scientific Reports. 8 (1), 10396(2018).
- Richter, K. N., et al. Glyoxal as an alternative fixative to formaldehyde in immunostaining and super-resolution microscopy. The EMBO Journal. 37 (1), 139-159 (2018).
- Rinschen, M. M., et al. Quantitative deep mapping of the cultured podocyte proteome uncovers shifts in proteostatic mechanisms during differentiation. American Journal of Physiology-Cell Physiology. 311 (3), 404-417 (2016).
- Asano, S. M., et al. Expansion microscopy: protocols for imaging proteins and RNA in cells and tissues. Current Protocols in Cell Biology. 80 (1), 56(2018).
- Wen, G., et al. Evaluation of direct grafting strategies via trivalent anchoring for enabling lipid membrane and cytoskeleton staining in expansion microscopy. ACS Nano. 14 (7), 7860-7867 (2020).
- Chen, F., et al. Nanoscale imaging of RNA with expansion microscopy. Nature Methods. 13 (8), 679-684 (2016).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nature Methods. 16 (1), 71-74 (2019).
- Zhao, Y., et al. Nanoscale imaging of clinical specimens using pathology-optimized expansion microscopy. Nature Biotechnology. 35 (8), 757-764 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten