Method Article
Imagerie des protéines podocytiques Nephrin, Actin et Podocin avec microscopie d’expansion
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
La méthode présentée permet la visualisation de protéines cellulaires étiquetées fluorescentes avec microscopie d’expansion conduisant à une résolution de 70 nm sur un microscope conventionnel.
Résumé
La perturbation du filtre glomerular composé de l’endothélium glomerular, de la membrane glomerular de sous-sol et des podocytes, a comme conséquence l’albuminuria. Les processus de pied de podocyte contiennent des faisceaux d’actine qui se lient aux protéines cytosquelettiques d’adaptateur telles que la podocine. Ces protéines adaptateurs, comme la podocine, relient l’épine dorsale du diaphragme fente glomérique, comme la néphrine, au cytosquelette d’actine. L’étude de la localisation et de la fonction de ces protéines podocytiques et d’autres est essentielle à la compréhension du rôle du filtre glomerular dans la santé et la maladie. Le protocole présenté permet à l’utilisateur de visualiser l’actine, la podocine et la néphrine dans les cellules avec imagerie à super résolution sur un microscope conventionnel. Premièrement, les cellules sont tachées d’une technique conventionnelle d’immunofluorescence. Toutes les protéines de l’échantillon sont ensuite ancrées de façon covalente dans un hydrogel gonflé. Grâce à la digestion avec la proteinase K, les protéines structurelles sont fendillées permettant un gonflement isotropical du gel dans la dernière étape. La dialyse de l’échantillon dans l’eau entraîne une expansion de 4 à 4,5 fois de l’échantillon et l’échantillon peut être photographié au microscope à fluorescence conventionnel, ce qui rend une résolution potentielle de 70 nm.
Introduction
L’albuminurie est un paramètre de substitution du risque cardiovasculaire et résulte de la perturbation du filtre glomerular1. Le filtre glomerular est composé de l’endothélium fenestrated, de la membrane glomerular de sous-sol et du diaphragme de fente formé par des podocytes. Les processus primaires et secondaires de pied des podocytes enveloppent autour de la paroi capillaire du glomerulum2. La structure délicate des processus de pied est maintenue par des faisceaux corticals d’actine qui servent également d’ancres pour les protéines multiples de diaphragme de fente et d’autres protéines d’adaptateur2. La protéine dorsale du diaphragme fendue est appelée néphrine et interagit de manière homophilique avec les molécules de néphrine des podocytes opposés. Grâce à diverses protéines adaptateurs, la néphrine est liée au cytosquelette d’actine2,3. Les mutations dans le gène d’encodage de néphrine NPHS1 mènent au syndrome néphrotique du typefinlandais 4.
L’une des protéines en interaction de la néphrine est la podocine, une protéine en épingle à cheveux de la famille des stomatines3. Podocin recrute de la néphrine aux radeaux lipidiques et la relie au cytosquelette d’actine5. La podocine est codée par le gène NPHS2. Les mutations dans NPHS2 mènent au syndrome néphrotique stéroïde-résistant6.
Pour visualiser et co-localisé les protéines adaptateurs de l’actine, des techniques d’immunofluorescence peuvent être utilisées. Malheureusement, la barrière de diffraction de la lumière limite la résolution des microscopes conventionnels de fluorescence à 200-350 nm7. De nouvelles techniques de microscopie, par exemple, l’épuisement des émissions stimulées (STED)8,la microscopie de localisation photoactivée (PALM)9,la microscopie de reconstruction optique stochastique (STORM ou dSTORM) ou la microscopie de suppression de l’état au sol suivie d’un retour individuel des molécules (GSDIM)9,10,11, permettent une résolution jusqu’à environ 10 nm. Cependant, ces techniques de super résolution nécessitent des microscopes très coûteux, du personnel bien formé et ne sont donc pas disponibles dans de nombreux laboratoires.
La microscopie d’expansion (ExM) est une technique nouvelle et simple qui permet l’imagerie par super résolution avec des microscopes conventionnels et qui est potentiellement disponible pour une grande communauté derecherche 12. Dans la microscopie d’expansion de rétention de protéine (proExM), l’échantillon d’intérêt (cellules ou tissu) est fixé et souillé avec des fluorophores13. Les protéines de l’échantillon sont ensuite ancrées de façon covalente par une petite molécule (6-((Acryloyl)amino)acide hexanoïque, succinimidyl ester, AcX) dans un hydrogel gonflé13. Grâce à la digestion enzymatique avec proteinase K (ProK), les protéines et les fluorophores maintiennent leur position relative dans le gel après expansion13. Après gonflement du gel, l’échantillon se dilate jusqu’à 4,5 fois (expansion volumétrique de 90 fois) menant à une résolution latérale efficace d’environ 60-70 nm (300 nm/4.5). Les modifications de cette technique peuvent même permettre une expansion 10 fois (expansion volumétrique 1000 fois), rendant une résolution de 20-30 nm sur les microscopes conventionnels14,15,16.
Des structures glomerular de souris et de reins humains ont été visualisées par l’intermédiaire d’ExM17. Dans cet article, nous présentons un protocole proExM détaillé pour visualiser des images de super résolution de F-actin et de la podocine de protéine d’actine-adaptateur dans les cellules utilisant un microscope conventionnel de fluorescence.
Protocole
1. Fractionnement et ensemencement des cellules
- Réchauffez le milieu de l’aigle modifié (DMEM) stérile de Dulbecco, y compris le sérum fœtal de veau (FCS) à 10 %, la solution saline tamponnée de phosphate stérile (PBS) et la trypsine stérile à 37 °C. Activez le banc propre.
- Préparer une plaque de 6 puits en ajoutant un glissement stérile de couvercle en verre (10 mm) à chaque puits à l’aide de forceps stériles.
- Mettez un plat de culture cellulaire de 10 cm avec des cellules Cos7 sous le banc propre. Sous le banc propre, aspirer le milieu des cellules à l’aide d’un dispositif à vide.
- Tenez le plat de culture cellulaire angulé dans une main et ajoutez 10 mL de PBS stérile sur le côté du plat de culture cellulaire pour éviter de rincer les cellules. Ez le plat de culture cellulaire vers le bas de sorte que PBS rince le plat complet de culture cellulaire.
- Retirer le PBS et ajouter 1 mL de trypsine au milieu du plat de culture cellulaire et incuber pendant 5 min à 37 °C.
- Arrêtez la réaction de trypsinisation en ajoutant 10 mL de DMEM dont 10% de FCS et pipette la solution cellulaire de haut en bas avec un pipettor pour séparer manuellement les cellules.
- Analyser le numéro de cellule par millilitre à l’aide d’une chambre de comptage.
- Ensemencer 68 000 cellules dans un milieu de 2 mL par puits sur le couvercle en verre glisse dans la plaque de 6 puits. Répartir les cellules dans la plaque de 6 puits en secouant prudemment la plaque horizontalement et verticalement.
REMARQUE : Les cellules doivent être dispersées. - Incuber les cellules pendant la nuit dans un incubateur de 37 °C avec 5 % de CO2.
2. Transfection des cellules
- Préparer deux tubes stériles de 1,5 mL (l’un pour l’ADN dilué (A) et l’autre pour le réagencé dilué pour la transfection lipidique cationique (B)). Pipette 0,75 μg de néphrine et 0,75 μg de plasmide d’expression podocine cDNA par puits dans un tube de 1,5 mL et le diluer en 100 μL de sérum moyen réduit par puits. Ajouter 3 μL de réaccfection cationique par puits au deuxième tube de 1,5 mL et diluer avec 100 μL de sérum moyen réduit. Incuber les deux réactions pendant 5 min à température ambiante.
- Combinez les deux réactions (A et B) à un complexe ADN-lipide et incubez pendant 20 min à température ambiante. Ajouter prudemment 200 μL du complexe ADN-lipides à chaque puits.
- Incuber les cellules transfectées à 37 °C avec 5 % de CO2 pendant 48 h sans changer le milieu.
3. Immunolabeling des structures cellulaires
- Préparer une solution de fixation (4% (w/v) paraformaldéhyde en PBS, 1 mL/puits), la solution de perméabilisation (0,5% (w/v) Triton X-100 en PBS, 1 mL/puits), et une solution de blocage (5% (v/v) albumine de sérum bovin en PBS, 1 mL/puits).
- Retirez le milieu à l’l’l’insage d’un dispositif sous vide. Ajouter 2 mL de PBS à chaque puits pour enlever le milieu supplémentaire. Aspirez complètement le PBS.
REMARQUE: Pour éviter de laver les cellules avec PBS, assurez-vous de pipette PBS pas sur le couvercle en verre glisser directement. - Fixer les cellules avec 4% (w/v) paraformaldéhyde (PFA) dissous dans PBS pendant 10 min à température ambiante.
REMARQUE : Alternativement, fixez des cellules dans 3% (v/v) glyoxal-éthanol18. - Jetez la PFA et lavez les cellules deux fois dans PBS (2 mL chacune) en évitant le pipetage direct sur les feuillets de couverture en verre. Perméabilizez les cellules fixes avec Triton X-100 0,5% (w/v) en PBS pendant 10 min à température ambiante.
- Retirez la solution de perméabilisation et lavez-la deux fois avec pbs comme indiqué dans l’étape 3.2.
- Bloquez les cellules en ajoutant 1 mL de 5% (v/v) BSA dans PBS pendant 1 h à température ambiante. Incuber les cellules avec 200 μL de l’anticorps primaire (anticorps anti-podocine 1:200 en 1% (v/v) BSA en PBS) pendant la nuit à 4 °C.
REMARQUE : Alternativement, incuber l’anticorps primaire pendant 1 h à température ambiante. - Retirez l’anticorps primaire et lavez-le trois fois avec du PBS comme à l’étape 3.2. Ajouter l’anticorps secondaire (chèvre anti-lapin Alexa 488 1:1000 en 1% (v/v) BSA en PBS) pendant 1 h à température ambiante. Gardez les cellules dans l’obscurité à l’aide d’une boîte.
- Jetez l’anticorps secondaire et lavez-le avec du PBS trois fois.
- Retirer le PBS et incuber les cellules avec 200 μL d’anticorps anti-néphrine 1:100 en 1% (v/v) BSA en PBS pendant 1 h à température ambiante. Laver avec PBS trois fois. Gardez les cellules dans l’obscurité à l’aide d’une boîte.
- Retirer le PBS et incuber avec l’anticorps secondaire âne anti-cobaye 633, 1:200, pendant 1 h à température ambiante. Laver avec PBS trois fois. Gardez les cellules dans l’obscurité à l’aide d’une boîte.
4. Microscopie d’expansion
- préparation
- Pour former les entretentres pour la chambre de gelation, coupez les glissements de couverture en verre #1,0 et #1,5 en rayures de 5 mm (quatre chacune par glissière de verre) à l’aide d’un couteau en diamant. Placez les bandes de glissement de couverture de #1,5 mm de sorte qu’elles forment un carré de 2,5 cm de longueur sur une glissière de verre et placez-les dans une chambre de coloration(figure 2A1-C1). Pipette une gouttelette de ddH2O dans les coins de la place pour adhérer à la couverture de verre glisser rayures les uns aux autres et à la glissade de verre (Figure 2A2-C2).
REMARQUE : Évitez le séchage complet du ddH2O car la force d’adhérence se perdra. Si nécessaire, appliquez plus de gouttelettes de ddH2O. Attendez environ 20 min pour que les feuillets de couverture de #1,5 mm soient fixés de façon stably avant de commencer par l’étape 4.1.2. - Placez quatre #1.0 bandes de glissement de couverture en verre par glissement de couverture sur les #1 de couverture de 0,5 mm. Adhérez aux rayures en pipetant une gouttelette sur chaque #1,5 mm de glissement de couverture(figure 2A3-C3).
REMARQUE : L’épaisseur du gel doit être d’au moins 0,15 mm pour faciliter la manipulation dans l’avancement du protocole. Cependant, pour éviter le gel excessif sur le dessus des cellules, maintenez la hauteur d’espacement près du substrat cellulaire. En utilisant #1.5 et #1.0 bandes de glissement de couverture comme espaceurs, la hauteur des entretaires sera d’environ 0,3 mm. Les cellules adhèrent au verre de couverture (hauteur 0,12 mm). Par conséquent, le gel sera assez épais pour manipuler, mais gel excessif sur le dessus des cellules est évité en utilisant #1,0 et #1,5 bandes de verre de couverture comme espaceurs. - Pour le couvercle de la chambre de gelation, envelopper un verre de couverture (#1,5) avec du film de paraffine. Évitez les plis ou la saleté sur le film de paraffine(figure 2A5-C5).
- Pour former les entretentres pour la chambre de gelation, coupez les glissements de couverture en verre #1,0 et #1,5 en rayures de 5 mm (quatre chacune par glissière de verre) à l’aide d’un couteau en diamant. Placez les bandes de glissement de couverture de #1,5 mm de sorte qu’elles forment un carré de 2,5 cm de longueur sur une glissière de verre et placez-les dans une chambre de coloration(figure 2A1-C1). Pipette une gouttelette de ddH2O dans les coins de la place pour adhérer à la couverture de verre glisser rayures les uns aux autres et à la glissade de verre (Figure 2A2-C2).
- Ancrage et polymérisation (gelation)
- Préparer le tampon d’ancrage (voir le tableau 1). Pour le traitement d’ancrage, remplacez PBS par 250 μL de tampon d’ancrage par puits directement sur le bordereau de couverture en verre et incubez pendant 3 h à température ambiante. Gardez le substrat dans l’obscurité à l’aide d’une boîte.
REMARQUE : Alternativement, incuber toute la nuit à température ambiante. Préparez un tampon d’ancrage frais pour chaque expérience et attendez 10-15 min jusqu’à ce qu’il soit dissous correctement. 6-((Acryloyl)amino)acide hexanoïque, ester succinimidyl (AcX) doit être remplacé tous les 4-5 mois pour assurer un ancrage adéquat. - Retirer le tampon d’ancrage et laver une fois avec 1,5 mL de PBS par puits.
- Pour tacher les fibres d’actine, décongeler une solution de phalloidine compatible ExM et incuber la phalloidine (5 μL de phalloidine diluée en 195 μL de 1 % (v/v) BSA en PBS/puits) pendant 45 min à température ambiante. Gardez les échantillons dans l’obscurité à l’aide d’une boîte.
- Entre-temps, dissoudre l’acrylate de sodium en ddH 2 Oà l’aided’un dispositif d’agitation. Sur la glace, préparer la solution monomère (voir le tableau 1).
REMARQUE : L’acrylate de sodium dissous devrait être une solution claire et incolore. Si la solution est jaune, remplacer par un nouvel acrylate de sodium. - Préparer la solution de gelling sur la glace (voir le tableau 1). Pipette ammoniumperoxidsulfate (APS) dans la solution de gelage juste avant que la solution de gelling soit appliquée à la chambre de gelation.
- Retirer la phalloidine des cellules et laver avec 1,5 mL de PBS deux fois à température ambiante. Laissez 1,5 mL de PBS dans le puits pour faciliter l’enlèvement de l’échantillon sur la feuille de couverture en verre.
- Placez les cellules sur le verre de couverture dans la chambre de hongre à l’aide de forceps et d’une canule pour soulever le glissement de verre de couverture de la plaque de 6 puits.
REMARQUE : Les cellules doivent être sur le dessus de la feuille de couverture en verre. Le slip de couvercle en verre ne doit pas toucher les entretaires. - Ajouter aps à la solution de gelling et vortex sous peu. Pipette 200 μL de solution de gelling sur l’échantillon( Figure 2A4-C4). Fermez prudemment la chambre de gelation en évitant les bulles d’air dans le gel (Figure 2A5-C5).
- Incuber la chambre gélissante pendant au moins 1 h à 37 °C pour polymériser le gel dans la chambre de coloration humide.
- Préparer le tampon d’ancrage (voir le tableau 1). Pour le traitement d’ancrage, remplacez PBS par 250 μL de tampon d’ancrage par puits directement sur le bordereau de couverture en verre et incubez pendant 3 h à température ambiante. Gardez le substrat dans l’obscurité à l’aide d’une boîte.
- Homogénéisation (digestion)
- Sortez la chambre de coloration de l’incubateur. Pour ouvrir le couvercle de la chambre de gelation, introduisez une lame de rasoir entre le couvercle et l’espaceur. Retirez le couvercle prudemment. Retirez les entretaires avec la lame de rasoir et éliminez tout gel supplémentaire en le coupant avec la lame de rasoir.
- Mettre la diapositive avec le gel et couvrir le verre dans un plat rempli de PBS. En secouant doucement, retirer le verre détaché du gel. Pour retirer facilement le gel du plat, placez la lame sous le gel pour fixer le gel à la lame.
- Avec le gel sur la lame, diviser le gel en petits morceaux (le quart du gel est divisé en deux à trois morceaux) à l’aide de la lame de rasoir. Poussez doucement un morceau de gel dans un puits d’une plaque de 6 puits avec fond de verre et pliez-le par un pinceau. Gardez le gel hydraté avec un peu de PBS à l’aide du pinceau pour éviter la déshydratation du gel.
REMARQUE : Les cellules font face vers le bas. - À l’aide d’un microscope inversé, prenez des images d’aperçu avec une faible ouverture numérique pour déterminer le facteur d’expansion après expansion.
- Préparer le tampon de digestion (voir le tableau 1). Diluer proteinase K à 4 U/mL dans le tampon de digestion pour recevoir la solution de digestion.
REMARQUE : Le tampon de digestion sans Proteinase K peut être stocké à 4 °C pendant 1 à 2 semaines. - Ajouter 500 μL de la solution de digestion à chaque puits et plonger le gel dans la solution. Laissez-le digérer toute la nuit à température ambiante et fermez le couvercle en gardant les échantillons dans l’obscurité.
REMARQUE : Alternativement, laissez-le digérer à 37 °C pendant 1 h.
- expansion
- Retirer la solution de digestion à l’aide d’une pipette et la jeter. Ajouter 1 mL de ddH2O. Incuber le gel immergé pendant 10 min à température ambiante.
- Retirer l’eau et ajouter 1 mL de ddHfrais 2O. Attendre 10 minutes et continuer à échanger de l’eau toutes les 10 minutes jusqu’à ce qu’un plateau d’expansion soit atteint.
REMARQUE : L’expansion de l’échantillon jusqu’à 4,5 fois est réalisable. Le gel devient optiquement clair.
- imagerie
- Retirer l’eau du gel et démarrer directement la microscopie. À l’aide d’un microscope inversé, utilisez principalement un objectif d’air (faible grossissement) pour trouver des cellules imageuses dans l’état de pré-expansion (étape 4.3.4).
- Passez à un objectif 40x (huile/eau) et 63x pour une meilleure résolution. Excitez avec la longueur d’onde de l’intérêt et de prendre l’image via la caméra.
- validation
- Prenez une image de vue d’ensemble de l’échantillon. Trouvez et assortir les mêmes structures dans l’échantillon qui ont été illustrées à l’étape 4.3.4. Utilisez le canal avec le meilleur rapport signal-bruit pour validation(figure 3 A-B).
REMARQUE : Ajustez les paramètres d’imagerie pour obtenir une luminosité similaire à celle de l’image acquise à l’état non élargi (étape 4.3.4). - Superposez les images avant et après l’expansion en les tournant et en les déplaçant avec ImageJ. Utilisez l’outil de mesure de distance dans ImageJ pour mesurer les distances entre les structures clairement identifiables. Mesurez au moins 10 structures différentes.
REMARQUE : Alternativement, utilisez un script Python pour mesurer les distances décrites dans14. - Calculez le facteur d’expansion en divisant les mesures post-/pré-expansion.
- Pour déterminer les distorsions, prenez des images avec une ouverture numérique plus élevée (Figure 4A-B). Superposez ces images et analysez-les avec ImageJ ou comme décritdans 15.
REMARQUE : La détermination des distorsions doit être effectuée régulièrement, mais pas nécessairement sur chaque échantillon.
- Prenez une image de vue d’ensemble de l’échantillon. Trouvez et assortir les mêmes structures dans l’échantillon qui ont été illustrées à l’étape 4.3.4. Utilisez le canal avec le meilleur rapport signal-bruit pour validation(figure 3 A-B).
Résultats
Le concept et le moment de ce protocole proExM sont représentés à la figure 1. Le jour 5, les cellules transfectées sont fixées et tachées d’anticorps fluorescents ciblant la protéine d’intérêt (Figure 1A,B). Le jour 6, le traitement par AcX conduit à la formation de groupes d’amine sur toutes les protéines (y compris les fluorophores) (Figure 1A,B)12. Lors de la polymérisation de l’hydrogel, ces groupes d’amine se lient covalently à l’hydrogel (jour 6). Après la polymérisation du gel, l’homogénéisation (digestion) est effectuée avec la proteinase K entraînant la destruction des protéines structurelles de la cellule (jour 6, figure 1A,B). Les anticorps étiquetés fluorescents restent pour la plupart conservés après digestion. En raison de la perturbation des protéines structurelles, la dialyse de l’eau de l’hydrogel entraîne l’expansion isotropique de la cellule dans l’hydrogel le jour 7 (Figure 1A,B). L’imagerie de l’échantillon est effectuée au microscope à fluorescence conventionnel( figure 1A). La validation des données pour déterminer le facteur d’expansion et exclure les distorsions doit être effectuée( figure 1A).
Pour effectuer l’expansion de la cellule isotropiquement, l’étape de gelation est essentielle. La figure 2 montre la vue latérale et supérieure d’une chambre de gelation. Les feuillets de couverture en verre construisent les entretentres de la chambre de gelation(figure 2A1-3/C1-3). Le verre de couverture avec les cellules fixes et souillées est placé avec les cellules vers le haut sur une glissière en verre(figure 2A4-C4). Le couvercle de la chambre de gelation est enveloppé de parafilm et est fermé sans bulles (Figure 2A5-C5).
Ce protocole ExM permet d’être multiplié par quatre. Pour déterminer le facteur d’expansion, il est essentiel d’imager les cellules avant et après l’expansion( Figure 3A + B). Un ancrage et une homogénéisation insuffisants peuvent entraîner des distorsions et des ruptures de cellules. La figure 4A + B montre des exemples représentatifs de cellules rompues dans différentes images de grossissement.
Cette méthode peut être utilisée pour étudier la co-localisation des protéines d’adaptateur f-actine et d’actine, par exemple la podocine et la néphrine (figure 5). Podocin est représenté en vert tandis que l’actine est étiquetée en bleu (Figure 5). La néphrine est marquée en vert. Les zones blanches indiquent une co-localisation.
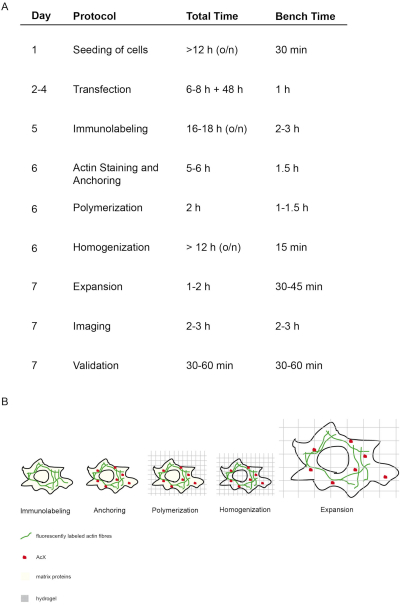
Figure 1: Concept et calendrier de ce protocole ExM. (A) Dans la colonne « protocole », chaque étape du protocole est décrite. (A + B) Après l’ensemencement et la transfecting des cellules, l’étiquetage immunofluorescent est effectué (Immunolabeling). (A + B) La petite molécule AcX (point rouge) se lie à toutes les protéines et les ancre à l’hydrogel (Ancrage). (A + B) Par polymérisation, toutes les protéines, y compris les fluorophores, sont liées de façon covalente via AcX à l’hydrogel (polymérisation). (A + B) L’homogénéisation conduit à la digestion des protéines matricielles structurelles. (A + B) L’expansion est obtenue par dialyse dans l’eau. (A) L’imagerie et la validation de l’imagerie finalisent l’expérience. (A) L’ensemble du protocole nécessite 7 jours (colonne « jour ») avec de nombreuses étapes d’incubation (temps total par colonne de jour « temps total »), mais le temps réel banc est beaucoup moins comme indiqué dans la colonne respective « temps de banc ». Modifié à partirde 14. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
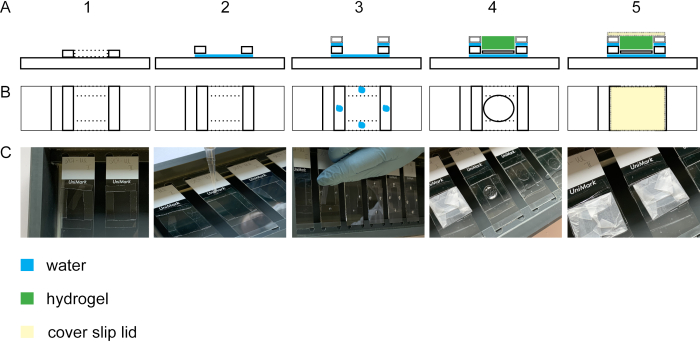
Figure 2 :Construction de la chambre de gelation. Vue latérale (A1) et vue supérieure (B1 + C1) d’une glissade en verre avec quatre #1,5 bandes de couverture. En ajoutant une gouttelette d’eau entre la glissière de verre et les rayures de glissement de couverture, les rayures adhéreront à la glissière enverre (vue latérale A2, vue supérieure B2, C2). Gouttelettes d’eau sur les bandes de couverture #1,5 conduire à l’adhésion de #1,0 bandes de couverture posées sur le dessus des bandes de couverture #1,5(vue latérale A3, vue du haut B3, C3). L’échantillon sur le bordereau de couverture est placé au milieu du rectangle à l’aide de forceps. Le gel est pipetted sur le dessus(vue latérale A4, vue supérieure B4, C4). (A5) Vue latérale et vue supérieure (B5 et C5) de la chambre de gelation assemblée comprenant le couvercle fermé qui est construit à partir d’un glissement de couverture enveloppé dans le parafilm. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
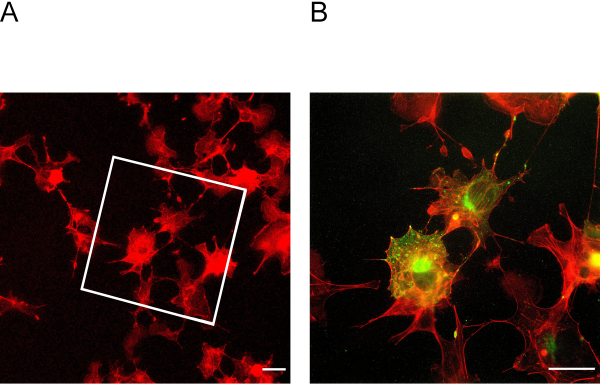
Figure 3 :Cellules avant et après l’expansion. ( A )Cellulesavant expansion souillées pour l’actine. La boîte indique dans quelle zone se trouvent les cellules élargies de la figure 3B. (B) Cellules après expansion souillées pour l’actine en rouge et podocine en vert. Podocin co-localise avec l’actine dans la périphérie cellulaire. Barre d’échelle = 5 μm, facteur d’expansion = 2. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 4: Distorsions et ruptures des cellules. ( A+ B) Images microscopiques représentatives des cellules cos7 immuno-tachées d’actine (rouge). Les cellules étaient fixes, tachées, ancrées, digérées et élargies. (A) Ruptures de cellules. Les flèches indiquaient des zones rompues. Barre d’échelle = 5 μm, facteur d’expansion = 4. (B) Ruptures et distorsion des cellules. Les flèches blanches indiquaient des zones rompues. Barre d’échelle = 5 μm, facteur d’expansion = 4. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
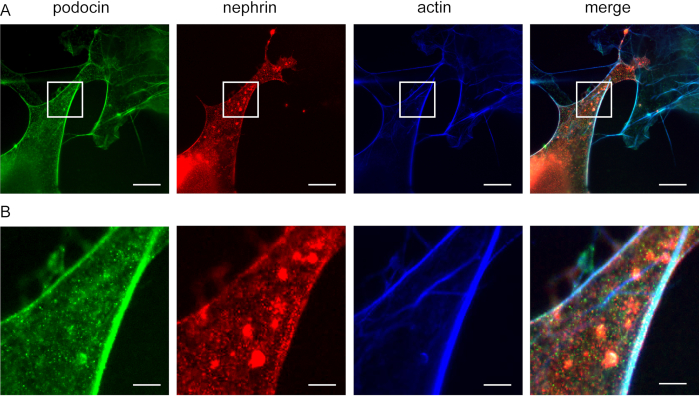
Figure 5: Podocin co-localise avec la néphrine et l’actine. Cellules cos7 immunofluorescentes étiquetées pour la podocine, l’actine et la néphrine. (A) Cellules Cos7 tachées de podocine (vert), d’actine (bleu) et de néphrine (rouge) avec ExM. Podocin co-localise avec l’actine et la néphrine. Barre d’échelle = 200 nm, facteur d’expansion = 4. (B) Grossissement de la zone indiquée dans (A), Barre d’échelle = 40 nm. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
| Tampon d’ancrage | concentration finale | ||
| NaHCO3 NaHCO3 | 150 mM | ||
| Acryloyl-X, SE (AcX) | 0,1 mg/ml | ||
| Solution monomère | Concentration de solution de stock g/100 ml | quantité (ml) | concentration finale (g/100 ml) |
| acrylate de sodium | 38 | 2.25 | 8.6 |
| acrylamide | 50 | 0.5 | 2.5 |
| N,N'-Méthylènebisacrylamide | 2 | 0.75 | 0.15 |
| chlorure de sodium | 29.2 | 4 | 11.7 |
| Pbs | 10x (10x) | 1 | 1x (1x) |
| Eau | 0.75 | ||
| total | 9.4 | ||
| Solution gelling | Concentration de solution de stock | montant (μl) | concentration finale (mg/ml) |
| solution monomère | Na | 190 | Na |
| Aps | 10% | 4 | 0.1 |
| TEMED (TEMED) | >99% | 4 | 0.1 |
| Eau | Na | 2 | Na |
| total | 200 | ||
| Solution de digestion | concentration finale | ||
| Tris Cl, pH 8,0 | 1 M | ||
| PH EDTA 8.0 | 0,5 M | ||
| Triton X-100 | 0.005% | ||
| Guanidin HCL | 8 M | ||
| Eau | |||
| proteinase K | 4 U/ml |
Tableau 1 : Solutions pour ExM.
Discussion
La méthode présentée permet à l’investigateur de visualiser les protéines cellulaires, p. ex., la podocine, la néphrine et les composants cytosquelettiques, p. ex., la F-actine. Dans ce protocole, les cellules cos7 transfectées sont utilisées comme modèle pour étudier l’interaction des protéines de diaphragme fendue avec la F-actine. Malheureusement, les lignées cellulaires immortalisées des podocytes n’expriment pas suffisamment de quantités endogènes de protéines diaphragmes fendues19.
Avec cette méthode, les protéines cellulaires peuvent être visualisées avec une résolution nanométrique à l’aide d’un microscope à fluorescence conventionnel. Les étapes les plus critiques du protocole sont les suivantes : 1) ancrage suffisant des groupes d’amine protéiques à l’hydrogel avec AcX, 2) polymérisation adéquate de l’hydrogel, 3) moment optimal pour la digestion et 4) sélection de fluorophores compatibles.
L’ancrage des protéines cellulaires à l’hydrogel est essentiel pour cette méthode afin de préserver la position de la protéine dans l’hydrogel pendant l’expansion. AcX est une petite molécule qui se lie à des groupes d’amine de protéines dans les cellules et les tissus. AcX crée une double bande carbone-carbone avec des protéines, permettant l’incorporation des protéines dans l’hydrogel dans l’étape de polymérisation20. AcX intègre également des anticorps afin que l’étiquetage avec des anticorps d’immunofluorescence puisse être effectué avant le traitement d’AcX. Un ancrage insuffisant peut entraîner des ruptures et des distorsions des cellules. En raison de la modification des groupes d’amine par fixatifs, il faut optimiser le fixatif ou le temps de fixation. De plus, un stockage insuffisant ou des conditions d’ancrage non optimisées peuvent entraîner des ruptures et des distorsions. Sur la base de notre expérience, AcX perd son effet optimal lorsqu’il est utilisé pendant plus de 3-4 mois.
La polymérisation du gel dépend de la température. Nous recommandons donc de conserver les solutions de polymérisation sur la glace avant de les achemmer dans la chambre de gelation. En outre, le temps de manipulation de l’étape de gelation doit être maintenu court (moins de 5 min) afin d’éviter la formation prématurée de gel. Le mélange complet de la solution de polymérisation empêche la polymérisation inégale. Les bulles d’air affecteront le processus d’expansion lors du toucher de l’échantillon et peuvent être évitées en ajoutant plus de solution de polymérisation.
Après l’incorporation des protéines cellulaires dans l’hydrogel, l’étape d’homogénéisation mécanique (ou digestion) est nécessaire pour assurer l’expansion. Différentes méthodes, par exemple, la chaleur et le détergent ou la digestion enzymatique, existent et doivent être personnalisées à l’échantillonétudié 12,14,20. Dans ce protocole, la protéase Proteinase K est utilisée pour la digestion enzymatique. Proteinase K est appliqué à un dosage suffisant pour détruire les protéines structurelles tout en préservant la plupart des autres protéines, y compris les anticorps fluorescents12. Si la digestion est incomplète, l’expansion de l’échantillon est insuffisante. De plus, l’échantillon peut se déchirer pendant le processus d’expansion( figure 3). Si une expansion inadéquate de l’échantillon s’est produite, le remplacement de l’eau est recommandé. Alternativement, le temps pour la digestion enzymatique peut être ajusté ou un nouvel aliquot de la Proteinase K ouvert.
Si l’échantillon est sur-digéré, les signaux de fluorescence seront diminués. Dans ce cas, le temps de digestion doit être réduit. Dans ExM en général, l’intensité du signal de fluorescence par unité de volume est réduite en raison de l’expansion volumétrique del’échantillon 14. Par conséquent, des temps d’exposition plus longs pendant l’imagerie doivent être pris en considération.
Il est essentiel de sélectionner des fluorophores compatibles ExM. Les dyes de cyanine sont dégradés pendant l’étape de polymérisation13. Les protéines de fluorescence à base de bactériophytochromes sont également en grande partie détruites13. Cependant, la plupart des protéines de la GFP seront conservées13. En outre, la streptavidine peut également être appliquée avant expansion, étiquetant les modifications post-translationnelles telles que la s-nitrolysation via une petite moléculetag 13.
La phalloidine, une petite molécule d’étiquetage pour cibler le cytosquelette d’actine, n’est pas compatible avec ExM21. Pour surmonter l’ancrage insuffisant de la phalloidine, l’ancrage trivalent (TRITON) a étéintroduit 21. Cette approche offre le ciblage simultané, l’étiquetage et la greffe de biomolécules21.
Cette méthode peut être modifiée pour tacher les molécules d’ARN (ExFish)22. Dans la microscopie d’expansion itérative (iExM) ou la microscopie X10, la résolution de 60-70 nm peut être étendue à environ 25 nm en appliquant un deuxième gel habitable dans le premier hydrogel élargi ou en effectuant une seule étape d’expansion utilisant un hydrogeldifférent 15,16. La microscopie d’expansion ultrastructure (U-ExM) permet une super résolution des protéines préservant leur attribution à un élément ultrastructural (par exemple, mitochrondria, microtubules)23. Une combinaison d’ExFish (ARN et ADN) et de méthodes proExM ont déjà été réaliséesainsi que 22,24. Le protocole présenté utilise des cellules cos7 transfectées comme modèle pour étudier les protéines du diaphragme fendue. Nous nous attendons à ce que d’autres cellules rénales de culture résidente, par exemple les cellules HEK293T, puissent être utilisées de la même façon pour ce protocole. Selon la lignée cellulaire, des ajustements peuvent être nécessaires pour les différentes conditions de culture et de transfection.
ExM améliore la résolution des échantillons immuno-souillés d’environ 4 fois atteignant une résolution spatiale latérale de 70 nm13. Comparé à d’autres techniques de super-résolution, ExM est exécuté sur un microscope conventionnel de fluorescence13,14. Par conséquent, aucun équipement coûteux ou personnel spécialement formé n’est nécessaire pour effectuer la méthode ExM14. Même si tous les fluorophores ne sont pas compatibles avec ExM, il existe généralement de nombreux anticorps disponibles avec des fluorophores optimisés avec des propriétés photophysiques nécessaires pour la microscopie à super résolution14. Le principal inconvénient de cette méthode est qu’ExM est incompatible avec les échantillons vivants12,14.
À l’avenir, l’amélioration de la composition chimique de l’hydrogel pourrait conduire à une résolution spatiale encore plusélevée 12. La combinaison de différents protocoles peut également permettre la visualisation des protéines, de l’ARN, de l’ADN ou des lipides dans les complexes d’un même échantillon avec une tellehaute résolution 12.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs remercient Blanka Duvnjak et Nikola Kuhr pour leur excellente assistance technique.
matériels
| Name | Company | Catalog Number | Comments |
| Acrylamide >99% | Sigma-Aldrich | A3553-100G | |
| 6-((Acryloyl)amino)hexanoic acid, succinimidyl ester, Acryloyl-X, SE | invitrogen | A-20770 | store up to 4 months |
| APS | Sigma-Aldrich | A3678-25G | |
| Deckgläser (cover glasses) | Engelbrecht | K12432 | 24x32mm #1.0 |
| Diamont cutter | VWR | 201-0392 | for cutting the cover slips |
| Guanidine HCl | Sigma-Aldrich | G3272-100G | 8M Stock can be kept at RT |
| Marten hair paintbrush | Leon Hardy | 3 (770) | |
| "Menzel" Deckgläser (cover glasses) | Thermo Fischer | 15654786 | 24x24mm #1.5 |
| N,N`-Methylenbisacrylamide | Sigma-Aldrich | M7256-25G | |
| Objektträger UniMark | Marienfeld | 703010 | |
| Proteinase K | New England Biolabs | P8107S | |
| Sodium Acrylate | Sigma-Aldrich | 408220 | check purity |
| Sodium Bicarbonate | Sigma-Aldrich | S5761 | |
| Staining chamber | produced at the university's workshop | ||
| TEMED | ROTH | 2367.1 | |
| 6-Well glass bottom plates | Cellvis | P06-1.5H-N | |
| Antibodies | |||
| Actin-ExM 546 | chrometra | non-available | 1:40 |
| Anti Podocin produced in rabbit | Sigma | P-0372-200UL | 1:200 |
| Donkey anti guinea-pig CF633 | Sigma | SAB4600129-50UL | 1:200 |
| Goat anti rabbit 488 | Life Technologies | A11034 | 1:1000 |
| Guinea pig anti nephrin | Origene | BP5030 | 1:100 |
| Software | |||
| FIJI | |||
| Visiview | |||
| microscope | |||
| AXIO Observer Z1 | Zeiss | non-available |
Références
- Matsushita, K., et al. Association of estimated glomerular filtration rate and albuminuria with all-cause and cardiovascular mortality in general population cohorts: a collaborative meta-analysis. Lancet. 375 (9731), 2073-2081 (2010).
- Faul, C., Asanuma, K., Yanagida-Asanuma, E., Kim, K., Mundel, P. Actin up: regulation of podocyte structure and function by components of the actin cytoskeleton. Trends in Cell Biology. 17 (9), 428-437 (2007).
- Saleem, M. A., et al. Co-localization of nephrin, podocin, and the actin cytoskeleton - Evidence for a role in podocyte foot process formation. American Journal of Pathology. 161 (4), 1459-1466 (2002).
- Kestila, M., et al. Positionally cloned gene for a novel glomerular protein - nephrin - is mutated in congenital nephrotic syndrome. Molecular Cell. 1 (4), 575-582 (1998).
- Huber, T. B., et al. Podocin-mediated recruitment of nephrin into lipid rafts is required for efficient nephrin signaling. Journal of the American Society of Nephrology. 14, 8(2003).
- Boute, N., et al. NPHS2, encoding the glomerular protein podocin, is mutated in autosomal recessive steroid-resistant nephrotic syndrome. Nature Genetics. 24 (4), 349-354 (2000).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. Journal of Cell Science. 124 (10), 1607-1611 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440 (7086), 935-939 (2006).
- van de Linde, S., et al. Direct stochastic optical reconstruction microscopy with standard fluorescent probes. Nature Protocols. 6 (7), 991-1009 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Testa, I., et al. Multicolor fluorescence nanoscopy in fixed and living cells by exciting conventional fluorophores with a single wavelength. Biophysical Journal. 99 (8), 2686-2694 (2010).
- Wassie, A. T., Zhao, Y., Boyden, E. S. Expansion microscopy: principles and uses in biological research. Nature Methods. 16 (1), 33-41 (2019).
- Tillberg, P. W., et al. Protein-retention expansion microscopy of cells and tissues labeled using standard fluorescent proteins and antibodies. Nature Biotechnology. 34 (9), 987-992 (2016).
- Truckenbrodt, S., Sommer, C., Rizzoli, S. O., Danzl, J. G. A practical guide to optimization in X10 expansion microscopy. Nature Protocols. 14 (3), 832-863 (2019).
- Truckenbrodt, S., et al. X10 expansion microscopy enables 25-nm resolution on conventional microscopes. Embo Reports. 19 (9), (2018).
- Chang, J. B., et al. Iterative expansion microscopy. Nature Methods. 14 (6), 593-599 (2017).
- Chozinski, T. J., et al. nanoscale optical imaging of mouse and human kidney via expansion microscopy. Scientific Reports. 8 (1), 10396(2018).
- Richter, K. N., et al. Glyoxal as an alternative fixative to formaldehyde in immunostaining and super-resolution microscopy. The EMBO Journal. 37 (1), 139-159 (2018).
- Rinschen, M. M., et al. Quantitative deep mapping of the cultured podocyte proteome uncovers shifts in proteostatic mechanisms during differentiation. American Journal of Physiology-Cell Physiology. 311 (3), 404-417 (2016).
- Asano, S. M., et al. Expansion microscopy: protocols for imaging proteins and RNA in cells and tissues. Current Protocols in Cell Biology. 80 (1), 56(2018).
- Wen, G., et al. Evaluation of direct grafting strategies via trivalent anchoring for enabling lipid membrane and cytoskeleton staining in expansion microscopy. ACS Nano. 14 (7), 7860-7867 (2020).
- Chen, F., et al. Nanoscale imaging of RNA with expansion microscopy. Nature Methods. 13 (8), 679-684 (2016).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nature Methods. 16 (1), 71-74 (2019).
- Zhao, Y., et al. Nanoscale imaging of clinical specimens using pathology-optimized expansion microscopy. Nature Biotechnology. 35 (8), 757-764 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon