Method Article
낮은 산소 환경에서 인간 대 식 세포 분극의 Proteomic 분석
요약
우리는 인간의 세포의 proteomic 서명을 macrophage 분극에 낮은 산소 환경 영향의 결정에이 적용 하는 프로토콜을 제시.
초록
대 식 세포는 타고 난 면역 세포 조직의 항상성 전염 성 병원 균에 대 한 응답에서 배열 하는 생리 기능 수에 관련 된. 이러한 셀의 다양 한 기능 활성화 상태로 또한 양극 화 라는 관련이 있습니다. 이러한 다양 한 분극의 정확한 분자 설명 대 식 세포 생물학의 분야에서 우선 순위입니다. 그것은 현재 다차원 접근은 분극 환경 신호에 의해 제어 되는 방법을 설명 하는 데 필요한 인정 했다. 이 보고서에서 우리는 인간의 세포에 다양 한 분극의 proteomic 서명을 얻기 위해 설계 된 프로토콜을 설명 합니다. 이 프로토콜은 대 식 세포 단백질 표정에-젤 분류 한 내용과 리스 C/트립 신-소화 세포 세포의 용 해에서 얻은의 레이블 없는 정량화를 기반으로 합니다. 우리는 또한 프로토콜 기반 솔루션에서 소화 하 고 전자 안으로 사용 하는 분류를 초점을 제공 합니다. 산소 농도 조직에 관련 된 환경 매개 변수, 때문에 우리는 어떻게 대기 구성 탐험이 프로토콜을 사용 또는 저 산소 환경에 영향을 미치는 macrophage 분극의 분류.
서문
대 식 세포는 여러 전염 성 병원 균에 대 한 응답에서 조직의 항상성, apoptotic 세포의 제거를 포함 하 여, 세포 외 기질1의 리 모델링에 이르기까지 생리 기능에 타고 난 면역 세포. 이 세포는 강한 phenotypic가 소성2 분극 라고도 하는 많은 가능한 활성화 상태를 변환 하는 특징. 이러한 다양 한 분극의 정확한 분자 설명 대 식 세포 생물학3의 분야에서 우선 순위입니다. 그것은 소위 M1/M2 이분법, 있는 m 1 프로-염증 성 나타내고 M2 염증 세포를 사용 하 여 이러한 분극 분류 제안 되었습니다. 이 모델은 급성 감염, 알레르기, 비만4같은 다양 한 병 적인 상황에 잘 맞는. 그러나, 만성 염증된 조직 및 암, 그것은 증명 되었습니다이 분류는 대 식 세포 특정 세포 환경5,6, 에서 제공 하는 광범위 한 phenotypic 레 퍼 토리를 파악 수 없습니다. 7. 현재 일치는 대 식 세포 양극 화 더 나은 설명 다차원 모델을 사용 하 여 특정 microenvironmental 신호8을 통합 하는. 이 결론은 M1/M2 모델은 획득된 분극9에 비효율적 보여주는 인간 대 식 세포의 transcriptomic 분석을 통해 확인 됐다.
연구를 인간의 세포에 다양 한 분극의 proteomic 서명 프로토콜을 제공 하는 것을 목표로 제시. 우리는 다양 한 산소 레벨의 환경에 인간의 세포를 분화 하 고 레이블 없는 정량화를 수행 하기 위해 전체 대 식 세포 프로테옴에서 펩 티 드를 얻을 하는 방법을 설명 합니다. 이 정량화 다양 한 단백질의 표현 레벨의 비교를 허용 한다. 줄기 세포에 대 한 연구 환경 키 매개 변수10으로 산소의 중요성을 계시 했다, 우리는이 조직 매개 변수 인 간에 있는 대 식 세포 분극 영향을 미칠 수 어떻게 이해를 추구 합니다. 산소의 부분 압력에서에서 발견 되었습니다 범위 3 (총 대기 압력)의 20% ~ 20% (정확한 값은 약 18.6% 물의 존재 하면서 세포 문화 인큐베이터에서 일반적으로 사용 되는 약 해당 인체 에 계정).
이전 작품과 형태학의 조회11 지점과 이러한 차이 아마 부분적으로는 그들은 노출된12다른 산소 수준 때문에 치경 다 기능에서 중간 세포를 보이고 있다. 또한, 골 수 유래 세포 박테리아12낮은 산소 환경 노출 되 면 phagocytize를 증가 능력을 보여줍니다. 인간의 세포 THP1 차별화 된13, 반대의 효과가 발견 되었습니다 있지만 이러한 결과 산소 대 식 세포 생물학의 레 귤 레이 터는 고 인간의 세포에 분자 수준에서이 역할을 명확히 하는 데 필요한 아이디어를 지 원하는. 이전 연구에서 우리는 이러한 문제를 해결 하는 proteomics 접근 방식을 적용 했습니다. 단백질의 수천에 대 한 식 레벨을 동시에 측정 하 여 우리는 분극에 산소의 영향을 강조 하 고 새로운 분자 마커의 목록을 제공. 우리는 또한 일부 세포 기능에 이러한 연구 결과 관련 된 수 있습니다. 특히, 우리는 apoptotic 세포의 식 균 작용의 속도 증가 되었다 IL4/IL13-편광 세포에서 밝혀 proteomic 분석14ALOX15의 upregulation에 연결 했다 발견. 현재 연구에서 우리는 이러한 분석을 수행 하는 방법을 설명 합니다.
프로토콜
건강 하 고, 취소 확인 된 기증자 로부터 인간의 혈액 샘플 (LRSC) 인증된 프로토콜 (CODECOH DC-2018-3114)의 일환으로 EFS (프랑스 국가 혈액 서비스)에서 얻은 했다. 기증자는 혈액의 사용에 대 한 서명된 동의 했다.
1. 미디어와 버퍼 준비
- 대 식 세포 매체 [RPMI glutamax + 10 mM HEPES + 1 x 비 본질적인 아미노산 (NEAA)]를 준비 하 고 37 ° c.에 따뜻한
- 대 식 세포 매체 + AB 플라즈마 (SAB)에서 10% 인간의 혈 청을 준비, 그것은 (0.22 μ m 필터) 필터 다음 37 ° C에 따뜻한 (macrophage 매체 + 10%로이 SAB).
- 정렬 버퍼 버퍼링 하는 인산 염 (PBS) + 0.5% 소 혈 청 알 부 민 (BSA) + 2 mM ethylenediaminetetraacetic 산 (EDTA) x [1]를 준비 하 고, 그것은 (0.22 μ m 필터), 필터링 하 고 4 ° c.에 그것을 유지합니다
2. 말 초 혈액 단 세포 (PBMCs) Leukoreduction 시스템 챔버 (LRSC)에서 격리
- 밀도 그라데이션 세포 분리 솔루션의 15 mL를 넣어 ( 재료의 표참조) 50 mL에 원심 튜브는 LRSC를 수신 하기 전에 실내 온도 (RT)에 따뜻한 수 있습니다 그래서.
참고: 밀도 온도에 따라 다릅니다. 이 단계 실시간에 equilibrate 수 있도록 사전에 수행 해야 합니다이 제품을 4 ° C에서 저장으로 - 50 mL 원심 분리 관으로는 LRSC를 빈 1 x PBS의 최대 50 mL를 추가 하 고 혼합. 아주 천천히, 혼합 밀도 그라데이션 솔루션 2.1 단계 워밍업의 15 mL 위에 2.2 단계 준비의 25 mL를 추가 합니다.
참고: 수 단계가 단계를 혼합 하지 않도록 주의 하십시오. 혈액이 단계의 어떤 소요도 없이 밀도 그라데이션 솔루션에 추가 되어야 합니다. - 휴식 없이 700 x g에 25 분 동안 원심 분리 튜브 두 원심
참고: 조밀도 기온 변화도 원심 분리의 끝에, 맨 아래쪽에서 레이어는: 적혈구와 granulocytes 펠 릿, 밀도 그라데이션 솔루션 위상, PBMCs, 그리고 플라즈마의 레이어를 형성. - 피펫으로와 함께 그것을 발음 하지 않고 플라즈마 단계를 통과 하 고 새로운 50 mL 원심 분리 관으로 PBMC 레이어를 수집 합니다. 세척 단계 및 300 x g에서 10 분 원심 분리기로 서 PBMCs에 1 x PBS의 최대 50 mL를 추가 합니다.
- 발음은 상쾌한 고 대 식 세포 매체의 40 mL에 펠 릿을 resuspend.
3. 자기 라벨의 및 절연 CD14+ 세포 (Monocytes)
- Malassez 챔버에 PBMCs를 계산 합니다. PBMCs (일반적으로 100 ~ 300 x 106 셀) 실험 수행에 필요한 금액을 철회, 원심 관, 및 300 x g에서 10 분 원심 분리기에 넣어.
- 발음은 상쾌한 고 펠 릿 10 당 1.3 단계 준비 정렬 버퍼의 80 µ L에서 resuspend7 PBMCs. 추가 20 µ L 10 당 CD14 microbeads의7 PBMCs 믹스 잘 하 고 지속적인 동요에서 4 ° C에서 15 분 동안 품 어.
- 세척 단계 및 300 x g.에서 10 분 원심 분리기는 상쾌한 발음 하 고 펠 릿 당 108 PBMCs 버퍼를 정렬의 500 µ L에서 resuspend 107 PBMCs 당 버퍼를 정렬의 1 mL를 추가 합니다.
- 자기 필드 구분 기호에서 열을 배치 합니다. 버퍼를 정렬의 3 mL와 함께 그것을 rinsing 하 여 열을 준비 합니다.
- 열에 세포 현 탁 액을 적용 합니다. 열 격리 되어야 하는 셀의 수에 따라 달라 집니다 (여기, 최대 109 PBMCs LS 열 사용 됩니다). 포함 레이블된 셀 흐름을 통해 수집 합니다.
참고:이 단계에서 시작, 모든 튜브 (부정과 긍정적인 선택) cytometry에 의해 나중의 다른 단계 검사에 대 한 보관 됩니다. - 열 정렬 버퍼의 3 x 3 mL을 씻어. 레이블이 없는 세포 단계의 3.12 같은 튜브를 통해 전달 수집 합니다. 추가 정렬 버퍼에 의해 세척 단계를 수행, 열 건조 하지 않도록 주의 하십시오. 열 컬렉션 튜브를 놓고 구분 기호에서 그것을 제거 합니다.
- 버퍼 열에 정렬의 5 mL 플라스틱 즉시 단단히 열에 플런저를 추진 하 여 자석으로 레이블이 지정 된 셀에 밖으로 플러시. CD14의 순도 높이기 위해+ 두 번째 열에 셀, eluted 분수 농축입니다.
- 새로운 열 3.4 3.7 단계 반복 합니다.
4입니다. 도금 Monocytes의
- Malassez 챔버에 monocytes를 계산 합니다. CD14의 순도 확인+ cytometry에 의해 세포. Monocytes는 실험에 필요한 금액을 철회 하 고 원심 분리 관에 넣어.
- 10 분에 300 x g. Aspirate는 상쾌한 centrifuge 고 대 식 세포 매체에 monocyte 펠 릿 resuspend. 셀 접시와 SAB + 25 ng/mL 대 식 세포 콜로 니 자극 인자 (M-CSF) 차별화를 유도 하는 것 1 h. 50 분 매체를 발음 하 고 대 식 세포 매체와 + 10%에 대 한 합의 하자.
5입니다. 하루 6 대 식 세포의 양극 화
- 매체 발음 대 식 세포 매체 + 10% 대체 SAB 다양 한 자극으로. 예를 들어 10 ng/mL 인터페론 감마 (INFγ) + 1 ng/mL lipopolysaccharide M1 양극 화를 (LPS) 또는 20 ng/mL 인터 루 킨 4 (IL4) + 20 ng/mL 인터 루 킨 (IL13) 13 m 2 양극 화에 대 한 추가 합니다.
참고: 자극 사이 24 및 48 h 다른 테스트를 진행 하기 전에 수행할 수 있습니다. - 파견 솔루션 또는 셀 스 크레이 퍼를 사용 하 여 셀을 수확.
6. 낮은 산소 조건 세포 배양
- 4 단계에서 starting, monocytes 그리고 대 식 세포 hypoxic 조건 분석을 수행 하는 산소 제어 환경에서 유지 관리 합니다. Hypoxia 작업 역을 사용 하 여 실험 기간 동안 원하는 산소 부분 압력 하에서 세포를 유지 하기 위하여.
참고: 낮은 산소 압력 하에서 작업 하는 경우 그것은 중요 한 모든 미디어와는 역 아래 세척 버퍼를 준비 하 고 액체에서 올바른 부분 압력을 얻기 위해 충분히 기다릴. 예를 들어 60 mm 페 트리 접시에에서 10 mL PBS의 대략 2 h O2 부분 압력 시작 대기 압력에서 (우리 광섬유 산소 센서를 사용 하 여 측정)에 대 한 25 mmHg에 도달 필요 합니다. 많은 hypoxic 역 또는 incubators, 산소 압력 대기압의 백분율로 설정 됩니다. 정확한 측량이 필요한 경우 직접 mmHg에 산소 압력을 설정 하는 권한을 부여 하는 역을 사용 하 여이 낫다.
7. 세포와에서 젤 소화 (프로토콜 1)
참고: 에이 다음 섹션, 펩 티 드를 가져오고 LC-MS/MS 분석을 수행 하는 데 사용 하는 두 개의 프로토콜을 설명 합니다. 세포 세포의 용 해 및 젤에 분류 및 소화, 프로토콜 1 설명 하 고 솔루션에서 소화 및 분류는 전자 초점 메서드를 사용 하 여 솔루션에 셀 세포 프로토콜 2에 설명 합니다.
- 세포 세포의 용 해 Laemmli 버퍼 [234 mM Tris-HCL (pH 6.8), 7.5 %SDS, 37% 글리세롤, 33.3% (v/v) β-mercaptoethanol, bromophenol 파랑 0.2 %w / v]를 수행 합니다. 4-12% 두번째-트리 스 아크릴 아 미드 젤에 각 샘플에 대 한 300000 셀에 해당 하는 단백질을 로드 합니다.
- 각 단백질 견본 그림3에서 exemplified 6 젤 대역으로 분할 될 수 있도록 전기 이동 이동의 기간을 제어 합니다.
- (30% 에탄올 + 7.5% 초 산 20 분), 고정 솔루션 젤 수정 다음 얼룩 솔루션 (R-250 Coomassie 파란 45 분)을 추가 합니다. (30% 에탄올 + 7.5% 아세트산 밴드 표시 될 때까지) destaining 솔루션 추가 단백질 밴드 깨끗 한 메스로 절 개 하기 전에.
- 주사위 각 500 µ L 튜브에 소개 전에 밴드 excised. 깨끗 한 유리 표면 keratins (이온된 수에 5 %SDS 솔루션 표면 청소를 사용할 수 있습니다)와 오염을 피하기 위하여 보증 된다.
- 워시 젤 조각 한 워시 25 m m 염화 중 탄산염에 이기 (50 %v / v) 뒤 37 ° C에서 20 분 동안 25 m m 염화 중 탄산염의 200 µ L에서 3 번. 10 분 100% 이기의 200 µ L 젤 조각을 탈수.
- 각 젤 조각 10 m 55 m m iodoacetamide 25 m m 염화 중 탄산염 (200 µ L)에 실시간에 어둠 속에서 35 분 뒤 56 ° C (200 µ L)에서 45 분 동안 25 m m 염화 중 탄산염에 DTT (dithiothreitol)를 품 어
- 알을 막으려고 품 어 실시간 세척에서 10 분 동안 25 m m 염화 중 탄산염에서 10 mM DTT의 200 µ L와 각 젤 조각 25 m m 염화 중 탄산염의 200 µ L에서 젤 조각 다음 10 분 동안 100% 이기의 200 µ L로 탈수.
- 제조업체의 지침에 따라 트립 신/리스-C 믹스와 함께 37 ° C에서 하룻밤 단백질 소화.
- 추가 하 여 15 분, 다음 15 분 동안 5% 포 름 산의 50 μ에 대 한 50% 이기의 50 μ 그리고 마지막으로, 50 μ 15 분 수영장에 대 한 100% 이기의 젤 조각에서 결과 펩 티 드를 추출 하 고 펩 티 드의 흡착을 제한 하는 낮은 흡수 관에 각 분수를 건조 하 고 샘플 손실입니다. 추가 분석까지-80 ° C에서 샘플을 저장 합니다.
8. 단백질 추출 및 솔루션에 소화 (프로토콜 2)
- 다음 세포의 용 해 버퍼의 150 µ L로 세포 세포의 용 해 (2 x 106 셀)를 수행:
- 7 M 요소, 2 M thiourea, 40mm 트리 스, 그리고 4% 챕 스, 프로 테아 제 억제제 (완전 한 미니, EDTA 무료 프로 테아 제 억제 물 칵테일)와 보충.
- thermoshaker와 RT에 30 분 동안 해결책 균질 20 분 RT 13,800 x g에서 centrifuge 고는 상쾌한을 유지.
- 2D 정리 키트와 오염 물질을 제거:
- 키트에는 precipitant 솔루션, 공동 비례적 솔루션, 워시 버퍼, 그리고 세척 첨가제 포함 되어 있습니다.
- Precipitant 솔루션의 300 µ L을 추가 하 고 잘 혼합. 15 분 추가 300 µ L 공동 비례적 솔루션의 ice에 품 어. 12000 x g 5 분에서 (적어도) 튜브를 원심. 작은 펠 릿은 표시 되어야 합니다. 빠르게 물의 resuspension 또는 펠 릿의 분산을 피하기 위해 다음 단계를 진행 합니다. 펠 릿을 방해 하지 않고는 상쾌한을 제거 합니다.
- 다시 뚜껑 경첩 및 펠 렛 튜브의 바닥에 남아 있는 액체를가지고 밖으로 직면 튜브 원심 짧은 펄스는 충분 합니다. 아니 보이는 액체 튜브에 남아 있어야 합니다.
- 펠 릿을 방해 하지 않고 공동 비례적 솔루션의 40 µ L를 추가 합니다. 튜브 5 분 5 분 동안 원심 분리기에 대 한 얼음에 앉아 다음 제거 하 고 세척을 삭제 하자. 드 이온의 25 µ L를 추가 합니다. 각 동 5-10에 대 한 관. 펠 릿 분산 해야 하지만 물에 분해 되지.
- (사전-20 ° C에서 1 시간 이상 냉장) 워시 버퍼의 1 mL 및 세척 첨가물의 5 µ L를 추가 합니다. 와 동 펠 릿 완전히 분산 될 때까지 20-30 s 마다 10 분 이상 30 분 소용돌이-20 ° C에서 튜브를 품 어
참고: 튜브 저장할 수 있습니다-20 ° C에서 최대 1 주일 최소한의 단백질 저하 또는 수정. - 원심 관 (이상) 12000 × g에서 5 분 신중 하 게 제거 하 고 삭제는 상쾌한. 흰색 공 표시 되어야 합니다. (해당 되는 경우 펠 릿이 너무 건조 하 고, resuspend 하는 것이 어려울 것 이다) airdry 더 이상 5 분 동안에 펠 릿을 허용 합니다.
- 8 M 요소와 0.1 M 염화 중 탄산염의 300 µ L에서 단백질 펠 릿을 resuspend. 1 분에 강하게 소용돌이 색도계 분석 결과 사용 하 여 단백질 농도 결정 합니다.
9. 솔루션 소화 (프로토콜 2)
- 700 mM DTT 수성 해결책의 5.1 µ L을 추가 하 여 이황화 다리를 감소 (최종 농도 12.5 m m)에서 resuspended 단백질에 8.4 단계와 thermoshaker와 함께 30 분 동안 37 ° C에서 품 어. 700 m m iodoacetamide 용액의 20.3 µ L을 추가 하 여 시스테인 잔류물을 알 (최종 농도 40 m m)는 thermoshaker와 함께 어둠 속에서 30 분 25 ° C에서 배양 하 고.
- 샘플을 0.1 M 염화 중 탄산염의 990 µ L를 추가 합니다. 트립 신/리스-C 믹스 (효소: 기판 비율 1: 100 w/w)의 해당 볼륨을 추가 합니다. Thermoshaker는 37 ° C 하룻밤에 품 어.
10. 청소 카트리지 (프로토콜 2)
- 젖은 1 열-볼륨 (1 mL) 메탄올의 카트리지. 1 열-볼륨 (1 mL) 80% 이기/HPLC 급 물 카트리지를 청소 하 고 흐름을 통해 삭제. 4 열-볼륨 (4 mL) 0.1% 포 름 산/HPLC-등급 물 카트리지 equilibrate 그리고 흐름을 통해 삭제.
- 샘플 10% 개미 산 성 또는 pH 2-3 (pH 산도 표시기 확인)에 물 90 µ L 시 어 지 다. Acidified 샘플을 로드 하 고 흐름을 통해 수집 합니다. 자형 (not 유지 펩 티 드 포함) 다시 로드 합니다. 6 열-볼륨 (6 mL) 0.1% 포 름 산/HPLC-등급 물 카트리지를 씻어.
- 0.1% 개미의 acid/50% 이기/HPLC 급 물 펩 티 드 1 열-볼륨 (1 mL)으로 카트리지의 elute. Microcentrifuge 1.5 mL 튜브에 전송. 진공 농축 기 (150 x g, 160 mBar에서 진공)를 사용 하 여 샘플을 집중 한다.
11. 전자 초점 (프로토콜 2) 분류
참고: 펩 티 드 3에서 10의 pH 범위 13 cm 스트립 오프 젤 fractionator를 사용 하 여 그들의 전자 포인트에 따라 구분 됩니다. (아래 요약) 공급 업체에서 제공 하는 다음 프로토콜을 사용 했습니다.
- 준비 하는 다음과 같은 솔루션: 솔루션은 (600 µ L의 글리세롤 솔루션, 60 OFFGEL 버퍼의 µ L, 초순의 4.34 mL) 및 솔루션 (해결책의 1.776 mL)-초순의 444 µ L B.
- IPG 스트립, 프레임 및 제조업체의 지침에 따라 전극 조립.
- B 각 잘으로 솔루션의 솔루션 B. 추가 40 µ L의 1.8 mL과 샘플 resuspend 각 잘으로 150 µ L의 샘플을 로드 합니다.
- 펩 티 드의 기본 방법 선택: OG12PE00 (3100 OFFGEL 낮은 해상도 키트, pH 3-10, 12-잘 프레임 사용에 대 한 펩 티 드 OFFGEL 기본 방법. 이 방법은 완성 된 (~ 20 h) 되었습니다 때까지 기다립니다. 제대로 분류 관에 분수를 수집 합니다.
12. 청소 하버드 장치 열 역 C18 게시물-IEF (프로토콜 2)
- 1%의 한 번에 몇 μ를 점진적으로 추가 TFA 각 분수 드 이온된 수에 샘플을 시 어 지 다 하. PH는 약 3 또는 아래 pH 종이 사용 하 여 확인 합니다.
- 준비 하는 다음과 같은 솔루션: 솔루션 1 (이기의 5 mL, 10 µ L의 포 름 산, 초순 4.99 mL) 및 솔루션 2 (이기의 0.5 mL, 포 름 산, 초순 9.49 mL의 10 µ L).
- -스핀 열 솔루션 1의 150 µ L 젖은. 원심 분리기 90 g와 흐름을 통해 삭제 x 750에서 s. 솔루션 2의 150 μ와 스핀 열 세척. 원심 분리기 90 g와 흐름을 통해 삭제 x 750에서 s.
- 열을 통해 분수를 전달 합니다. 원심 분리기 90 g와 흐름을 통해 삭제 x 750에서 s. 솔루션 2의 150 μ 씻어. 원심 분리기 90 g와 흐름을 통해 삭제 x 750에서 s.
- 솔루션 1의 50 μ와 분수 elute 90에 대 한 원심 분리기 s 750 x g.에서 한 번 더 이러한 단계를 반복.
- 드라이-분수 (150 x g, 진공 160 mBar) 진공 집중 장치를 사용 하 여-80 ° C에서 저장
13. Proteomic 데이터와 생물 정보학18 의 분석
- 나노-LC MS에 의해 얻은 데이터를 분석/MS 질량 분석기 MaxQuant (버전 1.5.2.8) 등 정량화 소프트웨어 안드로메다 검색 엔진을 사용 하 여.
- 틀린 발견 비율 (FDR) 1% 단백질 및 펩 티 드와 아미노산 7의 최소 길이를 설정 합니다. C 터미널으로 효소 특이성을 설정 Arg를 리스. 프롤린 채권에 2 놓친된 분열을 허용 합니다. 변수 수정으로 고정된 수정 및 N 맨끝 단백질 acetylation 및 메티오닌 산화 시스테인의 carbamidomethylation를 선택 합니다.
- 추가 통계 분석 소프트웨어와 데이터를 분석 합니다. FunRich 소프트웨어 (www.funrich.org/)를 사용 하 여 기능 농축 분석을 수행 합니다. 데이비드 소프트웨어 (https://david.ncifcrf.gov/)를 사용 하 여 유전자 온톨로지 농축 분석을 수행 합니다.
결과
주변 혈액 단 세포 (PBMCs) 차등 원심 분리 하 여 얻은에서 시작, 프로토콜 허용 CD14의 인구를 얻는+ monocytes cytometry (그림 1)에 의해 98% 이상의 평가 순도 함께. 이러한 monocytes secondarily 다양 한 분극 (그림 2)으로 구분 됩니다. 젤에 분류 선택, SDS 페이지 젤에 마이그레이션 원하는 밴드의 번호에 맞게 이며 밴드 excised 됩니다 (그림 3). 소화는 젤의 삭제 밴드에서 secondarily 수행 후는 펩 티 드 추출 됩니다. 나노-LC (액체 크로마토그래피)를 사용 하는 펩 티 드 분석-MS/MS 질량 분 서 계. MS/MS 스펙트럼 알려진된 펩 티 드 (그림 4A)에 대 한 얻은 스펙트럼의 주석에 따라 다양 한 단백질의 id를 제공 합니다. 단백질의 풍부의 정량화 다음 게시 된 소프트웨어를 사용 하 여 데이터베이스15,16단백질에서 오는 확인 된 펩 티 드의 수량 관련 하 여 계산 됩니다. 이 프로토콜에서 젤 소화 약 4000 확인 된 단백질을 제공 하 고 동적 범위 커버 5 로그 눈금 단위 (그림 4B) 발견 되었습니다. 이 확인 된 단백질의 차동 식의 분석 결정 다른 산소 환경 하에서 다양 한 분극의 클러스터링을 사용할 수 있습니다.
이 방법으로, 우리 또한 최대-통제 된다 (그림 5, 표 1) 3%의 낮은 산소 농도에 노출 될 때 단백질의 클러스터를 인식할 수 있습니다. 소화의 효율성을 평가 하기 위해 불가능에 젤 프로토콜을 사용 하는 경우 우리 제안 인간의 세포 (그림 6A)에 적응 된 솔루션에서 소화 방법. 이 방법으로, 우리가 쉽게 (후에 솔루션 소화) 얻을 수 있습니다 분류, IEF 것 그 분류를 현명 하 게 의미 없이 3600 단백질의 증가이 (그림 6B).

그림 1: 표현의 CD14 PBMC 정렬 (왼쪽된 패널) 전후 (오른쪽 패널) 정렬 cytometry 분석 흐름 자석 구슬 선택 후 얻은 순도 보여주는. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2: 두 개의 다른 분극에 대 한 획득된 형태학의이 보여주는 차별화 된 인간 세포의 단계 대조 이미지. 눈금 막대 나타냅니다 50 µ m. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3: 스테인드 Coomassie 파란의 이미징 젤 excised 됩니다 다양 한 밴드를 보여주는 [여기, M(Ø) 세포에서 6 밴드] 대 식 세포는 저 산소 환경에 노출의 5 분극에 대 한. IC = 면역성이 있는 복합물, DXM dexamethasone =. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
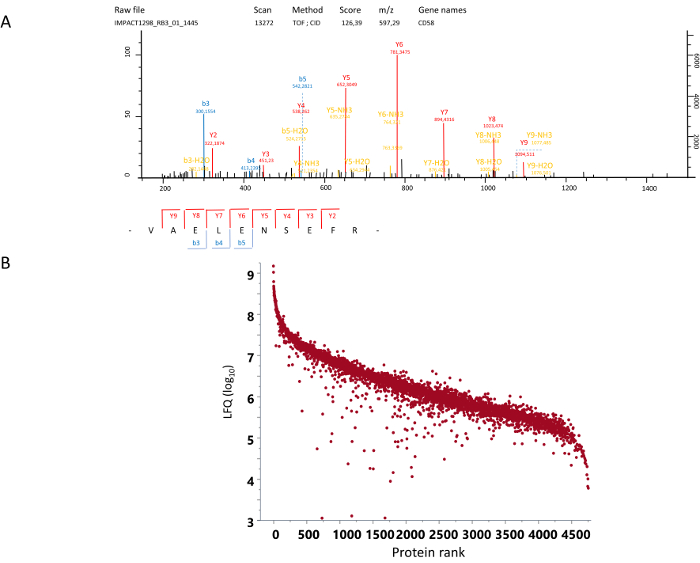
그림 4: MS/MS 스펙트럼 및 정량화. (A)는 MS/MS 스펙트럼의 예입니다. 여기에 표시 된 + 2 전기 요금 MS 스펙트럼에 m/z 597.29에 펩 티 드의 CID (충돌 유도 된 분리) 스펙트럼이 이다. 해당 시퀀스 CD58 단백질에서 Val-Ala-Glu-Leu-Glu-Asn-Ser-Glu-Phe-Arg로이 스펙트럼에서 결정 했다. (B) 순위 (로그인10 LFQ) 확인 된 단백질의 각 레이블 없는 정량화를 주문 했다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
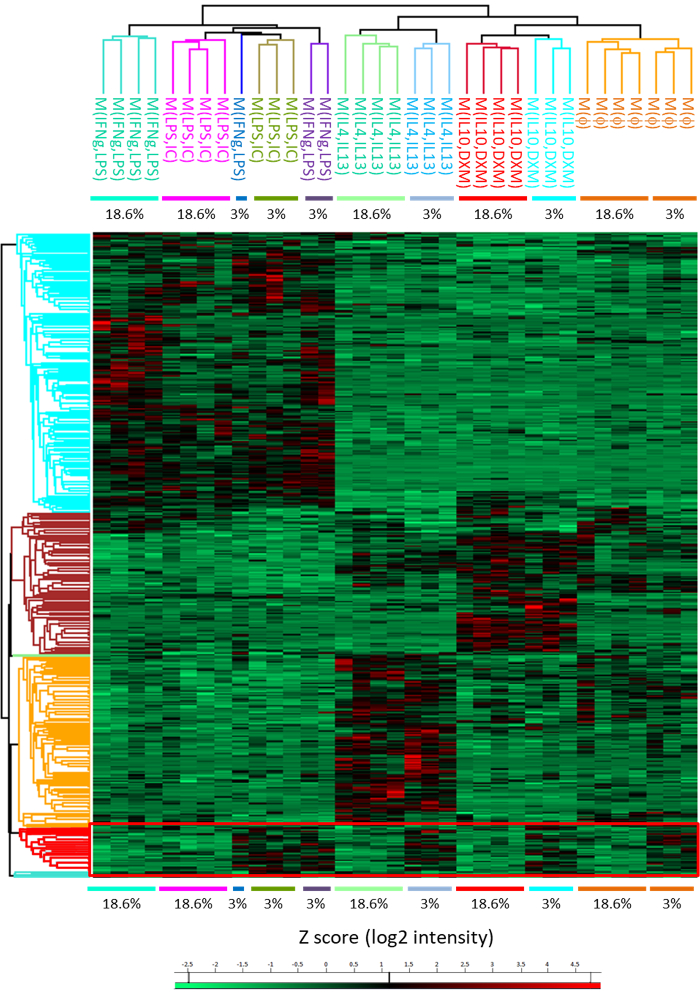
그림 5: 단백질 표현 사용 하 여 차동 열 지도 대표 하는 모든 분극의 계층적 클러스터링 상태. 분석 3% O2 상태 (빨간 사각형)에 있는 모든 분극에 overexpressed 단백질의 클러스터를 보여준다. 색 눈금은 z-점수 (log2 강도)을 나타냅니다. 각 행은 단백질 이며 각 열은 샘플입니다. 이 그림은 이전 게시14에서 유래. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 6: SDS 페이지 및 크로마. (A)은 스테인드 SDS 페이지 젤 세포 동안 저하의 부재와 소화의 효율을 보여주는 솔루션에 소화 후 단백질 세포 세포의 용 해에서와. (B)에서 분별 없이 솔루션에 소화 후 얻은 크로마. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| 클러스터 | proteinID | 단백질 이름 | 유전자 이름 | 펩 티 드 | 면도기 + 독특한 펩 티 드 | 고유한 펩 티 드 | 단백질 Id | ||
| 레드 | P0DMV9 | 열 충격 70 kDa 단백질 1B | HSPA1A | 40 | 37 | 4 | P0DMV9; P0DMV8; A8K5I0; Q59EJ3; B4DNT8; B4DWK5; B4DFN9; B4DI39; B4E1S9; B4DVU9; V9GZ37; B3KTT5; B4DNX1; B4E1T6; B4DNV4; Q9UQC1 | ||
| 레드 | P54709 | 나트륨/칼륨 수송 ATPase 소 단위 베타-3 | ATP1B3 | 8 | 8 | 8 | P54709; D3DNF9; C9JXZ1; C9JA36; H7C547; F8WBY4; Q58I18 | ||
| 레드 | O00462 | 베타-mannosidase | 의학 | 14 | 13 | 13 | O00462; A7LFP5; A8K6D3; E9PFW2; B4DT18; Q59EG5 | ||
| 레드 | Q8NAS7 | NADH 효소 [ubiquinone] 철-황 단백질 7, 미토 콘 드리 아 | NDUFS7 | 4 | 4 | 4 | Q8NAS7; Q6ZQU6; F5H5N1; B7Z1U1; A8K0V6; Q7LD69; F5GXJ1; O75251; B7Z4P1; Q9H3K5; A0A087WXF6; A0A087WTI3; Q6ZS38 | ||
| 레드 | Q8WWQ0 | 상호 작용 하는 PH 단백질 | PHIP | 5 | 5 | 4 | Q8WWQ0; Q9NWP3 | ||
| 레드 | E5RHK8 | Dynamin-3 | DNM3 | 11 | 2 | 2 | E5RHK8; Q9UQ16; B3KPF2; E5RIK2 | ||
| 레드 | A0A024QZ64 | 과 당-bisphosphate aldolase C | ALDOC | 18 | 14 | 7 | A0A024QZ64; P09972; B7Z1Y2; B7Z3K9; B7Z1N6; B7Z3K7; A8MVZ9; J3KSV6; J3QKP5; B7Z1L5; C9J8F3; K7EKH5; J3QKK1; B7Z1H6 | ||
| 레드 | O75489 | NADH 효소 [ubiquinone] 철-황 단백질 3, 미토 콘 드리 아 | NDUFS3 | 12 | 12 | 12 | O75489; Q9UF24; Q53FM7; E9PS48; E9PKL8; G3V194 | ||
| 레드 | P21912 | 호박 효소 [ubiquinone] 철 황 소 단위, 미토 콘 드리 아 | SDHB | 11 | 11 | 11 | P21912; A0A087WXX8; A0A087WWT1 | ||
| 레드 | A0A024R1Y7 | GH3 도메인에 포함 된 단백질 | LGP1; GHDC | 7 | 7 | 7 | A0A024R1Y7; Q8N2G8; B3KVB0; K7ESN3; K7EJT7; K7EQ41; K7EL54 | ||
| 레드 | E5KRK5 | NADH ubiquinone oxidoreductase 75 kDa 소 단위, 미토 콘 드리 아 | NDUFS1 | 29 | 29 | 29 | E5KRK5; P28331; B4DJ81; Q9P1A0; C9JPQ5; F8WDL5 | ||
| 레드 | A0A024QZ30 | 호박 효소 [ubiquinone] flavoprotein 소 단위, 미토 콘 드리 아 | SDHA | 17 | 17 | 17 | A0A024QZ30; P31040; D6RFM5; B3KT34; A0A087X1I3; Q0QF12; B4DYN5; B3KYA5; B4DNB2; H0Y8X1; B7Z6J5 | ||
| 레드 | A0A024R2F9 | 막 횡단 단백질 43 | TMEM43 | 16 | 16 | 16 | A0A024R2F9; Q9BTV4; Q8TEP9; V9GY05 | ||
| 레드 | A0A024R5K3 | NADH 효소 [ubiquinone] 철-황 단백질 8, 미토 콘 드리 아 | NDUFS8 | 5 | 5 | 5 | A0A024R5K3; E9PPW7; O00217; E9PN51; F8W9K7; E9PKH6; B4DYI3; Q53G17 | ||
| 레드 | O76003 | Glutaredoxin-3 | GLRX3 | 13 | 13 | 13 | O76003 | ||
| 레드 | Q1HBJ4 | 물질 활성화 한 단백질 키 니 아 제; 물질 활성화 한 단백질 키 니 아 제 1 | MAPK1 | 26 | 26 | 21 | Q1HBJ4; P28482; B4DHN0; Q499G7; K7ELV1; K7EN18; B4E104; P31152; Q16659 | ||
| 레드 | Q13151 | 이종 핵 ribonucleoprotein A0 | HNRNPA0 | 8 | 8 | 8 | Q13151 | ||
| 레드 | V9HWN7 | 과 당-bisphosphate aldolase A | ALDOA | 36 | 36 | 14 | V9HWN7; P04075; J3KPS3; H3BQN4; H3BUH7; H3BR04; H3BMQ8; H3BU78; H3BR68; A4UCS9; A4UCT0 | ||
| 레드 | B4DVJ0 | 포도 당-6-인산 isomerase | GPI | 32 | 32 | 23 | B4DVJ0; P06744; B4DE36; A0A0A0MTS2; K7EQ48; K7EP41; K7EPY4; K7ELR7; K7ERC6; K7ENA0; K7ESF4; K7EIL4; K7ERK8; Q59F85 | ||
| 레드 | V9HWB9 | L-젖 산 효소; L-젖 산 효소 체인 | LDHA | 36 | 36 | 34 | V9HWB9; P00338; B4DJI1; F5GXY2; F5GYU2; F5GXH2; F5H5J4; F5H6W8; F5GZQ4; F5H8H6; F5GXC7; F5GWW2; F5GXU1; A0A087WUM2; Q96L19; Q6ZMR3 | ||
| 레드 | P13674 | Prolyl hydroxylase 4 소 단위 알파-1 | P4HA1 | 26 | 26 | 26 | P13674; Q5VSQ6 | ||
| 레드 | Q6FHV6 | 감마-enolase; Enolase | ENO2 | 18 | 15 | 14 | Q6FHV6; P09104; A8K3B0; F5H0C8; F5H1C3; U3KQP4; U3KQQ1; Q9NPL4 | ||
| 레드 | Q99798 | Aconitate hydratase, 미토 콘 드리 아 | ACO2 | 40 | 40 | 40 | Q99798; B4DZ08; B2RBW5; A2A274; B4DJW1; B4DLY4; Q71UF1; B4DEC3; B4DW08; O75944 | ||
| 레드 | P17858 | ATP-종속 6-phosphofructokinase, 간 유형 | PFKL | 32 | 29 | 28 | P17858; Q7L2M7; Q9BSP4; B3KNQ7; B4E108; Q59GI2; F8WEU2; Q7Z3R9; Q6MZK4 | ||
| 레드 | A0A024R872 | 싱 같은 단백질 1 | FAM129B | 32 | 32 | 32 | A0A024R872; Q96TA1; Q9H8K1; Q2YD88; Q9H6L6 | ||
| 레드 | A0A024RC61 | Aminopeptidase N | ANPEP | 58 | 58 | 25 | A0A024RC61; P15144; Q59E93; B4DV63; B4DP01; B4DP96; H0YKT6; H0YLZ8; Q71E46; H0YMC1; Q8IVL7 | ||
| 레드 | V9HWF4 | Phosphoglycerate 니; Phosphoglycerate 키 니 아 제 1 | PGK1 | 41 | 41 | 35 | V9HWF4; P00558; B4E1H9; B4DHM5; B4DHB3; B4DWQ3; Q16444 | ||
| 레드 | Q12882 | Dihydropyrimidine 효소 [NADP(+)] | DPYD | 44 | 44 | 44 | Q12882; B4DML1 | ||
| 레드 | B4DEQ0 | 전자 전송 flavoprotein ubiquinone oxidoreductase, 미토 콘 드리 아 | ETFDH | 9 | 9 | 9 | B4DEQ0; Q547S8; A7UNU5; Q16134; D6RAD5 | ||
| 레드 | D9UAX9 | MHC 종류 I 항 원 | HLA-B | 13 | 3 | 2 | D9UAX9 | ||
| 레드 | V9HWK1 | Triosephosphate isomerase | TPI1 | 29 | 29 | 17 | V9HWK1; P60174; Q53HE2; B4DUI5; U3KPZ0; U3KQF3; U3KPS5 | ||
| 레드 | Q96HE7 | ERO1 같은 단백질 알파 | ERO1L | 28 | 28 | 28 | Q96HE7; G3V3E6; G3V5B3; G3V2H0; G3V503; Q5TAE8; B2RD00; Q86YB8 | ||
| 레드 | P14868 | Aspartate-tRNA 리가, 세포질 | DARS | 29 | 29 | 2 | P14868; Q53T60; Q53R85; H7BZ35; H7C278 | ||
| 레드 | P36871 | Phosphoglucomutase-1 | PGM1 | 24 | 24 | 5 | P36871; B4DFP1; Q9H1D2 | ||
표 1: 각 분극 낮은 산소 긴장의 밑에 일반적인 인간의 세포에 대 한-표현된 단백질의 목록입니다.
토론
Proteomics 전체 셀 또는 subcellular 구획에서 다른 단백질의 표현을 공부 하는 강력한 도구 이므로 세포 세포의 용 해 프로토콜의 최적화 및 단백질의 소화 연구의 숫자에 의해 수정 되었습니다. 젤 소화 (polyacrylamide 젤 매트릭스에 단백질의 소화)17, 솔루션18 및 필터 지원 샘플 준비19에서 소화를 포함 하는 방법의 세 가지 주요 클래스 있다. 이 마지막 방법은 보편적으로 설명 하는 처음에 낮은 재현성 및 필터20에 단백질의 가능한 손실에 보고 되었습니다. 젤 소화 가능한 경우 소화의 효율성을 평가 하는 것은 쉽게, 어렵고 불리 될 수 있는 강력한 방법입니다. 솔루션에서 소화 소화와 IEF 후 샘플을 청소 하지만이 가능성을 제공 합니다. 비교 될 때 이러한 두 메서드는 동일한 샘플 사이, IEF 분별 프로토콜 솔루션에 소화 보다 젤 소화21(와 같은 수의 분수) 확인 된 단백질의 높은 번호를 생성 합니다.
이 장점에도 불구 하 고 (특히 골수성 세포)에 세포내 프로 테아 제 때문에 솔루션 세포 중 가능한 단백질 저하를 고려할 필요가 있다. 이러한 기술을 기반으로 단백질 소화에서 중요 하 고 특정 분열 사이트 트립 신을 제시 하는 단백질을 분석할 수 이기도 합니다. 이 소화 제약을 해소 하지만 데이터 분석 단계와 생물 정보학 능숙해22추가 내려 proteomic 접근을 사용 하 여 가능 하다. 다양 한 세포질 구획에서 단백질의 가용 화 세포 프로테옴의 통제 샘플링을 선도 하는 플라즈마 세포 막에서 특히 얻기도 어려울 수 있습니다. 샘플의 나노-LC-MS/MS 질량 분석기 분석으로 진행 하기 위해서는 펩 티 드 질량 분석기 사용에 따라 수의 충분 한 수량을 얻을 하는 것이 중요 하다 (일반적으로, 총 단백질 시작 있어야 조건에 대 한 적어도 1 µ g 및 그것은 암시 IEF와 함께 사용 하는 분수의 수에 따라이 수량 증가). 이 제약 조건은 단점은 공부 되 고 세포 인구 부족, 경우 proteomic 게놈 기술 원료의 증폭 가능 하다에서 분화 하 수 있습니다.
더 부유한과 동료23 , 패 커와 Fuehr24의 정액 작품 후에 세포 배양에서 산소의 중요성을 충분히 인식 하고있다 하지. 우리는 지금 그 접착, 수명과 호의 낮은 산소 농도에서 세포 배양 알고 있다. 이 줄기 세포 연구25에 최대 중요성은 인식 된다. 제어 산소 조건에서 세포 배양에 대 한 주요 기술 문제는 전체 실험 기간 동안 원하는 산소 농도의 유지 보수 관련. 이 녹은 산소의 방출을 방지 하 고 사용 하 hypoxic 작업 스테이션의 낮은 산소 (글러브 박스와 챔버 처리)에서 세포의 조작을 허용 하 고 높은 산소 조건에 일시적인 폭발 방지 모든 미디어의 사전 부 화 필요 .
설명된 프로토콜의 인간 monocyte 파생 된 대 식 세포의 다양 한 분극 분자 서명이 서명에 산소 변조 효과 공부 하 사용 되었다. 이 연구에 그의 분극의 설명 통찰력을 부여 하고있다와 일부 기능 결과 드러났다. 예를 들어 우리는 efferocytosis에 관련 된 많은 단백질 저 산소 환경에 의해 변조 했다 발견. 설명 된 프로토콜에 따라이 proteomic 접근 방법과 이러한 신호 사용하실 수 있는 새로운 치료 접근14디자인 환경 매개 변수는 대 식 세포 기능을 수정 하는 방법을 탐구 하는 기회를 선물 한다.
이 작품에서 설명 proteomic 접근은 인간 대 식 세포 양극 화 연구의 분야에서 최근 몇 년 동안 사용 된 게놈 접근을 보완. Proteomics는 포스트 번역 상 수정 때문에 그들의 대응 mRNAs 보다 다른 식을 제시 하 고 새로운 바이오 마커의 발견으로 이어질 수 있습니다 단백질 정량화의 이점을 제공 합니다. 이러한 장점에도 불구 하 고 proteomic 데이터는 일반적으로 질량 분석, 매우 복잡 한 MS 스펙트럼 및 펩 티 드의 거짓인 긍정적인 탐지의 높은 감도 때문에 해석 하기가. 최근, 분석 소프트웨어는 이것을 방지 하기 위해 효율을 얻고 있다. Proteomics 또한 유전체학26 보다 낮은 재현성을 직면 하 고 다른 기술 (cytometry immunoblotting) 단백질의 양적 수정 확인을 사용 하 여 유효성 검사 단계와 연결 된 변화 상황 인 경우에 식 수준입니다.
공개
저자는 관심 없음 충돌 선언합니다.
감사의 말
오전 국가 리그 죄수 르 암과 라 Fondation 아크 부 라 검색 쉬르 르 암 라 하 여 젊은 그룹 지도자 프로그램 (ATIP/미래 Inserm-CNRS)에 의해 자금이 다. 우리는 생물학 플랫폼 (UTECHS MSBIO, 파스퇴르 연구소, 파리)에 대 한 질량 분석에서 Mariette Matondo 감사합니다. 우리는 원고의 그녀의 독서에 대 한 로렌 앤더슨을 감사합니다.
자료
| Name | Company | Catalog Number | Comments |
| Hypoxia Working Station | Oxford Optronix | Hypoxylab | |
| C6 Flow cytometer | BD | Accuri C6 | |
| Urea | Agilent Technologies | 5188-6435 | |
| Formic acid (FA) | ARISTAR | 450122M | |
| R-250 Coomassie blue | Biorad | 1,610,436 | |
| Lipopolysaccharide, E.Coli (LPS) | Calbiochem | 437627 | |
| 2D clean-up kit | GE Healthcare | 80-6484-51 | |
| RPMI 1640 medium, glutamax supplement | Gibco | 61870044 | |
| HEPES 1 M | Gibco | 15630-080 | |
| MEM Non-Essential Amino Acids (NEAA) Solution 100X | Gibco | 11140-035 | |
| Phosphate Buffered Saline (PBS) 1X | Gibco | 14190-094 | |
| Harvard Apparatus column Reverse C18 micro spin column | Harvard Apparatus | 74-4601 | |
| EDTA 0.5 M, pH 8.0 | Invitrogen | AM9260G | |
| NuPAGE Bis-Tris 4-12% | Life Technologies SAS | NP0321 BOX | |
| CD14 Microbeads human | Miltenyi Biotec | 130-050-201 | |
| MACS separation column LS | Miltenyi Biotec | 130-042-401 | |
| Macrophage colony-stimulating factor (M-CSF) | Miltenyi Biotec | 130-096-485 | |
| Interleukin 4 (IL4) | Miltenyi Biotec | 130-093-917 | |
| Interleukin 13 (IL13) | Miltenyi Biotec | 130-112-410 | |
| Interferon gamma (INFγ) | Miltenyi Biotec | 130-096-482 | |
| CD14-FITC (clone TÜK4) | Miltenyi Biotec | 130-080-701 | |
| MACSmix Tube Rotator | Miltenyi Biotec | 130-090-753 | |
| Trifluoroacetic Acid (TFA) | Pierce | 28904 | |
| Trypsin/Lys-C Mix | PROMEGA | V5073 | |
| Complete Mini, EDTA-free Protease Inhibitor cocktail | Roche | 11836170001 | |
| Density Gradient Solution (Histopaque 1077) | Sigma Aldrich | 10771-100ML | |
| Accumax | Sigma Aldrich | A7089-100ML | |
| Human Serum from human male AB plasma (SAB) | Sigma Aldrich | H4522-100ML | |
| Bovine Serum Albumin (BSA) solution 30% | Sigma Aldrich | A9576-50ML | |
| Trisma-base | Sigma Aldrich | T1503 | |
| Glycerol | Sigma Aldrich | 49767 | |
| β-Mercaptoethanol | Sigma Aldrich | M3148 | |
| Bromophenol blue | Sigma Aldrich | 114405 | |
| Sodium Dodecyl Sulfate (SDS) 20% | Sigma Aldrich | 5030 | |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | |
| Acetonitrile | Sigma Aldrich | 34888 | |
| Dithiothreitol | Sigma Aldrich | 43819 | |
| Iodoacetamide | Sigma Aldrich | 57670 | |
| Thiourea | Sigma Aldrich | T8656 | |
| CHAPS | Sigma Aldrich | C9426 | |
| Micro BCA Assay Kit | ThermoFisher | 23235 | |
| 5 mL sterile plastic pipette | VWR | 612-1685 | |
| Thermomixer C Eppendorf | VWR | 460-0223 | |
| Sep-Pak tC18 reverse phase cartridges, 100 mg | Waters | WAT036820 |
참고문헌
- Okabe, Y., Medzhitov, R. Tissue biology perspective on macrophages. Nature Immunology. 17 (1), 9-17 (2016).
- Sica, A., Mantovani, A. Macrophage plasticity and polarization: in vivo veritas. The Journal of Clinical Investigation. 122 (3), 787-795 (2012).
- Murray, P. J. Macrophage Polarization. Annual Review of Physiology. 79, 541-566 (2017).
- Chinetti-Gbaguidi, G., Staels, B. Macrophage polarization in metabolic disorders: functions and regulation. Current Opinion in Lipidology. 22 (5), 365-372 (2011).
- Chow, A., Brown, B. D., Merad, M. Studying the mononuclear phagocyte system in the molecular age. Nature Reviews Immunology. 11 (11), 788-798 (2011).
- Mosser, D. M., Edwards, J. P. Exploring the full spectrum of macrophage activation. Nature Reviews Immunology. 8 (12), 958-969 (2008).
- Murray, P. J., Wynn, T. A. Protective and pathogenic functions of macrophage subsets. Nature Reviews Immunology. 11 (11), 723-737 (2011).
- Ginhoux, F., Schultze, J. L., Murray, P. J., Ochando, J., Biswas, S. K. New insights into the multidimensional concept of macrophage ontogeny, activation and function. Nature Immunology. 17 (1), 34-40 (2016).
- Xue, J., et al. Transcriptome-based network analysis reveals a spectrum model of human macrophage activation. Immunity. 40 (2), 274-288 (2014).
- Csete, M. Oxygen in the cultivation of stem cells. Annals of the New York Academy of Sciences. 1049, 1-8 (2005).
- Johansson, A., et al. Functional, morphological, and phenotypical differences between rat alveolar and interstitial macrophages. American Journal of Respiratory Cell and Molecular Biology. 16 (5), 582-588 (1997).
- Pfau, J. C., Schneider, J. C., Archer, A. J., Sentissi, J., Leyva, F. J., Cramton, J. Environmental oxygen tension affects phenotype in cultured bone marrow-derived macrophages. American Journal of Physiology. Lung Cellular and Molecular Physiology. 286 (2), L354-L362 (2004).
- Grodzki, A. C. G., Giulivi, C., Lein, P. J. Oxygen tension modulates differentiation and primary macrophage functions in the human monocytic THP-1 cell line. PLoS One. 8 (1), e54926(2013).
- Court, M., Petre, G., Atifi, M. E., Millet, A. Proteomic Signature Reveals Modulation of Human Macrophage Polarization and Functions Under Differing Environmental Oxygen Conditions. Molecular & Cellular Proteomics. 16 (12), 2153-2168 (2017).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Cox, J., Neuhauser, N., Michalski, A., Scheltema, R. A., Olsen, J. V., Mann, M. Andromeda: a peptide search engine integrated into the MaxQuant environment. Journal of Proteome Research. 10 (4), 1794-1805 (2011).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols. 1 (6), 2856-2860 (2006).
- Court, M., et al. Toward a standardized urine proteome analysis methodology. Proteomics. 11 (6), 1160-1171 (2011).
- Wiśniewski, J. R., Zougman, A., Nagaraj, N., Mann, M. Universal sample preparation method for proteome analysis. Nature Methods. 6 (5), 359-362 (2009).
- Liebler, D. C., Ham, A. -J. L. Spin filter-based sample preparation for shotgun proteomics. Nature Methods. 6 (11), 785-786 (2009).
- Hubner, N. C., Ren, S., Mann, M. Peptide separation with immobilized pI strips is an attractive alternative to in-gel protein digestion for proteome analysis. Proteomics. 8 (23-24), 4862-4872 (2008).
- Park, J., et al. Informed-Proteomics: open-source software package for top-down proteomics. Nature Methods. 14 (9), 909-914 (2017).
- Richter, A., Sanford, K. K., Evans, V. J. Influence of oxygen and culture media on plating efficiency of some mammalian tissue cells. Journal of the National Cancer Institute. 49 (6), 1705-1712 (1972).
- Packer, L., Fuehr, K. Low oxygen concentration extends the lifespan of cultured human diploid cells. Nature. 267 (5610), 423-425 (1977).
- Lengner, C. J., et al. Derivation of pre-X inactivation human embryonic stem cells under physiological oxygen concentrations. Cell. 141 (5), 872-883 (2010).
- Sidoli, S., Kulej, K., Garcia, B. A. Why proteomics is not the new genomics and the future of mass spectrometry in cell biology. Journal of Cell Biology. 216 (1), 21-24 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유