Method Article
Proteomic Analysen der menschlichen Makrophagen Polarisierung in einer sauerstoffarmen Umgebung
In diesem Artikel
Zusammenfassung
Wir präsentieren Ihnen ein Protokoll zur Proteomic Unterschriften von menschlichen Makrophagen und wenden Sie diese zur Bestimmung der Auswirkungen einer sauerstoffarmen Umgebung auf Makrophagen Polarisation.
Zusammenfassung
Makrophagen sind angeborene Immunzellen beteiligt in einer Reihe von physiologischen Funktionen von Antworten auf infektiöse Erreger bis hin zu Gewebe Homöostase. Die verschiedenen Funktionen dieser Zellen beziehen sich auf deren Aktivierungsstatus die Polarisation auch genannt. Der genaue molekulare Beschreibung dieser verschiedenen Polarisationen ist eine Priorität im Bereich der Makrophagen Biologie. Anerkanntermaßen ist derzeit die ein mehrdimensionaler Ansatz ist notwendig, um zu beschreiben wie Polarisation durch Umweltsignale gesteuert wird. In diesem Bericht beschreiben wir ein Protokoll entwickelt, um die Proteomik-Signatur von verschiedenen Polarisationen in menschliche Makrophagen zu erhalten. Dieses Protokoll basiert auf einem markierungsfreie Quantifizierung der Makrophagen-Protein-Expression von fraktioniert auf dem Gel und Lys C/Trypsin-verdaut zellulären Lyse Inhalt erhalten. Wir bieten auch ein Protokoll basierend auf in-Lösung Verdauung und isoelektrischen Fokussierung Fraktionierung, als Alternative zu verwenden. Da Sauerstoff-Konzentration einen relevanten ökologischen Parameter im Gewebe ist, verwenden wir dieses Protokoll, um wie atmosphärische Zusammensetzung zu erkunden oder eine sauerstoffarmen Umgebung wirkt sich auf die Klassifizierung von Makrophagen Polarisation.
Einleitung
Makrophagen sind angeborene Immunzellen beteiligt in einer Reihe von physiologischen Funktionen von Antworten auf infektiöse Erreger bis hin zu Gewebe Homöostase, einschließlich der Entfernung des apoptotischen Zellen und der extrazellulären Matrix1Umbau. Diese Zellen zeichnen sich durch eine starke phänotypische Plastizität2 , die in einem viele mögliche Aktivierung Staaten übersetzt die Polarisationen auch genannt werden. Der genaue molekulare Beschreibung dieser verschiedenen Polarisationen ist eine Priorität im Bereich der Makrophagen Biologie3. Es wurde vorgeschlagen, diese mit Hilfe der sogenannten M1/M2 Dichotomie, in denen M1 steht Pro-inflammatorischen und M2 für entzündungshemmende Makrophagen Polarisationen zu klassifizieren. Dieses Modell passt gut in verschiedenen pathologischen Situationen wie akute Infektionen, Allergien und Übergewicht4. Allerdings wurde in chronisch entzündeten Geweben und Krebs nachgewiesen, dass diese Klassifizierung nicht in der Lage ist, das breite phänotypische Repertoire zu erfassen, das Makrophagen in bestimmten zellulären Umgebungen5,6, zu präsentieren 7. der aktuelle Konsens ist, dass Makrophagen Polarisation besser ein mehrdimensionales Modell beschrieben ist, um spezifische microenvironmental Signale8zu integrieren. Diese Schlussfolgerung wurde bestätigt durch die transkriptomischen Analyse der menschlichen Makrophagen, die zeigen, dass das Modell M1/M2 bei der Beschreibung der erhaltenen Polarisationen9ineffizient ist.
Die Studie präsentiert zielt darauf ab, ein Protokoll zur Proteomic Unterschriften von verschiedenen Polarisationen in menschliche Makrophagen zu erhalten. Wir beschreiben, wie menschliche Makrophagen im Umfeld der verschiedenen Sauerstoff-Niveaus zu unterscheiden und das ganze Makrophagen Proteom markierungsfreie Quantifizierung durchführen Peptide einzuholen. Diese Quantifizierung ermöglicht den Vergleich der Ausdruck verschiedener Proteine. Wie die Forschung an Stammzellen die Bedeutung von Sauerstoff als eine ökologische Schlüsselparameter10gezeigt hat, versuchen wir zu verstehen, wie dieser Parameter Gewebe Makrophagen Polarisierung in den Menschen beeinflussen kann. Der Partialdruck des Sauerstoffs ist Reichweite von 3 bis 20 % (des gesamten atmosphärischen Drucks) im menschlichen Körper gefunden worden wo entspricht in etwa 20 %, was in einer Zelle Kultur Inkubator verbreitet ist (der genaue Wert ist rund 18,6 % während der Einnahme der Anwesenheit von Wasser nicht berücksichtigt).
Bisherigen Arbeit hat gezeigt, dass alveoläre unterscheiden sich von interstitielle Makrophagen aus funktionellen und morphologischen Ansichten11 und diese Unterschiede wahrscheinlich teilweise aufgrund der unterschiedlichen Sauerstoff-Niveaus sind, die sie ausgesetzt12sind. Darüber hinaus zeigen dem Knochenmark stammenden Makrophagen eine erhöhte Fähigkeit, Bakterien bei einer sauerstoffarmen Umgebung12phagocytize. Der gegenteiligen Effekt hat für THP1 differenziert menschliche Makrophagen13gefunden worden, aber diese Ergebnisse unterstützen die Idee, dass Sauerstoff ein Regulator von Makrophagen Biologie ist, es ist zu klären, diese Rolle auf molekularer Ebene in menschliche Makrophagen. In einer früheren Studie haben wir einen Proteomics-Ansatz zur Behebung dieser Probleme angewendet. Durch gleichzeitige Messung Ausdruck Niveaus für Tausende von Proteinen, wir haben die Auswirkungen von Sauerstoff auf Polarisierung und stellte eine Liste von neuen molekularen Markern. Wir waren auch in der Lage, diese Erkenntnisse zu einigen Funktionen der Makrophagen zu beziehen. Vor allem fanden wir, dass die Rate der Phagozytose apoptotischer Zellen im IL4/IL13-polarisierten Makrophagen erhöht wurde, die an die Hochregulation des ALOX15 gebunden war, wie der Proteomik Analyse14offenbart. In der vorliegenden Studie beschreiben wir wie eine solche Analyse durchführen.
Protokoll
Menschlichen Blutproben (LRSC) von gesunden, anonymisierte Spender wurden von EFS (Französisch nationalen Blut Service) im Rahmen eines genehmigten Protokolls (CODECOH DC-2018-3114) erhalten. Gebern gab unterschriebene Zustimmung für die Verwendung von Blut.
(1) Medien und Puffer-Vorbereitung
- Bereiten Sie die Makrophagen Medium [RPMI Glutamax + 10 mM HEPES + 1 X nicht-essentiellen Aminosäuren (NEAA)] und auf 37 ° c warm
- Vorbereiten der Makrophagen Medium + 10 % humanem Serum aus AB Plasma (SAB), Filtern (0,22 µm Filter), dann auf 37 ° C warm (bezeichnet als Makrophagen Medium + 10 % SAB im folgenden).
- Vorbereiten des Sortierung Puffers [1 x Phosphat gepufferte Kochsalzlösung (PBS) + 0,5 % Rinderserumalbumin (BSA) + 2 mM Ethylenediaminetetraacetic Säure (EDTA)], Filtern (0,22 µm Filter) und pflegen sie bei 4 ° c
2. Isolierung der peripheren mononukleären Blutzellen (PBMCs) aus Leukoreduction System Kammer (LRSC)
- 15 mL Dichte Gradienten Zelle Trennung Lösung setzen (siehe Tabelle der Materialien) in einem 50 mL tube Zentrifugation so dass es auf Raumtemperatur (RT) vor dem Erhalt der LRSC wärmen kann.
Hinweis: Dichte hängt von Temperatur. Da dieses Produkt bei 4 ° C gelagert wird, muss dieser Schritt im Voraus erfolgen, damit es zu RT equilibrate können - Leeren Sie die LRSC in ein 50 mL-Röhrchen Zentrifugation, fügen Sie bis zu 50 mL 1 X PBS und mischen. Hinzugeben Sie 25 mL der Mischung während Schritt 2.2 auf 15 mL Dichte Gradienten Lösung erwärmt während Schritt 2.1 vorbereitet sehr langsam.
Hinweis: Achten Sie darauf, die Phasen während dieses Schrittes zu mischen. Das Blut muss auf die Dichte Gradienten Lösung ohne Störung dieser Phase hinzugefügt werden. - Zentrifugieren Sie beide Zentrifugation Rohre für 25 min bei 700 X g ohne Pausen.
Hinweis: Am Ende der Dichte-Gradienten-Zentrifugation, sind die Ebenen von unten nach oben: die Erythrozyten und Granulozyten bilden die Pellet, Dichte Gradienten Lösungsphase, Schicht von PBMCs und Plasma. - Mit einem Pipettieren der Plasma-Phase durchlaufen Sie, ohne es Absaugen und sammeln Sie die PBMC Schicht in eine neue 50 mL Zentrifugation Rohr. Die PBMCs als Waschschritt und Zentrifuge für 10 min bei 300 X g bis zu 50 mL 1 X PBS hinzufügen.
- Aspirieren Sie überstand und Aufschwemmen Sie das Pellet in 40 mL Makrophagen Medium.
3. magnetische Kennzeichnung und Isolierung von CD14+ Zellen (Monozyten)
- Zählen Sie die PBMCs in einer Malassez-Kammer. Abheben Sie der Betrag der PBMCs notwendig für die Durchführung des Experiments (in der Regel 100 bis 300 x 106 Zellen), legen Sie sie in eine Zentrifugation Rohr- und Zentrifuge für 10 min bei 300 X g.
- Den Überstand abgesaugt und erneut das Pellet in 80 µL des Puffers Sortierung während Schritt 1.3 pro 10 vorbereitet7 PBMCs Add 20 µL CD14 Microbeads pro 107 PBMCs. Mix gut und inkubieren Sie für 15 min bei 4 ° C unter ständiger Bewegung.
- Fügen Sie 1 mL Puffer pro 107 PBMCs zu sortieren, als Waschschritt und Zentrifuge für 10 min bei 300 X g. überstand Aspirieren und das Pellet in 500 µL Puffer pro 108 PBMCs Sortierung aufzuwirbeln.
- Legen Sie eine Spalte in das Magnetfeld des Separators. Bereiten Sie die Spalte durch Spülen mit 3 mL Puffer zu sortieren.
- Anwenden der Zellsuspension auf die Säule. Die Spalte hängt von der Anzahl der Zellen isoliert werden (hier LS Spalten für bis zu 109 PBMCs verwendet werden). Durchfluss mit unmarkierten Zellen zu sammeln.
Hinweis: Ab diesem Schritt, sind alle Rohre (negative und positive Selektionen) gehalten für die später Überprüfung der verschiedenen Schritte Durchflusszytometrie. - Waschen Sie die Spalte mit 3 x 3 mL Puffer zu sortieren. Unbeschriftete Zellen, die durch das gleiche Rohr aus Schritt 3.12 zu sammeln. Durchführen Sie Waschschritte durch Hinzufügen von Sortierung Puffer, achten Sie darauf, Ihre Spalte trocken. Legen Sie ein Sammelröhrchen unter der Spalte und entfernen Sie sie aus dem Abscheider.
- Pipette 5 mL Puffer in die Spalte zu sortieren. Sofort die magnetisch markierten Zellen fest drücken Sie den Kolben in die Spalte auszuwaschen. Erhöhung der Reinheit der CD14+ Zellen, der eluierten Bruch wird über eine zweite Spalte bereichert.
- Wiederholen Sie die Schritte 3.4 bis 3.7 mit einer neuen Spalte.
(4) Beschichtung von Monozyten
- Zählen Sie die Monozyten in einer Malassez-Kammer. Überprüfen Sie die Reinheit der CD14+ Zellen durch Durchflusszytometrie. Zurückziehen Sie die Höhe der Monozyten notwendig für das Experiment und legen Sie sie in ein Röhrchen Zentrifugation.
- Zentrifugieren Sie für 10 min bei 300 X g. Aspirat überstand und Aufschwemmen der Monocyte Pellet in Makrophagen Medium. Platte der Zellen und lassen sie die Regeln für 50 min bis 1 h aspirieren Sie das Medium und ersetzen Sie es mit Makrophagen Medium + 10 %, dass SAB + 25 ng/mL Makrophagen Kolonie stimulierende Faktor (M-CSF) um die Differenzierung zu induzieren.
(5) Polarisation von Makrophagen an Tag 6
- Aspirieren Sie das Medium. Ersetzen Sie ihn durch Makrophagen Medium + 10 % SAB mit verschiedenen Reizen. Beispielsweise fügen Sie 10 ng/mL Interferon Gamma (INFγ) + 1 ng/mL Lipopolysaccharid (LPS), M1 Polarisation zu erhalten oder 20 ng/mL Interleukin 4 (IL4) + 20 ng/mL Interleukin 13 (IL13) für M2 Polarisation.
Hinweis: Die Stimulation kann zwischen 24 und 48 Stunden, bevor Sie fortfahren, um weitere Tests durchgeführt werden. - Ernten Sie Zellen mit einer trennen Lösung oder Schabeisen Zelle.
(6) Zellkultur unter sauerstoffarmen Bedingungen
- Ab Schritt 4, pflegen Sie die Monozyten und Makrophagen in einer Sauerstoff-kontrollierten Umgebung hypoxischen Zustandsanalyse durchzuführen. Verwenden Sie eine Hypoxie Arbeitsplatz um Zellen unter der gewünschten Sauerstoffpartialdruck während des Experiments zu erhalten.
Hinweis: Beim Arbeiten unter niedrigen Sauerstoffdruck ist es wichtig, alle Medien und waschen Puffer unter dem Bahnhof vorzubereiten und ausreichend warten, bis der richtige Partialdruck in der Flüssigkeit zu erhalten. Zum Beispiel erfordert 10 mL PBS in einer 60 mm Petrischale etwa 2 h, 25 MmHg für O2 Partialdruck ab atü betragen (wie wir sie mit einem Glasfaser-Sauerstoffsensor gemessen haben) zu erreichen. In vielen hypoxischen Stationen oder Inkubatoren wird als prozentualer Anteil des atmosphärischen Drucks der Sauerstoffdruck festgelegt. Präzise Messungen erforderlich sind, ist es besser, verwenden Sie eine Station autorisieren, direkt den Sauerstoffdruck in MmHg festzulegen.
(7) lyse und In Gel Verdauung (Protokoll 1)
Hinweis: In diesem und den folgenden Abschnitten werden zwei Protokolle verwendet, um zu erhalten Peptide und LC-MS/MS-Analyse durchführen beschrieben. Protokoll Nr. 1 beschreibt Zelle Lysis und Fraktionierung in Gel und Verdauung und Protokoll 2 beschreibt in Lösungszelle lyse in Lösung Verdauung und Fraktionierung mit einem isoelektrischen Fokussierung Verfahren folgten.
- Lyse der Zelle im Laemmli-Puffer [234 mM Tris-HCL (pH 6,8), 7,5 % SDS, 37 % Glycerin, 33,3 % (V/V) β-Mercaptoethanol, Bromophenol blue 0,2 % w/V] durchführen. Laden Sie das Protein-Äquivalent von 300.000 Zellen für jede Probe auf 4-12 % Bis-Tris Acrylamid Gele.
- Steuern Sie die Dauer der elektrophoretische Migration erlauben jedes Protein Sample 6 Gel Bänder aufgeteilt werden soll, wie in Abbildung 3veranschaulicht.
- Befestigen Sie das Gel mit Festlegunglösung (30 % Ethanol + 7,5 % Essigsäure für 20 min), dann fügen Sie die Färbelösung (R-250 Coomassie Blau für 45 min). Fügen Sie die destaining-Lösung (30 % Ethanol + 7,5 % Essigsäure bis Streifen erscheinen) vor den Protein-Bands mit einem sauberen Skalpell besteuert.
- Jeder Würfel herausgeschnitten Band vor Einführung in 500 µL Röhren. Eine saubere Glasoberfläche ist berechtigt, zur Vermeidung von Kontaminationen mit Keratine (5 % SDS-Lösung in entionisiertem Wasser kann verwendet werden, um Oberflächen zu reinigen).
- Waschen Sie die Gel-Scheiben 3mal in 200 µL 25 mM Ammonium Bicarbonat für 20 min bei 37 ° C, gefolgt von einem waschen in 25 mM Ammonium Bicarbonat und Acetonitril (50 % V/V). Die Gel-Stücke mit 200 µL des 100 % Acetonitril für 10 min zu entwässern.
- Inkubieren Sie jedes Stück Gel mit 10 mM DTT (Dithiothreitol) in 25 mM Ammonium Bicarbonat für 45 min bei 56 ° C (200 µL), gefolgt von 55 mM Iodoacetamide in 25 mM Ammonium Bicarbonat (200 µL) für 35 min im Dunkeln bei RT
- Alkylierung zu stoppen, brüten jedes Gel mit 200 µL 10 mM DTT in 25 mM Ammonium Bicarbonat für 10 min bei RT. Wash Gel Stücke in 200 µL von 25 mM Ammonium Bicarbonat und dann mit 200 µL 100 % Acetonitril für 10 min zu entwässern.
- Die Proteine über Nacht bei 37 ° C mit Trypsin/Lys-C-Mix entsprechend den Anweisungen des Herstellers zu verdauen.
- Die daraus resultierenden Peptide aus Gel Stücke durch Zugabe von 50 μL der 50 % Acetonitril für 15 min, dann 50 μL der 5 % Ameisensäure für 15 min und schließlich 50 μL der 100 % Acetonitril für 15 min. Pool zu extrahieren und trocknen jede Fraktion in niedrig-Absorption Röhren, Adsorption von Peptiden zu begrenzen und Probenverlust. Speichern Sie die Proben bei-80 ° C bis zur weiteren Analyse.
8. die Proteingewinnung und In Lösung Verdauung (Protokoll Nr. 2)
- Durchführen Sie lyse der Zelle (2 x 10-6 -Zellen) mit 150 µL des Puffers folgende Lyse:
- 7 M Harnstoff, 2 M Thioharnstoff, 40 mM Tris und 4 % CHAPS, ergänzt mit Protease-Inhibitoren (komplette Mini, EDTA-freie Protease-Inhibitor cocktail).
- Die Lösungen für 30 min bei RT mit einem Thermoshaker zu homogenisieren. Zentrifugieren bei 13.800 x g für 20 min RT und den Überstand zu halten.
- Entfernen Sie Verschmutzungen mit einem 2D Aufräum Kit:
- Der Bausatz enthält Fällungsmittel Lösung, Co Fällungsmittel Lösung und Waschpuffer waschen Additiv.
- 300 µL des Fällungsmittels Lösung hinzufügen und gut verrühren. Inkubieren Sie für 15 min. hinzufügen 300 µL Co Fällungsmittel Lösung auf Eis. Zentrifugieren Sie die Rohre (mindestens) bei 12.000 x g für 5 min. Einen kleinen Pellets sollte sichtbar sein. Fahren Sie schnell zum nächsten Schritt Wiederfreisetzung oder Dispersion des Geschosses zu vermeiden. Den Überstand ohne zu stören das Pellet zu entfernen.
- Zentrifugieren Sie die Rohre wieder mit der Kappe-Scharnier und Pellet nach außen um jede restliche Flüssigkeit auf den Boden des Rohres zu bringen. Ein kurzer Impuls genügt. Es sollte keine sichtbare Flüssigkeit verbleibenden in den Rohren.
- Fügen Sie ohne zu stören das Pellet, 40 µL Co Fällungsmittel Lösung. Lassen Sie das Rohr sitzen auf Eis für 5 min. Zentrifuge für 5 min, dann entfernen und entsorgen der Wäsche. 25 µL deionisiertes Wasser hinzufügen. Vortex jede Röhre für 5-10 s. Die Pellets sollten zerstreuen aber nicht im Wasser auflösen.
- Fügen Sie 1 mL Waschpuffer (vorab gekühlt mindestens 1 h bei-20° C) und 5 µL waschen Additiv. Wirbel, bis das Pellet vollständig dispergiert ist. Inkubieren Sie die Röhrchen bei-20 ° C für mindestens 30 min. Vortex für 20-30 s alle 10 min
Hinweis: Die Rohre können bei-20 ° C bis zu 1 Woche mit minimalen Proteinabbau oder Änderung gespeichert werden. - Zentrifugieren Sie die Rohre (mindestens) bei 12.000 × g für 5 min. vorsichtig entfernen und entsorgen den überstand. Ein weißes Granulat sollte sichtbar sein. Erlauben Sie das Pellet, Airdry für nicht mehr als 5 min (wenn das Pellet zu trocken, es wird schwierig ist, Aufschwemmen werden).
- Das Protein Pellet in 300 µL 8 M Harnstoff und 0,1 M Ammonium Bicarbonat aufzuwirbeln. Wirbel stark für 1 min. bestimmen die Konzentration des Proteins mit einer kolorimetrischen Assay.
9. in-Lösung Verdauung (Protokoll Nr. 2)
- Disulfidbrücken durch Zugabe von 5.1 µL einer 700 mM DTT wässrigen Lösung reduzieren (Endkonzentration 12,5 mM) auf die resuspendierte Proteine aus Schritt 8,4 und Inkubation bei 37 ° C für 30 min mit einem Thermoshaker. Alkylat Cystein Rückstände durch Zugabe von 20,3 µL einer wässrigen Lösung von 700 mM Iodoacetamide (Endkonzentration 40 mM) und Inkubation bei 25° C für 30 min in der Dunkelheit mit einem Thermoshaker.
- 990 µL 0.1 M Ammonium Bicarbonat zur Probe hinzufügen. Fügen Sie einem entsprechenden Volumen von Trypsin/Lys-C Mix (Enzymsubstrat: Verhältnis 1: 100 w/w). Inkubation bei 37° C über Nacht mit einem Thermoshaker.
10. Bereinigung Patrone (Protokoll Nr. 2)
- Befeuchten Sie eine Patrone mit 1 Spalte-Volumen (1 mL) von Methanol. Reinigen Sie die Kartusche mit 1 Spalte-Volumen (1 mL) von 80 % Acetonitril, HPLC-Grade Wasser und entsorgen Sie den Durchfluss. Equilibrate der Kartusche mit 4 Spalte-Wassermengen (4 mL) 0,1 % Ameisensäure/HPLC-Klasse und der durchströmten entsorgen.
- Ansäuern Sie Proben mit 90 µL 10 % Ameisensäure oder Wasser bis pH 2-3 (überprüfen Sie den pH-Wert mit einem pH-Indikator). Laden Sie die gesäuerte Proben und die Durchströmung zu sammeln. Laden Sie den Durchfluss (enthält Peptide nicht beibehalten). Waschen Sie die Kartusche mit 6 Spalte-Wassermenge (6 mL) 0,1 % Ameisensäure/HPLC-Klasse.
- Eluieren Sie Peptide aus der Patrone mit 1 Spalte-Bände (1 mL) 0,1 % Ameisensäure acid/50% Acetonitril, HPLC-Grade Wasser. Auf einem 1,5 mL Microcentrifuge Schlauch übertragen. Die Probe mit einem Vakuum Konzentrator (150 X-g, bei 160 mBar Vakuum) zu konzentrieren.
11. die Fraktionierung von isoelektrischen Fokussierung (Protokoll Nr. 2)
Hinweis: Peptide sind getrennt nach ihrem isoelektrischen Punkte mit einem off-Gel Plasmafraktionierer auf einem 13 cm Streifen mit einem pH-Bereich von 3 bis 10. Wir benutzten das folgende Protokoll vom Lieferanten (zusammengefasst):
- Bereiten Sie die folgenden Lösungen: Lösung (600 µL Glycerin Lösung, 60 µL OFFGEL-Puffer, 4,34 mL Reinstwasser) und B (1,776 mL der Lösung A) und 444 µL Reinstwasser.
- Montieren Sie die IPG-Streifen, Rahmen und Elektroden gemäß den Anweisungen des Herstellers.
- Die Probe mit 1,8 mL der Lösung b hinzufügen 40 µL Lösung B in jede Vertiefung aufzuwirbeln. Laden Sie 150 µL der Probe, in jede Vertiefung.
- Wählen Sie die Standardmethode für Peptide: OG12PE00 (OFFGEL-Standard-Methode für Peptide für die Verwendung mit einem 3100 OFFGEL Low Res Kit, pH 3 bis 10, 12-Well Frames. Warten Sie, bis diese Methode abgeschlossen (~ 20 h) gewesen ist. Die Fraktionen in ordentlich beschrifteten Röhrchen zu sammeln.
12. Sanierung Harvard Apparat Spalte Reverse C18 Post-IEF (Protokoll Nr. 2)
- Fügen Sie ein paar μL schrittweise in einer Zeit von 1 % TFA in deionisiertes Wasser, jede Fraktion zum Ansäuern der Probenmaterials. Prüfung mit pH-Papier, das der pH-Wert über 3 oder darunter liegt.
- Bereiten Sie die folgenden Lösungen: Lösung 1 (5 mL Acetonitril, 10 µL der Ameisensäure, 4,99 mL Reinstwasser) und 2 (0,5 mL Acetonitril, 10 µL der Ameisensäure, 9,49 mL Reinstwasser).
- Vornässen der Spin-Spalte mit 150 µL Lösung 1. Zentrifuge für 90 s 750 x g und entsorgen der durchströmten. Waschen Sie die Spin-Spalte mit 150 μL der Lösung 2. Zentrifuge für 90 s 750 x g und entsorgen der durchströmten.
- Passieren Sie den Bruch durch die Säule. Zentrifuge für 90 s 750 x g und entsorgen der durchströmten. Waschen Sie mit 150 μL der Lösung 2. Zentrifuge für 90 s 750 x g und entsorgen der durchströmten.
- Eluieren Sie den Bruch mit 50 μL der Lösung 1. Zentrifuge für 90 s bei 750 X g. Wiederholen Sie diese Schritte noch einmal.
- Trocken-Fraktion mit einem Vakuum Konzentrator (150 X g, Vakuum 160 mBar) und bei-80 ° C lagern
13. Auswertung der Daten Proteomic und Bioinformatik18
- Analysieren Sie Daten, die von einem Nano-LC-MS/MS mass Spektrometer mit Quantifizierung Software wie MaxQuant (Version 1.5.2.8) und die Andromeda-Suchmaschine.
- Stellen Sie false Discovery Rate (FDR) auf 1 % für Proteine und Peptide und eine Mindestlänge von 7 Aminosäuren. Legen Sie Enzym Spezifität als C-terminale zu Arg und Lys. 2 verpasste Spaltungen an Prolin Bindungen zu ermöglichen. Wählen Sie Carbamidomethylation Cystein als feste Modifikation und N-terminale Protein Acetylierung und Methionin Oxidation als Variable Änderungen.
- Die Daten mit statistischen Analyse-Software weiter zu analysieren. Führen Sie eine funktionale Anreicherung Analyse mit FunRich Software (www.funrich.org/). Führen Sie eine Ontologie Bereicherung Genanalyse mit DAVID Software (https://david.ncifcrf.gov/).
Ergebnisse
Ausgehend von peripheren mononukleären Blutzellen (PBMCs) durch differentielle Zentrifugation gewonnen, das Protokoll ermöglicht den Erhalt einer Population von CD14+ Monozyten mit einem veranlagten Reinheit von mehr als 98 % von Durchflusszytometrie (Abbildung 1). Diese Monozyten werden sekundär gegenüber verschiedenen Polarisationen (Abbildung 2) unterschieden. Wenn eine Fraktionierung auf Gel gewählt, die Migration auf die SDS-Page Gelen angepasst ist, die Anzahl der gewünschten Bänder zu erhalten, und die Bands sind herausgeschnitten (Abbildung 3). Die Verdauung erfolgt sekundär in den ausgeschnittenen Bands des Gels, dann die Peptide werden extrahiert. Die Peptide sind mit einer Nano-LC (Flüssigkeitschromatographie) analysiert-MS/MS mass Spectrometer. MS/MS-Spektren geben die Identität verschiedener Proteine entsprechend die Kommentierung der Spektren für bekannte Peptide (Abb. 4A) erhalten. Die Quantifizierung der Fülle eines Proteins errechnet sich dann im Zusammenhang mit der Menge der identifizierten Peptide aus dem Protein mit veröffentlichte Software und Datenbanken15,16. Dieses Protokoll mit der Verdauung-Gel gibt etwa 4000 identifizierte Proteine und der dynamische Bereich festgestellt wurde, um 5 logarithmischen Einheiten (Abbildung 4 b) zu decken. Analyse der differentiellen Expression dieser identifizierte Proteine kann verwendet werden, um festzustellen, die Bündelung von verschiedenen Polarisationen unter verschiedenen Sauerstoff-Umgebungen.
Mit dieser Methode erkennen wir auch Gruppen von Proteinen, die Up-reguliert werden bei einer niedrigen Sauerstoffkonzentration von 3 % (Abbildung 5, Tabelle 1). Zur Beurteilung der Effizienz der Verdauung, das ist nicht möglich, wenn eine in-Gel-Protokoll verwendet wird, haben wir vorgeschlagen, eine in-Lösung Verdauung-Methode, die menschliche Makrophagen (Abb. 6A) angepasst wurde. Mit dieser Methode können wir problemlos erhalten (nach in-Lösung Verdauung) Identifizierung von 3600 Proteine ohne Fraktionierung, was bedeutet, dass Fraktionierung mit IEF Willen vernünftig erhöhen Sie diesen Wert (Abb. 6 b).

Abbildung 1: Flow Cytometry Analyse von CD14 Ausdruck von PBMC vor dem Sortieren (links) und nach dem Sortieren (rechte Abbildung) zeigt die erhaltenen Reinheit nach dem magnetischen Beads Auswahl. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: Phasenkontrast-Bilder von differenzierten menschlichen Makrophagen zeigt die Heterogenität der erhaltenen Morphologien für zwei unterschiedliche Polarisationen. Maßstabsleiste stellt 50 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Darstellung der Coomassie Blau gefärbt Gel zeigt die verschiedenen Bands, die herausgeschnitten werden [hier, 6 Bands in M(Ø) Makrophagen] für 5 Polarisationen von Makrophagen einer sauerstoffarmen Umgebung ausgesetzt. IC = Immunkomplexe, DXM = Dexamethason. Bitte klicken Sie hier für eine größere Version dieser Figur.
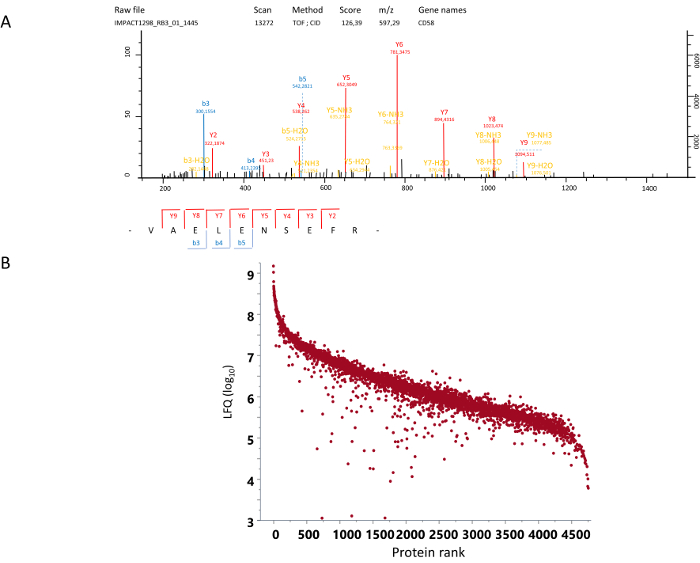
Abbildung 4: MS/MS-Spektrum und Quantifizierung. (A) ein Beispiel für eine MS/MS-Spektrum. Hier wird das CID (Kollision-induzierte Dissoziation) Spektrum eines Peptids fand bei m/Z 597.29 auf dem MS-Spektrum mit einer elektrischen Ladung von + 2. Die entsprechende Sequenz wurde aus diesem Spektrum als Val-Ala-Glu-Leu-Glu-Asn-Ser-Glu-Phe-Arg aus dem Protein CD58 ermittelt. (B) Rang bestellt markierungsfreie Quantifizierung aller identifizierten Proteine (log10 LFQ). Bitte klicken Sie hier für eine größere Version dieser Figur.
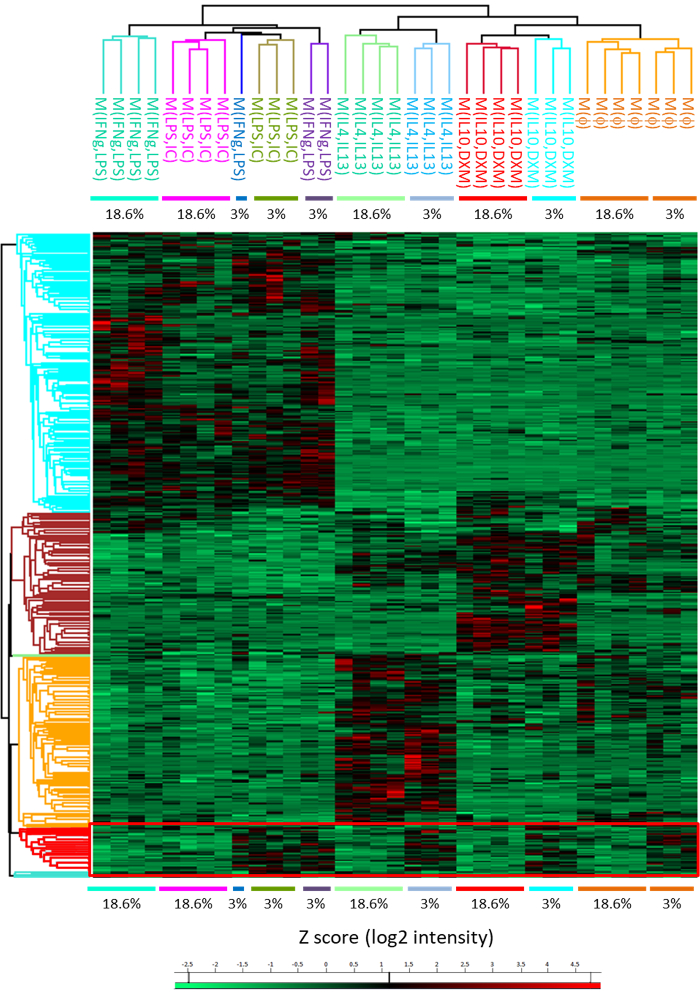
Abbildung 5: Heatmap repräsentieren die hierarchische Clusterung von alle Polarisation Staaten mit differenziell ausgedrückt Proteine. Analyse zeigt eine Gruppe von Proteinen in alle Polarisationen im 3 % O2 Zustand (rotes Rechteck) überexprimiert. Die Farbskala ist Z-Scores (log2 Intensität). Jede Zeile ist ein Protein und jede Spalte ist ein Beispiel. Diese Zahl stammt aus einer früheren Veröffentlichung14. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 6: SDS-PAGE und Chromatogramm. (A) Silber-gebeizt SDS-PAGE Gelen mit Protein aus Zelle Lysis und nach-Lösung Verdauung zeigt das Fehlen des Abbaus während lyse und Effizienz der Verdauung. (B) nach der Verdauung in Lösung ohne Fraktionierung gewonnenen Chromatogramm. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Cluster | proteinID | Protein-Namen | Gen Namen | Peptide | Rasierer + einzigartige Peptide | Einzigartige Peptide | Protein-IDs | ||
| Rot | P0DMV9 | Heat shock 70 kDa-Protein-1 b | HSPA1A | 40 | 37 | 4 | P0DMV9; P0DMV8; A8K5I0; Q59EJ3; B4DNT8; B4DWK5; B4DFN9; B4DI39; B4E1S9; B4DVU9; V9GZ37; B3KTT5; B4DNX1; B4E1T6; B4DNV4; Q9UQC1 | ||
| Rot | P54709 | Natrium/Kalium-Transport-ATPase Untereinheit Beta-3 | ATP1B3 | 8 | 8 | 8 | P54709; D3DNF9; C9JXZ1; C9JA36; H7C547; F8WBY4; Q58I18 | ||
| Rot | O00462 | Beta-mannosidase | MANBA | 14 | 13 | 13 | O00462; A7LFP5; A8K6D3; E9PFW2; B4DT18; Q59EG5 | ||
| Rot | Q8NAS7 | NADH Dehydrogenase [Ubiquinon] Eisen-Schwefel Protein 7, mitochondriale | NDUFS7 | 4 | 4 | 4 | Q8NAS7; Q6ZQU6; F5H5N1; B7Z1U1; A8K0V6; Q7LD69; F5GXJ1; O75251; B7Z4P1; Q9H3K5; A0A087WXF6; A0A087WTI3; Q6ZS38 | ||
| Rot | Q8WWQ0 | PH-Interaktion protein | PHIP | 5 | 5 | 4 | Q8WWQ0; Q9NWP3 | ||
| Rot | E5RHK8 | Dynamin-3 | DNM3 | 11 | 2 | 2 | E5RHK8; Q9UQ16; B3KPF2; E5RIK2 | ||
| Rot | A0A024QZ64 | Fruktose-Bisphosphate Aldolase C | ALDOC | 18 | 14 | 7 | A0A024QZ64; P09972; B7Z1Y2; B7Z3K9; B7Z1N6; B7Z3K7; A8MVZ9; J3KSV6; J3QKP5; B7Z1L5; C9J8F3; K7EKH5; J3QKK1; B7Z1H6 | ||
| Rot | O75489 | NADH Dehydrogenase [Ubiquinon] Eisen-Schwefel Protein 3, mitochondriale | NDUFS3 | 12 | 12 | 12 | O75489; Q9UF24; Q53FM7; E9PS48; E9PKL8; G3V194 | ||
| Rot | P21912 | Succinat-Dehydrogenase [Ubiquinon] Eisen-Schwefel Untereinheit, mitochondriale | SDHB | 11 | 11 | 11 | P21912; A0A087WXX8; A0A087WWT1 | ||
| Rot | A0A024R1Y7 | GH3 Domäne-haltige protein | LGP1; GHDC | 7 | 7 | 7 | A0A024R1Y7; Q8N2G8; B3KVB0; K7ESN3; K7EJT7; K7EQ41; K7EL54 | ||
| Rot | E5KRK5 | NADH-Ubiquinon Oxidoreductase 75 kDa Untereinheit, mitochondriale | NDUFS1 | 29 | 29 | 29 | E5KRK5; P28331; B4DJ81; Q9P1A0; C9JPQ5; F8WDL5 | ||
| Rot | A0A024QZ30 | Succinat-Dehydrogenase [Ubiquinon] Flavoprotein Untereinheit, mitochondriale | SDHA | 17 | 17 | 17 | A0A024QZ30; P31040; D6RFM5; B3KT34; A0A087X1I3; Q0QF12; B4DYN5; B3KYA5; B4DNB2; H0Y8X1; B7Z6J5 | ||
| Rot | A0A024R2F9 | Transmembranen Protein 43 | TMEM43 | 16 | 16 | 16 | A0A024R2F9; Q9BTV4; Q8TEP9; V9GY05 | ||
| Rot | A0A024R5K3 | NADH Dehydrogenase [Ubiquinon] Eisen-Schwefel Protein 8, mitochondriale | NDUFS8 | 5 | 5 | 5 | A0A024R5K3; E9PPW7; O00217; E9PN51; F8W9K7; E9PKH6; B4DYI3; Q53G17 | ||
| Rot | O76003 | Glutaredoxin-3 | GLRX3 | 13 | 13 | 13 | O76003 | ||
| Rot | Q1HBJ4 | Mitogen-activated Proteinkinase; Mitogen-activated Proteinkinase 1 | MAPK1 | 26 | 26 | 21 | Q1HBJ4; P28482; B4DHN0; Q499G7; K7ELV1; K7EN18; B4E104; P31152; Q16659 | ||
| Rot | Q13151 | Heterogene nuclear Ribonucleoprotein A0 | HNRNPA0 | 8 | 8 | 8 | Q13151 | ||
| Rot | V9HWN7 | Fruktose-Bisphosphate Aldolase A | ALDOA | 36 | 36 | 14 | V9HWN7; P04075; J3KPS3; H3BQN4; H3BUH7; H3BR04; H3BMQ8; H3BU78; H3BR68; A4UCS9; A4UCT0 | ||
| Rot | B4DVJ0 | Glukose-6-Phosphat-isomerase | GPI | 32 | 32 | 23 | B4DVJ0; P06744; B4DE36; A0A0A0MTS2; K7EQ48; K7EP41; K7EPY4; K7ELR7; K7ERC6; K7ENA0; K7ESF4; K7EIL4; K7ERK8; Q59F85 | ||
| Rot | V9HWB9 | L-Lactat-Dehydrogenase; L-Lactat-Dehydrogenase eine Kette | LDHA | 36 | 36 | 34 | V9HWB9; P00338; B4DJI1; F5GXY2; F5GYU2; F5GXH2; F5H5J4; F5H6W8; F5GZQ4; F5H8H6; F5GXC7; F5GWW2; F5GXU1; A0A087WUM2; Q96L19; Q6ZMR3 | ||
| Rot | P13674 | Prolyl-4-Hydroxylase Untereinheit Alpha-1 | P4HA1 | 26 | 26 | 26 | P13674; Q5VSQ6 | ||
| Rot | Q6FHV6 | Gamma-Enolase; Enolase | ENO2 | 18 | 15 | 14 | Q6FHV6; P09104; A8K3B0; F5H0C8; F5H1C3; U3KQP4; U3KQQ1; Q9NPL4 | ||
| Rot | Q99798 | Aconitate Hydratase, mitochondriale | ACO2 | 40 | 40 | 40 | Q99798; B4DZ08; B2RBW5; A2A274; B4DJW1; B4DLY4; Q71UF1; B4DEC3; B4DW08; O75944 | ||
| Rot | P17858 | ATP-abhängige 6-Phosphofructokinase, Leber-Typ | PFKL | 32 | 29 | 28 | P17858; Q7L2M7; Q9BSP4; B3KNQ7; B4E108; Q59GI2; F8WEU2; Q7Z3R9; Q6MZK4 | ||
| Rot | A0A024R872 | Niban-Like Protein 1 | FAM129B | 32 | 32 | 32 | A0A024R872; Q96TA1; Q9H8K1; Q2YD88; Q9H6L6 | ||
| Rot | A0A024RC61 | Aminopeptidase N | ANPEP | 58 | 58 | 25 | A0A024RC61; P15144; Q59E93; B4DV63; B4DP01; B4DP96; H0YKT6; H0YLZ8; Q71E46; H0YMC1; Q8IVL7 | ||
| Rot | V9HWF4 | Phosphoglycerate Kinase; Phosphoglycerate Kinase 1 | PGK1 | 41 | 41 | 35 | V9HWF4; P00558; B4E1H9; B4DHM5; B4DHB3; B4DWQ3; Q16444 | ||
| Rot | Q12882 | Dihydropyrimidine-Dehydrogenase [NADP(+)] | DPYD | 44 | 44 | 44 | Q12882; B4DML1 | ||
| Rot | B4DEQ0 | Electron Transfer Flavoprotein Ubiquinon Oxidoreductase, mitochondriale | ETFDH | 9 | 9 | 9 | B4DEQ0; Q547S8; A7UNU5; Q16134; D6RAD5 | ||
| Rot | D9UAX9 | MHC Klasse I antigen | HLA-B | 13 | 3 | 2 | D9UAX9 | ||
| Rot | V9HWK1 | Triosephosphate isomerase | TPI1 | 29 | 29 | 17 | V9HWK1; P60174; Q53HE2; B4DUI5; U3KPZ0; U3KQF3; U3KPS5 | ||
| Rot | Q96HE7 | ERO1-Like Protein alpha | ERO1L | 28 | 28 | 28 | Q96HE7; G3V3E6; G3V5B3; G3V2H0; G3V503; Q5TAE8; B2RD00; Q86YB8 | ||
| Rot | P14868 | Aspartate--cytoplasmatischen tRNA-ligase | DARS | 29 | 29 | 2 | P14868; Q53T60; Q53R85; H7BZ35; H7C278 | ||
| Rot | P36871 | Phosphoglucomutase-1 | PGM1 | 24 | 24 | 5 | P36871; B4DFP1; Q9H1D2 | ||
Tabelle 1: Liste der stark exprimierten Proteine für menschliche Makrophagen üblich, jede Polarisierung unter sauerstoffarmen Spannung.
Diskussion
Da Proteomics ein mächtiges Werkzeug ist, um den Ausdruck verschiedener Proteine aus einer ganzen Zelle oder subzelluläre Kompartimente zu studieren, wurde Optimierung des Protokolls Zelle Lysis und Verdauung der Proteine durch eine Reihe von Studien behoben. Es gibt drei Hauptklassen von Methoden, die im Gel Verdauung (Verdauung von Proteinen in Polyacrylamid Gelmatrix)17, Verdauung in Lösung18 und Filter-gestützte Probe Vorbereitung19enthalten. Diese letzte Methode zunächst als universelle, beschrieben wurde berichtet, niedrige Reproduzierbarkeit und möglichen Verlust von Proteinen auf die Filter-20auszustellen. Im Gel Verdauung ist eine robuste Methode, die Zeit- und nachteilig sein kann, insofern als Bewertung der Effizienz der Verdauung ist nicht einfach, wenn möglich. In Lösung Verdauung erfordert die Reinigung der Proben nach der Verdauung und IEF aber bietet diese Möglichkeit. Wenn diese beiden Methoden zwischen der gleichen Probe verglichen werden, ergibt sich in Lösung Verdauung mit IEF Fraktionierung Protokoll eine höhere Anzahl von identifizierten Proteine (mit der gleichen Anzahl von Brüchen) als in-Verdauung21Gel.
Trotz dieses Vorteils ist es notwendig, die möglichen Proteinabbau während in-Lösung lyse durch intrazelluläre Proteasen (insbesondere myeloische Zellen) zu betrachten. Es ist auch wichtig zu bedenken, dass diese Techniken auf Proteinverdauung basieren und nur in der Lage, Proteine präsentieren Trypsin spezifische Spaltstellen zu analysieren. Es ist möglich, einen Top-Down-Proteomik-Ansatz zu verwenden, der diese Einschränkung Verdauung entlastet, aber fügt Daten Analyseschritte und Bioinformatik Ressourcen22. Die Solubilisierung von Proteinen aus verschiedenen zellulären Kompartimenten kann auch schwer zu bekommen, vor allem von Plasmamembranen, führt zu einer unkontrollierten Probenahme von zellulären Proteom sein. Um mit einem Massenspektrometer Nano-LC-MS/MS-Analyse der Proben fortzufahren, ist es wichtig, eine ausreichende Menge von Peptiden, die zu erhalten, von dem Massenspektrometer verwendet abhängig können (in der Regel ab Gesamt-Protein sollte mindestens 1 µg für eine Bedingung und Es wird angedeutet, um diese Menge entsprechend der Anzahl der Bruch mit IEF verwendet zu erhöhen). Diese Einschränkung möglicherweise ein Nachteil, wenn die Zell-Population untersucht knapp ist, ist, der Proteomik von genomischen Techniken unterscheidet in der Verstärkung des Rohstoffs möglich ist.
Auch nach der bahnbrechenden Werke des Richer und Kollegen23 und Packer und Fuehr24wurde die Bedeutung von Sauerstoff in Zellkulturen nicht ausreichend erkannt. Wir wissen jetzt, dass der Kultivierung von Zellen unter niedrigen Sauerstoffkonzentrationen Adhäsion, Lebensdauer und Sparte begünstigt. Es ist anerkannt, dass dies von größter Bedeutung in der Stammzell-Forschung25. Die wichtigste technische Frage bei Zellkulturen unter kontrollierten Sauerstoffbedingungen bezieht sich auf Wartung der gewünschten Sauerstoffkonzentration während des gesamten Experiments. Dies erfordert Vorinkubation von allen Medien, Freisetzung von gelöstem Sauerstoff verhindern und Einsatz von hypoxischen Arbeitsstationen erlauben die Manipulation von Zellen unter sauerstoffarmen (Verarbeitung Kammer mit Glove-Box) und vorübergehende Exposition gegenüber hohen Sauerstoffbedingungen verhindern .
Das beschriebene Protokoll wurde verwendet, um erhalten die molekularen Signaturen von verschiedenen Polarisationen der menschliche Monocyte abgeleiteten Makrophagen und Studie über die Auswirkungen von Sauerstoff Modulation auf diese Signaturen. Diese Studie hat Einblick auf die Beschreibung dieser Polarisationen und hat einige funktionelle Konsequenzen ergeben. Zum Beispiel fanden wir, dass viele Proteine, die in Efferocytosis von einer sauerstoffarmen Umgebung moduliert wurden. Dieser Proteomic Ansatz, basierend auf dem beschriebenen Protokoll bietet die Möglichkeit zu erkunden, wie Umweltparameter Makrophagen Funktionen ändern und wie diese Signale verwendet werden können, um neue therapeutische Ansätze14zu entwerfen.
In dieser Arbeit beschriebene Proteomik-Ansatz ist komplementär zur genomischen Ansätze, die in den letzten Jahren auf dem Gebiet der menschlichen Makrophagen Polarisation Studien verwendet wurden. Proteomics bieten den Vorteil der Protein Quantifizierung, die einen anderen Ausdruck als ihre entsprechenden mRNAs durch post-translationalen Modifikationen zu präsentieren und zur Entdeckung neuer Biomarker führen kann. Trotz dieses Vorteils ist Proteomic Daten in der Regel schwer zu interpretieren, teilweise wegen der hohen Empfindlichkeit der Massenspektrometrie, was zu sehr komplexen MS Spektren und falsch positiven Nachweis von Peptiden. Vor kurzem hat Analysesoftware Effizienz gewonnen, um dies zu verhindern. Auch wenn es eine veränderte Situation, Proteomics auch Gesichter niedriger Reproduzierbarkeit als Genomics26 und ist verbunden mit Validierungsschritte mit anderen Techniken (Durchflusszytometrie, Immunoblotting), quantitative Veränderungen des Proteins zu bestätigen Ausdruck-Ebenen.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
AM la Ligue Nationale Contre le Cancer und la Fondation ARC pour la recherche Sur le Cancer vom Young Group Leader Programm (ATIP/Avenir Inserm-CNRS) finanziert. Wir bedanken uns bei Mariette Matondo von Massenspektrometrie für Biologie-Plattform (UTECHS MSBIO, Institut Pasteur, Paris). Wir danken Lauren Anderson für ihre Lektüre des Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| Hypoxia Working Station | Oxford Optronix | Hypoxylab | |
| C6 Flow cytometer | BD | Accuri C6 | |
| Urea | Agilent Technologies | 5188-6435 | |
| Formic acid (FA) | ARISTAR | 450122M | |
| R-250 Coomassie blue | Biorad | 1,610,436 | |
| Lipopolysaccharide, E.Coli (LPS) | Calbiochem | 437627 | |
| 2D clean-up kit | GE Healthcare | 80-6484-51 | |
| RPMI 1640 medium, glutamax supplement | Gibco | 61870044 | |
| HEPES 1 M | Gibco | 15630-080 | |
| MEM Non-Essential Amino Acids (NEAA) Solution 100X | Gibco | 11140-035 | |
| Phosphate Buffered Saline (PBS) 1X | Gibco | 14190-094 | |
| Harvard Apparatus column Reverse C18 micro spin column | Harvard Apparatus | 74-4601 | |
| EDTA 0.5 M, pH 8.0 | Invitrogen | AM9260G | |
| NuPAGE Bis-Tris 4-12% | Life Technologies SAS | NP0321 BOX | |
| CD14 Microbeads human | Miltenyi Biotec | 130-050-201 | |
| MACS separation column LS | Miltenyi Biotec | 130-042-401 | |
| Macrophage colony-stimulating factor (M-CSF) | Miltenyi Biotec | 130-096-485 | |
| Interleukin 4 (IL4) | Miltenyi Biotec | 130-093-917 | |
| Interleukin 13 (IL13) | Miltenyi Biotec | 130-112-410 | |
| Interferon gamma (INFγ) | Miltenyi Biotec | 130-096-482 | |
| CD14-FITC (clone TÜK4) | Miltenyi Biotec | 130-080-701 | |
| MACSmix Tube Rotator | Miltenyi Biotec | 130-090-753 | |
| Trifluoroacetic Acid (TFA) | Pierce | 28904 | |
| Trypsin/Lys-C Mix | PROMEGA | V5073 | |
| Complete Mini, EDTA-free Protease Inhibitor cocktail | Roche | 11836170001 | |
| Density Gradient Solution (Histopaque 1077) | Sigma Aldrich | 10771-100ML | |
| Accumax | Sigma Aldrich | A7089-100ML | |
| Human Serum from human male AB plasma (SAB) | Sigma Aldrich | H4522-100ML | |
| Bovine Serum Albumin (BSA) solution 30% | Sigma Aldrich | A9576-50ML | |
| Trisma-base | Sigma Aldrich | T1503 | |
| Glycerol | Sigma Aldrich | 49767 | |
| β-Mercaptoethanol | Sigma Aldrich | M3148 | |
| Bromophenol blue | Sigma Aldrich | 114405 | |
| Sodium Dodecyl Sulfate (SDS) 20% | Sigma Aldrich | 5030 | |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | |
| Acetonitrile | Sigma Aldrich | 34888 | |
| Dithiothreitol | Sigma Aldrich | 43819 | |
| Iodoacetamide | Sigma Aldrich | 57670 | |
| Thiourea | Sigma Aldrich | T8656 | |
| CHAPS | Sigma Aldrich | C9426 | |
| Micro BCA Assay Kit | ThermoFisher | 23235 | |
| 5 mL sterile plastic pipette | VWR | 612-1685 | |
| Thermomixer C Eppendorf | VWR | 460-0223 | |
| Sep-Pak tC18 reverse phase cartridges, 100 mg | Waters | WAT036820 |
Referenzen
- Okabe, Y., Medzhitov, R. Tissue biology perspective on macrophages. Nature Immunology. 17 (1), 9-17 (2016).
- Sica, A., Mantovani, A. Macrophage plasticity and polarization: in vivo veritas. The Journal of Clinical Investigation. 122 (3), 787-795 (2012).
- Murray, P. J. Macrophage Polarization. Annual Review of Physiology. 79, 541-566 (2017).
- Chinetti-Gbaguidi, G., Staels, B. Macrophage polarization in metabolic disorders: functions and regulation. Current Opinion in Lipidology. 22 (5), 365-372 (2011).
- Chow, A., Brown, B. D., Merad, M. Studying the mononuclear phagocyte system in the molecular age. Nature Reviews Immunology. 11 (11), 788-798 (2011).
- Mosser, D. M., Edwards, J. P. Exploring the full spectrum of macrophage activation. Nature Reviews Immunology. 8 (12), 958-969 (2008).
- Murray, P. J., Wynn, T. A. Protective and pathogenic functions of macrophage subsets. Nature Reviews Immunology. 11 (11), 723-737 (2011).
- Ginhoux, F., Schultze, J. L., Murray, P. J., Ochando, J., Biswas, S. K. New insights into the multidimensional concept of macrophage ontogeny, activation and function. Nature Immunology. 17 (1), 34-40 (2016).
- Xue, J., et al. Transcriptome-based network analysis reveals a spectrum model of human macrophage activation. Immunity. 40 (2), 274-288 (2014).
- Csete, M. Oxygen in the cultivation of stem cells. Annals of the New York Academy of Sciences. 1049, 1-8 (2005).
- Johansson, A., et al. Functional, morphological, and phenotypical differences between rat alveolar and interstitial macrophages. American Journal of Respiratory Cell and Molecular Biology. 16 (5), 582-588 (1997).
- Pfau, J. C., Schneider, J. C., Archer, A. J., Sentissi, J., Leyva, F. J., Cramton, J. Environmental oxygen tension affects phenotype in cultured bone marrow-derived macrophages. American Journal of Physiology. Lung Cellular and Molecular Physiology. 286 (2), L354-L362 (2004).
- Grodzki, A. C. G., Giulivi, C., Lein, P. J. Oxygen tension modulates differentiation and primary macrophage functions in the human monocytic THP-1 cell line. PLoS One. 8 (1), e54926(2013).
- Court, M., Petre, G., Atifi, M. E., Millet, A. Proteomic Signature Reveals Modulation of Human Macrophage Polarization and Functions Under Differing Environmental Oxygen Conditions. Molecular & Cellular Proteomics. 16 (12), 2153-2168 (2017).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Cox, J., Neuhauser, N., Michalski, A., Scheltema, R. A., Olsen, J. V., Mann, M. Andromeda: a peptide search engine integrated into the MaxQuant environment. Journal of Proteome Research. 10 (4), 1794-1805 (2011).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols. 1 (6), 2856-2860 (2006).
- Court, M., et al. Toward a standardized urine proteome analysis methodology. Proteomics. 11 (6), 1160-1171 (2011).
- Wiśniewski, J. R., Zougman, A., Nagaraj, N., Mann, M. Universal sample preparation method for proteome analysis. Nature Methods. 6 (5), 359-362 (2009).
- Liebler, D. C., Ham, A. -J. L. Spin filter-based sample preparation for shotgun proteomics. Nature Methods. 6 (11), 785-786 (2009).
- Hubner, N. C., Ren, S., Mann, M. Peptide separation with immobilized pI strips is an attractive alternative to in-gel protein digestion for proteome analysis. Proteomics. 8 (23-24), 4862-4872 (2008).
- Park, J., et al. Informed-Proteomics: open-source software package for top-down proteomics. Nature Methods. 14 (9), 909-914 (2017).
- Richter, A., Sanford, K. K., Evans, V. J. Influence of oxygen and culture media on plating efficiency of some mammalian tissue cells. Journal of the National Cancer Institute. 49 (6), 1705-1712 (1972).
- Packer, L., Fuehr, K. Low oxygen concentration extends the lifespan of cultured human diploid cells. Nature. 267 (5610), 423-425 (1977).
- Lengner, C. J., et al. Derivation of pre-X inactivation human embryonic stem cells under physiological oxygen concentrations. Cell. 141 (5), 872-883 (2010).
- Sidoli, S., Kulej, K., Garcia, B. A. Why proteomics is not the new genomics and the future of mass spectrometry in cell biology. Journal of Cell Biology. 216 (1), 21-24 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten