Method Article
Analyse protéomique de polarisation des macrophages humains dans un environnement pauvre en oxygène
Dans cet article
Résumé
Nous présentons un protocole visant à obtenir les signatures de protéomique des macrophages humains et cela s’applique à la détermination de l’impact d’un environnement pauvre en oxygène sur la polarisation de macrophages.
Résumé
Les macrophages sont des cellules immunitaires innées impliqués dans un certain nombre de fonctions physiologiques allant de réactions aux agents pathogènes infectieux à l’homéostasie tissulaire. Les différentes fonctions de ces cellules sont liées à leurs États d’activation, qui est aussi appelé polarisation. La description moléculaire précise de ces polarisations différentes est une priorité dans le domaine de la biologie du macrophage. Il est actuellement reconnu qu’une approche multidimensionnelle est nécessaire pour décrire comment la polarisation est contrôlée par des signaux environnementaux. Dans ce rapport, nous décrivons un protocole visant à obtenir la signature de la protéomique des polarisations différentes dans les macrophages humains. Ce protocole est basé sur une quantification exempte d’étiquette d’expression de protéine macrophage provenant de fractionnés en gel et le contenu de lyse cellulaire Lys C/trypsine-digéré. Nous fournissons également un protocole basé sur la solution en digestion et isoélectrique mise au point de fractionnement à utiliser comme solution de rechange. Parce que la concentration en oxygène est un paramètre environnemental pertinent dans les tissus, nous permet de découvrir la composition de l’atmosphère comment ce protocole ou un environnement pauvre en oxygène influe sur la classification de la polarisation du macrophage.
Introduction
Les macrophages sont des cellules immunitaires innées impliqués dans un certain nombre de fonctions physiologiques allant de réactions aux agents pathogènes infectieux à l’homéostasie tissulaire, y compris l’enlèvement de cellules apoptotiques et remodelage de la matrice extracellulaire1. Ces cellules sont caractérisées par une forte plasticité phénotypique2 qui se traduit par un nombreux États d’activation possible, qui sont aussi appelés les polarisations. La description moléculaire précise de ces polarisations différentes est une priorité dans le domaine des macrophages biologie3. Il a été proposé de classer ces polarisations à l’aide de la dichotomie de M1/M2 ce qu’on appelle, dans laquelle M1 représente pro-inflammatoires et M2 macrophages anti-inflammatoires. Ce modèle s’intègre bien dans diverses situations pathologiques comme les infections aiguës, allergies et obésité4. Toutefois, dans les tissus chroniquement enflammés et le cancer, il a été démontré que cette classification est incapable de comprendre le large répertoire phénotypique que macrophages présent dans certains environnements cellulaires5,,6, 7. le consensus actuel est que polarisation macrophage est mieux décrit en utilisant un modèle multidimensionnel pour intégrer des signaux micro-environnementales spécifiques8. Cette conclusion a été confirmée par analyse transcriptomique des macrophages humains montrant que le modèle M1/M2 est inefficace en décrivant les polarisations obtenu9.
L’étude présentée a pour but de fournir un protocole afin d’obtenir les signatures de protéomique de polarisations différentes dans les macrophages humains. Nous décrivons comment différencier les macrophages humains dans des environnements de divers niveaux d’oxygène et d’obtenir des peptides du protéome macrophage ensemble pour effectuer une quantification exempte d’étiquette. Cette quantification permet la comparaison des niveaux d’expression de diverses protéines. Recherche sur les cellules souches a révélé l’importance de l’oxygène comme un paramètre key environmental10, nous cherchons à comprendre comment ce paramètre tissu peut influencer polarisation macrophages chez les humains. La pression partielle d’oxygène a été trouvée à intervalle de 3 à 20 % (de la pression atmosphérique totale) dans le corps humain, où 20 % correspond à peu près à ce qui est couramment utilisé dans un incubateur de culture cellulaire (la valeur exacte est d’environ 18,6 % tout en prenant la présence d’eau en compte).
Travaux antérieurs ont montré qu’alvéolaire diffèrent des macrophages interstitielles de fonctionnelles et morphologique point de vues11 et que ces différences sont probablement partiellement en raison des niveaux d’oxygène différents auxquels ils sont exposés12. En outre, les dérivés de la moelle osseuse montrent une capacité accrue de phagocyter les bactéries lorsqu’ils sont exposés à un environnement de faible teneur en oxygène12. L’effet inverse a été trouvé pour les macrophages humains dissociés THP113, mais ces résultats appuient l’idée que l’oxygène est un organisme de réglementation de la biologie du macrophage et qu’il est nécessaire de préciser ce rôle au niveau moléculaire dans les macrophages humains. Dans une étude précédente, nous avons appliqué une approche protéomique pour résoudre ces problèmes. En mesurant les niveaux d’expression pour des milliers de protéines en même temps, nous avons mis en évidence l’impact de l’oxygène sur la polarisation ainsi qu’une liste de nouveaux marqueurs moléculaires. Nous avons également pu faire le lien entre ces résultats à certaines fonctions de macrophages. Notamment, nous avons constaté que le taux de la phagocytose des cellules apoptotiques a augmenté dans les macrophages IL4/IL13-polarisé, qui était liée à la stimulation de l’expression de ALOX15 tel que révélé par l' analyse de protéomique14. Dans la présente étude, nous décrivons comment effectuer une telle analyse.
Protocole
Des échantillons de sang humain (LRSC) provenant de donneurs sains, dépersonnalisées proviennent de l’EFS (Français National Blood Service) dans le cadre d’un protocole autorisé (CODECOH DC-2018-3114). Donateurs ont donné le consentement signé pour l’utilisation du sang.
1. médias et préparation du tampon
- Préparer le milieu de macrophage [RPMI glutamax + 10 mM HEPES + 1 x non essentiels des acides aminés (NEAA)] et elle chauffé à 37 ° C.
- Préparer le milieu macrophage + 10 % de sérum humain du plasma AB (SAB), filtrer (filtre de 0,22 µm), puis le réchauffer à 37 ° C (dénommé moyen de macrophage + 10 % SAB ci-après).
- Préparer le tampon tri [1 x en solution saline tamponnée au phosphate (PBS) + 0,5 % d’albumine sérique bovine (BSA) + acide éthylènediaminetétraacétique de 2 mM (EDTA)], la filtrer (filtre de 0,22 µm) et maintenir à 4 ° C.
2. isolement des cellules mononucléaires de sang périphérique (PBMC) de déleucocytation système chambre (LRSC)
- Mettre 15 mL de solution de séparation densité cellulaire dégradé (voir la Table des matières) dans 50 mL de centrifugation tube donc il peut réchauffer à température ambiante (RT) avant de recevoir le LRSC.
Remarque : La densité dépend de la température. Que ce produit est stocké à 4 ° C, cette étape doit être faite à l’avance donc il peut équilibrer RT. - Vider la LRSC dans un tube de centrifugation de 50 mL, ajoutez à 50 mL de PBS 1 x et mélangez. Très lentement, ajouter 25 mL du mélange préparé au cours de l’étape 2.2 sur le dessus de 15 mL de solution de gradient de densité réchauffée au cours de l’étape 2.1.
Remarque : Veillez à ne pas mélanger les phases au cours de cette étape. Le sang doit être ajouté à la solution de gradient de densité sans toute perturbation de cette phase. - Centrifuger les deux tubes à centrifugation pendant 25 min à 700 x g sans pauses.
Remarque : À la fin de la centrifugation en gradient de densité, les couches de bas en haut sont : les érythrocytes et granulocytes formant le pellet, la phase de solution de gradient de densité, la couche de PBMC et le plasma. - Avec une pipette, passer par la phase plasma sans aspirer il et recueillir la couche PBMC dans un nouveau tube de centrifugation de 50 mL. Ajoutez à 50 mL de solution 1 PBS x pour les PBMC comme une étape de lavage et centrifuger pendant 10 min à 300 x g.
- Aspirer le surnageant et resuspendre le culot dans 40 mL de milieu de macrophages.
3. magnétique d’étiquetage et de l’isolement de CD14+ cellules (Monocytes)
- Compter les PBMC dans une chambre de Malassez. Retirer le montant de PBMC nécessaires à la conduite de l’expérience (généralement de 100 à 300 x 106 cellules), les placer dans un tube de centrifugation et centrifuger pendant 10 min à 300 x g.
- Aspirer le surnageant et resuspendre le culot dans 80 µL du tampon tri préparé au cours de l’étape 1.3 par 107 PBMC. ajouter 20 µL de CD14 microbeads par 107 Mix PBMC. puits et incuber pendant 15 min à 4 ° C sous agitation constante.
- Ajouter 1 mL de tampon par 107 PBMC de tri comme une étape de lavage et centrifuger pendant 10 min à 300 x g. aspirer le surnageant et resuspendre le culot dans 500 µL de tampon par 108 PBMC de tri.
- Placez une colonne dans le champ magnétique du séparateur. Préparer la colonne en les rinçant avec 3 mL de tampon de tri.
- Appliquer la suspension de cellules dans la colonne. La colonne dépend du nombre de cellules d’isolement (ici, les colonnes de LS pour jusqu'à 109 PBMC sont utilisées). Prélever des cellules intermédiaires contenant sans étiquette.
NOTE : À partir de cette étape, tous les tubes (sélections négatives et positives) sont conservées pour vérifier plus tard des différentes étapes par cytométrie en flux. - Laver la colonne avec 3 x 3 mL de tampon de tri. Prélever des cellules non marquées en passant par le même tube de l’étape 3.12. Effectuer les étapes de lavage en ajoutant tampon tri, veillez à ne pas faire sécher votre colonne. Placer un tube de prélèvement au titre de la colonne et retirez-la du séparateur.
- Pipette 5 mL de tampon de tri dans la colonne. Rincer immédiatement les cellules magnétiquement étiquetées en appuyant fermement sur le piston dans la colonne. Pour augmenter la pureté de la CD14+ cellules, la fraction d’élution est enrichie sur une seconde colonne.
- Répétez les étapes 3,4 à 3,7 avec une nouvelle colonne.
4. électrodéposition de Monocytes
- Compter les monocytes dans une chambre de Malassez. Vérifier la pureté de la CD14+ cellules par cytométrie en flux. Retirer le montant des monocytes nécessaires pour l’expérience et les placer dans un tube de centrifugation.
- Centrifuger pendant 10 min à 300 x g. aspirer le surnageant et resuspendre le culot de monocytes dans le milieu du macrophage. Les cellules de la plaque et les laisser s’installer pour 50 min à 1 h. aspirer le milieu et remplacez-le par le moyen de macrophage + 10 % SAB + 25 ng/mL macrophage colony stimulant facteur (M-CSF) pour induire la différenciation.
5. polarisation des Macrophages au jour 6
- Aspirez le milieu. Remplacez-la par moyen de macrophage + 10 % SAB avec divers stimuli. Par exemple, ajoutez 10 gamma d’interféron ng/mL (INFγ) ou 20 ng/mL interleukine 4 (IL4) + 1 lipopolysaccharide (LPS) pour obtenir M1 polarisation, de ng/mL et 20 ng/mL interleukine 13 (IL13) pour polarisation M2.
Remarque : La stimulation peut être effectuée entre 24 et 48 heures avant de procéder à d’autres tests. - Récolte des cellules à l’aide d’une solution de décollement ou un grattoir de cellules.
6. cellule Culture dans des Conditions de faible teneur en oxygène
- À partir de l’étape 4, maintenir les monocytes et les macrophages dans un environnement contrôlé en oxygène pour réaliser une analyse des conditions hypoxiques ensuite. Utiliser une station de travail hypoxie afin de maintenir les cellules sous la pression partielle d’oxygène désiré pendant l’expérience.
Remarque : Lorsque vous travaillez sous la pression de l’oxygène, il est important de préparer tous les supports et les tampons de lavage sous la gare et attendre suffisamment pour obtenir la bonne pression partielle dans le liquide. Par exemple, 10 mL de PBS dans une boîte de pétri de 60 mm nécessite environ 2 h pour atteindre 25 mm Hg pour la pression partielle de2 O à partir de pressions atmosphériques (comme nous l’avons mesuré à l’aide d’un capteur à fibre optique d’oxygène). Dans de nombreux postes hypoxiques ou incubateurs, la pression d’oxygène est définie comme un pourcentage de la pression atmosphérique. Si des mesures précises sont nécessaires, il est préférable d’utiliser une station autorisant directement régler la pression de l’oxygène en mmHg.
7. lyse et Digestion de dans-Gel (protocole 1)
Remarque : En cela et les sections qui suivent, deux protocoles utilisés pour obtenir des peptides et effectuer des analyses LC-MS/MS sont décrites. Protocole 1 décrit la lyse cellulaire et le fractionnement en gel et la digestion, et protocole 2 la lyse des cellules en solution suivie de la digestion en solution et de fractionnement à l’aide d’une méthode mise au point isoélectrique.
- Effectuer la lyse des cellules dans le tampon de Laemmli [234 mM Tris-HCL (pH 6,8), 7,5 % SDS, 37 % de glycérol, 33,3 % (v/v) de β-mercaptoéthanol, bleu de bromophénol 0,2 % p/v]. Charger l’équivalent protéique de 300 000 cellules de chaque échantillon de 4 à 12 % bis-Tris gels d’acrylamide.
- Contrôler la durée de la migration électrophorétique pour permettre à chaque échantillon de protéine à se diviser en gel 6 bandes, comme illustré à la Figure 3.
- Fixer le gel avec une solution de fixation (30 % éthanol + 7,5 % d’acide acétique pendant 20 min), puis ajouter la solution colorante (R-250 Coomassie bleu pendant 45 min). Ajoutez la solution de décoloration (30 % éthanol + 7,5 % acétique jusqu'à apparaissent des bandes) avant d’exciser les bandes de protéines avec un scalpel propre.
- Couper en dés chaque bande excisée avant introduction dans des tubes de 500 µL. Une surface de verre propre est justifiée pour éviter toute contamination avec Kératines (solution SDS 5 % dans l’eau désionisée utilisable pour nettoyer les surfaces).
- Laver les tranches de gel 3 fois dans 200 µL de bicarbonate d’ammonium 25 mM pendant 20 min à 37 ° C, suivie d’un lavage au bicarbonate d’ammonium 25 mM et l’acétonitrile (50 % v/v). Déshydrater les morceaux de gel avec 200 µL de 100 % d’acétonitrile pendant 10 min.
- Incuber à chaque morceau de gel avec 10 mM TNT (dithiothréitol) en 25 mM d’ammonium bicarbonate pendant 45 min à 56 ° C (200 µL), suivi par l’iodoacétamide 55 mM à 25 mM de bicarbonate d’ammonium (200 µL) pendant 35 min à l’obscurité à température ambiante.
- Pour arrêter l’alkylation, incuber chaque morceau de gel avec 200 µL de 10 mM TNT de bicarbonate d’ammonium 25 mM pendant 10 min à RT. Wash les morceaux de gel dans 200 µL de bicarbonate d’ammonium 25 mM, puis déshydrater avec 200 µL d’acétonitrile 100 % pendant 10 min.
- Digérer les protéines pendant une nuit à 37 ° C avec la trypsine/Lys-C mélange selon les instructions du fabricant.
- Extraire les peptides résultants du gel de morceaux en ajoutant 50 μL de 50 % d’acétonitrile pendant 15 min, puis 50 μL d’acide formique 5 % pendant 15 min et enfin, 50 μL d’acétonitrile 100 % pendant 15 min. piscine et chaque fraction dans des tubes de faible absorption de limiter l’adsorption des peptides sèche et perte de l’échantillon. Conserver les échantillons à-80 ° C jusqu'à l’analyse ultérieure.
8. protein Extraction et Digestion en Solution (protocole 2)
- Effectuer la lyse cellulaire (2 x 106 cellules) avec 150 µL de la mémoire tampon de lyse suivants :
- Urée de 7 M, 2 M thiourée, 40 mM Tris et 4 % CHAPS, complétées par des inhibiteurs de protéase (mini complet, sans EDTA inhibiteur de protéase cocktail).
- Homogénéiser les solutions pendant 30 min à RT avec un thermoshaker. Centrifuger à 13 800 x g pendant 20 min RT et garder le liquide surnageant.
- Retirer les contaminants avec un kit de nettoyage 2D :
- Le kit contient la solution precipitant, solution co précipitant, tampon de lavage et additif de lavage.
- Ajouter 300 µL de solution precipitant et bien mélanger. Incuber sur glace pour 15 min. ajouter 300 µL de solution co précipitant. Centrifuger les tubes (au moins) à 12 000 x g pendant 5 min. Une petite pastille devrait être visible. Procéder rapidement à l’étape suivante pour éviter la remise en suspension ou la dispersion de la pastille. Retirez le surnageant sans déranger le culot.
- Centrifuger les tubes avec le cap-charnière et pellet vers l’extérieur pour mettre n’importe quel liquide restant au fond du tube. Une brève impulsion suffit. Il ne devrait y avoir aucun liquide visible restant dans les tubes.
- Sans déranger le culot, ajouter 40 µL de solution de co précipitant. Laisser le tube s’asseoir sur la glace pendant 5 min. centrifuger pendant 5 min, puis retirer et jeter le lavage. Ajouter 25 µL d’eau déionisée. Vortex de chaque tube pour 5-10 s. Le culot doit se disperser mais ne se dissolvent pas dans l’eau.
- Ajouter 1 mL de tampon de lavage (préalablement réfrigéré pendant au moins 1 h à-20 ° C) et 5 µL d’additif de lavage. Vortex jusqu'à ce que la pastille est entièrement dispersée. Incuber les tubes à-20 ° C pendant au moins 30 min. Vortex pour 20-30 s toutes les 10 min
Remarque : Les tubes peuvent être stockés à-20 ° C pendant 1 semaine avec la dégradation des protéines minimale ou la modification. - Centrifuger les tubes (au moins) à 12 000 × g pour 5 min. Retirez avec précaution et éliminer le surnageant. Une pastille blanche doit être visible. Laisser le diabolo à airdry pendant pas plus de 5 min (si la pastille est trop sec, qu'il sera difficile de remettre en suspension).
- Resuspendre le culot de protéine dans 300 µL de 8 M urée et 0,1 M de bicarbonate d’ammonium. Vortex fortement pendant 1 min. déterminer la concentration de la protéine à l’aide d’un dosage colorimétrique.
9. en-Solution Digestion (protocole 2)
- Réduire les ponts disulfures en ajoutant 5.1 µL d’une solution aqueuse de 700 mM DTT (concentration finale 12,5 mM) aux protéines remises en suspension d’étape 8,4 et incuber à 37 ° C pendant 30 min avec un thermoshaker. Alkylat résidus cystéine en ajoutant 20,3 µL d’une solution aqueuse de 700 mM iodoacétamide (concentration finale 40 mM) et en incubation à 25° C pendant 30 min dans le noir avec une thermoshaker.
- Ajouter 990 µL de bicarbonate d’ammonium 0.1 M dans l’échantillon. Ajouter un volume correspondant de trypsine/Lys-C mix (substrat de l’enzyme : ratio 1/100 w/w). Incuber à 37° C pendant la nuit avec un thermoshaker.
10. nettoyage cartouche (protocole 2)
- Mouiller une cartouche et 1 colonne-volume (1 mL) de méthanol. Nettoyer la cartouche et 1 colonne-volume (1 mL) d’eau de 80 % d’acétonitrile/qualité-HPLC et jeter le cheminement. Equilibrez la cartouche avec 4 colonne-volumes (4 mL) d’eau de 0,1 % d’acide formique/qualité-HPLC et jeter le cheminement.
- Acidifier les échantillons avec 90 µL d’eau à un pH de 2-3 (vérifier le pH avec un indicateur de pH) ou de 10 % d’acide formique. Charger les échantillons acidifiés et collecter le cheminement. Recharger le cheminement (contenant les peptides non retenu). Laver la cartouche avec 6 colonne-volume (6 mL) d’eau de l’acide formique/HPLC-grade de 0,1 %.
- Éluer les peptides de la cartouche avec 1 colonne-volumes (1 mL) d’eau de qualité/acétonitrile-HPLC acid/50% formique 0,1 %. Transférer dans un tube de microtubes de 1,5 mL. Concentrer l’échantillon à l’aide d’une pompe à vide (150 x g, sous vide à 160 mBar).
11. fractionnement par focalisation isoélectrique (protocole 2)
Remarque : Les Peptides sont séparés selon leurs points isoélectriques utilisant un fractionnateur hors-gel sur une bande de 13 cm couvrant une plage de pH de 3 à 10. Nous avons utilisé le protocole suivant fourni par le fournisseur (résumé ci-dessous) :
- Préparer les solutions suivantes : solution un (600 µL de solution de glycérol, 60 µL de tampon de OFFGEL, 4,34 mL d’eau ultrapure) et B (1,776 mL de solution A) et 444 µL d’eau ultrapure.
- Assembler les bandes IPG, cadres et les électrodes selon les instructions du fabricant.
- Remettre en suspension l’échantillon avec 1.8 mL de solution B. ajouter 40 µL de solution B dans chaque puits. Charger 150 µL de l’échantillon dans chaque puits.
- Sélectionnez le mode par défaut pour les peptides : OG12PE00 (OFFGEL méthode par défaut de peptides pour une utilisation avec un 3100 OFFGEL Low Res Kit, cadres de 12 puits de 3 à 10, pH. Attendez jusqu'à ce que cette méthode a été complété (~ 20 h). Recueillir les fractions en tubes correctement étiquetées.
12. nettoyage Harvard appareil colonne inverse C18 Post-IEF (protocole 2)
- Ajouter progressivement de quelques μL à la fois de 1 % TFA dans de l’eau déminéralisée à chaque fraction s’acidifier l’échantillon. Vérifier à l’aide de papier pH que le pH est d’environ 3 ou ci-dessous.
- Préparer les solutions suivantes : solution 1 (5 mL d’acétonitrile, 10 µL d’acide formique, 4,99 mL d’eau ultrapure) et 2 (0,5 mL d’acétonitrile, 10 µL de l’acide formique, 9,49 mL d’eau ultrapure).
- Pré humide la colonne avec 150 µL de la solution 1. Centrifuger pendant 90 s à 750 x g et jeter le cheminement. Laver la colonne avec 150 μl de solution 2. Centrifuger pendant 90 s à 750 x g et jeter le cheminement.
- Passez la fraction par le biais de la colonne. Centrifuger pendant 90 s à 750 x g et jeter le cheminement. Laver avec 150 μl de solution 2. Centrifuger pendant 90 s à 750 x g et jeter le cheminement.
- Éluer les fractions contenant 50 μL de la solution 1. Centrifuger pendant 90 s à 750 x g. Répétez ces étapes une fois de plus.
- Sec-fraction à l’aide d’une pompe à vide (150 x g, sous vide 160 mBar) et stocker à-80 ° C
13. l’analyse des données protéomiques et bioinformatique18
- Analyser les données obtenues par un nano-LC-MS/MS masse spectromètre à l’aide de logiciels de quantification telles que MaxQuant (version 15.2.8) et le moteur de recherche d’Andromède.
- Définir le taux de fausse découverte (FDR) à 1 % de protéines et de peptides et d’une longueur minimale de 7 acides aminés. Définir la spécificité de l’enzyme comme C-terminal pour Arg et Lys. Qu’il soit 2 clivages manqués proline obligations. Sélectionnez carbamidomethylation de cystéine comme modification fixe et oxydation de l’acétylation et méthionine des protéines N-terminal variable modifications.
- Poursuivre l’analyse des données avec le logiciel d’analyse statistique. Effectuer une analyse fonctionnelle de l’enrichissement à l’aide du logiciel FunRich (www.funrich.org/). Effectuer une analyse d’enrichissement ontologie génique à l’aide de logiciels de DAVID (https://david.ncifcrf.gov/).
Résultats
À partir de cellules mononucléaires du sang périphérique (PBMC) obtenus par centrifugation différentielle, le protocole permet l’obtention d’une population de CD14+ monocytes avec une pureté mises en recouvrement de plus de 98 % par cytométrie en flux (Figure 1). Ces monocytes sont différencient secondairement vers diverses polarisations (Figure 2). Quand un fractionnement sur gel est choisi, la migration sur gel SDS-page est adaptée pour obtenir le nombre de bandes souhaitées et les bandes sont excisées (Figure 3). La digestion est réalisée secondairement dans les bandes excisés du gel, puis les peptides sont extraits. Les peptides sont analysés en utilisant une (chromatographie en phase liquide) nano-LC-MS/MS de spectromètre de masse. Les spectres MS/MS donnent l’identité de diverses protéines selon l’annotation des spectres obtenus pour les peptides connus (Figure 4 a). La quantification de l’abondance d’une protéine est ensuite calculée en rapport avec la quantité de peptides identifiés provenant de la protéine à l’aide de logiciels publiés et bases de données15,16. Ce protocole avec la digestion en gel donne environ 4000 protéines identifiées, et la gamme dynamique a été trouvée pour couvrir les 5 unités de l’échelle logarithmique (Figure 4 b). Analyse de l’expression différentielle de ces protéines identifiées peut être utilisée pour déterminer le regroupement des diverses polarisations dans des environnements différents de l’oxygène.
Avec cette méthode, nous pouvons aussi reconnaître les amas de protéines qui sont régulée lorsqu’ils sont exposés à une concentration faible teneur en oxygène 3 % (Figure 5, tableau 1). Afin d’évaluer l’efficacité de la digestion, ce qui est impossible quand un protocole en gel est utilisé, nous a proposé une méthode de digestion en solution qui a été adaptée pour les macrophages humains (Figure 6 a). Avec cette méthode, on peut facilement obtenir (après digestion solution) identification de 3600 protéines sans fractionnement, ce qui signifie que le fractionnement avec la volonté de l’IEF sensiblement augmenter ce nombre (Figure 6 b).

Figure 1 : analyse d’écoulement cytometry de CD14 expression de PBMC avant le tri (panneau de gauche) et après le tri (panneau de droite) montrant la pureté obtenue après sélection de billes magnétiques. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : images de contraste de Phase des macrophages humains différenciés, montrant l’hétérogénéité de la morphologie obtenus pour les deux polarisations différentes. Barre d’échelle représente 50 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 : imagerie de bleu de Coomassie teinté gel montrant les différentes bandes qui vont être excisées [ici, 6 bandes dans les macrophages M(Ø)] pour 5 polarisations des macrophages exposés à un environnement pauvre en oxygène. IC = complexes immuns, DXM = dexaméthasone. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
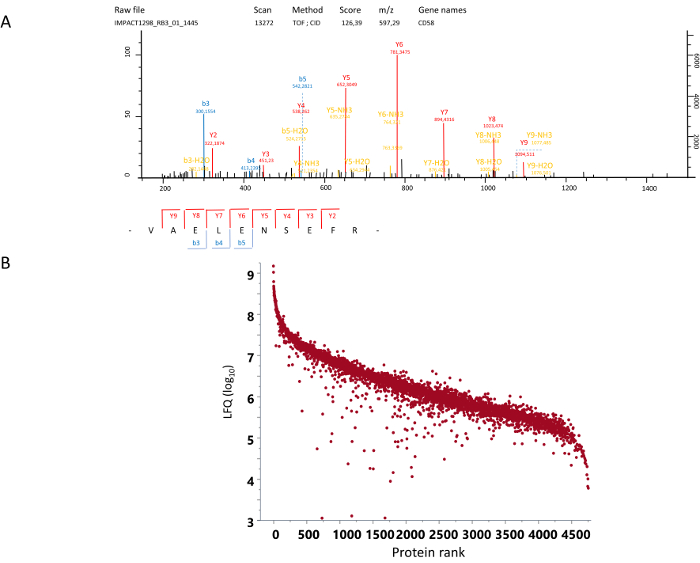
Figure 4 : spectre MS/MS et quantification. (A) un exemple d’un spectre MS/MS. Montré ici est le spectre de CID (dissociation induite par collision) d’un peptide à m/z 597.29 sur le spectre MS avec une charge électrique de + 2. La séquence correspondante a été déterminée à partir de cette partie du spectre comme Val-Ala-Glu-Leu-Glu-Asn-Ser-Glu-Phe-Arg de la protéine CD58. B grade ordre exempt d’étiquette de quantification pour chacune des protéines identifiées (log10 LFQ). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
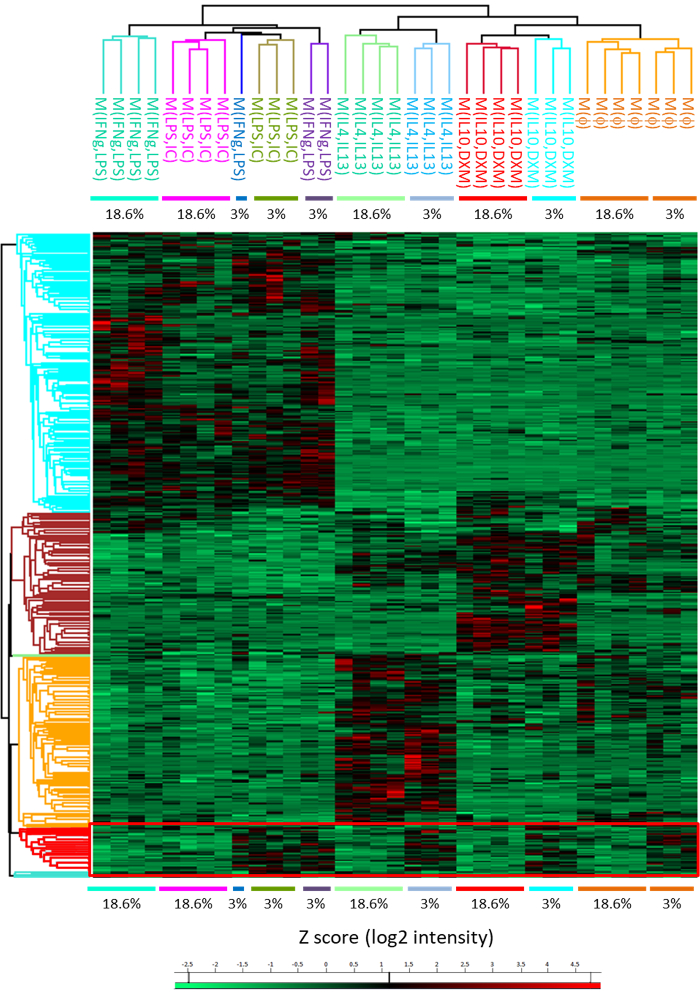
Figure 5 : Heatmap représentant le clustering hiérarchique de polarisation tous les États utilisant différemment les protéines exprimées. L’analyse révèle un amas de protéines surexprimés dans toutes les polarisations dans la condition de2 3 % O (rectangle rouge). L’échelle de couleurs représente z-scores (log2 intensité). Chaque ligne est une protéine, et chaque colonne est un échantillon. Ce chiffre provient d’une précédente publication14. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 6 : SDS-PAGE et chromatogramme. (A) gels de SDS-PAGE argent-colorés avec des protéines de la lyse cellulaire et après digestion de solution en montrant l’absence de dégradation lors de la lyse et l’efficacité de la digestion. (B) le chromatogramme obtenu d’après digestion solution sans fractionnement. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Cluster | proteinID | Noms de protéine | Noms de gènes | Peptides | Rasoir + peptides uniques | Peptides uniques | ID de la protéine | ||
| Rouge | P0DMV9 | Heat shock 70 kDa protéine 1 b | HSPA1A | 40 | 37 | 4 | P0DMV9 ; P0DMV8 ; A8K5I0 ; Q59EJ3 ; B4DNT8 ; B4DWK5 ; B4DFN9 ; B4DI39 ; B4E1S9 ; B4DVU9 ; V9GZ37 ; B3KTT5 ; B4DNX1 ; B4E1T6 ; B4DNV4 ; Q9UQC1 | ||
| Rouge | P54709 | Sodium/potassium-transport ATPase sous-unités bêta-3 | ATP1B3 | 8 | 8 | 8 | P54709 ; D3DNF9 ; C9JXZ1 ; C9JA36 ; H7C547 ; F8WBY4 ; Q58I18 | ||
| Rouge | O00462 | Bêta-mannosidase | MANBA | 14 | 13 | 13 | O00462 ; A7LFP5 ; A8K6D3 ; E9PFW2 ; B4DT18 ; Q59EG5 | ||
| Rouge | Q8NAS7 | Protéine fer-soufre de NADH déshydrogénase [ubiquinone] 7, mitochondriale | NDUFS7 | 4 | 4 | 4 | Q8NAS7 ; Q6ZQU6 ; F5H5N1 ; B7Z1U1 ; A8K0V6 ; Q7LD69 ; F5GXJ1 ; O75251 ; B7Z4P1 ; Q9H3K5 ; A0A087WXF6 ; A0A087WTI3 ; Q6ZS38 | ||
| Rouge | Q8WWQ0 | PH-interacting protein | PHIP | 5 | 5 | 4 | Q8WWQ0 ; Q9NWP3 | ||
| Rouge | E5RHK8 | Dynamine-3 | DNM3 | 11 | 2 | 2 | E5RHK8 ; Q9UQ16 ; B3KPF2 ; E5RIK2 | ||
| Rouge | A0A024QZ64 | Fructose-Diphosphate aldolase C | ALDOC | 18 | 14 | 7 | A0A024QZ64 ; P09972 ; B7Z1Y2 ; B7Z3K9 ; B7Z1N6 ; B7Z3K7 ; A8MVZ9 ; J3KSV6 ; J3QKP5 ; B7Z1L5 ; C9J8F3 ; K7EKH5 ; J3QKK1 ; B7Z1H6 | ||
| Rouge | O75489 | Protéine fer-soufre de NADH déshydrogénase [ubiquinone] 3, mitochondriale | NDUFS3 | 12 | 12 | 12 | O75489 ; Q9UF24 ; Q53FM7 ; E9PS48 ; E9PKL8 ; G3V194 | ||
| Rouge | P21912 | Succinate déshydrogénase [ubiquinone] fer-soufre sous-unité mitochondriale | SDHB | 11 | 11 | 11 | P21912 ; A0A087WXX8 ; A0A087WWT1 | ||
| Rouge | A0A024R1Y7 | GH3 contenant du domaine protéine | LGP1 ; GHDC | 7 | 7 | 7 | A0A024R1Y7 ; Q8N2G8 ; B3KVB0 ; K7ESN3 ; K7EJT7 ; K7EQ41 ; K7EL54 | ||
| Rouge | E5KRK5 | Sous-unité NADH-ubiquinone oxydoréductase 75 kDa, mitochondriale | NDUFS1 | 29 | 29 | 29 | E5KRK5 ; P28331 ; B4DJ81 ; Q9P1A0 ; C9JPQ5 ; F8WDL5 | ||
| Rouge | A0A024QZ30 | Succinate déshydrogénase [ubiquinone] flavoprotéine sous-unité mitochondriale | SDHA | 17 | 17 | 17 | A0A024QZ30 ; P31040 ; D6RFM5 ; B3KT34 ; A0A087X1I3 ; Q0QF12 ; B4DYN5 ; B3KYA5 ; B4DNB2 ; H0Y8X1 ; B7Z6J5 | ||
| Rouge | A0A024R2F9 | Protéine transmembranaire 43 | TMEM43 | 16 | 16 | 16 | A0A024R2F9 ; Q9BTV4 ; Q8TEP9 ; V9GY05 | ||
| Rouge | A0A024R5K3 | Protéine fer-soufre de NADH déshydrogénase [ubiquinone] 8, mitochondriale | NDUFS8 | 5 | 5 | 5 | A0A024R5K3 ; E9PPW7 ; O00217 ; E9PN51 ; F8W9K7 ; E9PKH6 ; B4DYI3 ; Q53G17 | ||
| Rouge | O76003 | Glutarédoxine-3 | GLRX3 | 13 | 13 | 13 | O76003 | ||
| Rouge | Q1HBJ4 | Mitogène-protéine kinase ; Mitogène-protéine kinase 1 | MAPK1 | 26 | 26 | 21 | Q1HBJ4 ; P28482 ; B4DHN0 ; Q499G7 ; K7ELV1 ; K7EN18 ; B4E104 ; P31152 ; Q16659 | ||
| Rouge | Q13151 | Ribonucléoprotéine nucléaire hétérogène A0 | HNRNPA0 | 8 | 8 | 8 | Q13151 | ||
| Rouge | V9HWN7 | Fructose-Diphosphate aldolase A musculaire | CINDY | 36 | 36 | 14 | V9HWN7 ; P04075 ; J3KPS3 ; H3BQN4 ; H3BUH7 ; H3BR04 ; H3BMQ8 ; H3BU78 ; H3BR68 ; A4UCS9 ; A4UCT0 | ||
| Rouge | B4DVJ0 | Glucose-6-phosphate isomérase | GPI | 32 | 32 | 23 | B4DVJ0 ; P06744 ; B4DE36 ; A0A0A0MTS2 ; K7EQ48 ; K7EP41 ; K7EPY4 ; K7ELR7 ; K7ERC6 ; K7ENA0 ; K7ESF4 ; K7EIL4 ; K7ERK8 ; Q59F85 | ||
| Rouge | V9HWB9 | L-lactate déshydrogénase ; L-lactate déshydrogénase une chaîne | LDHA | 36 | 36 | 34 | V9HWB9 ; P00338 ; B4DJI1 ; F5GXY2 ; F5GYU2 ; F5GXH2 ; F5H5J4 ; F5H6W8 ; F5GZQ4 ; F5H8H6 ; F5GXC7 ; F5GWW2 ; F5GXU1 ; A0A087WUM2 ; Q96L19 ; Q6ZMR3 | ||
| Rouge | P13674 | Prolyl hydroxylase-4 sous-unités alpha-1 | P4HA1 | 26 | 26 | 26 | P13674 ; Q5VSQ6 | ||
| Rouge | Q6FHV6 | Gamma-énolase ; Énolase | ENO2 | 18 | 15 | 14 | Q6FHV6 ; P09104 ; A8K3B0 ; F5H0C8 ; F5H1C3 ; U3KQP4 ; U3KQQ1 ; Q9NPL4 | ||
| Rouge | Q99798 | Aconitate hydratase, mitochondriale | ACO2 | 40 | 40 | 40 | Q99798 ; B4DZ08 ; B2RBW5 ; A2A274 ; B4DJW1 ; B4DLY4 ; Q71UF1 ; B4DEC3 ; B4DW08 ; O75944 | ||
| Rouge | P17858 | Type 6-phosphofructokinase, foie de ATP-dépendante | PFKL | 32 | 29 | 28 | P17858 ; Q7L2M7 ; Q9BSP4 ; B3KNQ7 ; B4E108 ; Q59GI2 ; F8WEU2 ; Q7Z3R9 ; Q6MZK4 | ||
| Rouge | A0A024R872 | Niban-comme la protéine 1 | FAM129B | 32 | 32 | 32 | A0A024R872 ; Q96TA1 ; Q9H8K1 ; Q2YD88 ; Q9H6L6 | ||
| Rouge | A0A024RC61 | Aminopeptidase N | ANPEP | 58 | 58 | 25 | A0A024RC61 ; P15144 ; Q59E93 ; B4DV63 ; B4DP01 ; B4DP96 ; H0YKT6 ; H0YLZ8 ; Q71E46 ; H0YMC1 ; Q8IVL7 | ||
| Rouge | V9HWF4 | Phosphoglycérate kinase ; Phosphoglycérate kinase 1 | PGK1 | 41 | 41 | 35 | V9HWF4 ; P00558 ; B4E1H9 ; B4DHM5 ; B4DHB3 ; B4DWQ3 ; Q16444 | ||
| Rouge | Q12882 | Dihydropyrimidine déshydrogénase [NADP(+)] | DPDY | 44 | 44 | 44 | Q12882 ; B4DML1 | ||
| Rouge | B4DEQ0 | Oxydoréductase d’ubiquinone-flavoprotéine transfert d’électron, mitochondriale | DÉFICIT | 9 | 9 | 9 | B4DEQ0 ; Q547S8 ; A7UNU5 ; Q16134 ; D6RAD5 | ||
| Rouge | D9UAX9 | CMH de classe I antigène | HLA-B | 13 | 3 | 2 | D9UAX9 | ||
| Rouge | V9HWK1 | Triosephosphate isomérase | TPI1 | 29 | 29 | 17 | V9HWK1 ; P60174 ; Q53HE2 ; B4DUI5 ; U3KPZ0 ; U3KQF3 ; U3KPS5 | ||
| Rouge | Q96HE7 | Alpha de la protéine ERO1 | ERO1L | 28 | 28 | 28 | Q96HE7 ; G3V3E6 ; G3V5B3 ; G3V2H0 ; G3V503 ; Q5TAE8 ; B2RD00 ; Q86YB8 | ||
| Rouge | P14868 | Aspartate--ligase d’ARNt, cytoplasmique | DARS | 29 | 29 | 2 | P14868 ; Q53T60 ; Q53R85 ; H7BZ35 ; H7C278 | ||
| Rouge | P36871 | Phosphoglucomutase-1 | PGM1 | 24 | 24 | 5 | P36871 ; B4DFP1 ; Q9H1D2 | ||
Tableau 1 : Liste des protéines surexprimés pour les macrophages humains communs à chaque polarisation sous faible tension d’oxygène.
Discussion
Parce que la protéomique est un outil puissant pour étudier l’expression des différentes protéines d’une cellule entière ou compartiments subcellulaires, optimisation du protocole de la lyse cellulaire et la digestion des protéines a été traitées par un certain nombre d’études. Il y a trois grandes classes de méthodes, qui incluent la digestion en gel (digestion des protéines en gel de polyacrylamide matrice)17, digestion en solution18 et filtre assistée par exemple préparation19. Cette dernière méthode, dans un premier temps décrit comme universelle, a été signalée à pièce faible reproductibilité et une éventuelle perte de protéines sur le filtre20. Digestion de dans-gel est une méthode robuste qui peut être fastidieux et désavantageux qu’évaluer l’efficacité de la digestion n’est pas facile, si possible. Digestion en solution offre cette possibilité, mais nécessite le nettoyage des échantillons après digestion et IEF. Lorsque ces deux méthodes sont comparées entre le même échantillon, digestion en solution avec protocole de fractionnement IEF donne un nombre plus élevé de protéines identifiées (avec le même nombre de fractions) que digestion21en gel.
Malgré cet avantage, il est nécessaire de tenir compte de la dégradation des protéines possible lors de la lyse solution en raison des protéases intracellulaires (en particulier dans les cellules myéloïdes). Il est également important de garder à l’esprit que ces techniques sont basées sur la digestion des protéines et seulement en mesure d’analyser les protéines présentant la trypsine sites de clivage spécifiques. Il est possible d’utiliser une approche descendante protéomiques qui soulage cette contrainte de digestion, mais ajoute des étapes d’analyse de données et de ressources de bioinformatique22. La solubilisation des protéines de différents compartiments cellulaires peut être également difficile à obtenir, surtout à partir de membranes plasmiques, conduisant à un échantillonnage non contrôlé de proteome cellulaire. Afin de procéder à une analyse de spectromètre de masse de nano-LC-MS/MS des échantillons, il est important d’obtenir une quantité suffisante de peptides, qui peuvent compter sur le spectromètre de masse utilisé (généralement, à partir de protéine totale doit être au moins 1 µg pour un état, et Il est implicite pour augmenter cette quantité en fonction du nombre de fraction utilisée avec IEF). Cette contrainte peut être un inconvénient si la population de cellules étudiée vient à manquer, ce qui différencie la protéomique de techniques génomiques dans lequel l’amplification de la matière première est possible.
Même après les ouvrages de Richer et ses collègues23 et Packer et Fuehr24, l’importance de l’oxygène dans des cultures cellulaires a été insuffisamment reconnu. Nous savons maintenant que la mise en culture des cellules sous de faibles concentrations d’oxygène favorise les adhésion, durée de vie et la division. Il est reconnu qu’il s’agit de la plus haute importance dans la recherche de cellules souches25. Le principal problème technique pour des cultures de cellules dans des conditions contrôlées d’oxygène est lié à l’entretien de la concentration en oxygène désiré pendant l’expérience entière. Cela nécessite la préincubation de tous les médias d’empêcher la libération d’oxygène dissous et utiliser des postes de travail hypoxique pour permettre la manipulation de cellules sous faible teneur en oxygène (traitement chambre avec boîte à gants) et exposition transitoire à des conditions d’oxygène élevée .
Le protocole décrit a été utilisé pour obtenir les signatures moléculaires de diverses polarisations des macrophages dérivés de monocytes humains et d’étudier les effets de modulation de l’oxygène sur ces signatures. Cette étude a donné un aperçu sur la description de ces polarisations et a révélé quelques conséquences fonctionnelles. Par exemple, nous avons constaté que plusieurs protéines impliquées dans efferocytosis ont été modulées par un environnement pauvre en oxygène. Cette approche protéomique, basée sur le protocole décrit, présente l’occasion d’explorer comment les paramètres environnementaux modifier les fonctions du macrophage et comment ces signaux peut être utilisés pour concevoir de nouvelles approches thérapeutiques14.
L’approche protéomique décrit dans ce travail est complémentaire des approches génomiques qui ont été utilisés au cours de ces dernières années dans le domaine des études de polarisation des macrophages humains. Protéomique offre l’avantage de la quantification des protéines, qui peut-être présenter une expression différente que leurs ARNm correspondants en raison de modifications post-traductionnelles et conduire à la découverte de nouveaux biomarqueurs. Malgré cet avantage, des données protéomiques sont généralement difficiles à interpréter, en partie en raison de la forte sensibilité de spectrométrie de masse, entraînant les spectres MS très complexes et fausses détections positives de peptides. Récemment, logiciel d’analyse a gagné en efficacité pour empêcher cela. Même si c’est une évolution de la situation, protéomique a également fait face à plus faible reproductibilité de génomique26 et est associée à des étapes de validation à l’aide d’autres techniques (cytométrie en flux, immunoblotting) pour confirmer les modifications quantitatives des protéines niveaux d’expression.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêt.
Remerciements
AM est financé par le programme Leader du groupe Young (ATIP/Avenir Inserm-CNRS), de la Ligue Nationale contre le Cancer et la Fondation ARC versez la recherche sur le Cancer. Nous remercions Mariette Matondo de la spectrométrie de masse pour plateforme de biologie (UTECHS MSBIO, Institut Pasteur, Paris). Nous remercions Lauren Anderson pour sa lecture du manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| Hypoxia Working Station | Oxford Optronix | Hypoxylab | |
| C6 Flow cytometer | BD | Accuri C6 | |
| Urea | Agilent Technologies | 5188-6435 | |
| Formic acid (FA) | ARISTAR | 450122M | |
| R-250 Coomassie blue | Biorad | 1,610,436 | |
| Lipopolysaccharide, E.Coli (LPS) | Calbiochem | 437627 | |
| 2D clean-up kit | GE Healthcare | 80-6484-51 | |
| RPMI 1640 medium, glutamax supplement | Gibco | 61870044 | |
| HEPES 1 M | Gibco | 15630-080 | |
| MEM Non-Essential Amino Acids (NEAA) Solution 100X | Gibco | 11140-035 | |
| Phosphate Buffered Saline (PBS) 1X | Gibco | 14190-094 | |
| Harvard Apparatus column Reverse C18 micro spin column | Harvard Apparatus | 74-4601 | |
| EDTA 0.5 M, pH 8.0 | Invitrogen | AM9260G | |
| NuPAGE Bis-Tris 4-12% | Life Technologies SAS | NP0321 BOX | |
| CD14 Microbeads human | Miltenyi Biotec | 130-050-201 | |
| MACS separation column LS | Miltenyi Biotec | 130-042-401 | |
| Macrophage colony-stimulating factor (M-CSF) | Miltenyi Biotec | 130-096-485 | |
| Interleukin 4 (IL4) | Miltenyi Biotec | 130-093-917 | |
| Interleukin 13 (IL13) | Miltenyi Biotec | 130-112-410 | |
| Interferon gamma (INFγ) | Miltenyi Biotec | 130-096-482 | |
| CD14-FITC (clone TÜK4) | Miltenyi Biotec | 130-080-701 | |
| MACSmix Tube Rotator | Miltenyi Biotec | 130-090-753 | |
| Trifluoroacetic Acid (TFA) | Pierce | 28904 | |
| Trypsin/Lys-C Mix | PROMEGA | V5073 | |
| Complete Mini, EDTA-free Protease Inhibitor cocktail | Roche | 11836170001 | |
| Density Gradient Solution (Histopaque 1077) | Sigma Aldrich | 10771-100ML | |
| Accumax | Sigma Aldrich | A7089-100ML | |
| Human Serum from human male AB plasma (SAB) | Sigma Aldrich | H4522-100ML | |
| Bovine Serum Albumin (BSA) solution 30% | Sigma Aldrich | A9576-50ML | |
| Trisma-base | Sigma Aldrich | T1503 | |
| Glycerol | Sigma Aldrich | 49767 | |
| β-Mercaptoethanol | Sigma Aldrich | M3148 | |
| Bromophenol blue | Sigma Aldrich | 114405 | |
| Sodium Dodecyl Sulfate (SDS) 20% | Sigma Aldrich | 5030 | |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | |
| Acetonitrile | Sigma Aldrich | 34888 | |
| Dithiothreitol | Sigma Aldrich | 43819 | |
| Iodoacetamide | Sigma Aldrich | 57670 | |
| Thiourea | Sigma Aldrich | T8656 | |
| CHAPS | Sigma Aldrich | C9426 | |
| Micro BCA Assay Kit | ThermoFisher | 23235 | |
| 5 mL sterile plastic pipette | VWR | 612-1685 | |
| Thermomixer C Eppendorf | VWR | 460-0223 | |
| Sep-Pak tC18 reverse phase cartridges, 100 mg | Waters | WAT036820 |
Références
- Okabe, Y., Medzhitov, R. Tissue biology perspective on macrophages. Nature Immunology. 17 (1), 9-17 (2016).
- Sica, A., Mantovani, A. Macrophage plasticity and polarization: in vivo veritas. The Journal of Clinical Investigation. 122 (3), 787-795 (2012).
- Murray, P. J. Macrophage Polarization. Annual Review of Physiology. 79, 541-566 (2017).
- Chinetti-Gbaguidi, G., Staels, B. Macrophage polarization in metabolic disorders: functions and regulation. Current Opinion in Lipidology. 22 (5), 365-372 (2011).
- Chow, A., Brown, B. D., Merad, M. Studying the mononuclear phagocyte system in the molecular age. Nature Reviews Immunology. 11 (11), 788-798 (2011).
- Mosser, D. M., Edwards, J. P. Exploring the full spectrum of macrophage activation. Nature Reviews Immunology. 8 (12), 958-969 (2008).
- Murray, P. J., Wynn, T. A. Protective and pathogenic functions of macrophage subsets. Nature Reviews Immunology. 11 (11), 723-737 (2011).
- Ginhoux, F., Schultze, J. L., Murray, P. J., Ochando, J., Biswas, S. K. New insights into the multidimensional concept of macrophage ontogeny, activation and function. Nature Immunology. 17 (1), 34-40 (2016).
- Xue, J., et al. Transcriptome-based network analysis reveals a spectrum model of human macrophage activation. Immunity. 40 (2), 274-288 (2014).
- Csete, M. Oxygen in the cultivation of stem cells. Annals of the New York Academy of Sciences. 1049, 1-8 (2005).
- Johansson, A., et al. Functional, morphological, and phenotypical differences between rat alveolar and interstitial macrophages. American Journal of Respiratory Cell and Molecular Biology. 16 (5), 582-588 (1997).
- Pfau, J. C., Schneider, J. C., Archer, A. J., Sentissi, J., Leyva, F. J., Cramton, J. Environmental oxygen tension affects phenotype in cultured bone marrow-derived macrophages. American Journal of Physiology. Lung Cellular and Molecular Physiology. 286 (2), L354-L362 (2004).
- Grodzki, A. C. G., Giulivi, C., Lein, P. J. Oxygen tension modulates differentiation and primary macrophage functions in the human monocytic THP-1 cell line. PLoS One. 8 (1), e54926(2013).
- Court, M., Petre, G., Atifi, M. E., Millet, A. Proteomic Signature Reveals Modulation of Human Macrophage Polarization and Functions Under Differing Environmental Oxygen Conditions. Molecular & Cellular Proteomics. 16 (12), 2153-2168 (2017).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Cox, J., Neuhauser, N., Michalski, A., Scheltema, R. A., Olsen, J. V., Mann, M. Andromeda: a peptide search engine integrated into the MaxQuant environment. Journal of Proteome Research. 10 (4), 1794-1805 (2011).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols. 1 (6), 2856-2860 (2006).
- Court, M., et al. Toward a standardized urine proteome analysis methodology. Proteomics. 11 (6), 1160-1171 (2011).
- Wiśniewski, J. R., Zougman, A., Nagaraj, N., Mann, M. Universal sample preparation method for proteome analysis. Nature Methods. 6 (5), 359-362 (2009).
- Liebler, D. C., Ham, A. -J. L. Spin filter-based sample preparation for shotgun proteomics. Nature Methods. 6 (11), 785-786 (2009).
- Hubner, N. C., Ren, S., Mann, M. Peptide separation with immobilized pI strips is an attractive alternative to in-gel protein digestion for proteome analysis. Proteomics. 8 (23-24), 4862-4872 (2008).
- Park, J., et al. Informed-Proteomics: open-source software package for top-down proteomics. Nature Methods. 14 (9), 909-914 (2017).
- Richter, A., Sanford, K. K., Evans, V. J. Influence of oxygen and culture media on plating efficiency of some mammalian tissue cells. Journal of the National Cancer Institute. 49 (6), 1705-1712 (1972).
- Packer, L., Fuehr, K. Low oxygen concentration extends the lifespan of cultured human diploid cells. Nature. 267 (5610), 423-425 (1977).
- Lengner, C. J., et al. Derivation of pre-X inactivation human embryonic stem cells under physiological oxygen concentrations. Cell. 141 (5), 872-883 (2010).
- Sidoli, S., Kulej, K., Garcia, B. A. Why proteomics is not the new genomics and the future of mass spectrometry in cell biology. Journal of Cell Biology. 216 (1), 21-24 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon