Method Article
低酸素環境下におけるヒトのマクロファージ偏光のプロテオーム解析
要約
人間の大食細胞のプロテオーム署名を取得し、これをマクロファージ偏波に低酸素環境の影響の定量に適用するためのプロトコルを提案します。
要約
大食細胞は自然免疫系細胞数感染病原体に応答から組織恒常性に至るまでの生理機能に関与します。これらの細胞の様々 な機能のアクティブ化の状態、また偏光と呼ばれる関連しています。これらの各種偏波の精密分子説明が大食細胞生物学の分野で優先されます。それは現在環境信号により偏光を制御する方法を説明する必要がある、多次元的なアプローチを認められています。本報告では、ヒトのマクロファージで各種偏波のプロテオームの署名を取得するためのプロトコルについて述べる.このプロトコルは、ゲル分別と Lys C/トリプシン消化細胞換散コンテンツから得られる大食細胞タンパク質発現のラベル無料数量に基づいています。またプロトコルに基づいてソリューションの消化との代替として使用する分画を中心に等電点を提供します。酸素濃度は、組織に関連する環境パラメーターは、このプロトコルを使用してどのように大気組成を探索するまたは低酸素環境マクロファージ偏波の分類に影響を与えます。
概要
大食細胞は自然免疫系細胞の数組織の恒常性は、細胞外マトリックス1の改造とアポトーシス細胞の除去を含む感染病原体に応答に至る生理機能に関与します。これらの細胞は、多くの可能なアクティベーション状態、偏波とも呼ばれますに変換する強力な表現型可塑性2によって特徴付けられます。これらの各種偏波の精密分子説明が大食細胞生物学3の分野で優先されます。それはいわゆる M1 ・ M2 の二分法、M1 プロ炎症性を表し M2 マクロファージ炎症を使用してこれらの分極を分類するために提案されている.このモデルは、急性感染症、アレルギー、肥満4など様々 な病理学的状況で適合します。しかし、慢性的な炎症を起こしている組織やがん、それ実証されていますこの分類は特定セルラ環境5,6,に存在するマクロファージ幅広い表現レパートリーを把握することができます。7します。 現在のコンセンサスは、多次元モデルを使用して、特定のアイソレータケージ信号8を統合するマクロファージの偏波がよりよく記述されています。この結論は、M1 ・ M2 モデルが得られた偏波9の記述で効率的であることを示す人間の大食細胞のトランスクリプトーム解析により確認されています。
研究は、ヒトのマクロファージで各種偏波のプロテオームの署名を取得するためのプロトコルを提供することを目的を発表しました。我々 は様々 な酸素濃度の環境下でヒトのマクロファージを区別し、ラベル無料定量化を実行する全体のマクロファージ プロテオームからペプチドを取得する方法について説明します。この数量は、様々 なタンパク質の発現レベルの比較をできます。幹細胞の研究は、環境キー パラメーター10として酸素の重要性を明らかにしたと私たちはこの組織パラメーターができるヒトのマクロファージ偏波にどのように影響を与えるかを理解ましょう。酸素の分圧は、20% がおよそ (正確な値は約 18.6% 水の存在をしながら細胞文化のインキュベーターでよく使用されるものに、人間の体で 3 (合計の大気圧) の 20% からの範囲に発見されています。考慮)。
前作は、肺胞間質性マクロファージ機能とは異なる、これらの違いが部分的に露出12が異なる酸素濃度のためおそらくビュー11の形態のポイントを示しています。さらに、骨髄由来マクロファージは、細菌12低酸素環境にさらされたときを phagocytize する能力の増大を示します。THP1 区別ヒトマクロファージ13、反対の効果が発見されているが、これらの結果は、大食細胞生物学のレギュレータを酸素には、人間の大食細胞の分子レベルでこの役割を明らかにする必要の考えを支持します。以前の研究では、これらの問題に対処するためのプロテオミクス手法を用いています。たくさんの蛋白質の発現量を同時に測定することにより我々 は偏波上の酸素の影響を強調表示され、新しい分子マーカーのリストを提供します。また、いくつかのマクロファージ機能にこれらの結果を関連することができた。特に、IL4 IL13 偏極マクロファージ、プロテオーム解析14によって明らかにされた ALOX15 のアップレギュレーションにリンクされていた」アポトーシス細胞の貪食能の率は増加したが分かった。本研究で我々 はこのような分析を実行する方法をについて説明します。
プロトコル
ド識別、健康なドナーから血液サンプル (LRSC) は、承認されたプロトコル (CODECOH DC-2018-3114) の一部として EFS (フランス語国民の血サービス) から得られました。ドナーは、血液を使用するため署名された同意書を与えた。
1. メディアとバッファーの準備
- [RPMI glutamax + 10 mM HEPES + 1 x 非必須アミノ酸 (NEAA)] マクロファージ媒体を準備し、37 ° C に温める
- マクロファージ中 + AB プラズマ (SAB) から 10% 血清を準備、それ (0.22 μ m フィルター) をフィルタ リングし、37 ° C に温めて (マクロファージ中 + 10% と呼ばれる SAB 以下)。
- [リン酸緩衝生理食塩水 (PBS) + 0.5% ウシ血清アルブミン (BSA) + 2 mM エチレンジアミン四酢酸 (EDTA) x 1] の並べ替えのバッファーを準備、フィルター (0.22 μ m フィルター)、および 4 ° C で維持
2. 末梢血単核球 (PBMCs) Leukoreduction システム室 (LRSC) からの分離
- 密度勾配細胞分離液 15 mL を入れて (材料表参照) 50 ml 遠心チューブ、LRSC を受信する前に部屋の温度 (RT) に温めてくれるので。
注: 密度は温度に依存します。それは右に釣り合うことができますので、この手順必要があります事前に行われるこの製品は 4 ° C で保存、 - 50 mL の遠心管に、LRSC を空、1 × PBS の最大 50 mL を加え、混ぜます。非常にゆっくりと準備段階 2.2 手順 2.1 の時にウォーム アップを密度勾配液の 15 mL の上にミックスの 25 mL を追加します。
注: このステップの間に段階を混在させないよう注意します。血液は、密度の勾配ソリューションのこのフェーズの妨害なしに追加する必要があります。 - 遠心分離機の両方の遠心分離チューブ 700 x g 区切りなしで 25 分。
注: 先頭に下からレイヤー密度勾配遠心法末、: 赤血球および顆粒球のペレット、密度勾配ソリューション フェーズ、PBMCs とプラズマの層を形成します。 - 、ピペットを使い、それを吸引せずにプラズマ相を通過し、新しい 50 mL 遠心チューブに PBMC 層を収集します。洗浄工程と 300 x g で 10 分間遠心 PBMCs に 50 mL の 1x PBS までを追加します。
- 上清を吸引し、マクロファージ中の 40 mL にペレットを再懸濁します。
3. 磁気ラベリングと CD14 の分離+細胞 (単球)
- マラッセ室 PBMCs をカウントします。PBMCs (通常 100 ~ 300 × 106セル) の実験を行なうために必要な量を撤回は、遠心分離の管の 300 x g で 10 分間遠心します。
- 上清を吸引し、10 ごとの 1.3 のステップ中に準備した並べ替えバッファーの 80 μ L でペレットを再懸濁します7 /10 CD14 マイクロ ビーズの PBMCs。 追加 20 μ l7 PBMCs ミックスも、一定の攪拌下の 4 ° C で 15 分間インキュベートします。
- 洗浄工程と 300 x g. で 10 分間遠心、上清を吸引し、500 μ L の 108 PBMCs あたりバッファーの並べ替えでペレットを再懸濁します 107 PBMCs あたりバッファーの並べ替えの 1 つの mL を追加します。
- 区切り文字の磁気フィールドに列を配置します。バッファーの並べ替えの 3 mL と洗浄によって列を準備します。
- 列に細胞懸濁液を適用します。列に分離する細胞の数によって異なります (ここでは、最大 109 PBMCs の LS の列が使用されます)。フローを含むラベルのない細胞を収集します。
注意: からこのステップでは、すべてのチューブ (正と負の選択) は、フローサイトメトリーによる別の手順のチェック後に保持されます。 - 3 x 3 ml のバッファーの並べ替えの列を洗います。3.12 のステップから同じチューブを通る細胞を収集します。追加の並べ替えバッファーして洗浄手順を実行、列が乾燥しないよう注意してください。列の下のコレクション チューブを置き、区切り文字から削除します。
- バッファーを列に並べ替えの 5 mL のピペットします。しっかりと列にプランジャーを押すことにより磁気標識細胞をすぐにフラッシュします。CD14 の純度を高めるため+ 2 番目の列にセル、溶出画分が濃縮されています。
- 新しい列に 3.7 3.4 の手順を繰り返します。
4. 単球へのめっき
- マラッセの商工会議所の球をカウントします。CD14 の純度+フローサイトメトリーによる細胞します。実験に必要な単球の量を撤回し、遠心分離の管の配置します。
- 300 x g 吸引上清中に 10 分間遠心し、マクロファージの中で単球ペレットを再懸濁します。細胞をプレートし、分化誘導に SAB + 25 ng/mL 大食細胞のコロニー刺激要因を (M-CSF) 1 h 50 分培地を吸引し、マクロファージ中 + 10% にそれを置き換えるを我慢させます。
5. 6 日目でのマクロファージの偏光
- 培地を吸引します。マクロファージ中 + 10% に置き換える様々 な刺激と SAB。たとえば、10 ng/mL インターフェロン γ (INFγ) + 1 ng/mL リポ多糖 (LPS) M1 偏光を取得するまたは 20 ng/mL のインターロイキン 4 (IL4) + M2 偏波 20 ng/mL インターロイキン 13 (IL13) を追加します。
メモ: 24 と 48 他のテストに進む前に h の刺激を実行できます。 - デタッチ ソリューションまたは細胞スクレーパーを使用してセルを収穫します。
6. 低酸素条件下で培養
- 手順 4 から始まって、単球や、低酸素の状態分析を行う酸素制御環境でマクロファージを維持します。実験中に必要な酸素分圧下で細胞を維持するために低酸素症ワーキング ステーションを使用します。
注意: 低酸素分圧下で作業する場合、すべてのメディアと駅下洗浄バッファーを準備し、液体で正しい圧を得るに十分に待つ重要です。たとえば、10 mL の PBS で 60 mm のペトリ皿 (我々 がファイバー光学酸素センサーを用いた測定) 大気圧から始まって O2分圧 25 mmHg に到達する約 2 時間が必要です。多くの低酸素ステーションまたはインキュベーター、酸素分圧は大気圧の割合として設定されます。精密測定が必要な場合は、直接 mmHg の酸素分圧を設定するを許可するステーションを使用することをお勧めします。
7. 換散およびゲルの消化力 (プロトコル 1)
注:次のセクションでは、ペプチドを取得し、クロマトグラフィー-タンデム質量分析を実行するために使用する 2 つのプロトコルを説明します。プロトコル 1 セル換散およびゲルの分別と消化を説明し、プロトコル 2 続いてソリューションで消化と等電点焦点法による分画溶液セル換散を説明します。
- 塩化物イオンとバッファー [234 mM トリス-HCL (pH 6.8) 7.5 %sds、37% のグリセロール、33.3% (v/v) β-メルカプトエタノール、ブロモフェノール ブルー 0.2 %w/v] のセル換散を実行します。4-12% ビス トリス アクリルアミドゲルの各サンプルの 300,000 の細胞のタンパク質と同等をロードします。
- 図 3に代表されるゲル 6 バンドに分割する各蛋白質のサンプルを許可する電気泳動の移行の期間を制御します。
- (30% エタノール + 20 分の 7.5% 酢酸)、定着液とゲルを修正し、染色液 (R-250 Coomassie 青い 45 分) を追加します。(30% エタノール + 7.5% 酢酸バンドが表示されるまで) 脱染め色液を追加をきれいなメスのタンパク バンドを切除する前に。
- サイコロ各 500 μ L 管の導入の前にバンドを摘出しました。きれいなガラス表面はケラチン (脱イオン水で 5 %sds の溶液は表面をきれいにする使用ことができます) との汚染を避けるために保証されます。
- 200 μ L の重炭酸アンモニウム 25 mM 25 mM 重炭酸アンモニウムとアセトニ トリル (50 %v/v) でワンウォッシュ続いて 37 ° C で 20 分間で 3 回のゲルのスライスを洗浄します。10 分間 100% アセトニ トリル 200 μ L 入りは、ゲルを脱水します。
- 10 mm DTT (ジチオトレイトール) 56 ° C (200 μ L) の 45 分の重炭酸アンモニウム 25 mM の室温暗闇の中で 35 分 25 mM 重炭酸アンモニウム (200 μ L) で 55 mM ヨードアセトアミド続いて各ゲル部分を孵化させなさい
- アルキル化を停止し、インキュベートした洗浄で 10 分の重炭酸アンモニウム 25 mM で 10 mM DTT の 200 μ L で各ゲル部分 25 mM 重炭酸アンモニウム、200 μ L のゲル部分 10 分間 100% アセトニ トリル 200 μ L で脱水します。
- 製造元の指示に従ってトリプシン/Lys-C ミックス 37 ° C で一晩蛋白質を消化します。
- 15 分後、15 分間 5% ギ酸の 50 μ L の 50% アセトニ トリル 50 μ L と 100% アセトニ トリル 15 分プールの最後に、50 μ L の追加によってゲル部分から生成されるペプチドを抽出し、ペプチドの吸着を抑える低吸収管に各画分を乾燥し、サンプルの損失。さらに詳しい分析まで-80 ° c のサンプルを格納します。
8. 蛋白質の抽出とソリューションの消化 (プロトコル 2)
- 次の換散バッファーの 150 μ L でセル換散 (2 x 10 の6セル) を実行します。
- 7 M 尿素、チオ尿素 2 M、40 mM、トリスとプロテアーゼ阻害剤 (完全なミニ ・ EDTA 無料プロテアーゼインヒビター カクテル) を添加した 4% チャップス。
- Thermoshaker と常温 30 分のためのソリューションをホモジナイズしてください。20 分 RT 13,800 × g で遠心し、上清を保ちます。
- 2 D のクリーン アップ キットに汚染物質を削除します。
- キットには、沈殿ソリューション、共同鉛塩法ソリューション、洗浄バッファー、および洗浄添加剤が含まれています。
- 沈殿溶液 300 μ L を加え、よく混ぜます。鉛塩法共同ソリューションの 15 分追加 300 μ L の氷の上を孵化させなさい。チューブを (少なくとも) 5 分間 12,000 × g で遠心分離機します。小さなペレットが見えるはずです。巻き上がりやペレットの分散を避けるために次のステップへ急速に進みます。ペレットを乱すことがなく上澄みを削除します。
- キャップ ヒンジとペレットが管の底に残りの液体をもたらす外向きで再びチューブを遠心します。短いパルスで十分です。必要がありますいないチューブの液残りの表示。
- ペレットを妨げないで、鉛塩法共同ソリューションの 40 μ L を追加します。5 分 5 分の遠心分離機のための氷の上に座るを削除し、破棄、洗浄チューブをしましょう。25 μ L の脱イオン水を追加します。各渦管 5-10 s。ペレットは、分散、水には溶けません。
- 1 mL の洗浄バッファー (-20 ° C で少なくとも 1 時間冷蔵前) と洗浄添加剤の 5 μ L を追加します。渦ペレットを完全に分散するまで。20-30 秒毎 10 分の少なくとも 30 分渦の-20 ° C でチューブを孵化させなさい
注: チューブは-20 ° C で最小限のタンパク質分解または変更と 1 週間まで保存できます。 - 遠心分離機管 (少なくとも) 12,000 × g で 5 分は慎重に削除し、上澄みを廃棄します。白いペレットが見えるはずです。(再懸濁しますが困難になるが、余りに乾燥ペレットの場合) 5 分以上センサーにペレットを許可します。
- 8 M 尿素と 0.1 M 炭酸水素アンモニウムの 300 μ L で蛋白ペレットを再懸濁します。1 分強の渦は、比色定量法を用いたタンパク質濃度を決定します。
9. ソリューション消化 (プロトコル 2)
- 700 mM DTT 水溶液 5.1 μ L を追加することによってジスルフィドを減らす (最終濃度 12.5 mM) 8.4 をステップから再懸濁の蛋白質と、thermoshaker で 30 分の 37 ° C で孵化させなさい。700 mM ヨードアセトアミド水溶液の 20.3 μ L を追加することによってシステイン残基をアルキレート (最終濃度 40 mM) と 25 の ° c、thermoshaker で暗闇の中で 30 分間インキュベートします。
- 0.1 M 炭酸水素アンモニウムの 990 μ L をサンプルに追加します。トリプシン/Lys-C ミックス (酵素: 基板の比 1: 100 w/w) の対応するボリュームを追加します。37 ° C、thermoshaker で一晩で孵化させなさい。
10. クリーン アップ カートリッジ (プロトコル 2)
- メタノールの 1 列-ボリューム (1 mL) のカートリッジを濡れています。1 列-水量 (1 mL) 80% アセトニ トリル/HPLC グレードのカートリッジをクリーニングし、流れを破棄します。0.1% ギ酸/HPLC グレードの水の 4 列-ボリューム (4 mL) とカートリッジを平衡させ、流れを破棄します。
- 10% ギ酸の ph (酸塩基指示薬と pH チェック) 2-3 水 90 μ L 試料を酸性化します。酸性のサンプルを読み込むし、流れを収集します。(ない保持ペプチッドを含んでいる) 流れを再読み込みします。6 列-水量 (6 mL) 0.1% ギ酸/HPLC グレードのカートリッジを洗ってください。
- 0.1% ギ酸 acid/50% アセトニ トリル/HPLC グレードの水の 1 列ボリューム (1 mL) のカートリッジからペプチドを溶出します。1.5 mL 遠心チューブに転送します。真空濃縮 (150 x g, 160 mBar で真空) を使用してサンプルを集中します。
11. 等電集中 (プロトコル 2) による分別
注: ペプチドは、3 から 10 までの pH 範囲をカバー 13 cm 帯のゲルを分留を使用して彼らの等電点に従って分かれています。(以下に要約) サプライヤーによって提供される次のプロトコルを使用しました。
- 次のソリューションの準備: ソリューション (グリセロールの解決、60 μ L OFFGEL バッファーの超純水の 4.34 mL 600 μ) とソリューション (ソリューション a 1.776 mL) と純水の 444 μ B。
- IPG のストリップ、フレーム、および製造元の指示に従って電極を組み立てます。
- 1.8 ml の液を各ウェルと B のソリューション * 追加 40 μ L のサンプルを再懸濁します。各ウェルに 150 μ L のサンプルをロードします。
- ペプチドの既定の方法を選択: OG12PE00 (ペプチド 3100 OFFGEL 低い解像度キット、pH 3-10、12 ウェル フレームで使用するための既定の方法を OFFGEL。このメソッドが完了した (~ 20 h) になるまで待ちます。正しくラベル付けされたチューブの分画を収集します。
12. クリーンアップ ハーバード装置列逆 C18 ポスト IEF (プロトコル 2)
- 徐々 に 1% の時に数 μ L を追加サンプルを酸性化する各画分をイオン交換水で TFA。Ph 試験紙、pH は 3 程度以下を使用してチェックします。
- 次のソリューションの準備: ソリューション (アセトニ トリル 5 mL、10 のギ酸の μ L、純水の 4.99 mL) 1 と解決方法 2 (0.5 mL のアセトニ トリル、ギ酸、純水の 9.49 mL の 10 μ L)。
- 事前解決策 1 の 150 μ L とスピン列を濡れています。遠心分離 90 x g と流れを破棄 750 s。ソリューション 2 の 150 μ L とスピン列を洗います。遠心分離 90 x g と流れを破棄 750 s。
- 列を端数を渡します。遠心分離 90 x g と流れを破棄 750 s。ソリューション 2 の 150 μ L で洗浄します。遠心分離 90 x g と流れを破棄 750 s。
- 解決策 1 の 50 μ L の割合を溶出します。遠心分離 90 s 750 x g にはこれらの手順をもう一度繰り返します。
- (150 x g、真空 160 mBar) 真空濃縮を使用してドライ分数、-80 ° C で保存
13. プロテオーム データおよび生物情報学の18の解析
- ナノ LC MS によって得られるデータを分析/MS 質量 MaxQuant (バージョン 1.5.2.8) など定量化ソフトウェアとアンドロメダの検索エンジンを使用しています。
- タンパク質とペプチドとアミノ酸 7 の最小の長さの両方の 1% に虚偽の発見率 (FDR) を設定します。C ターミナルとして酵素特異性を設定 Arg とリス。プロリン債で 2 逃した劈開を許可します。変数の変更として固定変更および N 末端タンパク質アセチル化およびメチオニン酸化としてシステインの carbamidomethylation を選択します。
- さらに統計解析ソフトウェアを使用してデータを分析します。FunRich ソフトウェア (www.funrich.org/) を使用して機能性濃縮分析を実行します。デビッド ・ ソフトウェア (https://david.ncifcrf.gov/) を使用して遺伝子オントロジー濃縮分析を実行します。
結果
差動遠心分離によって得られた末梢血単核球 (PBMCs) から始まって、プロトコルにより、CD14 の人口の取得+単球フローサイトメトリー (図 1) で 98% 以上の純度の評価。これらの単球は第二各種偏波 (図 2) に区別されます。ゲルの分別を選択、SDS ページのゲルの移行が目的のバンド数を取得する適応とバンドを切除 (図 3)。消化は第二のゲルの摘出のバンドで実行されますし、ペプチドを抽出します。ペプチドはナノ LC (液体クロマトグラフィー) を使用して分析-タンデム質量分析計。MS/MS スペクトルは、知られていたペプチッド (図 4 a) のスペクトルの注釈によると様々 な蛋白質のアイデンティティを与えます。豊富なタンパク質の定量は、公開されたソフトウェアとデータベース15,16を使用して蛋白質から来る特定のペプチドの量に関連して計算されます。ゲルの消化力でこのプロトコルは、約 4000 の識別された蛋白質を与えるし、ダイナミック レンジをカバー 5 の対数スケール単位 (図 4 b) が判明しています。これらの識別された蛋白質の発現の解析は、異なる酸素環境下で各種偏波のクラスタ リングを使用できます。
この方法では、我々 が調整された低酸素濃度 3% (図 5表 1) に露出されたとき蛋白質群を識別できます。消化の効率を評価するためにゲルのプロトコルを使用すると、可能ではない手法を提案ソリューションで消化人間の大食細胞 (図 6 a) に適応されています。この方法では、我々 は簡単に (後でソリューションの消化力で) 入手できます分別、分別 IEF はその分別を意味なく 3600 タンパク質の同定を増やすこの番号 (図 6 b)。

図 1: フローサイトメトリー解析 (左側のパネル) を並べ替える前に、(右側のパネル) を並べ替え後に PBMC の CD14 式の磁気ビーズを選択した後, 得られた純度を示すします。この図の拡大版を表示するのにはここをクリックしてください。

図 2: 2 つの異なる偏波の得られた形態の不均一性を示す差別化された人間の大食細胞の位相差像。スケール バーを表します 50 μ m.この図の拡大版を表示するのにはここをクリックしてください。

図 3: イメージング Coomassie 青いステンド摘出されます様々 なバンドを示す [ここでは、M(Ø) マクロファージの 6 バンド] のためのゲル低酸素環境にさらされるマクロファージの 5 偏。IC = 免疫複合体、DXM デキサメタゾンを =。この図の拡大版を表示するのにはここをクリックしてください。
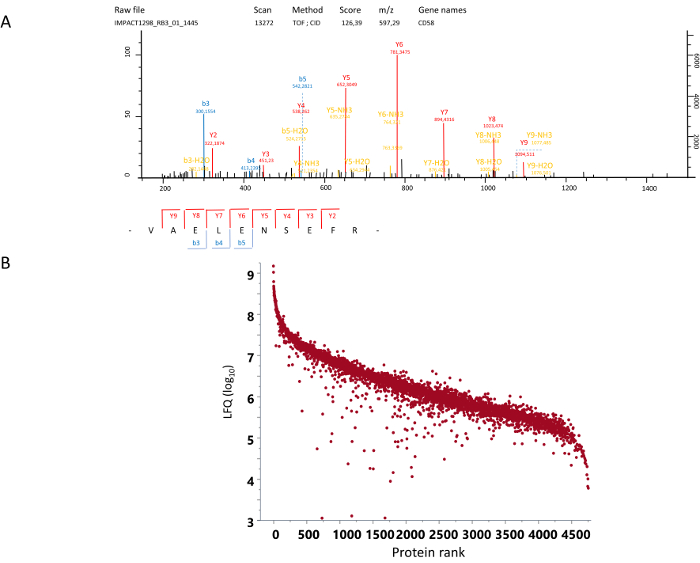
図 4: MS/MS スペクトルと定量化します。(A) MS/MS スペクトルの例。+2 の電荷を持つ MS スペクトルのm/z 597.29 は、ペプチドの CID (衝突誘起解離) スペクトルは、ここに示します。対応するシーケンスをタンパク質 CD58 から Val-Ala-Glu-Leu-Glu-Asn-Ser-Glu-Phe-Arg としてこのスペクトルから求めた。(ランク B) は、(ログ10 LFQ) 識別された蛋白質の各ラベル無料定量化を命じた。この図の拡大版を表示するのにはここをクリックしてください。
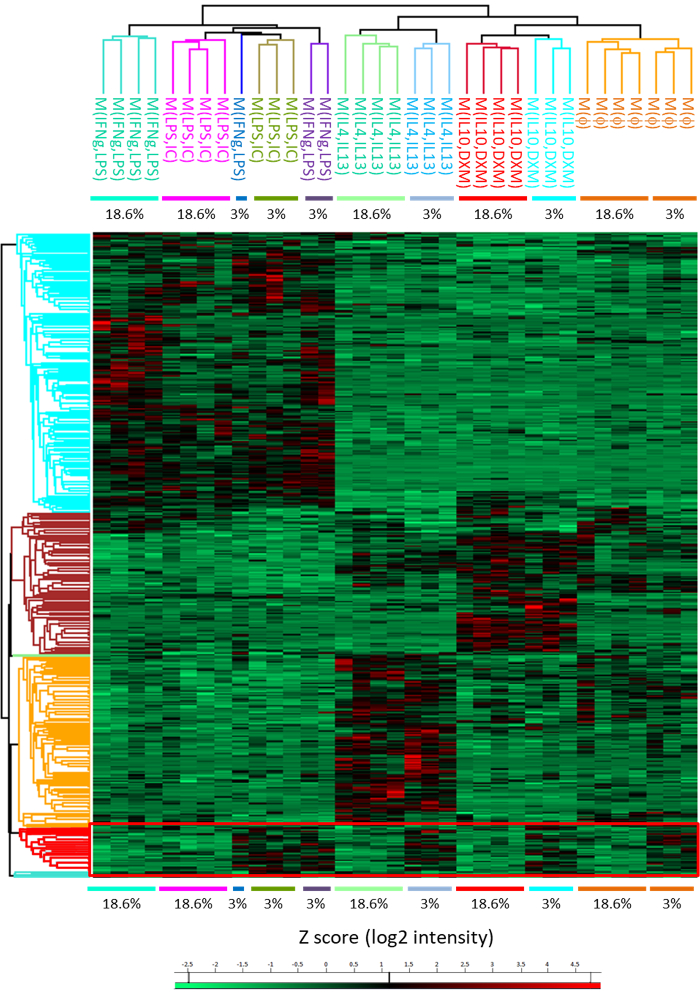
図 5: ヒート マップ全偏波の階層的クラスタ リングを表す状態特異的を使用して蛋白質を表現した。分析では、3% O2状態 (赤い四角形) ですべての偏波で過剰に発現するタンパク質群を明らかにします。カラー スケールは、z-スコア (log2 強度) を表します。各行、各列はサンプル蛋白質。この図は、以前の文書14に由来します。この図の拡大版を表示するのにはここをクリックしてください。

図 6: SDS-PAGE およびクロマト グラム。(A) 銀染色溶解中の劣化の有無と消化の効率を示すソリューションで消化後セル換散から蛋白質が付いている SDS-PAGE ゲル。(B) 分別せずソリューションで消化後から得られるクロマト グラム。この図の拡大版を表示するのにはここをクリックしてください。
| クラスター | proteinID | タンパク質名 | 遺伝子名 | ペプチド | かみそり + ユニークなペプチド | ユニークなペプチド | 蛋白質 Id | ||
| 赤 | P0DMV9 | 熱衝撃 70 kDa タンパク質 1B | HSPA1A | 40 | 37 | 4 | P0DMV9;P0DMV8;A8K5I0;Q59EJ3;B4DNT8;B4DWK5;B4DFN9;B4DI39;B4E1S9;B4DVU9;V9GZ37;B3KTT5;B4DNX1;B4E1T6;B4DNV4;Q9UQC1 | ||
| 赤 | P54709 | ナトリウム/カリウム輸送 atp アーゼ サブユニット β-3 | ATP1B3 | 8 | 8 | 8 | P54709;D3DNF9;C9JXZ1;C9JA36;H7C547;F8WBY4;Q58I18 | ||
| 赤 | O00462 | Β-マンノシダーゼ | 万場 | 14 | 13 | 13 | O00462;A7LFP5;A8K6D3;E9PFW2;B4DT18;Q59EG5 | ||
| 赤 | Q8NAS7 | NADH 脱水素酵素 [ユビキノン] 鉄硫黄タンパク質 7、ミトコンドリア | NDUFS7 | 4 | 4 | 4 | Q8NAS7;Q6ZQU6;F5H5N1;B7Z1U1;A8K0V6;Q7LD69;F5GXJ1;O75251;B7Z4P1;Q9H3K5;A0A087WXF6;A0A087WTI3;Q6ZS38 | ||
| 赤 | Q8WWQ0 | PH と相互作用するタンパク質 | PHIP | 5 | 5 | 4 | Q8WWQ0;Q9NWP3 | ||
| 赤 | E5RHK8 | ダイナミン 3 | DNM3 | 11 | 2 | 2 | E5RHK8;Q9UQ16;B3KPF2;E5RIK2 | ||
| 赤 | A0A024QZ64 | フルクトース-ビスリン酸アルドラーゼ C | ALDOC | 18 | 14 | 7 | A0A024QZ64;P09972;B7Z1Y2;B7Z3K9;B7Z1N6;B7Z3K7;A8MVZ9;J3KSV6;J3QKP5;B7Z1L5;C9J8F3;K7EKH5;J3QKK1;B7Z1H6 | ||
| 赤 | O75489 | NADH 脱水素酵素 [ユビキノン] 鉄硫黄蛋白質 3、ミトコンドリア | NDUFS3 | 12 | 12 | 12 | O75489;Q9UF24;Q53FM7;E9PS48;E9PKL8;G3V194 | ||
| 赤 | P21912 | コハク酸脱水素酵素 [ユビキノン] 鉄硫黄サブユニット、ミトコンドリア | SDHB | 11 | 11 | 11 | P21912;A0A087WXX8;A0A087WWT1 | ||
| 赤 | A0A024R1Y7 | GH3 ドメインを含むタンパク質 | LGP1;GHDC | 7 | 7 | 7 | A0A024R1Y7;Q8N2G8;B3KVB0;K7ESN3;K7EJT7;K7EQ41;K7EL54 | ||
| 赤 | E5KRK5 | ミトコンドリア NADH-ユビキノンの酸化環元酵素 75 kDa のサブユニット、 | NDUFS1 | 29 | 29 | 29 | E5KRK5;P28331;B4DJ81;Q9P1A0;C9JPQ5;F8WDL5 | ||
| 赤 | A0A024QZ30 | コハク酸脱水素酵素 [ユビキノン] フラボタンパク質サブユニット、ミトコンドリア | SDHA | 17 | 17 | 17 | A0A024QZ30;P31040;D6RFM5;B3KT34;A0A087X1I3;Q0QF12;B4DYN5;B3KYA5;B4DNB2;H0Y8X1;B7Z6J5 | ||
| 赤 | A0A024R2F9 | 膜貫通型タンパク質 43 | TMEM43 | 16 | 16 | 16 | A0A024R2F9;Q9BTV4;Q8TEP9;V9GY05 | ||
| 赤 | A0A024R5K3 | NADH 脱水素酵素 [ユビキノン] 鉄硫黄蛋白質 8、ミトコンドリア | NDUFS8 | 5 | 5 | 5 | A0A024R5K3;E9PPW7;O00217;E9PN51;F8W9K7;E9PKH6;B4DYI3;Q53G17 | ||
| 赤 | O76003 | Glutaredoxin 3 | GLRX3 | 13 | 13 | 13 | O76003 | ||
| 赤 | Q1HBJ4 | 分裂促進因子活性化タンパク質キナーゼ;分裂促進因子活性化タンパク質キナーゼ 1 | MAPK1 | 26 | 26 | 21 | Q1HBJ4;P28482;B4DHN0;Q499G7;K7ELV1;K7EN18;B4E104;P31152;Q16659 | ||
| 赤 | Q13151 | 異種核リボ核蛋白質 A0 | HNRNPA0 | 8 | 8 | 8 | Q13151 | ||
| 赤 | V9HWN7 | フルクトース-ビスリン酸アルドラーゼ A | ALDOA | 36 | 36 | 14 | V9HWN7;P04075;J3KPS3;H3BQN4;H3BUH7;H3BR04;H3BMQ8;H3BU78;H3BR68;A4UCS9;A4UCT0 | ||
| 赤 | B4DVJ0 | グルコース-6-リン酸イソメラーゼ | GPI | 32 | 32 | 23 | B4DVJ0;P06744;B4DE36;A0A0A0MTS2;K7EQ48;K7EP41;K7EPY4;K7ELR7;K7ERC6;K7ENA0;K7ESF4;K7EIL4;K7ERK8;Q59F85 | ||
| 赤 | V9HWB9 | L-乳酸デヒドロゲナーゼ;L-乳酸デヒドロゲナーゼ チェーン | 発現 | 36 | 36 | 34 | V9HWB9;P00338;B4DJI1;F5GXY2;F5GYU2;F5GXH2;F5H5J4;F5H6W8;F5GZQ4;F5H8H6;F5GXC7;F5GWW2;F5GXU1;A0A087WUM2;Q96L19;Q6ZMR3 | ||
| 赤 | P13674 | プロリル 4-ヒドロキシラーゼ サブユニット α-1 | P4HA1 | 26 | 26 | 26 | P13674;Q5VSQ6 | ||
| 赤 | Q6FHV6 | ガンマ-エノラーゼ;エノラーゼ | ENO2 | 18 | 15 | 14 | Q6FHV6;P09104;A8K3B0;F5H0C8;F5H1C3;U3KQP4;U3KQQ1;Q9NPL4 | ||
| 赤 | Q99798 | Aconitate ニトリルヒドラターゼ、ミトコンドリア | ACO2 | 40 | 40 | 40 | Q99798;B4DZ08;B2RBW5;A2A274;B4DJW1;B4DLY4;Q71UF1;B4DEC3;B4DW08;O75944 | ||
| 赤 | P17858 | ATP 依存性 6-ホスホフルクトキナーゼ、肝型 | PFKL | 32 | 29 | 28 | P17858;Q7L2M7;Q9BSP4;B3KNQ7;B4E108;Q59GI2;F8WEU2;Q7Z3R9;Q6MZK4 | ||
| 赤 | A0A024R872 | 二のようなタンパク質 1 | FAM129B | 32 | 32 | 32 | A0A024R872;Q96TA1;Q9H8K1;Q2YD88;Q9H6L6 | ||
| 赤 | A0A024RC61 | アミノペプチダーゼ N | ANPEP | 58 | 58 | 25 | A0A024RC61;P15144;Q59E93;B4DV63;B4DP01;B4DP96;H0YKT6;H0YLZ8;Q71E46;H0YMC1;Q8IVL7 | ||
| 赤 | V9HWF4 | ホスホ グリセリン酸キナーゼ;ホスホ グリセリン酸キナーゼ 1 | PGK1 | 41 | 41 | 35 | V9HWF4;P00558;B4E1H9;B4DHM5;B4DHB3;B4DWQ3;Q16444 | ||
| 赤 | Q12882 | ジヒドロピリミジン脱水素酵素 [NADP(+)] | DPYD | 44 | 44 | 44 | Q12882;B4DML1 | ||
| 赤 | B4DEQ0 | 電子伝達フラボタンパク質-ユビキノン酸化還元酵素、ミトコンドリア | ETFDH | 9 | 9 | 9 | B4DEQ0;Q547S8;A7UNU5;Q16134;D6RAD5 | ||
| 赤 | D9UAX9 | MHC のクラス I の抗原 | HLA B | 13 | 3 | 2 | D9UAX9 | ||
| 赤 | V9HWK1 | Triosephosphate 異性化酵素 | TPI1 | 29 | 29 | 17 | V9HWK1;P60174;Q53HE2;B4DUI5;U3KPZ0;U3KQF3;U3KPS5 | ||
| 赤 | Q96HE7 | ERO1 様タンパク質アルファ | ERO1L | 28 | 28 | 28 | Q96HE7;G3V3E6;G3V5B3;G3V2H0;G3V503;Q5TAE8;B2RD00;Q86YB8 | ||
| 赤 | P14868 | アスパラギン酸 - 細胞質 tRNA のリガーゼ | ダーズ | 29 | 29 | 2 | P14868;Q53T60;Q53R85;H7BZ35;H7C278 | ||
| 赤 | P36871 | ホスホグルコムターゼ 1 | PGM1 | 24 | 24 | 5 | P36871;B4DFP1;Q9H1D2 | ||
表 1: ヒトマクロファージ低酸素分圧下で各偏波に共通の過剰に表現された蛋白質のリスト。
ディスカッション
プロテオミクスは全細胞または細胞レベル下コンパートメントからの異なった蛋白質の表現を勉強する強力なツールで、セル換散のプロトコルの最適化とタンパク質の消化は、多くの研究によって対処されています。ゲルの消化力 (ポリアクリルアミド ゲルのマトリックス蛋白の消化)17、ソリューション18とフィルター支援サンプル準備19消化を含むメソッドの 3 つの主要なクラスがあります。最初は普遍的ないわばこの最後の方法は、低再現性とフィルター20の蛋白質の損失の可能性を展示する報告されています。ゲル内消化は可能であれば消化の効率を評価することは容易ではないという点で時間がかかり、不利にすることができます堅牢なメソッドです。ソリューションで消化はこの可能性を提供していますが、消化、IEF 後サンプルのクリーニングが必要です。これら 2 つのメソッドは、同じサンプル間比較されます、IEF 分別のプロトコルとソリューションの消化はよりゲル消化21(分数の同じ数) と特定されるタンパク質の数を生成します。
この利点にもかかわらず、(特に骨髄細胞) の細胞内プロテアーゼによるソリューションの換散の間に可能なタンパク質分解を考慮する必要です。これらのテクニックは、タンパク質の消化に基づいていることを覚えておくことが重要とだけ提示トリプシン特定胸の谷間サイト蛋白質を分析することができるがいます。この消化の制約を軽減が追加データ解析手順およびバイオインフォマティクス ressources22トップダウン プロテオミクスの手法を使用することが可能です。様々 な細胞コンパートメントからタンパク質の可溶化は、細胞膜、細胞のプロテオームの制御不能なサンプリングにつながるから特に入手が困難なことができます。試料のナノ-クロマトグラフィー-タンデム質量質量分析を進めるにあたっては、ペプチドは、使用する質量分析計に依存することの十分な量を得ることが重要だ (通常、総蛋白を開始条件を少なくとも 1 μ g をする必要があり、それは暗黙の IEF で使用する分数の数に応じてこの量を増加する)。この制約ゲノム技術原料の増幅が可能であるからプロテオームを区別する欠点研究されている細胞集団が不足している場合があります。
豊富な同僚の23のパッカーと Fuehr の24の精液の作品後も培養細胞における酸素の重要性認識されている十分にありません。密着性、寿命、および部門を支持低酸素濃度下で細胞を培養、今知っています。幹細胞研究25において最も重要であると認識されます。制御された酸素条件下での細胞培養の主な技術的な問題は、全体の実験中に必要な酸素濃度の保守に関連です。これは溶存酸素の放出を防ぐ (グローブ ボックスと商工会議所の処理) 低酸素下における細胞操作を許可する酸素ワーキング ステーションの使用し、高酸素状態に一時的な博覧会を防ぐためにすべてのメディアの前培養を必要とします。.
記述のプロトコルを使用して各種偏波のひと単球由来マクロファージの分子シグネチャを取得し、これらの署名に酸素変調の効果を研究します。本研究では、これらの分極の説明に洞察力を与えているし、いくつかの機能的な結果を明らかにしました。たとえば、efferocytosis に関与する多くのタンパク質が低酸素環境による変調がわかった。記述されているプロトコルに基づいて、このプロテオミクスの手法は、環境パラメーターは、マクロファージの機能を変更する方法、および新しい治療上のアプローチ14を設計するこれらの信号を使用ことができますを探索する機会を提示します。
この作品で説明したプロテオミクスの手法は人間の大食細胞の分極の研究の分野で近年中に使用されているゲノム科学のアプローチを補完するものです。プロテオミクスは、ポスト翻訳の修正のため、対応する Mrna のよりも異なる表現を提示し、新しいバイオ マーカーの発見につながる可能性のあるタンパク質定量の利点を提供しています。この利点にもかかわらず、プロテオーム データは高感度の質量分析法は、非常に複雑な MS スペクトルとペプチドの誤検出につながるため、一部を解釈する通常困難です。最近では、解析ソフトウェアは、この問題を回避するために効率を得ています。情勢の変化は、たとえプロテオミクスはまたゲノミクス26より低再現性に直面しているし、(フローサイトメトリー、イムノブロット) 蛋白質の量的な変更を確認する他の技術を使用しての検証手順に関連付けられています。表現のレベル。
開示事項
著者は利益相反を宣言しません。
謝辞
午前リーグ国立 contre le がんとラ財団アーク注ぐラ凝ったシュル ルがんラで若いグループ リーダー プログラム (ATIP/アベニール Inserm CNRS)、によって資金を供給します。生物学のプラットフォーム (UTECHS MSBIO、パスツール研究所、パリ) の質量分析からマリエット Matondo に感謝いたします。我々 はローレン ・ アンダーソン原稿の彼女の読書をありがちましょう。
資料
| Name | Company | Catalog Number | Comments |
| Hypoxia Working Station | Oxford Optronix | Hypoxylab | |
| C6 Flow cytometer | BD | Accuri C6 | |
| Urea | Agilent Technologies | 5188-6435 | |
| Formic acid (FA) | ARISTAR | 450122M | |
| R-250 Coomassie blue | Biorad | 1,610,436 | |
| Lipopolysaccharide, E.Coli (LPS) | Calbiochem | 437627 | |
| 2D clean-up kit | GE Healthcare | 80-6484-51 | |
| RPMI 1640 medium, glutamax supplement | Gibco | 61870044 | |
| HEPES 1 M | Gibco | 15630-080 | |
| MEM Non-Essential Amino Acids (NEAA) Solution 100X | Gibco | 11140-035 | |
| Phosphate Buffered Saline (PBS) 1X | Gibco | 14190-094 | |
| Harvard Apparatus column Reverse C18 micro spin column | Harvard Apparatus | 74-4601 | |
| EDTA 0.5 M, pH 8.0 | Invitrogen | AM9260G | |
| NuPAGE Bis-Tris 4-12% | Life Technologies SAS | NP0321 BOX | |
| CD14 Microbeads human | Miltenyi Biotec | 130-050-201 | |
| MACS separation column LS | Miltenyi Biotec | 130-042-401 | |
| Macrophage colony-stimulating factor (M-CSF) | Miltenyi Biotec | 130-096-485 | |
| Interleukin 4 (IL4) | Miltenyi Biotec | 130-093-917 | |
| Interleukin 13 (IL13) | Miltenyi Biotec | 130-112-410 | |
| Interferon gamma (INFγ) | Miltenyi Biotec | 130-096-482 | |
| CD14-FITC (clone TÜK4) | Miltenyi Biotec | 130-080-701 | |
| MACSmix Tube Rotator | Miltenyi Biotec | 130-090-753 | |
| Trifluoroacetic Acid (TFA) | Pierce | 28904 | |
| Trypsin/Lys-C Mix | PROMEGA | V5073 | |
| Complete Mini, EDTA-free Protease Inhibitor cocktail | Roche | 11836170001 | |
| Density Gradient Solution (Histopaque 1077) | Sigma Aldrich | 10771-100ML | |
| Accumax | Sigma Aldrich | A7089-100ML | |
| Human Serum from human male AB plasma (SAB) | Sigma Aldrich | H4522-100ML | |
| Bovine Serum Albumin (BSA) solution 30% | Sigma Aldrich | A9576-50ML | |
| Trisma-base | Sigma Aldrich | T1503 | |
| Glycerol | Sigma Aldrich | 49767 | |
| β-Mercaptoethanol | Sigma Aldrich | M3148 | |
| Bromophenol blue | Sigma Aldrich | 114405 | |
| Sodium Dodecyl Sulfate (SDS) 20% | Sigma Aldrich | 5030 | |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | |
| Acetonitrile | Sigma Aldrich | 34888 | |
| Dithiothreitol | Sigma Aldrich | 43819 | |
| Iodoacetamide | Sigma Aldrich | 57670 | |
| Thiourea | Sigma Aldrich | T8656 | |
| CHAPS | Sigma Aldrich | C9426 | |
| Micro BCA Assay Kit | ThermoFisher | 23235 | |
| 5 mL sterile plastic pipette | VWR | 612-1685 | |
| Thermomixer C Eppendorf | VWR | 460-0223 | |
| Sep-Pak tC18 reverse phase cartridges, 100 mg | Waters | WAT036820 |
参考文献
- Okabe, Y., Medzhitov, R. Tissue biology perspective on macrophages. Nature Immunology. 17 (1), 9-17 (2016).
- Sica, A., Mantovani, A. Macrophage plasticity and polarization: in vivo veritas. The Journal of Clinical Investigation. 122 (3), 787-795 (2012).
- Murray, P. J. Macrophage Polarization. Annual Review of Physiology. 79, 541-566 (2017).
- Chinetti-Gbaguidi, G., Staels, B. Macrophage polarization in metabolic disorders: functions and regulation. Current Opinion in Lipidology. 22 (5), 365-372 (2011).
- Chow, A., Brown, B. D., Merad, M. Studying the mononuclear phagocyte system in the molecular age. Nature Reviews Immunology. 11 (11), 788-798 (2011).
- Mosser, D. M., Edwards, J. P. Exploring the full spectrum of macrophage activation. Nature Reviews Immunology. 8 (12), 958-969 (2008).
- Murray, P. J., Wynn, T. A. Protective and pathogenic functions of macrophage subsets. Nature Reviews Immunology. 11 (11), 723-737 (2011).
- Ginhoux, F., Schultze, J. L., Murray, P. J., Ochando, J., Biswas, S. K. New insights into the multidimensional concept of macrophage ontogeny, activation and function. Nature Immunology. 17 (1), 34-40 (2016).
- Xue, J., et al. Transcriptome-based network analysis reveals a spectrum model of human macrophage activation. Immunity. 40 (2), 274-288 (2014).
- Csete, M. Oxygen in the cultivation of stem cells. Annals of the New York Academy of Sciences. 1049, 1-8 (2005).
- Johansson, A., et al. Functional, morphological, and phenotypical differences between rat alveolar and interstitial macrophages. American Journal of Respiratory Cell and Molecular Biology. 16 (5), 582-588 (1997).
- Pfau, J. C., Schneider, J. C., Archer, A. J., Sentissi, J., Leyva, F. J., Cramton, J. Environmental oxygen tension affects phenotype in cultured bone marrow-derived macrophages. American Journal of Physiology. Lung Cellular and Molecular Physiology. 286 (2), L354-L362 (2004).
- Grodzki, A. C. G., Giulivi, C., Lein, P. J. Oxygen tension modulates differentiation and primary macrophage functions in the human monocytic THP-1 cell line. PLoS One. 8 (1), e54926(2013).
- Court, M., Petre, G., Atifi, M. E., Millet, A. Proteomic Signature Reveals Modulation of Human Macrophage Polarization and Functions Under Differing Environmental Oxygen Conditions. Molecular & Cellular Proteomics. 16 (12), 2153-2168 (2017).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Cox, J., Neuhauser, N., Michalski, A., Scheltema, R. A., Olsen, J. V., Mann, M. Andromeda: a peptide search engine integrated into the MaxQuant environment. Journal of Proteome Research. 10 (4), 1794-1805 (2011).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols. 1 (6), 2856-2860 (2006).
- Court, M., et al. Toward a standardized urine proteome analysis methodology. Proteomics. 11 (6), 1160-1171 (2011).
- Wiśniewski, J. R., Zougman, A., Nagaraj, N., Mann, M. Universal sample preparation method for proteome analysis. Nature Methods. 6 (5), 359-362 (2009).
- Liebler, D. C., Ham, A. -J. L. Spin filter-based sample preparation for shotgun proteomics. Nature Methods. 6 (11), 785-786 (2009).
- Hubner, N. C., Ren, S., Mann, M. Peptide separation with immobilized pI strips is an attractive alternative to in-gel protein digestion for proteome analysis. Proteomics. 8 (23-24), 4862-4872 (2008).
- Park, J., et al. Informed-Proteomics: open-source software package for top-down proteomics. Nature Methods. 14 (9), 909-914 (2017).
- Richter, A., Sanford, K. K., Evans, V. J. Influence of oxygen and culture media on plating efficiency of some mammalian tissue cells. Journal of the National Cancer Institute. 49 (6), 1705-1712 (1972).
- Packer, L., Fuehr, K. Low oxygen concentration extends the lifespan of cultured human diploid cells. Nature. 267 (5610), 423-425 (1977).
- Lengner, C. J., et al. Derivation of pre-X inactivation human embryonic stem cells under physiological oxygen concentrations. Cell. 141 (5), 872-883 (2010).
- Sidoli, S., Kulej, K., Garcia, B. A. Why proteomics is not the new genomics and the future of mass spectrometry in cell biology. Journal of Cell Biology. 216 (1), 21-24 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved