Analisi dell'RNA di campioni ambientali mediante RT-PCR
Panoramica
Fonte: Laboratori del Dr. Ian Pepper e del Dr. Charles Gerba - Università dell'Arizona
Autore dimostrativo: Bradley Schmitz
La reazione a catena della trascrizione inversa-polimerasi (RT-PCR) coinvolge lo stesso processo della PCR convenzionale : temperatura ciclica per amplificare gli acidi nucleici. Tuttavia, mentre la PCR convenzionale amplifica solo gli acidi desossiribonucleici (DNA), la RT-PCR consente l'amplificazione degli acidi ribonucleici (RNA) attraverso la formazione di DNA complementare (cDNA). Ciò consente di analizzare gli organismi a base di RNA trovati nell'ambiente utilizzando metodi e tecnologie progettati per il DNA.
Molti virus presenti nell'ambiente usano l'RNA come materiale genetico. Diversi patogeni virali a base di RNA, come il Norovirus,e organismi indicatori, come il virus della chiazze lievi del pepe (PMMoV), non hanno metodi di rilevamento basati sulla coltura per la quantificazione. Al fine di rilevare la presenza di questi virus a RNA in campioni ambientali provenienti da suolo, acqua, agricoltura, ecc.,I saggi molecolari si basano sulla RT-PCR per convertire l'RNA in DNA. Senza RT-PCR, i microbiologi non sarebbero in grado di saggiare e ricercare numerosi virus a base di RNA che presentano rischi per la salute umana e ambientale.
La RT-PCR può anche essere impiegata come strumento per misurare l'attività microbica nell'ambiente. L'RNA messaggero (mRNA) è il modello a singolo filamento per la traduzione delle proteine e la misurazione dei livelli di diversi mRNA indica quali geni da cui i microbi vengono espressi nell'ambiente. L'analisi dell'espressione genica fornisce indizi su quali percorsi biologici vengono utilizzati dagli organismi per sopravvivere in diverse condizioni ambientali. In alcuni casi, l'espressione genica può essere utilizzata per determinare quali organismi possono sopravvivere meglio in condizioni difficili e hanno capacità di biorisanamento del suolo o dell'acqua contaminati.
Principi
La PCR si basa sull'amplificazione di modelli di DNA, che ne limita l'uso nel rilevamento di RNA dagli organismi. Tuttavia, RT-PCR fornisce un mezzo per utilizzare l'RNA per produrre cDNA utilizzando enzimi specializzati, noti come trascrittasi inversa (RT). Questo cDNA può quindi essere utilizzato come modello di partenza per la successiva amplificazione con PCR convenzionale (Figura 1).
La trascrizione inversa può essere controllata per amplificare solo i prodotti desiderati o un'intera comunità di acidi nucleici trovati all'interno di un campione ambientale, a seconda dei primer utilizzati per la sintesi del cDNA. Questo è importante, poiché i campioni di suolo e acqua sono spesso saturi di vari acidi nucleici che non sono desiderati per analisi specifiche. I primer casuali, che possono legarsi a sequenze di RNA presenti in qualsiasi tipo di microbi, possono essere utilizzati in RT-PCR per rilevare la maggior parte dell'RNA, in modo che il campione possa essere analizzato per la presenza e l'abbondanza relativa di più organismi nell'ambiente. D'altra parte, i primer specifici della sequenza avviano la sintesi del cDNA per sequenze precise che si trovano solo in uno o pochi organismi. Ciò consente di testare un campione ambientale per uno scopo specifico, ad esempio determinare se il Norovirus,che può causare malattie gastrointestinali nell'uomo, è presente nell'acqua.

Figura 1. Processo passo-passo di analisi RT-PCR di campioni di RNA ambientale.
Procedura
1. Raccolta del campione: campione di terreno
- Trova una posizione di esempio tramite GPS, coordinate o vista.
- Per il campionamento casuale, scegli punti casuali all'interno di un'area per ottenere un censimento generale degli habitat microbici. Il campionamento del transetto raccoglie da punti lungo una linea retta, ad esempio, adiacenti a un letto di torrente. I campioni della griglia vengono prelevati sistematicamente da punti a intervalli regolari e sono utili per mappare le comunità microbiche in un'area sconosciuta o variabile.
- Il campionamento all'interno di una profondità di 3-6 pollici (7,5-15 cm) fornisce l'accesso all'attività microbica più abbondante che è vicina, ma non all'interno, la rizosfera (la stretta regione del suolo direttamente interessata dalle radici delle piante e dai loro microrganismi associati).
- Per raccogliere il campione di terreno, spingere e ruotare una coclea a mano nel terreno alla profondità predeterminata.
- Sollevare la coclea. Il terreno si trova all'interno del gambo cavo della coclea.
- Raschiare il terreno sul fondo della coclea in un sacchetto di raccolta del terreno. Assicurati di non toccare o contaminare il terreno.
- Etichetta correttamente la borsa con posizione, nome, data e ora.
- In laboratorio, passare il terreno attraverso un setaccio di 2 mm per rimuovere ghiaia e roccia.
- Analizzare una parte del terreno per il contenuto di umidità del suolo. Per i dettagli di questo passaggio, fare riferimento al video di JoVE Science Education sull'umidità del suolo.
2. Raccolta del campione: campione d'acqua
- Trova la posizione di esempio tramite GPS, coordinate o vista.
- Raccogli l'acqua in una bottiglia di stoccaggio. Registrare il volume d'acqua raccolto; se i microbi nel campione sono analizzati quantitativamente, la concentrazione microbica può essere determinata in base al volume raccolto.
- Testare immediatamente l'acqua per eventuali parametri richiesti per l'esperimento (temperatura, pH, conducibilità, salinità, contenuto di azoto e fosforo, ecc.) utilizzando le apposite sonde elettroniche.
- Posizionare la bottiglia contenente il campione d'acqua in un dispositivo di raffreddamento con ghiaccio. Trasferire il dispositivo di raffreddamento in laboratorio.
3. Estrazione dell'RNA
- Per raccogliere e concentrare i microrganismi dal campione ambientale, fare riferimento al video Di educazione scientifica JoVE sull'estrazione di acidi nucleici della comunità.
- Estrarre l'RNA dai virus utilizzando un kit di estrazione commerciale secondo le istruzioni del produttore.
- In breve, prima mescolare i campioni con tampone di lisi integrato con etanolo, quindi aggiungere i campioni in colonne di spin.
- Centrifugare le colonne a circa 12.000 x g, scartare il flowthrough. Aggiungere il tampone di lavaggio alle colonne e centrifugare di nuovo.
- Aggiungere acqua priva di RNasi alle colonne e centrifugare per 30 s per eluire l'RNA in tubi sterili da microfuga a bassa adesione da 1,5 mL.
4. Trascrizione inversa - PCR
- Recuperare i seguenti reagenti dal congelatore a -20 °C: dNTP, buffer di trascrizione inversa concentrato(ad esempio,10x), primer (in questo esempio, primer casuali). Scongelarli sul ghiaccio o a temperatura ambiente all'interno di una cappa pulita e tenerli sul ghiaccio una volta scongelati. Recupera anche la trascrittasi inversa e l'inibitore della RNasi e tienili sul ghiaccio.
- Calcolare i volumi di reagenti necessari per realizzare un "master mix" che combini tutti i reagenti costanti tra ogni reazione (vedere tabella 1 per una reazione del campione). Preparare abbastanza master mix per ogni campione, così come un controllo positivo (utilizzando un modello di trascrizione noto) e un controllo negativo(ad esempio,senza trascrittasi inversa, con solo acqua come modello, ecc.) reazioni. Includi un ulteriore 10% in volume.
- Assemblare la miscela principale in tubi microfuga a bassa adesione da 1,5 ml. Ciò riduce al minimo il legame delle molecole di reagente alle pareti di plastica dei tubi.
- Quando i reagenti vengono scongelati, aggiungere volumi calcolati al tubo del microfuga. Vortice delicato e centrifuga ogni tubo prima dell'aggiunta. Assicurarsi di cambiare le punte delle pipette tra l'aggiunta di ciascun reagente per evitare la contaminazione.
- Dopo aver aggiunto tutti i reagenti, vortice e centrifuga la miscela principale per garantire una miscela omogenea. Rimettere i reagenti in deposito a -20 °C.
- Preparare ed etichettare strisce PCR a 8 tubi, designando una provetta per ogni campione o reazione di controllo.
- Aliquota di un volume uguale di miscela master in ciascun tubo. Quindi, aggiungere componenti specifici della reazione, come gli estratti di RNA.
- Posizionare saldamente il tappo della striscia sulla striscia PCR e centrifugare in una minicentrifuga con un adattatore per tubo strisciante per assicurarsi che tutto il liquido venga raccolto sul fondo di ciascun tubo (Figura 2).
- Posizionare saldamente la striscia PCR nel termociclatore. Premere verso il basso per assicurarsi che i tubi siano fissati.
- Impostare il termiciclo per eseguire il programma appropriato per l'RT utilizzato (vedere la Tabella 2 per un protocollo di esempio). Al termine del programma, i tubi conterranno prodotti cDNA, che potranno quindi essere sottoposti ad amplificazione PCR. Per i dettagli, fare riferimento ai video di JoVE Science Education sulla PCR e la PCR quantitativa. Conservare il cDNA a -20 °C fino all'uso.

Figura 2. Striscia a 8 tubi con cappuccio contenente miscela principale ed estratto.
| Reagente | Volume per 1 reazione (μL) |
| Buffer RT 10x | 2.0 |
| 25x dNTP | 0.8 |
| 10x Primer casuale | 2.0 |
| Multiscrivito | 1.0 |
| Inibitore della rnasi | 1.0 |
| Grado molecolare H2O | 3.2 |
| Volume totale | 10 |
Tabella 1. RT Master Mix Ingredienti.
| Fase 1 | Fase 2 | Fase 3 | Fase 4 |
| 25 °C , 10 min | 37 °C , 120 min | 85 °C , 5 min | 4 °C , ∞ |
Tabella 2. Programma termociclatore a reazione RT.
Risultati
Quando rt-PCR è completa, parte del prodotto PCR può essere separato e visualizzato su un gel di acarosio (Figura 3). In questo esempio, è stato utilizzato un primer gene-specifico per rilevare la presenza di un virus a RNA. Bande della dimensione prevista sono ottenute da due dei campioni e dalla reazione di controllo positiva, ma non dal controllo negativo, indicando la presenza di questo virus in due dei campioni di acqua sottoposti a test.
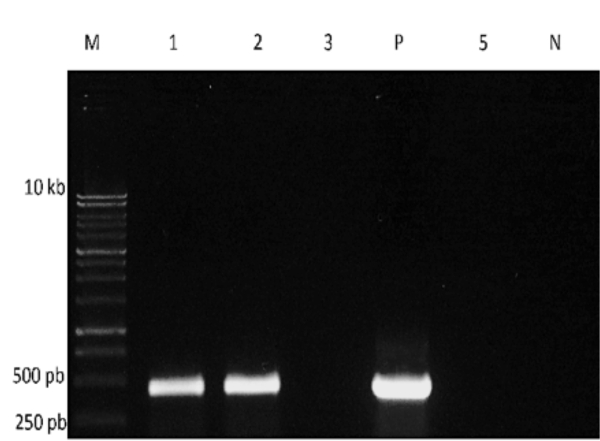
Figura 3. Elettroforesi su gel di prodotti RT-PCR. M: marcatore delle dimensioni del DNA; P: controllo positivo; N: controllo negativo. Le reazioni che utilizzavano l'RNA di quattro campioni d'acqua sono state eseguite nelle corsie 1, 2, 3 e 5.
Applicazione e Riepilogo
RT-PCR è necessario per creare cDNA da un modello di RNA. Ciò consente di analizzare i microrganismi a base di RNA utilizzando saggi molecolari sviluppati per il DNA. Una volta sintetizzato il cDNA, i saggi PCR possono determinare la presenza o l'assenza di microrganismi a base di RNA all'interno di un campione ambientale. Ciò consente ulteriori analisi a valle per determinare l'ecologia microbica, i rischi per la salute e i rischi ambientali.
RT-PCR può anche essere utilizzato per saggiare l'mRNA come mezzo per osservare quali geni vengono espressi in un ambiente. Ciò fornisce informazioni su quali proteine e percorsi i microbi fanno affidamento per sopravvivere in particolari condizioni ambientali. Le analisi di espressione genica possono identificare percorsi microbici che scompoggono i contaminanti ambientali come idrocarburi o solventi clorurati e i microbi con questi percorsi possono essere sfruttati per il biorisanamento.
La valutazione del rischio incorpora la RT-PCR al fine di analizzare i rischi per la salute umana e ambientale. La combinazione di RT con PCR quantitativa consente di enumerare i virus a RNA all'interno dei campioni, in modo che l'esposizione umana e ambientale possa essere calcolata ai fini della valutazione quantitativa del rischio microbico (QMRA).
Vai a...
Video da questa raccolta:

Now Playing
Analisi dell'RNA di campioni ambientali mediante RT-PCR
Environmental Microbiology
40.4K Visualizzazioni

Determinazione del contenuto di umidità del suolo
Environmental Microbiology
359.4K Visualizzazioni

Tecnica asettica nelle scienze ambientali
Environmental Microbiology
126.4K Visualizzazioni

Colorazione di Gram dei batteri provenienti da fonti ambientali
Environmental Microbiology
100.2K Visualizzazioni

Visualizzazione dei microrganismi del suolo tramite il test su vetrino a contatto e microscopia
Environmental Microbiology
42.2K Visualizzazioni

Funghi filamentosi
Environmental Microbiology
57.3K Visualizzazioni

Estrazione del DNA di comunità da colonie batteriche
Environmental Microbiology
28.8K Visualizzazioni

Rilevamento dei microrganismi ambientali tramite PCR ed elettroforesi su gel
Environmental Microbiology
44.6K Visualizzazioni

Quantificazione di microrganismi e virus ambientali mediante qPCR
Environmental Microbiology
47.8K Visualizzazioni

Analisi della qualità dell'acqua tramite organismi indicatori
Environmental Microbiology
29.5K Visualizzazioni

Isolamento di batteri fecali da campioni d'acqua mediante filtrazione
Environmental Microbiology
39.3K Visualizzazioni

Rilevamento di batteriofagi in campioni di matrici ambientali
Environmental Microbiology
40.7K Visualizzazioni

Coltura e conta di batteri da campioni di suolo
Environmental Microbiology
184.3K Visualizzazioni

Analisi della curva di crescita batterica e sue applicazioni ambientali
Environmental Microbiology
296.0K Visualizzazioni

Enumerazione delle alghe tramite metodo di coltivazione
Environmental Microbiology
13.8K Visualizzazioni