Estrazione del DNA di comunità da colonie batteriche
Panoramica
Fonte: Laboratori del Dr. Ian Pepper e del Dr. Charles Gerba - Università dell'Arizona
Autore dimostrativo: Luisa Ikner
I metodi tradizionali di analisi per le comunità microbiche all'interno dei suoli hanno solitamente coinvolto saggi culturali che utilizzano la metodologia di diluizione e placcatura su mezzi selettivi e differenziali o saggi di conteggio diretto. I conteggi diretti offrono informazioni sul numero totale di batteri presenti, ma non forniscono informazioni sul numero o sulla diversità delle popolazioni presenti all'interno della comunità. I conteggi delle piastre consentono l'enumerazione delle popolazioni culturali totali o selezionate e quindi forniscono informazioni sulle diverse popolazioni presenti. Tuttavia, poiché meno dell'1% dei batteri del suolo sono facilmente coltivabili, le informazioni culturali offrono solo una parte del quadro. La frazione effettiva della comunità che può essere coltivata dipende dal mezzo scelto per i conteggi culturali. Ogni singolo mezzo selezionerà per le popolazioni che sono più adatte a quel particolare mezzo.
Negli ultimi anni, i vantaggi di studiare il DNA della comunità estratto da campioni di suolo sono diventati evidenti. Si ritiene che questo approccio non basato sulla cultura sia più rappresentativo della comunità reale presente rispetto agli approcci basati sulla cultura. Oltre a fornire informazioni sui tipi di popolazioni presenti, questo approccio può anche fornire informazioni sul loro potenziale genetico. Come con qualsiasi tecnica, ci sono limitazioni ai dati che possono essere ottenuti con l'estrazione del DNA. Pertanto, molti ricercatori ora utilizzano l'estrazione del DNA in combinazione con conteggi diretti e culturali per massimizzare i dati ottenuti da un campione ambientale.
Principi
L'estrazione del DNA dal suolo può essere condotta in due modi (Tabella 1). Nel metodo in situ, viene utilizzata una combinazione di tecniche chimiche e meccaniche. Per questa estrazione, una massa di terreno è combinata con un volume equivalente di un tampone di estrazione. Le perle di vetro vengono quindi aggiunte alla sospensione insieme a un volume di detergente (in genere viene utilizzato il solfato di dodecile di sodio, o SDS) e il campione viene miscelato per facilitare la separazione dalle particelle del suolo seguita da incubazione a una temperatura elevata per promuovere la lisi cellulare. Dopo la centrifugazione, il surnatante viene sottoposto a ulteriori fasi di estrazione e incubazione al fine di purificare il prodotto DNA.
In alternativa, le cellule possono prima essere frazionate (o separate) dalla matrice del suolo prima dell'estrazione del materiale genetico. Una massa di campione di terreno subisce cicli successivi di miscelazione e centrifugazione lenta. La fase di battitura delle perle viene eliminata qui, tuttavia, al fine di mantenere intatte le cellule, che vengono centrifugate per ottenere un pellet. Un'estrazione a base di lisozima viene quindi eseguita in concomitanza con l'incubazione per interrompere le pareti cellulari e liberare il DNA per la purificazione.
Questo manoscritto e video dimostreranno il metodo in situ di estrazione del DNA dal suolo, poiché questa procedura ha dimostrato di produrre maggiori concentrazioni di DNA da campioni di suolo rispetto al metodo di frazionamento cellulare.
| Questione | Frazionamento batterico | In situ Lisi |
| Resa del DNA | 1-5 μg/g | 1-20 μg/g |
| Rappresentante della comunità | Meno rappresentativo a causa dell'assorbimento cellulare | Assorbimento cellulare più rappresentativo e non influenzato |
| Fonte di DNA recuperato | Solo batteri | Per lo più batteri ma anche funghi e protozoi |
| Grado di tranciatura del DNA | Meno cesoiatura | Più cesoiatura |
| Dimensione media dei frammenti di DNA | 50 kb | 25 kb |
| Grado di contaminazione utica | Meno contaminato | Più contaminato |
| Facilità di metodologia | Basso, laborioso | Più veloce, meno laborioso |
Tabella 1. Confronto tra metodologie di frazionamento batterico e lisi in situ per il recupero del DNA dal suolo.
Procedura
1. Estrazione del DNA della comunità batterica
- Per iniziare la procedura, pesare 100 g di terreno setacciato. Aggiungi questo a un recipiente di polipropilene e aggiungi 100 ml di tampone di estrazione composto da tampone Tris modificato con EDTA per promuovere il rilascio di batteri dalla matrice del suolo, quindi stringi la mano.
- Quindi, pesare 100 g di perle di vetro e aggiungerle al recipiente di miscelazione. Agitare il campione per 5 minuti utilizzando un dispositivo di battitura delle pere o uno shaker meccanico ad azione da polso per 15 minuti. Aggiungere 10 ml di dodecil solfato di sodio al 20%, o SDS, alla miscela, quindi agitare per un ulteriore minuto. Incubare ad una temperatura elevata di 60 - 65 °C per 60 min.
- Distribuire equamente il campione tra tubi separati da 50 ml e centrifugare per 10 minuti a 6.000 x g. Trasferire il surnatante dai tubi in un unico contenitore sterile. Quindi, ripetere l'estrazione sul pellet di terreno come descritto in precedenza, utilizzando un volume fresco di tampone di estrazione.
- Quindi, aggiungere il volume totale del surnatante lavorato, circa 200 ml, in un tubo pulito da 50 ml riempito a metà volume con una soluzione di polietilenglicole al 30% e cloruro di sodio 1,6 M. Invertire le bottiglie più volte a mano per mescolare e incubare a temperatura ambiente per 2 ore. Centrifugare i campioni a 10.000 x g per 20 minuti per pellettizzare il DNA.
- Rimuovere accuratamente il surnatante dal tubo della centrifuga, lasciando dietro di sé il pellet di acido nucleico parzialmente purificato. Aggiungere 20 mL di TE Buffer e 1,5 mL di una soluzione di acetato di potassio da 7,5 M per sospendare il pellet, quindi vortice. Posizionare la sospensione sul ghiaccio per 5 min. Centrifugare a 16.000 x g per 30 min a 4 °C per precipitare proteine e polisaccaridi.
- Quindi, aggiungere una RNAsi e una proteinasi K al campione, mescolare delicatamente a mano e lasciare riposare per un momento. Aggiungere un volume equivalente di fenolo:cloroformio:alcool isoamilico (rapporto miscela di 25:24:1) alla sospensione da estrarre e mescolare delicatamente a mano. Centrifugare la preparazione per 10 min a 13.000 x g. Rimuovere con attenzione il recipiente dalla centrifuga e annotare i due strati.
- Lo strato inferiore, più pesante, è composto dal fenolo: cloroformio: alcool isoamilico e detriti estratti, e lo strato superiore è l'acquoso e contiene il DNA. Posizionare la fase acquosa in un recipiente sterile, aggiungere un volume equivalente di isopropanolo e invertire delicatamente per avviare la precipitazione del DNA. Incubare la sospensione a temperatura ambiente per 2 ore. Pellet il DNA purificato mediante centrifugazione a 16.000 x g per 30 min. Rimuovere con attenzione il surnatante poiché il DNA pellettato può o non può essere visibile sul fondo del vaso, quindi ricaspendare in 1 mL di TE Buffer.
- Utilizzando uno spettrofotometro o un fluorimetro di quantificazione DNA/RNA, misurare il livello di DNA estratto dal campione. La quantità di DNA è stimata dalla lettura di 260 nm. Una lettura di assorbanza di 1,0 equivale a 50 μg di DNA per mL di soluzione. Se la concentrazione è troppo alta per letture accurate, diluire la sospensione da 1 a 10 o da 1 a 100 utilizzando acqua di grado molecolare.
- La purezza del DNA è stimata dal rapporto tra la lettura a 260 nm a quella a 280 nm. Un valore > 1,7 indica DNA relativamente puro. Il valore teorico massimo è 2.0.
Applicazione e Riepilogo
Il DNA comunitario proveniente da colonie coltivate o estratto dal suolo può essere sottoposto ad approcci bioinformatici e "omici" che consentono la caratterizzazione dei batteri originali all'interno del campione. Gli approcci omici includono la metagenomica – determinazione di "chi" è all'interno della comunità tramite il sequenziamento dell'rRNA 16S. Questo dà una stima della diversità all'interno della comunità.
È anche possibile calcolare il numero di cellule batteriche nel campione di terreno originale. Il DNA comunitario viene estratto da un suolo e quantificato mediante analisi spettroscopiche. La quantità stimata di DNA misurata come μg di DNA per mL di soluzione è correlata al volume totale di DNA estratto in soluzione per dare una quantità totale di DNA per g di suolo. Conoscendo il valore teorico del DNA per cellula, è possibile calcolare il numero totale di cellule per g di suolo.
Esempio
Un suolo ha 0,12 μg di DNA per g di terreno
Se ogni cellula ha 4 fg di DNA
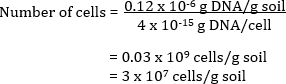
Il DNA della comunità estratto può essere sottoposto ad analisi PCR utilizzando primer specifici per determinare se una particolare specie è presente all'interno della comunità. Gli esempi includono specifici patogeni batterici come Clostridium perfringens o Bacillus anthracis.
Vai a...
Video da questa raccolta:

Now Playing
Estrazione del DNA di comunità da colonie batteriche
Environmental Microbiology
28.8K Visualizzazioni

Determinazione del contenuto di umidità del suolo
Environmental Microbiology
359.3K Visualizzazioni

Tecnica asettica nelle scienze ambientali
Environmental Microbiology
126.4K Visualizzazioni

Colorazione di Gram dei batteri provenienti da fonti ambientali
Environmental Microbiology
100.2K Visualizzazioni

Visualizzazione dei microrganismi del suolo tramite il test su vetrino a contatto e microscopia
Environmental Microbiology
42.2K Visualizzazioni

Funghi filamentosi
Environmental Microbiology
57.3K Visualizzazioni

Rilevamento dei microrganismi ambientali tramite PCR ed elettroforesi su gel
Environmental Microbiology
44.6K Visualizzazioni

Analisi dell'RNA di campioni ambientali mediante RT-PCR
Environmental Microbiology
40.4K Visualizzazioni

Quantificazione di microrganismi e virus ambientali mediante qPCR
Environmental Microbiology
47.8K Visualizzazioni

Analisi della qualità dell'acqua tramite organismi indicatori
Environmental Microbiology
29.5K Visualizzazioni

Isolamento di batteri fecali da campioni d'acqua mediante filtrazione
Environmental Microbiology
39.3K Visualizzazioni

Rilevamento di batteriofagi in campioni di matrici ambientali
Environmental Microbiology
40.7K Visualizzazioni

Coltura e conta di batteri da campioni di suolo
Environmental Microbiology
184.3K Visualizzazioni

Analisi della curva di crescita batterica e sue applicazioni ambientali
Environmental Microbiology
295.9K Visualizzazioni

Enumerazione delle alghe tramite metodo di coltivazione
Environmental Microbiology
13.8K Visualizzazioni