Method Article
Applicazione della colorazione dei mitocondri vivi nella selezione cellulare per purificare gli epatociti derivati da cellule staminali pluripotenti indotte umane
In questo articolo
Riepilogo
Gli epatociti derivati da cellule staminali pluripotenti possono essere purificati attraverso la selezione cellulare, utilizzando una combinazione di colorazione con molecole di adesione alle cellule mitocondriali e leucocitarie attivate (ALCAM, nota anche come CD166).
Abstract
Le cellule staminali embrionali umane (ES) e le cellule staminali pluripotenti indotte (iPS) hanno potenziali applicazioni nella medicina rigenerativa basata sulle cellule per il trattamento di organi gravemente malati grazie alla loro proliferazione illimitata e alle loro proprietà pluripotenti. Tuttavia, la differenziazione delle cellule ES/iPS umane in tipi di cellule bersaglio pure al 100% è impegnativa a causa della loro elevata sensibilità all'ambiente. La tumorigenesi dopo il trapianto è causata da cellule contaminate, proliferanti e indifferenziate, rendendo la tecnologia ad alta purificazione essenziale per la realizzazione sicura della medicina rigenerativa. Per mitigare il rischio di tumorigenesi, è stata sviluppata una tecnologia ad alta purificazione per gli epatociti derivati da cellule iPS umane. Il metodo utilizza FACS (fluorescence-activated cell sorting) utilizzando una combinazione di alto contenuto mitocondriale e il marcatore di superficie cellulare ALCAM (activated leukocyte cell adhesion molecule) senza modificazioni genetiche. Il 97% ± lo 0,38% (n = 5) degli epatociti purificati utilizzando questo metodo hanno mostrato espressione proteica di albumina. Questo articolo si propone di fornire procedure dettagliate per questo metodo, come applicato al più recente metodo di differenziazione bidimensionale per cellule iPS umane in epatociti.
Introduzione
Le cellule staminali embrionali e pluripotenti indotte (ES e iPS, rispettivamente) sono considerate fonti cellulari promettenti per le terapie rigenerative. Tuttavia, l'efficienza della differenziazione di queste cellule in specifici tipi di cellule bersaglio può variare, anche quando si utilizza la stessa linea cellulare, lo stesso protocollo e lo stesso sperimentatore 1,2,3,4. Questa variabilità può essere attribuita all'elevata sensibilità delle cellule ES/iPS umane al loro ambiente. Pertanto, attualmente è difficile ottenere costantemente cellule bersaglio pure. Per ottenere una medicina rigenerativa altamente sicura, è fondamentale eliminare le cellule proliferative e le cellule staminali indifferenziate nelle cellule terapeutiche ed è essenziale una tecnologia avanzata di purificazione per le cellule bersaglio 5,6,7.
Un selezionatore di cellule è un dispositivo che analizza istantaneamente le singole cellule e ordina le cellule vive di interesse in base all'intensità del segnale fluorescente, offrendo una soluzione promettente. Ciò può essere ottenuto attraverso la colorazione con anticorpi di marcatori di superficie specifici per il tipo di cellula o utilizzando espressioni geniche reporter specifiche per il tipo di cellula. Utilizzando questa tecnica, ci sono diversi rapporti su metodi per purificare i cardiomiociti 8,9,10 e gli epatociti11,12 derivati da cellule staminali pluripotenti. Hattori et al. hanno sviluppato un innovativo metodo di purificazione mitocondriale utilizzando un selezionatore di cellule13. Sfruttando il fatto che i cardiomiociti hanno un'elevata richiesta di energia attraverso l'attività mitocondriale, la colorazione delle cellule con il colorante mitocondriale vivo TMRM (estere metilmetilico-rodamina) può essere utilizzato per marcare e purificare altamente i cardiomiociti mediante FACS da corpi embrioidi derivati da cellule ES umane contenenti vari tipi di cellule. L'assenza di tumorigenicità è stata confermata da saggi di formazione di teratomi con i cardiomiociti purificati. Inoltre, Yamashita et al. hanno inaspettatamente scoperto un metodo per purificare gli epatociti da corpi embrioidi derivati da cellule ES umane isolando frazioni con elevata attività mitocondriale ed espressione ALCAM-positiva14. Il razionale di questo metodo è che gli epatociti hanno anche un numero relativamente alto di mitocondri a causa del loro elevato consumo di ATP per il metabolismo dei nutrienti e la disintossicazione15, e gli epatociti esprimono ALCAM, un membro della superfamiglia delle immunoglobuline, che svolge un ruolo nell'adesione e nella migrazione cellulare16.
I precedenti metodi altamente purificanti per gli epatociti derivati da cellule staminali pluripotenti richiedevano modificazioni genetiche e i metodi di purificazione non genetici avevano una bassa efficienza17. Il metodo non genetico mitocondriale ha il merito di raggiungere un'elevata purezza. Quando sono necessarie cellule progenitrici epatiche, è possibile scegliere i metodi CD133 e CD13 o Dlk1 18,19. Sebbene l'accuratezza della tecnologia di editing del genoma sia avanzata, il potenziale rischio di cambiamenti genomici imprevisti (ad esempio, la cancerogenesi) non può essere completamente eliminato. I metodi basati sull'attività mitocondriale, senza comportare modificazioni genetiche, possono essere esenti da tali rischi.
Come risultato dell'esame di vari indicatori mitocondriali nei cardiomiociti neonatali di ratto, è stato osservato che il TMRM scompare completamente entro 24 ore, mentre altri coloranti sono rimasti per almeno 5 giorni13. Inoltre, è importante notare che TMRM e JC-1 non hanno influenzato la vitalità cellulare quando valutati con il test 3-(4,5-dimetil-tiazol-2-il)-2,5-difeniltetrazolio bromuro (MTT), mentre altri coloranti hanno dimostrato effetti variabili sulla vitalità cellulare13. TMRM dimostra una maggiore sicurezza.
Ad oggi, sono stati sviluppati diversi metodi di differenziazione bidimensionale (2D) come approcci più efficienti per indurre la differenziazione rispetto alla formazione di corpi embrioidi tridimensionali (3D). Questo perché i composti o le citochine che inducono la differenziazione graduale possono essere somministrati uniformemente alle cellule su un piano 2D, piuttosto che in uno spazio 3D. In questo studio, il metodo20 precedentemente riportato è stato modificato per indurre la differenziazione in epatociti derivati da cellule iPS umane. Qui vengono descritti i dettagli delle procedure per la differenziazione e la purificazione 2D aggiornate di epatociti derivati da cellule iPS umane.
Protocollo
Questo studio ha utilizzato cellule iPS umane ottenute commercialmente (ceppo 253G1) (vedi Tabella dei materiali).
1. Mantenimento delle cellule iPS umane
- Mantenere le cellule iPS umane in condizioni prive di alimentatore in terreno AK02 su piastre di coltura rivestite con iMatrix-511 da 0,25 μg/cm2 (vedere la tabella dei materiali).
2. Differenziamento epatico di cellule iPS umane in colture 2D
NOTA: Fare riferimento alla Figura 1A per il programma giornaliero della differenziazione epatica. Durante tutto il processo, utilizzare piastre di coltura a 6 pozzetti e aggiungere 2 ml del terreno di lavaggio o coltura designato a ciascun pozzetto. Mantenere le condizioni di coltura cellulare al 100% di umidità, 5% di CO2 e 20% di O2 a 37 °C.
- Rivestire ciascun pozzetto di una piastra a 6 pozzetti con 1 mL di matrice a membrana basale (diluita 1/25 in PBS (-)) e incubare a 37 °C per 1 ora.
- Passaggio di cellule iPS umane sulla piastra rivestita a una densità di 20.000-30.000 cellule/cm² utilizzando un terreno AK02N integrato con un inibitore ROCK da 10 μM (Y-27632) (vedi Tabella dei materiali). Sostituire il terreno senza Y-27632 il giorno successivo e la coltura per altri 2 giorni.
- Lavare le celle una volta con il terreno RPMI-1640. Avviare la differenziazione dell'endoderma sostituendo il terreno con RPMI-B27-Glu (RPMI-1640 + 2% Insulina B27 + 1% Dipeptide L-Alanil-L-Glutammina) integrato con 3-6 μM di CHIR-99021 e 100 ng/mL di Activina A per 24 ore (vedi Tabella dei Materiali).
- Sostituire il terreno con RPMI-B27-Glu integrato con 50 ng/mL di Activina A per 24 ore.
- Per indurre la differenziazione dei progenitori epatici, sostituire il terreno con RPMI-B27-Glu integrato con dimetilsolfossido all'1% (DMSO) per 5 giorni.
- Per promuovere la maturazione degli epatociti, passare al terreno di maturazione degli epatociti: terreno L-15 di Leibovitz contenente 10 μM di idrocortisone 21-emiuccinato, 8,3% di brodo di triptosio fosfato, 100 nM di desametasone (DEX), 50 μg/mL di sodio-L-ascorbato, 0,58% di insulina-transferrina-selenio (ITS), 8,3% di siero fetale bovino (FBS), 2 mM di Dipeptide L-Alanil-L-Glutammina e 100 nM di N-Hexanoic-Try-Ile-(6)-ammino esanoide (Dihexa) (vedi Tabella dei materiali).
- Cambia il mezzo ogni 2 giorni per 20 giorni. Ordinare le cellule circa 27 giorni dopo l'inizio della differenziazione.
3. Preparazione cellulare per la purificazione di epatociti derivati da cellule iPS umane
- Lavare le cellule differenziate epatiche con 2 mL di PBS (-) per pozzetto.
- Aggiungere 1 mL di soluzione enzimatica per pozzetto, che include 0,1% di collagenasi, 0,083% di tripsina, 10 μM di inibitore ROCK (Y-27632), 20 nM di ciclosporina A e 50 μg/mL di sodio-L-ascorbato nel tampone Ads (116 mM di NaCl, 12,5 mM di NaH2PO4, 20 mM di HEPES, 5,4 mM di KCl, 5,6 mM di glucosio e 0,8 mM di MgSO4; pH 7,35) (vedere la Tabella dei materiali). Staccare le celle dalle piastre ruotandole orizzontalmente a 37 °C per circa 2 ore.
- Dopo aver verificato che le cellule sono staccate e arrotondate al microscopio, disperderle in singole cellule mediante pipettaggio delicato e raccoglierle in una provetta da 50 ml.
- Centrifugare le celle a 200 x g per 5 minuti a 18 °C per pellettizzarle. Rimuovere il surnatante utilizzando un aspiratore.
- Colorare i mitocondri delle cellule disperdendole in 5 mL di terreno di maturazione degli epatociti contenente 100 nM di TMRM (tetrametilrodamina, estere di methl) (vedi Tabella dei materiali). Incubare per 30 minuti a 37 °C proteggendo dalla luce avvolgendo in un foglio di alluminio.
- Centrifugare le celle a 200 x g per 5 minuti a 18 °C per pellettizzarle. Rimuovere il surnatante.
- Risospendere le cellule con 1-5 mL di tampone Ads freddo contenente il 2% di FBS.
- Contare il numero di cellule utilizzando una soluzione di blu di tripano allo 0,4% e una piastra per il conteggio delle cellule.
- Centrifugare le celle a 200 x g per 5 minuti a 4 °C per pellettizzarle. Rimuovere il surnatante.
- Diluire un anticorpo anti-ALCAM (anticorpo primario, vedere la Tabella dei materiali) in un rapporto 1:50 nel tampone Ads e aggiungere 100 μl di anticorpo diluito per 106 cellule. Colorare le celle per 50 minuti con ghiaccio. Preparare le cellule che non sono colorate con l'anticorpo primario come controllo negativo.
- Lavare due volte le celle con tampone Ads freddo contenente il 2% di FBS.
- Diluire un Alexa Fluor 488 donkey anti-goat IgG (anticorpo secondario, vedere la Tabella dei materiali) in un rapporto 1:100 nel buffer Ads e aggiungere 100 μl di anticorpo diluito per 106 cellule. Colorare le celle per 30 minuti con ghiaccio. Colorare le cellule che non sono state colorate con l'anticorpo primario utilizzando l'anticorpo secondario.
- Lavare due volte le celle con tampone Ads freddo contenente il 2% di FBS.
- Risospendere le celle nel buffer Ads e filtrarle attraverso un filtro a scatto (maglia da 35 μm) appena prima di ordinarle in base al FACS per rimuovere grandi grumi di celle e detriti.
4. Analisi FACS e smistamento di epatociti derivati da cellule iPS umane
- Impostare la macchina FACS per lo smistamento delle celle seguendo le istruzioni del produttore (vedere la tabella dei materiali).
NOTA: Utilizzare un ugello da 85 μm o 100 μm e un filtro ND da 2,0 per gli epatociti. Utilizza il canale FITC per rilevare Alexa Flour 488 per ALCAM e il canale PE per rilevare la fluorescenza TMRM per i mitocondri. - Regolare le tensioni FSC (forward scatter) e SSC (side scatter) per visualizzare correttamente la popolazione cellulare.
- Regolare le tensioni dei canali fluorescenti FITC e PE. Inizia con le celle non etichettate e centra la popolazione cellulare nella parte inferiore di ogni grafico del canale. Assicurati che le celle etichettate siano posizionate in modo appropriato all'interno del grafico.
- Usa una strategia di gating comune per eliminare i doppietti: Gate il principale FSC-A vs. Popolazione SSC-A (Figura 2A), seguita da FSC-H vs. -W, e poi SSC-H vs. -W (Figura 2B,C).
- Gate la popolazione TMRMhi e ALCAM+ (P2 in Figura 2E) per selezionare gli epatociti. Gate della popolazione TMRMhi e ALCAM- (P1 in Figura 2E) per selezionare i non epatociti. Raccogliere gli epatociti e i non epatociti selezionati in provette da 15 mL contenenti terreno di maturazione degli epatociti.
- Centrifugare la provetta di raccolta contenente le cellule selezionate a 200 x g per 5 minuti a 18 °C.
- Rimuovere il surnatante e risospendere le cellule nel terreno di maturazione degli epatociti.
- Seminare le cellule su fibroblasti embrionali di topo (MEF) trattati con mitomicina C o su una matrice di membrana basale gelatinosa con terreno di maturazione degli epatociti integrato con 10 μM di Y-27632 e 20 nM di ciclosporina A. Coltivare le cellule in un incubatore a 37 °C di CO2 per 5 giorni.
5. Immunocitochimica per la rilevazione di epatociti derivati da cellule iPS umane
- Lavare le cellule coltivate per 5 giorni dopo la cernita con PBS (-).
- Fissare le cellule con paraformaldeide al 4% per 5 minuti a temperatura ambiente.
- Lavare due volte le cellule con tampone TBS-T (1x TBS con 1% Tween 20) per rimuovere la paraformaldeide.
- Incubare le cellule con tampone TBS-T diluito Triton X-100 allo 0,1% per 5 minuti a temperatura ambiente.
- Lavare due volte le celle con il tampone TBS-T per rimuovere Triton X-100.
- Aggiungere la soluzione bloccante disponibile in commercio (vedere la Tabella dei materiali) alle celle per 30 minuti a temperatura ambiente.
- Aggiungere anticorpi primari contro l'albumina (marcatore degli epatociti) e l'antigene nucleare umano (hNA, vedere la Tabella dei materiali), diluiti 1:50 in soluzione bloccante.
- Incubare le cellule a 4 °C per una notte. Lavare due volte le celle con il tampone TBS-T.
- Aggiungere anticorpi secondari: Alexa Fluor 488 donkey anti-rabbit IgG e Alexa Fluor 546 donkey anti-mouse IgG, diluiti 1:100 in soluzione bloccante (vedi Tabella dei materiali).
- Incubare le cellule a temperatura ambiente per 30 minuti. Lavare due volte le celle con il tampone TBS-T.
- Valutare la purezza degli epatociti contando le cellule albumina-positive e hNA-positive al microscopio a fluorescenza. Calcolare la percentuale di cellule positive all'albumina per cellula hNA-positiva per entrambe le popolazioni P1 e P2.
Risultati
Viene mostrata la cronologia dei processi che inducono le cellule iPS umane a differenziarsi in epatociti attraverso la coltura 2D (Figura 1A) e le caratteristiche cellulari rappresentative (Figura 1B). Approssimativamente il 12° giorno di differenziazione, le cellule hanno iniziato a mostrare forme cellulari poligonali e nuclei rotondi, caratteristici degli epatociti. Alcuni epatociti mostravano anche multinucleazione.
L'analisi FACS è stata eseguita utilizzando le cellule il giorno 27 di differenziazione. Per identificare la popolazione ALCAM+ , i grafici comparativi con e senza il primo anticorpo anti-ALCAM sono mostrati rispettivamente nella Figura 2D, E. Inducendo la differenziazione epatica attraverso la coltura 2D, la popolazione di epatociti (P2) è risultata essere del 10,1% in questo esperimento, rappresentando un aumento significativo rispetto allo 0,8% (dati non mostrati) trovato utilizzando il metodo di formazione del corpo embrioide 3D.
Le cellule P1 e P2 che erano state selezionate sono state coltivate su fibroblasti embrionali di topo trattati con mitomicina C. Le cellule P2 mostravano una formazione di colonie sferiche compatte, mentre le cellule P1 erano disperse e avevano una morfologia più piatta. La colorazione immunoistochimica per l'antigene nucleare umano (hNA) e l'albumina hanno confermato l'arricchimento di cellule simili agli epatociti in P2, come illustrato nella Figura 3. Per valutare la purezza degli epatociti nelle cellule P1 e P2, è stato calcolato il rapporto tra le cellule positive all'albumina e le cellule hNA-positive (n = 5). I risultati hanno rivelato che lo 0% delle cellule P1 e il 97% ± lo 0,38% delle cellule P2 derivavano da iPSC umane e mostravano caratteristiche simili agli epatociti (Figura 3).
Per confermare il successo dello smistamento degli epatociti durante l'esperimento FACS, si raccomanda di co-colorare ASGR1 con il suo anticorpo specifico coniugato con diversi fluorofori e confermare che P2 contiene cellule ASGR1-positive.
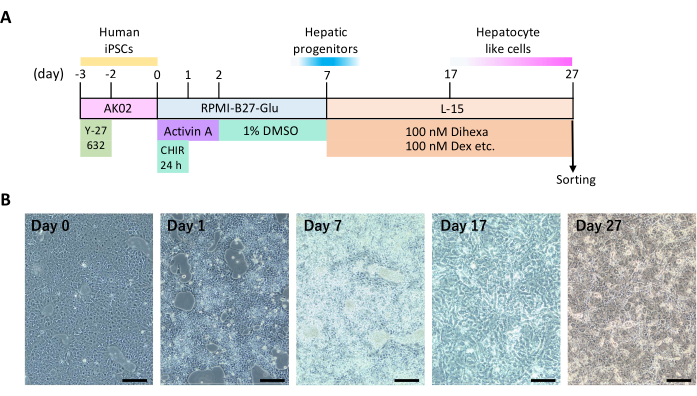
Figura 1: Differenziazione epatica 2D di iPSC umane. (A) La cronologia del differenziamento epatico. Questa figura è adattata da Yamashita et al.14. (B) Modificazioni morfologiche durante il differenziamento epatico. Barre di scala = 200 μm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Strategia di gating per lo smistamento di epatociti e non epatociti in cellule epatiche differenziate (giorno 27). (A) Gating della popolazione di cellule bersaglio utilizzando l'FSC-A vs. Grafico SSC-A. (B) Espansione della popolazione recintata dal passaggio (A) utilizzando l'FSC-H vs. FSC-W traccia ed esclude le cellule doppiette. (C) Espandere ulteriormente la popolazione recintata dal passaggio (B) utilizzando l'SSC-H vs. Grafico SSC-W ed escludendo le cellule doppiette. (D) e (E) Espansione della popolazione recintata dal passaggio (C) utilizzando il FITC-A (ALCAM) vs. Trama PE-A (TMRM). (D) Cellule colorate con TMRM e il 2° anticorpo . (E) Cellule colorate con TMRM e il 1° e il 2° anticorpo . Clicca qui per visualizzare una versione più grande di questa figura.
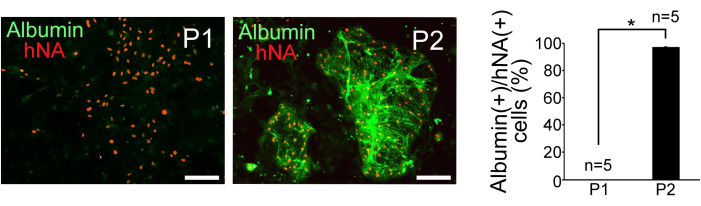
Figura 3: Risultati dell'immunoistochimica. Rilevamento immunoistochimico dell'albumina e dell'antigene nucleare umano (hNA) in cellule P1 e P2 selezionate e quantificazione della frazione cellulare di albumina (+) in cellule hNA (+). Barre di scala = 100 μm. I dati sono rappresentati come media ± deviazione standard (SD) (n = 5). L'analisi statistica è stata condotta utilizzando il test t di Student. *P < 0,05. Questa figura è adattata da Yamashita et al.14. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
A causa delle loro funzioni nel metabolismo dei nutrienti e nella disintossicazione, gli epatociti possiedono un numero relativamente elevato di mitocondri rispetto ad altri tipi di cellule15. ALCAM è un membro della superfamiglia delle immunoglobuline e svolge un ruolo nell'adesione e nella migrazione cellulare. È espresso in vari tipi di cellule, tra cui cellule epatiche, epiteliali, linfocitiche, mieloidi, fibroblasti e neuronali16. Utilizzando una combinazione del metodo basato sui mitocondri e dell'anticorpo ALCAM, sono stati identificati con successo gli epatociti derivati da cellule iPS umane.
Sebbene non siano stati eseguiti i saggi per caratterizzare la funzione secretoria o l'attività enzimatica degli epatociti purificati, è stata verificata l'espressione dell'mRNA del gene CYP3A4 negli epatociti purificati derivati da cellule iPS umane. Inoltre, la popolazione di epatociti con TMRM (alto) e ALCAM (+) isolati da corpi embrioidi derivati da cellule ES umane, ha osservato un'espressione più elevata di set di geni associati agli epatociti (VTN, SERPINA1, CYP1A1, FGB, FGA, FGG, AFP, ALB e APOB) rispetto ai cardiomiociti derivati da cellule ES14.
Un passaggio fondamentale all'interno del protocollo consiste nel staccare delicatamente le cellule differenziate epatiche 2D dai piatti di coltura con danni minimi, garantendo al contempo la conservazione dell'attività mitocondriale per la colorazione TMRM. A seconda dello stato di differenziazione, gli epatociti possono mostrare resistenza al distacco dalla piastra anche con trattamento enzimatico, una sfida che può essere efficacemente affrontata con l'aggiunta di 0,25 mM di EDTA.
I metodi esistenti per la purificazione degli epatociti tramite FACS prevedono l'introduzione di geni reporter a valle dei promotori specifici dei geni espressi dagli epatociti. Tuttavia, questo approccio comporta il potenziale rischio di effetti collaterali associati alle modificazioni genetiche. Il metodo mitocondriale, d'altra parte, elimina tali preoccupazioni. Sono stati identificati diversi marcatori di superficie cellulare di cellule staminali/progenitrici epatiche adulte o fetali18 e le cellule progenitrici epatiche derivate da cellule staminali pluripotenti umane potrebbero essere selezionate utilizzando la popolazione CD13+CD133+19. D'altra parte, tra gli epatociti derivati da cellule staminali pluripotenti, una piccola frazione degli epatociti maturi potrebbe essere selezionata utilizzando l'anticorpo ASGR1 (recettore 1 dell'asialoglicoproteina 1), che è l'unico marcatore di superficie cellulare specifico per gli epatociti. È stato dimostrato che la popolazione di cellule che mostravano un'elevata positività mitocondriale e ALCAM conteneva sia popolazioni marcate con cellule CD13+CD133+ che cellule ASGR1+. Ciò suggerisce che le popolazioni ad alto contenuto di mitocondriali e ALCAM-positive possono contenere un'ampia gamma di epatociti con varie maturità14. Di conseguenza, questo metodo può consentire la purificazione di una popolazione di epatociti con poche perdite.
L'analisi dei visceri interi fetali di topo al giorno embrionale 14,5 utilizzando questo metodo ha rivelato l'espressione di ALCAM sia nei miociti che negli epatociti14. Altri studi precedenti hanno riportato l'espressione di ALCAM nelle cellule progenitrici miocardiche murine21. Nel caso di cellule staminali pluripotenti umane, i cardiomiociti sono stati identificati nella popolazione ALCAM negativa. Questi dati mostrano che l'espressione di ALCAM nelle linee cardiache ed epatiche differisce a seconda sia della specie che degli stadi di maturità cellulare. Tuttavia, a causa della più alta frazione mitocondriale nei cardiomiociti rispetto agli epatociti, è stato effettivamente possibile separare i due tipi di cellule mediante FACS anche da cellule miste derivate dall'intero embrione di topo. Ciò suggerisce che il metodo proposto potrebbe essere utile per purificare epatociti e cardiomiociti da altre specie animali.
Si prevede che la tecnologia di purificazione degli epatociti qui descritta svolgerà un ruolo fondamentale nel progresso del campo della medicina rigenerativa, mitigando il potenziale rischio di formazione di tumori associati al trapianto di epatociti derivati da cellule iPS umane. Tuttavia, non è stata effettuata la negazione di una capacità di formazione di teratomi nella popolazione di epatociti purificata con questa tecnica. Devono essere condotti ulteriori studi per la garanzia della sicurezza.
Le cellule grandi e aderenti come gli epatociti sono altamente vulnerabili ai danni meccanici durante lo smistamento FACS, con conseguente perdita significativa di cellule vitali. Per le applicazioni cliniche, è essenziale sviluppare un nuovo metodo di purificazione che stabilisca un ambiente di coltura che consenta la sopravvivenza esclusivamente degli epatociti, come il metodo del lattato per la purificazione dei cardiomiociti22.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dal Ministero dell'Istruzione, della Cultura, dello Sport, della Scienza e della Tecnologia numero di sovvenzione [23390072].
Materiali
| Name | Company | Catalog Number | Comments |
| 253G1 human iPS cell line | RIKEN BioResource Research Center | HPS0002 | |
| 4% paraformaldehyde | FUJIFILM Wako Pure Chemical Corporation | 163-20145 | |
| Activin A Solution, Human, Recombinant | NACALAI TESQUE, INC. | 18585 | |
| Anti-Nuclei Antibody, clone 235-1 | Chemicon | MAB1281 | Antibody against human nuclear antigen |
| B-27 Supplement (50x), serum free | Thermo Fisher Scientific | 17504044 | |
| BD FACSAria III | BD Biosciences | Cell sorter | |
| CHIR-99021 | MedChemExpress | HY-10182 | |
| Ciclosporin A | FUJIFILM Wako Pure Chemical Corporation | 035-18961 | |
| Collagenase | FUJIFILM Wako Pure Chemical Corporation | 034-22363 | |
| Corning Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | Corning | 354230 | Gel-like basement membrane matrix |
| CultureSure Y-27632 | FUJIFILM Wako Pure Chemical Corporation | 036-24023 | ROCK inhibitor |
| Dexamethasone | FUJIFILM Wako Pure Chemical Corporation | 047-18863 | |
| Dimethyl sulfoxide (DMSO) | FUJIFILM Wako Pure Chemical Corporation | 047-29353 | |
| Donkey anti-Goat IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo Fisher Scientific | A-11055 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 546 | Thermo Fisher Scientific | A10036 | |
| Donkey anti-Rabbit IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo Fisher Scientific | A-21206 | |
| Falcon 5 mL Round Bottom Polystyrene Test Tube, with Cell Strainer Snap Cap | Corning | 352235 | |
| Fetal Bovine Serum | Biowest | 51820-500 | |
| GlutaMAX Supplement | Thermo Fisher Scientific | 35050061 | Dipeptide L-Alanyl-L-Glutamine |
| Human/Mouse/Rat/Canine ALCAM/CD166 Antibody | R&D Systems | AF1172 | |
| Hydrocortisone 21-hemisuccinate sodium salt | Sigma-Aldrich | H2270 | |
| iMatrix-511 silk | Nippi | 892021 | |
| ImmunoBlock | KAC | CTKN001 | Blocking solution |
| ITS-G Supplement(×100) | FUJIFILM Wako Pure Chemical Corporation | 090-06741 | |
| Leibovitz's L-15 Medium | FUJIFILM Wako Pure Chemical Corporation | 128-06075 | |
| N-Hexanoic-Try-Ile-(6)-amino Hexanoic amide (Dihexa) | Toronto Research Chemicals | H293745 | |
| Polyclonal Rabbit Anti-Human Albumin | Dako | A0001 | |
| Polyoxyethylene Sorbitan Monolaurate (Tween 20) | NACALAI TESQUE, INC. | 28353-85 | |
| RPMI-1640 | FUJIFILM Wako Pure Chemical Corporation | 189-02025 | |
| Sodium L-Ascorbate | NACALAI TESQUE, INC. | 03422-32 | |
| StemFit AK02N | REPROCELL | RCAK02N | |
| TBS (10x) | NACALAI TESQUE, INC. | 12748-31 | |
| Tetramethylrhodamine, methyl ester (TMRM) | Thermo Fisher Scientific | T668 | |
| Triton X-100 | NACALAI TESQUE, INC. | 28229-25 | |
| Trypan Blue Solution | NACALAI TESQUE, INC. | 20577-34 | |
| TRYPSIN 250 | Difco | 215240 | |
| Tryptose phosphate broth solution | Sigma-Aldrich | T8159 |
Riferimenti
- Yamamoto, T., et al. Differentiation potential of pluripotent stem cells correlates to the level of CHD7. Scientific Reports. 8 (1), 241 (2018).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Reports. 10 (6), 1851-1866 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Reports. 16 (6), 1446-1457 (2021).
- Chen, C. X. -. Q., et al. Standardized quality control workflow to evaluate the reproducibility and differentiation potential of human iPSCs into neurons. bioRxiv. , (2021).
- Duinsbergen, D., Salvatori, D., Eriksson, M., Mikkers, H. Tumors originating from induced pluripotent stem cells and methods for their prevention. Annals of the New York Academy of Sciences. 1176, 197-204 (2009).
- Nori, S., et al. Long-term safety issues of iPSC-based cell therapy in a spinal cord injury model: oncogenic transformation with epithelial-mesenchymal transition. Stem Cell Reports. 4 (3), 360-373 (2015).
- Hentze, H., et al. Teratoma formation by human embryonic stem cells: evaluation of essential parameters for future safety studies. Stem Cell Research. 2 (3), 198-210 (2009).
- Anderson, D., et al. Transgenic enrichment of cardiomyocytes from human embryonic stem cells. Molecular Therapy. 15 (11), 2027-2036 (2007).
- Hidaka, K., et al. Chamber-specific differentiation of Nkx2.5-positive cardiac precursor cells from murine embryonic stem cells. The FASEB Journal. 17 (6), 740-742 (2003).
- Gassanov, N., Er, F., Zagidullin, N., Hoppe, U. C. Endothelin induces differentiation of ANP-EGFP expressing embryonic stem cells towards a pacemaker phenotype. The FASEB Journal. 18 (14), 1710-1712 (2004).
- Duan, Y., et al. Differentiation and enrichment of hepatocyte-like cells from human embryonic stem cells in vitro and in vivo. Stem Cells. 25 (12), 3058-3068 (2007).
- Takayama, K., et al. Enrichment of high-functioning human iPS cell-derived hepatocyte-like cells for pharmaceutical research. Biomaterials. 161, 24-32 (2018).
- Hattori, F., et al. Nongenetic method for purifying stem cell-derived cardiomyocytes. Nature methods. 7, 61-66 (2009).
- Yamashita, H., Fukuda, K., Hattori, F. Hepatocyte-like cells derived from human pluripotent stem cells can be enriched by a combination of mitochondrial content and activated leukocyte cell adhesion molecule. JMA Journal. 2 (2), 174-183 (2019).
- Fernandez-Vizarra, E., Enríquez, J., Pérez-Martos, A., Montoya, J., Fernández-Silva, P. Tissue-specific differences in mitochondrial activity and biogenesis. Mitochondrion. 11, 207-213 (2010).
- Swart, G. W. M. Activated leukocyte cell adhesion molecule (CD166/ALCAM): Developmental and mechanistic aspects of cell clustering and cell migration. European Journal of Cell Biology. 81 (6), 313-321 (2002).
- Peters, D. T., et al. Asialoglycoprotein receptor 1 is a specific cell-surface marker for isolating hepatocytes derived from human pluripotent stem cells. Development. 143 (9), 1475-1481 (2016).
- Kamiya, A., Inagaki, Y. Stem and progenitor cell systems in liver development and regeneration. Hepatology Research. 45 (1), 29-37 (2015).
- Yanagida, A., Ito, K., Chikada, H., Nakauchi, H., Kamiya, A. An In vitro expansion system for generation of human ips cell-derived hepatic progenitor-like cells exhibiting a bipotent differentiation potential. PLOS One. 8 (7), e67541 (2013).
- Siller, R., Greenhough, S., Naumovska, E., Sullivan, G. J. Small-molecule-driven hepatocyte differentiation of human pluripotent stem cells. Stem Cell Reports. 4 (5), 939-952 (2015).
- Hirata, H., et al. ALCAM (CD166) is a surface marker for early murine cardiomyocytes. Cells, Tissues, Organs. 184 (3-4), 172-180 (2006).
- Tohyama, S., et al. Distinct metabolic flow enables large-scale purification of mouse and human pluripotent stem cell-derived cardiomyocytes. Cell Stem Cell. 12 (1), 127-137 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon