Method Article
Anwendung der Färbung lebender Mitochondrien bei der Zellsortierung zur Aufreinigung von Hepatozyten, die aus humanen induzierten pluripotenten Stammzellen gewonnen wurden
In diesem Artikel
Zusammenfassung
Hepatozyten, die aus pluripotenten Stammzellen gewonnen werden, können durch Zellsortierung aufgereinigt werden, wobei eine Kombination aus mitochondrialer und aktivierter Leukozytenzelladhäsionsmolekülfärbung (ALCAM, auch bekannt als CD166) verwendet wird.
Zusammenfassung
Humane embryonale Stammzellen (ES) und induzierte pluripotente Stammzellen (iPS) haben aufgrund ihrer unbegrenzten Proliferation und pluripotenten Eigenschaften potenzielle Anwendungen in der zellbasierten regenerativen Medizin zur Behandlung schwer erkrankter Organe. Die Differenzierung von humanen ES/iPS-Zellen in 100 % reine Zielzelltypen ist jedoch aufgrund ihrer hohen Empfindlichkeit gegenüber der Umwelt eine Herausforderung. Die Tumorgenese nach einer Transplantation wird durch kontaminierte, proliferierende und undifferenzierte Zellen verursacht, so dass die Hochreinigungstechnologie für die sichere Realisierung der regenerativen Medizin unerlässlich ist. Um das Risiko der Tumorentstehung zu verringern, wurde eine Hochreinigungstechnologie für aus humanen iPS-Zellen gewonnene Hepatozyten entwickelt. Das Verfahren verwendet FACS (Fluoreszenz-aktivierte Zellsortierung) unter Verwendung einer Kombination aus hohem mitochondrialem Gehalt und dem Zelloberflächenmarker ALCAM (aktiviertes Leukozytenzelladhäsionsmolekül) ohne genetische Veränderung. 97% ± 0,38% (n = 5) der gereinigten Hepatozyten mit dieser Methode zeigten eine Albuminproteinexpression. In diesem Artikel sollen detaillierte Verfahren für diese Methode vorgestellt werden, wie sie auf die aktuellste zweidimensionale Differenzierungsmethode für humane iPS-Zellen in Hepatozyten angewendet wird.
Einleitung
Embryonale und induzierte pluripotente Stammzellen (ES bzw. iPS) gelten als vielversprechende Zellquellen für regenerative Therapien. Die Effizienz der Differenzierung dieser Zellen in spezifische Zielzelltypen kann jedoch variieren, selbst wenn die gleiche Zelllinie, das gleiche Protokoll und der gleiche Experimentator verwendetwerden 1,2,3,4. Diese Variabilität kann auf die hohe Empfindlichkeit menschlicher ES/iPS-Zellen gegenüber ihrer Umgebung zurückgeführt werden. Daher ist es derzeit schwierig, konsistent reine Zielzellen zu gewinnen. Um eine hochsichere regenerative Medizin zu erreichen, ist es von entscheidender Bedeutung, proliferative Zellen und undifferenzierte Stammzellen in therapeutischen Zellen zu eliminieren, und eine fortschrittliche Reinigungstechnologie für Zielzellen ist unerlässlich 5,6,7.
Ein Zellsortierer ist ein Gerät, das einzelne Zellen sofort analysiert und lebende Zellen von Interesse basierend auf den Fluoreszenzsignalstärken sortiert und damit eine vielversprechende Lösung bietet. Dies kann durch die Antikörperfärbung von zelltypspezifischen Oberflächenmarkern oder durch die Verwendung zelltypspezifischer Reportergenexpressionen erreicht werden. Unter Verwendung dieser Technik gibt es mehrere Berichte über Methoden zur Reinigung von aus pluripotenten Stammzellen gewonnenen Kardiomyozyten 8,9,10 und Hepatozyten11,12. Hattori et al. entwickelten ein innovatives mitochondriales Aufreinigungsverfahren unter Verwendung eines Zellsortierers13. Unter Ausnutzung der Tatsache, dass Kardiomyozyten durch mitochondriale Aktivität einen hohen Energiebedarf haben, kann die Färbung der Zellen mit dem lebenden Mitochondrien-indikativen Farbstoff TMRM (Tetramethylrhodaminmethylester) zur Markierung und hochreinen Kardiomyozyten mittels FACS aus humanen ES-Zell-abgeleiteten Embryoidkörpern mit verschiedenen Zelltypen verwendet werden. Das Fehlen von Tumorigenität wurde durch Teratombildungsassays mit den gereinigten Kardiomyozyten bestätigt. Darüber hinaus entdeckten Yamashita et al. unerwartet eine Methode zur Reinigung von Hepatozyten aus humanen ES-Zell-abgeleiteten Embryoidkörpern durch Isolierung von Fraktionen mit hoher mitochondrialer Aktivität und ALCAM-positiver Expression14. Die Begründung für diese Methode ist, dass Hepatozyten aufgrund ihres hohen Verbrauchs an ATP für den Nährstoffstoffwechsel und die Entgiftung auch eine relativ hohe Anzahl von Mitochondrien aufweisen15, und Hepatozyten exprimieren ALCAM, ein Mitglied der Immunglobulin-Superfamilie, das eine Rolle bei der Zelladhäsion und -migration spielt16.
Frühere hochaufreinigende Methoden für aus pluripotenten Stammzellen gewonnene Hepatozyten erforderten genetische Modifikationen, und nicht-genetische Aufreinigungsmethoden wiesen eine geringe Effizienz auf17. Die mitochondriale nicht-genetische Methode zeichnet sich dadurch aus, dass sie eine hohe Reinheit erreicht. Wenn hepatische Vorläuferzellen benötigt werden, können CD133- und CD13- oder Dlk1-basierte Methoden18,19 gewählt werden. Obwohl die Genauigkeit der Genom-Editing-Technologie fortgeschritten ist, kann das potenzielle Risiko unvorhergesehener genomischer Veränderungen (z. B. Karzinogenese) nicht vollständig ausgeschlossen werden. Methoden, die auf mitochondrialer Aktivität basieren, ohne genetische Veränderungen zu beinhalten, können frei von solchen Risiken sein.
Als Ergebnis der Untersuchung verschiedener mitochondrialer Indikatoren in neonatalen Kardiomyozyten von Ratten wurde beobachtet, dass TMRM innerhalb von 24 Stunden vollständig verschwand, während andere Farbstoffe mindestens 5 Tage lang blieben13. Darüber hinaus ist es wichtig zu beachten, dass TMRM und JC-1 die Zellviabilität nicht beeinflussten, wenn sie mit dem 3-(4,5-Dimethylthiazol-2-yl)-2,5-diphenyltetrazoliumbromid (MTT)-Assay bewertet wurden, während andere Farbstoffe unterschiedliche Auswirkungen auf die Zellviabilität zeigten13. TMRM weist eine höhere Sicherheit auf.
Bisher wurden mehrere zweidimensionale (2D) Differenzierungsmethoden entwickelt, die im Vergleich zur dreidimensionalen (3D) Embryoidkörperbildung effizientere Ansätze zur Induktion der Differenzierung darstellen. Dies liegt daran, dass differenzierungsinduzierende Verbindungen oder Zytokine den Zellen einheitlich auf einer 2D-Ebene und nicht in einem 3D-Raum verabreicht werden können. In dieser Studie wurde die zuvor beschriebene Methode20 modifiziert, um die Differenzierung in humane iPS-Zell-abgeleitete Hepatozyten zu induzieren. Im Folgenden werden die Verfahren zur zeitgemäßen 2D-Differenzierung und -Reinigung von Hepatozyten, die aus humanen iPS-Zellen gewonnen werden, detailliert beschrieben.
Protokoll
In dieser Studie wurden kommerziell hergestellte humane iPS-Zellen (Stamm 253G1) verwendet (siehe Materialtabelle).
1. Erhaltung humaner iPS-Zellen
- Humane iPS-Zellen werden unter feederfreien Bedingungen im AK02-Medium auf Kulturschalen aufbewahrt, die mit 0,25 μg/cm2 iMatrix-511 beschichtet sind (siehe Materialtabelle).
2. Leberdifferenzierung von humanen iPS-Zellen in 2D-Kulturen
HINWEIS: In Abbildung 1A finden Sie den Tagesablauf der Leberdifferenzierung. Verwenden Sie während des gesamten Prozesses 6-Well-Kulturplatten und geben Sie 2 ml des dafür vorgesehenen Wasch- oder Kulturmediums in jede Vertiefung. Halten Sie den Zellkulturzustand bei 100 % Luftfeuchtigkeit, 5 % CO2 und 20 % O2 bei 37 °C aufrecht.
- Jede Vertiefung einer 6-Well-Platte mit 1 ml Basalmembranmatrix (verdünnt 1/25 in PBS ()) beschichten und 1 h bei 37 °C inkubieren.
- Humane iPS-Zellen werden mit einer Dichte von 20.000-30.000 Zellen/cm² unter Verwendung von AK02N-Medium, das mit einem 10 μM ROCK-Inhibitor (Y-27632) ergänzt wird, in die beschichtete Schale gebracht (siehe Materialtabelle). Ersetzen Sie das Medium am nächsten Tag ohne Y-27632 und kultivieren Sie es für weitere 2 Tage.
- Waschen Sie die Zellen einmal mit RPMI-1640 Medium. Initiieren Sie die Endodermdifferenzierung, indem Sie das Medium durch RPMI-B27-Glu (RPMI-1640 + 2% B27 Insulin + 1% Dipeptid L-Alanyl-L-Glutamin) ersetzen, ergänzt mit 3-6 μM CHIR-99021 und 100 ng/ml Activin A für 24 h (siehe Materialtabelle).
- Ersetzen Sie das Medium durch RPMI-B27-Glu, ergänzt mit 50 ng/mL Activin A für 24 h.
- Um die Differenzierung der hepatischen Vorläuferzellen zu induzieren, ersetzen Sie das Medium 5 Tage lang durch RPMI-B27-Glu, das mit 1 % Dimethylsulfoxid (DMSO) ergänzt ist.
- Um die Hepatozytenreifung zu fördern, wechseln Sie zu Hepatozyten-Reifungsmedium: L-15-Medium von Leibovitz mit 10 μM Hydrocortison-21-hemisuccinat, 8,3 % Tryptosephosphat-Brühe, 100 nM Dexamethason (DEX), 50 μg/ml Natrium-L-Ascorbat, 0,58 % Insulin-Transferrin-Selen (ITS), 8,3 % fötales Rinderserum (FBS), 2 mM Dipeptid L-Alanyl-L-Glutamin und 100 nM N-Hexanosäure-Try-Ile-(6)-aminohexansäureamid (Dihexa) (siehe Materialtabelle).
- Wechseln Sie das Medium alle 2 Tage für 20 Tage. Sortieren Sie die Zellen etwa 27 Tage nach Beginn der Differenzierung.
3. Zellvorbereitung für die Aufreinigung von Hepatozyten, die aus humanen iPS-Zellen gewonnen wurden
- Waschen Sie hepatische differenzierte Zellen mit 2 ml PBS (-) pro Well.
- Fügen Sie 1 ml Enzymlösung pro Vertiefung hinzu, die 0,1 % Kollagenase, 0,083 % Trypsin, 10 μM ROCK-Inhibitor (Y-27632), 20 μM Ciclosporin A und 50 μg/ml Natrium-L-Ascorbat in Ads-Puffer (116 mM NaCl, 12,5 mM NaH2PO4, 20 mM HEPES, 5,4 mM KCl, 5,6 mM Glukose und 0,8 mM MgSO4; pH 7,35) enthält (siehe Materialtabelle). Die Zellen werden von den Platten gelöst, indem sie sie ca. 2 h lang bei 37 °C horizontal drehen.
- Nachdem Sie unter einem Mikroskop bestätigt haben, dass die Zellen abgetrennt und gerundet sind, dispergieren Sie sie durch vorsichtiges Pipettieren in einzelne Zellen und sammeln Sie sie in einem 50-ml-Röhrchen.
- Zellen bei 200 x g für 5 min bei 18 °C zentrifugieren, um sie zu pelletieren. Entfernen Sie den Überstand mit einem Aspirator.
- Färben Sie die Mitochondrien der Zellen, indem Sie sie in 5 ml Hepatozyten-Reifungsmedium dispergieren, das 100 nM TMRM (Tetramethylrhodamin, Mehlester) enthält (siehe Materialtabelle). 30 min bei 37 °C inkubieren und dabei vor Licht schützen, indem in Alufolie eingewickelt werden.
- Zentrifugieren Sie die Zellen bei 200 x g für 5 min bei 18 °C, um sie zu pelletieren. Entfernen Sie den Überstand.
- Resuspendieren Sie die Zellen mit 1-5 ml kaltem Ads-Puffer mit 2% FBS.
- Zählen Sie die Zellzahl mit einer 0,4%igen Trypanblau-Lösung und einer Zellzählplatte.
- Zentrifugieren Sie die Zellen bei 200 x g für 5 min bei 4 °C, um sie zu pelletieren. Entfernen Sie den Überstand.
- Verdünnen Sie einen Anti-ALCAM-Antikörper (Primärantikörper, siehe Materialtabelle) im Verhältnis 1:50 in Ads-Puffer und fügen Sie 100 μl des verdünnten Antikörpers pro 106 Zellen hinzu. Färben Sie die Zellen 50 Minuten lang auf Eis. Bereiten Sie Zellen vor, die nicht mit dem primären Antikörper als Negativkontrolle gefärbt sind.
- Waschen Sie die Zellen zweimal mit kaltem Ads-Puffer mit 2% FBS.
- Verdünnen Sie ein Alexa Fluor 488 Esel-Anti-Ziegen-IgG (Sekundärantikörper, siehe Materialtabelle) im Verhältnis 1:100 in Ads-Puffer und fügen Sie 100 μl des verdünnten Antikörpers pro 106 Zellen hinzu. Färben Sie die Zellen 30 Minuten lang auf Eis. Färben Sie Zellen, die nicht mit dem Primärantikörper gefärbt wurden, mit dem Sekundärantikörper.
- Waschen Sie die Zellen zweimal mit kaltem Ads-Puffer mit 2% FBS.
- Resuspendieren Sie die Zellen in Ads-Puffer und filtern Sie sie durch ein Schnappkappen-Zellsieb (35 μm Netz), kurz bevor Sie nach FACS sortieren, um große Zellklumpen und Ablagerungen zu entfernen.
4. FACS-Analysen und Sortierung von aus humanen iPS-Zellen gewonnenen Hepatozyten
- Richten Sie die FACS-Maschine für die Zellensortierung gemäß den Anweisungen des Herstellers ein (siehe Materialtabelle).
HINWEIS: Verwenden Sie eine Düsengröße von 85 μm oder 100 μm und einen ND-Filter von 2,0 für Hepatozyten. Verwenden Sie den FITC-Kanal zum Nachweis von Alexa Flour 488 für ALCAM und den PE-Kanal zum Nachweis von TMRM-Fluoreszenz für Mitochondrien. - Passen Sie die Spannungen FSC (Vorwärtsstreuung) und SSC (Seitenstreuung) an, um die Zellpopulation richtig zu visualisieren.
- Passen Sie die Spannungen der FITC- und PE-Leuchtstoffkanäle an. Beginnen Sie mit unbeschrifteten Zellen, und zentrieren Sie die Zellpopulation am unteren Rand jedes Kanaldiagramms. Stellen Sie sicher, dass die beschrifteten Zellen im Diagramm richtig positioniert sind.
- Verwenden Sie eine gängige Gating-Strategie, um Dubletten zu eliminieren: Gate die wichtigsten FSC-A vs. SSC-A-Population (Abbildung 2A), gefolgt von FSC-H vs. -W und dann SSC-H vs. -W (Abbildung 2B,C).
- Gate der TMRMhi- und ALCAM+-Population (P2 in Abbildung 2E), um Hepatozyten zu sortieren. Gate der TMRMhi- und ALCAM-Population (P1 in Abbildung 2E), um Nicht-Hepatozyten zu sortieren. Sammeln Sie sortierte Hepatozyten und Nicht-Hepatozyten in 15-ml-Entnahmeröhrchen, die Hepatozyten-Reifungsmedium enthalten.
- Das Sammelröhrchen mit den sortierten Zellen bei 200 x g für 5 min bei 18 °C zentrifugieren.
- Entfernen Sie den Überstand und resuspendieren Sie die Zellen in Hepatozyten-Reifungsmedium.
- Aussaat der Zellen auf Mitomycin C-behandelten embryonalen Mausfibroblasten (MEFs) oder gelartiger Basalmembranmatrix mit Hepatozyten-Reifungsmedium, ergänzt mit 10 μM Y-27632 und 20 nM Ciclosporin A. Kulturieren Sie die Zellen in einem 37 °CCO 2 -Inkubator für 5 Tage.
5. Immunzytochemie zum Nachweis von Hepatozyten, die aus humanen iPS-Zellen gewonnen wurden
- Waschen Sie die kultivierten Zellen nach der Sortierung mit PBS (-) 5 Tage lang.
- Fixieren Sie die Zellen mit 4% Paraformaldehyd für 5 min bei Raumtemperatur.
- Waschen Sie die Zellen zweimal mit TBS-T-Puffer (1x TBS mit 1% Tween 20), um das Paraformaldehyd zu entfernen.
- Inkubieren Sie die Zellen mit 0,1 % Triton X-100 verdünntem TBS-T-Puffer für 5 Minuten bei Raumtemperatur.
- Waschen Sie die Zellen zweimal mit TBS-T-Puffer, um Triton X-100 zu entfernen.
- Die handelsübliche Blockierungslösung (siehe Materialtabelle) für 30 min bei Raumtemperatur in die Zellen geben.
- Primäre Antikörper gegen Albumin (Hepatozytenmarker) und humanes nukleäres Antigen (hNA, siehe Materialtabelle) zugeben, verdünnt 1:50 in Blockierungslösung.
- Inkubieren Sie die Zellen über Nacht bei 4 °C. Waschen Sie die Zellen zweimal mit TBS-T-Puffer.
- Sekundärantikörper hinzufügen: Alexa Fluor 488 Anti-Kaninchen-IgG und Alexa Fluor 546 Esel-Anti-Maus-IgG, 1:100 in Blockierungslösung verdünnt (siehe Materialtabelle).
- Inkubieren Sie die Zellen 30 Minuten lang bei Raumtemperatur. Waschen Sie die Zellen zweimal mit TBS-T-Puffer.
- Bewerten Sie die Reinheit von Hepatozyten, indem Sie albuminpositive und hNA-positive Zellen unter einem Fluoreszenzmikroskop zählen. Berechnen Sie den Prozentsatz der Albumin-positiven Zellen pro hNA-positiver Zelle sowohl für die P1- als auch für die P2-Population.
Ergebnisse
Die Zeitleiste der Prozesse, die humane iPS-Zellen dazu veranlassen, sich durch 2D-Kultur (Abbildung 1A) und repräsentative Zellmerkmale (Abbildung 1B) in Hepatozyten zu differenzieren, wird gezeigt. Ungefähr am 12. Tag der Differenzierung begannen die Zellen polygonale Zellformen und runde Zellkerne zu zeigen, die für Hepatozyten charakteristisch sind. Einige Hepatozyten zeigten auch eine Mehrkernigkeit.
Die FACS-Analyse wurde mit den Zellen am Differenzierungstag 27 durchgeführt. Zur Identifizierung der ALCAM+-Population sind in Abbildung 2D,E Vergleichsdiagramme mit und ohne den ersten Anti-ALCAM-Antikörper dargestellt. Durch die Induktion der hepatischen Differenzierung durch 2D-Kultur wurde festgestellt, dass die Population der Hepatozyten (P2) in diesem Experiment 10,1 % betrug, was einen signifikanten Anstieg gegenüber den 0,8 % (Daten nicht gezeigt) darstellt, die mit der 3D-Methode zur Bildung des Embryoidkörpers gefunden wurden.
P1- und P2-Zellen, die sortiert worden waren, wurden an embryonalen Fibroblasten der Maus kultiviert, die mit Mitomycin C behandelt wurden. P2-Zellen zeigten eine kompakte, kugelförmige Koloniebildung, während P1-Zellen dispergiert waren und eine flachere Morphologie aufwiesen. Immunhistochemische Färbungen für humanes nukleäres Antigen (hNA) und Albumin bestätigten die Anreicherung von hepatozytenähnlichen Zellen in P2, wie in Abbildung 3 dargestellt. Um die Reinheit der Hepatozyten in P1- und P2-Zellen zu bestimmen, wurde das Verhältnis von Albumin-positiven Zellen zu hNA-positiven Zellen berechnet (n = 5). Die Ergebnisse zeigten, dass 0% der P1-Zellen und 97% ± 0,38% der P2-Zellen aus humanen iPSCs stammten und hepatozytenähnliche Eigenschaften aufwiesen (Abbildung 3).
Um die erfolgreiche Sortierung der Hepatozyten während des FACS-Experiments zu bestätigen, wird empfohlen, ASGR1 mit seinem spezifischen Antikörper, der mit verschiedenen Fluorophoren konjugiert ist, co-zu färben und zu bestätigen, dass P2 ASGR1-positive Zellen enthält.
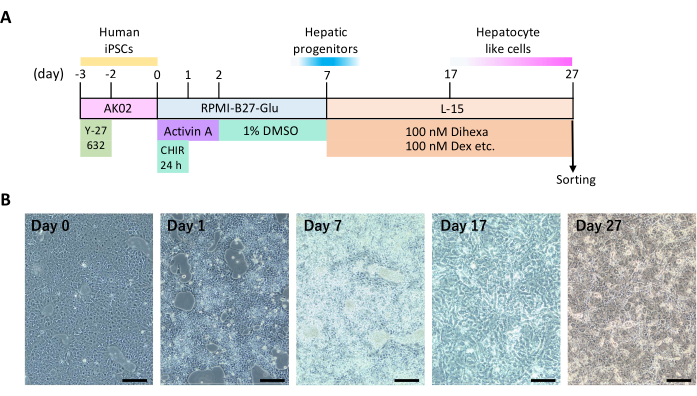
Abbildung 1: 2D-hepatische Differenzierung von humanen iPSCs. (A) Der zeitliche Ablauf der hepatischen Differenzierung. Diese Abbildung ist eine Adaption von Yamashita et al.14. (B) Morphologische Veränderungen während der hepatischen Differenzierung. Maßstabsbalken = 200 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Gating-Strategie zur Sortierung von Hepatozyten und Nicht-Hepatozyten in hepatischen differenzierten Zellen (Tag 27). (A) Gating der Zielzellpopulation mit dem FSC-A vs. SSC-A-Handlung. (B) Erweiterung der Gated Population aus Schritt (A) unter Verwendung des FSC-H vs. FSC-W zeichnet Dublettenzellen auf und schließt sie aus. (C) Weitere Erweiterung der Gated Population ab Schritt (B) unter Verwendung des SSC-H vs. SSC-W-Diagramm und ohne Dublettenzellen. (D) und (E) Erweiterung der Gated Population aus Schritt (C) unter Verwendung des FITC-A (ALCAM) vs. PE-A (TMRM)-Diagramm. (D) Gefärbte Zellen mit TMRM und dem 2. Antikörper. (E) Gefärbte Zellen mit TMRM und den 1. und 2. Antikörpern. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
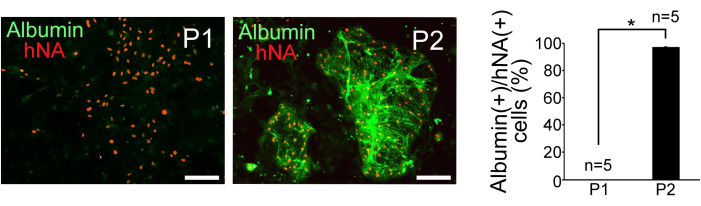
Abbildung 3: Immunhistochemische Ergebnisse. Immunhistochemischer Nachweis von Albumin und humanem nukleärem Antigen (hNA) in sortierten P1- und P2-Zellen und Quantifizierung der Albumin-(+)-Zellfraktion in hNA-(+)-Zellen. Maßstabsleisten = 100 μm. Die Daten werden als Mittelwert ± Standardabweichung (SD) (n = 5) dargestellt. Die statistische Analyse wurde mit dem Student's t-Test durchgeführt. *P < 0,05. Diese Abbildung ist eine Adaption von Yamashita et al.14. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Aufgrund ihrer Funktionen im Nährstoffstoffwechsel und in der Entgiftung besitzen Hepatozyten im Vergleich zu anderen Zelltypen eine relativ große Anzahl an Mitochondrien15. ALCAM ist ein Mitglied der Immunglobulin-Superfamilie und spielt eine Rolle bei der Zelladhäsion und -migration. Es wird in verschiedenen Zelltypen exprimiert, darunter hepatische, epitheliale, lymphozytäre, myeloische, Fibroblasten- und neuronale Zellen16. Durch die Verwendung einer Kombination aus der mitochondrienbasierten Methode und dem ALCAM-Antikörper konnten humane iPS-Zell-abgeleitete Hepatozyten erfolgreich identifiziert werden.
Während die Assays zur Charakterisierung der sekretorischen Funktion oder Enzymaktivität der gereinigten Hepatozyten nicht durchgeführt wurden, wurde die mRNA-Expression des CYP3A4-Gens in humanen iPS-Zell-abgeleiteten Hepatozyten, die gereinigt wurden, verifiziert. Darüber hinaus wurde bei Hepatozytenpopulationen mit TMRM (hoch) und ALCAM (+), die aus embryoiden Körpern isoliert wurden, die von humanen ES-Zellen abgeleitet wurden, eine höhere Expression von Hepatozyten-assoziierten Gensätzen (VTN, SERPINA1, CYP1A1, FGB, FGA, FGG, AFP, ALB und APOB) im Vergleich zu den aus ES-Zellen gewonnenen Kardiomyozytenbeobachtet 14.
Ein kritischer Schritt innerhalb des Protokolls besteht darin, differenzierte 2D-Leberzellen mit minimaler Beschädigung vorsichtig und mit minimaler Beschädigung von den Kulturschalen zu lösen und gleichzeitig die Erhaltung der mitochondrialen Aktivität für die TMRM-Färbung zu gewährleisten. Abhängig vom Differenzierungsstadium können Hepatozyten auch bei Enzymbehandlung eine Resistenz gegen die Ablösung von der Schale aufweisen, eine Herausforderung, die durch die Zugabe von 0,25 mM EDTA wirksam angegangen werden kann.
Bestehende Methoden zur Aufreinigung von Hepatozyten mittels FACS beinhalten die Einführung von Reportergenen stromabwärts von Promotoren, die für Hepatozyten-exprimierte Gene spezifisch sind. Dieser Ansatz birgt jedoch das potenzielle Risiko von Nebenwirkungen, die mit genetischen Veränderungen verbunden sind. Die mitochondriale Methode hingegen beseitigt solche Bedenken. Es wurden mehrere Zelloberflächenmarker von adulten oder fetalen Leberstamm-/Vorläuferzellen identifiziert18, und hepatische Vorläuferzellen, die aus humanen pluripotenten Stammzellen stammen, konnten anhand der CD13+CD133+ Population sortiert werden19. Andererseits konnte unter den aus pluripotenten Stammzellen gewonnenen Hepatozyten ein kleiner Teil der gereiften Hepatozyten mit dem ASGR1 (Asialoglycoprotein-Rezeptor 1) Antikörper17 sortiert werden, der der einzige Zelloberflächenmarker ist, der für Hepatozyten spezifisch ist. Es wurde gezeigt, dass die Population von Zellen mit hoher mitochondrialer und ALCAM-Positivität sowohl Populationen enthielt, die mit CD13+CD133+-Zellen markiert waren, als auch ASGR1+-Zellen. Dies deutet darauf hin, dass hochgradig mitochondriale und ALCAM-positive Populationen ein breites Spektrum an Hepatozyten mit unterschiedlichen Reifegraden enthalten können14. Folglich kann diese Methode die Reinigung einer Hepatozytenpopulation mit geringem Verlust ermöglichen.
Die Analyse ganzer Eingeweide des Mausfötus am embryonalen Tag 14.5 mit dieser Methode zeigte die Expression von ALCAM sowohl in Myozyten als auch in Hepatozyten14. Andere frühere Studien haben über die ALCAM-Expression in murinen Myokard-Vorläuferzellen berichtet21. Im Falle von humanen pluripotenten Stammzellen, die aus humanen pluripotenten Stammzellen gewonnen wurden, wurden die Kardiomyozyten in einer ALCAM-negativen Population identifiziert. Diese Daten zeigen, dass die ALCAM-Expression in kardialen und hepatischen Linien sowohl von der Spezies als auch von den Zellreifestadien abhängt. Aufgrund des höheren mitochondrialen Anteils in Kardiomyozyten im Vergleich zu Hepatozyten gelang es jedoch, die beiden Zelltypen mittels FACS auch von Mischzellen, die aus dem gesamten Mausembryo gewonnen wurden, zu trennen. Dies deutet darauf hin, dass die vorgeschlagene Methode für die Reinigung von Hepatozyten und Kardiomyozyten anderer Tierarten nützlich sein könnte.
Es wird erwartet, dass die hierin beschriebene Hepatozytenreinigungstechnologie eine entscheidende Rolle bei der Weiterentwicklung des Gebiets der regenerativen Medizin spielen wird, indem sie das potenzielle Risiko der Tumorbildung im Zusammenhang mit der Transplantation von humanen iPS-Zell-abgeleiteten Hepatozyten verringert. Die Negation einer teratombildenden Fähigkeit in der Hepatozytenpopulation, die durch diese Technik gereinigt wurde, wurde jedoch nicht durchgeführt. Weitere Studien zur Gewährleistung der Sicherheit müssen durchgeführt werden.
Große und adhärente Zellen wie Hepatozyten sind während der FACS-Sortierung sehr anfällig für mechanische Beschädigungen, was zu einem erheblichen Verlust lebensfähiger Zellen führt. Für klinische Anwendungen ist es unerlässlich, eine neue Aufreinigungsmethode zu entwickeln, die eine Kulturumgebung schafft, die ausschließlich das Überleben von Hepatozyten ermöglicht, wie z. B. die Laktatmethode zur Kardiomyozytenreinigung22.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Diese Arbeit wurde vom Ministerium für Bildung, Kultur, Sport, Wissenschaft und Technologie mit der Fördernummer [23390072] unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 253G1 human iPS cell line | RIKEN BioResource Research Center | HPS0002 | |
| 4% paraformaldehyde | FUJIFILM Wako Pure Chemical Corporation | 163-20145 | |
| Activin A Solution, Human, Recombinant | NACALAI TESQUE, INC. | 18585 | |
| Anti-Nuclei Antibody, clone 235-1 | Chemicon | MAB1281 | Antibody against human nuclear antigen |
| B-27 Supplement (50x), serum free | Thermo Fisher Scientific | 17504044 | |
| BD FACSAria III | BD Biosciences | Cell sorter | |
| CHIR-99021 | MedChemExpress | HY-10182 | |
| Ciclosporin A | FUJIFILM Wako Pure Chemical Corporation | 035-18961 | |
| Collagenase | FUJIFILM Wako Pure Chemical Corporation | 034-22363 | |
| Corning Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | Corning | 354230 | Gel-like basement membrane matrix |
| CultureSure Y-27632 | FUJIFILM Wako Pure Chemical Corporation | 036-24023 | ROCK inhibitor |
| Dexamethasone | FUJIFILM Wako Pure Chemical Corporation | 047-18863 | |
| Dimethyl sulfoxide (DMSO) | FUJIFILM Wako Pure Chemical Corporation | 047-29353 | |
| Donkey anti-Goat IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo Fisher Scientific | A-11055 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 546 | Thermo Fisher Scientific | A10036 | |
| Donkey anti-Rabbit IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo Fisher Scientific | A-21206 | |
| Falcon 5 mL Round Bottom Polystyrene Test Tube, with Cell Strainer Snap Cap | Corning | 352235 | |
| Fetal Bovine Serum | Biowest | 51820-500 | |
| GlutaMAX Supplement | Thermo Fisher Scientific | 35050061 | Dipeptide L-Alanyl-L-Glutamine |
| Human/Mouse/Rat/Canine ALCAM/CD166 Antibody | R&D Systems | AF1172 | |
| Hydrocortisone 21-hemisuccinate sodium salt | Sigma-Aldrich | H2270 | |
| iMatrix-511 silk | Nippi | 892021 | |
| ImmunoBlock | KAC | CTKN001 | Blocking solution |
| ITS-G Supplement(×100) | FUJIFILM Wako Pure Chemical Corporation | 090-06741 | |
| Leibovitz's L-15 Medium | FUJIFILM Wako Pure Chemical Corporation | 128-06075 | |
| N-Hexanoic-Try-Ile-(6)-amino Hexanoic amide (Dihexa) | Toronto Research Chemicals | H293745 | |
| Polyclonal Rabbit Anti-Human Albumin | Dako | A0001 | |
| Polyoxyethylene Sorbitan Monolaurate (Tween 20) | NACALAI TESQUE, INC. | 28353-85 | |
| RPMI-1640 | FUJIFILM Wako Pure Chemical Corporation | 189-02025 | |
| Sodium L-Ascorbate | NACALAI TESQUE, INC. | 03422-32 | |
| StemFit AK02N | REPROCELL | RCAK02N | |
| TBS (10x) | NACALAI TESQUE, INC. | 12748-31 | |
| Tetramethylrhodamine, methyl ester (TMRM) | Thermo Fisher Scientific | T668 | |
| Triton X-100 | NACALAI TESQUE, INC. | 28229-25 | |
| Trypan Blue Solution | NACALAI TESQUE, INC. | 20577-34 | |
| TRYPSIN 250 | Difco | 215240 | |
| Tryptose phosphate broth solution | Sigma-Aldrich | T8159 |
Referenzen
- Yamamoto, T., et al. Differentiation potential of pluripotent stem cells correlates to the level of CHD7. Scientific Reports. 8 (1), 241 (2018).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Reports. 10 (6), 1851-1866 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Reports. 16 (6), 1446-1457 (2021).
- Chen, C. X. -. Q., et al. Standardized quality control workflow to evaluate the reproducibility and differentiation potential of human iPSCs into neurons. bioRxiv. , (2021).
- Duinsbergen, D., Salvatori, D., Eriksson, M., Mikkers, H. Tumors originating from induced pluripotent stem cells and methods for their prevention. Annals of the New York Academy of Sciences. 1176, 197-204 (2009).
- Nori, S., et al. Long-term safety issues of iPSC-based cell therapy in a spinal cord injury model: oncogenic transformation with epithelial-mesenchymal transition. Stem Cell Reports. 4 (3), 360-373 (2015).
- Hentze, H., et al. Teratoma formation by human embryonic stem cells: evaluation of essential parameters for future safety studies. Stem Cell Research. 2 (3), 198-210 (2009).
- Anderson, D., et al. Transgenic enrichment of cardiomyocytes from human embryonic stem cells. Molecular Therapy. 15 (11), 2027-2036 (2007).
- Hidaka, K., et al. Chamber-specific differentiation of Nkx2.5-positive cardiac precursor cells from murine embryonic stem cells. The FASEB Journal. 17 (6), 740-742 (2003).
- Gassanov, N., Er, F., Zagidullin, N., Hoppe, U. C. Endothelin induces differentiation of ANP-EGFP expressing embryonic stem cells towards a pacemaker phenotype. The FASEB Journal. 18 (14), 1710-1712 (2004).
- Duan, Y., et al. Differentiation and enrichment of hepatocyte-like cells from human embryonic stem cells in vitro and in vivo. Stem Cells. 25 (12), 3058-3068 (2007).
- Takayama, K., et al. Enrichment of high-functioning human iPS cell-derived hepatocyte-like cells for pharmaceutical research. Biomaterials. 161, 24-32 (2018).
- Hattori, F., et al. Nongenetic method for purifying stem cell-derived cardiomyocytes. Nature methods. 7, 61-66 (2009).
- Yamashita, H., Fukuda, K., Hattori, F. Hepatocyte-like cells derived from human pluripotent stem cells can be enriched by a combination of mitochondrial content and activated leukocyte cell adhesion molecule. JMA Journal. 2 (2), 174-183 (2019).
- Fernandez-Vizarra, E., Enríquez, J., Pérez-Martos, A., Montoya, J., Fernández-Silva, P. Tissue-specific differences in mitochondrial activity and biogenesis. Mitochondrion. 11, 207-213 (2010).
- Swart, G. W. M. Activated leukocyte cell adhesion molecule (CD166/ALCAM): Developmental and mechanistic aspects of cell clustering and cell migration. European Journal of Cell Biology. 81 (6), 313-321 (2002).
- Peters, D. T., et al. Asialoglycoprotein receptor 1 is a specific cell-surface marker for isolating hepatocytes derived from human pluripotent stem cells. Development. 143 (9), 1475-1481 (2016).
- Kamiya, A., Inagaki, Y. Stem and progenitor cell systems in liver development and regeneration. Hepatology Research. 45 (1), 29-37 (2015).
- Yanagida, A., Ito, K., Chikada, H., Nakauchi, H., Kamiya, A. An In vitro expansion system for generation of human ips cell-derived hepatic progenitor-like cells exhibiting a bipotent differentiation potential. PLOS One. 8 (7), e67541 (2013).
- Siller, R., Greenhough, S., Naumovska, E., Sullivan, G. J. Small-molecule-driven hepatocyte differentiation of human pluripotent stem cells. Stem Cell Reports. 4 (5), 939-952 (2015).
- Hirata, H., et al. ALCAM (CD166) is a surface marker for early murine cardiomyocytes. Cells, Tissues, Organs. 184 (3-4), 172-180 (2006).
- Tohyama, S., et al. Distinct metabolic flow enables large-scale purification of mouse and human pluripotent stem cell-derived cardiomyocytes. Cell Stem Cell. 12 (1), 127-137 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.
