Method Article
Application de la coloration des mitochondries vivantes dans le tri cellulaire pour purifier les hépatocytes dérivés de cellules souches pluripotentes induites par l’homme
Dans cet article
Résumé
Les hépatocytes dérivés de cellules souches pluripotentes peuvent être purifiés par tri cellulaire, en utilisant une combinaison de coloration de la molécule d’adhésion des cellules leucocytaires activées (ALCAM, également connue sous le nom de CD166).
Résumé
Les cellules souches embryonnaires humaines (SE) et les cellules souches pluripotentes induites (iPS) ont des applications potentielles en médecine régénérative cellulaire pour traiter les organes gravement malades en raison de leur prolifération illimitée et de leurs propriétés pluripotentes. Cependant, il est difficile de différencier les cellules ES/iPS humaines en types de cellules cibles 100 % pures en raison de leur grande sensibilité à l’environnement. La tumorigenèse après transplantation est causée par des cellules contaminées, proliférantes et indifférenciées, ce qui rend la technologie de haute purification essentielle pour la réalisation en toute sécurité de la médecine régénérative. Pour atténuer le risque de tumorigenèse, une technologie de purification élevée a été développée pour les hépatocytes dérivés de cellules iPS humaines. La méthode utilise le FACS (tri cellulaire activé par fluorescence) en utilisant une combinaison de contenu mitochondrial élevé et du marqueur de surface cellulaire ALCAM (molécule d’adhésion cellulaire leucocytaire activée) sans modification génétique. 97 % ± 0,38 % (n = 5) des hépatocytes purifiés utilisant cette méthode présentaient une expression protéique de l’albumine. Cet article vise à fournir des procédures détaillées pour cette méthode, telle qu’appliquée à la méthode de différenciation bidimensionnelle la plus récente pour les cellules iPS humaines en hépatocytes.
Introduction
Les cellules souches embryonnaires et pluripotentes induites (ES et iPS, respectivement) sont considérées comme des sources cellulaires prometteuses pour les thérapies régénératives. Cependant, l’efficacité de la différenciation de ces cellules en types de cellules cibles spécifiques peut varier, même en utilisant la même lignée cellulaire, le même protocole et le même expérimentateur 1,2,3,4. Cette variabilité peut être attribuée à la grande sensibilité des cellules ES/iPS humaines à leur environnement. Par conséquent, il est actuellement difficile d’obtenir de manière cohérente des cellules cibles pures. Pour parvenir à une médecine régénérative hautement sûre, il est crucial d’éliminer les cellules prolifératives et les cellules souches indifférenciées dans les cellules thérapeutiques, et une technologie de purification avancée pour les cellules cibles est essentielle 5,6,7.
Un trieur de cellules est un appareil qui analyse instantanément les cellules individuelles et trie les cellules vivantes d’intérêt en fonction de l’intensité du signal fluorescent, offrant ainsi une solution prometteuse. Cela peut être accompli par la coloration par anticorps de marqueurs de surface spécifiques au type cellulaire ou en utilisant des expressions de gènes rapporteurs spécifiques au type cellulaire. En utilisant cette technique, il existe plusieurs rapports sur les méthodes de purification des cardiomyocytes 8,9,10 et des hépatocytes11,12 dérivés de cellules souches pluripotentes. Hattori et al. ont développé une méthode innovante de purification mitochondriale à l’aide d’un trieur de cellules13. Tirant parti du fait que les cardiomyocytes ont des besoins énergétiques élevés par le biais de l’activité mitochondriale, la coloration des cellules avec le colorant TMRM (ester méthylique de tétraméthylrhodamine) peut être utilisée pour marquer et purifier fortement les cardiomyocytes par FACS à partir de corps embryoïdes dérivés de cellules ES humaines contenant divers types de cellules. L’absence de tumorigénicité a été confirmée par des tests de formation de tératomes avec les cardiomyocytes purifiés. De plus, Yamashita et al. ont découvert de manière inattendue une méthode pour purifier les hépatocytes des corps embryoïdes dérivés de cellules ES humaines en isolant des fractions à forte activité mitochondriale et à expression ALCAM-positive14. La raison de cette méthode est que les hépatocytes ont également un nombre relativement élevé de mitochondries en raison de leur forte consommation d’ATP pour le métabolisme des nutriments et la détoxification15, et les hépatocytes expriment ALCAM, un membre de la superfamille des immunoglobulines, qui joue un rôle dans l’adhésion et la migration cellulaires16.
Les méthodes précédentes de purification élevée pour les hépatocytes dérivés de cellules souches pluripotentes nécessitaient des modifications génétiques, et les méthodes de purification non génétiques avaient une faible efficacité17. La méthode mitochondriale non génétique a le mérite d’atteindre une grande pureté. Lorsque des cellules progénitrices hépatiques sont nécessaires, il est possible de choisir des méthodes basées sur CD133 et CD13 ou Dlk118,19. Bien que la précision de la technologie d’édition du génome ait progressé, le risque potentiel de changements génomiques imprévus (par exemple, la cancérogenèse) ne peut pas être entièrement éliminé. Les méthodes basées sur l’activité mitochondriale, sans impliquer de modification génétique, peuvent être exemptes de tels risques.
À la suite de l’examen de divers indicateurs mitochondriaux dans les cardiomyocytes néonatals de rats, on a observé que la TMRM disparaissait complètement en 24 heures, tandis que les autres colorants restaient pendant au moins 5 jours13. De plus, il est important de noter que TMRM et JC-1 n’ont pas eu d’impact sur la viabilité cellulaire lorsqu’ils ont été évalués à l’aide de l’essai au bromure de 3-(4,5-diméthyl-thiazol-2-yl)-2,5-diphényltétrazolium (MTT), tandis que d’autres colorants ont démontré des effets variables sur la viabilité cellulaire13. TMRM démontre une sécurité accrue.
À ce jour, plusieurs méthodes de différenciation bidimensionnelle (2D) ont été développées comme approches plus efficaces pour induire la différenciation par rapport à la formation de corps embryoïdes tridimensionnels (3D). En effet, les composés ou cytokines induisant la différenciation par étapes peuvent être administrés uniformément aux cellules sur un plan 2D, plutôt que dans un espace 3D. Dans cette étude, la méthode20 précédemment rapportée a été modifiée pour induire la différenciation en hépatocytes dérivés de cellules iPS humaines. Nous décrirons ici les détails des procédures de différenciation et de purification 2D modernes des hépatocytes dérivés de cellules iPS humaines.
Protocole
Cette étude a utilisé des cellules iPS humaines obtenues commercialement (souche 253G1) (voir le tableau des matériaux).
1. Maintenance des cellules iPS humaines
- Maintenir les cellules iPS humaines dans des conditions exemptes de feeder dans un milieu AK02 sur des boîtes de culture recouvertes de 0,25 μg/cm2 iMatrix-511 (voir le tableau des matériaux).
2. Différenciation hépatique des cellules iPS humaines dans des cultures 2D
REMARQUE : Reportez-vous à la figure 1A pour le calendrier quotidien de différenciation hépatique. Tout au long du processus, utilisez des plaques de culture à 6 puits et ajoutez 2 ml du milieu de lavage ou de culture désigné dans chaque puits. Maintenir l’état de culture cellulaire à 100 % d’humidité, 5 % de CO2 et 20 % d’O2 à 37 °C.
- Enduire chaque puits d’une plaque de 6 puits avec 1 mL de matrice de membrane basale (diluée 1/25 dans du PBS (-)), et incuber à 37 °C pendant 1 h.
- Passage de cellules iPS humaines sur la boîte enrobée à une densité de 20 000 à 30 000 cellules/cm² à l’aide d’un milieu AK02N complété par un inhibiteur de ROCK 10 μM (Y-27632) (voir le tableau des matériaux). Remplacer le milieu sans Y-27632 le lendemain et cultiver pendant 2 jours supplémentaires.
- Lavez les cellules une fois avec le milieu RPMI-1640. Amorcer la différenciation de l’endoderme en remplaçant le milieu par du RPMI-B27-Glu (RPMI-1640 + 2 % d’insuline B27 + 1 % de dipeptide L-Alanyl-L-Glutamine) complété par 3-6 μM de CHIR-99021 et 100 ng/mL d’Activin A pendant 24 h (voir le tableau des matériaux).
- Remplacer le milieu par du RPMI-B27-Glu complété par 50 ng/mL d’Activin A pendant 24 h.
- Pour induire la différenciation des progéniteurs hépatiques, remplacez le milieu par du RPMI-B27-Glu complété par 1 % de diméthylsulfoxyde (DMSO) pendant 5 jours.
- Pour favoriser la maturation des hépatocytes, passez au milieu de maturation des hépatocytes : le milieu L-15 de Leibovitz contenant 10 μM d’hydrocortisone 21-hémisuccinate, 8,3 % de bouillon de phosphate tryptose, 100 nM de dexaméthasone (DEX), 50 μg/mL de L-ascorbate de sodium, 0,58 % d’insuline-transferrine-sélénium (ITS), 8,3 % de sérum de veau fœtal (FBS), 2 mM de dipeptide L-Alanyl-L-glutamine et 100 nM de N-Hexanoic-Try-Ile-(6)-amino hexanoïque amide (Dihexa) (voir le tableau des matériaux).
- Changez de milieu tous les 2 jours pendant 20 jours. Triez les cellules environ 27 jours après le début de la différenciation.
3. Préparation cellulaire pour la purification d’hépatocytes dérivés de cellules iPS humaines
- Laver les cellules hépatiques différenciées avec 2 mL de PBS (-) par puits.
- Ajouter 1 mL de solution enzymatique par puits, qui comprend 0,1 % de collagénase, 0,083 % de trypsine, 10 μM d’inhibiteur de ROCK (Y-27632), 20 nM de ciclosporine A et 50 μg/mL de sodium-L-ascorbate dans le tampon Ads (116 mM de NaCl, 12,5 mM de NaH2PO4, 20 mM d’HEPES, 5,4 mM de KCl, 5,6 mM de glucose et 0,8 mM de MgSO4 ; pH de 7,35) (voir le tableau des matériaux). Détachez les cellules des plaques en les tournant horizontalement à 37 °C pendant environ 2 h.
- Après avoir vérifié que les cellules sont détachées et arrondies au microscope, dispersez-les dans des cellules uniques par pipetage doux et recueillez-les dans un tube de 50 ml.
- Centrifuger les cellules à 200 x g pendant 5 min à 18 °C pour les granuler. Retirez le surnageant à l’aide d’un aspirateur.
- Teinter les mitochondries des cellules en les dispersant dans 5 mL de milieu de maturation hépatocytaire contenant 100 nM de TMRM (tétraméthylrhodamine, ester de méthlique) (voir Tableau des matières). Incuber 30 min à 37 °C à l’abri de la lumière en l’enveloppant dans du papier aluminium.
- Centrifugez les cellules à 200 x g pendant 5 min à 18 °C pour les granuler. Retirez le surnageant.
- Remettre les cellules en suspension avec 1 à 5 ml de tampon Ads froid contenant 2 % de FBS.
- Comptez le numéro de cellule à l’aide d’une solution de bleu de trypan à 0,4 % et d’une plaque de numération cellulaire.
- Centrifugez les cellules à 200 x g pendant 5 min à 4 °C pour les granuler. Retirez le surnageant.
- Diluer un anticorps anti-ALCAM (anticorps primaire, voir tableau des matériaux) dans un rapport de 1:50 dans le tampon Ads, et ajouter 100 μL de l’anticorps dilué par 106 cellules. Teintez les cellules pendant 50 min sur de la glace. Préparez des cellules qui ne sont pas colorées avec l’anticorps primaire comme contrôle négatif.
- Lavez deux fois les cellules avec un tampon Ads froid contenant 2 % de FBS.
- Diluer une IgG anti-chèvre d’âne Alexa Fluor 488 (anticorps secondaire, voir tableau des matériaux) à un rapport de 1:100 dans le tampon Ads, et ajouter 100 μL de l’anticorps dilué par 106 cellules. Teignez les cellules pendant 30 min sur de la glace. Cellules colorantes qui n’ont pas été colorées avec l’anticorps primaire à l’aide de l’anticorps secondaire.
- Lavez deux fois les cellules avec un tampon Ads froid contenant 2 % de FBS.
- Résuspendez les cellules dans la mémoire tampon Ads et filtrez-les à l’aide d’une crépine de cellule à capuchon encliquetable (maille de 35 μm) juste avant le tri par FACS pour éliminer les gros amas de cellules et les débris.
4. Analyses FACS et tri des hépatocytes dérivés de cellules iPS humaines
- Configurez la machine FACS pour le tri des cellules en suivant les instructions du fabricant (voir le tableau des matériaux).
REMARQUE : Utilisez une buse de 85 μm ou 100 μm et un filtre ND de 2,0 pour les hépatocytes. Utilisez le canal FITC pour détecter Alexa Flour 488 pour ALCAM et le canal PE pour détecter la fluorescence TMRM pour les mitochondries. - Ajustez les tensions FSC (diffusion directe) et SSC (diffusion latérale) pour visualiser correctement la population de cellules.
- Ajustez les tensions des canaux fluorescents FITC et PE. Commencez par les cellules non étiquetées et centrez la population de cellules au bas de chaque graphique de canaux. Assurez-vous que les cellules étiquetées sont correctement positionnées dans le tracé.
- Utilisez une stratégie de contrôle courante pour éliminer les doublets : Gate le FSC-A majeur vs. Population du CSS-A (figure 2A), suivie de la population FSC-H c. -W, puis SSC-H vs. -W (Figure 2B,C).
- Gate la population TMRMhi et ALCAM+ (P2 sur la figure 2E) pour trier les hépatocytes. Gate la population TMRMhi et ALCAM- (P1 dans la figure 2E) pour trier les non-hépatocytes. Prélever les hépatocytes et les non-hépatocytes triés dans des tubes de collecte de 15 ml contenant du milieu de maturation des hépatocytes.
- Centrifuger le tube de prélèvement contenant les cellules triées à 200 x g pendant 5 min à 18 °C.
- Retirez le surnageant et remettez les cellules en suspension dans le milieu de maturation des hépatocytes.
- Ensemencez les cellules sur des fibroblastes embryonnaires de souris (MEF) traités à la mitomycine C ou sur une matrice de membrane basale semblable à un gel avec un milieu de maturation hépatocytaire complété par 10 μM d’Y-27632 et 20 nM de ciclosporine A. Cultivez les cellules dans un incubateur deCO2 à 37 °C pendant 5 jours.
5. Immunocytochimie pour la détection des hépatocytes dérivés de cellules iPS humaines
- Laver les cellules cultivées pendant 5 jours après le tri avec du PBS (-).
- Fixez les cellules avec 4% de paraformaldéhyde pendant 5 min à température ambiante.
- Lavez deux fois les cellules avec un tampon TBS-T (1x TBS avec 1% de Tween 20) pour éliminer le paraformaldéhyde.
- Incuber les cellules avec 0,1 % de tampon TBS-T dilué Triton X-100 pendant 5 min à température ambiante.
- Lavez deux fois les cellules avec le tampon TBS-T pour retirer le Triton X-100.
- Ajouter une solution de blocage disponible dans le commerce (voir le tableau des matériaux) dans les cellules pendant 30 minutes à température ambiante.
- Ajouter des anticorps primaires contre l’albumine (marqueur hépatocytaire) et l’antigène nucléaire humain (hNA, voir tableau des matériaux), dilués 1:50 dans une solution bloquante.
- Incuber les cellules à 4 °C pendant la nuit. Lavez deux fois les cellules avec le tampon TBS-T.
- Ajouter des anticorps secondaires : Alexa Fluor 488 âne anti-IgG lapin et Alexa Fluor 546 âne anti-souris IgG, dilués 1:100 dans une solution bloquante (voir tableau des matériaux).
- Incuber les cellules à température ambiante pendant 30 min. Lavez deux fois les cellules avec le tampon TBS-T.
- Évaluez la pureté des hépatocytes en comptant les cellules positives à l’albumine et aux hNA positives au microscope à fluorescence. Calculez le pourcentage de cellules positives à l’albumine par cellule hNA positive pour les populations P1 et P2.
Résultats
La chronologie des processus induisant les cellules iPS humaines à se différencier en hépatocytes par culture 2D (Figure 1A) et les caractéristiques cellulaires représentatives (Figure 1B) sont présentées. Approximativement au 12e jour de différenciation, les cellules ont commencé à présenter des formes cellulaires polygonales et des noyaux ronds, caractéristiques des hépatocytes. Certains hépatocytes ont également montré une multinucléation.
L’analyse FACS a été réalisée à l’aide des cellules au jour 27 de la différenciation. Pour identifier la population ALCAM+, des graphiques comparatifs avec et sans le premier anticorps anti-ALCAM sont présentés respectivement dans les figures 2D et E. En induisant la différenciation hépatique par culture 2D, la population d’hépatocytes (P2) s’est avérée être de 10,1 % dans cette expérience, ce qui représente une augmentation significative par rapport aux 0,8 % (données non présentées) trouvés en utilisant la méthode de formation du corps embryoïde 3D.
Les cellules P1 et P2 qui avaient été triées ont été cultivées sur des fibroblastes embryonnaires de souris traités avec de la mitomycine C. Les cellules P2 présentaient une formation de colonie compacte et sphérique, tandis que les cellules P1 étaient dispersées et avaient une morphologie plus plate. La coloration immunohistochimique de l’antigène nucléaire humain (hNA) et de l’albumine a confirmé l’enrichissement des cellules hépatocytaires en P2, comme le montre la figure 3. Pour évaluer la pureté des hépatocytes dans les cellules P1 et P2, le rapport entre les cellules positives à l’albumine et les cellules positives à l’hNA a été calculé (n = 5). Les résultats ont révélé que 0 % des cellules P1 et 97 % ± 0,38 % des cellules P2 étaient dérivées d’iPSC humaines et présentaient des caractéristiques semblables à celles des hépatocytes (Figure 3).
Pour confirmer le bon tri des hépatocytes au cours de l’expérience FACS, il est recommandé de co-colorer ASGR1 avec son anticorps spécifique conjugué à différents fluorophores et de confirmer que P2 contient des cellules ASGR1-positives.
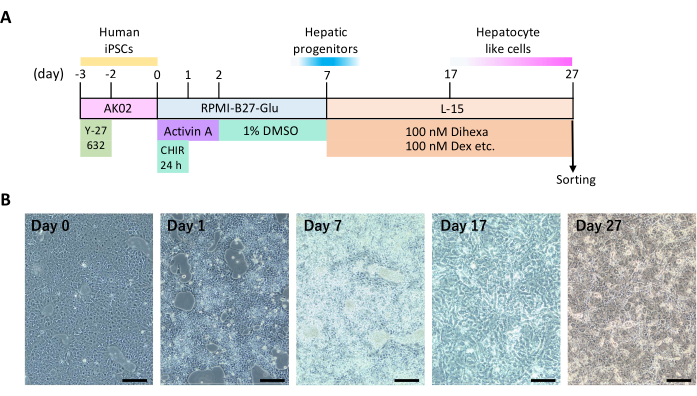
Figure 1 : Différenciation hépatique 2D des CSPi humaines. (A) La chronologie de la différenciation hépatique. Cette figure est adaptée de Yamashita et al.14. (B) Changements morphologiques au cours de la différenciation hépatique. Barres d’échelle = 200 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Stratégie de contrôle pour le tri des hépatocytes et des non-hépatocytes dans les cellules hépatiques différenciées (jour 27). (A) Déclenchement de la population cellulaire cible à l’aide du FSC-A vs. Complot SSC-A. (B) Augmenter la population fermée à partir de l’étape (A) à l’aide du FSC-H vs. FSC-W tracer et exclure les cellules doublets. (C) Augmenter davantage la population fermée à partir de l’étape (B) à l’aide du SSC-H vs. Tracé SSC-W et à l’exclusion des cellules doublets. (D) et (E) Expansion de la population fermée à partir de l’étape (C) à l’aide du FITC-A (ALCAM) vs. Parcelle PE-A (TMRM). (D) Cellules colorées avec TMRM et le 2èmeanticorps. (E) Cellules colorées avec TMRM et les anticorps 1er et 2e. Veuillez cliquer ici pour voir une version agrandie de cette figure.
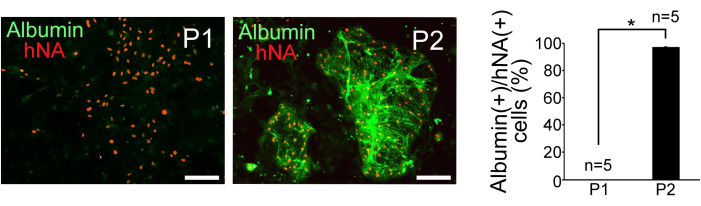
Figure 3 : Résultats d’immunohistochimie. Détection immunohistochimique de l’albumine et de l’antigène nucléaire humain (hNA) dans les cellules P1 et P2 triées, et quantification de la fraction cellulaire de l’albumine (+) dans les cellules hNA (+). Barres d’échelle = 100 μm. Les données sont représentées sous forme de moyenne ± d’écart-type (ET) (n = 5). L’analyse statistique a été effectuée à l’aide du test t de Student. *P < 0,05. Cette figure est adaptée de Yamashita et al.14. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
En raison de leurs fonctions dans le métabolisme des nutriments et la détoxification, les hépatocytes possèdent un nombre relativement important de mitochondries par rapport aux autres types de cellules15. ALCAM fait partie de la superfamille des immunoglobulines et joue un rôle dans l’adhésion et la migration cellulaires. Il est exprimé dans divers types de cellules, notamment les cellules hépatiques, épithéliales, lymphoïdes, myéloïdes, fibroblastiques et neuronales16. En utilisant une combinaison de la méthode basée sur les mitochondries et de l’anticorps ALCAM, les hépatocytes dérivés de cellules iPS humaines ont été identifiés avec succès.
Bien que les tests visant à caractériser la fonction sécrétoire ou l’activité enzymatique des hépatocytes purifiés n’aient pas été effectués, l’expression de l’ARNm du gène CYP3A4 dans les hépatocytes dérivés de cellules iPS humaines purifiés a été vérifiée. De plus, la population d’hépatocytes avec TMRM (élevée) et ALCAM (+) isolée à partir de corps embryoïdes dérivés de cellules ES humaines, qui a été observée des expressions plus élevées des ensembles de gènes associés aux hépatocytes (VTN, SERPINA1, CYP1A1, FGB, FGA, FGG, AFP, ALB et APOB) par rapport aux cardiomyocytes dérivés des cellules ES14.
Une étape critique du protocole consiste à détacher doucement les cellules hépatiques différenciées 2D des boîtes de culture avec un minimum de dommages tout en assurant la préservation de l’activité mitochondriale pour la coloration TMRM. Selon l’état de différenciation, les hépatocytes peuvent présenter une résistance au détachement de la boîte même avec un traitement enzymatique, un défi qui peut être efficacement résolu par l’ajout de 0,25 mM d’EDTA.
Les méthodes existantes de purification des hépatocytes via FACS impliquent l’introduction de gènes rapporteurs en aval des promoteurs spécifiques des gènes exprimés par les hépatocytes. Cependant, cette approche comporte un risque potentiel d’effets secondaires associés aux modifications génétiques. La méthode mitochondriale, en revanche, élimine ces préoccupations. Plusieurs marqueurs de surface cellulaire de cellules souches/progénitrices hépatiques adultes ou fœtales ont été identifiés18, et les cellules progénitrices hépatiques dérivées de cellules souches pluripotentes humaines ont pu être triées à l’aide de la population CD13+CD133+19. D’autre part, parmi les hépatocytes pluripotents dérivés de cellules souches, une petite fraction des hépatocytes matures a pu être triée à l’aide de l’anticorpsASGR1 (récepteur 1 de l’asialoglycoprotéine 1), qui est le seul marqueur de surface cellulaire spécifique aux hépatocytes. Il a été démontré que la population de cellules présentant une positivité mitochondriale élevée et ALCAM contenait à la fois des populations marquées par des cellules CD13+CD133+ et des cellules ASGR1+. Cela suggère que les populations mitochondriales élevées et ALCAM positives peuvent contenir une large gamme d’hépatocytes de différentes maturités14. Par conséquent, cette méthode peut permettre de purifier une population d’hépatocytes avec peu de pertes.
L’analyse de viscères entiers de fœtus de souris au jour embryonnaire 14,5 à l’aide de cette méthode a révélé l’expression d’ALCAM dans les myocytes et les hépatocytes14. D’autres études antérieures ont rapporté l’expression d’ALCAM dans les cellules progénitrices du myocarde murin21. Dans le cas de cellules dérivées de cellules souches pluripotentes humaines, les cardiomyocytes ont été identifiés dans la population ALCAM négative. Ces données montrent que l’expression d’ALCAM dans les lignées cardiaques et hépatiques diffère selon l’espèce et les stades de maturité cellulaire. Cependant, en raison de la fraction mitochondriale plus élevée dans les cardiomyocytes par rapport aux hépatocytes, il a été réussi à séparer les deux types de cellules par FACS, même à partir de cellules mixtes dérivées de l’embryon entier de souris. Cela suggère que la méthode proposée pourrait être utile pour purifier les hépatocytes et les cardiomyocytes d’autres espèces animales.
La technologie de purification des hépatocytes décrite dans le présent document devrait jouer un rôle central dans l’avancement du domaine de la médecine régénérative en atténuant le risque potentiel de formation de tumeurs associé à la transplantation d’hépatocytes dérivés de cellules iPS humaines. Cependant, la négation d’une capacité de formation de tératomes dans la population d’hépatocytes purifiée par cette technique n’a pas été faite. D’autres études doivent être menées pour assurer la sécurité.
Les cellules grandes et adhérentes telles que les hépatocytes sont très vulnérables aux dommages mécaniques lors du tri FACS, ce qui entraîne une perte importante de cellules viables. Pour les applications cliniques, il est essentiel de développer une nouvelle méthode de purification qui établit un environnement de culture permettant la survie des hépatocytes exclusivement comme la méthode du lactate pour la purification des cardiomyocytes22.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par le ministère de l’Éducation, de la Culture, des Sports, de la Science et de la Technologie n° de subvention [23390072].
matériels
| Name | Company | Catalog Number | Comments |
| 253G1 human iPS cell line | RIKEN BioResource Research Center | HPS0002 | |
| 4% paraformaldehyde | FUJIFILM Wako Pure Chemical Corporation | 163-20145 | |
| Activin A Solution, Human, Recombinant | NACALAI TESQUE, INC. | 18585 | |
| Anti-Nuclei Antibody, clone 235-1 | Chemicon | MAB1281 | Antibody against human nuclear antigen |
| B-27 Supplement (50x), serum free | Thermo Fisher Scientific | 17504044 | |
| BD FACSAria III | BD Biosciences | Cell sorter | |
| CHIR-99021 | MedChemExpress | HY-10182 | |
| Ciclosporin A | FUJIFILM Wako Pure Chemical Corporation | 035-18961 | |
| Collagenase | FUJIFILM Wako Pure Chemical Corporation | 034-22363 | |
| Corning Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | Corning | 354230 | Gel-like basement membrane matrix |
| CultureSure Y-27632 | FUJIFILM Wako Pure Chemical Corporation | 036-24023 | ROCK inhibitor |
| Dexamethasone | FUJIFILM Wako Pure Chemical Corporation | 047-18863 | |
| Dimethyl sulfoxide (DMSO) | FUJIFILM Wako Pure Chemical Corporation | 047-29353 | |
| Donkey anti-Goat IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo Fisher Scientific | A-11055 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 546 | Thermo Fisher Scientific | A10036 | |
| Donkey anti-Rabbit IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo Fisher Scientific | A-21206 | |
| Falcon 5 mL Round Bottom Polystyrene Test Tube, with Cell Strainer Snap Cap | Corning | 352235 | |
| Fetal Bovine Serum | Biowest | 51820-500 | |
| GlutaMAX Supplement | Thermo Fisher Scientific | 35050061 | Dipeptide L-Alanyl-L-Glutamine |
| Human/Mouse/Rat/Canine ALCAM/CD166 Antibody | R&D Systems | AF1172 | |
| Hydrocortisone 21-hemisuccinate sodium salt | Sigma-Aldrich | H2270 | |
| iMatrix-511 silk | Nippi | 892021 | |
| ImmunoBlock | KAC | CTKN001 | Blocking solution |
| ITS-G Supplement(×100) | FUJIFILM Wako Pure Chemical Corporation | 090-06741 | |
| Leibovitz's L-15 Medium | FUJIFILM Wako Pure Chemical Corporation | 128-06075 | |
| N-Hexanoic-Try-Ile-(6)-amino Hexanoic amide (Dihexa) | Toronto Research Chemicals | H293745 | |
| Polyclonal Rabbit Anti-Human Albumin | Dako | A0001 | |
| Polyoxyethylene Sorbitan Monolaurate (Tween 20) | NACALAI TESQUE, INC. | 28353-85 | |
| RPMI-1640 | FUJIFILM Wako Pure Chemical Corporation | 189-02025 | |
| Sodium L-Ascorbate | NACALAI TESQUE, INC. | 03422-32 | |
| StemFit AK02N | REPROCELL | RCAK02N | |
| TBS (10x) | NACALAI TESQUE, INC. | 12748-31 | |
| Tetramethylrhodamine, methyl ester (TMRM) | Thermo Fisher Scientific | T668 | |
| Triton X-100 | NACALAI TESQUE, INC. | 28229-25 | |
| Trypan Blue Solution | NACALAI TESQUE, INC. | 20577-34 | |
| TRYPSIN 250 | Difco | 215240 | |
| Tryptose phosphate broth solution | Sigma-Aldrich | T8159 |
Références
- Yamamoto, T., et al. Differentiation potential of pluripotent stem cells correlates to the level of CHD7. Scientific Reports. 8 (1), 241(2018).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Reports. 10 (6), 1851-1866 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Reports. 16 (6), 1446-1457 (2021).
- Chen, C. X. -Q., et al. Standardized quality control workflow to evaluate the reproducibility and differentiation potential of human iPSCs into neurons. bioRxiv. , (2021).
- Duinsbergen, D., Salvatori, D., Eriksson, M., Mikkers, H. Tumors originating from induced pluripotent stem cells and methods for their prevention. Annals of the New York Academy of Sciences. 1176, 197-204 (2009).
- Nori, S., et al. Long-term safety issues of iPSC-based cell therapy in a spinal cord injury model: oncogenic transformation with epithelial-mesenchymal transition. Stem Cell Reports. 4 (3), 360-373 (2015).
- Hentze, H., et al. Teratoma formation by human embryonic stem cells: evaluation of essential parameters for future safety studies. Stem Cell Research. 2 (3), 198-210 (2009).
- Anderson, D., et al. Transgenic enrichment of cardiomyocytes from human embryonic stem cells. Molecular Therapy. 15 (11), 2027-2036 (2007).
- Hidaka, K., et al. Chamber-specific differentiation of Nkx2.5-positive cardiac precursor cells from murine embryonic stem cells. The FASEB Journal. 17 (6), 740-742 (2003).
- Gassanov, N., Er, F., Zagidullin, N., Hoppe, U. C. Endothelin induces differentiation of ANP-EGFP expressing embryonic stem cells towards a pacemaker phenotype. The FASEB Journal. 18 (14), 1710-1712 (2004).
- Duan, Y., et al. Differentiation and enrichment of hepatocyte-like cells from human embryonic stem cells in vitro and in vivo. Stem Cells. 25 (12), 3058-3068 (2007).
- Takayama, K., et al. Enrichment of high-functioning human iPS cell-derived hepatocyte-like cells for pharmaceutical research. Biomaterials. 161, 24-32 (2018).
- Hattori, F., et al. Nongenetic method for purifying stem cell-derived cardiomyocytes. Nature methods. 7, 61-66 (2009).
- Yamashita, H., Fukuda, K., Hattori, F. Hepatocyte-like cells derived from human pluripotent stem cells can be enriched by a combination of mitochondrial content and activated leukocyte cell adhesion molecule. JMA Journal. 2 (2), 174-183 (2019).
- Fernandez-Vizarra, E., Enríquez, J., Pérez-Martos, A., Montoya, J., Fernández-Silva, P. Tissue-specific differences in mitochondrial activity and biogenesis. Mitochondrion. 11, 207-213 (2010).
- Swart, G. W. M. Activated leukocyte cell adhesion molecule (CD166/ALCAM): Developmental and mechanistic aspects of cell clustering and cell migration. European Journal of Cell Biology. 81 (6), 313-321 (2002).
- Peters, D. T., et al. Asialoglycoprotein receptor 1 is a specific cell-surface marker for isolating hepatocytes derived from human pluripotent stem cells. Development. 143 (9), Cambridge, England. 1475-1481 (2016).
- Kamiya, A., Inagaki, Y. Stem and progenitor cell systems in liver development and regeneration. Hepatology Research. 45 (1), 29-37 (2015).
- Yanagida, A., Ito, K., Chikada, H., Nakauchi, H., Kamiya, A. An In vitro expansion system for generation of human ips cell-derived hepatic progenitor-like cells exhibiting a bipotent differentiation potential. PLOS One. 8 (7), e67541(2013).
- Siller, R., Greenhough, S., Naumovska, E., Sullivan, G. J. Small-molecule-driven hepatocyte differentiation of human pluripotent stem cells. Stem Cell Reports. 4 (5), 939-952 (2015).
- Hirata, H., et al. ALCAM (CD166) is a surface marker for early murine cardiomyocytes. Cells, Tissues, Organs. 184 (3-4), 172-180 (2006).
- Tohyama, S., et al. Distinct metabolic flow enables large-scale purification of mouse and human pluripotent stem cell-derived cardiomyocytes. Cell Stem Cell. 12 (1), 127-137 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon