Method Article
Valutazione non distruttiva della densità cellulare regionale all'interno degli aggregati tumorali dopo il trattamento farmacologico
In questo articolo
Riepilogo
Il presente protocollo sviluppa una tecnica basata su immagini per la misurazione rapida, non distruttiva e priva di etichette della densità cellulare regionale e della vitalità all'interno di aggregati tumorali 3D. I risultati hanno rivelato un gradiente di densità cellulare, con densità cellulari più elevate nelle regioni centrali rispetto agli strati esterni nello sviluppo di aggregati e morte cellulare prevalentemente periferica negli aggregati HER2+ trattati con Trastuzumab.
Abstract
I modelli di sferoidi tumorali multicellulari (MCTS) hanno dimostrato una crescente utilità per lo studio in vitro della progressione del cancro e della scoperta di farmaci. Questi costrutti avascolare relativamente semplici imitano aspetti chiave dei tumori in vivo , come la struttura 3D e i gradienti fisiopatologici. I modelli MCTS possono fornire informazioni sul comportamento delle cellule tumorali durante lo sviluppo di sferoidi e in risposta ai farmaci; tuttavia, le loro dimensioni richieste limitano drasticamente gli strumenti utilizzati per la valutazione non distruttiva. L'imaging strutturale della tomografia a coerenza ottica e il software di analisi 3D Imaris sono esplorati per la misurazione rapida, non distruttiva e senza etichette della densità cellulare regionale all'interno degli MCTS. Questo approccio viene utilizzato per valutare gli MCTS in un periodo di maturazione di 4 giorni e durante un trattamento prolungato di 5 giorni con Trastuzumab, un farmaco anti-HER2 clinicamente rilevante. In breve, gli MCTS per il cancro al seno AU565 HER2+ sono stati creati tramite sovrapposizione liquida con o senza l'aggiunta di Matrigel (una matrice di membrana basale) per esplorare aggregati di diverse morfologie (aggregati 2.5D più spessi, simili a dischi o aggregati 2D piatti, rispettivamente). La densità cellulare all'interno della regione esterna, della regione di transizione e del nucleo interno è stata caratterizzata in MCTS maturi, rivelando un gradiente di densità cellulare con densità cellulari più elevate nelle regioni centrali rispetto agli strati esterni. L'aggiunta della matrice ha ridistribuito la densità cellulare e migliorato questo gradiente, diminuendo la densità della zona esterna e aumentando la compattazione cellulare nei nuclei. La densità cellulare è stata quantificata dopo il trattamento farmacologico (0 h, 24 h, 5 giorni) all'interno di zone progressivamente più profonde di 100 μm per valutare le potenziali differenze regionali nella risposta al farmaco. Nel punto temporale finale, quasi tutta la morte cellulare sembrava essere vincolata ai 200 μm esterni di ciascun aggregato, mentre le cellule più profonde nell'aggregato apparivano in gran parte inalterate, illustrando le differenze regionali nella risposta al farmaco, probabilmente a causa di limitazioni nella penetrazione del farmaco. L'attuale protocollo fornisce una tecnica unica per quantificare in modo non distruttivo la densità cellulare regionale all'interno dei tessuti cellulari densi e misurarla longitudinalmente.
Introduzione
I ricercatori si sono in gran parte rivolti a sistemi di coltura 3D da banco in vitro per studiare alcune delle caratteristiche chiave della progressione del tumore. Gran parte di questa ricerca è stata condotta dal riemergere di sferoidi tumorali multicellulari (MCTS) e organoidi più complessi 1,2. Sebbene questi modelli siano avascolare, forniscono un potente strumento per ricapitolare i processi fisiologici e patologici che si verificano in vivo 3,4,5. In particolare, i modelli di medie dimensioni (diametro 300-500 μm) possono imitare caratteristiche tumorali chiave come la struttura 3D, i gradienti fisiopatologici e la segnalazione metastatica dovuta all'ipossia all'interno del nucleo. È ben documentato che questi modelli mostrano i caratteristici strati concentrici osservati nei tumori vascolarizzati in vivo, vale a dire, uno strato esterno di cellule proliferative, uno strato transitorio di cellule senescenti / quiescenti e cellule che soffrono di ipossia nel nucleo 3,6,7,8,9 . Da questi modelli è possibile ottenere informazioni uniche caratterizzando il comportamento cellulare all'interno di questi strati, durante lo sviluppo e in risposta al farmaco. Tuttavia, la dimensione MCTS richiesta, necessaria per sviluppare i gradienti che li rendono modelli in vitro così potenti, limita drasticamente gli strumenti utilizzati per la valutazione non distruttiva. In effetti, una delle maggiori sfide con l'analisi non distruttiva degli MTCS è quantificare i dettagli della scala cellulare. La microscopia a campo luminoso e a contrasto di fase viene utilizzata abitualmente per valutare la crescita e lo sviluppo degli MCTS 3D in modo non distruttivo. Tuttavia, queste modalità sono limitate alle proiezioni 2D, mancando la capacità di visualizzare la struttura 3D cruciale di questi modelli 10,11,12,13. Le informazioni sulla citotossicità e sulla proliferazione cellulare sono tipicamente raccolte attraverso l'imaging fluorescente (cioè microscopia a foglio luminoso, microscopia confocale) o colorazione immunoistologica ex vivo 14,15,16. Mentre questi approcci forniscono preziose informazioni ad alta risoluzione sulla struttura del tessuto, la densità cellulare e la funzione cellulare, spesso richiedono la preparazione del campione come la compensazione ottica, il fissaggio / colorazione o l'incorporamento che impedisce le analisi longitudinali.
La tomografia a coerenza ottica (OCT) è una modalità di imaging strutturale non distruttiva che ha il potenziale per superare alcune delle sfide sopra menzionate. Vanta una risoluzione cellulare e un campo visivo sufficientemente ampio (fino a 10 mm x 10 mm) in grado di visualizzare interi aggregati multicellulari 17,18,19. È importante sottolineare che, a causa della natura visibile della luce utilizzata, questa tecnica è completamente non distruttiva e priva di etichette17. Inoltre, i campioni possono essere ripresi in situ senza richiedere la preparazione del campione, in modo tale che i campioni possano essere prelevati direttamente dall'incubatore, rapidamente scansionati con OCT (durata della scansione ~ 5-10 min), quindi restituiti all'incubatore, consentendo la caratterizzazione longitudinale. Molti studi che cercano di utilizzare OCT per analizzare il comportamento sferoide tumorale sono recentemente emersi. In una delle dimostrazioni più emozionanti, Huang et al. hanno utilizzato OCT per rilevare in modo non distruttivo nuclei necrotici all'interno di grandi modelli di sferoidi tumorali, osservando che le regioni delle cellule vive e morte possiedono differenze distinguibili nell'attenuazione ottica, che possono essere utilizzate per il monitoraggio della vitalità senza etichetta20. Allo stesso modo, Hari et al. hanno condotto misurazioni dell'indice di rifrazione (RI) degli sferoidi del cancro del colon umano (HCT116) ripresi con OCT per studiare la presenza di ipossia all'interno dei campioni21. Le loro misurazioni non erano sufficienti per le inferenze dirette, anche se hanno osservato un RI inferiore in posizioni correlate con il sito, anche se non le dimensioni, dei nuclei necrotici, successivamente identificati tramite microscopia confocale. Abd El-Sadek et al. hanno utilizzato OCT per visualizzare e quantificare la vitalità tissutale regionale dei modelli tumorali del cancro al seno22. Hanno riportato due metodi basati su OCT per visualizzare le dinamiche dei tessuti e hanno mostrato una moderata correlazione tra le differenze in queste metriche e le regioni identificate al microscopio di cellule vive / morte.
Il nostro lavoro pubblicato utilizzando OCT si è basato su questa letteratura precedente per stabilire un approccio quantitativo e non distruttivo per misurare la morfologia 3D e il conteggio cellulare all'interno di modelli di cancro al seno MCTS durante lo sviluppo10,23. Utilizzando il software di analisi delle immagini di rendering 3D Imaris per contare il numero di oggetti delle dimensioni di una cellula (cioè spot) ripresi all'interno delle scansioni del volume OCT, i conteggi delle cellule sono stati misurati in modo non distruttivo in MCTS che erano statisticamente simili a quelli determinati tramite emocitometro al momento della dissociazione aggregata. Tuttavia, a causa della natura strutturale dell'OCT, le membrane cellulari ancora presenti dopo la morte cellulare per necrosi possono essere erroneamente contate come cellule vive. Inoltre, questa caratterizzazione è stata estesa a monitorare in modo non distruttivo la vitalità cellulare all'interno di singoli aggregati sottoposti a un regime farmacologico con promettente successo10. È importante sottolineare che è stato notato che una simile vitalità cellulare è stata riportata dal nostro approccio OCT-Imaris con ciò che è stato confrontato all'interno di questi campioni al momento della dissociazione. Questo approccio cellulare non distruttivo e privo di etichette consente di contare le celle all'interno di costrutti 3D e aggregati densi longitudinalmente senza sacrificare la struttura di costruzione / aggregato.
Il presente lavoro riporta un approccio migliorato per quantificare direttamente la densità cellulare regionale all'interno di aggregati densi sfruttando la capacità di OCT-Imaris di misurare sia la morfologia aggregata 3D che il numero di cellule. Questo progresso metodologico fornisce un quadro più dettagliato della distribuzione spaziale e della proliferazione delle cellule all'interno dei caratteristici strati concentrici dei modelli MCTS. Piuttosto che calcolare semplicemente una densità di cella aggregata media complessiva, tali misurazioni della densità locale possono rivelare gradienti di densità cellulare, come quelli associati alla compattazione. Questa valutazione regionale viene applicata anche agli aggregati trattati con un chemioterapico per valutare la risposta al farmaco regionale, misurata dai cambiamenti nella densità cellulare locale. Questa combinazione di OCT e metodi avanzati di analisi di imaging fornisce la quantificazione della vitalità cellulare regionale, che può essere utilizzata per esplorare la penetrazione dei farmaci in base a quali regioni sperimentano diminuzioni della densità cellulare. Questo è il primo rapporto a quantificare in modo non distruttivo la densità e la vitalità delle cellule regionali in risposta al farmaco all'interno di tessuti cellulari densi e misurarlo longitudinalmente. Tale caratterizzazione della densità cellulare tridimensionale e della distribuzione spaziale attraverso interi MCTS può aiutare a ottimizzare la somministrazione di farmaci nel trattamento del cancro e migliorare la comprensione della progressione del modello di cancro.
Protocollo
Per il presente studio sono state utilizzate linee cellulari di carcinoma mammario AU565 (HER2+) e MDA-MB-231 (vedere Tabella dei materiali).
1. Preparazione degli aggregati tumorali
- Preparare au565 (HER2+) mezzo di crescita cellulare del cancro al seno utilizzando il mezzo basale Roswell Park Memorial Institute (RPMI) 1640 (+) in L-glutammina integrato con il 10% (v / v) di siero bovino fetale e l'1% di penicillina / streptomicina (vedi Tabella dei materiali).
- Preparare il mezzo di crescita cellulare del cancro al seno triplo negativo MDA-MB-231 utilizzando il Modified Eagle's Medium (DMEM) di Dulbecco integrato con il 10% (v / v) di siero bovino fetale (FBS), 100 U / mL di penicillina / streptomicina e 2 mM di L-glutammina (vedere Tabella dei materiali).
- Preparare colture cellulari confluenti al 70%-90% di entrambe le linee cellulari (~ 3-4 giorni di preparazione) in condizioni standard (37 °C, 5% CO2, 95% umidità relativa). Staccare i monostrati cellulari dai loro palloni di coltura seguendo il metodo di tripsinizzazione standard.
NOTA: Prevenire la sovraesposizione delle cellule alla tripsina, influenzando la loro vitalità.- Per le celle AU565, aspirare i mezzi della cella dal pallone usando una pipetta (o, se disponibile, usando una pompa per vuoto collegata tramite tubo a una punta di pipetta di vetro autoclavata) e sostituire con 2 ml di tripsina-EDTA. Lasciare il pallone per 3 minuti a temperatura ambiente, quindi spostarsi nell'incubatrice per altri 4 minuti. Successivamente, aggiungere 6 ml di terreno di crescita al pallone per neutralizzare la tripsina.
- Per le celle MDA-MB-231, aspirare i mezzi cellulari dal pallone nello stesso modo eseguito nel passaggio 1.3.1 e sostituirli con 1,5 mL di tripsina-EDTA. Dopo 7 minuti di incubazione, aggiungere 8,5 ml di terreno di crescita al pallone per neutralizzare la tripsina.
- Aggiungere 10 μL della sospensione cellulare a un emocitometro per determinare il numero di cellule nella sospensione24. Risospese le cellule in media alla concentrazione desiderata di 2,5 × 105 cellule/ml.
- Erogare 50 μL di sospensione cellulare in ciascun pozzetto di una piastra a fondo tondo, non aderente, a 96 pozzetti. Per le sospensioni preparate senza Matrigel (matrice di membrana basale, vedere Tabella dei materiali), aggiungere 50 μL di terreni di crescita semplici a ciascun pozzetto.
- Per le sospensioni preparate con la matrice a membrana basale, rimuovere il flaconcino della matrice da -20 °C e metterlo in frigorifero per scongelarlo durante la notte. Preparare un contenitore con mezzi di crescita e conservare in frigorifero per 10 minuti per raffreddare.
- Utilizzando una punta di pipetta congelata, aggiungere la matrice al mezzo refrigerato in modo tale che la concentrazione finale di questa soluzione sia del 5%. Aggiungere 50 μL di questo mezzo a ciascun pozzo, in modo tale che la concentrazione finale di matrice in questi pozzetti sia del 2,5%3.
NOTA: Nel lavoro precedente, la morfologia aggregata è stata valutata per queste linee cellulari10,25 e ha scoperto che gli aggregati MDA-MB-231 formano sferoidi con l'aggiunta della matrice di membrana, mentre le colture MDA-MB-231 senza la matrice e la matrice AU565+ / - formano tutti aggregati a forma di disco. La sfericità quantificata di >0,8 su una scala da 0 (piano) a 1,0 (sfera perfetta) è stata utilizzata per identificare aggregati sufficientemente sferici e quindi ci si aspettava che avessero il rapporto superficie-volume richiesto per un comportamento in vivo-simile a 26,27.
- Centrifugare le piastre a 123 x g per 10 minuti a temperatura ambiente subito dopo la semina per garantire la raccolta di un pellet cellulare sul fondo di ogni pozzetto.
NOTA: questo passaggio deve avvenire il più rapidamente possibile dopo la semina. Gli autori hanno osservato che troppo tempo tra questi passaggi consente alle cellule di depositarsi sul fondo e sui lati di ciascun pozzo, inibendo la loro capacità di raccogliere e aggregarsi sul fondo del pozzo. - Incubare le piastre per 4 giorni, a quel punto gli aggregati cellulari sono considerati maturi.
NOTA: gli aggregati AU565 preparati con la matrice della membrana basale richiedono un ulteriore periodo di coltura di 5 giorni per lo studio di risposta al farmaco (9 giorni totali, variazione dei media il giorno 4).
2. Somministrazione di Trastuzumab (TZM) a aggregati cellulari AU565
- Preparare 500 μg/mL di soluzione TZM (vedere Tabella dei materiali) in mezzi di crescita AU565. Il giorno 4, aggiungere 10 μL di questa soluzione a ciascun pozzetto, in modo tale che la concentrazione finale all'interno di ciascun pozzetto sia di 50 μg/ml.
- La cultura si aggrega per altri 5 giorni dopo l'aggiunta di TZM e valuta in momenti chiave.
NOTA: Per il presente studio, sono stati utilizzati punti temporali di 0 ore (immediatamente prima della somministrazione di farmaci), 24 ore e 120 ore dopo il farmaco per analizzare il farmaco.
3. Tomografia a coerenza ottica
NOTA: I campioni qui riportati sono stati ripresi con la tomografia a coerenza ottica (OCT) durante ogni giorno di maturazione (1-4), e poi di nuovo il giorno 5 (24 ore dopo l'aggiunta di farmaci) e il giorno 9 (120 ore dopo l'aggiunta di farmaci) per aggregati drogati selezionati. Per lo studio corrente è stato utilizzato un sistema commerciale spectral-Domain Optical Coherence Tomography (SDOCT, vedi Table of Materials) per l'imaging OCT. Sebbene questo approccio sia accettabile per quasi tutti i sistemi OCT e la procedura seguita sarà generalmente simile tra sistemi diversi, alcuni dei passaggi dettagliati che seguono sono specifici per l'apparecchiatura attuale.
- Utilizzare un sistema OCT per l'imaging strutturale. Impostare la frequenza di scansione A su 5,5 kHz per la raccolta di immagini ad alta risoluzione. Impostare l'indice di rifrazione a 1,33 per i campioni in un mezzo liquido. Impostare la dimensione del voxel su 1,10 x 1,10 x 2,58 μm3.
- Nella finestra Parametri immagine sul lato destro dello schermo, impostare il campo visivo (FOV) immettendo i valori X, Y e Z (in mm) in modo che il campione sia compreso in questa regione di interesse.
NOTA: FOV per questi studi era in genere impostato su 1,5 x 1,5 x 0,5 mm3. Assicurarsi che il campione rientri in questa regione alternando l'ingresso "Angolo" tra 0 e 90 gradi ed eseguendo la conferma visiva. - Fare clic su Modalità di acquisizione 3D, quindi fare clic su Registra per raccogliere la scansione del volume 3D del campione.
4. Analisi delle immagini
- Esportare il file dello Strumento di personalizzazione di Office nel formato software (Imaris, vedere Tabella dei materiali).
- Aprire il file dello Strumento di personalizzazione di Office nel kit di sviluppo software. Fare clic su Esporta, impostare il tipo di file su .jpg ed esportare le immagini in una cartella vuota.
- Aprire il software di elaborazione delle immagini (FIJI, vedere Tabella dei materiali) e importare la sequenza di immagini dalla cartella in cui sono memorizzati i JPEG esportati. Utilizzare il software per unire la sequenza di immagini e quindi salvare questo file come immagine TIFF.
- Creare una ricostruzione del volume seguendo i passaggi seguenti.
- Apri Imaris e vai al file TIFF convertito all'interno dell'Arena. Passare a Modifica proprietà immagine > e immettere la dimensione voxel (in μm) dall'immagine dello Strumento di personalizzazione di Office nelle caselle XYZ corrispondenti. Quindi, fare clic su OK.
NOTA: all'interno dell'albero degli oggetti di esempio sul lato sinistro dello schermo, deselezionare la scheda Volume per evitare ritardi software. - Fate clic su Aggiungi nuove superfici (Add New Surfaces ) sopra l'albero degli oggetti. Nel menu sotto l'albero, fai clic su Salta creazione automatica e modifica manualmente. All'interno della finestra Regolazione schermo , fate scorrere manualmente le frecce rosse e nere per migliorare il contrasto tra il campione e lo sfondo e migliorare la visualizzazione del campione.
- Regola la "Posizione della fetta" sulla fetta su un bordo del campione, cioè dove appare per la prima volta il segnale del campione. Utilizzare il tasto Esc per passare il mouse dalla modalità di navigazione alla modalità di selezione, quindi fare clic su Disegna. Tracciare manualmente il contorno della regione che visualizza il segnale.
- Avanzate la posizione della sezione immettendo la posizione successiva nella casella di input. Questa posizione successiva deve essere ≤100 fette più avanti nel campione rispetto alla precedente. Tracciare manualmente la regione che visualizza il segnale.
- Ripetere il passaggio 4.2.4 attraverso lo spessore del campione fino a raggiungere il bordo opposto del campione. Quindi, fai clic su Crea superficie nel menu a sinistra per cucire insieme queste sezioni e completare la ricostruzione del volume.
- Fare clic su Modifica, quindi fare clic su Selezione maschera. In questo modo viene creato un nuovo canale nella finestra Regolazione schermo contenente solo il campione isolato. Le caratteristiche morfologiche del campione sono ora disponibili nella scheda Statistiche > Dettagliata .
NOTA: Questo approccio è stato seguito per determinare le sfericità e i volumi riportati all'interno di questo studio corrente.
- Apri Imaris e vai al file TIFF convertito all'interno dell'Arena. Passare a Modifica proprietà immagine > e immettere la dimensione voxel (in μm) dall'immagine dello Strumento di personalizzazione di Office nelle caselle XYZ corrispondenti. Quindi, fare clic su OK.
- Ottenere la densità cellulare totale di un campione seguendo i passaggi seguenti.
- Sopra l'albero degli oggetti, selezionate Aggiungi nuovi punti. Nel menu Impostazioni algoritmo deselezionare tutte le caselle. Fare clic sulla freccia blu per passare alla schermata Canale sorgente.
- Dal menu a discesa visualizzato, selezionare il canale mascherato creato nel passaggio 5.2.8, in genere denominato Canale 2. Immettere il diametro medio della cella per il campione nella casella diametro XY. Assicurarsi che l'opzione Sottrazione in background sia selezionata.
NOTA: per questo lavoro con celle AU565 e MDA-MB-231, il diametro è stato impostato su 10 μm. Questa selezione si basa sulle dimensioni della cella (spiegata nella sezione Discussione).
- Dal menu a discesa visualizzato, selezionare il canale mascherato creato nel passaggio 5.2.8, in genere denominato Canale 2. Immettere il diametro medio della cella per il campione nella casella diametro XY. Assicurarsi che l'opzione Sottrazione in background sia selezionata.
- Fare clic sulla freccia blu per passare alla schermata Classifica punti . Nel grafico nella parte inferiore del menu, fare clic e trascinare il bordo sinistro della soglia gialla sul bordo sinistro del grafico, in modo che tutti gli oggetti siano inclusi nella soglia ombreggiata gialla. Quindi, fai clic sulla freccia verde per completare la creazione dello spot.
- Ottieni il numero di oggetti identificati (ad esempio, il conteggio delle celle campione) facendo clic su Statistiche > Numero complessivo > totale di punti.
- Per determinare la densità media delle celle aggregate, dividere il conteggio delle celle dell'aggregato (misurato nel passaggio 4.3.3) per il volume aggregato (determinato nel passaggio 4.2.6).
- Sopra l'albero degli oggetti, selezionate Aggiungi nuovi punti. Nel menu Impostazioni algoritmo deselezionare tutte le caselle. Fare clic sulla freccia blu per passare alla schermata Canale sorgente.
- Valutare la densità regionale in strati concentrici di aggregati sferici (Figura 1A).
- Dopo l'analisi morfologica e della densità cellulare, valutare la densità regionale. Fate clic sulla superficie creata al passaggio 4.2.5 e passate alla scheda Stile/Qualità superfici (Surfaces Style/Quality ). Cambia la selezione in Punto centrale e modifica la larghezza dei pixel su ≤20 per una migliore visibilità. Passare a Statistiche > Posizione > dettagliata e registrare la posizione del punto centrale.
- Selezionate Aggiungi nuovo fotogramma di riferimento dal menu sopra l'albero degli oggetti. Seleziona le caselle Visibile e Correggi accanto a XY nel menu. Fate clic e trascinate il centro dell'icona del fotogramma di riferimento in modo che sia in linea con il punto centrale. Deselezionate le caselle XY Visibile e Correggi (Fix) e selezionatele per XZ.
- Ancora una volta, fate clic e trascinate il centro dell'icona del fotogramma di riferimento in modo che sia in linea con il punto centrale. Infine, ripetete questa operazione per il piano YZ, alternando questi tre piani fissi fino a quando il sistema di riferimento non si allinea perfettamente con il punto centrale. Fate doppio clic sul quadro di riferimento nell'albero degli oggetti e rinominatelo Center o Similar.
- Fare clic sugli spot creati nel passaggio 4.3 e passare a Statistiche > Detailed > Position Reference Frame. Fate clic su Posiziona X Reference Frame finché non viene ordinato dal valore più alto a quello più basso. Registrare il valore più alto - questo indica il bordo più lontano del campione, cioè il raggio del campione.
NOTA: poiché i campioni utilizzati qui sono circolari nel piano XY, i calcoli possono essere eseguiti lungo l'asse X o Y con risultati simili. Per i campioni che non presentano simmetria X-Y (asimmetrica, ellissoide, geometrie irregolari, ecc.), si consiglia di eseguire queste analisi lungo più assi. - Passare a Statistiche. Nell'angolo in basso a destra del menu, fai clic su Esporta tutte le statistiche in file e salva i dati in un foglio di calcolo.
- Eseguite calcoli manuali per identificare i livelli concentrici lungo l'asse X.
NOTA: i campioni utilizzati in questo studio avevano un diametro di circa 500 μm; quindi, ci sono due zone concentriche esterne di 200 μm di spessore e una zona centrale interna di 100 μm di spessore. Si raccomanda di mantenere zone di 200 μm di spessore perché i comportamenti di diffusione dovrebbero cambiare su questa distanza 7,28,29,30. In linea con questo ragionamento, si raccomanda anche di utilizzare più zone concentriche per campioni più grandi e viceversa per campioni più piccoli. - Impostare una regione dello strato esterno di 200 μm sottraendo 200 μm dal raggio esterno trovato nel passaggio 4.4.3. Pertanto, la regione esterna comprenderà punti con posizioni X traraggio X e Xesterno, bordo interno (Figura 1A, regione arancione).
- Impostate una regione di transizione sottraendo 200 μm da Xesterno,bordo interno. La regione di transizione comprenderà punti con posizioni X tra Xesterno, bordo interno e Xtransizionale, bordo interno (Figura 1A, regione rosa).
- Impostare una regione centrale centrale che comprenda i punti rimanenti tra il centro del campione e ilbordo interno di transizione X (Figura 1A, regione blu).
- Aprire il file di foglio di calcolo salvato creato nel passaggio 4.4.4. Passare alla scheda Distanza dal quadro di riferimento di origine.
- Calcola il numero di punti nella regione principale utilizzando la funzione 'CONTA.SE (Colonnax, "< Xdi transizione,bordo interno"), dove colonnax è la colonna 'Distanza dal quadro di riferimento di origine'. Il valore risultante è il numero di cella in questa regione. Dividere questo valore per il volume della regione per ottenere la densità cellulare.
- In modo simile, calcolare il numero di celle nella regione di transizione utilizzando 'COUNTIF (Colonnax, "< Xesterno,bordo interno") - COUNTIF(Colonnax, "> Xtransitorio,bordo interno").
- Calcola il numero di celle nella regione esterna usando 'COUNTIF (Colonnax,"> Xtransitional,inner-edge").
- Valutare la densità cellulare regionale in campioni non sferici tramite "Spine regionali" (Figura 1B).
NOTA: per i campioni non sferici che non hanno strati concentrici, viene sviluppato il metodo "Regional Plug" per campionare la densità cellulare locale a varie profondità radiali in tutto l'aggregato.- Ripetere i passaggi 4.4.1-4.4.3 per impostare il sistema di riferimento centrale (Figura 1B, cerchio giallo centrale) e ottenere il raggio totale del campione.
- Eseguire calcoli manuali per identificare il posizionamento dei sistemi di riferimento all'interno delle regioni di transizione ed esterne in modo tale che la densità delle celle possa essere valutata nel campione in queste posizioni.
- Per calcolare la posizione esterna, sottrarre 50 μm dal valore del raggio del campione dal passaggio 4.5.1 e utilizzare questo valore come posizione X.
NOTA: utilizzare gli stessi valori di posizione Y e Z registrati nel passaggio 4.5.1 per tutti i tappi all'interno di un determinato campione per misurare la densità della cella lungo l'asse X del campione (Figura 1B, cerchio giallo più a sinistra). - Sopra l'albero degli oggetti, fai clic su Aggiungi nuovi punti e fai clic su Salta creazione automatica, Modifica manualmente. Con il puntatore in modalità "seleziona", tieni premuto maiusc + clic per posizionare un punto sullo schermo. Nel menu a sinistra, inserisci i valori di posizione XYZ calcolati sopra. Regola i diametri XY e Z su ≤20 per una migliore visibilità.
- Ripetete il passaggio 4.4.2 per aggiungere un fotogramma di riferimento in questa posizione spot. Rinominare questo fotogramma di riferimento nell'albero degli oggetti esterno o simile.
- Per calcolare la posizione di transizione (punto medio), individuate la media del valore della posizionecentrale X e i valori della posizioneesterna X. Utilizzate questo valore come posizione X per il sistema di riferimento di transizione. Come sopra, utilizzare gli stessi valori Y e Z registrati nel passaggio 4.5.1 per tutti i tappi all'interno di un determinato campione per misurare la densità della cella lungo l'asse X del campione (Figura 1B, cerchio giallo medio).
- Ripetere il passaggio 4.4.2 per posizionare il fotogramma di riferimento centrale in questa posizione, rinominandolo "transitorio" o simile una volta creato.
- Per calcolare la posizione esterna, sottrarre 50 μm dal valore del raggio del campione dal passaggio 4.5.1 e utilizzare questo valore come posizione X.
- Fai clic sugli spot creati nel passaggio 4.3 e vai a Statistiche. Nell'angolo in basso a destra del menu, fai clic su Esporta tutte le statistiche in file e salva i dati in un foglio di calcolo.
- Apri il foglio di calcolo. Passare alla scheda Distanza dal quadro di riferimento di origine. La distanza di ciascun oggetto dai fotogrammi di riferimento è mostrata nella colonna A e il raggruppamento dei fotogrammi di riferimento per ciascun oggetto è mostrato nella colonna G. Utilizzare la funzione 'MOD' per assegnare numericamente ogni distanza al corrispondente sistema di riferimento, dove 0 è il fotogramma centrale, 1 è la transizione e 2 è l'esterno.
- Filtrate i valori in questa colonna per lavorare con le distanze in ogni fotogramma di riferimento. Per ciascuno dei quadri di riferimento, calcolare il numero di oggetti entro 50 micrometri dal fotogramma utilizzando la funzione 'CONTA.SE (Colonnax, "≤50"), dove colonnax è la colonna 'Distanza dal quadro di riferimento di origine' per ciascun gruppo. Il valore risultante corrisponde al numero di celle in quel tappo regionale. Dividere questo valore per il volume del tappo da 100 μm per ottenere la densità della cella.
- Eseguire il metodo Spatially-Refined Regional Plug (Figura 1C).
NOTA: Questo approccio è stato utilizzato per valutare la vitalità cellulare regionale in risposta all'applicazione del farmaco. L'inclusione di più fotogrammi di riferimento migliora la risoluzione delle densità delle celle che possono essere calcolate in tutto lo spessore del modello.- Ripetere i passaggi 4.4.1-4.4.3 per impostare il sistema di riferimento centrale (Figura 1C, cerchio giallo centrale) e ottenere il raggio del campione.
- Eseguire calcoli manuali per determinare ulteriori posizioni di interesse. A tale scopo, aggiungere 100 μmal valore centrale X, quindi utilizzare i valori Y e Z del punto centrale per definire la prima posizione lungo l'asse centrale. Aggiungete un sistema di riferimento a questa posizione come nel passaggio 4.4.2 (Figura 1C, cerchio giallo adiacente al cerchio centrale).
NOTA: aggiungere meno μm in questo passaggio se si cerca un tracciamento della densità a risoluzione più elevata. - Ripetere i passaggi 4.6.2, aggiungendo 100 μm (o il numero desiderato di μm) a ciascuna posizione X sequenziale per stabilire un asse di tappi equamente distanziati attraverso lo spessore del campione. Posizionare l'ultimo sistema di riferimento ≤50 μm di distanza dal raggio esterno del campione (Figura 1C, cerchi gialli).
- Ripetere i passaggi 4.5.3-4.5.5 per ottenere conteggi cellulari all'interno di ogni tappo di diametro 100 μm. Dividere questi valori per il volume di ciascuna spina corrispondente per ottenere la densità della cella locale.
- Ripetere tutti i passaggi in questa sezione per i dati dello Strumento di personalizzazione di Office raccolti in ogni punto temporale della sperimentazione del farmaco. Il confronto della conta cellulare in ogni posizione attraverso il corso del tempo di trattamento dovrebbe rivelare dove la densità cellulare sta diminuendo (cioè le regioni che sperimentano la morte cellulare) e, di conseguenza, quanto in profondità il farmaco sta penetrando (cioè, quali strati vedono un calo della densità cellulare rispetto a quelli che mantengono o mostrano un aumento dei livelli di densità cellulare).
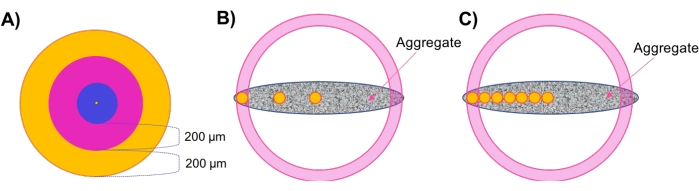
Figura 1: Illustrazione schematica. (A) L'approccio a strati concentrici (gusci) per la valutazione della densità cellulare regionale in aggregati sferici. (B) Il metodo del tappo regionale è stato sviluppato per valutare la densità cellulare locale in aggregati non sferici, dove piccoli tappi sferici (diametro 100 μm) (mostrati in giallo) sono utilizzati come indicatori di densità in ogni zona / spessore. (C) Il metodo della spina regionale spazialmente raffinato è impiegato per gli studi di penetrazione dei farmaci. Fare clic qui per visualizzare una versione più grande di questa figura.
Risultati
In una precedente pubblicazione, è stato stabilito un metodo per la misurazione non distruttiva della densità cellulare globale all'interno di aggregati cellulari utilizzando OCT10. Qui, questa tecnica viene estesa per valutare la densità cellulare regionale degli aggregati cellulari in via di sviluppo. La Figura 1 mostra uno schema di questa estensione, in cui la densità cellulare può essere valutata in strati concentrici di uno sferoide o più localmente osservando invece piccoli tappi sferici (diametro 100 μm), indicati dai cerchi gialli in Figura 1B,C. Gli sferoidi tumorali MDA-MB-231 sono stati valutati inizialmente impostando un punto centrale all'interno dell'aggregato e contando il numero di oggetti / cellule in strati concentrici sequenziali dello sferoide. Questi risultati sono presentati nella Figura 2. Il t-test dello studente ha rivelato una densità cellulare significativamente più elevata nel nucleo sferoide rispetto agli strati di transizione (p = 4,3e-4) ed esterni (p = 4,0e-6). Questo risultato indica la compattazione nel nucleo sferoide dopo 4 giorni.

Figura 2: Differenze nella densità regionale calcolate con l'approccio a strati concentrici per aggregati sferici MDA-MB-231. La figura illustra un gradiente di densità cellulare radiale con celle (n = 3) più densamente impacchettate nel nucleo aggregato e la densità cellulare locale che diminuisce con la distanza dal nucleo. Il conteggio totale medio delle celle viene visualizzato con una linea blu. La barra di scala per l'immagine inserita è di 100 μm. Dati indicati come media ± SD (*= p < 0,05, ***= p < 0,001). Fare clic qui per visualizzare una versione più grande di questa figura.
Tuttavia, una limitazione di questa tecnica di strato concentrico è che può essere utilizzata solo su aggregati sferici. Pertanto, l'approccio di conteggio delle cellule è stato adattato per stabilire un metodo Regional Plug, che campiona piccole zone in posizioni sequenziali attraverso l'aggregato, simile a una biopsia virtuale. Questi tappi forniscono misurazioni della densità della cella locale a profondità specifiche. Questa tecnica è stata convalidata rispetto all'approccio a strati concentrici eseguendo analisi sugli stessi sferoidi MDA-MB-231 (Figura 3). I risultati hanno mostrato un buon accordo tra gli approcci regionali a spina e a strato concentrico per questi aggregati sferici. I t-test dello studente non hanno rivelato differenze statistiche tra gli approcci per misurare gli strati esterni e di transizione (p = 0,243 e 0,484, rispettivamente) e una leggera ma significativa differenza nel calcolo delle densità al nucleo aggregato (p = 0,017).
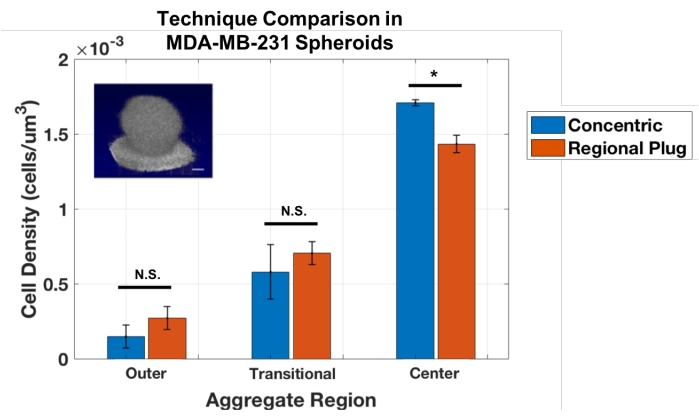
Figura 3: Gli approcci a zona concentrica e a spina regionale forniscono risultati simili per quantificare la densità cellulare negli aggregati sferici MDA-MB-231 (n = 3). Dati mostrati come SD ± media (* = p < 0,05). Fare clic qui per visualizzare una versione più grande di questa figura.
Questa tecnica è stata poi applicata per valutare aggregati tumorali sia sferici che non sferici il giorno 4 della maturità (Figura 4), osservando una tendenza simile verso la compattazione del nucleo per tutte le morfologie testate, indipendentemente dal tipo di cellula. Questa tendenza è stata più evidente nei campioni preparati con Matrigel (la matrice della membrana basale), un additivo noto per promuovere l'aggregazione ma i cui fattori esogeni e la cui composizione sono scarsamente caratterizzati 31,32,33. È interessante notare che l'aggiunta della matrice non sembra influenzare il volume o il conteggio delle cellule e quindi sembra avere un'influenza trascurabile sulla proliferazione cellulare. Piuttosto, l'aggiunta della matrice sembra ridistribuire la densità cellulare, promuovendo una significativa compattazione del nucleo e diminuendo la densità cellulare negli strati esterni. Questi risultati indicano anche che le cellule AU565 appaiono meno sensibili agli effetti di aggregazione mediati dalla matrice rispetto alle cellule MDA-MB-231. Questi risultati forniscono preziose informazioni sui meccanismi fisici attraverso i quali la matrice consente l'aggregazione cellulare in diverse linee cellulari di cancro al seno. È importante sottolineare che l'approccio a spina regionale dovrebbe adattarsi a qualsiasi MCTS che mostri geometrie aggregate sferiche o non sferiche.

Figura 4: L'aggiunta di matrice promuove una maggiore aggregazione e ridistribuisce la densità cellulare nelle linee cellulari tumorali. Per entrambe le linee cellulari MDA-MB-231 (A,B) e AU565 (C,D), l'aggiunta di matrice diminuisce la densità nella zona esterna e aumenta la compattazione al centro (n = 3) senza modificare sensibilmente il numero complessivo di cellule. Il conteggio totale medio delle celle viene mostrato con linee blu. Barre della scala inserite = 100 μm. Dati indicati come SD medi ± (*= p < 0,05, **= p < 0,01, ***= p < 0,001). Fare clic qui per visualizzare una versione più grande di questa figura.
Successivamente, questo approccio regionale a spina è stato utilizzato per tracciare la morte cellulare negli aggregati tumorali trattati con TZM in modo non distruttivo. Gli MCTS AU565 preparati con la matrice di membrana sono stati trattati con TZM il giorno 4 dello sviluppo e coltivati fino al giorno 9, con punti temporali chiave di imaging OCT a 0 h (pre-farmaco), 24 h e 120 h (5 giorni). Il metodo della spina regionale definito spazialmente è stato applicato in ogni punto temporale, con tappi impostati ogni 100 μm per l'intero spessore di ciascun aggregato, come mostrato dai cerchi gialli nella Figura 1C. La dimensione della spina centrale è stata mantenuta costante mentre la spina esterna è stata lasciata fluttuare di diametro, corrispondente alla modifica della dimensione aggregata. Piccole fluttuazioni della densità cellulare sono state osservate nel tempo entro i 500 μm interni di ciascun aggregato, indicando una morte cellulare minima (Figura 5). In effetti, la maggior parte delle morti cellulari si è verificata nei 200 μm esterni di ciascun aggregato, in particolare nei 100 μm più esterni, che sono scomparsi completamente entro il punto temporale di 120 ore per tutti gli aggregati analizzati. La visualizzazione della morte cellulare come risposta indicativa al farmaco principalmente negli strati esterni degli MCTS è coerente con i problemi di penetrazione del farmaco di TZM, un farmaco anticorpale clinicamente rilevante con un peso molecolare di 145 kDa34. In effetti, il farmaco si basa sulla diffusione passiva attraverso questi modelli cellulari densi, che dovrebbe sfidare la sua capacità di penetrare più in profondità di 200 μm nei modelli.
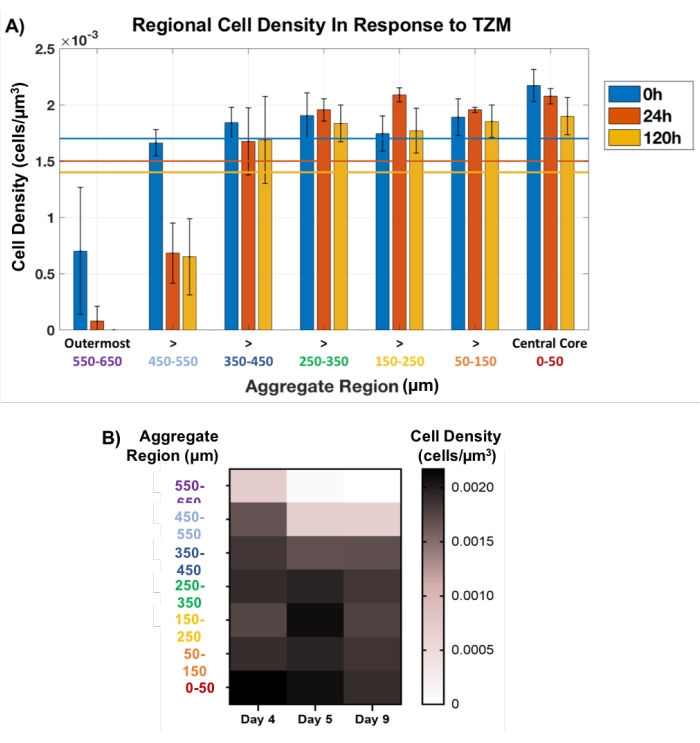
Figura 5: Vitalità cellulare in risposta al farmaco, misurata in tutto lo spessore aggregato. La spina al nucleo interno è stata mantenuta costante a 100 μm di diametro, mentre quella nella zona esterna è stata lasciata fluttuare con il variare delle dimensioni dell'aggregato. (A) La densità cellulare regionale in risposta all'aggiunta di TZM ha rivelato che la morte cellulare era in gran parte limitata ai 200 μm esterni, in particolare ai 100 μm esterni, che sono scomparsi completamente dopo 120 ore di trattamento. Lo spessore interno di 500 μm degli aggregati ha osservato un piccolo cambiamento nel conteggio delle cellule (n = 3). I conteggi totali medi delle celle sono mostrati con linee colorate corrispondenti per ogni punto temporale. I dati sono mostrati come media ± SD. (B) Grafico della mappa termica che rappresenta il cambiamento nella densità media delle cellule in risposta al farmaco in funzione dello spessore aggregato. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Significato
Gli sferoidi tumorali multicellulari (MCTS) sono potenti modelli 3D in vitro per lo studio della progressione tumorale e lo screening farmacologico 1,2,3. L'avanzamento dell'utilità di questi modelli aggregati relativamente semplici si basa fortemente sulla caratterizzazione delle loro caratteristiche chiave, come la morfologia e la densità cellulare, che sono note per influenzare sia la progressione del modello tumorale che la risposta terapeutica. Tuttavia, le loro dimensioni richieste introducono sfide nella valutazione di queste caratteristiche, in particolare per le analisi non distruttive. Il metodo presentato fornisce un nuovo strumento per la quantificazione longitudinale e senza etichette della densità e della vitalità delle cellule all'interno di regioni discrete di modelli aggregati 3D densi. Lo stesso aggregato può essere ripreso con la tomografia a coerenza ottica (OCT) per più giorni di sviluppo. Queste scansioni volumetriche possono essere analizzate per caratterizzare il modo in cui la densità cellulare si evolve a livello regionale durante la maturazione degli MCTS. Questi stessi principi si applicano all'analisi di aggregati drogati, che possono essere ripresi longitudinalmente in un determinato regime farmacologico per determinare dove la densità cellulare sta diminuendo, cioè dove il farmaco può uccidere attivamente le cellule. Questo approccio migliora significativamente rispetto ai metodi precedenti per ottenere informazioni simili su scala cellulare, che tradizionalmente richiedono fissazione, colorazione e / o sezionamento, precludendo così le analisi longitudinali. In effetti, questo strumento ha il potenziale per ridurre drasticamente il numero di campioni necessari per un determinato studio in quanto gli stessi campioni possono essere analizzati consecutivamente. Ci si aspetta anche che ciò introduca un valore aggiunto in ogni momento in quanto gli sviluppi possono essere monitorati all'interno di un singolo aggregato in quanto risponde a un determinato stimolo, piuttosto che fare affidamento su dati correlati da campioni terminali abbinati all'età. Oltre a questa applicazione MCTS qui dimostrata, questo metodo OCT-Imaris può studiare altri aggregati cellulari, corpi embrioidi, organoidi più complessi o campioni di tessuto fino a pochi millimetri di spessore. Questo protocollo migliorerà la comprensione della compattazione cellulare durante lo sviluppo aggregato e la risposta ai farmaci all'interno di modelli aggregati densi.
Modifiche
Il protocollo presentato è stato ottimizzato per l'analisi dei modelli MDA-MB-231 e AU565 MCTSS. Sono applicabili i modelli realizzati utilizzando altre linee cellulari; tuttavia, potrebbe essere necessaria una certa ottimizzazione del protocollo a causa di cambiamenti nella dimensione media delle cellule e nella morfologia aggregata. È stato condotto uno studio separato in cui le cellule MDA-MB-231 e AU565 sono state placcate in 2D e hanno ottenuto una dimensione media delle cellule tramite microscopia. Questo diametro è stato utilizzato come diametro XY all'interno della funzione "spot" di Imaris in modo tale che gli oggetti di questa dimensione approssimativa siano stati contati. La modifica di questo valore modifica il numero di oggetti che si trovano all'interno del campione10 e sono attesi risultati più accurati quando questo input corrisponde strettamente alle dimensioni della cella. Pertanto, un diametro XY adatto deve essere informato dalla dimensione media della cella per il tipo di cella utilizzato.
Passaggi critici e risoluzione dei problemi
Uno dei passaggi critici durante l'imaging dello Strumento di personalizzazione di Office è la selezione della risoluzione di scansione. La dimensione dei pixel impostata dall'utente deve essere sufficientemente piccola in modo che siano necessari più pixel per comprendere la dimensione media del tipo di cella utilizzato. Ciò migliora l'accuratezza dell'analisi degli "spot" all'interno di Imaris migliorando la risoluzione OCT delle cellule e riducendo la possibilità che il rumore stocastico dei pixel influisca negativamente sul conteggio delle cellule.
L'isolamento del campione all'interno del sistema di riferimento Imaris influenza notevolmente i valori di uscita per la densità della cella. Quando l'utente esegue questo passaggio, è accompagnato da un livello di soggettività e bias che deve essere applicato in modo coerente in tutte le analisi del campione. Quando si isola il campione all'interno della scansione del volume per iniziare l'analisi Imaris, è necessario prestare attenzione a tracciare accuratamente i contorni aggregati ed evitare l'inclusione di artefatti (ad esempio, riflessi da substrati o altezza del supporto).
Il corretto posizionamento dei sistemi di riferimento durante l'analisi delle spine regionali è un altro passo critico. Come affermato nell'introduzione, le tre zone critiche da analizzare durante lo sviluppo del modello sono la regione esterna proliferativa, la regione di transizione costituita da cellule senescenti / quiescenti e il nucleo ipossico. Il posizionamento dei sistemi di riferimento all'interno del centro di ciascun livello dovrebbe produrre la stima più accurata della densità delle celle all'interno di quella regione. Questo posizionamento del quadro di riferimento è ancora più importante per le analisi dettagliate dei tappi regionali utilizzate nel presente documento per lo studio del farmaco, poiché è necessario stabilire zone uniformemente distanziate per analizzare la risposta ai farmaci in modo più accurato.
Limiti e ricerca futura
La limitazione principale del metodo proposto è l'analisi slice-by-slice basata sull'utente eseguita all'interno di Imaris per isolare l'aggregazione all'interno dell'analisi del volume dello Strumento di personalizzazione di Office. Questo è un passo fondamentale per risultati accurati, che è in qualche modo soggettivo e, quindi, dovrebbe impartire un certo livello di variabilità tra gli utenti. Per risolvere questo problema, stiamo attualmente cercando di incorporare un algoritmo di rilevamento dei bordi eseguito all'interno di Matlab (o simile) prima di caricare la scansione in Imaris. Questo algoritmo dovrebbe identificare oggettivamente i bordi del campione in fette di scansione B progressive, dopo di che le analisi proposte possono essere eseguite su questa regione campione pre-isolata. Affrontare questa limitazione eliminerà la variabilità basata sull'utente e dovrebbe portare a una più ampia applicabilità di questo strumento basato sullo Strumento di personalizzazione di Office.
La capacità dell'OCT di visualizzare con precisione le cellule vive all'interno degli aggregati durante il trattamento farmacologico dipende dalla modalità di morte cellulare su cui opera il farmaco. Il lavoro precedente ha rivelato imprecisioni quantitative nel conteggio delle cellule vive riportate da OCT / Imaris in risposta alla doxorubicina, un noto farmaco anti-cancro che uccide le cellule attraverso la necrosi e l'apoptosi10. Si ipotizza che ciò sia dovuto alle membrane cellulari rimanenti durante la necrosi, che dovrebbero apparire come cellule vive durante l'imaging strutturale. TZM è noto per uccidere le cellule principalmente attraverso l'apoptosi35; pertanto, quando le cellule muoiono, devono scomporsi in pezzi sufficientemente piccoli da non essere tracciati/contati dall'OCT. Sebbene siano necessari ulteriori test di convalida con un farmaco apoptotico, i nostri primi esperimenti pilota mostrano l'eccellente accordo di OCT / Imaris sulla conta cellulare dissociata negli MCTS drogati con TZM (dati non pubblicati). Quindi, si prevede che i risultati delle cellule vive qui presentati siano più accurati dei farmaci che inducono la necrosi. Questa distinzione deve essere tenuta presente quando si applica questo approccio per i test di fattibilità in aggregati drogati.
La ricerca futura nella valutazione non distruttiva degli aggregati tumorali dovrebbe migliorare la comprensione di come si sviluppano e rispondono sia agli stimoli esterni che al trattamento farmacologico. Lo sviluppo di strumenti analitici, come quello qui presentato, dovrebbe espandere l'utilità del modello e migliorare l'accuratezza dei risultati, in particolare all'interno di applicazioni di impatto come lo screening dei farmaci e la valutazione della consegna / efficacia.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo studio è stato supportato da NIH R01 BRG CA207725 (MB/DTC) e NIH R01 CA233188 (MB). Vorremmo ringraziare AMC Pharmacy per il Trastuzumab fornito per questi esperimenti.
Materiali
| Name | Company | Catalog Number | Comments |
| 96 well plates | Greiner Bio-One | 650970 | CellStar Cell-Repellent Surface, https://shop.gbo.com/en/usa/products/bioscience/cell-culture-products/cellstar-cell-repellent-surface/ |
| 0.25% trypsin, 2.21 mM EDTA | Corning | 25-053-CI | |
| AU565 breast cancer cells | ATCC | ||
| Dulbecco's Modified Eagle's Medium | Corning | 10-013-CV | |
| Fetal Bovine Serum | ATCC | 30-2020 | |
| FIJI software | open-source | (Fiji Is Just) ImageJ v2.1/1.5.3j | Downloaded from https://imagej.net/software/fiji/ |
| Hemocytometer | Fisher Scientific | 0267151B | |
| Imaris image analysis software | Bitplane | Current version 9.8 | |
| L-glutamine | Lonza | 17-605E | |
| Matrigel | Corning | 354263 | |
| MDA-MB-231 breast cancer cells | ATCC | ||
| Microscope | Zeiss | Z1 AxioVision | |
| Penicilin streptomycin | Corning | 30-0002CI | |
| Plate centrifuge | Eppendorf | ||
| RPMI medium 1640 | Gibco | 11875-085 | |
| Spectral Domain Optical Coherence Tomography | ThorLabs | TEL220C1 | |
| T75 cell culture flasks | Greiner Bio-One | 658175 | |
| Trastuzumab | Remnant clinical samples of Trastuzumab were used in this study, generously gifted by the Albany Medical College Pharmacy. |
Riferimenti
- Sutherland, R., JA, M., Inch, W. Growth of multicell spheroids in tissue culture as a model of nodular carcinomas. Journal of the National Cancer Institute. 46 (1), 113-120 (1971).
- Sachs, N., et al. A living biobank of breast cancer organoids captures disease heterogeneity. Cell. 172 (1-2), 373-386 (2018).
- Nagelkerke, A., Bussink, J., Sweep, F. C. G. J., Span, P. N. Generation of multicellular tumor spheroids of breast cancer cells: How to go three-dimensional. Analytical Biochemistry. 437 (1), 17-19 (2013).
- Kunz-Schughart, L. A., Freyer, J. P., Hofstaedter, F., Ebner, R. The use of 3-D cultures for high-throughput screening: The multicellular spheroid model. Journal of Biomolecular Screening. 9 (4), 273-285 (2004).
- Hirschhaeuser, F., et al. Multicellular tumor spheroids: An underestimated tool is catching up again. Journal of Biotechnology. 148 (1), 3-15 (2010).
- Jiang, Y., Pjesivac-Grbovic, J., Cantrell, C., Freyer, J. P. A multiscale model for avascular tumor growth. Biophysical Journal. 89 (6), 3884-3894 (2005).
- Freyei, J. P., Sutherland, R. M. Regulation of growth saturation and development of necrosisin EMT6/R0 multicellular spheroids by the glucose and oxygen supply. Cancer Research. 46 (7), 3504-3512 (1986).
- Desoize, B., Jardillier, J. C. Multicellular resistance: a paradigm for clinical resistance. Critical Reviews in Oncology Hematology. 36 (2-3), 193-207 (2000).
- Mellor, H. R., Ferguson, D. J. P., Callaghan, R. A model of quiescent tumour microregions for evaluating multicellular resistance to chemotherapeutic drugs. British Journal of Cancer. 93 (3), 302-309 (2005).
- Roberge, C. L., et al. Non-destructive tumor aggregate morphology and viability quantification at cellular resolution, during development and in response to drug. Acta Biomaterialia. 117, 322-334 (2020).
- Piccinini, F., Tesei, A., Bevilacqua, A. Single-image based methods used for non-invasive volume estimation of cancer spheroids a practical assessing approach based on entry-level equipment. Computer Methods and Programs in Biomedicine. 135, 51-60 (2016).
- Imamura, Y., et al. Comparison of 2D- and 3D-culture models as drug-testing platforms in breast cancer. Oncology Reports. 33 (4), 1837-1843 (2015).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental & Clinical Cancer Research. 37 (1), 1-13 (2018).
- LaBarbera, D. V., Reid, B. G., Yoo, B. H. The multicellular tumor spheroid model for high-throughput cancer drug discovery. Expert Opinion on Drug Discovery. 7 (9), 819-830 (2012).
- Hakanson, M., Textor, M., Charnley, M. Engineered 3D environments to elucidate the effect of environmental parameters on drug response in cancer. Integrative Biology. 3 (1), 31-38 (2011).
- Pickl, M., Ries, C. H. Comparison of 3D and 2D tumor models reveals enhanced HER2 activation in 3D associated with an increased response to trastuzumab. Oncogene. 28 (3), 461-468 (2009).
- Huang, D., et al. Optical coherence tomography HHS public access. Science. 254 (5035), 1178-1181 (1991).
- Zhong, H. Q., et al. Enhancement of permeability of glycerol with ultrasound in human normal and cancer breast tissues in vitro using optical coherence tomography. Laser Physics Letters. 7 (5), 388-395 (2010).
- Fujimoto, J., Swanson, E. The development, commercialization, and impact of optical coherence tomography. Investigative Ophthalmology & Visual Science. 57 (9), (2016).
- Huang, Y., et al. Optical coherence tomography detects necrotic regions and volumetrically quantifies multicellular tumor spheroids. Cancer Research. 77 (21), 6011-6020 (2017).
- Hari, N., Patel, P., Ross, J., Hicks, K., Vanholsbeeck, F. Optical coherence tomography complements confocal microscopy for investigation of multicellular tumour spheroids. Scientific Reports. 9 (1), 1-11 (2019).
- El-Sadek, I. A., et al. Three-dimensional dynamics optical coherence tomography for tumor spheroid evaluation. Biomedical Optics Express. 12 (11), 6844(2021).
- Kingsley, D. M., et al. Laser-based 3D bioprinting for spatial and size control of tumor spheroids and embryoid bodies. Acta Biomateralia. 95, 357-370 (2019).
- Absher, M. Hemocytometer counting. Tissue Culture. , 395-397 (1973).
- Roberge, C. L., Rudkouskaya, A., Barroso, M., Corr, D. T. Longitudinal, label-free assessment of cell density and viability in multicellular tumor spheroids via optical coherence tomography. Summer Biomechanics, Bioengineering, and Biotransport Conference. , (2020).
- Bellotti, C., Duchi, S., Bevilacqua, A., Lucarelli, E., Piccinini, F. Long term morphological characterization of mesenchymal stromal cells 3D spheroids built with a rapid method based on entry-level equipment. Cytotechnology. 68 (6), 2479-2490 (2016).
- Noto, A., et al. Stearoyl-CoA desaturase-1 is a key factor for lung cancer-initiating cells. Cell Death & Disease. 4 (12), 947(2013).
- Riffle, S., Hegde, R. S. Modeling tumor cell adaptations to hypoxia in multicellular tumor spheroids. Journal of Experimental & Clinical Cancer Research. 36, 102(2017).
- Wilson, W. R., Hay, M. P. Targeting hypoxia in cancer therapy. Nature Reviews Cancer. 11, 393-410 (2011).
- Grimes, D. R., Kelly, C., Bloch, K., Partridge, M. A method for estimating the oxygen consumption rate in multicellular tumour spheroids. Journal of the Royal Society Interface. 11 (92), 20131124(2014).
- Nath, S., Devi, G. R. Three-dimensional culture systems in cancer research: Focus on tumor spheroid model. Pharmacology & Therapeutics. 163, 94-108 (2016).
- Pozzi, S., et al. Meet me halfway: Are in vitro 3D cancer models on the way to replace in vivo models for nanomedicine development. Advanced Drug Delivery Reviews. 175, 113760(2021).
- Nguyen, E. H., Daly, W. T., Belair, D. G., Le, N. N., Murphy, W. L. High throughput screening format identifies synthetic mimics of matrigel for tubulogenesis screening. , Available from: https://abstracts.biomaterials.org/data/papers/2015/abstracts/547.pdf (2015).
- Duchnowska, R., Szczylik, C. Central nervous system metastases in breast cancer patients administered trastuzumab. Cancer Treatment Reviews. 31 (4), 312-318 (2005).
- Zazo, S., et al. Generation, characterization, and maintenance of trastuzumab-resistant HER2+ breast cancer cell lines. American Journal of Cancer Research. 6 (11), 2661-2678 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon