Method Article
Microscopia elettronica seriale a scansione a blocchi (SBF-SEM) di campioni di tessuto biologico
In questo articolo
Riepilogo
Questo protocollo delinea un metodo di routine per l'utilizzo della microscopia elettronica seriale a scansione block-face (SBF-SEM), una potente tecnica di imaging 3D. L'applicazione di successo di SBF-SEM dipende da corrette tecniche di fissazione e colorazione dei tessuti, nonché da un'attenta considerazione delle impostazioni di imaging. Questo protocollo contiene considerazioni pratiche per l'intero processo.
Abstract
La microscopia elettronica seriale a scansione a blocchi (SBF-SEM) consente la raccolta da centinaia a migliaia di immagini ultrastrutturali registrate in serie, offrendo una visione tridimensionale senza precedenti della microanatomia tissutale. Mentre SBF-SEM ha visto un aumento esponenziale dell'uso negli ultimi anni, aspetti tecnici come una corretta preparazione dei tessuti e parametri di imaging sono fondamentali per il successo di questa modalità di imaging. Questo sistema di imaging beneficia della natura automatizzata del dispositivo, consentendo di lasciare il microscopio incustodito durante il processo di imaging, con la raccolta automatizzata di centinaia di immagini possibili in un solo giorno. Tuttavia, senza un'adeguata preparazione dei tessuti, l'ultrastruttura cellulare può essere modificata in modo tale da poter trarre conclusioni errate o fuorvianti. Inoltre, le immagini vengono generate scansionando la faccia a blocchi di un campione biologico incorporato nella resina e questo spesso presenta sfide e considerazioni che devono essere affrontate. L'accumulo di elettroni all'interno del blocco durante l'imaging, noto come "carica tissutale", può portare a una perdita di contrasto e all'incapacità di apprezzare la struttura cellulare. Inoltre, mentre l'aumento dell'intensità/tensione del fascio di elettroni o la diminuzione della velocità di scansione del fascio possono aumentare la risoluzione dell'immagine, questo può anche avere lo sfortunato effetto collaterale di danneggiare il blocco di resina e distorcere le immagini successive nella serie di imaging. Qui presentiamo un protocollo di routine per la preparazione di campioni di tessuto biologico che preserva l'ultrastruttura cellulare e diminuisce la carica tissutale. Forniamo anche considerazioni di imaging per la rapida acquisizione di immagini seriali di alta qualità con danni minimi al blocco tissutale.
Introduzione
La microscopia elettronica a scansione facciale a blocchi seriali (SBF-SEM) è stata descritta per la prima volta da Leighton nel 1981, dove ha modellato un microscopio elettronico a scansione potenziato con un microtomo incorporato in grado di tagliare e tagliare sottili sezioni di tessuto incorporate nella resina. Sfortunatamente, le limitazioni tecniche ne limitavano l'uso a campioni conduttivi, poiché campioni non conduttivi come il tessuto biologico accumulavano livelli inaccettabili di carica (accumulo di elettroni all'interno del campione di tessuto)1. Mentre rivesteva il blocco-faccia tra i tagli con la ricarica dei tessuti ridotta al carbonio evaporato, questo aumentò notevolmente il tempo di acquisizione dell'imaging e la memorizzazione delle immagini rimase un problema poiché la tecnologia informatica all'epoca era insufficiente per gestire le grandi dimensioni dei file create dal dispositivo. Questa metodologia è stata rivisitata da Denk e Horstmann nel 2004 utilizzando uno SBF-SEM dotato di una camera a pressionevariabile 2. Ciò ha permesso l'introduzione del vapore acqueo nella camera di imaging che riduce la carica all'interno del campione, rendendo praticabile l'imaging di campioni non conduttivi anche se con una perdita di risoluzione dell'immagine. Ulteriori miglioramenti nella preparazione dei tessuti e nei metodi di imaging ora consentono l'imaging utilizzando un alto vuoto e l'imaging SBF-SEM non si basa più sul vapore acqueo per dissiparela carica 3,4,5,6,7,8,9. Mentre SBF-SEM ha visto un aumento esponenziale dell'uso negli ultimi anni, aspetti tecnici come una corretta preparazione dei tessuti e parametri di imaging sono fondamentali per il successo di questa modalità di imaging.
SBF-SEM consente la raccolta automatizzata di migliaia di immagini di microscopia elettronica registrate in serie, con risoluzione planare fino a 3-5 nm10,11. Il tessuto, impregnato di metalli pesanti e incorporato nella resina, è posto all'interno di un microscopio elettronico a scansione (SEM) contenente un ultramicrotoma dotato di coltello a diamante. Una superficie piana viene tagliata con il coltello a diamante, il coltello viene retratti e la superficie del blocco viene scansionata in un motivo raster con un fascio di elettroni per creare un'immagine di ultrastruttura tissutale. Il blocco viene quindi sollevato una quantità specificata (ad esempio, 100 nm) nell'asse Z, noto come "passo z", e una nuova superficie viene tagliata prima che il processo si ripeta. In questo modo viene prodotto un blocco di immagini tridimensionale (3D) man mano che il tessuto viene tagliato. Questo sistema di imaging beneficia ulteriormente della natura automatizzata del dispositivo, consentendo di lasciare il microscopio incustodito durante il processo di imaging, con la raccolta automatizzata di centinaia di immagini possibili in un solo giorno.
Mentre l'imaging SBF-SEM utilizza principalmente elettroni retroscatterati per formare un'immagine della faccia del blocco, gli elettroni secondari vengono generati durante il processo di imaging12. Gli elettroni secondari possono accumularsi, insieme agli elettroni retroscatterati e a fascio primario che non sfuggono al blocco, e produrre "carica tissutale", che può portare a un campo elettrostatico localizzato nella faccia del blocco. Questo accumulo di elettroni può distorcere l'immagine o causare l'espulsa di elettroni dal blocco e contribuire al segnale raccolto dal rivelatore backscatter, diminuendo il rapporto segnale-rumore13. Mentre il livello di carica tissutale può essere diminuito riducendo la tensione o l'intensità del fascio di elettroni o riducendo il tempo di divaggio del fascio, ciò si traduce in un rapporto segnale-rumoreridotto 14. Quando si utilizza un fascio di elettroni di tensione o intensità inferiore, o il fascio può abitare all'interno di ogni spazio di pixel solo per un periodo di tempo più breve, meno elettroni retroscatterati vengono espulsi dal tessuto e catturati dal rivelatore di elettroni con conseguente segnale più debole. Denk e Horstmann affrontavano questo problema introducendo vapore acqueo nella camera, riducendo così la carica nella camera e sulla faccia del blocco a costo della risoluzione dell'immagine. Con una pressione della camera di 10-100 Pa, una porzione del fascio di elettroni è sparsa contribuendo al rumore dell'immagine e alla perdita di risoluzione, tuttavia questo produce anche ioni nella camera del campione che neutralizza la carica all'interno del bloccocampione 2. Metodi più recenti per neutralizzare la carica all'interno del blocco campione utilizzano l'iniezione focale di gas di azoto sulla faccia del blocco durante l'imaging, o introducendo tensione negativa allo stadio SBF-SEM per ridurre l'energia di carico sonda-fascio e aumentare ilsegnale raccolto 6,7,15. Piuttosto che introdurre distorsioni dello stadio, pressione della camera o iniezione di azoto localizzata per ridurre l'accumulo di carica sulla superficie del blocco, è anche possibile aumentare la conducibilità della resina introducendo carbonio nella miscela di resina consentendo impostazioni di imaging più aggressive16. Il seguente protocollo generale è un adattamento del protocollo Deerinck et al. Mentre il protocollo precedentemente menzionato si concentrava sull'elaborazione dei tessuti e sull'impregnazione di metalli pesanti, questo protocollo fornisce informazioni sul flusso di lavoro di imaging, analisi dei dati e ricostruzione inerente agli studi SBF-SEM. Nel nostro laboratorio, questo protocollo è stato applicato con successo e riproducibile a un'ampia varietà di tessuti tra cui cornea e strutture del segmento anteriore, palpebra, ghiandola lacrimale e harderiana, retina e nervo ottico, cuore, polmone e vie aeree, rene, fegato, muscolo cremastere e corteccia cerebrale / midollo, e in una varietà di specie tra cui topo, ratto, coniglio, porcellino d'India, pesce, monostrato e colture cellulari stratificate, maiale, primate non umano,nonché umano 20,21,22,23. Sebbene piccoli cambiamenti possano essere utili per tessuti e applicazioni specifici, questo protocollo generale si è dimostrato altamente riproducibile e utile nel contesto della nostra struttura di imaging di base.
Protocollo
Tutti gli animali sono stati trattati secondo le linee guida descritte nella Dichiarazione dell'Association for Research in Vision and Ofthalmology per l'uso degli animali nella ricerca sulla vista e sugli oftalmici e nelle linee guida per la manipolazione animale dell'University of Houston College of Optometry. Tutte le procedure per gli animali sono state approvate dalle istituzioni in cui sono state gestite: le procedure per topi, ratti, conigli, cavie e primati non umani sono state approvate dal Comitato per la cura e l'uso degli animali dell'Università di Houston, le procedure zebrafish sono state approvate dal Comitato per la cura e l'uso degli animali della DePauw University e le procedure suino sono state approvate dal Baylor College of Medicine Animal Care and Use Committee. Tutto il tessuto umano è stato trattato in conformità con la dichiarazione di Helsinki in merito alla ricerca sui tessuti umani ed è stata ottenuta un'adeguata approvazione del comitato di revisione istituzionale.
1. Lavorazione dei tessuti
- Preparare una soluzione stock di tampone di cacodilato di sodio da 0,4 M mescolando la polvere di cacodilato di sodio in ddH2O. Mescolare accuratamente tampone e pH regolare la soluzione a 7.3. Questo tampone viene utilizzato per rendere fissivo (composizione descritta di seguito nel passaggio 1.3), tampone di lavaggio, nonché soluzioni di osmio e ferrocianuro di potassio.
NOTA: La fissazione della perfusione è spesso il miglior metodo di fissazione per gli studi SBF-SEM, poiché la fissazione avviene rapidamente e in tutto il corpo. Se la fissazione della perfusione non è possibile all'interno del progetto di studio, passare al passaggio 1.3. - Eseguire la fissazione della perfusione con la pressione fisiologica appropriata perl'animale modello 24,25,26. Questo viene fatto tramite perfusione sequenziale transcardica con salina eparinizzata seguita da fissante, ognuno posto ad un'altezza specifica (ad esempio, 100 cm) sopra l'organismo (appropriato alla pressione fisiologica del sistema vascolare nel modello animale), con un flusso fisso nel ventricolo sinistro e uscendo da un'incisione fatta nell'atrio destro. Il tessuto di interesse diventerà pallido man mano che il sangue viene sostituito con fissativo, se tutto o una parte del tessuto non sbianca, il tessuto potrebbe non essere fissato in modo appropriato e l'ultrastruttura potrebbe non essere preservata.
- Utilizzare una lama di rasoio o bisturi affilato per tagliare campioni di tessuto in blocchi non più grandi di 2 mm x 2 mm x 2 mm. Se il passaggio 1.2 è stato saltato, farlo rapidamente in modo che il tessuto possa essere riparato il più rapidamente possibile.
- In alternativa, sezionare il tessuto sotto fissante e passare a un fresco fissatore per completare il processo di immersione. La composizione finale del fissatore consiste in 0,1 M di tampone di cacodilato di sodio contenente il 2,5% di glutaraldeide e 2 mM di cloruro di calcio. Lasciare procedere la fissazione per un minimo di 2 ore a temperatura ambiente e un massimo di pernottamento a 4 °C. Se possibile, utilizzare una piastra rocker/tilt per agitare delicatamente i campioni durante il fissaggio.
- In alternativa, se è disponibile un forno a microonde inverter, fissare il tessuto nel suddetto fissatore sotto vuoto a 150 watt per 4 cicli di 1 minuto di accensione, 1 minuto di riposo. La fissazione a microonde è il metodo preferito per il passaggio 1.3 in quanto fissa rapidamente i tessuti e preserva l'ultrastrutturatissutale 27.
NOTA: Il tessuto non deve mai essere lasciato asciugare durante questo protocollo, bisogna fare attenzione a trasferire rapidamente i tessuti da una soluzione all'altra.
- Lavare il tessuto fisso 5 volte per 3 minuti ciascuno (15 minuti totali) a temperatura ambiente in tampone di cacodilato di sodio da 0,1 M contenente cloruro di calcio da 2 mM.
- Rendere fresca la seguente soluzione di ferrocianuro di osmio, preferibilmente durante le fasi di lavaggio precedenti. Combinare una soluzione di tetrossido di osmio al 4% (preparata in ddH2O) con un volume uguale del 3% di ferrocianuro di potassio in tampone cacodilato da 0,2 M con cloruro di calcio da 4 mM. Dopo la precedente fase di lavaggio, posizionare il tessuto in questa soluzione per 1 ora sul ghiaccio al buio e nel cappuccio dei fumi.
NOTA: Il tetrossido di osmio è una sostanza cristallina gialla che viene fornita in un'ampola. Per creare la soluzione di tetrossido di osmio, aprire l'ampola, aggiungere ddH2O e sonicare per 3-4 ore al buio fino a quando i cristalli non sono completamente sciolti. La soluzione di tetrossido di osmio è una soluzione giallo chiaro, se la soluzione è nera l'osmio è stato ridotto e non deve più essere utilizzato. - Mentre il tessuto sta incubando nella soluzione di ferrocianuro di osmio, iniziare a preparare la soluzione di tiocarboidrazide (TCH). Preparare questa soluzione fresca e rla prontamente disponibile alla fine del periodo di fissazione dell'osmio ferrocianuro di 1 ora. Unire 0,1 g di tiocarboidrazide con 10 mL di ddH2O e posizionare questa soluzione in un forno a 60 °C per 1 ora. Per assicurarsi che la soluzione sia sciolta, ruotare delicatamente ogni 10 minuti. Prima dell'uso, filtrare questa soluzione attraverso un filtro siringa da 0,22 μm.
- Prima di incubare in TCH, lavare il tessuto con temperatura ambiente ddH2O 5x per 3 minuti ciascuno (15 minuti in totale).
- Posizionare il tessuto nella soluzione TCH filtrata per un totale di 20 minuti a temperatura ambiente (Figura 1A-C).
- Dopo l'incubazione in TCH, lavare il tessuto 5 volte per 3 minuti ciascuno (15 minuti totali) a temperatura ambiente ddH2O.
- Mettere il tessuto in ddH2O contenente il 2% di tetrossido di osmio (non osmio ridotto con ferrocianuro di potassio) per 30 minuti a temperatura ambiente. Questo dovrebbe essere fatto nella cappa dei fumi e al buio in quanto l'osmio può essere ridotto dalla luce (ad esempio, sotto un foglio dialluminio) (figura 1D-F).
- Dopo l'incubazione di tetrossido di osmio, lavare il tessuto 5 volte per 3 minuti ciascuno (15 minuti totali) a temperatura ambiente ddH2O.
- Mettere il tessuto in acetato di uro uro acquoso all'1% (polvere di acetato di uro di uro mescolato in ddH2O) durante la notte in frigorifero a 4 °C.
- Poco prima di rimuovere il tessuto dal frigorifero, preparare la soluzione aspartata di piombo di Walton fresca. Iniziare sciogliendo 0,066 g di nitrato di piombo in 10 mL di soluzione di acido aspartico da 0,03 M (0,04 g di acido aspartico in 10 mL di acqua distillata) e regolare il pH a 5,5 con 1 N KOH (0,5611 g in 10 mL di acqua distillata).
ATTENZIONE: Un precipitato può formarsi quando si regola il pH. Questo non è accettabile.- Utilizzando una barra di agitazione, aggiungere lentamente il dropwise da 1 N KOH durante il monitoraggio del pH. Preriscaldare la soluzione di aspartato di piombo chiaro finita in forno a 60 °C per 30 minuti. Se si forma un precipitato, la soluzione non può essere utilizzata e deve essere preparata un'altra soluzione.
- Rimuovere il tessuto dal frigorifero e lavare 5 volte per 3 minuti ciascuno (15 minuti totali) a temperatura ambiente ddH2O.
- Dopo il lavaggio, posizionare il tessuto nella soluzione di aspartato di piombo di Walton riscaldata per 30 minuti mantenendo la temperatura a 60 °C.
- Dopo l'incubazione nell'aspartato di piombo di Walton, lavare il tessuto 5 volte per 3 minuti ciascuno (15 minuti totali) a temperatura ambiente ddH2O(Figura 1G-I).
- Disidratare il tessuto attraverso una serie di acetone ghiacciato (30%, 50%, 70%, 95%, 95%, 100%, 100% e acetone al 100% (in ddH2O se applicabile) consentendo 10 minuti per ogni fase della serie.
- Dopo la serie di disidratazione ghiacciata, mettere il tessuto in acetone a temperatura ambiente per 10 minuti.
- Durante questo periodo, formulare la resina Embed 812 ACM. Usa la ricetta "hard mix" in quanto è più resistente ai danni al fascio. Mescolare accuratamente la resina e posizionare il tessuto in Embed 812:acetone (1:3 mix) per 4 ore, seguito da Embed 812:acetone (1:1 mix) per 8 ore o durante la notte, e infine Incorporare 812:acetone (3:1 mix) durante la notte. Eseguire questi passaggi di infiltrazione di resina a temperatura ambiente.
- Il giorno successivo, posiziona il tessuto in 100% Embed 812 per 4-8 ore, quindi in fresco 100% Embed 812 durante la notte e infine in fresco 100% Embed 812 per 4 ore. Eseguire questi passaggi di infiltrazione di resina a temperatura ambiente.
- Poco prima dell'incorporamento, posizionare una piccola quantità di resina in un contenitore di miscelazione e mescolare lentamente (un bastone di legno può essere utilizzato per mescolare) in polvere nera fumo fino a quando la resina non è satura della polvere ma è ancora fluida e non diventa granulosa. Dovrebbe assomigliare all'inchiostro spesso ed essere in grado di gocciolare lentamente dal bastone di legno senza grumi visibili.
- Orientare i campioni di tessuto in uno stampo in gomma siliconica e scattare una foto in modo che l'orientamento del campione all'interno del blocco di resina venga registrato e possa essere referenziato. Coprire i campioni in resina satura nera fumo sulla punta dello stampo in silicone e posizionare lo stampo in un forno per ~ 1 ora a 65 °C.
- Posizionare lo stampo su una pendenza per contenere la resina sulla punta dello stampo dove copre il campione di tessuto. Posizionare un'etichetta con un identificatore di campione esperimento/tessuto nello stampo all'estremità opposta della resina (Figura 2A).
- Rimuovere lo stampo in silicone dal forno e riempire il resto dello stampo con resina trasparente (senza nero fumo) assicurandosi che l'etichetta rimanga visibile. Curare la resina infusa con nero fumo abbastanza da non mescolare facilmente con la resina trasparente.
- Preparare un pozzo in più all'interno dello stampo che non contenga tessuto. A partire dal pozzo extra, riempire il resto dello stampo con resina trasparente.
- Se la resina infusa nero fumo inizia a sanguinare nella resina trasparente, riposizionare lo stampo in silicone nel forno per un ulteriore tempo (ad esempio, 15 minuti).
- Una volta che tutti i campioni di tessuto sono stati completati con resina trasparente, riposizionare lo stampo in silicone nel forno (piatto, senza inclinazione) a 65 °C per 48 ore per completare il processo di polimerizzazione.
2. Preparazione del blocco
NOTA: Il metodo dipenderà da come il campione è orientato nel blocco e da come deve avvenire il sessatura. Tuttavia, l'orientamento tissutale più comune trova il tessuto centrato nella punta del blocco di resina, perpendicolare all'estremità lunga del blocco di resina.
- Nella maggior parte dei casi, tagliare prima l'estremità del blocco per localizzare il tessuto posizionando il blocco del campione nel mandrino del microtomo con l'estremità affusolata che sporge circa 5-6 mm dal mandrino. Bloccarlo in posizione con la vite impostata e posizionarlo sotto una lampada termica.
- Dopo diversi minuti il blocco sarà malleabile e facile da tagliare. Posizionare il mandrino nel supporto stereomicroscopio e utilizzare una nuova lama di rasoio a doppio bordo per rendere le sezioni sottili parallele alla faccia del blocco fino a quando il tessuto non è visibile. Questo è meglio visto dalla luce di pesca attraverso la faccia del blocco, il campione di tessuto sarà meno riflettente e granulare rispetto a quelle porzioni della resina che sono prive di tessuto. Consultare la fotografia scattata di campioni di tessuto prima dell'introduzione della resina satura nera fumo per un'idea di come e dove si trova il tessuto.
- Mettere da parte un portaocca campione per il taglio. Questo portaoci non viene mai inserito nella camera SEM e può quindi essere maneggiato senza guanti, questo sarà indicato come il portaocca perno di rifilatura. Qualsiasi porta campioni destinato ad essere collocato nella camera di imaging non deve mai essere toccato senza guanti. In questo modo si evita di introdurre grasso e olio nella camera del microscopio.
- Posizionare un perno del campione di alluminio nel portaocca di rifilatura e stringere leggermente la vite impostata con la faccia (superficie piana) del perno tenuto 3-4 mm sopra il portaocito.
- Fai diversi graffi profondi e incrociati nella faccia del perno per fornire una superficie più ampia per la colla utilizzata per tenere il campione in posizione. Se si utilizza un perno in alluminio, per questo passaggio è consigliato un piccolo cacciavite a testa piatta in acciaio (Figura 2B).
- Posizionare il mandrino contenente il campione di tessuto sotto la lampada termica fino a quando la resina diventa morbida e malleabile, quindi posizionarlo nel contenitore del mandrino sotto lo stereomicroscopio.
- Utilizzo di una lama di rasoio a doppio taglio per tagliare via la resina in eccesso dalla porzione del blocco di resina contenente il campione di tessuto. In definitiva la dimensione del blocco tissutale attaccato al perno sarà di circa 3 mm di diametro e 2-3 mm di altezza.
- Spingere con cura il rasoio direttamente nel blocco di resina di circa 1-2 mm, quindi spingere con attenzione il rasoio orizzontalmente nel blocco di resina a una profondità pari al taglio precedente. Fallo lentamente e con grande cura, in quanto è possibile danneggiare o tagliare la porzione del blocco contenente il campione di tessuto. Man mano che i due tagli si incontrano, la resina in eccesso si separerà dal blocco. Continuare a rimuovere la resina fino a quando rimane solo un'area rialzata di 3 mm x 3 mm.
- Dopo questo taglio iniziale, posizionare il blocco (ancora nel mandrino) sotto la lampada termica per diversi minuti.
- Una volta che la resina diventa morbida e malleabile, riposizionare il blocco sotto lo stereomicroscopio. Utilizzando una nuova lama a doppio bordo, tagliare la parte superiore del blocco di resina, circa 1 mm sotto la porzione rifilata, con un unico taglio liscio. Una superficie piana è preferibile in quanto verrà incollata al perno del campione. Fare attenzione a non permettere che il campione si perdi, poiché questo passaggio richiede una certa forza che può essere trasferito nella parte rimossa del blocco e farlo volare via. Mettere da parte il campione tagliato e tagliato.
- Posizionare il portaocito perno di rifilatura contenente il perno di alluminio tagliato nel contenitore stereomicroscopio. Applicare un sottile strato di colla cianoacrilato sulla faccia del perno in modo che copra completamente il perno senza formare un menisco visibile. Raccogliere il pezzo tagliato del blocco di tessuto con pin e posizionarlo sulla faccia del perno. Centrare il campione di tessuto sul perno del campione. Spingilo verso il basso e tienilo premuto per diversi secondi. Lasciare impostare la colla per diversi minuti.
- Quando la colla è completamente asciutta, riposizionare il portaocca perno di rifilatura sotto lo stereomicroscopio. Utilizzando un file fine, filettare la resina in eccesso in modo che nessuna resina sia sporgente sul perno. La forma della resina dovrebbe assomigliare alla testa circolare del perno.
- Individuare il tessuto sulla parte sollevata del blocco di resina, l'illuminazione obliqua è utile per questo. Utilizzando un rasoio a doppio bordo, la porzione sollevata della resina contenente il campione di tessuto deve essere tagliata in un'area non superiore a 1 mm2. Se possibile, la faccia del blocco può essere tagliata ancora più piccola, questo ridurrà lo stress sul coltello a diamante e migliorerà la sua longevità.
- Rimuovere quanta più resina in eccesso possibile, lasciando il blocco leggermente più lungo in una dimensione. Questo viene fatto lentamente e con cura, in quanto è possibile che la resina contenente il campione di tessuto si rompa se viene applicata troppa forza. Mentre si consiglia un rasoio, è possibile utilizzare un file di metallo fine per questo passaggio.
- Con un angolo di file metallico fine la resina in eccesso, nell'area esterna alla porzione sollevata contenente il campione di tessuto, verso il bordo del perno (Figura 2C).
- Rimuovere le particelle di resina e la polvere dal campione preparato prima di applicare vernice argentata seguita dallo sputtering d'oro. Mescolare l'argento con acetone in modo che sia un liquido facilmente spalmabile, simile allo smalto per unghie (ma non così sottile da gocciolare dall'applicatore) e applicare un rivestimento sottile sull'intera superficie del blocco del campione. L'acetone evapora rapidamente, quindi potrebbe essere necessario aggiungere acetone aggiuntivo man mano che la vernice argentata inizia ad addensarsi.
- Lasciare asciugare la vernice argentata durante la notte prima di caricarsi al microscopio.
NOTA: Questo strato d'argento deve essere sottile per evitare di espandere la faccia del blocco oltre 1 mm x 1 mm e, mentre la vernice argentata non ha mai danneggiato il coltello a diamante, si consigliano ancora facce di blocco più piccole per preservare la longevità del coltello diamanta. L'acetone miscelato con l'argento deve evaporare completamente prima dello sputtering dell'oro o del caricamento del campione al microscopio per evitare di introdurre vapore acetone nella camera di imaging. - Dopo l'applicazione della vernice argentata, applicare un sottile strato di oro sul blocco campione. Utilizzando un dispositivo standard di sputtering sottovuoto dotato di un bersaglio standard in lamina d'oro, una pressione della camera di 200 milliTorr (gas Argon) e 40 milliambi in funzione per 2 minuti si tradurrà in un rivestimento in oro spesso 20 nm.
- Lasciare asciugare la vernice argentata durante la notte prima di caricarsi al microscopio.
- Dopo il rivestimento, posizionare il blocco montato e tagliato in un tubo con l'etichetta di esperimento appropriata attaccata. Creare tubi personalizzati utilizzando pipette di trasferimento usa e getta.
- Tagliare la pipetta di trasferimento appena sotto la lampadina, lasciando una breve porzione del tubo di pipetta di trasferimento attaccato sotto l'estremità bulbosa. Accorciare la porzione tubolare che è stata tagliata e tagliare la punta della pipetta abbastanza indietro in modo che il perno del campione di alluminio possa essere spinto in modo snuggly all'interno di esso.
- Posizionare l'estremità contenente il perno del campione di alluminio all'interno dell'estremità bulbosa della pipetta di trasferimento modificata.
- Prima di caricare un blocco di tessuto preparato, tagliare con cura la vernice argentata in eccesso dalla superficie della faccia del blocco.
3. Impostazioni SEM per l'imaging della faccia del blocco
NOTA: Le impostazioni di imaging che seguono sono state prodotte sul dispositivo utilizzato dagli autori, che è elencato nella tabella dei materiali fornita. Mentre questo dispositivo è in grado di imaging a pressione variabile, i migliori risultati sono stati catturati sotto alto vuoto.
- Tempo di dimora: Utilizzare 12 μs/px durante la sessatura seriale. Quando è stata identificata una regione di interesse, un'immagine a risoluzione più elevata può essere acquisita a 32 μs/px.
- Impostazioni vuoto: Utilizzare una pressione del cannone di 9e-008 Pa, una pressione della colonna di 1,1e-004 Pa e una pressione della camera di 9,5e-002 Pa.
- Tempo di acquisizione: Con le impostazioni di cui sopra, acquisisci uno stack di immagini px 2048x2048 a una velocità di 50 secondi per immagine. Le immagini a risoluzione più elevata delle regioni di interesse possono essere acquisite a 4096x4096 px a poco meno di 9 minuti per immagine.
- Spessore sezione: Utilizzare 100-200 nm. Meno è possibile, ma può richiedere una tensione del fascio inferiore, intensità o tempo di dimora.
- Alta tensione (HV): Utilizzare 7-12 kV. Mentre l'aumento della tensione riduce le dimensioni dello spot e aumenta la risoluzione, introduce più possibilità di danni al fascio. KV più alto aumenta la penetrazione del fascio che si traduce in perdita di dettagli. Tuttavia, l'abbassamento del kV degrada il rapporto segnale/rumore (Figura 3)14.
- Intensità fascio (BI): Il dispositivo SBF-SEM dell'autore offre una scala di intensità del fascio che va da 1 a 20. Su questa scala, i valori di 5-7 forniscono immagini di qualità senza carica eccessiva e danni al fascio. Maggiore è la BI maggiore è la risoluzione, tuttavia, ci sono maggiori possibilità di caricare e danneggiare il fascio14.
- Dimensioni spot e ingrandimento immagine: Determinare la dimensione del punto in base all'intensità del fascio e al livello di tensione. Idealmente, la dimensione del punto non dovrebbe essere maggiore della dimensione dei pixel utilizzata. La dimensione dei pixel è determinata dividendo il campo visivo (FOV) per il numero di pixel. Ad esempio, un FOV da 25 μm con una dimensione dell'immagine di 2048x2048 px darebbe 12,2 nm per pixel. Pertanto la dimensione dello spot non deve essere superiore a 12,2 nm. La figura 4 mostra come sono correlate HV, BI e dimensioni spot.
- Distanza di lavoro (WD) - Con l'imaging della faccia a blocchi la distanza di lavoro non è regolabile. È semplicemente un fattore di attenzione. Sarà quasi identico per tutti i blocchi di immagine. Sebbene la distanza di lavoro non sia regolabile, svolge un ruolo fondamentale nella risoluzione dell'immagine acquisita. Con la diminuzione della distanza di lavoro, aumenta il limite di risoluzione per le immagini acquisite. In alcuni casi può essere possibile ridurre la distanza di lavoro apportando modifiche all'interno della camera di imaging, tuttavia queste modifiche devono essere apportate a discrezione dell'utente. Al fine di ridurre la distanza di lavoro e aumentare la risoluzione dell'immagine, abbiamo allentato le viti del microtomo di montaggio della porta e riposizionato il microtomo in modo che riposava ~ 2 mm più vicino al rilevatore di travi dopo aver ritrasposizione le viti.
- Risoluzione - Utilizzando le impostazioni di cui sopra, è possibile una risoluzione x & y fino a 3,8 nm. È importante notare che la risoluzione è limitata dalle dimensioni del punto del fascio e la risoluzione in pixel delle acquisizioni di immagini (ad esempio, un campo visivo di 20 μm catturato in un'immagine pixel 2048x2048 ha una risoluzione in pixel di 9,8 nm, anche se è stata utilizzata una dimensione del punto di 3,8 nm). La risoluzione dell'immagine nel piano Z dipende dallo spessore della sezione, scopriamo che 100-200 nm funziona bene con questo protocollo.
Risultati
Cornea di topo
Questo protocollo è stato ampiamente applicato alla cornea di topo. Utilizzando l'imaging SBF-SEM, è stata dimostrata la soddisfazione di una rete di fasci di microfibrille senza elastina (EF PCB) all'interno della cornea di topo adulta. In precedenza si credeva che questa rete fosse presente solo durante lo sviluppo embrionale e postnatale precoce. SBF-SEM ha rivelato una vasta rete EFMB in tutta la cornea, con fibre individuali che hanno un diametro di 100-200 nm se misurate in sezione trasversale. Si è anche scoperto che questa rete EFMB era organizzata in strati distinti, con fibre strettamente associate ai cheratociti, anche all'interno di invaginazioni poco profonde sulla superficie del cheratocita (Figura 5). La scoperta di fibre EFMB nella cornea adulta ha portato alla microscopia elettronica a trasmissione immunogold-labeling (TEM), alla fluorescenza e agli studi confocali per comprendere ulteriormente la natura di questa rete23.
Un'ulteriore applicazione di questo protocollo portò alla scoperta di una popolazione precedentemente sconosciuta di nervi corneali centrali che si fondono con cellule epiteliali basali al confine stromale-epiteliale (Figura 6). In precedenza, si credeva che tutti i nervi che interagivano con l'epitelio a questo confine penetrassero nell'epitelio corneale e ramificassero producendo il plesso subbasale ed epiteliale. In questo studio, circa il 45% dei nervi centrali che interagiscono con l'epitelio basale sono stati sottoposti a fusione cellulare-cellula piuttosto che a una semplice penetrazione. Utilizzando metodi stereologici applicati ai set di dati SBF-SEM, è stato possibile mostrare che questa nuova popolazione nervosa aveva un rapporto superficie-volume circa la metà di quello dei nervi penetranti, coerente con il loro aspetto "gonfio" (Nerve Fusion - 3.32±0.25, Nerve Penetration - 1.39±0.14, p ≤ 0.05). Sono state create ricostruzioni 3D di fasci nervosi penetranti e fusi e dei loro mitocondri, evidenziando la mancanza di mitocondri in porzioni fuse dei fasci nervosi. La scoperta della fusione neuronale-epiteliale cellulare utilizzando SBF-SEM ha portato a studi di fluorescenza che hanno verificato la continuità della membrana tra le due cellule fuse21.
La cornea centrale è un tessuto avascolare, e come tale la vascolarizzazione limbale periferica è di particolare importanza per la salute generale della cornea. Le relazioni cellula-cellula e l'ultrastruttura di questa regione sono complesse; tuttavia, la capacità di apprezzare queste interazioni cellula-cellula e connessioni ultrastrutturali è stata limitata negli studi tem a fluorescenza e a sezione singola. Per questo motivo una pila di immagini SBF-SEM contenente vascucolatura limbale, fasci nervosi e cellule associate è stata segmentata manualmente per la ricostruzione 3D (Figura 7). In questa immagine si può vedere la stretta associazione tra le giunzioni endoteliali vascolari e un pericite sovrapposto, i singoli granuli di una cellula mastivascolare, il nucleo e il bordo anteriore di un neutrofilo che striscia lungo la superficie esterna della parete del vaso sanguigno, così come un fascio nervoso che passa.
Nel complesso, questo corpus di lavoro dimostra la capacità di questo protocollo di produrre set di dati di microscopia elettronica 3D di alta qualità in tessuti ricchi di matrice extracellulare ed epitelio, nonché vascucolatura e cellule associate.
Retina primate di ordine superiore - Plesso nervoso e rete vascolare
Lo strato di fibra nervosa retinica (RNFL) dei primati di ordine superiore contiene e dipende da una vasta rete vascolare. Spesso, le malattie della retina comportano cambiamenti in entrambi i parametri dello strato di fibre nervose retiniche e nella vascucolatura che si trova al suo interno. Comprendere la relazione tra la RNFL e la sua rete vascolare in tessuto sano e non patologico è il primo passo per comprendere eventuali cambiamenti che possono verificarsi a causa della malattia. Per comprendere meglio questa relazione, il protocollo SBF-SEM è stato applicato alla normale retina di primati di ordine superiore ed è stata eseguita la ricostruzione della rete vascolare e sono stati estratti dati volumetrici da questa ricostruzione (Figura 8). Questa regione di 4.642.307μm 3 della RNFL conteneva un letto vascolare di 1,207x10-4 μL in volume, che cova il 2,6% del volume totale della RNFL. Questo lavoro dimostra la capacità di questo protocollo di produrre set di dati di microscopia elettronica 3D di alta qualità in tessuto neurologico denso.
Zebrafish e Cuore gigante di Danio - Muscolo striato e vascucolatura in via di sviluppo
Sia il pesce zebra che il danio gigante sono modelli importanti per lo sviluppo e la rigenerazione del cuore. Storicamente, il cuore di zebrafish è considerato costituito da due segmenti miocardiali anatomicamente distinti che funzionano insieme a sostegno delle esigenze fisiologiche del pesce zebra. Tuttavia, l'interfaccia tra questi due strati ventricolari non era ben compresa. Utilizzando questo protocollo, è stata scoperta una regione giunzionale precedentemente non riconosciuta costituita da un sottile foglio di fibroblasti. Si è constatato che le aperture all'interno di questo foglio permettevano a due segmenti miocardici separati di entrare in contatto e formare complesse giunzioni di aderenza tra cui desmosomes e fascia adherens22.
Questo protocollo è stato utilizzato in ulteriori lavori di esame della rete vascolare del cuore gigante danio in via di sviluppo (Figura 9). Questo metodo consente l'apprezzamento 3D della rete di miociti cardiaci in via di sviluppo e la sua relazione con lo sviluppo della microvascolarizzazione. Nel complesso, questo lavoro dimostra la capacità di questo protocollo di produrre set di dati di microscopia elettronica 3D di alta qualità nei tessuti muscolari e altamente vascolarizzati.
Impostazioni dell'immagine, ricarica e risoluzione
Sebbene sia necessaria un'adeguata fissazione e colorazione dei metalli pesanti per l'imaging SBF-SEM di qualità, altrettanto importante è l'uso di resina conduttiva e impostazioni di imaging adeguate per le domande affrontate. In questo protocollo, l'uso del nero fumo viene utilizzato al fine di aumentare la conducibilità del blocco campione e fornire un condotto al perno di montaggio per il gioco degli elettroni secondari dalla faccia del blocco. Ciò si è dimostrato efficace nella lotta contro la carica tissutale che spesso degrada la qualità dell'immagine nei tessuti non preparati con nerofumo 16. Inoltre, la vernice argentata e lo sputtering d'oro applicato al blocco forniscono un percorso di dissipazione per l'accumulo di elettroni. Alcuni dispositivi consentono l'aggiunta di un compensatore di carica focale, che riduce la carica applicando uno sbuffo di azoto sulla faccia del blocco, tuttavia abbiamo avuto un successo simile con l'uso del nero fumo e l'applicazione di vernice argentata e sputtering oro al blocco15. La mancanza di conducibilità del campione può portare all'accumulo di elettroni visibile come carica tissutale (Figura 1), così come scariche visibili come bruschi spostamento e curvatura dell'immagine che riducono drasticamente la qualità dell'immagine(Figura 10B & F ). L'uso del nero fumo consente l'imaging sotto vuoto elevato e l'uso di impostazioni dell'immagine che si traducono in un elevato rapporto segnale-rumore e una migliore risoluzione dell'immagine. Una di queste impostazioni che porta a una migliore qualità dell'immagine è il tempo di ensità dei pixel. Il processo di imaging SBF-SEM prevede la scansione raster di un fascio di elettroni attraverso la superficie del campione per generare elettroni retroscatterati che il rivelatore del microscopio può raccogliere e interpretare come segnale. Il periodo di tempo in cui questo fascio può soffermarsi nello spazio di ogni pixel porta all'assegnazione di un valore di pixel più accurato a ciascuna posizione dei pixel (Figura 3A & B)2. C'è tuttavia un equilibrio che deve essere raggiunto tra l'aumento del segnale al rumore, la risoluzione e i danni infliti alla faccia del blocco. Il fascio irradia efficacemente la faccia del blocco con elettroni ad alta energia che possono abbattere e ammorbidire la resina con conseguente degradazione dell'immagine e complicazioni ditaglio (figura 10)28. Più sottile è la risoluzione z richiesta, più diventa difficile mantenere l'imaging ad alta risoluzione. Generalmente usiamo passaggi z di 100-200 nm, tuttavia le dimensioni del passaggio Z di 25-50 nm sono state segnalate5,29,30,31. Con gradini z di queste dimensioni, la rottura e l'ammorbidimento della resina a causa di danni al fascio possono portare a una compressione della resina che fa perdere al coltello un taglio o un taglio del blocco-faccia ma con "chiacchiere" dove il coltello salta sulla superficie del blocco creando increspature e bande13. Sebbene i piccoli passaggi z siano una prospettiva interessante, è importante tenere a mente la domanda di ricerca specifica quando si sceglie un passaggio z appropriato. L'over-sampling può portare a considerazioni sostanziali sull'archiviazione dei dati e a un aumento del tempo necessario per produrre ricostruzioni 3D.
Fissazione e colorazione dei tessuti
Prima dell'incubazione di metalli pesanti, i tessuti devono essere fissati in glutaraldeide. Mentre consigliamo vivamente la fissazione a microonde sotto vuoto per la conservazione dell'ultrastrutturatissutale 27,se non è disponibile un forno a microonde di grado di laboratorio, un forno a microonde inverter commerciale con potenzavariabile può essere sostituito 32,33,34,35. In questo caso, è necessario prestare particolare attenzione per garantire che la distorsione dei tessuti non si verifichi. Una fissazione impropria dei tessuti può causare una morfologia dei tessuti alterata, come si può vedere nella figura 10E. Questo protocollo, come la maggior parte dei moderni protocolli di colorazione SBF-SEM, è stato adattato dalla procedura di colorazione delineata da Deerinck nel2010 17,basata sulle macchie di osmio-tiocarboidrazide-osmio create da Willingham e Rutherford nel 198436. I metalli pesanti utilizzati in questo protocollo aggiungono contrasto alle strutture cellulari all'interno di un campione di tessuto (Figura 1). L'incubazione iniziale dell'osmio avviene con osmio ridotto che si lega ai legami C =C in grassi insaturi che portano alla colorazione di membrane e lipidi37,38. L'osmio è ridotto dal ferrocianuro di potassio, che aiuta nella colorazione dei lipidi saturi e agisce anche per stabilizzare i fosfolipidi39,40. Il tiocarboidrazide viene successivamente aggiunto come un mordente che si lega all'osmio dalla prima incubazione, fungendo da ponte su cui ulteriore osmio è legato in una fase successiva del protocollo41. L'acetato di uranile, che è un sale di uranio, è un efficace agente contrastante per lipidi, acidi nucleici e proteine, mentre il citrato di piombo migliora il contrasto di proteine e glicogeni. Le diverse affinità di questi agenti per i componenti cellulari migliorano ulteriormente il contrasto complessivo all'interno dei tessuti al di là delle incubazioni di osmio42.
Imaging del block-face
Figura 11, Figura 12, Figura 13 illustrano gli effetti combinati della tensione, del tempo di uscita dei pixel e dell'intensità del fascio. La pratica convenzionale suggerisce che una combinazione di bassa tensione, tempo di soffermazione breve e bassa intensità del fascio sono necessari per l'imaging ottimale e prevenire danni al fascio al blocco campione. Contrariamente a queste impostazioni, la Figura 11, la Figura 12, la Figura 13 illustrano che tensioni più elevate (ad esempio, 7 kV), tempi di soffermazione più lunghi (32 μs/px) e intensità del fascio più elevate (impostazione 6 nel nostro caso) possono produrre una qualità dell'immagine superiore rispetto alle impostazioni convenzionali.
SBF-SEM consente la raccolta di immagini seriali di microscopia elettronica che possono essere raccolte come set di dati 3D composto da voxel. Sebbene questo sia un uso incredibilmente potente di SBF-SEM, questo metodo consente anche l'imaging rapido e ripetibile di eventi biologici rari o cellule. L'acquisizione di immagini mediante SBF-SEM può essere monitorata per eventi rari e l'imaging messo in pausa al fine di raccogliere immagini di ingrandimento /risoluzione più elevate di questi eventi. Inoltre, il blocco può essere rimosso dalla camera del microscopio e dalla faccia del blocco sesata per l'imaging della microscopia elettronica a trasmissione (TEM). In questo modo è possibile raccogliere grandi set di dati di eventi rari utilizzando SBF-SEM e apprezzati su scala angstrom utilizzando TEM.

Figura 1: Raffronti SBF-SEM e TEM in varie fasi del protocollo. Questo protocollo contiene più passaggi in cui il tessuto campione è macchiato di metalli pesanti. Ciò influisce non solo sul contrasto tissutale e sull'apprezzamento delle strutture cellulari e degli organelli, ma anche sui livelli di carica che si verificano quando il tessuto viene immaginato. Questa figura contiene tre punti di vista distinti sul tessuto preparato: una vista a basso ingrandimento (A, D & G), una vista ad alto ingrandimento (B, E & H) e un confronto TEM della cornea di topo preparata (C, F & I). Si può notare che immagini di ingrandimento più elevate possono comportare un aumento della carica tissutale, poiché il fascio di elettroni è concentrato in una regione di tessuto più piccola. La fila superiore(A-C)è un campione rappresentativo di tessuto lavorato fino al completamento del passaggio 1.8, ed è stato impregnato di ferrocianuro di potassio, tetrossido di osmio e tiocarboidrazide. Le frecce nelle prime due colonne mostrano l'interfaccia epiteliale-stromica come punto di riferimento. Si noti il basso livello di contrasto rispetto alle due file inferiori, così come l'aumento dei livelli di carica tissutale. Il campione nella riga centrale (D-F) è stato elaborato attraverso il completamento del passaggio 1.10 e beneficia di un ulteriore passo di tetrossido di osmio ed è visibilmente più contrastato del campione nella riga superiore. Mentre le strutture cellulari sono percepibili, la ricarica è ancora presente. Il campione nella riga inferiore(G-I) beneficiadel protocollo di colorazione completo e ha una carica minima dei tessuti. L'imaging TEM rivela i livelli di contrasto tissutale impartiti dai metalli pesanti presenti ad ogni fase (colonna destra): gli organelli nell'endotelio corneale (*) sono più contrastati e apparenti man mano che la lavorazione tissutale continua attraverso il protocollo. Inoltre, i dettagli stromali di collagene e fibrillina diventano più visibili (punta di freccia) man mano che il protocollo viene completato. Pannello A, D & G scale bar = 50 μm. Pannello B, E & H scale bar = 10 μm. Pannello C, F & I barra di scala = 1 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Schema del bloccotissutale incorporato, del perno del campione e della preparazione finale. (A) Il tessuto deve essere posizionato in un orientamento noto sulla punta stessa dello stampo in resina e sul terzo superiore dello stampo riempito con resina satura nera fumo. La regione dello stampo più lontano dal tessuto dovrebbe rimanere chiara in modo che l'etichetta dell'esperimento possa essere chiaramente vista. (B) La superficie del perno del campione deve essere graffiata per produrre un motivo a griglia, il che consente una maggiore area di contatto per la colla cianoacrilato per indurirsi tra il blocco del campione preparato e il perno. (C) La resina saturo nera fumo deve creare un'ampia area di contatto con la testa del perno del campione, tuttavia la regione tagliata dal coltello a diamante non deve essere superiore a 1x1 mm. È buona pratica assotticcare il blocco verso la punta. Ciò riduce al minimo le forze di taglio sul coltello a diamante e, avendo una base più ampia, il blocco è più resistente alla separazione dal perno durante la sezionamento. Clicca qui per visualizzare una versione più grande di questa figura.
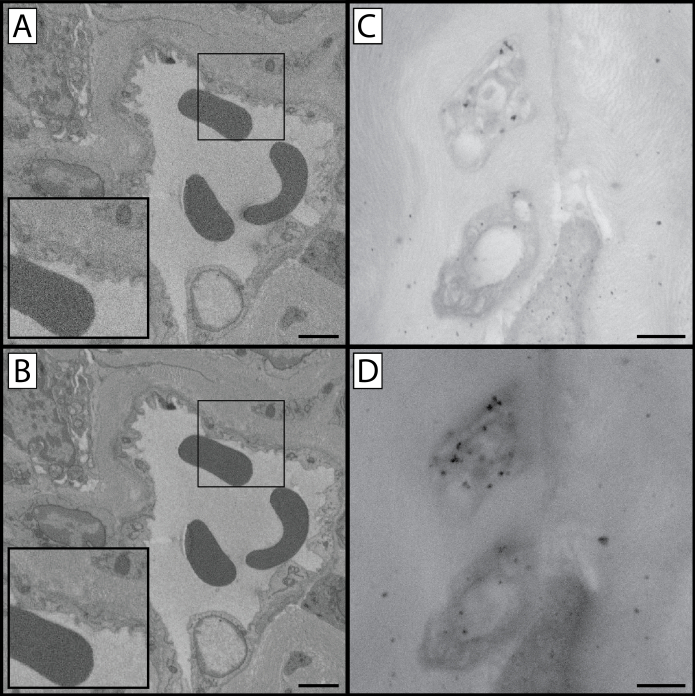
Figura 3: Confronto delle impostazioni di acquisizione dell'immagine. (A & B) I pannelli A e B confrontano la qualità e la risoluzione dell'immagine in funzione del tempo di tà dei pixel. Il pannello A è stato creato utilizzando un tempo di divagazione di 32 μs/pixel a 4 kV e soffre di un rapporto segnale/rumore diminuito come si evivi dall'aspetto "granuloso" dell'interno allargato. Il pannello B è stato creato utilizzando un tempo di divamperamento di 100 μs/pixel a 4 kV. L'aumento del tempo di dimora dei pixel aumenta il rapporto segnale/rumore e rivela un maggiore livello di dettaglio cellulare, tuttavia l'aumento del tempo di rigonfiamento dei pixel ha il potenziale per portare alla ricarica dei tessuti e / o all'accumulo di calore che ammorbidisce il blocco e introduce artefatti di taglio (chiacchiere) durante la sezionamento. I pannelli C e D confrontano le immagini catturate in condizioni di esposizione identiche ma con due diversi valori kV del fascio. Il tessuto in questi pannelli era impregnato di particelle nanogold tonica d'oro per rendere più evidenti le differenze nelle profondità di penetrazione del fascio. Il pannello C fu catturato a 9 kV mentre il pannello D fu catturato a 21 kV. L'aumento del kV ha il vantaggio di un maggiore contrasto (D), tuttavia i dettagli vengono persi a causa della raccolta di elettroni da una maggiore profondità di tessuto (C). Come risultato del campionamento di una sezione trasversale più ampia, un maggior numero di particelle immunogold specifiche per GAP 43 sono visibili mentre l'etichettatura non specifica rimane la stessa con conseguente aumento del rapporto segnale-rumore. Pannello A & B scale bar = 2 μm. Panel C & D scale bar = 1 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Intensità del fascio, kV e dimensione spot. (A) Al contatto con il campione di tessuto, il fascio di elettroni (azzurro) produce un volume di interazione a forma di goccia, dal quale vengono prodotte diverse forme di energia dall'interazione tra gli elettroni del fascio e il campione di tessuto. La forma a goccia è una funzione della densità dei tessuti e della colorazione dei metalli pesanti insieme all'energia del fascio e all'angolo di inclinazione del fascio dielettroni 43. Mentre i raggi X, gli elettroni di coclea e gli elettroni terziari sono prodotti durante l'imaging SBF-SEM, la preoccupazione principale è con elettroni retroscatterati (blu scuro) e secondari (verdi)13. L'immagine prodotta con l'imaging SBF-SEM è prodotta raccogliendo elettroni retroscatterati. Questi elettroni hanno origine da interazioni elastiche tra il fascio e il campione, e il segnale raccolto dipende fortemente dal numero atomico di atomi interagiti con - da qui la necessità di colorazione di metalli pesanti44. Gli elettroni secondari hanno origine da interazioni anelastiche tra il fascio e il campione e il rilevamento del loro segnale dipende fortemente dall'orientamento superficiale. Poiché la faccia del blocco è piatta in SBF-SEM, gli elettroni secondari non contribuiscono in modo significativo al segnale raccolto13. Infatti, l'accumulo secondario di elettroni sulla superficie del blocco può essere una delle principali fonti di ricarica e ha un effetto deleterio sulla qualità dell'immagine2. (B) Questo grafico mostra la relazione tra l'intensità del fascio, il kV del fascio e la dimensione dello spot. La dimensione dello spot è la risoluzione spaziale del fascio e determina il limite di risoluzione delle immagini prodotte. Abbassare kV aumenta la dimensione dello spot, ma diminuisce anche la profondità di imaging consentendo una migliore valutazione dei dettagli. Questo ha l'effetto di diminuire anche il segnale rilevabile. L'aumento dell'intensità del fascio offre un miglioramento iniziale delle dimensioni del punto e del rilevamento del segnale, ma aumenta rapidamente i livelli di ricarica dei tessuti. In definitiva, l'intensità del fascio e i valori kV scelti dipendono dal campione e sono meglio determinati empiricamente in relazione alla domanda scientifica posta. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Rete di fasci di microfibrille senza elastina nella cornea del mouse. ricostruzione 3D di microfibrille (bianche) strettamente associate ai cheratociti (giallo, arancione & verde) all'interno dello stroma corneale. Le microfibrille possono essere viste adiacenti e, in alcuni casi, all'interno di scanalature poco profonde, cheratociti corneali (frecce) (A). Questa rete di microfibrie senza elastina sono organizzate in strati distinti all'interno dello stroma corneale (B). Barra di scala = 2 μm. Il blocco di immagini ricostruito è di 45x45 μm nell'asse x & y e di 30 μm nell'asse z con voxel con una risoluzione di 22x22x100 nm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: Ricostruzione dei nervi corneali che passano attraverso lamina basale al confine stromale-epiteliale. Ricostruzione 3D di un nervo penetrante (viola) mentre passa attraverso la lamina basale (verde). Questo nervo può essere visto biforcare prima della penetrazione. Dopo essere penetrati nell'epitelio, entrambi i rami nervosi sono stati sottoposti a ramificazione. I mitocondri (giallo) sono visibili nelle porzioni stromali ed epiteliali del fascio nervoso. Barra di scala = 2,5 μm. Il blocco di immagini ricostruito è di 25x25 μm nell'asse x & y e di 14 μm nell'asse z con una risoluzione voxel di 12x12x100 nm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 7: Vascucolatura limbale e cellule associate nella cornea periferica del topo. Una singola immagine (A) da un blocco di immagini 3D (B) può essere vista attraverso la quale viaggiano un vaso, un fascio nervoso e le cellule associate. Il pannello C mostra un vaso ricostruito (rosso) con un pericita associato (grigio) avvolto intorno ad esso che copre le giunzioni cellulari endoteliali. Un fascio nervoso (blu) biforca nelle immediate vicinanze di questo vaso mentre viaggia attraverso il tessuto. Un neutrofilo (giallo) può essere visto parallelamente al lungo asse del vaso, con il suo nucleo polimorfico visibile all'interno del suo corpo cellulare e l'uropode finale visibile come sporgenza verso destra dell'immagine. Una mastociscosa (magenta) è visibile sul lato inferiore del vaso. Il pannello D isola questa mastocitta, dove i suoi granuli (verde) possono essere visti più facilmente sovrapponendo il nucleo (viola) all'interno della cellula. Il pannello E evidenzia le strutture cellulari sovrapposte alle ricostruzioni cellulari, con nuclei endoteliali denotati in blu e microparticelle aderenti visibili nel lume del vaso (arancione). Le frecce mostrano i bordi cellulare-cellulari tra le cellule endoteliali, che possono essere ulteriormente viste come creste sollevate che si estendono lungo le cellule sul lato luminare del vaso. Pannello Una barra di scala = 2 μm. Il blocco di immagini utilizzato per ricostruire queste cellule è 30x30 μm nell'asse x & y e 42,5 μm nell'asse z con una risoluzione voxel di 14,6x14,6x100 nm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 8: Rete vascolare ricostruita dello strato di fibre nervose retiniche primate non umane. (A) Un'immagine SBF-SEM 200x200 μm della retina del primate scattata a 8192x8192 px. La posizione campiozzata è di ~ 500 micron dal margine del bordo temporale inferiore di un occhio sano senza patologia. La serie di immagini ricostruita nei pannelli C & D è stata catturata a 2048x2048 px, con immagini sospese in modo che le regioni interessate potessero essere immagini a 8192x8192 px. Il pannello B è l'area intarsiata del pannello A, presa direttamente dall'immagine originale. Si noti il gran numero di assoni e i loro mitocondri. (C) Sezione di ortoslice attraverso un volume tissutale 200x200x200 μm di uno strato di fibra nervosa temporale inferiore dell'occhio di controllo, con vascucolatura segmentata. (D) Proiezione Z della vascucolatura dello strato di fibre nervose. Questa serie illustra la risoluzione possibile in un campo di grandi dimensioni utilizzando questa metodologia. Pannello Una barra di scala = 20 μm. Barra di scala panel B = 2 μm. La risoluzione voxel della serie di immagini è di 97,6x97,6x500 nm. La risoluzione dei pixel dell'area di interesse è di 24,4x24,4 nm. Clicca qui per visualizzare una versione più grande di questa figura.
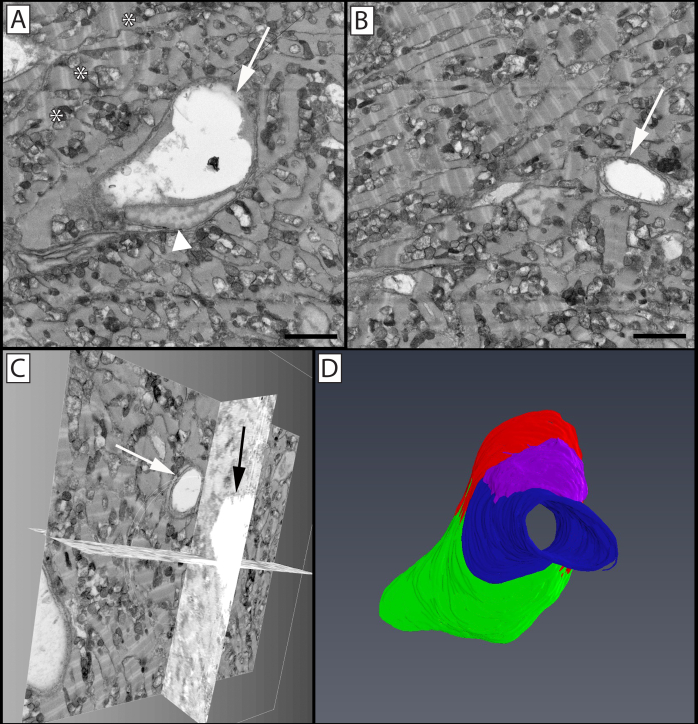
Figura 9: Segmentazione e rendering in volume 3D dei vasi nel cuore compatto del danio gigante (Devario malabaricus) (A) Micrografia bidimensionale in una pila di immagini, che mostra il profilo di un vaso centrale di dimensioni venulari (freccia) e di un nucleo endoteliale (punta di freccia), con miociti cardiaci circostanti ricchi di mitocondri e sarcomere ben organizzati (*). (B) Micrografia bidimensionale della pila di immagini con un recipiente di dimensioni capillari (freccia). (C) Proiezioni biorthogonali della pila di micrografi che mostrano il capillare nel pannello B proiettato attraverso una fetta ortogonale. (D) rendering 3D di cellule endoteliali segmentate che rivestono il recipiente ricostruito. Illustrate in verde, rosso, blu e viola sono quattro cellule endoteliali separate; la cella endoteliale raffigurata in blu può essere vista nella sezione trasversale nel pannello B (freccia), mentre le cellule endoteliali raffigurate in rosso (freccia) e verde (punta di freccia) sono viste nella sezione trasversale nel pannello A. Pannelli A & B barra di scala = 2 μm. Il blocco di immagini ricostruito è di 30x30 μm nell'asse x & y e di 16 μm nell'asse z con una risoluzione voxel di 14,6x14,6x100 nm. Clicca qui per visualizzare una versione più grande di questa figura.
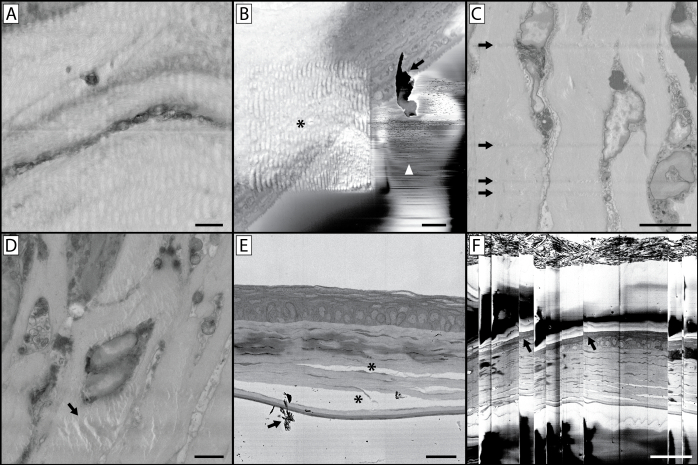
Figura 10: Complicazioni e artefatti dell'imaging. (A) La natura ondulata e distorta di questa immagine è il risultato dell'imaging utilizzando un tempo di divagazione dei pixel troppo lungo. Questo riscalda il blocco di resina, lasciando il blocco morbido e gommoso che si traduce in un'immagine distorta al taglio. (B) Questa immagine contiene una serie di manufatti. L'asterisco indica una distorsione ondulata causata dall'imaging precedente con un ingrandimento più elevato e simile al pannello A, concentrando il fascio su una regione più piccola con un tempo di dimora dei pixel più lungo ha ammorbidito la resina in questa regione di interesse. Mentre l'immagine di ingrandimento più elevata raccolta era priva di artefatti, ciò può portare a una serie successiva di immagini in cui il campione sottostante la regione di interesse appare distorto. Questo pannello illustra anche il problema dell'accumulo di detriti sulla faccia del blocco (freccia) durante l'imaging, indicato anche dalla freccia nel pannello E. Se questo diventa un problema di imaging persistente, sarà necessario rompere il vuoto, aprire la camera e soffiare via i detriti accumulati sul coltello diamanta e intorno al campione. Piccole scariche di elettroni dalla faccia del blocco possono portare ai rapidi cambiamenti di contrasto e alle linee denota dalla punta di freccia bianca. (C) Questa immagine illustra i graffi del coltello sulla faccia del blocco. Ciò può verificarsi a causa di un coltello danneggiato o dell'accumulo di detriti sul bordo del coltello. (D) Il manufatto indicato (freccia) è il risultato del fascio di elettroni focalizzato (senza sezionare) la faccia del blocco per un lungo periodo di tempo con il campione ancora nella camera di imaging. (E) Una fissazione impropria dei tessuti può portare alla separazione delle strutture cellulari e del tessuto connettivo (*). (F) Se si verifica una grande quantità di carica nel blocco di tessuto o resina, può verificarsi un successivo accumulo e scarico che porta l'immagine a "saltare" come si vede in questa immagine. Si noti la distorsione del tessuto nell'immagine in questi punti di salto (frecce). Pannello Una barra di scala = 1 μm. Barra di scala pannello B = 2 μm. Barra di scala del pannello C = 5 μm. Barra di scala pannello D = 2 μm. Barra di scala pannello E = 25 um. Barra di scala Pannello F = 50 um. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 11: Imaging del tessuto a 3 kV utilizzando vari tempi di abitare dei pixel e intensità del fascio. Tutte le immagini stavano raccogliendo utilizzando un fascio da 3 kV, l'intensità del fascio è su una scala specifica del dispositivo che va da 1 a 20. Il campo immaginato è del lume vascolare contenente globuli bianchi e rossi. A questo kV basso è difficile apprezzare i dettagli cellulari. Aumentare il tempo di rigonfiamento dei pixel ha avuto poco effetto. Aumentando l'intensità del fascio a 6 migliori contrasti dell'immagine. Clicca qui per visualizzare una versione più grande di questa figura.
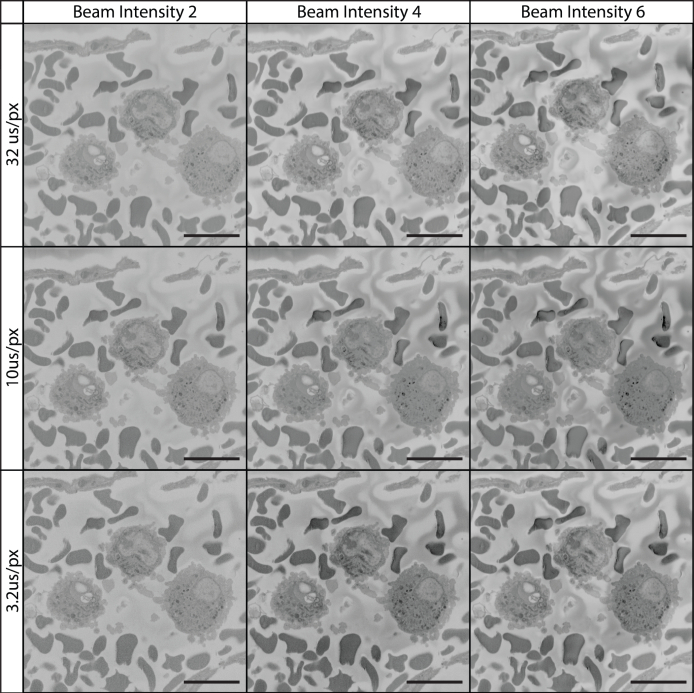
Figura 12: Imaging del tessuto a 7 kV utilizzando vari tempi di abitare dei pixel e intensità del fascio. Tutte le immagini sono state raccolte utilizzando un fascio da 7 kV, l'intensità del fascio è su una scala specifica del dispositivo che va da 1 a 20. Il campo immaginato è del lume vascolare contenente globuli bianchi e rossi. A 7 kV, l'aumento dell'intensità del fascio e del tempo di abitare dei pixel ha contribuito a un imaging di qualità superiore. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 13: Imaging del tessuto a 12 kV utilizzando vari tempi di abitare dei pixel e intensità del fascio. Tutte le immagini sono state raccolte utilizzando un fascio da 12 kV, l'intensità del fascio è su una scala specifica del dispositivo che va da 1 a 20. Il campo immaginato è del lume vascolare contenente globuli bianchi e rossi. A 12 kV, l'imaging è ottimizzato regolando il tempo di rigonfiamento dei pixel e l'intensità del fascio. La ricarica è ridotta/assente a tempi di rigonfiamento dei pixel più brevi, mentre i dettagli cellulari e il contrasto dell'immagine sono i migliori con un tempo di divaxamento dei pixel più lungo e un'intensità del fascio più elevata. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Lo scopo di questo documento sui metodi è quello di evidenziare la metodologia di preparazione e imaging dei tessuti che ha permesso al nostro laboratorio di catturare in modo affidabile immagini di microscopia elettronica seriale ad alta risoluzione e di evidenziare i passaggi critici che portano a questo risultato e potenziali insidie che possono verificarsi durante la conduzione dell'imaging SBF-SEM. Il successo nell'utilizzo di questo protocollo richiede una corretta fissazione dei tessuti, l'impregnazione di metalli pesanti nel campione, modifiche della resina incorporante per ridurre la carica, nonché una comprensione delle impostazioni del microscopio e dell'imaging utilizzate per raccogliere immagini. La massima, "qualità in, quality out" è un assioma appropriato per l'imaging SBF-SEM. Poiché l'obiettivo di SBF-SEM è spesso l'apprezzamento o la quantificazione dei dettagli ultrastrutturali, occorre prestare particolare attenzione alla strategia di fissazione al fine di garantire che la distorsione tissutale non si verifichi. Se il tessuto viene distorto in qualsiasi momento nella preparazione dei campioni (cioè subisce gonfiore, restringimento o interruzione della morfologia cellulare), la ricostruzione e la quantizzazione dei tessuti non produrranno dati accurati. Inoltre, l'uso di impostazioni di imaging errate può portare alla perdita di dati che non possono essere ricatturati poiché l'imaging SBF-SEM è un processo distruttivo. Inoltre, è necessario prestare attenzione quando si carica un campione di tessuto in quanto il delicato coltello diamanta può essere danneggiato da una preparazione del campione affrettata o errata. Ciò può causare schegge o rotture nel coltello, che possono lasciare segni di graffi visibili nelle immagini (Figura 10C). Il coltello diamanta può anche essere danneggiato da strutture calcificate, granuli duri o vetro incorporato accidentalmente (ad esempio, da ampule di reagente).
Mentre la maggior parte della letteratura SBF-SEM fino ad oggi utilizza tensioni di accelerazione del fascio nell'intervallo da 1 a 3 kV insieme a tempi di abitare in pixel più vicini a 1-5 μs / px (Figura 11)45,46,47,48,49, il protocollo corrente utilizza tensioni di accelerazione di 7-12 kV e un tempo di divaggio in pixel di 12 μs/px per l'imaging seriale e 32 μs/px per le regioni di imaging di interesse (Figure 12 & 13). Queste impostazioni, abbinate a uno spessore della fetta di 100-200 nm, consentono l'imaging di alta qualità e alta risoluzione di una vasta gamma di tessuti biologici. L'aumento della tensione di accelerazione consente un aumento del contrasto, della risoluzione e del rapporto segnale-rumore. L'aumento del tempo di dimora aumenta ulteriormente la risoluzione e il rapporto segnale-rumore, mentre l'aumento dello spessore della fetta porta a una riduzione della carica sulla superficie del blocco durante la sezionamento e combatte i danni indotti dal fascio nelle immagini successive14. Sebbene questo metodo di imaging possa differire dalla convenzione, le immagini e i set di dati prodotti parlano da soli. Se dovesse speculare sul motivo di questo successo, è possibile che sia il risultato della nostra combinazione unica di valori kV elevati, tempi di dimora dei pixel più lunghi e preparazione del blocco. L'aumento del kV di imaging si traduce in un aumento del volume di interazione tra il fascio di elettroni e il campione. Questo volume di interazione è sia più profondo che più ampio, con conseguente aumento teorico del numero di elettroni rilevati che provengono da più profondità all'interno del blocco campione, o da una sezione trasversale più ampia del tessuto man mano che la dimensione dello spot lacrima aumenta di diametro. Poiché SBF-SEM è interessato al dettaglio superficiale del blocco, ciò si traduce in una diminuzione teorica del rapporto segnale-rumore. Tuttavia, l'aumento del kV spinge anche gli elettroni più in profondità nel campione dove hanno meno probabilità di sfuggire al blocco e contribuire agli elettroni raccolti dal rivelatore. Con l'ulteriore vantaggio di un segnale maggiore attraverso tempi di divagazione dei pixel più lunghi e una maggiore intensità del fascio, è possibile che questo metodo di imaging si traduce in un maggiore aumento del segnale dalla superficie del campione in relazione al rumore originato all'interno del volume di interazione. Inoltre, l'aumento della conducibilità del campione introdotto con il rivestimento in nero fumo e argento e oro aiuta a migliorare l'accumulo di carica che ora si verifica più in profondità all'interno del blocco e più lontano dalla faccia del blocco. infatti Figura 11, Figura 12, Figura 13 mostrano che con l'aumento della carica del campione kV inizia a diminuire in quanto è potenzialmente spinto più in profondità nel blocco. I campioni con un basso ingrandimento possono essere acquisiti con un contrasto adeguato utilizzando le impostazioni convenzionali, tuttavia queste immagini spesso mancano di dettagli al momento di un'ispezione ravvicinata. I nostri dati mostrano chiaramente che quando si utilizza un ingrandimento relativamente elevato in cui l'obiettivo è il dettaglio cellulare, l'aumento delle impostazioni convenzionali può produrre risultati eccezionali. L'articolo 2020 di P. Goggin, et al fornisce una tabella utile che delinea l'effetto della modifica delle impostazioni di imaging sulla qualità finale dell'immagine ed è un utile riferimento da consultare se l'ottimizzazione del protocollo per i nuovi tessuti diventa necessaria14. Lo spessore della fetta di 100-200 nm consigliato in questo protocollo ha l'ulteriore vantaggio di consentire la raccolta di set di dati SBF-SEM di grandi dimensioni a una velocità rapida. Durante la raccolta di immagini a 12μs/px, ad esempio, l'imaging attraverso una profondità di 100 μm a 2048x2048 px richiede ~ 14 ore durante la sezione a 100nm / sezione, ma richiederebbe ~ 56 ore sessato a 25nm / sezione. Mentre la risoluzione x,y rimane invariata a causa dello spessore della sezione, non tenendo conto della maggiore capacità di immagine utilizzando valori kV più elevati e tempi di divagazione dei pixel che vengono con sezioni più grandi, è importante notare che la risoluzione lungo l'asse z ne risente. La perdita della risoluzione z è una considerazione importante e dovrebbe essere contemplata quando si decide come il tessuto dovrebbe essere orientato nel blocco di resina e in relazione al piano di imaging, e ha il potenziale per precludere lo studio di caratteristiche o interazioni cellulari più piccole (ad esempio, invaginazioni sinaptiche o caratteristiche intracellulari sulla scala di decine di nanometri). Tuttavia, oltre al tempo di imaging rapido, questo protocollo ha ulteriori vantaggi aggiuntivi in quanto produce rapidamente set di dati ideali per l'analisi stereologica e lo studio di eventi biologici rari o cellule. Uno spessore della sezione più grande può anche aiutare nella ricostruzione 3D manuale, poiché una regione di 100 μm se sesata a 100 nm / sezione richiederebbe la segmentazione manuale di 1.000 immagini, mentre la stessa regione sessata a 25 nm / sezione richiederebbe la segmentazione manuale di 4.000 immagini.
SBF-SEM ha il vantaggio di generare set di dati di grandi dimensioni in un periodo relativamente breve. Mentre l'analisi dei dati può essere eseguita utilizzando metodi quantitativi come la stereologia, che verrà discussa di seguito, spesso può essere informativa creare ricostruzioni 3D tramite segmentazione dell'immagine. Uno stack di immagini creato utilizzando SBF-SEM può essere pensato come una raccolta di voxel, mentre la segmentazione è il processo di assegnazione di questi voxel a oggetti definiti dall'utente creando così rappresentazioni 3D di strutture tissutali. Queste ricostruzioni forniscono spesso una prospettiva non vista sull'ultrastruttura dei tessuti e sull'interazione cellula-cellula(Figura 5, Figura 6, Figura 7, Figura 8, Figura 9). Inoltre, una volta create le ricostruzioni è possibile utilizzare i dati inerenti alle ricostruzioni per estrarre una ricchezza di informazioni dal tessuto segmentato. Parametri che vanno dalla superficie, al volume, alla lunghezza e alla distanza, nonché i dati angolari sono tutti prontamente disponibili una volta creata unaricostruzione 50,51. Sebbene ciò possa essere incredibilmente utile, soprattutto se associato a video e immagini estratti da set di dati ricostruiti, il tempo necessario per la segmentazione manuale è una considerazione importante quando si tenta di estrapolare i dati dai set di dati SBF-SEM. Attualmente sono disponibili una serie di software gratuiti e acquistabili per la segmentazione manuale e semi-manuale degli stack di immagini SBF-SEM. Un'opzione gratuita per il software di ricostruzione è il pacchetto di elaborazione delle immagini Fiji per ImageJ, un programma di elaborazione delle immagini open source, che contiene un plug-in dell'editor di segmentazione che consente lasegmentazione manuale 52,53. Inoltre, il software Reconstruct offre un'opzione alternativa di segmentazionegratuita 54 (Figura 8). Sebbene potenzialmente costose, le opzioni acquistabili spesso contengono set di funzionalità più robusti, come processi di segmentazione semi-automatizzati o suite di creazione di film e immagini. Una di queste opzioni è stata utilizzata per creare le ricostruzioni disponibili nella figura 5, figura 6, figura 7 e figura 9 (Dettagli disponibili nella tabella dei materiali). Inoltre, sono disponibili strumenti per la creazione, l'analisi e il rendering di ricostruzioni 3D basate sul contrasto utilizzando la realtà virtuale con il potenziale di accelerare notevolmente il processo diricostruzione 20,55. Sebbene non sempre disponibili per tutte le applicazioni, sono disponibili una serie di strumenti software per la segmentazione manuale assistita da computer che hanno il potenziale di ridurre notevolmente il tempo necessario per lasegmentazione 56,57,58. Indipendentemente dal software utilizzato, una notevole attenzione e una comprensione della domanda a cui viene data risposta, o una lacuna nelle conoscenze da colmare, dalle ricostruzioni seriali dovrebbero precedere la segmentazione, poiché il processo è laborioso e richiede molto tempo.
La produzione di ricostruzioni 3D ha le sue considerazioni. Con set di dati più grandi la potenza di elaborazione può essere un fattore limitante, quindi l'ottimizzazione dell'uso delle risorse di sistema può essere fondamentale per mantenere un flusso di lavoro produttivo e accelerare il processo di ricostruzione e rendering. Quando si esegue il rendering di una ricostruzione 3D, la maggior parte del software converte stack di immagini segmentate in una superficie composta da triangoli interconnessi. Se un progetto di ricostruzione è grande o intricato, il rendering di questi triangoli può richiedere una grande quantità di potenza di calcolo. Mentre si lavora a una ricostruzione 3D, può essere utile limitare il numero di triangoli che il software di ricostruzione può utilizzare per convertire le immagini segmentate in superfici ricostruite. Questo può essere utile per monitorare lo stato di avanzamento di una ricostruzione 3D durante il processo di segmentazione. Una volta completata la segmentazione, il limite del triangolo può essere rimosso prima di eseguire il rendering di immagini o video di ricostruzioni. In alternativa, e se il software di ricostruzione lo consente, abbiamo trovato successo nel monitorare l'avanzamento di una ricostruzione utilizzando il rendering del volume piuttosto che la generazione di superficie. Il rendering del volume, sebbene non sia adatto per immagini o video destinati alla pubblicazione o alla presentazione, richiede una potenza di elaborazione molto inferiore e come tale può essere utile per fornire un'esperienza fluida nella ricostruzione e nella preparazione di immagini e video di ricostruzioni. Inoltre, è buona pratica segmentare manualmente un set di dati SBF-SEM per definire ogni oggetto da ricostruire con il proprio identificatore univoco. Se un campo di cellule epiteliali viene ricostruito, ad esempio, piuttosto che assegnare tutte le cellule epiteliali a un gruppo voxel intitolato "epitelio", a ogni cellula epiteliale dovrebbe essere assegnato il proprio moniker (cioè Epi1, Epi2, Epi3, ecc.). Ciò offre una maggiore libertà quando la ricostruzione è completa, in quanto ogni cella può essere inclusa o esclusa dal rendering finale, assegnato a colori o trasparenze diversi o rimosso o introdotto singolarmente se viene prodotto un video. Inoltre, ciò consente di raccogliere metriche come l'area di superficie o il volume da ogni oggetto ricostruito anziché dal gruppo di oggetti nel suo complesso.
Un altro strumento incredibilmente potente per estrarre dati quantitativi dagli stack di immagini SBF-SEM è la pratica della stereologia. La stereologia sfrutta le relazioni matematiche intrinseche tra gli oggetti tridimensionali e le loro rappresentazioni bidimensionali (cioè le micrografie di elettroni). I set di dati SBF-SEM sono ideali per l'applicazione di stereologia, in quanto questo metodo per estrarre informazioni 3D da set di dati di grandi dimensioni richiede molto meno tempo e manodopera rispetto alla ricostruzione segmentata. La stereologia consiste generalmente nell'applicare griglie geometriche a immagini casuali e campionato uniformemente ed è stata ampiamente utilizzata negli ultimi 50 anni al fine di produrre stime accurate e imparziali del numero di cellule / organelli, lunghezza, superficie e volume21,59,60,61,62,63. Mentre le ricostruzioni 3D possono essere impressionanti e fornire una nuova prospettiva sui tessuti biologici, è spesso più veloce, più accurato, riproducibile e favorevole per campioni di grandi dimensioni utilizzare un approccio stereologico all'estrazione dei dati. Mentre ci sono molti articoli che discutono dell'applicazione pratica della stereologia64,65,66, un certo numero di libri di testo forniscono utili e approfondite panoramiche della metodologia e forniscono una serie di griglie stereologiche che possono essere applicate allo studio dell'ultrastrutturatissutale 67,68,69.
SBF-SEM è un potente metodo di imaging che consente l'apprezzamento tridimensionale dell'ultrastruttura tissutale. Mentre la capacità di creare set di dati 3D con risoluzione SEM mette domande precedentemente insopportabili alla nostra portata, una corretta preparazione dei tessuti e una comprensione dell'imaging SBF-SEM sono fondamentali per il successo degli studi che utilizzano questo metodo di microscopia. Speriamo che l'applicazione di questo protocollo agli studi futuri porti ad una visione sempre più approfondita dei misteri biologici che ci circondano e continui a spingerci più lontano nelle frontiere della conoscenza umana.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Vorremmo ringraziare il Dr. Sam Hanlon, Evelyn Brown e Margaret Gondo per la loro eccellente assistenza tecnica. Questa ricerca è stata sostenuta in parte dai National Institutes of Health (NIH) R01 EY-018239 e P30 EY007551 (National Eye Institute), in parte dalla Lion's Foundation for Sight, e in parte da NIH 1R15 HD084262-01 (National Institute of Child Health & Human Development).
Materiali
| Name | Company | Catalog Number | Comments |
| 1/16 x 3/8 Aluminum Rivets | Industrial Rivet & Fastener Co. | 6N37RFLAP/1100 | Used as specimen pins. |
| 2.5mm Flathead Screwdriver | Wiha Quality Tools | 27225 | |
| Acetone | Electron Microscopy Sciences | RT 10000 | Used to dilute silver paint. |
| Aspartic Acid | Sigma-Aldrich | A8949 | |
| Calcium Chloride | FisherScientific | C79-500 | |
| Conductive Silver Paint | Ted Pella | 16062 | |
| Denton Desk-II Vacuum Sputtering Device equipped with standard gold foil target | Denton Vacuum | N/A | This is the gold-sputtering device used by the authors, alternates are acceptable. |
| Double-edged Razors | Fisher Scientific | 50-949-411 | |
| Embed 812 | Electron Microscopy Sciences | 14120 | |
| Gatan 3View2 mounted in a Tescan Mira3 Field emission SEM | Gatan & Tescan | N/A | This is the SBF-SEM device used by the authors, alternates are acceptable. |
| Glass Shell Vials, 0.5 DRAM (1.8 ml) | Electron Microscopy Sciences | 72630-05 | |
| Gluteraldehyde | Electron Microscopy Sciences | 16320 | |
| Gorilla Super Glue - Impact Tough | NA | NA | Refered to as cyanoacrylate glue in text. |
| Ketjen Black | HM Royal | EC-600JD | Refered to as carbon black in text. |
| KOH | FisherScientific | 18-605-593 | |
| Lead Nitrate | Fisher Scientific | L62-100 | |
| Microwave | Pelco | BioWave Pro | This is the microwave used by the authors, alternates are acceptable. |
| Osmium Tetroxide | Sigma-Aldrich | 201030 | |
| Potassium Ferrocyanide | Sigma-Aldrich | P9387 | |
| Silicone Embedding Mold | Ted Pella | 10504 | |
| Sodium Cacodylate Trihydrate | Electron Microscopy Sciences | 12300 | |
| Samco Transfer Pipette | ThermoFisher Scientific | 202 | Used to make specimen pin storage tubes. |
| Swiss Pattern Needle Files | Electron Microscopy Sciences | 62115 | |
| Thiocarbohydrazide | Sigma-Aldrich | 223220 | |
| Uranyl Acetate | Polysciences, Inc. | 21447-25 | |
| Reconstruction Software | |||
| Amira Software | Thermo Scientific | N/A | Used to create the reconstructions found in figures 5-7 and 9. |
| Fiji (Fiji is Just ImageJ) | ImageJ.net | N/A | TrakEM2 can be added to Fiji to asist in manual segmentation. |
| Microscopy Image Browser (MIB) | University of Helsinki, Institute of Biotechnology | N/A | |
| Reconstuct Software | Neural Systems Lab | N/A | |
| SuRVoS Workbench | Diamond Light Source & The University of Nottingham | N/A | |
| SyGlass | IstoVisio, Inc. | N/A | Allows for reconstruction in virtual reality and histogram-based reconstruction methods. |
Riferimenti
- Leighton, S. B. SEM images of block faces, cut by a miniature microtome within the SEM - a technical note. Scanning Electron Microscopy. , Pt 2 73-76 (1981).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLOS Biology. 2 (11), 329(2004).
- He, Q., Hsueh, M., Zhang, G., Joy, D. C., Leapman, R. D. Biological serial block face scanning electron microscopy at improved z-resolution based on Monte Carlo model. Scientific Reports. 8 (1), 12985(2018).
- Zankel, A., Wagner, J., Poelt, P. Serial sectioning methods for 3D investigations in materials science. Micron. 62, 66-78 (2014).
- Titze, B., Genoud, C. Volume scanning electron microscopy for imaging biological ultrastructure. Biology of the Cell. 108 (11), 307-323 (2016).
- Ohta, K., et al. Beam deceleration for block-face scanning electron microscopy of embedded biological tissue. Micron. 43 (5), 612-620 (2012).
- Bouwer, J. C., et al. Deceleration of probe beam by stage bias potential improves resolution of serial block-face scanning electron microscopic images. Advanced Structural and Chemical Imaging. 2 (1), 11(2017).
- Kizilyaprak, C., Longo, G., Daraspe, J., Humbel, B. M. Investigation of resins suitable for the preparation of biological sample for 3-D electron microscopy. Journal of Structural Biology. 189 (2), 135-146 (2015).
- Kittelmann, M., Hawes, C., Hughes, L. Serial block face scanning electron microscopy and the reconstruction of plant cell membrane systems. Journal of Microscopy. 263 (2), 200-211 (2016).
- Biazik, J., Vihinen, H., Anwar, T., Jokitalo, E., Eskelinen, E. L. The versatile electron microscope: an ultrastructural overview of autophagy. Methods. 75, 44-53 (2015).
- Peddie, C. J., Collinson, L. M. Exploring the third dimension: Volume electron microscopy comes of age. Micron. 61, 9-19 (2014).
- Piňos, J., Mikmeková, Š, Frank, L. About the information depth of backscattered electron imaging. Journal of Microscopy. 266 (3), 335-342 (2017).
- Smith, D., Starborg, T. Serial block face scanning electron microscopy in cell biology: Applications and technology. Tissue Cell. 57, 111-122 (2019).
- Goggin, P., et al. Development of protocols for the first serial block-face scanning electron microscopy (SBF SEM) studies of bone tissue. Bone. 131, 115107(2020).
- Deerinck, T. J., et al. High-performance serial block-face SEM of nonconductive biological samples enabled by focal gas injection-based charge compensation. Journal of Microscopy. 270 (2), 142-149 (2018).
- Nguyen, H. B., et al. Conductive resins improve charging and resolution of acquired images in electron microscopic volume imaging. Scientific Reports. 6, 23721(2016).
- Deerinck, T. J., Bushong, E. A., Thor, A., Ellisman, M. H. NCMIR methods for 3D EM: a new protocol for preparation of biological specimens for serial block face scanning electron microscopy. National Center for Microscopy and Imaging Research. 6 (8), (2010).
- Deerinck, T. J., et al. Enhancing Serial Block-Face Scanning Electron Microscopy to Enable High Resolution 3-D Nanohistology of Cells and Tissues. Microscopy and Microanalysis. 16, 1138-1139 (2010).
- Kubota, Y. New developments in electron microscopy for serial image acquisition of neuronal profiles. Microscopy (Oxf). 64 (1), 27-36 (2015).
- Courson, J. A., et al. Serial block-face scanning electron microscopy: A provocative technique to define 3-dimensional ultrastructure of microvascular thrombosis. Thrombosis Research. 196, 519-522 (2020).
- Courson, J. A., et al. Serial block-face scanning electron microscopy reveals neuronal-epithelial cell fusion in the mouse cornea. PLoS One. 14 (11), 0224434(2019).
- Lafontant, P. J., et al. Cardiac Myocyte Diversity and a Fibroblast Network in the Junctional Region of the Zebrafish Heart Revealed by Transmission and Serial Block-Face Scanning Electron Microscopy. PLoS One. 8 (8), 72388(2013).
- Hanlon, S. D., Behzad, A. R., Sakai, L. Y., Burns, A. R. Corneal stroma microfibrils. Experimental Eye Research. 132, 198-207 (2015).
- Gage, G. J., Kipke, D. R., Shain, W. Whole animal perfusion fixation for rodents. Journal of Visualized Experiments. (65), e3564(2012).
- Davenport, A. T., Grant, K. A., Szeliga, K. T., Friedman, D. P., Daunais, J. B. Standardized method for the harvest of nonhuman primate tissue optimized for multiple modes of analyses. Cell Tissue Bank. 15 (1), 99-110 (2014).
- Schuster, A., et al. An isolated perfused pig heart model for the development, validation and translation of novel cardiovascular magnetic resonance techniques. Journal of Cardiovascular Magnetic Resonance. 12 (1), 53(2010).
- Hanlon, S. D., Patel, N. B., Burns, A. R. Assessment of postnatal corneal development in the C57BL/6 mouse using spectral domain optical coherence tomography and microwave-assisted histology. Experimental Eye Research. 93 (4), 363-370 (2011).
- Longiéras, N., Sebban, M., Palmas, P., Rivaton, A., Gardette, J. L. Multiscale approach to investigate the radiochemical degradation of epoxy resins under high-energy electron-beam irradiation. Journal of Polymer Science Part A: Polymer Chemistry. 44 (2), 865-887 (2006).
- Hashimoto, T., Thompson, G. E., Zhou, X., Withers, P. J. 3D imaging by serial block face scanning electron microscopy for materials science using ultramicrotomy. Ultramicroscopy. 163, 6-18 (2016).
- Rouquette, J., et al. Revealing the high-resolution three-dimensional network of chromatin and interchromatin space: A novel electron-microscopic approach to reconstructing nuclear architecture. Chromosome Research. 17 (6), 801(2009).
- Briggman, K. L., Helmstaedter, M., Denk, W. Wiring specificity in the direction-selectivity circuit of the retina. Nature. 471 (7337), 183-188 (2011).
- Katoh, K. Microwave-Assisted Tissue Preparation for Rapid Fixation, Decalcification, Antigen Retrieval, Cryosectioning, and Immunostaining. International Journal of Biochemistry & Cell Biology. 2016, 7076910(2016).
- Login, G. R., Dvorak, A. M. A review of rapid microwave fixation technology: its expanding niche in morphologic studies. Scanning. 15 (2), 58-66 (1993).
- Jamur, M. C., Faraco, C. D., Lunardi, L. O., Siraganian, R. P., Oliver, C. Microwave fixation improves antigenicity of glutaraldehyde-sensitive antigens while preserving ultrastructural detail. Journal of Histochemistry and Cytochemistry. 43 (3), 307-311 (1995).
- Leong, A. S., Sormunen, R. T. Microwave procedures for electron microscopy and resin-embedded sections. Micron. 29 (5), 397-409 (1998).
- Willingham, M. C., Rutherford, A. V. The use of osmium-thiocarbohydrazide-osmium (OTO) and ferrocyanide-reduced osmium methods to enhance membrane contrast and preservation in cultured cells. Journal of Histochemistry and Cytochemistry. 32 (4), 455-460 (1984).
- Khan, A. A., Riemersma, J. C., Booij, H. L. The reactions of osmium tetroxide with lipids and other compounds. Journal of Histochemistry and Cytochemistry. 9, 560-563 (1961).
- Belazi, D., Solé-Domènech, S., Johansson, B., Schalling, M., Sjövall, P. Chemical analysis of osmium tetroxide staining in adipose tissue using imaging ToF-SIMS. Histochemistry and Cell Biology. 132 (1), 105-115 (2009).
- Rivlin, P. K., Raymond, P. A. Use of osmium tetroxide-potassium ferricyanide in reconstructing cells from serial ultrathin sections. Journal of Neuroscience Methods. 20 (1), 23-33 (1987).
- Aguas, A. P. The use of osmium tetroxide-potassium ferrocyanide as an extracellular tracer in electron microscopy. Stain Technology. 57 (2), 69-73 (1982).
- Seligman, A. M., Wasserkrug, H. L., Hanker, J. S. A new staining method (OTO) for enhancing contrast of lipid--containing membranes and droplets in osmium tetroxide--fixed tissue with osmiophilic thiocarbohydrazide(TCH). Journal of Cell Biology. 30 (2), 424-432 (1966).
- Watson, M. L. Staining of tissue sections for electron microscopy with heavy metals. II. Application of solutions containing lead and barium. Journal of Biophysical and Biochemical Cytology. 4 (6), 727-730 (1958).
- Zhou, W., Apkarian, R., Wang, Z., Joy, D. Fundamentals of Scanning Electron Microscopy. Scanning Microscopy in Nanotechnology. , 1-40 (2006).
- Tapia, J. C., et al. High-contrast en bloc staining of neuronal tissue for field emission scanning electron microscopy. Nature Protocols. 7 (2), 193-206 (2012).
- Buchacker, T., et al. Assessment of the Alveolar Capillary Network in the Postnatal Mouse Lung in 3D Using Serial Block-Face Scanning Electron Microscopy. Frontiers in Physiology. 10, 1357(2019).
- Keeling, E., et al. 3D-Reconstructed Retinal Pigment Epithelial Cells Provide Insights into the Anatomy of the Outer Retina. International Journal of Molecular Sciences. 21 (21), (2020).
- Shang, P., et al. Chronic Alcohol Exposure Induces Aberrant Mitochondrial Morphology and Inhibits Respiratory Capacity in the Medial Prefrontal Cortex of Mice. Frontiers in Neuroscience. 14, 561173(2020).
- Pfeifer, C. R., et al. Quantitative analysis of mouse pancreatic islet architecture by serial block-face SEM. Journal of Structural Biology. 189 (1), 44-52 (2015).
- Wilke, S. A., et al. Deconstructing complexity: serial block-face electron microscopic analysis of the hippocampal mossy fiber synapse. Journal of Neuroscience. 33 (2), 507-522 (2013).
- Cocks, E., Taggart, M., Rind, F. C., White, K. A guide to analysis and reconstruction of serial block face scanning electron microscopy data. Journal of Microscopy. 270 (2), 217-234 (2018).
- Borrett, S., Hughes, L. Reporting methods for processing and analysis of data from serial block face scanning electron microscopy. Journal of Microscopy. 263 (1), 3-9 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529(2017).
- Fiala, J. C. Reconstruct: a free editor for serial section microscopy. Journal of Microscopy. 218, Pt 1 52-61 (2005).
- Pidhorskyi, S., Morehead, M., Jones, Q., Spirou, G., Doretto, G. syGlass: interactive exploration of multidimensional images using virtual reality Head-mounted displays. arXiv . , arXiv:1804.08197 (2018).
- Cardona, A., et al. TrakEM2 software for neural circuit reconstruction. PLoS One. 7 (6), 38011(2012).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy Image Browser: A Platform for Segmentation and Analysis of Multidimensional Datasets. PLOS Biology. 14 (1), 1002340(2016).
- Luengo, I., et al. SuRVoS: Super-Region Volume Segmentation workbench. Journal of Structural Biology. 198 (1), 43-53 (2017).
- Anderson, H. R., Stitt, A. W., Gardiner, T. A., Archer, D. B. Estimation of the surface area and volume of the retinal capillary basement membrane using the stereologic method of vertical sections. Analytical & Quantitative Cytology & Histology. 16 (4), 253-260 (1994).
- Gibbons, C. H., Illigens, B. M., Wang, N., Freeman, R. Quantification of sweat gland innervation: a clinical-pathologic correlation. Neurology. 72 (17), 1479-1486 (2009).
- Knust, J., Ochs, M., Gundersen, H. J., Nyengaard, J. R. Stereological estimates of alveolar number and size and capillary length and surface area in mice lungs. Anat Rec. 292 (1), Hoboken. 113-122 (2009).
- Mahon, G. J., et al. Chloroquine causes lysosomal dysfunction in neural retina and RPE: implications for retinopathy. Current Eye Research. 28 (4), 277-284 (2004).
- Michel, R. P., Cruz-Orive, L. M. Application of the Cavalieri principle and vertical sections method to lung: estimation of volume and pleural surface area. Journal of Microscopy. 150, Pt 2 117-136 (1988).
- Weibel, E. R. Stereological methods in cell biology: where are we--where are we going. Journal of Histochemistry and Cytochemistry. 29 (9), 1043-1052 (1981).
- Schmitz, C., Hof, P. R. Design-based stereology in neuroscience. Neuroscience. 130 (4), 813-831 (2005).
- Kristiansen, S. L., Nyengaard, J. R. Digital stereology in neuropathology. Apmis. 120 (4), 327-340 (2012).
- Howard, C. V., Reed, M. G. Unbiased Stereology. 2nd edn. , Garland Science/BIOS Scientific Publishers. (2005).
- Reith, A., Mayhew, T. M. Stereology and Morphometry in Electron Microscopy: Problems and Solutions. , Hemisphere Publishing Corporation. (1988).
- Mouton, P. R. Principles and practices of unbiased stereology: an introduction for bioscientists. , Johns Hopkins University Press. (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.