Method Article
Identificazione dei marcatori di superficie cellulare delle cellule staminali primarie e delle cellule progenitrici mediante l'etichettatura metabolica di Sialoglycan
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Presentato qui è un protocollo che combina un sistema di cocoltura neurale-endoteliale in vitro e l'incorporazione metabolica di sialoglycan con gruppi funzionali bioortogonali per espandere le cellule staminali primarie e progenitrici ed etichettare la loro superficie sialoglycoproteins per l'imaging o l'analisi della spettrometria di massa dei marcatori di superficie cellulare.
Abstract
Le cellule staminali neurali e progenitrici (NPG) sono la base cellulare per le strutture complesse e le funzioni del cervello. Si trovano in nicchie specializzate in vivo e possono essere isolati ed espansi in vitro,fungendo da risorsa importante per il trapianto di cellule per riparare i danni cerebrali. Tuttavia, gli NSPC sono eterogenei e non chiaramente definiti a livello molecolare o purificati a causa della mancanza di specifici marcatori di superficie cellulare. Il protocollo presentato, precedentemente riportato, combina un sistema di cocoltura neurale-endoteliale con un metodo di etichettatura glicana metabolica per identificare il sialoglycoproteooma superficiale dei NSPC primari. Il sistema di cocoltura nPC-endoteliale consente l'auto-rinnovamento e l'espansione dei NSPC primari in vitro,generando un numero sufficiente di NSPC. gruppi funzionali bioortogonali. Confrontando il sialoglybioteome da NPG auto-rinnovamento espanso in una co-coltura endoteliale con coltura neurale differenziante, identifichiamo un elenco di proteine di membrana che sono arricchite in NPC. Nel dettaglio, il protocollo prevede: 1) l'impostazione di una cocultura NSPC-endoteliale e la cultura differenziante NSPC; 2) etichettatura con azidosugar per-O-acetylated N-azidoacetylmannosamine (Ac4ManNAz); e 3) coniugazione di biotina a sialoglycan modificato per l'imaging dopo la fissazione della coltura neurale o l'estrazione di proteine dalla coltura neurale per l'analisi della spettrometria di massa. Quindi, i candidati marcatori di superficie arricchiti nsPC vengono selezionati mediante analisi comparativa dei dati della spettrometria di massa provenienti sia dal NSPC espanso che dalle colture neurali differenziate. Questo protocollo è altamente sensibile per identificare le proteine della membrana di scarsa abbondanza nei materiali di partenza e può essere applicato alla scoperta di marcatori in altri sistemi con modifiche appropriate
Introduzione
Le cellule staminali neurali sono definite come una popolazione cellulare multipotente che può auto-rinnovarsi per mantenere una piscina di cellule staminali e differenziarsi in neuroni e glia. Sono i principali tipi di cellule nel sistema nervoso e possono offrire un grande potenziale terapeutico nella medicina rigenerativa attraverso il trapianto di cellule in cervelli malati e feriti1,2. Come lo sviluppo procede, la popolazione di cellule staminali neurali diventa eterogenea3,4, e la proporzione di cellule staminali neurali nel cervello diminuisce gradualmente5. In generale, le cellule staminali neurali embrionali e altre cellule progenitrici neurali, collettivamente chiamate cellule staminali neurali e progenitrici (NPG), si trovano nelle zone germinali, nella zona ventricolare e nella zona subventricolare nei topi6. Nel cervello embrionale, le cellule staminali neurali generano neuroni direttamente o indirettamente attraverso cellule progenitrici intermedie (IPC), e in alcune specie attraverso la zona subventricolare esterna progenitori (oRG)7,8. La firma molecolare specifica, la morfologia, la posizione nella nicchia delle cellule staminali e il potenziale di differenziazione determinano il ruolo di ciascun sottotipo nell'organogenesi cerebrale e nelle applicazioni cliniche9. Tuttavia, i marcatori di superficie cellulare attualmente disponibili non possono inequivocabilmente discriminare e purificare diversi sottotipi di NSPC, limitando la comprensione e l'utilizzo di questi sottotipi.
L'identificazione dei marcatori di superficie primari dei NSPC è limitata da tre ostacoli principali. Il primo è il numero limitato di nSPC nel tessuto, rendendo difficile preparare campioni di proteine della superficie cellulare per l'analisi comune della spettrometria di massa. La seconda limitazione è la difficoltà di produrre sottotipi di cellule pure per la generazione di dati proteici di membrana specifici del sottotipo. Infine, la terza sfida è il basso rapporto delle proteine della superficie cellulare nelle proteine intere cellule, che ostacola la loro sensibilità di rilevamento mediante l'analisi della spettrometria di massa.
Per superare questi problemi, abbiamo sviluppato un approccio chemiopromico per arricchire e identificare selettivamente le proteine della superficie cellulare nelle NSPC primarie etichettando metabolicamente le sialoglycoproteine10. Per generare un numero sufficiente di NSPC, abbiamo approfittato di un protocollo stabilito per espandere e mantenere gli NSPC embrionali primari in stati indifferenziati in vitro, co-culcolando gli NSPC con linee cellulari endoteliali del cervello del topo utilizzando un supporto permeabile matrice di inserimento (ad esempio, transwell) sistema11. Al contrario, i PNG coltivati da soli senza cellule endoteliali generano progenie differenziata11,12. Pertanto, i campioni di proteine di questi due sistemi di coltura possono essere analizzati comparativamente per identificare le proteine che sono espresse in modo differenziale negli NPC e nei neuroni differenziati. Poiché la maggior parte delle proteine della superficie cellulare sono modificate dall'acido silico13, precursore acido silico innaturale analogico N-azidoacetylmannosamine-tetraacilato (Ac4ManNAz) è stato utilizzato per dirottare la via metabolica intrinseca in modo che endogeno, di recente i sialoglycan sitolizzati sono etichettati con gruppi di azido, generando una maniglia chimica14. Attraverso reazioni bioortogonali mediate azido-alkyne, che coniugano biotina ai sialoglycan, le proteine della superficie cellulare possono essere visualizzate e arricchite per l'identificazione proteomica attraverso un fluoroforo o una matrice accoppiata astampata da streptavidinoo matrice 14.
Qui, eseguiamo la colorazione dell'analisi del gel SDS-PAGE della superficie sialoglycoproteome da NSPC espansa in una co-coltura endoteliale e differenziando le cellule in un sistema non co-culturale. Purifichiamo anche selettivamente il sialoglycoproteome superficiale nei due sistemi di coltura per il confronto proteomico. Il nostro protocollo, rispetto ai tradizionali protocolli di purificazione della superficie cellulare basati sulla centrismola15, aumenta l'efficacia dell'estrazione delle proteine superficiali attraverso specifiche coniugazioni e affinità di tag Purificazione. Nel frattempo, aumenta la purezza di estrazione delle proteine della superficie cellulare sulla base della premessa che la verifica avviene principalmente alle proteine della superficie cellulare. Sebbene i fattori endoteliali non possano bloccare completamente la differenziazione degli NSPC espansi, lo studio comparativo tra una co-coltura e una cultura differenziata fornisce un metodo conveniente per individuare le proteine superficiali arricchite con cellule staminali senza la necessità di analizzare le proteine dai PNG purificate dal FACS16. Riteniamo che questo approccio possa essere applicato agli studi sulle proteine di superficie in altri sistemi con le modifiche appropriate.
Protocollo
Tutti i protocolli animali utilizzati in questo studio sono stati approvati dall'IACUC (Institutional Animal Care and Use Committee) dell'Università di Tsinghua ed eseguiti in conformità con le linee guida dell'IACUC. La struttura animale di laboratorio presso l'Università di Tsinghua è stata accreditata dall'AAALAC (Associazione per la valutazione e l'accreditamento di Laboratory Animal Care International). Per la messa in scena degli embrioni, la metà del giorno del tappo vaginale identificato è stata calcolata come giorno embrionale 0,5 (E0.5).
NOT: Tutte le cellule sono coltivate nell'incubatrice cellulare in condizioni di 37 e 5% di CO2.
1. Preparazione della cultura endoteliale del topo in inserti di supporto permeabile
NOT: Le celle BEND3 sono mantenute secondo le istruzioni del produttore.
- Preparare il supporto cellulare BEND3 (BM) aggiungendo 50 mL di FBS e 5 mL di penicillina-streptomicina in 500 mL di DMEM e mescolare bene.
- Aspirare il mezzo dal piatto e lavare la coltura delle cellule BEND3 con 1 mL di PBS una volta. Aggiungere 1 mL di 0,25% Trypsin-EDTA nelle cellule e incubare le cellule per 4 min a 37 gradi centigradi.
- Aggiungere 1 mL di BM nelle celle per neutralizzare Trypsin-EDTA e pipetta su e giù delicatamente per dissociare completamente le cellule. Trasferire la sospensione cellulare in un nuovo tubo conico da 15 mL e pellet per centrifugazione a temperatura ambiente (RT) per 5 min a 400 x g.
- Aspirare il supernatante dal tubo e risospendere le cellule con 9 mL di BM fresco, quindi aggiungere 1 mL di sospensione cellulare in un inserto supporto permeabile. Aggiungere altri 2 mL di BM fresco per bene nella camera inferiore della matrice. Continuare a coltura le cellule per un giorno.
2. Preparazione della cultura del corticale del mouse NSPC
-
Preparazione del piatto di coltura, mezzo di digestione della papaina, e mezzo di coltura corticale aderente (AM)
- Cappotto 6 pozzetti con poli-L-lysine (PLL) aggiungendo 1 mL di soluzione PLL per bene in piastre a 6 pozzetti. Quindi, incubare le piastre a RT per 30 min.
- Trasferire la soluzione PLL in un tubo conico da 15 mL. Lavare le piastre 3 volte con acqua doppia distillata. Asciugare le piastre e metterle da parte fino all'uso.
- Preparare il mezzo di digestione papaina sommando 50 U di papaina, 50 -L di L-glutamine e 50 -L di 100 mg/mL di acetil-L-cisteina in 5 mL di DMEM. Mescolare brevemente il mezzo e riscaldarlo a 37 gradi centigradi per 30 min per l'attivazione degli enzimi.
- Preparare il mezzo di coltura aderente alle cellule corticali (AM): aggiungere 500 500 l di pironevatodi di sodio, 500 l l di 100 mg/mL N-acetyl-L-Cysteine, 500 l di N2, 1 mL di B27 e 5 luna di 100 g/mL bFGF in 50 mL di DMEM. Mescolare bene il mezzo e scaldarlo a 37 gradi centigradi prima dell'uso.
-
Preparazione delle cellule corticali cerebrali primarie e successiva placcatura
- Sacrificare un topo incinta a tempo E10.5 con lussazione cervicale.
NOT: A E10.5, la maggior parte delle cellule sta proliferando NSPC nella corteccia cerebrale, dando origine a grandi cloni di progenie in vitro. - Sterilizzare l'addome del 75% di etanolo. Utilizzare forbici sottili e pinze microseghettate per aprire l'addome tagliando la pelle e il muscolo sottostante lungo il lato destro della linea mediana. Rimuovere delicatamente l'utero dalla cavità addominale con pinze seghettate e tagliarlo dalla cavità addominale con le forbici fini.
- Lavare l'utero con 40 mL di HBSS pre-raffreddato in piatto Petri da 10 cm. Quindi, trasferire l'utero in un nuovo piatto Petri 10 cm e lavarlo di nuovo con 40 mL di HBSS pre-raffreddato.
- Trasferire l'utero in una nuova teglia Petri di 10 cm con 40 mL di HBSS pre-raffreddato. Rimuovere gli embrioni dall'utero e dalla membrana amniotica, quindi tagliare le teste degli embrioni dai tronchi con microforzati Jeweler.
- Lavare le testine con 40 mL di HBSS pre-raffreddato e trasferire le testine in una nuova parabola Petri di 10 cm con 40 mL di HBSS pre-raffreddato. Usa le microcarpi Jewelers per staccare la pelle e la cartilagine che coprono il cervello, quindi taglia le cortice cerebrali e raccoglile in un tubo conico da 15 mL con HBSS pre-raffreddato.
- Pellet le cortice per centrifugazione per 3 min a 4 gradi centigradi e 300 x g. Aspirare il supernatante dal tubo, quindi aggiungere il mezzo di digestione papaina attivato e 15 -L di 4 mg/mL DNase I nel pellet di tessuto.
- Risospendere brevemente il pellet di tessuto con un leggero vortice. Incubare il tessuto a 37 gradi centigradi per 30 min. Durante questo periodo, allentare il tessuto da breve vortice ogni 10 min.
NOT: Alla fine della digestione, non ci dovrebbero essere pezzi di tessuto visibili nel tubo. - Pellet le cellule corticali per centrifugazione per 10 min a 4 gradi centigradi e 450 x g. Aspirare il supernatante dal tubo e lavare il pellet cellulare con DMEM pre-raffreddato. Ripetere questo passaggio una volta.
NOT: Durante la digestione e il lavaggio, prestare attenzione a non pipettare i tessuti e pellet cellulare grossolanamente per evitare di danneggiare le cellule con una forte forza di tosare. - Aspirare il supernatante dal tubo quindi aggiungere 1,5 mL di HBSS pre-raffreddato nel tubo. Dissociare il pellet di cellule corticali in singole celle con pipettaggio delicato. Contare il numero di cella con un emocitometro.
- Aggiungere 2 mL di AM e 2 x 104 cellule corticali per bene in piastre a 6 pozzetti. Incubare la piastra a 37 gradi centigradi e il 5% di CO2 per 3 h per consentire alle cellule di attaccarsi alla piastra.
- Sacrificare un topo incinta a tempo E10.5 con lussazione cervicale.
3. Creazione della cocultura neurale-endoteliale e del sistema di etichettatura Ac4ManNAz
- Un giorno dopo aver placcato le celle BEND3 negli inserti, aspirare delicatamente il mezzo nella camera inferiore prima, quindi gli inserti. Lavare l'infaccia degli inserti 3 volte con DMEM preriscaldato. Lavare la superficie esterna degli inserti risciacquando con DMEM preriscaldato.
- Aggiungere 1 mL di AM preriscaldato in un inserto, quindi trasferire gli inserti nei pozze con cellule corticali primarie. Incubare la co-cultura a 37 e 5% CO2 per 12 h.
- Sciogliere Ac4ManNAz in DMSO per raggiungere una concentrazione di scorte di 200 mM. 12 h dopo aver impostato la cocoltura neurale-endoteliale, aggiungere 1 -L di ac4pinze ManNAz per camera inferiore e 0,5 - L di stock per inserto nella co-cultura. Agitare immediatamente e delicatamente le piastre per mescolare bene il mezzo. Per le celle di controllo, aggiungere lo stesso volume di DMSO.
- Coltura le cellule per altri 5 giorni a 37 gradi centigradi e 5% di CO2. Preparare l'AM con 10x bFGF come mezzo di rialimentazione (RM). Durante questo periodo, aggiungere 100 L di RM per inserto e 200 L di RM per camera inferiore per alimentare nuovamente le cellule endoteliali e neurali ogni due giorni. Durante il refeeding, non fornire Ac4ManNAz o DMSO nella coltura.
4. Colorazione immunofluorescente delle sialoglycoproteine negli NSPC primari espansi e nei neuroni differenziati
- Preparare bTTAA-CuSO4 complesso 1 30x stock contenente 1,5 mCuSO4 e 9 m BTTAA in acqua a doppia distillazione. Preparare il buffer 1 appena coniugato a biotina contenente 50 m biotin-alkyne, 2,5 mm di ascorbate di sodio e 1complesso BTTAA-CuSO4 in PBS.
- Rimuovere gli inserti dalle piastre di cocoltura. Aspirare il mezzo di coltura dai pozzi inferiori e lavare le cellule neurali una volta con PBS preriscaldato.
- Aspira il PBS dai pozzi. Aggiungere 1 mL di soluzione PBS paraformaldeide pre-raffreddata 4% per bene nelle cellule e fissare le cellule a RT per 10 min. Quindi, lavare le cellule 3 volte con PBS pre-freddo.
- Aspirare PBS dai pozzi e aggiungere 1 mL di buffer a biotina appena preparato 1 per bene nelle cellule. Incubare le cellule a RT per 10 min.
- Aspirare il buffer di reazione dai pozzi. Lavare le cellule 3 volte con PBS. Preparare il buffer di colorazione contenente l'1% FBS e 1 g/mL Alexa Fluor 647-streptavidin. Aggiungere 1 mL di tampone di colorazione per bene nelle cellule e incubare le cellule a RT per 30 min.
- Aspirare il buffer di colorazione dai pozzi e le cellule lavate 3 volte con PBS pre-raffreddato. Preparare il buffer di blocco contenente 5% BSA e 0,3% detergente non ionico-100 in PBS. Aggiungere 1 mL di buffer di blocco per bene nelle cellule e incubare a RT per 10 min.
- Preparare una soluzione anticorpale primaria diluendo gli anticorpi anti-nestina e anti-tubulina III insieme nel buffer di blocco a rapporti di 1:20 e 1:1.000, rispettivamente. Rimuovere il buffer di blocco dai pozzetti e aggiungere 1 mL di soluzione anticorpale primaria per bene nelle cellule. Incubare le cellule a 4 gradi centigradi durante la notte.
- Rimuovere la soluzione anticorpale primaria dai pozzi. Lavare le cellule 3 volte con PBS pre-freddo. Preparare una soluzione anticorpale secondaria diluindo Alexa Fluor 488 capra anti-topo IgG1, Alexa Fluor 546 capra anti-topo IgG2b, e DAPI insieme nel buffer di blocco a una diluizione di 1:1,000. Aspirare il PBS dai pozzi e aggiungere 1 mL soluzione anticorpale secondaria per bene nelle cellule. Incubare le cellule a RT per 2 h.
- Aspirare la soluzione anticorpale dai pozzi e lavare le cellule 3 volte con PBS pre-raffreddato. Successivamente, le celle sono pronte per l'acquisizione dell'immagine.
5. Purificazione delle Sialoglycoproteine da NPC primari espansi e neuroni differenziati
- Preparare BTTAA-CuSO4 complesso 2 15x stock contenente 1,5 m CuSO4 e 3 mM BTTAA in acqua a doppia distillazione. Preparare il buffer di sospensione delle proteine A contenente il 4% SDS e 10 mM EDTA in acqua a doppia distillazione; tamburo di sospensione proteica B contenente 150 mM NaCl, trietanolamina 50 mM e 1% di etere oleyl in polioxyethylene (ad esempio Brij97) in acqua a doppia distillazione con pH 7.4. Prima dell'uso, mescolare il buffer A:buffer B : 1:8 (vol/vol) per preparare il buffer di sospensione proteica completo. Preparare il buffer di lavaggio delle proteine 1 contenente il 2% di SDS in PBS; tamburo di lavaggio delle proteine 2 contenente 8 M di urea in bicarbonato di ammonio 250 mM (ABC); e buffer di lavaggio proteico 3 contenente 2,5 M NaCl in PBS.
- Rimuovere gli inserti dalle piastre di cocoltura. Aspirare il mezzo di coltura dai pozzi inferiori e lavare le cellule neurali una volta con PBS pre-raffreddato.
- Aspirare il PBS dai pozzi e aggiungere 200 -L di tampone RIPA pre-raffreddato per bene nelle piastre. Incubare le piastre sul ghiaccio per 5 min. Raccogliere la lisi proteica in tubi da 1,5 mL. Pellet i detriti cellulari per centrifugazione per 10 min a 4 gradi centigradi e 12.000 x g.
- Trasferire il supernatante in nuovi tubi da 1,5 mL. Determinare la concentrazione proteica con il kit BCA secondo le istruzioni del produttore. Regolare la concentrazione proteica a 1 mg/mL.
- Aggiungere 100 alkyne-biotina, 2,5 m di ascorbate di sodio da 2,5 m e 1 x BTTAA-CuSO4 complesso 2 a 1 mL di lisi proteica e mescolare bene la soluzione. Incubare il mix a RT per 1 h.
- Trasferire la soluzione di reazione in 20 mL di metanolo pre-raffreddato in un tubo conico da 50 mL. Mescolare bene e incubare a -30 gradi durante la notte per far precipitare le proteine.
- Pellet la proteina precipita per centrifugazione per 15 min a 4 e 4.500 x g. Lavare il pellet proteico due volte con 20 mL di metanolo pre-raffreddato. Aspirare il super-attuoso dal tubo. Risospendere il pellet proteico con 4 mL di tampone proteico resuspension e trasferire la sospensione della proteina in un nuovo tubo conico da 15 mL.
- Prendere 50 - L di perline streptavidin e lavarle 3 volte con PBS. Aggiungere le perline lavate nella sospensione delle proteine. Incubare la soluzione a 4 gradi centigradi per 3 h su un rotatore verticale ad una velocità di rotazione di 20 giri/min.
- Lavare le perline in sequenza con 6 tipi di buffer: buffer di lavaggio delle proteine 1, buffer di lavaggio delle proteine 2, buffer di lavaggio delle proteine 3, 0,5 M ABC, 0,25 M ABC e 0,05 M ABC.
- Dopo il lavaggio, risospendere le perline con 20 gradi di PBS e trasferire le perline in un nuovo tubo da 1,5 mL. Aggiungete nelle perline 20 luna di 2x di proteine e trattate a 95 gradi centigradi per 10 min. I campioni di proteine devono quindi essere sottoposti a SDS-PAGE e macchiati con Coomassie brillante blu R-250 secondo le istruzioni del produttore. Tagliare le proteine in gel come indicato da Coomassie brillante blu R-250 per l'analisi della spettrometria di massa.
Risultati
L'intera procedura per l'espansione in vitro e l'etichettatura metabolica degli NSPN embrionali primari richiede 6 giorni(Figura 1A). La qualità della linea cellulare BEND3 e degli NSPC primari appena isolati sono la chiave per un esperimento di successo. Le cellule BEND3 sono la fonte di fattori solubili che stimolano l'auto-rinnovamento e la proliferazione degli NSPC. Va assicurato che le cellule BEND3 siano prive di qualsiasi contaminazione e si dividano attivamente con una minima morte cellulare prima della co-codifica con cellule neurali. Gli NSPC primari devono essere preparati con cura per evitare danni eccessivi durante la dissociazione. Gli NSPC danneggiati possono ancora crescere e differenziarsi; tuttavia, non sono in grado di rispondere a stimoli endoteliali bene per mantenere stelo ed espandersi. Occorre prestare particolare attenzione per essere asettico durante la coltura cellulare, in quanto il protocollo non suggerisce l'aggiunta di antibiotici al mezzo di coltura primaria.
La cocultura endoteliale di successo porterà gli NSPC a formare cloni di grandi dimensioni, simili a fogli. Tali forme clone in primo piano diventano evidenti al giorno 4 e sono molto tipiche al giorno 6. All'interno dei cloni, le cellule mantengono uno stretto contatto tra loro. L'immunostaining con anticorpi contro il marcatore NSPC Nestin e il marcatore neuronale z-tubulina III dovrebbe rivelare che nel clone, la maggior parte delle cellule sono Nestin- NSPC e pochissime sono cellule neuronali. Al contrario, la percentuale di cellule Nestin e z-tubulina III- le cellule neuronali in cloni formati nel sistema non co-culturale sono quasi la stessa (Figura 1B, 1De 1E).
Il reporter chimico, Ac4ManNAz, è un analogo metabolico e può essere incorporato nel percorso di sialazione della proteina intrinseca. Alte dosi di Ac4ManNAz sono tossiche per le cellule. Per ogni tipo specifico di cellula, la concentrazione di etichettatura di Ac4ManNAz deve essere pre-testata per ottenere la massima efficienza di etichettatura senza citotossicità significativa. In questo caso, la concentrazione ottimizzata di etichettatura di Ac4ManNAz per NSPC primaria è di 100 M. La valutazione combinatoria della morte cellulare e dei nuclei suggerisce che questa concentrazione di etichettatura non causa evidenti effetti citotossici ed è in grado di etichettare in modo efficiente gli NSPC(Figura 1C e 1D). La morfologia clonale, l'autorinnovamento e il potenziale di differenziazione degli NSPC sia nel sistema di cocultura endoteliale che nel sistema non co-culturale sono interessati (Figura 1C, 1De 1E).
L'etichettatura di successo degli NSPC di Ac4ManNAz può essere esaminata dopo aver coniugato biotina a una cultura mediata da una reazione bioortogonale tra azide e alkyne. Ogni cellula della cultura etichettata aAc4ManNAz viene macchiata e visualizzata con Alexa Fluor 647-streptavidin. Nessuna cellula è positiva per la colorazione Alexa Fluor 647-streptavidin nel gruppo di controllo DMSO. Inoltre, i campioni di proteine preparati dalla coltura etichettata aAc4ManNAz dalla coniugazione biotina e dalla purificazione delle perline di streptavidin mostrano un forte segnale di colorazione blu brillante Coomassie nei gel SDS-PAGE. Nel frattempo, c'erano solo sfondi di colorazione e segnali di legame non specifici nelle corsie caricate con campioni proteici dal gruppo di controllo DMSO. Ciò indica anche l'etichettatura efficiente dei NSPC da parte di Ac4ManNAz(Figura 1F).
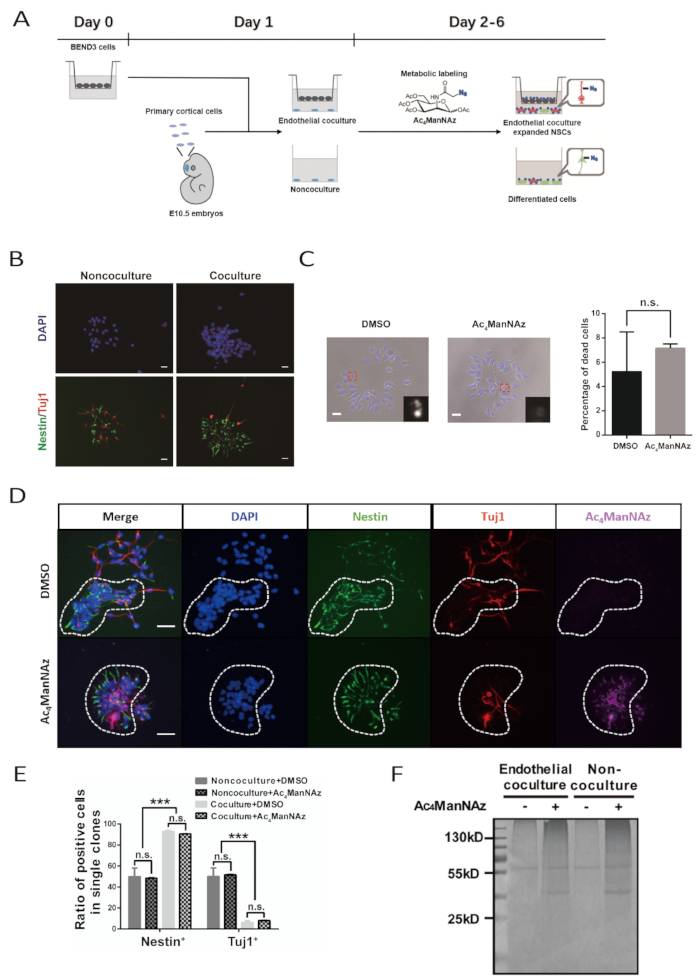
Figura 1 : Identificazione dei marcatori di superficie cellulare per i NPC primari assistiti dal sistema di cocoltura endoteliale e dall'etichettatura sialoglycan metabolica. (A) Schematico del flusso di lavoro per il protocollo. Questa cifra è stata modificata da Bai et al. 10. Le celle BEND3 vengono semiate in inserti a matrice su D0. La preparazione delle NSPC corticali primarie e l'impostazione del sistema di co-coltura vengono eseguite su D1. L'etichettatura metabolica della coltura dura da D2 a D6. La rialimentazione culturale viene effettuata su D3 e D5. (B) Le immagini immunofluorescenti per i cloni formati dai NPC primari dopo la coltura di 5 giorni con o senza cellule endoteliali. La barra di scala indica 20 immagini a campo luminoso per i cloni formati dai NSPC primari dopo una coltura di 5 giorni con Ac4ManNAz o DMSO. I nuclei sono stati controbilanciati da DAPI. La barra della scala indica 20 m. La barra di errore indica SEM (n.s. - non significativo). (D) Le immagini immunofluorescenti per NSPC formarono cloni nella co-cultura endoteliale con Ac4ManNAz o DMSO. Cerchio tratteggiato delimita un singolo clone neurale. La barra della scala indica una quantificazione di 50 m. (E) di NSPC e neuroni differenziati in cloni formati da NPC in sistemi di cocoltura endoteliale e non co-cultura con etichettatura Ac4ManNAz o controllo DMSO. La barra di errore indica SEM (Sezione : p < 0.0005; n.s. ) non significativo. (F) Coomassie brillante colorazione blu delle proteine purificate da perline streptavidine da cellule neurali etichettate con Ac4ManNAz o DMSO in co-cultura endoteliale e nonco-cultura sistema. La banda da 55 kD nei gruppi di etichettatura di controllo rappresenta proteine leganti non specifiche. (B, C, E e F) corrispondenti a questo protocollo sono stati adattati da Bai et al. 10. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
I marcatori di superficie sono comunemente utilizzati per etichettare e purificare specifici tipi di cellule in vitro e in vivo17,18. La scoperta dei marcatori di superficie contribuisce notevolmente alla medicina rigenerativa e alle ricerche sulle cellule staminali fornendo strumenti molecolari per arricchire selettivamente una popolazione di cellule staminali da tessuti e piatti di coltura normali o patologici, offrendo una cellula purificata per uso clinico o per lo studio delle proprietà biologiche. Tuttavia, i progressi nello sviluppo di marcatori di superficie per la ricerca sulle cellule staminali neurali sono stati lenti a causa della difficoltà nell'isolare le cellule staminali dai tessuti primari. Il protocollo qui descritto si basa su una piattaforma in vitro semplificata. Confrontando gli NPC primari espansi da una cocoltura endoteliale a una coltura neurale differenziante, le proteine espresse in modo differenziato nelle NPC espanse sono evidenziate e consentono un'ulteriore identificazione. Il nostro protocollo fornisce anche una strategia alternativa per purificare le proteine della superficie cellulare dirottando il percorso metabolico intrinseco per etichettare sialoglycan con gruppi bioortogonali. Rispetto ai protocolli tradizionali per la purificazione delle proteine della superficie cellulare, i vantaggi di questo protocollo sono sostenuti da due caratteristiche specifiche: 1) la prevalenza della verifica sulle proteine della superficie cellulare garantisce una copertura massima del proteoma della superficie cellulare, e 2) la specificità della reazione tra il gruppo bioortogonale e i suoi ligandi conferisce la purezza del proteoma superficiale acquisito. Pertanto, il nostro protocollo si traduce in un'analisi proteomica più sensibile nel caso di materiali di partenza meno. Abbiamo dimostrato la fattibilità di questo protocollo nei marcatori di superficie primari degli NSPC. Con le opportune modifiche sull'espansione delle cellule staminali in vitro, questo approccio chemioproomico può essere compatibile con l'identificazione dei marcatori di superficie di altri tipi di cellule staminali. È interessante notare che, poiché Ac4ManNAz è per-O-acetylated, potrebbe portare a S-glycosylation artificiale. L'uso di zuccheri innaturali senza ceto può evitare la formazione di artefatto e migliorare la specificità e la validità dell'etichettatura del glicano metabolico nelle cellule viventi19.
La preparazione delle cellule progenitrici neurali corticali primarie e delle cellule endoteliali sono passi critici del protocollo. In primo luogo, quando si digeriscono tessuti corticali embrionali, il tempo di digestione, la quantità di enzima e la forza di manipolazione devono essere attentamente controllati. Le forze eccessive di digestione e di tosatura meccanica danneggeranno l'integrità della membrana plasmatica e dei recettori della superficie cellulare che mediano la trasduzione del segnale per la sopravvivenza e la crescita delle cellule, e disturberanno anche la reattività degli NSPC alla stimolazione della stimolazione dei cellule endoteliali e la loro capacità di auto-rinnovamento. Per ottenere una corretta digestione, gli sperimentatori devono attivare completamente la papaina e fermare la digestione non appena i blocchi di tessuto scompaiono. In secondo luogo, le cellule BEND3 devono essere mantenute in uno stato sano per sostenere la secrezione. Si consiglia di utilizzare lotti di cellule BEND3 con meno passaggi e passare le cellule prima che raggiungano il 100% di confluenza. Ciò impedirà l'arresto del ciclo cellulare e la senescenza causata da danni al DNA accumulati durante il passaggio o dal contatto sovraffollato tra le cellule.
La tecnologia di sequenziamento ad alta produttività aumenta l'identificazione dei marcatori di superficie cellulare attraverso l'analisi dell'espressione dell'RNA, in particolare per i tipi di cellule, comprese le cellule staminali tissutali, che sono spesso presenti in vivo in quantità troppo piccole per eseguire l'analisi del proteoma Spettrometria. Anche se l'analisi RNA-seq può identificare geni specificamente espressi negli NSPC, potrebbe non riflettere realmente i livelli di espressione delle proteine, perché l'espressione dell'RNA non è sempre coerente con l'espressione proteica20. Inoltre, le biomolecole non proteiche che possono funzionare come marcatori di superficie non possono essere rilevate da studi trascrittomici. Ad esempio, l'oligosaccharide Lewis X è un noto produttore di superfici ampiamente utilizzato per etichettare cellule staminali embrionali umane e NSPC, anche se può essere associato a più proteine21. Pertanto, l'analisi della spettrometria di massa diretta non è ancora sostituibile e lo sviluppo di metodi che possono rendere l'analisi della spettrometria di massa più fattibile e conveniente è di grande interesse per studi futuri.
Oltre alla sialylation, altri tipi di modificazioni di proteine post-traduzionali svolgono un ruolo importante nella regolazione delle funzioni delle proteine modificate. Queste modifiche influenzano le proprietà delle proteine come la conformazione, l'emivita e la localizzazione subcellulare22,23. Diverse modifiche proteiche hanno specificità del tipo di cellula24,25,26. Con il crescente contenuto della cassetta degli attrezzi chimica, più tipi di modifica sono suscettibili di etichettatura metabolica con reporter chimici27. Di conseguenza, l'approccio chimico qui descritto può essere utilizzato per studiare altre differenze nella modificazione delle proteine tra cellule staminali e cellule differenziate, illustrando i meccanismi molecolari alla base del mantenimento delle proprietà delle cellule staminali e della differenziazione regolamento.
Divulgazioni
Gli autori non hanno nulla da divulgare.
Riconoscimenti
Le figure 1B, 1C, 1E e 1F sono riprodotte da Bai et al . 10 del sistema con il permesso della Royal Society of Chemistry. Ringraziamo Yi Hao nel laboratorio di X. C. per l'editing di figure. Questo lavoro è supportato dalla National Natural Science Foundation of China (da no. 91753206 a Q. S. e X. C., dal n. 31371093 al Q. S., e dal n. 21425204 e da 21672013 a X. C.).
Materiali
| Name | Company | Catalog Number | Comments |
| BEND3 | ATCC | CRL-229 | |
| DMEM | Gibco | 11960044 | |
| L-glutamine | Gibco | 25030081 | 1% |
| Sodium pyruvate | Sigma | P5280 | 1% |
| N2 supplement | Gibco | 17502048 | 1 to 100 |
| N-acetyl-L-cysteine | Sigma | A7250 | 1 mM |
| Papain | Worthington | LS003726 | 10 U/mL |
| B27 supplement | Gibco | 17504044 | 1 to 50 |
| Poly-L-lysine | Sigma | P4707 | |
| Basic Fibroblast growth factor | Gibco | PHG0261 | 10 ng/mL |
| Penicillin-Streptomycin | Gibco | 15140122 | 1% |
| Fetal bovine serum | Gibco | 10099141 | 10% |
| HBSS | Gibco | 14175095 | |
| Tripsin-EDTA, 0.25% | Gibco | 25200056 | |
| DPBS | Gibco | 14190094 | |
| Transwell | Corning | 3450 | |
| Paraformaldehyde | Sigma | 158127 | 4% |
| Sucrose | Sangon | A100335 | |
| DAPI | Gibco | 62248 | |
| RIPA buffer | Thermo Scientific | 89900 | |
| SDS-PAGE loading buffer 2x | Solarbio | P1018 | |
| 6-well plate | Corning | 3335 | |
| Tris-Glycine protein gel | invitrogen | xp00100box | |
| Mouse monoclonal anti-Nestin | Developmental Study Hybridoma Bank | Rat-401 | 1 to 20 |
| Mouse monoclonal anti-beta-tubulin III | Sigma | T8860 | 1 to 1,000 |
| Alexa Fluor 488 goat anti-mouse IgG1 | invitrogen | A-21121 | 1 to 1,000 |
| Alexa Fluor 546 goat anti-mouse IgG2b | invitrogen | A-21143 | 1 to 1,000 |
| Albumin Bovine V | Amresco | 0332 | |
| Triton X-100 | Amresco | 0694 | |
| BCA assay kit | Thermo Scientific | 23225 | |
| Dimethyl sulfoxide | Sigma | D2650 | |
| Brij97 | Aladdin | B129088 | |
| CuSO4 | Sigma | 209198 | |
| Alkyne-biotin | Click Chemistry Tools | TA105 | |
| BTTAA | Click Chemistry Tools | 1236 | |
| Ac4ManNAz | Click Chemistry Tools | 1084 | 100 µM |
| 9AzSia | synthesized in lab | ||
| Sodium ascorbate | Sigma | A4034 | |
| Methanol | Sigma | 34860 | |
| EDTA | Sangon | A100322 | |
| NaCl | Sangon | A100241 | |
| SDS | Sangon | A100227 | |
| Alexa Flour 647-conjugated streptavidin | invitrogen | S21374 | 1 to 1,000 |
| Triethanolamine | Sigma | V900257 | |
| Dynabeads M-280 Streptavidin | invitrogen | 60210 | |
| Ammonium bicarbonate | Sigma | 9830 | |
| Coomassie Brilliant Blue R-250 | Thermo Scientific | 20278 | |
| Isoflurane | RWD Life Science Co. | 970-00026-00 | |
| DNase I | Sigma | DN25 | 12 µg/mL |
| Urea | Sigma | U5378 |
Riferimenti
- Weissman, I. L. Stem Cells: Units of Development, Units of Regeneration, and Units in Evolution. Cell. 100, 157-168 (2000).
- Gage, F. H., Temple, S. Neural Stem Cells: Generating and Regenerating the Brain. Neuron. 80, 588-601 (2013).
- Gal, J. S. Molecular and Morphological Heterogeneity of Neural Precursors in the Mouse Neocortical Proliferative Zones. Journal of Neuroscience. 26, 1045-1056 (2006).
- Kawaguchi, A., et al. Single-cell gene profiling defines differential progenitor subclasses in mammalian neurogenesis. Development. 135, 3113-3124 (2008).
- Temple, S. The development of neural stem cells. Nature. 414, 112-117 (2001).
- Kwan, K. Y., Sestan, N., Anton, E. S. Transcriptional co-regulation of neuronal migration and laminar identity in the neocortex. Development. 139, 1535-1546 (2012).
- Kriegstein, A., Alvarez-Buylla, A. The Glial Nature of Embryonic and Adult Neural Stem Cells. Annual Review of Neuroscience. 32, 149-184 (2009).
- Wang, X., Tsai, J. W., LaMonica, B., Kriegstein, A. R. A new subtype of progenitor cell in the mouse embryonic neocortex. Nature Neuroscience. 14, 555-561 (2011).
- Taverna, E., Götz, M., Huttner, W. B. The Cell Biology of Neurogenesis: Toward an Understanding of the Development and Evolution of the Neocortex. Annual Review of Cell and Developmental Biology. 30, 465-502 (2014).
- Bai, Q. R., Dong, L., Hao, Y., Chen, X., Shen, Q. Metabolic glycan labeling-assisted discovery of cell-surface markers for primary neural stem and progenitor cells. Chemical Communications. 54, 5486-5489 (2018).
- Shen, Q., et al. Endothelial cells stimulate self-renewal and expand neurogenesis of neural stem cells. Science. 304, 1338-1340 (2004).
- Qian, X., et al. Timing of CNS cell generation: a programmed sequence of neuron and glial cell production from isolated murine cortical stem cells. Neuron. 28, 69-80 (2000).
- Varki, A. Glycan-based interactions involving vertebrate sialic-acid-recognizing proteins. Nature. 446, 1023-1029 (2007).
- Cheng, B., Xie, R., Dong, L., Chen, X. Metabolic Remodeling of Cell-Surface Sialic Acids: Principles, Applications, and Recent Advances. ChemBioChem. 17, 11-27 (2016).
- Lin, S. H., Guidotti, G. Purification of Membrane Proteins. Methods in Enzymology. 463, 619-629 (2009).
- Schmidt, J. R., et al. Pilot Study on Mass Spectrometry-Based Analysis of the Proteome of CD34+CD123+ Progenitor Cells for the Identification of Potential Targets for Immunotherapy in Acute Myeloid Leukemia. Proteomes. 6, (2018).
- Crisan, M., Dzierzak, E. The many faces of hematopoietic stem cell heterogeneity Development. Development. 144, 4195-4195 (2017).
- Uchida, N., et al. Direct isolation of human central nervous system stem cells. Proceedings of the National Academy of Sciences of the United States of America. 97, 14720-14725 (2000).
- Qin, W., et al. Artificial Cysteine S-Glycosylation Induced by Per-O-Acetylated Unnatural Monosaccharides during Metabolic Glycan Labeling. Angewandte Chemie International Edition. , (2018).
- Gry, M., et al. Correlations between RNA and protein expression profiles in 23 human cell lines. BMC Genomics. 10, 365 (2009).
- Hennen, E., et al. A LewisX Glycoprotein Screen Identifies the Low Density Lipoprotein Receptor-related Protein 1 (LRP1) as a Modulator of Oligodendrogenesis in Mice. Journal of Biological Chemistry. 288, 16538-16545 (2013).
- Seet, B. T., Dikic, I., Zhou, M. M., Pawson, T. Reading protein modifications with interaction domains. Nature Reviews Molecular Cell Biology. 7, 473-483 (2006).
- O’Brian, C. A., Chu, F. ReviewPost-translational disulfide modifications in cell signaling—role of inter-protein, intra-protein, S-glutathionyl, and S-cysteaminyl disulfide modifications in signal transmission. Free Radical Research. 39, 471-480 (2005).
- Williamson, A. J. K., Whetton, A. D. The requirement for proteomics to unravel stem cell regulatory mechanisms. Journal of Cellular Physiology. 226, 2478-2483 (2011).
- Christensen, B., et al. Cell Type-specific Post-translational Modifications of Mouse Osteopontin Are Associated with Different Adhesive Properties. Journal of Biological Chemistry. 282, 19463-19472 (2007).
- Yanagisawa, M., Yu, R. K. The expression and functions of glycoconjugates in neural stem cells. Glycobiology. 17, 57R-74R (2007).
- Best, M. D. Click Chemistry and Bioorthogonal Reactions: Unprecedented Selectivity in the Labeling of Biological Molecules. Biochemistry. 48, 6571-6584 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon