Method Article
Identifizieren von Zelloberflächenmarkern von primären neuralen Stamm- und Vorläuferzellen durch metabolische Kennzeichnung von Sialoglycan
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Präsentiert wird hier ein Protokoll, das ein in vitro neural-endotheliales Kokultursystem und die metabolische Einbeziehung von Sialoglycan mit bioorthogonalen funktionellen Gruppen kombiniert, um primäre neuronale Stamm- und Vorläuferzellen zu erweitern und ihre Oberfläche zu beschriften. sialoglycoproteinzur Bildgebung oder Massenspektrometrieanalyse von Zelloberflächenmarkern.
Zusammenfassung
Neurale Stamm- und Vorläuferzellen (NSPCs) sind die zelluläre Basis für die komplexen Strukturen und Funktionen des Gehirns. Sie befinden sich in spezialisierten Nischen in vivo und können isoliert und in vitroerweitert werden und dienen als wichtige Ressource für die Zelltransplantation, um Hirnschäden zu reparieren. NSPC sind jedoch heterogen und auf molekularer Ebene nicht klar definiert oder werden aufgrund des Fehlens spezifischer Zelloberflächenmarker gereinigt. Das vorgestellte Protokoll, das bereits berichtet wurde, kombiniert ein neuronal-endotheliales Kokultursystem mit einer metabolischen Glykan-Etikettierungsmethode, um das Oberflächen-Sialoglycoproteom der primären NSSPCs zu identifizieren. Das NSPC-endotheliale Kokultursystem ermöglicht die Selbsterneuerung und -erweiterung von primären NSPCs in vitround erzeugt eine ausreichende Anzahl von NSPCs. Sialoglycans in kultivierten NSPCs werden mit einem unnatürlichen Sialsäure-Metabolur-Reporter mit bioorthogonale funktionelle Gruppen. Durch den Vergleich der sialoglycoproteome aus sich selbst erneuernden NSPCs, die in einer endotheliaalen Kokultur erweitert wurden, mit der Differenzierung der neuronalen Kultur identifizieren wir eine Liste von Membranproteinen, die in NSPCs angereichert sind. Im Einzelnen umfasst das Protokoll: 1) Einrichtung einer NSPC-endotheliale Kokultur und NSPC-Differenzierungskultur; 2) Etikettierung mit Azidozucker pro-O-acetyliertem N-Azidoacetylmannosamin (Ac4ManNAz); und 3) Biotinkonjugation zu modifiziertem Sialoglycan zur Bildgebung nach Fixierung der neuronalen Kultur oder Proteinextraktion aus der neuronalen Kultur zur Massenspektrometrieanalyse. Anschließend werden die NSPC-angereicherten Oberflächenmarker-Kandidaten durch vergleichende Analyse von Massenspektrometriedaten sowohl aus dem erweiterten NSPC als auch aus differenzierten neuronalen Kulturen ausgewählt. Dieses Protokoll ist hochsensibel für die Identifizierung von Membranproteinen mit geringem Überfluss in den Ausgangsmaterialien und kann auf die Marker-Erkennung in anderen Systemen mit entsprechenden Modifikationen angewendet werden.
Einleitung
Neuronale Stammzellen sind definiert als eine multipotente Zellpopulation, die sich selbst erneuern kann, um einen Stammzellpool zu erhalten und sich in Neuronen und Glia zu differenzieren. Sie sind die wichtigsten Zelltypen im Nervensystem und können großes therapeutisches Potenzial in der regenerativen Medizin durch Zelltransplantation in kranke und verletzte Gehirnebieten 1,2. Mit der Entwicklung wird die neuronale Stammzellpopulation heterogen3,4, und der Anteil der neuronalen Stammzellen im Gehirn nimmt allmählich ab5. Im Allgemeinen befinden sich embryonale neuronale Stammzellen und andere neuronale Vorläuferzellen, kollektiv als neuronale Stamm- und Vorläuferzellen (NSPCs) bezeichnet, in den Keimzonen, der ventrikulären Zone und der subventrikulären Zone in Mäusen6. Im embryonalen Gehirn erzeugen neuronale Stammzellen Neuronen direkt oder indirekt über zwischengeschaltete Vorläuferzellen (IPCs) und bei einigen Arten durch die äußeren subventrikulären Zonenvorläufer (oRGs)7,8. Die spezifische molekulare Signatur, Morphologie, Lage in der Stammzellnische und Differenzierungspotenzial bestimmen die Rolle jedes Subtyps bei der Hirnorganogenese und klinischen Anwendungen9. Die derzeit verfügbaren Zelloberflächenmarker können jedoch verschiedene Subtypen von NSPCs nicht eindeutig unterscheiden und reinigen, was das Verständnis und die Nutzung dieser Subtypen einschränkt.
Die Identifizierung der primären NSPCs-Oberflächenmarker wird durch drei Haupthürden begrenzt. Die erste ist die begrenzte Zellanzahl von NSPCs im Gewebe, was es schwierig macht, Zelloberflächenproteinproben für eine gemeinsame Massenspektrometrieanalyse vorzubereiten. Die zweite Einschränkung ist die Schwierigkeit bei der Herstellung reiner Zellsubtypen zur Erzeugung subtypspezifischer Membranproteindaten. Die dritte Herausforderung schließlich ist das niedrige Verhältnis von Zelloberflächenproteinen in ganzen Zellproteinen, das ihre Nachweisempfindlichkeiten durch Massenspektrometrieanalyse behindert.
Um diese Probleme zu überwinden, entwickelten wir einen chemoprotemischen Ansatz zur selektiven Anreicherung und Identifizierung von Zelloberflächenproteinen in primären NSPCs durch metabolische Kennzeichnung der Sialoglycoproteine10. Um eine ausreichende Anzahl von NSPCs zu generieren, nutzten wir ein etabliertes Protokoll, um primäre embryonale NSPS in undifferenzierten Zuständen in vitro zu erweitern und zu erhalten, indem wir NSPCs mit Maushirn-Endothelzelllinien mit einer durchlässigen Unterstützung kokultieren. Matrixeinsatz(z.B. Transwell) System11. Im Gegensatz dazu erzeugen NPSCs, die allein ohne Endothelzellen kultiviert werden, eine differenzierte Nachkommenschaft11,12. So können Proteinproben aus diesen beiden Kultursystemen vergleichsweise analysiert werden, um Proteine zu identifizieren, die differenziell in NSPCs und differenzierten Neuronen exprimiert werden. Da die meisten Zelloberflächenproteine durch Sialinsäure13modifiziert werden, wurde der unnatürliche Sialinsäurevorläufer analog N-Azidoacetylmannosamin-Tetraacylatd (Ac4ManNAz) verwendet, um den intrinsischen Stoffwechselweg zu kapern, so dass endogene, neu synthetisierte Sialoglykane werden mit Azido-Gruppen beschriftet, wodurch ein chemischer Griff14erzeugt wird. Durch azido-alkyne-vermittelte bioorthogonale Reaktionen, die Biotin zu Sialoglycans konjugieren, können Zelloberflächenproteine visualisiert und zur proteomischen Identifizierung durch ein Streptavidin-gekoppeltes Fluorophor oder Matrix14angereichert werden.
Hier führen wir die Färbung der SDS-PAGE-Gelanalyse der Oberfläche sialoglycoproteome von NSPCs durch, die in einer endotheliaalen Kokultur erweitert sind und Zellen in einem Nicht-Kokultursystem differenzieren. Wir reinigen auch selektiv Oberflächensialoglycoproteome in den beiden Kultursystemen für den proteomischen Vergleich. Unser Protokoll, verglichen mit den traditionellen zentrifugationsbasierten Zelloberflächenreinigungsprotokollen15, erhöht die Extraktionswirksamkeit durch Reduzierung der Oberflächenproteinextraktionsverfahren durch spezifische Tag-Konjugation und Affinität reinigung. In der Zwischenzeit erhöht es die Extraktionsreinheit von Zelloberflächenproteinen auf der Grundlage der Prämisse, dass Sialylierung vor allem an den Zelloberflächenproteinen stattfindet. Obwohl endotheliale Faktoren die Differenzierung erweiterter NSPC nicht vollständig blockieren können, bietet die vergleichende Studie zwischen einer Kokultur und einer differenzierten Kultur eine bequeme Methode, um mit Stammzellen angereicherte Oberflächenproteine zu lokalisieren, ohne dass Proteine aus NPCs zu analysieren, die durch FACS16gereinigt werden. Wir glauben, dass dieser Ansatz auf Studien von Oberflächenproteinen in anderen Systemen mit den entsprechenden Modifikationen angewendet werden kann.
Protokoll
Alle in dieser Studie verwendeten Tierprotokolle wurden vom IACUC (Institutional Animal Care and Use Committee) der Tsinghua University genehmigt und in Übereinstimmung mit den Richtlinien des IACUC durchgeführt. Die Labortieranlage der Tsinghua University wurde von der AAALAC (Association for Assessment and Accreditation of Laboratory Animal Care International) akkreditiert. Für die Inszenierung von Embryonen wurde der am Mittag des identifizierten Vaginalsteckers als Embryonaltag 0,5 (E0.5) berechnet.
HINWEIS: Alle Zellen werden im Zellinkubator unter Bedingungen von 37 °C und 5%CO2kultiviert.
1. Vorbereitung der Maus Endothelkultur in durchlässigen Stützeinsätzen
HINWEIS: BEND3-Zellen werden gemäß den Anweisungen des Herstellers gepflegt.
- Bereiten Sie BEND3 Zellmedium (BM) vor, indem Sie 50 ml FBS und 5 ml Penicillin-Streptomycin in 500 ml DMEM einteilen und gut mischen.
- Das Medium aus der Schale absaugen und die BEND3-Zellenkultur einmal mit 1 ml PBS waschen. Fügen Sie 1 ml 0,25% Trypsin-EDTA in die Zellen und inkubieren Sie die Zellen für 4 min bei 37 °C.
- Fügen Sie 1 ml BM in die Zellen, um Trypsin-EDTA und Pipette sanft nach oben und unten zu neutralisieren, um die Zellen vollständig zu dissoziieren. Übertragen Sie die Zellsuspension in ein neues 15 ml konisches Rohr und Pellet durch Zentrifugation bei Raumtemperatur (RT) für 5 min bei 400 x g.
- Aspirieren Sie den Überstand aus dem Rohr und suspendieren Sie die Zellen mit 9 ml frischem BM, dann fügen Sie 1 ml Zellsuspension in einen durchlässigen Stützeinsatz. Fügen Sie weitere 2 ml frisches BM pro Brunnen in der unteren Kammer der Matrix hinzu. Fahren Sie mit der Kultur der Zellen für einen Tag fort.
2. Vorbereitung der primären kortikalen NSPC-Kultur der Maus
-
Zubereitung von Kulturplatte, Papainverdauungsmedium und kortikalhaftes Kulturmedium (AM)
- Mantel 6-Well-Platten mit Poly-L-Lysin (PLL) durch Zugabe von 1 ml PLL-Lösung pro Well in 6-Well-Platten. Dann inkubieren Sie die Platten bei RT für 30 min.
- Übertragen Sie die PLL-Lösung in ein 15 ml konisches Rohr. Waschen Sie die Platten 3 mal mit doppelt destilliertem Wasser. Die Platten lüften und bis zum Gebrauch beiseite legen.
- Bereiten Sie das Papain-Verdauungsmedium vor, indem Sie 50 U Papain, 50 L L L-Glutamin und 50 l 100 mg/ml Acetyl-L-Cystein in 5 ml DMEM einteilen. Mischen Sie das Medium kurz und erwärmen Sie es auf 37 °C für 30 min für die Enzymaktivierung.
- Bereiten Sie das kortikale Zellhafte Kulturmedium (AM): 500 l L-Glutamin, 500 l Natriumpyruvat, 500 l von 100 mg/ml N-Acetyl-L-Cystein, 500 l N2, 1 ml B27 und 5 l von 100 g/ml bFGF in 50 ml DMEM hinzufügen. Mischen Sie das Medium gut und erwärmen Sie es vor Gebrauch auf 37 °C.
-
Herstellung von primären zerebralen kortikalen Zellen und anschließender Beschichtung
- Opfern Sie eine e10.5 getächte schwangere Maus durch Zervix-Dislokation.
HINWEIS: Bei E10.5 vermehren sich die meisten Zellen in der Großhirnrinde, was zu großen Klonen von Nachkommen in vitro führt. - Sterilisieren Sie den Bauch um 75% Ethanol. Verwenden Sie feine Schere und mikrogezackte Zangen, um den Bauch zu öffnen, indem Sie die Haut und den darunter liegenden Muskel entlang der rechten Seite der Mittellinie schneiden. Entfernen Sie die Gebärmutter vorsichtig mit gezackten Zangen aus der Bauchhöhle und schneiden Sie sie mit einer feinen Schere aus der Bauchhöhle.
- Waschen Sie die Gebärmutter mit 40 ml vorgekühlten HBSS in 10 cm Petrischale. Dann die Gebärmutter in eine neue 10 cm Petrischale geben und mit 40 ml vorgekühltem HBSS wieder waschen.
- Die Gebärmutter in eine neue 10 cm Petrischale mit 40 ml vorgekühltem HBSS geben. Entfernen Sie die Embryonen aus der Gebärmutter und der Fruchtwassermembran, und schneiden Sie dann die Köpfe der Embryonen mit Jewelers Microforceps von den Stämmen ab.
- Die Köpfe mit 40 ml vorgekühltem HBSS waschen und die Köpfe auf eine neue 10 cm Petrischale mit 40 ml vorgekühltem HBSS übertragen. Verwenden Sie Jewelers Microforceps, um Haut und Knorpel, die das Gehirn bedecken, zu schälen, schneiden Sie dann die Zerebralparese ab und sammeln Sie sie in einem 15 ml konischen Rohr mit vorgekühltem HBSS.
- Pellet die Kortiken durch Zentrifugation für 3 min bei 4 °C und 300 x g. Aspirieren Sie den Überstand aus der Röhre, dann fügen Sie aktivierte Papain-Verdauungmittel und 15 l von 4 mg/ml DNase I in das Gewebepellet.
- Das Gewebepellet durch sanftes Wirbeln kurz aussetzen. Inkubieren Sie das Gewebe bei 37 °C für 30 min. Während dieser Zeit, lockern Sie das Gewebe durch kurze Wirbel alle 10 min.
HINWEIS: Am Ende der Verdauung sollten keine sichtbaren Gewebestücke in der Röhre vorhanden sein. - Pellet die kortikalen Zellen durch Zentrifugation für 10 min bei 4 °C und 450 x g. Den Überstand aus dem Rohr ansaugen und das Zellpellet mit vorgekühltem DMEM waschen. Wiederholen Sie diesen Schritt einmal.
HINWEIS: Achten Sie beim Verdauen und Waschen darauf, das Gewebe und das Zellpellet nicht grob zu pipetten, um eine Schädigung der Zellen mit einer starken Scherkraft zu vermeiden. - Aspirieren Sie den Überstand aus dem Rohr und fügen Sie dann 1,5 ml vorgekühlten HBSS in das Rohr. Dissoziieren Sie das kortikale Zellpellet in einzelne Zellen mit sanfter Pipettierung. Zählen Sie die Zellennummer mit einem Hämozytometer.
- Fügen Sie 2 ml AM und 2 x 104 kortikale Zellen pro Brunnen in 6-Well-Platten hinzu. Inkubieren Sie die Platte bei 37 °C und 5%CO2 für 3 h, um die Zellen an der Platte anbringen zu lassen.
- Opfern Sie eine e10.5 getächte schwangere Maus durch Zervix-Dislokation.
3. Aufbau von neural-endotheliale Kokultur und Ac4ManNAz Etikettiersystem
- Einen Tag nach dem Plattieren von BEND3-Zellen in den Einsätzen, saugen Sie das Medium in der unteren Kammer zuerst sanft an, dann die Einsätze. Waschen Sie die Einsender der Einsätze 3 mal mit vorgewärmtem DMEM. Waschen Sie die Außenfläche der Einsätze durch Spülen mit vorgewärmten DMEM.
- Fügen Sie 1 ml vorgewärmtes AM in einen Einsatz ein, und übertragen Sie die Einsätze dann mit primären kortikalen Zellen in die Brunnen. Inkubieren Sie die Co-Kultur bei 37 °C und 5%CO2 für 12 h.
- Ac4ManNAz in DMSO auflösen, um eine Lagerkonzentration von 200 mM zu erreichen. 12 h nach dem Einrichten der neuronalen endothelialen Kokultur, fügen Sie 1 l Ac4ManNAz-Bestand pro Bodenkammer und 0,5 l Lager pro Einsatz in die Co-Kultur hinzu. Schütteln Sie die Platten sofort und sanft, um das Medium gut zu mischen. Fügen Sie für die Steuerzellen das gleiche Volumen von DMSO hinzu.
- Kultur die Zellen für weitere 5 Tage bei 37 °C und 5%CO2. Bereiten Sie das AM mit 10x bFGF als Nachspeisemedium (RM) vor. Während dieser Zeit fügen Sie 100 l RM pro Einsatz und 200 l RM pro Bodenkammer hinzu, um die endothelialen und neuronalen Zellen jeden zweiten Tag wieder einzuspeisen. Während der Nachfütterung nicht Ac4ManNAz oder DMSO in die Kultur liefern.
4. Immunfluoreszenzfärbung von Sialoglycoproteinen in erweiterten primären NSPCs und differenzierten Neuronen
- Bereiten Sie BTTAA-CuSO4 Komplex 1 30x Vorrat mit 1,5 mM CuSO4 und 9 mMBTTAA in doppelt destilliertem Wasser vor. Bereiten Sie frisch biotinkonjugierten Puffer 1 mit 50 M Biotin-Alkyn, 2,5 mM Natriumascorbat und 1x BTTAA-CuSO4-Komplex in PBS vor.
- Entfernen Sie die Einsätze von den Co-Kultur-Platten. Saugen Sie das Kulturmedium aus den unteren Brunnen und waschen Sie die Nervenzellen einmal mit vorgewärmten PBS.
- Aspirieren Sie die PBS aus den Brunnen. Fügen Sie 1 ml vorgekühlte 4% Paraformaldehyd-PBS-Lösung pro Brunnen in die Zellen ein und fixieren Sie die Zellen bei RT für 10 min. Dann waschen Sie die Zellen 3 mal mit vorgekühlten PBS.
- PBS aus den Brunnen ansaugen und 1 ml frisch zubereiteten biotinkonjugierten Puffer 1 pro Brunnen in die Zellen geben. Inkubieren Sie die Zellen bei RT für 10 min.
- Saugen Sie den Reaktionspuffer aus den Brunnen. Waschen Sie die Zellen 3 Mal mit PBS. Bereiten Sie den Färbepuffer vor, der 1% FBS und 1 g/mL Alexa Fluor 647-streptavidin enthält. Fügen Sie 1 ml Färbepuffer pro Bohrung in die Zellen ein und inkubieren Sie die Zellen bei RT für 30 min.
- Saugen Sie den Färbepuffer aus den Brunnen und gewaschenen Zellen 3 mal mit vorgekühlten PBS. Bereiten Sie den Sperrpuffer vor, der 5% BSA und 0,3% nichtionisches Waschmittel-100 in PBS enthält. Fügen Sie 1 ml Sperrpuffer pro Bohrplatz in die Zellen ein und brüten Sie bei RT 10 min.
- Bereiten Sie eine primäre Antikörperlösung vor, indem Sie die Antikörper gegen Nestin und Anti-S-Tubulin III zusammen in den Blockierpuffer im Verhältnis von 1:20 bzw. 1:1.000 verdünnen. Entfernen Sie den Sperrpuffer aus den Brunnen und fügen Sie 1 ml primäre Antikörperlösung pro Bohrung in die Zellen ein. Inkubieren Sie die Zellen bei 4 °C über Nacht.
- Entfernen Sie die primäre Antikörperlösung aus den Brunnen. Waschen Sie die Zellen 3 Mal mit vorgekühltem PBS. Bereiten Sie eine sekundäre Antikörperlösung vor, indem Sie Alexa Fluor 488 Ziege Anti-Maus IgG1, Alexa Fluor 546 Ziege Anti-Maus IgG2b und DAPI zusammen in Blockierpuffer bei einer Verdünnung von 1:1.000 verdünnen. Aspirieren Sie die PBS aus den Brunnen und fügen Sie 1 ml sekundäre Antikörperlösung pro Brunnen in die Zellen. Inkubieren Sie die Zellen bei RT für 2 h.
- Die Antikörperlösung aus den Brunnen ansaugen und die Zellen 3 Mal mit vorgekühltem PBS waschen. Danach sind die Zellen für die Bildaufnahme bereit.
5. Reinigung von Sialoglycoproteinen aus erweiterten primären NSPCs und differenzierten Neuronen
- Bereiten Sie BTTAA-CuSO4 Komplex 2 15x Vorrat mit 1,5 mM CuSO4 und 3 mMBTTAA in doppelt destilliertem Wasser vor. Herstellung des Proteinresuspensionspuffers A mit 4% SDS und 10 mM EDTA in doppelt destilliertem Wasser; Protein-Resuspensionspuffer B mit 150 mM NaCl, 50 mM Triethanolamin und 1% Polyoxyethylenoleylether (z.B. Brij97) in doppelt destilliertem Wasser mit pH 7,4. Mischen Sie vor der Verwendung den Puffer A:puffer B = 1:8 (vol/vol), um den vollständigen Proteinresuspensionspuffer vorzubereiten. Herstellung von Proteinwaschpuffer 1 mit 2% SDS in PBS; Protein-Waschpuffer 2 mit 8 M Harnstoff in 250 mM Ammoniumbicarbonat (ABC); und Proteinwaschpuffer 3 mit 2,5 M NaCl in PBS.
- Entfernen Sie die Einsätze von den Co-Kultur-Platten. Das Kulturmedium aus den unteren Brunnen ansaugen und die Nervenzellen einmal mit vorgekühltem PBS waschen.
- Aspirieren Sie die PBS aus den Brunnen und fügen Sie 200 l vorgekühlten RIPA-Puffer pro Well in die Platten. Inkubieren Sie die Teller auf Eis für 5 min. Sammeln Sie die Proteinlyse in 1,5 ml Röhren. Pellet die Zellablagerungen durch Zentrifugation für 10 min bei 4 °C und 12.000 x g.
- Übertragen Sie den Überstand in neue 1,5 ml-Rohre. Bestimmen Sie die Proteinkonzentration mit dem BCA-Kit gemäß den Anweisungen des Herstellers. Stellen Sie die Proteinkonzentration auf 1 mg/ml ein.
- Fügen Sie 100 M Alkyn-Biotin, 2,5 mM Natriumascorbat und 1x BTTAA-CuSO4 Komplex 2 bis 1 ml Proteinlyse hinzu und mischen Sie die Lösung gut. Inkubieren Sie die Mischung bei RT für 1 h.
- Übertragen Sie die Reaktionslösung in 20 ml vorgekühltes Methanol in einem 50 ml konischen Rohr. Gut mischen und bei -30 °C über Nacht inkubieren, um die Proteine auszufällen.
- Pellet das Protein scheide durch Zentrifugieren 15 min bei 4 °C und 4.500 x gaus. Waschen Sie das Proteinpellet zweimal mit 20 ml vorgekühltem Methanol. Aspirieren Sie den Überstand aus der Röhre. Setzen Sie das Proteinpellet mit 4 ml Proteinresuspensionspuffer wieder auf und übertragen Sie die Proteinresuspension in eine neue 15 ml konische Röhre.
- Nehmen Sie 50 L Streptavidinperlen und waschen Sie sie 3 Mal mit PBS. Fügen Sie die gewaschenen Perlen in die Proteinresuspension. Inkubieren Sie die Lösung bei 4 °C für 3 h auf einem vertikalen Rotator bei einer Drehzahl von 20 Umdrehungen pro Minute.
- Waschen Sie die Perlen sequenziell mit 6 Arten von Puffern: Proteinwaschpuffer 1, Proteinwaschpuffer 2, Proteinwaschpuffer 3, 0,5 M ABC, 0,25 M ABC und 0,05 M ABC.
- Nach dem Waschen die Perlen mit 20 l PBS wieder aufhängen und die Perlen in ein neues 1,5 ml Rohr geben. 20 L 2x Protein-Ladepuffer in die Perlen geben und bei 95 °C 10 min behandeln. Die Proteinproben sollten dann SDS-PAGE unterzogen und nach herstellerspezifischen Anweisungen mit Coomassie brillantblau R-250 gefärbt werden. Schneiden Sie die Proteine in Gel, wie von Coomassie brillant blau R-250 für die Massenspektrometrie-Analyse angegeben.
Ergebnisse
Das gesamte Verfahren zur In-vitro-Expansion und metabolischen Kennzeichnung primärer embryonaler NSPCs dauert 6 Tage (Abbildung 1A). Die Qualität der BEND3-Zelllinie und frisch isolierte primäre NSSSPCs sind der Schlüssel zu einem erfolgreichen Experiment. BEND3-Zellen sind die Quelle von löslichen Faktoren, die die Selbsterneuerung und Proliferation von NSPCs stimulieren. Es sollte sichergestellt werden, dass die BEND3-Zellen frei von Kontamination sind und sich aktiv mit minimalem Zelltod teilen, bevor sie mit Nervenzellen kokultivieren. Die primären NSPC müssen sorgfältig vorbereitet sein, um übermäßige Schäden während der Dissoziation zu vermeiden. Beschädigte NSPC können immer noch wachsen und differenzieren; sie sind jedoch nicht in der Lage, auf endotheliale Reize gut zu reagieren, um die Haltigkeit aufrechtzuerhalten und sich auszudehnen. Besondere Vorsicht ist während der Zellkultivierung aseptisch zu sein, da das Protokoll keine Zugabe von Antibiotika zum primären Kulturmedium vorschlägt.
Erfolgreiche endotheliale Co-Kultur wird NSPCs dazu bringen, große, blattartige Klone zu bilden. Solche vorgestellten Klonformen werden an Tag 4 deutlich und sind an Tag 6 sehr typisch. Innerhalb der Klone halten die Zellen engen Kontakt zueinander. Die Immunfärbung mit Antikörpern gegen den NSPC-Marker Nestin und den neuronalen Marker -Tubulin III sollte zeigen, dass im Klon die meisten Zellen Nestin+ NSPCs und nur sehr wenige die Neuronenzellen von Stubulin III+ sind. Im Gegensatz dazu ist der Prozentsatz der Nestin+-Zellen und der Neuronalzellen im Klon, die im Nicht-Kokultursystem gebildet werden, nahezu gleich(Abbildung 1B, 1Dund 1E).
Der chemische Reporter, Ac4ManNAz, ist ein metabolisches Analogund und kann in den intrinsischen Protein-Sialylierungsweg integriert werden. Hohe Dosen von Ac4ManNAz sind toxisch für Zellen. Für jede bestimmte Zellart sollte die Etikettierungskonzentration von Ac4ManNAz vorgetestet werden, um die höchste Etikettiereffizienz ohne signifikante Zytotoxizität zu erreichen. Hier liegt die optimierte Etikettierungskonzentration von Ac4ManNAz für primäre NSPC bei 100 M. Die kombinatore Auswertung des Zelltodes, die durch Zell- und Kernmorphologie angezeigt wird, legt nahe, dass diese Etikettierungskonzentration keine offensichtlichen zytotoxischen Wirkungen verursacht und NSPCs effizient zu beschriften (Abbildung 1C und 1D). Die klonale Morphologie, Selbsterneuerung und Differenzierungspotenzial von NSPCs sowohl im endotheliale Kokultur- als auch im Nicht-Kokultursystem sind nicht betroffen (Abbildung 1C, 1Dund 1E).
Die erfolgreiche Kennzeichnung von NSPCs durch Ac4ManNAz kann nach der Konjugierung von Biotin zu einer Kultur untersucht werden, die durch eine bioorthogonale Reaktion zwischen Azid und Alkyne vermittelt wird. Jede Zelle in der Ac4ManNAz-beschrifteten Kultur wird mit Alexa Fluor 647-streptavidin befleckt und visualisiert. Keine Zelle ist positiv für Alexa Fluor 647-streptavidin Färbung in der DMSO-Kontrollgruppe. Darüber hinaus zeigen Proteinproben, die aus der Ac4ManNAz-gekennzeichneten Kultur durch Biotinkonjugation und Streptavidin-Perlenreinigung hergestellt werden, ein starkes Coomassie-brillantblaues Färbesignal in SDS-PAGE-Gelen. In den mit Proteinproben der DMSO-Kontrollgruppe beladenen Bahnen gab es nur Färbehintergrund und unspezifische Bindungssignale. Dies weist auch auf die effiziente Kennzeichnung von NSPCs durch Ac4ManNAz hin (Abbildung 1F).
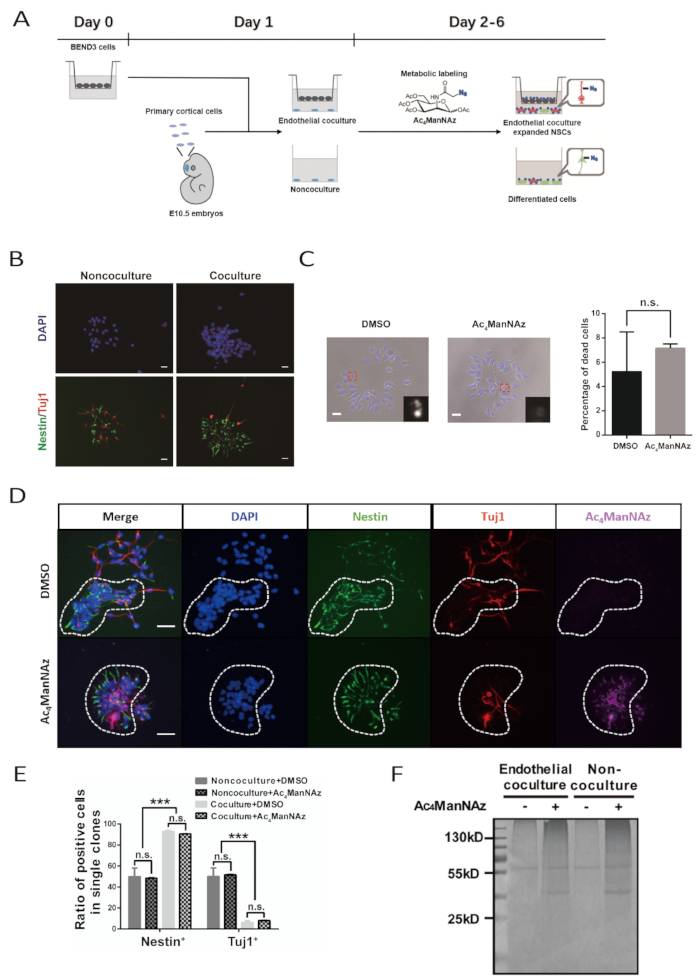
Abbildung 1 : Identifizierung von Zelloberflächenmarkern für primäre NSPC, unterstützt durch endotheliale Kokultursystem und metabolische Sialoglycan-Etikettierung. (A) Schematic des Workflows für das Protokoll. Diese Zahl wurde von Bai et al. geändert. 10. Die BEND3-Zellen werden in Matrixeinsätze auf D0 gesät. Die Vorbereitung der primären kortikalen NSPC und die Einrichtung eines Co-Kultur-Systems werden auf D1 durchgeführt. Die metabolische Kennzeichnung der Kultur dauert von D2 bis D6. Die Kulturnachfütterung erfolgt auf D3 und D5. (B) Die immunfluoreszierenden Bilder für Klone, die durch primäre NSPC nach 5-Tage-Kultur mit oder ohne Endothelzellen gebildet werden. Maßstabsleiste zeigt 20 m (C) Hellfeldbilder für Klone an, die von primären NSPCs nach einer 5-Tage-Kultur mit Ac4ManNAz oder DMSO gebildet werden. Die Kerne wurden von DAPI konterkariert. Der Maßstabsbalken zeigt 20 m an. Die Fehlerleiste gibt SEM an (n.s. = nicht signifikant). (D) Die immunfluoreszierenden Bilder für NSPC bildeten Klone in der endotheliale Kokultur mit Ac4ManNAz oder DMSO. Gestrichelter Kreis grenzt einen einzelnen neuronalen Klon ab. Der Maßstabsbalken zeigt 50 m .m. (E) Quantifizierung von NSPCs und differenzierten Neuronen in Klonen, die von NSPCs in endothelialen Kokultur- und Nicht-Co-Kultur-System mit Ac4ManNAz-Kennzeichnung oder DMSO-Steuerung gebildet werden. Die Fehlerleiste gibt SEM an (***p < 0.0005; n.s. = nicht signifikant). (F) Coomassie brillante blaue Färbung von Proteinen, die durch Streptavidinperlen aus Nervenzellen gereinigt werden, die mit Ac4ManNAz oder DMSO im endotheliaalen Kokultur- und Nichtkokultursystem gekennzeichnet sind. Das 55-kD-Band in Kontrollkennzeichnungsgruppen stellt unspezifische Bindungsproteine dar. (B, C, E und F), die diesem Protokoll entsprechen, wurden von Bai et al. 10. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Oberflächenmarker werden häufig verwendet, um bestimmte Zelltypen in vitro und in vivo17,18zu beschriften und zu reinigen. Die Entdeckung von Oberflächenmarkern trägt wesentlich zur regenerativen Medizin und Stammzellforschung bei, indem molekulare Werkzeuge zur selektiven Anreicherung einer Stammzellpopulation aus normalen oder pathologischen Geweben und Kulturgerichten zur Verfügung gestellt werden, die eine gereinigte Zelle anbieten. Ressourcen für die klinische Nutzung oder Untersuchung biologischer Eigenschaften. Die Entwicklung von Oberflächenmarkern für die forschung an neuronalen Stammzellen war jedoch nur langsam, da es schwierig war, Stammzellen aus Primärgeweben zu isolieren. Das hier beschriebene Protokoll basiert auf einer vereinfachten In-vitro-Plattform. Durch den Vergleich primärer NSPCs, die durch eine endotheliale Kokultur erweitert werden, mit einer differenzierenden neuronalen Kultur werden Proteine hervorgehoben, die in erweiterten NSSPCs differenziell exprimiert werden, und ermöglichen eine weitere Identifizierung. Unser Protokoll bietet auch eine alternative Strategie, um Zelloberflächenproteine zu reinigen, indem der intrinsische Stoffwechselweg zur Kennzeichnung von Sialoglycan mit bioorthogonalen Gruppen kapert wird. Im Vergleich zu herkömmlichen Protokollen zur Reinigung von Zelloberflächenproteinen werden die Vorteile dieses Protokolls durch zwei spezifische Merkmale untermauert: 1) die Prävalenz der Sialylierung auf Zelloberflächenproteinen gewährleistet eine maximale Abdeckung des Zelloberflächenproteoms, und 2) die Reaktionsspezifität zwischen der bioorthogonalen Gruppe und ihren Liganden gewährt reinheit des erworbenen Oberflächenproteoms. So führt unser Protokoll zu einer sensibleren proteomischen Analyse bei weniger Ausgangsstoffen. Wir haben die Durchführbarkeit dieses Protokolls in primären NSPCs-Oberflächenmarkern demonstriert. Mit den entsprechenden Modifikationen an expandierenden Stammzellen in vitro kann dieser chemoproteomische Ansatz mit der Identifizierung von Oberflächenmarkern anderer Stammzelltypen kompatibel sein. Es ist bemerkenswert, dass ac4ManNAz per-O-acetyliert ist, es könnte zu künstlicher S-Glykosylierung führen. Die Verwendung von unbefleckten unnatürlichen Zuckern kann die Artefaktbildung vermeiden und die Spezifität und Gültigkeit der metabolischen Glykan-Etikettierung in lebenden Zellenverbessern 19.
Die Vorbereitung von primären kortikalen neuronalen Vorläuferzellen und Endothelzellen sind kritische Schritte des Protokolls. Erstens müssen bei der Verdauung embryonaler kortikaler Gewebe die Verdauungszeit, die Menge des Enzyms und die Stärke der Handhabung sorgfältig kontrolliert werden. Übermäßige Verdauung und mechanische Scherkräfte schädigen die Integrität von Plasmamembran- und Zelloberflächenrezeptoren, die die Signaltransduktion für das Überleben und Wachstum der Zelle vermitteln, und sie stören auch die Reaktionsfähigkeit von NSPCs auf die Stimulation von Endothelzellen und ihre Selbsterneuerungsfähigkeit. Um eine ordnungsgemäße Verdauung zu erreichen, müssen die Experimentatoren das Papain vollständig aktivieren und die Verdauung stoppen, sobald die Gewebeblöcke verschwinden. Zweitens müssen BEND3-Zellen in einem gesunden Zustand gehalten werden, um die Sekretion zu unterstützen. Es wird empfohlen, BEND3-Zellchargen mit weniger Passagen und Durchgang seder Zellen zu verwenden, bevor sie 100% Zusammenfluss erreichen. Dadurch wird verhindert, dass der Zellzyklus verhaftet und durch DNA-Schäden verursacht wird, die sich während der Passage oder durch überfüllten Kontakt zwischen den Zellen angesammelt haben.
Die Hochdurchsatz-Sequenzierungstechnologie fördert die Identifizierung von Zelloberflächenmarkern durch die Analyse der RNA-Expression, insbesondere für Zelltypen einschließlich Gewebestammzellen, die oft in vivo in Mengen vorhanden sind, die zu klein sind, um Proteomanalysen durch Massenspektrometrie. Obwohl die RNA-Seq-Analyse Gene identifizieren kann, die speziell in NSPCs exprimiert werden, spiegelt sie möglicherweise nicht wirklich den Proteinexpressionsspiegel wider, da die RNA-Expression nicht immer mit der Proteinexpression20übereinstimmt. Darüber hinaus können Nicht-Protein-Biomoleküle, die als Oberflächenmarker wirken können, nicht durch transkriptomische Studien nachgewiesen werden. Zum Beispiel ist Oligosaccharid Lewis X ein bekannter Oberflächenhersteller, der weit verbreitet ist, um menschliche embryonale Stammzellen und NSPCs zu kennzeichnen, obwohl es mit mehreren Proteinen in Verbindung gebracht werden kann21. Daher ist die direkte Massenspektrometrieanalyse noch nicht substituierbar, und die Entwicklung von Methoden, die eine Analyse der Massenspektrometrie machbarer und bequemer machen können, ist für zukünftige Studien von großem Interesse.
Neben der Sialylierung spielen andere Arten von posttranslationalen Proteinmodifikationen eine wichtige Rolle bei der Regulierung von Funktionen modifizierter Proteine. Diese Modifikationen wirken sich auf Proteineigenschaften wie Konformation, Halbwertszeit und subzelluläre Lokalisation22,23aus. Mehrere Proteinmodifikationen haben die Zelltypspezifität24,25,26. Mit dem wachsenden Inhalt der chemischen Werkzeugkiste sind mehr Modifikationstypen für die metabolische Kennzeichnung mit chemischen Reporternzugänglich 27. Daher kann der hier beschriebene chemische Ansatz verwendet werden, um andere Unterschiede in der Proteinmodifikation zwischen Stammzellen und differenzierten Zellen zu untersuchen und die molekularen Mechanismen hinter der Aufrechterhaltung der Stammzelleigenschaften und der Differenzierung zu veranschaulichen. regelung.
Offenlegungen
Die Autoren haben nichts zu machen.
Danksagungen
Abbildung 1B, 1C, 1E und 1F werden aus Bai reproduziert et al . 10 mit Genehmigung der Royal Society of Chemistry. Wir danken Yi Hao im Labor von X. C. für die Schnitte. Diese Arbeit wird von der National Natural Science Foundation of China unterstützt (Nr. 91753206 bis Q. S. und X. C., Nr. 31371093 bis Q. S., und Nr. 21425204 und 21672013 bis X. C.).
Materialien
| Name | Company | Catalog Number | Comments |
| BEND3 | ATCC | CRL-229 | |
| DMEM | Gibco | 11960044 | |
| L-glutamine | Gibco | 25030081 | 1% |
| Sodium pyruvate | Sigma | P5280 | 1% |
| N2 supplement | Gibco | 17502048 | 1 to 100 |
| N-acetyl-L-cysteine | Sigma | A7250 | 1 mM |
| Papain | Worthington | LS003726 | 10 U/mL |
| B27 supplement | Gibco | 17504044 | 1 to 50 |
| Poly-L-lysine | Sigma | P4707 | |
| Basic Fibroblast growth factor | Gibco | PHG0261 | 10 ng/mL |
| Penicillin-Streptomycin | Gibco | 15140122 | 1% |
| Fetal bovine serum | Gibco | 10099141 | 10% |
| HBSS | Gibco | 14175095 | |
| Tripsin-EDTA, 0.25% | Gibco | 25200056 | |
| DPBS | Gibco | 14190094 | |
| Transwell | Corning | 3450 | |
| Paraformaldehyde | Sigma | 158127 | 4% |
| Sucrose | Sangon | A100335 | |
| DAPI | Gibco | 62248 | |
| RIPA buffer | Thermo Scientific | 89900 | |
| SDS-PAGE loading buffer 2x | Solarbio | P1018 | |
| 6-well plate | Corning | 3335 | |
| Tris-Glycine protein gel | invitrogen | xp00100box | |
| Mouse monoclonal anti-Nestin | Developmental Study Hybridoma Bank | Rat-401 | 1 to 20 |
| Mouse monoclonal anti-beta-tubulin III | Sigma | T8860 | 1 to 1,000 |
| Alexa Fluor 488 goat anti-mouse IgG1 | invitrogen | A-21121 | 1 to 1,000 |
| Alexa Fluor 546 goat anti-mouse IgG2b | invitrogen | A-21143 | 1 to 1,000 |
| Albumin Bovine V | Amresco | 0332 | |
| Triton X-100 | Amresco | 0694 | |
| BCA assay kit | Thermo Scientific | 23225 | |
| Dimethyl sulfoxide | Sigma | D2650 | |
| Brij97 | Aladdin | B129088 | |
| CuSO4 | Sigma | 209198 | |
| Alkyne-biotin | Click Chemistry Tools | TA105 | |
| BTTAA | Click Chemistry Tools | 1236 | |
| Ac4ManNAz | Click Chemistry Tools | 1084 | 100 µM |
| 9AzSia | synthesized in lab | ||
| Sodium ascorbate | Sigma | A4034 | |
| Methanol | Sigma | 34860 | |
| EDTA | Sangon | A100322 | |
| NaCl | Sangon | A100241 | |
| SDS | Sangon | A100227 | |
| Alexa Flour 647-conjugated streptavidin | invitrogen | S21374 | 1 to 1,000 |
| Triethanolamine | Sigma | V900257 | |
| Dynabeads M-280 Streptavidin | invitrogen | 60210 | |
| Ammonium bicarbonate | Sigma | 9830 | |
| Coomassie Brilliant Blue R-250 | Thermo Scientific | 20278 | |
| Isoflurane | RWD Life Science Co. | 970-00026-00 | |
| DNase I | Sigma | DN25 | 12 µg/mL |
| Urea | Sigma | U5378 |
Referenzen
- Weissman, I. L. Stem Cells: Units of Development, Units of Regeneration, and Units in Evolution. Cell. 100, 157-168 (2000).
- Gage, F. H., Temple, S. Neural Stem Cells: Generating and Regenerating the Brain. Neuron. 80, 588-601 (2013).
- Gal, J. S. Molecular and Morphological Heterogeneity of Neural Precursors in the Mouse Neocortical Proliferative Zones. Journal of Neuroscience. 26, 1045-1056 (2006).
- Kawaguchi, A., et al. Single-cell gene profiling defines differential progenitor subclasses in mammalian neurogenesis. Development. 135, 3113-3124 (2008).
- Temple, S. The development of neural stem cells. Nature. 414, 112-117 (2001).
- Kwan, K. Y., Sestan, N., Anton, E. S. Transcriptional co-regulation of neuronal migration and laminar identity in the neocortex. Development. 139, 1535-1546 (2012).
- Kriegstein, A., Alvarez-Buylla, A. The Glial Nature of Embryonic and Adult Neural Stem Cells. Annual Review of Neuroscience. 32, 149-184 (2009).
- Wang, X., Tsai, J. W., LaMonica, B., Kriegstein, A. R. A new subtype of progenitor cell in the mouse embryonic neocortex. Nature Neuroscience. 14, 555-561 (2011).
- Taverna, E., Götz, M., Huttner, W. B. The Cell Biology of Neurogenesis: Toward an Understanding of the Development and Evolution of the Neocortex. Annual Review of Cell and Developmental Biology. 30, 465-502 (2014).
- Bai, Q. R., Dong, L., Hao, Y., Chen, X., Shen, Q. Metabolic glycan labeling-assisted discovery of cell-surface markers for primary neural stem and progenitor cells. Chemical Communications. 54, 5486-5489 (2018).
- Shen, Q., et al. Endothelial cells stimulate self-renewal and expand neurogenesis of neural stem cells. Science. 304, 1338-1340 (2004).
- Qian, X., et al. Timing of CNS cell generation: a programmed sequence of neuron and glial cell production from isolated murine cortical stem cells. Neuron. 28, 69-80 (2000).
- Varki, A. Glycan-based interactions involving vertebrate sialic-acid-recognizing proteins. Nature. 446, 1023-1029 (2007).
- Cheng, B., Xie, R., Dong, L., Chen, X. Metabolic Remodeling of Cell-Surface Sialic Acids: Principles, Applications, and Recent Advances. ChemBioChem. 17, 11-27 (2016).
- Lin, S. H., Guidotti, G. Purification of Membrane Proteins. Methods in Enzymology. 463, 619-629 (2009).
- Schmidt, J. R., et al. Pilot Study on Mass Spectrometry-Based Analysis of the Proteome of CD34+CD123+ Progenitor Cells for the Identification of Potential Targets for Immunotherapy in Acute Myeloid Leukemia. Proteomes. 6, (2018).
- Crisan, M., Dzierzak, E. The many faces of hematopoietic stem cell heterogeneity Development. Development. 144, 4195-4195 (2017).
- Uchida, N., et al. Direct isolation of human central nervous system stem cells. Proceedings of the National Academy of Sciences of the United States of America. 97, 14720-14725 (2000).
- Qin, W., et al. Artificial Cysteine S-Glycosylation Induced by Per-O-Acetylated Unnatural Monosaccharides during Metabolic Glycan Labeling. Angewandte Chemie International Edition. , (2018).
- Gry, M., et al. Correlations between RNA and protein expression profiles in 23 human cell lines. BMC Genomics. 10, 365 (2009).
- Hennen, E., et al. A LewisX Glycoprotein Screen Identifies the Low Density Lipoprotein Receptor-related Protein 1 (LRP1) as a Modulator of Oligodendrogenesis in Mice. Journal of Biological Chemistry. 288, 16538-16545 (2013).
- Seet, B. T., Dikic, I., Zhou, M. M., Pawson, T. Reading protein modifications with interaction domains. Nature Reviews Molecular Cell Biology. 7, 473-483 (2006).
- O’Brian, C. A., Chu, F. ReviewPost-translational disulfide modifications in cell signaling—role of inter-protein, intra-protein, S-glutathionyl, and S-cysteaminyl disulfide modifications in signal transmission. Free Radical Research. 39, 471-480 (2005).
- Williamson, A. J. K., Whetton, A. D. The requirement for proteomics to unravel stem cell regulatory mechanisms. Journal of Cellular Physiology. 226, 2478-2483 (2011).
- Christensen, B., et al. Cell Type-specific Post-translational Modifications of Mouse Osteopontin Are Associated with Different Adhesive Properties. Journal of Biological Chemistry. 282, 19463-19472 (2007).
- Yanagisawa, M., Yu, R. K. The expression and functions of glycoconjugates in neural stem cells. Glycobiology. 17, 57R-74R (2007).
- Best, M. D. Click Chemistry and Bioorthogonal Reactions: Unprecedented Selectivity in the Labeling of Biological Molecules. Biochemistry. 48, 6571-6584 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten