Method Article
Identification des marqueurs de surface cellulaire des cellules neurales primaires de tige et de progéniture par l'étiquetage métabolique du sialoglycan
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Présenté ici est un protocole qui combine un système de co-culture neural-endothelial in vitro et l'incorporation métabolique du sialoglycan avec des groupes fonctionnels bioorthogonals pour étendre les cellules neurales primaires de tige et d'ancêtre et étiqueter leur surface sialoglycoproteins pour l'imagerie ou l'analyse de la spectrométrie de masse des marqueurs de surface cellulaire.
Résumé
Les cellules de tige et d'ancêtre neural (NSPc) sont la base cellulaire pour les structures et les fonctions complexes du cerveau. Ils sont situés dans des niches spécialisées in vivo et peuvent être isolés et étendus in vitro,servant de ressource importante pour la transplantation cellulaire pour réparer les lésions cérébrales. Cependant, les NSPC sont hétérogènes et ne sont pas clairement définis au niveau moléculaire ou purifiés en raison d'un manque de marqueurs de surface cellulaire spécifiques. Le protocole présenté, qui a été précédemment rapporté, combine un système de co-culture neural-endothelial avec une méthode métabolique d'étiquetage de glycan pour identifier le sialoglycoproteome de surface des NSPc primaires. Le système de co-culture endothéliale du NSPC permet l'auto-renouvellement et l'expansion des CNS PNS primaires in vitro,générant un nombre suffisant de NSPC. Les Sialoglycanes dans les NSPC cultivés sont étiquetés à l'aide d'un journaliste métabolique de l'acide sialique contre nature avec groupes fonctionnels bioorthogonal. En comparant le sialoglycoproteome des NSPC auto-renouvellements élargis dans une co-culture endothéliale avec la culture neuronale différenciante, nous identifions une liste de protéines membranaires qui sont enrichies dans les NSPC. Dans le détail, le protocole comprend : 1) la mise en place d'une co-culture endothéliale du NSPC et d'une culture différenciante du CNPC; 2) étiquetage avec azidosugar per-O-acetylated N-azidoacetylmannosamine (Ac4ManNAz); et 3) conjugaison de biotine au sialoglycan modifié pour la formation image après fixation de la culture neurale ou de l'extraction de protéine de la culture neurale pour l'analyse de spectrométrie de masse. Ensuite, les candidats marqueurs de surface enrichis par le NSPC sont sélectionnés par analyse comparative des données de spectrométrie de masse provenant à la fois du NSPC élargi et des cultures neuronales différenciées. Ce protocole est très sensible pour identifier les protéines membranaires de faible abondance dans les matériaux de départ, et il peut être appliqué à la découverte de marqueurs dans d'autres systèmes avec des modifications appropriées
Introduction
Les cellules souches neurales sont définies comme une population de cellules multipotentes qui peut se renouveler pour maintenir une piscine de cellules souches et se différencier en neurones et en gliales. Ils sont les principaux types de cellules dans le système nerveux et peuvent offrir un grand potentiel thérapeutique en médecine régénérative par la transplantation cellulaire dans les cerveaux malades et blessés1,2. Au fur et à mesure que le développement se poursuit, la population de cellules souches neurales devient hétérogène3,4, et la proportion de cellules souches neurales dans le cerveau diminue progressivement5. D'une manière générale, les cellules souches neurales embryonnaires et d'autres cellules progénitrices neurales, appelées cellules souches et progénitrices neurales (NSPC), sont situées dans les zones germinales, la zone ventriculaire et la zone sous-ventriculaire chez la souris6. Dans le cerveau embryonnaire, les cellules souches neurales génèrent des neurones directement ou indirectement par l'intermédiaire des cellules progénitrices intermédiaires (CIP), et chez certaines espèces par l'intermédiaire des progéniteurs de zone sous-ventriculaire externe (ORG)7,8. La signature moléculaire spécifique, la morphologie, l'emplacement dans la niche de cellules souches, et le potentiel de différenciation déterminent tous le rôle de chaque sous-type dans l'organogenèse du cerveau et les applications cliniques9. Cependant, les marqueurs de surface cellulaire actuellement disponibles ne peuvent pas discriminer et purifier sans équivoque différents sous-types de NSPC, limitant la compréhension et l'utilisation de ces sous-types.
L'identification des marqueurs de surface primaires des NSPC est limitée par trois obstacles majeurs. Le premier est le nombre limité de cellules de NSPcC dans le tissu, ce qui rend difficile la préparation d'échantillons de protéines de surface cellulaire pour l'analyse de spectrométrie de masse commune. La deuxième limitation est la difficulté à produire des sous-types de cellules pures pour générer des données de protéines membranaires spécifiques au sous-type. Enfin, le troisième défi est le faible rapport des protéines de surface cellulaire dans les protéines cellulaires entières, ce qui entrave leur sensibilité de détection par l'analyse de spectrométrie de masse.
Pour surmonter ces problèmes, nous avons développé une approche chimioprotéomique pour enrichir sélectivement et identifier les protéines de surface cellulaire dans les NSPC primaires en étiquetant métaboliquement les sialoglycoprotéines10. Pour générer un nombre suffisant de NSPC, nous avons profité d'un protocole établi pour étendre et maintenir les NSPC embryonnaires primaires dans des états indifférenciés in vitro, en co-ccultant des NSPc avec des lignées de cellules endothéliales de cerveau de souris utilisant un support perméable insert de matrice(par exemple, transwell) système11. En revanche, les NPSC cultivés seuls sans cellules endothéliales génèrent la progéniture différenciée11,12. Ainsi, les échantillons de protéines de ces deux systèmes de culture peuvent être analysés comparativement pour identifier les protéines qui sont exprimées différemment dans les NSPC et les neurones différenciés. Comme la plupart des protéines de surface cellulaire sont modifiées par l'acide sialique13, précurseur sialique contre nature précurseur analogique N-azidoacetylmannosamine-tétraacylated (Ac4ManNAz) a été utilisé pour détourner la voie métabolique intrinsèque de sorte que endogène, nouvellement les sialoglycas synthétisés sont étiquetés avec des groupes d'azido, générant une poignée chimique14. Grâce à des réactions bioorthogonal salkyne-négociées par azido, qui conjuguent la biotine aux sialoglycans, les protéines de surface cellulaire peuvent être visualisées et enrichies pour l'identification protéomique par un fluorophore couplé à la streptavidine ou une matrice14.
Ici, nous effectuons la coloration de l'analyse de gel SDS-PAGE du sialoglycoproteome de surface des NSPC étendus dans une co-culture endothéliale et des cellules différenciantes dans un système de non-co-culture. Nous purifions également sélectivement le sialoglycoproteome de surface dans les deux systèmes de culture pour la comparaison protéomique. Notre protocole, comparé aux protocoles traditionnels de purification de surface cellulaire basé sur la centrifugation15, augmente l'efficacité d'extraction en réduisant les procédures d'extraction de protéine de surface par la conjugaison et l'affinité spécifiques d'étiquette purification. Pendant ce temps, il augmente la pureté d'extraction des protéines de surface cellulaire basée sur la prémisse que la sialylation se produit principalement aux protéines de surface cellulaire. Bien que les facteurs endothéliaux ne puissent pas bloquer complètement la différenciation des NSPC élargis, l'étude comparative entre une co-culture et une culture différenciée fournit une méthode pratique pour identifier les protéines de surface enrichies en cellules souches sans avoir besoin de analyser les protéines des PNJ purifiées par FACS16. Nous croyons que cette approche peut être appliquée aux études des protéines de surface dans d'autres systèmes avec les modifications appropriées.
Protocole
Tous les protocoles animaux utilisés dans cette étude ont été approuvés par l'IACUC (Comité institutionnel de soins et d'utilisation des animaux) de l'Université de Tsinghua et exécutés conformément aux lignes directrices de l'IACUC. L'établissement d'animaux de laboratoire de l'Université de Tsinghua a été accrédité par l'AAALAC (Association for Assessment and Accreditation of Laboratory Animal Care International). Pour la mise en scène des embryons, la mi-journée du bouchon vaginal identifié a été calculée comme jour embryonnaire 0.5 (E0.5).
REMARQUE: Toutes les cellules sont cultivées dans l'incubateur cellulaire dans des conditions de 37 oC et 5 % de CO2.
1. Préparation de la culture endothéliale de souris dans les inserts de soutien perméables
REMARQUE: Les cellules BEND3 sont maintenues selon les instructions du fabricant.
- Préparer le milieu cellulaire BEND3 (BM) en ajoutant 50 ml de FBS et 5 ml de pénicilline-streptomycine dans 500 mL de DMEM et bien mélanger.
- Aspirer le milieu du plat et laver la culture des cellules BEND3 avec 1 ml de PBS une fois. Ajouter 1 ml de 0,25 % de trypsine-EDTA dans les cellules et incuber les cellules pendant 4 min à 37 oC.
- Ajouter 1 ml de BM dans les cellules pour neutraliser la trypsine-EDTA et pipette de haut en bas doucement pour dissocier complètement les cellules. Transférer la suspension cellulaire dans un nouveau tube conique de 15 ml et granule retifugation par centrifugation à température ambiante (RT) pendant 5 min à 400 x g.
- Aspirer le supernatant du tube et resuspendre les cellules avec 9 ml de BM frais, puis ajouter 1 ml de suspension cellulaire dans un insert de soutien perméable. Ajouter un autre 2 ml de BM frais par puits à la chambre inférieure de la matrice. Continuer à la culture des cellules pour une journée.
2. Préparation de la souris primaire Cortical NSPCs Culture
-
Préparation de la plaque de culture, du milieu de digestion de papain, et du milieu de culture adhérent corticale (AM)
- Enrober les plaques de 6 puits de poly-L-lysine (PLL) en ajoutant 1 ml de solution PLL par puits dans des assiettes de 6 puits. Ensuite, incuber les plaques à RT pendant 30 min.
- Transférer la solution PLL dans un tube conique de 15 ml. Laver les assiettes 3 fois avec de l'eau distillée double. Sécher les assiettes et les mettre de côté jusqu'à utilisation.
- Préparer le milieu de digestion papane en ajoutant 50 U de papaïne, 50 L de L-glutamine, et 50 L de 100 mg/mL d'acétyl-L-cystéine dans 5 mL de DMEM. Mélanger brièvement le milieu et le réchauffer à 37 oC pendant 30 min pour l'activation des enzymes.
- Préparer le milieu de culture adhérent des cellules corticales (AM) : ajouter 500 l de L-glutamine, 500 l de pyruvate de sodium, 500 l de 100 mg/mL N-acétyl-L-Cystéine, 500 oL de N2, 1 mL de B27 et 5 l de 100 g/mL bFGF en 50 mL de DMME. Bien mélanger le milieu et le réchauffer à 37 oC avant de l'utiliser.
-
Préparation des cellules corticales cérébrales primaires et placage subséquent
- Sacrifiez une souris enceinte e10.5 chronométrée par dislocation cervicale.
REMARQUE: À E10.5, une majorité de cellules prolifèrent des NSPC dans le cortex cérébral, donnant lieu à de grands clones de descendance in vitro. - Stériliser l'abdomen à 75 % d'éthanol. Utilisez des ciseaux fins et des forceps micro-serrénés pour ouvrir l'abdomen en coupant la peau et le muscle sous-jacent le long du côté droit de la ligne médiane. Retirez l'utérus de la cavité abdominale doucement avec des forceps dentelés et coupez-le de la cavité abdominale avec des ciseaux fins.
- Laver l'utérus avec 40 ml de HBSS préréfrigéré dans un plat Petri de 10 cm. Ensuite, transférer l'utérus dans un nouveau plat Petri de 10 cm et le laver à nouveau avec 40 ml de HBSS pré-réfrigéré.
- Transférer l'utérus dans un nouveau plat Petri de 10 cm avec 40 ml de HBSS préréfrigéré. Retirer les embryons de l'utérus et de la membrane amniotique, puis couper les têtes des embryons des troncs avec des microforceps de bijoux.
- Laver les têtes avec 40 ml de HBSS pré-réfrigéré et transférer les têtes à un nouveau plat Petri de 10 cm avec 40 ml de HBSS pré-réfrigéré. Utilisez des microforceps de bijoutiers pour éplucher la peau et le cartilage couvrant les cerveaux, puis couper les cortices cérébraux et les recueillir dans un tube conique de 15 ml avec HBSS pré-réfrigéré.
- Pelleter les cortices par centrifugation pendant 3 min à 4 oC et 300 x g. Aspirez le supernatant du tube, puis ajoutez le milieu de digestion de papain activé et 15 l de 4 mg/mL DNase I dans la granule de tissu.
- Resuspendre brièvement la pastille tissulaire par un doux tourbillonnement. Incuber le tissu à 37 oC pendant 30 min. Pendant ce temps, desserrer le tissu en faisant un bref vortex toutes les 10 minutes.
REMARQUE: À la fin de la digestion, il ne devrait pas y avoir de morceaux de tissu visibles dans le tube. - Pelleter les cellules corticales par centrifugation pendant 10 min à 4 oC et 450 x g. Aspirez le supernatant du tube et lavez la pastille cellulaire avec du DMEM pré-réfrigéré. Répétez cette étape une fois.
REMARQUE: Pendant la digestion et le lavage, prenez garde de ne pas pipette les tissus et les granulés de cellules à peu près pour éviter d'endommager les cellules avec une forte force de tonte. - Aspirez le supernatant du tube puis ajoutez 1,5 ml de HBSS pré-réfrigéré dans le tube. Dissocier la pastille de cellules corticales en cellules simples avec pipetting douce. Comptez le numéro de cellule avec un hémocytomètre.
- Ajouter 2 ml de AM et 2 x 104 cellules corticales par puits dans des plaques de 6 puits. Incuber la plaque à 37 oC et 5 % de CO2 pendant 3 h pour laisser les cellules se fixer à la plaque.
- Sacrifiez une souris enceinte e10.5 chronométrée par dislocation cervicale.
3. Mise en place de Neural-endothelial Co-culture et Ac4ManNAz Labeling System
- Un jour après le placage des cellules BEND3 dans les inserts, aspirez doucement le milieu dans la chambre inférieure d'abord, puis les inserts. Laver l'enface des inserts 3 fois avec dMEM pré-chauffé. Laver la surface extérieure des inserts en rinçant avec le DMEM préréchauffé.
- Ajouter 1 ml de AM pré-chauffé dans un insert, puis transférer les inserts dans les puits avec des cellules corticales primaires. Incuber la co-culture à 37 oC et 5 % de CO2 pour 12 h.
- Dissoudre Ac4ManNAz dans DMSO pour atteindre une concentration de stock de 200 mM. 12 h après la mise en place de la co-culture neural-endothéliale, ajouter 1 'L de stock Ac4ManNAz par chambre inférieure et 0,5 'L de stock par insert dans la co-culture. Agiter les assiettes immédiatement et délicatement pour bien mélanger le milieu. Pour les cellules témoins, ajouter un volume égal de DMSO.
- Culture des cellules pendant encore 5 jours à 37 oC et 5% CO2. Préparer le AM avec 10x bFGF comme moyen de réalimentation (RM). Pendant ce temps, ajoutez 100 L de RM par insert et 200 OL de RM par chambre inférieure pour renourrir les cellules endothéliales et neurales tous les deux jours. Pendant la réalimentation, ne pas fournir Ac4ManNAz ou DMSO dans la culture.
4. Coloration immunofluorescente des sialoglycoprotéines dans les NSPC primaires élargis et les neurones différenciés
- Préparer Le complexe BTTAA-CuSO4 1 30x contenant 1,5 mM CuSO4 et 9 mM BTTAA dans de l'eau à double distillation. Préparer un tampon fraîchement biotin-conjugué 1 contenant 50 M de biotine-alkyne, 2,5 mM d'ascorbate de sodium et 1 x Complexe BTTAA-CuSO4 dans le PBS.
- Retirer les inserts des plaques de co-culture. Aspirez le milieu de culture des puits inférieurs et lavez les cellules neurales une fois avec du PBS pré-chauffé.
- Aspirez le PBS des puits. Ajouter 1 mL de solution PBS de paraformaldéhyde préréfrigérée de 4 % par puits dans les cellules et fixer les cellules à RT pendant 10 min. Ensuite, lavez les cellules 3 fois avec du PBS pré-réfrigéré.
- Aspirez le PBS des puits et ajoutez 1 ml de tampon fraîchement préparé conjugué à la biotine 1 par puits dans les cellules. Incuber les cellules à RT pendant 10 min.
- Aspirez le tampon de réaction des puits. Laver les cellules 3 fois avec du PBS. Préparer le tampon de coloration contenant 1% FBS et 1 'g/mL Alexa Fluor 647-streptavidin. Ajouter 1 ml de tampon de coloration par puits dans les cellules et incuber les cellules à RT pendant 30 min.
- Aspirez le tampon de coloration des puits et des cellules lavées 3 fois avec pbS pré-réfrigéré. Préparer le tampon de blocage contenant 5% de BSA et 0,3% de détergent non ionique-100 dans PBS. Ajouter 1 ml de tampon de blocage par puits dans les cellules et incuber à RT pendant 10 minutes.
- Préparer une solution d'anticorps primaire en diluant les anticorps anti-nestin et anti-tubulin III ensemble dans le tampon de blocage à des ratios de 1:20 et 1:1,000, respectivement. Retirez le tampon de blocage des puits et ajoutez 1 ml de solution d'anticorps primaire par puits dans les cellules. Incuber les cellules à 4 oC pendant la nuit.
- Retirez la solution d'anticorps primaire des puits. Laver les cellules 3 fois avec du PBS préréfrigéré. Préparer une solution d'anticorps secondaire en diluant Alexa Fluor 488 chèvre anti-souris IgG1, Alexa Fluor 546 chèvre anti-souris IgG2b, et DAPI ensemble en bloquant tampon à une dilution de 1:1,000. Aspirez le PBS des puits et ajoutez 1 mL de solution d'anticorps secondaire par puits dans les cellules. Incuber les cellules à RT pendant 2 h.
- Aspirez la solution d'anticorps des puits et lavez les cellules 3 fois avec du PBS pré-réfrigéré. Ensuite, les cellules sont prêtes pour la capture d'image.
5. Purification des sialoglycoprotéines des NSPC primaires élargies et des neurones différenciés
- Préparer Le BTTAA-CuSO 4 complexe2 15x de bouillon contenant 1,5 mM CuSO4 et 3 mM BTTAA dans de l'eau double distillée. Préparer le tampon de résuspension des protéines A contenant 4 % de SDS et 10 mM EDTA dans de l'eau doubledistillée; tampon de résuspension des protéines B contenant 150 mM De NaCl, 50 mM de triéthanolamine et 1 % d'éther d'oléoyle polyoxyéthylène (p. ex. Brij97) dans de l'eau distillée double avec pH 7,4. Avant l'utilisation, mélanger le tampon A:buffer B à 1:8 (vol/vol) pour préparer le tampon de résuspension des protéines. Préparer un tampon de lavage de protéines 1 contenant 2 % de SDS dans le SDPB; tampon de lavage de protéines 2 contenant 8 M d'urée en 250 mM de bicarbonate d'ammonium (ABC); et tampon de lavage de protéine 3 contenant 2.5 M NaCl dans PBS.
- Retirer les inserts des plaques de co-culture. Aspirez le milieu de culture des puits inférieurs et lavez les cellules neurales une fois avec du PBS pré-réfrigéré.
- Aspirez le PBS des puits et ajoutez 200 l de tampon RIPA préréfrigéré par puits dans les plaques. Incuber les assiettes sur la glace pendant 5 min. Recueillir la lyse protéique dans des tubes de 1,5 ml. Pelleter les débris cellulaires par centrifugation pendant 10 min à 4 oC et 12 000 x g.
- Transférer le supernatant dans de nouveaux tubes de 1,5 ml. Déterminer la concentration en protéines avec le kit BCA selon les instructions du fabricant. Ajuster la concentration en protéines à 1 mg/ml.
- Ajouter 100 M d'alkyne-biotine, 2,5 mM d'ascorbate de sodium et 1 x BTTAA-CuSO4 complexe de 2 à 1 ml de lyse protéinée et bien mélanger la solution. Incuber le mix à RT pendant 1 h.
- Transférer la solution de réaction dans 20 ml de méthanol préréfrigéré dans un tube conique de 50 ml. Bien mélanger et incuber à -30 oC pendant la nuit pour précipiter les protéines.
- Pelleter la protéine précipite par centrifugation pendant 15 min à 4 oC et 4 500 x g. Laver le granule de protéines deux fois avec 20 ml de méthanol préréfrigéré. Aspirez le supernatant du tube. Resuspendre la pastille protéique avec 4 ml de tampon de résuspension des protéines et transférer la résuspension des protéines dans un nouveau tube conique de 15 ml.
- Prenez 50 l l de perles de streptavidin et lavez-les 3 fois avec du PBS. Ajouter les perles lavées dans la résuspension des protéines. Incuber la solution à 4 oC pour 3 h sur un rotateur vertical à une vitesse de rotation de 20 tr/min.
- Laver les perles de façon séquentielle avec 6 types de tampons: tampon de lavage des protéines 1, tampon de lavage des protéines 2, tampon de lavage des protéines 3, 0,5 M ABC, 0,25 M ABC et 0,05 M ABC.
- Après le lavage, resuspendre les perles avec 20 L de PBS et transférer les perles dans un nouveau tube de 1,5 ml. Ajouter 20 l de tampon de chargement de protéines 2x dans les perles et traiter à 95 oC pendant 10 min. Les échantillons de protéines doivent ensuite être soumis à SDS-PAGE et tachés de Coomassie bleu brillant R-250 selon les instructions du fabricant. Couper les protéines dans le gel comme indiqué par Coomassie bleu brillant R-250 pour l'analyse de spectrométrie de masse.
Résultats
L'ensemble de la procédure d'expansion in vitro et d'étiquetage métabolique des NSPC embryonnaires primaires prend 6 jours (figure 1A). La qualité de la lignée cellulaire BEND3 et des NSPC primaires fraîchement isolés est la clé d'une expérience réussie. Les cellules BEND3 sont la source de facteurs solubles qui stimulent l'auto-renouvellement et la prolifération des NSPC. Il faut s'assurer que les cellules BEND3 sont exemptes de toute contamination et se divisent activement avec une mort cellulaire minimale avant de co-cultiver avec des cellules neurales. Les PNSP primaires doivent être soigneusement préparés pour éviter les dommages excessifs pendant la dissociation. Les NSPC endommagés peuvent encore croître et se différencier; cependant, ils ne sont pas en mesure de répondre aux stimuli endothéliaux bien pour maintenir la tige et se dilater. Une prudence supplémentaire doit être prise pour être aseptique pendant la culture cellulaire, car le protocole ne suggère pas l'ajout d'antibiotiques au milieu de culture primaire.
Le succès de la co-culture endothéliale amènera les NSPC à former de grands clones en forme de feuilles. Ces formes de clones en vedette deviennent évidentes au jour 4 et sont très typiques au jour 6. Dans les clones, les cellules maintiennent un contact étroit les unes avec les autres. Immunostaining avec des anticorps contre le marqueur NSPC Nestin et le marqueur neuronal 'tubulin III devrait révéler que dans le clone, la plupart des cellules sont Nestin' NSPCs et très peu sont '-tubulin III' cellules neuronales. En revanche, le pourcentage de cellules DentineMD et de cellules neuronales de la tubuline III dans les clones formés dans un système non co-culture sont presque les mêmes (Figure 1B, 1D, et 1E).
Le reporter chimique, Ac4ManNAz, est un analogue métabolique et peut être incorporé dans la voie intrinsèque de sialylation de protéine. Des doses élevées d'Ac4ManNAz sont toxiques pour les cellules. Pour chaque type spécifique de cellule, la concentration d'étiquetage de l'Ac4ManNAz doit être pré-testée pour atteindre la plus haute efficacité d'étiquetage sans cytotoxicité significative. Ici, la concentration d'étiquetage optimisée de l'Ac4ManNAz pour le NSPC primaire est de 100 M. L'évaluation combinatoire de la mort cellulaire indiquée par la morphologie cellulaire et nucléi que cette concentration d'étiquetage ne cause pas d'effets cytotoxiques évidents et est efficacement les NSPC(figure 1C et 1D). La morphologie clonale, l'auto-renouvellement et le potentiel de différenciation des NSPC dans le système de coculture endothéliale et non-co-culture ne sont pas affectés (Figure 1C, 1D, et 1E).
L'étiquetage réussi des NSPC par L'ac4ManNAz peut être examiné après conjugaison de biotine à une culture médiée par une réaction bioorthogonal entre l'azide et l'alkyne. Chaque cellule de la culture étiquetée Ac4ManNAz est tachée et visualisée avec Alexa Fluor 647-streptavidin. Aucune cellule n'est positive pour Alexa Fluor 647-streptavidin coloration dans le groupe témoin DMSO. En outre, les échantillons de protéines préparés à partir de la culture Labeled Ac4ManNAz par la conjugaison de biotine et la purification de perles de streptavidin montrent fort Coomassie signal de coloration bleu brillant dans les gels SDS-PAGE. Pendant ce temps, il n'y avait que des antécédents de coloration et des signaux de liaison non spécifiques dans les voies chargées d'échantillons de protéines du groupe témoin DMSO. Cela indique également l'étiquetage efficace des NSPC par L'ac4ManNAz (figure 1F).
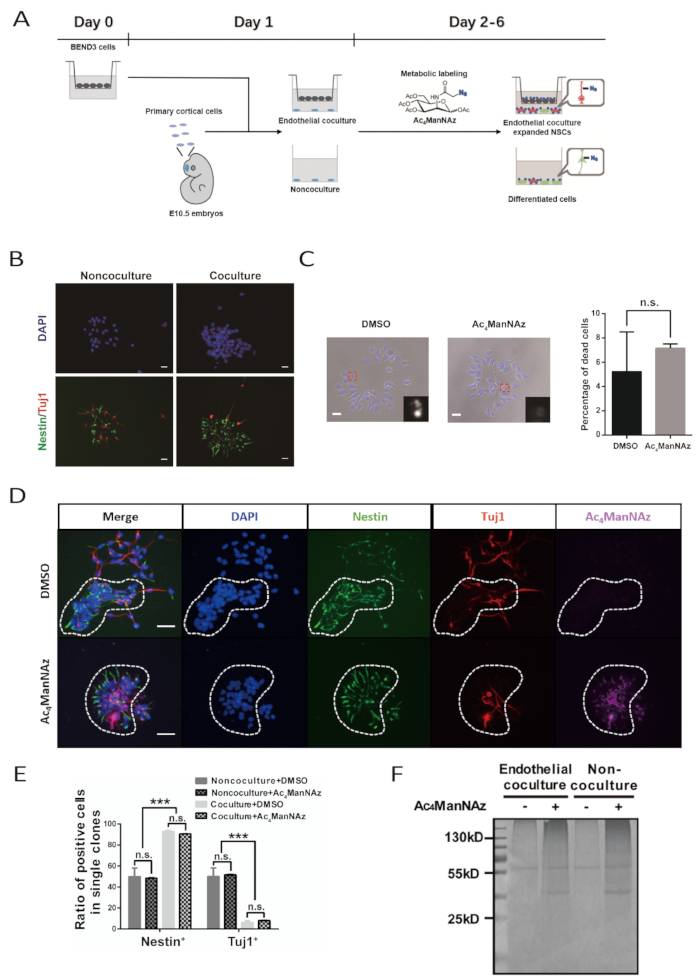
Figure 1 : Identification des marqueurs de surface cellulaire pour les NSPC primaires assistés par le système de co-culture endothéliale et l'étiquetage métabolique sialoglycan. (A) Schéma du flux de travail pour le protocole. Ce chiffre a été modifié à partir de Bai et al. 10. Les cellules BEND3 sont ensevoir dans des inserts de matrice sur D0. La préparation des NSPC corticaux primaires et la mise en place du système de co-culture sont effectuées sur D1. L'étiquetage métabolique de la culture dure de D2 à D6. Le refeeding culturel se fait sur D3 et D5. (B) Les images immunofluorescentes pour les clones formés par les NSPC primaires après la culture de 5 jours avec ou sans cellules endothéliales. La barre d'échelle indique 20 m. (C) Images de champ lumineux pour les clones formés par les NSPC primaires après une culture de 5 jours avec Ac4ManNAz ou DMSO. Les noyaux ont été contrecarrés par DAPI. La barre d'échelle indique 20 m. La barre d'erreur indique SEM (n.s. ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' (D) Les images immunofluorescentes pour NSPC ont formé des clones dans la co-culture endothéliale avec Ac4ManNAz ou DMSO. Le cercle en pointillé délimite un seul clone neuronal. La barre d'échelle indique 50 m. (E) Quantification des NSPC et des neurones différenciés dans les clones formés par les NSPC dans le système de co-culture endothéliale et non-co-culture avec l'étiquetage Ac4ManNAz ou le contrôle DMSO. La barre d'erreur indique SEM (lt; 0,0005; n.s. - non significatif). (F) Coomassie coloration bleue brillante de protéines purifiées par des perles de streptavidin à partir de cellules neurales étiquetées avec Ac4ManNAz ou DMSO dans la co-culture endothéliale et le système non-co-culture. La bande de 55 kD dans les groupes d'étiquetage témoin représente des protéines de liaison non spécifiques. (B, C, E et F) correspondant à ce protocole ont été adaptés de Bai et al. 10. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Discussion
Les marqueurs de surface sont couramment utilisés pour étiqueter et purifier des types de cellules spécifiques in vitro et in vivo17,18. La découverte de marqueurs de surface contribue grandement aux recherches sur la médecine régénérative et les cellules souches en fournissant des outils moléculaires pour enrichir sélectivement une population de cellules souches à partir de tissus normaux ou pathologiques et de plats de culture, offrant une cellule purifiée. pour une utilisation clinique ou l'étude des propriétés biologiques. Cependant, les progrès dans le développement de marqueurs de surface pour la recherche sur les cellules souches neurales ont été lents en raison de la difficulté à isoler les cellules souches des tissus primaires. Le protocole décrit ici est basé sur une plate-forme in vitro simplifiée. En comparant les NSPC primaires élargis par une co-culture endothéliale à une culture neuronale différenciante, les protéines exprimées différemment dans les NSPC élargis sont mises en évidence et permettent une identification plus approfondie. Notre protocole fournit également une stratégie alternative pour purifier les protéines de surface cellulaire en détournant la voie métabolique intrinsèque pour étiqueter le sialoglycan avec des groupes bioorthogonals. Par rapport aux protocoles traditionnels de purification des protéines de surface cellulaire, les avantages de ce protocole sont étayés par deux caractéristiques spécifiques : 1) la prévalence de la sialylation sur les protéines de surface cellulaire assure une couverture maximale du protéome de surface cellulaire, et 2) la spécificité de réaction entre le groupe bioorthogonal et ses ligands confère la pureté du protéome de surface acquis. Ainsi, notre protocole se traduit par une analyse protéomique plus sensible dans le cas de matériaux de démarrage moins. Nous avons démontré la faisabilité de ce protocole dans les marqueurs de surface des CNS PNS primaires. Avec les modifications appropriées sur l'expansion des cellules souches in vitro, cette approche chimioprotéomique peut être compatible avec l'identification des marqueurs de surface d'autres types de cellules souches. Il est à noter que comme Ac4ManNAz est per-O-acetylated il pourrait conduire à l'artificiel S-glycosylation. L'utilisation de sucres non aétélités non naturels peut éviter la formation d'artefacts et améliorer la spécificité et la validité de l'étiquetage métabolique du glycane dans les cellules vivantes19.
La préparation des cellules neurales neurales primaires et des cellules endothéliales sont des étapes critiques du protocole. Tout d'abord, lors de la digestion des tissus corticaux embryonnaires, le temps de digestion, la quantité d'enzymes et la force de manipulation doivent être soigneusement contrôlés. Une digestion excessive et des forces de cisaillement mécaniques endommageront l'intégrité de la membrane plasmatique et des récepteurs de surface cellulaire qui facilitent la transduction du signal pour la survie et la croissance des cellules, et ils perturberont également la réactivité des NSPC à la stimulation de cellules endothéliales et leur capacité d'auto-renouvellement. Pour obtenir une bonne digestion, les expérimentateurs doivent activer complètement la papaïne et arrêter la digestion dès que les blocs tissulaires disparaissent. Deuxièmement, les cellules BEND3 doivent être maintenues dans un état sain pour soutenir la sécrétion. Il est recommandé d'utiliser des lots de cellules BEND3 avec moins de passages et de passer les cellules avant qu'elles n'atteignent la confluence à 100%. Cela permettra d'éviter l'arrêt du cycle cellulaire et la sénescence causée par les dommages à l'ADN accumulés lors du passage ou par un contact surpeuplé entre les cellules.
La technologie de séquençage à haut débit stimule l'identification des marqueurs de surface cellulaire en analysant l'expression de l'ARN, en particulier pour les types de cellules, y compris les cellules souches tissulaires, qui sont souvent présentes in vivo en quantités trop petites pour effectuer l'analyse du protéome par spectrométrie de masse. Même si l'analyse RNA-seq peut identifier les gènes spécifiquement exprimés dans les NSPC, il peut ne pas vraiment refléter les niveaux d'expression des protéines, parce que l'expression de l'ARN n'est pas toujours compatible avec l'expression des protéines20. En outre, les biomolécules non protéiques qui peuvent fonctionner comme marqueurs de surface ne peuvent pas être détectées par des études transcriptomiques. Par exemple, l'oligosaccharide Lewis X est un fabricant de surface bien connu largement utilisé pour étiqueter les cellules souches embryonnaires humaines et les NSPC, même s'il peut être associé à plusieurs protéines21. Par conséquent, l'analyse directe de spectrométrie de masse n'est pas encore substituable, et le développement de méthodes qui peuvent rendre l'analyse de spectrométrie de masse plus faisable et pratique est d'un grand intérêt pour les études futures.
En plus de la sialylation, d'autres types de modifications de protéines post-traductionnelles jouent un rôle important dans la régulation des fonctions des protéines modifiées. Ces modifications affectent les propriétés protéiques telles que la conformation, la demi-vie et la localisation subcellulaire22,23. Plusieurs modifications de protéine ont la spécificité de type de cellule24,25,26. Avec le contenu croissant de la boîte à outils chimique, plus de types de modification sont favorables à l'étiquetage métabolique avec les journalistes chimiques27. Par conséquent, l'approche chimique décrite ici peut être utilisée pour étudier d'autres différences dans la modification des protéines entre les cellules souches et les cellules différenciées, illustrant les mécanismes moléculaires derrière le maintien des propriétés des cellules souches et la différenciation régulation.
Déclarations de divulgation
Les auteurs n'ont rien à divulguer.
Remerciements
Figure 1B, 1C, 1E et 1F sont reproduits à partir de Bai et coll. . 10 Ans et plus avec la permission de la Royal Society of Chemistry. Nous remercions Yi Hao dans le laboratoire de X. C. pour l'édition de chiffres. Ce travail est soutenu par la National Natural Science Foundation of China (no 91753206 à Q. S. et X. C., no 31371093 à Q. S., et nos 21425204 et 21672013 à X. C.).
matériels
| Name | Company | Catalog Number | Comments |
| BEND3 | ATCC | CRL-229 | |
| DMEM | Gibco | 11960044 | |
| L-glutamine | Gibco | 25030081 | 1% |
| Sodium pyruvate | Sigma | P5280 | 1% |
| N2 supplement | Gibco | 17502048 | 1 to 100 |
| N-acetyl-L-cysteine | Sigma | A7250 | 1 mM |
| Papain | Worthington | LS003726 | 10 U/mL |
| B27 supplement | Gibco | 17504044 | 1 to 50 |
| Poly-L-lysine | Sigma | P4707 | |
| Basic Fibroblast growth factor | Gibco | PHG0261 | 10 ng/mL |
| Penicillin-Streptomycin | Gibco | 15140122 | 1% |
| Fetal bovine serum | Gibco | 10099141 | 10% |
| HBSS | Gibco | 14175095 | |
| Tripsin-EDTA, 0.25% | Gibco | 25200056 | |
| DPBS | Gibco | 14190094 | |
| Transwell | Corning | 3450 | |
| Paraformaldehyde | Sigma | 158127 | 4% |
| Sucrose | Sangon | A100335 | |
| DAPI | Gibco | 62248 | |
| RIPA buffer | Thermo Scientific | 89900 | |
| SDS-PAGE loading buffer 2x | Solarbio | P1018 | |
| 6-well plate | Corning | 3335 | |
| Tris-Glycine protein gel | invitrogen | xp00100box | |
| Mouse monoclonal anti-Nestin | Developmental Study Hybridoma Bank | Rat-401 | 1 to 20 |
| Mouse monoclonal anti-beta-tubulin III | Sigma | T8860 | 1 to 1,000 |
| Alexa Fluor 488 goat anti-mouse IgG1 | invitrogen | A-21121 | 1 to 1,000 |
| Alexa Fluor 546 goat anti-mouse IgG2b | invitrogen | A-21143 | 1 to 1,000 |
| Albumin Bovine V | Amresco | 0332 | |
| Triton X-100 | Amresco | 0694 | |
| BCA assay kit | Thermo Scientific | 23225 | |
| Dimethyl sulfoxide | Sigma | D2650 | |
| Brij97 | Aladdin | B129088 | |
| CuSO4 | Sigma | 209198 | |
| Alkyne-biotin | Click Chemistry Tools | TA105 | |
| BTTAA | Click Chemistry Tools | 1236 | |
| Ac4ManNAz | Click Chemistry Tools | 1084 | 100 µM |
| 9AzSia | synthesized in lab | ||
| Sodium ascorbate | Sigma | A4034 | |
| Methanol | Sigma | 34860 | |
| EDTA | Sangon | A100322 | |
| NaCl | Sangon | A100241 | |
| SDS | Sangon | A100227 | |
| Alexa Flour 647-conjugated streptavidin | invitrogen | S21374 | 1 to 1,000 |
| Triethanolamine | Sigma | V900257 | |
| Dynabeads M-280 Streptavidin | invitrogen | 60210 | |
| Ammonium bicarbonate | Sigma | 9830 | |
| Coomassie Brilliant Blue R-250 | Thermo Scientific | 20278 | |
| Isoflurane | RWD Life Science Co. | 970-00026-00 | |
| DNase I | Sigma | DN25 | 12 µg/mL |
| Urea | Sigma | U5378 |
Références
- Weissman, I. L. Stem Cells: Units of Development, Units of Regeneration, and Units in Evolution. Cell. 100, 157-168 (2000).
- Gage, F. H., Temple, S. Neural Stem Cells: Generating and Regenerating the Brain. Neuron. 80, 588-601 (2013).
- Gal, J. S. Molecular and Morphological Heterogeneity of Neural Precursors in the Mouse Neocortical Proliferative Zones. Journal of Neuroscience. 26, 1045-1056 (2006).
- Kawaguchi, A., et al. Single-cell gene profiling defines differential progenitor subclasses in mammalian neurogenesis. Development. 135, 3113-3124 (2008).
- Temple, S. The development of neural stem cells. Nature. 414, 112-117 (2001).
- Kwan, K. Y., Sestan, N., Anton, E. S. Transcriptional co-regulation of neuronal migration and laminar identity in the neocortex. Development. 139, 1535-1546 (2012).
- Kriegstein, A., Alvarez-Buylla, A. The Glial Nature of Embryonic and Adult Neural Stem Cells. Annual Review of Neuroscience. 32, 149-184 (2009).
- Wang, X., Tsai, J. W., LaMonica, B., Kriegstein, A. R. A new subtype of progenitor cell in the mouse embryonic neocortex. Nature Neuroscience. 14, 555-561 (2011).
- Taverna, E., Götz, M., Huttner, W. B. The Cell Biology of Neurogenesis: Toward an Understanding of the Development and Evolution of the Neocortex. Annual Review of Cell and Developmental Biology. 30, 465-502 (2014).
- Bai, Q. R., Dong, L., Hao, Y., Chen, X., Shen, Q. Metabolic glycan labeling-assisted discovery of cell-surface markers for primary neural stem and progenitor cells. Chemical Communications. 54, 5486-5489 (2018).
- Shen, Q., et al. Endothelial cells stimulate self-renewal and expand neurogenesis of neural stem cells. Science. 304, 1338-1340 (2004).
- Qian, X., et al. Timing of CNS cell generation: a programmed sequence of neuron and glial cell production from isolated murine cortical stem cells. Neuron. 28, 69-80 (2000).
- Varki, A. Glycan-based interactions involving vertebrate sialic-acid-recognizing proteins. Nature. 446, 1023-1029 (2007).
- Cheng, B., Xie, R., Dong, L., Chen, X. Metabolic Remodeling of Cell-Surface Sialic Acids: Principles, Applications, and Recent Advances. ChemBioChem. 17, 11-27 (2016).
- Lin, S. H., Guidotti, G. Purification of Membrane Proteins. Methods in Enzymology. 463, 619-629 (2009).
- Schmidt, J. R., et al. Pilot Study on Mass Spectrometry-Based Analysis of the Proteome of CD34+CD123+ Progenitor Cells for the Identification of Potential Targets for Immunotherapy in Acute Myeloid Leukemia. Proteomes. 6, (2018).
- Crisan, M., Dzierzak, E. The many faces of hematopoietic stem cell heterogeneity Development. Development. 144, 4195-4195 (2017).
- Uchida, N., et al. Direct isolation of human central nervous system stem cells. Proceedings of the National Academy of Sciences of the United States of America. 97, 14720-14725 (2000).
- Qin, W., et al. Artificial Cysteine S-Glycosylation Induced by Per-O-Acetylated Unnatural Monosaccharides during Metabolic Glycan Labeling. Angewandte Chemie International Edition. , (2018).
- Gry, M., et al. Correlations between RNA and protein expression profiles in 23 human cell lines. BMC Genomics. 10, 365 (2009).
- Hennen, E., et al. A LewisX Glycoprotein Screen Identifies the Low Density Lipoprotein Receptor-related Protein 1 (LRP1) as a Modulator of Oligodendrogenesis in Mice. Journal of Biological Chemistry. 288, 16538-16545 (2013).
- Seet, B. T., Dikic, I., Zhou, M. M., Pawson, T. Reading protein modifications with interaction domains. Nature Reviews Molecular Cell Biology. 7, 473-483 (2006).
- O’Brian, C. A., Chu, F. ReviewPost-translational disulfide modifications in cell signaling—role of inter-protein, intra-protein, S-glutathionyl, and S-cysteaminyl disulfide modifications in signal transmission. Free Radical Research. 39, 471-480 (2005).
- Williamson, A. J. K., Whetton, A. D. The requirement for proteomics to unravel stem cell regulatory mechanisms. Journal of Cellular Physiology. 226, 2478-2483 (2011).
- Christensen, B., et al. Cell Type-specific Post-translational Modifications of Mouse Osteopontin Are Associated with Different Adhesive Properties. Journal of Biological Chemistry. 282, 19463-19472 (2007).
- Yanagisawa, M., Yu, R. K. The expression and functions of glycoconjugates in neural stem cells. Glycobiology. 17, 57R-74R (2007).
- Best, M. D. Click Chemistry and Bioorthogonal Reactions: Unprecedented Selectivity in the Labeling of Biological Molecules. Biochemistry. 48, 6571-6584 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon