Method Article
Protocole intégratif pour la génération d’organoïdes hépatiques humains 3D dérivés d’iPSC
Dans cet article
Résumé
Les organoïdes hépatiques 3D dérivés d’iPSC humains constituent un outil potentiel pour comprendre l’action de l’hormone thyroïdienne sur le développement du foie.
Résumé
L’obtention de cellules hépatiques stables en culture pose un défi important pour les études hépatiques. En gardant cela à l’esprit, une méthode optimisée est décrite en utilisant des cellules souches pluripotentes induites humaines (hiPSC) pour générer des cultures 3D d’organoïdes hépatiques humains (HHO). L’utilisation des OHH offre une approche précieuse pour comprendre le développement du foie, démêler les maladies du foie, mener des études à haut débit pour le développement de médicaments et explorer le potentiel de transplantation hépatique. Dans la première étude, à l’aide d’immunofluorescences et de techniques quantitatives de RT-PCR, la progression a été surveillée, identifiant la présence de diverses populations cellulaires, telles que les hépatoblastes et les deux types de cellules dérivées d’hépatoblastes : les cholangiocytes ou les cellules de type hépatocytaire, à différents stades de développement. Ce rapport présente un protocole 3D simple à partir des hiPSC pour acquérir des HHO qui reflètent les étapes du développement de l’embryon humain. Le protocole, d’une durée de 46 à 50 jours, comprend plusieurs étapes : (i) une gestion méticuleuse de la culture d’hiPSC pour générer des HHO, (ii) l’initiation de la différenciation cellulaire en 2D et la transition ultérieure vers la 3D, et (iii) une stratégie de dissociation optimisée pour décomposer les HHO en cellules uniques pour le séquençage de l’ARN sur cellule unique. À titre d’illustration des vastes applications de cette approche, le protocole actuel a déjà été appliqué pour démêler le rôle de la signalisation des hormones thyroïdiennes dans le développement des cellules hépatiques.
Introduction
Le foie remplit diverses fonctions métaboliques, telles que la régulation de la disponibilité de substrats énergétiques facilement utilisables comme le glucose et les corps cétoniques, ainsi que la détoxification des composés xénobiotiques. Ces dernières années, on a assisté à une augmentation significative de la prévalence des maladies du foie, largement attribuées à la stéatohépatite non alcoolique (NASH), qui, si elle n’est pas traitée, peut évoluer vers la cirrhose ou le cancer1. Par conséquent, il est impératif de comprendre les fonctions métaboliques du foie et des maladies qui y sont associées afin de faciliter le développement de traitements efficaces 2,3.
L’émergence de cultures tridimensionnelles (3D) a conduit à la création du modèle organoïde, représentant une approche révolutionnaire et innovante pour aborder la fonctionnalité et la complexité du développement des organes4. Les organoïdes sont définis comme des agrégats 3D auto-organisés de cellules différenciées qui imitent les fonctions et la cytoarchitecture de l’organe respectif5.
Au cours des dernières décennies, une myriade de protocoles d’organoïdes hépatiques humains (HHO) ont suscité un grand intérêt, allant de l’utilisation de diverses cellules humaines dérivées d’iPSC6 ou uniquement de cellules de type hépatocytes7 à l’incorporation d’une variété de microenvironnements complexes de facteurs de croissance ou d’inhibiteurs et de cellules progénitrices différenciatrices dans une monocouche7 ou 3D8. Ces approches se prêtent à une multitude d’objectifs potentiels, allant du criblage de médicaments à haut débit9 à l’obtention de connaissances plus approfondies sur les mécanismes sous-jacents aux maladies du foie10.
Ici, un protocole étape par étape de différenciation de l’OHH basé sur les indices chimiques mentionnés11 est effectué, avec des variations méthodologiques adaptées. Ce protocole commence par la manipulation et la culture appropriées de cellules souches pluripotentes induites humaines (hiPSC), détaillant les techniques de manipulation du gel de matrice extracellulaire, de passage cellulaire et de différenciation en HHO. Le processus commence par stimuler la différenciation des hiPSC en endoderme définitif (DE)12 , puis en imitant les effets in vivo du FGF et du BMP pour favoriser le développement de cellules monocouches de l’intestin antérieur postérieur (PFG)13. L’architecture 3D est réalisée au jour 10 lorsque les cellules PFG sont différenciées en une phase hépatique immature qui deviendra les hépatoblastes, la cellule précurseur fœtale des cholangiocytes et des hépatocytes2. Enfin, les structures 3D sont dissociées en cellules uniques pour les études de séquençage de l’ARN. À titre d’exemple de l’applicabilité de ce protocole, il a été démontré comment ce modèle HHO se prête à l’étude de l’action des hormones thyroïdiennes et de la déiodinase de type 2 (D2) sur le développement des hépatocytes et des cholangiocytes14.
Protocole
1. Prise en charge des hiPSC
REMARQUE : Les hiPSC (lignée cellulaire CS03iCTR-n3) ont été achetées dans le commerce. La gestion appropriée de l’enrobage de gel de la matrice extracellulaire et du milieu hiPSC est essentielle pour fixer les hiPSC aux plaques et les alimenter. Ici, les volumes nécessaires pour une plaque à 6 puits ont été décrits. Les hiPSC restants de la plaque à 6 puits, qui ne se différencient pas en organoïdes, peuvent être stockés dans de l’azote liquide pour un stockage à long terme.
- Distribution de gel de matrice extracellulaire et de milieu hiPSC
- Décongelez correctement la bouteille de gel de matrice extracellulaire en la plaçant dans un récipient avec de la glace à 4 °C pendant la nuit.
REMARQUE : Avant l’aliquotage, vérifiez le certificat d’analyse pour vérifier le volume du facteur de dilution. Cela représente la concentration en protéines du gel de la matrice extracellulaire ; Par conséquent, il varie d’un lot à l’autre. - Répartissez le gel de matrice extracellulaire dans des aliquotes appropriées en fonction du facteur de dilution (4x, 2x et 1x) à l’aide de tubes pré-refroidis et étiquetés. Ajouter un facteur de dilution de 4x de gel de matrice extracellulaire à 25 mL de DMEM/F12 froid pour enrober quatre plaques à 6 puits. Fermez rapidement les couvercles avec un film d’étanchéité de laboratoire et congelez-les à -80 °C. Évitez l’exposition prolongée à l’air et la création de bulles dans les aliquotes.
- Décongeler 100 ml de milieu de supplémentation hiPSC à 4 °C pendant la nuit. Ajoutez-le à 400 ml de milieu basal hiPSC. Une fois homogénéisé à l’aide d’une pipette sérologique de 50 mL, aliquote le milieu hiPSC dans des tubes de 40 mL et congeler à -20 °C jusqu’à utilisation.
- Décongelez correctement la bouteille de gel de matrice extracellulaire en la plaçant dans un récipient avec de la glace à 4 °C pendant la nuit.
- Revêtement de plaque pour la culture hiPSC
- Numérez et étiquetez la ou les plaques à 6 puits. Gardez une aliquote de gel de matrice extracellulaire à proximité, immergée dans de la glace sèche.
- Pour enrober une plaque à 6 puits, diluer une aliquote de gel de matrice extracellulaire (1x) dans 6,25 mL de DMEM/F12 froid (2x dans 12,5 mL et 4x dans 25 mL de DMEM/F12).
- Pipeter un petit volume de DMEM/F12 (~500 μL) dans le tube de gel de la matrice extracellulaire. Agitez de haut en bas pour décongeler et homogénéiser le gel de matrice extracellulaire avec du DMEM/F12 froid, en évitant la création de bulles, et renvoyez-le dans le reste du tube froid contenant du DMEM/F12 à l’aide d’une pipette sérologique pour mélanger complètement.
- Incorporer 1 mL de gel de matrice extracellulaire dans du DMEM/F12 dans un puits pour enrober la plaque à 6 puits (6 mL pour toute la plaque). Répartissez uniformément le 1 mL sur la surface en déplaçant, sans secouer, la plaque jusqu’à ce qu’elle soit complètement recouverte.
- Laisser reposer à température ambiante (RT) pendant 1 h avant utilisation. Si la plaque à 6 puits n’est pas entièrement utilisée, scellez-la avec un film d’étanchéité de laboratoire et conservez-la au réfrigérateur à 4 °C.
REMARQUE : Une plaque revêtue peut être conservée pendant 1 semaine au réfrigérateur à 4 °C.
- Décongélation et culture des hiPSC
- Calculer à l’avance le volume de milieu hiPSC et chaud à RT avec la plaque revêtue de 6 puits 1 h avant utilisation (2 mL par puits).
REMARQUE : En cas d’aliquotes froides ou congelées, plongez-les dans un incubateur à 37 °C ou un bain-marie 15 min avant utilisation. - Séparer 1 mL de hiPSC dans du N2liquide d’un cryoflacon en 3 puits d’une plaque à 6 puits (rapport 1:3). Pour une plaque entière à 6 puits, utilisez 2 cryoflacons. Ici, le protocole décrit la décongélation et la culture des hiPSC à partir d’un cryoflacon. Répétez le processus pour 2 cryoflacons.
- Introduisez le flacon cryogénique contenant environ 1 mL de cellules hiPSC congelées en le tenant à travers le capuchon et en le faisant glisser dans l’eau superficielle, le fond du flacon étant immergé dans un bain-marie à 37 °C jusqu’à la décongélation.
- Une fois décongelé, vaporisez le cryoflacon avec de l’éthanol et pipetez 1 mL de hiPSC. Distribuer lentement dans un tube conique vide de 15 ml. Ajouter 5 ml de hiPSC medium chaud goutte à goutte tout en secouant doucement le tube.
- Centrifuger le tube à 300 x g pendant 5 min à RT. Éliminer délicatement le surnageant en aspirant avec une pipette en verre reliée au vide sans perturber la pastille, en laissant un peu de milieu résiduel. Remettez délicatement la pastille en suspension dans 6 mL de milieu, puis ajoutez 2 mL par puits dans 3 puits de la plaque à 6 puits.
REMARQUE : Laissez de petits agrégats de hiPSC pour une croissance correcte. - Pliez la plaque à 6 puits et aspirez le gel de matrice extracellulaire de la plaque précédemment recouverte sans toucher la pointe au fond du puits.
- Transvaser 2 mL du milieu avec les hiPSC par puits. Secouez doucement la plaque vers l’avant, vers l’arrière et d’un côté à l’autre pour répartir uniformément les agrégats hiPSC sur la plaque.
- Placez la plaque à 6 puits dans un incubateur à 37 °C, 5 % de CO2 et 95 % d’humidité. Changez le milieu tous les jours (2 ml par puits), en vérifiant la croissance progressive.
- Passage des hiPSC lorsque 80 % de la confluence de croissance est acquise (après 4 à 5 jours ; voir Figure 1A). Les caractéristiques des hiPSC saines et prolifératives comprennent des frontières distinctes et un emballage serré de gros noyaux au centre des agrégations cellulaires en croissance.
- Calculer à l’avance le volume de milieu hiPSC et chaud à RT avec la plaque revêtue de 6 puits 1 h avant utilisation (2 mL par puits).
- Passage des hiPSC
- Une fois que les hiPSC atteignent une confluence de 80 %, ce qui indique environ 1,2 x10 cellules 6 par puits, passage dans une nouvelle plaque à 6 puits. Divisez selon un rapport de 1:3 (de 1 puits à 3), ou ajustez le rapport (1:4 ; 1:6) selon les exigences de l’expérience.
- Avant le passage, préparez l’enrobage de gel de matrice extracellulaire requis pour le nombre approprié de plaques à 6 puits, comme décrit à l’étape 1.2.
- Retirer le surnageant de chaque puits et ajouter 2 mL de PBS par puits. Aspirer le PBS, puis ajouter 1 mL de milieu de détachement hiPSC par puits pendant 1 min. Retirez ensuite 1 ml de milieu de détachement hiPSC.
- Placez la plaque à 6 puits dans un incubateur à 37 °C pendant 7 min. Ajouter 1 mL de milieu hiPSC par puits. Détachez et prélevez en pipetant 1 mL du milieu hiPSC avec des cellules dans le tube de 15 mL.
- Centrifuger le tube à 300 x g pendant 5 min à RT. Pour plaquer les cellules dans une plaque à 6 puits, répétez les étapes 1.3.5 à 1.3.7. Placez la plaque à 6 puits dans un incubateur à 37 °C, 5 % de CO2 et 95 % d’humidité. Passez à l’étape 1.5. pour congeler hiPSC si nécessaire.
- Congélation de hiPSC (facultatif)
REMARQUE : Le nombre de CSPh requis pour la promotion en HO peut donner lieu à des puits restants qui peuvent être stockés à long terme. S’il est nécessaire d’élargir le passage pour le stockage cryovial, les hiPSC peuvent être maintenus pendant de longues périodes.- Décongeler le volume requis de milieu de cryoconservation (1 mL par puits de cellules) dans de la glace à 4 °C. Étiquetez les cryotubes avec le nombre de passages, la date et la lignée cellulaire. Détachez les cellules avec le milieu de détachement hiPSC comme à l’étape 1.4. dans un tube conique de 15 ml.
- Centrifuger à 300 x g pendant 5 min à RT. Aspirer complètement le surnageant, en laissant soigneusement la pastille de cellule intacte. Remettre prudemment la pastille en suspension dans 1 mL de milieu de cryoconservation froid (par puits) en laissant les agrégats de hiPSC.
REMARQUE : Si les puits sont de faible densité, moins de 50 % de confluent, 1 mL de milieu de cryoconservation peut être utilisé pour chaque 2 puits. - Transvasez 1 mL de milieu de cryoconservation avec les hiPSC dans le cryotube. Agitez doucement le cryotube pour homogénéiser le contenu de la cellule.
REMARQUE : Si vous préparez plus de tubes à cryoconserver, laissez les tubes déjà préparés dans de la glace. - Placez les tubes dans le récipient de congélation à débit contrôlé à -80 °C pendant la nuit. Transférez les tubes dans le réservoir d’azote liquide après 24 h.
2. Différenciation étape par étape des hiPSC en organoïdes hépatiques
REMARQUE : La reconstitution des réactifs a été effectuée et suivie conformément aux directives du fabricant.
- Différenciation 2D de la CSPhiC en DE (jour 0-jour 4 (J-0 à J-4) ; Graphique 1B)
- Après 3 passages, si les cellules sont saines et se développent bien et rapidement, procédez à la différenciation de l’hiPSC. Enduire une plaque à 6 puits comme décrit à l’étape 1.2. Pour différencier les hiPSC en DE, utilisez le kit DE en suivant les instructions du fabricant.
- Avant de commencer la différenciation, cultivez les hiPSC dans un rapport de passage de 2:1 (de 6 puits à obtenir 3 pour commencer la différenciation) pendant 24 h pour atteindre la concentration cellulaire dans le puits indiquée dans les instructions DE.
- Effectuez la différenciation selon le protocole du fabricant avec les modifications suivantes.
- Augmenter la concentration d’Y-27632 ajoutée aux hiPSC de 10 μM à 50 μM (un jour avant de commencer la différenciation).
- Augmentez de 1,5 mL par puits le réactif de dissociation et également le volume de DMEM/F12 à la même quantité par puits que le réactif de dissociation. Ajouter 1 mL de DMEM/F12 (sur 1,5) dans chaque puits, et 0,5 mL supplémentaire accumulé dans un tube de 15 ou 50 mL.
- Différenciation des cellules DE en cellules PFG (J-4 à J-10 ; Graphique 1C)
- Préparez un milieu PFG (Advanced DMEM/F12 comme milieu basal, 1x supplément de Glutamax, 1x supplément de B27, 20 ng/mL de BMP4, 10 ng/mL de FGF2) pour la différenciation des cellules DE quelques jours avant l’induction dans le PFG et conservez-le à 4 °C.
REMARQUE : Le B27 contient du T3 dans sa composition. Dans le cas des études sur les hormones thyroïdiennes (TH), à partir de là, la B27 doit être remplacée par laB26-15 maison qui ne contient pas de TH. - Réchauffer le PFG moyen (2 mL par puits) et le DMEM/F12 avancé à 37 °C pendant 15 min ou RT pendant 1 h. Penchez-vous sur la plaque et aspirez le milieu DE des puits à l’aide d’une pipette en verre reliée à un vide (ou manuellement avec une pipette P1000) sans rayer les cellules.
- Ajouter 2 ml de DMEM/F12 avancé chaud par puits, puis aspirer. Ajouter 2 ml de PFG moyen chaud par puits dans la plaque à 6 puits. Changez le milieu tous les jours pendant les 6 prochains jours.
- Préparez un milieu PFG (Advanced DMEM/F12 comme milieu basal, 1x supplément de Glutamax, 1x supplément de B27, 20 ng/mL de BMP4, 10 ng/mL de FGF2) pour la différenciation des cellules DE quelques jours avant l’induction dans le PFG et conservez-le à 4 °C.
- Induction de cellules PFG 2D en organoïdes hépatiques immatures 3D (OHI ; D-10 à D-18)
REMARQUE : Près de 30 000 à 35 000 cellules sont nécessaires dans 30 μL ajoutés à chaque puits de la plaque ULA à 96 puits. Le rapport de passage est de 2:1, à partir de 2 puits (plaque à 6 puits) avec une monocouche de cellules PFG différenciées ; il fournira une plaque de fixation à ultra-basse température (ULA) complète de 96 puits (Figure 1D).- Avant d’initier les organoïdes 3D ou dans les premiers jours, préparez préalablement le milieu IHO (DMEM/F12 avancé comme milieu basal, 1x supplément de N2, 1x B27, 50 nM A83-01, 30 μM de dexaméthasone, 5 μM CHIR99021, 500 nM d’acide valproïque, 50 ng/mL de facteur de croissance épidermique humain (EGF), 20 ng/mL de facteur de croissance hépatocytaire humain (HGF), 40 ng/mL de Jagged-1, 300 ng/mL de N-6,20-O-dibutyryladenosine 30,50-cyclique 35 monophosphate de sodium (dbCAMP), 10 μM de nicotinamide) et stockez-le à 4 °C.
- Étiquetez une plaque à fixation ultra-basse (ULA) de 96 puits. Pour détacher les cellules PFG, utilisez l’enzyme de dissociation cellulaire selon les instructions du fabricant.
- Réchauffez l’enzyme de dissociation, le DMEM/F12 avancé et le PBS à 37 °C avant utilisation. Aspirez et jetez le milieu PFG des puits. Ajouter 2 mL de PBS par puits, puis aspirer.
- Ajouter 1,5 mL d’enzyme de dissociation cellulaire par puits à 37 °C pendant 7 minutes pour détacher les cellules du puits. Sans retirer l’enzyme de dissociation cellulaire, ajoutez 1 mL de DMEM avancé. Rassemblez toutes les cellules et ajoutez-les dans un tube de 50 ml.
- Comptez le nombre de cellules par mL à l’aide d’un compteur de cellules ou d’un hémocytomètre. Centrifugeuse à 150 x g pendant 8 min.
- À la pastille, ajoutez le volume approprié (3 ml de milieu IHO pour une plaque ULA entière de 96 puits) et 1 gel de matrice extracellulaire pour ajuster au nombre final de cellules nécessaires (30 000 à 35 000 cellules).
- Placez la plaque à 96 puits dans un incubateur réglé à 37 °C, 5 % de CO2 et 95 % d’humidité. Après 24 h (J-11), les cellules se regroupent en forme de cercle ; ajouter 50 μL par puits du milieu OHI.
- Le lendemain (J-12), augmenter à 100 μL de milieu OHI par puits. À J14, prélever ~100-120 μL de chaque puits soit avec la pipette en verre connectée à un vide, soit avec une pipette une par une. Veillez à ne pas aspirer les organoïdes. Ajouter 100 μL de milieu IHO à J-14 et J-16.
REMARQUE : Après J-16, en raison de ne pas être mis en mouvement, une croissance excessive apparaît qui disparaît après J18.
- Organoïdes d’hépatoblaste (HBO, D-18 à J-26)
REMARQUE : Dans cette étape, les OHI de la plaque ULA à 96 puits seront déplacées dans une plaque ULA à 6 puits. Pour 96 OHI de l’étape précédente, 10 organoïdes ont été placés par puits, représentant un total de 1 et 1/2 plaques ULA à 6 puits pour procéder à la maturation (Figure 1E).- Comme cela a été fait lors des étapes précédentes de différenciation, préparez le support de différenciation de HBO les jours précédents.
- Le cocktail de réactifs utilisé pour différencier les HBO est décrit à l’étape 2.3. Retirez l’acide valproïque et incorporez-les aux substances biochimiques suivantes : BMP7, BMP4 et FGF7. La composition finale est Advanced DMEM/F12 comme milieu basal, 1x supplément de N2, 1x B27, 50 nM A83-01, 30 μM de dexaméthasone, 5 μM CHIR99021, 500 nM d’acide valproïque, 50 ng/mL de facteur de croissance épidermique humaine (EGF), 20 ng/mL de facteur de croissance hépatocytaire humain (HGF), 40 ng/mL de Jagged-1, 300 ng/mL de sel de sodium monophosphate 30,50-cyclique de N-6,20-O-dibutyryladenosine 30,50-cyclique (dbCAMP), 10 μM de nicotinamide, 25 ng/mL FGF7, 50 ng/mL BMP4, 20 ng/mL BMP7.
- Réchauffer préalablement HBO medium (3 mL par puits) et Advanced DMEM/F12 (3 mL par puits) à 37 °C. Ajouter 3 ml de DMEM/F12 avancé chaud dans chaque puits.
- Répartissez 10 organoïdes OHI par puits à partir de la plaque de 96 puits avec une pointe de forage de 200 mL de large. Retirez le DMEM/F12 avancé en le penchant sur la plaque et en l’aspirant avec la pipette en verre connectée au vide (ou manuellement avec un P1000).
- Ajouter 3 ml de milieu HBO chaud par puits. Allumez et placez les plaques ULA à 6 puits sur un agitateur multiplateforme à 65 tr/min à l’intérieur de l’incubateur réglé à 37 °C, 5 % de CO2 et 95 % d’humidité. Après 4 jours (J-22) et 6 jours (J-24), remplacez le milieu HBO (3 mL par puits).
- Comme cela a été fait lors des étapes précédentes de différenciation, préparez le support de différenciation de HBO les jours précédents.
- Maturation en organoïdes hépatiques (OH) à travers 2 phases différentes (J-26 à J-46)
REMARQUE : Il s’agit du dernier processus de maturation qui se déroule en deux périodes de différenciation en raison du contenu et du type de réactifs dans le milieu.- Préparez à l’avance le premier milieu HO (HO1) et conservez-le à 4 °C. Supprimez Jagged1 et diminuez la concentration de CHIR99021 par rapport au milieu HBO. La composition finale est la suivante : DMEM/F12 avancé comme milieu basal, 1x supplément de N2, 1x B27, 500 nM A83-01, 30 μM de dexaméthasone, 2 μM CHIR99021, 50 ng/mL de facteur de croissance épidermique humaine (EGF), 20 ng/mL de facteur de croissance hépatocytaire humain (HGF), 300 ng/mL de N-6,20-O-dibutyryladenosine 30,50-cyclique 35 monophosphate de sel sodique (dbCAMP), 10 μM de nicotinamide, 25 ng/mL FGF7, 20 ng/mL BMP4, 20 ng/mL BMP7.
- Réchauffez les milieux DMEM/F12 et HO1 avancés à 37 °C avant utilisation. Dans la même plaque ULA à 6 puits, aspirez soigneusement le milieu HBO précédent en vous penchant sur la plaque et en plaçant l’extrémité de la pipette en verre reliée à un vide contre la paroi (ou manuellement avec une pipette P1000).
- Ajouter 3 ml de DMEM/F12 avancé chaud par puits et aspirer. Ajouter 3 ml de milieu HO1 chaud par puits. Changer le milieu HO1 tous les 3 jours (J-29, J-32, J-35, J-38) en même temps que le prélèvement de l’échantillon.
- À J 38, modifier le cocktail de réactifs de la seconde période de HO (HO2) en supprimant CHIR99021, BMP4, dbcAMP et FGF7 et en les remplaçant par FGF19 et DAPT. La composition finale du milieu est la suivante : Advanced DMEM/F12 comme milieu basal, 1x supplément de N2, 1x B27, 500 nM A83-01, 30 μM de dexaméthasone, 50 ng/mL de facteur de croissance épidermique humaine (EGF), 20 ng/mL de facteur de croissance hépatocytaire humain (HGF), 10 μM de nicotinamide, 20 ng/mL de BMP7, 25 ng/mL de FGF19, 5 μM DAPT.
- Réchauffer le DMEM/F12 Advanced et le milieu HO2 à 37 °C avant utilisation. Continuez avec la même plaque ULA à 6 puits et aspirez soigneusement le milieu HO1 précédent. Ajouter 3 ml de DMEM/F12 avancé par puits et retirer.
- Ajouter 3 ml de milieu HO2 chaud par puits. Changer le milieu HO2 tous les 4 jours (J-42, J-46, J-50, ...) et prélever des échantillons les mêmes jours que le milieu est changé.
3. Dissociation unicellulaire
REMARQUE : Cette étape est essentielle pour la technique de séquençage de l’ARN unicellulaire. Le nombre d’organoïdes peut varier en fonction de la taille, et plus le jour de la dissociation est ancien, plus la quantité de cellules dans les organoïdes est importante. Autrefois, le nombre d’organoïdes dissociés augmentait et les temps de dissociation diminuaient avec des quantités égales. La dissociation a été réalisée avec 10 organoïdes à J-14 et J-17, 8 organoïdes à J-23 et J-26, et 6 organoïdes à J-30 et J-45. Les procédures pour un groupe d’organoïdes sont détaillées (figure 1F).
- Préparation des réactifs
- Préparer l’Advanced-DMEM avec une concentration finale de 7,5 % d’albumine bovine fraction V (BSA) et filtrer après homogénéisation.
- Préparez 1 mL de milieu enzymatique de dissociation avec 1 mL de trypsine 0,5 % - EDTA (10x) comme milieu basal, 50 μM Y-27632, 40 U/μL d’inhibiteur de RNase et 1 U/μL de DNase I.
- Préparez le milieu d’inactivation de la trypsine avec 2 mL de DMEM/F12 avancé + 7,5 % de BSA comme milieu basal, 50 μM Y-27632, 40 U/μL d’inhibiteur de RNase et 1 U/μL de DNase I.
- Réchauffez le milieu et le PBS à 37 °C avant de commencer.
- Dissociation des OH
- Nettoyez le lieu de travail et les outils avec le nettoyant RNase. Recueillir le nombre d’organoïdes en fonction du jour de différenciation dans un tube en polypropylène de 15 mL avec une pointe grande ouverte.
- Laver avec du PBS 2x-3x. Ajouter 1 mL de milieu enzymatique de dissociation dans le tube de 15 mL avec les organoïdes collectés et maintenir à 37 °C dans une bascule à 30 tr/min pendant 5 min.
ATTENTION : Veillez à ne pas faire tomber le tube de la bascule. - Après 5 min, vérifiez d’abord que le capuchon ne contient pas d’organoïdes restants, puis perturbez mécaniquement les organoïdes avec un P1000 en tourbillonnant de haut en bas 10 fois par organoïde (60 fois au total). Répétez la même interruption avec un P200. Vérifiez le capuchon du tube au cas où les organoïdes se coinceraient dessus.
- Replacez le tube de 15 ml sur la bascule dans l’incubateur pendant encore 5 min. Répétez le processus du point 4, en commençant par faire tourner un P200 de haut en bas et en continuant avec une pipette sérologique de 1 mL reliée à un embout P10.
- Replacez le tube de 15 ml sur la bascule dans l’incubateur pendant les 5 dernières minutes. En cas d’absence de dissociation, laissez-le pendant 5 minutes supplémentaires et répétez la perturbation manuelle.
- Ajouter 1 mL de milieu d’inactivation de la trypsine et homogénéiser. Placez un nouveau tube en polypropylène de 15 ml avec une crépine à cellules de 40 μm sur le dessus. Filtrer les 2 mL (cellules avec le milieu enzymatique de dissociation et le milieu d’inactivation de la trypsine) à travers une passoire cellulaire de 40 μm dans le tube de 15 mL.
- Ajouter 1 mL de milieu d’inactivation de la trypsine pour récupérer les cellules restantes de la crépine de 40 μm.
- Prélever 5 μL de milieu unicellulaire mélangé à 5 μL d’une solution de colorant pour compter le nombre de cellules dissociées afin de procéder à leur fixation. Comptez les cellules après centrifugation et remise en suspension avec le premier réactif du kit de fixation et effectuez la filtration cellulaire à l’aide d’une crépine cellulaire de 40 μm.
Résultats
Chaque étape de ce protocole de différenciation par étapes des hiPSC en HHOs a été définie à l’aide de mesures quantitatives par qPCR et immunofluorescence de marqueurs connus spécifiques au stade de la bibliographie (Figure 2). L’étape par étape des deux techniques et les résultats obtenus en ce qui concerne la différenciation correcte en HO ont été décrits dans14. Dans l’étude précédente, les hiPSC ont été définies à partir des niveaux d’ARNm de POU5F1 (également connu sous le nom d’OCT4) et de SOX2, deux facteurs de Yamanaka16 bien connus, qui ont diminué au fil du temps (Figure 2A). Par la suite, les niveaux d’ARNm des marqueurs DE11 tels que OTX2, CER1 et FOXA2, et les marqueurs PFG11 HNF4A, CDX2 et TBX3, ainsi que l’expression localisée d’OTX2, HNF4A et TBX3 par immunofluorescence (Figure 2A,B). Après promotion 3D, pour suivre la progression de l’immunofluorescence de l’albumine et de HNF4a (Figure 2C) ; le marqueur prolifératif, MKI6717, et TBX3, important dans la régulation de l’hépatoblaste18 (Figure 2D) a été réalisé de J-18 à J-46. Notamment, l’albumine colocalisée avec HNF4a (Figure 2C) ; pendant ce temps, MKI67 est apparu sporadiquement à J-46 (Figure 2D). De plus, des cellules de type hépatocytes et cholangiocytes ont été identifiées grâce aux niveaux d’ARNm et à l’immunofluorescence de HNF4A19 et KRT718 à J-46 (Figure 2E).
Le volume des organoïdes en développement a augmenté progressivement de J-14 à J-38, comme l’indique l’augmentation de ~2 fois des niveaux d’ARNm pour le marqueur prolifératif MKI67 à J-22 par rapport à J-10. L’absence de T4 dans le milieu a entraîné une augmentation des niveaux d’ARNm MKI67 de ~20 % et ~75 % à J-14 et J-18, respectivement, par rapport au jour 10 (Figure 3A). Pour prouver la fonctionnalité de l’organoïde hépatique, les niveaux d’albumine, d’apolipoprotéine B et de A114 collectés dans les milieux des organoïdes hépatiques ont été mesurés à l’aide de l’ELISA. Aux jours J-42 et J-46, les niveaux d’ARNm ALB sont restés considérablement plus élevés chez les T4-HO par rapport aux T3-HOs ou V-Hos, respectivement de 3,0 fois et 2,5 fois (Figure 3B). D’autre part, les niveaux d’APOB étaient ~10 fois et 3 fois plus élevés (figure 3C), et les niveaux d’APOA1 étaient ~3 fois et ~2 fois plus élevés (figure 3D) à J-42 et J-46, respectivement.
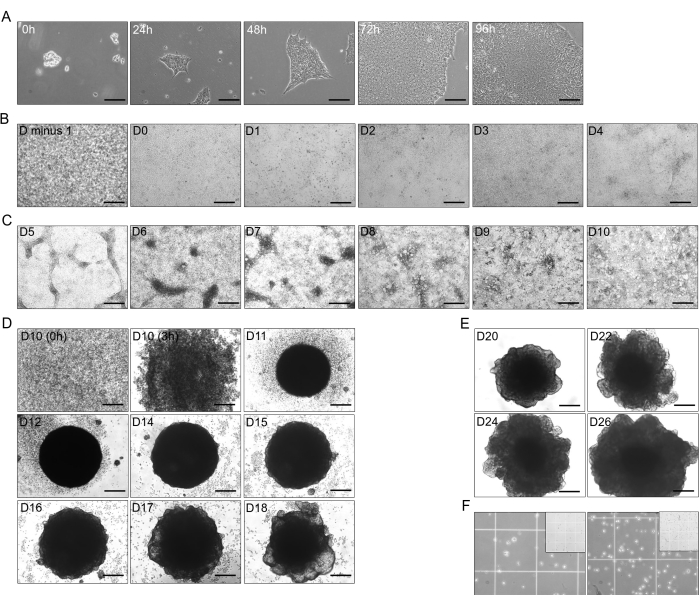
Figure 1 : Stades de différenciation des cellules hiPSC aux organoïdes hépatiques. (A) Croissance transitoire des hiPSC (ligne CS03iCTR-n3) de 0 h à 96 h, atteignant environ 70 %-80 % de confluence dans les puits d’une plaque. Barre d’échelle : 125 μm. (B) Progression de l’étape initiale de la différenciation des hiPSC en endoderme définitif (DE), montrant une confluence de 100 % la veille (J moins 1) après 24 h, et différenciation jusqu’à J4 (J-0 à J-4). Barre d’échelle : 300 μm. (C) Différenciation de l’DE (D-4) en intestin antérieur postérieur (PFG ; D-10) avec T4 ajouté B26. Barre d’échelle : 300 μm. (D) Transformation du 2D-PFG en organoïdes hépatiques immatures (OHI) 3D avec B26 ajouté en T4 à partir de J-10 sous forme de cellules uniques jusqu’à J-18, à l’exclusion de D-13. Barre d’échelle : 300 μm. (E) Entretien et croissance des organoïdes d’hépatoblaste (HBO) avec B26 ajouté en T4 à J-20, J-22, J-24 et J-26. Barre d’échelle : 300 μm. (F) Dissociation de HBO à J-26 (~2,5 x 105 cellules dans 750 μL ; à gauche) et de HO1 à J-30 (~4 x 105 cellules dans 750 μL ; à droite) en cellules simples avec B26 ajouté en T4. L’encart illustre la taille carrée de 1 mm de la chambre de Neuebauer, où le plus petit carré mesure 250 μm. Les images ont été transmises à des HHO dont la concentration libre de T4 utilisée dans les médias était de ~15 pM. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Suivi et caractérisation de la différenciation des OH. (A) Expression des niveaux d’ARNm des marqueurs hiPSC (POU5F1, SOX2), de l’endoderme défini (DE ; OTX2, CER1, FOXA2) et de l’intestin antérieur postérieur (PFG ; TBX3, HNF4A, CDX2). (iPSC, n = 8 ; DE et PFG n = 7 ; HNF4A dans PFG, n = 6). (B) Immunofluorescence au niveau de hiPSC, DE et PFG d’OTX2 (en haut, rouge), HNF4A (milieu, vert) et TBX3 (en bas, rouge). (C) « Immunofluorescence à J-18, J-26 et J-46 de l’albumine (rouge) et de HNF4A (vert). (D) Immunofluorescence à J-18, J-26 et J-46 de MKI67 (vert) et TBX3 (rouge). (E) Immunofluorescence à J-46 de l’albumine (marqueur hépatocytaire ; rouge) et KRT7 (marqueur cholangiocytaire ; vert). Les noyaux sont représentés avec du 4′,6-diamidino-2-fénilindol (DAPI). Cette figure a été modifiée au lieu de14. Veuillez cliquer ici pour l’agrandir et l’agrandir.

Figure 3 : Analyse de la maturation de l’HHO. (A) Taux relatifs d’ARNm du marqueur prolifératif MKI67 en l’absence de TH (noir) versus T4 libre à ~15 pM (rouge) dans le milieu de J-10 à J-46 (n=4 sauf T4-HOs à J-42, n=2 ; et à J-46, n=3). (B) Les niveaux d’albumine ont été mesurés dans le milieu de J-35 à J-50 dans trois conditions : absence de TH (noir), avec T4 libre à ~15 pM (rouge) et T3 libre à ~10 pM (bleu ; n = 4). (C, D) Les niveaux d’apolipoprotéine B (APOB) et A1 (APOA1) ont été mesurés dans le milieu de J-35 à J-50 dans trois conditions : absence de TH (noir) ; et avec T4 libre à ~15 pM (rouge). Données provenant de 10 organoïdes par puits (n=4). Le test t de Student bilatéral pour comparer les V-HO par rapport aux jours de T4-HOs et l’ANOVA à un facteur et le test de Tukey ont été utilisés pour les comparaisons multiples. Les données sont la moyenne des doublons, représentée par des diagrammes de points de dispersion alignés et leur moyenne. * : p<0,05 ; ** : p<0,01 ; : p<0.001 ; Réf. <0.0001. L’axe des x indique les jours de la différenciation. Cette figure a été modifiée au lieu de14. Veuillez cliquer ici pour l’agrandir et l’agrandir.
Discussion
Le protocole actuel offre divers détails méthodologiques sur la façon de manipuler les hiPSC et les cultures d’organoïdes 3D qui en découlent. Cela comprend les deux principales étapes critiques : (i) le détachement des cultures 2D puis leur développement en organoïdes hépatiques 3D après 10 jours, ainsi que (ii) la dissociation délicate d’une structure 3D en cellules uniques. Sur la base des informations disponibles, il s’agit du premier rapport d’un modèle 3D HHO pour étudier l’action des hormones thyroïdiennes, démontrant un pic d’expression de DIO2 dans les cellules de type hépatoblaste14 telles qu’identifiées à l’origine chez les souris hépatiques P120.
Comme pour de nombreuses autres techniques qui utilisent des échafaudages appliqués sur des cellules, le gel de matrice extracellulaire a été utilisé pour imiter la matrice extracellulaire21, facilitant ainsi l’assemblage dans la structure 3D caractéristique des organoïdes. La nature sensible à la chaleur de ce réactif souligne l’importance de sa manipulation pour le maintenir congelé et, lors de la manipulation, immergé dans la glace. Par conséquent, l’utilisation d’un gel de matrice extracellulaire différent est possible22, bien que les hiPSCs ne se soient pas fixées et n’aient pas proliféré dans une plaque d’absence d’échafaudage dans ce protocole, ce qui fait de sa bonne gestion un aspect crucial au début du protocole, parallèlement à la génération d’organoïdes 3D.
Après avoir terminé la différenciation des HO, une dissociation viable des organoïdes en cellules uniques représentait un autre obstacle majeur à surmonter, car une perturbation mécanique peut provoquer la mort cellulaire, d’où la qualité des données de séquençage de l’ARN23. Pour garantir des résultats optimaux, le présent protocole a montré que plus de 70 % de viabilité unicellulaire pouvait être atteinte, avec la dissociation de plus de 10cellules 5 avant et après la fixation pour un code-barres ultérieur.
Contrairement à d’autres manuscrits ne décrivant qu’un seul type de cellule dans les organoïdes7, la différenciation de l’HHO comprend au moins deux cellules dérivées d’hépatoblastes : les cellules de type hépatocytes et cholangiocytes. L’incorporation de deux réactifs responsables de leur formation, HFG24 et EGF25, ainsi que de la dexaméthasone26 qui favorise la maturation des hépatocytes, et de Jagged-118 qui active la signalisation Notch menant aux cholangiocytes. Ce réseau de signaux entrelacés dans le développement des HOs nous permet d’obtenir la structure 3D et d’imiter les expériences in vivo , en plus de surmonter l’inconvénient de la baisse de la différenciation des cultures à long terme d’hépatocytes primaires27. Il est à noter que malgré l’expression de marqueurs hépatiques adultes typiques, la présence de marqueurs fœtaux dans les phases avancées des organoïdes28 peut encore être détectée, comme le démontre ce protocole. Ceci, en fait, reflète le développement hépatique normal chez l’homme, chez qui les marqueurs hépatiques fœtaux peuvent normalement être détectés jusqu’à 1 an de vie29.
Un phénomène peu fréquent observé au cours du développement des hiPSC en HHO (en particulier pendant les périodes immatures des HOs, comme dans les derniers jours de l’OHI et de certains HBO et HO1) était l’apparition de structures kystiques surdéveloppées, qui finissaient par s’estomper une fois les plaques placées sur l’agitateur. Cette progression disproportionnée des structures kystiques suggère un noyau nécrotique à ce moment-là, la nécessité d’une irrigation plus élevéeavec des changements de milieu plus fréquents ou l’ajout de faibles volumes de gel de matrice extracellulaire pour éliminer la croissance excessive des kystes.
Contrairement à la publication originale, qui se concentrait sur un nombre accru d’organoïdes au cours de la dernière période de différenciation pour le criblage de médicaments à haut débit11, la méthode maintenant rapportée a introduit l’utilisation de plaques ULA pour favoriser la formation précoce d’organoïdes 3D de grande taille et faciliter la manipulation des HHO. Ces ajustements démontrent la souplesse et la vaste gamme d’études qui peuvent être réalisées grâce à ce protocole simple.
Déclarations de divulgation
Antonio C. Bianco est consultant pour Abbvie, Acella, Aligos, Synthonics. Les autres auteurs n’ont pas de divulgations pertinentes.
Remerciements
Ce travail a été soutenu par l’Institut national du diabète et des maladies digestives et rénales (NIDDK -DK58538, DK65066 DK77148 ; ACB).
matériels
| Name | Company | Catalog Number | Comments |
| 10 µL Universal Pipette Tips Filtered, Low rentention, Pre-sterile | VWR | 613-6462 | All procedures |
| 1000 µL Universal Pipette Tips Filtered, Low rentention, Pre-sterile | VWR | 613-6470 | All procedures |
| 15 mL Polypropilene Conical Tube | Falcon (Corning) | 352097 | Dissociation Hepatic Organoids |
| 200 µL Universal Pipette Tips Filtered, Low rentention, Pre-sterile | VWR | 613-6465 | All procedures |
| 3,3',5-Triiodo-L-thyronine | Sigma | T2877-100 | Hepatic Organoid differentiation |
| 40 μm Cell Strainer | Corning | 431750 | Dissociation Hepatic Organoids |
| 50 mL tube | Falcon (Corning) | 352070 | All procedures |
| 6 well-plate Nunc Cell-Culture Treated Multidishes | Thermo fisher scientific | 140675 | hiPSC maintenance |
| A83-01 | R&D Systems | 2939/10 | Hepatic Organoid differentiation |
| Advanced DMEM/F12 | Gibco | 12634010 | Hepatic Organoid differentiation |
| ART Wide Bore Filtered Pipette Tips | ART | 2069GPK | All procedures |
| B27 supplement | Gibco | 17504044 | Hepatic Organoid differentiation |
| BMP7 | R&D Systems | 354-BP-010/CF | Hepatic Organoid differentiation |
| Bovine Albumin Fraction V (7.5% solution) | Gibco | 15260037 | Dissociation Hepatic Organoids |
| BSA, Fraction V, Fatty Acid Free for Tissue Culture | GoldBio | A-421-100 | Dissociation Hepatic Organoids |
| CHIR99021 | R&D Systems | 4423/10 | Hepatic Organoid differentiation |
| Corning 96-well Clear Flat Bottom Ultra-Low Attachment | Corning | 3474 | Hepatic Organoid differentiation |
| Costar 6-well Clear Flat Bottom Ultra-Low Attachment | Corning | 3471 | Hepatic Organoid differentiation |
| CS03iCTR-n3 human induced Pluripotent Stem Cell line | Cedar-sinai | hiPSC maintenance | |
| DAPT | R&D Systems | 2634/10 | Hepatic Organoid differentiation |
| dbCAMP | Millipore Sigma | D0627-100MG | Hepatic Organoid differentiation |
| Dexamethasone | R&D Systems | 1126/100 | Hepatic Organoid differentiation |
| DMEM/F12 | Gibco | 11320033 | hiPSC maintenance |
| DNAse I, RNase-free, HC | Thermo Fisher scientific | EN0523 | Dissociation Hepatic Organoids |
| Falcon 10 mL Serological Pipet, Polystyrene, 0.1 Increments, Individually Packed, Sterile | Corning | 357551 | All procedures |
| Falcon 5 mL Serological Pipet, Polystyrene, 0.1 Increments, Individually Packed, Sterile | Corning | 357543 | All procedures |
| Falcon 50 mL Serological pipet, Polystyrene, 1.0 Increments, Individually Packed, Sterile | Corning | 357550 | All procedures |
| Gentle Cell Dissociation Reagent (GCDR) | Stemcell Technologies | 100-0485 | Hepatic Organoid differentiation |
| Glutamax supplement | Gibco | 35050061 | Hepatic Organoid differentiation |
| L-Thyroxine | Sigma | T1775-1G | Hepatic Organoid differentiation |
| Matrigel hESC-Qualified Matrix, LDEV-free, 5 mL | Corning | 354277 | Extracellular matrix gel |
| mFreSR | Stemcell Technologies | 5855 | hiPSC cryopreservation medium |
| mTeSR 5x Supplement | Stemcell Technologies | 100-0276 | hiPSC medium |
| mTeSR Plus | Stemcell Technologies | 100-0276 | hiPSC medium |
| Multi Platform Shaker | Fisherbrand (Thermo Fisher technologies) | 88861021 | Hepatic Organoid differentiation |
| N2 supplement | Gibco | 17502048 | Hepatic Organoid differentiation |
| Nicotinamide | R&D Systems | 4106/50 | Hepatic Organoid differentiation |
| PBS, pH 7.4 | Gibco | 10010023 | hiPSC maintenance |
| Recombinant human BMP4 | R&D Systems | 314-BP-010/CF | Hepatic Organoid differentiation |
| Recombinant human EGF | R&D Systems | 236-EG-200 | Hepatic Organoid differentiation |
| Recombinant human FGF basic/FGF2/bFGF | R&D Systems | 233-FB-010/CF | Hepatic Organoid differentiation |
| Recombinant human FGF19 | R&D Systems | 959-FG-025/CF | Hepatic Organoid differentiation |
| Recombinant human HGF | R&D Systems | 294-HG-005/CF | Hepatic Organoid differentiation |
| Recombinant Human Jagged-1 Fc Chimera | R&D Systems | 1277-JG-050 | Hepatic Organoid differentiation |
| Recombinant human KGF/FGF7 | R&D Systems | 251-KG-010/CF | Hepatic Organoid differentiation |
| ReLeSR | Stemcell Technologies | 100-0483 | hiPSC detaching medium |
| RNAse Inhibitor Ambion, cloned, 40 U/μL | Invitrogen | AM2682 | Dissociation Hepatic Organoids |
| RNase Zap | Invitrogen | AM9780 | Dissociation Hepatic Organoids |
| Sorvall Legend XT/XF Centrifuge Series | Thermo Fisher Scientific | 75004539 | All procedures |
| STEMdiff Definitive Endoderm Kit | Stemcell Technologies | 5110 | Hepatic Organoid differentiation |
| Trypan Blue solution (0.4%) | Gibco | 15250061 | Dye solution |
| TrypLE Express Enzyme | Gibco | 12604013 | Cell Dissociation enzyme |
| Trypsin 0.5% - EDTA (10X) | Gibco | 15400054 | Dissociation Hepatic Organoids |
| Valproic acid, sodium salt | R&D Systems | 2815/100 | Hepatic Organoid differentiation |
| Vari-Mix Platform Rocker | Thermo Fisher scientific | M79735Q | Dissociation Hepatic Organoids |
| Y-27632 dihydrochloride | R&D Systems | 1254 | Hepatic Organoid differentiation |
Références
- Peiseler, M., et al. Immune mechanisms linking metabolic injury to inflammation and fibrosis in fatty liver disease - novel insights into cellular communication circuits. J Hepatol. 77 (4), 1136-1160 (2022).
- Gordillo, M., Evans, T., Gouon-Evans, V. Orchestrating liver development. Development. 142 (12), 2094-2108 (2015).
- Lemaigre, F. P. Mechanisms of liver development: concepts for understanding liver disorders and design of novel therapies. Gastroenterology. 137 (1), 62-79 (2009).
- He, C., et al. Liver organoids, novel and promising modalities for exploring and repairing liver injury. Stem Cell Rev Rep. 19 (2), 345-357 (2023).
- Lancaster, M. A., Knoblich, J. A. Organogenesis in a dish: modeling development and disease using organoid technologies. Science. 345 (6194), 1247125 (2014).
- Takebe, T., et al. Vascularized and functional human liver from an iPSC-derived organ bud transplant. Nature. 499 (7459), 481-484 (2013).
- Si-Tayeb, K., et al. Highly efficient generation of human hepatocyte-like cells from induced pluripotent stem cells. Hepatology. 51 (1), 297-305 (2010).
- Wu, F., et al. Generation of hepatobiliary organoids from human induced pluripotent stem cells. J Hepatol. 70 (6), 1145-1158 (2019).
- Shinozawa, T., et al. High-fidelity drug-induced liver injury screen using human pluripotent stem cell-derived organoids. Gastroenterology. 160 (3), 831-846.e10 (2021).
- Ouchi, R., et al. Modeling steatohepatitis in humans with pluripotent stem cell-derived organoids. Cell Metab. 30 (2), 374-384.e6 (2019).
- Ramli, M. N. B., et al. Human pluripotent stem cell-derived organoids as models of liver disease. Gastroenterology. 159 (4), 1471-1486.e12 (2020).
- Shen, M. M. Nodal signaling: developmental roles and regulation. Development. 134 (6), 1023-1034 (2007).
- Ang, L. T., et al. A roadmap for human liver differentiation from pluripotent stem cells. Cell Rep. 22 (8), 2190-2205 (2018).
- Hidalgo-Álvarez, J., Salas-Lucia, F., Vera Cruz, D., Fonseca, T. L., Bianco, A. C. Localized T3 production modifies the transcriptome and promotes the hepatocyte-like lineage in iPSC-derived hepatic organoids. JCI Insight. 8 (23), e173780 (2023).
- . B27 Supplement Available from: https://www.weizmann.ac.il/molgen/hanna/sites/molgen.hanna/files/users/user52/HANNA-LAB-B22-B27-PROTOCOL-V3.pdf (2016)
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Zhu, X., Sun, J. CircHIPK3 regulates melanoma cell behaviors by binding with miR-215-5p to upregulate YY1. Mol Cell Probes. 53, 101644 (2020).
- Guan, Y., et al. Human hepatic organoids for the analysis of human genetic diseases. JCI Insight. 2 (17), e94954 (2017).
- Walesky, C., Apte, U. Role of hepatocyte nuclear factor 4α (HNF4α) in cell proliferation and cancer. Gene Expr. 16 (3), 101-108 (2015).
- Fonseca, T. L., et al. Hepatic inactivation of the type 2 deiodinase confers resistance to alcoholic liver steatosis. Alcohol Clin Exp Res. 43 (7), 1376-1383 (2019).
- Kleinman, H. K., Martin, G. R. Matrigel: basement membrane matrix with biological activity. Semin Cancer Biol. 15 (5), 378-386 (2005).
- Kozlowski, M. T., Crook, C. J., Ku, H. T. Towards organoid culture without Matrigel. Commun Biol. 4 (1), 1387 (2021).
- Arceneaux, D., et al. A contamination focused approach for optimizing the single-cell RNA-seq experiment. iScience. 26 (7), 107242 (2023).
- Matsumoto, K., Nakamura, T. Hepatocyte growth factor: molecular structure and implications for a central role in liver regeneration. J Gastroenterol Hepatol. 6 (5), 509-519 (1991).
- Kimura, M., Moteki, H., Ogihara, M. Role of hepatocyte growth regulators in liver regeneration. Cells. 12 (2), 208 (2023).
- Michalopoulos, G. K., Bowen, W. C., Mulè, K., Luo, J. HGF-, EGF-, and dexamethasone-induced gene expression patterns during formation of tissue in hepatic organoid cultures. Gene Expr. 11 (2), 55-75 (2003).
- Kaur, I., et al. Primary hepatocyte isolation and cultures: Technical aspects, challenges and advancements. Bioengineering (Basel). 10 (2), 131 (2023).
- Baxter, M., et al. Phenotypic and functional analyses show stem cell-derived hepatocyte-like cells better mimic fetal rather than adult hepatocytes. J Hepatol. 62 (3), 581-589 (2015).
- Blohm, M. E., Vesterling-Hörner, D., Calaminus, G., Göbel, U. Alpha 1-fetoprotein (AFP) reference values in infants up to 2 years of age. Pediatr Hematol Oncol. 15 (2), 135-142 (1998).
- Liu, Q., Zeng, A., Liu, Z., Wu, C., Song, L. Liver organoids: From fabrication to application in liver diseases. Front Physiol. 13, 956244 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon