Method Article
Reprogrammation du liquide amniotique primaire et cellules de Membrane de pluripotence dans des Conditions sans Xeno
Dans cet article
Résumé
Ce protocole décrit la reprogrammation des primaires amniotique fluide et membrane de cellules souches mésenchymateuses sur les cellules souches pluripotentes induites en utilisant une approche épisomiques sans intégration dans des conditions totalement chimiquement définies. Les procédures d’extraction, la culture, la reprogrammation et la caractérisation des cellules souches pluripotentes induites qui en résulte par une méthodologie rigoureuse sont reprises.
Résumé
Thérapies à base cellulaire autologues obtenu un pas de plus vers la réalité avec l’introduction de cellules souches pluripotentes induites. Les cellules souches fœtales, tels que le liquide amniotique et la membrane des cellules souches mésenchymateuses, représentent un type unique de cellules non différenciées avec promesse en génie tissulaire et de reprogrammation en iPSC pour futures interventions pédiatriques et cellules souches bancaire. Le protocole présenté ici décrit une méthode optimisée pour l’extraction et la mise en culture primaire amniotique fluide et membrane de cellules souches mésenchymateuses et générant épisomiques induite par les cellules souches pluripotentes de ces cellules en culture entièrement chimiquement définie conditions utilisant la vitronectine recombinante humaine et le milieu E8. Caractérisation des nouvelles lignes en appliquant des méthodes rigoureuses – cytométrie en flux, imagerie confocale, formation de tératome et profilage transcriptionnel – est également décrite. Les lignes nouvellement générés expriment des marqueurs de cellules souches embryonnaires – Oct3/4 a, Nanog, Sox2, TRA-1-60, TRA-1-81, SSEA-4 – tout en étant négatif pour le marqueur SSEA-1. Les lignées de cellules souches forment des tératomes chez la souris scid-beige dans 6 à 8 semaines et les tératomes contiennent des tissus représentatifs de tous les trois couches de germe. Profilage transcriptionnel des lignes par envoi de données microarray expression globale à un algorithme d’évaluation de pluripotence bioinformatic réputé toutes les lignes pluripotentes et par conséquent, cette approche est une alternative intéressante à l’expérimentation animale. Les nouvelles lignes iPSC utilisable facilement en aval expériences portant sur l’optimisation de la différenciation et l’ingénierie tissulaire.
Introduction
La technologie des cellules souches pluripotentes induites (iPSC) apporte sur les thérapies de remplacement cellulaire potentielle, maladie et modélisation du développement et drogues dépistage toxicologique1,2,3. Les thérapies de remplacement est possible sur le plan conceptuel par injection de cellules, différenciées implantation tissulaire (par exemple cardiaque "patches"), in vitro, ou régénération guidée au moyen de l’ingénierie tissulaire. Liquide amniotique (AFSC) et la membrane des cellules souches (AMSC) sont une excellente source de cellules pour ces interventions soit directement4,5,6,7 ou comme une population de cellules de départ pour la reprogrammation dans la pluripotence8,9,10,11,12.
Premières approches utilisées systèmes de culture non défini ou reprogrammation méthodes nécessitant l’intégration génomique comportent des constructions9,10,11,12. Une étude plus récente a employé un milieu exempt de xeno, même si une matrice d’attachement membrane basale moins définie (BMM) a utilisé, pour générer l’iPSC de cellules épithéliales du fluides amniotique. Cependant, l’analyse de formation tératome ne figurait pas dans l’étude avec une richesse d’in vitro et les données moléculaires. Cellules épithéliales fluides amniotiques ont été trouvés pour avoir une plus grande efficacité reprogrammation environ 8 fois par rapport aux fibroblastes néonatale13. Dans une autre étude, les cellules souches mésenchymateuses du liquide amniotique se retrouvent également à être reprogrammé dans iPSC avec un beaucoup l’efficacité supérieure12.
Cellules souches pluripotentes peuvent être différenciées en représentant de tissus de tous les 3 couches de germe et donc le potentiel plus large. Les patients pédiatriques pourraient bénéficier de la récolte, la reprogrammation et l’ingénierie tissulaire de leurs cellules de tige fluides amniotiques autologues avant la naissance et cellules de membrane amniotique souches durant la période périnatale. En outre, le niveau relativement faible de la différenciation de cellules souches fœtales (inférieurs à14,de cellules souches adultes15) pourrait théoriquement aider dans la lutte contre la rétention observée de partialité épigénétique des cellules source l’iPSC16.
Nous présentons un protocole pour la reprogrammation du liquide amniotique et membranaires des cellules souches pluripotence dans défini chimiquement milieu E8 xeno-sans le recombinant vitronectine17 (VTN) à l’aide de plasmides épisomiques18. Le principal avantage des cellules du liquide et la membrane amniotique comme source de cellules pour la reprogrammation réside dans leur disponibilité avant et Pendant la période périnatale et donc cette démarche profiterait principalement recherche en génie tissulaire pédiatrique.
Protocole
Le protocole suit les directives institutionnelles du Comité d’éthique pour la recherche. Consentement écrit du patient a été obtenue pour utiliser le liquide amniotique pour la recherche.
Ce protocole suit les politiques de l’animalier institutionnel et le Comité d’urbanisme de l’Université de South Alabama.
1. isolement et Culture des cellules souches mésenchymateuses primaires amniotique
-
Placage de cellules du liquides amniotique
- Obtenir un minimum de 2,5 mL de liquide amniotique récolté dans le processus de l’amniocentèse par un médecin.
Remarque : Tous les traitement de tissus et de cellules vivantes doit être exécutés dans une armoire de culture de tissus stériles et des équipements de protection individuelle approprié doivent être utilisé. Familiarité avec la culture cellulaire fondamentale et technique stérile est nécessaire. - Préparer le milieu culture cellulaire (AFMC) fluide et membrane amniotique : EBM-2 milieu basal, 15 % sérum fœtal (SVF), 20 ng/mL de protéine, 25 ng/mL d’EGF, 10 ng/mL de l’IGF. Pour la culture de cellules souches primaire de membrane amniotique mais aussi fluide amniotique, le milieu devrait être complété par une solution antibiotique-antimycosiques.
- Jour 0, mélanger 2,5 mL de liquide amniotique avec 3,5 mL de milieu de culture AFMC et plaque dans une fiole de T25. Incuber à 37 ° C et 5 % de CO2 pendant au moins 48 h d’intact avant de vérifier la présence de colonies.
- Le jour 5, les colonies de cellules adhérentes doivent être présents. Balançant doucement le flacon pour déloger les cellules qui ne pas pleinement respecter les bas et les débris et vide-aspirer le mélange de fluide amniotique/moyen usé à l’aide d’une pipette Pasteur. Remplacer par 5 mL de milieu AFMC frais.
- Culture pendant encore 5 jours. Moyen de changement tous les deux jours comme indiqué au point 1.1.4.
- Obtenir un minimum de 2,5 mL de liquide amniotique récolté dans le processus de l’amniocentèse par un médecin.
-
Isolement de primaire des cellules souches mésenchymateuses de l’amnios humain
- Obtenir des placentas dès que possible suivant la naissance, dans les 24 heures au plus tard, pour maximiser l’intégrité cellulaire. Couper un segment2 de 9 cm de l’amnios, enlever les caillots de sang et laver dans un tube à centrifuger 50 mL avec 30 mL de PBS additionné de solution antibiotique-antimycosiques.
- Émincer les membranes en utilisant une paire de scalpels de belles pièces dans un plat de vitroplants stérile de 10 cm. La digestion des membranes et l’extraction des cellules seront fera à l’aide d’un système de dissociation tissulaire. Suivre le protocole du fabricant.
NOTE : plus les pièces du tissu après hachage, plus le nombre de cellules récupéré après la digestion. - Transfert de la masse de tissu membrane hachées avec les lames de bistouri Swann-Morton dans tube de dissociation d’un tissu et mélanger avec 4,7 mL du milieu RPMI 1640. Incorporer les enzymes de dissociation (voir Table des matières).
- Monter les tubes sur le dissociator tissulaire et exécuter le programme « h_tumor_01 ». Incuber les tubes à 37 ° C sur une plate-forme bascule pendant 30 min.
- En outre, diluer les suspensions avec 35 mL de milieu RPMI 1640 et s’appliquent à un 70 µm/filtre placé sur un tube à centrifuger collection 50 mL.
- Centrifuger 5 min à 200 x g à la température ambiante, jeter surnageant, resuspendre le culot dans 5 mL de milieu RPMI 1640, compter les cellules à l’aide d’un Hémacytomètre et plaque à une densité de 10 000 cellules/cm2 dans des vaisseaux de culture de tissus traités avec fraîchement préparé AFMC additionné à la solution antibiotique-antimycosiques.
Remarque : Dans le cas d’une digestion incomplète, petits morceaux de tissu sera présents et cellules individuelles seront rares. Tourner de nouveau vers le bas et le culot de tout la plaque dans une fiole de T75.
-
Culture de primaire AFSC et CSEM
- Colonies de passage du CSFA/CSEM par aspiration-aspiration a passé à l’aide d’une pipette Pasteur à moyen et ajouter 2 mL d’enzyme détachement cellulaire dans le ballon. Incuber à 37 ° C pendant 5-8 min.
- Taper sur le ballon pour aider à déloger les cellules et mélanger la suspension avec un volume égal de support de l’AFMC (supplément antibiotique-antimycosiques ne devrait pas être nécessaire à partir de ce moment). Centrifuger à 200 g pour 4-5 min. Retirez le surnageant à l’aide soit une pipette Pasteur en verre ou tout simplement par le biais de renversant le tube et le vider dans un conteneur à déchets.
- Feuilleter le bas du tube de centrifugeuse pour casser vers le haut de l’extrait concentré en suspension monocellulaire dans la goutte reste de liquide et mélanger avec le milieu de l’AFMC pour l’électrodéposition. Plaque en T-flacons à une densité entre 2 500 et 5 000 cellules/cm2.
- Moyen de changement tous les deux jours. Pas la culture les lignées cellulaires au-delà de passage 6. Pour la reprogrammation des fins, utilisez aussi bas un passage possible.
- Préparer les stocks congelés de l’AFSC et AMSC comme sauvegardes à l’aide de congélation moyen. Récolter les cultures à l’aide d’une enzyme cellulaire de détachement, centrifuger à 200 g pour 4 ° C pendant 5 min.
- Retirez le surnageant à l’aide d’une pipette Pasteur et effleurer le fond du tube au singulier les cellules dans le culot. Remettre en suspension dans le milieu de congélation complet à une densité de 1 × 106par millilitre et partie aliquote dans cryovials. Stocker dans un récipient froid pendant la nuit à-80 ° C. Réglez à l’azote liquide pour le stockage à long terme.
2. reprogrammation dans pluripotence
- Obtenir les plasmides reprogrammation
- Achetez les plasmides reprogrammation via un référentiel de plasmide à but non lucratif. Un accord de transfert de matériel est nécessaire.
- Transformer les plasmides en cellules compétentes d’Escherichia Coli et isoler les plasmides à l’aide d’un kit d’extraction de plasmide commerciale. Suivez les instructions du fabricant.
- Mesurer la concentration d’ADN à l’aide d’un spectrophotomètre plasmidique. Viser une concentration élevée de plasmide qui en résulte, idéalement environ 1 µg/µL à éviter la dilution de l’échantillon au cours de la transfection.
- Mesurer les concentrations des plasmides individuels à l’aide d’un spectrophotomètre UV et l’aliquote eux individuellement.
- Mélangez ensemble 3 µg, 3 µg et 2 µg de plasmides EN2K, ET2K et M2L, respectivement. Il s’agit de la solution de plasmide reprogrammation. La quantité de solution de plasmide est suffisant pour transfecter 1 x 106 cellules. Préparer plusieurs de ces parties aliquotes.
- Stocker tous les aliquotes à-80 ° C.
- Préparer des plats de culture cible
- Recouvrir une plaque de 6 puits avec vitronectine – ajouter 1 mL de tampon de dilution de la vitronectine dans chaque puits et mélanger dans 40 µL de la solution mère de VTN (1 µg/cm2). Laisser à température ambiante (RT) ou dans l’incubateur à 37 ° C pendant 1 h.
- Vide-aspirer la solution à l’aide d’une pipette Pasteur et remplacez par 2 mL de milieu de l’AFMC dans chaque puits. Entreposer à 37 ° C jusqu'à ce que les cellules doivent être plaquées.
NOTE : Important : support de l’AFMC utilisé dans cette étape ne doit contenir aucune solution antibiotique ou antimycosique.
- Récoltes cultivées CSFA/CSEM primaire
NOTE : Développez l’AFSC/CSEM en culture assez pour faire des stocks congelés à un certain nombre de passage bas et dédier un flacon de T-75 pour la reprogrammation. Depuis aussi peu que 100 000 cellules sont suffisantes pour une expérience, le but de récolter environ 500 000 cellules pour compenser les pertes et si l’optimisation des paramètres de transfection ou les conditions de culture différentes est à tester.- Le plus tôt à un passage bas, récolter le CSFA/CSEM à l’aide de mélange enzymatique de cellule détachement comme indiqué au point 1.3.1 à 1.3.2. Après que les cellules ont été centrifugés, passez à l’étape suivante.
- Remettre le culot dans 1 mL de PBS et bien mélanger pour laver des composants du sérum. Compter les cellules à l’aide d’un Hémacytomètre. Ajuster la densité de la cellule à 100 000/mL de PBS et partie aliquote dans des tubes de microcentrifuge de 1,5 mL. Cela ne garantit qu’un temps de contact minimal entre les cellules et de la mémoire tampon utilisée pour la transfection.
- Placer le tube de microcentrifuge au sommet des tubes de polystyrène de 5 mL (comme les adaptateurs, permettra de centrifugation dans un rotor swing régulier) et centrifuger à 200 x g pendant 4 min à température ambiante. Inverser les tubes et éliminer le surnageant dans un conteneur à déchets. Ne pas utiliser un rotor à angle fixe.
- Effectuer une étape supplémentaire de centrifugation à 200 x g pendant 3 min à température ambiante. Ceci permettra le restant liquide des parois du tube pour recueillir au fond. Soigneusement aspirer tout cela à l’aide d’une pipette de 200 µL.
- Transfection avec reprogrammation des plasmides
- Pour la reprogrammation des expériences, un système de transfection (voir Table des matières) servira à fournir la reprogrammation des plasmides dans les cellules. Placez les conseils de transfection, tubes de transfection, tampon de resuspension et tampon électrolytique dans l’armoire de culture de tissus. Les réactifs du kit sont conservés au RT jusqu'à ce qu’ils sont ouverts, puis ils sont stockés à 4 ° C.
Remarque : Nous utilisons la version de 10 µL de la trousse. - Déplacer l’appareil de transfection près pour que sa station de métro peut être placée directement dans l’armoire. Remplir un tube de transfection avec 3 mL de tampon électrolytique et monter le tube dans la station en le poussant tout le chemin à l’intérieur de la fente.
- Prendre le plasmide reprogrammation aliquotes de solution préparées en 2.1.5 sortir de stockage de-80 ° C et laissez-les décongeler à RT dans la culture du cabinet.
- Sur le périphérique de transfection, sélectionnez les paramètres suivants de transfection : 950 V, 40 ms et 1 impulsion.
- Resuspendre le culot contenant 100 000 cellules dans 10 µL de tampon de resuspension. Travailler rapidement de ce point sur étant donné que le tampon de resuspension est légèrement toxique et un temps de pose accrue se traduit par une viabilité cellulaire sensiblement plus bas.
- Mélanger à 1/10 de la solution de plasmide reprogrammation (l’aliquote de solution a été préparée pour un total de 1 x 106 cellules).
- Monter un embout de transfection sur la pipette de transfection.
- Aspirer la suspension cellulaire dans la pointe de la transfection avec précaution, en évitant la formation de bulles d’air. Si on observe des bulles, expulser de la suspension et répéter l’aspiration. Bulles d’air n’entravera la transfection.
- Introduire la pipette de transfection dans le tube de transfection et appuyez sur le bouton « START » sur l’écran de l’appareil de transfection. Attendez le message de l’écran informant sur le succès de la transfection et retirez immédiatement la pipette du tube.
- Expulsez la suspension dans 1 puits de la plaque de 6 puits cible établie à la section 2.2. Mélanger dans le milieu d’un voisins bien et répartir la suspension égale dans les deux puits (la densité de cellules qui en résulte sera 50 000/puits).
- Répétez la transfection pour tous les tubes de microcentrifuge contenant CSFA/CSEM individuellement. Placer la plaque dans l’incubateur à 37 ° C et 5 % de CO2.
- Pour la reprogrammation des expériences, un système de transfection (voir Table des matières) servira à fournir la reprogrammation des plasmides dans les cellules. Placez les conseils de transfection, tubes de transfection, tampon de resuspension et tampon électrolytique dans l’armoire de culture de tissus. Les réactifs du kit sont conservés au RT jusqu'à ce qu’ils sont ouverts, puis ils sont stockés à 4 ° C.
- Culture de l’AFSC/CSEM transfectée
- La culture des cellules transfectées pendant 2 à 5 jours. Puis passer à la reprogrammation milieu composé de E8 additionné de 100 µM du butyrate de sodium au jour 3.
Remarque : Le passage secondaire peut être effectuée pour éviter la prolifération de la source de l’AFSC/CSEM. Toutefois, le passage désactive l’option de calculer l’efficacité reprogrammation correctement si ce paramètre est d’un intérêt. - Changer le milieu reprogrammation tous les jours à tous les deux jours pendant 10 jours. Changer le support de tous les jours du jour 10 sur.
- La culture des cellules transfectées pendant 2 à 5 jours. Puis passer à la reprogrammation milieu composé de E8 additionné de 100 µM du butyrate de sodium au jour 3.
- Cueillette manuelle des colonies entièrement reprogramed pour l’expansion clonale
- Entièrement reprogrammés colonies apparaissent autour du jour 14. Permettre aux colonies d’étendre dans la taille et deviennent compact. Ils peuvent être manuellement cueillis et transférés aux plaques fraîches dès le jour 15-16.
- 1 h avant la procédure de prélèvement, enduire plaques 24 puits 8 µL de VTN dans 300 µL de tampon de dilution de la vitronectine / puits (1 µg/cm2) et incuber à RT ou 37 ° C. Remplacer la solution avec E8 moyen sans le butyrate de sodium.
- Sélectionner les colonies d’une taille suffisante (idéalement plus de 400 µm de diamètre) dans une armoire de culture stérile. Un microscope à contraste de phase ou un stéréomicroscope peut être utilisé.
- Pour la cueillette, un microscope d’imagerie LCD placé dans l’armoire sera utilisé car son moniteur élimine le besoin d’oculaires. Stérilisez la platine du microscope avec l’éthanol à 70 %.
- À l’aide d’un microscope à contraste de phase régulière de culture cellulaire, sélectionnez marquer et notez le nombre de colonies à prélever. C’est important de s’assurer que le temps n’est pas perdu pour ce processus pendant la cueillette réelle.
- Remplir un certain nombre de tubes PCR qui est égal ou supérieur au nombre de colonies à prélever avec 30 µL de l’acide éthylènediaminetétraacétique (EDTA) de 0,5 mM dans du PBS. Colonies seront placés dans ces tubes pour la dissociation partielle avant l’ensemencement.
- Plan de choisir 5 colonies à la fois les plaques à l’aide d’une pipette de 10 µL définie sur 2 µL. tenir la pipette à un angle à la lisière de la colonie et délicatement et progressivement gratter toute la colonie sur la surface. Immédiatement aspirer toute la colonie dans l’embout de la pipette et transférer dans un des tubes PCR préparés avec de l’EDTA.
- Répéter l’opération avec les autres 4 colonies. Incuber à RT pendant 4 à 6 min.
- Pipette de la suspension et descendre doucement à l’aide d’un embout de la pipette plu décomposer la colonie en petites touffes. Éviter de créer une suspension monocellulaire.
- Plaque de la suspension directement dans une cible bien d’une plaque de 24 puits préparée à l’étape 2.6.2. Répéter l’opération avec les autres colonies.
- Répétez les étapes 2.6.6 par 2.6.10 si plus de 5 colonies sont à prélever, mais ne prenez pas plus de 5 colonies à la fois. Incuber à 37 ° C et 5 % de CO2.
- Expansion clonale et la maturation de l’iPSC
- Permettre aux colonies de grandir et de devenir compact. 3-6 jours suffisent. Changement du milieu de culture tous les jours. Utiliser entre 400 µL et 1 mL de milieu E8 basée sur la densité des cellules.
- Puits de la plaque 24 puits avec une densité suffisante de la colonie seront élargis en plaques 6 puits. 1 h avant le passage, enduire les plaques 6 puits avec VTN (comme au point 2.2.1). Remplacez ensuite la solution dans le puits avec 2 mL de milieu de E8 / puits.
- Aspirer le milieu usé provenant des puits de la source à l’aide d’une pipette 1 mL et remplacer par 300 µL de 0,5 mM EDTA pour laver. Aspirer immédiatement à l’aide de la pipette même remplacer encore une fois avec 300 µL de 0,5 mM EDTA, puis incuber à ta pendant 5 min. aspirer tout le liquide à l’aide d’une pipette de 1 mL.
- Définir la pipette de 1 mL de capacité, monter un bout de l’échelle-alésage 1 mL là-dessus et aspirer le milieu E8 de la cible dans la pointe. Laver la culture iPSC source avec un flux de données du milieu.
- Transférer la suspension dans la cible bien et la pipette de monter et descendre plusieurs fois pour briser les colonies dans les amas de cellules de 20-50. Assurez-vous qu’ils obtenir répartis uniformément dans le puits de doux balancement et secouant la plaque et incuber à 37 ° C et 5 % de CO2.
- Changer le moyen de tous les jours et le passage tous les 3 à 4 jours. Toute différenciation colonies peuvent être marqués sous le microscope à contraste de phase et supprimés à l’aide d’un embout de la pipette dans la culture du cabinet. Cela permet de multiplication sélective de la culture de l’iPSC pure de haute qualité.
- 1 h avant le passage, enduisez les puits d’une plaque de 6 puits avec VTN comme au point 2.2.1. Puis remplacer la solution 2 ml de milieu de E8 / puits.
- Passage systématique est similaire à l’initiale passant fait à l’aide de 0,5 mM EDTA (étapes 2.7.2 à 2.7.5). Remplacer moyen usé dans un puits d’une plaque de 6 puits avec 1 mL d’EDTA pour laver et éliminer.
- Ajouter 1 mL d’EDTA et incuber 5-7 min pour la dissociation partielle à RT. Optimiser le temps d’incubation si nécessaire, en vous assurant une suspension de près de 20 à 50 éléments touffes est produite. Éviter la dissociation en cellules individuelles.
- Jeter la solution d’EDTA et aspirer 1 mL de milieu E8 en utilisant une pipette 1 mL de la cible dans une échelle-alésage de pipette.
- Laver au large de la culture d’iPSC source avec un flux de données du milieu E8 à plusieurs reprises jusqu'à ce qu’une portion désirée de celui-ci a été libérée de la surface et passer dans la cible bien. Cette partie représente le coefficient de fractionnement (p. ex., 1/8 de la culture peuvent être transférées pour un ratio de 1:8.)
- Passage tous les 3-4 jours. Permettre à iPSC lines à mûrir en les cultivant au moins 15 passages avant de les utiliser dans des expériences en aval
3. caractérisation et Confirmation de pluripotence
Remarque : Consulter les fichiers supplémentaires pour plus de détails sur la cytométrie de flux et de microscopie confocale.
-
Test de formation de tératome
- Pour déterminer la capacité de l’iPSC se différencier en tissus représentatifs de chacune des trois couches germe par dosage de la formation de tératome, se conformer aux politiques institutionnelles concernant les soins aux animaux et l’utilisation, planifier laisser le temps pour déposer le protocole approprié documents. La formation de tératome chez la souris prendra entre 6 à 10 semaines.
- La culture de 4 puits d’une plaque de 6 puits par ligne iPSC pendant 4 jours. Le nombre approximatif des cellules injectées dans un flanc d’une souris est de 0,5 à 1 x 106 cellules. Facultatif : édition spéciale bien peut être dédié à déterminer un nombre représentatif de cellule dans un puits au moyen de la dissociation cellulaire unique avec une enzyme de détachement cellulaire et de comptage.
- Calculer le volume du mélange E8/BMM seront suspendus dans les touffes de l’iPSC : les deux flancs d’une souris sont injectés, chacune avec 150 µL de suspension de la touffe. Trois souris suffisent tester la formation de tératome de l’iPSC monotube. Inclure un supplément de 150 µL par aiguille dans le volume pour compenser la perte de volume mort. 1 aiguille par souris est utilisée. Le volume total est donc 3 * (150 µL * 2 + 150 µL) = 1350 µL
- Comme s’ils devaient être repiquées, tel que décrit dans les étapes 2.7.8 à 2.7.10, partiellement se dissocient les colonies iPSC avec EDTA. Il est important que les colonies se dissocient en touffes et pas divisés en cellules individuelles.
- Laver les colonies iPSC 675 µl de milieu E8 (la moitié du volume calculé à l’étape 3.3.3), en utilisant un embout large-alésage. Transférer la suspension de la touffe dans un tube de polystyrène de 5 mL. Placer sur la glace.
- Combiner avec 675 µL de BMM. Garder la suspension qui en résulte sur la glace jusqu'à ce que l’injection.
- Anesthésier les souris pour caler l’isoflurane. Cela devrait exécuter ou guidé par un personnel qualifié du vivarium.
- Vortex le polystyrène 5 mL tube brièvement et aspirer la suspension de la touffe (450 µL par souris) dans la seringue à une insuline équipé d’une aiguille de 22 G. Injecter par voie sous-cutanée 150 µL de la suspension cellulaire. Ce volume contient environ 1 x 106 cellules (voir étape 3.1.2).
- Injecter 3 souris par ligne iPSC. Suivre la pratique de soins appropriés aux animaux de toutes les méthodes.
- Surveiller la santé des souris tous les jours.
- Quand les tératomes atteignent un diamètre de point de terminaison de 1,5 à 2 cm, euthanasier les souris explantation les tératomes et stockez-les dans une solution de formol pour la fixation du tissu pendant 24 h.
- Apporter les tératomes fixes aux installations de base histologie de l’hématoxyline éosine (H & E) coloration. Un pathologiste évaluera la présence de tissus de tous les trois couches de germe.
Remarque pour Karyotyping : Live iPSC cultures doivent être expédiés à spécialisée cytogénétiques laboratoires pour la recherche de l’intégrité du caryotype. Il est recommandé que ce test tous les 5 passages.
-
Transcription de profilage
- Culture 2 puits d’une plaque de 6 puits pendant 3-4 jours obtenir un échantillon de RNA. Utilisez uniquement des cultures de haute qualité, une contamination mineure avec la différenciation des cellules peut être traitée en grattant à l’aide d’un embout de la pipette.
- Isoler l’ARN de cultures iPSC en utilisant un kit disponible dans le commerce suivant le protocole du fabricant. Expédier des échantillons à un laboratoire spécialisé central génomique.
- Obtenir les profils de transcriptional globales des lignes à l’aide de puces à ADN (voir Table des matières de choix pris en charge) ou séquençage de RNA iPSC.
- Soumettre des fichiers « *.idat » pour évaluation de bioinformatique de pluripotence grâce à une interface en ligne de l’Institut Coriell. Vous pouvez également soumettre « * .cel » fichiers de bioinformatic identification du type de cellule, y compris les cellules souches pluripotentes, grâce à une interface en ligne de l’Université Johns Hopkins. Voir Table des matières pour plus d’informations sur les types de données acceptent les épreuves individuelles de bioinformatique.
Résultats
Consentement éclairé a été obtenu chez les patients avant la récolte du liquide amniotique pour des fins de tests génétiques et de consacrer une petite aliquote du liquide pour la recherche. Aucun consentement n’est requis pour l’utilisation de la membrane amniotique dans la recherche car le placenta représente les déchets médicaux. Des cellules de tige de fluide et membrane amniotique Afficher Propriétés mésenchymateuses typiques, morphologiquement leurs cellules sont fusiformes et phase-lumineux. Lors de la reprogrammation, les cellules subissent mésenchymateuses-à-épithéliales transition (MET) et acquièrent la morphologie pavées et organisation spatiale des colonies, indication des propriétés épithéliales. Ce processus est lancé, dès 48 à 72 heures après l’introduction de la reprogrammation des plasmides épisomiques. Absence de ces colonies par jour 5 de reprogrammation semble indiquer l’échec de l’expérience. Les cellules des colonies MET prolifèrent et par conséquent, les colonies deviennent compacts, entre 5 et 14 jours. Colonies compactes de MET sont composées de cellules qui ne sont pas discernables facilement individuellement (Figure 1 a). Sur autour du jour 14, entièrement reprogrammés colonies apparaissent avec cellules disposées en une monocouche, transportant proéminentes, nucléoles et noyaux facilement discernables. Ils sont prêts à être mécaniquement isolé puis développée lorsque les colonies atteignent une taille convenable et devient compacts (Figure 1 b).
Reprogrammé entièrement et partiellement colonies sont présents dans les cultures sur toute la période de reprogrammation, bien que partiellement reprogrammés colonies n’acquièrent pas nécessairement pluripotence complet. La figure 2 illustre un représentant par cytométrie en flux analyse d’expression de marqueur de cellules souches embryonnaires (CSE) dans d’écoulement entièrement et partiellement pluripotentes colonies et leurs morphologies correspondantes. Pluripotence complet est associée à l’expression de Nanog, Oct4, Sox2, antigènes TRA et SSEA-4, tandis que SSEA-1 expression est négatif de19,20,21 (Figure 2 a). Partiellement les cellules pluripotentes, cependant, n’expriment pas Nanog et TRA antigènes20 (Figure 2 b). L’expression et la localisation des marqueurs ESC doivent être confirmées par immunocytochimiques coloration et photographié à l’aide d’un microscope à champ large ou confocal (Figure 3).
Une confirmation fonctionnelle de la pluripotence est obtenue en démontrant la capacité des lignes iPSC à tératomes de forme après une injection sous-cutanée des cellules dans des souris scid-beige. 6 à 8 semaines sont nécessaires pour les tératomes atteindre la taille de point final. H & E de coloration des tissus et l’examen par un pathologiste est ensuite effectuée pour confirmer la présence du tissus représentant tous les trois couches de germe – endoderme, neuroectoderme et mésoderme (Figure 4 a). Une alternative à l’expérimentation animale est d’analyser la signature transcriptionnelle associée pluripotence par des approches génomiques comme cDNA microarrays22,23. La proportion du profil transcriptionnel qui chevauche avec l’un d’un pool d’iPSC bien établie et les lignes de l’ESC peut ensuite être quantifiée par le logiciel d’évaluation de pluripotence bioinformatique en ligne sous la forme d’une parcelle de deux classificateurs – pluripotence et nouveauté (Figure 4 b). Plus le score de pluripotence, plus la requête iPSC ligne ressemble à des lignes établies. Une vingtaine de haute nouveauté, cependant, pourrait indiquer déviations ou des aberrations chromosomiques même, malgré une forte pluripotence score (p. ex. lignes tératocarcinome)22. Toutes les lignes de l’iPSC générés en suivant le protocole présenté ici ont été jugées pluripotentes par cytométrie en flux, imagerie, formation de tératome et méthodes d’analyse transcriptionnelle.

Figure 1 : évolution morphologique des cellules au cours de reprogrammation. (A) le liquide amniotique et la membrane des cellules souches, qui représentent les cellules source pour la reprogrammation, afficher une morphologie typique de mésenchymateuse, allongée et phase-lumineux (à gauche) jusqu'à ce qu’ils subissent la transition mésenchymateuse-à-épithéliales (MET) qui conduit à l’acquisition de propriétés épithéliales et de la formation de colonies avec des cellules de galets pierreuse (Centre). Ces colonies prolifèrent et créer des masses irrégulières cellulaires des cellules MET (à droite). (B) aux stades ultérieurs de reprogrammation (à partir d’autour du jour 14), des colonies de cellules entièrement reprogrammés émergent – individuellement discernables cellules avec des noyaux importants et nucléoles disposée en monocouches, avec des frontières bien définies (Centre) – et sont présents aux côtés des colonies MET qui sont les plus nombreux (gauche). Un clone de mature isolé entièrement reprogrammé est représenté sur la droite. Echelle = 100 µm s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
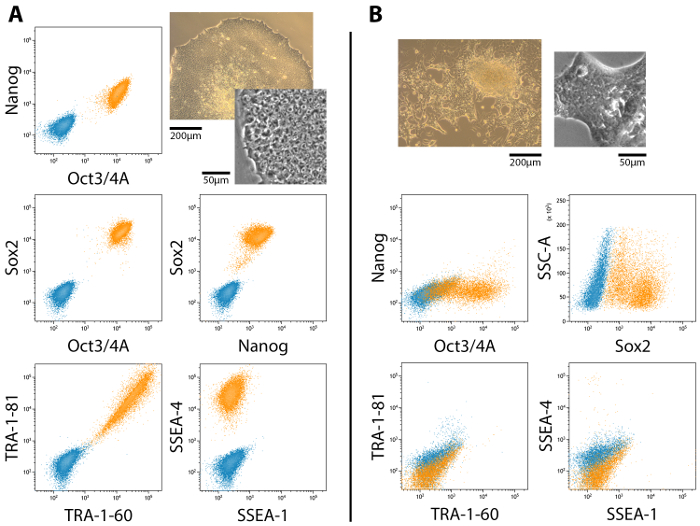
Figure 2 : analyse par cytométrie en flux de l’expression des marqueurs de l’ESC d’écoulement en totalité et en partie (MET) reprogrammé colonies cellulaires. (A) le profil d’expression pluripotentes est positif pour Oct4, Nanog, Sox2, TRA-1-60, TRA-1-81 et SSEA-4, tandis que négatif pour SSEA-1. (B) partiellement des colonies de cellules pluripotentes, ceux qui ont subi le MET, mais n’a pas pu évoluer vers la pluripotence complet, sont positives pour Oct4 et Sox2 mais Nanog, TRA et l’AESS antigènes sont absentes. Les morphologies associés sont inclus pour comparaison côte-à-côte. Barreaux de l’échelle = 200 µm et 50 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
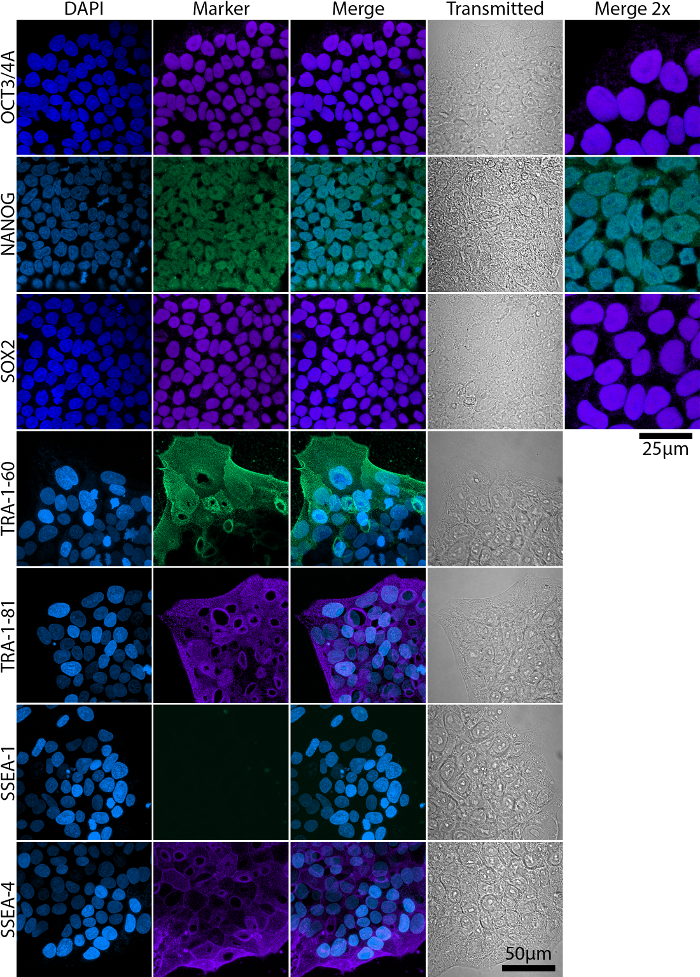
Figure 3 : analyse de l’expression des marqueurs de l’ESC dans mature CISP fluide amniotique d’imagerie confocale. Facteurs de transcription Oct3/4 a, Nanog et Sox2 sont localisées dans le noyau tandis que TRA et antigènes SSEA sont des glycoprotéines localisées sur la membrane. Echelle = 50 µm. les Images de plus fort grossissement (fusion 2 X) ont été inclus pour Oct3/4, Nanog et Sox2 pour meilleure visualisation de leur localisation nucléaire. Echelle = 25 µm. transmissibles – images acquises sur la lumière transmise. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : formation de tératome et profilage transcriptionnel dans mature CISP fluide et de membrane amniotique. (A) tératomes cultivés par voie sous-cutanée chez les souris scid-beige contiennent des tissus représentatifs de chacune des trois couches germinales (grossissement de X 100). (B) les profils d’expression globale « microarray » soumis au logiciel en ligne de pluripotence retourné une parcelle de deux classificateurs – pluripotence et nouveauté. Pluripotence high scores et les scores de nouveauté faible - rouges nuage - montrent un profil d’expression d’une ligne ESC/iPSC typique. Le nuage bleu représente une zone de cluster pour les cellules différenciées, alors que le nuage bleu pâle représente une zone de cluster pour partiellement les cellules pluripotentes. Le liquide amniotique (3 lignes) et iPSC membrane (4 lignes) ont été jugées pluripotentes par le test. Une ligne ESC WA25 a été incluse en tant que contrôle et est ici identifiée avec une flèche noire. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
La phase initiale de l’iPSC génération de cellules souches fœtales implique l’extraction de cellules source dans les tissus fœtaux, leur culture, expansion et introduction des plasmides reprogrammation épisomiques. Cette phase est suivie d’une période de culture d’environ 14 à 18 jours avant que les premières colonies entièrement reprogrammés peuvent être étendus. La phase finale est la maturation des clones iPSC. L’extraction initiale de cellules souches de membrane amniotique se faite au moyen d’une digestion enzymatique et mécanique combinée de l’amnios. Nous avons constaté qu’une durée d’incubation de 30 min a entraîné le plus grand nombre de cellules extraites à la viabilité de la plus haute. La procédure de la digestion peut produire des petits morceaux de tissus et cellules de touffes. Si la proportion de ceux-ci par rapport à simples cellules est élevée, nous vous recommandons de placage toutes les touffes et les cellules individuelles dans un récipient, car tous peuvent contribuer à des excroissances des cellules adhérentes. Placage des cellules souches fluides amniotique est simple car les cellules sont seulement mélangés avec le milieu de culture et incubés jusqu'à ce que les colonies de cellules adhérentes atteignent une taille suffisante. Régulière ustensiles en plastique imprégnées de culture tissulaire est parfaitement adapté et nous ne recommandons pas de surfaces spécialisées, même s’ils sont destinés pour la culture de l’amélioration des cellules primaires, car avec ces derniers, nous avons observé plus faible viabilité et difficultés avec le passage processus.
L’amniotique tige fluide et la membrane des cellules devraient être élargies et les stocks congelés mais, dans les meilleurs délais, les cellules peuvent être utilisés comme des cellules de source pour la reprogrammation. Aux fins de l’introduction des plasmides épisomiques dans les cellules, le système de transfection utilisé ici avec les paramètres de transfection valeur 950 V, 40 ms et 1 impulsion a très bien performé, avec toutes les lignes ont tenté en fin de compte avec succès reprogrammé) plus de 10 lignes). Le système de livraison principal concurrent fonctionnant sur un principe similaire n’a pas produit une expérience réussie de reprogrammation dans nos mains.
Les cellules transfectées sont ensemencées sur enduit de vitronectine vaiselle dans le milieu de l’AFMC pour des 3-5 premiers jours, puis le milieu est passé à E8 additionné le butyrate de sodium 100 µM. Cela augmente considérablement le taux d’acquisition de pluripotence complet. Les premiers signes de transformation morphologique peuvent être vu aussi tôt que 48 à 72 h. Les cellules de source subissent le MET et colonies de cellules avec une morphologie épithéliale apparaissent. Progressivement, ceux-ci prolifèrent et deviennent compacts. Un sous-ensemble des colonies va acquérir les caractéristiques morphologiques des totalement les cellules souches pluripotentes – individuellement discernables cellules avec des noyaux et des nucléoles, plats colonies avec des frontières bien définies, par opposition à des frontières floues chez partiellement pluripotentes MET colonies. Du moment de l’acquisition de pluripotence complet, les colonies de cellules MET compacts acquièrent des noyaux et les cellules individuelles deviennent perceptibles tout en créant un modèle morphologique unique. Pour un œil exercé, ce modèle est un signe évident de reprogrammation réussie. Cependant, à un enquêteur qui n’a pas de formation de la culture de la CFP, identification des colonies qui ont progressé avec succès à la pluripotence complet nécessite une évaluation soigneuse comme clones MET et iPSC peuvent être confondue avec l’autre. Figure 1 et Figure 2 fournissent des exemples des deux. Si MET clones sont cueillies au lieu de cela, analyse en cytométrie en flux approfondie révélera l’erreur, et en particulier, les antigènes TRA-1-60 et TRA-1-81 sera probablement absents comme illustré à la Figure 2. En effet, les antigènes TRA trouvées précédemment comme des marqueurs de pluripotence rigoureuses. Cependant, partiellement les cellules pluripotentes MET pourraient être d’intérêt dans le cancer recherche25.
Cette condition de culture n’est pas optimale pour la source de l’AFSC/CSEM et finalement, leur prolifération va ralentir et ils acquerront une morphologie plus plate, comme des fibroblastes. Les cellules source forment les tissus qui peuvent se détacher de la surface pendant les dernières étapes de reprogrammation, bien que cela n’affecte pas négativement le processus de reprogrammation. Au contraire, le processus conduit parfois à libérant un espace pour les colonies reprogrammés, tout en éliminant les matières cellulaires non reprogrammé indésirables. Tissus détachés peuvent facilement être jetées à l’aide d’un embout de la pipette stérile, laissant partiellement et complètement pluripotentes colonies derrière, qui simplifie la sélection manuelle en aval.
Pour la cueillette manuelle des colonies entièrement reprogrammés, nous utilisons un écran LCD système, qui peut être placé dans le cabinet de sécurité d’imagerie, dépourvu de toute pièce en saillie sur qui pourrait perturber le flux d’air. Autre que ce système d’imagerie, aucun équipement spécial n’est nécessaire car la cueillette elle-même peut être effectuée à l’aide de pipettes ordinaires. Les colonies cueillies sont partiellement dissociés en solution EDTA/PBS avant d’être plaqué dans les puits de cible à pousser comme des clones. Selon la ligne et le clone, pour plusieurs passages, les cultures peuvent être contaminés par spontanément différenciation des cellules. Manipulation manuelle et série passant généralement pour éliminer ce problème. Clones criblés de vaste différenciation doivent être jetées, cependant, clones précieux peuvent être récupérés avec divers degrés de succès par le biais de manuel répétée cueillette des colonies pluripotentes plutôt que de disposer de la différenciation des cellules. Les plasmides épisomiques apparaissaient à prendre environ 15 passages pour disparaître complètement de l' iPSC26. Par conséquent, il est conseillé d’autoriser les clones de croître au moins ce nombre de passages avant de les utiliser pour des applications en aval et des analyses, à l’exception de la surveillance systématique du caryotype et expression de l’antigène TRA. Expression de l’antigène TRA facilement peut être surveillée par cytométrie en flux, comme décrit ici dans le protocole, car le test nécessite environ 200 000 cellules seulement et peut être effectué chaque fois que les chercheurs sont dans le doute quant à savoir si les clones cultivés sont maintenant pluripotence correctement. Analyse en cytométrie en flux de l’expression du marqueur ESC ne constitue pas suffire à confirmer la pluripotence dans candidat lignes19.
Tératome formation test est le test de pluripotence concluante standard27. CFP cultivée dans des conditions chimiquement définies, xeno-free sont particulièrement sensibles à la dissociation induite par la mort et y injecter par voie sous-cutanée en touffes est donc nécessaire pour leur implantation réussie8,28. Après l’injection, généralement 4 à 6 semaines suffisent pour la croissance des xéno-greffes soit visible et avant la semaine 8, peuvent tous être récoltés, H & E tachés et analysées. Bien-être des animaux, le coût et tests longues périodes nécessaires sont des raisons pour le développement de méthodes alternatives. Les analyses génomiques combiné avec avancé, approches bioinformatiques alimenté par apprentissage machine peuvent fournir une évaluation précise des profils d’expression globale. Le coût d’obtention de ces données est comparable au coût de l’analyse de formation tératome, cependant, l’approche génomique est considérablement plus rapide et aucuns animaux ne doivent être utilisés. Une telle analyse est une de logiciels bioinformatiques pluripotence évaluation22. Il est implémenté comme une interface en ligne (Table des matières). La popularité croissante et le coût chute de volonté de séquençage de RNA assurent la continuité de cette approche. Une alternative à ce logiciel de pluripotence est disponible auprès de l’Université Johns Hopkins23 (cellnet.hms.harvard.edu) et est basée sur une approche similaire et est en mesure d’accepter les données microarray pour analyser le transcriptome d’échantillons humains. L’avantage de ce logiciel est qu’il a la capacité d’identifier non seulement les cellules souches pluripotentes mais également des cellules différenciées et, depuis ses datasets curated ont été dérivée de tissus primaires, le degré de similitude entre les cellules in vitro cultivé / tissus et tissus in vivo peuvent être déterminés, offrant un excellent contrôle de qualité pour l’élaboration de protocoles de différenciation ou génie tissulaire. Le test a la capacité de classer les requêtes en 20 cellules différentes ou les types de tissus. À l’heure actuelle, il nécessite des données microarray, mais les auteurs sont efforcent d’élargir les options de la plate-forme à l’ARN séquençage ainsi.
En suivant le protocole présenté, chercheurs peuvent générer iPSC lignées de cellules de tige fluide et membrane amniotiques avec une reproductibilité très élevée dans un milieu entièrement chimiquement défini et xeno-gratuit et en utilisant une méthode de reprogrammation sans intégration. Ces lignes peuvent être utilisés en recherche fondamentale afin d’optimiser les protocoles de différenciation et finalement dans la modélisation de la maladie, des médicaments tissus examen préalable, ou pédiatrique, études d’ingénierie.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun intérêt financier concurrentes.
Remerciements
Ce travail a été soutenu par le Fonds Medizinische Forschung à l’Université de Zurich, Forschungskredit de l’Université de Zurich, la NMS SCIEXCh sous ° 10.216 bourses et 12.176, The Swiss Society of Cardiology, The Swiss National Science Fondation au titre de la subvention [320030-122273] et [310030-143992], le 7e Programme-cadre, la soupape de vie, la Commission européenne au titre de la subvention [242008], la Fondation de Mayenfisch Olga, la Fondation EMDO, la subvention de démarrage 2012 de l’hôpital universitaire de Zurich, et le financement interne de l’Institut de Cancer de Mitchell.
matériels
| Name | Company | Catalog Number | Comments |
| Tumor Dissociation Kit, human | Miltenyi Biotec | 130-095-929 | tissue dissociation system, reagent kit, includes tissue dissociation tubes and tissue dissociation enzymes |
| gentleMACS Dissociator | Miltenyi Biotec | 130-093-235 | tissue dissociation system, dissociator |
| Thermo Scientific™ Shandon™ Disposable Scalpel No. 10, Sterile, Individually Wrapped, 5.75 (14.6cm) | Thermo-Fisher | 3120032 | |
| 70 µm cell strainers | Corning | 10054-456 | |
| RPMI 1640 medium | Thermo-Fisher | 32404014 | |
| rocking platform | VWR | 40000-300 | |
| 50 ml centrifuge tubes | Thermo-Fisher | 339652 | |
| 15 ml centrifuge tubes | Thermo-Fisher | 339650 | |
| EBM-2 basal medium | Lonza | CC-3156 | basal medium for AFMC medium |
| FGF 2 Human (expressed in E. coli, non-glycosylated) | Prospec Bio | CYT-218 | bFGF, supplement for AFMC medium |
| EGF Human, Pichia | Prospec Bio | CYT-332 | EGF, supplement for AFMC medium |
| LR3 Insulin Like Growth Factor-1 Human Recombinant | Prospec Bio | CYT-022 | IGF, supplement for AFMC medium |
| Fetal Bovine Serum, embryonic stem cell-qualified | Thermo-Fisher | 10439024 | FBS |
| Antibiotic-Antimycotic (100X) | Thermo-Fisher | 15240062 | for primary AFSC/AMSC, for routine AFSC/AMSC it should not be necessary, do not use in medium for transfected cells! |
| Accutase cell detachment solution | StemCell Technologies | 07920 | cell detachment enzyme |
| CryoStor™ CS10 | StemCell Technologies | 07930 | complete freezing medium |
| PBS, pH 7.4 | Thermo-Fisher Scientific | 10010023 | |
| EndoFree Plasmid Maxi Kit (10) | Qiagen | 12362 | for plasmid isolation |
| pEP4 E02S EN2K | Addgene | 20925 | EN2K, reprogramming factors Oct4+Sox2, Nanog+Klf4 |
| pEP4 E02S ET2K | Addgene | 20927 | ET2K, reprogramming factors Oct4+Sox2, SV40LT+Klf4 |
| pCEP4-M2L | Addgene | 20926 | M2L, reprogramming factors c-Myc+LIN28 |
| NanoDrop 2000c UV-Vis Spectrophotometer | Thermo-Fisher | ND-2000C | spectrophotometer |
| Neon® Transfection System | Thermo-Fisher | MPK5000 | transfection system, components: Neon pipette - transfection pipette Neon device - transfection device |
| Neon® Transfection System 10 µL Kit | Thermo-Fisher | MPK1025 | consumables kit for the Neon Transfection System, it contains: Neon tip - transfection tip Neon tube - transfection tube buffer R - resuspension buffer buffer E - electrolytic buffer |
| Stemolecule™ Sodium Butyrate | StemGent | 04-0005 | small molecule enhancer of reprogramming |
| TeSR-E8 | StemCell Technologies | 05940 | E8 medium |
| Vitronectin XF™ | StemCell Technologies | 07180 | VTN, stock concentration 250 µg/ml, used for coating at 1 µg/cm2 in vitronectin dilution (CellAdhere) buffer |
| CellAdhere™ Dilution Buffer | StemCell Technologies | 07183 | vitronectin dilution buffer |
| UltraPure™ 0.5M EDTA, pH 8.0 | Thermo-Fisher | 15575020 | dilute with PBS to 0.5 mM before use |
| EVOS® FL Imaging System | Thermo-Fisher Scientific | AMF4300 | LCD imaging microscope system |
| CKX53 Inverted Microscope | Olympus | phase contrast cell culture microscope | |
| Pierce™ 16% Formaldehyde (w/v), Methanol-free | Thermo-Fisher | 28908 | dilute to 4% with PBS before use, diluted can be stored at 2-8 °C for 1 week |
| Perm Buffer III | BD Biosciences | 558050 | permeabilization buffer, chill to -20 °C before use |
| Mouse IgG1, κ Isotype Control, Alexa Fluor® 488 | BD Biosciences | 557782 | isotype control for Oct3/4A, Nanog |
| Mouse IgG1, κ Isotype Control, Alexa Fluor® 647 | BD Biosciences | 557783 | isotype control for Sox2 |
| Mouse anti-human Oct3/4 (Human Isoform A), Alexa Fluor® 488 | BD Biosciences | 561628 | |
| Mouse anti-human Nanog, Alexa Fluor® 488 | BD Biosciences | 560791 | |
| Mouse anti-human Sox-2, Alexa Fluor® 647 | BD Biosciences | 562139 | |
| Mouse IgGM, κ Isotype Control, Alexa Fluor® 488 | BD Biosciences | 401617 | isotype control for TRA-1-60 |
| Mouse IgGM, κ Isotype Control, Alexa Fluor® 647 | BD Biosciences | 401618 | isotype control for TRA-1-81 |
| Mouse anti-human TRA-1-60, Alexa Fluor® 488 | BD Biosciences | 330613 | |
| Mouse anti-human TRA-1-81, Alexa Fluor® 647 | BD Biosciences | 330705 | |
| Mouse IgG1, κ Isotype Control, Alexa Fluor® 488 | BD Biosciences | 400129 | isotype control for SSEA-1 |
| Mouse IgG3, κ Isotype Control, Alexa Fluor® 647 | BD Biosciences | 401321 | isotype control for SSEA-4 |
| Mouse anti-human SSEA-1, Alexa Fluor® 488 | BD Biosciences | 323010 | |
| Mouse anti-human SSEA-4, Alexa Fluor® 647 | BD Biosciences | 330407 | |
| Affinipure F(ab')2 Fragment Goat Anti-Mouse IgG+IgM, Alexa Fluor® 488 | Jackson Immunoresearch | 115-606-068 | use at a dilution of 1:600 or further optimize |
| Affinipure F(ab')2 Fragment Goat Anti-Mouse IgG+IgM, Alexa Fluor® 647 | Jackson Immunoresearch | 115-546-068 | use at a dilution of 1:600 or further optimize |
| DAPI | Thermo-Fisher Scientific | D21490 | stock solution 10 mM, further dilute to 1:12.000 for a working solution |
| Corning® Matrigel® Growth Factor Reduced, Phenol Red-Free | Corning | 356231 | basement membrane matrix (BMM) |
| scid-beige mice, female | Taconic | CBSCBG-F | |
| RNeasy Plus Mini Kit (50) | Qiagen | 74134 | RNA isolation kit |
| T-25 flasks, tissue culture-treated | Thermo-Fisher | 156367 | |
| T-75 flasks, tissue culture-treated | Thermo-Fisher | 156499 | |
| Nunc™ tissue-culture dish | Thermo-Fisher | 12-567-650 | 10 cm tissue culture dish |
| 6-well plates, tissue-culture treated | Thermo-Fisher | 140675 | |
| Neubauer counting chamber (hemacytometer) | VWR | 15170-173 | |
| Mr. Frosty™ Freezing Container | Thermo-Fisher | 5100-0001 | freezing container |
| FACS tubes, Round Bottom Polystyrene Test Tube, 5ml | Corning | 352058 | 5 ml polystyrene tubes |
| Eppendorf tubes, 1.5 ml | Thermo-Fisher | 05-402-96 | 1.5 ml microcentrifuge tubes |
| PCR tubes, 200 µl | Thermo-Fisher | 14-222-262 | |
| pipette tips, 100 to 1250 µl | Thermo-Fisher | 02-707-407 | narrow-bore 1 mL tips |
| pipette tips, 5 to 300 µl | Thermo-Fisher | 02-707-410 | |
| pipette tips, 0.1 to 10 µl | Thermo-Fisher | 02-707-437 | |
| wide-bore pipette tips, 1000 µl | VWR | 89049-166 | wide-bore 1 mL tips |
| glass Pasteur pipettes | Thermo-Fisher | 13-678-20A | |
| ethanol, 200 proof | Thermo-Fisher | 04-355-451 | |
| vortex mixer | VWR | 10153-842 | |
| chambered coverglass, 8-well, 1.5mm borosilicate glass | Thermo-Fisher | 155409 | glass-bottom confocal-grade cultureware |
| 22G needles | VWR | 82002-366 | |
| insulin syringes | Thermo-Fisher | 22-253-260 | |
| Formalin solution, neutral buffered, 10% | Sigma-Aldrich | HT501128-4L | fixation of explanted teratomas |
| Illumina HT-12 v4 Expression BeachChip | Illumina | BD-103-0204 | expression microarray, supported by PluriTest, discontinued by manufacturer |
| PrimeView Human Genome U219 Array Plate | Thermo-Fisher | 901605 | expression microarray (formerly Affymetrix brand), soon to be supported by PluriTest |
| GeneChip™ Human Genome U133 Plus 2.0 Array | Thermo-Fisher | 902482 | expression microarray (formerly Affymetrix brand), supported by CellNet, soon to be supported by PluriTest |
| PluriTest® | Coriell Institute | www.pluritest.org, free service for bioinformatic assessment of pluripotency, accepts microarray data - *.idat files from HT-12 v4 platform, soon to support U133, U219 microarray and RNA sequencing data | |
| CellNet | Johns Hopkins University | cellnet.hms.harvard.edu, free service for bioinformatic identification of cell type, including plutipotent stem cells, based on U133 microarray data - *.cel files, soon to support RNA sequencing data |
Références
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126 (4), 663-676 (2006).
- Yu, J., et al. Induced Pluripotent Stem Cell Lines Derived from Human Somatic Cells. Science. 318 (5858), 1917-1920 (2007).
- Trounson, A., DeWitt, N. D. Pluripotent stem cells progressing to the clinic. Nat. Rev. Mol. Cell Biol. 17 (3), 194-200 (2016).
- Schmidt, D., et al. Prenatally fabricated autologous human living heart valves based on amniotic fluid derived progenitor cells as single cell source. Circulation. 116 (11 Suppl), I64-I70 (2007).
- Weber, B., Zeisberger, S. M., Hoerstrup, S. P. Prenatally harvested cells for cardiovascular tissue engineering: Fabrication of autologous implants prior to birth. Placenta. 32, S316-S319 (2011).
- Weber, B., et al. Prenatally engineered autologous amniotic fluid stem cell-based heart valves in the fetal circulation. Biomaterials. 33 (16), 4031-4043 (2012).
- Kehl, D., Weber, B., Hoerstrup, S. P. Bioengineered living cardiac and venous valve replacements: current status and future prospects. Cardiovasc. Pathol. 25 (4), 300-305 (2016).
- Slamecka, J., et al. Non-integrating episomal plasmid-based reprogramming of human amniotic fluid stem cells into induced pluripotent stem cells in chemically defined conditions. Cell Cycle. 15 (2), 234-249 (2016).
- Jiang, G., et al. Human Transgene-Free Amniotic-Fluid-Derived Induced Pluripotent Stem Cells for Autologous Cell Therapy. Stem Cells Dev. 23 (21), 2613-2625 (2014).
- Pipino, C., et al. Trisomy 21 mid-trimester amniotic fluid induced pluripotent stem cells maintain genetic signatures during reprogramming: implications for disease modeling and cryobanking. Cell. Reprogram. 16 (5), 331-344 (2014).
- Cai, J., et al. Generation of human induced pluripotent stem cells from umbilical cord matrix and amniotic membrane mesenchymal cells. J Biol. Chem. 285 (15), 11227-11234 (2010).
- Ge, X., et al. Human Amniotic Mesenchymal Stem Cell-Derived Induced Pluripotent Stem Cells May Generate a Universal Source of Cardiac Cells. Stem Cells Dev. 21 (15), 2798-2808 (2012).
- Drozd, A. M., Walczak, M. P., Piaskowski, S., Stoczynska-Fidelus, E., Rieske, P., Grzela, D. P. Generation of human iPSCs from cells of fibroblastic and epithelial origin by means of the oriP/EBNA-1 episomal reprogramming system. Stem Cell Res. Ther. 6 (1), (2015).
- Kang, N. -. H., et al. Potential antitumor therapeutic strategies of human amniotic membrane and amniotic fluid-derived stem cells. Cancer Gene Ther. 19 (8), 517-522 (2012).
- Moschidou, D., et al. Valproic Acid Confers Functional Pluripotency to Human Amniotic Fluid Stem Cells in a Transgene-free Approach. Mol. Ther. 20 (10), 1953-1967 (2012).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nature Methods. 8 (5), 424-429 (2011).
- Yu, J., et al. Human Induced Pluripotent Stem Cells Free of Vector and Transgene Sequences. Science. 324 (5928), 797-801 (2009).
- Martí, M., et al. Characterization of pluripotent stem cells. Nat. Protoc. 8 (2), 223-253 (2013).
- Chan, E. M., et al. Live cell imaging distinguishes bona fide human iPS cells from partially reprogrammed cells. Nat. Biotechnol. 27 (11), 1033-1037 (2009).
- Adewumi, O., et al. Characterization of human embryonic stem cell lines by the International Stem Cell Initiative. Nat. Biotechnol. 25 (7), 803-816 (2007).
- Müller, F. -. J., et al. A bioinformatic assay for pluripotency in human cells. Nature Methods. 8 (4), 315-317 (2011).
- Cahan, P., Li, H., Morris, S. A., Lummertz da Rocha, E., Daley, G. Q., Collins, J. J. CellNet: Network Biology Applied to Stem Cell Engineering. Cell. 158 (4), 903-915 (2014).
- Schopperle, W. M., DeWolf, W. C. The TRA-1-60 and TRA-1-81 Human Pluripotent Stem Cell Markers Are Expressed on Podocalyxin in Embryonal Carcinoma. STEM CELLS. 25 (3), 723-730 (2007).
- Ohnishi, K., et al. Premature Termination of Reprogramming In Vivo Leads to Cancer Development through Altered Epigenetic Regulation. Cell. 156 (4), 663-677 (2014).
- Schlaeger, T. M., et al. A comparison of non-integrating reprogramming methods. Nature Biotechnology. 33 (1), 58-63 (2014).
- Müller, F. -. J., Goldmann, J., Löser, P., Loring, J. F. A call to standardize teratoma assays used to define human pluripotent cell lines. Cell Stem Cell. 6 (5), 412-414 (2010).
- Beers, J., et al. Passaging and colony expansion of human pluripotent stem cells by enzyme-free dissociation in chemically defined culture conditions. Nat. Protoc. 7 (11), 2029-2040 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon