Method Article
Reprogrammierung primäre Fruchtwasser und Membrane Zellen zur Pluripotenz in Xeno-freien Bedingungen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Reprogrammierung von primären amniotische Flüssigkeit und Membran mesenchymalen Stammzellen in induzierte pluripotente Stammzellen unter Verwendung eines nicht-Integration von episomal Ansatzes in vollem Umfang chemisch definierten Bedingungen. Verfahren der Gewinnung, Kultur, Neuprogrammierung und Charakterisierung der daraus resultierenden induzierten pluripotenten Stammzellen durch strenge Methoden werden detailliert beschrieben.
Zusammenfassung
Autologe zellbasierte Therapien bekam einen Schritt näher an der Realität mit der Einführung von induzierten pluripotenten Stammzellen. Fetaler Stammzellen, wie Fruchtwasser und Membran mesenchymale Stammzellen, stellen eine einzigartige Art von undifferenzierten Zellen mit Versprechen in Gewebe-Engineering und Reprogrammierung in iPSC für zukünftige pädiatrische Interventionen und Stammzell-banking. Die hier vorgestellten Protokoll beschreibt ein optimiertes Verfahren zur Gewinnung und Kultivierung primäre amniotischen Flüssigkeit und Membran mesenchymalen Stammzellen und generiert episomal induzierte pluripotente Stammzellen aus diesen Zellen in vollem Umfang chemisch definierte Kultur Bedingungen, die Nutzung von menschlichen rekombinante Vitronectin und E8 Medium. Charakterisierung der neuen Linien durch die Anwendung strenger Methoden – Durchflusszytometrie, konfokale Bildgebung, Teratom Bildung und transkriptionelle profiling – wird ebenfalls beschrieben. Die neu erzeugten Linien auszudrücken Marker von embryonalen Stammzellen – Oct3/4A, Nanog, Sox2, TRA-1-60, TRA-1-81, SSEA-4 – wobei negativ für die SSEA-1-Markierung. Die Zelllinien bilden darum in scid-Beige Mäuse in ca. 6-8 Wochen und die darum enthalten Gewebe repräsentativ für alle drei Mikrobeschichten. Transkriptionelle Profilierung der Linien mit dem Absenden globalen Ausdruck Microarray Daten zu einem Bioinformatic Pluripotenz Bewertung Algorithmus als alle Linien pluripotenten und daher ist dieser Ansatz eine attraktive Alternative zu Tierversuchen. Die neuen iPSC-Linien können problemlos in nachgeschalteten Experimente mit der Optimierung von Differenzierung und Tissue Engineering verwendet werden.
Einleitung
Die Technologie von induzierten pluripotenten Stammzellen (iPSC) bringt über mögliche Zellersatztherapien, Krankheit, Medikament und Entwicklungsstörungen Modellierung und toxikologischen Screening1,2,3. -Ersatz-Therapien können konzeptionell durch Zelle Injektion erreicht werden, in-vitro-differenzierte Gewebe (z. B. kardiale Patches) Implantation oder geführte Regenerierung mittels Gewebetechnik. Fruchtwasser (AFSC) und Membran-Stammzellen (AMSC) sind eine ausgezeichnete Quelle von Zellen für diese Interventionen entweder direkt4,5,6,7 oder als ein Ausgangspunkt Zellpopulation Umprogrammierung in Pluripotenz8,9,10,11,12.
Frühe Ansätze verwendet nicht definierte Kultur-Systeme oder Neuprogrammierung Methoden, die beinhalten genomischen Integration von9,10,11,12konstruiert. Eine neuere Studie beschäftigt ein Xeno-freies Medium, obwohl eine weniger definierte Basalmembran Anlage Matrix (BMM) verwendet wurde, um iPSC aus amniotische Flüssigkeit Epithelzellen zu generieren. Teratom Bildung Assay war jedoch nicht in die Studie zusammen mit einer Fülle von in-vitro- und molekulare Daten enthalten. Amniotische flüssige epitheliale Zellen erwiesen sich als eine etwa 8-fold höhere Neuprogrammierung Effizienz im Vergleich zu Neugeborenen Fibroblasten13haben. In einer anderen Studie fanden mesenchymaler Stammzellen aus Fruchtwasser auch in iPSC mit einem viel höheren Wirkungsgrad12neu programmiert werden.
Pluripotente Stammzellen unterscheiden sich in Geweben repräsentativ für alle 3 Mikrobeschichten und somit das größte Potenzial haben. Pädiatrische Patienten könnten davon profitieren die Ernte, Neuprogrammierung und Tissue Engineering ihre autologe amniotische Flüssigkeit Stammzellen pränatal und perinatal amniotischen Membran-Stammzellen. Darüber hinaus konnte das relativ niedrige Niveau der Differenzierung von embryonalen Stammzellen (niedriger als adulte Stammzellen14,15) theoretisch helfen bei der Bewältigung des beobachteten Eigentumsvorbehalts epigenetische Voreingenommenheit von Quellzellen iPSC16.
Hier stellen wir Ihnen ein Protokoll für die Umprogrammierung Fruchtwasser und Membran Stammzellen zur Pluripotenz in chemisch Xeno-freie E8 Medium für rekombinante Vitronectin17 (VTN) mit episomal Plasmide18definiert. Der Hauptvorteil der amniotischen Flüssigkeit und Membran-Zellen als eine Quelle der Zellen für eine Anpassung liegt in ihrer Verfügbarkeit Pre und perinatal und somit würde dieser Ansatz vor allem Forschung zugute, in pädiatrischen Gewebetechnik.
Protokoll
Das Protokoll folgt institutionellen Richtlinien der Ethik-Kommission für Forschung am Menschen. Schriftliche Einwilligung des Patienten wurde für die Verwendung von Fruchtwasser für die Forschung erhalten.
Dieses Protokoll folgt die Politik der institutionellen Animal Care und Use Committee von der University of South Alabama.
1. Isolierung und Kultur der primären Fruchtwasser mesenchymalen Stammzellen
-
Beschichtung von amniotische Flüssigkeit Zellen
- Erhalten Sie ein Minimum von 2,5 mL Fruchtwasser geerntet in den Prozess der Amniozentese durch einen Arzt.
Hinweis: Alle Umgang mit lebenden Zellen und Gewebe muss in einem sterilen Gewebekultur Schrank durchgeführt werden und muss geeignete persönliche Schutzausrüstungen verwendet werden. Vertrautheit mit den grundlegenden Zellkultur und steriler Technik ist erforderlich. - Bereiten Sie die amniotische Flüssigkeit und Membran (AFMC) Zellkulturmedium: EBM-2 basal Medium, 15 % fetalen bovine Serum (FBS), 20 ng/mL von bFGF, 25 ng/mL des EGF, 10 ng/mL des IGF. Für die Kultur der primären amniotic Fluid sowie amniotischen Membran Stammzellen sollte das Medium mit Antibiotikum antimykotische Lösung ergänzt werden.
- Am Tag 0 mischen Sie 2,5 mL Fruchtwasser mit 3,5 mL Kulturmedium AFMC und Platte in ein T25-Fläschchen. Inkubation bei 37 ° C und 5 % CO2 für mindestens 48 Stunden vor der Prüfung auf das Vorhandensein von Kolonien ungestört.
- Am 5. Tag sollte Kolonien von adhärenten Zellen vorhanden sein. Schütteln Sie die Flasche um Zellen zu entfernen, die nicht in vollem Umfang haften am Boden und Schutt und Vakuum-Aspirat die abgebrannte Medium/amniotische flüssige Mischung mit einer Pasteurpipette. Ersetzen Sie mit 5 mL frischem AFMC Medium.
- Kultur für weitere 5 Tage. Verändern Sie jeden zweiten Tag, wie in Schritt 1.1.4 beschrieben Medium.
- Erhalten Sie ein Minimum von 2,5 mL Fruchtwasser geerntet in den Prozess der Amniozentese durch einen Arzt.
-
Isolierung der primären mesenchymalen Stammzellen aus menschlichen amnion
- Erhalten Sie die Plazenta so bald wie möglich nach der Geburt, innerhalb von 24 h spätestens zellulären Integrität zu maximieren. Schneiden Sie ein 9 cm2 Segment der Amnion, Blutgerinnsel entfernen und waschen in einem 50 mL Zentrifugenröhrchen mit 30 mL PBS mit Antibiotikum antimykotische Lösung ergänzt.
- Hacken Sie die Membranen mit einem Paar von Skalpelle Stücke in der Kulturschale steril 10 cm Gewebe fein. Die Verdauung der Membranen und die Gewinnung der Zellen werden mit einem Gewebe-Dissoziation-System erreicht werden. Protokoll des Herstellers zu folgen.
Hinweis: Je feiner die Stücke des Gewebes nach Wolfen, je höher die Anzahl der Zellen wiederhergestellt nach der Verdauung. - Übertragen Sie der Gewebemasse Hackfleisch / Membran mit dem Skalpellklingen in einem Gewebe Dissoziation Rohr und vermischen Sie sich mit 4,7 mL RPMI 1640 Medium. Die Dissoziation Enzyme (siehe Tabelle of Materials) mischen.
- Montieren Sie die Rohre auf die Gewebe Dissociator und führen Sie Programm "h_tumor_01". Inkubieren Sie die Röhrchen bei 37 ° C auf eine rockende Plattform für 30 min.
- Weiter verdünnt die Suspensionen mit 35 mL RPMI 1640 und gelten für ein 70 µm-Sieb über einem Zentrifugenröhrchen 50 mL Sammlung gelegt.
- Zentrifuge zubereitet für 5 min bei 200 X g bei Raumtemperatur, überstand verwerfen, das Pellet in 5 mL RPMI 1640 Aufschwemmen, zählen die Zellen unter Verwendung einer Hemacytometer und Platte bei einer Dichte von 10.000 Zellen/cm2 in Gewebekultur-behandelten Gefäße mit frisch AFMC Medium mit Antibiotikum antimykotische Lösung ergänzt.
Hinweis: Im Falle eines unvollständigen Verdauung, kleine Stücke des Gewebes werden anwesend sein und Einzelzellen werden knapp. Spin-down wieder und die ganze Tablette in einem T75 Kolben Platte.
-
Kultur der primären AFSC und AMSC
- Durchgang Kolonien des AFSC/AMSC durch Absaugen von Vakuum verbrachte mittlere mit einer Pasteurpipette und Zugabe von 2 mL der Zelle Ablösung Enzym in den Kolben. Inkubation bei 37 ° C für 5-8 Minuten.
- Tippen Sie auf die Flasche zu helfen, die Zellen zu verdrängen und mischen die Aufhängung mit einem gleichen Volumen des AFMC Mediums (Antibiotikum Antimykotikums Ergänzung sollte nicht von diesem Zeitpunkt an notwendig sein). Zentrifugieren Sie bei 200 g für ca. 4-5 min. Entfernen des Überstands mit entweder einem Glas Pasteurpipette oder einfach durch Umkehrung der Röhre und in einen Abfallbehälter entleert.
- Blättern Sie unten die Zentrifugenröhrchen zu brechen das Pellet in einer Einzelzelle Suspension in die restlichen Tropfen Flüssigkeit und mischen Sie mit AFMC Medium für Beschichtung. Platte in T-Flaschen mit einer Dichte zwischen 2.500 und 5.000 Zellen/cm2.
- Wechsel-Medium jeden zweiten Tag. Die Zell-Linien über Durchgang 6 nicht Kultur. Umprogrammierung Zwecke, als niedrige verwenden Sie eine Passage wie möglich.
- Bereiten Sie gefrorenen Vorräte an AFSC und AMSC als Backups mit eiskalten Medium. Kulturen, die mit einer Zelle Ablösung Enzym, Zentrifuge bei 200 g bei 4 ° C für 5 min zu ernten.
- Den Überstand mit einer Pasteurpipette zu entfernen und den Boden des Röhrchens auf die Zellen im Pellet singularisieren flick. Im kompletten Einfrieren Medium bei einer Dichte von 1 × 106/mL und Aliquot in Cryovials aufzuwirbeln. Lagern Sie in einem eiskalten Behälter über Nacht bei-80 ° C. Fahren Sie mit flüssigem Stickstoff für die langfristige Lagerung.
2. Reprogrammierung in Pluripotenz
- Erhalten Sie die Neuprogrammierung Plasmide
- Die Neuprogrammierung Plasmide durch ein Non-Profit-Plasmid-Repository zu kaufen. Ein Material Transfer Agreement ist erforderlich.
- Verwandeln Sie die Plasmide in kompetente E. Coli Zellen und isolieren Sie die Plasmide mit einem kommerziellen Plasmid-Extraktion-Kit. Folgen Sie den Anweisungen des Herstellers.
- Messen Sie die Konzentration von Plasmid DNA mit einem Spektralphotometer. Ziel für eine hohe Konzentration resultierende Plasmid, idealerweise ca. 1 µg/µL, Verdünnung der Probe während der Transfektion zu vermeiden.
- Die Konzentrationen der einzelnen Plasmide unter Verwendung eines UV-Spektralphotometer und aliquoten messen sie individuell.
- Mischen Sie 3 µg, 3 µg und 2 µg von EN2K, ET2K und M2L Plasmide, beziehungsweise. Dies ist die Neuprogrammierung Plasmid-Lösung. Die Menge der Plasmid-Lösung reicht, 1 x 106 Zellen zu transfizieren. Mehrere solcher Aliquote vorzubereiten.
- Speichern Sie alle Aliquote bei-80 ° C.
- Zieltafel Kultur vorbereiten
- Ein 6-Well-Platte mit Vitronectin beschichten – fügen Sie 1 mL Vitronectin Verdünnungspuffer in jede Vertiefung und mischen in 40 µL der Stammlösung VTN (1 µg/cm2). Lassen Sie bei Raumtemperatur (RT) oder im Brutschrank bei 37 ° C für 1 h.
- Vakuum-Aspirat die Lösung unter Verwendung einer Pasteurpipette und ersetzen durch 2 mL des AFMC Mediums in jede Vertiefung. Speichern Sie bei 37 ° C, bis die Zellen überzogen werden sollen.
Hinweis: Wichtig: The AFMC Medium verwendet in diesem Schritt sollte keine antibiotischen oder antimykotischen Lösungen enthalten.
- Ernte kultiviert primäre AFSC/AMSC
Hinweis: Vergrößern Sie AFSC/AMSC in Kultur genug machen gefrorenen Vorräte bei einer niedrigen Passagenanzahl und eine T-75 Flasche Umprogrammierung zu widmen. Seit sowenig wie sind 100.000 Zellen ausreichend für ein Experiment, Ziel bei der Ernte rund 500.000 Zellen, um Verluste auszugleichen und wenn Optimierung der Transfektion Parameter oder verschiedenen Kulturbedingungen sollen getestet werden.- Umgehend an einen niedrigen Gang ernten Sie die AFSC/AMSC Zelle Ablösung Enzym-Mix verwenden, wie unter Punkt 1.3.1, 1.3.2 beschrieben. Nachdem die Zellen zentrifugiert haben, fahren Sie mit dem nächsten Schritt fort.
- Das Pellet in 1 mL PBS und gut mischen, Serumkomponenten waschen Aufschwemmen. Zählen Sie die Zellen mit einem Hemacytometer. Passen Sie die Zelldichte bis 100.000/mL PBS und Aliquot in 1,5 mL Mikrozentrifugenröhrchen. Dadurch ist nur einen minimalen Kontaktzeit zwischen den Zellen und der Puffer für die Transfektion verwendet.
- Den Microcentrifuge Schlauch auf 5 mL Polystyrol Rohre (als Adapter, lässt Zentrifugation in eine normale Schaukel-Rotor) und Zentrifuge bei 200 X g für 4 min bei Raumtemperatur. Invertieren Sie der Rohre und entsorgen Sie den Überstand in einen Abfallbehälter. Verwenden Sie einen Rotor mit festen Winkeln nicht.
- Führen Sie eine zusätzliche Zentrifugationsschritt bei 200 X g für 3 min bei Raumtemperatur. Dadurch wird die verbleibende Flüssigkeit aus den Wänden des Rohres an der Unterseite zu sammeln. Abzusaugen Sie sorgfältig alle davon mit einer 200 µL Pipette.
- Transfektion mit Umprogrammierung Plasmide
- Umprogrammierung Experimente, werden eine Transfektion System (siehe Tabelle der Materialien) zur Neuprogrammierung Plasmide in die Zellen zu liefern. Platzieren Sie Transfektion Tipps, Transfektion Rohre, Wiederfreisetzung Puffer und elektrolytische Puffer in der Gewebekultur Schrank. Die Kit-Reagenzien sind bei RT gehalten bis sie geöffnet werden, dann sie bei 4 ° c gelagert werden
Hinweis: Wir verwenden die 10 µL-Version des Kit. - Verschieben Sie die Transfektion Gerät zu schließen, so dass die u-Bahnstation direkt in den Schrank gestellt werden kann. Füllen Sie eine Transfektion Röhre mit 3 mL elektrolytische Puffer und montieren Sie den Schlauch in die Station ganz in den Schlitz schieben.
- Nehmen Sie die Neuprogrammierung Plasmid Lösung Aliquote in 2.1.5 Rücksprung-80 ° C Lagerung vorbereitet und es ermöglichen Sie ihnen, bei RT in der Kultur Schrank Auftauen.
- Wählen Sie auf der Transfektion Gerät folgende Transfektion Parameter: 950 V, 40 ms und 1 Impuls.
- Aufzuwirbeln Sie das Pellet mit 100.000 Zellen in 10 µL Wiederfreisetzung Puffer. Arbeiten Sie schnell von diesem Punkt auf seit Wiederfreisetzung Puffer leicht giftig ist und eine erhöhte Belichtungszeit ergibt sich eine deutlich niedrigere Zellviabilität.
- Mischen Sie in 1/10 der Neuprogrammierung Plasmid-Lösung (die Lösung aliquoten bereitete für eine Gesamtmenge von 1 x 106 Zellen).
- Montieren Sie einen Transfektion Tipp auf die Transfektion Pipette.
- Aspirieren Sie die Zellsuspension in die Transfektion Spitze sorgfältig, Bildung von Luftblasen zu vermeiden. Wenn Bläschen beobachtet werden, die Aussetzung zu vertreiben und die Aspiration wiederholen. Luftblasen werden Transfektion behindern.
- Legen Sie die Transfektion Pipette in die Transfektion Röhre und drücken Sie die Schaltfläche "START" auf dem Bildschirm des Geräts Transfektion. Warten Sie, bis der Bildschirm-Meldung über den Erfolg der Transfektion und entfernen Sie die Pipette aus der Tube sofort.
- Vertreiben Sie die Aufhängung in 1 auch von der Ziel-6-Well-Platte in Abschnitt 2.2 vorbereitet. Das Medium von einem benachbarten gut mischen Sie und verteilen Sie die Suspension gleichmäßig in beide Vertiefungen (die resultierende Zelldichte werden 50.000/gut).
- Wiederholen Sie die Transfektion für alle Mikrozentrifugenröhrchen AFSC/AMSC individuell mit. Legen Sie die Platte in den Brutschrank bei 37 ° C und 5 % CO2.
- Umprogrammierung Experimente, werden eine Transfektion System (siehe Tabelle der Materialien) zur Neuprogrammierung Plasmide in die Zellen zu liefern. Platzieren Sie Transfektion Tipps, Transfektion Rohre, Wiederfreisetzung Puffer und elektrolytische Puffer in der Gewebekultur Schrank. Die Kit-Reagenzien sind bei RT gehalten bis sie geöffnet werden, dann sie bei 4 ° c gelagert werden
- Kultur der transfizierten AFSC/AMSC
- Kultur der transfizierten Zellen für 2-5 Tage. Wechseln Sie dann zur Neuprogrammierung Medium bestehend aus E8 ergänzt mit 100 µM von Natrium Butyrate am 3. Tag.
Hinweis: Zur Vermeidung von Überwucherung der Quelle AFSC/AMSC kann sekundäre Durchgang durchgeführt werden. Allerdings wird die Passagierung deaktivieren Sie die Option für die Neuprogrammierung Effizienz korrekt zu berechnen, wenn dieser Parameter von Interesse ist. - Ändern der Neuprogrammierung Mediums täglich, jeden zweiten Tag für 10 Tage. Wechseln Sie das Medium jeden Tag von Tag 10 auf.
- Kultur der transfizierten Zellen für 2-5 Tage. Wechseln Sie dann zur Neuprogrammierung Medium bestehend aus E8 ergänzt mit 100 µM von Natrium Butyrate am 3. Tag.
- Manuellen Kommissionierung komplett neuprogrammiert Kolonien für klonale expansion
- Voll umprogrammierten Kolonien erscheinen um 14. Tag. Kolonien zu erweitern in Größe und kompakter zu ermöglichen. Sie können manuell abgeholt und so früh wie Tag 15-16 auf frische Platten übertragen.
- 1 h vor der Kommissioniervorgang Mantel 24-Well-Platten mit 8 µL VTN in 300 µL Vitronectin Verdünnungspuffer pro Bohrloch (1 µg/cm2) und bei RT oder 37 ° c inkubieren Ersetzen Sie die Lösung mit E8 Medium ohne Natrium Butyrat.
- Wählen Sie Kolonien eine ausreichende Größe (im Idealfall über 400 µm im Durchmesser) in einer sterilen Kultur Kabinett. Eine Phase-Kontrast-Mikroskop oder einem Stereomikroskop kann verwendet werden.
- Für die Kommissionierung, wird ein LCD imaging Mikroskop in den Schrank gelegt verwendet werden, da der Monitor die Notwendigkeit für Okulare eliminiert. Sterilisieren Sie den Mikroskoptisch mit 70 % Ethanol.
- Mit einem regelmäßigen Phasenkontrast-Zelle Kultur Mikroskop, auswählen, markieren und notieren Sie die Anzahl der Kolonien abgeholt werden. Dies ist wichtig, um sicherzustellen, dass keine Zeit für diesen Prozess während die eigentliche Kommissionierung verschwendet wird.
- Eine Reihe von PCR-Röhrchen, die gleich oder größer als die Anzahl der Kolonien zu kommissionierenden mit 30 µL 0,5 mM Ethylenediaminetetraacetic Säure (EDTA) mit PBS-Puffer zu füllen. Kolonien werden in diese Rohre für partielle Dissoziation vor Beschichtung platziert werden.
- Planen, wählen Sie 5 Kolonien zu einem Zeitpunkt von den Platten mit einer 10 µL Pipette legen Sie auf 2 µL. die Pipettenspitze am Rande Kolonie schräg halten und vorsichtig und allmählich die ganze Kolonie von der Oberfläche zu kratzen. Sofort die ganze Kolonie in der Pipettenspitze Aspirieren und in eine der vorbereiteten PCR Röhren mit EDTA übertragen.
- Wiederholen Sie mit den restlichen 4 Kolonien. 4-6 min bei RT inkubieren.
- Pipettieren der Suspension nach oben und unten sanft mit einem größeren Pipettenspitze zu die Kolonie kleiner Klumpen brechen. Vermeiden Sie eine einzelne Zelle-Suspension.
- Platte die Suspension direkt in ein Ziel auch von einer 24-Well-Platte in Schritt 2.6.2 vorbereitet. Wiederholen Sie mit den übrigen Kolonien.
- Wiederholen Sie die Schritte 2.6.6 durch 2.6.10, wenn mehr als 5 Kolonien abgeholt werden, aber nicht mehr als 5 Kolonien zu einem Zeitpunkt abholen. Inkubation bei 37 ° C und 5 % CO2.
- Klonalen Expansion und Reifung der iPSC
- Kolonien zu wachsen und kompakter zu ermöglichen. 3-6 Tage sind ausreichend. Wechseln Sie täglich das Kulturmedium. 400 µL bis 1 mL der E8 Medium basierend auf die Zelldichte verwenden.
- Brunnen von 24-Well-Platte mit einer ausreichenden Dichte Kolonie werden in 6-Well Platten erweitert. 1 h vor Passagierung, bestreichen Sie die 6-Well-Platten mit VTN (wie in Schritt 2.2.1). Ersetzen Sie die Lösung in den Brunnen mit 2 mL E8 Medium pro Bohrloch.
- Das abgebrannte Medium aus der Quelle-Brunnen mit einer 1 mL-Pipette abzusaugen und ersetzen mit 300 µL 0,5 mM EDTA zu waschen. Aspirieren Sie sofort mit der gleichen Pipettenspitze und ersetzen mit 300 µL 0,5 mM EDTA wieder, dann bei RT inkubieren Sie für 5 min. aspirieren Sie alle Flüssigkeit, die mit einer 1-mL-Pipette.
- Set 1-mL-Pipette, Kapazität, einen 1 mL weite Bohrung Tipp drauf montieren und aspirieren E8 Medium aus dem Ziel weit in die Spitze. Die Source-iPSC-Kultur mit einem Strom des Mediums abwaschen.
- Übertragen Sie die Aussetzung in das Ziel gut und Pipettieren nach oben und unten mehrmals Kolonien in Klumpen von 20-50 Zellen aufzubrechen. Stellen Sie sicher, sie bekommen in den Brunnen durch sanftes Schaukeln und schütteln die Platte gleichmäßig verteilt und Inkubation bei 37 ° C und 5 % CO2.
- Änderung des Mediums täglich und Stelle alle 3 bis 4 Tage. Jede Differenzierung Kolonien können unter dem Phasenkontrast-Mikroskop markiert und mit einer Pipettenspitze in der Kultur Schrank entfernt. Dies ermöglicht eine selektive Vermehrung von qualitativ hochwertigen reinen iPSC-Kultur.
- 1 h vor Passagierung, Mantel der Brunnen von einem 6-Well-Platte mit VTN wie in Schritt 2.2.1. Ersetzen Sie die Lösung mit 2 ml E8 Medium pro Bohrloch.
- Routinemäßige Passagierung ist ähnlich der ersten Passagierung getan mit 0,5 mM EDTA (Schritte 2.7.2, 2.7.5). Ersetzen Sie verbrauchte Mittel in einen Brunnen von einem 6-Well-Platte mit 1 mL EDTA zu waschen und zu verwerfen.
- Fügen Sie 1 mL EDTA und 5-7 min für partielle Dissoziation bei RT inkubieren. Die Inkubationszeit zu optimieren wenn notwendig, sicherzustellen, dass eine Aussetzung der rund 20-50 Zellen Klumpen entsteht. Dissoziation in einzelnen Zellen zu vermeiden.
- Die EDTA-Lösung zu verwerfen und 1 mL des Mediums E8 mit einer 1-mL-Pipette aus dem Ziel gut in eine weite Bohrung Pipettenspitze Aspirieren.
- Abwaschen der Source-iPSC-Kultur mit einem Strom des Mediums E8 wiederholt, bis eine gewünschte Portion von der Oberfläche erschien und gut in das Ziel zu übertragen. Dieser Teil steht für das Split-Verhältnis (z.B., 1/8 der Kultur kann übertragen werden für ein Verhältnis von 1:8.)
- Durchgang alle 3-4 Tage. IPSC Linien Reifen durch Kultivierung sie für mindestens 15 Passagen vor der Verwendung in nachgeschalteten Experimente zu ermöglichen
3. Charakterisierung und Bestätigung der Pluripotenz
Hinweis: Beziehen sich auf die zusätzlichen Dateien für Details auf Durchflusszytometrie und konfokalen Mikroskopie.
-
Teratom Bildung assay
- Um die Kapazität der iPSC durch Teratom Bildung Assay in Geweben, die repräsentativ für alle drei Mikrobeschichten unterscheiden zu bestimmen, folgen Sie die institutionellen Richtlinien für Tierpflege und Nutzung, Vorausplanung, damit Zeit für die Einreichung des entsprechenden Protokolls Dokumente. Die Teratom Bildung bei Mäusen dauert zwischen 6 bis 10 Wochen.
- Kultur 4 Brunnen von einer 6-Well-Platte pro iPSC-Linie für 4 Tage. Die ungefähre Anzahl der Zellen in einer Flanke einer Maus injiziert ist 0,5 bis 1 x 106 Zellen. Optional: Aufpreis kann auch gewidmet werden Bestimmung einer Anzahl von repräsentativen Zellen in einem gut durch einzelne Zelle Dissoziation mit einer Zelle Ablösung Enzym und zählen.
- Berechnen Sie das Volumen der E8/BMM Mischung der iPSC-Klumpen in ausgesetzt werden: beide Flanken einer Maus injiziert werden, jeweils mit 150 µL Büschel Suspension. Drei Mäuse sind ausreichend, um Teratom Bildung von iPSC zeilenweise testen. Schließen Sie eine zusätzliche 150 µL pro Nadel in die resultierende Lautstärke zum Ausgleich Totvolumen. 1 Nadel pro Maus dient. Das Gesamtvolumen beträgt also 3 * (150 µL * 2 + 150 µL) = 1350 µL
- Distanzieren Sie die iPSC-Kolonien mit EDTA teilweise als wären sie zu passagiert werden, wie in den Schritten 2.7.8, 2.7.10 beschrieben. Es ist wichtig, dass die Kolonien werden in Klumpen getrennt und nicht in einzelne Zellen aufgeteilt.
- Verlauf der iPSC-Kolonien mit 675 µL E8 Medium (die Hälfte des berechneten ab Schritt 3.3.3), verwenden einen Wide-Bohrung-Tipp. Übertragen Sie die Büschel Suspension in einer 5 mL Polystyrol Rohr. Auf Eis legen.
- Kombinieren Sie mit 675 µL BMM. Halten Sie die entstandene Suspension auf Eis, bis die Injektion.
- Die Mäuse um sie zu immobilisieren zu betäuben mit Isofluran. Dies sollte durchgeführt oder geleitet von Fachkräften des Vivariums.
- Vortex 5 mL Polystyrol Rohr kurz und aspirieren die Büschel-Suspension (450 µL pro Maus) in einer Insulin-Spritze mit einer 22 G Nadel ausgestattet. 150 µL Zellsuspension subkutan zu injizieren. Dieser Band enthält ca. 1 x 106 Zellen (siehe Punkt 3.1.2).
- 3 Mäuse pro iPSC Zeile zu injizieren. Folgen Sie richtige Tierpflege Praxis für alle Verfahren.
- Der Zustand der Mäuse täglich überwacht.
- Wenn die darum einen Endpunkt von 1,5 bis 2 cm Durchmesser erreichen, einschläfern die Mäuse, explant der darum, und speichern sie in Formalin-Lösung für Gewebe Fixierung für 24 h.
- Eine Histologie zentrale Einrichtung für Hämatoxylin Eosin (H & E) Färbung bringen Sie der festen darum. Ein Pathologe wird das Vorhandensein von Geweben aller drei Mikrobeschichten benoten.
Hinweis für Karyotyping: Live iPSC Kulturen zu versendet werden sollten spezialisierte zytogenetischen Labors zur Prüfung der Integrität der Karyotyp. Es wird empfohlen, dass diese Tests durchgeführt werden, alle 5 Gänge.
-
Transkriptionelle Profilierung
- Kultur 2 Brunnen von einer 6-Well-Platte für 3-4 Tage eine RNA-Probe zu erhalten. Verwenden Sie nur qualitativ hochwertige Kulturen, geringfügige Verunreinigungen mit Differenzierung der Zellen durch Schaben sie mit einer Spitze Pipettieren angesprochen werden kann.
- Isolieren Sie RNA aus iPSC-Kulturen mit einem kommerziell erhältlichen Kit nach der Hersteller-Protokolls. Proben zu einer spezialisierten genomische Kern-Anlage zu versenden.
- Erhalten Sie die globale transcriptional Profile der iPSC Linien mittels Microarrays (siehe Tabelle der Materialien für die unterstützten Optionen) oder RNA Sequenzierung.
- "*.Idat" Dateien für bioinformatische Beurteilung der Pluripotenz durch eine Online-Schnittstelle am Coriell Institut einreichen. Alternativ senden "* .cel" Dateien für bioinformatische Identifikation des Zelltyps, einschließlich pluripotente Stammzellen durch eine Online-Schnittstelle an der Johns Hopkins University. Weitere Informationen über die Arten von Daten, die die einzelnen Bioinformatic Assays akzeptieren Tabelle der Materialien .
Ergebnisse
Informierter Zustimmung wurde von Patienten erhalten vor der Ernte Fruchtwasser für genetische Testzwecke und eine kleine aliquoten des Fluids für die Forschung zu widmen. Ohne Zustimmung ist erforderlich für die Nutzung der amniotic Membrane in der Forschung, da die Plazenta medizinischen Abfälle darstellt. Amniotische Flüssigkeit und Membran Stammzellen typische mesenchymalen Anzeigeeigenschaften, morphologisch ihre Zellen sind spindelförmig und Phase-hell. Bei der Umprogrammierung, die Zellen mesenchymalen-epithelialen (MET) Übergang zu unterziehen und Kopfsteinpflaster-ähnliche Morphologie und räumliche Organisation der Kolonien, unter Angabe der epitheliale Eigenschaften zu erwerben. Dieser Prozess ist bereits 48-72 h nach der Einführung der Neuprogrammierung episomal Plasmide eingeleitet. Fehlen diese Kolonien bis zum Tag 5 der Neuprogrammierung würde Scheitern des Versuchs zeigen. Die Zellen der MET Kolonien vermehren und infolgedessen die Kolonien zu kompakt, zwischen 5 und 14 Tag. Kompakte MET Kolonien bestehen aus Zellen, die nicht leicht individuell erkennbar (Abbildung 1A). Auf ganzen Tag 14, erscheinen voll umprogrammierten Kolonien mit Zellen in einem monomolekularen Film, mit Prominenten, angeordnet leicht erkennbaren Kernen und Nukleoli. Sie sind bereit, mechanisch zu isolieren und erweitert, wenn die Kolonien eine geeignete Größe erreichen und kompakter (Abbildung 1 b).
Voll und teilweise umprogrammiert Kolonien sind in den Kulturen während der gesamten Neuprogrammierung, obwohl teilweise umprogrammiert Kolonien nicht unbedingt vollständige Pluripotenz erwerben. Abbildung 2 zeigt ein Vertreter fließen durchflusszytometrischen Analyse von embryonalen Stammzellen (ESC) Marker Ausdruck in voll und teilweise pluripotenten Kolonien und ihre entsprechenden Morphologien. Volle Pluripotenz ist verbunden mit dem Ausdruck von Nanog, Oct4, Sox2, TRA Antigene und SSEA-4, während SSEA-1 Ausdruck negativer19,20,21 (Abbildung 2A). Teilweise ausdrücklich pluripotente Zellen, jedoch nicht Nanog und TRA Antigene20 (Abb. 2 b). Der Ausdruck und die Lokalisierung der ESC Marker sollte durch Immunocytochemical Färbung und abgebildet mit einem Weitfeld- oder confocal Mikroskop (Abbildung 3) bestätigt werden.
Eine funktionale Bestätigung der Pluripotenz wird erreicht durch den Nachweis der Fähigkeit der iPSC Linien, Form darum nach subkutane Injektion von Zellen in scid-Beige Mäuse. für die darum, die Endpunkt-Größe zu erreichen sind ca. 6-8 Wochen notwendig. H & E Färbung des Gewebes und der Untersuchung durch einen Pathologen wird dann durchgeführt, um das Vorhandensein von Geweben repräsentativ für alle drei Mikrobeschichten – Entoderm, Neuroectoderm und Mesoderm (Abb. 4A) bestätigen. Eine Alternative zu Tierversuchen ist zu analysieren, die transkriptionelle Signatur zugeordnete Pluripotenz von genomischer Ansätze wie cDNA Microarrays22,23. Der Anteil der transkriptionellen Profil, das überschneidet sich mit einem aus einem Pool von etablierten iPSC und ESC-Linien kann dann durch die Online-bioinformatische Pluripotenz Auswerte-Software in Form von einem Grundstück von zwei Klassifikatoren – Pluripotenz quantifiziert werden und Neuheit (Abbildung 4 b). Je höher die Punktzahl Pluripotenz, ähnelt das mehr der iPSC Abfragezeile der etablierten Linien. Eine hohe Neuheit Punktzahl deutet jedoch Abweichungen oder sogar Chromosomenaberrationen, trotz einer hohen Pluripotenz Punktzahl (z. B. Teratocarcinoma Linien)22. Alle iPSC-Linien generiert anhand der hier vorgestellten Protokoll gibt pluripotenten Durchflusszytometrie, Bildgebung, Teratom Bildung und transkriptionelle Analysemethoden gehalten wurden.

Abbildung 1: morphologische Fortschreiten der Zellen während der Reprogrammierung. (A) Fruchtwasser und Membran-Stammzellen, die Quellzellen Umprogrammierung darstellen, zeigt eine typische mesenchymale Morphologie, länglich und Phase-hell (links) erst mesenchymalen-epithelialen Übergang (MET) zu unterziehen, führt zum Erwerb von epithelialen Eigenschaften und die Bildung von Kolonien mit Kopfsteinpflaster Stein-ähnliche Zellen (Mitte). Diese Kolonien vermehren und unregelmäßige zelluläre Massen von MET Zellen (rechts) zu erstellen. (B) in den späteren Phasen der Neuprogrammierung (ab Tag 14) Kolonien vollständig reprogrammierten Zellen entstehen – einzeln erkennbare Zellen mit prominenten Kerne und Kernkörperchen in Monolayer, mit klar definierten Grenzen (Mitte) – angeordnet und sind Neben MET-Kolonien, die immer zahlreicher (links) sind vorhanden. Ein voll umprogrammierten isolierter Reifen Klon ist auf der rechten Seite dargestellt. Maßstabsleiste = 100 µm Klicken Sie bitte hier, um eine größere Version dieser Figur.
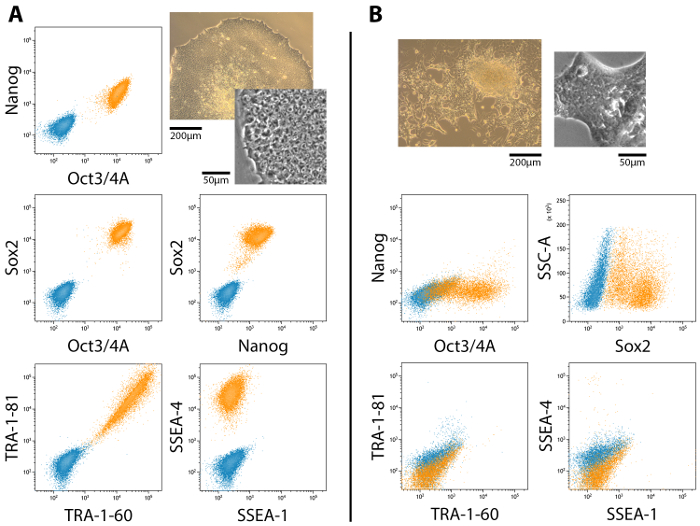
Abbildung 2: Flow durchflusszytometrischen Analyse des Ausdrucks des ESC-Marker in voll und teilweise umprogrammiert (MET) Zelle Kolonien. (A) die pluripotenten Expressionsprofil ist positiv für Oct4, Nanog, Sox2, TRA-1-60, TRA-1-81 und SSEA-4, während negative bei SSEA-1. (B) teilweise pluripotente Zelle Kolonien – jene, die die MET unterzogen, aber nicht die volle Pluripotenz Fortschritt – sind positiv für Oct4 und Sox2 aber Nanog, TRA und SSEA Antigene fehlen. Die damit verbundenen Morphologien sind für Side-by-Side-Vergleich enthalten. Skalieren von Balken = 200 µm und 50 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
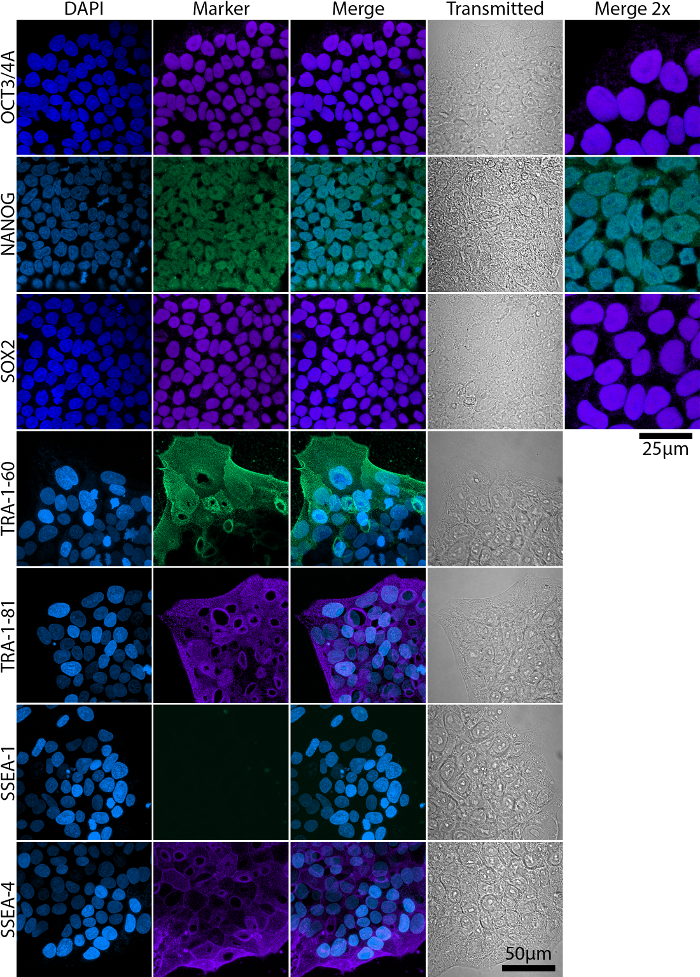
Abbildung 3: Confocal imaging Analyse des Ausdrucks der ESC-Marker in der ältere amniotische Flüssigkeit iPSC. Transkriptionsfaktoren Oct3/4A, Nanog und Sox2 sind in den Kernen während TRA lokalisiert und SSEA Antigene sind Glykoproteine auf der Membran lokalisiert. Maßstabsleiste = 50 µm. Bilder von stärkerer Vergrößerung (Merge 2 X) wurden zur besseren Visualisierung ihrer nuklearen Lokalisierung für Oct3/4, Nanog und Sox2 enthalten. Maßstabsleiste = 25 µm. übertragbare – Bilder auf Durchlicht erworben. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: Teratom Bildung und transkriptionelle Profilerstellung in Reife amniotische Flüssigkeit und Membran iPSC. (A) darum gewachsen in scid-Beige Mäusen subkutan enthalten Gewebe repräsentativ für alle drei Mikrobeschichten (100 X Vergrößerung). (B) die globale Expressionsprofile Microarray Online-Pluripotenz Software vorgelegt kehrte ein Grundstück von zwei Klassifikatoren – Pluripotenz und Neuheit. Hohe Pluripotenz Scores und niedrigen Neuheit Scores - red cloud - ein Expressionsprofil einer typischen ESC/iPSC-Zeile angeben. Die blaue Wolke stellt einen Cluster-Bereich für differenzierte Zellen, während die schwache blaue Wolke einen Cluster-Bereich für teilweise pluripotente Zellen. Das Fruchtwasser (3 Zeilen) und Membran (4 Zeilen) iPSC galten pluripotenten durch den Test. Eine ESC-Linie WA25 wurde als ein Steuerelement enthalten und wird hier mit einem schwarzen Pfeil gekennzeichnet. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Die erste Phase der iPSC-Generation von fetalen Stammzellen beinhaltet die Gewinnung von die Quellzellen aus fetaler Gewebe, ihre Kultur, Ausbau und Einführung von episomal Neuprogrammierung Plasmide. Dieser Phase folgt eine Kulturdauer von ca. 14-18 Tage, bevor die ersten voll umprogrammierten Kolonien erweitert werden können. Die Abschlussphase ist Reifung der iPSC-Klone. Die ersten Gewinnung von Stammzellen amniotischen Membran wird mittels einer kombinierten mechanische und enzymatische Verdauung von der Amnion erreicht. Wir fanden, dass die höchste Anzahl von Zellen mit der höchsten Rentabilität extrahiert eine Inkubationszeit von 30 min führte. Die Verdauung Verfahren produzieren kleine Stücke von Gewebe- und Klumpen. Wenn der Anteil dieser bezogen auf einzelne Zellen hoch ist, empfiehlt es sich, alle Klumpen und einzelne Zellen in einem Gefäß Überzug, da alle Auswüchse der adhärenten Zellen beitragen können. Plattieren amniotische Flüssigkeit Stammzellen ist einfach, wie die Zellen sind nur mit dem Kulturmedium gemischt und inkubiert bis Kolonien von adhärenten Zellen eine ausreichende Größe erreichen. Regelmäßige Gewebekultur behandelt Plasticware eignet sich hervorragend und wir empfehlen nicht spezielle Oberflächen, obwohl sie für verbesserte Primärzelle Kultur bestimmt sind, da mit diesen unteren künstliche und Schwierigkeiten mit der Passagierung zu beobachten Prozesses.
Die amniotische Flüssigkeit und Membran Stamm Zellen sollte erweitert werden und Aktien eingefroren aber umgehend, die Zellen können als Quellzellen Umprogrammierung verwendet werden. Für die Zwecke der Einführung des episomal Plasmide in die Zellen, die Transfektion Systematik hier mit der Transfektion Parametrierung auf 950 V, hat 40 ms und 1 Impuls sehr gut, mit allen Linien versuchte letztlich erfolgreich reprogrammierten ( mehr als 10 Zeilen). Die wichtigsten konkurrierende Liefersystem Betrieb auf einem ähnlichen Prinzip produzieren kein gelungenes Neuprogrammierung Experiment in unseren Händen.
Transfizierten Zellen sind für die ersten 3-5 Tage auf Vitronectin-beschichtete Gerichte in AFMC Medium ausgesät, dann das Medium wechselt zur E8 mit 100 µM Natrium Butyrat ergänzt. Dies erhöht die Rate der vollen Pluripotenz Erwerb. Die ersten Anzeichen der morphologischen Transformation können bereits 48-72 h gesehen werden. Die Quellzellen durchlaufen der MET und Kolonien von Zellen epithelialen Morphologie erscheinen. Diese allmählich vermehren und zu kompakt. Eine Teilmenge der Kolonien erwirbt die morphologischen Merkmale der voll pluripotente Stammzellen – einzeln erkennbaren Zellen mit prominenten Kerne und Nukleoli, beobachtet flache Kolonien mit klar definierten Grenzen, im Gegensatz zu den unscharfen Grenzen in teilweise Pluripotent MET Kolonien. Nach dem Moment des Erwerbs der vollen Pluripotenz kompakte MET Zelle Kolonien erwerben prominente Kerne und die einzelnen Zellen werden während der Erstellung eine einzigartige morphologische Struktur erkennbar. Dieses Muster ist ein geschultes Auge ein deutliches Zeichen für eine erfolgreiche Anpassung. Jedoch erfordert Identifikation der Kolonien, die zur vollständigen Pluripotenz erfolgreich geschafft haben, ein Ermittler, der PSC Kultur Ausbildung fehlt, sorgfältigen Auswertung wie MET und iPSC Klone miteinander verwechselt werden können. Abbildung 1 und Abbildung 2 geben Sie Beispiele für beide. Wenn MET Klone werden stattdessen gepflückt, gründliche Flow Cytometry-Analyse wird den Fehler offenbaren, und insbesondere die TRA-1-60 und TRA-1-81 Antigene höchstwahrscheinlich nicht vorhanden werden wie in Abbildung 2dargestellt. In der Tat fanden TRA Antigene zuvor strengen Pluripotenz Marker werden. Jedoch möglicherweise teilweise MET pluripotente Zellen des Interesses an Krebs Forschung25.
Diese Kultur Bedingung ist suboptimal für die Quelle AFSC/AMSC und schließlich ihre Vermehrung verlangsamt und erwerben sie eine flachere, Fibroblasten-ähnliche Morphologie. Die Quellzellen bilden Gewebe, die während der neueren Stadien der Neuprogrammierung, von der Oberfläche lösen können, obwohl dies die Neuprogrammierung Prozess nicht negativ beeinflussen. Im Gegenteil, führt der Prozess manchmal zu Freigabe von Speicherplatz für die umprogrammierten Kolonien, während die Beseitigung unerwünschter UN umprogrammierten Zellmaterial. Freistehende Gewebe können leicht mit einem sterilen Pipettenspitze, teilweise verlassen und vollständig pluripotenten Kolonien hinter, erheblich vereinfacht manuelle Auswahl flussabwärts verworfen werden.
Für die manuelle Kommissionierung der voll umprogrammierten Kolonien verwenden wir eine LCD imaging-System, das in den Sicherheitsschrank platziert werden kann, fehlen Teile herausragen, die den Luftstrom stören würde. Außer diesem imaging-System ist keine besondere Ausrüstung notwendig, da die Ernte selbst mit regelmäßigen Pipetten durchgeführt werden kann. Die gepflückten Kolonien sind teilweise in EDTA/PBS-Lösung dissoziiert, bevor Sie in die Ziel-Brunnen, als Klone wachsen überzogen wird. Abhängig von der Linie und der Klon, für einige Passagen können die Kulturen kontaminiert sein, mit Zellen spontan zu unterscheiden. Manuelle Manipulation und serielle Passagierung in der Regel dieses Problem beseitigen. Klone, die gespickt mit umfangreichen Differenzierung sollte weggeworfen werden, jedoch wertvolle Klone mit unterschiedlichem Erfolg durch wiederholte manuelle Kommissionierung von pluripotenten Kolonien statt Entsorgung der Differenzierung von Zellen geborgen werden können. Episomal Plasmide wurden gezeigt, um rund 15 Passagen verloren völlig aus der iPSC-26zu nehmen. Daher ist es ratsam, um die Klone für mindestens diese Anzahl von Passagen wachsen vor der Verwendung für downstream-Anwendungen und -Analysen, mit Ausnahme der Routineüberwachung der TRA-Antigen-Expression und Karyotyp zu ermöglichen. TRA-Antigen-Expression kann leicht durch Durchflusszytometrie überwacht werden, wie hier im Protokoll beschrieben, da der Test nur rund 200.000 Zellen erfordert und durchgeführt werden, kann wenn die Forscher in Zweifel sind, ob die kultivierten Klone pflegen Pluripotenz richtig. Flow-Zytometrie-Analyse des ESC Marker Ausdrucks gilt nicht ausreichen, um die Pluripotenz Kandidat Linien19bestätigen.
Teratom-Bildung-Assay ist der standard schlüssig Pluripotenz Test27. PSC in chemisch definierten, Xeno-freien Bedingungen gewachsen sind besonders anfällig für Dissoziation-induzierte Tod und Injektion von ihnen subkutan als Klumpen ist daher notwendig für ihre erfolgreiche Implantation8,28. Nach Injektion in der Regel 4-6 Wochen sind genug für das Wachstum der Xeno-Grafts sichtbar sein und vor der 8. Woche, alles kann sein geerntet, H & E gefärbt und ausgewertet. Tierschutz, Kosten und lange Zeiträume benötigt Tests sind Gründe für die Entwicklung von Alternativmethoden. Genomische Analysen kombiniert mit fortschrittlichen, Machine Learning betriebene bioinformatische Ansätze bieten eine genaue Bewertung der globalen Expressionsprofile. Die Kosten für den Erhalt solcher Daten ist vergleichbar mit den Kosten des Teratom Bildung Assays, jedoch der genomische Ansatz ist wesentlich schneller und keine Tiere müssen verwendet werden. Ein solcher Test ist ein Bioinformatic Pluripotenz Auswertung Software22. Es ist als eine Online-Schnittstelle (Table of Materials) implementiert. Die wachsende Popularität und sinkenden Kosten der RNA Sequenzierung wird Kontinuität dieses Ansatzes. Eine Alternative zu diesem Pluripotenz Software steht von der Johns Hopkins University23 (cellnet.hms.harvard.edu) und basiert auf einem ähnlichen Ansatz und kann Microarray Daten um das Transkriptom des humanen Proben analysieren zu akzeptieren. Der Vorteil dieser Software ist, dass es die Fähigkeit hat, nicht nur pluripotente Stammzellen zu identifizieren, sondern auch Zellen differenzierte und, seit seiner kuratierten Datensätze aus primären Gewebe, der Grad der Ähnlichkeit zwischen den Zellen in Vitro gewachsen stammen / Gewebe und in-Vivo -Gewebe bestimmt werden können, bietet eine hervorragende Qualitätskontrolle für die Entwicklung der Differenzierung Protokolle oder Gewebetechnik. Der Test hat die Fähigkeit, die Abfragen in 20 verschiedenen Zellen oder Gewebetypen zu klassifizieren. Derzeit, es erfordert Microarray Daten aber die Autoren arbeiten für den Ausbau der Plattformoptionen an RNA Sequenzierung sowie.
Anhand der vorgestellten Protokolls können Forscher iPSC Linien aus amniotische Flüssigkeit und Membran Stammzellen eine sehr hohe Reproduzierbarkeit in vollständig chemisch definierten und Xeno-freies Medium und mit einer nicht-Integration von Neuprogrammierung Methode generieren. Diese Zeilen können in der Grundlagenforschung zur Optimierung der Differenzierung Protokolle und letztlich in der Krankheit Modellierung, Drogen-Screening oder pädiatrischen Gewebetechnik Studien verwendet werden.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessenkonflikte.
Danksagungen
Diese Arbeit wurde durch den Fonds Medizinische Forschung an der UZH Forschungskredit der Universität Zürich, The SCIEX NMSCh unter Stipendien 10.216 und 12.176, der Schweizerischen Gesellschaft für Kardiologie, The Swiss National Science unterstützt. Stiftung unter Grant [320030-122273] und [310030-143992], das 7. Rahmenprogramm, Leben Ventil, Europäische Kommission unter Grant [242008], die Olga Mayenfisch Stiftung, die EMDO Stiftung, Gründungshilfe 2012 von der Universitätsklinik Zürich, und interne Finanzierung des Mitchell Cancer Institute.
Materialien
| Name | Company | Catalog Number | Comments |
| Tumor Dissociation Kit, human | Miltenyi Biotec | 130-095-929 | tissue dissociation system, reagent kit, includes tissue dissociation tubes and tissue dissociation enzymes |
| gentleMACS Dissociator | Miltenyi Biotec | 130-093-235 | tissue dissociation system, dissociator |
| Thermo Scientific™ Shandon™ Disposable Scalpel No. 10, Sterile, Individually Wrapped, 5.75 (14.6cm) | Thermo-Fisher | 3120032 | |
| 70 µm cell strainers | Corning | 10054-456 | |
| RPMI 1640 medium | Thermo-Fisher | 32404014 | |
| rocking platform | VWR | 40000-300 | |
| 50 ml centrifuge tubes | Thermo-Fisher | 339652 | |
| 15 ml centrifuge tubes | Thermo-Fisher | 339650 | |
| EBM-2 basal medium | Lonza | CC-3156 | basal medium for AFMC medium |
| FGF 2 Human (expressed in E. coli, non-glycosylated) | Prospec Bio | CYT-218 | bFGF, supplement for AFMC medium |
| EGF Human, Pichia | Prospec Bio | CYT-332 | EGF, supplement for AFMC medium |
| LR3 Insulin Like Growth Factor-1 Human Recombinant | Prospec Bio | CYT-022 | IGF, supplement for AFMC medium |
| Fetal Bovine Serum, embryonic stem cell-qualified | Thermo-Fisher | 10439024 | FBS |
| Antibiotic-Antimycotic (100X) | Thermo-Fisher | 15240062 | for primary AFSC/AMSC, for routine AFSC/AMSC it should not be necessary, do not use in medium for transfected cells! |
| Accutase cell detachment solution | StemCell Technologies | 07920 | cell detachment enzyme |
| CryoStor™ CS10 | StemCell Technologies | 07930 | complete freezing medium |
| PBS, pH 7.4 | Thermo-Fisher Scientific | 10010023 | |
| EndoFree Plasmid Maxi Kit (10) | Qiagen | 12362 | for plasmid isolation |
| pEP4 E02S EN2K | Addgene | 20925 | EN2K, reprogramming factors Oct4+Sox2, Nanog+Klf4 |
| pEP4 E02S ET2K | Addgene | 20927 | ET2K, reprogramming factors Oct4+Sox2, SV40LT+Klf4 |
| pCEP4-M2L | Addgene | 20926 | M2L, reprogramming factors c-Myc+LIN28 |
| NanoDrop 2000c UV-Vis Spectrophotometer | Thermo-Fisher | ND-2000C | spectrophotometer |
| Neon® Transfection System | Thermo-Fisher | MPK5000 | transfection system, components: Neon pipette - transfection pipette Neon device - transfection device |
| Neon® Transfection System 10 µL Kit | Thermo-Fisher | MPK1025 | consumables kit for the Neon Transfection System, it contains: Neon tip - transfection tip Neon tube - transfection tube buffer R - resuspension buffer buffer E - electrolytic buffer |
| Stemolecule™ Sodium Butyrate | StemGent | 04-0005 | small molecule enhancer of reprogramming |
| TeSR-E8 | StemCell Technologies | 05940 | E8 medium |
| Vitronectin XF™ | StemCell Technologies | 07180 | VTN, stock concentration 250 µg/ml, used for coating at 1 µg/cm2 in vitronectin dilution (CellAdhere) buffer |
| CellAdhere™ Dilution Buffer | StemCell Technologies | 07183 | vitronectin dilution buffer |
| UltraPure™ 0.5M EDTA, pH 8.0 | Thermo-Fisher | 15575020 | dilute with PBS to 0.5 mM before use |
| EVOS® FL Imaging System | Thermo-Fisher Scientific | AMF4300 | LCD imaging microscope system |
| CKX53 Inverted Microscope | Olympus | phase contrast cell culture microscope | |
| Pierce™ 16% Formaldehyde (w/v), Methanol-free | Thermo-Fisher | 28908 | dilute to 4% with PBS before use, diluted can be stored at 2-8 °C for 1 week |
| Perm Buffer III | BD Biosciences | 558050 | permeabilization buffer, chill to -20 °C before use |
| Mouse IgG1, κ Isotype Control, Alexa Fluor® 488 | BD Biosciences | 557782 | isotype control for Oct3/4A, Nanog |
| Mouse IgG1, κ Isotype Control, Alexa Fluor® 647 | BD Biosciences | 557783 | isotype control for Sox2 |
| Mouse anti-human Oct3/4 (Human Isoform A), Alexa Fluor® 488 | BD Biosciences | 561628 | |
| Mouse anti-human Nanog, Alexa Fluor® 488 | BD Biosciences | 560791 | |
| Mouse anti-human Sox-2, Alexa Fluor® 647 | BD Biosciences | 562139 | |
| Mouse IgGM, κ Isotype Control, Alexa Fluor® 488 | BD Biosciences | 401617 | isotype control for TRA-1-60 |
| Mouse IgGM, κ Isotype Control, Alexa Fluor® 647 | BD Biosciences | 401618 | isotype control for TRA-1-81 |
| Mouse anti-human TRA-1-60, Alexa Fluor® 488 | BD Biosciences | 330613 | |
| Mouse anti-human TRA-1-81, Alexa Fluor® 647 | BD Biosciences | 330705 | |
| Mouse IgG1, κ Isotype Control, Alexa Fluor® 488 | BD Biosciences | 400129 | isotype control for SSEA-1 |
| Mouse IgG3, κ Isotype Control, Alexa Fluor® 647 | BD Biosciences | 401321 | isotype control for SSEA-4 |
| Mouse anti-human SSEA-1, Alexa Fluor® 488 | BD Biosciences | 323010 | |
| Mouse anti-human SSEA-4, Alexa Fluor® 647 | BD Biosciences | 330407 | |
| Affinipure F(ab')2 Fragment Goat Anti-Mouse IgG+IgM, Alexa Fluor® 488 | Jackson Immunoresearch | 115-606-068 | use at a dilution of 1:600 or further optimize |
| Affinipure F(ab')2 Fragment Goat Anti-Mouse IgG+IgM, Alexa Fluor® 647 | Jackson Immunoresearch | 115-546-068 | use at a dilution of 1:600 or further optimize |
| DAPI | Thermo-Fisher Scientific | D21490 | stock solution 10 mM, further dilute to 1:12.000 for a working solution |
| Corning® Matrigel® Growth Factor Reduced, Phenol Red-Free | Corning | 356231 | basement membrane matrix (BMM) |
| scid-beige mice, female | Taconic | CBSCBG-F | |
| RNeasy Plus Mini Kit (50) | Qiagen | 74134 | RNA isolation kit |
| T-25 flasks, tissue culture-treated | Thermo-Fisher | 156367 | |
| T-75 flasks, tissue culture-treated | Thermo-Fisher | 156499 | |
| Nunc™ tissue-culture dish | Thermo-Fisher | 12-567-650 | 10 cm tissue culture dish |
| 6-well plates, tissue-culture treated | Thermo-Fisher | 140675 | |
| Neubauer counting chamber (hemacytometer) | VWR | 15170-173 | |
| Mr. Frosty™ Freezing Container | Thermo-Fisher | 5100-0001 | freezing container |
| FACS tubes, Round Bottom Polystyrene Test Tube, 5ml | Corning | 352058 | 5 ml polystyrene tubes |
| Eppendorf tubes, 1.5 ml | Thermo-Fisher | 05-402-96 | 1.5 ml microcentrifuge tubes |
| PCR tubes, 200 µl | Thermo-Fisher | 14-222-262 | |
| pipette tips, 100 to 1250 µl | Thermo-Fisher | 02-707-407 | narrow-bore 1 mL tips |
| pipette tips, 5 to 300 µl | Thermo-Fisher | 02-707-410 | |
| pipette tips, 0.1 to 10 µl | Thermo-Fisher | 02-707-437 | |
| wide-bore pipette tips, 1000 µl | VWR | 89049-166 | wide-bore 1 mL tips |
| glass Pasteur pipettes | Thermo-Fisher | 13-678-20A | |
| ethanol, 200 proof | Thermo-Fisher | 04-355-451 | |
| vortex mixer | VWR | 10153-842 | |
| chambered coverglass, 8-well, 1.5mm borosilicate glass | Thermo-Fisher | 155409 | glass-bottom confocal-grade cultureware |
| 22G needles | VWR | 82002-366 | |
| insulin syringes | Thermo-Fisher | 22-253-260 | |
| Formalin solution, neutral buffered, 10% | Sigma-Aldrich | HT501128-4L | fixation of explanted teratomas |
| Illumina HT-12 v4 Expression BeachChip | Illumina | BD-103-0204 | expression microarray, supported by PluriTest, discontinued by manufacturer |
| PrimeView Human Genome U219 Array Plate | Thermo-Fisher | 901605 | expression microarray (formerly Affymetrix brand), soon to be supported by PluriTest |
| GeneChip™ Human Genome U133 Plus 2.0 Array | Thermo-Fisher | 902482 | expression microarray (formerly Affymetrix brand), supported by CellNet, soon to be supported by PluriTest |
| PluriTest® | Coriell Institute | www.pluritest.org, free service for bioinformatic assessment of pluripotency, accepts microarray data - *.idat files from HT-12 v4 platform, soon to support U133, U219 microarray and RNA sequencing data | |
| CellNet | Johns Hopkins University | cellnet.hms.harvard.edu, free service for bioinformatic identification of cell type, including plutipotent stem cells, based on U133 microarray data - *.cel files, soon to support RNA sequencing data |
Referenzen
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126 (4), 663-676 (2006).
- Yu, J., et al. Induced Pluripotent Stem Cell Lines Derived from Human Somatic Cells. Science. 318 (5858), 1917-1920 (2007).
- Trounson, A., DeWitt, N. D. Pluripotent stem cells progressing to the clinic. Nat. Rev. Mol. Cell Biol. 17 (3), 194-200 (2016).
- Schmidt, D., et al. Prenatally fabricated autologous human living heart valves based on amniotic fluid derived progenitor cells as single cell source. Circulation. 116 (11 Suppl), I64-I70 (2007).
- Weber, B., Zeisberger, S. M., Hoerstrup, S. P. Prenatally harvested cells for cardiovascular tissue engineering: Fabrication of autologous implants prior to birth. Placenta. 32, S316-S319 (2011).
- Weber, B., et al. Prenatally engineered autologous amniotic fluid stem cell-based heart valves in the fetal circulation. Biomaterials. 33 (16), 4031-4043 (2012).
- Kehl, D., Weber, B., Hoerstrup, S. P. Bioengineered living cardiac and venous valve replacements: current status and future prospects. Cardiovasc. Pathol. 25 (4), 300-305 (2016).
- Slamecka, J., et al. Non-integrating episomal plasmid-based reprogramming of human amniotic fluid stem cells into induced pluripotent stem cells in chemically defined conditions. Cell Cycle. 15 (2), 234-249 (2016).
- Jiang, G., et al. Human Transgene-Free Amniotic-Fluid-Derived Induced Pluripotent Stem Cells for Autologous Cell Therapy. Stem Cells Dev. 23 (21), 2613-2625 (2014).
- Pipino, C., et al. Trisomy 21 mid-trimester amniotic fluid induced pluripotent stem cells maintain genetic signatures during reprogramming: implications for disease modeling and cryobanking. Cell. Reprogram. 16 (5), 331-344 (2014).
- Cai, J., et al. Generation of human induced pluripotent stem cells from umbilical cord matrix and amniotic membrane mesenchymal cells. J Biol. Chem. 285 (15), 11227-11234 (2010).
- Ge, X., et al. Human Amniotic Mesenchymal Stem Cell-Derived Induced Pluripotent Stem Cells May Generate a Universal Source of Cardiac Cells. Stem Cells Dev. 21 (15), 2798-2808 (2012).
- Drozd, A. M., Walczak, M. P., Piaskowski, S., Stoczynska-Fidelus, E., Rieske, P., Grzela, D. P. Generation of human iPSCs from cells of fibroblastic and epithelial origin by means of the oriP/EBNA-1 episomal reprogramming system. Stem Cell Res. Ther. 6 (1), (2015).
- Kang, N. -. H., et al. Potential antitumor therapeutic strategies of human amniotic membrane and amniotic fluid-derived stem cells. Cancer Gene Ther. 19 (8), 517-522 (2012).
- Moschidou, D., et al. Valproic Acid Confers Functional Pluripotency to Human Amniotic Fluid Stem Cells in a Transgene-free Approach. Mol. Ther. 20 (10), 1953-1967 (2012).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nature Methods. 8 (5), 424-429 (2011).
- Yu, J., et al. Human Induced Pluripotent Stem Cells Free of Vector and Transgene Sequences. Science. 324 (5928), 797-801 (2009).
- Martí, M., et al. Characterization of pluripotent stem cells. Nat. Protoc. 8 (2), 223-253 (2013).
- Chan, E. M., et al. Live cell imaging distinguishes bona fide human iPS cells from partially reprogrammed cells. Nat. Biotechnol. 27 (11), 1033-1037 (2009).
- Adewumi, O., et al. Characterization of human embryonic stem cell lines by the International Stem Cell Initiative. Nat. Biotechnol. 25 (7), 803-816 (2007).
- Müller, F. -. J., et al. A bioinformatic assay for pluripotency in human cells. Nature Methods. 8 (4), 315-317 (2011).
- Cahan, P., Li, H., Morris, S. A., Lummertz da Rocha, E., Daley, G. Q., Collins, J. J. CellNet: Network Biology Applied to Stem Cell Engineering. Cell. 158 (4), 903-915 (2014).
- Schopperle, W. M., DeWolf, W. C. The TRA-1-60 and TRA-1-81 Human Pluripotent Stem Cell Markers Are Expressed on Podocalyxin in Embryonal Carcinoma. STEM CELLS. 25 (3), 723-730 (2007).
- Ohnishi, K., et al. Premature Termination of Reprogramming In Vivo Leads to Cancer Development through Altered Epigenetic Regulation. Cell. 156 (4), 663-677 (2014).
- Schlaeger, T. M., et al. A comparison of non-integrating reprogramming methods. Nature Biotechnology. 33 (1), 58-63 (2014).
- Müller, F. -. J., Goldmann, J., Löser, P., Loring, J. F. A call to standardize teratoma assays used to define human pluripotent cell lines. Cell Stem Cell. 6 (5), 412-414 (2010).
- Beers, J., et al. Passaging and colony expansion of human pluripotent stem cells by enzyme-free dissociation in chemically defined culture conditions. Nat. Protoc. 7 (11), 2029-2040 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
ISSN 2578-2746
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.