Method Article
Native Polyacrylamidde Gel Electrophesis Análisis de la dimerización endógena IRF5
En este artículo
Resumen
Se describe un método nativo de mancha occidental para analizar la dmerización del factor regulador de interferón endógeno 5 en la línea de células dendríticas plasmáticas CAL-1. Este protocolo también se puede aplicar a otras líneas de celda.
Resumen
El factor regulador del interferón 5 (IRF5) es un factor de transcripción clave para regular la respuesta inmunitaria. Se activa aguas abajo de la vía de señalización de la respuesta primaria de diferenciación mieloide similar al de peaje 88 (TLR-MyD88). La activación de IRF5 implica fosforilación, dimerización y posterior translocación del citoplasma al núcleo, lo que a su vez induce la expresión génica de varias citoquinas proinflamatorias. Un ensayo de detección para la activación de IRF5 es esencial para estudiar las funciones IRF5 y sus vías relevantes. Este artículo describe un ensayo robusto para detectar la activación endógena de IRF5 en la línea de células dendríticas (pDC) plascitoide humanas CAL-1. El protocolo consiste en un ensayo de electroforesis no desnaturalizador modificado que puede distinguir IRF5 en sus formas monómero y dimer, proporcionando así un enfoque asequible y sensible para analizar la activación IRF5.
Introducción
El factor regulador del interferón 5 (IRF5) es un importante regulador de la transcripción que desempeña un papel destacado en la regulación de la respuesta inmunitaria, particularmente en la liberación de citoquinas proinflamatorias e interferones de tipo I (IFN)1,2 ,3. La desregulación de IRF5 es un factor que contribuye a numerosas enfermedades autoinmunes, como lo demuestran varios polimorfismos en el locus IRF5 que están asociados con lupus eritematoso sistémico, esclerosis múltiple, artritis reumatoide,etc. 4, 5,6,7,8,9,10. Por lo tanto, un ensayo de detección robusto para el estado de activación endógeno IRF5 es crucial para entender las vías regulatorias y los efectos posteriores de IRF5 en un contexto celular fisiológicamente relevante.
IRF5 se expresa constitutivamente en monocitos, células dendríticas (CC), células B y macrófagos1,11. Al igual que con otros factores de transcripción de la familia IRF, IRF5 reside en el citoplasma en su estado latente. Tras la activación, IRF5 es fosforilado y forma homodímeros, que luego se trasladan al núcleo y se unen a elementos reguladores específicos de genes que codifican IFN de tipo I y citoquinas proinflamatorias, induciendo finalmente la expresión de estos genes1 ,2,11,12,13. IRF5 regula las respuestas inmunitarias innatas aguas abajo de varios receptores similares a los peajes (TLR), como TLR7, TLR 8 y TLR 9, que se localizan en endosomas y utilizan MyD88 para la señalización1,11,14. Estos TLR reconocen principalmente especies de ácido nucleico extraños como EL ARN de una sola cadena (ssRNA) y el ADN CpG no metilado que son sintomáticos de una infección15,16,17,18. IRF5 ha demostrado regular las respuestas inmunitarias contra infecciones bacterianas, virales y fúngicas19,20,21. Teniendo en cuenta el papel influyente y diverso de IRF5 en el sistema inmunológico, mejorar o amortiguar la actividad del IRF5 podría servir como una vía novedosa para el desarrollo de agentes terapéuticos22. Por lo tanto, es fundamental desarrollar un protocolo para monitorear el estado de activación del IRF5 endógeno para permitir una investigación exhaustiva de las vías y mecanismos que regulan la actividad de IRF5 en diferentes tipos de células.
Hasta nuestro conocimiento, no se ha publicado ningún ensayo electroforético bioquímico o gel para la activación endógena de IRF5 antes del desarrollo de este protocolo. Se ha demostrado que la fosforilación es un primer paso importante de la activación del IRF5, y se desarrolló un anticuerpo fosfoespecífico IRF5 que condujo al descubrimiento y confirmación de un residuo de serina importante para la actividad13de IRF5. Sin embargo, mientras que el anticuerpo detecta claramente iRF5 fosforilado cuando se inmunoprecipita o sobreexpresa23, no detecta la fosforilación IRF5 en un lisato de células enteras en nuestras manos (datos no mostrados). La dimerización es el siguiente paso de la activación iRF5, y muchos estudios importantes hasta la fecha que investigan este paso se basaron en la sobreexpresión de IRF5 etiquetado con epítopos, a menudo en tipos de celda irrelevantes que normalmente no expresan IRF511,12 ,24,25. Estudios anteriores han demostrado que el Dimerizado IRF5 no siempre puede translocaren en el núcleo y por lo tanto no está necesariamente completamente activado25,26. Se desarrolló un ensayo para la localización nuclear endógena IRF5 para evaluar la activación de IRF5 mediante la citometría de flujo de imágenes27. Este ensayo se ha aplicado en estudios que fueron cruciales para entender la actividad del IRF5, especialmente en los tipos de células primarias o raras28,29 y avanzaron en gran medida el conocimiento en el campo. Sin embargo, este ensayo se basa en un instrumento especializado que no está ampliamente disponible para los investigadores. Además, a menudo es necesario investigar los pasos iniciales de la activación mientras se disecciónn las vías reguladoras IRF5 y se identifican los reguladores ascendentes y los componentes de la vía. Este estudio proporciona un ensayo bioquímico robusto y fiable para los eventos de activación temprana de IRF5 que se puede realizar en laboratorios equipados con herramientas de biología molecular. El protocolo descrito aquí será muy útil para investigar las vías y mecanismos de las acciones iRF5, especialmente cuando se combina con ensayos ortogonales como el análisis citométrico del flujo de imágenes de la localización nuclear IRF523, 27,28,30.
La electroforesis nativa de gel de poliacrilamida (PAGE nativa) es un método ampliamente utilizado para analizar complejos proteicos31,32. A diferencia de la electroforesis de gel de poliacrilamida de dodecilosulfato sódico (SDS-PAGE), PAGE nativa separa las proteínas en función de su forma, tamaño y carga. También conserva la estructura proteica autóctona sin desnaturalización31,33,34,35. El protocolo presentado aprovecha estas características de PAGE nativo y detecta las formas monoméricas y diminutas de IRF5. Este método es particularmente importante para detectar eventos de activación temprana porque no hay ningún anticuerpo disponible comercialmente adecuado que pueda detectar IRF5 fosforilado endógeno. Anteriormente, varios estudios publicados utilizaban PAGE nativo para evaluar la dimización de IRF5. Sin embargo, la mayoría de estos estudios dependían de la sobreexpresión de IRF5 con etiqueta epitope exógena para analizar el estado de activación2,13,24,36,37 . Este trabajo presenta un protocolo paso a paso para analizar la dimización endógena IRF5 a través de una técnica PAGE nativa modificada en una línea de célula dendrítica plasmacitoide humana (pDC), donde se ha demostrado que la actividad IRF5 es crucial para su función1, 38,39,40. Esta misma técnica se ha aplicado a otras líneas celulares23.
Protocolo
NOTA: El protocolo descrito aquí utiliza la línea celular pDC CAL-1 tratada con resiquimod (R848), un agonista para TLR7/8. Este protocolo se ha aplicado a otros tipos de células humanas y murinas, incluyendo RAW 264.7 (línea de macrófagos murinos), THP-1 (línea celular monocítica humana), BJAB (línea celular B humana), Ramos (línea celular B humana) y MUTZ-3 (línea de células dendríticas humanas)23.
1. Estimulación de células CAL-1
- Mantener el cultivo celular CAL-1 en un matraz T75 a 37oC y 5% deCO2 en condiciones estériles con 20 a 25 ml de medio RPMI 1640 que contiene un 5% de suero bovino fetal (FBS), 25 mM DE HEPES y 1x mercaptoetanol (es decir, medio RPMI 1640 completo).
- Transfiera las células a un tubo cónico de 50 ml.
NOTA: Las células CAL-1 no son adherentes. Para los tipos de células adherentes, se puede realizar la tripinización estándar para cosechar células. - Centrifugar las células a 200 x g durante 5 min a temperatura ambiente (RT). Retire el sobrenadante y resuspenda el pellet celular en 8 ml del medio RPMI 1640 completo para obtener una suspensión homogénea de una sola célula.
- Cuente las células usando un hemocaquímetro. Sembrar las células a una densidad de 1 x 106 células por pozo en una placa de 6 pozos con 4 ml de medio RPMI 1640 completo precalentado en cada pocto. Incubar durante 20 a 24 h a 37 oC y 5% deCO2 para permitir que la confluencia alcance el 90%-95% (correspondiente a aproximadamente 1,5 x 106 células).
- Estimular las células añadiendo 4 sL de 1 mg/ml R848 por pozo de la placa de 6 pocillos (concentración final de 1 g/ml). También configure un pozo de control no estimulado con células sin el tratamiento R848.
- Asegúrese de que el R848 esté uniformemente disperso balanceando suavemente la placa de lado a lado. A continuación, incubar las células durante 2 a 16 h en la incubadora a 37 oC y 5% deCO2.
2. Extracción de proteínas celulares
- Transfiera las suspensiones celulares de la placa de 6 pozos en tubos centrífugos de 5 ml.
- Centrífuga a 200 x g durante 5 min en RT. Retire el sobrenadante y vuelva a suspender el pellet celular en 1 ml de solución salina tamponada con fosfato (PBS) para obtener una suspensión homogénea de una sola célula.
- Transfiera la suspensión celular a un tubo centrífugo de 1,5 ml.
- Gire brevemente a 12.000 x g durante 0,5 x 1 min a 4 oC y retire cuidadosamente el sobrenadante.
- Preparar el tampón de lisis que contiene 6,25 ml de 1 M Tris-HCl pH 7,4 (concentración final de 25 mM), 7,5 ml de 5 M NaCl (concentración final de 150 mM), 0,5 ml de 0,5 M EDTA (concentración final de 1 mM), 2,5 ml de NP-40 (concentración final del 1%) y 7,5 ml de glicerol (concentración final del 5%) en 250 ml de agua desionizada (ddH2O). Añadir 100x cóctel de un solo uso inhibidor de la proteasa a una concentración final de 1x al tampón de lisis justo antes de su uso. Mantenga el tampón de lisis preparado en hielo.
NOTA: El tampón de lisis sin la proteasa se puede almacenar a 4 oC. - Resuspenda el gránulo celular en 30 ml de tampón de lisis helada y mezcle pipeteando hacia arriba y hacia abajo.
- Incubar sobre hielo durante 15 x 20 min.
- Aclare el lisado centrifugando a 12.000 x g durante 15 x 20 min a 4 oC. Transfiera el sobrenadante a un nuevo tubo de centrífuga pretallado de 1,5 ml. Mantenga los extractos en hielo en todo momento.
NOTA: Los líticos celulares se pueden almacenar a -20 oC o -80 oC. No hierva las muestras. - Mida la concentración de proteínas con el reactivo Bradford.
3. Análisis de la dimerización IRF5 por PAGE Nativo
- Prepare los amortiguadores de electroforesis de cámara superior (-) e inferior (+). El búfer de la cámara superior consiste en 0,3% de desoxicolato de sodio (NaDOC) en 1 búfer de ejecución PAGE nativo, y la cámara inferior consta de sólo 1 x búfer de ejecución PAGE nativo.
NOTA: Prepare un nuevo búfer de cámara superior para cada nueva carrera. - Enjuague bien un gel PAGE nativo de 3%-12% con agua sin distorsionar los pozos. Coloca el gel en el tanque de gel mini y retira el peine. Pre-ejecutar el gel en una cámara fría de 4oC o sobre hielo a 150 V durante 30 min.
NOTA: El prerunning elimina el exceso de amoníaco e iones de persulfato que pueden interferir con el funcionamiento del gel. - Durante el prerun, prepare las muestras para la carga mezclando las proteínas celulares guardadas en hielo con un búfer de muestra nativo 4x.
- Después de la pre-ejecución, cargue 10 a 15 g de proteína con un volumen final de 10 a 15 s por muestra.
NOTA: La sobrecarga de proteínas puede causar frotis. - Ejecutar gel a 85 V durante 30 min, luego 150 V durante 2 h.
NOTA: Teniendo en cuenta las diferencias en el equipo y las líneas celulares utilizadas por diferentes laboratorios, pequeñas modificaciones en la concentración de muestras de proteínas, voltaje y tiempo de funcionamiento pueden ser apropiadas para optimizar este protocolo. Reducir el voltaje de pre-ejecución y funcionamiento mientras aumenta el tiempo de funcionamiento puede ayudar a mejorar la resolución del dimer y la consistencia de los resultados. - Remoje el gel en tampón de carrera SDS (25 mM Tris pH 8.3, 250 mM de glicina, 0,1% SDS) durante 30 min a RT.
NOTA: No se requiere agitación. Ocasionalmente, la intensidad de la banda puede no ser proporcional a la cantidad de proteína cargada debido a la transferencia ineficiente en presencia de desoxicolato (DOC), que afecta principalmente a la forma monomérica de IRF5. Remojar el gel en el búfer de carrera SDS antes de la transferencia resuelve este problema. El gel es frágil. Manipule con extremo cuidado desde el extremo inferior (es decir, mayor porcentaje) del gel.
4. Análisis inmunoblot de IRF5
- Active la membrana de difluoruro de polivinilideno (PDVF) empapándola en metanol durante aproximadamente 5 minutos.
- Haga un corte en una esquina de la membrana para indicar su orientación. Montar el sándwich de transferencia de acuerdo con el orden secuencial detallado en el protocolo del fabricante con un cuidado adicional para asegurarse de que no hay burbujas de aire atrapados en el interior.
- Coloque el cassette de transferencia en el tanque y transfiera a 20 V durante 1 h sobre hielo.
NOTA: Realice todas las incubaciones y lavados en pasos posteriores con una coctelera mecedora. - Retire la membrana del cassette con fórceps de plástico una vez completada la transferencia. Bloquear la membrana en tampón de bloqueo (TBS) durante 45 minutos a RT.
NOTA: El búfer TBS con 5% de BSA también se puede utilizar como búfer de bloqueo. - Incubar la membrana con el anticuerpo primario indicado en la Tabla 1. Lavar la membrana con 1 tampón de lavado TBST (20 mM Tris, pH 7.0, 150 mM NaCl y 0.1% Tween 20) durante 3 min mientras se balancea. Repita el lavado 2x.
| Dillución | Búfer de dilatación | Incubación | Comentarios | |
| Anticuerpo primario (Anti-IRF5) | 1/1,000 | Búfer de bloqueo TBS | Noche a 4oC o 2 h a RT | Los anticuerpos diluidos se pueden reutilizar varias veces si se almacenan a 4 oC en presencia de 0,02% de azida sódica. |
| Anticuerpo secundario (Anticonejo) | 1/10,000 | Búfer de bloqueo TBS | 45 min a RT | Los anticuerpos diluidos se pueden reutilizar varias veces si se almacenan a 4 oC en presencia de 0,02% de azida sódica. |
| NOTA: La dilución debe optimizarse, ya que varía entre los fabricantes. | ||||
Tabla 1: Especificaciones de los anticuerpos utilizados en el procedimiento de inmunoblotting.
- Incubar la membrana con el anticuerpo secundario indicado en la Tabla 1. Lave las membranas durante 3 min en 1 tampón de lavado TBST. Repita el lavado 2x.
- Escanee la mancha utilizando un sistema de documentación de gel adecuado.
Resultados
El inmunoblot (IB) con un anticuerpo anti-IRF5 se realizó en células CAL-1 no estimuladas o estimuladas con 1 g/ml R848 durante 2 h(Figura 1). Se prepararon los lysates celulares y se realizó el PAGE nativo. En las células CAL-1 no estimuladas, IRF5 se detectó como una sola banda en la PÁGINA nativa, correspondiente a su forma monomérica. Tras el tratamiento de las células CAL-1 con R848 durante 2 h, el nivel de monómero IRF5 disminuyó con un aumento simultáneo en la acumulación de una banda de migración lenta que correspondía a la forma dimerica de IRF5.
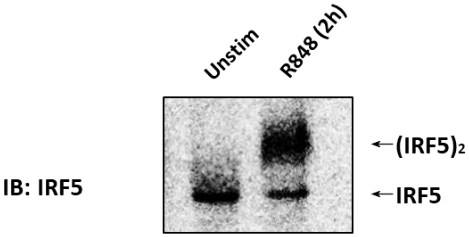
Figura 1: IRF5 endógeno dimerizado en células CAL-1 cuando se estimula con agonista TLR7/8. Las células CAL-1 no fueron tratadas o tratadas con R848 durante el tiempo indicado. Las muestras de proteínas se resolvieron mediante PAGE nativo y siguieron con el IB utilizando el anticuerpo anti-IRF5. Haga clic aquí para ver una versión más grande de esta figura.
El inmunoblot con anticuerpo anti-IRF5 se realizó en células 293T que sobreexpresan 293T no transtrinfectadas y transinfectadas con varias construcciones. Se prepararon los lysates celulares y se realizó PAGE nativo (Figura 2). No se detectó IRF5 en células 293T no transinfectadas, lo que demuestra la especificidad del anticuerpo anti-IRF5. Una sola banda correspondiente al IRF5 monomérico sólo se detectó en las células 293T sobreexpresando IRF5. Cuando se cotranspiraron las construcciones que codifican proteínas activadoras IRF5, incluyendo NRIG (constitutivamente activo RIG-I), MAVS e IKK, apareció una banda de migración lenta correspondiente a la forma dimerica de IRF5. Sin embargo, NMDA5 (constitutivamente activo MDA5), una proteína relacionada con RIG-I, no indujo la dimización de IRF5 cuando cotransfectó.

Figura 2: Cotransfección de factores activadores IRF5 indujeron la dimización IRF5 en células 293T. Las células 293T no estaban transtrinfectadas (carril 1) o transfiguradas con IRF5 (carril 2) junto con varios reguladores IRF5 (carriles 3 a 6). Las muestras de proteínas se resolvieron mediante PAGE nativo y siguieron con el IB utilizando el anticuerpo anti-IRF5. NRIG - N-terminal de RIG-I; NMDA5 - N-terminal de MDA5. (Publicado originalmente en The Journal of Immunology. KT Chow, C Wilkins, M Narita, R Green, M Knoll, YM Loo y M Gale Jr. 2018. Programas inmunes diferenciales y superpuestos regulados por IRF3 e IRF5 en células dendríticas plasmacitoides. J. Inmunol. 201 (10) 3036-3050. Copyright © 2018 La Asociación Americana de Inmunólogos, Inc.23). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El protocolo descrito aquí es un PAGE nativo modificado que distingue las formas monoméricas y dimericas de IRF5 endógeno. Ha habido pocos estudios que informan de la detección de activación endógena IRF5 utilizando la técnica especializada de citometría de flujo de imágenes23,27,28,30. Este protocolo utiliza una técnica común y reactivos y herramientas comunes para evaluar el estado de activación endógeno IRF5 durante los primeros eventos de activación. El protocolo implica modificaciones simples a un protocolo PAGE nativo estándar para permitir la distinción entre las formas monoméricas y diminutas de IRF5. Se puede adaptar fácilmente a los estudios utilizando otras líneas celulares23. Este protocolo PAGE nativo modificado puede resolver IRF5 endógeno en sus dos formas claramente sin interferencias proteicas no específicas(Figura 2). El IRF5 endógeno de células no estimuladas se detectó como una banda única transparente en este sistema de gel, mientras que el tratamiento con R848 durante 2 h dio lugar a la aparición de una banda correspondiente a los dimers IRF5(Figura 1).
Un ensayo nativo de dimerización PAGE para IRF3, un factor de transcripción similar al de IRF5 en la misma familia, se ha desarrollado y ampliamente utilizado en las últimas dos décadas32. A pesar de las pruebas exhaustivas y la solución de problemas, no pudimos aplicar el mismo protocolo que emplea el sistema Laemmli Tris-glycine para resolver el monómero y dimer IRF5. El protocolo descrito en este artículo utiliza geles de gradiente Bis-Tris, que tienen una química muy diferente a los geles Tris-glycine de porcentaje único utilizados en el protocolo IRF3. El diferente pH y la composición química de los sistemas electroforéticos de gel pueden ser cruciales para distinguir las diversas formas de IRF3 e IRF5. De hecho, IRF3 e IRF5, aunque similares, tienen diferentes propiedades (por ejemplo, puntos isoeléctricos y sitios de modificación) que probablemente resultan en un comportamiento diferente mientras se seseparan en diferentes sistemas de gel.
Se utilizó un búfer de ejecución PAGE nativo 1x que contiene DOC para la ejecución del gel. El tampón debe prepararse fresco o mantenerse en un ambiente limpio y libre de proteínas para evitar la aparición de precipitados blancos que nublan la solución en la cámara superior como resultado de la precipitación doc de proteínas no específicas. Se recomienda encarecidamente que las muestras Endógenas IRF5 extraídas se sometan a la PAGE nativa lo antes posible, preferiblemente dentro de una semana con ciclos mínimos de congelación-descongelación. De lo contrario, puede haber una degradación significativa de las proteínas. La degradación se observa con almacenamiento tanto a -80 oC como a -20 oC. Además, el pH del búfer de ejecución SDS y el búfer de lavado TBST deben ajustarse en RT.
El volumen final ideal de la muestra cargada en cada pozo era de 10 a 15 l, pero es posible que se requieran ligeros ajustes dependiendo de los diferentes tipos de celdas. Después de la carrera inicial a 85 V durante 30 minutos, se recomienda continuar ejecutando el gel a 150 V durante aproximadamente 2 a 3 h para lograr una separación y resolución distintas del monómero y dimer IRF5. Una vez finalizada la carrera, es de suma importancia manejar el gel meticulosamente desde su extremo inferior debido a sus niveles diferenciales de densidad, que van desde el 3% en la parte superior y aumentando hacia el 12% en la parte inferior. En este caso, es preferible retirar el gel de la placa sumergiéndolo en el tampón de carrera utilizado, que actúa como un amortiguador de impacto para minimizar la fricción y permite que el gel flote lejos de la placa para evitar roturas.
Algunos inconvenientes menores de este protocolo incluyen la selección limitada de geles disponibles para lograr los resultados deseados. Geles caseros y algunas otras marcas de geles disponibles comercialmente han sido probados sin éxito. En nuestras manos, el uso de un sistema comercial de tampón y gel de funcionamiento contribuyó a la robustez y reproducibilidad de este protocolo, aunque no se han realizado pruebas exhaustivas de tampones caseros. La atención al detalle es esencial, y la experiencia es clave para el éxito en la obtención de resultados claros. Por último, la resolución de IRF5 requirió mucho tiempo (es decir, 2 x 3 h) para obtener una separación ideal del monómero y el dimer. Una mayor mejora y modificación en el futuro puede mejorar la eficiencia y minimizar los inconvenientes de este protocolo.
En conclusión, este protocolo es un ensayo robusto para la detección de monómero y dimer Endógeno IRF5. Es adecuado para aplicaciones en varios tipos de células humanas y murinas que expresan IRF5 endógeno. Será una herramienta valiosa para estudiar las vías reguladoras IRF5 y los componentes de señalización en varios tipos de celdas.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El trabajo fue apoyado por la financiación de la Fundación Croucher y los fondos de inicio de la Universidad de la Ciudad. Agradecemos a todos los miembros del laboratorio Chow por su ayuda con el experimento y la lectura crítica del manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Life Technologies, HK | 21985023 | |
| 300 W/250 V power supply 230 V AC | Life Technologies, HK | PS0301 | |
| Anti-IRF5 antibody | Bethyl Laboratories, USA | A303-385 | |
| BIOSAN Rocker Shaker (cold room safe) | EcoLife, HK | MR-12 | |
| EDTA Buffer, pH 8, 0.5 M 4x 100 mL | Life Technologies | 15575020 | |
| Glycerol 500 mL | Life Technologies | 15514011 | |
| Glycine | Life Technologies, HK | 15527013 | |
| Goat anti-Mouse IgG DyLight 800 Conjugated Antibody | LAB-A-PORTER/Rockland, HK | 610-145-002-0.5 | |
| Goat anti-Rabbit IgG DyLight 800 Conjugated Antibody | LAB-A-PORTER/Rockland, HK | 611-145-002-0.5 | |
| Halt protease inhibitor cocktail (100x) | Thermo Fisher Scientific, HK | 78430 | |
| HEPES | Life Technologies, HK | 15630080 | |
| LI-COR Odyssey Blocking Buffer (TBS) | Gene Company, HK | 927-50000 | |
| Mini Tank blot module combo; Transfer module, accessories | Life Technologies, HK | NW2000 | |
| NativePAGE 3-12% gels, 10 well kit | Life Technologies, HK | BN1001BOX | |
| NativePAGE Running Buffer 20x | Life Technologies, HK | BN2001 | |
| NativePAGE Sample Buffer 4x | Life Technologies, HK | BN2003 | |
| NP-40 Alternative, Nonylphenyl Polyethylene Glycol | Tin Hang/Calbiochem, HK | #492016-100ML | |
| PBS 7.4 | Life Technologies, HK | 10010023 | |
| Polyvinylidene difluoride (PVDF) membrane | Bio-gene/Merck Millipore, HK | IPFL00010 | |
| Protein assay kit II (BSA) | Bio-Rad, HK | 5000002 | |
| R848 | Invivogen, HK | tlrl-r848 | |
| RPMI 1640 | Life Technologies, HK | 61870127 | |
| Sodium Chloride | ThermoFisher | BP358-1 | |
| Sodium deoxycholate ≥97% (titration) | Tin Hang/Sigma, HK | D6750-100G | |
| Tris | Life Technologies, HK | 15504020 | |
| TWEEN 20 | Tin Hang/Sigma, HK | #P9416-100ML |
Referencias
- Takaoka, A., et al. Integral role of IRF-5 in the gene induction programme activated by Toll-like receptors. Nature. 434 (7030), 243-249 (2005).
- Ren, J., Chen, X., Chen, Z. J. IKKbeta is an IRF5 kinase that instigates inflammation. Proceedings of the National Academy of Sciences of the United States of America. 111 (49), 17438-17443 (2014).
- Negishi, H., Taniguchi, T., Yanai, H. The Interferon (IFN) Class of Cytokines and the IFN Regulatory Factor (IRF) Transcription Factor Family. Cold Spring Harbor Perspective Biology. 10 (11), (2018).
- Clark, D. N., et al. Four Promoters of IRF5 Respond Distinctly to Stimuli and are Affected by Autoimmune-Risk Polymorphisms. Frontiers in Immunology. 4, 360 (2013).
- Bo, M., et al. Rheumatoid arthritis patient antibodies highly recognize IL-2 in the immune response pathway involving IRF5 and EBV antigens. Scientific Reports. 8 (1), 1789 (2018).
- Duffau, P., et al. Promotion of Inflammatory Arthritis by Interferon Regulatory Factor 5 in a Mouse Model. Arthritis and Rheumatolpgy. 67 (12), 3146-3157 (2015).
- Feng, D., et al. Irf5-deficient mice are protected from pristane-induced lupus via increased Th2 cytokines and altered IgG class switching. European Journal of Immunology. 42 (6), 1477-1487 (2012).
- Richez, C., et al. IFN regulatory factor 5 is required for disease development in the FcgammaRIIB-/-Yaa and FcgammaRIIB-/- mouse models of systemic lupus erythematosus. The Journal of Immunology. 184 (2), 796-806 (2010).
- Tada, Y., et al. Interferon regulatory factor 5 is critical for the development of lupus in MRL/lpr mice. Arthritis and Rheumatology. 63 (3), 738-748 (2011).
- Weiss, M., et al. IRF5 controls both acute and chronic inflammation. Proceedings of the National Academy of Sciences of the United States of America. 112 (35), 11001-11006 (2015).
- Schoenemeyer, A., et al. The interferon regulatory factor, IRF5, is a central mediator of toll-like receptor 7 signaling. Journal of Biological Chemistry. 280 (17), 17005-17012 (2005).
- Balkhi, M. Y., Fitzgerald, K. A., Pitha, P. M. Functional regulation of MyD88-activated interferon regulatory factor 5 by K63-linked polyubiquitination. Molecular and Cellular Biology. 28 (24), 7296-7308 (2008).
- Lopez-Pelaez, M., et al. Protein kinase IKKβ-catalyzed phosphorylation of IRF5 at Ser462 induces its dimerization and nuclear translocation in myeloid cells. Proceedings of the National Academy of Sciences of the United States of America. 111 (49), 17432-17437 (2014).
- McGettrick, A. F., O'Neill, L. A. Localisation and trafficking of Toll-like receptors: an important mode of regulation. Current Opinion Immunology. 22 (1), 20-27 (2010).
- Baccala, R., Hoebe, K., Kono, D. H., Beutler, B., Theofilopoulos, A. N. TLR-dependent and TLR-independent pathways of type I interferon induction in systemic autoimmunity. Nature Medicine. 13 (5), 543-551 (2007).
- Gilliet, M., Cao, W., Liu, Y. J. Plasmacytoid dendritic cells: sensing nucleic acids in viral infection and autoimmune diseases. Nature Reviews Immunology. 8 (8), 594-606 (2008).
- Kawai, T., Akira, S. Toll-like Receptors and Their Crosstalk with Other Innate Receptors in Infection and Immunity. Immunity. 34 (5), 637-650 (2011).
- Liu, Z., Davidson, A. Taming lupus-a new understanding of pathogenesis is leading to clinical advances. Nature Medicine. 18 (6), 871-882 (2012).
- del Fresno, C., et al. Interferon-beta production via Dectin-1-Syk-IRF5 signaling in dendritic cells is crucial for immunity to C. albicans. Immunity. 38 (6), 1176-1186 (2013).
- Wang, X., et al. Expression Levels of Interferon Regulatory Factor 5 (IRF5) and Related Inflammatory Cytokines Associated with Severity, Prognosis, and Causative Pathogen in Patients with Community-Acquired Pneumonia. Medical Science Monitor. 24, 3620-3630 (2018).
- Zhao, Y., et al. Microbial recognition by GEF-H1 controls IKKepsilon mediated activation of IRF5. Nature Communications. 10 (1), 1349 (2019).
- Almuttaqi, H., Udalova, I. A. Advances and challenges in targeting IRF5, a key regulator of inflammation. FEBS Journal. 286 (9), 1624-1637 (2019).
- Chow, K. T., et al. Differential and Overlapping Immune Programs Regulated by IRF3 and IRF5 in Plasmacytoid Dendritic Cells. The Journal of Immunology. 201 (10), 3036-3050 (2018).
- Cheng, T. F., et al. Differential Activation of IFN Regulatory Factor (IRF)-3 and IRF-5 Transcription Factors during Viral Infection. The Journal of Immunology. 176 (12), 7462-7470 (2006).
- Chang Foreman, H. C., Van Scoy, S., Cheng, T. F., Reich, N. C. Activation of interferon regulatory factor 5 by site specific phosphorylation. PLoS One. 7 (3), 33098 (2012).
- Lin, R., Yang, L., Arguello, M., Penafuerte, C., Hiscott, J. A CRM1-dependent nuclear export pathway is involved in the regulation of IRF-5 subcellular localization. Journal of Biological Chemistry. 280 (4), 3088-3095 (2005).
- Stone, R. C., et al. Interferon regulatory factor 5 activation in monocytes of systemic lupus erythematosus patients is triggered by circulating autoantigens independent of type I interferons. Arthritis and Rheumatology. 64 (3), 788-798 (2012).
- De, S., et al. B Cell-Intrinsic Role for IRF5 in TLR9/BCR-Induced Human B Cell Activation, Proliferation, and Plasmablast Differentiation. Frontiers in Immunology. 8, 1938 (2017).
- Fabie, A., et al. IRF-5 Promotes Cell Death in CD4 T Cells during Chronic Infection. Cell Reports. 24 (5), 1163-1175 (2018).
- Cushing, L., et al. IRAK4 kinase activity controls Toll-like receptor-induced inflammation through the transcription factor IRF5 in primary human monocytes. Journal of Biological Chemistry. 292 (45), 18689-18698 (2017).
- Li, C., Arakawa, T. Application of native polyacrylamide gel electrophoresis for protein analysis: Bovine serum albumin as a model protein. International Journal of Biological Macromolecules. 125, 566-571 (2019).
- Iwamura, T., et al. Induction of IRF-3/-7 kinase and NF-kappaB in response to double-stranded RNA and virus infection: common and unique pathways. Genes to Cells. 6 (4), 375-388 (2001).
- Subhadarshanee, B., Mohanty, A., Jagdev, M. K., Vasudevan, D., Behera, R. K. Surface charge dependent separation of modified and hybrid ferritin in native PAGE: Impact of lysine 104. Biochimica et Biophysica Acta - Proteins and Proteomics. 1865 (10), 1267-1273 (2017).
- Reynolds, J. A., Tanford, C. Binding of Dodecyl Sulfate to Proteins at High Binding Ratios - Possible Implications for State of Proteins in Biological Membranes. Proceedings of the National Academy of Sciences of the United States of America. 66 (3), 1002 (1970).
- Manning, M., Colon, W. Structural basis of protein kinetic stability: resistance to sodium dodecyl sulfate suggests a central role for rigidity and a bias toward beta-sheet structure. Biochemistry. 43 (35), 11248-11254 (2004).
- Balkhi, M. Y., Fitzgerald, K. A., Pitha, P. M. IKKalpha negatively regulates IRF-5 function in a MyD88-TRAF6 pathway. Cellular Signalling. 22 (1), 117-127 (2010).
- Paun, A., et al. Functional characterization of murine interferon regulatory factor 5 (IRF-5) and its role in the innate antiviral response. Journal of Biological Chemistry. 283 (21), 14295-14308 (2008).
- Yasuda, K., et al. Murine dendritic cell type I IFN production induced by human IgG-RNA immune complexes is IFN regulatory factor (IRF)5 and IRF7 dependent and is required for IL-6 production. The Journal of Immunology. 178 (11), 6876-6885 (2007).
- Steinhagen, F., et al. IRF-5 and NF-kappaB p50 co-regulate IFN-beta and IL-6 expression in TLR9-stimulated human plasmacytoid dendritic cells. European Journal of Immunology. 43 (7), 1896-1906 (2013).
- Gratz, N., et al. Type I interferon production induced by Streptococcus pyogenes-derived nucleic acids is required for host protection. PLoS Pathogens. 7 (5), 1001345 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados