Method Article
Caracterización citométrica de flujo simultáneo de múltiples tipos de células recuperadas del cerebro del ratón/cordón espinal a través de diferentes métodos de homogeneización
* Estos autores han contribuido por igual
En este artículo
Resumen
Presentamos un método de citometría de flujo para identificar simultáneamente diferentes tipos de células recuperadas del cerebro del ratón o de la médula espinal. Este método podría ser explotado para aislar o caracterizar las poblaciones de células puras en enfermedades neurodegenerativas o para cuantificar el alcance de la orientación celular a la administración in vivo de vectores virales o nanopartículas.
Resumen
Los recientes avances en las ciencias de vectores virales y nanomateriales han abierto el camino a nuevos enfoques de vanguardia para investigar o manipular el sistema nervioso central (SNC). Sin embargo, una mayor optimización de estas tecnologías se beneficiaría de métodos que permitan determinar rápida y agilizar el alcance del SNC y la orientación específica de las células en la administración de vectores virales o nanopartículas en el cuerpo. Aquí, presentamos un protocolo que aprovecha las capacidades de alto rendimiento y multiplexación de la citometría de flujo para permitir una cuantificación directa de diferentes subtipos celulares aislados del cerebro del ratón o de la médula espinal, a saber, microglia/macrophages, linfocitos, astrocitos, oligodendrocitos, neuronas y células endoteliales. Aplicamos este enfoque para resaltar las diferencias críticas entre dos métodos de homogeneización tisular en términos de rendimiento celular, viabilidad y composición. Esto podría indicar al usuario que elija el mejor método dependiendo de los tipos de celda de interés y la aplicación específica. Este método no es adecuado para el análisis de la distribución anatómica, ya que el tejido se homogeneiza para generar una suspensión de una sola célula. Sin embargo, permite trabajar con células viables y se puede combinar con la clasificación celular, abriendo el camino para varias aplicaciones que podrían ampliar el repertorio de herramientas en manos del neurocientífico, que van desde el establecimiento de cultivos primarios derivados de poblaciones de células puras, hasta análisis de expresión génica y ensayos bioquímicos o funcionales sobre subtipos celulares bien definidos en el contexto de enfermedades neurodegenerativas, sobre el tratamiento farmacológico o la terapia génica.
Introducción
Las tecnologías de generación de genes y fármacos (como los vectores virales y las nanopartículas) se han convertido en una poderosa herramienta que se puede aplicar para obtener mejores perspectivas sobre vías moleculares específicas alteradas en enfermedades neurodegenerativas y para el desarrollo de enfoques terapéuticos innovadores1,2,3. La optimización de estas herramientas se basa en la cuantificación de: (1) el grado de penetración en el SNC en diferentes vías de administración y (2) la focalización de poblaciones celulares específicas. Los análisis histológicos se aplican generalmente para visualizar genes de reporterofluorescentes o nanopartículas etiquetadas fluorescentes en diferentes áreas del SNC y en diferentes tipos de células, identificados por inmunomanchado para marcadores celulares específicos4,5. A pesar de que este enfoque proporciona información valiosa sobre la biodistribución del gen administrado o las herramientas de administración de fármacos, la técnica puede ser lenta e intensa en el trabajo de parto, ya que requiere: (1) fijación de tejidos, criopreservación o incrustación y corte de parafina; (2) la tinción de marcadores celulares específicos que a veces requieren la recuperación de antígenos; 3) adquisición por microscopía de fluorescencia, que normalmente permite el análisis de un número limitado de marcadores diferentes dentro del mismo experimento; (4) procesamiento de imágenes para permitir una cuantificación adecuada de la señal de interés.
La citometría de flujo se ha convertido en una técnica ampliamente utilizada que aprovecha marcadores fluorescentes muy específicos para permitir no sólo una evaluación cuantitativa rápida de diferentes fenotipos celulares en suspensiones celulares, basada en la expresión de antígenos superficiales o intracelulares, sino también mediciones funcionales (por ejemplo, tasa de apoptosis, proliferación, análisis del ciclo celular, etc.). El aislamiento físico de las células a través de la clasificación de células fluorescentes activadas también es posible, lo que permite aplicaciones posteriores (por ejemplo, cultivo celular, ARNseq, análisis bioquímicos, etc.) 6,7,8.
La homogeneización de tejidos es un paso crítico necesario para obtener una suspensión de una sola célula que permita evaluaciones citométricas de flujo descendente fiables y reproducibles. Se han descrito diferentes métodos para la homogeneización cerebro-tejido adulto, principalmente con el objetivo de aislar las células de microglia9,10,11; pueden clasificarse en general en dos categorías principales: (1) disociación mecánica, que utiliza la fuerza de molienda o cizallamiento a través de un homogeneizador de Dounce (DH) para separar las células de sus nichos y formar una suspensión de una sola célula relativamente homogeneizada, y (2) la digestión enzimática, que se basa en la incubación de trozos de tejido picado a 37 oC en presencia de enzimas proteolíticas, como la trippsina o la papaína, favoreciendo la degradación de la matriz extracelular para crear una suspensión celular bastante homogeneizada12.
Independientemente del método que se utilice, se recomienda un paso de purificación después de la homogeneización del tejido para eliminar la mielina a través de la centrifugación en un gradiente de densidad o por selección magnética9,12, antes de pasar a las aplicaciones aguas abajo.
Aquí, describimos un método de procesamiento de tejido basado en la digestión de la papaína (PD) seguido de purificación en un gradiente de densidad, optimizado para obtener suspensiones celulares heterogéneas viables del cerebro del ratón o de la médula espinal de una manera sensible al tiempo y adecuada para la citometría de flujo. Además, describimos un panel de citometría de flujo de 9 colores y la estrategia de gating que adoptamos en el laboratorio para permitir la discriminación simultánea de diferentes poblaciones de SNC, células vivas/muertas o positividad para reporteros fluorescentes como proteína fluorescente verde o tinte de rodamina. Mediante la aplicación de este análisis citométrico de flujo, podemos comparar diferentes métodos de procesamiento de tejido, es decir, PD frente a DH, en términos de preservación de la viabilidad celular y rendimientos de diferentes tipos de células.
Los detalles que proporcionamos aquí pueden instruir la decisión sobre el protocolo de homogeneización y la combinación de anticuerpos para utilizar en el panel de citometría de flujo, basado en el tipo de celda específico de interés y los análisis aguas abajo (por ejemplo, sensible a la temperatura seguimiento de marcadores fluorescentes específicos, cultivo in vitro, análisis funcionales).
Protocolo
Todos los métodos descritos aquí han sido aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC) del Dana Farber Cancer Institute (número de protocolo 16-024).
1. Preparación de las soluciones necesarias para el experimento
- Prepare 1 solución salina equilibrada de Hank (HBSS) diluyendo 10x HBSS con agua estéril. Enfríe previamente la solución sobre hielo. Se necesitan al menos 25 ml de solución para cada muestra.
- Preparar la solución isotónica de Percoll (IPS) mezclando 10x HBSS 1:10 estéril con medio de gradiente de densidad (es decir, Percoll). Pre-enfriado en hielo.
NOTA: IpS se puede almacenar durante un máximo de 30 días a 4 oC. - Preparar la solución de bloqueo de citometría de flujo (FACS) (BL) (1% de albúmina sérica bovina [BSA], suero bovino fetal al 5% [FBS] en solución salina con fosfato [PBS]). Pre-enfriado en hielo.
2. Eutanasia animal por perfusión intracardiaca y disección tisular
NOTA: En los experimentos se utilizaron ratones C57BL/6J de ocho semanas de edad, ya sea sexo. La perfusión con solución de PBS se realiza para eliminar la contaminación de la sangre de los órganos, antes de proceder con la digestión de los tejidos.
- Anestetizar el ratón mediante el uso de una mezcla de ketamina/xilazina (90-200 mg/kg de ketamina, 10 mg/kg de xilazina). Coloque el ratón en su espalda y pegue cada extremidad hasta el soporte. Verifique la profundidad adecuada de la anestesia comprobando el reflejo de abstinencia.
- Haga una incisión cutánea de línea media a nivel de la entrada torácica para exponer el esternón. Use fórceps para agarrar la punta del esternón, luego haga una incisión de 1 cm a cada lado de la caja torácica. Finalmente cortar a través del diafragma y abrir el esternón lo suficientemente suficiente como para visualizar el corazón.
- Usa fórceps para agarrar suavemente el corazón por el ventrículo derecho y elevarlo a la línea media y ligeramente fuera del pecho.
- Inserte una aguja de mariposa de 23 G en la punta del ventrículo izquierdo, hacia la aorta y sostenga firmemente.
- Inicie la perfusión con 1x PBS. Atraviesa la aurícula derecha usando tijeras para permitir que el perfusado salga de la circulación. Fije el caudal de PBS en 3 mL/min. Perpeto con al menos 15 mL de 1x PBS para asegurarse de que los tejidos estén limpios.
NOTA: El blanqueamiento del hígado y los vasos sanguíneos mesentéricos son signos de buena perfusión. Si es necesario, el volumen de la prefusión se puede aumentar hasta que el líquido que sale del corazón esté libre de sangre, momento en el que se puede detener la línea de descarga. - Después de la perfusión, corta el cerebro de la médula espinal y retira el cerebro del cráneo con tijeras y fórceps. Retire el pelaje para aumentar la visibilidad y el control durante la disección y evitar el transporte de contaminantes capilares. Saque la médula espinal de su columna utilizando una jeringa de 3 ml llena de PBS.
- Transfiera cada tejido en un pozo de una placa multipode 6po precargada con 2 ml de hielo-frío 1x HBSS y mantener en hielo hasta la digestión.
- Divida el cerebro y la médula espinal en dos mitades, a lo largo de la línea longitudinal.
NOTA: La mitad de cada tejido se homogeneiza (ver secciones a continuación) para permitir análisis citométricos de flujo; la otra mitad se puede asignar a diferentes procesos para análisis alternativos (por ejemplo, sumergido en solución fijativa paraparaformaldehídepara para histología).
3. Digestión enzimática del cerebro y la médula espinal
NOTA: Los volúmenes descritos en esta sección son suficientes para la digestión de la mitad del cerebro o la médula espinal.
- Usa un par de tijeras para picar los tejidos en trozos de 1 x 2 mm de espesor.
- Cortar la punta de una pipeta de 1000 l con un par de tijeras para que sea lo suficientemente grande como para permitir la recolección de las piezas de tejido. Enjuague previamente la punta de la pipeta con 1 hbSS. A continuación, utilice la pipeta para transferir los 2 ml de solución de HBSS que contiene el tejido picado a un tubo cónico de 15 ml.
NOTA: El pre-enrrecemiento de la punta de la pipeta es importante para evitar la adherencia de las piezas de tejido dentro de la punta. - Lavar el pozo con 2 ml adicionales de hielo frío 1x HBSS y transferir la solución al tubo cónico correspondiente de 15 ml que contiene las piezas de tejido.
- Centrifugar cada muestra durante 5 min a 250 x g a 4oC.
- Preparar la mezcla enzimática 1 del kit de disociación del tejido neural (NTDK; Tabla de Materiales) mezclando 50 ml de enzima P con 1900 l de tampón X por muestra. Mezcla enzimática cálida de 1 a 37oC en un baño de agua. Mezclar enzima sin usar 1 a 37 oC durante al menos 10 minutos antes de su uso para permitir la activación completa de la enzima.
- Aspirar el sobrenadante del tubo cónico de 15 ml y añadir 1,95 ml de mezcla enzimática 1 a cada muestra. Suavemente vórtice para asegurarse de que el pellet se resuspende.
- Incubar las muestras en una rueda o coctelera durante 15 min a 37 oC.
- Mientras tanto, prepare la mezcla enzimática 2 del NTDK mezclando 10 ml de enzima A con 20 ml de tampón Y por muestra; precalentar la solución a 37 oC en un baño de agua.
- Al final de la incubación con mezcla enzimática 1, añadir 30 l de mezcla enzimática 2 a cada muestra.
- Mezcle suavemente las muestras pipeteando hacia arriba y hacia abajo con una punta de pipeta de 1000 ml preenjugada con 1 HBSS.
- Incubar la muestra en una rueda o coctelera durante 15 min a 37 oC.
- Después de la incubación, añadir 10 ml de hielo-frío 1x HBSS a cada tubo para inactivar la mezcla enzimática 1 y la mezcla enzimática 2.
- Centrifugar cada muestra durante 10 min a 320 x g a 4oC.
- Desechar el sobrenadante; Añadir hielo frío 1x HBSS a cada tubo hasta un volumen final de 7 ml y resuspender suavemente el pellet por vórtice.
- Continúe con la sección 5 para la eliminación de escombros.
4. Homogeneización mecánica del cerebro y la médula espinal
NOTA: Los volúmenes descritos en esta sección son suficientes para la homogeneización de la mitad del cerebro o la médula espinal. El protocolo descrito en esta sección se puede utilizar como una alternativa de método al descrito en la sección 3, dependiendo de la necesidad del usuario como se describe a continuación.
- Enfríe previamente el mortero de vidrio de la trituradora de tejido Dounce(Tabla de Materiales)sobre hielo.
- Añadir 3 ml de 1x HBSS pre-enfriado al mortero.
- Transfiera el tejido (cerebro o médula espinal) desde el pozo de la placa de 6 pocillos en el mortero de vidrio asegurándose de que se sumerge en 1 x HBSS y se sienta en la parte inferior del mortero.
- Aplastar suavemente el tejido con 10 golpes de pestillo A seguido de 10 golpes de peste B. Transferir la mezcla homogeneizada en un nuevo tubo cónico de 15 ml.
- Llene el tubo a un volumen final de 10 ml utilizando 1x HBSS pre-enfriado y centrífuga durante 10 min a 320 x g a 4 oC.
- Aspirar el sobrenadante y añadir hielo-frío 1x HBSS a cada tubo hasta un volumen final de 7 mL y resuspender suavemente el pellet por vórtice.
- Continúe con la sección 5 para la eliminación de escombros.
5. Eliminación de escombros
NOTA: La eliminación de escombros, compuesta principalmente de tejido no digerido y vainas de mielina, es un paso crítico para permitir una tinción eficiente del homogeneizado tisular para análisis citométricos de flujo posteriores.
- Filtrar cada muestra a través de un colador de células de 70 m para eliminar cualquier trozo de tejido no digerido. Este paso es particularmente importante especialmente cuando se trabaja con tejidos de la médula espinal ya que estas muestras son más propensas a contener fragmentos nerviosos no digeridos o meninges que podrían afectar los pasos posteriores.
- Asegúrese de que el volumen final es de 7 ml en cada tubo de muestra. Si no es así, llene con hielo-frío 1x HBSS hasta 7 mL.
- Añadir 3 ml de IPS preenfriado a cada muestra para hacer un volumen final de 10 ml de una solución que contenga medio de gradiente de densidad a una concentración final del 30%. Retírese suavemente de las muestras para asegurarse de que se mezclan homogéneamente.
- Muestras de centrífuga durante 15 min a 700 x g a 18oC, asegurándose de ajustar la aceleración de la centrífuga a 7 y el freno a 0.
NOTA: La centrifugación debe tardar aproximadamente 30 minutos. - Retire delicadamente las muestras de la centrífuga.
NOTA: Un disco blanquecino compuesto de escombros y mielina debe ser visible flotando en la superficie de la solución. Un pellet (que contiene las células de interés) debe ser visible en la parte inferior del tubo. - Aspirar cuidadosamente todo el disco blanquecino de escombros y luego el resto del sobrenadante asegurándose de no desalojar el pellet. Deje alrededor de 100 l de solución encima del pellet celular para evitar el riesgo de desalojarlo inadvertidamente.
- Añadir 1 ml de FACS BL, resuspender el pellet pipeteando hacia arriba y hacia abajo con una punta de pipeta de 1000 ml y transfiera muestras a tubos de 1,5 ml.
- Centrífuga durante 5 min a 450 x g a temperatura ambiente (RT).
- Aspirar suavemente el sobrenadante y resuspender el pellet en un búfer adecuado compatible con los análisis aguas abajo (ver sección 6 para el protocolo utilizado para la evaluación citométrica de flujo de múltiples tipos de células).
6. Tinción para la evaluación citométrica de flujo de múltiples tipos de células
- Resuspenda el pellet obtenido en el paso 5.9 con 350 l de FACS BL. Añadir bloque Fc a cada muestra a una concentración final de 5 g/ml.
NOTA: Se deben utilizar al menos 100 l de la muestra para una tinción, para asegurarse de procesar suficientes células para permitir análisis fiables. - Incubar la muestra durante 10 min a 4oC antes de proceder con la tinción.
- Prepare una mezcla de anticuerpos de acuerdo con la Tabla 1.
- Añadir mezcla de anticuerpos a cada tubo, vórtice para 5 s e incubar las muestras durante 15 minutos a 4 oC en la oscuridad.
- Añadir 1 ml de PBS a cada tubo, vórtice y centrífuga durante 5 min a 450 x g en RT.
- Mientras tanto, prepare la mezcla de estreptavidina de acuerdo con la Tabla 1.
- Deseche el sobrenadante y vuelva a suspender el pellet en la mezcla de estreptavidina preparada en el paso 6.6. Para cada muestra, utilice el mismo volumen que el utilizado para la tinción en el paso 6.4.
- Vórtice durante 5 s e incubar las muestras durante 10 min a 4oC en la oscuridad.
- Añadir 1 ml de PBS a cada tubo, vórtice y centrífuga durante 5 min a 450 x g en RT.
- Desechar el sobrenadante y resuspender el pellet en FACS BL. Use 300 l de FACS BL para cada 100 l de muestra manchada.
- Añadir 7-amino-actinomicina D (7-AAD) solución a cada muestra. Utilice 5 l de 7-AAD por cada 300 l de muestra preparada en el paso 6.10.
- Almacene las muestras a 4 oC en la oscuridad hasta el análisis citofluorimétrico. Realice el análisis dentro de las 16 h de la preparación de la muestra, para garantizar >60% viabilidad celular.
Resultados
Comparamos dos métodos de homogeneización diferentes (DH versus PD) aplicados al cerebro del ratón y la médula espinal, para probar la eficiencia en la recuperación de diferentes tipos de células viables adecuadas para aplicaciones posteriores. Para ello, explotamos un panel de citometría de flujo de 9 colores diseñado para caracterizar, en la misma muestra, diferentes tipos de células del SNC, incluyendo microglia, linfocitos, neuronas, astrocitos, oligodendrocitos y endotelio.
Los tejidos cerebrales y de la médula espinal se recuperaron de ratones diferentes (n a 6), divididos en dos mitades longitudinalmente, pesados y procesados en paralelo mediante la aplicación de la interrupción mecánica utilizando el homogeneizador Dounce (método DH) o picados suavemente y digeridos enzimáticamente utilizando el NTDK comercial basado en papaína (método PD)(Figura 1A). Después de la eliminación de escombros, las células del cerebro o de la médula espinal se diluyeron 1/10 o 1/2-1/5, respectivamente, en azul Trypan para determinar el rendimiento celular y la viabilidad con una cámara Neubauer(Figura 1B,C). El método DH en general produjo un mayor rendimiento celular de cerebro y médula espinal. Sin embargo, la mayoría de las células recuperadas estaban muertas, lo que resulta en sólo el 13,8% a 3,3% de las células viables en el cerebro y el 10,5% a 1,5% en la médula espinal(Figura 1B). Muchas de las células muertas formaron agregados(Figura 1C); este fenómeno podría deberse a la presencia de redes celulares altamente interconectadas (como las células endoteliales y gliales que recubren la vasculatura del SNC) que no pudieron ser desagregadas por la fuerza de cizallamiento aplicada con la DH. Estos agregados de células de la muerte probablemente no fueron eliminados por el gradiente de densidad y terminaron en el pellet celular final utilizado para el análisis citofluorimétrico. Por el contrario, el método PD determinó una mejor preservación general de la viabilidad celular (90,6% a 0,6% en el cerebro y 85,2% a 2,8% en la médula espinal). La papaína es capaz de digerir la matriz extracelular y las uniones de célula a célula de manera eficiente, lo que conduce a una suspensión de una sola célula más uniforme. Algunas de las células que mueren durante el proceso de picado podrían ser digeridas aún más por la papaína, lo que conduce a la formación de desechos celulares que se separan más eficientemente a través del gradiente de densidad. En general, esto probablemente determinó una mejor preservación de la viabilidad celular con el método PD, a pesar de un rendimiento celular ligeramente menor en comparación con el método DH.
Una alícuota de 100 ol de las suspensiones de células cerebrales y de la médula espinal se tiñó con la mezcla de anticuerpos (Tabla 1) y se analizó mediante citometría de flujo con un panel de 9 colores. La Figura 2A muestra la estrategia de gating utilizada para identificar los diferentes tipos de células de las suspensiones del cerebro y de las células de la médula espinal. Brevemente, la primera puerta identifica a la población general de acuerdo con la dispersión hacia adelante (FSC) y la dispersión lateral (SSC), excluyendo los desechos de células pequeñas. Luego se identifican las células vivas (7-AAD-). Dentro de la población total de células vivas, se resaltan las células CD45+ y CD45-. Dentro de la puerta CD45+, se identifican los linfocitos CD45+CD11b+ microglia/macrophages y CD45+CD11b-. Dentro de la puerta CD45, las células son discriminadas según la positividad para ACSA2 (astrocitos) u O4 (oligodendrocitos). Las células CD45-ACS2-O4- se subdividen según la positividad para Ti1 (neuronas) o CD31 (endotelio). Las células Thy1-CD31 restantes se clasifican como "otros tipos de células", no contabilizadas por nuestra mezcla de anticuerpos.
Como se muestra en la Figura 2B, con el método DH alrededor del 38% de las células viables recuperadas del cerebro y alrededor del 32% de las células viables recuperadas de la médula espinal eran de origen hematopoyético (CD45+). Por otro lado, el método PD permitió recuperar un rendimiento significativamente alto de células viables en ambos tejidos, con una fracción muy grande representada por células CD45- no hematopoyéticas (alrededor del 82% en el cerebro y 92% en la médula espinal). Sorprendentemente, CD45+CD11b+ microglia/macrophages representaron la fracción celular viable más abundante con el método DH(Figura 2C). Sin embargo, el método PD produjo una representación más heterogénea de los tipos de células, incluyendo astrocitos ACSA+, oligodendrocitos O4+, células endoteliales CD31+ y neuronas Thy1+(Figura 2C). Curiosamente, las neuronas viables y las células endoteliales apenas eran detectables con el método DH.
El método DH se basa en la molienda mecánica del tejido entre el pestillo de vidrio y el mortero del homogeneizador Dounce para obtener homogeneización tisular. Esto podría causar cierto estrés de cizallamiento que probablemente dañará y afectará la viabilidad de células grandes o muy sensibles como las neuronas o células de la neurovasculatura. Evaluamos la viabilidad celular (porcentaje de células 7-AAD-) dentro de cada subpoblación celular identificada a través del panel de anticuerpos(Figura 3). Células hematopoyéticas (CD45+) aisladas del cerebro y la médula espinal, incluyendo microglia/macrofagos (CD45+CD11b+) y otras células no mieloides (CD45+Cd11b-), mostraron una viabilidad muy alta independientemente del método de homogeneización utilizado(Figura 3A). Por el contrario, el método DH determinó una reducción significativa de la viabilidad de las poblaciones de CD45 (Figura 3B),mientras que el método PD determinó una amplia preservación de los diferentes tipos de células del SNC. En detalle, las neuronas y las células endoteliales fueron las subpoblaciones más significativamente afectadas por la DH y preservadas por el método PD.
En la Figura 4se resume una presentación esquemática de los pasos críticos necesarios para la preparación adecuada de la muestra y la eliminación eficiente de los desechos.
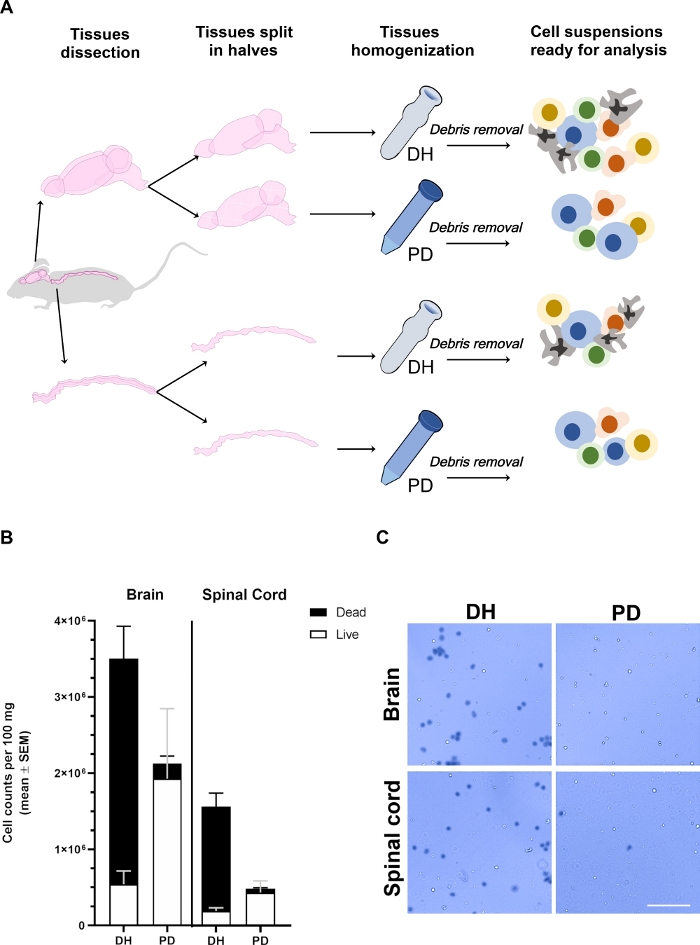
Figura 1: El rendimiento de las células recuperadas del cerebro y la médula espinal se ve afectado por el método de homogeneización.
(A) Esquema experimental. Los ratones fueron anestesiados e intracardiacalmente perfundidos con PBS para eliminar las células sanguíneas circulantes intravasculares. El cerebro y la médula espinal fueron cuidadosamente diseccionados y divididos en dos mitades longitudinalmente. Los tejidos se homogeneizaron utilizando el homogeneizador Dounce (DH) o la digestión de la papaína (PD) como se detalla en el texto principal. La mielina y los restos de tejido se eliminaron por centrifugación en una solución media de gradiente de densidad del 30%, lo que dio lugar a una suspensión celular heterogénea que contenía diferentes tipos de células que podían analizarse mediante citometría de flujo. (B) Histogramas que muestran el rendimiento de las células recuperadas del cerebro o de la médula espinal tras la homogeneización tisular con el método DH o PD. Se representa la media de SEM de al menos 6 animales por condición. (C) Fotomicrografías representativas del microscopio de campo brillante de células positivas azules de Trypan (muertas) y negativas (en vivo) recuperadas del cerebro o de la médula espinal por los dos métodos. Barra de escala a 100 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Las proporciones relativas de los diferentes tipos de células recuperadas del SNC se ven afectadas por el método de homogeneización tisular.
(A) Gráficas representativas de la citometría de flujo que muestran la estrategia de gating para identificar la subpoblación celular diferente dentro de las preparaciones celulares obtenidas del cerebro o la médula espinal: la población celular está cerrada en los parámetros físicos FSC y SSC, seguida de la selección de células vivas 7-AAD; luego las células son discriminadas de acuerdo con la positividad para el marcador CD45; microglia/macrophages se identifican como células CD11b+ dentro de la fracción CD45+, mientras que los linfocitos son CD11b-. Astrocitos, oligodendrocitos, células endoteliales y neuronales se identifican como células ACSA2+, O4+, CD31+ o Thy1+ dentro de CD45-, respectivamente. (B) Histogramas que muestran el porcentaje de células CD45+ y CD45- dentro de las poblaciones totales de células vivas o muertas, en el cerebro o la médula espinal tras la homogeneización con el método DH o PD. El análisis estadístico de los resultados mostrados en los gráficos se indica en la Tabla 2. (C) Gráficos circulares que muestran el porcentaje de diferentes subtipos celulares viables dentro de la población celular total, en el cerebro o la médula espinal tras la homogeneización con el método DH o PD. También se notifica el porcentaje del total de células muertas. N.o 6. CD45+CD11b+ - microglia/macrophages; CD45+CD11b- - linfocitos/células no mieloides; CD45-ACSA2+ - astrocitos; CD45-O4+ - oligodendrocitos; CD45-Thy1+ - neuronas; CD45-CD31+ - células endoteliales; Otras células negativas para todos los marcadores mencionados anteriormente. El análisis estadístico de los resultados mostrados en los gráficos se indica en la Tabla 2. Haga clic aquí para ver una versión más grande de esta figura.
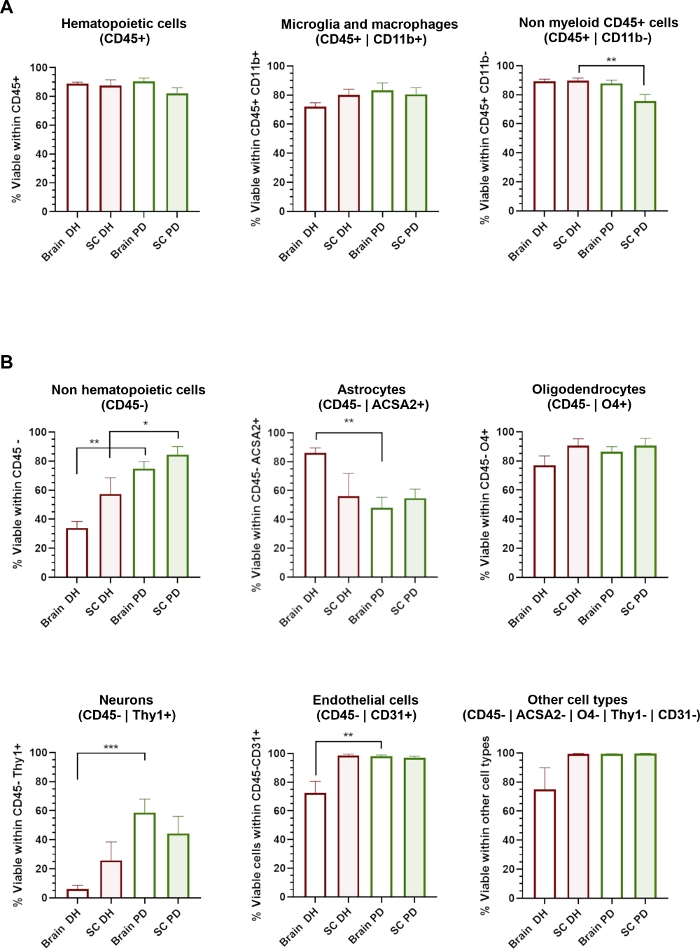
Figura 3: La viabilidad celular de diferentes tipos de células del SNC se ve afectada por el método de homogeneización aplicado.
(A) Histogramas que muestran el porcentaje de células vivas 7-AAD- dentro de las poblaciones hematopoyéticas CD45+incluyendo CD11b+ microglia/macrophages y CD11b- células no mieloides. (B) Histogramas que muestran el porcentaje de 7-AAD- células vivas dentro de poblaciones CD45- no hematopoyéticas incluyendo astrocitos, oligodendrocitos, neuronas, endoteliales y otros tipos de células. * & p < 0.05, ** á p < 0.01, Mann-Whitney entre DH y PD. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Representación esquemática de los pasos críticos necesarios para el procesamiento adecuado del tejido.
Se muestra una lista de los pasos más críticos necesarios para el procesamiento adecuado de tejidos y la eliminación eficiente de escombros. Es importante identificar correctamente el disco de escombros (flecha negra) y el pellet celular (flecha azul) formado después de la centrifugación de las muestras en el gradiente de densidad del 30%. El disco de escombros, junto con el resto del sobrenadante, debe retirarse cuidadosamente por aspiración sin desalojar el pellet celular para evitar la pérdida de muestra. Haga clic aquí para ver una versión más grande de esta figura.
| Mezcla de anticuerpos | Concentración inicial (g/mL) | Concentración final (g/mL) | Factor de dilución |
| anti CD45/BV510 | 200 | 2 | 100 |
| anti CD11b/APC.780 | 200 | 2 | 100 |
| anti CD31/BV421 | 200 | 2 | 100 |
| anti ACSA2/APC | 150 | 0.75 | 200 |
| anti O4/biotina | Na | Na | 40 |
| anti CD90.2/PE. Cy7 | 200 | 2 | 100 |
| Mezcla de Streptavidin | Concentración inicial (g/mL) | Concentración final (g/mL) | Factor de dilución |
| Streptavidin/Alexa 680 | 1000 | 1 | 1000 |
Tabla 1: Receta para la preparación de mezclas para la tinción de citometría de flujo. La tabla describe las concentraciones óptimas de anticuerpos y estreptavidina utilizadas para permitir análisis citométricos de flujo de múltiples tipos de células. Consulte la Tabla de materiales para obtener más información sobre los números de catálogo de cada reactivo mencionado en la tabla.
| Estadísticas de la Figura 2B | ||||||||||
| BRAIN (% de celdas) | ||||||||||
| CD45+ | CD45- | |||||||||
| Vivir | Muerto | Vivir | Muerto | |||||||
| Método | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | ||
| Dh | 15.20 | 2,32 euros | 1.90 | 0,30 euros | 24.78 | 4.045 euros | 51.58 | 6.033 euros | ||
| Pd | 15.20 | 2,65 euros | 2.33 | 1,10 euros | 68.53 | 3.618 euros | 13.93 | 2.180 euros | ||
| Mann-Whitney | Ns | Ns | *** | ** | ||||||
| valor p | 0.9989 | 0.738 | 0.0006 | 0.0015 | ||||||
| CORD SPINAL (% de células) | ||||||||||
| CD45+ | CD45- | |||||||||
| Vivir | Muerto | Vivir | Muerto | |||||||
| Método | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | ||
| Dh | 15.00 | 8,21 euros | 1.41 | 0,11 euros | 31.64 | 8,21 euros | 51.95 | 16,52 euros | ||
| Pd | 7.49 | 4,99 euros | 1.15 | 0,68 euros | 84.27 | 9,39 euros | 7.09 | 3,75 euros | ||
| Mann-Whitney | Ns | Ns | * | Ns | ||||||
| valor p | 0.5548 | 0.7236 | 0.0438 | 0.1144 | ||||||
| Estadísticas de la Figura 2C | |||||||||||||||||
| BRAIN (% de celdas) | |||||||||||||||||
| CD45+ | CD45- | ||||||||||||||||
| CD11b+ | CD11b- | ACSA2 | O4 | Thy1 | CD31 | Otro | Muerto | ||||||||||
| Método | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | |
| Dh | 19.32 | 3,88 euros | 1.17 | 0,27 euros | 9.52 | 2,68 euros | 3.41 | 1,01 | 1.39 | 0,77 euros | 0.48 | 0,29 euros | 10.52 | 4,49 euros | 53.83 | 5,79 euros | |
| Pd | 10.88 | 2,03 euros | 1.65 | 0,48 euros | 8.17 | 2,66 euros | 6.54 | 0,76 euros | 6.37 | 1,76 euros | 8.27 | 1,25 euros | 33.28 | 6,34 euros | 23.72 | 5,31 euros | |
| Mann-Whitney | Ns | Ns | Ns | * | ** | *** | ** | ** | |||||||||
| valor p | 0.1206 | 0.4819 | 0.5894 | 0.0264 | 0.0093 | 0.0003 | 0.0084 | 0.0022 | |||||||||
| CORD SPINAL (% de células) | |||||||||||||||||
| CD45+ | CD45- | ||||||||||||||||
| CD11b+ | CD11b- | ACSA2 | O4 | Thy1 | CD31 | Otro | Muerto | ||||||||||
| Método | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | Decir | • SEM | |
| Dh | 21.23 | 6,25 euros | 2.51 | 0,57 euros | 4.26 | 2,34 euros | 9.40 | 1,89 euros | 2.82 | 1,51 euros | 0.97 | 0,50 euros | 22.74 | 9,04 euros | 35.28 | 1,89 euros | |
| Pd | 9.63 | 1,67 euros | 2.77 | 0,48 euros | 4.23 | 1,59 euros | 28.62 | 3,57 euros | 1.26 | 0,49 euros | 6.94 | 2,14 euros | 26.39 | 8,17 euros | 19.09 | 4,76 euros | |
| Mann-Whitney | Ns | Ns | Ns | * | Ns | * | Ns | Ns | |||||||||
| valor p | 0.1905 | >0.9999 | 0.7302 | 0.0159 | 0.7302 | 0.0317 | 0.7302 | 0.1111 | |||||||||
Tabla 2: Análisis estadístico de las diferentes poblaciones recuperadas mediante la aplicación del método DH o PD. La tabla describe las estadísticas de los gráficos que se muestran en la Figura 2B y la Figura 2C. Se representa el promedio y el SEM de al menos seis muestras independientes. También se notifican el valor p y los detalles de la prueba estadística aplicada para cada comparación.
Discusión
Aquí describimos un protocolo para la co-purificación y el análisis citométrico de flujo simultáneo de algunas de las células del SNC más relevantes del cerebro del ratón y la médula espinal. Tradicionalmente, se han aplicado análisis histológicos para describir la distribución de nanopartículas o la eficiencia de transducción de vectores virales en el SNC5,13, o para proporcionar información sobre los cambios morfológicos y moleculares que se producen en tipos celulares específicos durante una patología o sobre el tratamiento farmacológico14. Sin embargo, la histología carece de procesatividad y no permite un examen exhaustivo de múltiples características en las mismas muestras histológicas, debido al número limitado de marcadores que se pueden analizar simultáneamente. Nuestro enfoque puede ser complementario a los análisis histológicos tradicionales y se puede combinar con varias aplicaciones posteriores (clasificación, cultivo primario, análisis bioquímicos o de secuenciación de próxima generación) para ampliar la compilación de información que se puede obtener a partir de muestras individuales. Sin embargo, se deben considerar algunos factores clave enumerados a continuación, ya que pueden afectar críticamente el éxito de este enfoque:
- Cantidad de tejido inicial. Hemos optimizado la separación celular para comenzar con tan poco como media médula espinal o mitad cerebro. En nuestra experiencia, el procesamiento de la mitad del cerebro o la mitad de la médula espinal de un ratón adulto de 8 semanas con el método PD produce 1 x 6 x 106 células viables del cerebro y alrededor de 0.1 x 0.5 x 106 células viables de la médula espinal después del paso de gradiente de densidad. No hemos medido el rendimiento de las células después de aplicar el método PD en tejidos de recién nacidos o ratones menores de 8 semanas. Sin embargo, en nuestras manos, el resultado es proporcional al peso del tejido inicial. Por lo tanto, para los animales más jóvenes (por ejemplo, cachorros de 10 días de edad) se debe procesar todo el cerebro o la médula espinal para garantizar un buen rendimiento celular para análisis descendentes fiables. Si es necesario, la agrupación de tejidos de varios animales ayudaría a aumentar el rendimiento celular. En nuestra experiencia, este protocolo también se puede aplicar sin modificaciones para aislar las células del cerebro o la médula espinal de las ratas, en la medida en que se respete la proporción de reactivos utilizados por peso del tejido inicial. Para los tejidos de más de 250 mg de peso, se sugiere la escala de reactivos hacia arriba o la división del tejido en múltiples muestras (cada ponderación <250 mg).
- Extracción de meninges/trozos de tejido residual antes del gradiente de densidad. En nuestra experiencia, el método PD descrito por la presente no es capaz de digerir eficientemente meninges o tejidos altamente mielinados como los nervios y las raíces nerviosas que emergen de la médula espinal. Cuando estos tejidos están presentes, algunas piezas pegajosas no digeridas pueden permanecer en la suspensión celular recuperada después de los pasos de digestión enzimática, antes de la eliminación de escombros. La eliminación de estos fragmentos filtrando la solución a través de un colador de células de 70 m (como se sugiere en el paso 5.1) es fundamental para una preparación de celda exitosa. De hecho, si no se eliminan, las meninges o piezas de tejido dificultarán la separación eficiente del gradiente de densidad, lo que resulta en rendimientos celulares deficientes.
- Temporización, temperatura y esterilidad. Es muy importante realizar todos los pasos de manera oportuna utilizando los ajustes de temperatura y las incubaciones correctas como se sugiere. Esto es fundamental para garantizar una alta viabilidad celular e integridad de la muestra. Dependiendo de la aplicación aguas abajo, es posible que sea necesario realizar todos los pasos bajo una campana estéril y con reactivos estériles (por ejemplo, establecer cultivos celulares primarios). La incubación prolongada en la solución digestiva enzimática más allá del tiempo sugerido (sección 3) podría resultar en una gota de viabilidad celular. Los epítopos de algunos antígenos superficiales podrían ser sensibles a la papaína, lo que resulta en la pérdida de señal en la citometría de flujo. Para aplicaciones específicas que requieren marcadores adicionales distintos de los descritos en este artículo, se recomienda probar el rendimiento de diferentes clones de anticuerpos antes de iniciar el experimento. Se ha informado de que el medio de gradiente de densidad podría contener algunos rastros de endotoxinas que pueden desencadenar la activación de las células inmunitarias (microglia/macrofagos). Siempre se deben añadir controles internos adecuados en los experimentos siempre que se analicen estas poblaciones, para excluir los posibles efectos inducidos por el procedimiento. Seguir con las temperaturas sugeridas y las sobras de medio de gradiente de densidad de lavado inmediatamente después del paso de eliminación de escombros es generalmente suficiente para evitar la activación de las células inmunitarias. Sin embargo, en caso de que las aplicaciones posteriores (por ejemplo, RNAseq o análisis funcionales) se vean afectadas por este paso, el usuario debe cambiar a una preparación de medio de gradiente de densidad de baja endotoxina (sugerido en la Tabla de Materiales).
- Anticuerpos y máquina sFACS. El protocolo de tinción de citometría de flujo presentado por el presente hace uso de concentraciones de anticuerpos y conjugaciones de color que funcionaron mejor con los rendimientos celulares recuperados en nuestra experiencia de laboratorio y con máquinas FACS disponibles en nuestro instituto. El usuario debe titer los anticuerpos en sus manos antes de comenzar un nuevo experimento, ya que la concentración podría necesitar ser ligeramente ajustada. Además, animamos a utilizar siempre controles de tinción de un solo color en cada experimento para verificar que todos los anticuerpos y la configuración de compensación de la máquina FACS están funcionando adecuadamente. Hay que tener en cuenta que el antígeno CD90 (Tu) utilizado para detectar neuronas existe en dos isotipos diferentes, a saber, CD90.2 o CD90.1 dependiendo de la tensión del ratón: las cepas de ratón más utilizadas, como C57BL6/J express CD90.2; cepas de ratón como AKR/J, PL y FVB/N express CD90.1. Por lo tanto, el usuario debe verificar cuidadosamente la tensión del ratón y elegir el anticuerpo anti-CD90 apropiado (como se sugiere en la Tabla de Materiales)antes de comenzar los experimentos.
En resumen, el protocolo aquí presentado aprovecha una suave digestión enzimática seguida de una tinción de 9 colores que permite una evaluación citométrica de flujo simultánea eficiente de diferentes tipos de células del cerebro del ratón y la médula espinal. El protocolo podría ser explotado para monitorear de manera general y completa la eficiencia de la focalización celular por nanopartículas o vectores virales administrados en el SNC15. Además, el protocolo podría adoptarse fácilmente para aplicaciones descendentes muy delicadas, como la clasificación celular, la subcultura ex vivo, el ARNAseq de una sola célula, como resultado de suma importancia no sólo para la evaluación preclínica de la orientación celular mediante terapias, sino también para la caracterización en profundidad de los procesos patológicos en enfermedades neurodegenerativas.
Una fracción de toda la población celular del SNC no es discriminada por este protocolo (véase "otros" tipos de células en la Figura 2); esto se puede explicar por la presencia de otros subtipos celulares que están presentes en el SNC pero no son capturados por los anticuerpos que utilizamos. En nuestros análisis preliminares, alrededor del 14% de la fracción de "otras células" es positiva para CD73, un marcador de células mesenquimales enriquecido en la neurovasculatura e implicado en varios procesos neuroinflamatorios16,17. Además, hipotetizamos que la fracción de "otras células" también podría comprender células menos diferenciadas, como progenitores en diferentes etapas de maduración, como células madre neurales nestin+, progenitores de glia radial nestin+ vimentin+, progenitores neuronales doublecortin+, células precursoras de oligodendrocitos NG2+, entre otras. Estos subtipos de células podrían ser fácilmente investigados mediante la aplicación de nuestro protocolo de citometría de flujo, ya que elegimos una configuración de tintes fluorescentes que permite acomodar hasta dos marcadores celulares adicionales conjugados con el isotiocianato de fluoresceína (FITC) o fluorofores (PE).
En general, nuestro enfoque podría proporcionar una nueva herramienta para realizar investigaciones más exhaustivas en el contexto del SNC (en salud y enfermedad) aprovechando una tecnología bien consolidada que permita evaluaciones cuantitativas cualitativas y de alto rendimiento como la citometría de flujo.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este estudio fue financiado por fondos de start-up del Boston Children's Hospital a A.B., ALSA grant nr. 17-IIP-343 a M.P., y la Oficina del Subsecretario de Defensa para Asuntos de Salud a través del Programa de Investigación de Esclerosis Lateral Amiotrófica bajo el Premio No. W81XWH-17-1-0036 a M.P. Reconocemos DFCI Flow Cytometry Core para soporte técnico.
Materiales
| Name | Company | Catalog Number | Comments |
| 10X HBSS (Calcium, Magnesium chloride, and Magnesium Sulfate-free) | Gibco | 14185-052 | |
| 70 mm Cell Strainer | Corning | 431751 | |
| ACSA/ACSA2 anti-mouse antibody | Miltenyi Biotec | 130-117-535 | APC conjugated |
| Bovine Serum Albumin | Sigma Aldrich | A9647-1KG | |
| CD11b rat anti-mouse antibody | Invitrogen | 47-0112-82 | APC-eFluor 780 conjugated |
| CD31 rat anti-mouse antibody | BD Bioscience | 562939 | BV421 conjugated |
| CD45 rat anti-mouse antibody | Biolegend | 103138 | Brilliant Violet 510 conjugated |
| CD90.1/Thy1.1 rat anti-mouse antibody | Biolegend | 202518 | PE/Cy7 conjugated |
| CD90.2/Thy1.2 rat anti-mouse antibody | Biolegend | 1005325 | PE/Cy7 conjugated |
| Conical Tubes (15 mL) | CellTreat | 229411 | |
| Conical Tubes (50 mL) | CellTreat | 229422 | |
| Dounce Tissue Grinder set (Includes Mortar as well as Pestles A and B) | Sigma-Aldrich | D9063-1SET | |
| Fc (CD16/CD32) Block rat anti-mouse antibody | BD Pharmingen | 553142 | |
| Fetal Bovine Serum | Benchmark | 100-106 | |
| Neural Tissue Dissociation Kit (P) | Miltenyi Biotec | 130-092-628 | |
| O4 anti mouse/rat/human antibody | Miltenyi Biotec | 130-095-895 | Biotin conjugated |
| Percoll | GE Healthcare | 10266569 | sold as not sterile reagent |
| Percoll | Sigma | 65455529 | sterile reagent (to be used for applications requiring sterility) |
| Percoll PLUS | Sigma | GE17-5445-01 | reagent containing very low traces of endotoxin |
| Streptavidin | Invitrogen | S3258 | Alexa Fluor 680 conjugated |
Referencias
- Deverman, B. E., Ravina, B. M., Bankiewicz, K. S., Paul, S. M., Sah, D. W. Y. Gene therapy for neurological disorders: progress and prospects. Nature Reviews Drug Discovery. 17 (9), 641-659 (2018).
- Teleanu, D., Negut, I., Grumezescu, V., Grumezescu, A., Teleanu, R. Nanomaterials for Drug Delivery to the Central Nervous System. Nanomaterials. 9 (3), 371(2019).
- Chen, S., et al. Recombinant Viral Vectors as Neuroscience Tools. Current Protocols in Neuroscience. 87 (1), 67(2019).
- Alves, S., et al. Ultramicroscopy as a novel tool to unravel the tropism of AAV gene therapy vectors in the brain. Scientific Reports. 6 (1), 28272(2016).
- Peviani, M., et al. Lentiviral vectors carrying enhancer elements of Hb9 promoter drive selective transgene expression in mouse spinal cord motor neurons. Journal of Neuroscience Methods. 205 (1), 139-147 (2012).
- Baumgarth, N., Roederer, M. A practical approach to multicolor flow cytometry for immunophenotyping. Journal of Immunological Methods. 243 (1-2), 77-97 (2000).
- Sykora, M. M., Reschke, M. Immunophenotyping of Tissue Samples Using Multicolor Flow Cytometry. Methods in Molecular Biology. 1953, 253-268 (2019).
- Legroux, L., et al. An optimized method to process mouse CNS to simultaneously analyze neural cells and leukocytes by flow cytometry. Journal of Neuroscience Methods. 247, 23-31 (2015).
- Lee, J. K., Tansey, M. G. Microglia Isolation from Adult Mouse Brain. Methods in Molecular Biology. 1041, 17-23 (2013).
- Grabert, K., McColl, B. W. Isolation and Phenotyping of Adult Mouse Microglial Cells. Methods in Molecular Biology. 1784, 77-86 (2018).
- Nikodemova, M., Watters, J. J. Efficient isolation of live microglia with preserved phenotypes from adult mouse brain. Journal of Neuroinflammation. 9 (1), 635(2012).
- Garcia, J. A., Cardona, S. M., Cardona, A. E. Isolation and analysis of mouse microglial cells. Current Protocols in Immunology. 104 (1), 1-15 (2014).
- Papa, S., et al. Selective Nanovector Mediated Treatment of Activated Proinflammatory Microglia/Macrophages in Spinal Cord Injury. ACS Nano. 7 (11), 9881-9895 (2013).
- Peviani, M., et al. Neuroprotective effects of the Sigma-1 receptor (S1R) agonist PRE-084, in a mouse model of motor neuron disease not linked to SOD1 mutation. Neurobiology of Disease. 62, 218-232 (2014).
- Peviani, M., et al. Biodegradable polymeric nanoparticles administered in the cerebrospinal fluid: Brain biodistribution, preferential internalization in microglia and implications for cell-selective drug release. Biomaterials. 209, 25-40 (2019).
- Meng, F., et al. CD73-derived adenosine controls inflammation and neurodegeneration by modulating dopamine signalling. Brain. 142 (3), 700-718 (2019).
- Nedeljkovic, N. Complex regulation of ecto-5'-nucleotidase/CD73 and A2AR-mediated adenosine signaling at neurovascular unit: A link between acute and chronic neuroinflammation. Pharmacological Research. 144, 99-115 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados