Method Article
Combinación de imágenes Raman y Análisis Multivariante para visualizar Lignina, Celulosa y Hemicelulosa en el Muro de la Célula Vegetal
En este artículo
Resumen
Este protocolo tiene como objetivo presentar un método general para visualizar la lignina, la celulosa y la hemicelulosa en las paredes celulares de las plantas usando imágenes Raman y análisis multivariante.
Resumen
La aplicación de la imagen Raman a la biomasa vegetal está aumentando porque puede ofrecer información espacial y composicional sobre soluciones acuosas. El análisis no suele requerir una preparación extensiva de la muestra; La información estructural y química se puede obtener sin etiquetado. Sin embargo, cada imagen Raman contiene miles de espectros; Esto plantea dificultades al extraer información oculta, especialmente para componentes con estructuras químicas similares. Este trabajo introduce un análisis multivariado para abordar esta cuestión. El protocolo establece un método general para visualizar los componentes principales, incluyendo lignina, celulosa y hemicelulosa dentro de la pared celular de la planta. En este protocolo, se describen procedimientos para la preparación de muestras, adquisición espectral y procesamiento de datos. Es altamente dependiente de la habilidad del operador en la preparación de la muestra y en el análisis de los datos. Mediante el uso de este enfoque, una investigación Raman puede ser realizada por un usuario no especialista paraH-calidad de datos y resultados significativos para el análisis de la pared celular de la planta.
Introducción
Plant biomass is the most abundant renewable resource on Earth; is mainly composed of lignin, cellulose, and hemicellulose; and is considered an attractive source of bioenergy and bio-based chemicals1. Unfortunately, it can resist degradation and confer hydrolytic stability or structural robustness to the plant cell wall. Such resistance is attributable to the accessible surface area, biomass particle size, degree of polymerization, cellulose crystallinity, and protective lignin2. A comprehensive understanding of the structural and chemical nature of the plant cell wall is thus significant from the viewpoint of plant biology and chemistry, as well as from that of commercial utilization. Commonly used wet chemistry analyses, such as chromatography, mass spectrometry, and nuclear magnetic resonance spectroscopy, only provide average compositional data of the measured sample. Furthermore, these methods are invasive and destroy the original structure of the plant tissue3.
The Raman imaging technique is a powerful tool for the nondestructive visualization of spatially resolved chemical information4. It uses a laser light to cause inelastic scattering with a photon and relies on changes in polarizability arising from the molecular vibrations. In this case, water causes weak Raman scattering, which makes this approach suitable for in situ investigations of biological samples5. The application of the Raman imaging technique to the plant cell wall can elucidate the structure and composition of plant cell walls in their native state, with the resolution on the scale of the single cell and even of the cell wall layers6. A typical Raman imaging analysis of a plant cell wall generally consists of three steps: 1) sample preparation, 2) spectral acquisition, and 3) data processing.
Although one of the major advantages of Raman imaging is the ability to achieve label-free and non-destructive spectra with minimal sample preparation, physical sample sectioning is still necessary to expose the surface of interest. This process should be performed carefully to obtain a flat surface, since the technique depends on maintaining optical focus7. Spectral acquisition requires a balance between image quality and extensive acquisition times8. Data processing aims to effectively extract the chemical information from the image data, especially for the components with similar chemical structures, such as cellulose and hemicellulose. Due to the strong spectral overlap, the exact spectra are difficult to discern. In this case, multivariate analysis is a straightforward approach to effectively uncover the hiding structural and chemical information9. This work presents a general protocol describing the use of Raman imaging to visualize the main components in plant cell walls, including lignin, cellulose, and hemicellulose.
Protocolo
1. Preparación de la muestra
- Corte un pequeño bloque de tejido (aproximadamente 3 mm x 3 mm x 5 mm) de la muestra de la planta ( por ejemplo, un tallo de álamo).
- Sumerja el tejido en agua desionizada hirviendo durante 30 min. Se transfiere inmediatamente a agua desionizada a temperatura ambiente (RT) durante 30 min. Repita este paso hasta que el tejido se hunde en el fondo del recipiente, lo que indica que el aire en el tejido se ha eliminado y que el tejido se ha ablandado.
Nota: Para las muestras que se hunden en el fondo antes de este paso, repita este ciclo generalmente 3-5 veces. - Preparar alícuotas de polietilenglicol (PEG) de 20, 50, 70, 90% (v / v) en agua desionizada, así como PEG puro y mantener las soluciones a 65 ° C.
- Incube el tejido en una serie de baños graduados de PEG para desplazar el agua y permitir que el PEG se infiltre.
Nota: Típicamente, se utiliza PEG con un peso molecular de 2.000 (PEG2000) para incrustar.- Procesar el tejido con g(C) 70% de PEG durante 2 h, (d) 90% de PEG durante 2 h, y (b) 50% de PEG durante 1,5 h, (E) 100% de PEG durante 10 h.
- Precalentar un casete a 65 ° C en un horno. Vierta el PEG que contiene el bloque en el casete y luego coloque el bloque de tejido en la posición deseada utilizando pinzas precalentadas o agujas.
- Enfriar lentamente el cassette y almacenar el tejido a RT hasta su uso.
- Diseccionar el bloque de PEG que contiene el tejido objetivo en un pequeño bloque (aproximadamente 1 cm x 1 cm x 2 cm) usando una cuchilla de afeitar afilada y montarlo en el micrótomo.
- Cortar secciones delgadas del bloque de PEG (típicamente 3-10 μm).
- Enjuague la sección con agua desionizada en un cristal de reloj 10 veces para eliminar el PEG del tejido.
- Sumergir las secciones con tolueno / etanol (2: 1, v / v) durante 6 h para eliminar los extractos. Preparar el líquido de reacción mezclando 65 ml de agua desionizada, 0,5 ml de ácido acético y 0,6 g de cloro sódicoRito en un vaso de precipitados. Añadir una sección y 3 ml de líquido de reacción a un vial de 5 ml. Atornille la parte superior del vial.
- Calentar el vial en un baño de agua a 75 ° C durante 2 h para eliminar la lignina del tejido. Enjuague la sección con agua desionizada en un cristal de reloj 10 veces.
- Transferir la sección no tratada / deslignificada a un portaobjetos de microscopio de vidrio. Despliegue la sección cuidadosamente con pinceles o agujas. Retire el agua desionizada extra con papel de seda.
- Sumergir la sección en D 2 O. Cubrir la muestra con un protector de vidrio. Sellar el protector con esmalte de uñas para evitar la evaporación del D 2 O.
2. Adquisición Espectral
- Abra el software de operación del instrumento del microscopio Raman confocal. Encienda el láser (longitud de onda = 532 nm), enfoque en la superficie del silicio cristalino con un objetivo de microscopio 100X y haga clic en el botón de calibración para calibrar el instrumento.
- Cambiar el instrumentoAl modo de microscopio óptico y encienda la lámpara del microscopio. Montar el portaobjetos del microscopio sobre el escenario, con el revestimiento de la cubierta frente al objetivo.
- Ver la muestra con un objetivo de microscopio 20X y localizar el área de interés. Aplique aceite de inmersión a la cubreobjetos y cambie al objetivo del microscopio de inmersión (60X, apertura numérica NA = 1.35). Enfoque en la superficie de la muestra.
- Cambie el instrumento al modo de prueba Raman y apague la lámpara del microscopio.
- Elija un área de asignación mediante una herramienta rectangular. Alterar el tamaño del paso para determinar el número de espectros obtenidos.
Nota: Tenga en cuenta el tamaño del paso (generalmente mayor que el diámetro del punto calculado por la apertura numérica del objetivo, teóricamente 1.22λ / NA). Los tamaños debajo de esto resultarán en sobremuestreo. - Establezca los parámetros espectrales óptimos para obtener la mejor relación señal-ruido (SNR) y calidad espectral en un tiempo de adquisición apropiado (generalmente <8 h), depeLa adecuación de la muestra. En general, introduzca los parámetros de imagen en el software del instrumento de la siguiente manera: láser (532 nm), filtro (100%), agujero (300), hendidura (100), espectrómetro (1.840 cm -1 ), ranuras (1200t) Aceite 60X) y tiempo de adquisición (2 s).
- Guarde los datos espectrales antes de procesar los datos y convertirlos a un formato universal ( por ejemplo, archivos TXT).
3. Análisis de datos
- Cargar los datos espectrales (archivos TXT) en el software de análisis de datos ( por ejemplo, Matlab). Aplicar una técnica de reducción de ruido en el conjunto de datos para mejorar la SNR ( por ejemplo, el algoritmo de Savitzsky-Golay o el algoritmo wavelet)
- Utilice muestras no tratadas para producir las imágenes de lignina y polisacáridos. Para la obtención de imágenes de lignina, considere el pico espectral alrededor de 1.600 cm -1 debido a la vibración de estiramiento simétrica del anillo aromático. Para la obtención de imágenes de polisacáridos (incluyendo celulosa y hemicelulosa), utilice el pico espectral aAlrededor de 2.889 cm -1 debido a los tramos CH y CH 2 .
- Utilizar muestras deslignificadas para generar las imágenes de celulosa y hemicelulosa. Realizar la auto-modelación de la curva de resolución (SMCR) en los espectros adquiridos para discriminar los espectros de celulosa y hemicelulosa y la imagen de sus distribuciones.
- Realizar análisis de componentes principales (PCA) y análisis de agrupamiento en los datos adquiridos para distinguir los espectros Raman de diferentes capas de pared celular.
Resultados
La Figura 1 presenta una visión general de un sistema micro-Raman típico para la imagen Raman de una pared celular de una planta. Como ejemplo, los espectros Raman originales del álamo ( Populus nigra L.) tienen deriva y espigas de línea de base significativas ( Figura 2a ). Después de realizar el método de preprocesamiento automático para el conjunto de datos de imágenes Raman (APRI), estos dos contaminantes espectrales se eliminan con éxito ( Figura 2b ). Un espectro Raman típico de álamo se muestra en la Figura 3 , y sus asignaciones de banda se enumeran en la Tabla 1 . La imagen Raman de lignina se genera por la integración de la región espectral de 1.550-1.650 cm -1 , que se atribuye a la estructura de anillo aromático ( Figura 4a ). La figura 4d muestra la imagen Raman de polisacáridos, whQue se consigue integrando el pico a 2889 cm $ ^ { - 1 } $ . Para la imagen de celulosa y hemicelulosa, se realiza SMCR sobre los datos de imagen Raman de una muestra deslignificada. Los espectros e imágenes correspondientes se muestran en la Figura 5 . La Figura 6 presenta los resultados obtenidos por PCA y análisis de agrupamiento.

Figura 1: Un Esquema de Imaging Raman para el Muro de Célula de Planta. La muestra de sección transversal (álamo como ejemplo) se mide mediante un sistema micro-Raman que acopla un microscopio óptico a un espectrómetro Raman de alta resolución con un detector de dispositivo de carga acoplada (CCD). En la imagen de campo luminoso, la pared celular de la planta está organizada en la esquina de la celda (CC), la lámina central compuesta (CML) y la pared secundaria (SW). Convencionalmente, una imagen Raman se genera mediante un solo picoIntegración o intensidad. En los datos originales se encuentran dos contaminantes espectrales ( es decir, desviaciones de línea de base y picos cósmicos). Haga clic aquí para ver una versión más grande de esta figura.
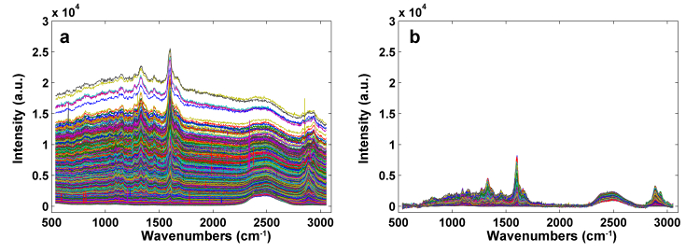
Figura 2: Espectros de Raman antes ( a ) y después ( b ) Pretratamiento por APRI. Se eliminan con éxito dos contaminantes espectrales. Haga clic aquí para ver una versión más grande de esta figura.
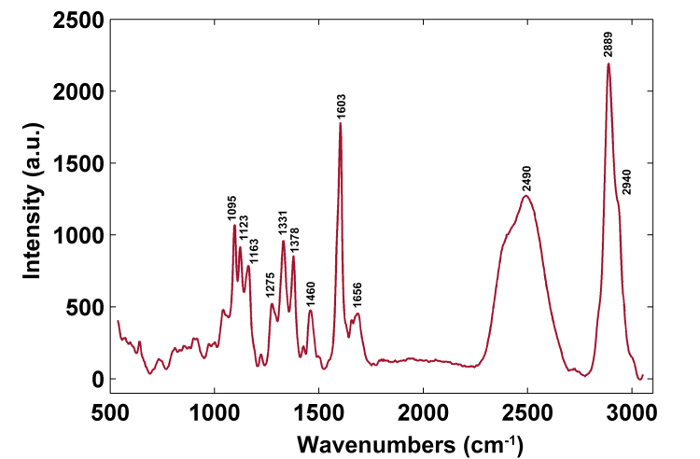
Figura 3: Espectro Raman típico de la pared celular del álamo. Haga clic aquí para ver una versión más grande de esta figura.
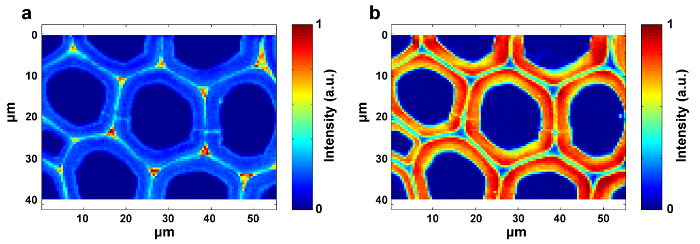
Figura 4: Imágenes Raman de Lignina ( a ) y Polisacáridos ( b ) dentro de la Malla Celular del Poplar. La imagen de lignina se genera integrando el pico alrededor de 1.600 cm -1 . La imagen del polisacárido se produce integrando el pico alrededor de 2.889 cm -1 . Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Imágenes Raman de celulosa ( a ) y hemicelulosa ( b ) dentro de la pared celular de álamo. SMCR se realiza sobre los datos de imagen Raman de una muestra deslignificada. La deslignificación contribuye a la exposición de las características espectrales de la celulosa y la hemicelulosa. La celulosa se concentra principalmente en el SO, mientras que la distribución de la hemicelulosa es casi uniforme en toda la pared celular del álamo. Haga clic aquí para ver una versión más grande de esta figura.
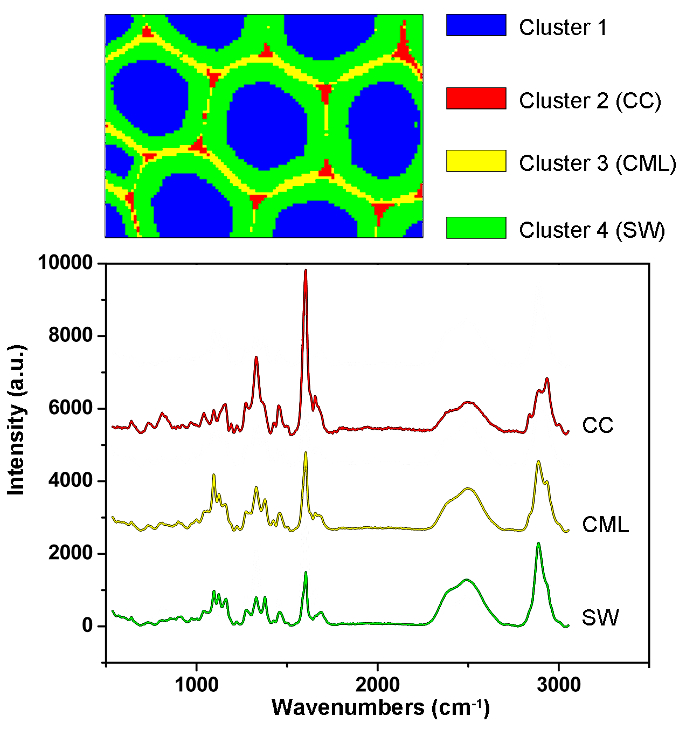
Figura 6: Resultados de análisis de PCA y agrupación para identificar automáticamente diferentes capas de pared celular en la pared celular de álamo. Mediante el uso de PCA y análisis de agrupación, los espectros se dividen en cuatro partes correspondientes a la luz celular, CC, CML y SW. A continuación se dan los espectros medios de estas capas. Los resultados revelaron que la lignina se concentra a lo largo de la CML y en el CC, mientras que los polisacáridos se concentran principalmente en el SW. Haga clic aquí para ver una versión más grande de esta figura.
| Número de ondas (cm ^ { - 1} ) | Componentes | Asignaciones |
| 1.095 | Celulosa, Hemicelulosa | Átomo pesado CC y CO estiramiento vibración |
| 1,123 | Celulosa, Hemicelulosa | Átomo pesado CC y CO estiramiento vibración |
| 1,163 | Celulosa, Hemicelulosa | Átomo pesado CC y vibración de estiramiento de CO más vibración de flexión HCC y HCO |
| 1,275 | Lignina | Aril-O de arilo OH y arilo O-CH3; Guaiacilo con el grupo C = O |
| 1.331 | Lignina, Celulosa, Hemicelulosa | Vibración de flexión HCC y HCO |
| 1.378 | Celulosa, Hemicelulosa | HCC, HCO y HOC vibración de flexión |
| 1,460 | Lignina, Celulosa, Hemicelulosa | Vibración de flexión HCH y HOC |
| 1.603 | Lignina | Anillo arilo vibración de estiramiento, vibración simétrica |
| 1.656 | Lignina | Anillo conjugado C = C vibración de estiramiento de alcohol coniferílico; C = O vibración de estiramiento de coniferaldehído |
| 2.889 | C H | CH y CH 2 vibración de estiramiento |
| 2,940 | L, C, H | CH vibración de estiramiento en OCH 3 vibración asimétrica |
Tabla 1: Posiciones máximas de Raman y asignaciones de bandas
Discusión
La pared celular de la planta es un compuesto que se organiza en varias capas, incluyendo la esquina de la celda (CC), la pared secundaria (SW, con las capas S1, S2 y S3) y la lámina central compuesta (CML, Pared), lo que hace difícil obtener una superficie plana durante la preparación de la muestra. Por lo tanto, las muestras de plantas, especialmente hierba, que tiene una estructura más complicada que la madera, a menudo necesitan solidificarse para permitir una sección fina. PEG es una matriz dura ideal para el corte y la investigación Raman, ya que es soluble en agua. Se puede eliminar fácilmente enjuagando con agua desionizada. PEG utilizado para la incrustación tiene diferentes pesos moleculares, que varían de 1.000-20.000. Cuanto mayor sea el peso molecular del PEG, menor será la capacidad de penetración 10 . D2O ayudará a reducir la fluorescencia de la lignina y tiene un pico marcado a 2.490 cm -1 , pero no eliminará la interferencia de fluorescencia. DosLas señales de ruido principal se derraman en los canales, junto con las señales reales: 1) la fluorescencia de la muestra y del fondo, así como las fluctuaciones térmicas del CCD, pueden dar lugar a deriva basal y 2) los rayos cósmicos pueden afectar notablemente a los detectores sensibles, que se manifiestan en Como anchos de banda estrecha. El método APRI se desarrolló para abordar estas cuestiones 11 . El APRI incluye los mínimos cuadrados penalizados por penetración (airPLS) y el análisis de componentes principales (PCA) para eliminar las derivaciones de línea de base y los picos cósmicos usando las características espectrales mismas.
Para lograr imágenes de distribución, se debe seleccionar la intensidad / integración de pico y la adaptación lineal de todo el espectro mediante métodos multivariados. El primero es adecuado para los componentes con picos Raman específicos ( por ejemplo, lignina), mientras que el segundo es apropiado para los componentes con fuerte solapamiento espectral ( por ejemplo, celulosa y hemicelulosa). La distriSe encuentran disponibles imágenes de lignina y polisacáridos integrando los picos específicos alrededor de 1.600 cm -1 y 2.889 cm -1 , respectivamente (Figura 4). Sin embargo, las imágenes de celulosa y hemicelulosa son difíciles de generar directamente por integración de picos debido a la fuerte superposición espectral. El análisis multivariado permite clasificar el contenido de información convoluto de acuerdo con la hipótesis de que los datos originales se reconstruyen a partir de un número limitado de factores significativos [ 12] . Por lo tanto, se puede aplicar para discriminar sus espectros y producir imágenes Raman correspondientes. Para muestras de plantas, la presencia de extractivos y lignina interfiere con la capacidad de reconstruir los espectros de celulosa y hemicelulosa. Es necesario eliminar estas interferencias antes del análisis SMCR de los datos de imagen. SMCR fue introducido por primera vez por Lawton y Sylvester como una técnica multivariante específicamente desarrollado para resolver un componente puro fDe un conjunto de espectros, sin ningún recurso a una biblioteca espectral, para analizar los datos de imagen 13 . La clasificación espectral es importante para comprender mejor la naturaleza estructural y química de la pared celular de la planta. Aquí, los datos de imagen han sido sometidos a análisis de componentes principales (PCA) y el análisis de agrupamiento para distinguir los espectros Raman de diferentes capas de la pared celular [ 14] .
Sin embargo, esta técnica tiene dos limitaciones. En primer lugar, el efecto Raman es débil: la sección transversal típica de dispersión Raman es ~ 10-29 cm 2 por molécula, lo que la hace vulnerable a la fluorescencia intensa 15 . Una posible manera de mejorar el defecto es preparando las secciones lo más delgada posible y aplicando un algoritmo de corrección de línea de base, pero las señales fluorescentes son difíciles de eliminar. En segundo lugar, el tratamiento químico es un procedimiento significativo cuando se consiguen imágenes Raman de célulasUlosa y hemicelulosa, y puede aumentar el riesgo de cambiar el contenido químico original. Por lo tanto, es mejor quitar los extractivos y la lignina tanto como sea posible, sin la estructura de la pared celular que se ve demasiado diferente de la no tratada.
En conclusión, este protocolo es adecuado para estudiar la distribución de lignina, celulosa y hemicelulosa dentro de la pared celular de la planta. El uso de imágenes Raman para obtener la información deseada depende en gran medida de la habilidad del operador en la preparación de la muestra y en el análisis de los datos. Una buena preparación de muestras es esencial para recoger espectros Raman de alta calidad. Un análisis apropiado de los datos proporciona información sobre los espectros a gran escala y extrae la información oculta de la imagen. Como método fundamental, este protocolo puede utilizarse para rastrear los cambios dinámicos de los principales componentes durante el tratamiento químico, físico o biológico a nivel micro.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Damos las gracias al Ministerio de Ciencia y Tecnología de China (2016YDF0600803) por el apoyo financiero.
Materiales
| Name | Company | Catalog Number | Comments |
| Microtome | Thermo Scientific | Microm HM430 | |
| Confocal Raman microscope | Horiba Jobin Yvon | Xplora | |
| Oven | Shanghai ZHICHENG | ZXFD-A5040 |
Referencias
- Gonzalo, G. D., et al. Bacterial Enzymes Involved in Lignin Degradation. J. Biotechnol. 236, 110-119 (2016).
- Rosatella, A. A., Afonso, C. A. M. Chapter 2. Ionic Liquids in the Biorefinery Concept: Challenges and Perspectives. , 38-64 (2016).
- Sun, L., et al. Understanding tissue specific compositions of bioenergy feedstocks Through hyperspectral Raman imaging. Bio. 108 (2), 286-295 (2009).
- Tolstik, T., et al. Classification and prediction of HCC tissues by Raman imaging with identification of fatty acids as potential lipid biomarkers. J. Cancer. Res. Clin. Oncol. 141 (3), 407-418 (2015).
- Schrader, B. . Infrared and Raman spectroscopy: methods and applications. , (2008).
- Gierlinger, N., et al. Imaging of plant cell walls by confocal Raman microscopy. Nat. Protoc. 7 (9), 1694-1708 (2012).
- Luca, A. C. D., et al. Online fluorescence suppression in modulated Raman spectroscopy. Anal. Chem. 82 (2), 738-745 (2009).
- Schlücker, S., et al. Raman microspectroscopy: a comparison of point, line, and wide-field imaging methodologies. Anal. Chem. 75 (16), 4312-4318 (2003).
- Cooper, J. B. Chemometric analysis of Raman spectroscopic data for process control applications. Chemometr. Intell. Lab. Syst. 46 (2), 231-247 (1999).
- Cheng, H. J., Hsiau, S. S. The study of granular agglomeration mechanism. Powder Technol. 199 (3), 272-283 (2010).
- Zhang, X., et al. Method for removing spectral contaminants to improve analysis of Raman imaging data. Sci. Rep. 6, 39891 (2016).
- Shinzawa, H., et al. Multivariate data analysis for Raman spectroscopic imaging. J. Raman Spectrosc. 40 (12), 1720-1725 (2009).
- Lawton, W. H., Sylvestre, E. A. Self modeling curve resolution. Technometrics. 13, 617-633 (1971).
- Zhang, X., et al. Method for automatically identifying spectra of different wood cell wall layers in Raman imaging data set. Anal. Chem. 87 (2), 1344-1350 (2015).
- Kudelski, A. Analytical application of Raman spectroscopy. Talanta. 76 (1), 1-8 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados