Method Article
Extraktion und Identifizierung von Mikrokernen aus humanen Lymphozyten des peripheren Blutes
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt ein Verfahren zur Extraktion und Reinigung von Mikrokernen aus menschlichen Lymphozyten mittels Saccharose-Dichtegradientenzentrifugation. Es bietet eine experimentelle Grundlage, um die Zusammensetzung und Funktion von Mikrokernen zu untersuchen.
Zusammenfassung
Ein Mikronukleus (MN) ist eine abnormale Kernstruktur, die sich in Zellen, insbesondere im Knochenmark oder in Blutzellen, bildet, wenn sie äußeren Schäden wie Strahlung aufgrund von ungelösten DNA-Schäden oder mitotischen Fehlern ausgesetzt wird. Einmal gebildet, können MNs aktiv zu verschiedenen krebserregenden Prozessen beitragen, einschließlich entzündlicher Signalwege und chromosomaler genetischer Umlagerungen. MNs enthalten Kern-DNA, Histone, Nukleoproteinfragmente und andere aktive Proteine, die eng mit ihren Funktionen verbunden sind. Die Untersuchung der Bildung und der Bestandteile von MN ist entscheidend für das Verständnis ihrer Rolle bei der Steuerung krebserregender Prozesse. Die Extraktion und Reinigung von MN sind für das Erreichen dieser Forschungsziele unerlässlich. Die Instabilität der MN-Kernmembran und ihre Anfälligkeit für Brüche machen diese Prozesse jedoch technisch anspruchsvoll. Derzeit gibt es nur wenige Studien, die über den Einsatz der Dichtegradientenzentrifugation für die MN-Trennung berichten. Diese Studie fasst die Prozesse der MN-Trennung und -Reinigung zusammen und vereinfacht sie. Humane periphere Blutlymphozyten, die der Strahlung ausgesetzt waren, wurden isoliert, und MNs wurden mit Saccharosepuffern unterschiedlicher Konzentrationen in einem zweistufigen Prozess getrennt und gereinigt. Die Integrität und Reinheit der MNs wurde verifiziert und bietet Forschern, die die Ursachen und Funktionen von MNs untersuchen, eine klare und praktische Demonstration des experimentellen Verfahrens.
Einleitung
Der Mikronukleus (MN) ist eine subzelluläre Struktur im Zytoplasma, die Kern-DNA, Histone und Nukleoproteinfragmente enthält, die von Membranstrukturen umgeben sind 1,2. Das MN ist vollständig vom Hauptkern getrennt und erscheint typischerweise in seiner Nähe in einer ovalen oder kreisförmigen Form mit einer Größe von 1/16 bis 1/3 des Durchmessers des Hauptkerns. Die Bildung von MNs ist in erster Linie mit azentrischen Chromosomenfragmenten, chromosomaler Missegregation, dizentrischem Chromosomenbruch, Chromosomeninstabilität und der Aggregation von Doppelminuten (DBs) verbunden3. MNs kommen in gesunden Zellen selten vor und werden überwiegend durch die Exposition gegenüber exogenen Genotoxinen verursacht, die zu DNA-Schäden oder mitotischen Fehlern führen 4,5. Strahlenschäden tragen erheblich zur MN-Bildung bei, und die Exposition des Menschen gegenüber ionisierender Strahlung (IR) hat mit den Fortschritten in klinischen und technologischen Anwendungen zugenommen 6,7. Die IR-Exposition induziert Einzelstrangbrüche (SSBs) und Doppelstrangbrüche (DSBs) in der zellulären DNA, die zum Zelltod oder zur Apoptose führen können 8,9,10. MNs sind Fragmente von Chromosomen, ganzen Chromosomen oder Chromatiden, die aufgrund einer nicht reparierten oder nicht übereinstimmenden DSB-Reparatur entstehen. Sie dienen als kritische Indikatoren für den Grad der Schädigung, die durch IR 7,11,12 verursacht werden. Ein umfassendes Verständnis der Komponenten in MNs ist unerlässlich, um die Nebenwirkungen der Strahlentherapie zu mildern und die Exposition der Öffentlichkeit gegenüber unerwünschter Strahlung zu minimieren 6,13. Die in MNs enthaltenen Proteine und Nukleinsäuren sind jedoch bis heute unvollständig charakterisiert.
Es wird angenommen, dass MNs aus der Induktion verschiedener Arten von DNA-Schäden resultieren. Ihre Replikationsmechanismen und DNA-Reparaturfähigkeiten sind beeinträchtigt, was innerhalb kurzer Zeit zu umfangreichen DNA-Schäden führt 3,14,15. MNs sind auch wichtige Indikatoren für die chromosomale Instabilität, die in Krebsvorstufen konsistent vorhanden ist 3,14,16,17,18,19. MNs werden seit langem als Biomarker für Genotoxizität, Tumorrisiko und Tumorgrad 3,20,21 verwendet. Einmal gebildet, können MN zahlreiche krebserregende Prozesse aktiv antreiben, einschließlich der Entzündungssignalisierung22,23 und der chromosomalen genetischen Umlagerung 24,25,26. Zum Beispiel initiieren MNs eine Entzündungskaskade, indem sie die zyklische GMP-AMP-Synthase (cGAS) des viralen Mustererkennungsrezeptors (PRR) aus dem Zytoplasma rekrutieren 4,22,23,27. Andere Proteine, die in MNs vorhanden sein können, sind die Exonukleasen28, die Transkriptionsmechanismen4 und die Translationskofaktoren. Es bleibt unklar, ob MNs ein einzigartiges, verzerrtes Proteinprofil aus der zytoplasmatischen Proteinbibliothek zusammensetzen oder passiv Proteine mit hoher Abundanz aus dem Zellkern und dem Zytoplasma während ihrer Bildung aufnehmen. Abgesehen von einzelnen Proteinen wie cGAS ist noch nicht bestimmt, inwiefern bestimmte Umgebungen die Gesamtzusammensetzung von MN beeinflussen. Das Verständnis der gesamten Mikrokernlandschaft ist begrenzt, da es keine öffentlich zugänglichen Datensätze für die Erkundung gibt. Daher ist die Extraktion vollständiger und gereinigter MNs für eine tiefgreifende und umfassende Analyse ihrer Bestandteile dringend erforderlich.
Ein Grund für eine verzögerte DNA-Replikation in MNs könnte Replikationsstress sein, der durch einen Mangel an Enzymen und Cofaktoren verursacht wird, die für die DNA-Synthese und -Reparatur erforderlich sind. Dieser Mangel kann aus einer fehlerhaften Assemblierung der MN-Kernhülle resultieren, die zum Fehlen eines Kernporenkomplexes20 führt. Folglich sind MNs nicht in der Lage, Schlüsselproteine zu importieren, die für die Aufrechterhaltung der Integrität der Kernmembran und der Genomstabilität unerlässlich sind 2,29,30,31. Die unvollständige MN-Kernmembran ist anfällig für Brüche, was die MN-Extraktion zu einer großen Herausforderung macht.
Derzeit werden MNs hauptsächlich mit Hilfe der Saccharose-Dichtegradientenzentrifugation27,32,33 extrahiert und durch Durchflusszytometrie34 gereinigt. In dieser Studie wurde der Trennungs- und Reinigungsprozess von MNs zusammengefasst und vereinfacht. Lymphozyten aus dem peripheren Blut von Menschen, die Strahlung ausgesetzt waren, wurden isoliert, und MNs wurden zweimal mit Saccharosepuffern unterschiedlicher Konzentrationen gereinigt. Die Integrität und Reinheit der MNs wurden verifiziert und den Forschern eine praktische und intuitive experimentelle Demonstration zur Untersuchung der Ursachen und Funktionen von MNs geboten.
Protokoll
Alle Experimente mit humanen peripheren Blutproben wurden in Übereinstimmung mit den einschlägigen Richtlinien und Vorschriften durchgeführt. Diese Studie wurde vom Ethical Committee of Nuclear Industry 416 Hospital, China (2020 Review [Nr. 48]), genehmigt und von allen Teilnehmern wurde eine Einverständniserklärung eingeholt. Zu den Ausschlusskriterien gehörten Personen mit schweren chronischen Krankheiten wie Tumoren, Gicht oder Bluterkrankungen sowie Personen, die in ihrem Beruf radioaktiven oder genotoxischen Substanzen ausgesetzt waren. Für diese Studie wurden 10 ml peripheres Blut von einem gesunden 28-jährigen Mann in ein Blutentnahmeröhrchen entnommen, das Heparin-Natrium als Antikoagulans enthielt. Die Probe wurde in vitro mit 60Co γ-Strahlen bei 37 °C unter Verwendung eines Standardgeräts für Gamma-Luft-Bewegungsenergie auf therapeutischem Niveau bestrahlt. Die Bestrahlungsdosis wurde auf 4,0 Gy mit einer Dosisleistung von 0,6350 Gy/min festgelegt. Nach der Bestrahlung wurde die Probe 2 h lang bei 37 °C inkubiert, in RPMI-1640-Medium inokuliert und 60 h lang bei 37 °C kultiviert. Einzelheiten zu den in dieser Studie verwendeten Reagenzien und Geräten sind in der Materialtabelle enthalten.
1. Präparation des Mikrokerns und des Zellkerns
- Geben Sie Cytochalasin B in das periphere Blutzellmedium, um eine Endkonzentration von 10 μg/ml zu erreichen. Die Mischung 45 min bei 37 °C inkubieren.
HINWEIS: Cytochalasin B ist ein Aktinpolymerisationsinhibitor, der die effiziente Trennung von Mikrokernen vom Zellkern vor der Zelllyse erleichtert35. - Zentrifugieren Sie die Blutzellen bei 200 × g für 5 min bei 4 °C.
- Die Blutzellen werden in 10 ml 0,01 M PBS resuspendiert, vorgekühlt bei 4 °C.
2. Isolierung von Lymphozyten
- Geben Sie 15 ml humane Lymphozytentrennungslösung in ein konisches 50-ml-Röhrchen. Schichten Sie die suspendierten peripheren Blutzellen vorsichtig auf die Trennlösung.
- Das Röhrchen bei 400 × g 30 min bei 4 °C zentrifugieren.
HINWEIS: Nach der Zentrifugation trennen sich die Blutzellen im Röhrchen von oben nach unten in vier verschiedene Schichten: Die erste Schicht ist die Plasmaschicht (Blutplättchen), die zweite Schicht ist die milchig-weiße Ring-Lymphozytenschicht, die dritte Schicht ist die transparente Trennflüssigkeitsschicht und die vierte Schicht ist die Schicht der roten Blutkörperchen. - Übertragen Sie die zweite Schicht (Lymphozyten) vorsichtig in ein separates Zentrifugenröhrchen. PBS (0,01 M) mit dem dreifachen Volumen der übertragenen Zellen zugeben, gut mischen und bei 200 × g für 10 min bei 4 °C zentrifugieren. Entsorgen Sie den Überstand mit einer Pipette.
3. Lyse von Lymphozyten
- Legen Sie das in der konischen Röhre gesammelte Zellpellet auf Eis.
- Fügen Sie 6 mL Kaltlysepuffer hinzu (Tabelle 1, Zusatzdatei 1) und resuspendieren Sie das Zellpellet im Lysepuffer.
HINWEIS: Der Lysepuffer sollte unmittelbar vor der Verwendung mit den in Tabelle 2, Zusatzdatei 1 aufgeführten Inhaltsstoffen ergänzt werden. - Übertragen Sie das Zelllysat in einen Glashomogenisator.
- Verwenden Sie vorsichtig einen losen Mahlgrad, um die Probe manuell zu homogenisieren, indem Sie den Homogenisator 10 Mal auf und ab bewegen.
- Übertragen Sie das homogenisierte Zelllysat zurück in das konische 50-ml-Röhrchen.
HINWEIS: Führen Sie alle Schritte vorsichtig durch und halten Sie die Probe auf Eis.
4. Initiale Isolierung von MNs über einen Saccharose-Dichtegradienten
- Mischen Sie 5 ml des Zelllysats mit einem gleichen Volumen von 1,8 M Saccharosepuffer (Tabelle 3, Zusatzdatei 1), um ein Verhältnis von Lysat zu Saccharosepuffer von 1:1 zu erreichen.
HINWEIS: Der 1,8 M Saccharosepuffer sollte unmittelbar vor der Verwendung mit den in Tabelle 4, Zusatzdatei 1 aufgeführten Bestandteilen ergänzt werden. - Bereiten Sie den Saccharose-Dichtegradienten vor: Geben Sie 15 mL 1,6 M Saccharosepuffer (Tabelle 5, Zusatzdatei 1) auf den Boden eines konischen 50-ml-Röhrchens. Geben Sie langsam 20 ml 1,8 M Saccharosepuffer (Tabelle 3, Zusatzdatei 1) auf den 1,6 M Saccharosepuffer.
HINWEIS: Sowohl der 1,6 M Saccharosepuffer als auch der 1,8 M Saccharosepuffer sollten unmittelbar vor der Anwendung mit den in Tabelle 4, Ergänzende Datei 1 aufgeführten Bestandteile ergänzt werden. Um die Saccharosepuffer hinzuzufügen, legen Sie die Spitze der Pipette gegen die obere Innenseite des konischen Röhrchens und pipettieren Sie die Lösung langsam. - Pipettieren Sie langsam 10 ml des Gemischs [Lysat 1:1 1,8 M Saccharosepuffer] auf den zweischichtigen Saccharosedichtegradienten.
HINWEIS: Fügen Sie die Schichten langsam hinzu, um sicherzustellen, dass sich die mittlere 1,8-M-Pufferschicht nicht mit der unteren 1,6-M-Pufferschicht vermischt und die obere 1:1-Mischung aus lysierten Zellen und 1,8 M-Saccharosepuffer sich nicht mit der 1,8 M-Pufferschicht vermischt. - Das Röhrchen wird in einer Horizontalrotorzentrifuge bei 1000 × g für 20 min bei 4 °C zentrifugiert.
HINWEIS: Nach der Zentrifugation trennt sich die Flüssigkeit in drei Schichten: Die obere Schicht enthält ca. 3 ml Zelltrümmer, die mittlere Schicht enthält ca. 3 mL Mikronuklei (MNs), die untere Schicht enthält ca. 39 mL Zellkerne. - Entfernen Sie die oberen 3 ml Flüssigkeit in der Nähe der Oberfläche und sammeln Sie die 3 ml ungereinigter Mikrokerne aus der mittleren Schicht für den zweiten Saccharose-Dichtegradienten.
5. Sekundärreinigung von MNs über einen Saccharose-Dichtegradienten
- Bereiten Sie den Saccharose-Dichtegradienten vor:
- Geben Sie 3 ml 1,8 M Saccharosepuffer (Tabelle 3, Zusatzdatei 1) auf den Boden eines konischen 15-ml-Röhrchens.
- 3 mL 1,5 M Saccharosepuffer (Tabelle 6, Zusatzdatei 1) zusätzlich zu dem 1,8 M Saccharosepuffer geben.
- Zum Schluss fügen Sie 3 mL 1,4 M Saccharosepuffer (Tabelle 7, Zusatzdatei 1) zu dem 1,5 M Saccharosepuffer hinzu.
HINWEIS: Positionieren Sie die Spitze der Pipette an der oberen Innenseite des konischen Röhrchens und pipettieren Sie die Lösung langsam.
- Pipettieren Sie langsam 3 mL der mittleren Schicht aus dem zweiten Saccharosedichtegradienten auf die Oberseite des dreischichtigen Saccharosedichtegradienten.
HINWEIS: Fügen Sie die Schichten langsam hinzu, um ein Vermischen zu vermeiden. - Das Röhrchen wird in einer Horizontalrotorzentrifuge bei 500 × g für 20 min bei 4 °C zentrifugiert.
HINWEIS: Nach der Zentrifugation enthält die obere Schicht 1-4 ml gereinigte Mikrokerne (MN) und die untere Schicht hauptsächlich 8 ml Zellkerne. - Bewahren Sie die oberen 1-4 ml der gereinigten Mikrokerne für die spätere Verwendung auf.
6. Identifizierung von multinationalen Finanzinstituten
- Nehmen Sie die ungereinigte Mikronukleuslösung (erhalten in Schritt 4) und die gereinigte Mikronukleuslösung (erhalten in Schritt 5) und bereiten Sie Zellabstriche auf dem Objektträger vor.
- Fügen Sie 4%ige Paraformaldehydlösung hinzu, um die Probe 15 Minuten lang bei Raumtemperatur (RT) zu fixieren.
- Waschen Sie die Objektträger zweimal mit PBS (0,01 m), 3 min pro Waschgang.
- Fügen Sie 0,1% Triton X-100 hinzu, um die Zellen bei RT für 5 min zu permeabilisieren.
- Blockieren Sie die Zellen in 5% BSA bei RT für 30 min.
- Den Primärantikörper gegen Anti-γ-H2AX (verdünnt in 5 % BSA, 1:200) zugeben und 1 h bei RT inkubieren.
- Waschen Sie die Objektträger zweimal mit PBS (0,01 m), 5 min pro Waschgang.
- Den Sekundärantikörper (verdünnt in 5 % BSA, 1:500) zugeben und 1 Stunde lang bei RT im Dunkeln inkubieren.
- Fügen Sie DAPI-Farbstofflösung hinzu und inkubieren Sie 15 Minuten lang bei RT.
- Waschen Sie die Objektträger zweimal mit PBS (0,01 m), 3 min pro Waschgang.
- Tragen Sie eine Dichtungslösung auf den Objektträger auf, um ihn abzudichten.
- Beobachten Sie die Fluoreszenz unter einem Fluoreszenzmikroskop (Vergrößerung: 400 ×, Autofokus und automatische Belichtung) und zählen Sie die Mikrokerne mit einer Zellzählplatte.
HINWEIS: 4 % Paraformaldehyd, 0,1 % Triton X-100 und 5 % BSA werden mit PBS (0,01 M) verdünnt. γ-H2AX (Gamma H2AX) ist eine phosphorylierte Form des Histon-H2AX-Proteins, die als Reaktion auf DNA-Schäden gebildet wird. Im Zellkern ist die Phosphorylierung von Histon H2AX in bestimmten Bereichen konzentriert, so dass die Anti-γ-H2AX-Fluoreszenz fleckig erscheinen kann. Die Phosphorylierung von Histon H2AX ist im Mikronukleus konzentriert, was zu einer insgesamt helleren Fluoreszenz mit deutlichen Fleckenführt 36. DAPI ist ein Fluoreszenzfarbstoff, der stark an die DNA bindet und häufig in der Fluoreszenzmikroskopie verwendet wird. Da Mikrokerne gebrochene DNA- und Histonfragmente enthalten, können sie mit anti-γ-H2AX-Fluoreszenzantikörpern und DAPI gefärbt werden. Die Fluoreszenz zeigt die Anzahl, Größe und Vollständigkeit der Mikrokerne an, hilft dabei, sie von den Zellkernen zu unterscheiden und die Reinigungswirksamkeitzu beurteilen 32.
Ergebnisse
Nach der Strahlenexposition wurde humanes peripheres Blut 60 Stunden lang mit RPMI-1640 inkubiert, und dann wurde Cytochalasin B zur Vorbereitung des Mikronukleus (MN) hinzugefügt. Die Lymphozyten wurden mit einer Lymphozytentrennungslösung isoliert, gefolgt von einer Lyse mit einem speziell konfigurierten Zelllysat und einer schonenden Homogenisierung in einem Glashomogenisator. Das Homogenat wurde 1:1 mit 1,8 M Saccharosepuffer gemischt. Ungereinigte Mikrokerne wurden nach der ersten Saccharose-Dichtegradientenzentrifugation erhalten, und gereinigte Mikrokerne wurden nach der zweiten Saccharose-Dichtegradientenzentrifugation erhalten (Abbildung 1).
Nach der ersten Saccharose-Dichtegradientenzentrifugation wurden ungereinigte Mikrokerne in Zellabstriche aufbereitet und einer Anti-γ-H2AX- und DAPI-Fluoreszenzfärbung unterzogen. Die Ergebnisse zeigten eine große Variation in der Größe der ungereinigten Mikrokerne, wobei einige größere Mikrokerne sichtbar waren (Abbildung 2A). Nach der zweiten Saccharose-Dichtegradientenzentrifugation wurden die gereinigten Mikrokerne in Zellabstriche aufbereitet und einer Anti-γ-H2AX- und DAPI-Fluoreszenzfärbung unterzogen. Die Ergebnisse zeigten, dass die gereinigten Mikrokerne kleiner waren und dass der Größenunterschied zwischen den Mikrokernen weniger ausgeprägt war, wobei keine großen Kerne beobachtet wurden (Abbildung 2B). Basierend auf 10 ml peripherem Blut können 100.000 bis 200.000 Mikrokerne gewonnen werden.
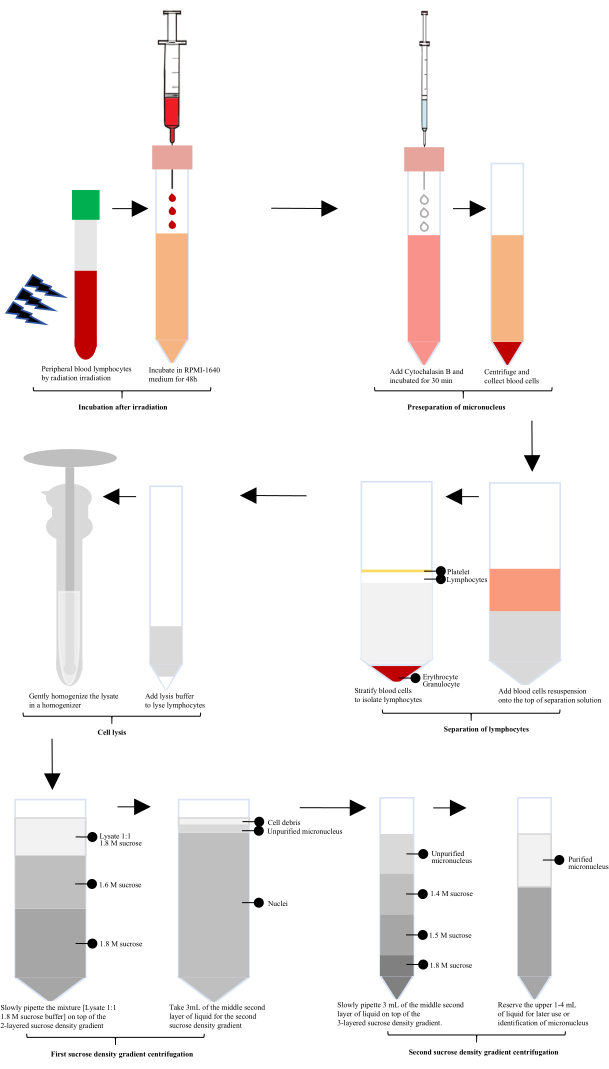
Abbildung 1: Schematische Darstellung der Extraktion und Reinigung von Lymphozyten-Mikrokernen nach Bestrahlung. Nach der Bestrahlung umfasst der Prozess eine Inkubation, eine Mikronukleus-Vortrennung, eine Isolierung von Lymphozyten, eine Zelllyse und zwei Runden der Saccharose-Dichtegradientenzentrifugation. In diesen Schritten werden dann gereinigte Mikrokerne erhalten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Identifizierung von Mikronukleen mittels Immunfluoreszenz. (A) Nach der ersten Saccharose-Dichtegradientenzentrifugation wurden ungereinigte Mikrokerne als Zellabstriche präpariert und einer Anti-γ-H2AX- (grün) und DAPI-Fluoreszenzfärbung (blau) unterzogen. Pfeile zeigen die Kerne an. (B) Nach der zweiten Saccharose-Dichtegradientenzentrifugation wurden gereinigte Mikrokerne als Zellabstriche präpariert und einer Anti-γ-H2AX- und DAPI-Fluoreszenzfärbung unterzogen. Maßstabsbalken: 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei 1: Reagenzien und Puffer, die in diesem Protokoll verwendet werden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Bei dieser Methode wurden bestrahlte humane Lymphozyten aus dem peripheren Blut zur Isolierung und Reinigung von Mikrokernen (MNs) verwendet. Viele frühere Studien haben über die detaillierten Schritte für die Zelllyse und die primäre Trennung der MNsberichtet 4,32,33,34. Die Zelllyselösung enthält typischerweise 0,1 % NP-40, um die Zellmembran zu zerstören, während sie eine minimale Wirkung auf die Kernmembran hat, wodurch die Integrität sowohl des Zellkerns als auch der MNs erhalten bleibt. Insbesondere dienen andere Komponenten des Lysats auch als Puffer 4,32,33,34. Aufgrund der unvollständigen Montage ist die MN-Membran anfällig für einen Bruch 2,29,30,31, was den Trenn- und Reinigungsprozess erschwert. Typischerweise werden 1,6 M und 1,8 M Saccharosepuffer in der Dichtegradientenzentrifugation für die Ersttrennung von MNs verwendet. Die Saccharosepuffer tragen zum Schutz und zur Pufferung der MNs bei, indem sie die Integrität der mikronuklearen Membran gewährleisten und gleichzeitig ihre Trennung über den Dichtegradienten 4,32,33,34 erleichtern. Bei dieser Methode wurden diese Schlüsselschritte für die Lymphozytenlyse und die primäre Trennung der MNs sorgfältig befolgt.
Im Gegensatz zu früheren Methoden der Mikronukleus-Trennung (MN) verbessert diese Methode den Reinigungsprozess. Über die Aufreinigung von MN durch Durchflusszytometrie wurde bereits berichtet33,34. MNs können jedoch aufgrund von Scherkräften während des Sortierprozesses reißen, was eine weitere Detektion von MN-Komponenten behindern kann. Darüber hinaus schränkt die begrenzte Verfügbarkeit von Durchflusszytometern die weit verbreitete Anwendung der Durchflusszytometrie für die MN-Sortierung ein. In anderen Studien wurde die sekundäre Saccharose-Dichtegradientenzentrifugation zur Aufreinigung von MN eingesetzt, aber vor der sekundären Zentrifugation wurde eine Ultrahochgeschwindigkeitszentrifugation verwendet4. Diese Hochgeschwindigkeitszentrifugation kann zum Bruch einiger MNs führen, was sich nachteilig auf die anschließende Analyse von MN-Komponenten auswirkt. Im Gegensatz dazu ist in diesem Protokoll die Isolierung und Reinigung von MN ein kritischer Schritt. Die sekundäre Saccharose-Dichtegradientenzentrifugation wird zur Reinigung von MN eingesetzt, wobei die MN-haltige Schicht, die aus der ersten Saccharose-Dichtegradientenzentrifugation gewonnen wird, direkt der zweiten Zentrifugation unterzogen wird. Dieser Ansatz vermeidet die Schädigung von MNs, die durch die Ultrahochgeschwindigkeitszentrifugation verursacht wird.
Diese Methode konzentriert sich ausschließlich auf die Isolierung und Reinigung von MNs aus humanen peripheren Blutlymphozyten und schließt andere Zelltypen, wie z. B. Tumorzellen, nicht ein. Zukünftige Studien werden darauf abzielen, die Aufreinigung von MNs aus verschiedenen Zelltypen zu demonstrieren, um die Universalität der Methode zu erhöhen. Darüber hinaus bleibt das Volumen des gereinigten MN-Gemisches relativ groß. Zukünftige Bemühungen werden sich auf die Optimierung des Saccharose-Dichtegradienten konzentrieren, um hochkonzentrierte MN-Lösungen zu erhalten, die weitere Untersuchungen der Zusammensetzung und Funktion von MNs erleichtern würden.
Das Erscheinungsbild von MNs spiegelt das Ausmaß der Chromosomen- und DNA-Schäden wider und spielt eine wichtige Rolle bei der Reparatur von Strahlenschäden und der Bildung von Krebszellen 7,11,12,13,14,16,17,18,19. Diese Prozesse sind eng mit den Rollen und Funktionen von Proteinen, Nukleinsäuren und anderen Komponenten in MNs verbunden. Daher ist die Reinigung von MNs entscheidend für die Untersuchung ihrer Rolle bei Krankheiten, die mit Chromosomen- und DNA-Schäden zusammenhängen. Diese Methode verbessert den sekundären Saccharose-Dichtegradienten-Zentrifugationsschritt im MN-Aufreinigungsprozess und demonstriert die Bildung von MNs in humanen peripheren Blutlymphozyten. Es umfasst auch die Zelllyse, die primäre Trennung unter Verwendung des ersten Saccharose-Dichtegradienten und die Reinigung mit dem zweiten Saccharose-Dichtegradienten. Dieser Ansatz bietet einen klaren, experimentellen Rahmen für Forscher, die die Ursachen und Funktionen von MNs untersuchen.
Offenlegungen
Nichts.
Danksagungen
Alle Abbildungen wurden von den Autoren über die Geschäftsstelle WPS erstellt. Diese Arbeit wurde von der Natural Science Foundation des Chengdu Medical College (CYZ19-38) und dem Sichuan Province Science and Technology Support Program (2024NSFSC0592) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | Thermo | 339650 | |
| 4% paraformaldehyde solution | Beyotime | P0099 | |
| 50 mL conical tube | Thermo | 339652 | |
| Anti-γ-H2AX antibody | Bioss | bsm-52163R | |
| Bovine serum albumin | Beyotime | ST023 | |
| Calcium chloride | Biosharp | BS249 | |
| Cytochalasin B | Macklin | 14930-96-2 | |
| DAPI dyeing solution | Bioss | S0001 | |
| Dithiothreitol (DTT) | Coolaber | CD4941 | |
| EDTA | Biosharp | BS107 | Ethylene Diamine Tetraacetic Acid |
| Fluorescence microscope | Nikon | Ti2-U | |
| Homogenizer | Ybscience | YB101103-1(20 mL) | |
| Horizontal controlled temperature centrifuge | Thermo | Sorvall ST1R plus | Brake speed is set to "5" |
| Human lymphocyte separation solution | Beyotime | C0025 | |
| Magnesium acetate | Macklin | M833330 | |
| NP-40 | Coolaber | SL9320 | 10% solution |
| Phosphate buffer solution(PBS) | HyClone | SH30256.01B | |
| Protease inhibitors | Coolaber | SL1086 | Cocktail (100×) |
| Secondary antibody | Bioss | Bs-0295G | Goat Anti-Rabbit IgG H&L/FITC |
| Spermidine | Coolaber | CS10431 | |
| Spermine | Coolaber | CS10441 | |
| Sucrose | Coolaber | CS10581 | |
| Tris-HCl | Biosharp | BS157 | |
| Triton X-100 | Beyotime | P0096 |
Referenzen
- Hatch, E. M., Fischer, A. H., Deerinck, T. J. Catastrophic nuclear envelope collapse in cancer cell micronuclei. Cell. 154, 47-60 (2013).
- Liu, S., Kwon, M., Mannino, N. Nuclear envelope assembly defects link mitotic errors to chromothripsis. Nature. 561, 551-555 (2018).
- Sommer, S., Buraczewska, I., Kruszewski, M. Micronucleus assay: The state of art, and future directions. Int J Mol Sci. 21 (4), 1534 (2020).
- MacDonald, K. M., et al. Antecedent chromatin organization determines cGAS recruitment to ruptured micronuclei. Nat. Commun. 14, 556 (2023).
- Abdisalaam, S., Mukherjee, S., Bhattacharya, S. NBS1-CtIP–mediated DNA end resection suppresses cGAS binding to micronuclei. Nucleic Acids Res. 50, 2681-2699 (2022).
- Li, L., Kim, H. S., Kwon, S. W. Inhibitory effects of saeu-jeot extract on NLRP3 inflammasome activation and radiation-induced micronucleus formation. Food Sci Nutr. 10 (6), 1921-1927 (2022).
- Qian, Q. Z., Cao, X., Ke, F. H., Shen, Effects of ionising radiation on micronucleus formation and chromosomal aberrations in Chinese radiation workers. Radiat Prot Dosimetry. 168 (2), 197-203 (2016).
- Rzeszowska-Wolny, J., Przybyszewski, W. M., Widel, M. Ionizing radiation-induced bystander effects, potential targets for modulation of radiotherapy. Eur J Pharmacol. 625 (1-3), 156-164 (2009).
- Zuo, Y. H., Dang, X. H., Zhang, H. F. Genomic instability induced by ionizing radiation in human hepatocytes. J Toxicol Environ Health A. 75 (12), 700-706 (2012).
- Eken, A., Aydin, A., Erdem, O. Cytogenetic analysis of peripheral blood lymphocytes of hospital staff occupationally exposed to low doses of ionizing radiation. Toxicol Ind Health. 26 (5), 273-280 (2010).
- Gutierrez-Enriquez, S., Ramon, Y. C. T., Alonso, C. Ionizing radiation or mitomycin-induced micronuclei in lymphocytes of BRCA1 or BRCA2 mutation carriers. Breast Cancer Res Treat. 127 (3), 611-622 (2011).
- Brzozowska, K., et al. Effect of temperature during irradiation on the level of micronuclei in human peripheral blood lymphocytes exposed to X-rays and neutrons. Int J Radiat Biol. 85 (10), 891-899 (2009).
- Yamini, K., Gopal, V. Natural radioprotective agents against ionizing radiation-an overview. Int J PharmTech Res. 2 (2), 1421-1426 (2010).
- Terradas, M., Martín, M., Genescà, A. Impaired nuclear functions in micronuclei results in genome instability and chromothripsis. Arch Toxicol. 90, 2657-2667 (2016).
- Crasta, K., Ganem, N. J., Dagher, R. DNA breaks and chromosome pulverization from errors in mitosis. Nature. 482, 53-58 (2012).
- Fenech, M., Morley, A. Measurement of micronuclei in lymphocytes. Mutat Res. 147, 29-36 (1985).
- Kirsch-Volders, M., Bonassi, S., Knasmueller, S. Commentary: Critical questions, misconceptions and a road map for improving the use of the lymphocyte cytokinesis-block micronucleus assay for in vivo biomonitoring of human exposure to genotoxic chemicals-a HUMN project perspective. Mutat Res Rev Mutat Res. 759, 49-58 (2014).
- Fenech, M., et al. Molecular mechanisms of micronucleus, nucleoplasmic bridge and nuclear bud formation in mammalian and human cells. Mutagenesis. 26, 125-132 (2011).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nat Protoc. 2, 1084-1104 (2007).
- Fenech, M. Cytokinesis-block micronucleus cytome assay evolution into a more comprehensive method to measure chromosomal instability. Genes (Basel). 11 (10), 1203 (2020).
- Kwon, M., Leibowitz, M. L., Lee, J. H. Small but mighty: The causes and consequences of micronucleus rupture. Exp Mol Med. 52 (11), 1777-1786 (2020).
- Harding, S. M., et al. Mitotic progression following DNA damage enables pattern recognition within micronuclei. Nature. 548, 466-470 (2017).
- Mackenzie, K. J., et al. cGAS surveillance of micronuclei links genome instability to innate immunity. Nature. 548, 461-465 (2017).
- Tang, S., Stokasimov, E., Cui, Y. Breakage of cytoplasmic chromosomes by pathological DNA base excision repair. Nature. 606, 930-936 (2022).
- Zhang, C. Z., et al. Chromothripsis from DNA damage in micronuclei. Nature. 522, 179-184 (2015).
- Umbreit, N. T., Zhang, C. Z. Z., Lynch, L. D. Mechanisms generating cancer genome complexity from a single cell division error. Science. 368, eaba0712 (2020).
- Li, Q. J., Wu, P., Du, Q. J. cGAS-STING, an important signaling pathway in diseases and their therapy. Med Comm. 5 (4), e511 (2024).
- Mohr, L., Toufektchan, E., von Morgen, P. ER-directed TREX1 limits cGAS activation at micronuclei. Mol Cell. 81, 724-738.e9 (2021).
- Okamoto, A., Utani, K., Shimizu, N. DNA replication occurs in all lamina positive micronuclei, but never in lamina negative micronuclei. Mutagenesis. 27, 323-327 (2012).
- Hatch, E. M., Fischer, A. H., Deerinck, T. J. Catastrophic nuclear envelope collapse in cancer cell micronuclei. Cell. 154, 47-60 (2013).
- Guo, X., Dai, X., Wu, X. Understanding the birth of rupture-prone and irreparable micronuclei. Chromosoma. 129 (3-4), 181-200 (2020).
- MacDonald, K. M., et al. The proteomic landscape of genotoxic stress-induced micronuclei. Molecular Cell. 84 (7), 1377-1391.e6 (2024).
- Agustinus, A. S., Al-Rawi, D., Dameracharla, B. Epigenetic dysregulation from chromosomal transit in micronuclei. Nature. 619 (7968), 176-183 (2023).
- Toufektchan, E., Maciejowski, J. Purification of micronuclei from cultured cells by flow cytometry. STAR Protoc. 2 (1), 100378 (2021).
- Shimizu, N., Kanda, T., Wahl, G. M. Selective capture of acentric fragments by micronuclei provides a rapid method for purifying extrachromosomally amplified DNA. Nat Genet. 12, 65 (1996).
- Samur, B. M., et al. Assessment of extracorporeal photopheresis related cell damage. Transfus Apher Sci. 61 (6), 103472 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten