Method Article
Quantitative Analyse von Vitamin-A-Metaboliten in okulärem und nicht-okulärem Gewebe von Mäusen mittels Hochleistungsflüssigkeitschromatographie
In diesem Artikel
Zusammenfassung
Hier wird ein Normalphasen-Hochleistungsflüssigkeitschromatographieverfahren beschrieben, um kritische Retinoide zu detektieren und zu quantifizieren, die an der Erleichterung der Sehfunktion sowohl im Augengewebe als auch im systemischen Gewebe beteiligt sind, im Rahmen der systemischen Vitamin-A-Versorgung zur Erzeugung des essentiellen lichtempfindlichen Rhodopsin-Chromophors 11-cis-Retinal.
Zusammenfassung
G-Protein-gekoppelte Rezeptoren (GPCRs) sind eine Superfamilie von Transmembranproteinen, die Signalkaskaden durch Aktivierung ihres G-Proteins bei Assoziation mit ihrem Liganden auslösen. In der Sicht aller Säugetiere ist Rhodopsin das GPCR, das für die Initiierung der Phototransduktionskaskade verantwortlich ist. Innerhalb der Photorezeptoren ist Rhodopsin an sein Chromophor 11-cis-Retinal gebunden und wird durch die lichtempfindliche Isomerisierung von 11-cis-Retinal zu all-trans-Retinal aktiviert, wodurch das Transducin-G-Protein aktiviert wird, was zur Phototransduktionskaskade führt.
Während die Phototransduktion gut verstanden ist, sind die Prozesse, die an der Versorgung mit Vitamin-A-Vorläufern in der Nahrung für die 11-cis-Retinal-Bildung im Auge beteiligt sind, sowie Krankheiten, die zu einer Störung dieser Versorgung führen, noch nicht vollständig verstanden. Sobald Vitamin-A-Vorläufer in den Darm aufgenommen wurden, werden sie in der Leber als Retinylester gespeichert und als All-trans-Retinol, das an das Retinol-bindende Protein 4 (RBP4) gebunden ist, in den Blutkreislauf abgegeben. Dieses zirkulatorische RBP4-Retinol wird von systemischen Organen wie Leber, Lunge, Niere und Auge aufgenommen. Daher ist eine Methode zur Quantifizierung der verschiedenen Metaboliten von Vitamin A in der Nahrung im Auge und in den systemischen Organen von entscheidender Bedeutung für die Untersuchung der ordnungsgemäßen Funktion von Rhodopsin GPCR.
In dieser Methode stellen wir eine umfassende Extraktions- und Analysemethode für die Vitamin-A-Analytik in murinem Gewebe vor. Durch die Normalphasen-Hochleistungsflüssigkeitschromatographie können alle relevanten Isomere von Retinaldehyden, Retinolen und Retinylestern gleichzeitig in einem einzigen Lauf nachgewiesen werden, was die effiziente Verwendung von Versuchsproben ermöglicht und die interne Zuverlässigkeit verschiedener Vitamin-A-Metaboliten innerhalb derselben Probe erhöht. Mit dieser umfassenden Methode werden die Forscher in der Lage sein, die systemische Vitamin-A-Versorgung in der GPCR-Funktion von Rhodopsin besser zu beurteilen.
Einleitung
G-Protein-gekoppelte Rezeptoren (GPCRs) sind eine der am besten untersuchten und charakterisierten Superfamilien von bekannten Proteinen. In ihrer bekanntesten Funktion dienen GPCRs als Zelloberflächenrezeptor bei der Signaltransduktion und initialisieren intrazelluläre Reaktionen bei der Bindung an einen bestimmten Liganden. GPCRs zeichnen sich durch sieben helikale Transmembrandomänen (TM) und sechs Gesamtschleifendomänen aus. Von den sechs Schleifen sind drei Schleifen extrazellulär orientiert, um die Ligandenbindung zu erleichtern, während die anderen drei intrazellulären Schleifen an ein heterotrimeres G-Protein gekoppelt sind, das aus den Untereinheiten Gα, Gβ und Gγbesteht 1,2.
GPCRs werden in mehrere Klassen eingeteilt, darunter Rhodopsin-ähnliche Klassen der Klasse A, Klasse-B-Secretin-Rezeptorfamilie, Klasse-C-Glutamat, Klasse-D-Pilzpaarungs-Pheromonrezeptoren, zyklische AMP-Rezeptoren der Klasse E und Klasse F gekräuselte/geglättete 3,4. Wie der Name schon sagt, umfasst die GPCR-Rhodopsin-ähnliche Unterklasse der Klasse A Rhodopsin, das kritische GPCR, das für die Phototransduktion und die Sehfunktion verantwortlich ist. Rhodopsin enthält alle relevanten Schlüsselmerkmale und Strukturelemente, die im kanonischen Modell der GPCRs zu finden sind, einschließlich der zuvor erwähnten sieben helikalen TM-Domänen, der sechs extrazellulären und intrazellulären Schleifen und der Assoziation mit einem heterotrimeren G-Protein, das auch als Transducin (G t) in den Photorezeptoren bekanntist 1,5,6,7. Innerhalb der Bindungstasche von Rhodopsin bindet 11-cis-Retinal, der lichtempfindliche Chromophor-Ligand, über eine kovalente Schiff-Basenbindung an Rhodopsin an Lysin 296 und bildet so 11-cis-Retinyliden 1,8. Bei der Absorption eines Photons wird 11-cis-Retinyliden zu all-trans-Retinyliden photoisomerisiert, wodurch eine Konformationsänderung innerhalb von Rhodopsin induziert wird. Daher ist der 11-cis-retinale Ligand entscheidend für die Funktion des Rhodopsin-GPCR, und eine robuste und effiziente Versorgung mit 11-cis-Retinal muss kontinuierlich aufrechterhalten werden, um die hohe Umsatzrate innerhalb der Photorezeptoren zu überwinden.
Retinaldehyde wie 11-cis-Retinal gehören zu einer Gruppe von Molekülen, die zusammen als Retinoide bezeichnet werden, und biologisch relevante Retinoide werden allgemein als Vitamin A bezeichnet. Retinoide zeichnen sich durch eine zyklische Endgruppe aus, die mit einer konjugierten Polyenkette verbunden ist, mit einer polaren Endgruppe am anderen Ende. Retinaldehyde und assoziierte Vitamere des Vitamin A sind keine Ausnahme von dieser Charakterisierung, die den β-Ionon-Ring als cyclische Endgruppe, eine Diterpen-Polyen-Kette und eine je nach Vitamer unterschiedliche polare Endgruppe enthalten, d.h. Aldehydgruppe für Retinaldehyde, Hydroxylgruppe für Retinole, Carboxylgruppe für Retinsäuren, Esterbindung für Retinylester, usw. (Abbildung 1)9,10.
Säugetiere können Vitamin A nicht de novo synthetisieren, Pflanzen jedoch schon; Daher müssen alle Retinoide in Säugetiersystemen aus der Ernährung pflanzlicher Produzenten bis zu den Verbrauchern in der Nahrungskette stammen. Im kanonischen Modell des Vitamin-A-Stoffwechsels wird β-Carotin, das archetypische pflanzliche Provitamin A, über den Scavenger-Rezeptor der Klasse B, Mitglied 1 (SCARB1) in die Darm-Enterozyten aufgenommen, das durch β-Carotin-Oxygenase 1 (BCO1/BCMO1) in zwei Moleküle von all-trans-Retinal gespalten wird, das an das Retinaldehyd-bindende Protein 2 (RBP2) bindet und zu all-trans reduziert wird-Retinol durch Retinol-Dehydrogenasen (RDH), umgewandelt durch Lecithin-Retinol-Acyltransferase (LRAT) in Retinylester und dann in den Blutkreislauf in Chylomikron 11,12,13,14. Retinylester, wie z.B. Retinylpalmitat, dienen hingegen als vorherrschendes Provitamin A aus tierischen Quellen. Retinylpalmitat aus dem Darmlumen wird durch die Carboxylesterase 1 (CES1) zu all-trans-Retinol hydrolysiert und diffundiert in den intestinalen Enterozyten15. Die Leber ist das primäre Speicher- und homöostatische Organ für die Vitamin-A-Homöostase, die die Retinylester in diesen Chylomikronen absorbiert, die durch Retinylester-Hydrolasen zu all-trans-Retinol hydrolysiert werden, das an das zelluläre Retinol-bindende Protein 1 (CRBP1) gebunden ist, in hepatische Sternzellen eindringt und von LRAT zur Lagerung wieder in Retinylester umgewandeltwird 13,16, 17. Urheberrecht Um einen homöostatischen Vitamin-A-Spiegel im Organismus aufrechtzuerhalten, setzt die Leber Vitamin A in Form von all-trans-Retinol frei, das an einen Serumtransportkomplex gebunden ist, der aus dem Retinol-bindenden Protein 4 (RBP4) und Transthyretin (TTR) besteht15,18,19. Dieser Komplex wird in diesem Manuskript als Holo-RBP4 bezeichnet.
Um diese systemische Vitamin-A-Versorgung im Blut nutzen zu können, muss systemisches Gewebe, einschließlich des Augengewebes, in dem eine robuste Vitamin-A-Quelle aufrechterhalten wird, über eine Methode zur Aufnahme von Holo-RBP4 in das Gewebe verfügen. In der photorezeptorreichen Netzhaut im Augengewebe ist der durch Retinsäure 6 (STRA6) stimulierte Membranrezeptor der Transporter, der an dieser Funktion beteiligt ist. In mechanistischen Studien wurde gezeigt, dass STRA6 in der Lage ist, die Aufnahme von extrazellulärem all-trans-Retinol aus Holo-RBP4 in das RPE20 zu erleichtern. Dieses importierte all-trans-Retinol tritt dann in den visuellen Zyklus ein, d. h. in den Prozess, durch den all-trans-Retinol innerhalb des RPE und des äußeren Photorezeptorsegments in 11-cis-Retinal umgewandelt wird, wodurch die Sehfunktion erleichtert wird, wenn es an Rhodopsin 9,21 gebunden ist.
Sobald all-trans-Retinol aus zirkulatorischem Holo-RBP4 über STRA6 die Blut-Netzhaut-Schranke in das RPE im Augengewebe überquert, wird all-trans-Retinol im RPE zunächst durch LRAT zu Retinylester verestert und dann durch das retinale Pigmentepithel-spezifische 65-kDa-Protein (RPE65) zu 11-cis-Retinol hydrolysiert. 11-cis-Retinol wird dann durch die Retinol-Dehydrogenase 5 in 11-cis-Retinal umgewandelt. Dieses 11-cis-Retinal wird dann durch das Interphotorezeptor-Retinoid-bindende Protein (IRBP) in das äußere Segment (OS) des Photorezeptors transportiert9,21. Im endoplasmatischen Retikulum, das den Photorezeptorkern innerhalb der äußeren Kernschicht (ONL) umgibt, werden die Opsin-GPCRs synthetisiert und über das verbindende Zilium (CC) transportiert. Die Motorproteine, die an diesem Transport durch das CC beteiligt sind, sind umstritten, aber aktuelle Hypothesen implizieren, dass der Intraflagellentransport (IFT) von Kinesin und Dynein oder der Myosin-basierte Transport wahrscheinlich die Ursache für diesen Prozess sind 14,22,23,24,25,26. Sobald diese beiden Komponenten in den membranösen Scheiben innerhalb des OS aufeinandertreffen, bilden 11-cis-Retinal und Opsin 11-cis-Retinyliden durch eine kovalente Bindung der Schiff-Base an Lysin 196 auf Rhodopsin, bereit für die Phototransduktion8.
Während die Expression von STRA6 im RPE der Netzhaut die Aufnahme von all-trans-Retinol aus Holo-RBP4 erleichtert, wurde festgestellt, dass STRA6 in der Leber nicht exprimiert wird, obwohl es als primäres homöostatisches Organ für Vitamin A fungiert und Fähigkeiten bei der Aufnahme von all-trans-Retinol aus Holo-RBP4 aufweist 15,19,27,28, 29,30,31. Schließlich wurde ein analoger Rezeptor namens Retinol-bindender Protein-4-Rezeptor 2 (RBPR2) entdeckt, der die Fähigkeit zeigt, all-trans-Retinol aus Holo-RBP4 aufzunehmen, ähnlich wie STRA6, aber im Lebergewebe exprimiertwird 32.
Daher erfordert ein vollständiges Verständnis der Rolle von Rhodopsin für die Sehfunktion ein Verständnis der biologischen Prozesse, die in der Regeneration des Sehpigments gipfeln. Dies steht wiederum in engem Zusammenhang mit den zuvor beschriebenen Prozessen, einschließlich des Metabolismus von Provitamin-A-Vorläufern, der Speicherung in der Leber, der Freisetzung von Holo-RBP4 durch die Leber und der eventuellen Aufnahme von Holo-RBP4 durch STRA6- und RBPR2-Membranrezeptoren. Wie oben erwähnt, sind Tiermodelle wie Mäuse nach wie vor eines der führenden Modelle bei der Erforschung solcher Prozesse. Daher möchten wir eine Extraktionsmethode für Retinoide in murinem Gewebe vorstellen, sowie eine Normalphasen-Hochleistungsflüssigkeitschromatographie (HPLC)-Methode, mit der diese Retinoide nachgewiesen und quantifiziert werden können. Mit diesen Methoden können die oben beschriebenen wichtigen Retinoide, wie z.B. der 11-cis-retinale Rhodopsin-Ligand oder das Haupttransportretinoid all-trans-Retinol, in okulären, hepatischen und systemischen Organen analysiert werden. Durch die Bewertung der Retinoidversorgung in murinem Gewebe kann unser Verständnis der Krankheitszustände und Pathologien im Zusammenhang mit der logistischen Versorgung mit Retinoiden weiter verbessert werden.
Neben ihrer Funktion als Chromophor in der visuellen Funktion durch Assoziation mit Opsin-GPCRs spielen Retinoide auch eine wichtige Rolle bei der Signalübertragung von Säugetierzellen durch Retinsäure-Signalwege, die durch zwei Familien von Kernrezeptoren erleichtert werden, Retinsäurerezeptoren (RARs) und Retinoid-X-Rezeptoren (RXRs), die direkt an die DNA binden und die Gentranskription regulieren33. Diese beiden Familien oder Rezeptoren verwenden beide Retinoide in Form von Retinsäuren als Liganden. Es wurde gezeigt, dass RARs eine Affinität sowohl für all-trans-Retinsäure als auch für 9-cis-Retinsäure aufweisen, während RXRs eine Affinität für nur 9-cis-Retinsäure aufweisen34,35. Retinsäuren in unkontrollierten Mengen sind teratogen, und die Retinsäure-Signalübertragung muss extrem streng kontrolliert werden36. Die Produktion von Retinsäuren für die Signalübertragung muss lokal und zu sehr spezifischen Zeitpunkten erfolgen, um die richtige Entwicklung von Geweben zu gewährleisten, wie z. B. bei der Entwicklung des Hinterhirns und der Gliedmaßen, aber unzählige andere Beispiele nutzen die Retinsäure-Signalgebung37,38. In Zellen, die an der Retinsäure-Signalübertragung beteiligt sind, werden Retinsäuren durch zwei Gruppen von Enzymen synthetisiert: Alkohol/Retinol-Dehydrogenasen (ADHs/RDHs), die die Oxidation von Retinolen, die von STRA6 oder RBPR2 aufgenommen werden, zu Retinaldehyden erleichtern, und Retinaldehyd-Dehydrogenasen (RALDHs), die die Oxidation von Retinaldehyden zu Retinsäuren erleichtern39. Obwohl Retinsäuren nicht an der GPCR-Signalübertragung an sich beteiligt sind, stellen sie dennoch ein wichtiges Retinoid dar, das auch als Ligand für Signalrezeptoren fungiert.
Auch wenn hier nicht näher beschrieben, möchten wir die bisher etablierten Methoden zum Retinoid-Nachweis mittels HPLC in verschiedenen Kontexten, wie z.B. in der Lebensmittelforschung und der Erforschung von mikrobiellem Rhodopsin, würdigen. Diese Methoden verfolgen unterschiedliche Ziele und Ansätze für den Retinoid-Nachweis, einschließlich der Verwendung von Umkehrphasentechniken, die weniger flüchtige und gefährliche mobile Phasen erfordern 40,41,42, des Nachweises von Retinsäuren und ihrer zugehörigen Isomere40,41 sowie der Reinigung und Extraktion aus verschiedenen biologischen Quellen43. Unsere Methode konzentriert sich speziell auf den Nachweis von Retinylpalmitat, Retinaldehyd-Isomeren und Retinol-Isomeren aus Säugetiergewebe. Unterschiedliche Protokolle sollten in Betracht gezogen werden, wenn sich der vorgesehene Anwendungsfall von dieser speziellen Anwendung unterscheidet.
Protokoll
HINWEIS: Alle Tierversuche wurden vom Institutional Animal Care and Use Committee (IACUC) der University of Minnesota (Protokoll # 2312-41637A) genehmigt und in Übereinstimmung mit der ARVO-Erklärung für die Verwendung von Tieren in der Augen- und Sehforschung durchgeführt. Führen Sie alle Extraktionen im Dunkeln unter einem schwachen roten Licht zur Beleuchtung durch. Achten Sie auf das Restlicht, das von Instrumentendisplays und Zubehör-LEDs abgegeben wird.
1. Erzeugung von spektrophotometrischen Retinoid-Standards und externen Standardkurven
HINWEIS: Bereiten Sie vor der Analyse mit der HPLC einen Trockeneisbehälter für die vorübergehende Lagerung von Retinoiden vor.
- Wiegen Sie eine beliebige, aber angemessene Menge an Retinoiden und lösen Sie sie in einem geeigneten Lösungsmittel auf, um sie durch Spektrophotometrie zu quantifizieren.
HINWEIS: Das in dieser Studie verwendete Lösungsmittel der Wahl ist Ethanol, und die molaren Absorptionswerte von in Ethanol gelösten Retinoiden sind in Tabelle 1 aufgeführt. Zum Auflösen von Standards und Proben muss wasserfreies Ethanol in HPLC-Qualität verwendet werden. Ethanol, das ausschließlich durch Destillation gereinigt wird, bildet ein azeotropes Gemisch mit Wasser, das etwa 4 Vol.-% Wasser enthält. Wasser ist nicht mischbar mit der hexanoischen mobilen Phase und führt zu einer Hydratation der stationären Phase der Kieselsäure, was schließlich zu einer Degradation der Säule führt. - Unter Verwendung des Beer-Lambert-Gesetzes und des molaren Absorptionsvermögens des entsprechenden Retinoids wird die Konzentration des erzeugten Standards quantifiziert (Tabelle 1).
Extinktion = molare Absorptionsfähigkeit (ε) × molare Konzentration × Weglänge - Führen Sie serielle Verdünnungen durch, um Konzentrationen zu erzeugen, die eine Standardkurve innerhalb des Quantifizierungsbereichs für das gewünschte Gewebe erzeugen können.
HINWEIS: Beachten Sie die grundlegenden Einschränkungen des Beer-Lambert-Gesetzes, wie z. B. seine mangelnde Gültigkeit bei hohen Konzentrationen von Analyt. Um dieses Problem zu vermeiden, sollten die verdünnten Retinoidstandards die Erzeugung von Absorptionswerten größer als 1 vermeiden. Für unsere Anwendungen in der Retinol- und Retinaldehyd-Quantifizierung in murinen Organen stellten wir fest, dass eine Kalibrierkurve mit einem Bereich von 1-10 ng in der Lage war, die typischen gefundenen Mengen abzudecken. Für die Quantifizierung von Retinylpalmitat aus der Leber von Mäusen fanden wir heraus, dass eine Kalibrierkurve mit einem Bereich von 20-80 μg in der Lage war, die typischen gefundenen Mengen abzudecken. - Durch inkrementelle Änderung des Injektionsvolumens aus der zuvor erstellten Stammlösung, wodurch die injizierte Menge an Retinoid schrittweise verändert wird, integrieren Sie die Peaks, um eine externe Standardkurve zu erzeugen, die für die Retinoid-Quantifizierung geeignet ist, wobei die Peakintegration direkt proportional zur Menge des injizierten Retinoids ist.
2. Gewebeentnahme und Probenentnahme
HINWEIS: Bereiten Sie einen Trockeneisbehälter für die vorübergehende Lagerung von Gewebe vor der Gewebehomogenisierung und Retinoidextraktion vor. Die empfohlenen Mengen an Gewebeentnahme sind in Tabelle 2 aufgeführt. Um Retinoidschwankungen aufgrund von Schwankungen des Blutgehalts für jedes Gewebe zu berücksichtigen, sollte die Gewebeextraktion an vollständig durchbluteten Mäusen und die Blutextraktion an separaten Mäusen durchgeführt werden.
- Euthanasieren Sie die Mäuse gemäß den Richtlinien des vom Institutional Animal Care and Use Committee (IACUC) diktierten Protokolls (hier CO 2-Erstickung).
- Blut: Enthaupten Sie die Mäuse unmittelbar nach der Euthanasie mit einer Schere und lassen Sie das Blut aus dem Hauptrumpf der Mäuse in ein 1,5-ml-Röhrchen ab.
- Auge: Entferne mit einer Pinzette die Augen vom enthaupteten Kopf.
- Gehirn: Schneide mit einer kleinen Schere in den enthaupteten Kopf und entferne das Gehirn mit einer Pinzette.
- Niere, Leber, Milz, Herz und Lunge: Machen Sie mit einer kleinen Schere einen Schnitt in den Bauch, schneiden Sie in die obere Richtung entlang der Mittellinie und schneiden Sie durch das Brustbein und den Brustkorb. Entferne das freiliegende Gewebe mit einer Pinzette.
3. Homogenisierung des Gewebes
HINWEIS: Wenn die Analyse kleinerer Organpartitionen gewünscht wird, z. B. in größeren Organen (z. B. Leber- oder Lungengewebe), sollte das gesamte Organ homogenisiert werden, um Unterschiede im Retinoidgehalt in verschiedenen Teilen des Gewebes zu vermeiden. Partitionieren Sie stattdessen das Homogenat, wenn kleinere Gewebemengen gewünscht sind. Ein Schema für das Protokoll ist in Abbildung 2 dargestellt. Dieses modifizierte Protokoll wurde von Kane und Napoliübernommen 44.
- Legen Sie das Gewebe zusammen mit 50 % eiskalter Kochsalzlösung (0,9 %) und 50 % Methanol in das Gefäßmahlröhrchen. In Tabelle 2 finden Sie das für jeden Gewebetyp verwendete Volumen.
- Setzen Sie den Stößel in das Mahlrohr ein und führen Sie langsam und vorsichtig fünf volle Umdrehungen mit dem Stößel durch, um ein Homogenat zu erhalten.
- Füllen Sie die Proben unmittelbar nach der Homogenisierung in 15-ml-Röhrchen um.
- Fügen Sie 2 mL Methanol hinzu und lassen Sie es 15 Minuten bei Raumtemperatur ruhen.
HINWEIS: Wenn eine Analyse von Retinaldehydoximderivaten gewünscht ist, fügen Sie 1 ml 0,1 M Hydroxylaminhydrochlorid in 0,1 M HEPES (pH 6,5) hinzu (Abbildung 1).
4. Retinoid-Extraktion
ACHTUNG: Hexan ist leicht entzündlich, leicht flüchtig und hochgiftig. Beim Umgang mit Hexan müssen vom National Institute for Occupational Safety and Health (NIOSH) zugelassene Atemschutzmasken, Augenschutz, Butylhandschuhe und ein Abzug verwendet werden. Beim Verdampfen von Hexan aus Proben wird eine Form von verstärkter Luftzirkulationsvorrichtung empfohlen, um die Bildung von Lösungsmitteldämpfen zu verhindern, z. B. eine Schnorchel-Saugvorrichtung.
- Geben Sie 10 mL Hexan zum Homogenat und mischen Sie das Röhrchen mindestens 10 s lang horizontal.
HINWEIS: Es ist wichtig, dass sich die Phasen vollständig mischen. - Das Homogenat/Hexan-Gemisch wird 3 Minuten lang bei 1.000 × g zentrifugiert, um die Phasentrennung zu erleichtern.
- Führen Sie die Extraktion 2x durch, um eine vollständige Extraktion der Retinoide aus dem Homogenat sicherzustellen. Wiederholen Sie die Schritte 4.1 und 4.2.
- Ziehen Sie die Hexanschicht mit einer Pipette ab und geben Sie die Hexanschicht zur Vakuumverdampfung in einen separaten Satz 15-ml-Glasröhrchen.
HINWEIS: Verwenden Sie zum Verdampfen GLASS 15 ml-Röhrchen, um ein Anhaften von Retinoiden an den Wänden des Röhrchens zu vermeiden. - Mit einer Vakuumzentrifuge das Hexan vollständig verdampfen.
5. Resuspensions- und HPLC-Analyse
HINWEIS: Da es sich bei dem in diesem Manuskript verwendeten HPLC-System um ein binäres Pumpensystem handelte, wurde die mobile Vierkomponenten-Phase vor der Operation in eine einzelne Flasche vorgemischt.
ACHTUNG: Alle vier organischen Lösungsmittel, die bei diesem Verfahren verwendet werden, sind leicht entzündlich, leicht flüchtig und hochgiftig. 1,4-Dioxan ist anfällig für die Bildung explosiver Peroxide, wenn es Sauerstoff ausgesetzt wird. Alle Gefäße, die 1,4-Dioxan enthalten, bei Nichtgebrauch geschlossen halten. Beim Umgang mit diesen Lösungsmitteln müssen vom National Institute for Occupational Safety and Health (NIOSH) zugelassene Atemschutzmasken, Augenschutz, Butylhandschuhe und ein Abzug verwendet werden. Während diese Lösungsmittel in einer HPLC betrieben werden, wird eine Form von verbesserter Luftzirkulation empfohlen, um die Ansammlung von Lösungsmitteldämpfen zu verhindern, z. B. ein Schnorchel-Sauggerät.
- Resuspendieren Sie das getrocknete 15-ml-Röhrchen mit 100 μl Hexan; Vortexen Sie gut, um sicherzustellen, dass alle Retinoide aufgelöst sind.
- Pipettieren Sie alle 100 μl Hexan in einen einzigen Glaseinsatz für die HPLC-Analyse.
- Aufbau des HPLC-Ablaufs (adaptiert von Landers und Olson45): mobile Phase: 85,4 % Hexan (v/v), 11,2 % Ethylacetat (v/v), 2 % Dioxan (v/v), 1,4 % 1-Octanol (v/v); Säule: zwei Säulen 4,6 mm ID x 250 nm, 5 μm, in Reihe geschaltet; ein mehrspaltiges Thermostat Temperatur: 25 °C; Injektionsvolumen: 100 μL; Durchflussrate: 1 mL/min; Laufzeit: 40 min. Verwenden Sie die Absorptionserkennung des UV-Spektrums; Lassen Sie die Option aktiviert, um ein UV-Spektrum von 200 nm bis 400 nm zu erfassen.
6. Peak-Identifizierung und -Integration
- Identifizieren Sie Peaks anhand der Retentionszeit und der UV-Spektren jedes Retinoids von Interesse, wie sie bei der Analyse von Retinoidstandards beobachtet wurden (Abbildung 3, Tabelle 3 und Tabelle 4).
- Unter Verwendung des chromatographischen Datensystems des gewählten HPLC-Systems werden die identifizierten Peaks integriert. Die Integration oder Fläche unter der Kurve ist direkt proportional zur Menge des Analyten. Referenzieren Sie die in Schritt 1 generierte externe Standardkurve, um den Analyten zu quantifizieren.
- Für die Analyse von Chromatogrammen, die aus biologischem Gewebe erzeugt werden, ist die manuelle Integration gegenüber der automatischen Integration zu verwenden, die von typischen chromatographischen Datensystemen angeboten wird, da bei solchen Proben häufig Variabilitäten bei Parametern wie der Retentionszeit beobachtet werden.
- Stellen Sie sicher, dass die Chromatogramme keine Unregelmäßigkeiten aufweisen, die auf Probleme während des Laufs hinweisen könnten, wie z. B. verrauschte Basislinien oder nicht-Gaußsche Peaks. Diese Probleme deuten auf Verunreinigungen in der HPLC oder Verschleiß an den Säulen hin und sollten für eine valide Analyse behoben werden.
Ergebnisse
Hier haben wir die oben beschriebene Methode zum Nachweis und zur Quantifizierung von Retinoiden in murinem okulärem und systemischem Gewebe verwendet und repräsentative Chromatogramme erstellt. Zusätzlich geben wir eine Zusammenfassung der typischen Retinoide, die in diesen Geweben nachgewiesen werden können.
Im Alter von 6 Monaten wurden die Mäuse durch CO2 -Erstickung eingeschläfert. Um den okulären Retinoidgehalt aufrechtzuerhalten, wurden die Mäuse vor der Euthanasation und Extraktion 2 Tage lang dunkeladaptiert. Zwei Augen, 0,2 g Leber und 75 μl Blut wurden für die Retinoidextraktion und die anschließende HPLC-Analyse entnommen. Für das Augengewebe wurde ein Auge ohne Zusatz von Hydroxylamin extrahiert, während das andere einer Hydroxylaminbehandlung unterzogen wurde. Für Leber und Blut war eine Hydroxylaminbehandlung nicht erforderlich, da Retinaldehyde in diesen Geweben normalerweise nicht nachgewiesen werden.
Im Chromatogramm des Mausauges, das nicht mit Hydroxylamin behandelt wurde, wurde 13-cis-Retinal, eines der beiden nicht-kanonischen Retinaldehyd-Isomere, identifiziert. Zusätzlich wurden sowohl 11-cis-Retinal als auch all-trans-Retinal, die beiden kanonischen Retinaldehyd-Isomere, identifiziert. In beiden Chromatogrammen wurden sowohl das visuelle Zyklus-Zwischenprodukt 11-cis-Retinol als auch der Hauptvitamin-A-Transport von all-trans-Retinol identifiziert (Abbildung 4A).
Im Chromatogramm des mit Hydroxylamin behandelten Mausauges waren noch alle zuvor erwähnten Retinaldehyd-Isomere vorhanden. Die Retentionszeiten für diese Isomere wurden jedoch deutlich erhöht. Darüber hinaus liegen diese Retinaldehyd-Isomere jetzt sowohl als syn- als auch als anti-Isomere vor. Bei der Quantifizierung durch Peak-Integration müssen sowohl die syn- als auch die anti-Peaks summiert werden, um einen genauen Integrationswert zu erhalten. Sowohl 11-cis-Retinol als auch all-trans-Retinol waren noch vorhanden (Abbildung 4B).
Im Chromatogramm von Lebergewebe von Mäusen wurden Retinylpalmitat und all-trans-Retinol in diesen Geweben identifiziert. Retinylpalmitat, ein Retinylester, dient als Hauptspeicherform von Vitamin A bei Säugetieren und kommt in erheblichen Mengen im Lebergewebe von Säugetieren vor. Die Leber gibt Vitamin A in systemisches Gewebe als Holo-RBP4 ab, das all-trans-Retinol innerhalb des RBP4-Transthryetin-Komplexes enthält. Anschließend kann in diesem Chromatogramm ein großer all-trans-Retinol-Peak identifiziert werden (Abbildung 4C).
Im Chromatogramm des Blutes von Mäusen wurde ein großer all-trans-Retinol-Peak identifiziert. Angesichts der Tatsache, dass Holo-RBP4 aus der Leber über das Kreislaufsystem an systemische Gewebe abgegeben wird, ist dies zu erwarten (Abbildung 4D). Die charakteristische UV-Spektrum-Absorption (~250-400 nm) der Retinoid-Isoformen auf den zeitabhängig aufgelösten HPLC-Peaks kann hilfreich sein, um die Qualität des Vorhandenseins der Isoform in einem bestimmten jeweiligen Peak zunächst zu bestätigen (Abbildung 5).

Abbildung 1: Chemische Strukturen von Retinoiden. (A) Diese Retinoide kommen typischerweise in extrahiertem Mausgewebe vor. Beachten Sie die unterschiedlichen polaren Endgruppen sowie die unterschiedlichen Positionen der cis-Doppelbindungen in Retinoid-Isomeren. (B) Die Umwandlung von Retinaldehyden in Netzhautoxime erhöht die Extraktionseffizienz, verhindert die Co-Elution von Peaks mit Retinylpalmitat sowie die Co-Elution innerhalb von Retinaldehyd-Isomeren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Arbeitsablauf für Gewebeentnahme, Homogenisierung, Retinoidextraktion und Analyse. Netzhaut- und Gewebeproben von dunkeladaptierten Mäusen wurden homogenisiert und extrahiert. Schritt 1: Nach der Euthanasie von Mäusen gemäß den Richtlinien werden Blut und verschiedene Gewebe entnommen. Schritte 2-4: Die Gewebehomogenisierung erfolgt dann mit Kochsalzlösung und Methanol, mit Hydroxylaminhydrochlorid für quantitative Analysen. Schritte 5-7: Retinoide werden mit Hexan behandelt, gefolgt von einer Verdampfung. Schritte 8,9: Resuspendiert mit 100 μl Hexan, um sicherzustellen, dass alle Retinoide aufgelöst sind. Schritt 10: Abschließend werden die Proben mittels HPLC analysiert. Abkürzung: HPLC = High-performance Liquid Chromatography. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Chromatogramm von Retinoidstandards und Hydroxylamin-behandelten Retinoidstandards. (A) Dieses Chromatogramm enthält Retinylpalmitat, 13-cis-Retinal, 11-cis-Retinal, all-trans-Retinal, 11-cis-Retinol, 13-cis-Retinol und all-trans-Retinol-Standards. (B) Dieses Chromatogramm enthält Retinylpalmitat, 13-cis-Retinal, 11-cis-Retinal, all-trans-Retinal, 11-cis-Retinol, 13-cis-Retinol und all-trans-Retinol-Standards. Dieses Chromatogramm enthält neben den Retinol-Isomeren auch die Syn- und Anti-Oxim-Retinaldehyd-Isomere. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Repräsentative Chromatogramme. (A) Repräsentatives Chromatogramm des Augengewebes der Maus. Dieses Chromatogramm enthält neben Retinol-Isomeren auch Retinoide, die typischerweise im Augengewebe von Mäusen vorkommen, einschließlich Retinaldehyd-Isomere. (B) Repräsentatives Chromatogramm von mit Hydroxylamin behandeltem Augengewebe von Mäusen. Es ist zu beachten, dass dieses Chromatogramm anstelle der nicht chemisch modifizierten Retinaldehyde, die in den vorherigen Chromatogrammen (Abbildung 4A) gefunden wurden, zusätzlich zu den Retinol-Isomeren die Syn- und Anti-Oxim-Retinaldeyde-Isomere enthält. (C) Repräsentatives Chromatogramm von Lebergewebe der Maus. Dieses Chromatogramm enthält Retinoide, die typischerweise im Lebergewebe von Mäusen vorkommen, einschließlich Retinylpalmitat und All-trans-Retinol. (D) Repräsentatives Chromatogramm von Mausblut. Dieses Chromatogramm enthält Retinoide, die typischerweise im Blut von Mäusen vorkommen, einschließlich all-trans-Retinol. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
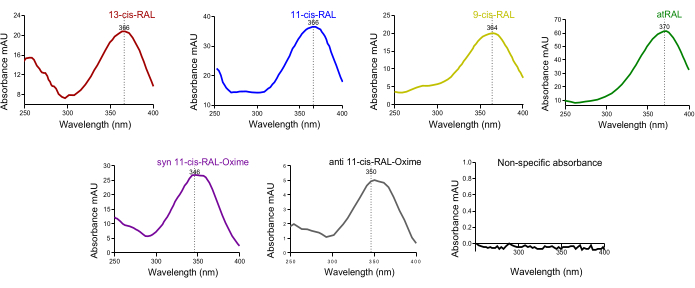
Abbildung 5: UV-Absorptionsspektrum von Retinaldehyd-Isomeren aus murinem Augengewebe. Absorptionsspektren von (A) 13-cis-Retinal, (B) 11-cis-Retinal, (C) 9-cis-Retinal, (D) all-trans-Retinal, (E) syn 11-cis-Retinaloxim, (F) Anti-11-cis-Retinaloxim. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Überblick über HPLC-Module, mobile Phasenströmung und andere Instrumente. Der mobile Phasenfluss beginnt bei den vorbereiteten Lösungsmittelflaschen und wird von der binären Pumpe mit hohem Druck in den Autosampler gepumpt, wo der Analyt in den Lösungsmittelstrom eingearbeitet wird. Die bewegliche Phase und der Analyt gelangen in das temperaturgeregelte Säulenfach und schließlich zur Messung in den Diodenarray-Detektor. (A) Rot getönter Monitorbildschirm zur Minimierung der Photoisomerisierung von Retinoiden. (B) Binäre Pumpe. (C) Probennehmer für Fläschchen. (D) Mehrsäulen-Thermostat. (E) Diodenarray-Detektor. (F) Analytischer Fraktionssammler. (G) Abfallbehälter aus Poly mit hoher Dichte. (H) Von links nach rechts, Bördelkappe aus Aluminium, 250-μl-Einsatz mit Polymerfüßen, gelbbraun getöntes Bördelfläschchen mit Bördelaufsatz, Endmontage des Injektionsfläschchens. (I) Links, gekühlte Zentrifuge. Rechts, Zentrifugenkonzentrator. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Verbindung | Molares Absorptionsvermögen (ε) | Lösungsmittel | Referenz |
| Retinylpalmitat | 49260 | Ethanol | Hubbard et al.49 |
| 13-cis-Retinal | 35500 | Ethanol | Hubbard et al.49 |
| 11-cis-Netzhaut | 24935 | Ethanol | Hubbard et al.49 |
| 9-cis-Retinal | 36100 | Ethanol | Robeson et al.51 |
| All-trans-Retinal | 42880 | Ethanol | Hubbard et al.49 |
| 11-cis-Retinol | 34890 | Ethanol | Hubbard et al.49 |
| 13-cis-Retinol | 48305 | Ethanol | Robeson et al.50 und Robeson et al.51 |
| All-Trans-Retinol | 52770 | Ethanol | Hubbard et al.49 |
Tabelle 1: Molares Absorptionsvermögen von Retinoiden in Ethanol. Die molaren Absorptionswerte von Retinoiden wurden von Kane und Napoli44, Hubbard et al.46, Robeson et al.47 und Robeson et al.48 erhoben. Das molare Absorptionsvermögen hängt vom Lösungsmittel ab. Die hier aufgeführten Werte sind spezifisch für in Ethanol gelöste Retinoide.
| Gewebe | Menge der Gewebeentnahme | Volumen für die Homogenisierung |
| Blut | 75 μL | N/A |
| Leber | 0,2 g | 2 mL |
| Niere | 2 ganze Nieren | 2 mL |
| Haut | 0,2 g | 1 mL – 1,5 mL |
| Milz | 1 ganze Milz | 1 mL |
| Auge | Beide Retina, Pool 4 Retina | 1 mL |
| Gehirn | 1 ganzes Gehirn | 2 mL |
| Herz | 1 Ganzes Herz | 1 mL |
| Lunge | 0,2 g | 2 mL |
Tabelle 2: Empfohlene Gewebemenge pro Analyse. Gewebemenge pro Analyse und Volumen von 50 % eiskalter Kochsalzlösung (0,9 %) und 50 % Methanol für die Gewebehomogenisierung, wie von Kane und Napoliempfohlen 44.
| Verbindung | Aufbewahrungszeit (min) | Maximale UV-Absorption (nm) |
| Retinylpalmitat | 5.177 | 326 |
| 13-cis-Retinal | 7.268 | 366 |
| 11-cis-Netzhaut | 7.691 | 366 |
| 9-cis-Retinal | 7.931 | 364 |
| All-trans-Retinal | 8.993 | 370 |
| 11-cis-Retinol | 17.003 | 318 |
| 13-cis-Retinol | 17.83 | 328 |
| All-Trans-Retinol | 23.933 | 326 |
Tabelle 3: Retentionszeiten und maximale Absorptionswellenlängen von Retinoiden. Diese Werte wurden bei Verwendung mit dem beschriebenen Verfahren und der mobilen Phase beobachtet, wie sie mit dem referenzierten Diodenarray-Detektor detektiert wurden.
| Verbindung | Aufbewahrungszeit (min) | Maximale UV-Absorption (nm) |
| Syn 11-cis-Retinaloxim | 9.59 | 346 |
| Syn All-trans-Retinales Oxim | 10.6 | 356 |
| Syn 13-cis-Retinaloxim | 11.3 | 352 |
| Anti-13-cis-Retinaloxim | 13 | 356 |
| Anti-11-cis-Retinaloxim | 15.1 | 350 |
| Anti All-trans-Retinales Oxim | 19.7 | 360 |
Tabelle 4: Retentionszeiten und maximale Absorptionswellenlängen von Hydroxylamin-behandelten Retinaldehyden, was zu Syn- und Anti-Oxim-Isomeren jedes Retinaldehyds führt. Diese Werte wurden bei Verwendung mit dem beschriebenen Verfahren und der mobilen Phase beobachtet, wie sie mit dem referenzierten Diodenarray-Detektor detektiert wurden.
Diskussion
Bei dieser Methode wird die Normalphasen-HPLC verwendet, um relevante Retinoide, einschließlich Retinylester, Retinaldehyde und Retinole, nachzuweisen und zu quantifizieren. Angesichts der Bedeutung von 11-cis-Retinal als kritischem Chromophor bei der Aktivierung des Rhodopsin-GPCR ist eine Methode, die die Metaboliten nachweisen kann, die mit der Produktion von 11-cis-Retinal in Verbindung stehen, entscheidend für die Untersuchung der gesamten Sehfunktion. Der Hauptvorteil dieser Methode besteht darin, dass alle relevanten Isomere sowohl von Retinaldehyden als auch von Retinolen gleichzeitig mit einem einzigen Lauf nachgewiesen und quantifiziert werden können. Angesichts der Tatsache, dass lebensfähige experimentelle Gewebe knapp sind, ermöglicht diese Methode einen effizienten Einsatz von Gewebe für den Retinoid-Nachweis. Während bei der Trennung von Retinoiden über die Verwendung von mobilen Phasen auf Hexan- und Ethylacetatbasis berichtet wurde49, verbessert die Zugabe von 1,4-Dioxan und 1-Octanol die Auflösung von Retinoid-Isomeren. 1,4-Dioxan ist für die Auflösung von 13-cis-Retinol und 11-cis-Retinol50 notwendig, während langkettige Alkohole wie 1-Octanol die Trennung von Retinol-Isomeren weiter verbessern 45,51,52.
Für die Quantifizierung von Retinaldehyd-Isomeren ist eine vorherige Umwandlung in die entsprechenden Retinaldehyd-Oxime mit Hydroxylamin erforderlich, da die reaktive Aldehydgruppe dazu neigt, mit anderen Biomolekülen wie Proteinen und Lipiden zu reagieren 45,53,54. Retinaldehydoxime werden leichter extrahiert und bewirken, dass Retinaldehyd-Peaks später im Lauf eluieren, wodurch eine Peak-Co-Elution mit der unbekannten Gruppe von Peaks und dem Retinylpalmitat-Peak, der bei ~5-8 min für biologisches Gewebe eluiert, verhindert wird. Die Behandlung von Retinaldehyden mit Hydroxylamin verhindert auch die Co-Elution von Retinaldehyden mit ähnlichen Elutionszeiten45. Bei der Umwandlung in Retinaldehydoxime werden sowohl syn- als auch anti-Isomere erzeugt, die zu zwei verschiedenen Zeitpunkten mit zwei unterschiedlichen Peaks für jedes Retinaldehyd-Isomer eluieren. Die Integration für beide Peaks muss summiert werden, um jedes Retinaldehyd-Isomer richtig zu quantifizieren. Neben der Retentionszeit bieten die UV-Spektren eine weitere Metrik, die bei der Identifizierung von Peaks verwendet werden kann. Jedes Retinoid weist ein leicht unterschiedliches UV-Absorptionsmaximum auf. Wenn diese beiden Metriken zusammen mit der Retentionszeit berücksichtigt werden, ermöglichen sie eine genaue Identifizierung von Peaks (Tabelle 3 und Tabelle 4).
Während das Vorhandensein von kanonischen Retinaldehyd-Isomeren wie 11-cis-Retinal und all-trans-Retinal im Augengewebe erwartet wird, wobei 11-cis-Retinal das kritische Rhodopsin-Liganden-Chromophor und all-trans-Retinal der photoisomerisierte Ligand ist, werden andere nicht-kanonische Retinaldehyd-Isomere mit diesem Verfahren nachgewiesen. Insbesondere konnte 13-cis-Retinal in HPLC-Läufen von murinem Augengewebe während unserer Analyse leicht nachgewiesen werden (Abbildung 4A-D). Das Auftreten dieser nicht-kanonischen Retinaldehyde ist jedoch nicht völlig unerwartet, da sowohl 13-cis-Retinal als auch 9-cis-Retinal bekannte Nebenprodukte sind, die aus lichtinduzierter Belastung der Netzhaut resultieren55,56. Diese Retinaldehyd-Isomere sind nicht am bekannten kanonischen Phototransduktionskaskadenweg beteiligt und ihr metabolisches Schicksal ist relativ unbekannt. Es wurde jedoch gezeigt, dass 9-cis-Retinal auch stabil an die Bindungstasche von Rhodopsin bindet und Isorhodopsin bildet. Die Rolle von Isorhodopsin muss noch vollständig charakterisiert werden, aber Studien haben gezeigt, dass Isorhodopsin ähnlich wie das 11-cis-Retinal-basierte Rhodopsin57,58 lichtempfindliche Fähigkeiten aufweist. Zumindest bietet die Fähigkeit von 9-cis-Retinal, stabil Isorhodopsin zu bilden und zu all-trans-Retinal zu photoisomerisieren, eine vernünftige Erklärung für sein Fehlen in unserer HPLC-Analyse56. Andererseits wurde nicht gezeigt, dass 13-cis-Retinal an einem bekannten biochemischen Signalweg des Auges beteiligt ist, und seine Akkumulation im Augengewebe könnte zu oxidativem Stress in der Netzhaut führen 55,59,60.
Obwohl 9-cis-Retinal typischerweise nicht in murinem Gewebe vorkommt, sollte erwähnt werden, dass die mit Hydroxylamin behandelte Methode keine praktikable Methode zur Trennung und Quantifizierung von 9-cis-Retinal ist. Die Peaks für syn 9-cis-retinal und syn 13-cis-retinal koelutieren nach 11,2 min. In Fällen, in denen signifikante Mengen an 9-cis-Retinal zu erwarten sind, z. B. wenn es exogen angewendet wird, ist nur die Quantifizierung von kombiniertem 9-cis-Retinal und 13-cis-Retinal möglich. Zur qualitativen Identifizierung ermöglicht die Durchführung der Methode ohne Behandlung des Gewebes mit Hydroxylamin die vollständige Trennung von 13-cis-retinalen und 9-cis-retinalen Peaks.
Neben dem Augengewebe kann diese Methode auch problemlos auf systemisches Gewebe angewendet werden. Während Retinaldehyde in ihren verschiedenen isomeren Formen typischerweise nicht in systemischem Gewebe vorkommen, ist all-trans-Retinol, das an das Retinol-bindende Protein 4 (Holo-RBP4) gebunden ist, die Haupttransportform von Vitamin A in Säugetierorganismen, und all-trans-Retinol kann mit dieser Methode leicht im Blut nachgewiesen werden. Neben dem Blut kann all-trans-Retinol typischerweise auch in allen systemischen Geweben nachgewiesen werden. Daher kann mit dieser Methode ein vollständiges systemisches gewebeweites Retinoidprofil erstellt werden.
Da Retinaldehyde, insbesondere der kritische Rhodopsin-Ligand 11-cis-Retinal, extrem anfällig für Photoisomerisierung sind, ist es von entscheidender Bedeutung, dass vorbeugende Maßnahmen ergriffen werden, um eine versehentliche Lichtexposition während eines der oben beschriebenen Schritte zu verhindern. Neben den offensichtlichen Quellen aus der Raumbeleuchtung und den Instrumententafeln haben wir festgestellt, dass fehlerhaftes Licht von Zubehör-LEDs in verschiedenen Instrumenten, wie z. B. hinter Instrumenten, einschließlich der HPLC selbst, die Hauptquellen für versehentliche Lichtexposition sind (Abbildung 6). Darüber hinaus haben wir festgestellt, dass eine spezielle Dunkelkammer für den ordnungsgemäßen Umgang mit Retinaldehyd-Isomeren und experimentellen Geweben von entscheidender Bedeutung ist. Neben der Lichtminimierung empfehlen wir, das Gewebe nach der Euthanasie und der Entnahme schnell und zeitnah zu verarbeiten, da der Abbau von Retinaldehyd-Isomeren auch in Abwesenheit von Licht erfolgt. Im Idealfall sollten die Gewebeentnahme, die Extraktion und die HPLC-Analyse am selben Tag abgeschlossen sein.
Gewebe, das nicht sofort verarbeitet wird, muss im Dunkeln bei -80 °C gelagert werden und sollte vor der Verarbeitung nicht länger als eine Woche gelagert werden. Wie bereits erwähnt, sind alle Lösungsmittel, die in der mobilen Phase und in den Extraktionsschritten des oben genannten Verfahrens verwendet werden, hochgiftig, flüchtig und brennbar. Beim Umgang mit diesen gefährlichen Lösungsmitteln müssen geeignete Verfahren und Geräte verwendet werden. Vor dem Umgang mit Lösungsmitteln muss geeignete persönliche Schutzausrüstung (PSA) wie Butylhandschuhe, NIOSH-zugelassene Atemschutzmasken und Augenschutz angelegt werden, und alle Lösungsmitteltransfers müssen in einem Abzug erfolgen. Beachten Sie, dass 1,4-Dioxan anfällig für die Bildung explosiver Peroxide ist, wenn es Sauerstoff ausgesetzt wird, und bewahren Sie alle Lösungsmittelbehälter mit 1,4-Dioxan dicht von der Luft entfernt auf.
Wenn eine Normalphasen-HPLC-Methode aufgrund unzureichender Materialien oder Geräte nicht praktikabel ist, gibt es typischere Umkehrphasenmethoden mit weniger flüchtigen Lösungsmitteln wie Acetonitril und Wasser zum Nachweis und zur Quantifizierung von Retinaldehyden und Retinolen. Diese Verfahren sind jedoch nicht in der Lage, Isomere zu trennen und können nur Gesamtretinaldehyde oder Gesamtretinole nachweisen44,54.
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Diese Arbeit wurde durch NIH-NEI-Zuschüsse (EY030889 und 3R01EY030889-03S1) und teilweise durch die Startkapitalhilfe der University of Minnesota für G.P.L. unterstützt. Wir möchten uns auch beim National Eye Institute für die Bereitstellung des in diesem Manuskript verwendeten 11-cis-Retinal-Standards bedanken.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagent | |||
| 1-Octanol, suitable for HPLC, ≥99.5% | Sigma-Aldrich, Millipore Sigma | 203-917-6 | |
| 1,4-Dioxane, suitable for HPLC, ≥99.5% | Sigma-Aldrich, Millipore Sigma | 204-661-8 | |
| 11-cis-retinal | National Eye Institute | N/A | |
| 11-cis-Retinol | Toronto Research Chemicals | TRC-R252105 | |
| 13-cis-retinal | Toronto Research Chemicals | TRC-R239900 | |
| 13-cis-retinol | Toronto Research Chemicals | TRC-R252110 | |
| All-trans-Retinal | Toronto Research Chemicals | TRC-R240000 | |
| All-trans-Retinol | Toronto Research Chemicals | TRC-R252002 | |
| Ethyl Acetate, suitable for HPLC, ≥99.7% | Sigma-Aldrich, Millipore Sigma | 205-500-4 | |
| Hexane, HPLC Grade | Fisher Scientific, Spectrum Chemical | 18-610-808 | |
| Methanol (HPLC) | Fisher Scienctific | A452SK-4 | |
| Retinyl Palmitate | Toronto Research Chemicals | TRC-R275450 | |
| Sodium Chloride (Crystalline/Certified ACS) | Fisher Scientific | S271-500 | |
| Instruments | |||
| 1260 Infinity II Analytical Fraction Collector | Agilent | G1364F | |
| 1260 Infinity II Binary Pump | Agilent | G7112B | |
| 1260 Infinity II Diode Array Detector | Agilent | G7115A | |
| 1260 Infinity II Multicolumn Thermostat | Agilent | G7116A | |
| 1260 Infinity II Vialsampler | Agilent | G7129A | |
| ST40R Refrigerated Centrifuge | Thermo Scientific | TSST40R | |
| Vacufuge Plus Centrifuge Concentrator | Eppendorf | 22820168 | |
| Consumables | |||
| 2 mL Amber Screw Top Vials | Agilent | 5188-6535 | |
| Crimp Cap with PTFE/red rubber septa, 11 mm | Agilent | 5183-4498 | |
| Disposable Glass Conical Centrifuge Tubes | Millipore Sigma | CLS9950215 | |
| Screw cap tube, 15 mL | Sarstedt | 62.554.502 | |
| Vial insert, 150 µL, glass with polymer feet | Agilent | 5183-2088 |
Referenzen
- Palczewski, K., et al. Crystal structure of rhodopsin: A G protein-coupled receptor. Science. 289 (5480), 739-745 (2000).
- Rosenbaum, D. M., Rasmussen, S. G. F., Kobilka, B. K. The structure and function of G-protein-coupled receptors. Nature. 459 (7245), 356-363 (2009).
- Alhosaini, K., Azhar, A., Alonazi, A., Al-Zoghaibi, F. GPCRs: The most promiscuous druggable receptor of the mankind. Saudi Pharm J. 29 (6), 539-551 (2021).
- Hu, G. -. M., Mai, T. -. L., Chen, C. -. M. Visualizing the GPCR network: Classification and evolution. Sci Rep. 7 (1), 15495 (2017).
- Lerea, C. L., Somers, D. E., Hurley, J. B., Klock, I. B., Bunt-Milam, A. H. Identification of specific transducin α subunits in retinal rod and cone photoreceptors. Science. 234 (4772), 77-80 (1986).
- Gao, Y., Hu, H., Ramachandran, S., Erickson, J. W., Cerione, R. A., Skiniotis, G. Structures of the rhodopsin-transducin complex: Insights into G protein activation. Mol Cell. 75 (4), 781-790.e3 (2019).
- Zhou, X. E., Melcher, K., Xu, H. E. Structure and activation of rhodopsin. Acta Pharmacol Sin. 33 (3), 291-299 (2012).
- Robinson, P. R., Cohen, G. B., Zhukovsky, E. A., Oprian, D. D. Constitutively active mutants of rhodopsin. Neuron. 9 (4), 719-725 (1992).
- Kiser, P. D., Golczak, M., Palczewski, K. Chemistry of the retinoid (visual) cycle. Chem Rev. 114 (1), 194-232 (2014).
- Sani, B. P., Hill, D. L. [3] Structural characteristics of synthetic retinoids. Methods Enzymol. 189, 43-50 (1990).
- Lobo, G. P., Amengual, J., Palczewski, G., Babino, D., von Lintig, J. Carotenoid-oxygenases: Key players for carotenoid function and homeostasis in mammalian biology. Biochim Biophys Acta. 1821 (1), 78-87 (2012).
- Amengual, J., et al. Two carotenoid oxygenases contribute to mammalian provitamin A metabolism. J Biol Chem. 288 (47), 34081-34096 (2013).
- Harrison, E. H. Mechanisms involved in the intestinal absorption of dietary vitamin A and provitamin A carotenoids. Biochim Biophys Acta. 1821 (1), 70-77 (2012).
- Leung, M., et al. The logistical backbone of photoreceptor cell function: Complementary mechanisms of dietary vitamin A receptors and rhodopsin transporters. Int J Mol Sci. 25 (8), 4278 (2024).
- Martin Ask, N., Leung, M., Radhakrishnan, R., Lobo, G. P. Vitamin A transporters in visual function: A mini review on membrane receptors for dietary vitamin A uptake, storage, and transport to the eye. Nutrients. 13 (11), 3987 (2021).
- Harrison, E. H. Carotenoids, β-apocarotenoids, and retinoids: The long and the short of it. Nutrients. 14 (7), 1411 (2022).
- Li, Y., Wongsiriroj, N., Blaner, W. S. The multifaceted nature of retinoid transport and metabolism. Hepatobiliary Surg Nutr. 3 (3), 126-139 (2014).
- D'Ambrosio, D. N., Clugston, R. D., Blaner, W. S. Vitamin A metabolism: An update. Nutrients. 3 (1), 63-103 (2011).
- Yamamoto, Y., et al. Interactions of transthyretin (TTR) and retinol-binding protein (RBP) in the uptake of retinol by primary rat hepatocytes. Exp Cell Res. 234 (2), 373-378 (1997).
- Kawaguchi, R., et al. A membrane receptor for retinol binding protein mediates cellular uptake of vitamin A. Science. 315 (5813), 820-825 (2007).
- Kiser, P. D., Golczak, M., Maeda, A., Palczewski, K. Key enzymes of the retinoid (visual) cycle in vertebrate retina. Biochim Biophys Acta. 1821 (1), 137-151 (2012).
- Radhakrishnan, R., et al. The role of motor proteins in photoreceptor protein transport and visual function. Ophthalmic Genet. 43 (3), 285-300 (2022).
- Solanki, A. K., et al. Loss of motor protein MYO1C causes rhodopsin mislocalization and results in impaired visual function. Cells. 10 (6), 1322 (2021).
- Liu, X., Udovichenko, I. P., Brown, S. D. M., Steel, K. P., Williams, D. S. Myosin VIIa participates in opsin transport through the photoreceptor cilium. J Neurosci. 19 (15), 6267-6274 (1999).
- Insinna, C., Besharse, J. C. Intraflagellar transport and the sensory outer segment of vertebrate photoreceptors. Dev Dyn. 237 (8), 1982-1992 (2008).
- Krock, B. L., Mills-Henry, I., Perkins, B. D. Retrograde intraflagellar transport by cytoplasmic dynein-2 is required for outer segment extension in vertebrate photoreceptors but not arrestin translocation. Invest Ophthalmol Vis Sci. 50 (11), 5463-5471 (2009).
- Blaner, W. S. STRA6, a cell-surface receptor for retinol-binding protein: The plot thickens. Cell Metab. 5 (3), 164-166 (2007).
- Bouillet, P., et al. Developmental expression pattern of Stra6, a retinoic acid-responsive gene encoding a new type of membrane protein. Mech Dev. 63 (2), 173-186 (1997).
- Blomhoff, R., Norum, K. R., Berg, T. Hepatic uptake of [3H]retinol bound to the serum retinol binding protein involves both parenchymal and perisinusoidal stellate cells. J Biol Chem. 260 (25), 13571-13575 (1985).
- Kelly, M., von Lintig, J. STRA6: role in cellular retinol uptake and efflux. Hepatobiliary Surg Nutr. 4 (4), 229-242 (2015).
- Quadro, L., et al. The role of extrahepatic retinol binding protein in the mobilization of retinoid stores. J. Lipid Res. 45 (11), 1975-1982 (2004).
- Alapatt, P., et al. Liver retinol transporter and receptor for serum retinol-binding protein (RBP4). J Biol Chem. 288 (2), 1250-1265 (2013).
- Chawla, A., Repa, J. J., Evans, R. M., Mangelsdorf, D. J. Nuclear receptors and lipid physiology: Opening the X-files. Science. 294 (5548), 1866-1870 (2001).
- Heyman, R. A., et al. 9-cis retinoic acid is a high affinity ligand for the retinoid X receptor. Cell. 68 (2), 397-406 (1992).
- Allenby, G., et al. Retinoic acid receptors and retinoid X receptors: interactions with endogenous retinoic acids. Proc Natl Acad Sci USA. 90 (1), 30-34 (1993).
- Das, B. C., et al. Retinoic acid signaling pathways in development and diseases. Bioorg Med Chem. 22 (2), 673-683 (2014).
- Hernandez, R. E., Putzke, A. P., Myers, J. P., Margaretha, L., Moens, C. B. Cyp26 enzymes generate the retinoic acid response pattern necessary for hindbrain development. Development. 134 (1), 177-187 (2007).
- Yashiro, K., et al. Regulation of retinoic acid distribution is required for proximodistal patterning and outgrowth of the developing mouse limb. Dev Cell. 6 (3), 411-422 (2004).
- Duester, G. Families of retinoid dehydrogenases regulating vitamin A function. Eur J Biochem. 267 (14), 4315-4324 (2000).
- Tatum, V., Chow, C. K. Rapid measurement of retinol, retinal, 13-cis-retinoic acid and all-trans-retinoic acid by high performance liquid chromatography. J Food Drug Anal. 13 (3), (2020).
- Teerlink, T., Copper, M. P., Klaassen, I., Braakhuis, B. J. M. Simultaneous analysis of retinol, all-trans- and 13-cis-retinoic acid and 13-cis-4-oxoretinoic acid in plasma by liquid chromatography using on-column concentration after single-phase fluid extraction. J Chromatogr B. Biomed Sci App. 694 (1), 83-92 (1997).
- Egberg, D. C., Heroff, J. C., Potter, R. H. Determination of all-trans and 13-cis vitamin A in food products by high-pressure liquid chromatography. J Agric Food Chem. 25 (5), 1127-1132 (1977).
- Sudo, Y., et al. A microbial rhodopsin with a unique retinal composition shows both sensory rhodopsin II and bacteriorhodopsin-like properties. J Biol Chem. 286 (8), 5967-5976 (2011).
- Kane, M. A., Napoli, J. L. Quantification of endogenous retinoids. Methods Mol Biol. 652, 1-54 (2010).
- Landers, G. M., Olson, J. A. Rapid, simultaneous determination of isomers of retinal, retinal oxime and retinol by high-performance liquid chromatography. J Chromatogr A. 438, 383-392 (1988).
- Hubbard, R., Brown, P. K., Bownds, D. [243] Methodology of vitamin A and visual pigments. Methods Enzymol. 18, 615-653 (1971).
- Robeson, C. D., et al. Chemistry of vitamin A. XXIV. The synthesis of geometric isomers of vitamin A via methyl β-methylglutaconate1. J Am Chem Soc. 77 (15), 4111-4119 (1955).
- Robeson, C. D., Blum, W. P., Dieterle, J. M., Cawley, J. D., Baxter, J. G. Chemistry of vitamin A. XXV. Geometrical isomers of vitamin A aldehyde and an isomer of its α-ionone analog1. J Am Chem Soc. 77 (15), 4120-4125 (1955).
- Hubinger, J. C. Determination of retinol, retinyl palmitate, and retinoic acid in consumer cosmetic products. J Cosmet Sci. 60 (5), 485-500 (2009).
- Bhat, P. V., Co, H. T., Lacroix, A. Effect of 2-alkanols on the separation of geometric isomers of retinol in non-aqueous high-performance liquid chromatography. J Chromatogr A. 260, 129-136 (1983).
- Stancher, B., Zonta, F. Quantitative high-performance liquid chromatographic method for determining the isomer distribution of retinol (vitamin A1) and 3-dehydroretinol (vitamin A2) in fish oils. J Chromatogr. 312, 423-434 (1984).
- Zonta, F., Stancher, B. High-performance liquid chromatography of retinals, retinols (vitamin A1) and their dehydro homologues (vitamin A2): improvements in resolution and spectroscopic characterization of the stereoisomers. J Chromatogr A. 301, 65-75 (1984).
- van Kuijk, F. J., Handelman, G. J., Dratz, E. A. Rapid analysis of the major classes of retinoids by step gradient reversed-phase high-performance liquid chromatography using retinal (O-ethyl) oxime derivatives. J Chromatogr. 348 (1), 241-251 (1985).
- Kane, M. A., Folias, A. E., Napoli, J. L. HPLC/UV quantitation of retinal, retinol, and retinyl esters in serum and tissues. Anal Biochem. 378 (1), 71-79 (2008).
- Widjaja-Adhi, M. A. K., Ramkumar, S., von Lintig, J. Protective role of carotenoids in the visual cycle. FASEB J. 32 (11), 6305-6315 (2018).
- Ramkumar, S., Jastrzebska, B., Montenegro, D., Sparrow, J. R., von Lintig, J. Unraveling the mystery of ocular retinoid turnover: Insights from albino mice and the role of STRA6. J Biol Chem. 300 (3), 105781 (2024).
- de Grip, W. J., Lugtenburg, J. Isorhodopsin: An undervalued visual pigment analog. Colorants. 1 (3), 256-279 (2022).
- Fan, J., Rohrer, B., Moiseyev, G., Ma, J., Crouch, R. K. Isorhodopsin rather than rhodopsin mediates rod function in RPE65 knock-out mice. Proc Natl Acad Sci USA. 100 (23), 13662-13667 (2003).
- Sparrow, J. R. Bisretinoids of RPE lipofuscin: Trigger for complement activation in age-related macular degeneration. Adv Exp Med Biol. 703, 63-74 (2010).
- Różanowska, M., Handzel, K., Boulton, M. E., Różanowski, B. Cytotoxicity of all-trans-retinal increases upon photodegradation. Photochem Photobiol. 88 (6), 1362-1372 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten