Method Article
In vitro Dreidimensionaler Keimtest der Angiogenese unter Verwendung embryonaler Stammzellen der Maus für die Modellierung von Gefäßerkrankungen und Arzneimitteltests
In diesem Artikel
Zusammenfassung
Dieser Assay verwendet embryonale Stammzellen der Maus, die in Embryoidkörperchen differenziert sind, die in 3D-Kollagengel kultiviert werden, um die biologischen Prozesse zu analysieren, die die Keimangiogenese in vitro steuern. Die Technik kann zum Testen von Medikamenten, zur Modellierung von Krankheiten und zur Untersuchung spezifischer Gene im Zusammenhang mit embryonal tödlichen Deletionen eingesetzt werden.
Zusammenfassung
Jüngste Fortschritte bei induzierten pluripotenten Stammzellen (iPSC) und Geneditierungstechnologien ermöglichen die Entwicklung neuartiger menschlicher zellbasierter Krankheitsmodelle für phänotypische Wirkstoffforschungsprogramme (PDD). Obwohl diese neuartigen Geräte die Sicherheit und Wirksamkeit von Prüfpräparaten beim Menschen genauer vorhersagen könnten, stützt sich ihre Entwicklung bis zur klinischen Entwicklung immer noch stark auf Daten von Säugetieren, insbesondere auf die Verwendung von Mauskrankheitsmodellen. Parallel zu humanen Organoid- oder Organ-on-Chip-Krankheitsmodellen ist die Entwicklung relevanter In-vitro-Mausmodelle daher ein ungedeckter Bedarf für die Bewertung direkter Arzneimittelwirksamkeits- und Sicherheitsvergleiche zwischen Spezies und In-vivo- und In-vitro-Bedingungen. In dieser Arbeit wird ein vaskulärer Keimtest beschrieben, der embryonale Stammzellen der Maus verwendet, die in Embryoidkörper (EBs) differenziert sind. Vaskularisierte EBs, die auf 3D-Kollagengel kultiviert werden, entwickeln neue Blutgefäße, die sich ausdehnen, ein Prozess, der als Keimangiogenese bezeichnet wird. Dieses Modell rekapituliert die wichtigsten Merkmale der in vivo sprießenden Angiogenese - Bildung von Blutgefäßen aus einem bereits bestehenden vaskulären Netzwerk - einschließlich der Selektion von Endothelspitzenzellen, der Migration und Proliferation von Endothelzellen, der Zellführung, der Röhrenbildung und der Rekrutierung von Muralzellen. Es ist für das Screening auf Medikamente und Gene geeignet, die die Angiogenese modulieren, und zeigt Ähnlichkeiten mit kürzlich beschriebenen dreidimensionalen (3D) vaskulären Assays, die auf humanen iPSC-Technologien basieren.
Einleitung
In den letzten drei Jahrzehnten wurde die zielgerichtete Wirkstoffforschung (TDD) von der pharmazeutischen Industrie in großem Umfang in der Arzneimittelforschung eingesetzt. TDD beinhaltet ein definiertes molekulares Ziel, das eine wichtige Rolle bei einer Krankheit spielt, und beruht auf der Entwicklung relativ einfacher Zellkultursysteme und Readouts für das Wirkstoff-Screening1. Zu den meisten typischen Krankheitsmodellen, die in TDD-Programmen verwendet werden, gehören traditionelle Zellkulturmethoden wie Krebszellen oder immortalisierte Zelllinien, die in künstlichen Umgebungen und unphysiologischen Substraten gezüchtet werden. Obwohl viele dieser Modelle praktikable Werkzeuge zur Identifizierung erfolgreicher Arzneimittelkandidaten zur Verfügung gestellt haben, kann der Einsatz solcher Systeme aufgrund ihrer geringen Krankheitsrelevanz fraglich sein2.
Bei den meisten Krankheiten sind die zugrunde liegenden Mechanismen in der Tat komplex und verschiedene Zelltypen, unabhängige Signalwege und mehrere Gensätze tragen oft zu einem bestimmten Krankheitsphänotyp bei. Dies gilt auch für Erbkrankheiten, bei denen die Hauptursache eine Mutation in einem einzigen Gen ist. Mit dem jüngsten Aufkommen von Technologien für humane induzierte pluripotente Stammzellen (iPSC) und Geneditierungswerkzeugen ist es nun möglich, 3D-Organoide und Organ-on-Chip-Krankheitsmodelle zu generieren, die die menschliche Komplexität in vivo besser rekapitulieren könnten 3,4. Die Entwicklung solcher Technologien ist mit einem Wiederaufleben des Interesses an phänotypischen Wirkstoffforschungsprogrammen (PDD) verbunden1. PDD kann mit empirischem Screening verglichen werden, da sie nicht auf dem Wissen über die Identität eines bestimmten Wirkstoffziels oder einer Hypothese über seine Rolle bei der Krankheit beruht. Der PDD-Ansatz wird heute zunehmend als wichtiger Beitrag zur Entdeckung von First-in-Class-Medikamenten anerkannt5. Da die Entwicklung menschlicher Organoid- und Organ-on-Chip-Technologien noch in den Kinderschuhen steckt, wird erwartet, dass iPSC-Modelle (ergänzt durch innovative Bildgebungs- und maschinelle Lernwerkzeuge 6,7) in naher Zukunft mehrere neuartige komplexe zellbasierte Krankheitsmodelle für das Wirkstoffscreening und die damit verbundenen PDD-Programme bereitstellen werden, um die geringe Produktivität des TDD-Ansatzes zu überwinden8. 9. Sonstiges
Während menschliche Organoid- und Organ-on-Chip-Modelle wichtige Einblicke in die Komplexität von Krankheiten und die Identifizierung neuer Medikamente liefern können, ist die Einführung von Medikamenten in die neue klinische Praxis auch stark auf Daten aus Tiermodellen angewiesen, um ihre Wirksamkeit und Sicherheit zu bewerten. Unter ihnen sind genetisch veränderte Mäuse sicherlich die am meisten bevorzugten Säugetiermodelle. Sie haben viele Vorteile, da sie eine relativ kurze Generationszeit für Säugetiere haben, viele ähnliche Phänotypen wie menschliche Krankheiten haben und leicht genetisch manipuliert werden können. Sie werden daher in großem Umfang in Arzneimittelforschungsprogrammen eingesetzt10. Die Überbrückung der Kluft zwischen Mäusen und Menschen bleibt jedoch eine wichtige Herausforderung11. Die Entwicklung von In-vitro-Mausmodellen, die humanen Organoid- und Organ-on-Chip-Modellen entsprechen, könnte diese Lücke zumindest teilweise schließen, da sie direkte Vergleiche der Wirksamkeit und Sicherheit von Medikamenten zwischen In-vivo-Maus- und In-vitro-Humandaten ermöglicht.
In dieser Arbeit wird ein vaskulärer Keimtest in Maus-Embryoidkörpern (EBs) beschrieben. Blutgefäße bestehen aus Endothelzellen (innere Auskleidung der Gefäßwände), Wandzellen (glatte Gefäßmuskelzellen und Perizyten)12. Dieses Protokoll basiert auf der Differenzierung von embryonalen Stammzellen (mES-Zellen) der Maus in vaskularisierte EBs unter Verwendung von hängenden Tröpfchen, die die de novo Endothelzell- und Muralzelldifferenzierung rekapitulieren13,14. ES-Zellen der Maus können leicht in Kultur aus isolierten Tag-3.5-Blastozysten der Maus mit unterschiedlichem genetischen Hintergrund etabliert werden15. Sie bieten auch Möglichkeiten für klonale Analysen, die Rückverfolgung von Abstammungslinien und können leicht genetisch manipuliert werden, um Krankheitsmodelle zu erstellen13,16.
Da Blutgefäße alle Organe ernähren, ist es nicht verwunderlich, dass viele, wenn nicht sogar alle, Krankheiten mit Veränderungen der Mikrogefäße verbunden sind. Unter pathologischen Zuständen können Endothelzellen einen aktivierten Zustand annehmen oder dysfunktional werden, was zum Absterben von Muralzellen oder zur Abwanderung von Blutgefäßen führt. Diese können zu einer übermäßigen Angiogenese oder zu einer Verdünnung der Gefäße führen, können einen abnormalen Blutfluss und eine defekte Blutgefäßbarriere induzieren, was zu einer Extravasation von Immunzellen und Entzündungen führt12,17,18,19. Die Forschung für die Entwicklung von Medikamenten, die Blutgefäße modulieren, ist daher hoch, und es wurden bereits mehrere molekulare Akteure und Konzepte für die therapeutische Ausrichtung identifiziert. In diesem Zusammenhang eignet sich das beschriebene Protokoll besonders für die Erstellung von Krankheitsmodellen und für die Erprobung von Medikamenten, da es die wichtigsten Merkmale der in vivo keimenden Angiogenese rekapituliert, einschließlich der Selektion von Endothelspitzen- und Stielzellen, der Migration und Proliferation von Endothelzellen, der Führung von Endothelzellen, der Röhrenbildung und der Rekrutierung von Muralzellen. Es zeigt auch Ähnlichkeiten mit kürzlich beschriebenen 3D-vaskulären Assays, die auf humanen iPSC-Technologien basieren20.
Protokoll
1. Medienvorbereitung und -kultur des mESC
- Bereiten Sie konditioniertes Medium +/- (CM+/-) mit dem Nahrungsergänzungsmittel 1x Glasgow MEM (G-MEM BHK-21) Medium mit 10% (vol/vol) hitzeinaktiviertem fötalem Kälberserum (FBS), 0,05 mM β-Mercaptoethanol, 1x nicht-essentiellen Aminosäuren (NEAA 1x), 2 mM L-Glutamin und 1 mM Natriumpyruvat zu.
- Bereiten Sie konditioniertes Medium +/+ (CM+/+) unter Verwendung des Nahrungsergänzungsmittels CM+/- medium mit Leukämie-Hemmfaktor (LIF) (1.500 U/ml) und 0,1 mM β-Mercaptoethanol zu.
- Konditioniertes Medium +/+ in Gegenwart von zwei Inhibitoren (CM+/+ 2i) unter Verwendung des Supplements CM+/+ Medium mit 1 μM PD0325901 und 3 μM CHIR99021 herstellen.
- Bereiten Sie eine 0,1%ige Gelatinelösung vor, indem Sie 25 ml vorgewärmte 2%ige Gelatinelösung in 500 ml phosphatgepufferter Kochsalzlösung ohne Calcium und Magnesium (DPBS) mischen.
- Bereiten Sie 10x Trypsin-Versenphosphat-Puffer (TVP 10x) vor, indem Sie Trypsin (2,5 %) mit TVP 1x (9,5 %, 10x DPBS, 1 mM EDTA, 0,01 % Hühnerserum, 0,01 % Trypsin (2,5 %) inH2O) mischen (Verhältnis 1:10, vol/vol).
Anmerkungen: Filtern Sie alle Lösungen durch einen 0,22-μm-Porenfilter. Lagern Sie das Nährmedium langfristig bei -20 °C und bewahren Sie andere Reagenzien bis zu 3 Wochen bei 4 °C auf (siehe Materialtabelle). - Zwei 12-Well-Zellkulturplatten mit 0,1%iger Gelatinelösung (500 μL pro Well) bestreichen und 30 min in einem CO 2 -Inkubator (37 °C, 5% CO2, feuchte Atmosphäre) inkubieren.
- Waschen Sie die gelatinebeschichteten Platten mit PBS und fügen Sie 500 μL CM+/- Medium hinzu.
- Tauen Sie zwei Fläschchen mit 1 x 106 kryokonservierten bestrahlten embryonalen Mausfibroblasten (MEFs) bei 37 °C auf und überführen Sie die Zellsuspension in ein konisches Röhrchen mit 5 ml CM+/- Medium.
- Zentrifugieren Sie die Zellen bei 200 x g für 5 min bei Raumtemperatur (RT). Saugen Sie das Medium an und resuspendieren Sie das Zellpellet vorsichtig in 12 ml CM+/- Medium mit einer Konzentration von 1,67 x 105 Zellen pro ml.
- Säen Sie 500 μl MEF-Suspension in eine 12-Well-Zellkulturplatte (2,4 x 10,5 Zellen procm2) und inkubieren Sie die Platte über Nacht (o/n) in einem CO2 -Inkubator.
- Eine Durchstechflasche mit kryokonservierten mES-Zellen (1 x 106) wird in CM+/+ 2i-Medium aufgetaut.
- Die mESC-Suspension wird auf einer vorgewaschenen 12-Well-Platte mit MEFs in 1 ml CM+/+ 2i-Medium ausgesät und die Platte in einen CO2 -Inkubator überführt. Frischen Sie das Medium täglich auf.
- Bei 70% Konfluenz werden die mESC-Kolonien mit DPBS gewaschen. Fügen Sie 150 μl warmen 10x TVP-Puffer hinzu und inkubieren Sie ihn 30 s lang bei RT, um die enzymatische Dissoziation zu initiieren.
- Entfernen Sie vorsichtig den TVP-Puffer, resuspendieren Sie die Zellen in 1 ml CM+/+ 2i-Medium und dissoziieren Sie die Kolonien durch sanftes Pipettieren in einzelne Zellen.
- Passage der Zellen, indem man sie auf eine neue 12-Well-Zellkulturplatte mit MEFs überführt (Spaltungsverhältnis: 1:3-1:5). Mischen Sie vorsichtig, um die Zellen zu verteilen, und inkubieren Sie sie in einem CO2 - Inkubator.
- Frischen Sie das Nährmedium (2-3 ml) auf und beobachten Sie täglich das Zellwachstum/die Zellmorphologie. Wiederholen Sie die serielle Übergabe bei 70 % Konfluenz alle 2 Tage. Wechseln Sie für zwei Durchgänge auf CM+/+ Medium, bevor Sie die Zelldifferenzierung einleiten.
2. EB-Bildung im hängenden Tropfen
- Stellen Sie frisches Mesodermdifferenzierungsmedium her, indem Sie das CM+/- Medium mit basischem Fibroblastenwachstumsfaktor (bFGF) (50 ng·mL-1) und mit Bone Morphogenetic Protein 4 (BMP-4) (5 ng·mL-1) ergänzen und bis zur Verwendung bei 4 °C halten.
- Eine Vertiefung der 6-Well-Zellkulturplatte mit 500 μL 0,1%iger Gelatinelösung bestreichen und für 30 min in einen CO2 -Inkubator geben.
- Waschen Sie die gelatinebeschichteten Platten mit PBS und fügen Sie 500 μL CM+/- Medium hinzu.
- Um eine reine Population von mES-Zellen zu erhalten, trypsinisieren Sie die Zellkulturplatte mit 10x TVP-Puffer für 30 s bei RT, resuspendieren Sie die Zellen in 1 mL Mesoderm-Differenzierungsmedium und übertragen Sie sie dann für 30 min auf die gelatinebeschichtete 6-Well-Platte, damit sich die MEFs anlagern können, während die mESCs in Suspension bleiben.
- Sammeln Sie die Zellsuspension in einem konischen 50-ml-Röhrchen und zählen Sie die Zellen mit einem Neubauer-Hämozytometer und einem Trypanblau-Farbstoff für einen Lebend-/Totzellausschluss.
- Zentrifugieren Sie die Zellen bei 200 x g für 5 min bei RT. Entfernen Sie den Überstand und resuspendieren Sie das Zellpellet in Mesodermdifferenzierungsmedium, um 4,55 x 104 Zellen pro ml zu erreichen.
- Füllen Sie den Boden von 94 mm Polystyrolschalen mit niedrigem Aufsatz mit 15 ml sterilem Wasser.
HINWEIS: Vier Schalen mit hängenden Tropfen (1,6 x 105 Zellen in 3,52 ml Medium) sind erforderlich, um eine bestimmte Erkrankung mit dem 3D-Keimungsangiogenese-Assay zu testen. - Übertragen Sie die Zellsuspension in ein steriles Kunststoffreservoir und beladen Sie vier Positionen einer Mehrkanalpipette mit 22 μl Zellsuspension pro Kanal (1 x 10,3 Zellen pro 22 μl Tropfen).
- Heben Sie den Deckel der 94-mm-Schüssel an, drehen Sie ihn um und legen Sie ihn mit der Innenseite nach oben auf die saubere Oberfläche des Durchlaufschranks.
- Geben Sie 40 Tropfen der Zellsuspension auf die Innenfläche jedes Deckels. Drehen Sie den Deckel vorsichtig und ungestört um und setzen Sie ihn wieder auf die Schale, so dass die Tropfen dem Wasser zugewandt sind.
- Inkubieren Sie die Gerichte in einem CO2 - Inkubator. Betrachten Sie dies als Differenzierungstag 0. Bewahren Sie die Platten 4 Tage lang auf, um EBs zu bilden.
3. Konkurrenz-Assay für die Position der Spitzenzelle
- Kultivieren Sie eine fluoreszierende und eine nicht fluoreszierende mESC-Linie, wie zuvor beschrieben13. Als Beispiel werden gelb markierte 7ACS/EYFP-mESCs und R1-mESCs verwendet.
- Bereiten Sie Mosaik-EBs vor, indem Sie die beiden mESC-Linien zu gleichen Teilen mischen (Verhältnis 1:1) und inkubieren Sie die hängenden Tropfenschalen in einem CO 2 -Inkubator, wie in Schritt2 beschrieben.
4. Schwimmende EBs-Kultur zur vaskulären Differenzierung
- Bevor Sie EBs aus den hängenden Tropfen sammeln, bereiten Sie Folgendes vor.
- Bereiten Sie 5%ige Agarlösung inH2Ovor und sterilisieren Sie sie durch Autoklavieren (20 min bei 120 °C).
- Verwenden Sie die warme 5%ige Agarlösung, um G-MEM BHK-21 Medium mit 1%igem Agar zuzubereiten, und gießen Sie schnell 3 ml in eine der 60-mm-Styroporschalen. Lassen Sie den Agar 1 h bei RT erstarren. Lagern Sie das Geschirr bis zur Verwendung bei 4 °C.
- Bereiten Sie frisches 2x vaskuläres Differenzierungsmedium zu, indem Sie das CM+/- Medium mit bFGF (100 ng·mL-1) und VEGF-A (50 ng·mL-1) ergänzen. Lagern Sie das Medium bis zur Verwendung bei 4 °C.
- Sammeln Sie die hängenden Tropfen in einem konischen 15-ml-Röhrchen mit einer P1000-Pipette und entfernen Sie den Überstand nach einigen Minuten EB-Sedimentation.
- Resuspendieren Sie die EBs in 3 mL 2x vaskulärem Differenzierungsmedium, überführen Sie die EB-Suspension in eine agarbeschichtete Schale und verteilen Sie die EBs homogen, um eine Aggregation zu vermeiden.
- Die Schalen werden im CO 2 -Inkubator inkubiert und das Medium alle2 Tage bis zum 9. Tag mit 1x vaskulärem Differenzierungsmedium in Gegenwart von bFGF (50 ng·mL-1) und VEGF-A (25 ng·mL-1) aufgefrischt.
- Alternativ kann Platelet Derived Growth Factor-BB (PDGF-BB) (10 ng·mL-1 an Tag 4 und 5 ng·mL-1 an Tag 6 und Tag 8) in das vaskuläre Differenzierungsmedium gegeben werden, um die Differenzierung der Muralzellen zu fördern.
5. Durchflusszytometrische Analyse
- Sammeln Sie die 9 Tage alten EBs in einem konischen 15-ml-Röhrchen mit einer P1000-Pipette und waschen Sie sie einmal mit warmem PBS.
- Fügen Sie 1 ml G-MEM BHK-21-Medium hinzu, das 0,2 mg·ml-1 Kollagenase A enthält, und inkubieren Sie die Zellen 5 Minuten lang in einem CO2 -Inkubator.
- Dissoziieren Sie die EBs, indem Sie mit einer P1000-Pipette vorsichtig auf und ab pipettieren.
- Stoppen Sie die Kollagenaseaktivität durch Zugabe von 1 ml kaltem G-MEM BHK-21-Medium mit 10 % FBS.
- Zentrifugieren Sie die Zellen bei 200 x g für 5 min bei RT.
- Resuspendieren Sie die Zellen in 500 μl PBS mit 2% FBS.
- Zählen Sie die Zellen mit einem Neubauer-Hämozytometer.
- Resuspendieren Sie 400.000 Zellen in 100 μl PBS mit 2 % FBS pro Färbebedingung.
- Die Zellen werden 45 min lang bei 4 °C mit folgenden Antikörpern inkubiert: APC-konjugierter Ratten-Anti-Maus-Anti-Maus-PECAM-1-Antikörper (Klon MEC13.3) und FITC-konjugierter Ratten-Anti-Maus-CD45 (Klon 30-F11) oder Isotyp-Kontroll-Antikörper.
- Waschen Sie die Zellen zweimal mit 1 ml PBS, das 2 % FBS enthält.
- Resuspendieren Sie die Zellen, um eine Endkonzentration von 5 x 106 Zellen pro ml zu erreichen.
- Filtern Sie die Zellen mit einem Reagenzglas aus Polystyrol mit rundem Boden und einem Schnappverschluss für das Zellsieb.
- Analysieren Sie 20.000 PECAM-1 (+)-Ereignisse mittels Durchflusszytometrie.
6.3D Assay zur Keimangiogenese und Immunfluoreszenzfärbung
- Bereiten Sie an Tag 9 ein Keimmedium vor, indem Sie 10 % FBS (vol/vol), bFGF (50 ng·mL-1), VEGF-A (25 ng·mL-1), humanes rekombinantes Erythropoetin (hEPO) (20 ng·mL-1), humanes Interleukin-6 (IL-6) (10 ng·mL-1), 0,05 mM β-Mercaptoethanol, NEAA (1x), L-Glutamin (1x), Natriumpyruvat (1x), Typ-I-Rattenschwanzkollagen (1,25 mg·ml-1), und NaOH (3,1 mM) auf GMEM BHK-1 Medium.
- Um eine Kollagengelierung zu vermeiden, bewahren Sie das Keimmedium bis zur Verwendung auf Eis auf.
- Um die Wirkung von pro/anti-angiogenen Molekülen zu bewerten, fügen Sie dem Keimmedium das ausgewählte Medikament oder Vehikel in der ausgewählten Konzentration hinzu.
- Sammeln Sie 9 Tage alte EBs aus einer 60-mm-Agarschale in einem konischen 15-ml-Röhrchen (entspricht einer Bedingung) und entfernen Sie den Überstand nach einigen Minuten Sedimentation.
- Decken Sie den Boden einer 35-mm-Kulturschale mit 1 ml des Keimmediums ab und inkubieren Sie ihn 5 Minuten lang bei 37 °C, um die Gelierung einzuleiten.
- Resuspendieren Sie die EBs in 2 ml kaltem Keimmedium.
- Übertragen Sie die Suspension in die 35-mm-Kulturschale, die mit der ersten Schicht Keimmedium beschichtet ist.
- Verteilen Sie die EBs auf der gesamten Platte und stellen Sie sicher, dass sie sich in gleichem Abstand zueinander befinden. Inkubieren Sie das Gericht in einem CO2 - Inkubator. Die erste Sprossenbildung erfolgt innerhalb von 24-48 h.
- An Tag 12 wird das EBs-haltige Kollagengel vorsichtig mit einem Spatel auf einen Objektträger (75 x 26 mm) übertragen.
- Entfernen Sie die überschüssige Flüssigkeit mit einer Pipette (P1000) und dehydrieren Sie das Gel, indem Sie eine Mullfolie aus Nylonleinen und saugfähige Filterkarten (Gellöschpapier) auf das Gel legen. Üben Sie Druck aus, indem Sie ein Gewicht (250 g) für 2 Minuten platzieren. Entfernen Sie vorsichtig das Nylon-/Filterpapier und lassen Sie die Objektträger 30 Minuten lang bei RT an der Luft trocknen.
- Waschen Sie die Objektträger dreimal mit PBS für 5 min bei RT.
- Fixieren Sie die EBs mit Zinklösung (siehe Materialtabelle) o/n bei 4 °C. Alternativ fixieren Sie die mosaikfluoreszierenden EBs mit Paraformaldehyd (PFA) (4%) o/n bei 4 °C im Dunkeln.
- Entferne das Fixiermittel. Waschen Sie die Objektträger fünfmal mit PBS für 5 min bei RT.
- Permeabilisieren Sie die EBs in PBS, die 0,1% Triton-X100 enthalten, für 15 min bei RT.
- Entfernen Sie die Permeabilisierungslösung. Waschen Sie die Objektträger fünfmal mit PBS für 5 Minuten.
- Inkubieren Sie die EBs im Blockierungspuffer (PBS mit 2% Kälberserumalbumin, BSA) für 1 h bei RT.
- Um die Endothelsprossen zu färben, verwenden Sie einen Anti-Maus-Anti-PECAM-1-Primärantikörper der Ratte (Verdünnung 1:100) im Blockierungspuffer o/n bei 4 °C.
- Waschen Sie die Objektträger fünfmal mit PBS für 5 Minuten.
- Inkubieren Sie die Objektträger mit dem Anti-Ratten-Sekundärantikörper Alexa 555 für Ziegen in Blockierungspuffer (1:250-Verdünnung) und bei Bedarf mit FITC-konjugiertem Anti-α-SMA-Antikörper im Blocking-Puffer (1:250-Verdünnung), um Wandzellen 2 h lang bei RT im Dunkeln zu färben.
- Waschen Sie die Objektträger dreimal 5 min lang mit PBS und einmal mit H20, bevor Sie sie montieren.
7. Konfokale Bildgebung, morphometrische und quantitative Analyse von EB-Endothelsprossen
- Hochauflösende Bilder der immungefärbten EBs durch Zusammenführung der Fokusebene (z-Stacking) mit einem konfokalen Mikroskop aufnehmen. Verwenden Sie ein Objektiv mit 10-facher Vergrößerung, um ganze EBs abzubilden.
- Analysieren Sie die mit ImageJ aufgenommenen Bilder, um die Morphologie zu bewerten und die Eigenschaften von PECAM-1-positiven Endothelsprossen gemäß etablierten Quantifizierungsmethoden zu quantifizieren13,14,21.
- Berechnen Sie die durchschnittliche Anzahl der Endothelsprossen pro EBs, indem Sie die Gesamtzahl der Sprossen pro einzelnen EBs manuell zählen.
- Messen Sie die einzelnen Sprossenlängen mit dem ImageJ-Zeichenwerkzeug. Definieren Sie die Basis des Endothelsprosses beginnend im EB-Kernbereich und ziehen Sie manuell eine Linie bis zum Ende der Sprossenspitze.
- Berechnen Sie die mittlere Anzahl der Spitzenzellen pro Spross, indem Sie die Anzahl der Spitzenzellen pro einzelnen Spross manuell zählen, und berechnen Sie dann den Mittelwert pro EB.
- Berechnen Sie die Ausrichtung der Filopodien, indem Sie die Achse des Elternsprosses manuell bestimmen, und messen Sie den spitzen Winkel zwischen ihnen mit dem Winkelwerkzeug der ImageJ-Software. Berechnen Sie die Anzahl der Sprossen mit einer Ausrichtung >50° und teilen Sie sie durch die Gesamtzahl der Sprossen des gewünschten EB.
HINWEIS: Die Winkel reichten immer von 0° bis 90°.
- Nehmen Sie hochauflösende Bilder der immungefärbten EBs mit einem konfokalen Mikroskop auf. Verwenden Sie ein Objektiv mit 40-facher Vergrößerung, um hochauflösende Bilder einzelner Endothelsprossen zu erstellen.
- Quantifizieren Sie die Gefäßabdeckung von PECAM-1-positiven EC-Sprossen, die von α-SMA-positiven MCs umgeben sind, mithilfe der ImageJ-Software.
- Teilen Sie die zusammengefügten Bilder in separate rote und grüne Kanäle auf.
- Konvertieren Sie die Bilder in ihre binäre Form.
- Messen Sie die gesamte Zellfläche des Sprosses, die separat von PECAM-1 (rot markierte Endothelzellen) und von α-SMA-positiven Zellen (grün markierte Wandzellen) besetzt ist.
- Generieren Sie das zusammengeführte Bild mit der Bildrechnerfunktion und dem AND-Operator. Messen Sie die gesamte zelluläre Fläche des Bildes. Um die Abdeckung zu berechnen, teilen Sie die Fläche des kolokalisierten Bildes durch die Fläche des PECAM-1-Binärbildes.
- Um die Zellkonkurrenz um die Position der Endothelspitzen-/Stielzellen von Sprossen, die von Mosaik-EBs entwickelt wurden, zu analysieren, wird die Anzahl der Spitzenzellen manuell bewertet und ihre genotypische Herkunft anhand des Fluoreszenzsignals markiert. Berechnen Sie die Mittelwerte pro EB.
Ergebnisse
Die Protokollübersicht des Blutgefäß-Sprossen-Assays ist in Abbildung 1 dargestellt. Neun Tage alte EBs, die aus drei unabhängigen 129/Ola mESC-Linien (Z/Red, R1 und E14) stammten, wurden mit Hilfe von Kollagenase A enzymatisch in einzelne Zellen dissoziiert. Die Zellen wurden auf PECAM-1 gefärbt und wie beschrieben mittels Fluoreszenz-aktivierter Zellsortierung (FACS) analysiert. Alle Zelllinien zeigten eine robuste Endotheldifferenzierung, und es wurden keine Unterschiede in ihrer Fähigkeit zur Differenzierung in Endothelzellen beobachtet. Alle Zelllinien produzierten etwa 10,5 % ± 1,3 % Endothelzellen (Abbildung 2A). Die relativen Expressionsniveaus von PAN-endotelialen Zellmarkern in den PECAM-1 (+) Zellpopulationen wurden ebenfalls quantifiziert. Die mRNA-Expressionsniveaus aller analysierten Marker (Flk1, Flt1, Flt4, Eng, Tie2 und Cdh5) waren zwischen den Zelllinien und den Experimenten vergleichbar, was die Robustheit des Differenzierungsprotokolls bestätigt (Abbildung 2B). PECAM-1 (+)-Zellpopulationen exprimierten nur sehr niedrige mRNA-Konzentrationen von arteriellen (Notch1 und Efnb2) oder venösen (Couptf2 und Ephb4) Markern, was den relativ unreifen Zustand der Endothelzellen unterstützt, die durch das Protokoll generiert wurden (Abbildung 2C).
Anschließend wurde die Fähigkeit des Gefäßkeim-EB-Modells demonstriert, nach Medikamenten zu suchen, die die Angiogenese modulieren (Abbildung 3). DC101 und DAPT (N-[N-(3,5-difluorphenacetyl)-Lalanyl]-S-phenylglycin t-butylester) wurden getestet. Diese Verbindungen werden in Mäusen häufig verwendet, um die Angiogenese zu blockieren, indem sie die VEGFR2-Aktivität hemmen, oder um die Differenzierung von Endothelspitzenzellen und die Bildung eines dichten vaskulären Plexus zu fördern, indem sie auf den Notch-Signalweg abzielen (Abbildung 3A). Sowohl VEGF- als auch Notch-Signalwege sind wichtige Regulatoren der keimenden Angiogenese in vivo. Die Effekte verschiedener Konzentrationen von DC101 und DAPT in EBs, die mit Kollagen I plattiert wurden, wurden untersucht. Hohe Dosen von DC101 im Bereich von 6-30 mg· L-1 hemmte sowohl die Anzahl als auch die Länge der Gefäßkeime, während DAPT bei einer Dosis von 1 μmol· gegenteilige Effekte hatte L-1 (Abbildung 3B-C). Stark vergrößerte Bilder der für PECAM-1 gefärbten Gefäßsprossen von EBs, die in Gegenwart von DAPT kultiviert wurden, sind ebenfalls vorhanden (Abbildung 3D). DAPT auch bei niedrigen Dosen von 0,5-1 μmol· L-1 erhöhte stark die Anzahl der Endothelspitzenzellen mit Gefäßsprossen, die einen fehlgeleiteten Phänotyp aufwiesen (Abbildung 3D-F). Eine hohe Dosis von DAPT führte auch zu einer Gefäßkoaleszenz und zur Bildung großer und flacher Endothelzellbereiche ohne Organisation (Abbildung 3A). Die Ergebnisse bestätigten die Fähigkeit des Modells, Medikamente zu testen, die die Angiogenese entweder fördern oder hemmen.
Um zu bestätigen, dass dieses Modell für die Nachahmung von Gefäßerkrankungen geeignet ist, werden die konfokalen Bilder von EBs bereitgestellt, die von Acvrl1+/- mESCs abgeleitet wurden. Das Acvrl1-Gen kodiert für ALK1 (Activin Receptor-like Kinase 1), einen Rezeptor, der spezifisch in Endothelzellen exprimiert wird und bei Mutation für die Entwicklung einer seltenen vaskulären Erkrankung mit Angiodysplasie verantwortlich ist, die als hereditäre hämorrhagische Teleangiektasie (HHT) bezeichnet wird. Ein stark vergrößertes Bild der Acvrl1+/- Endothelsprossen zeigte, dass sie mehr Endothelspitzenzellen und mehr Verzweigungen pro Spross aufwiesen, die in zufälligen Winkeln zu den Elterngefäßen standen. Diese bestätigten in vitro einen fehlgeleiteten Phänotyp, wie er bei HHT-Mäusen beobachtet wurde (Abbildung 4).
Durch die Bildung chimärer EBs, die differenzierte fluoreszenzmarkierte mESC und mESC eines bestimmten Genotyps enthalten, wird ein alternatives Protokoll zur Untersuchung des Prozesses der Selektion der Endothelspitze enthalten (Abbildung 5A-C). Eine konfokale Aufnahme von PECAM-1-markierten Gefäßsprossen identifizierte den genotypischen Ursprung der führenden Endothelspitzenzellen (Abbildung 5B). Mischungen einer Wildtyp-YFP-mESC-Linie (gelb fluoreszierendes Protein) im Verhältnis 1:1 mit einer unmarkierten mESC-Wildtyp-Linie führten durchweg zu einem gleichen Beitrag jeder Population zu den führenden Endothelspitzenzellen (Abbildung 5C).
Dieses Protokoll eignet sich auch zur Quantifizierung der Wandzellbedeckung des Gefäßkeims. EBs, die eine Angiogenese durchlaufen, wurden fixiert und für PECAM-1 (Endothelzellen, rot) und für α-Smooth Muscle Actin (α-SMA) (Muralzellen, grün) gefärbt (Abbildung 5D). Ein Bild mit hoher Vergrößerung zeigte, wie ein einzelner Gefäßspross von Wandzellen umgeben war (Abbildung 5E, links). Die binäre Transformation wurde nach der Farbkanaltrennung durchgeführt (Abbildung F-G), um das Verhältnis des PECAM-1 (+)-Gefäßes, das von α-SMA (+) Wandzellen bedeckt ist, mit der ImageJ-Software zu quantifizieren (Abbildung 5H).

Abbildung 1: Zeitlicher Ablauf der Protokollverfahren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
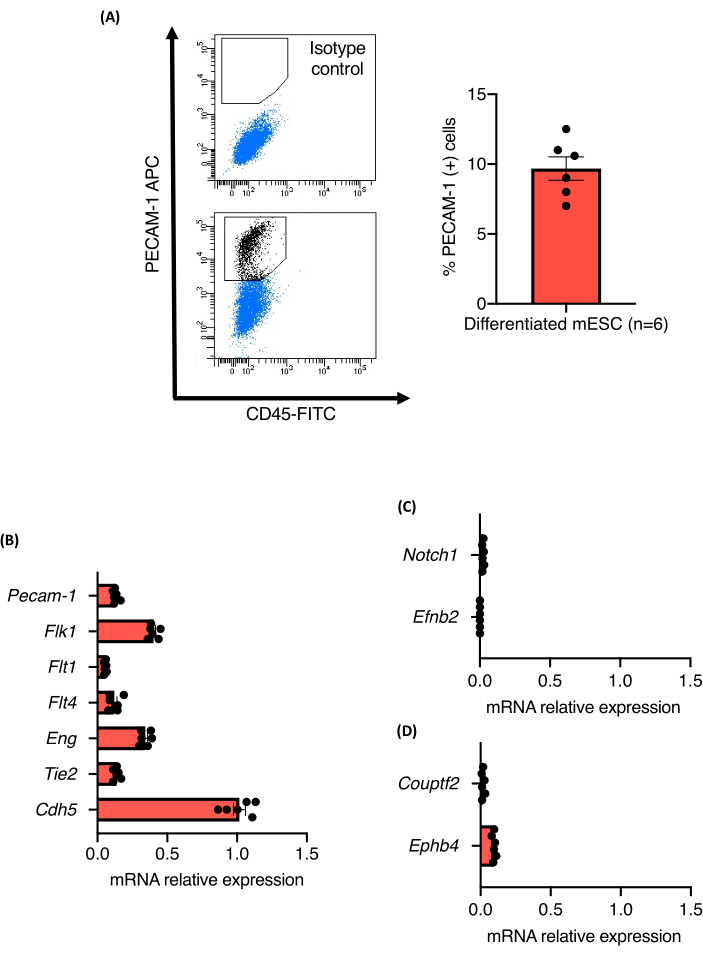
Abbildung 2: Charakterisierung von ECs, die aus der vaskulären Differenzierung von mES-Zellen innerhalb von EBs abgeleitet wurden. (A) Durchflusszytometrische Analyse der CD31-Expression aus 9 Tage alten EBs und Quantifizierung des Prozentsatzes der Pecam-1 (+)-Zellen. (B-D) mRNA-Expressionsniveaus von Pecam-1, Flk1, Flt1, Flt4, Eng, Tie2, Cdh5, Notch1, EfnB2, Couptf2 und EphB4 in sortierten Endothelzellen von 9 Tage alten EBs. Fehlerbalken stellen den Mittelwert ± den Standardfehler des Mittelwerts (SEM) dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
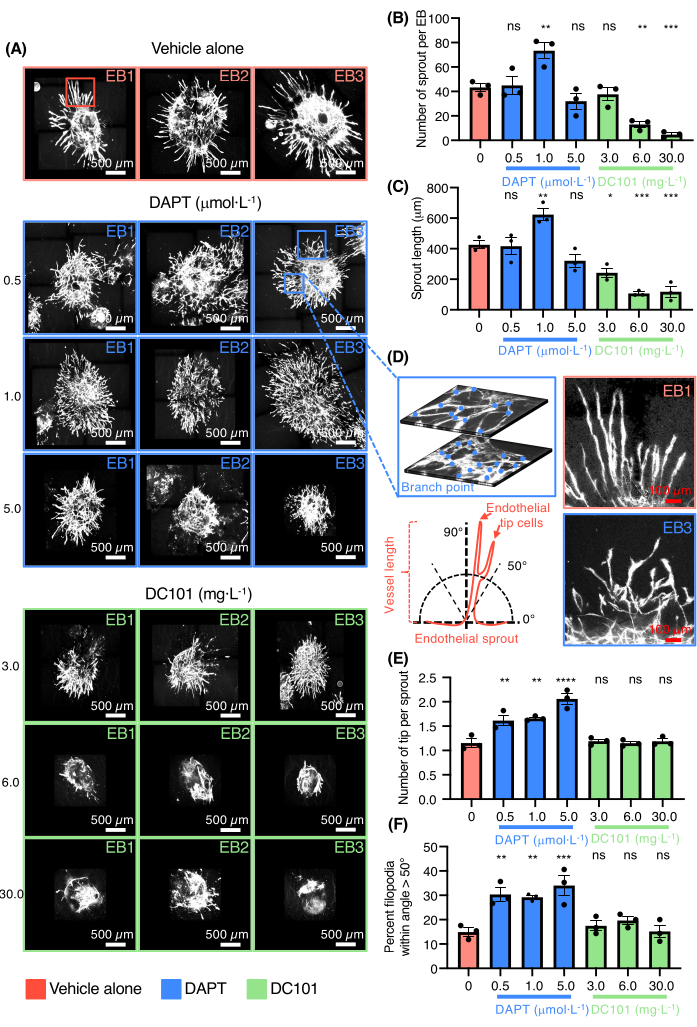
Abbildung 3: 3D-Keimungs-Angiogenese-Assay für Arzneimitteltests. (A) Konfokale Aufnahmen von drei repräsentativen EBs, die für Pecam-1 (weiße, Endothelzellen) gefärbt wurden, die nur mit Vehikel, DAPT (0,5 μmol· L-1, 1,0 μmol· L-1, 5,0 μmol· L-1) oder DC101 (3 mg· L-1, 6 mg· L-1, 30 mg· L-1). (B) Quantifizierung der Anzahl der Sprossen pro EB. (C) Quantifizierung der Sprossenlänge. (D) Oben links zeigt die hohe Vergrößerung des Endothelsprossen die Netzwerkkomplexität und den Verzweigungspunkt, der auf zwei verschiedenen Schichten desselben EB zählt, unten links eine schematische Darstellung, die die Messmethode der endothelialen Sprossenorientierung darstellt. Auf der oberen rechten Tafel eine hohe Vergrößerung der Endothelsprossen allein aus dem Fahrzeug und auf der unteren rechten Tafel eine hohe Vergrößerung der Endothelsprossen aus DAPT (0,5 μmol· L-1) Zustand. (E) Quantifizierung der Anzahl der Spitzenzellen pro Spross. (F) Quantifizierung des Prozentsatzes der Filopodien innerhalb eines Winkels >50°. Alle Balken stellen den Mittelwert ± SEM- und p-Werte aus dem ungepaarten einfaktoriellen ANOVA-Test dar. ns = nicht signifikant, *p < 0,05, **p < 0,01, ***p < 0,001 und ****p < 0,0001. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 4: Sprossendefekte Gefäße in hereditären hämorrhagischen Teleangiektasie-EBs. Auf der Oberseite sind konfokale Bilder von repräsentativen 12 Tage alten EBs aus den Genotypen Acvrl1+/+ und Acvrl1+/- zu sehen, die für Pecam-1 (weiße, Endothelzellen) gefärbt wurden. Auf der Unterseite ist eine hohe Vergrößerung der Acvrl1+/- Endothelsprossen (weißer Kasten von oben) zu sehen, die zahlreiche Spitzenzellen (rote Pfeile), Endothelverzweigungspunkte (blaue Punkte) und den Phänotyp der Triebfehlleitung (grüne Pfeile) zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 5: Untersuchung der Position der Spitzen-/Stielzellen und der Gefäßreifung mit dem 3D-Sprossen-Assay. (A) Konfokales Bild eines repräsentativen, 12 Tage alten chimären EB aus R1-Wildtyp-Zelllinie, gemischt 1:1 mit 7ACS/EYFP-Wildtyp-Zelllinie, gefärbt für Pecam-1 (rote, Endothelzellen). Rote Pfeile zeigen Spitzenzellen aus der R1-Zelllinie und grüne Pfeile Spitzenzellen aus der 7ACS/EYFP-Zelllinie an. (B) Hohe Vergrößerung eines Endothelsprosses, die die Verteilung von R1- und 7ACS/EYFP-Endothelzellen entlang des Sprosses zeigt. (C) Quantifizierung der relativen Genotypisierungsspitzenzellen aus repräsentativen Wildtyp-EBs. (D) Konfokale Aufnahme eines repräsentativen 12 Tage alten EB, gefärbt auf Pecam-1 (rot, Endothelzellen) und α-SMA (grün, Wandzellen). (E) Hohe Vergrößerung eines Endothelsprossen (weißer rechteckiger gepunkteter Kasten von D), der die Interaktion zwischen Wandbild und Endothelzellen zeigt. (F-G) Bilder des farbigen Endothelsprossen und der dazugehörigen binär transformierten Bilder. (H) Binäres Bild der kolokalisierten Pecam-1- und α-SMA-Färbung. Anteil der Endothelzellsprossen, die von Wandzellen bedeckt sind. Alle Balken stellen den Mittelwert ± SEM dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Diskussion
Dieses Protokoll beschreibt einen unvoreingenommenen, robusten und reproduzierbaren 3D-EB-basierten vaskulären Sprouting-Assay, der für das Screening auf Medikamente und Gene geeignet ist, die die Angiogenese modulieren. Diese Methode bietet Vorteile gegenüber vielen weit verbreiteten zweidimensionalen (2D) Assays, bei denen Endothelzellkulturen wie Human Nabelvenen-Endothelzellen (HUVECs) zur Überwachung der Migration (Lateral Scratch Assay oder der Boyden-Kammer-Assay)22,23 oder der Proliferation (Zählen der Zellanzahl, Nachweis der DNA-Synthese, Nachweis von Proliferationsmarkern oder metabolischen Assays)24 verwendet werden Insofern ermöglicht es auf einzigartige Weise die Untersuchung sowohl der Endothel- als auch der Muralzelldifferenzierung und ihrer Organisation in einem vaskulären Netzwerk, das wichtige Schritte der keimenden Angiogenese nachahmt. Diese Schritte umfassen die Selektion der Endothelspitzenzellen, die Proliferation der Stielzellen, die räumliche Orientierung und Migration des Gefäßsprosses und die Rekrutierung von Muralzellen in das entstehende Blutgefäß25. Es bietet auch Vorteile für viele 3D-Angiogenesemodelle. Der Fibrinbead 26,27 oder der Kollagengel-Assay28,29,30, der die Tubulogenese nachahmt, verwenden üblicherweise HUVECs oder Endothelial Colony Forming Cells derived Endothelial Cells (ECFC-EC), da sie eine hohe Proliferationsrate aufweisen, aber nicht für primäre Endothelzellen der Maus geeignet sind, die in Kultur schwer zu halten sind. Der ex vivo Retina-Explantat31 oder der vaskularisierte Mikroorgan-Assay32 kann alle Schritte der Blutgefäßbildung gut rekapitulieren, aber sie haben komplexe experimentelle Verfahren und sind nicht für ein Hochdurchsatz-Arzneimittel- oder genetisches Screening geeignet. Dies gilt auch für den ex vivo Aortenring-Assay 33,34 und für viele In-vivo-Assays, wie z. B. Tumorimplantate bei Mäusen oder Funktionsverluststudien bei Mäusen, die oft hohe Kosten und Schwierigkeiten bei der Gewinnung großer Datenmengen haben 35. Dieses Protokoll ergänzt auch ähnliche In-vitro-Angiogenese-Assays mit humanen iPS-Zellen und ermöglicht Vergleiche zwischen Maus- und Humandaten. Es ist jedoch wichtig zu beachten, dass aus menschlichen iPS-Zellen gewonnene Endothelzellen eine geringere Fähigkeit zum Keimen aufweisen als die Mauszellen36,37.
Die hier entwickelte Methode hat auch einige Einschränkungen. Es kann die Auswirkungen des Flüssigkeitsflusses auf die Reifung der Blutgefäße und die Durchlässigkeit der Gefäße nicht bewerten und produziert im Vergleich zu neueren mikrofabrizierten Geräten, die sich in der Entwicklung befinden, keine entstehenden Blutgefäße in einer bestimmten Gewebeumgebung. Tatsächlich können Organ-on-Chip-Technologien, die Mikrofluidik mit Tissue Engineering kombinieren, kultivierten Endothelzellen eine Mikroumgebung bieten, die der in vivo ähnelt 38,39. Mikrofluidische Systeme enthalten die richtige Zusammensetzung der extrazellulären Matrix und sind darauf ausgelegt, mechanische Signale wie Scherspannungen zu erzeugen. Einige sind so konzipiert, dass sie Wandzellen und andere unterstützende Zellen eines bestimmten Gewebes einbeziehen oder chemische Gradienten erzeugen können. Sie enthalten Netzwerke von flüssigkeitsgefüllten Kanälen im Mikrometerbereich, die in Größe und Struktur den Blutkapillaren ähneln. Die Organ-on-Chip-Technologien ermöglichen auch die Quantifizierung spezifischer Gefäßfunktionen, einschließlich der Permeabilität und des transendothelialen elektrischen Widerstands. Obwohl Organ-on-Chip-Technologien vielversprechend sind, gehen sie weit über die Forschungskompetenz der meisten Biologielabore hinaus, müssen immer noch angemessen standardisiert werden und erfordern spezielle Herstellungstechniken. Die Kommerzialisierung der Herstellung von Organ-on-Chip-Technologie steht erst am Anfang, und diese Systeme gelten für Pharmaunternehmen derzeit als unerschwinglich40,41.
Es gibt mehrere kritische Schritte, die berücksichtigt werden sollten. Verwenden Sie qualitativ hochwertige Zellen, die gut akzeptierte Marker (Nanog, Oct4, Sox2 und SSEA-1) des pluripotenten Zustands robust exprimieren. Es ist wichtig, ihr Wachstum, die Form der mES-Zellen sowie die Größe und Morphologie der mES-Kolonien sorgfältig zu überwachen. Da die karyotypische Stabilität in kultivierten Zellen stochastisch ist, ist eine Neubewertung nach umfangreicher Passage unerlässlich. Es wird empfohlen, Produkte zu verwenden, die bereits für die mESC-Kultur getestet wurden, und MEF-Futterautomaten, fetales Kälberserum und alle chemischen Verbindungen für mehrere Passagen zu testen, um festzustellen, ob mES-Zellen ihre zellulären Eigenschaften beibehalten oder ob sie differenzieren oder einen epiblastischen Phänotyp erwerben. Das Medium sollte täglich aufgefrischt werden und mESC-Kolonien sollten nicht zu groß und dicht werden. Schließlich müssen mES-Zellen vor der Differenzierung mindestens zwei Passagen ohne 2i kultiviert werden, um die beste Ausbeute an Endothelzellen zu gewährleisten.
Die Endotheldifferenzierung umfasst zwei wichtige Schritte: die Bildung von EBs mit der Hanging-Drop-Methode und deren Kultivierung unter schwimmenden Bedingungen in Gegenwart von vaskulärem Differenzierungsmedium13,14. Die Bewegung der hängenden Tropfenschalen sollte minimiert werden, um eine gleichmäßige Zellaggregation zu erreichen. Die Anzahl der mES-Zellen, die zur Bildung eines Aggregats verwendet werden, liegt in den meisten Fällen zwischen 800-1.000 Zellen, muss jedoch möglicherweise optimiert werden, wenn mES-Zellen einen anderen genetischen Hintergrund als 129/ola haben, um eine optimale Differenzierung in vaskularisierte EBs zu gewährleisten. Wenn EBs unter schwimmenden Bedingungen kultiviert werden, müssen sie sorgfältig verteilt werden und Bewegungen vermeiden, die die EB-Verklumpung begünstigen.
EBs werden schließlich in Kollagen-I-Gel kultiviert, um Gefäßsprossen zu bilden. Angiogenes Medium sollte frisch zubereitet werden und nach dem Mischen mit Kollagen muss I auf Eis gehalten werden, um eine spontane Gelierung zu vermeiden. Im Falle von Drogentests werden in diesem Schritt Medikamente in der richtigen Konzentration in die kalte Mischung gegeben. Die Anpassung des pH-Werts mit NaOH vor der Resuspendierung der EBs ist von entscheidender Bedeutung, da sonst die Kollagensäure zu Zelltoxizität führt. Schließlich sollten EBs in gleichem Abstand zueinander verteilt werden, um reproduzierbare Ergebnisse zu gewährleisten.
Zusammenfassend lässt sich sagen, dass diese Methode einen auf mESC basierenden 3D-Gefäßkeimungstest vorstellt, der die erforderliche Robustheit und Skalierbarkeit aufweist, um für das genetische Screening verwendet zu werden, wie es kürzlich von Elling U. et al. beschrieben wurde. die eine große Haplobank von hemi/homozygoten mutierten mESC16 generierte und für ein phänotypisches Wirkstoffforschungsprogramm diente.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde durch Zuschüsse der Nederlandse organisatie voor gezondheidsonderzoek en zorginnovatie (ZonMW 446002501), Health Holland (LSHM19057-H040), des Leading Fellows Programme Marie Skłodowska-Curie COFUND und der Association Maladie de Rendu-Osler (AMRO) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Milipore, Merck | 805740 | Biohazard: adequate safety instructions should be taken when handling |
| Agar Noble | Difco, BD Pharmigen | 214220 | |
| Alexa Fluo 555 goat anti rat IgG | Life technologies | A21434 | |
| APC conjugated rat anti-mouse PECAM-1 antibody (clone MEC13.3) | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control (Clone R35-95) | BD Biosciences | 553932 | |
| Axiovert 25 inverted phase contrast tissue culture microscope | ZEISS | ||
| Basic Fibroblast Growth Factor-2 (bFGF) | Peprotech | 450-33 | |
| Benchtop Centrifuge, Allegra X-15R | Beckman Coulter | 392932 | |
| Biosafety cabinet BioVanguard (Green Line) | Telstar | 133H401001 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A9418 | |
| Cell counting chamber, Buerker, 0.100mm | Marienfeld | 640211 | |
| Cell culture dishes 60 x 15mm | Corning | 353802 | |
| Cell culture dishes, 35 x 10 mm | Corning | 353801 | |
| Cell culture plates 12-well | Corning | 3512 | |
| CFX96 Touch Real-Time PCR Detection System | Biorad | 1855196 | |
| Chicken serum | Sigma-Aldrich | C5405 | |
| CHIR-99021 (CT99021) HCl | Selleckchem | S2924 | |
| Collagen I, High Concentration, Rat Tail, 100mg | Corning | 354249 | |
| Collagenase A | Roche | 10103586001 | |
| Confocal Laser Scanning Microscope, TCS SP5 | Leica | ||
| Cover glasses, 24 × 50 mm | Vwr | 631-0146 | |
| DAPT γ?secretase inhibitor | Sigma Aldrich | D5942 | |
| DC101 anti mouse VEGFR-2 Clone | BioXcell | BP0060 | |
| DC101 isotype rat IgG1 | BioXcell | BP0290 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2438-5X | Biohazard: adequate safety instructions should be taken when handling |
| DPBS (10x), no calcium, no magnesium | Gibco, Thermofisher scientific | 14200067 | |
| EDTA 40 mM | Gibco, Thermofisher scientific | 15575-038 | |
| Embryonic stem-cell Fetal Bovine Serum | Gibco, Thermofisher scientific | 16141-079 | Should be lot-tested for maximum ES cell viability and growth. Heat inactivate at 60°C and store at −20 °C for up to 1 year |
| Eppendorf Microcentrifuge 5415R | Eppendorf AG | Z605212 | |
| Erythropoietin, human (hEPO), 250 U (2.5 µg) (1 mL) | Roche | 11120166001 | |
| ESGRO Recombinant Mouse LIF Protein (10? units 1 mL) | Milipore, Merck | ESG1107 | |
| Falcon tubes 15 mL | Greiner Bio-One | 188271 | |
| Falcon tubes 50 mL | Greiner Bio-0ne | 227270 | |
| Filter tip ,clear ,sterile F.Gilson, P-200 | Greiner Bio-One | 739288 | |
| Filter tip ,clear ,sterile F.Gilson, P10 | Greiner Bio-One | 771288 | |
| Filter tip ,clear ,sterile F.Gilson, P1000 | Greiner Bio-One | 740288 | |
| FITC conjugated anti-α Smooth Muscle Actin (SMA) (clone 1A4) | Sigma Aldrich | F3777 | |
| FITC conjugated rat anti-mouse CD45 (clone 30-F11) | Biolegend | 103107 | |
| FITC Rat IgG2b, κ Isotype Ctrl Antibody (clone RTK4530) | Biolegend | 400605 | |
| Fluorscent mounting media | DAKO | S3023 | |
| Gascompress | Cutisoft | 45846 | |
| Gauze Cutisoft 10 x 10 cm | Bsn Medical | 45844_00 | |
| Gel blotting paper, Grade GB003 | Whatman | WHA10547922 | |
| Gelatin solution, type B | Sigma-Aldrich | G1393-100 ml | |
| Glasgow's MEM (GMEM) | Gibco, Thermofisher scientific | 21710082 | |
| IHC Zinc Fixative | BD Pharmigen | 550523 | |
| IncuSafe CO2 Incubator | PHCBi | MCO-170AICUV-PE | |
| Interleukin-6, human (hIL-6) | Roche | 11138600001 | |
| L-Glutamine 200 mM | Gibco, Thermofisher scientific | 25030-024 | |
| MEM Non-Essential Amino Acids Solution (100x) | Gibco, Thermofisher scientific | 11140035 | |
| Microscope slide box | Kartell Labware | 278 | |
| Microscope slide, Starfrost | Knittel glass | VS113711FKB.0 | |
| Mm_Cdh5_1_SG QuantiTect Primer Assay | Qiagen | QT00110467 | |
| Mm_Eng_1_SG QuantiTect Primer Assay | Qiagen | QT00148981 | |
| Mm_Epha4_1_SG QuantiTect Primer Assay | Qiagen | QT00093576 | |
| Mm_Ephb2_1_SG QuantiTect Primer Assay | Qiagen | QT00154014 | |
| Mm_Flt1_1_SG QuantiTect Primer Assay | Qiagen | QT00096292 | |
| Mm_Flt4_1_SG QuantiTect Primer Assay | Qiagen | QT00099064 | |
| Mm_Gapdh_3_SG QuantiTect Primer Assay | Qiagen | QT01658692 | |
| Mm_Kdr_1_SG QuantiTect Primer Assay | Qiagen | QT00097020 | |
| Mm_Notch1_1_SG QuantiTect Primer | Qiagen | QT00156982 | |
| Mm_Nr2f2_1_SG QuantiTect Primer Assay | Qiagen | QT00153104 | |
| Mm_Pecam1_1_SG QuantiTect Primer | Qiagen | QT01052044 | |
| Mm_Tek_1_SG QuantiTect Primer Assay | Qiagen | QT00114576 | |
| Mouse (ICR) Inactivated Embryonic Fibroblasts (2 M) | Gibco, Thermofisher scientific | A24903 | Store vials in liquid nitrogen (195.79 °C) indefinitely |
| Mouse embryonic stem cell line 7AC5/EYFP (ATCC SCRC-1033) | ATCC | SCRC-1033 | Generated by Dr A Nagy, Samuel Lunenfeld Research Institute, Mount Sinai Hospital, 600 University Ave, Toronto, Ontario, M5G 1X5, Canada. [Hadjantonakis, A. K., et al. Mechanisms of Development. 76 (1–2), 79–90 (1998)]. |
| Mouse embryonic stem cell lines Acvrl1 +/- and Acvrl1 +/+ | Generated at Leiden University Medical Centre [Thalgott, J.H. et al. Circulation. 138 (23), 2698–2712 (2018)]. | ||
| Mouse embryonic stem cells line E14 | Provided by M Letarte laboratory and generated according to Cho, S. K., et al. Blood. 98 (13), 3635–3642 (2001). | ||
| Mouse embryonic stem cells line R1 (ATCC SCRC-1011) | ATCC | SCRC-1011 | Generated by Dr A Nagy, Samuel Lunenfeld Research Institute, Mount Sinai Hospital, 600 University Ave, Toronto, Ontario, M5G 1X5, Canada. [Nagy, A., et al. Procedings of the National Academy of Sciences of the United States of America. 90 (18), 8424–8428 (1993)]. |
| Mouse embryonic stem cells line Z/Red (strain 129/Ola) | Generated by Dr A Nagy, Samuel Lunenfeld Research Institute, Mount Sinai Hospital, 600 University Ave, Toronto, Ontario, M5G 1X5, Canada [Vintersten, K., et al. Genesis. 40 (4), 241–246 (2004)]. | ||
| NanoDrop 1000 UV/VIS Spectrophotometer | Thermo Fischer Scientific | ND-1000 | |
| PD0325901 | Selleckchem | S1036 | |
| PDGF-BB, Recombinant Human | Peprotech | 100-14B | |
| Pecam-1 antibody, Rat Anti-Mouse | BD Biosciences | 550274 | |
| Penicillin-streptomycin (10,000 U/mL) | Gibco, Thermofisher scientific | 15140122 | |
| Petri dish, PS, 94/16 mm, standard ,with vents, sterile | Greiner Bio-One | 633181 | |
| Pipetboy acu 2 | Integra-Biosciences | 155 019 | |
| Pipetman G Multichannel P8 x 200G | Gilson | F144072 | |
| Pipetman G Starter Kit, 4 Pipette Kit, P2G, P20G, P200G, P1000G | Gilson | F167360 | |
| Recombinant Human BMP-4 Protein | R&D Systems | 314-BP | |
| RNeasy Plus mini Kit | QIAGEN | 74134 | |
| Serological pipettes, 10 mL | Greiner Bio-One | 607 180 | |
| Serological pipettes, 25 mL | Greiner Bio-One | 760 180 | |
| Serological pipettes, 5 mL | Greiner Bio-One | 606 180 | |
| Sodium hydroxide (NaOH) | Merck | 106498 | |
| Sodium pyruvate 100 mM | Gibco, Thermofisher scientific | 11360039 | |
| Test tubes 5ml round-bottom with cell-strainer cap | Corning | 352235 | |
| Thermal cycler, T100 | Biorad | 1861096 | |
| Triton X-100 (BioXtra) | Sigma Aldrich | T9284 | |
| Trypan Blue Solution, 0.4% | Gibco, Thermofisher scientific | 15250061 | |
| Trypsin (2.5%) | Gibco, Thermofisher scientific | 15090046 | |
| Vacuum Filter/Storage Bottle System, 500 mL | Corning | 430758 | |
| VEGFA165 , recombinant murine | Peprotech | 450-32 | |
| Water, Sterile | Fresenius-Kabi | B230531 | |
| Waterbath, Lab-Line Digital | Thermo Fischer Scientific | 18052A |
Referenzen
- Moffat, J. G., Vincent, F., Lee, J. A., Eder, J., Prunotto, M. Opportunities and challenges in phenotypic drug discovery: an industry perspective. Nature Reviews Drug Discovery. 16 (8), 531-543 (2017).
- Horvath, P., et al. Screening out irrelevant cell-based models of disease. Nature Reviews. Drug Discovery. 15 (11), 751-769 (2016).
- Low, L. A., Mummery, C., Berridge, B. R., Austin, C. P., Tagle, D. A. Organs-on-chips: into the next decade. Nature Reviews. Drug Discovery. , (2020).
- Ma, C., Peng, Y., Li, H., Chen, W. Organ-on-a-Chip: A new paradigm for drug development. Trends in Pharmacological Sciences. 42 (2), 119-133 (2021).
- Swinney, D. C., Anthony, J. How were new medicines discovered. Nature Reviews Drug Discovery. 10 (7), 507-519 (2011).
- Hussain, S., et al. High-content image generation for drug discovery using generative adversarial networks. Neural Networks: The Official Journal of the INternational Neural Network Society. 132, 353-363 (2020).
- Scheeder, C., Heigwer, F., Boutros, M. Machine learning and image-based profiling in drug discovery. Current Opinion in Systems Biology. 10, 43-52 (2018).
- Wagner, B. K., Schreiber, S. L. The power of sophisticated phenotypic screening and modern mechanism-of-action methods. Cell Chemical Biology. 23 (1), 3-9 (2016).
- Scannell, J. W., Bosley, J. When quality beats quantity: Decision theory, drug discovery, and the reproducibility crisis. PLoS One. 11 (2), 0147215 (2016).
- Webster, J. D., Santagostino, S. F., Foreman, O. Applications and considerations for the use of genetically engineered mouse models in drug development. Cell and Tissue Research. 380 (2), 325-340 (2020).
- Howland, D. S., Munoz-Sanjuan, I. Mind the gap: models in multiple species needed for therapeutic development in Huntington's disease. Movement Disorders: Official Journal of the Movement Disorder Scoiety. 29 (11), 1397-1403 (2014).
- Galaris, G., Thalgott, J. H., Lebrin, F. P. G. Pericytes in hereditary hemorrhagic telangiectasia. Advances in Experimental Medicine and Biology. 1147, 215-246 (2019).
- Thalgott, J. H., et al. Decreased expression of vascular endothelial growth factor receptor 1 contributes to the pathogenesis of hereditary hemorrhagic telangiectasia type 2. Circulation. 138 (23), 2698-2712 (2018).
- Lebrin, F., et al. Thalidomide stimulates vessel maturation and reduces epistaxis in individuals with hereditary hemorrhagic telangiectasia. Nature Medicine. 16 (4), 420-428 (2010).
- Czechanski, A., et al. Derivation and characterization of mouse embryonic stem cells from permissive and nonpermissive strains. Nature Protocols. 9 (3), 559-574 (2014).
- Elling, U., et al. A reversible haploid mouse embryonic stem cell biobank resource for functional genomics. Nature. 550 (7674), 114-118 (2017).
- Cheng, J., et al. Targeting pericytes for therapeutic approaches to neurological disorders. Acta Neuropathologica. 136 (4), 507-523 (2018).
- Chade, A. R. Small vessels, big role: Renal microcirculation and progression of renal injury. Hypertension. 69 (4), 551-563 (2017).
- Jourde-Chiche, N., et al. Endothelium structure and function in kidney health and disease. Nature Reviews. Nephrology. 15 (2), 87-108 (2019).
- van Duinen, V., et al. Standardized and scalable assay to study perfused 3D angiogenic sprouting of iPSC-derived endothelial cells in vitro. Journal of Visualized Experiment: JoVE. (153), e59678 (2019).
- Chappell, J. C., Taylor, S. M., Ferrara, N., Bautch, V. L. Local guidance of emerging vessel sprouts requires soluble Flt-1. Developmental Cell. 17 (3), 377-386 (2009).
- Sato, Y., Rifkin, D. B. Inhibition of endothelial cell movement by pericytes and smooth muscle cells: activation of a latent transforming growth factor-beta 1-like molecule by plasmin during co-culture. Journal of Cell Biology. 109 (1), 309-315 (1989).
- Tchaikovski, V., Olieslagers, S., Bohmer, F. D., Waltenberger, J. Diabetes mellitus activates signal transduction pathways resulting in vascular endothelial growth factor resistance of human monocytes. Circulation. 120 (2), 150-159 (2009).
- Staton, C. A., Reed, M. W., Brown, N. J. A critical analysis of current in vitro and in vivo angiogenesis assays. International Journal of Experimental Pathology. 90 (3), 195-221 (2009).
- Herbert, S. P., Stainier, D. Y. Molecular control of endothelial cell behaviour during blood vessel morphogenesis. Nature Reviews Molecular Cell Biology. 12 (9), 551-564 (2011).
- Nakatsu, M. N., Hughes, C. C. An optimized three-dimensional in vitro model for the analysis of angiogenesis. Methods in Enzymology. 443, 65-82 (2008).
- Nakatsu, M. N., Davis, J., Hughes, C. C. Optimized fibrin gel bead assay for the study of angiogenesis. Journal of Visualized Experiments: JoVE. (186), (2007).
- Gau, D., et al. Pharmacological intervention of MKL/SRF signaling by CCG-1423 impedes endothelial cell migration and angiogenesis. Angiogenesis. 20 (4), 663-672 (2017).
- Torres-Estay, V., et al. Androgens modulate male-derived endothelial cell homeostasis using androgen receptor-dependent and receptor-independent mechanisms. Angiogenesis. 20 (1), 25-38 (2017).
- Merjaneh, M., et al. Pro-angiogenic capacities of microvesicles produced by skin wound myofibroblasts. Angiogenesis. 20 (3), 385-398 (2017).
- Rezzola, S., et al. In vitro and ex vivo retina angiogenesis assays. Angiogenesis. 17 (3), 429-442 (2014).
- Wang, X., Phan, D. T. T., George, S. C., Hughes, C. C. W., Lee, A. P. 3D Anastomosed microvascular network model with living capillary networks and endothelial cell-lined microfluidic channels. Methods in Molecular Biology. 1612, 325-344 (2017).
- Nicosia, R. F., Ottinetti, A. Growth of microvessels in serum-free matrix culture of rat aorta. A quantitative assay of angiogenesis in vitro. Laboratory Investigation; A Journal of Technical Methods and Pathology. 63 (1), 115-122 (1990).
- Nicosia, R. F. The aortic ring model of angiogenesis: a quarter century of search and discovery. Journal of Cellular and Molecular Medicine. 13 (10), 4113-4136 (2009).
- Nowak-Sliwinska, P., et al. Consensus guidelines for the use and interpretation of angiogenesis assays. Angiogenesis. 21 (3), 425 (2018).
- Belair, D. G., Schwartz, M. P., Knudsen, T., Murphy, W. L. Human iPSC-derived endothelial cell sprouting assay in synthetic hydrogel arrays. Acta Biomaterialia. 39, 12-24 (2016).
- Bezenah, J. R., Kong, Y. P., Putnam, A. J. Evaluating the potential of endothelial cells derived from human induced pluripotent stem cells to form microvascular networks in 3D cultures. Scientific Reports. 8 (1), 2671 (2018).
- Henderson, A. R., Choi, H., Lee, E. Blood and lymphatic vasculatures on-chip platforms and their applications for organ-specific in vitro modeling. Micromachines (Basel). 11 (2), 147 (2020).
- Lin, D. S. Y., Guo, F., Zhang, B. Modeling organ-specific vasculature with organ-on-a-chip devices. Nanotechnology. 30 (2), 024002 (2019).
- Pollet, A., den Toonder, J. M. J. Recapitulating the vasculature using organ-on-chip technology. Bioengineering. 7 (1), 17 (2020).
- Cochrane, A., et al. Advanced in vitro models of vascular biology: Human induced pluripotent stem cells and organ-on-chip technology. Advanced Drug Delivery Reviews. 140, 68-77 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten