Method Article
Organotypische Schnittkulturen als präklinische Modelle der Tumormikroumgebung bei primärem Pankreaskarzinom und Metastasierung
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Aufbereitung von organotypischen Schnittkulturen (OTSCs). Diese Technik ermöglicht die ex vivo-Kultivierung von intaktem mehrzelligem Gewebe. OTSCs können sofort eingesetzt werden, um ihr jeweiliges Ansprechen auf Medikamente in einer multizellulären Umgebung zu testen.
Zusammenfassung
Realitätsnahe präklinische Modelle des primären Pankreaskarzinoms und der Metastasierung werden dringend benötigt, um das Therapieansprechen ex vivo zu testen und eine personalisierte Behandlung der Patienten zu ermöglichen. Das Fehlen einer tumorspezifischen Mikroumgebung in den derzeit verwendeten Modellen, z.B. bei patienteneigenen Zelllinien und Xenotransplantaten, erlaubt jedoch nur begrenzte prädiktive Erkenntnisse. Organotypische Schnittkulturen (OTSCs) bestehen aus intaktem mehrzelligem Gewebe, das schnell für die ortsaufgelöste Wirkstoffreaktionsprüfung verwendet werden kann.
Dieses Protokoll beschreibt die Erzeugung und Kultivierung lebensfähiger Tumorschnitte des Bauchspeicheldrüsenkarzinoms und dessen Metastasierung. Kurz gesagt, das Gewebe wird in Low Melt Agarose gegossen und in kaltem isotonischem Puffer gelagert. Anschließend werden Gewebeschnitte von 300 μm Dicke mit einem Vibratom erzeugt. Nach der Aufbereitung werden die Scheiben an einer Luft-Flüssig-Grenzfläche unter Verwendung von Zellkultureinsätzen und einem geeigneten Kultivierungsmedium kultiviert. Während der Kultivierung können Veränderungen der Zelldifferenzierung und der Lebensfähigkeit überwacht werden. Darüber hinaus ermöglicht diese Technik die Anwendung der Behandlung auf lebensfähiges menschliches Tumorgewebe ex vivo und nachfolgende nachgelagerte Analysen, wie z. B. Transkriptom- und Proteom-Profiling.
OTSCs bieten eine einzigartige Möglichkeit, das individuelle Ansprechen auf die Behandlung ex vivo zu testen und individuelle transkriptomische und proteomische Profile zu identifizieren, die mit dem jeweiligen Ansprechen verschiedener Schnitte eines Tumors verbunden sind. OTSCs können weiter erforscht werden, um therapeutische Strategien zur Personalisierung der Behandlung von primärem Bauchspeicheldrüsenkrebs und Metastasen zu identifizieren.
Einleitung
Bestehende präklinische Modelle des duktalen Adenokarzinoms der Bauchspeicheldrüse (PDAC) und der entsprechenden Metastasen sind schlechte Prädiktoren für das Ansprechen auf die Behandlung bei Patienten, was ein großer Nachteil bei der Arzneimittelentwicklung und der Identifizierung prädiktiver Biomarker darstellt1. Obwohl Modelle wie patienteneigene Organoide und patienteneigene Xenotransplantate vielversprechend sind, ist ihr Einsatz nach wie vor begrenzt2. Die Haupteinschränkungen dieser In-vitro-Modelle sind das Fehlen der Tumormikroumgebung und die Xenotransplantation bei nicht-humanen immungeschwächten Spezies. Insbesondere bei PDAC und seinen Metastasen hat die Tumormikroumgebung in den letzten Jahren aufgrund ihrer entscheidenden Funktionen in der Tumorbiologie erheblich an Interesse gewonnen. Es besteht aus zellulären und azellulären Komponenten, wie z.B. (Myo-)Fibroblasten, Pankreas-Sternzellen, Immunzellen, Blutgefäßen, extrazellulärer Matrix, Zytokinen und Wachstumsfaktoren3. Diese Mikroumgebung ist keine nicht-funktionelle Tumorkomponente, sondern induziert die Tumorprogression und Metastasierung und scheint wesentlich zur Strahlen- und Chemotherapieresistenz beizutragen4. Die PDAC-Mikroumgebung beeinträchtigt nicht nur mechanisch die Wirkstoffabgabe, sondern besitzt auch eine Immun- und Wirkstofffängeraktivität 5,6,7. Daher werden präklinische Modelle, die das komplexe Zusammenspiel von Tumorzellen und der Tumormikroumgebung widerspiegeln, dringend benötigt, um das Ansprechen der Patienten auf die Behandlung ex vivo angemessen zu testen und eine individualisierte klinische Behandlung zu leiten.
Ex-vivo-Kulturen frischer Tumorproben stellen eine große Annäherung an den Tumor in situ dar. Organotypische Schnittkulturen (OTSCs) wurden kürzlich entwickelt und für verschiedene Tumoren wie Kopf-, Hals-, Brust-, Prostata-, Lungen-, Dickdarm- und Bauchspeicheldrüsenkrebs untersucht 8,9,10,11,12. Es wurde gezeigt, dass OTSCs ihre Ausgangsmorphologie, proliferative Aktivität und Mikroumgebung während der Kultivierung für einen definierten, gewebeabhängigen Zeitraum beibehalten 11,12,13. OTSCs von PDACs behielten ihre Lebensfähigkeit, Morphologie und die meisten Bestandteile ihrer Tumormikroumgebung für 4-9 Tage in mehreren In-vitro-Studien bei 5,12,14. Perspektivisch ermöglicht diese Technik eine unmittelbare Anwendung der Behandlung auf lebensfähiges menschliches Tumorgewebe ex vivo und nachfolgende Analysen, wie z. B. die Profilerstellung des Transkriptoms und des Proteoms.
Die Etablierung von OTSCs bietet eine einzigartige Möglichkeit, das Ansprechen auf die Behandlung ex vivo zeitnah nach der Operation zu testen. Auf diese Weise werden OTSCs prospektiv die Identifizierung therapeutischer Strategien zur Personalisierung der Behandlung von metastasierten Erkrankungen ermöglichen. Dieses Protokoll beschreibt die Generierung und Kultivierung lebensfähiger OTSCs für Bauchspeicheldrüsenkrebs.
Protokoll
Die Gewebeproben wurden nach Genehmigung durch die örtliche Ethikkommission der Universität zu Lübeck entnommen und verarbeitet (# 16-281).
1. Entnahme und Handhabung von frischem Gewebe
HINWEIS: Jede nicht fixierte menschliche Gewebeprobe sollte mit Vorsicht behandelt werden, um das Risiko einer Infektion durch durch Blut übertragbare Krankheitserreger zu vermeiden. Alle Patienten sollten vor der Gewebeaufbereitung auf HIV, HBV und HCV negativ getestet werden. Tragen Sie einen Schutzmantel und fassen Sie menschliche Gewebeproben mit Handschuhen an.
- Entnehmen Sie unmittelbar nach der Operation frische, unfixierte und ungefrorene PDAC-Gewebeproben mit einer Mindestgröße von 0,4 x 0,4 cm und transportieren Sie die Probe in einer Gewebeaufbewahrungslösung ins Labor.
- Wenn möglich, das frische Tuch sofort verarbeiten.
- Alternativ können Sie das Taschentuch über Nacht in einer Aufbewahrungslösung auf nassem Eis bei 4 °C lagern. Die Lagerung von Gewebe kann jedoch zu einer Beeinträchtigung der Lebensfähigkeit führen und sollte im Allgemeinen vermieden werden.
2. Vorbereitung
- Niedrigschmelzende Agarosezubereitung
- Bereiten Sie 100 mL niedrigschmelzende Agarose (8%) vor, indem Sie 8 g Agarose in 100 mL vorgewärmter Ringer-Lösung auflösen und bis zur Verwendung bei 4 °C lagern.
- Nach Ankündigung der Tumorresektion die Agarose in der Mikrowelle schmelzen.
- Legen Sie die Agarose in ein vorgewärmtes Wasserbad (37 °C) und lassen Sie sie vor der Zubereitung auf physiologische Temperaturen abkühlen.
- Vibratome-Setup
- Setzen Sie eine Rasierklinge in die Halterung des Vibratoms ein und führen Sie ggf. eine automatische Winkelverstellung nach Herstellerangaben durch.
- Kühlen Sie den Mantel der Schneidkammer mit einem Kühlaggregat oder nassem Eis ab.
- Füllen Sie die Schneidkammer mit ca. 100 mL einer physiologischen Schneidlösung (z. B. Ringer-Lösung).
- Legen Sie die montierte Rasierklinge in die vorgekühlte Schneidlösung und lassen Sie die Rasierklinge abkühlen.
3. Einbettung von Gewebe in niedrigschmelzende Agarose
- Waschen Sie die Gewebeprobe mit gekühltem (4 °C) PBS und legen Sie das Gewebe in PBS auf eine große (~14 cm) Petrischale auf Eis.
- Makroskopisch sichtbares überschüssiges Bindegewebe auf Eis mit einem Skalpell entfernen, da dies die Schneidleistung beeinträchtigen könnte.
- Legen Sie das Taschentuch in eine kleine Petrischale (~3 cm).
- Passen Sie die Gewebeausrichtung so an, dass das verbleibende makroskopisch sichtbare Bindegewebe die gleiche Ausrichtung hat wie die Ebene des Bodens der Petrischale. Der Boden der Petrischale hat die gleiche Ausrichtung wie die Schnittebene.
- Die vorbereitete niedrigschmelzende Agarose in die kleine Petrischale geben.
- Passen Sie die Ausrichtung des Gewebes bei Bedarf mit einer Pinzette an.
- Stellen Sie die Petrischale auf nasses Eis, um die Agarose schneller auszuhärten.
- Schneiden Sie das Gewebe vorsichtig mit einem Skalpell und lassen Sie auf jeder Seite des Gewebes mindestens 5 mm Agarose umgeben.
- Übertragen Sie das eingebettete Gewebe vorsichtig und kleben Sie es mit Sekundenkleber auf den Probenhalter.
- Setzen Sie nach einigen Sekunden den Probenhalter in die Schneidkammer ein.
- Passen Sie bei Bedarf die Ausrichtung des Taschentuchs in Richtung der Rasierklinge an.
4. Schneiden des in Agarose eingebetteten Gewebes mit einem Vibratom
- Definieren Sie die äußeren Grenzen des Schnittbereichs (y-Achse) entsprechend der Größe der Gewebeprobe.
- Richten Sie die Klinge nach oben auf den Gewebeblock aus.
- Stellen Sie die Schnittgeschwindigkeit auf 0,04 mm/s, die Schnittamplitude auf 1 mm und die Scheibendicke auf 300 μm ein.
- Schneiden Sie die ersten Scheiben vorsichtig ab und geben Sie die Scheiben in ein separates Gefäß mit vorgekühlter (4 °C) Schneidlösung auf nassem Eis.
5. Kultur organotypischer Scheibenkulturen
- Bereiten Sie eine 6-Well-Platte mit 1 mL des entsprechenden Kultivierungsmediums pro Well vor.
- Medium A: Fortgeschrittenes DMEM/F12, 10 % FBS, 1 % Penicillin/Streptomycin.
- Medium B: RPMI 1640, 10 % FBS, 1 % Penicillin/Streptomycin, 4 μg/ml Insulin, 8 ng/ml EGF, 0,3 μg/ml Hydrocortison.
HINWEIS: Das Medium für optimale Kulturbedingungen kann je nach Gewebe und Patient variieren. Es wurden zwei verschiedene Gewebekulturmedien verglichen, eines auf Basis von DMEM/F12 (Medium A), das zweite auf Basis von RPMI (Medium B). Es wurden keine wesentlichen Unterschiede zwischen diesen Medien festgestellt. Für alle in diesem Protokoll gezeigten Versuche wurde Medium A verwendet.
- Legen Sie die 6-Well-Platte mit dem Medium in einen Inkubator und lassen Sie die Temperatur und den pH-Wert vor der Kultivierung anpassen.
- Platzieren Sie die Scheiben mit einem Blickfilter auf Zellkultureinsätzen (z. B. hydrophile PTFE-Einsätze mit 0,4 μm Porengröße).
- Entfernen Sie überschüssige Schneidlösung, indem Sie den geladenen Filter auf ein steriles Tuch legen.
- Legen Sie den geladenen Filter in die vorbereitete 6-Well-Platte. Fügen Sie der Einlage kein zusätzliches Medium hinzu.
- Stellen Sie die 6-Well-Platte in einen Inkubator (37 °C, 5 % CO2).
- Wechseln Sie das Medium alle 2 Tage, indem Sie die Schritte 5.1, 5.2 und 5.5 mit einer neuen 6-Well-Platte wiederholen.
HINWEIS: Organotypische Schnittkulturen können je nach individueller Forschungsfrage über verschiedene Zeiträume kultiviert werden.
6. Viabilitätstest von Resazurin
HINWEIS: Der Resazurin-Viabilitätsassay misst die allgemeine metabolische Aktivität der organotypischen Schnittkulturen basierend auf der Reduktion von nicht fluoreszierendem blauem Resazurin zu rot fluoreszierendem Resorufin in lebenden Zellen15. Der Assay hat keine toxischen Wirkungen auf Zellen und kann je nach individueller Fragestellung wiederholt auf die Kulturen angewendet werden. Die Lebensfähigkeit wurde alle 2-3 Tage mit dem Resazurin-Assay gemessen.
- Herstellung von Resazurin-Stammlösung
- Schalten Sie das Licht der sterilen Haube aus, da Resazurin-Stammlösung lichtempfindlich ist.
- Bereiten Sie eine Stammlösung mit 10 mg/ml Resazurin-Natriumsalz in 1x PBS vor.
- Bewahren Sie die Stammlösung bis zur Verwendung in lichtgeschützten Aliquoten bei 4 °C im Kühlschrank auf.
- Bewertung der allgemeinen Lebensfähigkeit der Scheibe
- Schalten Sie das Licht der sterilen Haube aus, da Resazurin-Stammlösung lichtempfindlich ist.
- Die Resazurin-Stammlösung 1:250 mit einem geeigneten Medium verdünnen.
HINWEIS: Das für die Verdünnung verwendete Medium sollte das gleiche sein wie für die Kultivierung (Medium A oder Medium B). - Bereiten Sie 1 ml der endgültigen Resazurin-Lösung pro Scheibe vor und fügen Sie weitere 1 ml für die Blindkontrolle hinzu, z. B. verdünnen Sie für 6 Scheiben 28 μl Resazurin-Stammlösung in 7 mL Medium.
- Geben Sie die Resazurin-Lösung in 6-Well-Platten mit 1 ml der verdünnten Resazurin-Lösung pro Vertiefung ab.
- Übertragen Sie die Kultivierungsfilter mit den Scheiben in die Vertiefungen mit der Resazurin-Lösung. Eine Vertiefung mit der Resazurin-Lösung wird als Blindkontrolle leer gehalten.
HINWEIS: Um das experimentelle Verfahren zu vereinfachen, kann dieser Schritt mit dem Wechsel des Mediums kombiniert werden (Schritt 5.7). Falls jedoch zusätzliche Viabilitätsmessungen erforderlich sind, kann der Assay jederzeit während der Kultivierung der Schnittkulturen durchgeführt werden. - Legen Sie die Gewebescheiben für 1 h bei 37 °C und 5 % CO2 in einen Inkubator.
- Bereiten Sie neue 6-Well-Platten mit dem Kulturmedium vor, wenn die Schnittkultur fortgesetzt wird (siehe Schritte 5.1 und 5.2).
- Entfernen Sie die Kultivierungsfilter mit den Scheiben aus der Resazurin-Lösung und entfernen Sie die überschüssige Lösung, indem Sie den geladenen Filter auf ein steriles Tuch legen.
- Übertragen Sie die Kultivierungsfilter mit den Scheiben auf die zuvor vorbereitete Kulturplatte.
- Entnehmen Sie aus jeder 6-Well-Platte 100 μl Resazurin-Lösung und geben Sie sie auf eine 96-Well-Platte. Platzieren Sie bei der Blindkontrolle drei Proben (3 x 100 μl) in separaten Vertiefungen der 96-Well-Platte.
- Quantifizieren Sie die Extinktion mit einem Plattenphotometer gemäß den Anweisungen des Herstellers. Die Anregungswellenlänge ist auf 545 nm und die Emissionswellenlänge auf 600 nm eingestellt.
7. Formalinfixierung und Paraffineinbettung von OTSCs
- Übertragen Sie die kultivierten Gewebescheiben vorsichtig in eine Einbettkassette aus Kunststoff. Führen Sie dazu die folgenden Schritte aus.
- Platzieren Sie den Kultivierungsfilter mit der montierten Scheibe auf einer Petrischale.
- Schneiden Sie mit einem Skalpell die Filtermembran vorsichtig mit der montierten Gewebescheibe aus.
- Übertragen Sie die Filtermembran mit der montierten Scheibe vorsichtig in einen Biopsie-Nylonbeutel und legen Sie ihn in eine Einbettkassette.
- Übertragen Sie anschließend die Einbettkassetten aus Kunststoff in einen Behälter mit vorgekühltem (4 °C) 4,5 % Formalin. Die Scheiben können bis zur weiteren Verwendung in Formalinlösung bei 4 °C aufbewahrt werden, sollten jedoch mindestens 24 Stunden lang inkubiert werden.
HINWEIS: OTSCs müssen mit großer Vorsicht übertragen werden, da sie leicht auseinanderreißen.
- Spülen Sie die formalinfixierte Schnittkultur vorsichtig 1,5 h lang mit fließendem Leitungswasser aus.
- Dehydrieren Sie die formalinfixierte Gewebescheibe durch Inkubation in 70% Ethanol (2x für 3 h), 95% Ethanol (1x für Nacht, 1x für 3 h), gefolgt von absolutem Ethanol (1x für 3 h, 1x für nacht).
- Reinigen Sie die Formalin-fixierte Gewebescheibe durch zweimalige 3-stündige Inkubation in Xylol.
- Tauchen Sie das Gewebe bei 60 °C (1x für über Nacht, 1x für 2 h) in Paraffin. Betten Sie das Gewebe in einen Paraffinblock in eine Gewebeeinbettungsform ein.
- Schneiden Sie den in Paraffin eingebetteten Gewebeblock in 4 μm Dicke mit einem Mikrotom und schwimmen Sie in einem 40 °C warmen Wasserbad mit destilliertem Wasser. Übertragen Sie die Ausschnitte auf Objektträger.
- Paraffinabschnitte 1 h bei 60 °C inkubieren, um das Gewebe mit dem Glas zu verbinden. Inkubieren Sie die Objektträger über Nacht bei 37 °C.
8. Hämatoxylin- und Eosin-Färbung (H&E)
- Schnitte aus Schritt 7.7 durch Inkubation in Xylol (3x für 5 min) entparaffinieren.
- Rehydrieren Sie durch Inkubation in absolutem Alkohol (2x für 5 min), 95% Alkohol (2x für 5 min) und 70% Alkohol (1x für 5 min). Kurz mit destilliertem Wasser nachspülen.
- In Mayer Hämatoxylin-Lösung 5 min einfärben. 10 Min. mit fließendem Leitungswasser nachspülen.
- Gegenfärbung in 0,5%iger Eosin-Lösung für 40 s. Mit destilliertem Wasser abspülen.
- Dehydrieren durch Inkubation in 70%igem Alkohol (maximal 1 min), 95%igem Ethanol (2x für 3 min) bzw. absolutem Alkohol (2x für 3 min).
- Klären Sie in drei Xylolwechseln (jeweils einige Sekunden). Geben Sie einen Tropfen Montagemedium auf und decken Sie die Objektträger mit einem Deckglas ab.
9. Immunhistochemie von OTSCs
- Entparaffinisieren Sie die Schnitte durch Inkubation in Xylol 2x für 10 min, gefolgt von 1:1 Ethanol/Xylol für 10 min.
- Übertragen Sie die Objektträger auf 100% Ethanol (2x für 3 min), 96% Ethanol (2x für 3 min), 70% Ethanol (1x für 3 min) und dann auf 50% Ethanol (1x für 3 min).
- Führen Sie eine Antigen-Retrieval durch, um das antigene Epitop zu entlarven. Die Objektträger in der Mikrowelle in Citratpuffer 5 min bei 900 W erhitzen, anschließend 2x für 8 min bei 600 W. Die Objektträger 20 min auf Raumtemperatur abkühlen lassen.
- 3x in PBS 3x für 3 min auf einem Shaker waschen.
- Führen Sie die Permeabilisierung der Zellmembranen durch Inkubation in 0,1 % Triton X-100 in PBS (200 μl pro Objektträger) in einer befeuchteten Kammer bei Raumtemperatur für 10 Minuten durch.
- Waschen Sie in PBS für drei Wechsel à 3 min auf dem Shaker.
- Inkubieren Sie Schnitte mit 3%iger H2O2-Lösung in Methanol (200 μl pro Objektträger) in einer befeuchteten Kammer bei Raumtemperatur für 10 Minuten, um die endogene Peroxidaseaktivität zu blockieren.
- 3x in PBS 3x für 3 min auf einem Shaker waschen.
- 200 μl Blockierungspuffer (1:50 Pferdeserum in PBS) zugeben und in einer befeuchteten Kammer bei Raumtemperatur 25 Minuten lang inkubieren.
- Lassen Sie den blockierenden Puffer von den Objektträgern ab, indem Sie den Objektträger auf ein Papiertaschentuch kippen.
- 200 μl angemessen verdünnter Primärantikörper in Antikörperverdünnungsmittel auf die Objektträger geben und in einer befeuchteten Kammer bei 4 °C über Nacht inkubieren. Als Negativkontrolle ist eine geeignete Isoform von Maus-Immunglobulinen in der gleichen Verdünnung wie der Primärantikörper zu verwenden.
- 3x 3x für 3 min in PBS auf einem Shaker waschen.
- 200 μl biotinylierter Sekundärantikörper (1:50 Lösung in PBS) auf die Objektträger geben und in einer befeuchteten Kammer bei Raumtemperatur für 30 min inkubieren.
- 3x 3x für 3 min in PBS auf einem Shaker waschen.
- Bereiten Sie den Avidin/Biotin-basierten Peroxidase-Komplex vor der Anwendung gemäß den Anweisungen des Herstellers vor. 200 μl Avidin-Biotin-Peroxidase-Komplex auf die Objektträger geben und in einer befeuchteten Kammer bei Raumtemperatur 30 min inkubieren.
- 3x 3x für 3 min in PBS auf einem Shaker waschen.
- Tragen Sie 200 μl DAB-Substratlösung auf (direkt vor Gebrauch frisch hergestellt: 1 Tropfen DAB auf 1 mL Substrat), 200 μl pro Objektträger. Lassen Sie die Farbentwicklung 1-3 min einwirken, bis die gewünschte Farbintensität erreicht ist.
- 10 Min. mit fließendem Leitungswasser nachspülen.
- Färben Sie die Objektträger, indem Sie die Seiten 5 Minuten lang in Hämatoxylin eintauchen.
- 10 Min. mit fließendem Leitungswasser nachspülen.
- Decken Sie die Objektträger mit wässrigem Eindeckmedium und Deckgläsern ab. Die gerahmten Dias können dauerhaft bei Raumtemperatur gelagert werden.
Ergebnisse
Abbildung 1 gibt einen Überblick über den Arbeitsablauf zur Kultivierung von OTSCs aus frischem, ungefrorenem Tumorgewebe. Proben von primären PDACs und Metastasen wurden direkt nach der chirurgischen Resektion entnommen und über Nacht auf Nasseis bei 4 °C in der Gewebeaufbewahrungslösung gelagert. Die Proben wurden verarbeitet und die Scheiben wurden wie im Protokoll beschrieben kultiviert. Die makroskopische Morphologie der einzelnen OTSC veränderte sich während der Kultivierung nicht wesentlich. Die Größe der Oberfläche der OTSCs nahm jedoch im Laufe der Zeit ab, wie beispielhaft in Abbildung 2A während der Kultivierung über 6 Tage gezeigt. Die Gesamtviabilität von OTSC wurde durch Resazurin-Viabilitätstest an den Tagen 0, 2/3 und 6 beurteilt (Abbildung 2B). Der Resazurin-Viabilitätsassay misst die allgemeine metabolische Aktivität des OTSC basierend auf der Reduktion von nicht fluoreszierendem blauem Resazurin zu rot fluoreszierendem Resorufin in lebenden Zellen15. Abbildung 2B zeigt den Vergleich der Viabilität, gemessen durch Resazurinreduktion nach der Kultivierung von OTSCs aus zwei repräsentativen Primärtumoren in zwei verschiedenen Medien. OTSCs wurden entweder in Medium A (fortgeschrittenes DMEM/F12, 10 % FBS, 1 % Penicillin/Streptomycin) oder Medium B (RPMI 1640, 10 % FBS, 1 % Penicillin/Streptomycin, 4 μg/mL Insulin, 8 ng/mL EGF, 0,3 μg/mL Hydrocortison) kultiviert, was unabhängig vom Kulturmedium zu einer ähnlichen Gesamtviabilität führte. Bemerkenswert ist, dass eine Abnahme der Lebensfähigkeit nach Tag 0 häufig beobachtet werden kann und aufgrund des Schneideverfahrens und der Anpassung an die Kulturbedingungen zu erwarten ist. Wir beobachteten jedoch auch eine erhöhte Lebensfähigkeit, wie beispielhaft im rechten Bild gezeigt (Abbildung 2B).
Nach definierten Kultivierungszeiträumen in Medium A wurden OTSCs zur weiteren immunhistologischen Charakterisierung (IHC) in Formalin fixiert. Nach der Formalinfixierung wurden OTSCs paraffineingebettet und geschnitten. H&E-gefärbte Schnitte zeigten, dass die Gesamtstruktur des Gewebes über die gesamte Zeit der Kultivierung ex vivo erhalten blieb (Abbildung 3). Tumor- und Stromazellen wurden mittels IHC auf Cytokeratin 7 bzw. Vimentin unterschieden. Während der Kultivierung wurden keine wesentlichen Veränderungen des Tumor-Stroma-Verhältnisses festgestellt.
Die mikroskopische histopathologische Auswertung von H&E-Schnitten ergab keinen signifikanten Anstieg der Nekrose aller kultivierten Gewebe während der Kultivierung (Abbildung 3, Abbildung 4, Abbildung 5). Zusätzlich wurden Ki-67 und gespaltene Caspase 3 zur Beurteilung der Proliferation bzw. Apoptose gefärbt. Auch hier konnten wir keine groben Veränderungen der Proliferation und Apoptose während des Kulturzeitraums von 6 Tagen feststellen (Abbildung 3). Der Anteil apoptotischer Zellen nimmt jedoch im Laufe der Zeit bei längeren Kultivierungsperioden zu, gemessen durch gespaltene Caspase-3-Färbung. Abbildung 4 zeigt den Anstieg der gespaltenen Caspase-3-positiven Zellen nach 15-tägiger Kultivierung eines primären PDAC. Abbildung 5 zeigt die Histopathologie einer Peritonealmetastase eines PDAC. Dieses Experiment zeigt eine hohe intratumorale Heterogenität zwischen den einzelnen Schichten in Bezug auf den Tumor-/Stromagehalt sowie die natürlich vorkommende Apoptose, gemessen durch gespaltene Caspase-3-Färbung. Daher ist die histopathologische Bewertung von kultivierten OTSCs wichtig für die Beurteilung der Tumor/Stroma-Architektur und der Lebensfähigkeit jedes kultivierten Tumors bzw. seiner Metastasen.
Abbildung 6 zeigt die Histopathologie einer PDAC-Metastasierung der Bauchdecke. Sowohl die H&E-Färbung als auch die IHC für Cytokeratin 7 zeigten, dass die abgeleiteten OTSCs keine Tumorzellen enthielten, sondern nur aus Bindegewebe bestanden, das teilweise nekrotisch war. Ein Nachteil der OTSC-Technologie besteht darin, dass frisches, unfixiertes und ungefrorenes Gewebe vor der Kultivierung nicht auf seinen Tumorzellgehalt untersucht werden kann.
Insgesamt zeigen diese Daten, dass sich die multizelluläre Architektur eines Tumors und seine jeweilige Metastasierung, die aus unterschiedlichen Zelltypen besteht, in OTSCs widerspiegeln. Insbesondere Tumor-Stroma-Wechselwirkungen bleiben erhalten.
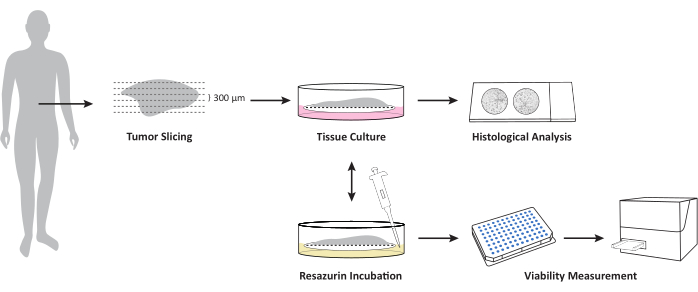
Abbildung 1: Überblick über den Workflow für die Kultivierung von OTSCs aus frischen, ungefrorenen Gewebetumor-/Metastasenproben. Die frische, ungefrorene Gewebeprobe wird mit einem Vibratom geschnitten und an der Luft-Flüssig-Grenzfläche auf PTFE-Zellkultureinsätzen kultiviert. Die allgemeine Lebensfähigkeit kann zu definierten Zeitpunkten mit einem Resazurin-Viabilitätstest gemessen werden. Dieser Assay ermöglicht eine weitere Kultivierung nach der Viabilitätsmessung. Die Scheiben werden zu definierten Zeitpunkten für die weitere histopathologische Untersuchung formalinfixiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
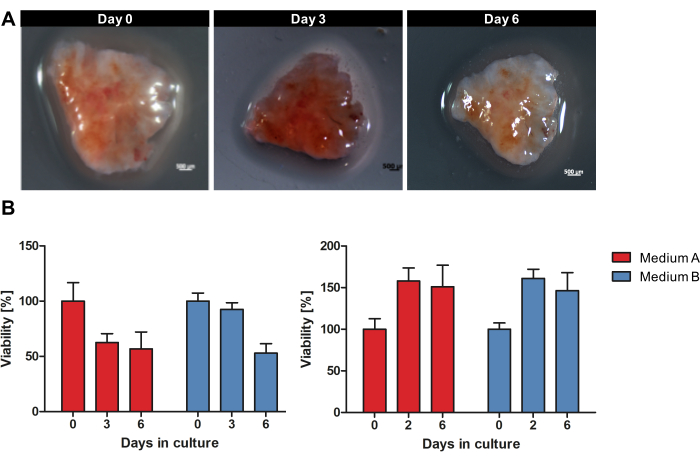
Abbildung 2: Makroskopische Morphologie und Lebensfähigkeit von OTSCs während der Kultivierung. Ein PDAC-Exemplar wurde 6 Tage lang als OTSC kultiviert. (A) Die makroskopische Morphologie der einzelnen OTSC veränderte sich während des Anbaus nicht wesentlich. Die Größe der Oberfläche der OTSC nahm jedoch im Laufe der Zeit ab. (B) Die Gesamtlebensfähigkeit wurde durch einen Resazurin-Viabilitätstest in Medium A und B quantifiziert. Eine Abnahme der Lebensfähigkeit kann häufig nach Tag 0 aufgrund des Schnittverfahrens und der Anpassung an die Kulturbedingungen beobachtet werden. Wir beobachteten jedoch auch eine Steigerung der Lebensfähigkeit, wie beispielhaft im rechten Bild dargestellt. Jedes Panel repräsentiert eine individuelle Tumorprobe mit unterschiedlichen Ausbeuten an abgeleiteten Schnittkulturen (linkes Panel n = 10, rechtes Panel n = 10). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Histopathologische Bewertung von OTSCs nach einer Gesamtkultivierungszeit von 6 Tagen. OTSCs (n = 6) wurden in Medium A kultiviert. Die Gewebemorphologie wurde durch H&E-Färbung beurteilt. Tumor- und Stromazellen wurden mittels IHC auf Cytokeratin 7 bzw. Vimentin unterschieden. Ki-67 und gespaltene Caspase 3 wurden zur Beurteilung der Proliferation bzw. Apoptose gefärbt. Über einen Gesamtkultivierungszeitraum von 6 Tagen wurde kein signifikanter Anstieg der Apoptose beobachtet. Jeder Maßstabsbalken steht für 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Histopathologische Bewertung von OTSCs nach einer Gesamtkultivierungszeit von 15 Tagen. OTSCs (n = 8) wurden in Medium A kultiviert. Die Gewebemorphologie wurde durch H&E-Färbung beurteilt. Tumor- und Stromazellen wurden mittels IHC auf Cytokeratin 7 bzw. Vimentin unterschieden. Ki-67 und gespaltene Caspase 3 wurden zur Beurteilung der Proliferation bzw. Apoptose gefärbt. Die Apoptose stieg nach 15 Tagen der Kultivierung erheblich an, wie z. B. die Färbung von gespaltener Caspase-3 zeigt. Jeder Maßstabsbalken steht für 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Histopathologische Bewertung von OTSCs einer Peritonealmetastasierung eines PDAC. OTSCs (n = 8) wurden in Medium A kultiviert. Die Gewebemorphologie wurde durch H&E-Färbung beurteilt. Tumor- und Stromazellen wurden mittels IHC auf Cytokeratin 7 bzw. Vimentin unterschieden. Ki-67 und gespaltene Caspase 3 wurden zur Beurteilung der Proliferation bzw. Apoptose gefärbt. Die histopathologische Evaluation ergab eine hohe intratumorale Heterogenität zwischen den einzelnen Scheiben hinsichtlich des Tumor-/Stromagehalts sowie der natürlich vorkommenden Apoptose, gemessen durch gespaltene Caspase-3-Färbung. Jeder Maßstabsbalken steht für 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Histopathologische Abklärung von OTSCs einer PDAC-Metastasierung der Bauchdecke. OTSCs (n = 5) wurden in Medium A kultiviert. Die Gewebemorphologie wurde durch H&E-Färbung beurteilt. Tumor- und Stromazellen wurden mittels IHC auf Cytokeratin 7 bzw. Vimentin unterschieden. Ki-67 und gespaltene Caspase 3 wurden zur Beurteilung der Proliferation bzw. Apoptose gefärbt. Die histopathologische Untersuchung ergab das Fehlen von Tumorzellen in der kultivierten Metastasierung und partiellen Nekrose, das vor der Kultivierung nicht festgestellt werden konnte. Jeder Maßstabsbalken steht für 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
OTSCs von frischen Tumorproben sind eine enge Annäherung an den Tumor in situ. Sie behalten ihre Ausgangsmorphologie, ihre proliferative Aktivität und ihr Mikromilieu während der Kultivierung für einen definierten, gewebeabhängigen Zeitraum bei 11,12,13. Diese Technik ermöglicht die unmittelbare Anwendung der Behandlung auf lebensfähiges menschliches Tumorgewebe ex vivo und nachfolgende Analysen, wie z. B. die Profilerstellung des Transkriptoms und des Proteoms. Ein besonderer Vorteil von OTSCs besteht darin, dass ortsaufgelöste Downstream-Analysen wie die MALDI-Bildgebung auf intaktes mehrzelliges Tumorgewebe angewendet werden können.
Im Folgenden wird eine Methode zur Generierung, Kultivierung und histopathologischen Bewertung von OTSCs der PDAC und ihrer Metastasen beschrieben. Das Schlüsselproblem bei dieser Technik besteht darin, frisches Gewebe ohne Gefrierverfahren und ohne Kultivierung an einer Luft-Flüssigkeits-Grenzfläche zu schneiden. Eine Einschränkung dieser Methode besteht darin, dass frisches, unfixiertes und ungefrorenes Gewebe vor der Kultivierung nicht auf seinen Tumorzellgehalt untersucht werden kann. Wie in unseren Ergebnissen beispielhaft gezeigt, können OTSCs zeigen, dass sie aus Gewebekomponenten bestehen, die nicht von schnellen Schnitten anderer Proben desselben Tumors erwartet werden. Eine histopathologische Bewertung der OTSCs nach der Kultivierung ist für die Datenauswertung einzelner Scheibenkulturen unerlässlich. Der beschriebene Viabilitätstest erlaubt nur eine allgemeine Beurteilung der allgemeinen Lebensfähigkeit jedes OTSC. Grundsätzlich kann die beschriebene Methode in jedem Labor implementiert werden, das mit einer Vibratom- und Gewebekultureinheit ausgestattet ist. Aufgrund der Einfachheit dieser Technik kann sie leicht an verschiedene Tumore und Forschungsfragen angepasst werden.
Es wurden mehrere Methoden entwickelt, um menschliches Tumorgewebe ex vivo zu kultivieren, z. B. primäre Zelllinien, von Patienten stammende Organoide und Xenotransplantate. Ihr Einsatz für die Wirkstoffentwicklung und die Identifizierung prädiktiver Biomarker bleibt jedoch vor allem aufgrund des Verlusts des Gewebekontexts, d.h. der Interaktion von Stroma und Tumorzellen, begrenzt2.
Obwohl die vorgestellte Methode der organotypischen Schnittkulturetablierung die multizelluläre Gewebearchitektur beibehält, müssen einige Einschränkungen berücksichtigt werden. Zunächst muss ein erhebliches Maß an (Intra-)Tumorheterogenität berücksichtigt werden. Unterschiedliche OTSCs, die aus einer einzigen Tumorbiopsie stammen, können erhebliche Unterschiede in ihren Anteilen an Tumor- und Stromazellen aufweisen. Neben der allgemeinen Gewebequalität könnte dies ein Grund für die unterschiedliche Lebensfähigkeit der Scheiben während der Kultur sein. Zweitens kann das Medium für optimale Kulturbedingungen je nach Gewebe und Patient variieren. Wir verglichen zwei verschiedene Gewebekulturmedien: das erste auf DMEM/F12 (Medium A) und das zweite auf RPMI (Medium B). Jede Probe von Primärtumoren oder deren Metastasen kann modifiziert werden. Drittens kann eine Tumornekrose bereits in situ16 auftreten und ist nicht notwendigerweise auf unzureichende Kulturbedingungen der OTSCs zurückzuführen. Ein Vergleich mit der Histopathologie des Primärtumors sollte in Betracht gezogen werden.
Die Etablierung von OTSCs, wie in diesem Protokoll beschrieben, bietet die Möglichkeit, das Ansprechen auf die Behandlung zeitnah nach der Operation in einem ex vivo-Modellsystem zu testen, das die Mikroumgebung des Tumors konserviert. In der Zukunft werden OTSCs die Entwicklung individueller therapeutischer Strategien zur Personalisierung der Behandlung von metastasierten Erkrankungen erleichtern.
Offenlegungen
Die Autoren legten keine potenziellen Interessenkonflikte offen.
Danksagungen
R. Braun wurde gefördert durch die Clinician Scientist School Lübeck (DFG #413535489) und das Junior Funding Program der Universität zu Lübeck.
Materialien
| Name | Company | Catalog Number | Comments |
| Advanced DMEM/F-12 Medium | Gibco | 12634028 | |
| Agarose Low Melt | Roth | 6351.2 | 8% in Ringer solution |
| Antibody Diluent, Background Reducing | Dako | S3022 | |
| AquaTex | Merck | 108562 | |
| Bioethanol (99%, denatured) | CHEMSolute | 2,21,19,010 | |
| Citric Acid monohydrate | Sigma Aldrich | C7129 | |
| Cleaved Caspase-3 (Asp175) (5A1E) Rabbit mAb | Cell Signalling Technology | 9664 | 1:400 dilution |
| Derby Extra Double Edge Safety Razor Blades | Derby Tokai | ||
| Embedding cassettes | Roth | H579.1 | |
| Eosin Y-solution 0,5% aqueous | Merck | 10,98,44,100 | |
| Eukitt Quick hardening mounting medium | Sigma-Aldrich | 3989 | |
| Fetal bovine serum | Gibco | 10270106 | |
| Formaldehyde solution 4,5%, buffered | Büfa Chemikalien | B211101000 | |
| Hem alum solution acid acc. to Mayer | Roth | T865 | |
| Human EGF | Milteniy Biotec | 130-097-794 | |
| Hydrocortisone | Sigma Aldrich (Merck) | H0888 | |
| Hydrogen peroxide 30% | Merck | 1,08,59,71,000 | |
| Insulin human | Sigma Aldrich (Merck) | 12643 | |
| Liquid DAB+ Substrate Chromogen System | Dako | K3468 | |
| MACS Tissue Storage Solution | Milteniy Biotech | 130-100-008 | |
| Methanol | Merck | 10.600.092.500 | |
| Microscope Slides Superfrost Plus | Thermo Scientific | J1800AMNZ | |
| Millicell Cell Culture Insert, 30 mm, hydrophilic PTFE, 0.4 µm | Millipore (Merck) | PICM0RG50 | |
| Monoclonal mouse anti-human Cytokeratin 7 (Clone OV-TL 12/30) | Dako | M7018 | 1:200 dilution |
| Monoclonal mouse anti-human Ki67 Clone MIB-1 | Dako | M7240 | 1:200 dilution |
| Monoclonal mouse Anti-vimentin (Clone V9) | Dako | M0725 | 1:200 dilution |
| Negative control Mouse IgG2a | Dako | X0943 | 1:200 dilution |
| Negative control Mouse IgG1 | Dako | X093101-2 | 1:200 dilution |
| Paraffin (melting temperature 56°- 58°) | Merck | 10,73,37,100 | |
| Penicillin-Streptomycin (10.000 U/ml) | Gibco | 15140122 | |
| PBS pH 7,4 (1x) Flow Cytometry Grade | Gibco | A12860301 | |
| Resazurin sodium salt; 10 mg/ml in PBS | Sigma Aldrich | R7017 | 1:250 dilution |
| Ringer's solution | Fresenius Kabi | 2610813 | |
| RPMI-1640 Medium | Sigma Aldrich (Merck) | R8758 | |
| Tissue culture testplate 6 | TPP | 92006 | |
| Triton X-100 | Sigma Aldrich | 9002-93-1 | |
| VECTASTAIN Elite ABC-Peroxidase Kit | Vector Laboratories | PK-6200 | |
| Xylene (extra pure) | J.T.Baker | 8,11,85,000 | |
| Equipment | |||
| ClarioStar Microplate Reader | BMG Labtech | ||
| Paraffin Embedding Center E61110 | Leica | ||
| Rotary Microtome Microm HM355S | Thermo Scientific | ||
| Section Transfer System Microm STS | Thermo Scientific | ||
| VT 1200S Vibratom | Leica |
Referenzen
- Nevala-Plagemann, C., Hidalgo, M., Garrido-Laguna, I. From state-of-the-art treatments to novel therapies for advanced-stage pancreatic cancer. Nature Reviews. Clinical Oncology. 17 (2), 108-123 (2020).
- Tiriac, H., et al. Organoid profiling identifies common responders to chemotherapy in pancreatic cancer. Cancer Discovery. 8 (9), 1112-1129 (2018).
- Waghray, M., Yalamanchili, M., di Magliano, M. P., Simeone, D. M. Deciphering the role of stroma in pancreatic cancer. Current Opinion in Gastroenterology. 29 (5), 537-543 (2013).
- Hwang, R. F., et al. Cancer-associated stromal fibroblasts promote pancreatic tumor progression. Cancer Research. 68 (3), 918-926 (2008).
- Jiang, X., et al. Long-lived pancreatic ductal adenocarcinoma slice cultures enable precise study of the immune microenvironment. Oncoimmunology. 6 (7), 1333210 (2017).
- Hessmann, E., et al. Fibroblast drug scavenging increases intratumoural gemcitabine accumulation in murine pancreas cancer. Gut. 67 (3), 497-507 (2018).
- Feig, C., et al. The pancreas cancer microenvironment. Clinical Cancer Research. 18 (16), 4266-4276 (2012).
- Koerfer, J., et al. Organotypic slice cultures of human gastric and esophagogastric junction cancer. Cancer Medicine. 5 (7), 1444-1453 (2016).
- Gerlach, M. M., et al. Slice cultures from head and neck squamous cell carcinoma: a novel test system for drug susceptibility and mechanisms of resistance. British Journal of Cancer. 110 (2), 479-488 (2014).
- Holliday, D. L., et al. The practicalities of using tissue slices as preclinical organotypic breast cancer models. Journal of Clinical Pathology. 66 (3), 253-255 (2013).
- Vaira, V., et al. Preclinical model of organotypic culture for pharmacodynamic profiling of human tumors. Proceedings of the National Academy of Sciences of the United States of America. 107 (18), 8352-8356 (2010).
- Lim, C. Y., et al. Organotypic slice cultures of pancreatic ductal adenocarcinoma preserve the tumor microenvironment and provide a platform for drug response. Pancreatology. 18 (8), 913-927 (2018).
- Brandenburger, M., et al. Organotypic slice culture from human adult ventricular myocardium. Cardiovascular Research. 93 (1), 50-59 (2012).
- Misra, S., et al. Ex vivo organotypic culture system of precision-cut slices of human pancreatic ductal adenocarcinoma. Science Reports. 9 (1), 2133 (2019).
- O'Brien, J., Wilson, I., Orton, T., Pognan, F. Investigation of the Alamar Blue (resazurin) fluorescent dye for the assessment of mammalian cell cytotoxicity. European Journal of Biochemistry. 267 (17), 5421-5426 (2000).
- Beutler, B., Cerami, A. Tumor necrosis, cachexia, shock, and inflammation: a common mediator. Annual Review of Biochemisty. 57, 505-518 (1988).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten