Method Article
Erken Zebra Balığı Embriyoları için Işık Levha Mikroskobu Görüntüleme ve Montaj Stratejileri
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Erken zebra balığı embriyolarının bir ışık tabakası mikroskobu kullanılarak bozulmamış bir koryon içinde görüntülenmesi için bir örnek hazırlama stratejisi açıklanmaktadır. Embriyoların %70 epiboli ve tomurcuk aşamalarında koryon içinde edindiği farklı yönelimleri analiz eder ve ışık tabakası sistemini kullanarak embriyo boyunca hücresel ölçekte çözünürlük elde etmek için görüntüleme stratejilerini detaylandırır.
Özet
Işık tabakası mikroskobu, zebra balığı embriyolarının minimum fototoksisite ile uzun zaman ölçeklerinde canlı görüntülenmesi için tercih edilen metodoloji haline gelmiştir. Özellikle, numune rotasyonuna izin veren bir çoklu görüntüleme sistemi, tüm embriyoların farklı açılardan görüntülenmesini sağlar. Bununla birlikte, çoklu görüntüleme sistemine sahip çoğu görüntüleme oturumunda, numuneler genellikle bir polimer tüp içinde hazırlandığından, numune montajı zahmetli bir süreçtir. Bu sürece yardımcı olmak için bu protokol, %70 epiboli ve erken somit aşamaları arasındaki erken zebra balığı gelişimini görüntülemek için temel montaj stratejilerini açıklar. Spesifik olarak, çalışma, embriyoların koryon içindeki% 70 epiboli ve tomurcuk aşamalarında varsayılan olarak bulunduğu çeşitli pozisyonlar hakkında istatistikler sağlar. Ayrıca, gelişimin erken aşamalarında bütün zebra balığı embriyolarını görüntülemek için gereken optimum açı sayısını ve açılar arasındaki aralığı tartışır, böylece hücresel ölçekli bilgiler farklı görünümleri birleştirerek çıkarılabilir. Son olarak, embriyo, hücresel ölçekte bir çözünürlük elde etmek için gerekli olan kameranın tüm görüş alanını kapsadığından, bu protokol, farklı görünümlerin kaydedilmesi için embriyonun üstünden veya altından boncuk bilgilerinin kullanılması sürecini detaylandırır.
Giriş
Minimal fototoksisitenin sağlanması, uzun süreler boyunca yüksek uzay-zamansal çözünürlüğe sahip canlı embriyoların görüntülenmesi için önemli bir gerekliliktir. Son on yılda, ışık tabakası mikroskobu bu gereksinimi karşılamak için tercih edilen metodoloji haline gelmiştir 1,2,3,4,5,6,7. Kısaca ilk kez 2004 yılında gelişimsel süreçleri yakalamak için kullanılan bu teknikte8, embriyonun içinden karşılıklı uçlardan hizalanmış ince iki lazer tabakası geçerek sadece ilgilenilen düzlemi aydınlatır. Bir algılama hedefi ortogonal olarak yerleştirilir ve ardından numunedeki tüm aydınlatılmış noktalardan yayılan floresan ışık aynı anda toplanır. Daha sonra embriyonun statik ışık tabakası boyunca sırayla hareket ettirilmesiyle bir 3D görüntü elde edilir.
Ek olarak, bu metodolojinin çoklu görüntü ışık tabakası mikroskobu olarak adlandırılan özel bir biçiminde, numuneler, aynı embriyonun birden fazla açıdan görüntülenmesini sağlayan bir rotor kullanılarak döndürülebilen bir polimer tüp içinde askıya alınabilir 9,10,11. Görüntülemeyi takiben, çoklu açılardan gelen görüntüler, tipik olarak embriyo içinde (örneğin, çekirdekler) veya tüpte (örneğin, floresan boncuklar) küresel floresan belirteçler olan kayıt belirteçlerine dayalı olarak birleştirilir. Çoklu görüntüleme görüntüleme ve füzyon, eksenel çözünürlüğü önemli ölçüde iyileştirerek her üç boyuttada izotropik çözünürlük sağlar 12. Bu büyük bir avantaj olsa da, çoklu görüntüleme metodolojisinin en büyük zorluğu, embriyoların tüm görüntüleme süresi boyunca tüplere monte edilmesi ve yerinde tutulması gereken numune montajıdır.
Çok görüntülü görüntüleme yapmak, embriyoları yerinde tutmak ve görüntüleme sırasında hareketi önlemek için embriyolar agaroz içine gömülebilir. Bununla birlikte, bu genellikle, özellikle burada tartışılan model sistem olan erken evre zebra balığı embriyoları13 için zararlı büyüme ve gelişmeye yol açar. İkinci bir montaj stratejisi, embriyonun çapından sadece biraz daha büyük olan ince bir tüp kullanmaktır, burada embriyo, embriyo ortamı ile birlikte tüpe çekilebilir, ardından tüpün altını bir agaroz tıkaç14 ile kapatmaktır. Bu yöntemde, tüp embriyo ortamı ile doldurulduğundan, farklı görünümlerin füzyonu için floresan boncuklar gibi kayıt belirteçleri kullanılamaz ve bu nedenle kayıt, embriyo içindeki belirteçlere bağlıdır. Genel olarak, boncuklar, herhangi bir mikroskobun hem aydınlatma hem de algılama sınırlamaları nedeniyle, embriyo içindeki belirteçlerin sinyali, numunenin daha derinlerine inildiğinde bozulduğunda daha iyi kayıt belirteçleri olarak işlev görür.
Bu nedenle, burada detaylandırılacak olan ve daha önce 5,13,14,15,16 kullanılan üçüncü bir yaklaşım, erken zebra balığı embriyolarının sağlam bir koryon ile görüntülenmesi ve tüpün, kayıt belirteçleri olarak boncuklar içeren minimum oranda agaroz ile doldurulmasıdır. Bu senaryoda, embriyoları bir koryon içinde konumlandırmak için manuel müdahale mümkün olmadığından, bu çalışma, özellikle %70 epiboli ve tomurcuk aşamalarına odaklanarak, erken zebra balığı embriyolarının düştüğü varsayılan yönelim hakkında istatistikler sağlar. Daha sonra, erken evre embriyoları hücresel ölçekte çözünürlükte görüntülemek için gereken en uygun görüntüleme sayısını tartışır ve FIJI tabanlı bir eklentiolan BigStitcher'ı kullanarak füzyon sürecini detaylandırır 10,17,18. Birlikte, 20x/1 NA objektifi kullanan bu protokol, zebra balığı embriyologlarının, gastrulasyondan erken somit evrelerine kadar çekirdek ve zar belirteçleri olan embriyoları görüntülemek için çok görüntülü ışık tabakası sistemlerini kullanmalarını kolaylaştırmayı amaçlamaktadır.
Protokol
Bu çalışmada kullanılan zebra balığı bakım ve deneysel prosedürleri, kurumsal hayvan etik komitesi tarafından onaylanmıştır, vide Referans TIFR/IAEC/2023-1 ve TIFR/IAEC/2023-5. Tg(actb2:GFP-Hsa.UTRN)19 eksprese eden heterozigot balıkların çaprazlanmasıyla elde edilen embriyolara tek hücreli aşamada H2A-mCherry mRNA (30 pg) enjekte edildi. H2A-mCherry mRNA, in vitro transkripsiyon ile pCS2+ H2A-mCherry plazmidi (Oates laboratuvarı, EPFL'den bir hediye) kullanılarak sentezlendi. Protokolün geri kalanında sırasıyla Utr-GFP ve H2A-mCherry olarak adlandırılan her iki belirteci de eksprese eden embriyolar,% 70 epiboli ve tomurcuk aşamalarında görüntülendi. Çalışmada kullanılan reaktiflerin ve ekipmanların detayları Malzeme Tablosunda listelenmiştir.
1. Çoklu görüntüleme için numune hazırlama
- FEP/PTFE tüplerin hazırlanması
- FEP/PTFE tüplerini daha önce açıklanan prosedürleri izleyerek temizleyin14.
- Temizledikten sonra, tüpleri ihtiyaca göre 2-2,5 cm uzunluklarda kesin ve çift distile su içeren 2 mL mikrosantrifüj tüplerinde saklayın. Tüpler bu şekilde yaklaşık bir ay saklanabilir.
- Numune hazırlamadan önce, tüpleri düzleştirmek için tüpleri 70-75 °C'de yaklaşık 25 dakika ısıtın. Topaklanmayı önlemek için her bir mikrosantrifüj tüpünde aşırı kalabalıktan kaçının ve etkili tüp düzleştirme için yeterli alan sağlayın.
NOT: Tüpler üzerinde kalan herhangi bir parmak izi/toz parçacığı görüntülemeyi engelleyebileceğinden, tüm tüp taşıma prosedürü boyunca eldiven giyin.
- Agaroz hazırlanması
- E3 tampon 50x stok çözeltilerini aşağıdaki gibi hazırlayın:
- Stok 1 - 3.252 g Na2HPO4, 0.285 g KH2PO4, 11.933 g NaCl, 0.477 g KCl 1 L deiyonize su
- Stok 2 - 2.426 g CaCl2, 1 L deiyonize suda 4.067 g MgS04.
- 1x E3 tamponu yapmak için, 960 mL deiyonize suya stok 1 ve 2 çözeltilerinin her birine 20 mL ekleyin.
NOT: Işığın saçılmasına neden olduğu için görüntüleme için kullanılan E3 tamponuna metilen mavisi eklemeyin. - Düşük erime noktalı agarozu, bir ısı bloğu üzerinde 70-75 °C'de ısıtarak 1x E3 içinde çözün ve çözelti topak veya kristal olmadan berraklaşana kadar karıştırın.
- Çok görüntülü görüntüleme için, analiz sırasında görüntü kaydını sağlayan agaroz çözeltisine ticari olarak temin edilebilen floresan boncuklar ( Malzeme Tablosuna bakın) ekleyin. Boncukları ayırmak için bir su banyosu ultrasonicator'da 20-30 dakika boyunca 40 kHz frekansında boncukları oda sıcaklığında sonikasyon.
- Sonikasyondan sonra, 15 mL'lik bir tüpte 10 mL agaroz çözeltisine 1 μL boncuk ekleyin ve boncukların düzgün dağılmasını sağlamak için tüpü iyice vorteksleyin.
NOT: Agaroza eklenecek boncuk çözeltisinin hacmi, stok çözeltisine ve üreticiye bağlıdır. Bir dizi hacim deneyin ve hafif levha sisteminde görüntülendiğinde boncukların iyi dağılmış göründüğü bir konsantrasyon seçin. Sonikasyon ve girdaplamaya rağmen çok fazla boncuk toplanabilirken, çok az boncuk görüntü kaydını etkileyebilir. - Eklenen boncuklarla birlikte agaroz tüpünü, numune hazırlamadan önce en az 30 dakika boyunca 37 °C'de tutulan kapalı bir su banyosunda tutun. Bu, agarozun sıcaklığının 70 °C'den 37 °C'ye düşmesini sağlar.
- E3 tampon 50x stok çözeltilerini aşağıdaki gibi hazırlayın:
- Numune hazırlama
- Görüntüleme gününden önceki akşam balıkları bölücülerle çiftler halinde yerleştirin. Bölücüleri yaklaşık 15 dakika çıkarın ve standart prosedür20'yi kullanarak embriyoları toplayın.
- Embriyolar numune hazırlama aşamasına geldiğinde, 37 ° C'de tutulan 10 mL agarozun tamamını (boncuklu) 6 cm'lik bir Petri kabına dökün.
- 10 ila 15 embriyoyu agaroz çözeltisine aktarmadan önce sıcaklığın 33 ° C'nin altına düşmesi için yaklaşık 2-3 dakika bekleyin.
NOT: Bu, agaroza transfer edilen embriyolara olası bir ısı şokunu önlemek için önemli bir adımdır. - Embriyoları transfer ederken agaroz çözeltisine mümkün olduğunca az E3 tamponu eklendiğinden emin olun. Petri kabını, aktarılacak olan küçük tamponun iyice dağılması için döndürün.
- Numune hazırlama prosedürünün geri kalanında eldiven giyin.
- Uygun bir pipet ucu ile 200 μL'lik bir mikropipet alın ve mikrosantrifüj tüpünden çıkarılan temizlenmiş düz bir tüpü Şekil 1A,B'de gösterildiği gibi pipet ucuna yerleştirin.
- Tüpe biraz agaroz aspire edin, ardından mikropipet kullanarak 2-3 embriyo elde edin (Şekil 1C,D). Embriyoların tüpün dibine yakın olduğundan emin olun (Şekil 1E), bu görüntüleme yapılırken önemli hale gelecektir. Ayrıca, farklı embriyolar arasında biraz agaroz olduğundan emin olun (Şekil 1E), böylece bu bölgedeki boncuklar kayıt belirteçleri olarak kullanılabilir.
- Pipetteki basıncı serbest bırakmadan, tüpü pipet ucundan ayırın ve agarozun katılaşması için E3 ile doldurulmuş petri kabının kapağına yerleştirin (Şekil 1F).
- Petri kabındaki tüm embriyolar tüplere aktarılana kadar 1.3.6 ila 1.3.8 arasındaki adımları tekrarlayın.
- Agaroz katılaştıktan sonra (5-10 dakika sürebilir ve Petri kabındaki agaroz kontrol edilerek doğrulanabilir), tüpleri E3 ile doldurulmuş 2 mL'lik bir mikrosantrifüj tüpüne aktarın (Şekil 1G).
- Çoklu görüntülemeye geçmeden önce, embriyoların bulunduğu tüpleri gerektiği gibi 28 °C veya 33 °C'lik bir inkübatörde saklayın.
2. Çoklu görüntüleme görüntüleme
NOT: Bu adım, zebra balığı embriyolarının erken gelişim aşamalarında çoklu görüntüleme için genel bir prosedür sunar. Aşağıda ayrıntıları verilen yöntem, herhangi bir çoklu görüntü ışık tabakası mikroskobu sistemine kolayca uyarlanabilir.
- Embriyonun yerinin belirlenmesi
NOT: Mikroskobun numune haznesini embriyo ortamı20 ile doldurun ve numune haznesinin sıcaklığını, görüntülemeden en az 10 dakika önce ilgilenilen sıcaklığa ayarlayın.- Numune tutucuyu daha önceaçıklandığı gibi 21 monte edin, ancak bir değişiklikle. Görüntüleme tüp içinden yapılacağı için, embriyoların bulunduğu tüpü, agarozu dışarı itmeden ve alttaki embriyoları rahatsız etmeden yerinde tutulacak şekilde doğrudan doğru çaptaki bir kılcal damara yerleştirin (Şekil 1H).
NOT: Numune haznesi tanımlanmış bir yüksekliğe sahip olduğundan, numune hazırlama sırasında embriyoların tüpün dibine doğru yerleştirildiğinden emin olmak (adım 1.3.7'de belirtildiği gibi), embriyonun odanın tabanına çarpmadan görüş alanına yerleştirilmesini sağlayacaktır. - Numune tutucuyu çoklu görüntüleme sistemine yerleştirin ve embriyoyu görüş alanının merkezine getirmek için x ve y kontrollerini kullanın.
- Daha sonra embriyoyu odak noktasına getirmek için örnek navigatörünün z kontrolünü kullanın.
- Bu noktada, embriyonun koryon içindeki oryantasyonu açıkça görülecektir ve tatmin edici değilse, yeni bir tüp yerleştirin ve ilgilenilen bir oryantasyona sahip embriyoyu seçin.
- Numune tutucuyu daha önceaçıklandığı gibi 21 monte edin, ancak bir değişiklikle. Görüntüleme tüp içinden yapılacağı için, embriyoların bulunduğu tüpü, agarozu dışarı itmeden ve alttaki embriyoları rahatsız etmeden yerinde tutulacak şekilde doğrudan doğru çaptaki bir kılcal damara yerleştirin (Şekil 1H).
- Işık levhalarının hizalanması
- Embriyo yerleştirildikten sonra, tüm deneysel ayarları ayarlayın - lazerler, ışık yolu, filtre, ışın ayırıcı ve kamera.
- Alım parametrelerini ayarlarken, yazılım pivot tarama için bir seçenek sunuyorsa, onu seçin. Bir pivot taraması, numunedeki herhangi bir yapı veya opak bölgeyle karşılaştığında ışık tabakasından çıkan gölgeleme etkisini azaltacaktır.
- Numunenin canlı taramasını başlatın. Işık tabakasının kalınlığını ve dolayısıyla eksenel çözünürlüğü belirleyecek olan bit boyutunu, lazer gücünü ve istenen yakınlaştırmayı ayarlayın.
- Işık levhalarını hizalamak için önce tek taraflı aydınlatmaya geçin ve iki ışık levhasının (sol ve sağ) ayarlarını sırayla değiştirin. Numunenin net ilgi yapılarının görülebildiği bir bölgesine dijital olarak yakınlaştırın.
NOT: Bu çalışmada, tabakaları hizalamak için bir nükleer (H2A-mCherry) veya bir membran (Utr-GFP) işaretleyici kullanılmıştır. İlgili bölgede en keskin sinyal kontrastının elde edilmesi için yazılımdaki ışık sayfası ayarlarının değiştirilmesi önerilir. - En iyi sinyali elde etmek için iki ışık levhası ayrı ayrı optimize edildikten sonra, her iki ışık levhasını da açın. İkinci bir hizalama turu gerçekleştirin ve titremenin minimum düzeyde olduğundan emin olun, bu da iki sayfanın oldukça iyi hizalandığını gösterir.
- Hizalama yapıldıktan sonra, yazılım izin veriyorsa, iki sayfa tarafından oluşturulan görüntüleri otomatik olarak birleştirme seçeneğini etkinleştirin. Alternatif olarak, önce numunenin etrafındaki boncuklara odaklanarak hafif tabakaları kaba bir şekilde hizalayabilir ve ardından numuneden gelen bilgileri kullanarak hizalamada ince ayar yapılabilir.
NOT: İki florofor (nükleer ve membran belirteçleri gibi) ile görüntüleme yaparken, optimal hizalama genellikle iki yapı için marjinal olarak farklıdır. Bu senaryoda, aşağı akış işlemedeki kısıtlamalara dayalı bir ayar seçin. Örneğin, hücre sınırları bölümlere ayrılacaksa, ki bu nispeten daha yoğun işlem gerektirir, ışık tabakalarını zar işaretleyicisine göre hizalayın.
- Çoklu görüntüleme görüntülemeyi ayarlama
- Işık levhalarını hizaladıktan sonra, çoklu görüntüleme görüntülemenin yanı sıra z-stack gerçekleştirme seçeneklerini etkinleştirin.
- Canlı taramayı başlatın ve ilk görünümü seçmek için numune navigatörünü kullanarak gezinin. Bu görünümde, dilim aralığını ve ardından z yığınının ilk ve son dilimlerini ayarlayın. Bu konumu, bu görünüm için konum ve z yığını bilgilerini not alan çoklu görünüm iletişim kutusuna ekleyin.
- Numune gezginindeki açıyı değiştirerek bir sonraki görünüme geçin. İkinci görünüm için z-stack'i ayarlayın ve bilgileri çoklu görüntü iletişim kutusuna ekleyin.
- Ek görünümler için de aynısını tekrarlayın.
NOT: Farklı görünümler arasında yeterli örtüşme olması için her görünümde belirli bir minimum sayıda dilimin görüntülendiğinden emin olun, bu da işleme sırasında görünümlerin kaydedilmesine yardımcı olur. Minimum dilim sayısı, yakınlaştırma faktörünün yanı sıra görüntüleme için seçilen açı sayısına da bağlı olacaktır. - Tüm görünümleri ayarladıktan sonra, bu bilgileri, z-yığını bilgilerini, tüpün (x, y, z) koordinatlarını ve her görünüm için açı özelliklerini içerecek bir metin dosyasına (örneğin 'embriyo pozisyonları' olarak adlandırılır) kaydedin. Kaydettikten sonra, çoklu görüntü iletişim kutusundan tüm konumları temizleyin.
- Görüntüleme nispeten büyük olan sağlam bir koryon ile yapıldığından, tüp içindeki boncukları aynı ayarlarla görüntülemek mümkün değildir. Bu nedenle, boncuk bilgisinin tüp içinde farklı bir konumdan elde edilmesi gerekir. Bunu yapmak için, boncukların göründüğü embriyodan uzakta farklı bir 'y' koordinatına çevirin. Bu konumu çoklu görüntü iletişim kutusuna ekleyin ve bu konumu bir metin dosyasına kaydedin (örneğin, 'beads-y' olarak adlandırılır).
NOT: İki pozisyondaki tüp eğriliğindeki olası farklılıkların etkilerini en aza indirmek için boncukları embriyo örneğine mümkün olduğunca yakın görüntüleyin. Bu nedenle, tüpe birden fazla embriyo monte edilmişse, boncukları oluşturmak için tüpler arasında biraz agaroz bırakmak önemlidir (adım 1.3.7'de belirtildiği gibi). - Metin dosyalarının kaydedildiği klasöre gidin ve 'embriyo pozisyonları' dosyasını 'boncuk pozisyonları' adlı yeni bir dosyaya çoğaltın. 'beads-positions' dosyasındaki tüm görünümler için y koordinatını 'beads-y' dosyasındaki y koordinatıyla değiştirin. Bu, boncukların aynı sayıda görünüm, z-yığını ve x-koordinatlarıyla, ancak tüpte farklı bir y konumunda görüntülenmesini sağlayacaktır.
- Görüntüleme yazılımına geri dönün ve yazılıma 'boncuk pozisyonları' dosyasını yükleyin. Zaman serisi seçeneği etkinleştirilirse, bir döngü seçin ve denemeyi başlatın. Boncuk görüntülerini, görüntü işleme sırasında farklı görünümleri kaydetmek için kullanılacak olan 'boncuklar' olarak kaydedin.
- Boncuk görüntülerini çektikten sonra, pozisyonları temizleyin ve yazılıma ilk 'embriyo pozisyonları' dosyasını yükleyin. İstenilen döngü sayısını uygun bir zaman aralığı ile ayarlayın ve deneyi başlatın.
3. Çoklu görüntü analizi
NOT: Çoklu görüntüleme görüntülerini kaynaştırmak için, Çoklu Görüntü Yeniden Yapılandırma eklentisinin en son sürümü olan bir FIJI eklentisi olan BigStitcher kullanılır 10,17,18. Eklenti, 'Yardım' menüsü altındaki 'Güncelle' seçeneğinden erişilebilen 'Güncelleme Sitelerini Yönet' işlevine BigStitcher eklentisi eklenerek yüklenebilir. Eklenti kurulduktan sonra 'Eklentiler' menüsü altında görünecektir. Füzyonda yer alan geniş adımlar aşağıdaki gibidir: (1) Hem boncuklar hem de embriyolar için .xml/.h5 dosya çiftlerini tanımlayın; (2) Tüm görünümleri boncuk dosyasıyla kaydedin; (3) Dekonvolüsyon için kullanılabilen boncuklar için Ekstraksiyon Noktası Yayılma Fonksiyonu (PSF) (Şekil 2A); (4) Kayıt ve PSF bilgilerini boncuk dosyasından embriyo dosyasına aktarın ve çoklu görüntü evrişimini başlatın. Bu adımların çoğu daha önce ayrıntılıolarak açıklanmıştır 21 ve burada farklı şekilde işlenen adımlar açıklanmaktadır.
- .xml veri kümesi tanımlama
- Daha önce açıklandığı gibi hem boncuklar hem de embriyo için sırasıyla 'beads.xml' ve 'embryo.xml' olarak yeni bir veri seti tanımlayın21.
- İlgi alanlarını kullanarak tespit ve kayıt
- beads.xml dosyasının başarılı bir şekilde dışa aktarılmasının ardından, 'Çoklu Görünüm Gezgini' iletişim kutusunda kayıt için gerekli olan görünümleri seçin (Şekil 2B). Sağ tıklayın ve İlgi Alanlarını Algıla'yı seçin. Açıklanan adımlaradevam edin 21.
- İlgi alanlarını belirledikten sonra, tüm görünümleri seçin, sağ tıklayın ve İlgi Alanı Puanlarını Kullanarak Kaydol'u seçin. Açıklanan protokolü izleyin21.
- Boncuk kaydının başarıyla çalışıp çalışmadığını dikkatlice kontrol edin. Başarılı bir kayıttan sonra, farklı görünümlerden üst üste binen boncuklar üst üste bindirilir (Şekil 2C). Kaydın ne kadar hassas bir şekilde çalıştığını kontrol etmek için, art arda iki görünüm seçin, çakışan bölgeye gidin ve farklı açılardan görüntülenen boncukların üst üste bindirilip bindirilmediğini gözlemlemek için iki görünüm arasında geçiş yapın. Bunu art arda her görünüm için tekrarlayın.
- Çakışma kesin değilse, kayıt için gereken önemi ve/veya kabul edilebilir çakışma hatasını ('Kayıt' iletişim kutusunda 'RANSAC hatası' olarak adlandırılır) gevşeterek kaydı yeniden deneyin.
- Başarılı bir kaydın ardından, güncellenmiş .xml dosyasını kaydetmek için 'Multiview Explorer' penceresinde Kaydet'e tıklayın.
NOT: Kayıt verimliliği hakkında daha ayrıntılı bilgi içerdiğinden günlük dosyasını da kaydedin. Kayıt bilgilerini boncuklardan embriyoya çevirmek için aşağıda açıklanan adımları izleyin. - bead.xml dosyasını tercih ettiğiniz bir metin düzenleyicide açın ve tüm bloğu "ViewRegistrations" altında kopyalayın.
- embryo.xml açın ve "ViewRegistrations" bloğunu boncuk dosyasından kopyalanan blokla değiştirin. Birden fazla kanal varsa, her kanal için kayıt bilgilerini yukarıdaki gibi değiştirin. Kayıt bilgileri manuel olarak veya buradan indirilebilen özel olarak yazılmış MATLAB kodu kullanılarak aktarılabilir: https://github.com/sundar07/Multiview_analysis
- embryo.xml dosyasını "BigStitcher"da açın ve kaydın embriyo için başarıyla çalışıp çalışmadığını dikkatlice kontrol edin. Her iki ardışık görünümde ilgilenilen yapıların örtüşmesini kontrol ederek boncuklar için yapılanın aynısını tekrarlayın.
NOT: Bazen, boncukların mükemmel bir şekilde kaydedilmesine rağmen embriyo kaydı arzu edilen kadar olmayabilir. Bu, boncukların görüntülendiği pozisyondan embriyo pozisyonuna kadar tüp eğriliğinde küçük değişiklikler varsa mümkündür. Ek olarak, koryon içinde serbestçe yüzdüğü için embriyonun görünümler arasında ince hareketleri olabilir. Bu durumda, embriyoda bir nükleer belirteç kullanarak ikinci bir kayıt turu gerçekleştirin. - Bunu yapmak için, embryo.xml dosyasını "BigStitcher"da açın, nükleer işaretleyiciye sahip tüm görünümlere sağ tıklayın ve İlgi Noktalarını Tespit Et'i seçin.
- İlgi noktalarını 'çekirdek' olarak yeniden adlandırın ve boncuklar için gerçekleştirilen adımlara devam edin.
- 'Gauss Farkı' parametrelerini ayarlarken, çekirdeğin tamamının olmasa da çoğunun tespit edildiğinden ve ektopik nükleer tespit olmadığından emin olun. Ardından, Bitti'ye tıklayın.
- Bunu takiben, İlgi Alanlarını kullanarak kaydol'u seçerek bu görünümleri kaydedin ve Kesin tanımlayıcı tabanlı (çeviri değişmez) seçeneğini seçin. 'Tüm görüntülemeleri ve ilgi noktalarını karşılaştır' seçeneğinin seçili olduğundan emin olun ve ilgi noktası olarak 'çekirdekler' kullanın. İlk görünümü düzeltme seçeneğini kullanın ve geri eşlemeyin.
- Kayıt için, katı düzenlemeli bir afin model ve eklentideki varsayılan parametreler kullanın.
- Her iki ardışık görünümü karşılaştırarak kaydın başarısını yeniden kontrol edin.
- Başarılı bir kaydın ardından, güncellenmiş .xml dosyasını kaydetmek için 'Multiview Explorer' penceresinde Kaydet'e tıklayın.
- embryo.xml dosyasını tercih ettiğiniz bir metin düzenleyicide açın ve kayıt bilgilerini nükleer belirteçlerden diğer kanallara kopyalayın.
- Nokta yayılma fonksiyonu çıkarma ve atama
- Çoklu görüntü dekonvolüsyonunu gerçekleştirmek için, görüntüleme sisteminin PSF'sini kayıtlı boncuk veri setinden çıkarın ve embriyo dosyasına uygulayın.
- Bu bilgileri almak için, boncuk dosyasındaki tüm görünümleri seçin ve ardından sağ tıklatıp Nokta Yayılma İşlevleri ve Ayıkla seçeneklerini belirleyin.
- Görünen iletişim kutusunda, Karşılık Gelen İlgi Noktalarını Kullan ve PSF'den minimum yoğunluk projeksiyonlarını kaldır seçeneklerinin işaretli olduğundan emin olun, varsayılan PSF boyutlarıyla ilerleyin ve Tamam'a tıklayın.
NOT: PSF ayıklama işlemi tüm görünümler için başarılı olursa, günlük dosyasında 'Çıkarılan n/n PSF'ler' görüntülenir - Bunu takiben, .xml dosyasını yeniden kaydedin. 'Multiview Explorer' iletişim kutusunun PSF sütununda işaretli bir onay kutusu görünecek ve çıkarılan tüm PSF'lerle birlikte ilgili klasörde bir 'psf' klasörü oluşturulacaktır.
- embryo.xml dosyasını açın ve her görünüm için PSF'yi ayrı ayrı atayın. "Bir görünüme" sağ tıklayın → Nokta Yayılma İşlevleri'ne tıklayın → Ata'yı seçin → Gelişmiş'i seçin, ardından Tüm seçili görünümlere yeni PSF ata'yı seçin. Gözat'a tıklayın, .xml dosya yoluna gidin ve psf klasörünü açın.
- Seçilen görünümde karşılık gelen kimliğe sahip eşleşen PSF'yi seçin ve Tamam'a tıklayın, ardından 'Çoklu Görünüm Gezgini' penceresinde PSF onay kutusu işaretli olarak görünecektir.
- Diğer tüm görünümler için işlemi tekrarlayın.
- Çoklu görüntü füzyonu ve evrişim çözme
- Tüm görünümler için nokta yayılma işlevi atandıktan sonra, sağ tıklayın ve Çoklu Görüntü Evrişimini Çözme'yi seçin.
- Sınırlayıcı kutuyu şu anda seçili görünümler olarak seçin. Bu iş için, varsayılan OSEM hızlandırması ve yineleme sayısı iyi çalışır.
- Daha hızlı hesaplama isteniyorsa veya CPU belleği sınırlanıyorsa görüntüleri gerektiği gibi alt örnekleyin.
NOT: Gerekli RAM mevcut belleği aşarsa, pencerenin altında kırmızı renkli bir uyarı hata mesajı açılır. Bu uyarı açılırsa evrişimi kaldırmayı başlatmayın, çünkü eklenti işleme sırasında bir noktada duracak ve yanıt vermeyi durduracaktır. - Evrişimin ilerlemesini değerlendirmek için, her 5 yinelemede bir sonuçları görüntüleyecek olan günlük dosyasını kontrol edin.
- Hesaplama hızını artırmak için, daha önce kuruluysa GPU'da çoklu görüntü evrişimini çözme işlemi gerçekleştirin.
NOT: Bu işlemin sonunda, tiff dosyası olarak kaydedilebilen kaynaşmış bir görüntü penceresi açılacaktır.
Sonuçlar
Numuneyi hassas bir şekilde yönlendirmek, bir mikroskopi kurulumunu verimli bir şekilde kullanmanın hayati bir parçasıdır. Bununla birlikte, numunelerin bir tüpte hazırlanması gerekliliği göz önüne alındığında, çok görüntülü bir ışık tabakası sistemi kullanılırken numunelerin manuel olarak yönlendirilmesi genellikle mümkün değildir. Bu nedenle, embriyoların koryon içinde aldığı stereotipik pozisyonlar olup olmadığını kontrol etmek için, zebra balığı embriyoları %70 epibolide (döllenmeden yaklaşık 7 saat sonra (hpf)) görüntülendi, çünkü gastrulasyondan erken somit aşamalarına kadar hızlandırılmış görüntüleme bu çalışmanın odak noktasıydı. Numuneler% 70 epibolide görüntülemeden hemen önce hazırlandığında, embriyolar numuneler arasında sıklıkla gözlenen spesifik bir yönelim göstermedi. Bu genellikle istenen bir durum olmadığından, örnekler gastrulasyondan çok önce hazırlandı ve görüntülemenin başlangıcına kadar uygun sıcaklıkta mikrosantrifüj tüplerinde saklandı. Bu koşullar altında,% 70 epibolide (N = 3; n = 87 embriyo), embriyo yönelimleri şu şekilde sınıflandırılabilir: (1) Embriyonun hayvan-bitkisel (AV) ekseni polimer tüpün uzun eksenine dik olduğunda yatay, (2) AV ekseni polimer tüpün uzun eksenine paralel olduğunda dikey ve, (3) Eğik, AV ekseni dar bir açıda olduğunda (Şekil 3A). Yatay konum en az temsil edilirken, dikey ve eğik konumlar eşit olarak gözlendi (Şekil 4).
Embriyolar tüplerde bırakıldığında, embriyolar% 90 epiboliye kadar bu ilgili pozisyonlarda stabildi, daha sonra çoğu embriyo yönelimlerini değiştirdi. Bu nedenle, değişen yönelimleri hesaba katmak için embriyogenezin tomurcuk aşamasında (yaklaşık 10 hpf) ikinci bir yönelim dokümantasyonu turu gerekliydi. Bu, bağımsız olarak hazırlanmış numuneler için gerçekleştirildi. Erken somit evrelerinin görüntülenmesi için, daha önce, polimer tüpün uzun eksenine ortogonal notokordlu bir embriyonun, eksen15 boyunca oluşan çoklu bilateral somitlerin görüntülenmesine izin verdiği için ideal yönelim olduğu bildirilmişti. Örnekler gastrulasyondan önce hazırlandığında (N = 3, n = 93 embriyo), embriyoların yaklaşık% 25'i bu yönelimi sergiledi (Şekil 4) ve bu embriyolar, önceki raporlarla tutarlı olarak en az 8 somit aşamasına kadar bu yönelimde sabit kaldı15. Embriyoların geri kalanı tomurcuk aşamasında çeşitli başka yönelimler sergiledi ( Şekil 3B ve Şekil 4'te sınıflandırılmıştır); Bununla birlikte, birçoğu erken somit oluşumu sırasında yatay konuma yeniden yönlendirildi. İlginç bir şekilde, embriyoların benzer bir yüzdesi, numunenin gastrulasyondan önce, %70 epibolide veya tomurcuk aşamasından hemen önce hazırlanıp hazırlanmadığına bakılmaksızın tomurcuk aşamasında yatay bir yönelim sergiledi. Bu nedenle, numune hazırlama zamanlaması, %70 epibolide görüntüleme için gözlemlenenin aksine, somit aşamalarının görüntülenmesi için ilgilenilen yatay yönelim için daha az kritik görünmektedir.
Çoklu görüntüleme sisteminin avantajı, aynı numuneyi birden fazla açıdan görüntüleme yeteneğidir. Bununla birlikte, bahsedilen aşamalardaki zebra balığı embriyoları için, tüm embriyo boyunca hücresel çözünürlük elde etmek için gereken görüntüleme sayısı net değildir. Bunu karakterize etmek için, zebra balığı embriyoları, 4.57 μm'lik bir hafif tabaka kalınlığına karşılık gelen 1'lik bir yakınlaştırma faktörü ile algılama kolunda 20x/1 NA objektif kullanılarak görüntülendi. Bu ayarlar altında, embriyo, 6.5 μm piksel boyutuna ve 1920 x 1920 piksellik bir alana sahip bir sCMOS kameranın tüm görüş alanını kapladı. Aktin filamentlerini (Utr-GFP) işaretleyen transgenik bir çizgiden elde edilen 1 hücreli evre embriyolarda histon etiketli bir florofor (H2A-mCherry) için mRNA'nın mikroenjeksiyonu, embriyodaki hem çekirdeklerin hem de hücre zarlarının görüntülenmesine izin verdi. Farklı açısal aralıklara sahip embriyoların çoklu görünüm füzyonunu gerçekleştirmek için, çift transgenik embriyoların yanı sıra tüpteki boncukların 360° edinimi her 30° (% 70 epibolide n = 3 embriyo; tomurcuk aşamasında n = 3 embriyo) veya 45 ° (% 70 epibolide n = 3 embriyo; tomurcuk aşamasında n = 3 embriyo) aralıklarla, her açıda yaklaşık 100 dilim ve 2 μm'lik bir dilim aralığı ile gerçekleştirildi. Görüntü işleme sırasında alternatif açıları atlayarak, 30° ve 45° alımları ek olarak 60° ve 90° veri setlerini sağladı.
Elde edilen görüntüler daha sonra boncuk bilgileri kullanılarak kaydedildi ve kayıt detayları protokol bölümünde anlatıldığı gibi embriyo veri setine aktarıldı. BigStitcher eklentisi kullanılarak başarılı bir kayıt, 90° veri seti dışındaki farklı alımlar için elde edildi, bu da muhtemelen numunenin her açıdan daha az kapsanmasından kaynaklanıyor. Bunun üstesinden gelmek için, embriyolar her açıda yaklaşık 400 dilim ve 2 μm'lik bir dilim aralığı ile her 90°'de bir, hem %70 epiboli hem de tomurcuk aşamalarında görüntülendi ve bunlar başarıyla kaydedildi (her aşama için n = 3 embriyo).
Bir sonraki adım, kayıtlı veri kümelerinin çoklu görüntüleme füzyonunu ve evrişimini çözmeyi gerçekleştirmekti. Bu, hesaplamayı hızlandırmak için 4x altörnekleme ile yapıldı. Bireysel görünümlerden ve çok görüntülü yeniden yapılandırılmış bir embriyodan çekirdeklerin temsilinde görüldüğü gibi (Şekil 5), bireysel görünümler daha küçük bir görüş alanını kapsar ve bu da füzyon üzerine tüm embriyonun bir görüntüsünü verir. Temsil için, çekirdekler, FIJI'deki "Yardım" menüsünün "Güncelleme Sitelerini Yönet" bölümüne eklenebilen ve eklendikten sonra eklentiler menüsünden erişilebilen FIJI tabanlı bir eklenti olan Mastodon (https://github.com/mastodon-sc/mastodon) kullanılarak tespit edildi. Algılama için, ilgili görüntüler önce XML/hdf5 formatına dönüştürüldü ve ardından Mastodon'un bir DoG dedektörü (Çap 6 μm ve Kalite Eşiği 80) ile 'Algılama' eklentisi kullanılarak nükleer algılama gerçekleştirildi.
Farklı kaynaşmış görüntüler arasında, 90° veri seti, numunenin daha derinlerinde çok yüksek bir arka plan gösterdi ve bu da onu herhangi bir nicelik tespiti yapmak için uygun olmayan hale getirdi. Bu nedenle, diğer çalışmalarda sıklıkla 90° aralıklarla görüntülenen Drosophila embriyoları gibi daha küçük örneklerin aksine 9,22, 20x/1 NA objektif kullanılarak erken zebra balığı embriyolarının görüntülenmesi için aynı şey önerilmez. 30 °, 45 ° ve 60 ° kaynaşmış veri setleri arasında, niteliksel olarak, çekirdek bilgisinde önemli bir fark yoktu (Şekil 6B, üst sıra); bununla birlikte, hücre sınırları gibi daha ince yapılar, geri kalanına kıyasla 30° kaynaşmış veri seti ile çok daha iyi çözülmüş göründü (Şekil 6C, üst satır).
Bu gözlemi doğrulamak için Mastodon, her 30°, 45° ve 60°'de bir görüntülemeden elde edilen kaynaşmış görüntülerdeki çekirdekleri tespit etmek için kullanıldı. Kaynaşmış görüntülerde, her biri embriyo yüzeyinden 20 μm derinlikte (Şekil 6A), 50 μm ve 100 μm derinlikte olmak üzere üç bölge analiz için seçildi. Görüntüler arasında çekirdek tespitinin verimliliğini karşılaştırmak için, algılama, farklı açısal aralıklardan kaynaşmış görüntülerde aynı parametrelerle yukarıda açıklandığı gibi gerçekleştirildi. Analiz edilen tüm bölgelerde, her 30°, 45° veya 60°'de bir görüntülemeden elde edilen kaynaşmış görüntülerden bağımsız olarak her çekirdek tespit edildi (Şekil 6B, alt sıra). Böylece, çekirdekler gibi küresel yapılar, yukarıdaki açısal aralıkların herhangi birinde bilgi kaybı olmadan görüntülenebilir.
Hücre sınırlarını analiz etmek için,24,25,26 hücrelerini segmentlere ayırmak için rutin olarak kullanılan bir FIJI eklentisi olan Doku Analizörü23 kullanıldı. Mastodon'a benzer şekilde, Doku Analiz Cihazı eklentisi FIJI'deki "Yardım" menüsünün "Güncelleme Sitelerini Yönet" bölümüne eklenebilir ve eklendikten sonra eklentiler menüsünden erişilebilir. Hücre sınırları, varsayılan parametreler ve doku derinliğine bağlı olarak 1,5 ile 2 arasında değişen güçlü bir bulanıklık ve 1'lik zayıf bir bulanıklık ile havza algoritması kullanılarak bölümlere ayrıldı. Bu parametreler tüm analizlerde sabit tutuldu ve bu da basit bir karşılaştırmayı kolaylaştırdı. Bölümlere ayrılmış görüntüler orijinal giriş görüntüleriyle manuel olarak karşılaştırıldığında, yazılımın bir hücre sınırını algılayamadığı veya var olmayan hücre sınırları çizdiği hatalar gözlemlendi (Şekil 6C, alt satır). Doku analizörü eklentisi tarafından yapılan hata sayısı, bölgede tespit edilen toplam bağ sayısına normalize edildi ve 'Sınır segmentasyon hatası' olarak hesaplandı. Bu hatalar analiz edilen tüm bölgelerde mevcut olmakla birlikte, 45° ve 60° açısal aralıklarla görüntülemeden elde edilen kaynaşmış görüntülerde hata sayısı 30°'ye kıyasla büyük ölçüde artmıştır (Şekil 6D). Bu, kaynaşmış görüntülerdeki çözünürlüğün, açısal aralık arttırıldığında giderek daha kötü olduğunu gösterdi. Bu nedenle, hücre sınırları gibi daha ince yapıları segmentlere ayırmak için daha sıkı bir açısal aralık, aşağı akış işlemlerini kolaylaştırır.
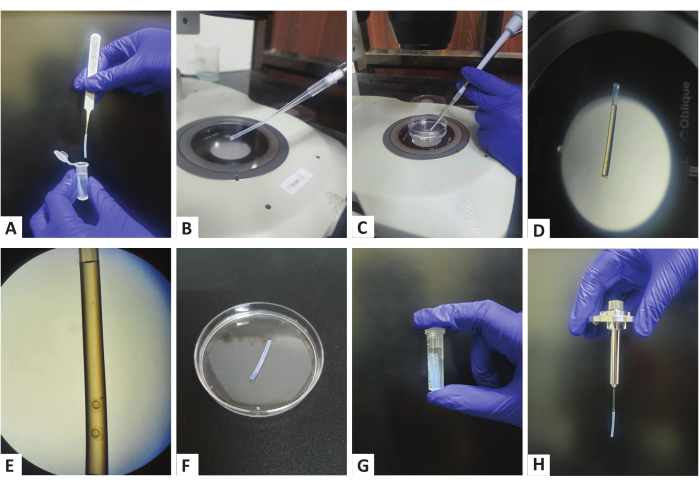
Şekil 1: Polimer tüpler kullanılarak numune hazırlama. (A) Depolanan mikrosantrifüj tüplerinden bir polimer tüp forseps kullanılarak alınır. (B) Tüpü 200 μL'lik bir mikropipetin ucuna takmak. (C) Embriyoların bir pipet yardımıyla tüp içine aspire edilmesi. (D,E) Tüpün dibine doğru görülebilen tomurcuk aşaması embriyolarına sahip bir polimer tüp. (F) Polimer tüpler, agarozu katılaştırmak için E3 içeren bir Petri kabına yerleştirilir. (G) Polimer tüplerin E3 ile doldurulmuş bir mikrosantrifüj tüpünde saklanması. (H) Monte edilmiş polimer tüp ile monte edilmiş numune tutucu. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2: Boncuklar kullanarak çoklu görüntü analizi iş akışı ve kaydı. (A) Çoklu görüntü analizi boru hattı. (B) Resim, bir FIJI eklentisi olan BigStitcher'da, her görünümün tek bir satır olarak göründüğü ve açı, kanal, kayıt, ilgi noktaları ve PSF hakkında bilgi içerdiği "Çoklu Görünüm Gezgini" penceresini gösterir. Protokolde tartışılan tüm komutlar, açılır menüde gösterildiği gibi bir görünüm seçildiğinde ve ardından sağ tıklama yapıldığında görünür. Seçilen görünümler, gösterildiği gibi BigDataViewer penceresinde görselleştirilebilir. (C) 30° açısal aralıklarla görüntülemeden elde edilen kayıttan önce (solda) ve kayıttan sonra (sağda) temsili bir boncuk görüntüsü. Tüm açısal görünümler seçildi ve görüntülendi. Ölçek çubukları: 75 μm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 3: Varsayılan embriyo oryantasyonlarına genel bakış. Embriyoların %70 epiboli (A) ve tomurcuk aşamalarında (B) koryon içinde içine düştüğü oryantasyonların temsili görüntüleri. Her paneldeki üst sıra, ışıklı levha sisteminden elde edilen parlak alan görüntülerini, alt sıra ise temsili karikatürleri gösterir. (B) 'deki oklar, oryantasyonu tanımlamak için kullanılan notokordun konumunu gösterir. Ap, hayvan direği; Vp, bitkisel direk; A, ön; P, arka. Ölçek çubukları: 100 μm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 4: Farklı embriyo yönelimlerinin istatistikleri. (A) Yığılmış sütun, gastrulasyondan önce örnekler hazırlandığında %70 epibolide belirtilen yönlere düşen embriyoların yüzdelerini gösterir. (B) Yığılmış sütunlar, numunelerin gastrulasyondan önce hazırlandığı tomurcuk aşamalarında (solda), %70 epibolide (ortada) ve tomurcuk aşamalarında (sağda) belirtilen yönlere düşen embriyoların yüzdelerini gösterir. N, embriyoların elde edildiği bağımsız kavramaların sayısı; n, toplam embriyo sayısı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 5: Çok görünümlü, yeniden yapılandırılmış bir embriyodan alınan çekirdeklerin temsili. (Bir)3D saçılma grafiği, 30̛° açısal aralıklarla görüntülenen çok görüntülü yeniden yapılandırılmış bir embriyoda tespit edilen çekirdekleri temsil eder. Her daire bir çekirdeği temsil eder ve çekirdek konumlarının merkezi çizilir. Nükleer koordinatlar, bir FIJI eklentisi olan Mastodon kullanılarak elde edildi. (B) Aynı embriyonun üç temsili görünümünden elde edilen 3B saçılma çekirdek grafikleri - tasvirler 60° aralıklıdır. Her çekirdek için renkler rastgele atandı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 6: Kaynaşmış görüntülerdeki hücresel ölçekli bilgilerin karşılaştırılması. (A) Soldaki görüntü, yüzeyden 20 μm derinlikte çok görüntülü yeniden yapılandırılmış bir embriyodan bir anlık görüntüyü göstermektedir. Sarı daire, daha fazla analiz için kullanılan bölgeyi gösterir. Ölçek çubukları: 50 μm (tüm panellerde). (B) Sırasıyla her 30 °, 45 ° ve 60 ° 'de bir görüntülenen üç veri setinden embriyonun yüzeyinden 20 μm derinlikte çekirdeklerin temsili ham görüntüleri (üstte). Bir FIJI eklentisi olan Mastodon tarafından aynı görüntülerde (yeşil renkte) çekirdek tespit edildi (altta). (C) Aktin markörü, Utr-GFP (üstte) kullanılarak sırasıyla her 30 °, 45 ° ve 60 ° 'de bir görüntülenen üç veri setinden embriyonun yüzeyinden 20 μm derinlikte temsili ham görüntüler. Bir FIJI eklentisi olan Tissue Analyzer tarafından segmentlere ayrılan sınırlar aynı görüntüler için gösterilir (altta). Oklar, Doku Analizörü tarafından sınır segmentasyonunda yapılan hataları gösterir, sarı ok eksik bir sınırı temsil eder ve beyaz ok, görsel olarak hiçbir sınır yokmuş gibi göründüğünde yanlış algılanan sınırları temsil eder. (D) Kutu çizimi, Doku Analiz Cihazının, her 30 °, 45 ° ve 60 ° 'de farklı derinliklerde görüntülenen çoklu görüntü yeniden yapılandırılmış embriyolar arasında yaptığı hataların yüzdesini gösterir. Hata çubukları, çeyrekler arası aralığın 1,5 katını gösterir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Bir embriyoyu ilgilenilen bölgeyi görüntülemek için doğru yönde konumlandırmak, bir kullanıcı için genellikle başarısız bir mikroskopi seansı ile sonuçlanan hız sınırlayıcı adımlardan biridir. Bu, numuneler bir tüp içine gömüldüğü için oryantasyonun manuel olarak manipüle edilmesinin zor olduğu çok görüşlü bir ışık tabakası mikroskobunda daha fazladır. Bu sürece yardımcı olmak için, bu çalışma, embriyolu polimer tüp bir mikrosantrifüj tüpünde dik tutulduğunda, bir zebra balığı embriyosunun bir koryon içinde% 70 epiboli ve erken somit aşamaları arasında aldığı çeşitli pozisyonların istatistiklerini bildirmektedir.
% 70 epibolide, dikey veya yatay olarak yönlendirilmiş embriyolar, tüm embriyo boyunca olayların tamamlayıcı bilgilerini sağlar. Yatay oryantasyon, hayvan kutbundaki hücresel dinamiklerin yanı sıra vegetal kutuptan bakıldığında epiboli geçiren hücrelerin dinamiklerinin görselleştirilmesine izin verirken, dikey oryantasyon, dorsal organizatördeki hücresel dinamikler, gastrulasyon sırasında hücrelerin içselleştirilmesi ve yakınsama-ekstansiyon akışları dahil olmak üzere embriyonun dorsal ve ventral taraflarının görselleştirilmesine izin verir. Bu çalışma, örneklerin görüntülemeden birkaç saat önce hazırlanması durumunda, tamamen şans eseri dikey yönelimde bir embriyo elde etme olasılığının yüksek olduğunu göstermektedir; Bununla birlikte, yatay yönelim bu aşamalarda çok daha az yaygın görünmektedir. %70 epiboliden önceki aşamalar için, kullanıcıların hızlandırılmış görüntülemeye devam etmeden önce koryon içindeki benzer embriyo oryantasyon istatistiklerini belgelemeleri önerilir, çünkü bu bilgilere önceden sahip olmak uzun vadede zaman kazandırır.
Tomurcuk aşamasından itibaren, görüntüleme için en iyi oryantasyon, yatay olarak konumlandırılmış bir embriyodur, çünkü bu, embriyoların tüm anteroposterior vücut ekseni boyunca hücresel dinamiklerin takip edilmesine izin verir15. Bu oryantasyonda bir embriyo elde etmek nispeten basittir, çünkü embriyoların yaklaşık% 25'i, numunenin ne zaman hazırlandığına bakılmaksızın bu stabil pozisyona düşer. Genel olarak, ilgilenilen herhangi bir aşamayı görüntülemek için, her tüpte iki ila üç embriyo bulunan yaklaşık 15 tüpün hazırlanması önerilir, bu da istenen oryantasyonda embriyo elde etme olasılığının yüksek olmasını ve dolayısıyla herhangi bir günde başarılı bir mikroskopi seansı yapılmasını sağlayacaktır. Ek olarak, hızlandırılmış görüntüleme için doğru oryantasyona sahip bir embriyo seçmek için tüplerin ilgilenilen aşamadan yaklaşık bir saat önce sıralanması önerilir.
Doğru yönde bir embriyo elde ettikten sonra, görüntüleme için görüntüleme sayısını ve görünümler arasındaki açı aralığını hesaba katmak önemlidir. Bu, gereken minimum uzamsal ve zamansal çözünürlüğe bağlıdır. Embriyonun belirli bir yapısı veya bölgesi ne kadar çok açıdan görüntülenirse, uzamsal çözünürlük o kadar iyi olur ( Şekil 6'da gösterildiği gibi), ancak bu zamansal çözünürlük üzerinde bir uzlaşmaya yol açar. Bu protokolde, iki aydınlatma ve bir algılama koluna sahip bir sistemle, 30°'lik bir açısal aralıkla tam 360° boyunca iki kanalın ve 2 μm'lik bir dilim aralığı ile her açıda yaklaşık 100 z-diliminin görüntülenmesi, bir kerelik nokta için yaklaşık 3 dakika sürdü. Bu, erken embriyonik aşamalardaki hücreleri izlemek için yeterli olsa da, daha hızlı dinamiklere sahip olaylar yakalanacaksa, daha az sayıda açı elde edilmelidir, bu da daha iyi zamansal çözünürlük sağlar, ancak uzamsal çözünürlükten ödün verilir, özellikle de hücre sınırları gibi daha ince yapıların bölümlere ayrılması gerekiyorsa. Zamansal çözünürlüğü iyileştirmek için göz önünde bulundurulması gereken ikinci bir seçenek, zebra balığı embriyolarında gastrulasyon sırasında hücre hareketlerini izlemek için yakın zamanda kullanılan edinme hızını önemli ölçüde artıracak iki algılama koluna sahip bir çoklu görüntüleme sistemi kullanmaktır16. Ek olarak, bu protokolde yalnızca 20x/1 NA hedefi test edilmiş olsa da, görüntülenen örneğe, gerekli uzamsal çözünürlüğe ve görüş alanına bağlı olarak doğru hedefin dikkate alınması gerekir. Birlikte ele alındığında, belirli bir numune ve görüş alanı için gerekli aşağı akış işleme ve miktar tayinine bağlı olarak, görüntüleme sayısının ve açı aralığının dikkatli bir şekilde seçilmesi gerekir. Bununla birlikte, tüm erken zebra balığı embriyolarının, özellikle birden fazla açıdan hücresel ölçekte çözünürlükle görüntülenmesi için, 20x/1 NA hedefi optimal görünmektedir, çünkü daha düşük büyütmeli bir hedef ve NA (örneğin, 10x/0.5 NA objektif) çok daha düşük çözünürlüğe sahip olacaktır, bu da muhtemelen daha fazla segmentasyon hatasına yol açarken, daha yüksek büyütme ve NA olan bir objektif, kameranın görüş alanındaki tüm embriyoyu kaplamak için uygun olmayacaktır.
Bu çalışmada, montaj ve görüntüleme stratejilerini tartışmak için, hücre sınırlarının izlenmesi için bir vekil olarak kullanılabilecek bir aktin markörü ve çekirdekleri tespit etmek ve izlemek için bir histon markörü kullanılmıştır. Aslında, çok görüşlü bir ışık tabakası sistemi kullanan daha önce yayınlanmış çalışmaların çoğu, tercihen benzer belirteçleri 5,7,15,16,27 kullanmıştır. Bu belirteçlerin seçilmesinin ana nedeni, çoklu görüntü ışık tabakası mikroskobunun büyük ölçüde, doku ölçeklerinde veya birkaç saatten günlere kadar uzanan uzun süreler boyunca tüm embriyo boyunca hücresel ölçekli dinamikleri (örneğin, hücre şekli ve hücrenin yeniden düzenlenmesi) takip etmek için kullanılmasıdır. Öte yandan, hücre altı çözünürlük elde etmeye ihtiyaç duyulursa, ilgilenilen bölgeyi işaretleyen farklı bir transgenik çizgi kullanılabilir, ancak bu senaryoda, çok görüşlü bir ışık tabakası mikroskobu en iyi alet seçimi olmayabilir ve daha ziyade bir konfokal, süper çözünürlüklü bir sistem veya hatta bir kafes ışık tabakası mikroskobu28, 29 araştırılan soruna daha uygun olabilir.
Tartışılan protokol, gastrulasyondan 15 somit aşamalı zebra balığı embriyolarına30 (yaklaşık 17 hpf) herhangi bir değişiklik yapmadan iyi çalışır ve burada test edilmemesine rağmen, gastrulasyon aşamasından önce daha erken embriyolar için de işe yaraması muhtemeldir. 15 somit aşamasının ötesinde, spontan kas kasılmaları30,31 başlar ve bu, tüpe ve numune odasına bir anestezik olan trikserin eklenerek bastırılabilir. Ek olarak, yaklaşık 18 hpf'de, yumurta sarısından uzaklaşan kuyruk versiyonu30 başlar ve bu da embriyoyu görüş alanından uzaklaştırır. Bununla ilgilenmek için, embriyoyu odakta tutmak için uzayan ucu izleyen bir izleme algoritmasının kullanılması gerekir32. Ayrıca, nöral gelişimi takip etmek gibi bir koryondan yoksun olduklarında daha sonraki evre embriyoları görüntülemek için, yakın zamanda gerçekleştirilen gibi alternatif bir montaj stratejisinin kullanılması gerekir33.
Sağlam bir koryona sahip zebra balığı embriyolarını görüntülemek için, hem kayıt belirteçleri olarak işlev gören boncukları hem de aynı görüş alanındaki embriyoyu yakalamak 20x/1 NA objektif kullanılarak mümkün değildir. Bu çalışma, numunenin üstünde veya altında bulunan boncuklardan elde edilen bilgilerle farklı görünümleri kaydederek ve ardından işleme sırasında kaydı boncuklardan embriyoya kaydırarak basit bir alternatif sunmaktadır. Embriyo numunesi bu şekilde kaydedildikten sonra, ilk kaydın daha da ince ayar yapılması için numune içinde bulunan kayıt belirteçleri olarak çekirdekler kullanılarak ikinci bir kayıt turu gerçekleştirilebilir. Hızlandırılmış görüntüleme yapılırken, çekirdek bilgileri, sırayla, daha önce21 bildirildiği gibi ardışık zaman noktalarının kaydedilmesi için kullanılabilir. Bu protokolün bir alternatifi, boncuklardan vazgeçmek ve bunun yerine ilk kayıt için çekirdek bilgilerini kullanmaktır. Ancak, BigStitcher eklentisinde, muhtemelen işleme sırasında embriyoda daha derinde tespit edilmeyen birçok çekirdeğin bir kombinasyonu ve ayrıca belirli açılardan bakıldığında embriyoda bulunan nispeten daha az sayıda çekirdek nedeniyle (örneğin, geç epiboli aşamalarında ventral olarak bakıldığında) kayıt genellikle başarısız oldu (burada gösterilmemiştir).
Bu protokolde, kayıt için numunenin üstünden veya altından alınan boncuk bilgileri kullanıldığından ve numunenin görüntülenmesi ile aynı görünümde boncukların görüntülenmesine gerek olmadığından, bu protokol tüm embriyo yerine sadece belirli ilgi alanlarını görüntülemek için ancak farklı açılardan yüksek büyütme ile kullanılabilir. Ayrıca, kayıt için numuneler arasındaki boncuk bilgilerini kullanarak tüpte dikey olarak istiflenmiş birden fazla numuneyi görüntülemek için bu protokolü uyarlamak kolaydır.
Açıklamalar
Yazarlar herhangi bir rekabet çıkarı beyan etmemektedir.
Teşekkürler
Balık tesisinin bakımı için Dr. Kalidas Kohale ve ekibine ve ışık tabakası mikroskobunun bakımı için KV Boby'ye teşekkür ederiz. SRN, Hindistan Hükümeti Atom Enerjisi Departmanı'ndan (DAE) mali destek aldığını kabul eder (Proje Tanımlama no. RTI4003, 11.02.2020 tarihli ve 1303/2/2019/R\&D-II/DAE/2079 sayılı DAE OM), Max Planck Society Ortak Grup programı (M.PG. A MOZG0010) ve Bilim ve Mühendislik Araştırma Kurulu Başlangıç Araştırma Hibesi (SRG/2023/001716).
Malzemeler
| Name | Company | Catalog Number | Comments |
| Agarose, low gelling temperature | Sigma-Aldrich | A9414 | |
| Calcium Chloride dihydrate | Sigma-Aldrich | 12022 | |
| FIJI | Version: ImageJ 1.54f | ||
| Latex beads, carboxylate-modified polystyrene, fluorescent red, 0.5 μm mean particle size, aqueous suspension | Sigma-Aldrich | L3280 | |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | |
| mMESSAGE mMACHINE SP6 Transcription kit | ThermoFischer Scientific | AM1340 | For in vitro transccription of H2A-mCherry plasmid |
| Potassium Chhloride | Sigma-Aldrich | P9541 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P0662 | |
| PTFE Sleeving AWG 15L - 1.58 mm ID x 0.15 mm Wall +/-0.05 | Adtech Innovations in Fluoroplastics | STW15 | PTFE tubes |
| Sodium Chloride | Sigma-Aldrich | S3014 | |
| Sodium phosphate dibasic | Sigma-Aldrich | 71640 | |
| Ultrasonic Cleaner | Labman | LMUC3 | Ultrasonicator |
| Zeiss LightSheet 7 System | Zeiss |
Referanslar
- Wan, Y., McDole, K., Keller, P. J. Light sheet microscopy and its potential for understanding developmental processes. Annu Rev Cell Dev Biol. 35, 655-681 (2019).
- Keller, P. J., Schmidt, A. D., Wittbrodt, J., Stelzer, E. H. Reconstruction of zebrafish early embryonic development by scanned light sheet microscopy. Science. 322 (5904), 1065-1069 (2008).
- Keller, P. J., et al. high-contrast imaging of animal development with scanned light sheet-based structured-illumination microscopy. Nat Methods. 7 (8), 637-642 (2010).
- Keller, P. J. Imaging morphogenesis: Technological advances and biological insights. Science. 340 (6137), 1234168(2013).
- Schmid, B., et al. High-speed panoramic Light sheet microscopy reveals global endodermal cell dynamics. Nat Commun. 4 (1), 2207(2013).
- Strnad, P., et al. Inverted Light sheet microscope for imaging mouse pre-implantation development. Nat Methods. 13 (2), 139-142 (2016).
- McDole, K., et al. In toto imaging and reconstruction of post-implantation mouse development at the single-cell level. Cell. 175 (3), 859-876 (2018).
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Krzic, U., Gunther, S., Saunders, T. E., Streichan, S. J., Hufnagel, L. Multiview Light sheet microscope for rapid in toto imaging. Nat Methods. 9 (7), 730-733 (2012).
- Preibisch, S., Saalfeld, S., Schindelin, J., Tomancak, P. Software for bead-based registration of selective plane illumination microscopy data. Nat Methods. 7 (6), 418-419 (2010).
- Swoger, J., Verveer, P., Greger, K., Huisken, J., Stelzer, E. H. K. Multiview image fusion improves resolution in three-dimensional microscopy. Opt Express. 15 (13), 8029-8042 (2007).
- Swoger, J., Huisken, J., Stelzer, E. H. Multiple imaging axis microscopy improves resolution for thick-sample applications. Opt Lett. 28 (18), 1654-1656 (2003).
- Kaufmann, A., Mickoleit, M., Weber, M., Huisken, J. Multilayer mounting enables long-term imaging of zebrafish development in a light sheet microscope. Development. 139 (17), 3242-3247 (2012).
- Weber, M., Mickoleit, M., Huisken, J. Multilayer mounting for long-term light sheet microscopy of zebrafish. J Vis Exp. (84), e51119(2014).
- Naganathan, S. R., Popović, M., Oates, A. C. Left-right symmetry of zebrafish embryos requires somite surface tension. Nature. 605 (7910), 516-521 (2022).
- Shah, G., et al. Multi-scale imaging and analysis identify pan-embryo cell dynamics of germlayer formation in zebrafish. Nat Commun. 10 (1), 5753(2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Hörl, D., et al. BigStitcher: Reconstructing high-resolution image datasets of cleared and expanded samples. Nat Methods. 16 (9), 870-874 (2019).
- Behrndt, M., et al. Forces driving epithelial spreading in zebrafish gastrulation. Science. 338 (6104), 257-260 (2012).
- Westerfield, M. The Zebrafish Book: A Guide for the Laboratory Use of Zebrafish (Danio Rerio). , University of Oregon Press. (2000).
- Icha, J., et al. Using light sheet fluorescence microscopy to image zebrafish eye development. J Vis Exp. (110), e53966(2016).
- Tomer, R., Khairy, K., Amat, F., Keller, P. J. Quantitative high-speed imaging of entire developing embryos with simultaneous multiview Light sheet microscopy. Nat Methods. 9 (7), 755-763 (2012).
- Aigouy, B., Prud'homme, B. Segmentation and quantitative analysis of epithelial tissues. Methods Mol Biol. 2540, 387-399 (2022).
- Mancini, L., et al. Apical size and deltaA expression predict adult neural stem cell decisions along lineage progression. Science Adv. 9, 7519(2023).
- Piscitello-Gomez, R., Mahmoud, A., Dye, N., Eaton, S. Sensitivity of the timing of Drosophila pupal wing morphogenesis to external perturbations. bioRxiv. , (2023).
- Tsuboi, A., Fujimoto, K., Kondo, T. Spatiotemporal remodeling of extracellular matrix orients epithelial sheet folding. Science Adv. 9, 2154(2023).
- Fu, Q., Martin, B. L., Matus, D. Q., Gao, L. Imaging multicellular specimens with real-time optimized tiling Light sheet selective plane illumination microscopy. Nat Commun. 7 (1), 11088(2016).
- Chen, B. C., et al. Lattice Light sheet microscopy: imaging molecules to embryos at high spatiotemporal resolution. Science. 346 (6208), 1257998(2014).
- York, H. M., et al. Deterministic early endosomal maturations emerge from a stochastic trigger-and-convert mechanism. Nat Commun. 14 (1), 4652(2023).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Melançon, E., Liu, D. W. C., Westerfield, M., Eisen, J. S. Pathfinding by identified zebrafish motoneurons in the absence of muscle pioneers. J Neurosci. 17 (20), 7796(1997).
- Rohde, L. A., et al. Cell-autonomous timing drives the vertebrate segmentation clock's wave pattern. eLife. , 13:RP93764 (2024).
- Haynes, E. M., et al. KLC4 shapes axon arbors during development and mediates adult behavior. eLife. 11, e74270(2022).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır