Method Article
Lichtblattmikroskopie: Bildgebung und Montagestrategien für frühe Zebrafischembryonen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Eine Probenvorbereitungsstrategie für die Abbildung früher Zebrafischembryonen in einem intakten Chorion mit einem Lichtblattmikroskop wird beschrieben. Es analysiert die verschiedenen Orientierungen, die Embryonen innerhalb des Chorions in den 70% Epibolie- und Knospenstadien annehmen, und beschreibt bildgebende Strategien, um mit Hilfe des Lichtblattsystems eine zelluläre Auflösung im gesamten Embryo zu erzielen.
Zusammenfassung
Die Lichtblattmikroskopie hat sich zur Methode der Wahl für die Live-Bildgebung von Zebrafischembryonen über lange Zeiträume mit minimaler Phototoxizität entwickelt. Insbesondere ein Multiview-System, das eine Probenrotation ermöglicht, ermöglicht die Bildgebung ganzer Embryonen aus verschiedenen Blickwinkeln. Bei den meisten Bildgebungssitzungen mit einem Multiview-System ist die Probenmontage jedoch ein mühsamer Prozess, da die Proben in der Regel in einem Polymerröhrchen vorbereitet werden. Um diesen Prozess zu unterstützen, beschreibt dieses Protokoll grundlegende Aufstiegsstrategien für die Abbildung der frühen Entwicklung von Zebrafischen zwischen dem 70%-Epibolie- und dem frühen Somitenstadium. Konkret liefert die Studie Statistiken über die verschiedenen Positionen, in denen sich die Embryonen in den 70% Epibolie- und Knospenstadien innerhalb des Chorions befinden. Darüber hinaus wird die optimale Anzahl von Winkeln und das Intervall zwischen den Winkeln diskutiert, die für die Abbildung ganzer Zebrafischembryonen in den frühen Entwicklungsstadien erforderlich sind, damit durch Verschmelzung der verschiedenen Ansichten Informationen auf zellulärer Ebene extrahiert werden können. Da der Embryo schließlich das gesamte Sichtfeld der Kamera abdeckt, was erforderlich ist, um eine Auflösung im Zellmaßstab zu erhalten, beschreibt dieses Protokoll den Prozess der Verwendung von Perleninformationen von oberhalb oder unterhalb des Embryos für die Registrierung der verschiedenen Ansichten.
Einleitung
Die Gewährleistung einer minimalen Phototoxizität ist eine wichtige Voraussetzung für die Bildgebung lebender Embryonen mit hoher räumlich-zeitlicher Auflösung über lange Zeiträume. In den letzten zehn Jahren hat sich die Lichtblattmikroskopie zur Methode der Wahl entwickelt, um diese Anforderung zu erfüllen 1,2,3,4,5,6,7. Kurzgesagt, bei dieser Technik, die erstmals 2004 zur Erfassung von Entwicklungsprozessen 8 eingesetzt wurde, passieren zwei ausgerichtete dünne Laserschichten den Embryo von gegenüberliegenden Enden und beleuchten nur die interessierende Ebene. Ein Detektionsobjektiv wird orthogonal platziert, und dann wird das emittierte Fluoreszenzlicht von allen beleuchteten Punkten in der Probe gleichzeitig gesammelt. Ein 3D-Bild wird dann erhalten, indem der Embryo nacheinander durch das statische Lichtblatt bewegt wird.
Darüber hinaus können in einer speziellen Form dieser Methodik, die als Multiview-Lichtblattmikroskopie bezeichnet wird, die Proben in einem Polymerröhrchen suspendiert werden, das mit Hilfe eines Rotors gedreht werden kann, was die Abbildung desselben Embryos aus mehreren Winkeln ermöglicht 9,10,11. Nach der Bildgebung werden die Bilder aus mehreren Blickwinkeln auf der Grundlage von Registrierungsmarkern fusioniert, bei denen es sich in der Regel um kugelförmige Fluoreszenzmarker im Embryo (z. B. Zellkerne) oder in der Röhre (z. B. fluoreszierende Kügelchen) handelt. Multiview-Bildgebung und -Fusion verbessern die axiale Auflösung erheblich und bieten eine isotrope Auflösung über alle drei Dimensionen12. Dies ist zwar ein großer Vorteil, aber eine große Herausforderung der Multiview-Methodik ist die Probenaufnahme, bei der die Embryonen während des gesamten Verlaufs der Bildgebung in den Eileitern montiert und an Ort und Stelle gehalten werden müssen.
Für die Durchführung von Multiview-Bildgebungen, um die Embryonen an Ort und Stelle zu halten und Bewegungen während der Bildgebung zu verhindern, können Embryonen in Agarose eingebettet werden. Dies führt jedoch oft zu einem nachteiligen Wachstum und einer nachteiligen Entwicklung, insbesondere bei Zebrafischembryonen im Frühstadium13, dem Modellsystem, das hier diskutiert wird. Eine zweite Montagestrategie besteht darin, einen dünnen Schlauch zu verwenden, der nur geringfügig größer als der Durchmesser des Embryos ist, wobei der Embryo zusammen mit dem Embryomedium in den Schlauch gezogen werden kann, gefolgt von dem Verschließen des Bodens des Schlauches mit einem Agarose-Stopfen14. Da bei dieser Methode das Röhrchen mit Embryomedium gefüllt ist, können Registrierungsmarker wie fluoreszierende Kügelchen nicht für die Verschmelzung der verschiedenen Ansichten verwendet werden, und die Registrierung ist daher auf Marker innerhalb des Embryos angewiesen. Im Allgemeinen fungieren Kügelchen als bessere Registrierungsmarker, da das Signal der Marker im Embryo abnimmt, wenn es tiefer in die Probe eindringt, was sowohl auf Beleuchtungs- als auch auf Detektionseinschränkungen eines Mikroskops zurückzuführen ist.
Ein dritter Ansatz, der hier detailliert beschriebenund zuvor 5,13,14,15,16 verwendet wurde, besteht darin, frühe Zebrafischembryonen mit einem intakten Chorion abzubilden und die Röhre mit einem minimalen Prozentsatz an Agarose zu füllen, die Kügelchen als Registrierungsmarker enthält. Da in diesem Szenario ein manueller Eingriff zur Positionierung von Embryonen innerhalb eines Chorions nicht möglich ist, liefert diese Studie Statistiken über die Standardorientierung, in die frühe Zebrafischembryonen fallen, wobei der Schwerpunkt auf 70% der Epibolie- und Knospenstadien liegt. Anschließend wird die optimale Anzahl von Ansichten diskutiert, die für die Abbildung von Embryonen im Frühstadium mit zellulärer Auflösung erforderlich sind, und der Fusionsprozess mit BigStitcher, einem auf den Philippinen basierenden Plugin 10,17,18, detailliert beschrieben. Zusammen zielt dieses Protokoll, das ein 20x/1 NA-Objektiv verwendet, darauf ab, Zebrafischembryologen die Verwendung von Multiview-Lichtblattsystemen für die Bildgebung von Embryonen mit Zellkernen und Membranmarkern von der Gastrulation bis zu den frühen Somitenstadien zu erleichtern.
Protokoll
Die in dieser Studie verwendeten Erhaltungs- und Versuchsverfahren für Zebrafische wurden von der institutionellen Tierethikkommission unter den Aktenzeichen TIFR/IAEC/2023-1 und TIFR/IAEC/2023-5 genehmigt. Embryonen, die durch Kreuzung heterozygoter Fische gewonnen wurden, die Tg(actb2:GFP-Hsa.UTRN)19 exprimieren, wurden im Einzelzellstadium mit H2A-mCherry-mRNA (30 pg) injiziert. Die H2A-mCherry-mRNA wurde mit dem pCS2+ H2A-mCherry-Plasmid (ein Geschenk des Labors von Oates, EPFL) durch in vitro-Transkription synthetisiert. Embryonen, die beide Marker exprimierten, die im Rest des Protokolls als Utr-GFP bzw. H2A-mCherry bezeichnet werden, wurden im 70%-Epibolie- und Knospenstadium abgebildet. Die Einzelheiten zu den in der Studie verwendeten Reagenzien und Geräten sind in der Materialtabelle aufgeführt.
1. Probenvorbereitung für die Multiview-Bildgebung
- Vorbereitung von FEP/PTFE-Schläuchen
- Reinigen Sie FEP/PTFE-Schläuche gemäß den zuvor beschriebenen Verfahren14.
- Schneiden Sie die Röhrchen nach der Reinigung je nach Bedarf in Längen von 2-2,5 cm und lagern Sie sie in 2-ml-Mikrozentrifugenröhrchen mit doppelt destilliertem Wasser. Die Röhrchen können auf diese Weise etwa einen Monat lang gelagert werden.
- Erhitzen Sie die Röhrchen vor der Probenvorbereitung ca. 25 Minuten lang auf 70-75 °C, um die Röhrchen zu begradigen. Vermeiden Sie eine Überfüllung in jedem Mikrozentrifugenröhrchen, um ein Verklumpen zu verhindern und ausreichend Platz für eine effektive Röhrchenbegradigung zu gewährleisten.
HINWEIS: Tragen Sie während der gesamten Handhabung des Röhrchens Handschuhe, da Fingerabdrücke/Staubpartikel auf den Röhrchen die Bildgebung beeinträchtigen können.
- Zubereitung von Agarose
- Bereiten Sie die E3-Puffer 50x Stammlösungen wie folgt vor:
- Stamm 1 - 3,252 g Na2HPO4, 0,285 g KH2PO4, 11,933 g NaCl, 0,477 g KCl in 1 l entionisiertem Wasser
- Stamm 2 - 2,426 g CaCl2, 4,067 g MgSO4 in 1 L deionisiertem Wasser.
- Um 1x E3-Puffer herzustellen, fügen Sie jeweils 20 mL Lösungen aus Stamm 1 und 2 in 960 mL deionisiertem Wasser hinzu.
HINWEIS: Geben Sie kein Methylenblau in den E3-Puffer, der für die Bildgebung verwendet wird, da dies zu Lichtstreuung führt. - Niedrigschmelzende Agarose in 1x E3 durch Erhitzen bei 70-75 °C auf einem Heizblock unter Rühren auflösen, bis die Lösung klar und ohne Klumpen oder Kristalle ist.
- Für die Multiview-Bildgebung fügen Sie der Agaroselösung handelsübliche fluoreszierende Kügelchen (siehe Materialtabelle) hinzu, die eine Bildregistrierung während der Analyse ermöglichen. Beschallen Sie die Kügelchen bei Raumtemperatur mit einer Frequenz von 40 kHz für 20-30 min in einem Wasserbad-Ultraschallgerät, um die Kügelchen zu disaggregieren.
- Geben Sie nach der Beschallung 1 μl Kügelchen zu 10 ml Agaroselösung in ein 15 ml-Röhrchen und wirbeln Sie das Röhrchen gut durch, um eine gleichmäßige Dispersion der Kügelchen zu gewährleisten.
HINWEIS: Das Volumen der Bead-Lösung, die der Agarose zugesetzt werden soll, hängt von der Stammlösung und dem Hersteller ab. Probieren Sie eine Reihe von Volumina aus und wählen Sie eine Konzentration, bei der die Kügelchen gut dispergiert erscheinen, wenn sie im Lichtblattsystem abgebildet werden. Zu viele Kügelchen können sich trotz Beschallung und Wirbelbildung ansammeln, während zu wenige Kügelchen die Bildregistrierung beeinträchtigen können. - Bewahren Sie das Agaroseröhrchen mit den zugesetzten Kügelchen mindestens 30 Minuten lang in einem geschlossenen Wasserbad bei 37 °C auf, bevor Sie die Probe vorbereiten. Dadurch wird sichergestellt, dass die Temperatur der Agarose von 70 °C auf 37 °C gesenkt wird.
- Bereiten Sie die E3-Puffer 50x Stammlösungen wie folgt vor:
- Probenvorbereitung
- Setzen Sie die Fische am Abend vor dem Tag der Bildgebung paarweise mit Trennwänden auf. Entfernen Sie die Trennwände für ca. 15 Minuten und entnehmen Sie die Embryonen mit dem Standardverfahren20.
- Sobald die Embryonen das Stadium erreicht haben, das für die Probenvorbereitung von Interesse ist, gießen Sie die gesamten 10 ml Agarose (mit Kügelchen), die bei 37 °C aufbewahrt werden, auf eine 6 cm große Petrischale.
- Warten Sie etwa 2-3 Minuten, um die Temperatur unter 33 °C zu bringen, bevor Sie 10 bis 15 Embryonen in die Agaroselösung übertragen.
HINWEIS: Dies ist ein wichtiger Schritt, um einen möglichen Hitzeschock für Embryonen zu verhindern, die auf die Agarose übertragen wurden. - Stellen Sie sicher, dass der Agaroselösung während des Embryotransfers so wenig E3-Puffer wie möglich zugesetzt wird. Schwenken Sie die Petrischale so, dass der kleine Puffer, der übertragen worden wäre, gut verteilt wird.
- Tragen Sie für den Rest der Probenvorbereitung Handschuhe.
- Nehmen Sie eine 200-μl-Mikropipette mit einer geeigneten Pipettenspitze und führen Sie ein gereinigtes gerades Röhrchen, das aus dem Mikrozentrifugenröhrchen entnommen wurde, in die Pipettenspitze ein, wie in Abbildung 1A,B gezeigt.
- Aspirieren Sie etwas Agarose in das Röhrchen, gefolgt von 2-3 Embryonen mit der Mikropipette (Abbildung 1C,D). Stellen Sie sicher, dass sich die Embryonen in der Nähe des Bodens der Röhre befinden (Abbildung 1E), was bei der Einrichtung der Bildgebung wichtig wird. Stellen Sie außerdem sicher, dass zwischen den verschiedenen Embryonen etwas Agarose vorhanden ist (Abbildung 1E), damit die Kügelchen in dieser Region als Registrierungsmarker verwendet werden können.
- Lösen Sie das Röhrchen, ohne den Druck von der Pipette zu lösen, von der Pipettenspitze und legen Sie es auf den Deckel der mit E3 gefüllten Petrischale, damit sich die Agarose verfestigen kann (Abbildung 1F).
- Die Schritte 1.3.6 bis 1.3.8 werden wiederholt, bis alle Embryonen in der Petrischale in die Röhrchen überführt sind.
- Sobald die Agarose erstarrt ist (was 5-10 Minuten dauern kann und durch Überprüfen der Agarose in der Petrischale bestätigt werden kann), übertragen Sie die Röhrchen in ein mit E3 gefülltes 2-ml-Mikrozentrifugenröhrchen (Abbildung 1G).
- Lagern Sie die Eileiter mit den Embryonen je nach Bedarf in einem 28 °C oder 33 °C heißen Inkubator, bevor Sie mit der Multiview-Bildgebung fortfahren.
2. Multiview-Bildgebung
HINWEIS: In diesem Schritt wird ein allgemeines Verfahren für die Multiview-Bildgebung von Zebrafischembryonen in den frühen Entwicklungsstadien vorgestellt. Die unten beschriebene Methode kann leicht an jedes Multiview-Lichtblattmikroskopiesystem angepasst werden.
- Lokalisierung des Embryos
HINWEIS: Füllen Sie die Probenkammer des Mikroskops mit dem Embryomedium20 und stellen Sie die Temperatur der Probenkammer mindestens 10 Minuten vor der Bildgebung auf die interessierende Temperatur ein.- Der Probenhalter ist wie zuvor beschrieben21 zu montieren, jedoch mit einer Änderung. Da die Bildgebung durch die Röhre erfolgt, führen Sie die Röhre mit den Embryonen direkt in eine Kapillare mit dem richtigen Durchmesser ein, so dass sie an Ort und Stelle bleibt, ohne die Agarose herauszudrücken und die Embryonen am Boden zu stören (Abbildung 1H).
HINWEIS: Da die Probenkammer eine definierte Höhe hat, würde die Sicherstellung, dass sich die Embryonen während der Probenvorbereitung am Boden des Röhrchens befinden (wie in Schritt 1.3.7 erwähnt), eine Positionierung des Embryos im Sichtfeld ermöglichen, ohne den Boden der Kammer zu berühren. - Setzen Sie den Probenhalter in das Multiview-System ein und bringen Sie den Embryo mit den x- und y-Reglern in die Mitte des Sichtfeldes.
- Verwenden Sie dann die z-Steuerung des Probennavigators, um den Embryo in den Fokus zu bringen.
- Zu diesem Zeitpunkt ist die Ausrichtung des Embryos innerhalb des Chorions deutlich sichtbar, und wenn sie nicht zufriedenstellend ist, führen Sie eine neue Röhre ein und wählen Sie den Embryo mit einer interessanten Ausrichtung.
- Der Probenhalter ist wie zuvor beschrieben21 zu montieren, jedoch mit einer Änderung. Da die Bildgebung durch die Röhre erfolgt, führen Sie die Röhre mit den Embryonen direkt in eine Kapillare mit dem richtigen Durchmesser ein, so dass sie an Ort und Stelle bleibt, ohne die Agarose herauszudrücken und die Embryonen am Boden zu stören (Abbildung 1H).
- Ausrichten der Lichtbleche
- Sobald der Embryo in Position ist, richten Sie alle experimentellen Einstellungen ein - Laser, Strahlengang, Filter, Strahlteiler und Kamera.
- Wenn die Software beim Einrichten der Erfassungsparameter eine Option für den Pivot-Scan bereitstellt, wählen Sie diese aus. Ein Pivot-Scan reduziert den Schatteneffekt, der vom Lichtblatt ausgeht, wenn es auf Strukturen oder undurchsichtige Bereiche in der Probe trifft.
- Starten Sie das Live-Scannen der Probe. Stellen Sie die Bitgröße, die Laserleistung und den gewünschten Zoom ein, die die Dicke des Lichtblatts und damit die axiale Auflösung bestimmen.
- Um die Lichtblätter auszurichten, wechseln Sie zunächst auf einseitige Beleuchtung und ändern Sie nacheinander die Einstellungen der beiden Lichtblätter (links und rechts). Zoomen Sie digital in einen Bereich der Probe, in dem klare Strukturen sichtbar sind.
HINWEIS: In dieser Studie wurde entweder ein nukleärer (H2A-mCherry) oder ein Membranmarker (Utr-GFP) verwendet, um die Blätter auszurichten. Es wird empfohlen, die Einstellungen des Lichtblatts in der Software zu ändern, damit der schärfste Signalkontrast im interessierenden Bereich erreicht wird. - Sobald die beiden Leuchtfolien individuell optimiert sind, um das beste Signal zu erzielen, schalten Sie beide Lichtfolien ein. Führen Sie eine zweite Ausrichtungsrunde durch, und stellen Sie sicher, dass das Flackern minimal ist, was auf eine recht gute Ausrichtung der beiden Blätter hinweist.
- Sobald die Ausrichtung abgeschlossen ist, aktivieren Sie die Option zum automatischen Zusammenführen der von den beiden Blättern erzeugten Bilder, sofern die Software dies zulässt. Alternativ kann man die Lichtblätter zunächst grob ausrichten, indem man sich auf Perlen um die Probe herum konzentriert, und dann die Ausrichtung anhand von Informationen aus der Probe feinabstimmen.
HINWEIS: Bei der Bildgebung mit zwei Fluorophoren (z. B. Kern- und Membranmarkern) unterscheidet sich die optimale Ausrichtung für die beiden Strukturen in der Regel geringfügig. Wählen Sie in diesem Szenario eine Einstellung aus, die auf Einschränkungen für die Downstreamverarbeitung basiert. Sollen beispielsweise Zellgrenzen segmentiert werden, was relativ prozessintensiver ist, richten Sie die Lichtschichten anhand des Membranmarkers aus.
- Einrichten von Multiview-Imaging
- Aktivieren Sie nach dem Ausrichten der Lichtfolien die Optionen zum Ausführen eines Z-Stapels sowie einer Multiview-Bildgebung.
- Starten Sie das Live-Scannen und navigieren Sie mit dem Probennavigator, um die erste Ansicht auszuwählen. Richten Sie in dieser Ansicht das Slice-Intervall ein, gefolgt vom ersten und letzten Slice des Z-Stapels. Fügen Sie diese Position im Dialogfeld "Mehrfachansicht" hinzu, in dem die Positions- und Z-Stapel-Informationen für diese Ansicht notiert werden.
- Wechseln Sie zur nächsten Ansicht, indem Sie den Winkel im Proben-Navigator ändern. Richten Sie den Z-Stapel für die zweite Ansicht ein, und fügen Sie die Informationen im Dialogfeld für die Mehrfachansicht hinzu.
- Wiederholen Sie dies für weitere Ansichten.
HINWEIS: Stellen Sie sicher, dass in jeder Ansicht eine bestimmte Mindestanzahl von Segmenten abgebildet wird, damit genügend Überlappung zwischen den verschiedenen Ansichten vorhanden ist, was schließlich bei der Registrierung der Ansichten während der Verarbeitung hilfreich ist. Die minimale Anzahl der Slices hängt sowohl vom Zoomfaktor als auch von der Anzahl der für die Bildgebung gewählten Winkel ab. - Nachdem Sie alle Ansichten eingestellt haben, speichern Sie diese Informationen in einer Textdatei (z. B. mit dem Namen "embryo-positions"), die z-Stapel-Informationen, (x, y, z), die Koordinaten der Röhre sowie die Angabe des Winkels für jede Ansicht enthält. Löschen Sie nach dem Speichern alle Positionen aus dem Multiview-Dialogfeld.
- Da die Bildgebung mit einem intakten Chorion erfolgt, das relativ groß ist, ist es nicht möglich, die Kügelchen in der Röhre mit den gleichen Einstellungen zu betrachten. Daher müssen die Raupeninformationen von einer anderen Position im Rohr aus erfasst werden. Um dies zu tun, übersetzen Sie in eine andere "y"-Koordinate weg vom Embryo, wo die Kügelchen sichtbar sind. Fügen Sie diese Position zum Multiview-Dialogfeld hinzu und speichern Sie diese Position in einer Textdatei (z. B. mit dem Namen "beads-y").
HINWEIS: Bilden Sie die Kügelchen so nah wie möglich an der Embryoprobe ab, um die Auswirkungen möglicher Unterschiede in der Eileiterkrümmung an den beiden Positionen zu minimieren. Wenn mehrere Embryonen in die Röhre eingesetzt werden, ist es daher wichtig, ein wenig Agarose zwischen den Röhren zu lassen, um die Kügelchen abzubilden (wie in Schritt 1.3.7 erwähnt). - Gehen Sie in den Ordner, in dem die Textdateien gespeichert sind, und duplizieren Sie die Datei 'embryo-positions' in eine neue Datei mit dem Namen 'beads-positions'. Ersetzen Sie die y-Koordinate für alle Ansichten in der Datei 'beads-positions' durch die y-Koordinate aus der Datei 'beads-y'. Dadurch wird sichergestellt, dass die Perlen mit der gleichen Anzahl von Ansichten, Z-Stapeln und X-Koordinaten, aber an einer anderen Y-Position in der Röhre abgebildet werden.
- Kehren Sie zur Bildgebungssoftware zurück und laden Sie die Datei "bead-positions" in die Software. Wenn die Option Zeitreihen aktiviert ist, wählen Sie einen Zyklus aus und starten Sie das Experiment. Speichern Sie Raupenbilder als "Perlen", die für die Registrierung der verschiedenen Ansichten während der Bildverarbeitung verwendet werden.
- Nachdem Sie die Perlenbilder aufgenommen haben, löschen Sie die Positionen und laden Sie die anfängliche Datei "embryo-positions" in die Software. Stellen Sie die gewünschte Anzahl von Zyklen mit einem geeigneten Zeitintervall ein und starten Sie das Experiment.
3. Multiview-Bildanalyse
HINWEIS: Für die Verschmelzung der Multiview-Bilder wird ein FIJI-Plugin, BigStitcher, verwendet, die neueste Version des Multiview-Rekonstruktions-Plugins, 10,17,18. Das Plugin kann installiert werden, indem das BigStitcher-Plugin in der Funktion "Update-Sites verwalten" hinzugefügt wird, die über die Option "Update" im Menü "Hilfe" aufgerufen werden kann. Nach der Installation wird das Plugin im Menü "Plugins" angezeigt. Die groben Schritte bei der Fusion sind wie folgt: (1) Definieren Sie .xml/.h5-Dateipaare sowohl für die Kügelchen als auch für die Embryonen; (2) Registrieren Sie alle Ansichten in der Perlendatei; (3) Extraktionspunkt-Spreizfunktion (PSF) für die Kügelchen, die zur Dekonvolution verwendet werden kann (Abbildung 2A); (4) Übertragen Sie die Registrierungs- und PSF-Informationen aus der Kügelchendatei in die Embryodatei und beginnen Sie mit der Multiview-Dekonvolution. Die meisten dieser Schritte wurden bereits ausführlichbeschrieben 21, und hier werden die Schritte beschrieben, die unterschiedlich verarbeitet werden.
- Definieren eines .xml Datensatzes
- Definieren Sie einen neuen Datensatz sowohl für die Kügelchen als auch für den Embryo als "beads.xml" bzw. "embryo.xml", wie zuvor beschrieben21.
- Erkennung und Registrierung anhand von Interessenpunkten
- Nach dem erfolgreichen Export der beads.xml Datei wählen Sie im Dialogfeld "Multiview Explorer" die Ansichten aus, die für die Registrierung erforderlich sind (Abbildung 2B). Klicken Sie mit der rechten Maustaste, und wählen Sie Interessenpunkte erkennen aus. Fahren Sie mit den Schritten wie beschriebenfort 21.
- Nachdem Sie Interessenpunkte erkannt haben, wählen Sie alle Ansichten aus, klicken Sie mit der rechten Maustaste, und wählen Sie Mit Interessenpunkten registrieren aus. Befolgen Sie das Protokoll wie beschrieben21.
- Überprüfen Sie sorgfältig, ob die Perlenregistrierung erfolgreich funktioniert hat. Nach erfolgreicher Registrierung werden überlappende Perlen aus verschiedenen Ansichten überlagert (Abbildung 2C). Um zu überprüfen, wie genau die Registrierung funktioniert hat, wählen Sie zwei aufeinanderfolgende Ansichten aus, wechseln Sie zum überlappenden Bereich und wechseln Sie zwischen den beiden Ansichten, um zu beobachten, ob sich die aus verschiedenen Winkeln abgebildeten Perlen überlagern. Wiederholen Sie diesen Vorgang für jede nachfolgende Ansicht.
- Wenn die Überlappung nicht genau ist, wiederholen Sie die Registrierung, indem Sie die erforderliche Signifikanz für die Registrierung und/oder den akzeptablen Überlappungsfehler (im Dialogfeld "Registrierung" als "RANSAC-Fehler" bezeichnet) lockern.
- Klicken Sie nach erfolgreicher Registrierung im Fenster "Multiview Explorer" auf Speichern , um die aktualisierte .xml Datei zu speichern.
HINWEIS: Speichern Sie auch die Protokolldatei, da sie weitere detaillierte Informationen über die Effizienz der Registrierung enthält. Um die Registrierungsinformationen von den Kügelchen in den Embryo zu übertragen, befolgen Sie die unten beschriebenen Schritte. - Öffnen Sie die bead.xml Datei in einem Texteditor Ihrer Wahl und kopieren Sie den gesamten Block unter "ViewRegistrations".
- Öffnen Sie die embryo.xml und ersetzen Sie den Block "ViewRegistrations" durch den Block, der aus der Beads-Datei kopiert wurde. Wenn mehrere Kanäle vorhanden sind, ersetzen Sie die Registrierungsinformationen für jeden Kanal wie oben. Die Registrierungsinformationen können entweder manuell oder mit dem eigens geschriebenen MATLAB-Code übertragen werden, der hier heruntergeladen werden kann: https://github.com/sundar07/Multiview_analysis
- Öffnen Sie die embryo.xml Datei in "BigStitcher" und prüfen Sie sorgfältig, ob die Registrierung für den Embryo erfolgreich funktioniert hat. Wiederholen Sie den gleichen Vorgang wie für die Perlen, indem Sie die Überlappung der interessierenden Strukturen in jeweils zwei aufeinanderfolgenden Ansichten überprüfen.
HINWEIS: Gelegentlich kann es vorkommen, dass die Registrierung des Embryos nicht so wünschenswert ist, obwohl die Kügelchen perfekt registriert wurden. Dies ist möglich, wenn sich die Krümmung der Röhre von der Position, an der die Kügelchen abgebildet werden, zur Embryonalposition geringfügig ändert. Darüber hinaus kann es zu subtilen Bewegungen des Embryos zwischen den Ansichten kommen, da er frei im Chorion schwebt. In diesem Fall führen Sie eine zweite Registrierungsrunde mit einem Kernmarker im Embryo durch. - Öffnen Sie dazu die embryo.xml Datei in "BigStitcher", klicken Sie mit der rechten Maustaste auf alle Ansichten, die den Kernmarker haben, und wählen Sie Interessenpunkte erkennen.
- Benennen Sie die Interessenpunkte in "Kerne" um und fahren Sie mit den Schritten fort, die für Kügelchen ausgeführt wurden.
- Stellen Sie beim Einstellen der Parameter "Differenz der Gauß" sicher, dass die meisten, wenn nicht sogar alle Kerne nachgewiesen werden und dass kein ektopischer Kernnachweis erfolgt. Klicken Sie anschließend auf Fertig.
- Registrieren Sie anschließend diese Ansichten, indem Sie Mit Zinspunkten registrieren wählen und sich für die Option Präziser Deskriptor basiert (translationsinvariant) entscheiden. Stellen Sie sicher, dass die Option "Alle Ansichten und Interessenpunkte vergleichen" ausgewählt ist, und verwenden Sie "Kerne" als Interessenpunkte. Verwenden Sie die Option zum Fixieren der ersten Ansicht und zum Zuordnen nicht zurück.
- Verwenden Sie für die Registrierung ein affines Modell mit starrer Regularisierung und den Standardparametern im Plugin.
- Überprüfen Sie den Erfolg der Registrierung erneut, indem Sie alle zwei aufeinanderfolgenden Ansichten vergleichen.
- Klicken Sie nach erfolgreicher Registrierung im Fenster "Multiview Explorer" auf Speichern , um die aktualisierte .xml Datei zu speichern.
- Öffnen Sie die embryo.xml Datei in einem Texteditor Ihrer Wahl und kopieren Sie die Registrierungsinformationen von den nuklearen Markern auf die anderen Kanäle.
- Extraktion und Zuweisung von Punktspreizungsfunktionen
- Um eine Multiview-Dekonvolution durchzuführen, extrahieren Sie die PSF des Bildgebungssystems aus dem registrierten Kügelchendatensatz und wenden Sie sie auf die Embryodatei an.
- Um diese Informationen zu erhalten, wählen Sie alle Ansichten in der Bead-Datei aus, klicken Sie mit der rechten Maustaste, und wählen Sie die Optionen Punktverteilungsfunktionen und Extraktion aus.
- Stellen Sie im angezeigten Dialogfeld sicher, dass die Optionen "Entsprechende Interessenpunkte verwenden " und "Min. Intensitätsprojektionen aus PSF entfernen " aktiviert sind, fahren Sie mit den Standard-PSF-Größen fort und klicken Sie auf OK.
HINWEIS: Wenn die PSF-Extraktion für alle Ansichten erfolgreich ist, wird in der Protokolldatei "Extrahierte n/n PSFs" angezeigt. - Speichern Sie anschließend die .xml Datei erneut. Ein aktiviertes Kontrollkästchen in der PSF-Spalte des Dialogfelds "Multiview Explorer" wird angezeigt, und ein "psf"-Ordner wird in dem entsprechenden Ordner mit allen extrahierten PSFs generiert.
- Öffnen Sie die embryo.xml Datei und weisen Sie die PSF für jede Ansicht separat zu. Klicken Sie mit der rechten Maustaste auf "eine Ansicht" → klicken Sie auf Punktspreizungsfunktionen → wählen Sie Zuweisen → wählen Sie Erweitert, gefolgt von Neue PSF allen ausgewählten Ansichten zuweisen. Klicken Sie auf Durchsuchen, gehen Sie zum .xml Dateipfad und öffnen Sie den PSF-Ordner .
- Wählen Sie die passende PSF mit der entsprechenden ID in der ausgewählten Ansicht aus und klicken Sie auf OK, woraufhin das PSF-Kontrollkästchen im Fenster "Multiview Explorer" aktiviert erscheint.
- Wiederholen Sie den Vorgang für alle anderen Ansichten.
- Multiview-Fusion und -Dekonvolution
- Nachdem die Punktverteilungsfunktion für alle Ansichten zugewiesen wurde, klicken Sie mit der rechten Maustaste, und wählen Sie Multiview-Entfaltung.
- Wählen Sie den Begrenzungsrahmen als aktuell ausgewählte Ansichten aus. Für diese Arbeit funktionieren die standardmäßige OSEM-Beschleunigung und die Anzahl der Iterationen gut.
- Führen Sie bei Bedarf ein Downsampling der Bilder durch, wenn eine schnellere Berechnung gewünscht wird oder wenn der CPU-Speicher begrenzt ist.
HINWEIS: Wenn der erforderliche Arbeitsspeicher den vorhandenen Speicher übersteigt, wird am unteren Rand des Fensters eine Warnmeldung in roter Farbe angezeigt. Starten Sie die Dekonvolution nicht, wenn diese Warnung erscheint, da das Plugin irgendwann während der Verarbeitung ins Stocken gerät und nicht mehr reagiert. - Um den Fortschritt der Dekonvolution zu beurteilen, überprüfen Sie die Protokolldatei, in der alle 5 Iterationen Ergebnisse angezeigt werden.
- Um die Rechengeschwindigkeit zu erhöhen, führen Sie eine Multiview-Dekonvolution in der GPU durch, falls zuvor installiert.
HINWEIS: Am Ende dieses Vorgangs öffnet sich ein verschmolzenes Bildfenster, das als TIFF-Datei gespeichert werden kann.
Ergebnisse
Die präzise Ausrichtung der Probe ist ein wesentlicher Bestandteil der effizienten Nutzung eines Mikroskopie-Setups. Bei der Verwendung eines Multiview-Lichtplattensystems ist das manuelle Ausrichten der Proben jedoch oft nicht möglich, da die Proben in einem Röhrchen vorbereitet werden müssen. Um zu überprüfen, ob es stereotype Positionen gibt, die Embryonen innerhalb des Chorions einnehmen, wurden Zebrafischembryonen mit 70% Epibolie (etwa 7 h nach der Befruchtung (hpf)) abgebildet, da die Zeitrafferbildgebung von der Gastrulation bis zu den frühen Somitenstadien im Mittelpunkt dieser Studie stand. Als die Proben unmittelbar vor der Bildgebung bei 70% Epibolie vorbereitet wurden, zeigten die Embryonen keine spezifische Orientierung, die häufig bei Proben beobachtet wird. Da dies oft nicht erwünscht ist, wurden die Proben lange vor der Gastrulation vorbereitet und in Mikrozentrifugenröhrchen bei der entsprechenden Temperatur bis zum Beginn der Bildgebung gelagert. Unter diesen Bedingungen konnten die Embryoorientierungen bei 70 % Epibolie (N = 3; n = 87 Embryonen) eingeteilt werden in (1) Horizontal, wenn die tierisch-pflanzliche (AV) Achse des Embryos orthogonal zur Längsachse des Polymerschlauchs war, (2) Vertikal, wenn die AV-Achse parallel zur Längsachse des Polymerschlauchs war, und (3) Schräg, wenn sich die AV-Achse in einem spitzen Winkel befand (Abbildung 3A). Die horizontale Position war am wenigsten vertreten, während die vertikale und die schräge Position gleichermaßen beobachtet wurden (Abbildung 4).
Wenn die Embryonen in den Eileitern belassen wurden, waren die Embryonen in diesen jeweiligen Positionen bis zu 90% Epibolie stabil, danach änderten die meisten Embryonen ihre Ausrichtung. Daher war eine zweite Runde der Dokumentation der Orientierungen im Knospenstadium der Embryogenese (ca. 10 hpf) erforderlich, um die veränderten Orientierungen zu berücksichtigen. Dies wurde an unabhängig voneinander präparierten Proben durchgeführt. Für die Bildgebung früher Somitenstadien wurde zuvor berichtet, dass ein Embryo mit seinem Notochord orthogonal zur Längsachse der Polymerröhre die ideale Orientierung darstellt, da er die Visualisierung mehrerer bilateraler Somiten ermöglicht, die sich entlang der Achse15 bilden. Bei der Probenvorbereitung vor der Gastrulation (N = 3, n = 93 Embryonen) wiesen etwa 25 % der Embryonen diese Ausrichtung auf (Abbildung 4) und diese Embryonen blieben in dieser Ausrichtung mindestens bis zum 8-Somiten-Stadium stabil, was mit früheren Berichtenübereinstimmt 15. Die übrigen Embryonen zeigten im Knospenstadium verschiedene andere Orientierungen (klassifiziert in Abbildung 3B und Abbildung 4); Viele von ihnen orientierten sich jedoch während der frühen Somitenbildung in die horizontale Position um. Interessanterweise zeigte ein ähnlicher Prozentsatz der Embryonen im Knospenstadium eine horizontale Ausrichtung, unabhängig davon, ob die Probe vor der Gastrulation, bei 70% Epibolie oder unmittelbar vor dem Knospenstadium vorbereitet wurde. Daher scheint der Zeitpunkt der Probenvorbereitung für die horizontale Ausrichtung, die für die Bildgebung von Somitenstadien von Interesse ist, weniger kritisch zu sein, im Gegensatz zu dem, was bei der Bildgebung bei 70% Epibolie beobachtet wurde.
Der Vorteil eines Multiview-Systems ist die Möglichkeit, dieselbe Probe aus mehreren Blickwinkeln zu betrachten. Bei Zebrafischembryonen in den genannten Stadien ist jedoch nicht klar, wie viele Aufrufe erforderlich sind, um eine zelluläre Auflösung über den gesamten Embryo zu erhalten. Um dies zu charakterisieren, wurden Zebrafischembryonen mit einem 20x/1 NA-Objektiv im Detektionsarm mit einem Zoomfaktor von 1 abgebildet, was einer Lichtschichtdicke von 4,57 μm entsprach. Unter diesen Einstellungen deckte der Embryo das gesamte Sichtfeld einer sCMOS-Kamera mit einer Pixelgröße von 6,5 μm und einer Fläche von 1920 x 1920 Pixeln ab. Die Mikroinjektion von mRNA für ein histonmarkiertes Fluorophor (H2A-mCherry) in Embryonen im 1-Zell-Stadium, die aus einer transgenen Linie mit markierten Aktinfilamenten (Utr-GFP) gewonnen wurden, ermöglichte die Visualisierung sowohl der Zellkerne als auch der Zellmembranen im Embryo. Um die Multiview-Fusion von Embryonen mit unterschiedlichen Winkelintervallen durchzuführen, wurde eine 360°-Aufnahme der doppelt transgenen Embryonen sowie der Kügelchen in der Röhre in allen 30° (n = 3 Embryonen bei 70% Epibolie; n = 3 Embryonen im Knospenstadium) oder 45° (n = 3 Embryonen bei 70% Epibolie; n = 3 Embryonen im Knospenstadium) in Abständen von etwa 100 Scheiben in jedem Winkel und einem Schnittintervall von 2 μm durchgeführt. Durch das Überspringen alternativer Winkel während der Bildverarbeitung lieferten die 30°- und 45°-Aufnahmen zusätzlich die 60°- und 90°-Datensätze.
Die aufgenommenen Bilder wurden dann anhand der Perleninformationen registriert, und die Registrierungsdetails wurden in den Embryodatensatz übertragen, wie im Protokollabschnitt beschrieben. Die erfolgreiche Registrierung mit dem BigStitcher-Plugin wurde für die verschiedenen Erfassungen mit Ausnahme des 90°-Datensatzes erreicht, was wahrscheinlich auf eine geringere Abdeckung der Probe aus jedem Winkel zurückzuführen ist. Um dies zu überwinden, wurden die Embryonen alle 90° mit etwa 400 Scheiben in jedem Winkel und einem Schnittabstand von 2 μm sowohl im Epibolie- als auch im Knospenstadium abgebildet, was erfolgreich registriert wurde (n = 3 Embryonen pro Stadium).
Der nächste Schritt bestand darin, eine Multiview-Fusion und Dekonvolution der registrierten Datensätze durchzuführen. Dies geschah mit 4-fachem Downsampling, um die Berechnung zu beschleunigen. Wie bei der Darstellung von Zellkernen aus Einzelansichten und einem rekonstruierten Embryo mit mehreren Ansichten zu sehen ist (Abbildung 5), decken die Einzelansichten ein kleineres Sichtfeld ab, das bei der Fusion ein Bild des gesamten Embryos ergab. Für die Darstellung wurden Kerne mit Mastodon (https://github.com/mastodon-sc/mastodon) detektiert, einem auf Fidschi basierenden Plugin, das in Fidschi im Abschnitt "Manage Update Sites" des Menüs "Hilfe" hinzugefügt und nach dem Hinzufügen unter dem Plugins-Menü aufgerufen werden kann. Für die Detektion wurden zunächst die entsprechenden Bilder in das XML/hdf5-Format konvertiert und dann die nukleare Detektion mit dem 'Detection'-Plugin von Mastodon mit einem DoG-Detektor (Durchmesser 6 μm und Qualitätsschwelle 80) durchgeführt.
Unter den verschiedenen fusionierten Bildern zeigte der 90°-Datensatz einen sehr hohen Hintergrund tiefer in der Probe, was ihn für eine Quantifizierung ungeeignet machte. Im Gegensatz zu kleineren Proben wie Drosophila-Embryonen, die in anderen Studien häufig mit einem 90°-Intervall abgebildet wurden 9,22, wird dies daher nicht für die Bildgebung früher Zebrafischembryonen mit einem 20x/1 NA-Objektiv empfohlen. Zwischen den 30°-, 45°- und 60°-fusionierten Datensätzen gab es qualitativ keinen wesentlichen Unterschied in der Kerninformation (Abbildung 6B, obere Reihe); feinere Strukturen wie Zellgrenzen schienen jedoch mit dem 30°-fusionierten Datensatz im Vergleich zum Rest viel besser aufgelöst zu sein (Abbildung 6C, obere Reihe).
Um diese Beobachtung zu bestätigen, wurde Mastodon verwendet, um Kerne in den fusionierten Bildern zu erkennen, die aus der Bildgebung alle 30°, 45° und 60° erhalten wurden. Für die Analyse wurden drei Regionen in den fusionierten Bildern ausgewählt, jeweils eine in einer Tiefe von 20 μm (Abbildung 6A), 50 μm und 100 μm von der Oberfläche des Embryos. Um die Effizienz der Kerndetektion über Bilder hinweg zu vergleichen, wurde die Detektion wie oben beschrieben durchgeführt, mit identischen Parametern über fusionierte Bilder aus verschiedenen Winkelintervallen. In allen analysierten Regionen wurde jeder Kern unabhängig von fusionierten Bildern detektiert, die aus der Bildgebung alle 30°, 45° oder 60° gewonnen wurden (Abbildung 6B, untere Reihe). Somit können kugelförmige Strukturen wie Kerne in jedem der oben genannten Winkelintervalle ohne Informationsverlust abgebildet werden.
Zur Analyse von Zellgrenzen wurde der Tissue Analyzer23 verwendet, ein FIJI-Plugin, das routinemäßig zur Segmentierung der Zellen24, 25 und 26 verwendet wird. Ähnlich wie Mastodon kann das Tissue Analyzer-Plugin im Abschnitt "Manage Update Sites" des Menüs "Hilfe" in FIJI hinzugefügt und nach dem Hinzufügen unter dem Plugins-Menü aufgerufen werden. Die Zellgrenzen wurden unter Verwendung des Watershed-Algorithmus mit Standardparametern und einer starken Unschärfe von 1,5 bis 2 je nach Gewebetiefe und einer schwachen Unschärfe von 1 segmentiert. Diese Parameter wurden über alle Analysen hinweg konstant gehalten, was einen einfachen Vergleich ermöglicht. Beim manuellen Vergleich der segmentierten Bilder mit den ursprünglichen Eingabebildern wurden Fehler beobachtet, bei denen die Software entweder eine Zellgrenze nicht erkannte oder nicht vorhandene Zellgrenzen zeichnete (Abbildung 6C, untere Reihe). Die Anzahl der Fehler, die vom Gewebeanalyse-Plugin gemacht wurden, wurde auf die Gesamtzahl der in der Region erkannten Bindungen normalisiert und als "Grenzsegmentierungsfehler" berechnet. Während diese Fehler in allen analysierten Bereichen vorhanden waren, stieg die Anzahl der Fehler in fusionierten Bildern, die aus der Bildgebung in Winkelintervallen von 45° und 60° gewonnen wurden, im Vergleich zu 30° drastisch an (Abbildung 6D). Dies deutete darauf hin, dass die Auflösung in den fusionierten Bildern mit zunehmendem Winkelintervall zunehmend schlechter wurde. Für die Segmentierung feinerer Strukturen wie Zellgrenzen ermöglicht ein engeres Winkelintervall eine einfachere Weiterverarbeitung.
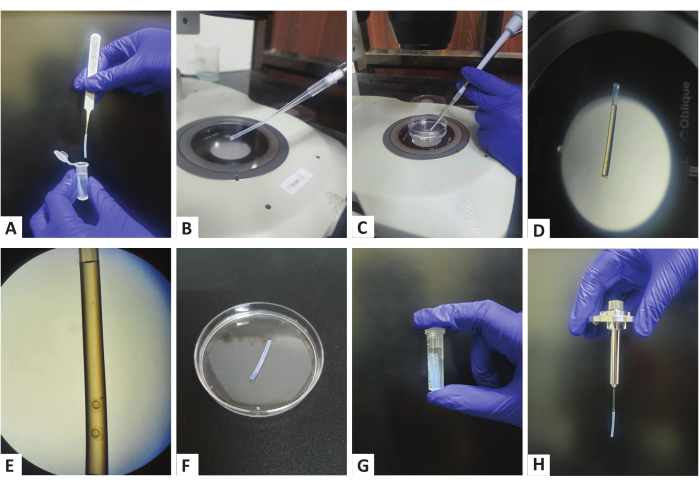
Abbildung 1: Probenvorbereitung mit Polymerröhrchen. (A) Ein Polymerröhrchen aus den gelagerten Mikrozentrifugenröhrchen wird mit einer Pinzette entnommen. (B) Befestigen Sie das Röhrchen an der Spitze einer 200-μl-Mikropipette. (C) Aspirieren von Embryonen mit Hilfe einer Pipette in das Röhrchen. (D,E) Ein Polymerröhrchen mit Embryonen im Knospenstadium, die am Boden des Röhrchens sichtbar sind. (F) Die Polymerröhrchen werden mit E3 auf eine Petrischale gelegt, um die Agarose zu verfestigen. (G) Lagerung der Polymerröhrchen in einem mit E3 gefüllten Mikrozentrifugenröhrchen. (H) Zusammengebauter Probenhalter mit dem montierten Polymerröhrchen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Workflow für die Multiview-Bildanalyse und Registrierung mithilfe von Perlen. (A) Die Multiview-Bildanalyse-Pipeline. (B) Das Bild zeigt das "Multiview Explorer"-Fenster in BigStitcher, einem FIJI-Plugin, in dem jede Ansicht als einzelne Zeile angezeigt wird und Informationen über den Winkel, den Kanal, die Registrierung, die Interessenpunkte und die PSF enthält. Alle im Protokoll besprochenen Befehle werden angezeigt, wenn eine Ansicht ausgewählt und mit der rechten Maustaste geklickt wird, wie im Popup-Menü dargestellt. Die ausgewählten Ansichten können im BigDataViewer-Fenster visualisiert werden, wie gezeigt. (C) Ein repräsentatives Bead-Bild vor (links) und nach der Registrierung (rechts), das aus der Bildgebung in 30°-Winkelabständen gewonnen wurde. Alle Winkelansichten wurden ausgewählt und angezeigt. Maßstabsbalken: 75 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Überblick über die Standard-Embryoorientierungen. Repräsentative Bilder der Orientierungen, in die die Embryonen innerhalb des Chorions bei 70% Epibolie (A) und Knospenstadien (B) fallen. Die obere Reihe in jedem Panel zeigt Hellfeldbilder, die aus dem Lichtblattsystem gewonnen wurden, und die untere Reihe zeigt repräsentative Cartoons. Die Pfeile in (B) zeigen die Position des Notochords an, der zur Identifizierung der Ausrichtung verwendet wurde. Ap, Tierstange; Vp, pflanzlicher Pol; A, anterior; P, posterior. Maßstabsbalken: 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Statistik der verschiedenen Embryonenorientierungen. (A) Die gestapelte Spalte zeigt den prozentualen Anteil der Embryonen an, die bei der Probenvorbereitung vor der Gastrulation mit 70 % Epiboly in die angegebenen Orientierungen fallen. (B) Die gestapelten Säulen zeigen den prozentualen Anteil der Embryonen, die in den Knospenstadien bei der Probenvorbereitung vor der Gastrulation (links), bei 70 % Epibolie (Mitte) und im Knospenstadium (rechts) in die angegebenen Orientierungen fallen. N, die Anzahl der unabhängigen Gelege, aus denen Embryonen gewonnen wurden; n, die Gesamtzahl der Embryonen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Darstellung der Zellkerne eines rekonstruierten Embryos mit mehreren Ansichten. (ein)Das 3D-Streudiagramm stellt Zellkerne dar, die in einem mit Multiview rekonstruierten Embryo nachgewiesen wurden, der im Winkelintervall von 30̛° abgebildet wurde. Jeder Kreis stellt einen Kern dar, und der Schwerpunkt der Kernpositionen wird dargestellt. Die nuklearen Koordinaten wurden mit Mastodon, einem FIJI-Plugin, ermittelt. (B) 3D-Streudiagramme von Zellkernen aus drei repräsentativen Ansichten desselben Embryos - die Darstellungen sind 60° voneinander entfernt. Die Farben für jeden Zellkern wurden nach dem Zufallsprinzip zugewiesen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Vergleich von Informationen auf zellulärer Skala in fusionierten Bildern. (A) Das Bild links zeigt einen Schnappschuss eines mit Multiview rekonstruierten Embryos in einer Tiefe von 20 μm über der Oberfläche. Der gelbe Kreis zeigt die Region, die für die weitere Analyse verwendet wird. Maßstabsleisten: 50 μm (in allen Panels). (B) Repräsentative Rohbilder von Zellkernen in einer Tiefe von 20 μm von der Oberfläche des Embryos aus drei Datensätzen, die alle 30°, 45° bzw. 60° abgebildet wurden (oben). Zellkerne, die in den gleichen Bildern von Mastodon, einem FIJI-Plugin (unten), entdeckt wurden (in grün). (C) Repräsentative Rohbilder in einer Tiefe von 20 μm von der Oberfläche des Embryos aus drei Datensätzen, die alle 30°, 45° bzw. 60° unter Verwendung des Aktinmarkers Utr-GFP (oben) abgebildet wurden. Grenzen, die von Tissue Analyzer, einem FIJI-Plugin, segmentiert wurden, werden für dieselben Bilder angezeigt (unten). Pfeile zeigen Fehler an, die der Tissue Analyzer bei der Grenzsegmentierung gemacht hat, wobei der gelbe Pfeil eine fehlende Begrenzung und der weiße Pfeil falsch erkannte Grenzen darstellt, wenn visuell keine Grenzen zu existieren scheinen. (D) Der Boxplot zeigt den prozentualen Prozentsatz der Fehler, die der Gewebeanalysator zwischen rekonstruierten Embryonen gemacht hat, die alle 30°, 45° und 60° in verschiedenen Tiefen abgebildet wurden. Fehlerbalken zeigen das 1,5-fache des Interquartilsabstands an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Positionierung eines Embryos in der richtigen Ausrichtung, um die interessierende Region abzubilden, ist einer der geschwindigkeitsbegrenzenden Schritte, der oft zu einer fehlgeschlagenen Mikroskopiesitzung für eine Anwenderin führt. Dies gilt umso mehr für ein Multiview-Lichtblattmikroskop, bei dem die manuelle Manipulation der Ausrichtung schwierig ist, da die Proben in eine Röhre eingebettet sind. Um diesen Prozess zu unterstützen, berichtet diese Studie über die Statistik verschiedener Positionen, die ein Zebrafischembryo zwischen 70% epibolen und frühen Somitenstadien innerhalb eines Chorions einnimmt, wenn das Polymerröhrchen mit den Embryonen in einem Mikrozentrifugenröhrchen aufrecht gehalten wird.
Bei 70% Epibolie liefern vertikal oder horizontal ausgerichtete Embryonen komplementäre Informationen über das Geschehen im gesamten Embryo. Während die horizontale Ausrichtung die Visualisierung der zellulären Dynamik im tierischen Pol sowie der Dynamik von Zellen, die sich in der Epibolie befinden, aus der Sicht des pflanzlichen Pols ermöglicht, ermöglicht die vertikale Ausrichtung die Visualisierung der dorsalen und ventralen Seite des Embryos, einschließlich der zellulären Dynamik im dorsalen Organizer, der Internalisierung von Zellen während der Gastrulation und der Konvergenz-Extensions-Strömungen. Diese Studie zeigt, dass es eine hohe Wahrscheinlichkeit gibt, einen Embryo in rein vertikaler Ausrichtung zu erhalten, wenn die Proben einige Stunden vor der Bildgebung vorbereitet werden; Allerdings scheint die horizontale Ausrichtung in diesen Phasen viel weniger vorherrschend zu sein. Für Stadien vor 70% Epibolie wird empfohlen, dass Benutzer ähnliche Statistiken der Embryonenorientierung im Chorion dokumentieren, bevor sie mit der Zeitrafferbildgebung fortfahren, da diese Informationen auf lange Sicht Zeit sparen.
Ab dem Knospenstadium ist die beste Orientierung für die Bildgebung ein horizontal positionierter Embryo, da dies die Verfolgung der zellulären Dynamik entlang der gesamten anteroposterioren Körperachse von Embryonen ermöglicht15. Einen Embryo in dieser Orientierung zu bekommen ist relativ einfach, da etwa 25% der Embryonen in diese stabile Position fallen, unabhängig davon, wann die Probe vorbereitet wird. Im Allgemeinen wird für die Bildgebung in jedem interessierenden Stadium die Vorbereitung von etwa 15 Eileitern mit zwei bis drei Embryonen in jedem Eileiter empfohlen, was eine hohe Wahrscheinlichkeit gewährleistet, Embryonen in der gewünschten Ausrichtung zu erhalten, und somit eine erfolgreiche Mikroskopiesitzung an einem bestimmten Tag. Darüber hinaus wird empfohlen, die Eileiter etwa eine Stunde vor dem interessierenden Stadium zu sortieren, um einen Embryo mit der richtigen Ausrichtung für die Zeitrafferbildgebung auszuwählen.
Nach der Gewinnung eines Embryos in der richtigen Ausrichtung ist es wichtig, die Anzahl der Ansichten und den Winkelabstand zwischen den Ansichten für die Bildgebung zu berücksichtigen. Dies hängt von der minimal erforderlichen räumlichen und zeitlichen Auflösung ab. Je mehr Winkel eine bestimmte Struktur oder Region des Embryos abgebildet wird, desto besser ist die räumliche Auflösung (wie in Abbildung 6 gezeigt), was jedoch zu einem Kompromiss bei der zeitlichen Auflösung führt. Bei diesem Protokoll dauerte die Abbildung von zwei Kanälen über volle 360° mit einem Winkelintervall von 30° und etwa 100 Z-Schichten in jedem Winkel mit einem Schichtabstand von 2 μm etwa 3 Minuten für einen einmaligen Punkt. Dies ist zwar ausreichend, um Zellen in frühen Embryonalstadien zu verfolgen, aber wenn Ereignisse mit schnellerer Dynamik erfasst werden sollen, muss eine geringere Anzahl von Winkeln erfasst werden, um eine bessere zeitliche Auflösung zu gewährleisten, aber mit einem Kompromiss bei der räumlichen Auflösung, insbesondere wenn feinere Strukturen wie Zellgrenzen segmentiert werden müssen. Eine zweite Option, die zur Verbesserung der zeitlichen Auflösung in Betracht gezogen werden sollte, ist die Verwendung eines Multiview-Systems mit zwei Detektionsarmen, das die Erfassungsgeschwindigkeit erheblich erhöht, wie sie kürzlich zur Verfolgung von Zellbewegungen während der Gastrulation in Zebrafischembryonen verwendet wurde16. Obwohl in diesem Protokoll nur das 20x/1 NA-Objektiv getestet wurde, muss je nach abzubildender Probe, der erforderlichen räumlichen Auflösung und dem Sichtfeld das richtige Objektiv berücksichtigt werden. Basierend auf der erforderlichen Downstream-Verarbeitung und Quantifizierung für eine bestimmte Probe und ein bestimmtes Sichtfeld müssen die Anzahl der Ansichten und das Winkelintervall sorgfältig ausgewählt werden. Für die Abbildung ganzer früher Zebrafischembryonen, insbesondere mit zellulärer Auflösung aus mehreren Winkeln, scheint jedoch ein 20x/1 NA-Objektiv optimal zu sein, da ein Objektiv mit geringerer Vergrößerung und NA (z. B. 10x/0,5 NA-Objektiv) eine viel geringere Auflösung aufweist, was wahrscheinlich zu mehr Segmentierungsfehlern führt, während ein Objektiv mit höherer Vergrößerung und NA nicht geeignet ist, den gesamten Embryo im Sichtfeld der Kamera abzudecken.
In dieser Studie wurden zur Diskussion von Mounting- und Bildgebungsstrategien ein Aktinmarker, der als Proxy für die Verfolgung von Zellgrenzen verwendet werden kann, und ein Histonmarker zum Nachweis und zur Verfolgung von Zellkernen verwendet. Tatsächlich haben die meisten der zuvor veröffentlichten Studien, die ein Multiview-Lichtblattsystem verwendet haben, bevorzugt ähnliche Marker 5,7,15,16,27 verwendet. Der Hauptgrund für die Wahl dieser Marker ist, dass die Multiview-Lichtblattmikroskopie hauptsächlich zur Verfolgung der Dynamik auf zellulärer Skala (z. B. Zellform und Zellumlagerung) entweder im Gewebemaßstab oder über den gesamten Embryo über lange Zeiträume von mehreren Stunden bis Tagen verwendet wird. Auf der anderen Seite, wenn es notwendig ist, eine subzelluläre Auflösung zu erreichen, könnte eine andere transgene Linie verwendet werden, die den interessierenden Bereich markiert, aber in diesem Szenario ist ein Mehrsicht-Lichtblattmikroskop möglicherweise nicht die beste Wahl des Instruments, sondern eher ein konfokales, ein superauflösendes System oder sogar ein Gitterlichtblattmikroskop28. 29 könnte besser zu dem zu untersuchenden Problem passen.
Das besprochene Protokoll funktioniert gut von der Gastrulation bis zu Zebrafischembryonen im 15-Somitenstadium30 (ca. 17 hpf) ohne jegliche Modifikationen, und obwohl es hier nicht getestet wurde, funktioniert es wahrscheinlich auch bei früheren Embryonen vor dem Gastrulationsstadium. Jenseits des 15-Somiten-Stadiums beginnen spontane Muskelkontraktionen30,31, die durch Zugabe von Tricain, einem Anästhetikum, sowohl in der Sonde als auch in der Probenkammer unterdrückt werden können. Darüber hinaus beginnt bei etwa 18 hpf die Schwanzeversion weg vom Eigelb30, wodurch der Embryo aus dem Sichtfeld verschoben wird. Um dies zu gewährleisten, muss ein Tracking-Algorithmus eingesetzt werden, der das verlängerte Ende verfolgt, um den Embryo im Fokus zu halten32. Darüber hinaus muss für die Bildgebung von Embryonen in späteren Stadien, wenn sie frei von einem Chorion sind, wie z. B. zur Verfolgung der neuronalen Entwicklung, eine alternative Aufstiegsstrategie angewendet werden, wie sie kürzlich durchgeführt wurde33.
Für die Bildgebung von Zebrafischembryonen mit intaktem Chorion ist es mit einem 20x/1 NA-Objektiv nicht möglich, sowohl die Kügelchen, die als Registrierungsmarker dienen, als auch den Embryo im selben Sichtfeld zu erfassen. Diese Studie bietet eine einfache Alternative, indem die verschiedenen Ansichten mit Informationen von Kügelchen registriert werden, die über oder unter der Probe vorhanden sind, gefolgt von der Verschiebung der Registrierung von den Kügelchen auf den Embryo während der Verarbeitung. Sobald die Embryonenprobe auf diese Weise registriert ist, kann eine zweite Registrierungsrunde mit Zellkernen als Registrierungsmarker durchgeführt werden, die in der Probe vorhanden sind, um die Erstregistrierung weiter zu verfeinern. Bei der Durchführung von Zeitraffer-Bildgebung können die Kerninformationen wiederum für die Registrierung aufeinanderfolgender Zeitpunkte verwendet werden, wie zuvor berichtet wurde21. Eine Alternative zu diesem Protokoll besteht darin, auf Kügelchen zu verzichten und stattdessen die Zellkerninformationen für die Erstregistrierung zu verwenden. Aber die Registrierung schlug oft fehl (hier nicht gezeigt) im BigStitcher-Plugin, möglicherweise aufgrund einer Kombination von vielen Kernen, die während der Verarbeitung tiefer im Embryo nicht entdeckt wurden, und auch aufgrund einer relativ geringeren Anzahl von Kernen, die im Embryo vorhanden sind, wenn sie aus bestimmten Blickwinkeln betrachtet werden (z. B. wenn sie ventral in späten Epibolienstadien betrachtet werden).
Da bei diesem Protokoll Beadsinformationen von oberhalb oder unterhalb der Probe für die Registrierung verwendet werden und es nicht erforderlich ist, die Kügelchen in der gleichen Ansicht wie die Probe abzubilden, kann man dieses Protokoll nur für die Bildgebung bestimmter interessierender Regionen verwenden, jedoch mit hoher Vergrößerung aus verschiedenen Winkeln anstelle des gesamten Embryos. Darüber hinaus ist es einfach, dieses Protokoll für die Abbildung mehrerer vertikal im Röhrchen gestapelter Proben anzupassen, indem Bead-Informationen zwischen den Proben für die Registrierung verwendet werden.
Offenlegungen
Die Autoren erklären, dass keine konkurrierenden Interessen bestehen.
Danksagungen
Wir danken Dr. Kalidas Kohale und seinem Team für die Instandhaltung der Fischanlage und KV Boby für die Wartung des Lichtblattmikroskops. SRN dankt der finanziellen Unterstützung durch das Department of Atomic Energy (DAE) der indischen Regierung (Projektidentifikationsnummer RTI4003, DAE OM Nr. 1303/2/2019/R\&D-II/DAE/2079 vom 11.02.2020), das Programm der Partnergruppe der Max-Planck-Gesellschaft (M.PG. A MOZG0010) und dem Science and Engineering Research Board Start-up Research Grant (SRG/2023/001716).
Materialien
| Name | Company | Catalog Number | Comments |
| Agarose, low gelling temperature | Sigma-Aldrich | A9414 | |
| Calcium Chloride dihydrate | Sigma-Aldrich | 12022 | |
| FIJI | Version: ImageJ 1.54f | ||
| Latex beads, carboxylate-modified polystyrene, fluorescent red, 0.5 μm mean particle size, aqueous suspension | Sigma-Aldrich | L3280 | |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | |
| mMESSAGE mMACHINE SP6 Transcription kit | ThermoFischer Scientific | AM1340 | For in vitro transccription of H2A-mCherry plasmid |
| Potassium Chhloride | Sigma-Aldrich | P9541 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P0662 | |
| PTFE Sleeving AWG 15L - 1.58 mm ID x 0.15 mm Wall +/-0.05 | Adtech Innovations in Fluoroplastics | STW15 | PTFE tubes |
| Sodium Chloride | Sigma-Aldrich | S3014 | |
| Sodium phosphate dibasic | Sigma-Aldrich | 71640 | |
| Ultrasonic Cleaner | Labman | LMUC3 | Ultrasonicator |
| Zeiss LightSheet 7 System | Zeiss |
Referenzen
- Wan, Y., McDole, K., Keller, P. J. Light sheet microscopy and its potential for understanding developmental processes. Annu Rev Cell Dev Biol. 35, 655-681 (2019).
- Keller, P. J., Schmidt, A. D., Wittbrodt, J., Stelzer, E. H. Reconstruction of zebrafish early embryonic development by scanned light sheet microscopy. Science. 322 (5904), 1065-1069 (2008).
- Keller, P. J., et al. high-contrast imaging of animal development with scanned light sheet-based structured-illumination microscopy. Nat Methods. 7 (8), 637-642 (2010).
- Keller, P. J. Imaging morphogenesis: Technological advances and biological insights. Science. 340 (6137), 1234168(2013).
- Schmid, B., et al. High-speed panoramic Light sheet microscopy reveals global endodermal cell dynamics. Nat Commun. 4 (1), 2207(2013).
- Strnad, P., et al. Inverted Light sheet microscope for imaging mouse pre-implantation development. Nat Methods. 13 (2), 139-142 (2016).
- McDole, K., et al. In toto imaging and reconstruction of post-implantation mouse development at the single-cell level. Cell. 175 (3), 859-876 (2018).
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Krzic, U., Gunther, S., Saunders, T. E., Streichan, S. J., Hufnagel, L. Multiview Light sheet microscope for rapid in toto imaging. Nat Methods. 9 (7), 730-733 (2012).
- Preibisch, S., Saalfeld, S., Schindelin, J., Tomancak, P. Software for bead-based registration of selective plane illumination microscopy data. Nat Methods. 7 (6), 418-419 (2010).
- Swoger, J., Verveer, P., Greger, K., Huisken, J., Stelzer, E. H. K. Multiview image fusion improves resolution in three-dimensional microscopy. Opt Express. 15 (13), 8029-8042 (2007).
- Swoger, J., Huisken, J., Stelzer, E. H. Multiple imaging axis microscopy improves resolution for thick-sample applications. Opt Lett. 28 (18), 1654-1656 (2003).
- Kaufmann, A., Mickoleit, M., Weber, M., Huisken, J. Multilayer mounting enables long-term imaging of zebrafish development in a light sheet microscope. Development. 139 (17), 3242-3247 (2012).
- Weber, M., Mickoleit, M., Huisken, J. Multilayer mounting for long-term light sheet microscopy of zebrafish. J Vis Exp. (84), e51119(2014).
- Naganathan, S. R., Popović, M., Oates, A. C. Left-right symmetry of zebrafish embryos requires somite surface tension. Nature. 605 (7910), 516-521 (2022).
- Shah, G., et al. Multi-scale imaging and analysis identify pan-embryo cell dynamics of germlayer formation in zebrafish. Nat Commun. 10 (1), 5753(2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Hörl, D., et al. BigStitcher: Reconstructing high-resolution image datasets of cleared and expanded samples. Nat Methods. 16 (9), 870-874 (2019).
- Behrndt, M., et al. Forces driving epithelial spreading in zebrafish gastrulation. Science. 338 (6104), 257-260 (2012).
- Westerfield, M. The Zebrafish Book: A Guide for the Laboratory Use of Zebrafish (Danio Rerio). , University of Oregon Press. (2000).
- Icha, J., et al. Using light sheet fluorescence microscopy to image zebrafish eye development. J Vis Exp. (110), e53966(2016).
- Tomer, R., Khairy, K., Amat, F., Keller, P. J. Quantitative high-speed imaging of entire developing embryos with simultaneous multiview Light sheet microscopy. Nat Methods. 9 (7), 755-763 (2012).
- Aigouy, B., Prud'homme, B. Segmentation and quantitative analysis of epithelial tissues. Methods Mol Biol. 2540, 387-399 (2022).
- Mancini, L., et al. Apical size and deltaA expression predict adult neural stem cell decisions along lineage progression. Science Adv. 9, 7519(2023).
- Piscitello-Gomez, R., Mahmoud, A., Dye, N., Eaton, S. Sensitivity of the timing of Drosophila pupal wing morphogenesis to external perturbations. bioRxiv. , (2023).
- Tsuboi, A., Fujimoto, K., Kondo, T. Spatiotemporal remodeling of extracellular matrix orients epithelial sheet folding. Science Adv. 9, 2154(2023).
- Fu, Q., Martin, B. L., Matus, D. Q., Gao, L. Imaging multicellular specimens with real-time optimized tiling Light sheet selective plane illumination microscopy. Nat Commun. 7 (1), 11088(2016).
- Chen, B. C., et al. Lattice Light sheet microscopy: imaging molecules to embryos at high spatiotemporal resolution. Science. 346 (6208), 1257998(2014).
- York, H. M., et al. Deterministic early endosomal maturations emerge from a stochastic trigger-and-convert mechanism. Nat Commun. 14 (1), 4652(2023).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Melançon, E., Liu, D. W. C., Westerfield, M., Eisen, J. S. Pathfinding by identified zebrafish motoneurons in the absence of muscle pioneers. J Neurosci. 17 (20), 7796(1997).
- Rohde, L. A., et al. Cell-autonomous timing drives the vertebrate segmentation clock's wave pattern. eLife. , 13:RP93764 (2024).
- Haynes, E. M., et al. KLC4 shapes axon arbors during development and mediates adult behavior. eLife. 11, e74270(2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten