Method Article
初期ゼブラフィッシュ胚のライトシート顕微鏡イメージングとマウント戦略
要約
ライトシート顕微鏡を使用して、無傷の絨毛膜内の初期のゼブラフィッシュ胚をイメージングするためのサンプル調製戦略について説明します。これは、70%エピボリーおよび芽の段階で胚が絨毛膜内で獲得するさまざまな向きを分析し、ライトシートシステムを使用して胚全体で細胞スケールの分解能を得るためのイメージング戦略を詳しく説明します。
要約
ライトシート顕微鏡は、ゼブラフィッシュの胚を最小限の光毒性で長期間にわたってライブイメージングするための最適な方法論となっています。特に、サンプルの回転が可能なマルチビューシステムにより、胚全体をさまざまな角度からイメージングすることができます。しかし、マルチビューシステムを使用するほとんどのイメージングセッションでは、サンプルは通常ポリマーチューブ内で調製されるため、サンプルのマウントは面倒なプロセスです。このプロセスを支援するために、このプロトコルでは、70%エピボリー期と初期体節期の間の初期のゼブラフィッシュの発達をイメージングするための基本的なマウント戦略について説明します。具体的には、この研究では、絨毛膜内の70%エピボリー期と芽期における胚のデフォルト位置に関する統計を提供しています。さらに、発生の初期段階でゼブラフィッシュの胚全体をイメージングするために必要な最適な角度の数と角度の間隔について説明し、異なるビューを融合させることで細胞スケールの情報を抽出できるようにします。最後に、胚はカメラの視野全体をカバーし、これは細胞スケールの解像度を得るために必要であるため、このプロトコルでは、異なるビューの登録のために胚の上または下からのビーズ情報を使用するプロセスを詳述します。
概要
光毒性を最小限に抑えることは、生きた胚を高い時空間分解能で長期間イメージングするための主要な要件です。過去10年間で、ライトシート顕微鏡は、この要件を満たすための最適な方法論になりました1,2,3,4,5,6,7。簡単に言うと、2004年に発生過程を捉えるために初めて使用されたこの技術8では、整列した2枚の薄いレーザーシートが対向する端から胚を通過し、目的の平面のみを照らします。検出対物レンズを直交して配置し、サンプル中のすべての照らされた点から放出された蛍光灯を同時に収集します。次に、静的ライトシートを介して胚を順次移動させることにより、3D画像が得られます。
さらに、この方法論の特定の形態では、マルチビューライトシート顕微鏡法と呼ばれ、サンプルをローターを使用して回転させることができるポリマーチューブに懸濁することができ、複数の角度から同じ胚のイメージングを可能にする9、10、11。イメージング後、複数の角度からの画像は、レジストレーションマーカー(通常は胚内(核など)またはチューブ内(蛍光ビーズなど)の球状蛍光マーカー)に基づいて融合されます。マルチビューイメージングとフュージョンにより、軸方向の解像度が大幅に向上し、3次元すべてで等方性分解能が得られます12。これは大きな利点ですが、マルチビュー法の大きな課題は、イメージングの全時間を通じて胚をチューブ内にマウントし、所定の位置に保持する必要があるサンプルマウントです。
マルチビューイメージングを行うために、胚を所定の位置に保持し、イメージング中の動きを防ぐために、胚をアガロースに埋め込むことができます。しかし、これはしばしば有害な成長と発達につながり、特にここで議論されているモデルシステムである初期段階のゼブラフィッシュ胚13にとっては有害です。第2の取り付け戦略は、胚の直径よりわずかに大きいだけの細いチューブを使用することであり、そこでは、胚を胚培地と共にチューブ内に引き込むことができ、続いて、チューブの底部をアガロースプラグ14で閉じることができる。この方法では、チューブが胚培地で満たされているため、蛍光ビーズなどのレジストレーションマーカーを異なるビューの融合に使用できず、したがってレジストレーションは胚内のマーカーに依存します。一般に、ビーズは、顕微鏡の照明と検出の両方の制限により、胚内のマーカーの信号がサンプルの奥深くに移動すると劣化するため、より優れたレジストレーションマーカーとして機能します。
したがって、ここで詳しく説明し、以前に5,13,14,15,16使用した3番目のアプローチは、初期のゼブラフィッシュの胚を無傷の絨毛膜でイメージングし、レジストレーションマーカーとしてビーズを含む最小限の割合のアガロースをチューブに充填することです。このシナリオでは、絨毛膜内に胚を配置するための手動介入が不可能であるため、この研究では、初期のゼブラフィッシュの胚が陥るデフォルトの向きに関する統計を提供し、特に 70% のエピボリーと芽の段階に焦点を当てています。次に、細胞スケールの解像度で初期段階の胚をイメージングするために必要な最適なビュー数について説明し、FIJIベースのプラグインであるBigStitcherを使用した融合のプロセスを詳しく説明します10,17,18。20x/1 NA対物レンズを使用するこのプロトコルは、ゼブラフィッシュの発生学者が原腸陥入から早期体節期までの核および膜マーカーを持つ胚をイメージングするためのマルチビューライトシートシステムの使用を容易にすることを目的としています。
プロトコル
この研究で使用されたゼブラフィッシュの飼育および実験手順は、TIFR/IAEC/2023-1およびTIFR/IAEC/2023-5を参照し、施設の動物倫理委員会によって承認されました。 Tg(actb2:GFP-Hsa.UTRN)19を発現するヘテロ接合魚類を交配して得られた胚に、H2A-mCherry mRNA(30 pg)を1細胞期に注入した。H2A-mCherry mRNAは、pCS2+ H2A-mCherryプラスミド(オーツ研究所、EPFLからの寄贈品)を用いて in vitro 転写により合成しました。プロトコルの残りの部分で、それぞれUtr-GFPおよびH2A-mCherryと呼ばれる両方のマーカーを発現する胚を、70%エピボリーおよびバドステージでイメージングしました。本試験で使用した試薬および機器の詳細は、 資料表に記載されています。
1. マルチビューイメージング用のサンプル調製
- FEP/PTFEチューブの調製
- 前述の手順に従ってFEP / PTFEチューブを清掃します14.
- 洗浄後、必要に応じてチューブを2〜2.5cmの長さに切断し、二重蒸留水を含む2mLの微量遠心チューブに保管します。チューブは、この方法で約1ヶ月間保存することができます。
- サンプル調製の前に、チューブを70〜75°Cで約25分間加熱して、チューブをまっすぐにします。各微量遠心チューブの過密を避けて凝集を防ぎ、効果的なチューブの矯正のための十分なスペースを確保します。
注意: チューブに残った指紋/ほこりの粒子がイメージングを妨げる可能性があるため、チューブの取り扱い手順全体を通して手袋を着用してください。
- アガロースの調製
- E3バッファー50xストック溶液を次のように調製します。
- ストック 1 - 3.252 g の Na2HPO4、0.285 g の KH2PO4、11.933 g の NaCl、0.477 g の KCl を 1 L の脱イオン水に溶かします。
- ストック2 - 2.426 gのCaCl2、4.067 gのMgSO4、1 Lの脱イオン水。
- 1x E3バッファーを作製するには、ストック1と2の溶液をそれぞれ20 mLずつ960 mLの脱イオン水に加えます。
注:イメージングに使用するE3バッファーにメチレンブルーを添加すると、光の散乱を引き起こすため、添加しないでください。 - 低融点アガロースを1x E3に溶解し、ヒートブロック上で70〜75°Cで加熱し、溶液が塊や結晶なしで透明になるまで攪拌します。
- マルチビューイメージングを行うには、市販の蛍光ビーズ( 材料表を参照)をアガロース溶液に添加すると、解析中の画像レジストレーションが可能になります。ビーズを40kHzの周波数で室温で20〜30分間、水浴超音波装置で超音波処理し、ビーズを解凝集します。
- 超音波処理後、15mLチューブ内の10mLのアガロース溶液に1μLのビーズを加え、チューブをしっかりとボルテックスしてビーズが均一に分散するようにします。
注:アガロースに添加するビーズ溶液の量は、原液とメーカーによって異なります。さまざまな容量を試し、ライトシートシステムでイメージングしたときにビーズが十分に分散しているように見える濃度を選択してください。超音波処理やボルテックスにもかかわらず、ビーズが多すぎると凝集する可能性がありますが、ビーズが少なすぎると画像のレジストレーションに影響を与える可能性があります。 - ビーズを添加したアガロースチューブを、サンプル調製前に少なくとも30分間37°Cに維持した密閉水浴に保管してください。これにより、アガロースの温度が70°Cから37°Cに下がります。
- E3バッファー50xストック溶液を次のように調製します。
- サンプル調製
- イメージングの日の前夜に仕切り付きのペアで魚をセットアップします。仕切りを約15分間取り外し、標準的な手順20を使用して胚を採取します。
- 胚がサンプル調製の対象となる段階に達したら、37°Cに保たれた10 mLのアガロース(ビーズ付き)全体を6 cmのシャーレに注ぎます。
- 約2〜3分待って温度を33°C未満にしてから、10〜15個の胚をアガロース溶液に移します。
注:これは、アガロースに移植された胚への熱ショックを防ぐための重要なステップです。 - 胚移植時にアガロース溶液に添加するE3バッファーをできるだけ少なくしてください。ペトリ皿を渦巻かせて、転送されたであろう小さなバッファーが十分に分散されるようにします。
- サンプル調製手順の残りの部分では手袋を着用してください。
- 適切なピペットチップが付いた200μLのマイクロピペットを取り、 図1A、Bに示すように、マイクロ遠心チューブから取り出した洗浄済みのストレートチューブをピペットチップに挿入します。
- 少量のアガロースをチューブに吸引し、続いてマイクロピペットを使用して2〜3個の胚を吸引します(図1C、D)。胚がチューブの底に近づいていることを確認してください(図1E)これは、イメージングを設定する際に重要になります。また、異なる胚の間に小さなアガロースがあることを確認して(図1E)、この領域のビーズをレジストレーションマーカーとして使用できるようにします。
- ピペットからの圧力を解放せずに、チューブをピペットチップから取り外し、E3で満たされたシャーレの蓋に置いて、アガロースを固めます(図1F)。
- ペトリ皿内のすべての胚がチューブに移されるまで、手順1.3.6から1.3.8を繰り返します。
- アガロースが固まったら(これには5〜10分かかり、ペトリ皿内のアガロースを確認することで確認できます)、チューブをE2で満たされた3mL微量遠心チューブに移します(図1G)。
- マルチビューイメージングに進む前に、必要に応じて、胚の入ったチューブを28°Cまたは33°Cのインキュベーターに保管してください。
2. マルチビューイメージング
注:このステップでは、ゼブラフィッシュの胚の初期発生段階におけるマルチビューイメージングの一般的な手順を示します。以下に詳述する方法は、あらゆるマルチビューライトシート顕微鏡システムに簡単に適応させることができます。
- 胚の位置を特定する
注:顕微鏡のサンプルチャンバーに胚培地20を満たし、イメージングの少なくとも10分前にサンプルチャンバーの温度を目的の温度に設定する。- ただし、サンプルホルダーは、前述した21のように、1つの変更を加えて組み立てます。イメージングはチューブを介して行われるため、胚が入ったチューブを適切な直径の毛細血管に直接挿入し、アガロースを押し出して底部の胚を乱さないように所定の位置に保持します(図1H)。
注:サンプルチャンバーには高さが定義されているため、サンプル調製中に胚がチューブの底に向かって配置されていることを確認すると(ステップ1.3.7で述べたように)、チャンバーの床にぶつかることなく胚を視野に配置することができます。 - サンプルホルダーをマルチビューシステムに挿入し、XコントロールとYコントロールを使用して胚を視野の中心に近づけます。
- 標本ナビゲーターのzコントロールを使用して、胚に焦点を合わせます。
- この時点で、絨毛膜内の胚の向きがはっきりと見えるようになり、満足できない場合は、新しいチューブを挿入して、関心のある向きの胚を選択します。
- ただし、サンプルホルダーは、前述した21のように、1つの変更を加えて組み立てます。イメージングはチューブを介して行われるため、胚が入ったチューブを適切な直径の毛細血管に直接挿入し、アガロースを押し出して底部の胚を乱さないように所定の位置に保持します(図1H)。
- ライトシートの位置合わせ
- 胚が所定の位置に配置されたら、レーザー、光路、フィルター、ビームスプリッター、カメラなど、すべての実験設定を設定します。
- 取得パラメータの設定中に、ソフトウェアに ピボットスキャンのオプションがある場合は、それを選択します。ピボットスキャンは、サンプル内の構造や不透明な領域に遭遇したときにライトシートから現れるシャドウイング効果を軽減します。
- サンプルのライブスキャンを開始します。ビットサイズ、レーザー出力、および目的のズームを設定することで、ライトシートの厚さ、つまり軸方向の解像度が決まります。
- ライトシートを揃えるには、まず 片面照明 に切り替えて、 2枚のライトシート (左右)の設定を順番に変更してください。サンプルの領域にデジタルでズームインし、目的の明確な構造が見えます。
注:この研究では、核マーカー(H2A-mCherry)または膜マーカー(Utr-GFP)のいずれかを使用してシートを整列させました。ソフトウェアでライトシートの設定を変更すると、関心領域で信号のコントラストが最もシャープになります。 - 2つのライトシートが個別に最適化され、最適な信号が得られたら、両方のライトシートをオンにします。2回目のアライメントを行い、ちらつきが最小限に抑えられていること、つまり2枚のシートがかなり良好にアライメントされていることを確認します。
- 位置合わせが完了したら、ソフトウェアで許可されている場合は、2つのシートによって生成された画像を自動的に融合するオプションを有効にします。あるいは、最初にサンプルの周りのビーズに焦点を合わせてライトシートを粗く整列させ、次にサンプルからの情報を使用して整列を微調整することもできます。
注:2つの蛍光色素(核マーカーや膜マーカーなど)でイメージングする場合、通常、最適なアライメントは2つの構造でわずかに異なります。このシナリオでは、ダウンストリーム処理の制約に基づいて設定を選択します。たとえば、細胞の境界をセグメント化する場合、これは比較的処理負荷がかかりますが、メンブレンマーカーに基づいてライトシートを整列させます。
- マルチビューイメージングの設定
- ライトシートを整列させた後、zスタックとマルチビューイメージングを実行するオプションをアクティブにします。
- ライブスキャンを開始し、標本ナビゲーターを使用してナビゲートし、最初のビューを選択します。このビューでは、スライス間隔を設定し、その後に z スタックの最初と最後のスライスを設定します。この位置をマルチビューダイアログボックスに追加すると、このビューの位置とZスタック情報が記録されます。
- 試料ナビゲーターで角度を変更して、次のビューに移動します。2 番目のビューの Z スタックを設定し、マルチビュー ダイアログ ボックスに情報を追加します。
- 追加のビューについても同じことを繰り返します。
注: 各ビューで特定の最小数のスライスがイメージ化されるようにして、異なるビュー間に十分なオーバーラップを確保し、最終的に処理中にビューを登録するのに役立ちます。スライスの最小数は、ズーム係数とイメージング用に選択した角度の数によって異なります。 - すべてのビューを設定した後、この情報をテキストファイル(たとえば、「embryo-positions」という名前)に保存します。これには、zスタック情報、チューブの(x、y、z)座標、および各ビューの角度の仕様が含まれます。保存後、マルチビューダイアログボックスからすべての位置をクリアします。
- イメージングは比較的大きい無傷の絨毛膜で行われるため、同じ設定でチューブ内のビーズを見ることはできません。そのため、ビーズ情報はチューブ内の異なる位置から取得する必要があります。これを行うには、ビーズが見える胚から離れた別の「y」座標に変換します。この位置をマルチビューダイアログボックスに追加し、この位置をテキストファイル(たとえば、「beads-y」という名前)に保存します。
注:2つの位置でのチューブの曲率の違いによる影響を最小限に抑えるために、ビーズをできるだけ胚サンプルの近くに画像化します。したがって、チューブ内に複数の胚がマウントされている場合は、ビーズを画像化するためにチューブの間に小さなアガロースを残すことが重要です(ステップ1.3.7で述べたように)。 - テキストファイルが保存されているフォルダに移動し、「embryo-positions」ファイルを「beads-positions」という名前の新しいファイルに複製します。'beads-positions' ファイル内のすべてのビューの y 座標を 'beads-y' ファイルの y 座標で置き換えます。これにより、ビーズは同じビュー数、zスタック、x座標で、チューブ内の異なるy位置で画像化されます。
- イメージングソフトウェアに戻り、ソフトウェアに「bead-positions」ファイルをロードします。時系列オプションがアクティブになっている場合は、1 つのサイクルを選択して実験を開始します。ビーズ画像を「ビーズ」として保存し、画像処理中にさまざまなビューを登録するために使用されます。
- ビーズ画像を撮影した後、位置をクリアし、ソフトウェアに最初の「胚位置」ファイルをロードします。適切な時間間隔で必要なサイクル数を設定し、実験を開始します。
3. マルチビュー画像解析
注:マルチビュー画像を融合するために、Multiview Reconstructionプラグインの最新バージョンであるFIJIプラグインであるBigStitcherが利用されます10,17,18。このプラグインをインストールするには、「ヘルプ」メニューの「更新」オプションでアクセスできる「更新サイトの管理」機能にBigStitcherプラグインを追加します。インストールが完了すると、プラグインは「プラグイン」メニューの下に表示されます。融合に関与する広範なステップは次のとおりです:(1)ビーズと胚の両方について.xml/.h5ファイルペアを定義します。(2)すべてのビューをビーズファイルに登録します。(3)デコンボリューションに使用できるビーズの抽出点像分布関数(PSF)(図2A)。(4)登録情報とPSF情報をビーズファイルから胚ファイルに移し、マルチビューデコンボリューションを開始します。これらのステップの大部分は、詳細21で以前に説明されており、ここでは、異なる方法で処理されるステップが説明されている。
- .xmlデータセットの定義
- ビーズと胚の両方について、新しいデータセットをそれぞれ「beads.xml」と「embryo.xml」として定義する(前述の21)。
- インタレストポイントを使用した検出と登録
- beads.xmlファイルが正常にエクスポートされたら、[Multiview Explorer]ダイアログボックスで登録に必要なビューを選択します(図2B)。右クリックして [Detect Interest Points] を選択します。21 で説明されている手順に進みます。
- 関心ポイントを検出したら、すべてのビューを選択して右クリックし、[ Register using Interest Points] を選択します。21で説明されているプロトコルに従います。
- ビードの登録が正常に機能したかどうかを慎重に確認してください。レジストレーションが成功すると、異なるビューからの重なり合うビーズが重ね合わせられます(図2C)。レジストレーションがどの程度正確に機能したかを確認するには、2つの連続するビューを選択し、重なり合う領域に移動し、2つのビューを切り替えて、異なる角度から画像化されたビードが重ね合わされているかどうかを確認します。これを連続するビューごとに繰り返します。
- オーバーラップが正確でない場合は、登録に必要な意味やオーバーラップの許容誤差 ([登録] ダイアログ ボックスでは "RANSAC エラー" と呼ばれます) を緩和して登録を再試行します。
- 登録が成功したら、「Multiview Explorer」ウィンドウで 「保存 」をクリックして、更新された.xmlファイルを保存します。
注: 登録の効率に関する詳細な情報が含まれているため、ログ ファイルも保存してください。ビーズから胚への登録情報を翻訳するには、以下の手順に従います。 - 選択したテキストエディタで bead.xml ファイルを開き、[ViewRegistrations]の下のブロック全体をコピーします。
- embryo.xmlを開き、「ViewRegistrations」ブロックをビーズファイルからコピーしたブロックに置き換えます。複数のチャンネルがある場合は、上記のように各チャンネルの登録情報を置き換えてください。登録情報は、手動で転送することも、ここからダウンロードできるカスタム記述の MATLAB コードを使用して転送することもできます https://github.com/sundar07/Multiview_analysis
- 「BigStitcher」で embryo.xml ファイルを開き、胚の登録が正常に機能したかどうかを注意深く確認してください。ビーズに対して行ったのと同じことを繰り返し、連続する2つのビューごとに対象の構造の重なりを確認します。
注:時折、ビーズが完全に登録されているにもかかわらず、胚の登録がそれほど望ましくない場合があります。これは、ビーズが画像化された位置から胚の位置までチューブの湾曲にわずかな変化がある場合に可能です。さらに、胚は絨毛膜内を自由に浮遊しているため、視線間で微妙な動きがある可能性があります。この場合、胚の核マーカーを使用して2回目の登録を行います。 - これを行うには、「BigStitcher」で embryo.xml ファイルを開き、核マーカーを含むすべてのビューを右クリックして、[ インタレストポイントの検出]を選択します。
- 関心点の名前を「nuclei」に変更し、ビーズに対して実行した手順に進みます。
- 「ガウスの差」パラメータを設定するときは、すべてではないにしてもほとんどの原子核が検出され、異所性核が検出されないことを確認してください。次に、[ 完了]をクリックします。
- これに続いて、これらのビューを登録するには、 Register using Interest points を選択し、 Precise descriptor based (translation-invariant) オプションを選択します。[すべてのビューと関心ポイントを比較] オプションが選択されていることを確認し、関心ポイントとして 'nuclei' を使用します。最初のビューを固定するオプションを使用し、マップし直さないでください。
- 登録には、厳密な正則化とプラグインの既定のパラメーターを持つアフィン モデルを使用します。
- 2つの連続したビューごとに比較して、登録の成功を再確認します。
- 登録が成功したら、「Multiview Explorer」ウィンドウで 「保存 」をクリックして、更新された.xmlファイルを保存します。
- 選択したテキストエディタでembryo.xmlファイルを開き、核マーカーから他のチャンネルに登録情報をコピーします。
- 点像分布関数の抽出と割り当て
- マルチビューデコンボリューションを実行するには、登録したビーズデータセットからイメージングシステムのPSFを抽出し、それを胚ファイルに適用します。
- この情報を取得するには、ビード ファイル内のすべてのビューを選択し、右クリックして[ ポイント スプレッド関数 ]と [抽出 ]オプションを選択します。
- 表示されるダイアログボックスで、[ 対応するインタレストポイントを使用 ]と[ PSFから最小強度投影を削除 ]がオンになっていることを確認し、デフォルトのPSFサイズで続行し、[ OK]をクリックします。
注: すべてのビューで PSF の抽出が成功した場合、ログ ファイルには「抽出された n/n 個の PSF」と表示されます - その後、.xmlファイルを再保存します。「Multiview Explorer」ダイアログボックスのPSF列にチェックマークが付けられたチェックボックスが表示され、抽出されたすべてのPSFとともにそれぞれのフォルダに「psf」フォルダが生成されます。
- embryo.xml ファイルを開き、各ビューに PSF を個別に割り当てます。「ビュー」を右クリックして→「Point Spread Functions」をクリックし→「Assign」を選択し、「Advanced」を選択してから、「選択したすべてのビューに新しいPSF→割り当てる」を選択します。[参照]をクリックし、.xmlファイルパスに移動して、psfフォルダを開きます。
- 選択したビューで対応するIDと一致するPSFを選択し、[ OK]をクリックすると、[マルチビューエクスプローラー]ウィンドウにPSFチェックボックスがチェックされて表示されます。
- 他のすべてのビューに対してこのプロセスを繰り返します。
- マルチビューフュージョンとデコンボリューション
- ポイント スプレッド関数をすべてのビューに割り当てたら、右クリックして[ マルチビュー デコンボリューション]を選択します。
- 現在選択されているビューとして 境界ボックス を選択します。この作業では、デフォルトのOSEMアクセラレーションと反復回数が適切に機能します。
- より高速な計算が必要な場合、または CPU メモリが制限されている場合は、必要に応じて画像をダウンサンプリングします。
注意: 必要なRAMが既存のメモリを超えると、ウィンドウの下部に赤色の警告エラーメッセージがポップアップ表示されます。この警告が表示された場合は、処理中のある時点でプラグインが停止して応答を停止するため、デコンボリューションを開始しないでください。 - デコンボリューションの進行状況を評価するには、5 回の反復ごとに結果が表示されるログ ファイルを確認します。
- 計算速度を上げるには、GPU でマルチビューデコンボリューションを実行します (以前にインストールされている場合)。
注:このプロセスの最後に、融合した画像ウィンドウがポップアップ表示され、tiffファイルとして保存できます。
結果
サンプルを正確に方向付けることは、顕微鏡セットアップを効率的に使用するために不可欠な部分です。ただし、マルチビューライトシートシステムを使用する場合、チューブ内でサンプルを調製する必要があるため、サンプルを手動で方向付けすることは不可能な場合がよくあります。そこで、絨毛膜内で胚が占めるステレオタイプな位置があるかどうかを調べるため、原腸陥入から早期体節までのタイムラプスイメージングが本研究の焦点であったため、ゼブラフィッシュの胚を70%エピボリー(受精後約7時間(hpf)で画像化しました。70%エピボリーでイメージングする直前にサンプルを調製したとき、胚はサンプル間で頻繁に観察される特定の配向を示さなかった。これは望ましくない場合が多いため、サンプルは原腸陥入のかなり前に調製し、イメージングが開始されるまで適切な温度で微量遠心チューブに保管しました。これらの条件下では、70%エピボリー(N = 3;n = 87個の胚)で、胚の向きは、(1)胚の動物-植物(AV)軸がポリマーチューブの長軸に直交している場合の水平、(2)AV軸がポリマーチューブの長軸に平行な場合の垂直、および(3)斜め、 AV軸が鋭角にあったとき(図3A)。水平位置は最も少なく、垂直位置と斜め位置は等しく観察されました(図4)。
胚をチューブに残したとき、胚はこれらのそれぞれの位置で90%エピボリーまで安定しており、その後、ほとんどの胚は向きを変えました。したがって、胚形成の芽の段階での向きの2回目の文書化(約10 hpf)は、向きの変化を説明するために必要でした。これは、独立して調製されたサンプルに対して実行されました。初期の体節期のイメージングには、ポリマーチューブの長軸に直交する脊索を持つ胚が、軸15に沿って形成される複数の両側体節を視覚化できるため、理想的な配向であることが以前に報告されていました。原腸陥入前にサンプルを調製した場合(N = 3、n = 93胚)、胚の約25%がこの向きを示し(図4)、これらの胚は少なくとも8体節期までこの向きで安定しており、以前の報告15と一致しています。残りの胚は、芽の段階で他のさまざまな向きを示しました( 図3B および 図4に分類)。しかし、それらの多くは、体節形成の初期に水平位置に向きを変えました。興味深いことに、サンプルが原腸陥入前、70%エピボリー、または芽期の直前に調製されたかどうかに関係なく、芽の段階で同様の割合の胚が水平方向を示しました。したがって、サンプル調製のタイミングは、70%エピボリーでのイメージングで観察されたものとは異なり、体節ステージのイメージングの対象の水平方向にとってそれほど重要ではないようです。
マルチビューシステムの利点は、同じサンプルを複数の角度から表示できることです。しかし、上記の段階のゼブラフィッシュの胚については、胚全体にわたって細胞分解能を得るために必要なビューの数は明らかではありません。これを特徴付けるために、ゼブラフィッシュの胚を検出アームの20x/1 NA対物レンズを使用して、ズーム係数1でイメージングしました。これは、4.57 μmの薄いシートの厚さに相当します。これらの設定では、胚は6.5μmのピクセルサイズと1920×1920ピクセルの領域を持つsCMOSカメラの視野全体を覆いました。アクチンフィラメント(Utr-GFP)を標識したトランスジェニック系統から得られた1細胞期胚にヒストンタグ付き蛍光色素(H2A-mCherry)にmRNAをマイクロインジェクションすると、胚の核と細胞膜の両方を可視化することができました。異なる角度間隔の胚のマルチビュー融合を行うために、ダブルトランスジェニック胚およびチューブ内のビーズの360°取得を、30°間隔(n = 70%エピボリーの胚、n = 芽期の3胚)または45°(n = 70%エピボリーの3つの胚、n = 芽期の3胚)ごとに、各角度で約100スライス、スライス間隔2μmで行った。画像処理中に交互の角度をスキップすることで、30°と45°の取り込みはさらに60°と90°のデータセットを提供しました。
次いで、取得した画像をビーズ情報を用いて登録し、登録詳細をプロトコールのセクションに記載したように胚データセットに転送した。BigStitcherプラグインを使用したレジストレーションは、90°データセットを除いて、さまざまな取得で成功しましたが、これは各角度からのサンプルのカバレッジが少ないためである可能性があります。これを克服するために、胚を90°ごとに、各角度で約400スライス、スライス間隔2μmで、70%エピボリーステージとバッドステージの両方でイメージングし、成功裏に登録されました(各ステージでn = 3胚)。
次のステップは、登録されたデータセットのマルチビューフュージョンとデコンボリューションを実行することでした。これは、計算を高速化するために4倍のダウンサンプリングで行われました。個々の視点からの核の表現とマルチビュー再構築された胚(図5)に見られるように、個々のビューはより小さな視野をカバーしており、融合すると胚全体の画像が得られました。表現のために、核はFIJIベースのプラグインであるMastodon(https://github.com/mastodon-sc/mastodon)を使用して検出され、これはFIJIの「ヘルプ」メニューの「更新サイトの管理」セクションに追加でき、追加するとプラグインメニューからアクセスできます。検出については、まずそれぞれの画像をXML/hdf5形式に変換し、次にMastodonの「Detection」プラグインとDoG検出器(Diameter 6 μm、Quality Threshold 80)を使用して核検出を行いました。
さまざまな融合画像の中で、90°のデータセットはサンプルの深部で非常に高いバックグラウンドを示しており、定量化の実行には適していませんでした。したがって、ショウジョウバエの胚のような小さなサンプルは、他の研究で90°間隔で頻繁に画像化されています9,22とは異なり、20x/1 NA対物レンズを使用して初期のゼブラフィッシュ胚をイメージングする場合には推奨されません。30°、45°、60°の融合データセット間では、定性的には、核情報に大きな違いはありませんでした(図6B、上段)。しかし、セル境界などのより微細な構造は、他のデータセットと比較して、30°融合データセットの方がはるかによく分解されているように見えました(図6C、上段)。
この観察結果を確認するために、Mastodonを使用して、30°、45°、および60°ごとのイメージングから得られた融合画像中の核を検出しました。融合した画像内の3つの領域(胚の表面から20μm(図6A)、50μm、および100μmの深さにそれぞれ1つずつ)を解析用に選択しました。画像間での核検出の効率を比較するために、検出は上記のように、異なる角度間隔からの融合画像間で同一のパラメータを使用して実行しました。解析したすべての領域で、30°、45°、または60°ごとのイメージングから得られた融合画像に関係なく、すべての核が検出されました(図6B、下段)。したがって、原子核のような球状構造は、情報を失うことなく、上記の角度間隔のいずれでも画像化することができます。
細胞境界を解析するために、細胞24、25、26のセグメント化に日常的に使用されるFIJIプラグインであるTissue Analyzer23を使用した。Mastodonと同様に、Tissue Analyzerプラグインは、FIJIの「ヘルプ」メニューの「更新サイトの管理」セクションに追加でき、追加するとプラグインメニューからアクセスできます。細胞境界は、デフォルトのパラメータと、組織の深さに応じて1.5から2の範囲の強いぼかしと1の弱いぼかしを使用して、流域アルゴリズムを使用してセグメント化されました。これらのパラメータはすべての分析で一定に保たれ、簡単な比較が容易になりました。セグメント化された画像を手動で元の入力画像と比較すると、ソフトウェアがセル境界の検出に失敗したか、存在しないセル境界を描画するエラーが観察されました(図6C、下段)。Tissue Analyzer プラグインによって行われたエラーの数は、その領域で検出された結合の総数に正規化され、「境界セグメンテーションエラー」として計算されました。これらの誤差は解析したすべての領域に存在しましたが、45°および60°の角度間隔でのイメージングから得られた融合画像では、30°と比較して誤差の数が大幅に増加しました(図6D)。これは、角度間隔が長くなると、融合した画像の解像度がますます悪くなることを示しています。したがって、細胞境界などのより微細な構造をセグメント化する場合、角度間隔が狭いほど、下流の処理が容易になります。
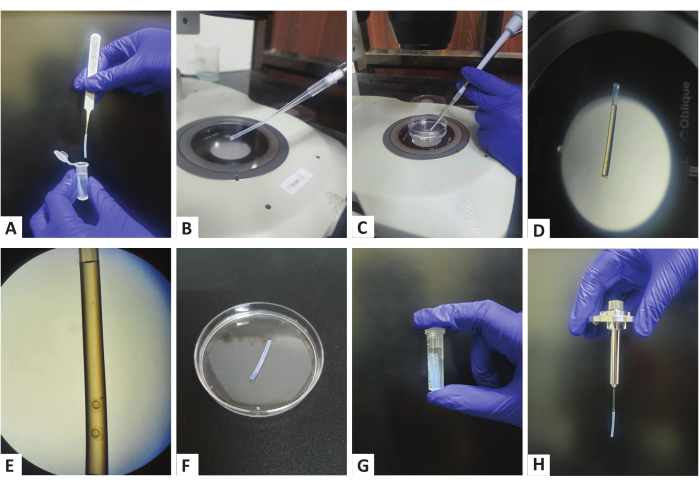
図1:ポリマーチューブを使用したサンプル調製。 (A)保管された微量遠心チューブからポリマーチューブを鉗子を用いて採取する。(B)チューブを200μLマイクロピペットの先端に取り付けます。(C)ピペットの助けを借りて胚をチューブに吸引します。(D,E)チューブの底に向かって芽期の胚が見えるポリマーチューブ。(F)ポリマーチューブをE3の入ったペトリ皿に載せて、アガロースを固めます。(G)E3を充填した微量遠心チューブにポリマーチューブを保管します。(H)ポリマーチューブが取り付けられたサンプルホルダー。 この図の拡大版を表示するには、ここをクリックしてください。

図2: ビーズを使用したマルチビュー画像解析のワークフローとレジストレーション。 (A)マルチビュー画像解析パイプライン。(B)画像は、FIJIプラグインであるBigStitcherの「Multiview Explorer」ウィンドウを示しており、各ビューは1行として表示され、角度、チャネル、登録、関心ポイント、およびPSFに関する情報が含まれています。プロトコルで説明されているすべてのコマンドは、ポップアップメニューに示すように、ビューを選択して右クリックすると表示されます。選択したビューは、次に示すように BigDataViewer ウィンドウで視覚化できます。(C)30°角間隔でのイメージングから得られたレジストレーション前(左)とレジストレーション後(右)の代表的なビーズ画像。すべての角度ビューが選択され、表示されています。スケールバー:75μmこの 図の拡大版を表示するには、ここをクリックしてください。

図3: デフォルトの胚の向きの概要。胚が70%エピボリー(A)および芽期(B)で絨毛膜内に収まる向きの代表的な画像。各パネルの一番上の行はライトシートシステムから取得した明視野画像を示し、下の行は代表的な漫画を示しています。(B)の矢印は、向きの識別に使用された脊索の位置を示しています。AP、動物の棒;Vp、植物ポール;A、前方;P、後部。スケールバー:100μmこの 図の拡大版を表示するには、ここをクリックしてください。

図4: さまざまな胚の向きの統計。(A)積み重ねられた列は、原腸陥入前にサンプルを調製したときに70%エピボリーで示された配向に落ちる胚の割合を示しています。(B)積み重ねられた列は、原腸陥入前にサンプルを調製した場合の芽期(左)、70%エピボリー(中央)、および芽期(右)で示された配向に該当する胚の割合を示しています。N、胚が得られた独立したクラッチの数。n、胚の総数。 この図の拡大版を表示するには、ここをクリックしてください。

図5: マルチビュー再構成胚からの核の表現。 (ア)3D散布図は、 30°の角度間隔で画像化されたマルチビュー再構成胚で検出された核を表しています。各円は原子核を表し、原子核の位置の重心がプロットされます。核座標は、FIJIのプラグインであるMastodonを使用して取得しました。(B)同じ胚の3つの代表的なビューからの核の3D散布図-描写は60°離れています。各原子核の色はランダムに割り当てられました。 この図の拡大版を表示するには、ここをクリックしてください。

図6: 融合画像におけるセルラースケール情報の比較。(A)左の画像は、表面から20μmの深さでマルチビュー再構成された胚のスナップショットを示しています。黄色の円は、詳細な解析に使用される領域を示しています。スケールバー:50μm(すべてのパネル)。(B)胚の表面から深さ20μmの核の代表的な生画像で、それぞれ30°、45°、60°ごとに画像化されたデータセットから得られたもの(上)。同じ画像で検出された核(緑色)は、FIJIのプラグインであるMastodon(下)です。(C)アクチンマーカーであるUtr-GFPを使用して、30°、45°、60°ごとに画像化された3つのデータセットから、胚の表面から深さ20μmの深さでの代表的な生画像(上)。FIJIのプラグインであるTissue Analyzerによってセグメント化された境界は、同じ画像に対して表示されます(下)。矢印は、Tissue Analyzer が境界セグメンテーションで行ったエラーを示しており、黄色の矢印は欠落している境界を表し、白い矢印は、視覚的に境界が存在しないように見える場合に誤って検出された境界を表しています。(D)箱ひげ図は、Tissue Analyzerが30°、45°、60°ごとに異なる深さで画像化されたマルチビュー再構築胚間で行ったエラーの割合を示しています。エラーバーは、四分位範囲の1.5倍を示します。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
関心領域を画像化するために胚を正しい向きに配置することは、ユーザーの顕微鏡セッションが失敗することが多い律速ステップの1つです。これは、サンプルがチューブ内に埋め込まれているため、向きを手動で操作することが難しいマルチビューライトシート顕微鏡ではなおさらです。このプロセスを支援するために、この研究では、胚の入ったポリマーチューブを微量遠心チューブ内で直立させた場合に、ゼブラフィッシュの胚が絨毛膜内の70%のエピボリー段階と初期の体節期の間で占めるさまざまな位置の統計を報告しています。
70%がエピボリーで、垂直または水平に配向した胚は、胚全体にわたるイベントの補完的な情報を提供します。水平方向は、動物極の細胞ダイナミクスと、植物極から見たエピボリーを受けている細胞のダイナミクスを視覚化することを可能にしますが、垂直方向は、背側オーガナイザーの細胞ダイナミクス、原腸陥入中の細胞の内在化、収束-伸展の流れなど、胚の背側と腹側の視覚化を可能にします。この研究は、サンプルがイメージングの数時間前に準備されている場合、純粋に偶然に垂直方向の胚を得る可能性が高いことを示しています。ただし、これらの段階では、水平方向はあまり一般的ではないようです。エピボリーが70%より前の段階では、タイムラプスイメージングに進む前に、絨毛膜内の胚の向きに関する同様の統計を記録することをお勧めします。
芽の段階から、イメージングのための最良の向きは水平に配置された胚であり、これにより胚15の前後体軸全体に沿った細胞動態を追うことができる。この向きで胚を得るのは比較的簡単で、サンプルがいつ準備されるかに関係なく、胚の約25%がこの安定した位置に収まります。一般に、関心のある任意の段階をイメージングするには、各チューブに2〜3個の胚を入れた約15本のチューブを準備することが推奨されます。これにより、目的の向きの胚を得る高い確率が保証され、したがって、任意の日に顕微鏡セッションが成功します。さらに、タイムラプスイメージングに適した向きの胚を選択するために、目的のステージの約1時間前にチューブを選別することをお勧めします。
正しい向きの胚を取得した後、イメージングのためにビューの数とビュー間の角度間隔を考慮することが重要です。これは、最低限必要な空間分解能と時間分解能によって異なります。胚の特定の構造または領域を画像化する角度が多ければ多いほど、空間分解能は向上しますが( 図6を参照)、これは時間分解能の妥協につながります。このプロトコルでは、2つの照明アームと1つの検出アームを備えたシステムを使用して、角度間隔30°の360°全体にわたって2つのチャネルをイメージングし、スライス間隔を2μmにして各角度で約100個のzスライスをイメージングすると、1回のポイントで約3分かかりました。これは初期胚期の細胞を追跡するには十分ですが、より速いダイナミクスを持つイベントを捕捉する場合は、取得する角度の数を減らして時間分解能を向上させる必要がありますが、特に細胞境界などの微細な構造をセグメント化する必要がある場合は、空間分解能が損なわれます。時間分解能を向上させるために考慮すべき第2の選択肢は、2つの検出アームを備えたマルチビューシステムを使用することであり、これにより、ゼブラフィッシュ胚16の原腸陥入中の細胞運動を追跡するために最近使用されたように、取得速度が大幅に向上します。さらに、このプロトコルでは20x/1 NA対物レンズのみがテストされましたが、イメージングするサンプル、必要な空間分解能、および視野に応じて、適切な対物レンズを考慮する必要があります。まとめると、特定のサンプルと視野に必要なダウンストリーム処理と定量に基づいて、視野の数と角度間隔を慎重に選択する必要があります。しかし、初期のゼブラフィッシュの胚全体を、特に複数の角度から細胞スケールの解像度でイメージングするには、倍率が低くNAの対物レンズ(例えば、10x/0.5 NAの対物レンズ)は解像度がはるかに低くなり、セグメンテーションエラーが増える可能性が高いため、20x/1 NAの対物レンズが最適であるように思われます。一方、高倍率とNAの対物レンズは、カメラの視野内で胚全体をカバーするのに適していません。
本研究では、マウンティングとイメージングの戦略を検討するために、細胞境界をトレースするためのプロキシとして使用できるアクチンマーカーと、核を検出および追跡するためのヒストンマーカーを使用しました。実際、マルチビューライトシートシステムを使用した以前に発表された研究のほとんどは、同様のマーカー5、7、15、16、27を優先的に使用しています。これらのマーカーを選択する主な理由は、マルチビューライトシート顕微鏡法が主に、組織スケールまたは数時間から数日にわたる長期間にわたる胚全体にわたる細胞スケールのダイナミクス(細胞形状や細胞再配列など)を追跡するために使用されることです。一方、細胞内分解能を得る必要がある場合には、関心領域をマーキングする異なるトランスジェニックラインを使用することができるが、このシナリオでは、マルチビューライトシート顕微鏡は装置の最良の選択ではなく、むしろ共焦点、超解像システム、あるいは格子ライトシート顕微鏡28、29は、調査されている問題により適している可能性があります。
議論されたプロトコルは、原腸陥入から15体節期のゼブラフィッシュ胚30(約17 hpf)まで、何の変更もなく良好に機能し、ここではテストされていませんが、原腸陥入期の前の以前の胚に対しても機能する可能性があります。15体節期を超えると、自発的な筋肉収縮が始まり30,31、これは、チューブとサンプルチャンバーに麻酔薬であるトリカインを追加することで抑制できます。さらに、約18 hpfで、卵黄から離れる尾の反転が始まり30、これにより胚は視野から遠ざかります。これに対処するためには、胚32に焦点を合わせ続けるために、伸長する端部を追跡する追跡アルゴリズムを採用する必要がある。さらに、神経発達を追うためなど、絨毛膜を欠損した後期胚のイメージングには、最近行われたように別のマウント戦略を採用する必要がある33。
ゼブラフィッシュの胚を無傷の絨毛膜でイメージングする場合、レジストレーションマーカーとして機能するビーズと胚の両方を同じ視野で捕捉することは、20x/1 NA対物レンズを使用しても不可能です。この研究では、サンプルの上または下に存在するビーズからの情報に異なるビューを登録し、その後、処理中にビーズから胚に登録をシフトすることにより、簡単な代替手段を提供します。この方法で胚サンプルが登録されると、サンプル内に存在する核をレジストレーションマーカーとして使用して、初期レジストレーションをさらに微調整するための2回目のレジストレーションを行うことができます。タイムラプスイメージングを行う場合、核情報は、次に、以前に報告されたように連続した時点の登録に用いることができる21。このプロトコルの1つの代替手段は、ビーズを放棄し、代わりに初期登録に核情報を使用することです。しかし、BigStitcherプラグインでは、処理中に多くの核が胚の深部で検出されないことや、特定の角度から見たときに胚に存在する核の数が比較的少ないため(例えば、エピボリー後期に腹側から見た場合など)により、BigStitcherプラグインで登録がしばしば失敗しました(ここには示されていません)。
このプロトコルでは、サンプルの上または下からのビーズ情報がレジストレーションに使用され、サンプルをイメージングするのと同じビューでビーズをイメージングする必要がないため、このプロトコルを使用して、特定の関心領域のみをイメージングすることができますが、胚全体ではなく異なる角度から高倍率でイメージングできます。さらに、レジストレーション用のサンプル間からのビーズ情報を使用することにより、チューブ内に垂直に積み重ねられた複数のサンプルをイメージングするために、このプロトコルを容易に適応させることができます。
開示事項
著者は、競合する利益を宣言しません。
謝辞
魚類施設のメンテナンスにはKalidas Kohale博士と彼のチーム、ライトシート顕微鏡のメンテナンスにはKV Bobyに感謝します。SRNは、インド政府原子力省(DAE)からの財政支援を認めています(プロジェクト識別番号。RTI4003、DAE OM no. 1303/2/2019/R\&D-II/DAE/2079 (2020年2月11日付)、マックスプランク協会パートナーグループプログラム(M.PG.A MOZG0010)および理工学研究委員会スタートアップ研究助成金(SRG/2023/001716)。
資料
| Name | Company | Catalog Number | Comments |
| Agarose, low gelling temperature | Sigma-Aldrich | A9414 | |
| Calcium Chloride dihydrate | Sigma-Aldrich | 12022 | |
| FIJI | Version: ImageJ 1.54f | ||
| Latex beads, carboxylate-modified polystyrene, fluorescent red, 0.5 μm mean particle size, aqueous suspension | Sigma-Aldrich | L3280 | |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | |
| mMESSAGE mMACHINE SP6 Transcription kit | ThermoFischer Scientific | AM1340 | For in vitro transccription of H2A-mCherry plasmid |
| Potassium Chhloride | Sigma-Aldrich | P9541 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P0662 | |
| PTFE Sleeving AWG 15L - 1.58 mm ID x 0.15 mm Wall +/-0.05 | Adtech Innovations in Fluoroplastics | STW15 | PTFE tubes |
| Sodium Chloride | Sigma-Aldrich | S3014 | |
| Sodium phosphate dibasic | Sigma-Aldrich | 71640 | |
| Ultrasonic Cleaner | Labman | LMUC3 | Ultrasonicator |
| Zeiss LightSheet 7 System | Zeiss |
参考文献
- Wan, Y., McDole, K., Keller, P. J. Light sheet microscopy and its potential for understanding developmental processes. Annu Rev Cell Dev Biol. 35, 655-681 (2019).
- Keller, P. J., Schmidt, A. D., Wittbrodt, J., Stelzer, E. H. Reconstruction of zebrafish early embryonic development by scanned light sheet microscopy. Science. 322 (5904), 1065-1069 (2008).
- Keller, P. J., et al. high-contrast imaging of animal development with scanned light sheet-based structured-illumination microscopy. Nat Methods. 7 (8), 637-642 (2010).
- Keller, P. J. Imaging morphogenesis: Technological advances and biological insights. Science. 340 (6137), 1234168(2013).
- Schmid, B., et al. High-speed panoramic Light sheet microscopy reveals global endodermal cell dynamics. Nat Commun. 4 (1), 2207(2013).
- Strnad, P., et al. Inverted Light sheet microscope for imaging mouse pre-implantation development. Nat Methods. 13 (2), 139-142 (2016).
- McDole, K., et al. In toto imaging and reconstruction of post-implantation mouse development at the single-cell level. Cell. 175 (3), 859-876 (2018).
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Krzic, U., Gunther, S., Saunders, T. E., Streichan, S. J., Hufnagel, L. Multiview Light sheet microscope for rapid in toto imaging. Nat Methods. 9 (7), 730-733 (2012).
- Preibisch, S., Saalfeld, S., Schindelin, J., Tomancak, P. Software for bead-based registration of selective plane illumination microscopy data. Nat Methods. 7 (6), 418-419 (2010).
- Swoger, J., Verveer, P., Greger, K., Huisken, J., Stelzer, E. H. K. Multiview image fusion improves resolution in three-dimensional microscopy. Opt Express. 15 (13), 8029-8042 (2007).
- Swoger, J., Huisken, J., Stelzer, E. H. Multiple imaging axis microscopy improves resolution for thick-sample applications. Opt Lett. 28 (18), 1654-1656 (2003).
- Kaufmann, A., Mickoleit, M., Weber, M., Huisken, J. Multilayer mounting enables long-term imaging of zebrafish development in a light sheet microscope. Development. 139 (17), 3242-3247 (2012).
- Weber, M., Mickoleit, M., Huisken, J. Multilayer mounting for long-term light sheet microscopy of zebrafish. J Vis Exp. (84), e51119(2014).
- Naganathan, S. R., Popović, M., Oates, A. C. Left-right symmetry of zebrafish embryos requires somite surface tension. Nature. 605 (7910), 516-521 (2022).
- Shah, G., et al. Multi-scale imaging and analysis identify pan-embryo cell dynamics of germlayer formation in zebrafish. Nat Commun. 10 (1), 5753(2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Hörl, D., et al. BigStitcher: Reconstructing high-resolution image datasets of cleared and expanded samples. Nat Methods. 16 (9), 870-874 (2019).
- Behrndt, M., et al. Forces driving epithelial spreading in zebrafish gastrulation. Science. 338 (6104), 257-260 (2012).
- Westerfield, M. The Zebrafish Book: A Guide for the Laboratory Use of Zebrafish (Danio Rerio). , University of Oregon Press. (2000).
- Icha, J., et al. Using light sheet fluorescence microscopy to image zebrafish eye development. J Vis Exp. (110), e53966(2016).
- Tomer, R., Khairy, K., Amat, F., Keller, P. J. Quantitative high-speed imaging of entire developing embryos with simultaneous multiview Light sheet microscopy. Nat Methods. 9 (7), 755-763 (2012).
- Aigouy, B., Prud'homme, B. Segmentation and quantitative analysis of epithelial tissues. Methods Mol Biol. 2540, 387-399 (2022).
- Mancini, L., et al. Apical size and deltaA expression predict adult neural stem cell decisions along lineage progression. Science Adv. 9, 7519(2023).
- Piscitello-Gomez, R., Mahmoud, A., Dye, N., Eaton, S. Sensitivity of the timing of Drosophila pupal wing morphogenesis to external perturbations. bioRxiv. , (2023).
- Tsuboi, A., Fujimoto, K., Kondo, T. Spatiotemporal remodeling of extracellular matrix orients epithelial sheet folding. Science Adv. 9, 2154(2023).
- Fu, Q., Martin, B. L., Matus, D. Q., Gao, L. Imaging multicellular specimens with real-time optimized tiling Light sheet selective plane illumination microscopy. Nat Commun. 7 (1), 11088(2016).
- Chen, B. C., et al. Lattice Light sheet microscopy: imaging molecules to embryos at high spatiotemporal resolution. Science. 346 (6208), 1257998(2014).
- York, H. M., et al. Deterministic early endosomal maturations emerge from a stochastic trigger-and-convert mechanism. Nat Commun. 14 (1), 4652(2023).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Melançon, E., Liu, D. W. C., Westerfield, M., Eisen, J. S. Pathfinding by identified zebrafish motoneurons in the absence of muscle pioneers. J Neurosci. 17 (20), 7796(1997).
- Rohde, L. A., et al. Cell-autonomous timing drives the vertebrate segmentation clock's wave pattern. eLife. , 13:RP93764 (2024).
- Haynes, E. M., et al. KLC4 shapes axon arbors during development and mediates adult behavior. eLife. 11, e74270(2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved