Method Article
Imagem de microscopia de folha de luz e estratégias de montagem para embriões iniciais de peixe-zebra
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Uma estratégia de preparação de amostras para imagens de embriões iniciais de peixe-zebra dentro de um córion intacto usando um microscópio de folha de luz é descrita. Ele analisa as diferentes orientações que os embriões adquirem dentro do córion nos estágios de 70% de epibólia e gema e detalha estratégias de imagem para obter resolução em escala celular em todo o embrião usando o sistema de folha de luz.
Resumo
A microscopia de folha de luz tornou-se a metodologia de escolha para imagens ao vivo de embriões de peixe-zebra em longas escalas de tempo com fototoxicidade mínima. Em particular, um sistema multiview, que permite a rotação da amostra, permite a obtenção de imagens de embriões inteiros de diferentes ângulos. No entanto, na maioria das sessões de imagem com um sistema multiview, a montagem da amostra é um processo problemático, pois as amostras geralmente são preparadas em um tubo de polímero. Para auxiliar nesse processo, este protocolo descreve estratégias básicas de montagem para imagens do desenvolvimento inicial do peixe-zebra entre os estágios de 70% de epicolia e somite inicial. Especificamente, o estudo fornece estatísticas sobre as várias posições padrão dos embriões nos estágios de 70% de epibólio e botão dentro do córion. Além disso, discute o número ideal de ângulos e o intervalo entre os ângulos necessários para a obtenção de imagens de embriões inteiros de peixe-zebra nos estágios iniciais de desenvolvimento, para que as informações em escala celular possam ser extraídas pela fusão das diferentes visualizações. Finalmente, como o embrião cobre todo o campo de visão da câmera, que é necessário para obter uma resolução em escala celular, este protocolo detalha o processo de uso de informações de esferas de cima ou de baixo do embrião para o registro das diferentes visualizações.
Introdução
Garantir o mínimo de fototoxicidade é um requisito importante para a obtenção de imagens de embriões vivos com alta resolução espaço-temporal por longos períodos. Na última década, a microscopia de folha de luz tornou-se a metodologia de escolha para atender a esse requisito 1,2,3,4,5,6,7. Resumidamente, nesta técnica que foi usada pela primeira vez em 2004 para capturar processos de desenvolvimento8, duas folhas finas de laser alinhadas passam pelo embrião de extremidades opostas, iluminando apenas o plano de interesse. Uma objetiva de detecção é colocada ortogonalmente e, em seguida, a luz fluorescente emitida de todos os pontos iluminados da amostra é coletada simultaneamente. Uma imagem 3D é então obtida movendo sequencialmente o embrião através da folha de luz estática.
Além disso, em uma forma específica dessa metodologia, denominada microscopia de folha de luz multiview, as amostras podem ser suspensas em um tubo de polímero que pode ser girado usando um rotor, permitindo a imagem do mesmo embrião de vários ângulos 9,10,11. Após a imagem, as imagens de vários ângulos são fundidas com base em marcadores de registro, que normalmente são marcadores fluorescentes globulares dentro do embrião (por exemplo, núcleos) ou no tubo (por exemplo, esferas fluorescentes). A imagem e a fusão multiview melhoram significativamente a resolução axial, fornecendo resolução isotrópica em todas as três dimensões12. Embora esta seja uma grande vantagem, um grande desafio da metodologia multiview é a montagem de amostras, onde os embriões devem ser montados e mantidos no lugar nos tubos durante todo o curso de imagem.
Para realizar imagens multiview, para manter os embriões no lugar e evitar o movimento durante a imagem, os embriões podem ser incorporados em agarose. No entanto, isso geralmente leva a um crescimento e desenvolvimento prejudiciais, particularmente para embriões de peixe-zebra em estágio inicial13, o sistema modelo discutido aqui. Uma segunda estratégia de montagem é usar um tubo fino que seja apenas um pouco maior que o diâmetro do embrião, onde o embrião pode ser puxado para dentro do tubo junto com o meio embrionário, seguido pelo fechamento do fundo do tubo com um tampão de agarose14. Nesse método, como o tubo é preenchido com meio embrionário, marcadores de registro, como contas fluorescentes, não podem ser usados para a fusão das diferentes visualizações e, portanto, o registro depende de marcadores dentro do embrião. Em geral, as contas atuam como melhores marcadores de registro, pois o sinal dos marcadores dentro do embrião se degrada ao se aprofundar na amostra devido às limitações de iluminação e detecção de qualquer microscópio.
Assim, uma terceira abordagem, que será detalhada aqui e usada anteriormente 5,13,14,15,16, é a imagem de embriões iniciais de peixe-zebra com um córion intacto e o preenchimento do tubo com uma porcentagem mínima de agarose, que contém contas como marcadores de registro. Nesse cenário, como a intervenção manual para posicionar embriões dentro de um córion não é possível, este estudo fornece estatísticas sobre a orientação padrão em que os embriões iniciais de peixe-zebra se enquadram, concentrando-se particularmente em 70% dos estágios de epíbulo e brotamento. Em seguida, discute o número ideal de visualizações necessárias para a imagem de embriões em estágio inicial com resolução em escala celular e detalha o processo de fusão usando o BigStitcher, um plug-in baseado em FIJI 10,17,18. Juntos, este protocolo, que usa uma objetiva de 20x/1 NA, visa facilitar os embriologistas de peixe-zebra no uso de sistemas de folha de luz multiview para imagens de embriões com núcleos e marcadores de membrana desde a gastrulação até os estágios iniciais de somito.
Protocolo
A manutenção do peixe-zebra e os procedimentos experimentais utilizados neste estudo foram aprovados pelo comitê institucional de ética animal, vide Referência TIFR/IAEC/2023-1 e TIFR/IAEC/2023-5. Embriões obtidos pelo cruzamento de peixes heterozigotos expressando Tg(actb2:GFP-Hsa.UTRN)19 foram injetados com mRNA H2A-mCherry (30 pg) no estágio unicelular. O mRNA de H2A-mCherry foi sintetizado usando o plasmídeo pCS2+ H2A-mCherry (um presente do laboratório de Oates, EPFL) por transcrição in vitro . Embriões que expressam ambos os marcadores, referidos como Utr-GFP e H2A-mCherry, respectivamente, no restante do protocolo, foram fotografados nos estágios de 70% de epibólia e gema. Os detalhes dos reagentes e equipamentos usados no estudo estão listados na Tabela de Materiais.
1. Preparação da amostra para imagens multiview
- Preparação de tubos FEP/PTFE
- Limpe os tubos FEP/PTFE seguindo os procedimentos descritos anteriormente14.
- Após a limpeza, corte os tubos em comprimentos de 2-2,5 cm conforme a necessidade e armazene-os em tubos de microcentrífuga de 2 mL contendo água destilada duplamente. Os tubos podem ser armazenados dessa maneira por cerca de um mês.
- Antes da preparação da amostra, aqueça os tubos a 70-75 °C por aproximadamente 25 min para endireitar os tubos. Evite a superlotação em cada tubo de microcentrífuga para evitar aglomeração, garantindo espaço suficiente para um endireitamento eficaz do tubo.
NOTA: Use luvas durante todo o procedimento de manuseio do tubo, pois qualquer impressão digital/partícula de poeira deixada nos tubos pode interferir na imagem.
- Preparação de agarose
- Prepare as soluções de estoque de buffer E3 50x da seguinte forma:
- Estoque 1 - 3,252 g de Na2HPO4, 0,285 g de KH2PO4, 11,933 g de NaCl, 0,477 g de KCl em 1 L de água deionizada
- Estoque 2 - 2.426 g de CaCl2, 4.067 g de MgSO4 em 1 L de água deionizada.
- Para fazer 1x tampão E3, adicione 20 mL de cada uma das soluções estoque 1 e 2 em 960 mL de água deionizada.
NOTA: Não adicione azul de metileno no tampão E3 usado para imagens, pois isso causa dispersão da luz. - Dissolver a agarose de baixo ponto de fusão em 1x E3 aquecendo a 70-75 °C num bloco de calor com agitação até que a solução fique límpida, sem grumos ou cristais.
- Para imagens multiview, adicione esferas fluorescentes disponíveis comercialmente (consulte a Tabela de Materiais) à solução de agarose, o que permite o registro da imagem durante a análise. Sonicar os grânulos à temperatura ambiente com frequência de 40 kHz por 20-30 min em um ultrassônico em banho-maria para desagregar os grânulos.
- Após a sonicação, adicione 1 μL de grânulos a 10 mL de solução de agarose em um tubo de 15 mL e vortex o tubo bem para garantir a dispersão uniforme dos grânulos.
NOTA: O volume da solução de esferas a ser adicionada à agarose depende da solução estoque e do fabricante. Experimente uma variedade de volumes e escolha uma concentração em que as contas apareçam bem dispersas quando fotografadas no sistema de folha de luz. Muitas contas podem se agregar apesar da sonicação e do vórtice, enquanto poucas contas podem afetar o registro da imagem. - Manter o tubo de agarose com esferas adicionadas num banho-maria fechado mantido a 37 °C durante, pelo menos, 30 minutos antes da preparação da amostra. Isso garante que a temperatura da agarose caia de 70 °C para 37 °C.
- Prepare as soluções de estoque de buffer E3 50x da seguinte forma:
- Preparação da amostra
- Configure os peixes em pares com divisórias à noite antes do dia da imagem. Remova as divisórias por cerca de 15 min e colete os embriões usando o procedimento padrão20.
- Quando os embriões atingirem o estágio de interesse para a preparação da amostra, despeje todos os 10 mL de agarose (com contas) mantidos a 37 ° C em uma placa de Petri de 6 cm.
- Aguarde cerca de 2-3 minutos para que a temperatura desça a 33 °C antes de transferir 10 a 15 embriões para a solução de agarose.
NOTA: Este é um passo importante para evitar qualquer possível choque térmico aos embriões transferidos para a agarose. - Certifique-se de que o mínimo possível de tampão E3 seja adicionado à solução de agarose durante a transferência de embriões. Gire a placa de Petri para que o pequeno tampão que teria sido transferido fique bem disperso.
- Use luvas para o resto do procedimento de preparação da amostra.
- Pegue uma micropipeta de 200 μL com uma ponta de pipeta apropriada e insira um tubo reto limpo retirado do tubo da microcentrífuga na ponta da pipeta, conforme mostrado na Figura 1A, B.
- Aspire um pouco de agarose para o tubo, seguido por 2-3 embriões usando a micropipeta (Figura 1C, D). Certifique-se de que os embriões estejam próximos ao fundo do tubo (Figura 1E), o que se tornará importante durante a configuração da imagem. Além disso, certifique-se de que haja um pouco de agarose entre os diferentes embriões (Figura 1E) para que as contas nesta região possam ser usadas como marcadores de registro.
- Sem liberar a pressão da pipeta, retire o tubo da ponta da pipeta e coloque-o na tampa da placa de Petri cheia de E3 para que a agarose solidifique (Figura 1F).
- Repita as etapas 1.3.6 a 1.3.8 até que todos os embriões da placa de Petri sejam transferidos para os tubos.
- Assim que a agarose solidificar (o que pode levar de 5 a 10 minutos e pode ser confirmado verificando a agarose na placa de Petri), transfira os tubos para um tubo de microcentrífuga de 2 mL cheio de E3 (Figura 1G).
- Armazene os tubos com os embriões em uma incubadora a 28 ° C ou 33 ° C, conforme necessário, antes de prosseguir para a imagem multiview.
2. Imagem multiview
NOTA: Esta etapa apresenta um procedimento geral para imagens multivisualização de embriões de peixe-zebra em seus estágios iniciais de desenvolvimento. O método detalhado abaixo pode ser facilmente adaptado a qualquer sistema de microscopia de folha de luz multiview.
- Localizando o embrião
NOTA: Encha a câmara de amostra do microscópio com o meio embrionário20 e ajuste a temperatura da câmara de amostra para a temperatura de interesse pelo menos 10 min antes da imagem.- Monte o porta-amostras conforme descrito anteriormente21, no entanto, com uma modificação. Como a imagem será realizada através do tubo, insira o tubo com embriões diretamente em um capilar de diâmetro correto, de modo que seja mantido no lugar sem empurrar a agarose para fora e perturbar os embriões na parte inferior (Figura 1H).
NOTA: Como a câmara de amostra tem uma altura definida, garantir que os embriões estejam localizados na parte inferior do tubo durante a preparação da amostra (conforme mencionado na etapa 1.3.7) permitiria posicionar o embrião no campo de view sem atingir o chão da câmara. - Insira o suporte de amostra no sistema multiview e use os controles x e y para trazer o embrião para o centro do campo de view.
- Use o controle z do navegador de amostras para colocar o embrião em foco.
- Neste ponto, a orientação do embrião dentro do córion será claramente visível e, se não for satisfatória, insira um novo tubo e escolha o embrião com uma orientação de interesse.
- Monte o porta-amostras conforme descrito anteriormente21, no entanto, com uma modificação. Como a imagem será realizada através do tubo, insira o tubo com embriões diretamente em um capilar de diâmetro correto, de modo que seja mantido no lugar sem empurrar a agarose para fora e perturbar os embriões na parte inferior (Figura 1H).
- Alinhando as folhas de luz
- Quando o embrião estiver em posição, configure todas as configurações experimentais - lasers, caminho de luz, filtro, divisor de feixe e câmera.
- Ao configurar os parâmetros de aquisição, se o software fornecer uma opção para varredura dinâmica, selecione-a. Uma varredura de pivô reduzirá o efeito de sombreamento que emerge da folha de luz à medida que encontra quaisquer estruturas ou regiões opacas na amostra.
- Inicie a varredura ao vivo da amostra. Configure o tamanho do bit, a potência do laser e o zoom desejado, que determinarão a espessura da folha de luz e, portanto, a resolução axial.
- Para alinhar as folhas de luz, primeiro mude para a iluminação de um lado e altere as configurações das duas folhas de luz (esquerda e direita) sequencialmente. Amplie digitalmente uma região da amostra onde estruturas claras de interesse são visíveis.
NOTA: Neste estudo, um marcador nuclear (H2A-mCherry) ou um marcador de membrana (Utr-GFP) foi usado para alinhar as folhas. Recomenda-se alterar as configurações da folha de luz no software para que o contraste de sinal mais nítido seja obtido na região de interesse. - Uma vez que as duas folhas de luz são otimizadas individualmente para obter o melhor sinal, ligue ambas as folhas de luz. Execute uma segunda rodada de alinhamento e certifique-se de que a cintilação seja mínima, o que indica um alinhamento bastante bom das duas folhas.
- Feito o alinhamento, ative a opção de fundir automaticamente as imagens geradas pelas duas folhas, se permitido pelo software. Alternativamente, pode-se primeiro alinhar grosseiramente as folhas de luz, concentrando-se nas contas ao redor da amostra e, em seguida, ajustar o alinhamento usando as informações da amostra.
NOTA: Durante a aquisição de imagens com dois fluoróforos (como marcadores nucleares e de membrana), o alinhamento ideal geralmente é marginalmente diferente para as duas estruturas. Nesse cenário, escolha uma configuração com base nas restrições do processamento downstream. Por exemplo, se os limites das células forem segmentados, o que é relativamente mais intensivo em processamento, alinhe as folhas de luz com base no marcador de membrana.
- Configurando a imagem multiview
- Depois de alinhar as folhas de luz, ative as opções para executar uma pilha z, bem como imagens multivisualização.
- Inicie a varredura ao vivo e navegue usando o navegador de amostras para selecionar a primeira visualização. Nessa visualização, configure o intervalo de fatia, seguido pela primeira e última fatias da pilha z. Adicione essa posição na caixa de diálogo de múltiplas visualizações, que anota as informações de posição e pilha z para essa visualização.
- Mova para a próxima vista alterando o ângulo no navegador de amostras. Configure a pilha z para a segunda visualização e adicione as informações na caixa de diálogo de visualização múltipla.
- Repita o mesmo para visualizações adicionais.
NOTA: Certifique-se de que um determinado número mínimo de fatias seja visualizado em cada exibição para que haja sobreposição suficiente entre as diferentes exibições, o que eventualmente ajudará a registrar as exibições durante o processamento. O número mínimo de fatias dependerá do fator de zoom, bem como do número de ângulos escolhidos para a imagem. - Depois de definir todas as visualizações, salve essas informações em um arquivo de texto (chamado 'posições embrionárias', por exemplo), que conterá informações da pilha z, coordenadas (x, y, z) do tubo, bem como especificações do ângulo para cada visualização. Depois de salvar, limpe todas as posições da caixa de diálogo multivisualização.
- Como a imagem é feita com um córion intacto, que é relativamente grande, não é possível visualizar as contas no tubo com as mesmas configurações. Portanto, as informações do cordão devem ser adquiridas de uma posição diferente no tubo. Para fazer isso, traduza para uma coordenada 'y' diferente do embrião onde as contas são visíveis. Adicione esta posição à caixa de diálogo multivisualização e guarde esta posição num ficheiro de texto (denominado 'beads-y', por exemplo).
NOTA: Visualize as contas o mais próximo possível da amostra de embrião para minimizar os efeitos de possíveis diferenças na curvatura do tubo nas duas posições. Portanto, se vários embriões forem montados no tubo, é importante deixar um pouco de agarose entre os tubos para obter a imagem das contas (conforme mencionado na etapa 1.3.7). - Vá para a pasta onde os arquivos de texto são salvos e duplique o arquivo 'embryo-positions' para um novo arquivo chamado 'beads-positions'. Substitua a coordenada y para todas as vistas no arquivo 'beads-positions' pela coordenada y do arquivo 'beads-y'. Isso garantirá que as contas sejam visualizadas com o mesmo número de visualizações, pilhas z e coordenadas x, mas em uma posição y diferente no tubo.
- Retorne ao software de imagem e carregue o arquivo 'bead-positions' no software. Se a opção de série temporal estiver ativada, selecione um ciclo e inicie o experimento. Salve as imagens de contas como 'contas', que serão usadas para registrar as diferentes visualizações durante o processamento da imagem.
- Depois de tirar as imagens do grânulo, limpe as posições e carregue o arquivo inicial de 'posições embrionárias' no software. Defina o número desejável de ciclos com um intervalo de tempo adequado e inicie o experimento.
3. Análise de imagem multivisualização
NOTA: Para fundir as imagens multiview, um plugin FIJI, BigStitcher, que é a versão mais recente do plugin Multiview Reconstruction, é utilizado 10,17,18. O plug-in pode ser instalado adicionando o plug-in BigStitcher na função 'Gerenciar sites de atualização', que pode ser acessada na opção 'Atualizar' no menu 'Ajuda'. Uma vez instalado, o plug-in aparecerá no menu 'Plugins'. As etapas gerais envolvidas na fusão são as seguintes: (1) Defina pares de arquivos .xml/.h5 para os grânulos e os embriões; (2) Registre todas as vistas com o arquivo de contas; (3) Função de propagação do ponto de extração (PSF) para os grânulos, que pode ser usada para deconvolução (Figura 2A); (4) Transfira as informações de registro e PSF do arquivo de contas para o arquivo de embrião e inicie a deconvolução multiview. A maioria dessas etapas foi descrita anteriormente em detalhes21 e, aqui, as etapas que são processadas de forma diferente são descritas.
- Definindo um conjunto de dados .xml
- Defina um novo conjunto de dados para as contas e o embrião como 'beads.xml' e 'embryo.xml', respectivamente, conforme descrito anteriormente21.
- Detecção e registro usando pontos de interesse
- Após a exportação bem-sucedida do arquivo beads.xml, selecione as visualizações necessárias para o registro na caixa de diálogo 'Multiview Explorer' (Figura 2B). Clique com o botão direito do mouse e escolha Detectar pontos de interesse. Prossiga com as etapas conforme descrito21.
- Depois de detectar pontos de interesse, selecione todas as visualizações, clique com o botão direito do mouse e escolha Registrar usando pontos de interesse. Siga o protocolo conforme descrito21.
- Verifique cuidadosamente se o registro do cordão funcionou com sucesso. Após o registro bem-sucedido, contas sobrepostas de diferentes visualizações são sobrepostas (Figura 2C). Para verificar a precisão com que o registro funcionou, selecione duas visualizações consecutivas, vá para a região sobreposta e alterne entre as duas visualizações para observar se as contas fotografadas de diferentes ângulos estão sobrepostas. Repita isso para cada exibição consecutiva.
- Se a sobreposição não for precisa, tente novamente o registro relaxando o significado necessário para o registro e/ou o erro aceitável de sobreposição (denominado 'Erro RANSAC' na caixa de diálogo 'Registro').
- Após o registro bem-sucedido, clique em Salvar na janela 'Multiview Explorer' para salvar o arquivo .xml atualizado.
NOTA: Salve o arquivo de log também, pois ele contém informações mais detalhadas sobre a eficiência do registro. Para traduzir as informações de registro das contas para o embrião, siga as etapas descritas abaixo. - Abra o arquivo bead.xml em um editor de texto de sua escolha e copie o bloco inteiro em "ViewRegistrations".
- Abra o embryo.xml e substitua o bloco "ViewRegistrations" pelo bloco copiado do arquivo beads. Se houver vários canais, substitua as informações de registro de cada canal conforme acima. As informações de registro podem ser transferidas manualmente ou usando o código MATLAB personalizado que pode ser baixado aqui: https://github.com/sundar07/Multiview_analysis
- Abra o arquivo embryo.xml no "BigStitcher" e verifique cuidadosamente se o registro funcionou com sucesso para o embrião. Repita o mesmo que foi feito para as contas, verificando a sobreposição de estruturas de interesse a cada duas vistas consecutivas.
NOTA: Ocasionalmente, o registro de embriões pode não ser tão desejável, apesar de as contas serem registradas perfeitamente. Isso é possível se houver pequenas mudanças na curvatura do tubo da posição em que as contas são fotografadas para a posição do embrião. Além disso, pode haver movimentos sutis do embrião entre as vistas, pois ele está flutuando livremente dentro do córion. Nesse caso, faça uma segunda rodada de registro usando um marcador nuclear no embrião. - Para fazer isso, abra o arquivo embryo.xml em "BigStitcher", clique com o botão direito do mouse em todas as visualizações que possuem o marcador nuclear e escolha Detectar pontos de interesse.
- Renomeie os pontos de interesse como 'núcleos' e prossiga com as etapas executadas para contas.
- Ao definir os parâmetros de 'Diferença de Gaussiano', certifique-se de que a maioria, se não todos, os núcleos sejam detectados e que não haja detecção nuclear ectópica. Em seguida, clique em Concluído.
- Em seguida, registre essas visualizações escolhendo Registrar usando pontos de interesse e opte pela opção Baseado em descritor preciso (invariante de tradução). Certifique-se de que a opção 'comparar todas as visualizações e pontos de interesse' esteja selecionada e use 'núcleos' como pontos de interesse. Use a opção para corrigir a primeira visualização e não mapeie de volta.
- Para o registro, use um modelo afim com regularização rígida e os parâmetros padrão no plugin.
- Verifique novamente o sucesso do registro comparando a cada duas visualizações consecutivas.
- Após o registro bem-sucedido, clique em Salvar na janela 'Multiview Explorer' para salvar o arquivo .xml atualizado.
- Abra o arquivo embryo.xml em um editor de texto de sua escolha e copie as informações de registro dos marcadores nucleares para os outros canais.
- Extração e atribuição de função de propagação de pontos
- Para realizar a deconvolução multiview, extraia o PSF do sistema de imagem do conjunto de dados de esferas registradas e aplique-o ao arquivo de embrião.
- Para obter essas informações, selecione todas as vistas no arquivo de cordão e, em seguida, clique com o botão direito do mouse e escolha as opções Funções de difusão de ponto e Extrair .
- Na caixa de diálogo exibida, certifique-se de que as projeções Usar pontos de interesse correspondentes e Remover intensidade mínima do PSF estejam marcadas, prossiga com os tamanhos padrão do PSF e clique em OK.
NOTA: Se a extração de PSF for bem-sucedida para todas as visualizações, o arquivo de log exibirá 'PSFs n/n extraídos' - Depois disso, salve novamente o arquivo .xml. Uma caixa de seleção marcada na coluna PSF da caixa de diálogo 'Multiview Explorer' aparecerá e uma pasta 'psf' será gerada na respectiva pasta com todos os PSFs extraídos.
- Abra o arquivo embryo.xml e atribua o PSF para cada exibição separadamente. Clique com o botão direito do mouse em "uma exibição" → clique em Funções de propagação de pontos → escolha Atribuir → selecione Avançado, seguido por Atribuir novo PSF a todas as exibições selecionadas. Clique em Procurar, vá para o caminho do arquivo .xml e abra a pasta psf .
- Escolha o PSF correspondente com o ID correspondente na visualização selecionada e clique em OK, após o que a caixa de seleção PSF aparecerá marcada na janela 'Multiview Explorer'.
- Repita o processo para todas as outras exibições.
- Fusão e deconvolução multivisualização
- Depois que a função de difusão de pontos tiver sido atribuída a todas as vistas, clique com o botão direito do mouse e escolha Deconvolução de múltiplas vistas.
- Selecione a caixa delimitadora como vistas selecionadas no momento. Para este trabalho, a aceleração padrão do OSEM e o número de iterações funcionam bem.
- Reduza a resolução das imagens conforme necessário se a computação mais rápida for desejada ou se a memória da CPU for limitante.
NOTA: Se a RAM necessária exceder a memória existente, uma mensagem de erro de aviso na cor vermelha aparecerá na parte inferior da janela. Não inicie a deconvolução se este aviso aparecer, pois o plug-in irá parar e parar de responder em algum momento durante o processamento. - Para avaliar o progresso da deconvolução, verifique o arquivo de log, que exibirá os resultados a cada 5 iterações.
- Para aumentar a velocidade computacional, execute a deconvolução de visualização múltipla na GPU, se instalada anteriormente.
NOTA: No final deste processo, uma janela de imagem fundida aparecerá, que pode ser salva como um arquivo tiff.
Resultados
Orientar a amostra de maneira precisa é uma parte vital do uso eficiente de uma configuração de microscopia. No entanto, a orientação manual das amostras muitas vezes não é possível ao usar um sistema de folha de luz multiview, dada a necessidade de preparar as amostras em um tubo. Portanto, para verificar se existem posições estereotipadas que os embriões assumem dentro do córion, os embriões de peixe-zebra foram fotografados em 70% de epiboly (cerca de 7 h pós-fertilização (hpf)), uma vez que a imagem de lapso de tempo da gastrulação aos estágios iniciais de somitos foi o foco deste estudo. Quando as amostras foram preparadas imediatamente antes da imagem a 70% de epibolia, os embriões não mostraram orientação específica que é frequentemente observada nas amostras. Como isso geralmente não é desejável, as amostras foram preparadas bem antes da gastrulação e armazenadas em tubos de microcentrífuga na temperatura apropriada até o início da imagem. Nessas condições, a 70% de epibólia (N = 3; n = 87 embriões), as orientações embrionárias podem ser classificadas em (1) Horizontal, quando o eixo animal-vegetal (AV) do embrião era ortogonal ao eixo longo do tubo polimérico, (2) Vertical, quando o eixo AV era paralelo ao eixo longo do tubo polimérico, e, (3) Oblíquo, quando o eixo AV estava em ângulo agudo (Figura 3A). A posição horizontal foi a menos representada, enquanto as posições vertical e oblíqua foram igualmente observadas (Figura 4).
Quando os embriões foram deixados nas trompas, os embriões ficaram estáveis nessas respectivas posições até 90% epiboly, após o que a maioria dos embriões mudou de orientação. Portanto, uma segunda rodada de documentação de orientações no estágio de brotamento da embriogênese (cerca de 10 hpf) foi necessária para levar em conta as orientações alteradas. Isso foi realizado para amostras preparadas independentemente. Para a imagem dos estágios iniciais do somito, foi relatado anteriormente que um embrião com sua notocorda ortogonal ao eixo longo do tubo de polímero era a orientação ideal, pois permite a visualização de múltiplos somitos bilaterais se formando ao longo do eixo15. Quando as amostras foram preparadas antes da gastrulação (N = 3, n = 93 embriões), cerca de 25% dos embriões exibiram essa orientação (Figura 4) e esses embriões permaneceram estáveis nessa orientação até pelo menos o estágio de 8-somito, consistente com relatos anteriores15. O restante dos embriões exibiu várias outras orientações no estágio de brotamento (classificados na Figura 3B e Figura 4); no entanto, muitos deles foram reorientados para a posição horizontal durante a formação inicial do somito. Curiosamente, uma porcentagem semelhante de embriões apresentou uma orientação horizontal no estágio de gema, independentemente de a amostra ter sido preparada antes da gastrulação, a 70% de epibolia ou imediatamente antes do estágio de gema. Assim, o tempo de preparação da amostra parece menos crítico para a orientação horizontal de interesse para os estágios de imagem do somito, ao contrário do que foi observado para a imagem em 70% de epibólia.
A vantagem de um sistema multiview é a capacidade de visualizar a mesma amostra de vários ângulos. No entanto, para embriões de peixe-zebra nos estágios mencionados, o número de visualizações necessárias para obter a resolução celular em todo o embrião não é claro. Para caracterizar isso, os embriões de peixe-zebra foram fotografados usando uma objetiva de 20x/1 NA no braço de detecção com um fator de zoom de 1, que correspondeu a uma espessura de folha leve de 4,57 μm. Sob essas configurações, o embrião cobriu todo o campo de visão de uma câmera sCMOS com um tamanho de pixel de 6,5 μm e uma área de 1920 por 1920 pixels. A microinjeção de mRNA para um fluoróforo marcado com histonas (H2A-mCherry) em embriões de estágio de 1 célula obtidos de uma linhagem transgênica que marcou filamentos de actina (Utr-GFP) permitiu a visualização de núcleos e membranas celulares no embrião. Para realizar a fusão multiview de embriões com diferentes intervalos angulares, a aquisição de 360° dos embriões transgênicos duplos, bem como dos grânulos no tubo, foi realizada a cada 30° (n = 3 embriões a 70% de epibolia; n = 3 embriões em estágio de gema) ou 45° (n = 3 embriões em 70% de epíbulo; n = 3 embriões em 70% de epíbulo; n = 3 embriões em fase de gema) intervalos com cerca de 100 cortes em cada ângulo e um intervalo de corte de 2 μm. Ao pular ângulos alternados durante o processamento da imagem, as aquisições de 30° e 45° forneceram adicionalmente os conjuntos de dados de 60° e 90°.
As imagens adquiridas foram então registradas usando as informações do grânulo e os detalhes do registro foram transferidos para o conjunto de dados do embrião, conforme descrito na seção do protocolo. O registro bem-sucedido usando o plug-in BigStitcher foi alcançado para as diferentes aquisições, exceto para o conjunto de dados de 90°, o que provavelmente se deve à menor cobertura da amostra de cada ângulo. Para superar isso, os embriões foram fotografados a cada 90 ° com cerca de 400 fatias em cada ângulo e um intervalo de fatia de 2 μm, nos estágios de 70% de epibólia e gema, que foram registrados com sucesso (n = 3 embriões para cada estágio).
O próximo passo foi realizar a fusão multiview e a deconvolução dos conjuntos de dados registrados. Isso foi feito com redução de 4x para acelerar a computação. Como visto na representação de núcleos a partir de vistas individuais e um embrião reconstruído com múltiplas visualizações (Figura 5), as vistas individuais cobrem um campo de visão menor, que, após a fusão, produziu uma imagem de todo o embrião. Para a representação, os núcleos foram detectados usando o Mastodon (https://github.com/mastodon-sc/mastodon), um plugin baseado em FIJI, que pode ser adicionado na seção "Gerenciar sites de atualização" do menu "Ajuda" em FIJI e acessado no menu de plugins uma vez adicionado. Para a detecção, as respectivas imagens foram primeiro convertidas para o formato XML / hdf5 e, em seguida, a detecção nuclear foi realizada usando o plug-in 'Detecção' do Mastodon com um detector DoG (Diâmetro 6 μm e Limite de Qualidade 80).
Entre as diferentes imagens fundidas, o conjunto de dados de 90° mostrou um fundo muito alto mais profundo na amostra, tornando-o inadequado para realizar qualquer quantificação. Assim, ao contrário de amostras menores, como embriões de Drosophila, que frequentemente foram fotografados com um intervalo de 90° em outros estudos 9,22, o mesmo não é recomendado para imagens de embriões de peixe-zebra iniciais usando uma objetiva de 20x/1 NA. Entre os conjuntos de dados fundidos de 30°, 45° e 60°, qualitativamente, não houve diferença substancial nas informações dos núcleos (Figura 6B, linha superior); no entanto, estruturas mais finas, como limites de células, pareciam muito melhor resolvidas com o conjunto de dados fundidos de 30 ° em comparação com o restante (Figura 6C, linha superior).
Para confirmar essa observação, o Mastodon foi usado para detectar núcleos nas imagens fundidas obtidas a partir de imagens a cada 30°, 45° e 60°. Três regiões nas imagens fundidas, uma a uma profundidade de 20 μm (Figura 6A), 50 μm e 100 μm da superfície do embrião, foram escolhidas para a análise. Para comparar a eficiência da detecção de núcleos entre imagens, a detecção foi realizada conforme descrito acima, com parâmetros idênticos em imagens fundidas de diferentes intervalos angulares. Em todas as regiões analisadas, todos os núcleos foram detectados, independentemente das imagens fundidas obtidas a cada 30°, 45° ou 60° (Figura 6B, linha inferior). Assim, estruturas globulares, como núcleos, podem ser visualizadas em qualquer um dos intervalos angulares acima sem perda de informação.
Para analisar os limites celulares, foi utilizado o Tissue Analyzer23, um plugin FIJI usado rotineiramente para segmentar células 24,25,26. Semelhante ao Mastodon, o plug-in Tissue Analyzer pode ser adicionado na seção "Gerenciar sites de atualização" do menu "Ajuda" em FIJI e acessado no menu de plug-ins uma vez adicionado. Os limites das células foram segmentados usando o algoritmo de bacia hidrográfica com parâmetros padrão e um forte desfoque variando de 1,5 a 2, dependendo da profundidade do tecido, e um desfoque fraco de 1. Esses parâmetros foram mantidos constantes em todas as análises, facilitando uma comparação direta. Quando as imagens segmentadas foram comparadas manualmente com as imagens de entrada originais, foram observados erros, onde o software falhou em detectar um limite de célula ou desenhou limites de célula inexistentes (Figura 6C, linha inferior). O número de erros cometidos pelo plug-in do analisador de tecidos foi normalizado para o número total de ligações detectadas na região e calculado como 'Erro de segmentação de limite'. Embora esses erros estivessem presentes em todas as regiões analisadas, o número de erros aumentou drasticamente nas imagens fundidas obtidas a partir de imagens em intervalos angulares de 45° e 60°, em comparação com 30° (Figura 6D). Isso indicou que a resolução nas imagens fundidas era cada vez pior quando o intervalo angular era aumentado. Assim, para segmentar estruturas mais finas, como limites de células, um intervalo angular mais estreito permite um processamento mais fácil a jusante.
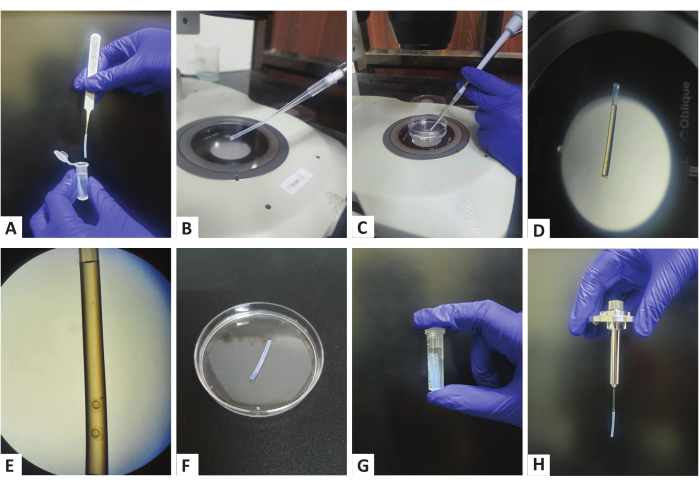
Figura 1: Preparo de amostras usando tubos de polímero. (A) Um tubo de polímero dos tubos de microcentrífuga armazenados é retirado usando uma pinça. (B) Conectando o tubo à ponta de uma micropipeta de 200 μL. (C) Aspiração de embriões com a ajuda de uma pipeta para dentro do tubo. (D, E) Um tubo de polímero com embriões em estágio de botão visíveis na parte inferior do tubo. (F) Os tubos de polímero são colocados em uma placa de Petri com E3 para solidificar a agarose. (G) Armazenamento dos tubos de polímero em um tubo de microcentrífuga preenchido com E3. (H) Suporte de amostra montado com o tubo de polímero montado. Clique aqui para ver uma versão maior desta figura.

Figura 2: Fluxo de trabalho de análise de imagem multivisualização e registro usando contas. (A) O pipeline de análise de imagem multivisualização. (B) A imagem mostra a janela "Multiview Explorer" no BigStitcher, um plugin FIJI, onde cada visualização aparece como uma única linha e contém informações sobre o ângulo, canal, registro, pontos de interesse e PSF. Todos os comandos discutidos no protocolo aparecem quando uma visualização é selecionada e seguida por um clique com o botão direito, conforme ilustrado no menu pop-up. As visualizações selecionadas podem ser visualizadas na janela BigDataViewer, como mostrado. (C) Uma imagem representativa do grânulo antes (esquerda) e depois do registro (direita) obtida a partir de imagens em intervalos angulares de 30 °. Todas as vistas angulares foram selecionadas e exibidas. Barras de escala: 75 μm. Clique aqui para ver uma versão maior desta figura.

Figura 3: Visão geral das orientações padrão do embrião. Imagens representativas das orientações em que os embriões se enquadram no córion em 70% dos estágios de epibólia (A) e botão (B). A linha superior em cada painel mostra imagens de campo claro obtidas do sistema de folha de luz, e a linha inferior mostra desenhos animados representativos. As setas em (B) indicam a posição da notocorda, que foi usada para identificar a orientação. Ap, poste animal; Vp, pólo vegetal; A, anterior; P, posterior. Barras de escala: 100 μm. Clique aqui para ver uma versão ampliada desta figura.

Figura 4: Estatísticas de diferentes orientações embrionárias. (A) A coluna empilhada indica as porcentagens de embriões que se enquadram nas orientações indicadas em 70% epiboly quando as amostras são preparadas antes da gastrulação. (B) As colunas empilhadas indicam as porcentagens de embriões que se enquadram nas orientações indicadas nos estágios de gema, quando as amostras são preparadas antes da gastrulação (esquerda), em 70% de epiboly (meio) e nos estágios de gema (direita). N, o número de ninhadas independentes das quais os embriões foram obtidos; n, o número total de embriões. Clique aqui para ver uma versão maior desta figura.

Figura 5: Representação de núcleos de um embrião reconstruído multiview. (UMA)O gráfico de dispersão 3D representa núcleos detectados em um embrião reconstruído com múltiplas visualizações fotografado em um intervalo angular de 30̛°. Cada círculo representa um núcleo e o centróide das posições dos núcleos é plotado. As coordenadas nucleares foram obtidas usando o Mastodon, um plugin FIJI. (B) Gráficos de dispersão 3D de núcleos de três vistas representativas do mesmo embrião - as representações estão separadas por 60 °. As cores para cada núcleo foram atribuídas aleatoriamente. Clique aqui para ver uma versão maior desta figura.

Figura 6: Comparação de informações em escala celular em imagens fundidas. (A) A imagem à esquerda mostra um instantâneo de um embrião reconstruído com múltiplas visualizações a uma profundidade de 20 μm da superfície. O círculo amarelo representa a região usada para análise posterior. Barras de escala: 50 μm (em todos os painéis). (B) Imagens brutas representativas de núcleos a 20 μm de profundidade da superfície do embrião a partir de três conjuntos de dados fotografados a cada 30 °, 45 ° e 60 °, respectivamente (topo). Núcleos detectados (em verde) nas mesmas imagens pelo Mastodon, um plugin FIJI (abaixo). (C) Imagens brutas representativas a 20 μm de profundidade da superfície do embrião a partir de três conjuntos de dados fotografados a cada 30 °, 45 ° e 60 °, respectivamente, usando o marcador de actina, Utr-GFP (topo). Os limites segmentados pelo Tissue Analyzer, um plug-in FIJI, são mostrados para as mesmas imagens (parte inferior). As setas indicam erros cometidos pelo Tissue Analyzer na segmentação de limites, com a seta amarela representando um limite ausente e a seta branca representando limites detectados incorretamente quando visualmente nenhum limite parece existir. (D) O gráfico de caixa mostra a porcentagem de erros que o Tissue Analyzer cometeu entre embriões reconstruídos multiview fotografados a cada 30°, 45° e 60° em diferentes profundidades. As barras de erro indicam 1,5 vezes o intervalo interquartil. Clique aqui para ver uma versão maior desta figura.
Discussão
Posicionar um embrião na orientação correta para obter imagens da região de interesse é uma das etapas de limitação de taxa que geralmente resulta em uma sessão de microscopia com falha para um usuário. Isso é mais verdade em um microscópio de folha de luz multiview, onde a manipulação manual da orientação é difícil, pois as amostras são incorporadas em um tubo. Para auxiliar nesse processo, este estudo relata as estatísticas de várias posições que um embrião de peixe-zebra ocupa entre 70% dos estágios epiboly e inicial de somitos dentro de um córion quando o tubo de polímero com embriões é mantido na posição vertical em um tubo de microcentrífuga.
Com 70% de epíbulo, embriões orientados vertical ou horizontalmente fornecem informações complementares de eventos em todo o embrião. Enquanto a orientação horizontal permite a visualização da dinâmica celular no pólo animal, bem como a dinâmica das células em epíbula vistas do pólo vegetal, a orientação vertical permite a visualização dos lados dorsal e ventral do embrião, incluindo a dinâmica celular no organizador dorsal, internalização das células durante a gastrulação e fluxos de convergência-extensão. Este estudo mostra que há uma alta probabilidade de obter um embrião na orientação vertical puramente por acaso se as amostras forem preparadas algumas horas antes da imagem; no entanto, a orientação horizontal parece ser muito menos prevalente nesses estágios. Para estágios anteriores a 70% de epiboly, recomenda-se que os usuários documentem estatísticas semelhantes de orientação do embrião dentro do córion antes de prosseguir com a imagem de lapso de tempo, pois ter essas informações de antemão economiza tempo a longo prazo.
A partir do estágio de brotamento, a melhor orientação para a imagem é um embrião posicionado horizontalmente, pois isso permite acompanhar a dinâmica celular ao longo de todo o eixo ântero-posterior do corpo dos embriões15. Obter um embrião nessa orientação é relativamente simples, pois cerca de 25% dos embriões caem nessa posição estável, independentemente de quando a amostra é preparada. Em geral, para a obtenção de imagens de qualquer estágio de interesse, recomenda-se a preparação de cerca de 15 tubos com dois a três embriões em cada tubo, o que garantirá uma alta probabilidade de obtenção de embriões na orientação desejada e, portanto, uma sessão de microscopia bem-sucedida em um determinado dia. Além disso, recomenda-se que os tubos sejam classificados cerca de uma hora antes do estágio de interesse para escolher um embrião com a orientação correta para imagens de lapso de tempo.
Depois de obter um embrião na orientação correta, é importante levar em consideração o número de visualizações e o intervalo de ângulo entre as visualizações para imagens. Isso depende da resolução espacial e temporal mínima necessária. Quanto mais ângulos a partir dos quais uma determinada estrutura ou região do embrião é visualizada, melhor é a resolução espacial (como mostrado na Figura 6), mas isso leva a um compromisso na resolução temporal. Neste protocolo, com um sistema que possui dois braços de iluminação e um de detecção, a geração de imagens de dois canais em 360° com um intervalo angular de 30° e cerca de 100 fatias z em cada ângulo com um espaçamento entre fatias de 2 μm, levou cerca de 3 minutos para um ponto único. Embora isso seja suficiente para rastrear células em estágios embrionários iniciais, se eventos com dinâmica mais rápida forem capturados, um número menor de ângulos deve ser adquirido, garantindo melhor resolução temporal, mas com um compromisso na resolução espacial, especialmente se estruturas mais finas, como limites de células, tiverem que ser segmentados. Uma segunda opção a ser considerada para melhorar a resolução temporal é usar um sistema multiview com dois braços de detecção, o que aumentará substancialmente a velocidade de aquisição, como usado recentemente para rastrear os movimentos celulares durante a gastrulação em embriões de peixe-zebra16. Além disso, embora apenas a objetiva 20x/1 NA tenha sido testada neste protocolo, dependendo da amostra que está sendo fotografada, da resolução espacial necessária e do campo de visão, a objetiva correta precisa ser considerada. Em conjunto, com base no processamento e quantificação a jusante necessários para uma amostra e campo de visão específicos, o número de visualizações e o intervalo de ângulo precisam ser cuidadosamente escolhidos. Para imagens de embriões inteiros de peixe-zebra inicial, particularmente com resolução em escala celular de vários ângulos, no entanto, uma objetiva de 20x/1 NA parece ser ideal, pois uma objetiva com menor ampliação e NA (por exemplo, objetiva de 10x/0,5 NA) terá resolução muito menor, provavelmente levando a mais erros de segmentação, enquanto uma objetiva com maior ampliação e NA não será viável para cobrir todo o embrião no campo de visão da câmera.
Neste estudo, para discutir estratégias de montagem e imagem, foram utilizados um marcador de actina, que pode ser usado como proxy para traçar os limites celulares, e um marcador de histona, para detectar e rastrear núcleos. De fato, a maioria dos estudos publicados anteriormente que usaram um sistema de folha de luz multiview usaram preferencialmente marcadores semelhantes 5,7,15,16,27. A principal razão para escolher esses marcadores é que a microscopia de folha de luz multiview é usada principalmente para seguir a dinâmica em escala celular (por exemplo, forma celular e rearranjo celular) em escalas de tecido ou em todo o embrião por longos períodos de tempo que abrangem várias horas a dias. Por outro lado, se houver necessidade de obtenção de resolução subcelular, uma linha transgênica diferente marcando a região de interesse poderia ser usada, mas neste cenário, um microscópio de folha de luz multiview pode não ser a melhor escolha de instrumento, e sim um confocal, um sistema de super-resolução ou mesmo um microscópio de folha de luztreliçada 28, 29 pode se adequar melhor ao problema que está sendo investigado.
O protocolo discutido funciona bem desde a gastrulação até embriões de peixe-zebra em estágio de 15 somitos30 (cerca de 17 hpf) sem nenhuma modificação e, embora não tenha sido testado aqui, é provável que funcione também para embriões anteriores antes do estágio de gastrulação. Além do estágio de 15 somitos, iniciam-se contrações musculares espontâneas30,31, que podem ser suprimidas pela adição de tricaína, um anestésico, no tubo e na câmara de amostra. Além disso, por volta de 18 hpf, a eversão da cauda para longe da gema começa30, o que afasta o embrião do campo de visão. Para cuidar disso, um algoritmo de rastreamento que rastreia a extremidade alongada precisa ser empregado para manter o embrião em foco32. Além disso, para imagens de embriões em estágio avançado quando eles são desprovidos de um córion, como para seguir o desenvolvimento neural, uma estratégia de montagem alternativa precisa ser empregada como realizada recentemente33.
Para obter imagens de embriões de peixe-zebra com um córion intacto, não é possível capturar as contas, que atuam como marcadores de registro, e o embrião no mesmo campo de visão usando uma objetiva de 20x/1 NA. Este estudo fornece uma alternativa simples, registrando as diferentes visualizações com informações de contas que estão presentes acima ou abaixo da amostra, seguido pela mudança do registro das contas para o embrião durante o processamento. Uma vez que a amostra de embrião é registrada dessa forma, uma segunda rodada de registro usando núcleos como marcadores de registro, que estão presentes dentro da amostra, para ajustar ainda mais o registro inicial. Ao realizar imagens de lapso de tempo, as informações dos núcleos, por sua vez, podem ser usadas para registro de pontos de tempo consecutivos, como foi relatado anteriormente21. Uma alternativa a este protocolo é renunciar aos beads e, em vez disso, usar as informações dos núcleos para o registro inicial. Mas o registro muitas vezes falhou (não mostrado aqui) no plug-in BigStitcher, possivelmente devido a uma combinação de muitos núcleos que não são detectados mais profundamente no embrião durante o processamento e também por causa de um número relativamente menor de núcleos que estão presentes no embrião quando vistos de certos ângulos (por exemplo, quando vistos ventralmente em estágios epibólicos tardios).
Neste protocolo, como as informações do grânulo de cima ou de baixo da amostra são usadas para registro e não há necessidade de imagens de grânulos na mesma visão que a imagem da amostra, pode-se usar este protocolo para obter imagens apenas de regiões específicas de interesse, mas em alta ampliação de diferentes ângulos em vez de todo o embrião. Além disso, é fácil adaptar este protocolo para imagens de várias amostras empilhadas verticalmente no tubo usando informações de esferas entre as amostras para registro.
Divulgações
Os autores declaram não haver interesse conflitante.
Agradecimentos
Agradecemos ao Dr. Kalidas Kohale e sua equipe pela manutenção das instalações de peixes e à KV Boby pela manutenção do microscópio de folha de luz. A SRN reconhece o apoio financeiro do Departamento de Energia Atômica (DAE), Governo da Índia (Identificação do Projeto nº. RTI4003, DAE OM nº 1303/2/2019/R\&D-II/DAE/2079 de 11.02.2020), o programa Max Planck Society Partner Group (M.PG. A MOZG0010) e a Bolsa de Pesquisa Inicial do Conselho de Pesquisa em Ciência e Engenharia (SRG/2023/001716).
Materiais
| Name | Company | Catalog Number | Comments |
| Agarose, low gelling temperature | Sigma-Aldrich | A9414 | |
| Calcium Chloride dihydrate | Sigma-Aldrich | 12022 | |
| FIJI | Version: ImageJ 1.54f | ||
| Latex beads, carboxylate-modified polystyrene, fluorescent red, 0.5 μm mean particle size, aqueous suspension | Sigma-Aldrich | L3280 | |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | |
| mMESSAGE mMACHINE SP6 Transcription kit | ThermoFischer Scientific | AM1340 | For in vitro transccription of H2A-mCherry plasmid |
| Potassium Chhloride | Sigma-Aldrich | P9541 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P0662 | |
| PTFE Sleeving AWG 15L - 1.58 mm ID x 0.15 mm Wall +/-0.05 | Adtech Innovations in Fluoroplastics | STW15 | PTFE tubes |
| Sodium Chloride | Sigma-Aldrich | S3014 | |
| Sodium phosphate dibasic | Sigma-Aldrich | 71640 | |
| Ultrasonic Cleaner | Labman | LMUC3 | Ultrasonicator |
| Zeiss LightSheet 7 System | Zeiss |
Referências
- Wan, Y., McDole, K., Keller, P. J. Light sheet microscopy and its potential for understanding developmental processes. Annu Rev Cell Dev Biol. 35, 655-681 (2019).
- Keller, P. J., Schmidt, A. D., Wittbrodt, J., Stelzer, E. H. Reconstruction of zebrafish early embryonic development by scanned light sheet microscopy. Science. 322 (5904), 1065-1069 (2008).
- Keller, P. J., et al. high-contrast imaging of animal development with scanned light sheet-based structured-illumination microscopy. Nat Methods. 7 (8), 637-642 (2010).
- Keller, P. J. Imaging morphogenesis: Technological advances and biological insights. Science. 340 (6137), 1234168(2013).
- Schmid, B., et al. High-speed panoramic Light sheet microscopy reveals global endodermal cell dynamics. Nat Commun. 4 (1), 2207(2013).
- Strnad, P., et al. Inverted Light sheet microscope for imaging mouse pre-implantation development. Nat Methods. 13 (2), 139-142 (2016).
- McDole, K., et al. In toto imaging and reconstruction of post-implantation mouse development at the single-cell level. Cell. 175 (3), 859-876 (2018).
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Krzic, U., Gunther, S., Saunders, T. E., Streichan, S. J., Hufnagel, L. Multiview Light sheet microscope for rapid in toto imaging. Nat Methods. 9 (7), 730-733 (2012).
- Preibisch, S., Saalfeld, S., Schindelin, J., Tomancak, P. Software for bead-based registration of selective plane illumination microscopy data. Nat Methods. 7 (6), 418-419 (2010).
- Swoger, J., Verveer, P., Greger, K., Huisken, J., Stelzer, E. H. K. Multiview image fusion improves resolution in three-dimensional microscopy. Opt Express. 15 (13), 8029-8042 (2007).
- Swoger, J., Huisken, J., Stelzer, E. H. Multiple imaging axis microscopy improves resolution for thick-sample applications. Opt Lett. 28 (18), 1654-1656 (2003).
- Kaufmann, A., Mickoleit, M., Weber, M., Huisken, J. Multilayer mounting enables long-term imaging of zebrafish development in a light sheet microscope. Development. 139 (17), 3242-3247 (2012).
- Weber, M., Mickoleit, M., Huisken, J. Multilayer mounting for long-term light sheet microscopy of zebrafish. J Vis Exp. (84), e51119(2014).
- Naganathan, S. R., Popović, M., Oates, A. C. Left-right symmetry of zebrafish embryos requires somite surface tension. Nature. 605 (7910), 516-521 (2022).
- Shah, G., et al. Multi-scale imaging and analysis identify pan-embryo cell dynamics of germlayer formation in zebrafish. Nat Commun. 10 (1), 5753(2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Hörl, D., et al. BigStitcher: Reconstructing high-resolution image datasets of cleared and expanded samples. Nat Methods. 16 (9), 870-874 (2019).
- Behrndt, M., et al. Forces driving epithelial spreading in zebrafish gastrulation. Science. 338 (6104), 257-260 (2012).
- Westerfield, M. The Zebrafish Book: A Guide for the Laboratory Use of Zebrafish (Danio Rerio). , University of Oregon Press. (2000).
- Icha, J., et al. Using light sheet fluorescence microscopy to image zebrafish eye development. J Vis Exp. (110), e53966(2016).
- Tomer, R., Khairy, K., Amat, F., Keller, P. J. Quantitative high-speed imaging of entire developing embryos with simultaneous multiview Light sheet microscopy. Nat Methods. 9 (7), 755-763 (2012).
- Aigouy, B., Prud'homme, B. Segmentation and quantitative analysis of epithelial tissues. Methods Mol Biol. 2540, 387-399 (2022).
- Mancini, L., et al. Apical size and deltaA expression predict adult neural stem cell decisions along lineage progression. Science Adv. 9, 7519(2023).
- Piscitello-Gomez, R., Mahmoud, A., Dye, N., Eaton, S. Sensitivity of the timing of Drosophila pupal wing morphogenesis to external perturbations. bioRxiv. , (2023).
- Tsuboi, A., Fujimoto, K., Kondo, T. Spatiotemporal remodeling of extracellular matrix orients epithelial sheet folding. Science Adv. 9, 2154(2023).
- Fu, Q., Martin, B. L., Matus, D. Q., Gao, L. Imaging multicellular specimens with real-time optimized tiling Light sheet selective plane illumination microscopy. Nat Commun. 7 (1), 11088(2016).
- Chen, B. C., et al. Lattice Light sheet microscopy: imaging molecules to embryos at high spatiotemporal resolution. Science. 346 (6208), 1257998(2014).
- York, H. M., et al. Deterministic early endosomal maturations emerge from a stochastic trigger-and-convert mechanism. Nat Commun. 14 (1), 4652(2023).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Melançon, E., Liu, D. W. C., Westerfield, M., Eisen, J. S. Pathfinding by identified zebrafish motoneurons in the absence of muscle pioneers. J Neurosci. 17 (20), 7796(1997).
- Rohde, L. A., et al. Cell-autonomous timing drives the vertebrate segmentation clock's wave pattern. eLife. , 13:RP93764 (2024).
- Haynes, E. M., et al. KLC4 shapes axon arbors during development and mediates adult behavior. eLife. 11, e74270(2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados