Method Article
RNA 'nın ayak Izleri tanımlaması: proteinli kompleksleri RNA Immunoprecipitation ile tandem sonra sıralamayı (RIPiT-seq)
Bu Makalede
Özet
Burada, endojen RNA bağlayıcı siteleri veya RNA: protein (RNP) kompleksler "ayak izleri" mammalin hücrelerden zenginleştirmek için bir protokol sunuyoruz. Bu yaklaşım RNP alt paketleri iki immünoprecipitations içerir ve bu nedenle tandem RNA immünopresipitasyon dubbed (ripit).
Özet
Tandem içinde RNA immünopyemek (RIPiT) RNA: protein (RNP) kompleksinde bir çift proteinin RNA ayak izlerini zenginleştirmeye yarayan bir yöntemdir. RipIt iki arıtma adımlarını kullanır. İlk olarak, etiketli bir RNP altbirim immünoprecipitation hafif RNase sindirim ve sonraki Non-denaturing benzeşme elüsyon tarafından izlenir. Başka bir RNP altbirim ikinci bir immünoprecipitation tanımlanmış bir kompleks zenginleştirme sağlar. Rnas ve proteinlerin denatüre bir elüsyonu takiben, RNA ayak izleri yüksek verim DNA sıralı kütüphaneler dönüştürülür. Daha popüler ultraviyole aksine (UV) çapraz immünoprecipitation takip (CLIP) yaklaşım RBP bağlama siteleri zenginleştirin, ripit UV-çapraz bağımsız. Bu nedenle RIPiT RNA interactomunda mevcut olan çok sayıda proteinlere uygulanabilir ve bunun ötesinde RNA Yönetmeliği için önemlidir, ancak RNA veya UV-Crosslink ' e doğrudan temas etmez. RipIt 'deki iki arıtma adımları, başka bir cofactor ile ortaklık içinde ilgi gören bir protein olan bağlayıcı siteleri tanımlamanın ek bir avantajı sağlar. Çift arıtma stratejisi de arka plan sınırlandırarak sinyali artırmak için hizmet vermektedir. Burada, RIPiT 'i gerçekleştirmek ve yalıtılmış RNA ayak izlerinden yüksek verimlilik sıralaması kitaplıkları oluşturmak için adım akıllı bir prosedür sağlıyoruz. Ayrıca, RIPiT 'in avantajları ve uygulamalarını özetlemektedir ve bazı sınırlamalarını tartışırız.
Giriş
Hücreler içinde RNA, RNA: protein kompleksler (RNPs) oluşturmak için proteinler ile kompleks içinde bulunmaktadır. RNPs RNA bağlayıcı proteinleri (RBPs, doğrudan RNA bağlamak olanlar) etrafında monte edilir ama aynı zamanda olmayan RBPs (Bu bind RBPs ama RNA değil) oluşur ve genellikle doğada dinamik. Rbps ve onların Kofaktörler düzenleyici işlevleri yürütmek için rnps içinde topluca işlev. Örneğin, anlamsız aracılı mRNA çürüğü (NMD) yolu, UPF proteinleri (UPF1, UPF2, ve UPF3b) zamanından önce sonlandırılmış ribosome tanır. UPF proteinlerinin her biri RNA 'ya bağlanabilirler, ancak sadece aktif bir NMD kompleksinin formaya başladığı zaman birlikte birleştiriyorlar. Bu kompleks içinde, UPF1 daha fazla olmayan bir RBP SMG1 tarafından fosforilasyon tarafından aktive edilir, ve böyle UPF1 aktivasyon sonunda mRNA çürüme İndükleme faktörleri1,2işe yol açar. Bu örnekte, rbps işe alma ve NMD tetikleyen RNP kompleks aktivasyonu için RBP Kofaktörler gerektirir. Ancak RNPs 'nin başka bir özelliği de kompozisyonlu heterojenliği. Farklı E, A, B veya C kompleksler içinde bulunan spliceosome düşünün. Farklı spliceosome kompleksleri çakışan ve farklı proteinlere sahiptir3. RNP fonksiyonlarını incelemek için, Rnas 'ın bir RBP ve ilişkili proteinlerle bağlı olduğu aydınlatmak önemlidir. Farklı avantajları ve dezavantajları4,5,6,7olan her yaklaşımla, bunu başarmak için birçok yöntem var.
RBP bağlayıcı siteleri tanımlamak için yaygın popüler Yöntemler-çapraz bağlama immünoprecipitation (CLıP) ve çeşitli varyasyonları izledi-ultraviyole güveniyor (UV) RNA için bir RBP Crosslink ışık,8. Ancak bu, RNPs içinde RNA 'ya doğrudan başvurmayan RBPs için etkili bir yaklaşım değildir. Burada, RNA bağlayıcı sitelerini izole etmek ve tanımlamak için RBPs ve RBPs olmayan benzer alternatif bir yaklaşım açıklanmaktadır. Bu yaklaşım tandem (ripit) RNA immünoprecipitation, tek bir arıtma (Şekil 1)9,10ile karşılaştırıldığında daha yüksek özgüllük elde etmenize yardımcı olan iki immünopyemek adımları oluşur. Bireysel immünopresipitasyon (IP) adımları klip ile karşılaştırıldığında daha düşük bir katı olarak gerçekleştirilebilir gibi, RipIt immünoprecipitation sırasında güçlü deterjanlar varlığına dayanabilir antikorların mevcudiyeti bağlı değildir. RIPiT 'in en eşsiz avantajı, iki farklı proteini iki arıtma adımında hedefleme yeteneğidir; Bu, diğer benzer kompleksleri11komkonumlu farklı bir RNP kompleksi zenginleştirmek için güçlü bir yol sağlar.
RipIt prosedürünü küçük varyasyonlar RNP zenginleştirme daha da artırabilir. Örneğin, RNPs içindeki bazı RNA-protein veya protein-protein etkileşimleri geçici ve bu tür kompleksleri ayak izlerini verimli bir şekilde arındırmak zor olabilir. Bu tür etkileşimleri stabilize etmek için, RNPs hücre liziz ve RIPiT öncesinde formaldehit içeren hücreler arasında çapraz bağlanabilir. Örneğin, eXoN kavşak kompleksi (EJC) çekirdek faktörü, EIF4AIII ve EJC demontaj faktörü arasında zayıf bir etkileşimin, PYM12 ' nin daha fazla RNA ayak izi zenginleştirilmiş olması gibi formaldehit tedavisi ile stabilize edilebilir olduğunu gözlemledik (veri gösterilir). Hücre hasat ve RIPiT önce, hücreler de stabilize etmek veya belirli bir durumda RNPs zenginleştirmek için ilaçlar ile tedavi edilebilir. Örneğin, tercüme sırasında mRNA 'dan çıkarılan proteinleri okurken (örn., EJC13, UPF114), puromisin, sikloheksimit veya harringtonin gibi çeviri inhibitörleriyle tedavi, RNAs üzerinde proteinler.
RIPiT 'ten kurtarılan RNA miktarı genellikle düşüktür (0.5-10 pmoles, i.e., 10-250 ng RNA, 75 NT ortalama RNA uzunluğunu dikkate alınarak). Bunun birincil nedeni, belirli bir proteinin sadece küçük bir kısmını RNPs içindeki diğer proteinler ile karmaşık mevcut olduğu (herhangi bir "ücretsiz" protein ilk adımda ıp'ed ikinci IP sırasında kaybolur). Bu RNA 'dan RNA-Seq kütüphaneleri oluşturmak için, Ayrıca burada daha önce yayımlanan protokolün bu kadar düşük RNA girişleri için uygun bir uyarlamasını özetlemektedir15,16 (Şekil 2), hangi verir yüksek verimlilik sıralaması hazır örnekleri 3 Gün.
Protokol
1. kararlı HEK293 hücre hatları tetracycline indüklenebilir bayrak-etiket faiz (POI) proteini Ifade kurulması
- Büyüme ortamında 10 x 104 hücre/ml yoğunluklu bir stabil entegre FLP rekombinasyon hedef (frt) sitesi Ile tohum HEK293 hücreler (Dulbecco modifiye kartal orta [dmem] + 10% fetal Sığır serum [FBS] + 1% penisilin-streptomisin [Penn/strep]) 6- iyi plakalar. Hücrelerin, 37 °C ve% 5 CO2 ' de nemlendirilmiş bir kuluçta gece büyüymesine izin verin (sonraki tüm adımlar için standart büyüme koşulları).
- Ertesi gün, hücreler ~ 70% confluent olmalıdır. Transfeksiyon reaktif protokolüne göre, pcDNA5-TETO-FLAG-POI: pOG44 9:1 oranında FRT site içeren HEK293 hücreleri transfekt.
Not: FLAG etiketi seri motif DYKDDDDK (D = aspartik asit, Y = tirozin ve K = lizin) vardır. - 24 saat sonra, antibiyotik seçimine başlayın. Medya çıkarın ve 100 μg/mL higromisin ile tamamlayıcı taze büyüme orta ekleyin. 72 h içinde, untransfected hücreler ölmek başlamalıdır.
- Her 48-72 h, değişim büyüme medya ve taze higromisin ile ek.
- Seçim ~ 2 hafta sonra, stabil transfekte hücrelerin ayrık koloniler görünmeye başlayacaktır. Koloniler çıplak gözle görünürken, plakaya 1 mL tripsin ekleyin ve 37 °C ' de 5 dakika boyunca inküye yapın. DMEM hücrelerinde resuspend, yeni bir 10 cm plaka içine hücre transferi ve 10 mL taze büyüme orta 100 μg/mL higromisin ile takviye hacmi ayarlayın. Plaka kalıcı stokları hazırlamak için daha fazla hücre genişletmek için ~ 80% konfluency ulaşmak için izin verin.
- Deneme için FLAG-POı ifadesinin en uygun seviyesini elde etmek için gereken tetrasiklin (Tet) miktarını belirleyin. 12-Well formatında hücreler büyümek ve 16-24 h için 0-1000 ng/ml aralığı arasında Tet bir titrasyon yürütmek. POI 'ye karşı antikor ile titrasyon örnekleri üzerinde Batı lekesi gerçekleştirin.
Not: ~ 60 kDa 'ye kadar olan proteinler için, bayrak etiketli ve proteinin endojen kopyası sodyum Dodesil sülfat-Poliakrilamid jel elektroforez (SDS-Page) üzerinde çözülebilir, bayrak-POI ' i n endojen meslektaşı ile ifade düzeylerini karşılaştırın. Daha büyük proteinler için, Tet kaynaklı örneklerde sinyal yoğunluğu indüklenmiş örnekle karşılaştırılabilir. Tetrasiklin stok çözeltisi% 100 etanol içinde 1 mg/mL 'de hazırlanabilir. Hücre kültürü çalışmaları için Stoktaki dilüsyonlar steril su veya fosfat-tamponlu tuzlu (PBS) olmalıdır. Tetracycline stok her ay bir kez taze hazırlanmalıdır.
2. tetracycline Indüksiyon ve RIPiT prosedür için kültür hücreleri
- Tohum stabil entegre Flag-POI HEK293 bir yoğunlukta 3 x 105 hücreler/ml büyüme ortamında 15 cm plakalar. Hücrelerin 37 °C ve% 5 CO2' de büyümesine izin verin.
Not: Genel olarak, üç ila 5 15 cm plakaları faiz RNP bolluğuna bağlı olarak RNA ayak izleri ~ 2-20 pmol verecektir. RNPs formaldehit olacak ve sıkı koşullar altında temizleşmiş, o zaman iki kat daha fazla giriş gerekebilir. - Önceden işaretli-POI (bkz: adım 1,6) 16-24 h hücre hasat olacak önce ifade ikna etmek için medyada optimum konsantrasyon tespit tetrasiklin ekleyin.
Not: Büyüme ortamını değiştirmek gerekli değildir. Hücreler, tohumlama sonrası ya da plaka ~ 80% confluent olduğunda yaklaşık 72 h hasat etmeye hazır olmalıdır. Hücrelerin confluent olmaktan kaçınmak önemlidir.
3. hücre hasat, formaldehit tedavisi, ve hücre Lysis
- Her 15 cm plaka için 15 ml soğutulmuş PBS ile Tek tabakalı hücrelerini yavaşça yıkayın. Sonra 30 mL PBS hücrelerinde kazımak. Eğer hücreler hasat öncesi ilaçlar ile tedavi edildi, ilaç ile ek PBS. 50 mL konik tüp içine tüm hücreleri toplayın.
Not: Zayıf etkileşimleri korumak için, hücreler formaldehit ile çapraz bağlı olabilir: adım 3,1 son konsantrasyon 0,1% için hücre süspansiyona formaldehit ekleyin ve 10 dakika oda sıcaklığında rocker üzerinde kuluçka. su giderici tamponunun 3 ml 'ini ekleyin (Tablo 1 ) ve kaya 5 ek dakika için. - 4 °C ' de 10 dakika için 400 x g 'de Pellet hücreleri ve supernatant atın.
- Lyse hücreleri 4 mL buz-soğuk hipotonik lizis tamponu (HLB; Tablo 1). Hücreleri yeniden pelletini için bir P1000 pipet kullanın. 5 mL 'Lik bir tüpe aktarın ve 10 dakika boyunca buz üzerinde lysate inküye.
- Lysate 'yi bir buz banyosuna yerleştirin ve 2 s duraklama ile 1 s darbeleri ile 30 s için% 10 amplitüstte sonikat yapın. Daha sonra, 108 μL 5 M NaCl ekleyerek tuz konsantrasyonunu 150 mM 'ye ayarlayın.
Not: Formaldehit çapraz bağlı numuneler, lizis tamponunun% 0,1 ' sini SDS ve sodyum deoksikolatın dahil ederek daha sıkı liziz ve arıtmaya tabi olabilir. - 21.000 x g 'de 4 °c ' de 10 dakika santrifüjleme ile lysate temizleyin. Girişte protein düzeylerinin Batı leke analizi için etiketlenmiş bir tüpte 20 μl süpernatant (hücre özü) toplayın (bkz. Şekil 3a).
Not: Lysate santrifüjte iken, flag agaroz boncuklar yıkanabilir (bkz. Adım 4,1). Flag agaroz boncuk ön yıkanmalıdır 3x 4 ml buz-soğuk izotonik yıkama tampon (ısowb, Tablo 1).
4. bayrak Immunoprecipitation
- Adım 3,5 için kalan süpernatant uygulamak 750 μL önceden yıkanmış Flag agaroz boncuk 5 ml tüp (yatak hacmi yıkanmış Flag agaroz boncuk olacak 375 μL). İşaret agaroz boncuk ve hücre özü 1-3 h için 4 °c hafif karıştırma ile.
Not: Flag agaroz boncuk bu hacim protein geniş bir yelpazede için yeterli olmalıdır, ancak gerekirse, optimize edilebilir. - Pellet Flag agaroz boncuklar 400 x g 'de santrifüjleme ile 4 °c ' de 1 dak. Tükenmiş hücre ekstresi protein düzeylerinin Batı leke analizi için etiketlenmiş bir tüpte süpernatant 20 μl toplayın (bkz. Şekil 3a). Kalan supernatant atın.
- Bayrak agaroz boncuk yıkamak için, 4 ml ısowb ve resuspend ekleyin. 4 °C ' de 1 dak için 400 x g 'de Pellet boncuklar. Dikkatle supernatant kaldırın. 4X tekrarlayın.
Not: Formaldehit çapraz bağlı IP 'lerin sıkı yıkamalar için,% 0,1 SDS ve sodyum deoksikolatın her biri ilk iki yıkama basamakları için yıkama tampona dahil edilebilir.
5. RNase ı sindirim
- 750 μL 'de (istenilen ayak izi boyutları için uygun konsantrasyon, ampirik olarak belirlenmelidir), RNase ı ile 0.002-0.01 adet/mL arasında seyreltilebilir. Isowb-RNase I ' I yıkayıp bayrak agaroz boncuklar ekleyin ve 10 dakika boyunca 4 °c ' de nazik karıştırma ile inküye yapın.
- 4 °C ' de 1 dak için 400 x g 'de Pellet boncuklar. Batı leke analizi için etiketli bir tüpte 20 μl süpernatant (RNase ı elüsyon) toplayın. Isowb-RNase I ve Wash bayrağı agaroz boncuk 4X ısowb ile adım 4,3 açıklandığı gibi atın.
6. yakınlık elüsyonu
- Bir elüsyon tampon stokları hazırlayın (FLAG peptid at 250 ng/mL ısowb). 375 μL 'yi Flag agaroz boncuk için Uygula ve 1-2 h için 4 °c ' de yavaşça sallayın. Pellet Flag agaroz boncuklar ve Flag IP (bkz. Şekil 3a) Batı leke proteinleri için bir 15 μL kısım toplamak.
Not: Benzerliği devam ettiğinde, Bölüm 7 gerçekleştirilebilir.
7. manyetik boncuk-Antibody konjugasyon
- Wash 50 μL manyetik boncuk (örn., Dynabeads; Malzeme tablosu) 3x içinde 1 mL ısowb içinde 1,5 mL tüp. 100 μL konjugasyon tamponunun içinde manyetik boncuk resuspend. Antikor uygun miktarda ekleyin (IP için kullanılacak antikor tam miktarı her antikor için ampirik olarak belirlenmelidir).
Not: Protein A manyetik boncuklar tavşan üretilen antikorlar için optimum ise protein G manyetik boncuklar fare antikorları için daha uygundur. Manyetik boncuk uyumluluk şeması her antikor için uygun boncuk seçmek için tedarikçi Web sitesinde mevcuttur. - Manyetik boncuk 2x konjugasyon tampon (Tablo 1) yıkayın. Karışımı oda sıcaklığında en az 10 dk. 375 μL 'de RIPiT seyreltme tamponunda resuspend manyetik boncuklar. Bir sonraki adıma kadar buzda saklayın.
8. Ikinci Immünoprecipitation
- Adım 6,1 ' dan kalan FLAG benzeşimi elüsyonu, ilgi proteinlerine karşı antikorlarla birleştirilebilecek manyetik boncuklarla uygulayın. 1-2 h için 4 °c ' de nazik karıştırma ile kulkarın. bir mıknatıs üzerinde manyetik boncuk yakalayın ve Batı Blot yoluyla ilişkisiz proteinlerin analizi için 15 μL süpernatant toplayın. 1 mL ısowb ile manyetik boncuk 7x yıkayın.
9. denaturing elüsyon
- 100 μL şeffaf örnek tampon (Tablo 1) manyetik boncuk ve P200 pipet ile pelletini ekleyin. 10 dakika boyunca buzun üzerine inküyün. düzenli olarak boncuklar pelletini hafifçe Flick.
- Mıknatıs üzerinde manyetik boncuk yakalayın ve Batı leke yoluyla ripit elüsyon proteinleri analizi için 15 μL elüsyon toplayın (bkz. Şekil 3a). Kalan elüsyonu yeni bir etiketli 1,5 mL tüpüne aktarın.
Not: Numuneler formaldehit crosslinked olsaydı, daha sonra numuneler 65 °C ' de 1 saat boyunca çapraz bağlantı yapmak için inkübe edilmelidir. - Çeşitli adımlarda toplanan örneklere Batı lekesi gerçekleştirin (giriş, bayrak IP tükenmesi, flag IP, ikinci IP tükenmesi, ikinci IP elüsyon). İki yem proteinine karşı antikorları olan Blot, biliniyorsa diğer interaktörler ve negatif kontrol olarak en az bir etkileşmeyen RBP (Şekil 3a).
10. RNA ekstraksiyon ve son kür
- RIPiT elüsyona, 320 μL RNase-Free ddH2O, 400 μL Phenol-Chloroform isoamill alkol (pcıaa, pH 4,5) ekleyin ve 30 s için Vortex ve 5 dakika için 12.000 x g oda sıcaklığında spin. ayrı bir tüpün içinde 350 μL sulu faz toplayın. 35 μL 3 M sodyum asetat, 1 μL 1 M MgCl2, 10 μg glycogen ve 1 ml 100% etanol ekleyin. Gece-20 °C ' de inküyün.
- RNA Pelet için, 12.000 x g 'de 4 °c ' de 30 dakika Santrifüjü. 70% etanol içinde yıkama RNA.
- RNase I dekolsiyon sonrası RNA 'da kalan 3 ' fosfat 'i kaldırmak için, 17 μL RNase-Free ddH2O 'da Ise 2 μL 10X T4 polinükleotid KINAZ (PNK) tampon (malzeme tablosu) ve 1 μL T4 PNK ekleyin. 37 °C ' de 30 dakika boyunca inkübe
Not: T4 PNK 'nin 3 ' fosfataz aktivitesi pH 617' de optimum aktiviteye sahiptir. PNK reaksiyon tampon T4 PNK 5 ' kinaz aktivite için optimize edilmiştir ve 7,6 pH vardır. Son kür reaksiyonu pH ayarı girişiminde bulunulurken, bu adım daha da optimize edilebilir. - 380 μL RNase-Free ddH2O ve 400 ΜL PCıAA pH 4,5 ' ı boruya ekleyin. 30 s için Vortex, 12.000 x g 'de Santrifüjlü 5 dk. sulu faz toplayın ve 35 μl 3 m sodyum asetat, 1 μL 1 m MgCl2, 10 μg glycogen ve 1 ml% 100 etanol ekleyin.
- Gece-20 °C ' de inküyün. % 70 etanol ile Pellet ve yıkama RNA yukarıda. 4,5 μL RNase-ücretsiz su içinde resuspend RNA.
11. RNA ayak Izi büyüklüğü ve bolluk tahmini
-
Başarılı bir RIPiT 1 pmol veya daha fazla RNA parçası verim bekleniyor. Gerçek verimi ölçmek için, 0,7 μL RIPiT RNA (~ 1/6 toplam verim) yeni bir tüpe aktarın. 2 μL 10X T4 PNK tampon, 1 μL 1 mM ATP, 40 Μcı γ32P-ATP (stoktaki 0.5 − 1.0 μL) ve 1 μL T4 PNK ekleyin. Hacmi 10 μL olarak ayarlayın ve 37 °C ' de 30 dakika boyunca inküye yapın.
- Paralel PNK reaksiyonları, bir boyut ve miktar standartları kullanmak için bir sentetik RNA veya DNA oligo (20-40 NT) düşük menzilli DNA Ladder ve 0,1 pmol etiket.
- % 26 üre-sayfa jel (20 x 27 x 0,45 mm3) ÜZERINDE etiketli RNA/DNA çözün. Jel Pre-Run olmalıdır 30 dakika 35 W. Flush kuyuları öncesi çalıştırmadan önce ve örnekleri yüklemeden önce ve çalıştırmak 35 W kadar bromophenol mavi boya ön neredeyse jel sonuna ulaştı.
- Jel cam plakalardan 8 x 11 inç filtre kağıdının bir parçasına dikkatle çıkarın. Kağıt üstünde jel ile, jel kurutma aparatı yer ve plastik wrap bir parça ile kapak. 80 °C ' de kuru jel, vakum ile 1 saat.
- Kurutulmuş jeli bir fosfora bir gecede veya yeterli sinyal tespit edilinceye kadar açığa çıkarın. Görüntü fosforuydu. İyi kalite RNA bir RIPiT şerit içinde bir smear olarak görünmelidir, minimal belirgin bantları ile (Şekil 3B). RNA 'ya ölçmek için, RIPiT şeridinde istenilen boyutta RNA parçalarının sinyal yoğunluğunu etiketli sentetik oligo 0,1 pmol sinyaline göre karşılaştırın.
Not: Alternatif olarak, RNA ayak izi boyutu ve miktarları yüksek hassasiyetli biyoanaliz cihazı kullanılarak doğrulanabilir (Şekil 3C).
12. adaptör ligasyonu
- 3,8 μL su içinde en az 3 pmol RNA çözülür gibi RIPiT RNA hazırlayın.
- 0,2 mL polimeraz zincir reaksiyonu (PCR) tüpünde, 3,8 μL RNA, 1 μL MIR-CAT-33 pre-adenylated adaptör (7 μM) (malzeme tablosu) birleştirir. Karışımı, 65 °c ' de 10 dk, 16 °C için 5 dakika, sonra 4 °C ' de tutun.
Not: Pre-adenylated bağlayıcı ya oligo sentezleme hizmetinden sipariş edilebilir, ya da herhangi bir oligo sentezi hizmetinden özel bir unadenylated DNA oligo MTH RNA ligaz kullanılarak adenylated olabilir (malzeme tablosu) ve jel saflaştırılmış. - Aynı tüp için 1,5 μL 10X T4 RNA ligaz tampon, 7,5 μL% 50 Polietilen glikol 8000 (Peg-8000), 0,75 μL 20 mm ditiyotreitol (DTT) ve 0,45 μL T4 RNL2 tr. K227Q (malzeme tablosu) ekleyin.
Not: 50% PEG-8000 RNA ligaz ile birlikte gelir ve tampon satın alınır. PEG-8000 çözeltileri viskoz ve yavaşça pipetleme yapılmalıdır. - 6 h için 30 °C ' de termal bisikletçiye reaksiyonunu ele getirin, ısı 65 °C ' de ligaz 10 dakika süreyle inaktive et, ardından 4 °C ' de tutun.
13. Ters transkripsiyon
- Adım 12,4 ligasyon karışımı ile tüp için, eklemek 11,25 μL 4X deoxynucleotide trifosfat (dntp) Mix, normal ve biyotinlenmiş dntps bir karışımını içerir (bkz: Tablo 1), 1,0 μL 10 μM RT astar (malzeme tablosu), ve 6,8 μL RNase-ücretsiz su. 65 °C ' de 5 dakika boyunca inküye yapın, ardından 4 °C ' de tutun.
- Tüpleri buza aktarın ve MgCl2 (Tablo 1), 2,25 μL, 100 mM DTT, 1,2 μL 'nin son hacmi olan 45 μL (malzeme tablosu) ile 9,0 μL 'LIK 5x ilk iplikli (FS) tampon ekleyin.
- 55 ° c 'de bir termal bisikletçi olarak 30-60 dk. ısı devre dışı ters transkriptaz, 70 °c ' de 15 dakika ve numuneyi 4 °c ' de tutun.
14. RT ürününün arıtılması
- 45 μL 'yi RT reaksiyonuna 2x üre yük tamponu (Tablo 1) ekleyin. 45 μL 'de düşük menzilli DNA merdiven 1 μg seyreltmeli ve 45 μL 2x üre yük tamponu ekleyin.
- Hazırlamak 10% üre-sayfa jel (20 x 28 x 0,15 cm3; Tablo 1) 8-iyi tarak ile. Şırınga veya pipet kullanarak, 0.5 x Tris/borate/EDTA (TBE) tampon ile Flush kuyuları.
Not: Yukarıdaki ev yapımı jelleri, genişletilmiş RT ürününü genişletilemez RT astarından ayıran daha iyi çözünürlük sunar. Alternatif olarak, önceden cast Urea-sayfa jelleri de kullanılabilir (malzeme tablosu). Ön dökme jellerin iyi başına daha küçük maksimum hacimler izin olarak, bu nedenle numuneler birden fazla kuyular bölünmesi gerekir. Ön döküm jeller 150 − 200 V 'de çalıştırılmalıdır. - 30 dakika boyunca jel 35 W 'de önceden çalıştırın. yine floş kuyuları, yük örnekleri ve çalışan jel 35 W içinde bromophenol mavi boya ön kadar göç etti 1 jel sonundan inç.
Not: Ön çalıştırma sırasında metal ısı emici kullanın ve jel aşırı ısınmasını önlemek için son çalışma. - 0,5 x TBE 'de hazırlanan 1x altın nüklik asit jel leke çözeltisi ile 5 dakika boyunca jeli lekeleyin. Bu boya ışık duyarlı, bu yüzden ışık pozlama kaçının.
- 520 nm uyarma ve 580 nm Emisyon filtreleri kullanarak dokümantasyon amacıyla floresan tarayıcıdaki görüntü jeli. Floresan tarayıcı kullanılamıyorsa, mavi ışık transluminator kullanın. RT ürünü, genişletilemez RT astar (Şekil 4) ' ün üzerinde başlayarak bir smear olarak görünmelidir.
Not: Altın nüklik asit jel boyama boyası, UV-ışık kaynağına sahip bir jel doc üzerinde kolayca görselleştirilse de, hasar görmesini önlemek için değerli RT ürününü UV 'ye maruz bırakmanın hayati önem taşımaktadır. - Mavi ışık transaydınlatıcında jeli görselleştirin ve RT ürününü jelden çıkarın. 30-200 NT (Şekil 4) arasında değişen UZANTıLARLA DNA 'yı kesmeleri tavsiye edilir. Temiz bir yüzeye eksiz jel parçaları yerleştirin ve yüzey alanı artırmak için küçük parçalar halinde dilim kıyma. 1,5 mL tüpüne dikkatle aktarın ve 800 μL DNA elüsyon tamponunu (Tablo 1) ekleyin.
- Gece boyunca oda sıcaklığında DNA elüsyonu tamponu ile yumuşak karıştırma ile jel parçalarını kulyın.
- 2 mL 'Lik bir toplama tüpüne yerleştirilen selüloz asetat filtre sütunu (malzeme tablosu) aracılığıyla Bulamaç geçirerek jel tamponunu jelden ayırın.
- 1,5 mL tüpte, 500 μL streptavidin boncuk yıkama tamponu ile 10 μL streptavidin manyetik boncuk yıkama (Tablo 1). Üç toplam yıkanıyor için tekrarlayın. Boncuk kurumasına izin vermeyin. 10 μL DNA elüsyon tampon (Tablo 1) içinde resuspend boncuk.
- 14,8 adımda jel parçalarından ayrılan erüsyon tamponunu, yıkanmış streptavidin manyetik boncukları içeren tüpten aktarın.
- Oda sıcaklığında en az 8 saat boyunca nazik karıştırma ile inküye yapın. Bir mıknatıs üzerinde boncuk yakalama, süpernatant kaldırmak ve 10 μL RNase-Free su ve 0,2 ml PCR tüp transfer manyetik boncuk pelletini.
15. RT ürününün döngülerleşmesi
- Streptavidin boncuklar üzerinde yakalanan RT ürünleri boncuk bağlı iken circularized vardır. Manyetik boncuk Bulamaç için, 2,0 μL 10X döngü reaksiyonu tampon ekleyin, 1,0 μL 1 mM ATP, 1,0 μL, 50 mM MnCl2, 4,0 μL 5 M betaine, 1,0 μL ssDNA ligaz ı (malzeme tablosu) ve 1,0 μL RNase-Free su.
- 4 h için 60 °C ' de termal bisikletçi üzerinde olan döngü reaksiyonunu intutma. ısı, ssDNA ligaz ı, 80 °C ' de 10 dakika ısıtarak 4 °C ' de tutun.
16. test PCR
- Büyük ölçekli bir PCR 'ye geçmeden önce, her örnek için ideal amplifikasyon döngülerinin sayısını belirlemek üzere bir parça dairesel ürün kullanın. Bu adım, aşırı amplifikasyon önlemeye yardımcı olur ve PCR reaksiyon bileşenleri daha yüksek PCR döngüleri sınırlanması gibi PCR eserler sınırlamak.
- A 45 μL PCR reaksiyonu ile hazırlama 4,0-6,0 μL-den döngüsel ürün, adım 15,2, 9,0 μL 5x reaksiyon tampon, 0,9 μL 10 M dNTPs, 2,25 μL 10 μM PE 1.0 astar (malzeme tablosu), 2,25 μL 10 μm PE 2.0 astar (malzeme tablosu) ve 0,045 μL yüksek doğruluk DNA polimeraz (malzeme tablosu) ve su.
- Karışım reaksiyonu iyi ve 3 15 μL reaksiyonlarına ayrılır. Bu üç reaksiyonlardan her biri, değişken sayıda PCR döngüsüne tabi olacaktır. İdeal döngüleri sayısı 7 ile 14 arasında olması bekleniyor. Böylece 8, 11 ve 14 döngüleri için test PCRs gerçekleştirin.
- Aşağıdaki PCR koşullarını kullanın: 98 °C-30 s; 98 °C-5 s; 65 °C-10 s; 72 °C-15 s; 72 °C-2 dak; 12 °C-tutun.
- 3 μL 6x jel yükleme boya ekleyin ve mavi boya ön jel 3/4 göç edene kadar% 10 yerel sayfa jel çözmek. 14,4 ve 14,5 (Şekil 5) adımda olduğu gibi altın nüklik asit jel leke ve görüntü kullanarak jel leke.
- İdeal PCR döngüsü sayısını seçmek için, PCR ürünlerini artan döngü sayısını karşılaştırın. Aşırı amplifikasyon eserler olmadan beklenen boyuttaki en büyük ürün miktarını veren döngü numarasını seçin (örn., beklenen ürüne göre çok daha büyük DNA smear, ve PE 1.0 ve PE 2.0 astarların hiçbir farkedilemez tükenmesi görülür (kırmızı oka bakınız Şekil 5).
17. büyük ölçekli PCR
- 16,2 adımda olduğu gibi 45 μL PCR reaksiyonu hazırlayın ve PCR 'i tekrarlayın. 150 V 'de% 10 yerel sayfada PCR çözme, mavi ışık transilluminator üzerinde 1x altın nüklik asit jel leke ve görüntü ile leke.
- PCR ürününü jelden çıkarın ve 3 mL şırıngaya aktarın. Jel ve ekstrüzyon bir 1,5 ml tüp içine ezmek için şırınga kullanın.
Not: Devre dışı RT ürünü, 151 BP 'nin PCR ürününü verir. Böylece, bu adımda bir boyutu 151 BP 'den daha büyük olan seçme ürünleri olmalıdır (Şekil 6). - 900 μL DNA elüsyon tamponunu ekleyin ve gece boyu yumuşak karıştırma ile Oda sıcaklığında inküye yapın.
- Jel Bulamaç 2 mL toplama tüpüne yerleştirilen bir selüloz asetat filtre sütununa aktarın. Spin at 12.000 x g 3 dakika, taze bir tüp içine süpernatant toplama.
- Ezilmiş jel için başka bir 400 μL DNA eklemi tampon ekleyin ve 1,5 mL tüpüne aktarın. İkinci bir elüsyon için ek 4 saat boyunca nazik karıştırma ile inkübe.
- Havuz tüm elutions ve bölünmüş 3 tüpler ile 400 μL her. 1 mL 100% etanol ve 10 μg glikojen ekleyerek DNA 'Yı çökeltir. Vortex ve en az 2 saat-20 °C ' de inkük.
- 4 °C ' de 30 dakika için 12.000 x g 'de Pellet DNA 'sı. 70% etanol ile yıkayın DNA Pelet.
- Dikkatle pipetleme ile tüm etanol kaldırmak ve hızlı bir şekilde 20 μl su içinde DNA Pelet pelletini.
Not: Bu aşamada, DNA Pelet kuru haline izin vermek önemli değildir, olarak kurutmak, onu ortaya çıkarabilir. - Fluorometre ve yüksek hassasiyet DNA biyoanalizörü ile PCR ürününün boyutunu ve konsantrasyonunu belirlemek için DNA örneğinin küçük bir kısmını kullanın. Örnekler artık platformlardan birinde sıralama için sunulabilir.
- Sıralı okuma (örneğin, bağdaştırıcı kaldırma, dizileri > 30 phred puanı tutmak için kırpma), referans genomuna hizalanır ve UCSC genom tarayıcısı gibi bir tarayıcıda görselleştirilebilir (Şekil 7).
Sonuçlar
Başarılı bir RIPiT, hem ilgi hem de diğer bilinen etkileşen proteinlerin immünoptitasyonu ve etkileşimsiz proteinlerin olmaması ile sonuçlanır. Şekil 3A'da görüldüğü gibi, hem MAGOH hem de EIF4AIII ripit elution 'da tespıt edildi, ancak HNRNPA1 (şerit 6) değildi. Paralel olarak, RNP kompleksleri ile birlikte arıtılmış RNA ayak izleri otoradyografi tekniklerinde (Şekil 3B) veya biyoanalizör (Şekil 3C) ile tespit edildi. Puromisindir tedavisinin RNA 'da EJC doluluk sıklığını arttırılması bekleniyor ve Şekil 3B 'de puromisin tedavi edilen ripit 'te daha güçlü RNA ayak izi sinyali izlendi (şerit 2 ve 3 ' ü karşılaştırın). Derin sıralamanın numunelerini oluşturmak için RNA 'ya bir adaptör bağlanması ve ardından adaptör sırasına göre bir astar kullanarak RNA 'yı DNA 'ya transkriptmak gerekir. Ters transkripsiyon adımı, Ters transkripsiyon ürününün arıtılması için biyotinlenmiş nükleotid içerir. Ters transkripsiyon sonrasında, ürün Urea-PAGE (Şekil 4) tarafından Genişletilebilir adaptörden ayrılmalıdır. Ters transkripsiyon ürünü daha sonra döngüsel ve PCR amplifikatörüdür. Uygun sayıda PCR döngüsü, döngüsel ürünü aşırı yükseltmez. Aşırı amplifikasyon, primer tükenme ve anormal PCR ürününe neden olacaktır (bkz. Şekil 5, şerit 4). Aşırı amplifikasyon kanıtı olmadan en büyük amplifikasyon ile döngü sayısı büyük ölçekli bir PCR için kullanmak en uygun (Şekil 5 şerit 3 ve Şekil 6).

Şekil 1 : RIPiT 'deki ana adımları özetleyen şematik. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
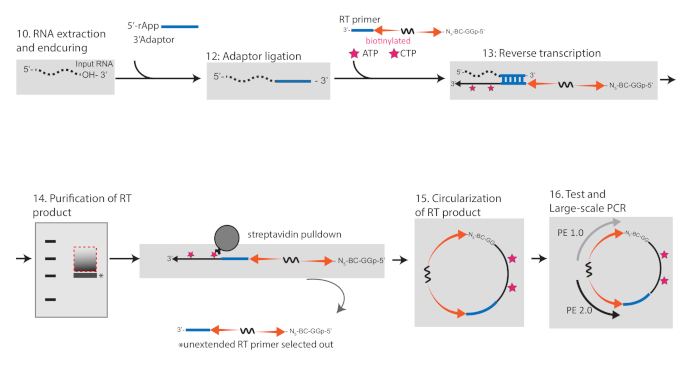
Şekil 2 : Yüksek verimlilik sıralaması Için RIPiT RNA 'nın kütüphanelere dönüştürülmesi için iş akışını tasvir eden şematik. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
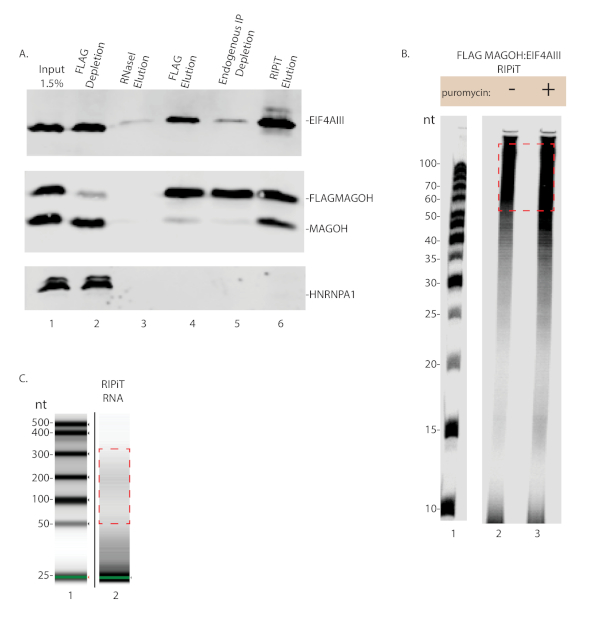
Şekil 3 : RNA ve protein verimleri RIPiT 'Den tahmin edilir. (A) ripit prosedürün her önemli adımından arındırılmış proteinlerin Batı leke. (B) bir bayrağın RNA ayak izlerinin autoradiograph görüntüsü: EIF4AIII ripit, tedavi edilen ve tedavi edilmeyen hücreleri karşılaştıran puromisindir. Kırmızı kutu, sonuçta sıralı kitaplıklarına dönüştürülecek RNA ayak izi boyutunu gösterir. (C) bir biyoanalizör kullanılarak görselleştirildiğinde, B panelindeki şerit 2 ' de RipIt 'den gelen RNA 'nın profili. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 4 : Ters transkripsiyon (RT) ürün% 10 üre-sayfa jel üzerinde çözüldü ve altın nüklik asit jel leke ile lekelenmiş. Kırmızı kutu, genişletilmiş RT ürünlerinin jel arıtma için eksiz jel bölgesini gösterir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
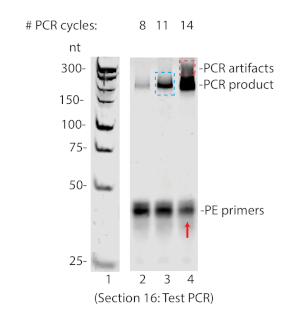
Şekil 5 : Test PCR% 8 olmayan denaturing sayfa üzerinde çözüldü. 14 döngüde (kırmızı kutu) ve aşırı amplifikasyon göstergesi olan astar (kırmızı ok) paralellerinin paralel olarak tükenmesine neden olan belirsiz büyük PCR ürünlerini unutmayın. Bu örnek için, büyük ölçekli PCR (mavi kutu) için 11 döngü seçilmiştir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
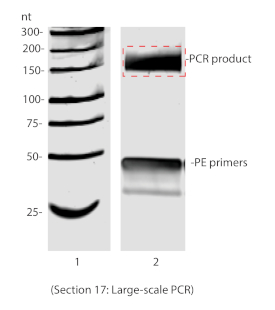
Şekil 6 : Büyük ölçekli PCR% 8 ' i denatsız sayfada çözüldü. Kırmızı kutu, jel arıtma için uyarılmış jel parçasını gösterir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 7 : MAPK1 GENI bayrak-MAGOH dağılımı gösteren Genome tarayıcı ekran görüntüsü: bir RIPiT temsili sonuçları olarak EIF4AIII ayak izleri. Kırmızı oklar, beklenen kurallı EJC bağlama sitelerini gösterir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
| Pbs | 137 mM NaCl 2,7 mM KCl 10 mM na2HPO4· 7h2O KH2Po4 pH 7,4 | | |
| Tampon söndürme | 2,5 M Glycine tarafından 2,5 mM Tris tabanı | |
| Hipotonik lizis tamponu (HLB) | 20 mM Tris-HCl pH 7,5 15 mM NaCl 10 mM EDTA 0,5% ıGEPAL 0,1% Triton-X-100 1x Aprotinin * 1x Leupeptin * 1x Pepstatin * 1 mM PMSF (phenylmethylsulfonyl fluorid) * | * Her seferinde taze eklenmesi gerekir |
| İzotonik yıkama tamponu (ısowb) | 20 mM Tris-HCl pH 7,5 150 mM NaCl 0,1% ıGEPAL | |
| Konjugasyon tampon | 0,02% polysorbate-20 1x PBS | |
| Örnek arabelleği temizle | 100 mM Tris-HCl pH 6,8 4% SDS 10 mM EDTA | |
| 4xdNTPmix | 0,25 mM dGTP 0,25 mM dTTP 0,175 mM dATP 0,1625 mM dCTP 0,075 mM biotin-dATP 0,0875 mM biotin-dCTP | |
| 5x Ilk Iplikli tampon w/o MgCl2 | 250 mM Tris-HCl pH 8 375 mM KCl | |
| RIPiT seyreltme tamponu (1 mL) | 1 mL ısowb 5 μL 200x BSA 20 μL 10% Triton-X-100 20 μL 0.5 M EDTA 1x Aprotinin * 1x Leupeptin * 1x Pepstatin * 1 mM PMSF * | * Her seferinde taze eklenmesi gerekir |
| 2x denaturing yük tamponu | 3 mL 5x TBE 1,8 g Ficoll türü 400 6,3 g üre 3 mg bromophenol mavi 3 mg ksilen cyanol Hacmi 15 mL ddH2O olarak ayarlayın | Çözelti içine almak için, bir kabı içinde su tüp yer ve sıcak plaka üzerinde kaynatın 10 – 15 dk. hacmi 15 ml 'ye ayarladıktan sonra boyalar ekleyin |
| Urea-sayfa jeli | 6 M üre Akrilamid: bisacrylamid (40% [w/v]) uygun yüzde 0.5 x TBE 0,01% TEMED | |
| DNA elüsyon tampon | 300 mM NaCl 1 mM EDTA | |
| Streptavidin boncuk yıkama tamponu | 0,5 M NaOH 20 mM Tris-HCl pH 7,5 1 mM EDTA |
Tablo 1: arabellekleri.
Tartışmalar
Biz burada bazı önemli hususlar başarıyla RIPiT gerçekleştirmek için tartışmak. Her şeyden önce, her adımda mümkün olan en yüksek verimliliği elde etmek için bireysel IPS optimize edilmelidir. Burada açıklanan hücre giriş sayısı için Flag agaroz boncuk miktarı test ettiğimiz çeşitli proteinler için sağlam olduğu kanıtlanmıştır. Ortak proteinlerin sadece küçük bir kısmı olarak bayrak proteini ile Co-immunoprecip, etkili ikinci IP için gerekli antikor miktarı genellikle düşüktür (daha az 10 μg). Küçük ölçekli ripit (1 10 cm plaka) sonra iki immünoprecipitation adımlar sırasında her fraksiyonda proteinlerin Batı leke doğrulama takip son derece verimlilik ve ölçeklendirme önce prosedürün özgüllüğü değerlendirmek için yararlı olduğunu kanıtlamak. Hem hedeflenen proteinlerin hem de kompleksin diğer beklenen etkileşim proteinlerinin elüsyonda saptanmalıdır. Aynı zamanda, tükenmiş endoglikozidazları (Flag-agarose veya manyetik boncuk için ilişkisiz) protein için tahlil için yararlıdır immünoprecipitation verimliliği ve bir kompleks içine montaj proteinlerin yüzdesi iyi bir tahmin var. Ayrıca, bu analiz, RNase sindirim koşullarının RNA 'ya bağımlı etkileşimleri bir RNP içindeki RNA bağımsız etkileşimlerinden ayırmak için yeterli olup olmadığını da bildirir. Bu nedenle, negatif bir denetim, ideal bir RBP ilgi RNP ilgisiz dahil etmek için önemlidir. Örneğin, Şekil 3A'da, HNRNPA1 giriş var, ancak ripit elution algılanmaz. HNRNPA1, EJC ile doğrudan etkileşime girmiyor ancak EJC ve HNRNPA1 aynı RNA molekülüne bağlıysa EJC ile dolaylı etkileşen bir RBP 'dir. Elüsyonda negatif kontrol proteininin algılanması, zayıf RIPiT özgüllüğü veya yetersiz RNA dipbaskı gösterir. Böyle bir durumda, elde edilen RNA ayak izleri, ilgi proteininin ayak izlerini tamamen yansıtmaz. Sonraki RNA-Seq için boyut 50-200 NT ayak izleri tavsiye edilir. süre RNase ı tedavi veya kullanılan enzim miktarı istenilen boyutta ayak izleri elde etmek için optimize edilebilir. En iyi durum senaryosu istenen boyut aralığında iyi sinyal elde etmek için olacak ve en uygun koşullarda bile daha uzun ve kısa RNAs olması kaçınılmaz olduğunu unutmayın. RIPiT, tek bir RBP 'nin bağlayıcı sitelerini elde etmek için de kullanılabilir. Böyle bir durumda, aynı protein iki farklı antikorlar ile immünoprecip, olabilir, ilk bir benzeşme etiketi karşı bir antikor kullanarak ve daha sonra protein kendisi karşı antikorlar18. Son olarak, negatif bir kontrol RipIt ikinci IP ilgisiz bir protein karşı bir protein karşı antikor ile birlikte bir bayrak etiketli kontrol proteini (örneğin, yeşil floresan protein) ifade hücrelerinden paralel olarak gerçekleştirilebilir.
Birçok avantajlarına rağmen, RIPiT yaklaşımı ve olası ilaçlar bazı sınırlamaları dikkate almak önemlidir. İlk arıtma sonra benzeşme gereksinimi etiketli bir protein ifade etmek için biyolojik kaynak gerektirir. Bir siteye özgü rekombinasyon sistemi hücre satırında veya ilgi organizma mevcut değilse, bir FLAG etiketi (8 amino asitler) gibi kısa bir benzeşimi etiketi CRISPR/CAS tabanlı genom düzenleme yaklaşımı19kullanarak endojen gen Locus tanıtılabilir. Flag etiketi bu yaklaşım için ideal bir epitopu, çünkü Flag antikor benzeşme için iyi uygundur ve formaldehit Crosslinking ile birlikte kullanılabilecek yüksek iyonik güçlü ve hafif denatlık koşullara dayanabilir. RipIt yaklaşımı başka bir sınırlama hücresel malzeme büyük bir giriş için gereksinimdir. Bu, bir RBP 'nin sadece küçük bir yüzdesi olarak RNP 'deki diğer proteinlerle etkileşen bir ölçüde kaçınılmaz kalabilir. Hala geliştirilmiş Kütüphane hazırlama yaklaşımlar büyük giriş gereksinimi aşağı getirmek için yardımcı olabilir. Bu adımların daha da kolaylaştırması için olası fikirler, ikinci IP yıkandıktan hemen sonra ve RNP 'nin son elüsyonu öncesinde manyetik boncuklar üzerinde RNA 3 ' lü olan ve adaptör ligasyonu gerçekleştirmesini içerir. Böyle bir yaklaşım, geçerli KLIP-Seq yordamlarda ve son zamanlarda açıklanan RipIt20varyasyonunda başarıyla uygulanır. Bu değişiklikler ayrıca kütüphane hazırlama prosedürün erken aşamalarından birkaç zaman alan RNA arıtma adımlarını da kaldıracaktır. Ayrıca, RNA 'da bir RBP 'nin çapraz sitesinin nükleotid seviyesi çözünürlüğü sağlayan CLIP 'nin aksine, ripit ayak izlerinin çözünürlüğü onlarca nükleotiid seviyesinde kalacaktır. Son olarak, RNPs birden fazla RBPs içerebilir gibi, RIPiT zenginleştirilmiş RNA siteleri birçok RBPs bağlayıcı sitelerin bir karışımını içerir. Bireysel rbps tarafından bağlı fikir birliği dizileri hızla artan hızda açığa ediliyor ve şimdi kolayca kullanılabilir21,22,23, bu bilgileri RBP sitelerin çeşitliliği deconvolve için yararlanarak olabilir RIPiT çıkışlarında zenginleştirilmiştir. Bu zorluklara rağmen, RIPiT-seq, hücresel kontrol eden RNA makinelerinin iç işleyişlerine benzersiz Öngörüler sağlayabilen, dinamik, heterojen ve hatta geçici RNP kompleksler RNA ayak izlerini yakalamak için etkili bir işlemdir Işlev.
Açıklamalar
Yazarların ifşa etmesi gereken hiçbir şey yok.
Teşekkürler
Bu çalışma NıH Grant GM120209 (GS) tarafından destekleniyordu. Yazarlar, Hizmetleri için OSUCCC Genomics Shared Resources Core 'a teşekkür ederler (CCC destek Grant NCı P30 CA16058).
Malzemeler
| Name | Company | Catalog Number | Comments |
| Anti-FLAG Affinity Gel | Sigma | A2220 | |
| ATP, [γ-32P]- 3,000 Ci/mmol 10 mCi/mL EasyTide, 250 µCi | PerkinElmer | BLU502A250UC | |
| BD Disposable Syringes with Luer-Lok Tips (200) | Fisher | 14-823-435 | |
| Betaine 5M | Sigma | B0300 | |
| biotin-dATP | TriLink | N-5002 | |
| biotin-dCTP | Perkin Elmer | NEL540001EA | |
| Branson Sonifier, Model SSE-1 | Branson | ||
| CircLigase I | VWR | 76081-606 | ssDNA ligase I |
| DMEM, High Glucose | ThermoFisher | 11995-065 | |
| DNA load buffer NEB | NEB | ||
| Dynabeads Protein A | LifeTech | 10002D | |
| Flp-In-T-REx 293 Cell Line | ThermoFisher | R78007 | |
| GeneRuler Low Range DNA Ladder | ThermoScientific | FERSM1203 | |
| Hygromycin B | ThermoFisher | 10687010 | |
| Mini-PROTEAN TBE Gel 10 well | Bio-Rad | 4565013 | |
| Mini-PROTEAN TBE-Urea Gel | Bio-Rad | 4566033 | |
| miRCAT-33 adapter 5′-TGGAATTCTCGGGTGCCAAGGddC-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Mirus transIT-X2 transfection reagent | Mirus | MIR 6004 | |
| Mth RNA ligase | NEB | E2610S | |
| PE1.0 5′-AATGATACGGCGACCACCGAGATCTACACT CTTTCCCTACACGACGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| PE2.0 5′-CAAGCAGAAGACGGCATACGAGATCGGTCTC GGCATTCCTGCTGAACCGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Phenol/Chloroform/Isoamyl Alcohol (25:24:1, pH 6.7, 100 mLl) | Fisher | BP1752I-100 | |
| Purple Gel Loading Dye (6x) | NEB | NEB #7025 | |
| Q5 DNA Polymerase | NEB | M0491S/L | |
| RNase I, E. coli, 1,000 U | Eppicenter | N6901K | |
| SPIN-X column | Corning | CLS8160-24EA | |
| Streptavidin beads | ThermoFisher | 60210 | |
| Superscript III (SSIII) | ThermoScientific | 18080044 | reverse transcriptase enzyme |
| SybrGold | ThermoFisher | S11494 | gold nucleic acid gel stain |
| T4 Polynucleotide Kinase-2500U | NEB | M0201L | |
| T4RNL2 Tr. K227Q | NEB | M0351S | |
| Tetracycline | Sigma | 87128 | |
| Thermostable 5´ App DNA/RNA Ligase | NEB | M0319S | |
| TruSeq_SE1 5′-pGGCACTANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE10 5′-pGGTGTTCNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE11 5′-pGGTAAGTNNNNNAGATCGGAA GAGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE12 5′-pGGAGATGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE2 5′-pGGGTAGCNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE35′-pGGTCGATNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE4 5′-pGGCCTCGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE5 5′-pGGTGACANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE6 5′-pGGTAGACNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE7 5′-pGGGCCCTNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCT TCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE8 5′-pGGATCGGNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTT CCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE9 5′-pGGACTGANNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Typhoon 5 Bimolecular Imager | GE Healthcare Life Science | 29187191 |
Referanslar
- Karousis, E. D., Nasif, S., Mühlemann, O. Nonsense-mediated mRNA decay: novel mechanistic insights and biological impact. Wiley Interdisciplinary Reviews: RNA. 7 (5), 661-682 (2016).
- Ivanov, P. V., Gehring, N. H., Kunz, J. B., Hentze, M. W., Kulozik, A. E. Interactions between UPF1, eRFs, PABP and the exon junction complex suggest an integrated model for mammalian NMD pathways. The EMBO Journal. 27 (5), 736-747 (2008).
- Papasaikas, P., Valcárcel, J. The Spliceosome: The Ultimate RNA Chaperone and Sculptor. Trends in Biochemical Sciences. 41 (1), 33-45 (2016).
- Jensen, K. B., Darnell, R. B. CLIP: Crosslinking and ImmunoPrecipitation of In Vivo RNA Targets of RNA-Binding Proteins. Methods in Molecular Biology. 488, 85-98 (2008).
- Garzia, A., Morozov, P., Sajek, M., Meyer, C., Tuschl, T. PAR-CLIP for Discovering Target Sites of RNA-Binding Proteins. mRNA Decay: Methods and Protocols. 1720, 55-75 (2018).
- Konig, J., et al. iCLIP - Transcriptome-wide Mapping of Protein-RNA Interactions with Individual Nucleotide Resolution. Journal of Visualized Experiments. (50), e2638 (2011).
- Sibley, C. R. Individual Nucleotide Resolution UV Cross-Linking and Immunoprecipitation (iCLIP) to Determine Protein-RNA Interactions. RNA Detection: Methods and Protocols. 1649, 427-454 (2018).
- Wheeler, E. C., Van Nostrand, E. L., Yeo, G. W. Advances and challenges in the detection of transcriptome‐wide protein-RNA interactions. Wiley Interdisciplinary Reviews: RNA. 9 (1), (2018).
- Singh, G., et al. The Cellular EJC Interactome Reveals Higher-Order mRNP Structure and an EJC-SR Protein Nexus. Cell. 151, 750-764 (2012).
- Singh, G., Ricci, E. P., Moore, M. J. RIPiT-Seq: A high-throughput approach for footprinting RNA:protein complexes. Methods. 65, 320-332 (2014).
- Mabin, J. W., et al. The Exon Junction Complex Undergoes a Compositional Switch that Alters mRNP Structure and Nonsense-Mediated mRNA Decay Activity. Cell Reports. 25 (9), 2431-2446 (2018).
- Gehring, N. H., Lamprinaki, S., Kulozik, A. E., Hentze, M. W. Disassembly of Exon Junction Complexes by PYM. Cell. 137 (3), 536-548 (2009).
- Dostie, J., Dreyfuss, G. Translation Is Required to Remove Y14 from mRNAs in the Cytoplasm. Current Biology. 12 (13), 1060-1067 (2002).
- Zünd, D., Gruber, A. R., Zavolan, M., Mühlemann, O. Translation-dependent displacement of UPF1 from coding sequences causes its enrichment in 3' UTRs. Nature Structural & Molecular Biology. 20 (8), 936-943 (2013).
- Gangras, P., Dayeh, D. M., Mabin, J. W., Nakanishi, K., Singh, G. Cloning and Identification of Recombinant Argonaute-Bound Small RNAs Using Next-Generation Sequencing. Argonaute Proteins: Methods and Protocols. 1680, 1-28 (2018).
- Heyer, E. E., Ozadam, H., Ricci, E. P., Cenik, C., Moore, M. J. An optimized kit-free method for making strand-specific deep sequencing libraries from RNA fragments. Nucleic Acids Research. 43 (1), 2 (2015).
- Cameron, V., Uhlenbeck, O. C. 3'-Phosphatase activity in T4 polynucleotide kinase. Biochemistry. 16 (23), 5120-5126 (1977).
- Ricci, E. P., et al. Staufen1 senses overall transcript secondary structure to regulate translation. Nature Structural & Molecular Biology. 21 (1), 26-35 (2014).
- Lackner, D. H., et al. A generic strategy for CRISPR-Cas9-mediated gene tagging. Nature Communications. 6, 10237 (2015).
- Metkar, M., et al. Higher-Order Organization Principles of Pre-translational mRNPs. Molecular Cell. 72 (4), 715-726 (2018).
- Giudice, G., Sánchez-Cabo, F., Torroja, C., Lara-Pezzi, E. ATtRACT-a database of RNA-binding proteins and associated motifs. Database: The Journal of Biological Databases and Curation. 2016, (2016).
- Paz, I., Kosti, I., Ares, M., Cline, M., Mandel-Gutfreund, Y. RBPmap: a web server for mapping binding sites of RNA-binding proteins. Nucleic Acids Research. 42, 361-367 (2014).
- Sundararaman, B., et al. Resources for the Comprehensive Discovery of Functional RNA Elements. Molecular Cell. 61 (6), 903-913 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır