Method Article
Identificação de pegadas de RNA: complexos proteicos via imunoprecipitação de RNA em tandem seguido de sequenciamento (RIPiT-Seq)
Neste Artigo
Resumo
Aqui, nós apresentamos um protocolo para enriquecer locais de ligação do RNA endógeno ou "pegadas" de complexos do RNA: proteína (RNP) das pilhas de mamífero. Essa abordagem envolve duas imunoprecipitações de subunidades de RNP e, portanto, é apelidado de imunoprecipitação de RNA em tandem (RIPiT).
Resumo
A imunoprecipitação do RNA em tandem (RIPiT) é um método para enriquecer pegadas do RNA de um par de proteínas dentro de um complexo do RNA: proteína (RNP). O RIPiT emprega duas etapas de purificação. Primeiramente, a imunoprecipitação de um subunidade etiquetado de RNP é seguida pela digestão suave de RNase e pela eluição não-desnaturando subseqüente da afinidade. Uma segunda imunoprecipitação de outra subunidade da RNP permite o enriquecimento de um complexo definido. Depois de uma eluição de desnaturação de RNAs e proteínas, as pegadas de RNA são convertidas em bibliotecas de sequenciamento de DNA de alta produtividade. Ao contrário do mais popular ultravioleta (UV) reticulação seguido por imunoprecipitação (clip) abordagem para enriquecer RBP sites de ligação, RipIt é UV-reticulação independente. Daqui o ripit pode ser aplicado às proteínas numerosas atuais no de do RNA e além de que são essenciais à regulação do RNA mas não contatam diretamente o RNA ou o UV-Crosslink mal ao RNA. As duas etapas da purificação em RIPiT fornecem uma vantagem adicional de identificar os locais obrigatórios onde uma proteína do interesse actua na parceria com um outro cofactor. A dupla estratégia de purificação também serve para melhorar o sinal, limitando o fundo. Aqui, fornecemos um procedimento passo-sábio para executar o RIPiT e para gerar bibliotecas de sequenciamento de alto débito de pegadas de RNA isoladas. Também esboçamos as vantagens e aplicações do RIPiT e discutimos algumas das suas limitações.
Introdução
Dentro das células, o RNA existe no complexo com proteínas para formar os complexos RNA: protein (RNPs). RNPs são montados em torno das proteínas de ligação do RNA (RBPs, aqueles que ligam diretamente o RNA) mas igualmente compreendem de non-RBPs (aqueles que ligam RBPs mas não o RNA), e são frequentemente dinâmicos na natureza. Rbps e seus cofatores funcionam coletivamente dentro de rnps para executar funções regulatórias. Por exemplo, na via de deterioração de mRNA mediada por disparates (NMD), as proteínas UPF (UPF1, UPF2 e UPF3b) reconhecem o Ribossome prematuramente finalizado. Cada uma das proteínas de UPF pode ligar ao RNA, mas é somente quando montam junto que um complexo ativo de NMD começa a se formar. Dentro deste complexo, UPF1 é ativado mais pela fosforilação por um SMG1 não-RBP, e essa ativação UPF1 eventualmente leva ao recrutamento de fatores Indutores de decaimento de mRNA1,2. Neste exemplo, o RBPs requer cofatores não RBP para recrutamento e ativação do complexo RNP que aciona a NMD. No entanto, outra propriedade de RNPs é a sua heterogeneidade composicional. Considere o spliceosome, que existe em distintos complexos de E, A, B ou C. Os complexos spliceossoma diferentes têm as proteínas de sobreposição e distintas3. Para estudar as funções da RNP, é importante elucidar quais RNAs são vinculados por uma RBP e suas proteínas associadas. Existem muitos métodos para fazer isso, com cada abordagem tendo suas distintas vantagens e desvantagens4,5,6,7.
Os métodos amplamente populares para identificar sítios de ligação da RBP — reticulação seguida de imunoprecipitação (CLIP) e suas várias variações-dependem da luz ultravioleta (UV) para vincular uma RBP ao RNA8. Entretanto, esta não é uma aproximação eficaz para non-RBPs dentro de RNPs, que não contatam o RNA diretamente. Aqui, nós descrevemos uma aproximação alternativa que seja aplicável aos RBPs e não-RBPs igualmente, para isolar e identificar seus locais da ligação do RNA. Essa abordagem denominada imunoprecipitação de RNA em tandem (ripit) consiste em duas etapas de imunoprecipitação, que auxiliam na obtenção de maior especificidade em relação a uma única purificação (Figura 1)9,10. Como as etapas individuais da imunoprecipitação (IP) podem ser executadas em uma mais baixa rigor em comparação ao grampo, o ripit não depende da disponibilidade dos anticorpos que podem suportar a presença de detergentes fortes durante a imunoprecipitação. A vantagem a mais original de RIPiT é a habilidade de alvejar duas proteínas diferentes em duas etapas da purificação; Isto fornece uma maneira poderosa de enriquecer um complexo composicionalmente distinto de RNP de outros complexos similares11.
Pequenas variações no procedimento do RIPiT podem aumentar ainda mais o enriquecimento de RNP. Por exemplo, algumas interações RNA-proteína ou proteína-proteína dentro de RNPs são transitórios e pode ser difícil purificar eficientemente pegadas de tais complexos. Para estabilizar essas interações, os RNPs podem ser reticulados dentro de células com formaldeído antes da lise celular e do RIPiT. Por exemplo, nós observamos que uma interação fraca entre o fator de núcleo complexo da junção do exon (EJC), EIF4AIII e o fator da desmontagem de EJC, PYM12 pode ser estabilizado com o tratamento do formaldehyde tal que mais pegadas do RNA são enriquecidas (dados não mostrado). Antes da colheita da pilha e do RIPiT, as pilhas podem igualmente ser tratadas com as drogas para estabilizar ou enriquecer RNPs em um estado particular. Por exemplo, ao estudar proteínas que são removidas do mRNA durante a tradução (por exemplo, a EJC UPF114), o tratamento com inibidores de tradução como a puromicina, cicloheximida ou harringtonina pode levar ao aumento da ocupação de proteínas em RNAs.
A quantidade de RNA recuperado de RIPiT é geralmente baixa (0.5-10 pmoles, isto é, 10-250 ng RNA considerando um comprimento médio do RNA de 75 NT). A principal razão para isso é que apenas uma pequena fração de uma determinada proteína está presente no complexo com outras proteínas dentro de RNPs (qualquer "livre" proteína IP'ed na primeira etapa é perdida durante o segundo IP). Para gerar bibliotecas de RNA-Seq deste RNA, nós igualmente esboçamos aqui uma adaptação do protocolo previamente publicado apropriado para tais baixas entradas do RNA15,16 (Figura 2), que rende amostras prontas de sequenciamento de alta produtividade em 3 Dias.
Protocolo
1. estabelecimento de linhas de célula HEK293 estáveis expressando tetraciclina-inducible FLAG-Tagged proteína de interesse (POI)
- Sementes HEK293 células com um local de meta de recombinação de FLP (FRT) de forma estável em uma densidade de 10 x 104 células/ml em meio de crescimento (Dulbecco ' s modificado médio de Eagle [DMEM] + 10% soro fetal bovino [FBS] + 1% penicilina-estreptomicina [Penn/Strep]) em 6- placas bem. Permita que as pilhas cresçam durante a noite em uma incubadora humidificada em 37 ° c e em 5% CO2 (condições de crescimento padrão para todas as etapas subseqüentes).
- No dia seguinte, as células devem ser ~ 70% confluentes. De acordo com o protocolo do reagente do transfection, transfecção o local FRT-contendo HEK293 pilhas com uma relação 9:1 de pcDNA5-teto-Flag-POI: pOG44.
Nota: A tag FLAG tem o motivo da sequência DYKDDDDK (D = ácido aspático, Y = tirosina e K = lisina). - Após 24 h, comece a seleção antibiótica. Remova a mídia e adicione um meio de crescimento fresco suplementado com 100 μg/mL de higromicina. Dentro 72 h, as pilhas untransfected devem começar a morrer.
- Cada 48-72 h, mudança de mídia de crescimento e suplemento com hygromycin fresco.
- Após ~ 2 semanas da seleção, as colônias discretas de pilhas estàvel transfected começarão a aparecer. Uma vez que as colônias são visíveis a olho nu, adicione 1 mL de tripsina à placa e incubar por 5 min a 37 ° c. Reressuscitem as células em DMEM, transfira as células para uma nova placa de 10 cm e ajuste o volume para um meio de crescimento fresco de 10 mL suplementado com 100 μg/mL de higromicina. Permita que a placa alcangue a confluência de ~ 80% para expandir mais pilhas para preparar estoques permanentes.
- Determine a quantidade de tetraciclina (Tet) necessária para obter o nível ideal de expressão FLAG-POI para o experimento. Cresça pilhas no formato 12-well e conduza um titulação de Tet entre 0-1000 ng/ml escala para 16-24 h. realize manchas ocidentais nas amostras de titulação com anticorpos contra o POI.
Nota: Para proteínas de até ~ 60 kDa, a FLAG-Tagged e a cópia endógena da proteína pode ser resolvida na eletroforese de gel de dodecilo sulfato-poliacrilamida de sódio (SDS-PAGE) para comparar os níveis de expressão de FLAG-POI à sua contraparte endógena. Para proteínas maiores, a intensidade do sinal em amostras induzidas por Tet pode ser comparada à amostra não induzida. A solução de estoque de tetraciclina pode ser preparada em 1 mg/mL em etanol a 100%. As diluições do estoque para o trabalho da cultura de pilha devem estar na água estéril ou no Saline fosfato-tamponado (PBS). O estoque de tetraciclina deve ser preparado fresco uma vez a cada mês.
2. cultivo de células para a indução de tetraciclina e procedimento de RIPiT
- Semente estàvel integrada bandeira-POI HEK293 em uma densidade de 3 x 105 Cells/ml no meio do crescimento em placas de 15 cm. Permitir que as células cresçam a 37 ° c e 5% CO2.
Nota: Em geral, três a 5 15 cm placas renderá ~ 2-20 pmol de pegadas de RNA dependendo da abundância do RNP de interesse. Se RNPs será formaldeído reticulado e purificado condições rigorosas, então duas vezes mais entrada pode ser necessária. - Adicione a tetraciclina à concentração óptima pré-determinada aos meios para induzir a expressão do FLAG-POI (ver passo 1,6) 16-24 h antes de as células serem colhidas.
Nota: Não é necessário mudar o meio de crescimento. As células devem estar prontas para colher cerca de 72 h após a semeadura ou quando as placas são ~ 80% confluente. É importante evitar que as células se tornem confluentes.
3. colheita da pilha, tratamento do formaldehyde, e lysis da pilha
- Lave suavemente as células monocamada com 15 mL de PBS refrigerada para cada placa de 15 cm. Em seguida, raspe as células em 30 mL de PBS. Se as células foram tratadas com drogas antes da colheita, suplemento de PBS com a droga. Colete todas as células em 50 mL de tubo cônico.
Nota: Para preservar as interações fracas, as células podem ser reticuladas com formaldeído: Adicionar formaldeído à suspensão celular da etapa 3,1 para uma concentração final de 0,1% e incubar em um rocker à temperatura ambiente por 10 min. adicionar 3 mL do tampão de têmpera (tabela 1 ) e rock por 5 minutos adicionais. - Células da pelota em 400 x g por 10 min a 4 ° c e descartar sobrenadante.
- Células de lyse em 4 mL de tampão de Lise hipotônica gelada (HLB; Tabela 1). Use uma pipeta P1000 para resuspender as células. Transferir para um tubo de 5 mL e incubar lisado no gelo durante 10 min.
- Coloque o lisado em um banho de gelo e proceda a 10% de amplitude para 30 s com 1 s pulsos com 2 s pausas. Em seguida, ajuste a concentração de sal para 150 mM adicionando 108 μL de NaCl de 5 M.
Nota: As amostras reticuladas de formaldeído podem estar sujeitas a Lise e purificação mais rigorosas, incluindo 0,1% de SDS e desoxicolato de sódio no tampão de Lise. - Limpe o lisado por centrifugação a 21.000 x g durante 10 min a 4 ° c. Colete 20 μL de sobrenadante (extrato celular) em um tubo rotulado para análise de Western blot dos níveis de proteína na entrada (ver Figura 3a).
Nota: Quando o lisado estiver no centrifugador, os grânulos do agarose da bandeira podem ser lavados (veja a etapa 4,1). Os grânulos do agarose da bandeira devem ser 3x Pre-washed em 4 ml do amortecedor isotônica Ice-Cold da lavagem (isowb, tabela 1).
4. SINALIZAR imunoprecipitação
- Aplique o sobrenadante restante da etapa 3,5 a 750 μL de grânulos pré-lavados do agarose da bandeira em um tubo de 5 mL (o volume da cama de grânulos lavados do agarose da bandeira será 375 μL). Incubar as esferas do agarose da bandeira e o extrato da pilha para 1-3 h em 4 ° c com mistura delicada.
Nota: Este volume de grânulos do agarose da bandeira deve ser suficiente para uma largo-escala das proteínas, mas pode ser aperfeiçoado, se necessário. - Grânulos do agarose da bandeira da pelota pela centrifugação em 400 x g por 1 minuto em 4 ° c. Colete 20 μL do sobrenadante em um tubo rotulado para análise de Western blot dos níveis de proteína no extrato de células empobrecido (ver Figura 3a). Descarte o sobrenadante restante.
- Para lavar a bandeira de agarose contas, adicione 4 mL de IsoWB e ressuscitem. Grânulos da pelota em 400 x g por 1 minuto em 4 ° c. Remova cuidadosamente o sobrenadante. Repita 4x.
Nota: Para as lavagens rigorosas do formaldeído reticulado IPS, 0,1% cada um de SDS e desoxicolato de sódio pode ser incluído no tampão de lavagem para os dois primeiros passos de lavagem.
5. RNase I digestão
- Diluir RNase I para 0,002-0,01 unidades/mL em 750 μL de IsoWB (a concentração adequada para tamanhos de pegada desejados deve ser determinada empiricamente). Adicionar IsoWB-RNase I para as esferas de agarose de bandeira lavada e incubar com mistura suave a 4 ° c durante 10 min.
- Grânulos da pelota em 400 x g por 1 minuto em 4 ° c. Colete 20 μL de sobrenadante (RNase I eluição) em um tubo rotulado para análise de Western Blot. Rejeite IsoWB-RNase I e o agarose da bandeira da lavagem Perla 4x com IsoWB como descrito na etapa 4,3.
6. eluição de afinidade
- Prepare um estoque de tampão de eluição (peptídeo FLAG em 250 ng/mL em IsoWB). Aplique 375 μL de tampão de eluição a esferas de agarose de FLAG e agite suavemente a 4 ° c por 1-2 h. grânulos de agarose da bandeira da pelota e colete uma alíquota de 15 μL da eluição para o borrão ocidental das proteínas no IP da bandeira (veja Figura 3a).
Nota: Quando a eluição de afinidade está em andamento, a seção 7 pode ser realizada.
7. grânulo magnético-conjugação de anticorpos
- Lave 50 μL de grânulos magnéticos (i.e., Dynabeads; Tabela de materiais) 3x em 1 mL de IsoWB em 1,5 mL de tubo. Ressuscitem grânulos magnéticos em 100 μL de tampão de conjugação. Adicione a quantidade apropriada de anticorpo (a quantidade exata de anticorpo a usar-se para o IP precisará de ser determinada empiricamente para cada anticorpo).
Nota: As esferas magnéticas da proteína A são ideais para os anticorpos produzidos no coelho visto que os grânulos magnéticos da proteína G são mais apropriados para anticorpos do rato. Magnetic contas de compatibilidade gráfico está disponível no site do fornecedor para escolher contas apropriadas para cada anticorpo. - Lave os grânulos magnéticos 2x em tampão de conjugação (tabela 1). Incubar a mistura à temperatura ambiente durante pelo menos 10 min. Ressuspender os grânulos magnéticos em 375 μL de tampão de diluição do RIPiT. Armazene no gelo até o próximo passo.
8. segunda imunoprecipitação
- Aplique a eluição restante da afinidade da bandeira da etapa 6,1 aos grânulos magnéticos acoplados aos anticorpos de encontro à proteína do interesse. Incubar com mistura delicada em 4 ° c para 1-2 h. Capture grânulos magnéticos em um ímã e colete 15 μl do sobrenadante para a análise de proteínas não acoplado através do borrão ocidental. Lave os grânulos magnéticos 7x com 1 mL de IsoWB.
9. eluição de desnaturação
- Adicione 100 μL de tampão de amostra transparente (tabela 1) a grânulos magnéticos e ressuspender com uma pipeta P200. Incubar no gelo por 10 min. Mexa suavemente para Ressuspender as contas periodicamente.
- Capture grânulos magnéticos em um ímã e colete 15 μL da eluição para a análise das proteínas na eluição do RIPiT através do borrão ocidental (veja Figura 3a). Transfira a eluição restante em um tubo 1,5 mL novo etiquetado.
Nota: Se as amostras foram reticuladas com formaldeído, as amostras devem ser incubados a 65 ° c por 1 h para inverter a reticulação. - Realize manchas ocidentais em amostras coletadas em várias etapas (entrada, depleção de IP de FLAG, sinalizador IP, segunda depleção de IP, segunda eluição de IP). Blot com anticorpos contra as duas proteínas de isca, seus outros interatores se conhecidos, e pelo menos uma RBP não-interagindo como um controle negativo (Figura 3a).
10. extração do RNA e cura da extremidade
- Para a eluição de RIPiT, adicione 320 μL de ddH RNase-livre2O, 400 μL de álcool isoamílico de fenol-clorofórmio (PCIAA, pH 4,5) e vórtice por 30 s e gire à temperatura ambiente em 12.000 x g por 5 min. colete 350 μL de fase aquosa em um tubo separado. Adicionar 35 μL de acetato de sódio a 3 M, 1 μL de 1 M de MgCl2, 10 μg de glicogênio e 1 ml de etanol a 100%. Incubar durante a noite a-20 ° c.
- Para o RNA da pelota, centrifugue em 12.000 x g por 30 minutos em 4 ° c. Lavagem de RNA em 70% etanol.
- Para remover 3 ' fosfato esquerdo no RNA após a clivagem de RNase I, Ressuspender a pelota do RNA em 17 μL de DDH RNase-livre2O, e adicionar 2 μl do amortecedor da quinase do polinucleotídeo de 10x T4 (PNK) (tabela dos materiais) e de 1 μL de T4 PNK. Incubar a 37 ° c durante 30 min.
Nota: A atividade de 3 ' fosfatase de T4 PNK tem a atividade óptima em pH 617. O tampão da reação de PNK é aperfeiçoado para a atividade de 5 ' quinase de T4 PNK e tem um pH de 7,6. Ao ajustar o pH da reação de cura final foi tentado, este passo pode ser mais otimizado. - Adicionar 380 μL de ddH RNase-Free2o e 400 ΜL de Pciaa pH 4,5 para o tubo. Vortex por 30 s, centrifugador a 12.000 x g durante 5 min. recolha a fase aquosa e adicione 35 μL de acetato de sódio a 3 m, 1 μL de 1 m de MgCl2, 10 μg de glicogênio e 1 ml de 100% de etanol.
- Incubar durante a noite a-20 ° c. Pellet e lavagem RNA com 70% etanol como acima. Ressuspender o RNA em 4,5 μL de água livre de RNase.
11. estimativa do tamanho e abundância da pegada do RNA
-
Um RIPiT bem-sucedido é esperado para produzir 1 pmol ou mais de fragmentos de RNA. Para quantificar o rendimento real, transfira 0,7 μL do RNA do RIPiT (~ 1/6 do rendimento total) em um tubo novo. Adicionar 2 μL de tampão PNK 10x T4, 1 μL de 1 mM ATP, 40 μCi γ32P-ATP (0,5 − 1,0 μl do estoque) e 1 μL de T4 PNK. Ajustar o volume para 10 μL e incubar a 37 ° c durante 30 min.
- Em reações paralelas de PNK, rotule uma escada do ADN da escala baixa e 0,1 pmol de um oligo sintético do RNA ou do ADN (20-40 NT) para usar padrões do tamanho e da quantidade.
- Resolva o RNA/ADN etiquetado no gel da Urea-página de 26% (20 x 27 x 0,45 milímetros3). O gel deve ser Pre-Run por 30 minutos em 35 w. poços nivelado antes de Pre-Run e antes de carregar amostras e funcionar em 35 W até que a parte dianteira azul do corante do bromofenol quase alcangou a extremidade do gel.
- Retire cuidadosamente o gel das placas de vidro para uma peça de papel de filtro de 8 x 11 polegadas. Com gel na parte superior do papel, coloc no instrumento de secagem do gel e cubra-o com uma parte de envoltório plástico. Gel seco a 80 ° c por 1 h com vácuo.
- Expor o gel secado a um phosphoscreen durante a noite ou até que o sinal adequado esteja detectado. Imagem fosfoscreen. O RNA de boa qualidade de um RIPiT deve aparecer como um esfregaço na pista, com bandas de destaque mínimas (Figura 3B). Para quantificar o RNA, compare a intensidade do sinal dos fragmentos de RNA do tamanho desejado na pista de RIPiT ao sinal de 0,1 pmol do oligo sintético etiquetado.
Nota: Alternativamente, o tamanho e as quantidades da pegada do RNA podem ser verificados usando um Bioanalyzer da elevado-sensibilidade (Figura 3C).
12. ligadura do adaptador
- Prepare o RNA do RIPiT de tal forma que pelo menos 3 pmol de RNA seja dissolvido em 3,8 μL de água.
- Em um tubo da reacção em cadeia do polymerase de 0,2 mL (PCR), combine 3,8 μL do RNA, 1 μL do adaptador de miR-CAT-33 pre-adenylated (7 μM) (tabela dos materiais). Incubar a mistura num termociclador a 65 ° c durante 10 min, 16 ° c durante 5 min e, em seguida, segure a 4 ° c.
Nota: O linker pré-adenylated pode ser requisitado do serviço da síntese do oligo, ou um oligo unadenylated feito encomenda do ADN de todo o serviço da síntese do oligo pode ser adenylated usando a ligase do RNA de mth (tabela dos materiais) e o gel purified. - Para o mesmo tubo, adicione 1,5 μL de tampão de ligase de RNA de 10x T4, 7,5 μL de 50% de polietileno glicol 8000 (PEG-8000), 0,75 μL de ditiothreitol de 20 mM (TDT) e 0,45 μL de T4 RNL2 TR. K227Q (tabela de materiais).
Nota: 50% PEG-8000 vem com RNA ligase e tampão comprado. As soluções PEG-8000 são viscosas e devem ser pipetadas lentamente. - Incubar a reação no Thermal cycler em 30 ° c por 6 h, o calor inativar a ligase em 65 ° c por 10 minutos, a seguir prenda em 4 ° c.
13. transcrição reversa
- Para o tubo com a mistura de ligadura do passo 12,4, adicionar 11,25 μL de 4x dos desoxinucleótidos trifosfato (dNTP) Mix, que contém uma mistura de dNTPs regulares e biotinylated (ver tabela 1), 1,0 μL de 10 μm RT primers (tabela de materiais), e 6,8 μL de Água RNase-livre. Incubar a 65 ° c durante 5 min e, em seguida, segure a 4 ° c.
- Transfira os tubos para o gelo e adicione 9,0 μL de tampão de primeira vertente 5x (FS) sem MgCl2 (tabela 1), 2,25 ΜL de 100 mM DTT, 1,2 μL de enzima de transcriptase reversa para um volume final de 45 μl (tabela de materiais).
- Incubar num termociclador a 55 ° c por 30-60 min. o calor inactiva a transcriptase reversa a 70 ° c durante 15 min e mantém a amostra a 4 ° c.
14. purificação do produto RT
- Adicionar 45 μL de tampão de carga de ureia 2x (tabela 1) à reacção RT. Diluir 1 μg de uma escada de ADN de baixa gama em 45 μL e adicionar 45 μL de tampão de carga de 2x ureia.
- Prepare 10% de ureia-gel de página (20 x 28 x 0,15 cm3; Tabela 1) com 8-bem pente. Usando a seringa ou o Pipet, os poços nivelados com 0.5 x Tris/Borate/EDTA (TBE) tampão.
Nota: Os géis caseiros acima oferecem uma melhor resolução na separação do produto RT estendido do primer RT não estendido. Como alternativa, os géis pre-cast da ureia-página podem igualmente ser usados (tabela dos materiais). Como os géis pré-fabricados permitem volumes máximos menores por poço, para que as amostras precisem ser divididas em vários poços. Os géis pré-fabricados devem ser executados a 150 − 200 V. - Pre-Run o gel em 35 w por 30 min. poços de descarga novamente, amostras de carga e executar gel em 35 W até bromofenol frente corante azul migrou para cerca de 1 polegada a partir do final do gel.
Nota: Use o dissipador de calor do metal durante o Pre-Run e o funcionamento final para impedir o superaquecimento do gel. - Manchar o gel para 5 min em 1x ouro ácido nucleico gel mancha solução preparada em 0,5 x TBE. Este corante é sensível à luz, para evitar a exposição à luz.
- Gel de imagem no scanner fluorescente para fins de documentação usando 520 nm excitação e 580 nm filtros de emissão. Se um scanner fluorescente não estiver disponível, use um transiluminador de luz azul. O produto RT deve aparecer como um esfregaço a partir do primer RT não estendido (Figura 4).
Nota: Embora o gel do ácido nucleico do ouro que mancha a tintura seja visualizado facilmente em um doc do gel com fonte da UV-luz, é vital não expor o produto valioso do RT ao UV para impedir dano. - Visualize o gel em um transiluminador de luz azul e extirpar o produto RT do gel. Recomenda-se cortar o ADN com as extensões que variam de 30-200 NT (Figura 4). Coloc as partes extirpada do gel em uma superfície limpa e mince a fatia em partes pequenas para aumentar a área de superfície. Transfira cuidadosamente para um tubo de 1,5 mL e adicione 800 μL de tampão de eluição de ADN (tabela 1).
- Incubar as partes do gel com mistura delicada com o amortecedor da eluição do ADN sobre a noite na temperatura ambiente.
- Separe o tampão de eluição do gel passando a pasta através de uma coluna de filtro de acetato de celulose (tabela de materiais) colocada num tubo de recolha de 2 ml.
- No tubo 1,5 mL, lave 10 μL de grânulos magnéticos de estreptavidina com 500 μL de tampão de lavagem do grânulo de estreptavidina (tabela 1). Repita para três laves totais. Não deixe os grânulos secarem. Ressuspender grânulos em 10 μL de tampão de eluição de DNA (tabela 1).
- Transfira o tampão de eluição separado das partes do gel na etapa 14,8 ao tubo que contem as contas magnéticas estreptavidina lavadas.
- Incubar com mistura suave durante pelo menos 8 h à temperatura ambiente. Capture grânulos em um ímã, remova sobrenadantes e ressuscite grânulos magnéticos em 10 μL de água RNase-livre e transfira a um tubo do PCR de 0,2 mL.
15. circularização do produto RT
- Os produtos de RT capturados em grânulos de estreptavidina molde quando encadernado em grânulos. Para a pasta de cordão magnético, adicione 2,0 μL de tampão de reação de circularização 10x, 1,0 μL de 1 mM ATP, 1,0 μL de 50 mM MnCl2, 4,0 μL de 5 M de betaína, 1,0 μL de ssDNA ligase I (tabela de materiais) e 1,0 μL de água livre de RNase.
- Incubar a reacção de circularização num termociclador a 60 ° c durante 4 h. o calor inactiva a ligase de ssDNA I aquecendo-se a 80 ° c durante 10 min e, em seguida, segure a 4 ° c.
16. teste de PCR
- Antes de proceder a uma PCR em grande escala, use uma porção de produto circularizado para determinar o número ideal de ciclos de amplificação para cada amostra. Esta etapa ajuda a evitar a sobreamplificação e limitar os artefatos de PCR, pois os componentes da reação de PCR se limitam a ciclos de PCR maiores.
- Prepare uma reacção de PCR de 45 μL com 4,0-6,0 μL do produto circularizado a partir do passo 15,2, 9,0 μL de tampão de reacção 5x, 0,9 μL 10 M dNTPs, 2,25 μL de 10 μM PE 1.0 Primer (tabela de materiais), 2,25 μL de 10 μm PE 2.0 Primer (tabela de materiais) e 0, 45 μL de DNA polimerase de alta fidelidade (tabela de materiais) e água.
- Misture bem a reacção e divida em 3 15 μL de reacções. Cada uma destas três reacções estará sujeita a um número variável de ciclos de PCR. Espera-se que o número ideal de ciclos entre 7 e 14. Então, execute PCRs de teste para 8, 11 e 14 ciclos.
- Use as seguintes condições do PCR: 98 ° c-30 s; 98 ° c-5 s; 65 ° c-10 s; 72 ° c-15 s; 72 ° c-2 min; 12 ° c-segurar.
- Adicione 3 μL de corante de carregamento de gel 6x e resolva em 10% gel de página nativa até que a frente de corante azul migre 3/4 do gel. Manchar o gel usando a mancha e a imagem do gel do ácido nucleico do ouro como nas etapas 14,4 e 14,5 (Figura 5).
- Para escolher o número ideal de ciclos do PCR, compare produtos do PCR do número crescente de ciclos. Escolha o número do ciclo que produz a maior quantidade de produto do tamanho esperado sem artefatos de superamplificação (por exemplo, esfregaço de DNA muito maior do que o esperado do produto, e onde não há depleção apreciável dos primers PE 1.0 e PE 2.0 é visto (ver seta vermelha em Figura 5).
17. PCR em grande escala
- Prepare uma reacção de PCR de 45 μL, como na etapa 16,2 e repita o PCR. Resolva o PCR na página nativa de 10% em 150 V, mancha com a mancha do gel do ácido nucleico do ouro 1x e a imagem em um transilluminator claro azul.
- Extirpar o produto de PCR do gel e transferir para uma seringa de 3 mL. Use a seringa para esmagar o gel e a extrusão em um tubo de 1,5 mL.
Nota: O produto RT não estendido em cima da circularização rende o produto do PCR de 151 BP. Assim, nesta etapa um deve dimensionar os produtos seleto que são maiores do que 151 BP (Figura 6). - Adicionar 900 μL de tampão de eluição de ADN e incubar à temperatura ambiente durante a noite com mistura suave.
- Transfira a pasta de gel para uma coluna de filtro de acetato de celulose colocada num tubo de recolha de 2 mL. Gire em 12.000 x g por 3 min, coletando sobrenadante em um tubo fresco.
- Adicione outro 400 μL de tampão de eluição de ADN ao gel esmagado e transfira para um tubo de 1,5 mL. Incubar com mistura delicada por 4 horas adicionais para uma segunda eluição.
- Piscina todas as elutions e dividido em 3 tubos com 400 μL cada. Precipitate DNA adicionando 1 mL de 100% de etanol e 10 μg de glicogênio. Vortex e incubar pelo menos 2 h a-20 ° c.
- Pellet DNA em 12.000 x g por 30 min a 4 ° c. Lave a pelota do ADN com 70% de etanol.
- Retire cuidadosamente todo o etanol através de pipetagem e rapidamente ressuscitar o pellet de ADN em 20 μL de água.
Nota: Nesta fase, é importante não deixar a pelota do DNA se tornar seca, como a secagem de DNA pode desnaturá-lo. - Use uma parcela pequena da amostra do ADN para determinar o tamanho e a concentração do produto do PCR através do fluorômetro e do Bioanalyzer elevado do ADN da sensibilidade. As amostras agora podem ser submetidas para sequenciamento em uma das plataformas.
- As leituras seqüenciadas podem ser processadas (por exemplo, remoção do adaptador, aparar para manter sequências > 30 Pontuação PHRED), alinhadas ao genoma de referência e visualizadas em um navegador como o navegador do genoma UCSC (Figura 7).
Resultados
Um RIPiT bem sucedido conduzirá à imunoprecipitação de ambas as proteínas do interesse e de outras proteínas de interação conhecidas, e a ausência de proteínas não-interagindo. Como observado na Figura 3a, ambos os magoh e EIF4AIII foram detectados na eluição do RIPIT, mas HNRNPA1 não foi (pista 6). Paralelamente, as pegadas de RNA que foram copurificadas com os complexos RNP foram detectadas via autoradiografia (Figura 3B) ou Bioanalyzer (Figura 3C). O tratamento com puromicina é esperado para aumentar a ocupação de EJC no RNA, e um sinal mais forte da pegada do RNA foi observado no ripit Tratado puromicina na Figura 3B (compare as pistas 2 e 3). A geração de amostras para seqüenciamento profundo requer a ligação de um adaptador ao RNA e, em seguida, reverter a transcrição do RNA para o DNA usando uma cartilha que reagrupa a seqüência do adaptador. A etapa reversa da transcrição incorpora nucleotides biotinylated, para a purificação do produto reverso da transcrição. Após a transcrição reversa, o produto deve ser separado do adaptador não estendido pela ureia-PAGE (Figura 4). O produto reverso da transcrição é molde então e PCR amplificado. O número apropriado de ciclos do PCR não deve overamplify o produto molde. A superamplificação resultará na depleção de primer e no produto de PCR aberrante (ver Figura 5, pista 4). O número de ciclos com maior amplificação sem evidência de superamplificação é mais adequado para o uso de PCR em grande escala (Figura 5 Lane 3 e Figura 6).

Figura 1 : Esquema delineando as etapas principais no RIPiT. Por favor clique aqui para ver uma versão maior desta figura.
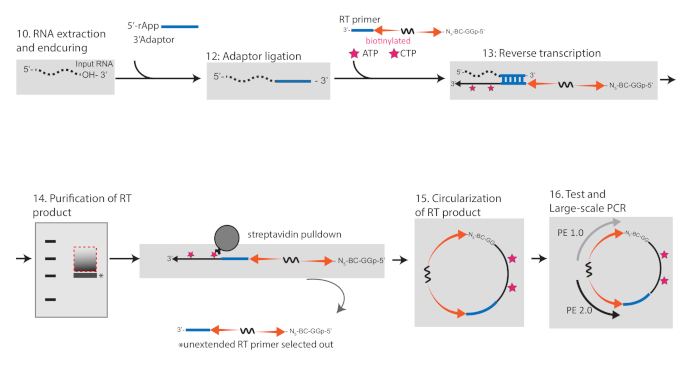
Figura 2 : Esquemático representando o fluxo de trabalho para conversão de RNA RIPiT em bibliotecas para sequenciamento de alta taxa de transferência. Por favor clique aqui para ver uma versão maior desta figura.
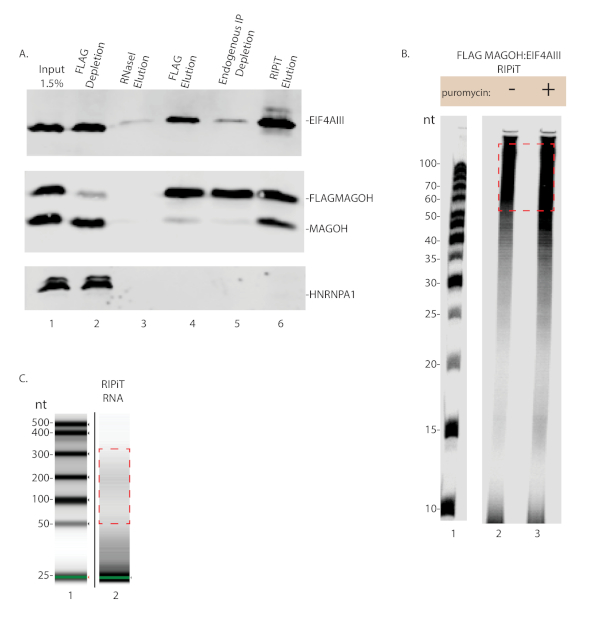
Figura 3 : Estimativa de RNA e rendimento protéico do RIPiT. (A) mancha ocidental das proteínas purificadas de cada etapa principal no procedimento de ripit. (B) imagem autoradiografia de pegadas de RNA de uma bandeira-magoh: EIF4AIII ripit comparando células de puromicina tratadas e não tratadas. A caixa vermelha indica o tamanho da pegada do RNA que será convertido finalmente em bibliotecas de sequenciamento. (C) perfil de RNA eluído do ripit como na pista 2 no painel B quando visualizado utilizando um bioanalisador. Por favor clique aqui para ver uma versão maior desta figura.

Figura 4 : O produto reverso da transcrição (RT) resolveu em um gel da Urea-página de 10% e manchado com mancha do gel do ácido nucleico do ouro. A caixa vermelha indica a região do gel extirpada para a purificação do gel de produtos estendidos de RT. Por favor clique aqui para ver uma versão maior desta figura.
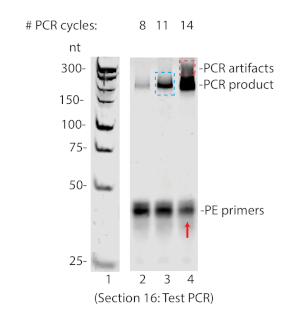
Figura 5 : Teste de PCR resolvido em 8% não-desnaturação Page. Observe os produtos de PCR aberrantemente grandes que aparecem em 14 ciclos (caixa vermelha) e a depleção paralela de primers (seta vermelha) indicativos de superamplificação. Para esta amostra, foram escolhidos 11 ciclos para a PCR em grande escala (caixa azul). Por favor clique aqui para ver uma versão maior desta figura.
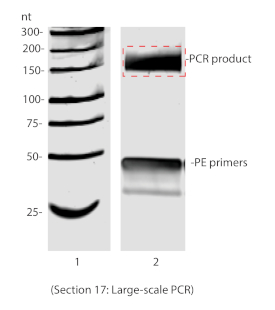
Figura 6 : O PCR em grande escala resolveu em 8% não-desnaturando a página. A caixa vermelha indica a parte do gel extirpada para a purificação do gel. Por favor clique aqui para ver uma versão maior desta figura.

Figura 7 : Screenshot do navegador do genoma do gene MAPK1 que mostra a distribuição de Flag-MAGOH: EIF4AIII pegadas como resultados representativos de um RIPiT. As setas vermelhas denotam os locais de ligação de EJC canônico esperados. Por favor clique aqui para ver uma versão maior desta figura.
| Pbs | 137 mM NaCl 2,7 milímetros KCl 10 mM na2HPO4· 7h2O KH2po4 pH 7,4 | |
| Tampão de têmpera | 2,5 M glicina 2,5 mM TRIS-base | |
| Tampão de Lise hipotônica (HLB) | 20 mM Tris-HCl pH 7,5 NaCl de 15 mM EDTA de 10 mM 0,5% IGEPAL 0,1% Triton-X-100 1x Aprotinin * 1x Leupeptina * 1x Pepstatin * 1 mM PMSF (fluoreto de fenilmetilsulfonilo) * | * deve ser adicionado fresco cada vez |
| Tampão de lavagem isotonic (IsoWB) | 20 mM Tris-HCl pH 7,5 150 mM NaCl 0,1% IGEPAL | |
| Buffer de conjugação | 0, 2% Polysorbate-20 1X PBS | |
| Limpar amostra de buffer | 100 mM Tris-HCl pH 6,8 SDS DE 4% EDTA de 10 mM | |
| 4xdNTPmix | 0,25 mM dGTP 0,25 mM dTTP 0,175 mM dATP 0,1625 mM dCTP 0, 75 mM biotina-dATP 0, 875 mM biotina-dCTP | |
| 5x primeiro-Strand buffer w/o MgCl2 | 250 mM Tris-HCl pH 8 375 milímetros KCl | |
| Tampão de diluição do RIPiT (1 mL) | 1 mL de IsoWB 5 μL 200x BSA 20 μL 10% Triton-X-100 20 μL 0.5 M EDTA 1x Aprotinin * 1x Leupeptina * 1x Pepstatin * 1 mM PMSF * | * deve ser adicionado fresco cada vez |
| Amortecedor de carga de desnaturação 2x | 3 mL 5x TBE 1,8 g Ficoll tipo 400 6,3 g de ureia 3 mg de bromofenol azul 3 mg de xileno cianol Ajustar o volume para 15 mL ddH2O | Para entrar em solução, coloque o tubo em água em um copo e deixe ferver na chapa quente por 10 – 15 min. Adicione corantes depois de ajustar o volume para 15 mL |
| Gel de uréia-página | 6 M de ureia Acrilamida: bisacrilamida (40% [w/v]) para a percentagem adequada 0.5 x TBE 0, 1% TEMED | |
| Tampão da eluição do ADN | 300 mM NaCl 1 mM EDTA | |
| Streptavidin talão tampão de lavagem | 0,5 M NaOH 20 mM Tris-HCl pH 7,5 1 mM EDTA |
Tabela 1: buffers.
Discussão
Discutimos aqui algumas considerações-chave para executar com sucesso o RIPiT. Acima de tudo, IPs individuais devem ser otimizados para alcançar a maior eficiência possível em cada etapa. A quantidade de grânulos do agarose da bandeira para o número de entrada das pilhas descreveu aqui provou ser robusta para uma vasta gama de proteínas que nós testamos. Como apenas uma pequena fração das proteínas parceiras é coimunoprecipitada com a proteína FLAG, a quantidade de anticorpos necessários para o segundo IP eficiente é geralmente baixa (menos de 10 μg). O RIPiT de pequena escala (da placa de 1 10 cm) seguido pela verificação ocidental do borrão das proteínas em cada fração durante as duas etapas da imunoprecipitação provam extremamente útil avaliar a eficiência e a especificidade do procedimento antes de escalar acima. Ambas as proteínas alvejadas assim como todas as outras proteínas de interação esperadas no complexo devem ser detectadas na eluição. Também é benéfico para o ensaio de proteínas nos lisados empobrecidos (não ligado à bandeira-agarose ou grânulos magnéticos) para ter uma boa estimativa da eficiência da imunoprecipitação e da percentagem de proteínas que se montam em um complexo. Além disso, esta análise também informa se as condições de digestão do RNase são suficientes para separar as interações RNA-dependentes de interações RNA-independentes dentro de um RNP. Conseqüentemente, é apenas como importante incluir um controle negativo, idealmente um RBP não relacionado ao RNP do interesse. Por exemplo, na Figura 3a, HNRNPA1 está presente na entrada, mas não é detectado na eluição do ripit. HNRNPA1 é um RBP que não interage diretamente com o EJC mas interage indiretamente com o EJC quando o EJC e o HNRNPA1 são limitados à mesma molécula do RNA. A deteção da proteína de controle negativo na eluição indica a especificidade pobre do RIPiT ou o footprinting insuficiente do RNA. Nesse caso, as pegadas de RNA obtidas não refletirão completamente as pegadas da proteína de interesse. As pegadas do tamanho 50-200 NT são recomendadas para o RNA-Seq subseqüente. duração do tratamento de RNase I ou a quantidade de enzima usada pode ser otimizada para obter pegadas de tamanho desejadas. Observe que o cenário de melhor caso será obter um bom sinal na faixa de tamanho desejada, e é inevitável ter RNAs mais longas e mais curtas, mesmo nas condições mais ideais. RIPiT também pode ser usado para obter sites de ligação de um único RBP. Em tal caso, a mesma proteína pode ser immunoprecipitated com dois anticorpos diferentes, primeiramente usando um anticorpo de encontro a uma etiqueta da afinidade e então com os anticorpos de encontro à proteína própria18. Finalmente, um controle negativo RIPiT pode ser realizado em paralelo a partir de células que expressam uma proteína de controle FLAG-Tagged (por exemplo, proteína verde fluorescente) em combinação com o anticorpo contra uma proteína contra uma proteína não relacionada no segundo IP.
Apesar de suas muitas vantagens, é importante considerar algumas limitações da abordagem do RIPiT, e possíveis remédios. A exigência da eluição da afinidade após a primeira purificação necessita a fonte biológica de expressar uma proteína etiquetada. Se um sistema de recombinação específico do site não estiver disponível na linha celular ou organismo de interesse, uma marca de afinidade curta, como uma tag FLAG (8 aminoácidos), pode ser introduzida no locus gênico endógeno usando a abordagem de edição de genoma baseada em CRISPR/CAS19. A tag FLAG é um epítopo ideal para esta abordagem, porque o anticorpo FLAG é bem adequado para eluição de afinidade e pode suportar altas forças iônicas e condições de desnaturação leves que podem ser usadas em combinação com reticulação de formaldeído. Outra limitação da abordagem do RIPiT é a exigência de uma grande entrada de material celular. Isto pode permanecer inevitável até certo ponto como somente uma porcentagem pequena de um RBP provavelmente interage com outras proteínas no RNP. As abordagens de preparação de biblioteca ainda melhoradas podem ajudar a derrubar o requisito de entrada grande. As idéias possíveis para agilizar ainda mais estas etapas incluem a realização do desfosforilação do RNA 3 '-End e a ligadura do adaptador nos grânulos magnéticos imediatamente depois do segundo lava do IP e antes da eluição final de RNP. Essa abordagem é implementada com êxito nos procedimentos atuais do CLIP-Seq e em uma variação descrita recentemente do RIPiT20. Tais mudanças removerão também diversos passos demorados da purificação do RNA das fases adiantadas do procedimento da preparação da biblioteca. Mais, ao contrário do grampo, que fornece uma definição nivelada do nucleotide do local de reticulação de um RBP no RNA, a definição de pegadas do RIPiT permanecerá no nível de dezenas de nucleotides. Finalmente, porque os RNPs podem incluir RBPs múltiplos, os locais enriquecidos do RNA de RIPiT incluem uma mistura de locais de ligação de muitos RBPs. Como as seqüências de consenso vinculadas por rbps individuais estão sendo descobertos em um ritmo rapidamente crescente e agora estão prontamente disponíveis21,22,23, esta informação pode ser aproveitada para desvolve a variedade de sites RBP enriquecido em saídas de RIPiT. Não obstante estes desafios, RIPiT-seq é um procedimento eficaz para capturar pegadas do RNA de complexos dinâmicos, heterogêneos, e mesmo transientes de RNP, que podem fornecer introspecções originais no funcionamento interno de machineries do RNA que controlam o celular Função.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pela concessão GM120209 de NIH (GS). Os autores agradecem o núcleo de recursos compartilhados da OSUCCC Genomics para seus serviços (CCC support Grant NCI p30 CA16058).
Materiais
| Name | Company | Catalog Number | Comments |
| Anti-FLAG Affinity Gel | Sigma | A2220 | |
| ATP, [γ-32P]- 3,000 Ci/mmol 10 mCi/mL EasyTide, 250 µCi | PerkinElmer | BLU502A250UC | |
| BD Disposable Syringes with Luer-Lok Tips (200) | Fisher | 14-823-435 | |
| Betaine 5M | Sigma | B0300 | |
| biotin-dATP | TriLink | N-5002 | |
| biotin-dCTP | Perkin Elmer | NEL540001EA | |
| Branson Sonifier, Model SSE-1 | Branson | ||
| CircLigase I | VWR | 76081-606 | ssDNA ligase I |
| DMEM, High Glucose | ThermoFisher | 11995-065 | |
| DNA load buffer NEB | NEB | ||
| Dynabeads Protein A | LifeTech | 10002D | |
| Flp-In-T-REx 293 Cell Line | ThermoFisher | R78007 | |
| GeneRuler Low Range DNA Ladder | ThermoScientific | FERSM1203 | |
| Hygromycin B | ThermoFisher | 10687010 | |
| Mini-PROTEAN TBE Gel 10 well | Bio-Rad | 4565013 | |
| Mini-PROTEAN TBE-Urea Gel | Bio-Rad | 4566033 | |
| miRCAT-33 adapter 5′-TGGAATTCTCGGGTGCCAAGGddC-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Mirus transIT-X2 transfection reagent | Mirus | MIR 6004 | |
| Mth RNA ligase | NEB | E2610S | |
| PE1.0 5′-AATGATACGGCGACCACCGAGATCTACACT CTTTCCCTACACGACGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| PE2.0 5′-CAAGCAGAAGACGGCATACGAGATCGGTCTC GGCATTCCTGCTGAACCGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Phenol/Chloroform/Isoamyl Alcohol (25:24:1, pH 6.7, 100 mLl) | Fisher | BP1752I-100 | |
| Purple Gel Loading Dye (6x) | NEB | NEB #7025 | |
| Q5 DNA Polymerase | NEB | M0491S/L | |
| RNase I, E. coli, 1,000 U | Eppicenter | N6901K | |
| SPIN-X column | Corning | CLS8160-24EA | |
| Streptavidin beads | ThermoFisher | 60210 | |
| Superscript III (SSIII) | ThermoScientific | 18080044 | reverse transcriptase enzyme |
| SybrGold | ThermoFisher | S11494 | gold nucleic acid gel stain |
| T4 Polynucleotide Kinase-2500U | NEB | M0201L | |
| T4RNL2 Tr. K227Q | NEB | M0351S | |
| Tetracycline | Sigma | 87128 | |
| Thermostable 5´ App DNA/RNA Ligase | NEB | M0319S | |
| TruSeq_SE1 5′-pGGCACTANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE10 5′-pGGTGTTCNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE11 5′-pGGTAAGTNNNNNAGATCGGAA GAGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE12 5′-pGGAGATGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE2 5′-pGGGTAGCNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE35′-pGGTCGATNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE4 5′-pGGCCTCGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE5 5′-pGGTGACANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE6 5′-pGGTAGACNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE7 5′-pGGGCCCTNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCT TCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE8 5′-pGGATCGGNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTT CCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE9 5′-pGGACTGANNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Typhoon 5 Bimolecular Imager | GE Healthcare Life Science | 29187191 |
Referências
- Karousis, E. D., Nasif, S., Mühlemann, O. Nonsense-mediated mRNA decay: novel mechanistic insights and biological impact. Wiley Interdisciplinary Reviews: RNA. 7 (5), 661-682 (2016).
- Ivanov, P. V., Gehring, N. H., Kunz, J. B., Hentze, M. W., Kulozik, A. E. Interactions between UPF1, eRFs, PABP and the exon junction complex suggest an integrated model for mammalian NMD pathways. The EMBO Journal. 27 (5), 736-747 (2008).
- Papasaikas, P., Valcárcel, J. The Spliceosome: The Ultimate RNA Chaperone and Sculptor. Trends in Biochemical Sciences. 41 (1), 33-45 (2016).
- Jensen, K. B., Darnell, R. B. CLIP: Crosslinking and ImmunoPrecipitation of In Vivo RNA Targets of RNA-Binding Proteins. Methods in Molecular Biology. 488, Clifton, N.J. 85-98 (2008).
- Garzia, A., Morozov, P., Sajek, M., Meyer, C., Tuschl, T. PAR-CLIP for Discovering Target Sites of RNA-Binding Proteins. mRNA Decay: Methods and Protocols. 1720, 55-75 (2018).
- Konig, J., et al. iCLIP - Transcriptome-wide Mapping of Protein-RNA Interactions with Individual Nucleotide Resolution. Journal of Visualized Experiments. (50), e2638(2011).
- Sibley, C. R. Individual Nucleotide Resolution UV Cross-Linking and Immunoprecipitation (iCLIP) to Determine Protein-RNA Interactions. RNA Detection: Methods and Protocols. 1649, 427-454 (2018).
- Wheeler, E. C., Van Nostrand, E. L., Yeo, G. W. Advances and challenges in the detection of transcriptome‐wide protein-RNA interactions. Wiley Interdisciplinary Reviews: RNA. 9 (1), (2018).
- Singh, G., et al. The Cellular EJC Interactome Reveals Higher-Order mRNP Structure and an EJC-SR Protein Nexus. Cell. 151, 750-764 (2012).
- Singh, G., Ricci, E. P., Moore, M. J. RIPiT-Seq: A high-throughput approach for footprinting RNA:protein complexes. Methods. 65, 320-332 (2014).
- Mabin, J. W., et al. The Exon Junction Complex Undergoes a Compositional Switch that Alters mRNP Structure and Nonsense-Mediated mRNA Decay Activity. Cell Reports. 25 (9), 2431-2446 (2018).
- Gehring, N. H., Lamprinaki, S., Kulozik, A. E., Hentze, M. W. Disassembly of Exon Junction Complexes by PYM. Cell. 137 (3), 536-548 (2009).
- Dostie, J., Dreyfuss, G. Translation Is Required to Remove Y14 from mRNAs in the Cytoplasm. Current Biology. 12 (13), 1060-1067 (2002).
- Zünd, D., Gruber, A. R., Zavolan, M., Mühlemann, O. Translation-dependent displacement of UPF1 from coding sequences causes its enrichment in 3' UTRs. Nature Structural & Molecular Biology. 20 (8), 936-943 (2013).
- Gangras, P., Dayeh, D. M., Mabin, J. W., Nakanishi, K., Singh, G. Cloning and Identification of Recombinant Argonaute-Bound Small RNAs Using Next-Generation Sequencing. Argonaute Proteins: Methods and Protocols. 1680, 1-28 (2018).
- Heyer, E. E., Ozadam, H., Ricci, E. P., Cenik, C., Moore, M. J. An optimized kit-free method for making strand-specific deep sequencing libraries from RNA fragments. Nucleic Acids Research. 43 (1), 2(2015).
- Cameron, V., Uhlenbeck, O. C. 3'-Phosphatase activity in T4 polynucleotide kinase. Biochemistry. 16 (23), 5120-5126 (1977).
- Ricci, E. P., et al. Staufen1 senses overall transcript secondary structure to regulate translation. Nature Structural & Molecular Biology. 21 (1), 26-35 (2014).
- Lackner, D. H., et al. A generic strategy for CRISPR-Cas9-mediated gene tagging. Nature Communications. 6, 10237(2015).
- Metkar, M., et al. Higher-Order Organization Principles of Pre-translational mRNPs. Molecular Cell. 72 (4), 715-726 (2018).
- Giudice, G., Sánchez-Cabo, F., Torroja, C., Lara-Pezzi, E. ATtRACT-a database of RNA-binding proteins and associated motifs. Database: The Journal of Biological Databases and Curation. 2016, (2016).
- Paz, I., Kosti, I., Ares, M., Cline, M., Mandel-Gutfreund, Y. RBPmap: a web server for mapping binding sites of RNA-binding proteins. Nucleic Acids Research. 42, Web Server issue 361-367 (2014).
- Sundararaman, B., et al. Resources for the Comprehensive Discovery of Functional RNA Elements. Molecular Cell. 61 (6), 903-913 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados