Method Article
Identifizierung von Fußabdrücken von RNA:Protein-Komplexe über RNA-Immunpräzipitation im Tandem gefolgt von Sequenzierung (RIPiT-Seq)
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Anreicherung endogener RNA-Bindungsstellen oder "Footprints" von RNA:Protein(RNP)-Komplexen aus Säugetierzellen vor. Dieser Ansatz umfasst zwei Immunpräzipitationen von RNP-Untereinheiten und wird daher als RNA-Immunpräzipitation im Tandem (RIPiT) bezeichnet.
Zusammenfassung
RNA-Immunpräzipitation im Tandem (RIPiT) ist eine Methode zur Anreicherung von RNA-Footprints eines Proteinpaares innerhalb eines RNA:Protein-Komplexes (RNP). RIPiT verwendet zwei Reinigungsschritte. Zunächst folgt auf die Immunpräzipitation einer markierten RNP-Untereinheit eine milde RNase-Verdauung und die anschließende nicht-denaturing-Affinitätselution. Eine zweite Immunpräzipitation einer anderen RNP-Untereinheit ermöglicht die Anreicherung eines definierten Komplexes. Nach einer denaturierenden Elution von RNAs und Proteinen werden die RNA-Footprints in DNA-Sequenzierungsbibliotheken mit hohem Durchsatz umgewandelt. Im Gegensatz zum populäreren ultravioletten (UV) Vernetzung gefolgt von Immunpräzipitation (CLIP) Ansatz zur Anreicherung von RBP-Bindungsstellen, RIPiT ist UV-Vernetzung unabhängig. Daher kann RIPiT auf zahlreiche Proteine angewendet werden, die in der RNA-Interactome und darüber hinaus vorhanden sind und für die RNA-Regulierung unerlässlich sind, aber nicht direkt mit der RNA oder dem UV-Kreuzvernetzen schlecht mit RNA in Kontakt treten. Die beiden Reinigungsschritte in RIPiT bieten einen zusätzlichen Vorteil bei der Identifizierung von Bindungsstellen, an denen ein Protein von Interesse in Partnerschaft mit einem anderen Cofaktor agiert. Die Doppelreinigungsstrategie dient auch dazu, das Signal durch Begrenzung des Hintergrunds zu verbessern. Hier bieten wir ein schrittweises Verfahren zur Durchführung von RIPiT und zur Generierung von Sequenzierungsbibliotheken mit hohem Durchsatz aus isolierten RNA-Footprints. Wir skizzieren auch die Vorteile und Anwendungen von RIPiT und besprechen einige seiner Einschränkungen.
Einleitung
Innerhalb von Zellen existiert RNA in komplexen Proteinen, um RNA:Protein-Komplexe (RNPs) zu bilden. RNPs werden um RNA-bindende Proteine (RBPs, die RNA direkt binden) zusammengesetzt, bestehen aber auch aus Nicht-RBPs (die RBPs binden, aber nicht RNA) und sind oft dynamischer Natur. RBPs und ihre Cofactors arbeiten gemeinsam innerhalb von RNPs, um regulierungsrechtliche Funktionen auszuführen. Beispielsweise erkennen die UPF-Proteine (UPF1, UPF2 und UPF3b) im unsinnvermittelten mRNA-Zerfallsweg (NMD) das vorzeitig beendete Ribosom. Jedes der UPF-Proteine kann an RNA binden, aber erst wenn sie sich zusammensetzen, beginnt sich ein aktiver NMD-Komplex zu bilden. Innerhalb dieses Komplexes wird UPF1 durch Phosphorylierung durch ein Nicht-RBP SMG1 weiter aktiviert, und eine solche UPF1-Aktivierung führt schließlich zur Rekrutierung von mRNA-Zerfallsinduzierenden Faktoren1,2. In diesem Beispiel benötigen RBPs Nicht-RBP-Cofaktoren für die Rekrutierung und Aktivierung des RNP-Komplexes, der NMD auslöst. Eine weitere Eigenschaft von RNPs ist ihre kompositorische Heterogenität. Betrachten wir den Spliceosome, der in unterschiedlichen E-, A-, B- oder C-Komplexen existiert. Verschiedene spliceosome komplexe haben überlappende und unterschiedliche Proteine3. Um RNP-Funktionen zu untersuchen, ist es wichtig zu klären, welche RNAs durch ein RBP und die zugehörigen Proteine gebunden sind. Viele Methoden existieren, um dies zu erreichen, wobei jeder Ansatz seine deutlichen Vor- und Nachteile4,5,6,7hat.
Die weit verbreiteten Methoden zur Identifizierung von RBP-Bindungsstellen – Vernetzung gefolgt von Immunpräzipitation (CLIP) und seinen verschiedenen Variationen – verlassen sich auf ultraviolettes (UV) Licht, um ein RBP mit RNA8zu vernetzen. Dies ist jedoch kein effektiver Ansatz für Nicht-RBPs innerhalb von RNPs, die die RNA nicht direkt kontaktieren. Hier beschreiben wir einen alternativen Ansatz, der für RBPs und Nicht-RBPs gleichermaßen gilt, um ihre RNA-Bindungsstellen zu isolieren und zu identifizieren. Dieser Ansatz, der als RNA-Immunpräzipitation im Tandem (RIPiT) bezeichnet wird, besteht aus zwei Immunpräzipitationsschritten, die dazu beitragen, eine höhere Spezifität im Vergleich zu einer einzigen Reinigung zu erreichen (Abbildung 1)9,10. Da die einzelnen Immunpräzipitationsschritte (IP) mit einer geringeren Stringenz im Vergleich zu CLIP durchgeführt werden können, hängt RIPiT nicht von der Verfügbarkeit von Antikörpern ab, die das Vorhandensein starker Detergenzien während der Immunpräzipitation aushalten können. Der einzigartigste Vorteil von RIPiT ist die Fähigkeit, zwei verschiedene Proteine in zwei Reinigungsschritten anzuvisieren; Dies bietet eine leistungsstarke Möglichkeit, einen kompositorisch unterschiedlichen RNP-Komplex aus anderen ähnlichen Komplexen zu bereichern11.
Kleine Variationen des RIPiT-Verfahrens können die RNP-Anreicherung weiter verbessern. Zum Beispiel sind einige RNA-Protein- oder Protein-Protein-Wechselwirkungen innerhalb von RNPs vorübergehend und es kann schwierig sein, Fußabdrücke solcher Komplexe effizient zu reinigen. Um solche Wechselwirkungen zu stabilisieren, können RNPs innerhalb von Zellen mit Formaldehyd vor der Zelllyse und RIPiT vernetzt werden. Zum Beispiel haben wir beobachtet, dass eine schwache Wechselwirkung zwischen dem Exon-Junction-Komplex (EJC)-Kernfaktor EIF4AIII und dem EJC-Demontagefaktor PYM12 mit Formaldehyd-Behandlung stabilisiert werden kann, so dass mehr RNA-Fußabdrücke angereichert werden (Daten nicht angezeigt). Vor der Zellernte und RIPiT können Zellen auch mit Medikamenten behandelt werden, um RNPs in einem bestimmten Zustand zu stabilisieren oder anzureichern. Wenn beispielsweise Proteine untersucht werden, die während der Translation aus der mRNA entfernt werden (z. B. eJC13, UPF114), kann die Behandlung mit Translationshemmern wie Puromycin, Cycloheximid oder Harringtonin zu einer erhöhten Belegung von Proteine auf RNAs.
Die Menge der von RIPiT zurückgewonnenen RNA ist in der Regel gering (0,5-10 Pmole, d.h. 10-250 ng RNA unter Berücksichtigung einer durchschnittlichen RNA-Länge von 75 nt). Der Hauptgrund dafür ist, dass nur ein kleiner Bruchteil eines bestimmten Proteins in Komplex mit anderen Proteinen innerhalb von RNPs vorhanden ist (jedes "freie" Protein IP'ed im ersten Schritt geht während des zweiten IP verloren). Um aus dieser RNA RNA-Seq-Bibliotheken zu generieren, skizzieren wir hier auch eine Anpassung des zuvor veröffentlichten Protokolls, das für solche niedrigen RNA-Eingänge geeignet ist15,16 (Abbildung 2), was zu hochdurchsatzbereiten Proben in 3 Tage.
Protokoll
1. Aufbau stabiler HEK293-Zelllinien, die Tetracyclin-induzierbares FLAG-markiertes Protein von Interesse (POI) ausdrücken
- Seed HEK293 Zellen mit einer stabil integrierten Flp-Rekombinationsziel -Site (FRT) mit einer Dichte von 10 x 104 Zellen/ml im Wachstumsmedium (Dulbeccos modifiziertes Eagle-Medium [DMEM] + 10% fetales Rinderserum [FBS] + 1% Penicillin-Streptomycin [penn/strep]) in 6- brunnen Platten. Lassen Sie Zellen über Nacht in einem befeuchteten Inkubator bei 37 °C und 5%CO2 wachsen (Standardwachstumsbedingungen für alle nachfolgenden Schritte).
- Am nächsten Tag sollten die Zellen zu 70 % konfluent sein. Transfektionsreagenzprotokoll, transfekt die FRT-standorthaltigen HEK293-Zellen mit einem Verhältnis von 9:1 von pcDNA5-TETO-FLAG-POI:pOG44.
HINWEIS: Das FLAG-Tag hat das Sequenzmotiv DYKDDDDK (D = Aspartinsäure, Y = Tyrosin und K = Lysin). - Nach 24 h beginnen Sie mit der Antibiotikaauswahl. Entfernen Sie die Medien und fügen Sie frisches Wachstumsmedium hinzu, das mit 100 g/ml Hygromycin ergänzt wird. Innerhalb von 72 h sollten untransfizierte Zellen zu sterben beginnen.
- Alle 48-72 h, ändern Wachstumsmedien und ergänzen mit frischem Hygromycin.
- Nach 2 Wochen der Selektion beginnen diskrete Kolonien von stabil transfizierten Zellen zu erscheinen. Sobald Kolonien mit bloßem Auge sichtbar sind, 1 ml Trypsin auf die Platte geben und 5 min bei 37 °C brüten. Zellen in DMEM resuspendieren, Zellen in eine neue 10 cm Platte übertragen und das Volumen auf 10 ml frisches Wachstumsmedium einstellen, ergänzt durch 100 g/ml Hygromycin. Lassen Sie die Platte 80 % Konfluenz erreichen, um Zellen weiter zu erweitern, um dauerhafte Bestände vorzubereiten.
- Bestimmen Sie die Menge an Tetracyclin (Tet), die erforderlich ist, um den optimalen Flag-POI-Ausdruck für das Experiment zu erhalten. Wachsen Sie Zellen im 12-Well-Format und führen Sie eine Titration von Tet zwischen 0-1.000 ng/ml Bereich für 16-24 h. Führen Sie western Blots auf den Titrationsproben mit Antikörper gegen die POI.
HINWEIS: Bei Proteinen bis zu 60 kDa können die FLAG-markierte und die endogene Kopie des Proteins auf dem Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE) aufgelöst werden, um die Expressionsniveaus von FLAG-POI mit seinem endogenen Gegenstück zu vergleichen. Bei größeren Proteinen kann die Signalintensität in Tet-induzierten Proben mit nicht induzierten Proben verglichen werden. Die Tetracyclin-Stammlösung kann mit 1 mg/ml in 100% Ethanol hergestellt werden. Verdünnungen des Bestandes für zellkulturbezogene Arbeit sollten in sterilem Wasser oder phosphatgepufferter Salzsäure (PBS) erfolgen. Der Tetracyclinbestand sollte einmal im Monat frisch zubereitet werden.
2. Culturing Cells for Tetracyclininduction and RIPiT Procedure
- Seed stabil integrierte FLAG-POI HEK293 mit einer Dichte von 3 x 105 Zellen/ml im Wachstumsmedium in 15 cm Platten. Zellen bei 37 °C und 5%CO2wachsen lassen.
HINWEIS: Im Allgemeinen ergeben drei bis fünf 15 cm Platten je nach Häufigkeit des RNP von Interesse 2-20 pmol RNA-Fußabdrücke. Wenn RNPs formaldehyd vernetzt und unter strengen Bedingungen gereinigt werden, kann eine doppelt so hohe Inanspruchnahme erforderlich sein. - Fügen Sie Tetracyclin zu einer vorbestimmten optimalen Konzentration in das Medium ein, um die Expression des FLAG-POI zu induzieren (siehe Schritt 1.6) 16-24 h, bevor die Zellen geerntet werden.
HINWEIS: Es ist nicht notwendig, das Wachstumsmedium zu ändern. Die Zellen sollten nach der Aussaat etwa 72 h oder bei einem Zufluss von 80 % zur Ernte bereit sein. Es ist wichtig zu vermeiden, dass Zellen konfluent werden.
3. Zellernte, Formaldehyd-Behandlung und Zelllyse
- Waschen Sie die Monolayer-Zellen sanft mit 15 ml gekühltem PBS für jede 15 cm Platte. Dann abkratzen Zellen in 30 ml PBS. Wenn Zellen vor der Ernte mit Medikamenten behandelt wurden, ergänzen Sie PBS mit dem Medikament. Sammeln Sie alle Zellen in 50 ml konische Röhre.
HINWEIS: Zur Aufrechterhaltung schwacher Wechselwirkungen können Zellen mit Formaldehyd vernetzt werden: Formaldehyd von Schritt 3.1 auf eine Endkonzentration von 0,1% eintragen und auf einer Wippe bei Raumtemperatur für 10 min inkubieren. 3 ml des Abschreckungspuffers hinzufügen (Tabelle1 ) und Rock für 5 zusätzliche Minuten. - Pelletzellen bei 400 x g für 10 min bei 4 °C und abwerfen Überstand.
- Lysezellen in 4 ml eiskaltem hypotonischer Lysepuffer (HLB; Tabelle 1). Verwenden Sie eine P1000-Pipette, um Zellen wieder aufzuhängen. Auf ein 5 ml Rohr übertragen und 10 min auf Eis bebrüten.
- Das Lysat in ein Eisbad geben und bei 10% Amplitude für 30 s mit 1 s Impulsen mit 2 s Pausen beschallen. Passen Sie dann die Salzkonzentration auf 150 mM an, indem Sie 108 l von 5 M NaCl hinzufügen.
HINWEIS: Formaldehyd-vernetzte Proben können einer strengeren Lyse und Reinigung unterzogen werden, indem jeweils 0,1 % SDS und Natriumdeoxycholat in den Lysepuffer einbeziehen. - Löschen Sie das Lysat durch Zentrifugation bei 21.000 x g für 10 min bei 4 °C. Sammeln Sie 20 L Überstand (Zellextrakt) in einem beschrifteten Rohr für die Western-Blot-Analyse des Proteinspiegels im Input (siehe Abbildung 3A).
HINWEIS: Während Lysat in zentrifugiert ist, können FLAG-Agaroseperlen gewaschen werden (siehe Schritt 4.1). FLAG Agarose Perlen sollten 3x in 4 ml eiskalten isotonischen Waschpuffer vorgewaschen werden (IsoWB, Tabelle 1).
4. FLAG Immunpräzipitation
- Tragen Sie den restlichen Überstand aus Schritt 3,5 bis 750 l vorgewaschenen FLAG-Agaroseperlen in einem 5 ml-Rohr auf (Bettvolumen der gewaschenen FLAG-Agaroseperlen beträgt 375 l). INkubieren FLAG Agarose Perlen und Zellextrakt für 1-3 h bei 4 °C mit sanfter Mischung.
HINWEIS: Dieses Volumen an FLAG-Agaroseperlen sollte für eine breite Palette von Proteinen ausreichen, kann aber bei Bedarf optimiert werden. - Pellet FLAG Agarose Perlen durch Zentrifugation bei 400 x g für 1 min bei 4 °C. Sammeln Sie 20 L des Überstandes in einer beschrifteten Röhre für die Western-Blot-Analyse des Proteinspiegels in erschöpftem Zellextrakt (siehe Abbildung 3A). Verwerfen Sie den verbleibenden Überstand.
- Um FLAG Agarose Perlen zu waschen, fügen Sie 4 ml IsoWB und resuspend. Pelletperlen bei 400 x g für 1 min bei 4 °C. Entfernen Sie vorsichtig Überstand. Wiederholen Sie 4x.
HINWEIS: Bei strengen Waschungen der formaldehydvernetzten IPs können jeweils 0,1% SDS und Natriumdeoxycholat in den Waschpuffer für die ersten beiden Waschschritte aufgenommen werden.
5. RNase I Verdauung
- Verdünnung der RNase I auf 0,002-0,01 Einheiten/ml in 750 l IsoWB (die entsprechende Konzentration für gewünschte Footprintgrößen muss empirisch bestimmt werden). IsoWB-RNase I zu gewaschenen FLAG-Agaroseperlen hinzufügen und mit sanftem Mischen bei 4 °C 10 min bebrüten.
- Pelletperlen bei 400 x g für 1 min bei 4 °C. Sammeln Sie 20 L Überstand (RNase I Elution) in einem beschrifteten Rohr für die Western Blot-Analyse. IsoWB-RNase I entsorgen und FLAG Agarose Perlen 4x mit IsoWB waschen, wie in Schritt 4.3 beschrieben.
6. Affinitäts-Elution
- Bereiten Sie einen Vorrat an Elutionspuffer (FLAG-Peptid bei 250 ng/ml in IsoWB) vor. 375 L Elutionspuffer auf FLAG-Agaroseperlen auftragen und bei 4 °C für 1-2 h sanft schütteln. Pellet FLAG Agarose perlen und sammeln Sie einen 15 L Aliquot der Elution für Western-Blot von Proteinen in FLAG IP (siehe Abbildung 3A).
HINWEIS: Wenn die Affinitäts-Elution im Gange ist, kann Abschnitt 7 durchgeführt werden.
7. Magnetischer Bead-Antikörper Konjugation
- Waschen Sie 50 L magnetische Perlen (d. h. Dynabeads; Materialtabelle) 3x in 1 ml IsoWB in 1,5 ml Rohr. Setzen Sie magnetische Perlen in 100 l Konjugationspuffer aus. Fügen Sie eine angemessene Menge an Antikörpern hinzu (die genaue Menge an Antikörpern, die für IP verwendet werden sollen, muss empirisch für jeden Antikörper bestimmt werden).
HINWEIS: Protein A Magnetperlen sind optimal für Antikörper, die bei Kaninchen produziert werden, während Protein G Magnetperlen besser für Mausantikörper geeignet sind. Magnetische Perlen Kompatibilitätstabelle ist auf der Lieferanten-Website verfügbar, um Perlen zu wählen, die für jeden Antikörper geeignet sind. - Magnetische Perlen 2x im Konjugationspuffer waschen (Tabelle 1). Inkubationsgemisch bei Raumtemperatur für mindestens 10 min. Magnetische Perlen in 375 l RIPiT-Verdünnungspuffer resuspendieren. Auf Eis bis zum nächsten Schritt aufbewahren.
8. Zweite Immunitizipation
- Tragen Sie die verbleibende FLAG-Affinitätselution ab Schritt 6.1 auf magnetische Perlen auf, die an Antikörper gegen das Protein von Interesse gekoppelt sind. Mit sanftem Mischen bei 4 °C für 1-2 h inkubieren. Magnetische Perlen auf einem Magneten erfassen und 15 L Überstand für die Analyse ungebundener Proteine über Western Blot sammeln. Magnetische Perlen 7x mit 1 ml IsoWB waschen.
9. Entsende-Elution
- Fügen Sie 100 l durchklaren Probenpuffer (Tabelle 1) zu magnetischen Perlen hinzu und suspendieren Sie mit einer P200-Pipette. 10 min. auf Eis bebrüten. Flick sanft, um Perlen regelmäßig wieder auszusetzen.
- Erfassen Sie magnetische Perlen auf einem Magneten und sammeln Sie 15 L Elution für die Analyse von Proteinen in DER RIPiT-Elution über Western Blot (siehe Abbildung 3A). Restelution in ein neues, beschriftetes 1,5 ml-Rohr übertragen.
HINWEIS: Wenn Proben formaldehyd vernetzt waren, müssen Proben bei 65 °C für 1 h inkubiert werden, um die Vernetzung umzukehren. - Führen Sie westliche Blots auf Proben aus, die in verschiedenen Schritten gesammelt wurden (Eingabe, FLAG IP-Erschöpfung, FLAG IP, zweite IP-Erschöpfung, zweite IP-Elution). Blot mit Antikörpern gegen die beiden Köderproteine, deren andere Interaktoren, sofern bekannt, und mindestens einem nicht interagierenden RBP als Negativkontrolle (Abbildung 3A).
10. RNA-Extraktion und Endhärtung
- Zur RIPiT-Elution 320 l RNase-frei ddH2O, 400 l Phenol-Chlorform-Isoamylalkohol (PCIAA, pH 4,5) hinzufügen und bei Raumtemperatur bei 12.000 x g für 5 min wirbeln. Sammeln Sie 350 l wässrige Phase in ein separates Rohr. Fügen Sie 35 l 3 M Natriumacetat, 1 l 1 M MgCl2, 10 g Glykogen und 1 ml 100% Ethanol hinzu. Über Nacht bei -20 °C inkubieren.
- Zur Pellet-RNA zentrifugieren Sie bei 12.000 x g 30 min bei 4 °C. Waschen Sie RNA in 70% Ethanol.
- Um 3' Phosphat, das nach der RNase I-Spaltung auf der RNA zurückbleibt, zu entfernen, setzen Sie das RNA-Pellet in 17 l RNase-frei ddH2O wieder auf, und fügen Sie 2 l 10x T4-Polynukleotid-Kinase (PNK) Puffer(Materialtabelle)und 1 l T4 PNK hinzu. Bei 37 °C für 30 min inkubieren.
HINWEIS: Die 3'phosphatase Aktivität von T4 PNK hat eine optimale Aktivität bei pH 617. Der PNK-Reaktionspuffer ist für die 5' Kinase-Aktivität von T4 PNK optimiert und hat einen pH-Wert von 7,6. Während der pH-Wert der Endhärtungsreaktion angepasst wurde, kann dieser Schritt weiter optimiert werden. - Fügen Sie 380 l RNase-freie ddH2O und 400 l PCIAA pH 4.5 in das Rohr. Wirbel für 30 s, Zentrifuge bei 12.000 x g für 5 min. Wässerphase sammeln und 35 l 3 M Natriumacetat, 1 l von 1 M MgCl2,10 g Glykogen und 1 ml 100% Ethanol hinzufügen.
- Über Nacht bei -20 °C inkubieren. Pellet und waschen RNA mit 70% Ethanol wie oben. Resuspend-RNA in 4,5 l RNase-freiem Wasser.
11. Schätzung der Größe und Häufigkeit des RNA-Fußabdrucks
-
Ein erfolgreiches RIPiT wird voraussichtlich 1 pmol oder mehr von RNA-Fragmenten ergeben. Um die tatsächliche Ausbeute zu quantifizieren, übertragen Sie 0,7 l RIPiT-RNA (ca. 1/6 der Gesamtausbeute) in ein neues Rohr. Fügen Sie 2 l von 10x T4 PNK Puffer, 1 l von 1 mM ATP, 40 ,Ci32P-ATP (0,5 x 1,0 l des Bestands) und 1 l T4 PNK hinzu. Die Lautstärke auf 10 l einstellen und 30 min bei 37 °C inkubieren.
- In parallelen PNK-Reaktionen beschriften Sie eine DNA-Leiter mit niedrigem Bereich und 0,1 pmol eines synthetischen RNA- oder DNA-Oligos (20-40 nt), um Größen- und Mengenstandards zu verwenden.
- Aufsetzen auf beschriftete RNA/DNA auf 26% Harnstoff-PAGE-Gel (20 x 27 x 0,45 mm3). Gel muss 30 min bei 35 W vorlaufen. Vorlauf und vor dem Beladen von Proben vorlaufen und bei 35 W laufen, bis die Bromphenol-Blaufarbstofffront fast das Ende des Gels erreicht hat.
- Entfernen Sie das Gel vorsichtig von Glasplatten auf ein Stück 8 x 11 Zoll Filterpapier. Mit Gel auf Papier, in Gel-Trocknungsapparate legen und mit einem Stück Plastikfolie abdecken. Trockenes Gel bei 80 °C für 1 h mit Vakuum.
- Trockengel über Nacht oder bis zum Erkennen eines ausreichenden Signals einem Phosphoscreen aussetzen. Bild Phosphoscreen. Gute RNA von einem RIPiT sollte als Abstrich in der Spur erscheinen, mit minimalen prominenten Bändern (Abbildung 3B). Um die RNA zu quantifizieren, vergleichen Sie die Signalintensität der gewünschten RNA-Fragmente in der RIPiT-Spur mit dem Signal von 0,1 pmol markierten synthetischen Oligos.
HINWEIS: Alternativ können RNA-Fußabdruckgröße und -mengen mit einem hochempfindlichen Bioanalyzer überprüft werden (Abbildung 3C).
12. Adapter Ligation
- Bereiten Sie RIPiT-RNA so vor, dass mindestens 3 pmol RNA in 3,8 l Wasser gelöst wird.
- In einem 0,2 ml Polymerase-Kettenreaktions-Rohr (PCR) 3,8 l RNA, 1 L miR-CAT-33 voradenyliertenAdapter (7 'M) (Materialtabelle) kombinieren. Inkubieren Sie die Mischung auf einem thermischen Cycler bei 65 °C für 10 min, 16 °C für 5 min, dann halten Sie bei 4 °C.
HINWEIS: Der voradenylierte Linker kann entweder aus dem Oligosynthese-Service bestellt werden, oder ein benutzerdefiniertes unadenyliertes DNA-Oligo von jedem Oligo-Synthese-Service kann mit Mth-RNA-Ligase (Tabelle derMaterialien) und Gel gereinigt werden. - Zur gleichen Röhre fügen Sie 1,5 l 10x T4-RNA-Ligasepuffer, 7,5 l 50% Polyethylenglykol 8000 (PEG-8000), 0,75 l von 20 mM Dithiothreitol (DTT) und 0,45 l T4 RNL2 Tr. K227Q(Materialtabelle )hinzu.
HINWEIS: 50% PEG-8000 wird mit RNA-Ligase und Puffer gekauft. PEG-8000 Lösungen sind zähflüssig und sollten langsam pipettiert werden. - Inkubationsreaktion im Thermischen Cycler bei 30 °C für 6 h, Hitze inaktivieren die Ligase bei 65 °C für 10 min, dann bei 4 °C halten.
13. Umgekehrte Transkription
- In das Rohr mit dem Ligationsmix aus Schritt 12.4 11,25 l 4x Desoxynukleotidtriphosphat (dNTP) geben, der eine Mischung aus regulären und biotinylierten dNTPsenthält (siehe Tabelle 1), 1,0 l mit 10 RNasefreies Wasser. Bei 65 °C 5 min inkubieren, dann bei 4 °C halten.
- Übertragen Sie die Rohre auf Eis und fügen Sie 9,0 l 5x 1.-Strang-Puffer (FS) ohne MgCl2 (Tabelle 1), 2,25 l von 100 mM DTT, 1,2 l Reverse-Transkriptase-Enzym zu einem Endvolumen von 45 l(Materialtabelle)hinzu.
- Inkubieren Sie in einem thermischen Cycler bei 55 °C für 30-60 min. Inaktivieren Sie die Umgekehrte Transkriptase bei 70 °C für 15 min und halten Sie die Probe bei 4 °C.
14. Reinigung des RT-Produkts
- Fügen Sie der RT-Reaktion 45 L 2x Harnstoff-Lastpuffer (Tabelle 1) hinzu. Verdünnen Sie 1 g einer DNA-Leiter mit niedrigem Bereich in 45 l und fügen Sie 45 l 2x Harnstoff-Lastpuffer hinzu.
- Zubereitung 10% Harnstoff-PAGE Gel (20 x 28 x 0,15 cm3; Tabelle 1) mit 8-Well-Kamm. Mit Spritze oder Pipetten, Spülbrunnen mit 0,5x Tris/Borate/EDTA (TBE) Puffer.
HINWEIS: Die oben genannten hausgemachten Gele bieten eine bessere Auflösung bei der Trennung des erweiterten RT-Produkts von der unerweiterten RT-Grundierung. Alternativ können auch vorgegossene Harnstoff-PAGE-Gele verwendet werden (Tabelle der Materialien). Da Vorguss-Gele kleinere maximale Volumina pro Bohrkörper zulassen, müssen Proben in mehrere Bohrungen unterteilt werden. Vorgegossene Gele sollten bei 150 bis 200 V betrieben werden. - Führen Sie das Gel bei 35 W für 30 min vor. Spülen Sie brunnen wieder, laden Sie Proben und laufen Gel bei 35 W, bis Bromphenol blau Farbstoff front auf etwa 1 Zoll vom Ende des Gels migriert ist.
HINWEIS: Verwenden Sie Metall-Kühlkörper während der Vorlauf und den letzten Lauf, um Gelüberhitzung zu verhindern. - Das Gel für 5 min in 1x Gold Nukleinsäure-Gel-Färbung in 0,5x TBE hergestellt. Dieser Farbstoff ist lichtempfindlich, so vermeiden Lichtbelichtung.
- Bildgel auf Fluoreszenzscanner zu Dokumentationszwecken mit 520 nm Anregung und 580 nm Emissionsfiltern. Wenn ein Fluoreszenzscanner nicht verfügbar ist, verwenden Sie einen Blaulichttransilluminator. Das RT-Produkt sollte als Abstrich über dem nicht verlängerten RT-Primer erscheinen (Abbildung 4).
HINWEIS: Obwohl der Goldnukleinsäure-Gel-Färbefarbstoff leicht auf einem Gel-Doc mit UV-Lichtquelle visualisiert werden kann, ist es wichtig, das wertvolle RT-Produkt nicht UV-Aussetzen, um Schäden zu verhindern. - Visualisieren Sie das Gel auf einem blaulichten Transilluminator und verbrauchen Sie das RT-Produkt aus dem Gel. Es wird empfohlen, DNA mit Erweiterungen von 30-200 nt zu schneiden (Abbildung 4). Legen Sie die ausgeschnittenen Gelstücke auf eine saubere Oberfläche und zerkleinern Sie die Scheibe in kleine Stücke, um die Oberfläche zu vergrößern. Vorsichtig in ein 1,5 ml-Rohr übertragen und 800 l DNA-Elutionspuffer hinzufügen (Tabelle 1).
- Inkubieren Sie die Gelstücke mit sanftem Mischen mit DNA-Elutionspuffer über Nacht bei Raumtemperatur.
- Trennen Sie den Elutionspuffer vom Gel, indem Sie die Gülle durch eine Celluloseacetat-Filtersäule(Materialtabelle)in einem 2 ml Sammelrohr übergeben.
- In 1,5 ml Rohr 10 l Streptavidin-Magnetperlen mit 500 l Streptavidin-Perlen-Waschpuffer waschen (Tabelle 1). Wiederholen Sie dies für drei Gesamtwältsjahre. Lassen Sie die Perlen nicht austrocknen. Resuspend Perlen in 10 l DNA-Elutionspuffer (Tabelle 1).
- Übertragen Sie den von den Gelstücken getrennten Elutionspuffer in Schritt 14.8 in das Rohr, das die gewaschenen Streptavidin-Magnetperlen enthält.
- Mit sanftem Mischen mindestens 8 h bei Raumtemperatur inkubieren. Erfassen Sie Perlen auf einem Magneten, entfernen Sie Überstand und suspendieren Sie magnetische Perlen in 10 l RNase-freiem Wasser und übertragen Sie sie auf ein 0,2 ml PCR-Rohr.
15. Zirkularisierung des RT-Produkts
- RT-Produkte, die auf Streptavidinperlen gefangen werden, werden kreisförmigen, während sie an Perlen gebunden sind. Zur magnetischen Perlenschlämme 2,0 l 10x Kreisförmigereaktionspuffer, 1,0 l mit 1 mM ATP, 1,0 l 50 mM MnCl2, 4,0 l 5 m Betain, 1,0 l ssDNA ligase I (Materialtabelle)und 1,0 l RNase-freies Wasser hinzufügen.
- Inkubieren Sie die Zirkelungsreaktion auf einem Thermischen Cycler bei 60 °C für 4 h. Die ssDNA Ligase I durch Erhitzen bei 80 °C für 10 min inaktivieren und dann bei 4 °C halten.
16. PCR testen
- Bevor Sie zu einer großen PCR übergehen, verwenden Sie einen Teil des kreisförmigen Produkts, um die ideale Anzahl von Amplifikationszyklen für jede Probe zu bestimmen. Dieser Schritt hilft, eine Überverstärkung zu verhindern und PCR-Artefakte zu begrenzen, da PCR-Reaktionskomponenten bei höheren PCR-Zyklen einschränkend werden.
- Bereiten Sie eine 45-L-PCR-Reaktion mit 4,0-6,0 l des kreisförmigen Produkts ab Schritt 15,2, 9,0 l 5x Reaktionspuffer, 0,9 l 10 M dNTPs, 2,25 l mit 10 -M PE1,0-Grundierung(Materialtabelle),2,25 l mit 10 ,0-M-PE2.0-Grundierung (Materialtabelle ) vor. und 0,045 l hochtreue DNA-Polymerase (Materialtabelle) und Wasser.
- Mischen Sie die Reaktion gut und teilen Sie sich in drei 15-L-Reaktionen auf. Jede dieser drei Reaktionen unterliegt einer variablen Anzahl von PCR-Zyklen. Die ideale Anzahl von Zyklen wird voraussichtlich zwischen 7 und 14 liegen. Führen Sie also Test-PCRs für 8, 11 und 14 Zyklen durch.
- Verwenden Sie die folgenden PCR-Bedingungen: 98 °C - 30 s; 98 °C - 5 s; 65 °C - 10 s; 72 °C - 15 s; 72 °C - 2 min; 12 °C - halten.
- Fügen Sie 3 l 6x Gel-Ladefarbstoff hinzu und lösen Sie auf 10% natives PAGE-Gel auf, bis die blaue Farbstofffront 3/4 des Gels migriert hat. Färben Sie das Gel mit Goldnukleinsäure-Gel-Färbung und Bild wie in den Schritten 14.4 und 14.5 (Abbildung 5).
- Um die ideale Anzahl von PCR-Zyklen zu wählen, vergleichen Sie PCR-Produkte aus der zunehmenden Anzahl von Zyklen. Wählen Sie die Zyklusnummer, die die größte Menge an Produkt der erwarteten Größe ohne ÜberverstärkungArtefakte ergibt (z. B. DNA-Abstrich viel größer als erwartet, und wo keine nennenswerte Erschöpfung der PE1.0- und PE2.0-Primer zu sehen ist (siehe roter Pfeil in Abbildung 5).
17. Große PCR
- Bereiten Sie eine 45-L-PCR-Reaktion vor, wie in Schritt 16.2, und wiederholen Sie die PCR. Lösen Sie PCR auf 10% native PAGE bei 150 V, Fleck mit 1x Gold Nukleinsäure Gel Fleck und Bild auf einem blauen Licht Transilluminator.
- Das PCR-Produkt aus dem Gel ausnehmen und auf eine 3 ml Spritze übertragen. Verwenden Sie die Spritze, um das Gel zu zerkleinern und in ein 1,5 ml-Rohr zu extrudieren.
HINWEIS: Das unerweiterte RT-Produkt bei Zirkularisierung ergibt PCR-Produkt von 151 bp. Daher sollte man in diesem Schritt die Größe der Auswahl von Produkten auswählen, die größer als 151 bp sind (Abbildung 6). - Fügen Sie 900 l DNA-Elutionspuffer hinzu und inkubieren Sie bei Raumtemperatur über Nacht mit sanftem Mischen.
- Die Gelschlämme in eine Celluloseacetat-Filtersäule in einem 2 ml Sammelrohr übertragen. Drehen Sie bei 12.000 x g für 3 min, sammeln Überstand in einem frischen Rohr.
- Fügen Sie dem zerkleinerten Gel weitere 400 l DNA-Elutionspuffer hinzu und übertragen Sie es in ein 1,5 ml-Rohr. Mit sanftem Mischen für weitere 4 Stunden für eine zweite Elution inkubieren.
- Bündeln Sie alle Elutionen und teilen Sie sich in 3 Röhren mit jeweils 400 l. Niederschlagen Sie die DNA, indem Sie 1 ml 100% Ethanol und 10 g Glykogen hinzufügen. Wirbel und inkubieren mindestens 2 h bei -20 °C.
- Pellet-DNA bei 12.000 x g für 30 min bei 4 °C. Waschen Sie DNA-Pellets mit 70% Ethanol.
- Entfernen Sie sorgfältig alle Ethanol durch Pipettieren und schnell wieder aufsetzen DNA-Pellet in 20 l Wasser.
HINWEIS: In diesem Stadium ist es wichtig, das DNA-Pellet nicht trocken werden zu lassen, da das Austrocknen der DNA sie denaturieren kann. - Verwenden Sie einen kleinen Teil der DNA-Probe, um Größe und Konzentration des PCR-Produkts über Fluortometer und hochempfindlichen DNA-Bioanalysator zu bestimmen. Beispiele können nun zur Sequenzierung auf einer der Plattformen eingereicht werden.
- Die sequenzierten Lesevorgänge können verarbeitet (z. B. Adapterentfernung, Trimmen, um Sequenzen >30 Phred-Score zu halten), am Referenzgenom ausgerichtet und in einem Browser wie dem UCSC-Genombrowser visualisiert werden (Abbildung 7).
Ergebnisse
Ein erfolgreiches RIPiT führt zur Immunpräzipitation sowohl von Proteinen von Interesse als auch von anderen bekannten interagierenden Proteinen und dem Fehlen nicht interagierender Proteine. Wie in Abbildung 3Adargestellt, wurden sowohl Magoh als auch EIF4AIII in der RIPiT-Elution nachgewiesen, HNRNPA1 jedoch nicht (Spur 6). Parallel dazu wurden RNA-Footprints, die mit den RNP-Komplexen kogereinigt wurden, mittels Autoradiographie (Abbildung 3B) oder Bioanalyzer (Abbildung 3C) nachgewiesen. Die Puromycin-Behandlung wird voraussichtlich die EJC-Belegung auf RNA erhöhen, und ein stärkeres RNA-Fußabdrucksignal wurde in der mit Puromycin behandelten RIPiT in Abbildung 3B beobachtet (Vergleich der Bahnen 2 und 3). Das Generieren von Proben für die tiefe Sequenzierung erfordert die Liganderung eines Adapters an die RNA und umgekehrtes Transkribieren der RNA in DNA mit einem Primer, der der Adaptersequenz anneals entspricht. Der umgekehrte Transkriptionsschritt beinhaltet biotinylierte Nukleotide zur Reinigung des Reverse-Transkriptionsprodukts. Nach der umgekehrten Transkription muss das Produkt durch Harnstoff-PAGE vom unerweiterten Adapter getrennt werden (Abbildung 4). Das Reverse-Transkriptionsprodukt wird dann zirkuliert und PCR verstärkt. Die entsprechende Anzahl von PCR-Zyklen darf das zirkulierte Produkt nicht überverstärken. Eine Überverstärkung führt zu einer Erschöpfung der Grundierung und einem ableitenden PCR-Produkt (siehe Abbildung 5, Spur 4). Die Anzahl der Zyklen mit der größten Verstärkung ohne Nachweis einer Überverstärkung ist am besten geeignet, um für eine große PCR zu verwenden (Abbildung 5 Spur 3 und Abbildung 6).

Abbildung 1 : Schematisch, der die wichtigsten Schritte in RIPiT umreißt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
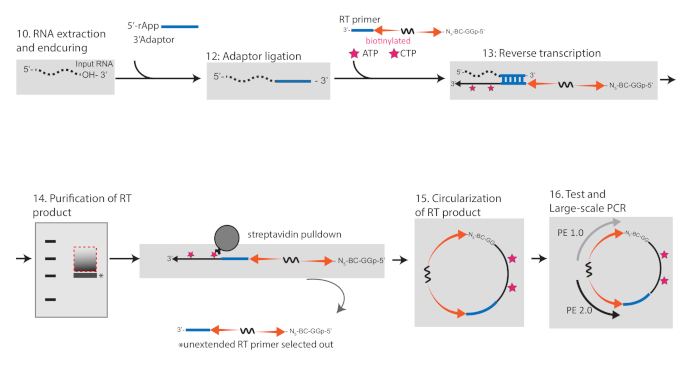
Abbildung 2 : Schematische Darstellung des Workflows für die Konvertierung von RIPiT-RNA in Bibliotheken für die Sequenzierung mit hohem Durchsatz. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
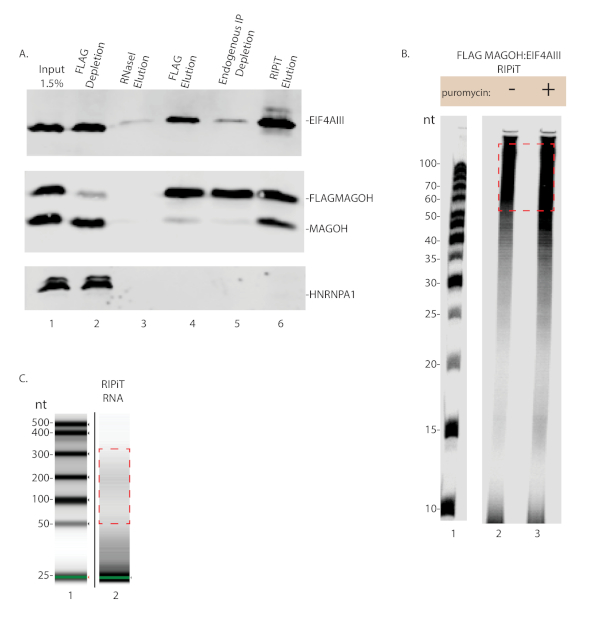
Abbildung 3 : Schätzung der RNA- und Proteinerträge von RIPiT. (A) Westlicher Fleck der Proteine, die aus jedem wichtigen Schritt des RIPiT-Verfahrens gereinigt werden. (B) Autoradiographbild von RNA-Fußabdrücken aus einem FLAG-MAGOH:EIF4AIII RIPiT zum Vergleich von puromycin behandelten und unbehandelten Zellen. Rotes Feld zeigt die RNA-Footprintgröße an, die letztendlich in Sequenzierungsbibliotheken umgewandelt wird. (C) Profil der RNA, die von RIPiT wie in Spur 2 in Panel B eluiert wird, wenn sie mit einem Bioanalyzer visualisiert wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4 : Reverse Transkriptionsprodukt (RT) auf einem 10% Harnstoff-PAGE-Gel gelöst und mit Goldnukleinsäure-Gel-Färbung gebeizt. Rote Box zeigt Gel-Region für Gel-Reinigung von erweiterten RT-Produkten verbrauchen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
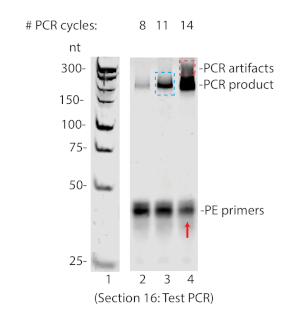
Abbildung 5 : Test PCR aufgelöst auf 8% nicht-denaturing PAGE. Beachten Sie die aberrant großen PCR-Produkte, die bei 14 Zyklen (roter Kasten) erscheinen, und die parallele Erschöpfung der Primer (roter Pfeil), die auf eine Überverstärkung hindeuten. Für dieses Beispiel wurden 11 Zyklen für die großflächige PCR (blaue Box) ausgewählt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
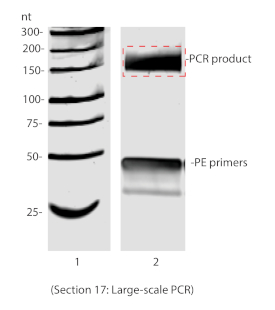
Abbildung 6 : Großflächige PCR aufgelöst auf 8% nicht-denaturing PAGE. Rote Box zeigt das Gelstück für Gelreinigung verbrauchen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 7 : Genom-Browser-Screenshot des MAPK1-Gens, der die Verteilung von FLAG-MAGOH:EIF4AIII Fußabdrücken als repräsentative Ergebnisse eines RIPiT zeigt. Rote Pfeile bezeichnen die erwarteten kanonischen EJC-Bindungsstellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Pbs | 137 mM NaCl 2,7 mM KCl 10 mM Na2HPO4bei 7H2O KH2PO4 pH 7,4 | |
| Quenching-Puffer | 2.5 M Glycin 2,5 mM Tris-Base | |
| Hypotonischer Lysepuffer (HLB) | 20 mM Tris-HCl pH 7,5 15 mM NaCl 10 mM EDTA 0,5% IGEPAL 0,1% Triton-X-100 1x Aprotinin* 1x Leupeptin* 1x Pepstatin* 1 mM PMSF (Phenylmethylsulfonylfluorid)* | *muss jedes Mal frisch hinzugefügt werden |
| Isotonic Wash Buffer (IsoWB) | 20 mM Tris-HCl pH 7,5 150 mM NaCl 0,1% IGEPAL | |
| Konjugationspuffer | 0,02% Polysorbat-20 1x PBS | |
| Löschen des Beispielpuffers | 100 mM Tris-HCl pH 6,8 4% SDS 10 mM EDTA | |
| 4xdNTPmix | 0,25 mM dGTP 0,25 mM dTTP 0,175 mM dATP 0,1625 mM dCTP 0,075 mM Biotin-dATP 0,0875 mM Biotin-dCTP | |
| 5x First-Strand Puffer w/o MgCl2 | 250 mM Tris-HCl pH 8 375 mM KCl | |
| RIPiT Verdünnungspuffer (1 ml) | 1 mL IsoWB 5 l 200x BSA 20 l 10% Triton-X-100 20 l 0,5 Mio. EDTA 1x Aprotinin* 1x Leupeptin* 1x Pepstatin* 1 mM PMSF* | *muss jedes Mal frisch hinzugefügt werden |
| 2x Entsendelastpuffer | 3 mL 5x TBE 1.8 g Ficoll Typ 400 6,3 g Harnstoff 3 mg Bromphenolblau 3 mg Xylolcyanol Lautstärke auf 15 ml ddH2O einstellen | Um in lösungsweise in Sendezeit zu gelangen, das Rohr in Wasser in ein Becherglas geben und 10–15 min auf der Kochplatte kochen. Färbe nach Einstellung des Volumens auf 15 ml hinzufügen |
| Harnstoff-PAGE Gel | 6 M Harnstoff Acrylamid:Bisacrylamid (40% [w/v]) zu einem angemessenen Prozentsatz 0.5x TBE 0,01% TEMED | |
| DNA-Elutionspuffer | 300 mM NaCl 1 mM EDTA | |
| Streptavidin Bead Wash Buffer | 0,5 M NaOH 20 mM Tris-HCl pH 7,5 1 mM EDTA |
Tabelle 1: Puffer.
Diskussion
Wir besprechen hier einige wichtige Überlegungen, um RIPiT erfolgreich durchzuführen. Vor allem müssen einzelne IPs optimiert werden, um bei jedem Schritt eine höchstmögliche Effizienz zu erzielen. Die Menge an FLAG-Agarose-Perlen für die hier beschriebene Eingangsanzahl von Zellen hat sich für eine Vielzahl von Proteinen, die wir getestet haben, als robust erwiesen. Da nur ein kleiner Bruchteil der Partnerproteine mit dem FLAG-Protein ko-immunpräzipiert wird, ist die Menge an Antikörpern, die für eine effiziente zweite IP benötigt werden, in der Regel gering (weniger als 10 g). Kleine RIPiT (von einer 10 cm Platte) gefolgt von Western Blot Überprüfung von Proteinen in jeder Fraktion während der beiden Immunpräzipitungsschritte erweisen sich als äußerst nützlich, um die Effizienz und Spezifität des Verfahrens vor der Skalierung zu bewerten. Sowohl gezielte Proteine als auch alle anderen erwarteten interagierenden Proteine im Komplex sollten in der Elution nachgewiesen werden. Es ist auch vorteilhaft, proteine in den erschöpften Lysaten (ungebunden an die FLAG-Agarose oder magnetische Perlen) zu prüfen, um eine gute Schätzung der Immunpräzipitationseffizienz und des Prozentsatzes der Proteine zu haben, die sich zu einem Komplex zusammenfügen. Darüber hinaus informiert diese Analyse auch, ob die RNase-Verdauungsbedingungen ausreichen, um RNA-abhängige Wechselwirkungen von RNA-unabhängigen Wechselwirkungen innerhalb eines RNP zu trennen. Daher ist es genauso wichtig, eine negative Kontrolle einzubeziehen, idealerweise ein RBP, der nicht mit dem RNP von Interesse in Verbindung steht. In Abbildung 3Aist beispielsweise HNRNPA1 im Eingang vorhanden, wird aber in der RIPiT-Elution nicht erkannt. HNRNPA1 ist ein RBP, das nicht direkt mit dem EJC interagiert, sondern indirekt mit dem EJC interagiert, wenn EJC und HNRNPA1 an dasselbe RNA-Molekül gebunden sind. Der Nachweis des Negativkontrollproteins in der Elution deutet entweder auf eine schlechte RIPiT-Spezifität oder eine unzureichende RNA-Fußabdruckung hin. In einem solchen Fall spiegeln die erhaltenen RNA-Fußabdrücke die Fußabdrücke des Proteins nicht vollständig wider. Fußabdrücke der Größe 50-200 nt werden für die nachfolgende RNA-Seq empfohlen. Die Dauer der RNase I-Behandlung oder die Menge des verwendeten Enzyms kann optimiert werden, um die gewünschte Größe Fußabdrücke zu erhalten. Beachten Sie, dass das Beste-Case-Szenario sein wird, ein gutes Signal im gewünschten Größenbereich zu erhalten, und es ist unvermeidlich, längere und kürzere RNAs auch unter den optimalsten Bedingungen zu haben. RIPiT kann auch verwendet werden, um Bindungsstellen eines einzelnen RBP zu erhalten. In einem solchen Fall kann dasselbe Protein mit zwei verschiedenen Antikörpern immunisiert werden, zuerst mit einem Antikörper gegen ein Affinitätstag und dann mit Antikörpern gegen das Protein selbst18. Schließlich kann eine negative Kontrolle RIPiT parallel von Zellen durchgeführt werden, die ein FLAG-markiertes Kontrollprotein (z. B. grünes fluoreszierendes Protein) in Kombination mit Antikörper gegen ein Protein gegen ein unabhängiges Protein im zweiten UZ exzieren.
Trotz seiner vielen Vorteile ist es wichtig, einige Einschränkungen des RIPiT-Ansatzes und mögliche Abhilfemaßnahmen in Betracht zu ziehen. Das Erfordernis der Affinitätselution nach der ersten Reinigung erfordert die biologische Quelle, um ein markiertes Protein auszudrücken. Wenn ein standortspezifisches Rekombinationssystem in der Zelllinie oder dem von Interesse lebenden Organismus nicht verfügbar ist, kann ein kurzes Affinitäts-Tag wie ein FLAG-Tag (8 Aminosäuren) am endogenen Gen-Locus mit CRISPR/Cas-basiertem Genom-Editing-Ansatz19eingeführt werden. Das FLAG-Tag ist ein ideales Epitop für diesen Ansatz, da der FLAG-Antikörper für die Affinitätselution gut geeignet ist und hohen Ionenstärken und milden Denaturierungsbedingungen standhält, die in Kombination mit formaldehyd-Vernetzung verwendet werden können. Eine weitere Einschränkung des RIPiT-Ansatzes ist die Anforderung an einen großen Input von zellulärem Material. Dies kann bis zu einem gewissen Grad unvermeidbar bleiben, da nur ein kleiner Prozentsatz eines RBP wahrscheinlich mit anderen Proteinen im RNP interagiert. Noch verbesserte Bibliotheksvorbereitungsansätze können dazu beitragen, den großen Inputbedarf zu senken. Mögliche Ideen zur weiteren Rationalisierung dieser Schritte sind die Durchführung der RNA 3'-End-Dephosphorylierung und Adapterligation auf den magnetischen Perlen unmittelbar nach der zweiten IP-Wäsche und vor der endgültigen Elution von RNP. Ein solcher Ansatz wird erfolgreich in aktuellen CLIP-Seq-Verfahren und in einer kürzlich beschriebenen Variante von RIPiT20implementiert. Solche Änderungen werden auch mehrere zeitaufwändige RNA-Reinigungsschritte aus den frühen Phasen des Bibliotheksvorbereitungsverfahrens entfernen. Im Gegensatz zu CLIP, das eine Nukleotid-Auflösung der Vernetzungsstelle eines RBP auf der RNA bietet, bleibt die Auflösung von RIPiT-Footprints auf der Ebene von Dutzenden von Nukleotiden. Da RNPs mehrere RBPs enthalten können, enthalten die RIPiT-angereicherten RNA-Sites eine Mischung aus Bindungsstellen vieler RBPs. Da Konsenssequenzen, die von einzelnen RBPs gebunden sind, in einem rasant wachsenden Tempo aufgedeckt werden und jetzt leicht verfügbar sind21,22,23, können diese Informationen genutzt werden, um das Sortiment der RBP-Sites zu dekonvolvenieren. in RIPiT-Ausgängen angereichert. Ungeachtet dieser Herausforderungen ist RIPiT-Seq ein effektives Verfahren zur Erfassung von RNA-Fußabdrücken dynamischer, heterogener und sogar vorübergehender RNP-Komplexe, die einzigartige Einblicke in das Innenleben von RNA-Maschinen geben können, die zelluläre funktion.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde durch den NIH-Zuschuss GM120209 (GS) unterstützt. Die Autoren danken dem OSUCCC Genomics Shared Resources Core für ihre Dienste (CCC Support Grant NCI P30 CA16058).
Materialien
| Name | Company | Catalog Number | Comments |
| Anti-FLAG Affinity Gel | Sigma | A2220 | |
| ATP, [γ-32P]- 3,000 Ci/mmol 10 mCi/mL EasyTide, 250 µCi | PerkinElmer | BLU502A250UC | |
| BD Disposable Syringes with Luer-Lok Tips (200) | Fisher | 14-823-435 | |
| Betaine 5M | Sigma | B0300 | |
| biotin-dATP | TriLink | N-5002 | |
| biotin-dCTP | Perkin Elmer | NEL540001EA | |
| Branson Sonifier, Model SSE-1 | Branson | ||
| CircLigase I | VWR | 76081-606 | ssDNA ligase I |
| DMEM, High Glucose | ThermoFisher | 11995-065 | |
| DNA load buffer NEB | NEB | ||
| Dynabeads Protein A | LifeTech | 10002D | |
| Flp-In-T-REx 293 Cell Line | ThermoFisher | R78007 | |
| GeneRuler Low Range DNA Ladder | ThermoScientific | FERSM1203 | |
| Hygromycin B | ThermoFisher | 10687010 | |
| Mini-PROTEAN TBE Gel 10 well | Bio-Rad | 4565013 | |
| Mini-PROTEAN TBE-Urea Gel | Bio-Rad | 4566033 | |
| miRCAT-33 adapter 5′-TGGAATTCTCGGGTGCCAAGGddC-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Mirus transIT-X2 transfection reagent | Mirus | MIR 6004 | |
| Mth RNA ligase | NEB | E2610S | |
| PE1.0 5′-AATGATACGGCGACCACCGAGATCTACACT CTTTCCCTACACGACGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| PE2.0 5′-CAAGCAGAAGACGGCATACGAGATCGGTCTC GGCATTCCTGCTGAACCGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Phenol/Chloroform/Isoamyl Alcohol (25:24:1, pH 6.7, 100 mLl) | Fisher | BP1752I-100 | |
| Purple Gel Loading Dye (6x) | NEB | NEB #7025 | |
| Q5 DNA Polymerase | NEB | M0491S/L | |
| RNase I, E. coli, 1,000 U | Eppicenter | N6901K | |
| SPIN-X column | Corning | CLS8160-24EA | |
| Streptavidin beads | ThermoFisher | 60210 | |
| Superscript III (SSIII) | ThermoScientific | 18080044 | reverse transcriptase enzyme |
| SybrGold | ThermoFisher | S11494 | gold nucleic acid gel stain |
| T4 Polynucleotide Kinase-2500U | NEB | M0201L | |
| T4RNL2 Tr. K227Q | NEB | M0351S | |
| Tetracycline | Sigma | 87128 | |
| Thermostable 5´ App DNA/RNA Ligase | NEB | M0319S | |
| TruSeq_SE1 5′-pGGCACTANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE10 5′-pGGTGTTCNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE11 5′-pGGTAAGTNNNNNAGATCGGAA GAGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE12 5′-pGGAGATGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE2 5′-pGGGTAGCNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE35′-pGGTCGATNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE4 5′-pGGCCTCGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE5 5′-pGGTGACANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE6 5′-pGGTAGACNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE7 5′-pGGGCCCTNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCT TCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE8 5′-pGGATCGGNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTT CCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE9 5′-pGGACTGANNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Typhoon 5 Bimolecular Imager | GE Healthcare Life Science | 29187191 |
Referenzen
- Karousis, E. D., Nasif, S., Mühlemann, O. Nonsense-mediated mRNA decay: novel mechanistic insights and biological impact. Wiley Interdisciplinary Reviews: RNA. 7 (5), 661-682 (2016).
- Ivanov, P. V., Gehring, N. H., Kunz, J. B., Hentze, M. W., Kulozik, A. E. Interactions between UPF1, eRFs, PABP and the exon junction complex suggest an integrated model for mammalian NMD pathways. The EMBO Journal. 27 (5), 736-747 (2008).
- Papasaikas, P., Valcárcel, J. The Spliceosome: The Ultimate RNA Chaperone and Sculptor. Trends in Biochemical Sciences. 41 (1), 33-45 (2016).
- Jensen, K. B., Darnell, R. B. CLIP: Crosslinking and ImmunoPrecipitation of In Vivo RNA Targets of RNA-Binding Proteins. Methods in Molecular Biology. 488, Clifton, N.J. 85-98 (2008).
- Garzia, A., Morozov, P., Sajek, M., Meyer, C., Tuschl, T. PAR-CLIP for Discovering Target Sites of RNA-Binding Proteins. mRNA Decay: Methods and Protocols. 1720, 55-75 (2018).
- Konig, J., et al. iCLIP - Transcriptome-wide Mapping of Protein-RNA Interactions with Individual Nucleotide Resolution. Journal of Visualized Experiments. (50), e2638(2011).
- Sibley, C. R. Individual Nucleotide Resolution UV Cross-Linking and Immunoprecipitation (iCLIP) to Determine Protein-RNA Interactions. RNA Detection: Methods and Protocols. 1649, 427-454 (2018).
- Wheeler, E. C., Van Nostrand, E. L., Yeo, G. W. Advances and challenges in the detection of transcriptome‐wide protein-RNA interactions. Wiley Interdisciplinary Reviews: RNA. 9 (1), (2018).
- Singh, G., et al. The Cellular EJC Interactome Reveals Higher-Order mRNP Structure and an EJC-SR Protein Nexus. Cell. 151, 750-764 (2012).
- Singh, G., Ricci, E. P., Moore, M. J. RIPiT-Seq: A high-throughput approach for footprinting RNA:protein complexes. Methods. 65, 320-332 (2014).
- Mabin, J. W., et al. The Exon Junction Complex Undergoes a Compositional Switch that Alters mRNP Structure and Nonsense-Mediated mRNA Decay Activity. Cell Reports. 25 (9), 2431-2446 (2018).
- Gehring, N. H., Lamprinaki, S., Kulozik, A. E., Hentze, M. W. Disassembly of Exon Junction Complexes by PYM. Cell. 137 (3), 536-548 (2009).
- Dostie, J., Dreyfuss, G. Translation Is Required to Remove Y14 from mRNAs in the Cytoplasm. Current Biology. 12 (13), 1060-1067 (2002).
- Zünd, D., Gruber, A. R., Zavolan, M., Mühlemann, O. Translation-dependent displacement of UPF1 from coding sequences causes its enrichment in 3' UTRs. Nature Structural & Molecular Biology. 20 (8), 936-943 (2013).
- Gangras, P., Dayeh, D. M., Mabin, J. W., Nakanishi, K., Singh, G. Cloning and Identification of Recombinant Argonaute-Bound Small RNAs Using Next-Generation Sequencing. Argonaute Proteins: Methods and Protocols. 1680, 1-28 (2018).
- Heyer, E. E., Ozadam, H., Ricci, E. P., Cenik, C., Moore, M. J. An optimized kit-free method for making strand-specific deep sequencing libraries from RNA fragments. Nucleic Acids Research. 43 (1), 2(2015).
- Cameron, V., Uhlenbeck, O. C. 3'-Phosphatase activity in T4 polynucleotide kinase. Biochemistry. 16 (23), 5120-5126 (1977).
- Ricci, E. P., et al. Staufen1 senses overall transcript secondary structure to regulate translation. Nature Structural & Molecular Biology. 21 (1), 26-35 (2014).
- Lackner, D. H., et al. A generic strategy for CRISPR-Cas9-mediated gene tagging. Nature Communications. 6, 10237(2015).
- Metkar, M., et al. Higher-Order Organization Principles of Pre-translational mRNPs. Molecular Cell. 72 (4), 715-726 (2018).
- Giudice, G., Sánchez-Cabo, F., Torroja, C., Lara-Pezzi, E. ATtRACT-a database of RNA-binding proteins and associated motifs. Database: The Journal of Biological Databases and Curation. 2016, (2016).
- Paz, I., Kosti, I., Ares, M., Cline, M., Mandel-Gutfreund, Y. RBPmap: a web server for mapping binding sites of RNA-binding proteins. Nucleic Acids Research. 42, Web Server issue 361-367 (2014).
- Sundararaman, B., et al. Resources for the Comprehensive Discovery of Functional RNA Elements. Molecular Cell. 61 (6), 903-913 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten