Method Article
RNA足迹的识别:通过RNA免疫沉淀在串联中的蛋白质复合物,然后测序(RIPiT-Seq)
摘要
在这里,我们提出一个协议,以丰富内源RNA结合位点或"足迹"的RNA:蛋白质(RNP)复合物从哺乳动物细胞。这种方法涉及RNP亚单位的两个免疫沉淀,因此被称为RNA免疫沉淀串联(RIPiT)。
摘要
RNA免疫沉淀串联(RIPiT)是一种在RNA:蛋白(RNP)复合物内丰富一对蛋白质的RNA足迹的方法。RIPiT 采用两个纯化步骤。首先,标记的RNP亚单位的免疫沉淀后是轻度RNase消化和随后的非变性亲和洗脱。另一个RNP亚单位的第二次免疫沉淀允许扩充定义的复合物。在RNA和蛋白质变性后,RNA足迹被转换成高通量的DNA测序库。与更流行的紫外线 (UV) 交联后免疫沉淀 (CLIP) 方法以丰富 RBP 结合位点不同,RIPiT 是紫外线交联独立。因此,RIPiT可应用于RNA相互作用中存在的众多蛋白质,这些蛋白质对RNA调控至关重要,但不直接接触RNA或紫外线交联,与RNA接触不良。RIPiT 中的两个纯化步骤提供了另一个优势,用于识别感兴趣的蛋白质与另一个共同因子合作作用的结合位点。双重纯化策略也有助于通过限制背景来增强信号。在这里,我们提供了一个逐步的过程来执行RIPiT,并从分离的RNA足迹生成高通量测序库。我们还概述了 RIPiT 的优势和应用,并讨论了它的一些局限性。
引言
在细胞内,RNA与蛋白质复合体存在,形成RNA:蛋白质复合物(RNPs)。RNPs围绕RNA结合蛋白(RBPs,那些直接结合RNA的蛋白质)进行组装,但也包括非限制性核糖核酸(那些结合RPS但非RNA的),并且在性质上通常是动态的。RP 及其辅助因素在 RnP 中共同发挥作用,以执行监管职能。例如,在无意义介导的mRNA衰变(NMD)通路中,UPF蛋白(UPF1、UPF2和UPF3b)识别过早终止的核糖体。每个UPF蛋白都可以与RNA结合,但只有当它们聚集在一起时,活性NMD复合物才会开始形成。在这个复合体中,UPF1通过非RBP SMG1进一步通过磷酸化激活,这种UPF1激活最终导致mRNA衰变诱导因子1的招募,2。在此示例中,RBP 需要非 RBP 辅助因素来招募和激活触发 NMD 的 RNP 复合物。然而,RnPs的另一个属性是它们的组成异质性。考虑存在于不同E、A、B或C复合物中的拼接体。不同的拼接体复合物具有重叠和不同的蛋白质3。为了研究RNP功能,必须阐明哪些RNA被RBP及其相关蛋白质所束缚。有许多方法可以实现它,每种方法都有其明显的优点和缺点4,5,6,7。
广泛流行的方法来识别RBP结合位点 - 交联后免疫沉淀(CLIP)及其各种变异 - 依靠紫外线(UV)光将RBP与RNA8交联。然而,对于RNP中不直接接触RNA的非RRNA,这不是一种有效的方法。在这里,我们描述了一种适用于RSP和非限制性商业惯例的替代方法,用于分离和识别其RNA结合位点。这种方法称为RNA免疫沉淀串联(RIPiT)由两个免疫沉淀步骤组成,与单个纯化相比,这有助于实现更高的特异性(图1)9,10。与 CLIP 相比,单个免疫沉淀 (IP) 步骤可以以较低的严格性执行,因此 RIPiT 不依赖于在免疫沉淀期间能够承受强洗涤剂存在的抗体的可用性。RIPiT最独特的优势是能够以两个不同的纯化步骤靶向两种不同的蛋白质;这提供了一个强大的方法来丰富一个成分独特的RNP复合物从其他类似的复合物11。
对 RIPiT 程序的微小变化可以进一步增强 RNP 富集性。例如,RNA-蛋白质或蛋白质-蛋白质-蛋白质在RNPs内的一些相互作用是暂时的,可能难以有效地纯化这种复合物的足迹。为了稳定这种相互作用,在细胞裂解和RIPiT之前,RNPs可以在细胞内与甲醛交联。例如,我们观察到,外子结复合体 (EJC) 核心因子 EIF4AIII 和 EJC 拆解因子之间的弱相互作用,PYM12可以通过甲醛处理来稳定,以便更多 RNA 足迹得到丰富(数据不如图所示)。在细胞收获和RIPiT之前,细胞也可以用药物治疗,以稳定或丰富特定状态的RNP。例如,在研究在翻译过程中从mRNA中去除的蛋白质(例如,EJC 13,UPF114)时,使用翻译抑制剂(如紫霉素、环霉素或哈林托宁)进行治疗可导致增加的占用率。RNA上的蛋白质。
从RIPiT中恢复的RNA量通常较低(0.5-10 pmoles,即10-250 ng RNA,考虑到平均RNA长度为75 nt)。主要原因是,只有一小部分特定蛋白质与RNPs内的其他蛋白质复合存在(第一步中任何"自由"蛋白IP在第二个IP期间丢失)。为了从这个RNA生成RNA-Seq库,我们在此还概述了以前发布的协议,适用于这种低RNA输入15,16(图2),在3中产生高通量测序就绪样品。天。
研究方案
1. 建立稳定的 HEK293 细胞系,表达四环素诱导 FLAG 标记感兴趣的蛋白质 (POI)
- 种子 HEK293 细胞具有一个稳定增长的 Flp 重组靶 (FRT) 位点,在生长培养基的密度为 10 x 104细胞/mL(Dulbeco 的改性 Eagle 的培养基 [DMEM] = 10% 胎儿牛血清 [FBS] = 1% 青霉素-链球菌素 [penn/链球菌]井板。允许细胞在37°C和5%CO2(所有后续步骤的标准生长条件)的加湿培养箱中一夜之间生长。
- 第二天,细胞应为+70%的汇入。根据转染试剂协议,转染含有FRT位点的HEK293细胞,PCDNA5-TETO-FLAG-POI:pOG44的比率为9:1。
注:FLAG 标记具有序列主题 DYKDDDDK(D = 阿斯巴酸、Y = 酪氨酸和 K = 莱氨酸)。 - 24小时后,开始选择抗生素。去除介质,加入新的生长介质,辅以100微克/mL湿霉素。在72小时内,未转染的细胞应开始死亡。
- 每48-72小时,改变生长介质和补充新鲜湿霉素。
- 经过2周的选择后,可连续转染细胞的离散菌落将开始出现。一旦肉眼看到菌落,在板中加入1 mL的胰蛋白酶,并在37°C下孵育5分钟。在DMEM中重新悬浮细胞,将细胞转移到新的10厘米板中,并将体积调节到10mL新鲜生长培养基,辅以100μg/mL湿霉素。允许板达到+80%的汇合,以进一步扩大细胞,以准备永久库存。
- 确定获得实验 FLAG-POI 表达的最佳水平所需的四环素 (Tet) 量。以12孔形式生长细胞,并在0-1000纳克/mL范围内进行16-24小时的Tet滴定。
注:对于高达+60 kDa的蛋白质,PAGE标记的蛋白质和内源拷贝可在硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)上解决,以比较FLAG-POI的表达水平与其内源性对应物。对于较大的蛋白质,Tet诱导样品的信号强度可以与非诱导样品进行比较。四环素库存溶液可在100%乙醇中制备1mg/mL。用于细胞培养工作的库存稀释应在无菌水或磷酸盐缓冲盐水 (PBS) 中。四环素股票应每月新鲜准备一次。
2. 四环素诱导和RIPiT程序的培养细胞
- 种子在15厘米板的生长培养基中,以3 x 105细胞/mL的密度稳稳地集成FLAG-POI HEK293。允许细胞在37°C和5%CO2下生长。
注:一般来说,3至5个15厘米的板会产生±2-20 pmol的RNA足迹,这取决于感兴趣的RNP的丰度。如果RNP在严格的条件下是甲醛交联和纯化的,那么可能需要两倍的输入。 - 在细胞收获前,将四环素加入预先确定的最佳浓度,以诱导FLAG-POI的表达(参见步骤1.6)16-24小时。
注:没有必要改变增长介质。细胞应在播种后或板体为±80%时,准备收获约72小时。重要的是要避免细胞变得汇入。
3. 细胞收获、甲醛处理和细胞解理
- 用每15厘米板15 mL的冷冻PBS轻轻清洗单层细胞。然后在30 mL的PBS中刮掉细胞。如果细胞在收获前用药物治疗,用药物补充PBS。将所有细胞收集到50 mL锥形管中。
注:为了保持弱相互作用,细胞可与甲醛交联:将甲醛从步骤3.1添加到细胞悬浮液中,最终浓度为0.1%,并在室温下在摇臂上孵育10分钟。加入3 mL的淬火缓冲液(表1))和岩石5分钟。 - 在400 x g下粒细胞在4°C下10分钟,并丢弃上清液。
- 4 mL 的冰冷下发性莱沙缓冲液 (HLB;表 1)。使用 P1000 移液器重新悬浮细胞。转移到5 mL管,在冰上孵育10分钟。
- 将液兰在冰浴中放置,以 10% 振幅声波 30 秒,1 s 脉冲,暂停 2 秒。然后,通过加入108 μL的5M NaCl,将盐浓度调整到150 mM。
注:甲醛交联样品可以通过在解液缓冲液中分别包括0.1%的SDS和脱氧胆酸钠,进行更严格的解糖和纯化。 - 在 4°C 下通过离心在 21,000 x g下清除分热10分钟。在标记管中收集20μL上清液(细胞提取物),用于输入中蛋白质水平的西方斑点分析(见图3 A)。
注:当莱沙在离心机中时,可以清洗FLAG腺珠(参见步骤4.1)。FLAG 角胶珠应在 4 mL 的冰冷等位洗液缓冲液中预洗 3x(IsoWB,表 1)。
4. 标志免疫沉淀
- 在 5 mL 管中涂抹步骤 3.5 至 750 μL 预洗的 FLAG agarose 珠子的剩余上清液(洗过的 FLAG agarose 珠的床体积为 375 μL)。在4°C下孵育FLAG角珠和细胞提取物1-3小时,温和混合。
注:这种量的FLAG agarose珠应该足以用于广泛的蛋白质,但可以优化,如果需要的话。 - 在 4°C 下,在 400 x g下离心 1 分钟,通过小球角角角。在标记管中收集20μL的上清液,用于西方血泡分析耗尽细胞提取物中的蛋白质水平(见图3 A)。丢弃剩余的上清液。
- 要洗FLAG agarose 珠子,请加入 4 mL 的 IsoWB 并重新悬浮。在 4°C 下,在 400 x g处的颗粒珠 1 分钟。小心地去除上清液。重复 4 次。
注:对于严格洗涤甲醛交联IP,0.1%的SDS和脱氧乙酸钠可以包含在洗涤缓冲液的前两个洗涤步骤。
5. RNase I 消化
- 在 750 μL 的 IsoWB 中稀释 RNase I 至 0.002-0.01 单位/mL(需要根据经验确定所需占地面积尺寸的适当浓度)。将 IsoWB-RNase I 加入洗涤 FLAG 角质珠,并在 4°C 下温和混合孵育 10 分钟。
- 在 4°C 下,在 400 x g处的颗粒珠 1 分钟。在标记管中收集 20 μL 上清液 (RNase I 洗脱), 用于西斑分析。丢弃 IsoWB-RNase I 并按照步骤 4.3 中所述使用 IsoWB 清洗 FLAG 角胶珠 4x。
6. 亲和力洗脱
- 准备洗脱缓冲液(在IsoWB中为250纳克/mL的FLAG肽)。将375 μL的洗脱缓冲液涂至FLAG角胶珠子上,在4°C下轻轻摇动1-2小时。 Pellet FLAG角质角胶珠子,并收集FLAG IP中西方蛋白斑的15μL等分(见图3A)。
注:当进行亲和洗脱时,可以执行第 7 节。
7. 磁珠抗体结合
- 清洗 50 μL 磁珠(即动态珠;材料表)3x 在 1 mL IsoWB 中,在 1.5 mL 管中。在100 μL的偶联缓冲液中重新悬浮磁珠。添加适当数量的抗体(用于 IP 的抗体的确切数量需要为每个抗体进行经验确定)。
注:蛋白质 A 磁珠最适合兔子产生的抗体,而蛋白质 G 磁珠更适合小鼠抗体。供应商网站上提供磁珠兼容性图,以选择适合每种抗体的珠子。 - 在结合缓冲液中清洗磁珠2倍(表1)。在室温下孵育混合物至少10分钟。在375μL的RIPiT稀释缓冲液中重新悬浮磁珠。储存在冰上,直到下一步。
8. 第二次免疫沉淀
- 将步骤 6.1 中剩余的 FLAG 亲缘洗脱应用于与抗体耦合的磁珠,以对抗感兴趣的蛋白质。在4°C下温和混合孵育1-2小时,在磁铁上捕获磁珠,并收集15μL的上清液,通过西斑分析未结合的蛋白质。用 1 mL 的 IsoWB 清洗磁珠 7 倍。
9. 变性洗脱
- 向磁珠中加入100 μL的透明样品缓冲液(表1),用P200移液器重新悬浮。在冰上孵育10分钟,轻轻轻拂,定期重新悬浮珠子。
- 在磁体上捕获磁珠并收集 15 μL 的洗脱,通过西斑中分析 RIPiT 洗脱中的蛋白质(参见图 3A)。将剩余的洗脱转移到新的标有 1.5 mL 的管中。
注:如果样品是甲醛交联,则必须在65°C下孵育样品1小时,以反向交联。 - 对以不同步骤收集的样本(输入、FLAG IP 损耗、FLAG IP、第二次 IP 损耗、第二次 IP 洗脱)执行西方印迹。带抗体的Blot对两种诱饵蛋白,其另一个相互作用者,如果已知,和至少一个非相互作用的RBP作为阴性对照(图3A)。
10. RNA提取和末端固化
- 在RIPiT洗脱中,加入320μL的无RNase ddH2O、400μL的苯基异醇(PCIAA,pH 4.5),在室温下旋转30秒,在室温下旋转5分钟。 加入35μL的3M醋酸钠,1μL的1MMgCl2,10μg的糖原,和1mL的100%乙醇。在-20°C下孵育过夜。
- 对于颗粒RNA,在12,000 x g的离心机在4°C下30分钟。在70%乙醇中清洗RNA。
- 要去除RNase I裂解后RNA上留下的3'磷酸盐,在17μL无RN酶ddH2O中重新悬浮RNA颗粒,并加入2μL的10xT4多核苷酸激酶(PNK)缓冲液(材料表)和1μL的T4 PNK。在37°C孵育30分钟。
注:T4 PNK的3'磷酸酶活性在pH617时具有最佳活性。PNK反应缓冲液针对T4 PNK的5'激酶活性进行了优化,pHH为7.6。虽然已经尝试调整末端固化反应的pH,但可以进一步优化此步骤。 - 在管中加入 380 μL 的无 RNase ddH2O 和 400 μL PCIAA pH 4.5。涡旋30秒,在12,000 x g下离心5分钟。收集水相,加入35μL的3M醋酸钠,1μL的1MMgCl2,10μg的糖原,1mL的100%乙醇。
- 在-20°C下孵育过夜。上面用70%乙醇进行颗粒和清洗RNA。在4.5 μL无RNase水中重新悬浮RNA。
11. RNA足迹大小和丰度估计
-
成功的RIPiT有望产生1个或更多的RNA片段。要量化实际产量,将 0.7 μL 的 RIPiT RNA(总产量的 ±1/6)转移到新管中。加入 2 μL 的 10x T4 PNK 缓冲液,1 μL 的 1 mM ATP,40 μCi =32P-ATP(0.5±1.0 μL 的库存),以及 1 μL 的 T4 PNK。将体积调节至10μL,在37°C下孵育30分钟。
- 在平行的PNK反应中,将合成RNA或DNA寡核糖核酸(20-40 nt)的低范围DNA梯和0.1pmol贴上标签,以使用尺寸和数量标准。
- 在 26% 尿素-PAGE 凝胶(20 x 27 x 0.45 mm3) 上解决标记的 RNA/DNA。凝胶必须在预运行前和装载样品之前在 35 W 的 Flush 井上预运行 30 分钟,并在 35 W 处运行,直到溴酚蓝色染料正面几乎到达凝胶的末端。
- 小心地将玻璃板上的凝胶取出到一块 8 x 11 英寸滤纸上。将凝胶放在纸张上,放入凝胶干燥装置中,并盖上一块塑料包装。在80°C干燥凝胶1小时,真空。
- 将干燥的凝胶暴露在磷化物中过夜或直到检测到足够的信号。图像磷光屏。来自RIPiT的优质RNA应作为涂片出现在车道上,具有最小的突出带状(图3B)。要量化RNA,请将RIPiT通道中所需大小RNA片段的信号强度与标记合成寡核苷酸0.1pmol的信号进行比较。
注:或者,RNA足迹大小和量可以使用高灵敏度生物分析仪(图3C)进行验证。
12. 适配器连接
- 制备RIPiT RNA,使至少3pmol的RNA溶解在3.8μL的水中。
- 在0.2 mL聚合酶链反应(PCR)管中,结合3.8μL的RNA,1μL的miR-CAT-33预脱脂适配器(7μM)(材料表)。在 65°C 的热循环器上孵育混合物 10 分钟,16°C 孵育 5 分钟,然后在 4°C 下保持。
注:预脱酸链接器可以从寡聚物合成服务订购,也可以从任何寡核聚子合成服务中定制未脱酸的DNA寡聚子,可以使用Mth RNA胶合酶(材料表)和凝胶纯化进行脱酸。 - 在同一管中,加入1.5μL的10x T4 RNA胶合酶缓冲液,7.5μL的50%聚乙烯乙二醇8000(PEG-8000),0.75 μL的20m二硫二醇(DTT),和0.45μL的T4 RNL2 Tr. K227Q(材料表)。
注:50% PEG-8000随附RNA连带和缓冲液。PEG-8000 溶液具有粘性,应缓慢移液。 - 在热循环器中孵育反应在30°C下6小时,在65°C下加热灭活10分钟,然后在4°C下保持。
13. 反向转录
- 在步骤12.4起的结扎混合物中,加入11.25 μL的4x脱氧核苷酸三磷酸(dNTP)混合物,其中含有常规和生物素化dNTP(见表1)、1.0μL的10μM RT引物(材料表)和6.8μL 无RNase水。在65°C孵育5分钟,然后在4°C下孵育。
- 将管转移到冰上,并在无MgCl2(表1)、2.25 μL的100mM DTT、1.2 μL逆转录酶的5x第一链(FS)缓冲液中加入9.0 μL(材料表)。
- 在55°C的热循环器中孵育30-60分钟。热在70°C下灭活逆转录酶15分钟,并将样品保持在4°C。
14. RT产品的净化
- 在RT反应中加入45μL的2倍尿素负载缓冲液(表1)。在45μL中稀释1μg低范围DNA阶梯,并加入45μL的2倍尿素负载缓冲液。
- 准备10%尿素-PAGE凝胶(20 x 28 x 0.15厘米3;表 1)用8孔梳子。使用注射器或移液器,用0.5x Tris/硼酸盐/EDTA(TBE)缓冲液冲洗井。
注:上述自制凝胶在将扩展RT产品与未扩展的RT底漆分离时,具有更好的分辨率。作为替代,预铸尿素-PAGE凝胶也可以使用(材料表)。由于预铸凝胶允许每口井的最大体积较小,因此样品需要分成多个井。预铸造凝胶应在 150–200 V 下运行。 - 在 35 W 下预运行凝胶 30 分钟。再次冲洗井,加载样品并在 35 W 下运行凝胶,直到溴酚蓝色染料正面从凝胶末端移至约 1 英寸。
注:在预运行和最终运行期间使用金属散热器,以防止凝胶过热。 - 在以0.5x TBE制备的1x金核酸凝胶染色溶液中染色凝胶5分钟。这种染料对光敏感,所以避免光曝光。
- 使用 520 nm 激发和 580 nm 发射过滤器在荧光扫描仪上成像凝胶,用于记录目的。如果荧光扫描仪不可用,请使用蓝光透射器。RT 产品应显示为从未扩展的 RT 引漆上方开始的涂片 (图 4)。
注:虽然金核酸凝胶染色剂很容易在带有紫外线光源的凝胶文档上可视化,但重要的是不要将有价值的 RT 产品暴露在紫外线下以防止损坏。 - 在蓝光透射器上可视化凝胶,并从凝胶中去除RT产物。建议切割DNA,其延伸范围为30-200 nt(图4)。将切除的凝胶片放在干净的表面上,将切片切成小块以增加表面积。小心地转移到1.5 mL管中,并加入800μL的DNA洗脱缓冲液(表1)。
- 在室温下,夜间与DNA洗脱缓冲液温和混合,孵育凝胶片。
- 通过将浆料通过放置在 2 mL 收集管中的醋酸纤维素滤芯(材料表),将洗脱缓冲液与凝胶分离。
- 在1.5 mL管中,用500μL的链球菌珠洗涤缓冲液清洗10μL链球菌(表1)。重复三次总的分。不要让珠子干涸。在10μL的DNA洗脱缓冲液中重新悬浮珠子(表1)。
- 将步骤 14.8 中从凝胶片中分离出来的洗脱缓冲液转移到包含已洗过的链球菌磁珠的管中。
- 在室温下,用温和混合孵育至少8小时。在磁体上捕获磁珠,去除上清液,在 10 μL 无 RNase 水中重新悬浮磁珠,并转移到 0.2 mL PCR 管中。
15. RT产品的循环化
- 在链球菌珠上捕获的RT产品在结合在珠子上时被循环化。在磁珠浆中,加入2.0μL的10x循环反应缓冲液,1.0μL的1mM ATP,1.0μL的50mMMnCl2,4.0μL的5M贝塔,1.0μL的ssDNA连带I(材料表),1.0μL的无RNase水。
- 在60°C下孵育热循环反应4小时。热在80°C加热10分钟,然后保持在4°C处,使ssDNA连带I失去活性。
16. 测试 PCR
- 在继续进行大规模PCR之前,使用一部分循环产物来确定每个样品的扩增周期的理想数量。此步骤有助于防止过度扩增并限制 PCR 伪影,因为 PCR 反应组分在较高的 PCR 周期时变得有限。
- 从步骤 15.2 起,用 4.0-6.0 μL 的循环产物制备 45 μL PCR 反应,从步骤 15.2,9.0 μL 的 5x 反应缓冲液,0.9 μL 10 M dNTP,2.25 μL 的 10 μM PE1.0 底漆 (材料表),2.25 μL 的 10 μM PE2.0 底漆 (材料表)和0.045 μL的高保真DNA聚合酶(材料表)和水。
- 混合反应良好,分成三个15μL反应。这三种反应中的每一个都将受到多变的PCR周期的影响。理想的周期数预计在 7 到 14 之间。因此,对 8、11 和 14 个周期执行测试 PCR。
- 使用以下 PCR 条件: 98 °C - 30 s;98 °C - 5 s;65 °C - 10 s;72 °C - 15 s;72 °C - 2 分钟;12 °C - 保持。
- 加入3 μL的6x凝胶加载染料,并在10%原生PAGE凝胶上解决,直到蓝色染料正面迁移3/4凝胶。使用金核酸凝胶染色和图像染色凝胶,如步骤14.4和14.5(图5)。
- 要选择理想的 PCR 周期数,请比较从不断增加的循环数中产生的 PCR 产品。选择产生最大数量的预期尺寸的积数的周期数,而不产生过度放大伪影(例如,DNA涂片比预期产品大得多,并且看不到 PE1.0 和 PE2.0 引漆的明显损耗(参见 中的红色箭头)图 5.
17. 大规模PCR
- 准备45μL PCR反应,如步骤16.2,并重复PCR。在 150 V 下在 10% 原生 PAGE 上解决 PCR,在蓝光转导器上染色 1x 金核酸凝胶染色和图像。
- 从凝胶中去除PCR产物,并转移到3mL注射器。使用注射器粉碎凝胶并挤出到 1.5 mL 管中。
注:循环后未扩展的RT产品产生151 bp的PCR产物。因此,在此步骤中,应调整选择大于 151 bp 的产品的大小(图 6)。 - 加入900 μL的DNA洗脱缓冲液,在室温下孵育过夜,温和混合。
- 将凝胶浆料转移到放置在 2 mL 收集管中的醋酸纤维素滤芯柱上。以 12,000 x g旋转 3 分钟,将上清液收集到新鲜管中。
- 在粉碎的凝胶中再加入400 μL的DNA洗脱缓冲液,并转移到1.5 mL管中。用温和混合孵育,再孵育4小时,进行第二次洗脱。
- 汇集所有洗脱液,分成3个管,每个管400μL。加入1mL的100%乙醇和10μg的糖原,沉淀DNA。涡旋,在-20°C下孵育至少2小时。
- 在4°C下,在12,000 x g下粒DNA30分钟。用70%乙醇清洗DNA颗粒。
- 通过移液小心地去除所有乙醇,并在20μL水中快速重新悬浮DNA颗粒。
注:在这个阶段,重要的是不要让DNA颗粒变干,因为干燥的DNA可以变性它。 - 使用一小部分DNA样品通过荧光计和高灵敏度DNA生物分析仪确定PCR产物的大小和浓度。现在,可以在其中一个平台上提交样本进行排序。
- 可处理序列读取(例如,适配器删除、修剪以保持序列 >30 Phred 分数)、与参考基因组对齐,并在 UCSC 基因组浏览器等浏览器上可视化(图 7)。
结果
成功的RIPiT将导致感兴趣的蛋白质和其他已知相互作用蛋白的免疫沉淀,并且不存在非相互作用的蛋白质。如图3 A所示,在RIPiT洗脱中检测到马戈和EIF4AIII,但HNRNPA1没有(通道6)。同时,通过自光成像(图3B)或生物分析仪(图3C)检测出与RNP复合物共同纯化的RNA足迹。Puromycin治疗有望增加EJC对RNA的占用率,在图3 B(比较通道2和3)中,在治疗的用于治疗的RIPiT中观察到了更强的RNA足迹信号。为深度测序生成样本需要将适配器与RNA进行分离,然后使用引板将RNA逆转转录到DNA中。逆转录步骤采用生物素化核苷酸,用于纯化逆转录产物。逆转录后,产品必须通过尿素-PAGE与未扩展的适配器分离(图4)。然后,逆转录产物被循环和PCR放大。适当数量的PCR周期不得过度放大循环产品。过度放大将导致引注损耗和异常 PCR 产物(参见图5,通道 4)。在无过度扩增证据的情况下,放大次数最大的周期数最适合用于大规模 PCR(图 5通道 3 和图6)。

图 1:概述 RIPiT 中主要步骤的架构。请点击此处查看此图的较大版本。
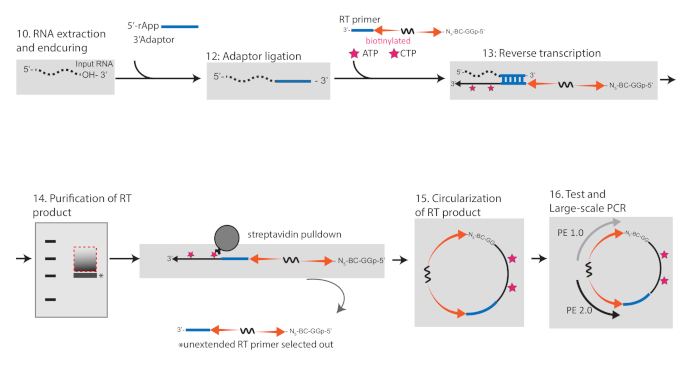
图 2:描述将RIPiT RNA转换为库进行高通量测序的工作流程的架构。请点击此处查看此图的较大版本。
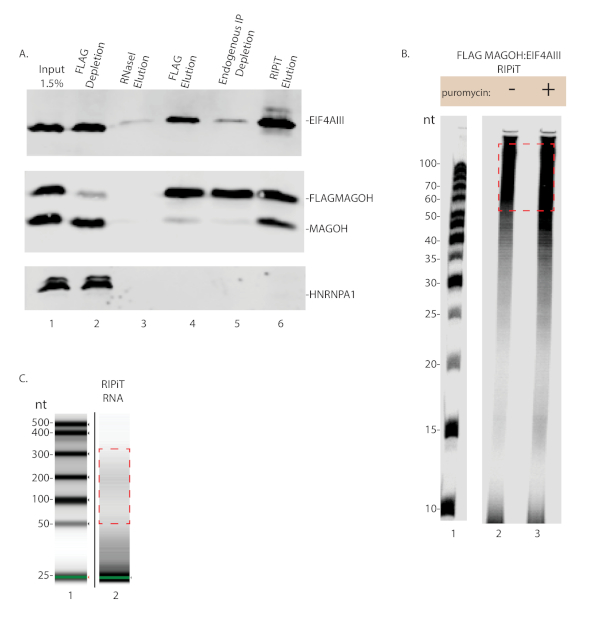
图 3:从RIPiT估计RNA和蛋白质产量。(A) 从 RIPiT 程序的每个主要步骤中纯化的蛋白质的西方斑点。(B) FLAG-MAGOH:EIF4AIII RIPiT 的RNA足迹的自动放射成像,比较已处理的和未经处理的细胞。红色框表示最终将转换为测序库的 RNA 足迹大小。(C) 使用生物分析仪进行可视化时,从 RIPiT 洗脱的RNA的轮廓在面板 B 中的通道 2 中。请点击此处查看此图的较大版本。

图 4:逆转录(RT)产物在10%尿素PAGE凝胶上解决,并沾染金核酸凝胶染色。红盒表示为延长RT产品的凝胶纯化而切除的凝胶区域。请点击此处查看此图的较大版本。
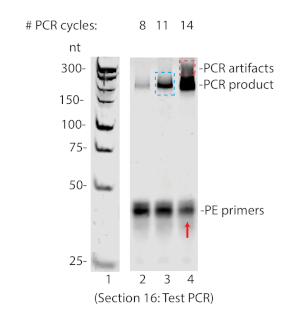
图 5:在8%非变性PAGE上解决的PCR测试。请注意,在 14 个周期(红色框)处出现的异常大 PCR 产品以及指示过度放大的引体(红色箭头)的平行损耗。对于此示例,为大型 PCR(蓝色框)选择了 11 个周期。请点击此处查看此图的较大版本。
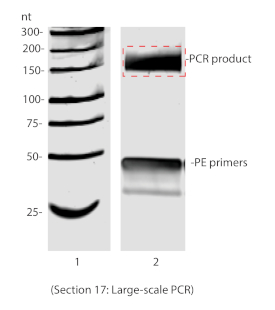
图 6:在8%非变性PAGE上解析的大规模PCR。红色框表示为凝胶纯化而切除了的凝胶片。请点击此处查看此图的较大版本。

图 7:MAPK1基因的基因组浏览器截图,显示FLAG-MAGOH:EIF4AIII足迹的分布,作为RIPiT的代表性结果。红色箭头表示预期的规范 EJC 绑定站点。请点击此处查看此图的较大版本。
| Pbs | 137 mM NaCl 2.7 mM KCl 10 mM Na2HPO4+7H2O KH2PO4 pH 7.4 | |
| 淬火缓冲器 | 2.5 M 甘氨酸 2.5 mM 三元底座 | |
| 低通性莱沙缓冲液 (HLB) | 20 mM Tris-HCl pH 7.5 15 mM 纳Cl 10 mM EDTA 0.5% IGEPAL 0.1% 特里顿-X-100 1x 阿普罗蒂宁* 1x 勒肽* 1x 肽* 1 mM PMSF(苯基硫磺酸氟化物)* | *每次必须添加新鲜 |
| 等子洗涤缓冲液 (IsoWB) | 20 mM Tris-HCl pH 7.5 150 mM 纳Cl 0.1% IGEPAL | |
| 共偶缓冲器 | 0.02% 聚二沙酯-20 1x PBS | |
| 清除样本缓冲区 | 100 mM Tris-HCl pH 6.8 4% SDS 10 mM EDTA | |
| 4xdNTPmix | 0.25 mM dGTP 0.25 mM dTTP 0.175 mM dATP 0.1625 mM dCTP 0.075 mM 生物锡-dATP 0.0875 mM 生物锡-dCTP | |
| 5x 一线缓冲器,带 MgCl2 | 250 mM Tris-HCl pH 8 375 mM KCl | |
| RIPiT 稀释缓冲液 (1 mL) | 1 mL IsoWB 5 μL 200x BSA 20 μL 10% 特里顿-X-100 20 μL 0.5M EDTA 1x 阿普罗蒂宁* 1x 勒肽* 1x 肽* 1 mM PMSF* | *每次必须添加新鲜 |
| 2x 变性负载缓冲器 | 3 mL 5x TBE 1.8 g 菲科尔类型 400 6.3克尿素 3毫克溴酚蓝 3毫克二甲二醇 将音量调节到 15 mL ddH2O | 要进入溶液中,将管子放入烧杯中,在热板上煮10-15分钟。 |
| 尿素-页凝胶 | 6 M 尿素 丙烯酰胺:二甲酰胺(40%[w/v])至适当百分比 0.5 x TBE 0.01% 特姆德 | |
| 脱氧核糖核酸洗脱缓冲液 | 300 mM 纳Cl 1 mM EDTA | |
| 链球菌珠洗液缓冲液 | 0.5 M NaOH 20 mM Tris-HCl pH 7.5 1 mM EDTA |
表 1:缓冲区。
讨论
在这里,我们将讨论一些成功执行 RIPiT 的关键注意事项。首先,必须优化单个 IP,以便在每个步骤中实现尽可能高的效率。此处描述的输入细胞的FLAG agarose 珠子的量已被证明对于我们测试过的各种蛋白质是可靠的。由于只有一小部分伙伴蛋白与 FLAG 蛋白共同免疫,因此高效第二 IP 所需的抗体量通常较低(小于 10 μg)。小规模的RIPiT(从一个10厘米的板)然后西方印版验证蛋白质在每个部分在两个免疫沉淀步骤证明非常有用,以评估效率和特殊性的程序之前扩大。应在洗脱中检测靶向蛋白和复合物中任何其他预期相互作用的蛋白质。对耗尽的解结中的蛋白质(未绑定到FLAG-agarose或磁珠)进行检测也有利于对免疫沉淀效率和组合成复合物的蛋白质的百分比进行很好的估计。此外,此分析还告知 RNase 消化条件是否足以将 RNA 依赖性相互作用与 RNP 内的 RNA 独立相互作用分离。因此,包含负控制同样重要,理想情况下,RBP 与感兴趣的 RNP 无关。例如,在图 3A中 ,HNRNPA1 存在于输入中,但在 RIPiT 洗脱中未检测到。HNRNPA1 是一种 RBP,它不直接与 EJC 相互作用,但当 EJC 和 HNRNPA1 与同一 RNA 分子绑定时,与 EJC 间接交互。在洗脱中检测负对照蛋白表明RIPiT特异性较差或RNA足迹不足。在这种情况下,获得的RNA足迹不会完全反映感兴趣的蛋白质的足迹。建议在后续RNA-Seq中使用尺寸为50-200 nt的足迹。RNase I治疗的持续时间或使用的酶量可以进行优化,以获得所需的尺寸足迹。请注意,最佳方案是在所需的大小范围内获得良好的信号,即使在最佳条件下,也不可避免地拥有更长和更短的 RNA。RIPiT 还可用于获取单个 RBP 的绑定站点。在这种情况下,同一蛋白质可以用两种不同的抗体进行免疫沉淀,首先使用抗体来对抗亲和体标记,然后用抗体对抗蛋白质本身。最后,可以从表达FLAG标记控制蛋白(例如绿色荧光蛋白)的细胞中并行执行负对照RIPiT,结合抗体对抗第二IP中不相关的蛋白质。
尽管RIPiT方法有许多优点,但必须考虑一些局限性和可能的补救措施。第一次纯化后亲和洗脱的要求要求生物来源来表达标记的蛋白质。如果感兴趣的细胞系或生物体中不存在站点特定的重组系统,可以使用CRISPR/Cas的基因组编辑方法19在内源基因位点引入一个短的亲和标记,如FLAG标记(8个氨基酸)。FLAG 标签是这种方法的理想表位,因为 FLAG 抗体非常适合亲和洗脱,可以承受高离子强度和温和的变性条件,可与甲醛交联结合使用。RIPiT 方法的另一个限制是需要大量输入蜂窝材料。这在一定程度上可能仍然是不可避免的,因为只有一小部分的RBP可能与RNP中的其他蛋白质相互作用。仍然改进的库准备方法可以帮助降低大量输入需求。进一步简化这些步骤的可能想法包括:在第二次 IP 洗洗后和 RNP 最终洗脱之前,立即对磁珠进行 RNA 3'端脱磷酸化和适配器结扎。这种方法在目前的CLIP-Seq过程中和最近描述的RIPiT20变体中成功实现。这些变化还将从图书馆制备程序的早期阶段去除几个耗时的RNA纯化步骤。此外,与CLIP不同,CLIP在RNA上提供RBP交联位点的核苷酸水平分辨率,而RIPiT足迹的分辨率将保持在几十个核苷酸的水平。最后,由于RNP可能包括多种限制性商业惯例,RIPiT富集RNA位点包括许多限制性商业惯例结合位点的混合。由于由个别限制性商业惯例所约束的共识序列正以迅速的速度被发现,现在随时可用21、22、23,这些信息可以利用这些信息来减少RBP站点的分类在 RIPiT 输出中丰富。尽管存在这些挑战,RIPiT-Seq 是捕获动态、异构甚至瞬态 RNP 复合物的 RNA 足迹的有效程序,它可以为控制细胞的 RNA 机的内部工作提供独特的见解功能。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了NIH赠款GM120209(GS)的支持。作者感谢OSUCCC基因组共享资源核心的服务(CCC支持赠款NCI P30 CA16058)。
材料
| Name | Company | Catalog Number | Comments |
| Anti-FLAG Affinity Gel | Sigma | A2220 | |
| ATP, [γ-32P]- 3,000 Ci/mmol 10 mCi/mL EasyTide, 250 µCi | PerkinElmer | BLU502A250UC | |
| BD Disposable Syringes with Luer-Lok Tips (200) | Fisher | 14-823-435 | |
| Betaine 5M | Sigma | B0300 | |
| biotin-dATP | TriLink | N-5002 | |
| biotin-dCTP | Perkin Elmer | NEL540001EA | |
| Branson Sonifier, Model SSE-1 | Branson | ||
| CircLigase I | VWR | 76081-606 | ssDNA ligase I |
| DMEM, High Glucose | ThermoFisher | 11995-065 | |
| DNA load buffer NEB | NEB | ||
| Dynabeads Protein A | LifeTech | 10002D | |
| Flp-In-T-REx 293 Cell Line | ThermoFisher | R78007 | |
| GeneRuler Low Range DNA Ladder | ThermoScientific | FERSM1203 | |
| Hygromycin B | ThermoFisher | 10687010 | |
| Mini-PROTEAN TBE Gel 10 well | Bio-Rad | 4565013 | |
| Mini-PROTEAN TBE-Urea Gel | Bio-Rad | 4566033 | |
| miRCAT-33 adapter 5′-TGGAATTCTCGGGTGCCAAGGddC-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Mirus transIT-X2 transfection reagent | Mirus | MIR 6004 | |
| Mth RNA ligase | NEB | E2610S | |
| PE1.0 5′-AATGATACGGCGACCACCGAGATCTACACT CTTTCCCTACACGACGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| PE2.0 5′-CAAGCAGAAGACGGCATACGAGATCGGTCTC GGCATTCCTGCTGAACCGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Phenol/Chloroform/Isoamyl Alcohol (25:24:1, pH 6.7, 100 mLl) | Fisher | BP1752I-100 | |
| Purple Gel Loading Dye (6x) | NEB | NEB #7025 | |
| Q5 DNA Polymerase | NEB | M0491S/L | |
| RNase I, E. coli, 1,000 U | Eppicenter | N6901K | |
| SPIN-X column | Corning | CLS8160-24EA | |
| Streptavidin beads | ThermoFisher | 60210 | |
| Superscript III (SSIII) | ThermoScientific | 18080044 | reverse transcriptase enzyme |
| SybrGold | ThermoFisher | S11494 | gold nucleic acid gel stain |
| T4 Polynucleotide Kinase-2500U | NEB | M0201L | |
| T4RNL2 Tr. K227Q | NEB | M0351S | |
| Tetracycline | Sigma | 87128 | |
| Thermostable 5´ App DNA/RNA Ligase | NEB | M0319S | |
| TruSeq_SE1 5′-pGGCACTANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE10 5′-pGGTGTTCNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE11 5′-pGGTAAGTNNNNNAGATCGGAA GAGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE12 5′-pGGAGATGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE2 5′-pGGGTAGCNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE35′-pGGTCGATNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE4 5′-pGGCCTCGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE5 5′-pGGTGACANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE6 5′-pGGTAGACNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE7 5′-pGGGCCCTNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCT TCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE8 5′-pGGATCGGNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTT CCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE9 5′-pGGACTGANNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Typhoon 5 Bimolecular Imager | GE Healthcare Life Science | 29187191 |
参考文献
- Karousis, E. D., Nasif, S., Mühlemann, O. Nonsense-mediated mRNA decay: novel mechanistic insights and biological impact. Wiley Interdisciplinary Reviews: RNA. 7 (5), 661-682 (2016).
- Ivanov, P. V., Gehring, N. H., Kunz, J. B., Hentze, M. W., Kulozik, A. E. Interactions between UPF1, eRFs, PABP and the exon junction complex suggest an integrated model for mammalian NMD pathways. The EMBO Journal. 27 (5), 736-747 (2008).
- Papasaikas, P., Valcárcel, J. The Spliceosome: The Ultimate RNA Chaperone and Sculptor. Trends in Biochemical Sciences. 41 (1), 33-45 (2016).
- Jensen, K. B., Darnell, R. B. CLIP: Crosslinking and ImmunoPrecipitation of In Vivo RNA Targets of RNA-Binding Proteins. Methods in Molecular Biology. 488, 85-98 (2008).
- Garzia, A., Morozov, P., Sajek, M., Meyer, C., Tuschl, T. PAR-CLIP for Discovering Target Sites of RNA-Binding Proteins. mRNA Decay: Methods and Protocols. 1720, 55-75 (2018).
- Konig, J., et al. iCLIP - Transcriptome-wide Mapping of Protein-RNA Interactions with Individual Nucleotide Resolution. Journal of Visualized Experiments. (50), e2638 (2011).
- Sibley, C. R. Individual Nucleotide Resolution UV Cross-Linking and Immunoprecipitation (iCLIP) to Determine Protein-RNA Interactions. RNA Detection: Methods and Protocols. 1649, 427-454 (2018).
- Wheeler, E. C., Van Nostrand, E. L., Yeo, G. W. Advances and challenges in the detection of transcriptome‐wide protein-RNA interactions. Wiley Interdisciplinary Reviews: RNA. 9 (1), (2018).
- Singh, G., et al. The Cellular EJC Interactome Reveals Higher-Order mRNP Structure and an EJC-SR Protein Nexus. Cell. 151, 750-764 (2012).
- Singh, G., Ricci, E. P., Moore, M. J. RIPiT-Seq: A high-throughput approach for footprinting RNA:protein complexes. Methods. 65, 320-332 (2014).
- Mabin, J. W., et al. The Exon Junction Complex Undergoes a Compositional Switch that Alters mRNP Structure and Nonsense-Mediated mRNA Decay Activity. Cell Reports. 25 (9), 2431-2446 (2018).
- Gehring, N. H., Lamprinaki, S., Kulozik, A. E., Hentze, M. W. Disassembly of Exon Junction Complexes by PYM. Cell. 137 (3), 536-548 (2009).
- Dostie, J., Dreyfuss, G. Translation Is Required to Remove Y14 from mRNAs in the Cytoplasm. Current Biology. 12 (13), 1060-1067 (2002).
- Zünd, D., Gruber, A. R., Zavolan, M., Mühlemann, O. Translation-dependent displacement of UPF1 from coding sequences causes its enrichment in 3' UTRs. Nature Structural & Molecular Biology. 20 (8), 936-943 (2013).
- Gangras, P., Dayeh, D. M., Mabin, J. W., Nakanishi, K., Singh, G. Cloning and Identification of Recombinant Argonaute-Bound Small RNAs Using Next-Generation Sequencing. Argonaute Proteins: Methods and Protocols. 1680, 1-28 (2018).
- Heyer, E. E., Ozadam, H., Ricci, E. P., Cenik, C., Moore, M. J. An optimized kit-free method for making strand-specific deep sequencing libraries from RNA fragments. Nucleic Acids Research. 43 (1), 2 (2015).
- Cameron, V., Uhlenbeck, O. C. 3'-Phosphatase activity in T4 polynucleotide kinase. Biochemistry. 16 (23), 5120-5126 (1977).
- Ricci, E. P., et al. Staufen1 senses overall transcript secondary structure to regulate translation. Nature Structural & Molecular Biology. 21 (1), 26-35 (2014).
- Lackner, D. H., et al. A generic strategy for CRISPR-Cas9-mediated gene tagging. Nature Communications. 6, 10237 (2015).
- Metkar, M., et al. Higher-Order Organization Principles of Pre-translational mRNPs. Molecular Cell. 72 (4), 715-726 (2018).
- Giudice, G., Sánchez-Cabo, F., Torroja, C., Lara-Pezzi, E. ATtRACT-a database of RNA-binding proteins and associated motifs. Database: The Journal of Biological Databases and Curation. 2016, (2016).
- Paz, I., Kosti, I., Ares, M., Cline, M., Mandel-Gutfreund, Y. RBPmap: a web server for mapping binding sites of RNA-binding proteins. Nucleic Acids Research. 42, 361-367 (2014).
- Sundararaman, B., et al. Resources for the Comprehensive Discovery of Functional RNA Elements. Molecular Cell. 61 (6), 903-913 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。