Method Article
Identification des empreintes de pas de l'ARN: Complexes protéiques par l'intermédiaire de l'immunoprécipitation d'ARN dans Tandem suivi du séquençage (RIPiT-Seq)
Dans cet article
Résumé
Ici, nous présentons un protocole pour enrichir les sites endogènes de liaison d'ARN ou « empreintes » des complexes d'ARN:protéines (RNP) des cellules mammifères. Cette approche implique deux immunoprécipitations des sous-unités de RNP et est donc surnommée immunoprécipitation d'ARN en tandem (RIPiT).
Résumé
L'immunoprécipitation d'ARN en tandem (RIPiT) est une méthode pour enrichir des empreintes d'ARN d'une paire de protéines dans un complexe d'ARN:protéine (RNP). RIPiT emploie deux étapes de purification. Tout d'abord, l'immunoprécipitation d'une sous-unité marquée de RNP est suivie de la digestion douce de RNase et de l'élution d'affinité non dénaturante suivante. Une deuxième immunoprécipitation d'une autre sous-unité RNP permet l'enrichissement d'un complexe défini. À la suite d'une suppression dénaturante des ARN et des protéines, les empreintes d'ARN sont converties en bibliothèques de séquençage de l'ADN à haut débit. Contrairement à l'ultraviolet plus populaire (UV) crosslinking suivie par l'immunoprécipitation (CLIP) approche pour enrichir les sites de liaison RBP, RIPiT est UV-crosslinking indépendant. Par conséquent RIPiT peut être appliqué à de nombreuses protéines présentes dans l'interagiome ARN et au-delà qui sont essentiels à la régulation de l'ARN, mais ne pas entrer directement en contact avec l'ARN ou UV-crosslink mal à l'ARN. Les deux étapes de purification du RIPiT offrent un avantage supplémentaire d'identifier les sites de liaison où une protéine d'intérêt agit en partenariat avec un autre cofactor. La stratégie de double purification sert également à améliorer le signal en limitant le fond. Ici, nous fournissons une procédure progressive pour effectuer RIPiT et pour générer des bibliothèques de séquençage à haut débit à partir d'empreintes d'ARN isolées. Nous exposons également les avantages et les applications du RIPiT et discutons de certaines de ses limites.
Introduction
Dans les cellules, l'ARN existe dans le complexe avec des protéines pour former des complexes ARN:protéines (RNP). Les RRP sont assemblés autour de protéines liant l'ARN (RBPs, celles qui lient directement l'ARN) mais comprennent également des non-RRP (ceux qui lient les RRP mais pas l'ARN), et sont souvent de nature dynamique. Les RRP et leurs cofacteurs fonctionnent collectivement au sein des RNi pour exécuter des fonctions réglementaires. Par exemple, dans la voie de désintégration de l'ARNm médiée par l'absurdité (NMD), les protéines UPF (UPF1, UPF2 et UPF3b) reconnaissent le ribosome prématurément terminé. Chacune des protéines de l'UPF peut se lier à l'ARN, mais ce n'est que lorsqu'elles s'assemblent ensemble qu'un complexe actif de NMD commence à se former. Dans ce complexe, UPF1 est encore activé par la phosphorylation par un SMG1 non-RBP, et une telle activation UPF1 conduit finalement au recrutement de la désintégration de l'ARNm induisant des facteurs1,2. Dans cet exemple, les RRP exigent des cofacteurs non-RBP pour le recrutement et l'activation du complexe RNP qui déclenche la MNM. Une autre propriété des RNP est leur hétérogénéité compositionnelle. Considérez le spliceosome, qui existe dans les complexes E, A, B ou C distincts. Différents complexes épiscédsome ont des protéines qui se chevauchent et des protéines distinctes3. Pour étudier les fonctions RNP, il est important d'élucider quels ARN sont liés par un RBP et ses protéines associées. De nombreuses méthodes existent pour y parvenir, chaque approche ayant ses avantages et inconvénients distincts4,5,6,7.
Les méthodes très populaires pour identifier les sites de liaison RBP — liaisons croisées suivies par l'immunoprécipitation (CLIP) et ses diverses variations - s'appuient sur la lumière ultraviolette (UV) pour relier un RBP à l'ARN8. Cependant, ce n'est pas une approche efficace pour les non-RPP dans les RNP, qui ne contactent pas l'ARN directement. Ici, nous décrivons une approche alternative qui s'applique aux RRP et aux non-RPR, pour isoler et identifier leurs sites de liaison d'ARN. Cette approche appelée immunoprécipitation d'ARN en tandem (RIPiT) se compose de deux étapes d'immunoprécipitation, qui aident à atteindre une spécificité plus élevée par rapport à une seule purification (Figure 1)9,10. Comme les étapes individuelles d'immunoprécipitation (IP) peuvent être effectuées à une chaîne inférieure par rapport à CLIP, RIPiT ne dépend pas de la disponibilité d'anticorps qui peuvent résister à la présence de détergents forts pendant l'immunoprécipitation. L'avantage le plus unique de RIPiT est la capacité de cibler deux protéines différentes en deux étapes de purification; ceci fournit un moyen puissant d'enrichir un complexe RNP compositionnellement distinct d'autres complexes similaires11.
De petites variations à la procédure RIPiT peuvent encore améliorer l'enrichissement RNP. Par exemple, certaines interactions ARN-protéines ou protéines-protéines au sein des RNP sont transitoires et il peut être difficile de purifier efficacement les empreintes de ces complexes. Pour stabiliser de telles interactions, les RNR peuvent être reliés entre eux à l'intérieur des cellules avec du formaldéhyde avant la lyse cellulaire et le RIPiT. Par exemple, nous avons observé qu'une faible interaction entre le facteur de base du complexe de jonction d'exon (EJC), EIF4AIII et le facteur de désassemblage de l'EJC, PYM12 peut être stabilisée avec un traitement au formaldéhyde de telle sorte que plus d'empreintes d'ARN sont enrichies (données non montré). Avant la récolte cellulaire et le RIPiT, les cellules peuvent également être traitées avec des médicaments pour stabiliser ou enrichir les RNi dans un état particulier. Par exemple, lors de l'étude des protéines qui sont retirées de l'ARNm pendant la traduction (p. ex., l'EJC13, UPF114), le traitement avec des inhibiteurs de traduction tels que la puromycine, le cycloheximide ou la harringtonine peut entraîner une augmentation de l'occupation de protéines sur les ARN.
La quantité d'ARN récupérée dans le RIPiT est généralement faible (0,5-10 pmoles, c'est-à-d.- 10-250 ng ARN compte tenu d'une longueur moyenne d'ARN de 75 nt). La principale raison en est que seule une petite fraction d'une protéine donnée est présente en complexe avec d'autres protéines dans les RNP (toute protéine ip'ed "libre" dans la première étape est perdue au cours de la deuxième PI). Pour générer des bibliothèques RNA-Seq à partir de cet ARN, nous avons également décrit ici une adaptation du protocole précédemment publié adapté à ces faibles entrées d'ARN15,16 (Figure 2), qui donne des échantillons prêts à sequencing à haut débit dans 3 Jours.
Protocole
1. Établissement de lignées cellulaires STABLES HEK293 exprimant des protéines d'intérêt (POI) inducibles par la tétracycline
- Cellules HEK293 de semences avec un site de recombinaison Flp (FRT) stablement intégré à une densité de 10 x 104 cellules/mL dans le milieu de croissance (le milieu d'aigle modifié de Dulbecco [DMEM] - sérum bovin fœtal de 10 % [FBS] - 1 % pénicilline-streptomycine [penn/strep]) en 6- plaques de puits. Permettre aux cellules de se développer pendant la nuit dans un incubateur humidifié à 37 oC et 5 % de CO2 (conditions de croissance standard pour toutes les étapes ultérieures).
- Le lendemain, les cellules devraient être de 70 % de confluents. Selon le protocole de réactif de transfection, transfect les cellules DE HEK293 contenant le site de FRT avec un rapport 9:1 de pcDNA5-TETO-FLAG-POI:pOG44.
REMARQUE: L'étiquette FLAG a le motif de séquence DYKDDDDK (D - acide aspartique, Y - tyrosine, et K - lysine). - Après 24 h, commencer la sélection d'antibiotiques. Enlever les milieux et ajouter un milieu de croissance frais complété par une hygromycine de 100 g/mL. Dans les 72 h, les cellules non transfectes devraient commencer à mourir.
- Tous les 48-72 h, changer les médias de croissance et compléter avec de l'hygromycine fraîche.
- Après 2 semaines de sélection, des colonies discrètes de cellules transfectées stables commenceront à apparaître. Une fois que les colonies sont visibles à l'œil nu, ajouter 1 ml de trypsine à l'assiette et incuber pendant 5 min à 37 oC. Resuspendre les cellules dans DMEM, transférer les cellules dans une nouvelle plaque de 10 cm, et ajuster le volume à 10 ml de milieu de croissance frais complété par 100 g/mL d'hygromycine. Permettre à la plaque d'atteindre 80 % de confluence pour élargir davantage les cellules afin de préparer des stocks permanents.
- Déterminer la quantité de tétracycline (Tet) nécessaire pour obtenir le niveau optimal d'expression FLAG-POI pour l'expérience. Cultivez des cellules en format 12 puits et effectuez une titration de Tet entre 0-1 000 ng/mL de portée pour 16-24 h. Effectuer des taches occidentales sur les échantillons de titration avec anticorps contre le POI.
REMARQUE: Pour les protéines jusqu'à 60 kDa, l'électrophorèse de gel FLAG et la copie endogène de la protéine peuvent être résolues sur l'électrophorede de gel de sulfate-polyacrylamide de dodécyl de sodium (SDS-PAGE) pour comparer les niveaux d'expression de FLAG-POI à son homologue endogène. Pour les protéines plus grosses, l'intensité du signal dans les échantillons induits par Tet peut être comparée à l'échantillon non induit. La solution de stock de tétracycline peut être préparée à 1 mg/mL en éthanol 100%. Les dilutions du stock pour le travail de culture cellulaire devraient être dans l'eau stérile ou saline tamponnée de phosphate (PBS). Le stock de tétracycline doit être préparé frais une fois par mois.
2. Cellules de culture pour l'induction de tétracycline et la procédure de RIPiT
- Graine intégrée de façon stable FLAG-POI HEK293 à une densité de 3 x 105 cellules/mL dans le milieu de croissance dans des plaques de 15 cm. Permettre aux cellules de se développer à37 oC et 5 % de CO 2.
REMARQUE: En général, trois à cinq plaques de 15 cm donneront environ 2 à 20 pmol d'empreintes d'ARN selon l'abondance du RNP d'intérêt. Si les rNRp seront reliés entre le formaldéhyde et purifiés dans des conditions strictes, alors deux fois plus d'entrée peut être nécessaire. - Ajouter de la tétracycline à une concentration optimale prédéterminée dans le média pour induire l'expression du FLAG-POI (voir l'étape 1.6) 16-24 h avant que les cellules ne soient récoltées.
REMARQUE: Il n'est pas nécessaire de changer le milieu de croissance. Les cellules doivent être prêtes à récolter environ 72 h après l'ensemencement ou lorsque les plaques sont des confluents de 80 %. Il est important d'éviter que les cellules ne deviennent confluentes.
3. Récolte cellulaire, traitement du formaldéhyde et lyse cellulaire
- Laver délicatement les cellules monocouches avec 15 ml de PBS réfrigéré pour chaque plaque de 15 cm. Ensuite, gratter les cellules dans 30 ml de PBS. Si les cellules ont été traitées avec des médicaments avant la récolte, compléter PBS avec le médicament. Recueillir toutes les cellules dans un tube conique de 50 ml.
REMARQUE: Pour préserver les interactions faibles, les cellules peuvent être reliées à un formaldéhyde : ajouter du formaldéhyde à la suspension cellulaire de l'étape 3.1 à une concentration finale de 0,1 % et incuber sur un rocker à température ambiante pendant 10 min. Ajouter 3 ml du tampon d'étanchéité (tableau1 ) et la roche pendant 5 minutes supplémentaires. - Pelleter les cellules à 400 x g pendant 10 min à 4 oC et jeter le supernatant.
- Lyse les cellules dans 4 ml de tampon de lyse hypotonique glacée (HLB; Tableau 1). Utilisez une pipette P1000 pour resuspendre les cellules. Transférer dans un tube de 5 ml et incuber le lysate sur la glace pendant 10 min.
- Placer le lysate dans un bain de glace et sonicate à 10% d'amplitude pour 30 s avec des impulsions de 1 s avec 2 pauses. Ensuite, ajuster la concentration de sel à 150 mm en ajoutant 108 'L de 5 M NaCl.
REMARQUE: Les échantillons de formaldéhyde croisés peuvent être soumis à une lyse et à une purification plus strictes en incluant 0,1 % chacun des SDS et du désoxycholate de sodium dans le tampon de lyse. - Effacer le lysate par centrifugation à 21 000 x g pendant 10 min à 4 oC. Recueillir 20 Supernatantls (extrait de cellules) dans un tube étiqueté pour l'analyse de la tache occidentale des niveaux de protéines dans l'entrée (voir Figure 3A).
REMARQUE: Pendant que le lysate est en centrifugeuse, les perles d'agarose DE FLAG peuvent être lavées (voir l'étape 4.1). Les perles d'agarose DE FLAG doivent être prélavées 3x dans 4 ml de tampon de lavage isotonique glacé (IsoWB, tableau 1).
4. Immunoprécipitation DE FLAG
- Appliquer le reste des supernatants de l'étape 3,5 à 750 l de perles d'agarose FLAG prélavées dans un tube de 5 ml (le volume de lit des perles d'agarose FLAG lavées sera de 375 l). Incuber les perles d'agarose FLAG et l'extrait de cellules pendant 1-3 h à 4 oC avec un mélange doux.
REMARQUE: Ce volume de perles d'agarose DE FLAG devrait être suffisant pour un large éventail de protéines, mais peut être optimisé, si nécessaire. - Pellet FLAG perles d'agarose par centrifugation à 400 x g pendant 1 min à 4 oC. Recueillir 20 l l du supernatant dans un tube étiqueté pour l'analyse de la tache occidentale des niveaux de protéines dans l'extrait de cellules appauvries (voir Figure 3A). Jeter le reste du supernatant.
- Pour laver les perles d'agarose FLAG, ajouter 4 ml d'IsoWB et le suspendre à nouveau. Pellet perles à 400 x g pendant 1 min à 4 oC. Retirez soigneusement le supernatant. Répéter 4x.
REMARQUE: Pour les lavages rigoureux des ADRESSEs IP croisées au formaldéhyde, 0,1 % de sDS et de désoxycholate de sodium peuvent être inclus dans le tampon de lavage pour les deux premières étapes de lavage.
5. RNase I Digestion
- Diluer RNase I à 0,002-0,01 unités/mL dans 750 'L d'IsoWB (la concentration appropriée pour les tailles d'empreintes désirées doit être déterminée empiriquement). Ajouter IsoWB-RNase I aux perles lavées FLAG agarose et incuber avec un mélange doux à 4 oC pendant 10 min.
- Pellet perles à 400 x g pendant 1 min à 4 oC. Recueillir 20 Supernatant L (RNase I elution) dans un tube étiqueté pour l'analyse de tache occidentale. Jetez IsoWB-RNase I et lavez les perles FLAG agarose 4x avec IsoWB comme décrit à l'étape 4.3.
6. Élution d'affinité
- Préparer un stock de tampon d'élution (PEPtide FLAG à 250 ng/mL à IsoWB). Appliquer 375 ll de tampon d'élution sur les perles d'agarose FLAG et secouer doucement à 4 oC pendant 1 à 2 h. Pellet FLAG perles d'agarose et recueillir un aliquot de 15 l de l'élution pour la tache occidentale de protéines dans FLAG IP (voir Figure 3A).
REMARQUE: Lorsque l'élution d'affinité est en cours, la section 7 peut être effectuée.
7. Conjugaison magnétique de perle-anticorps
- Laver 50 perles magnétiques L (c.-à-d. Dynabeads; Tableau des matériaux) 3x en 1 ml IsoWB en tube de 1,5 ml. Resuspendre les perles magnétiques dans 100 l de tampon de conjugaison. Ajouter une quantité appropriée d'anticorps (la quantité exacte d'anticorps à utiliser pour la propriété intellectuelle devra être déterminée empiriquement pour chaque anticorps).
REMARQUE: Les perles magnétiques protéiques A sont optimales pour les anticorps produits chez le lapin tandis que les perles magnétiques protéiques G sont plus appropriées pour les anticorps de souris. Le tableau de compatibilité des perles magnétiques est disponible sur le site Web du fournisseur pour choisir des perles appropriées pour chaque anticorps. - Laver les perles magnétiques 2x dans le tampon de conjugaison (tableau 1). Incuber le mélange à température ambiante pendant au moins 10 min. Resuspendre les perles magnétiques dans 375 'L de tampon de dilution RIPiT. Conserver sur la glace jusqu'à la prochaine étape.
8. Deuxième immunoprécipitation
- Appliquer l'élution d'affinité FLAG restante de l'étape 6.1 aux perles magnétiques couplées aux anticorps contre la protéine d'intérêt. Incuber avec un mélange doux à 4 oC pendant 1-2 h. Capturer les perles magnétiques sur un aimant et recueillir 15 l de supernatant pour l'analyse des protéines non liées via western blot. Laver les perles magnétiques 7x avec 1 ml d'IsoWB.
9. Dénaturation de l'élution
- Ajouter 100 l de tampon d'échantillon clair (tableau 1) aux perles magnétiques et le suspendre à l'intérieur avec une pipette P200. Incuber sur la glace pendant 10 min. Faites glisser doucement pour resuspendre les perles périodiquement.
- Capturer les perles magnétiques sur un aimant et recueillir 15 l d'élution pour l'analyse des protéines dans l'élution RIPiT via la tache occidentale (voir la figure 3A). Transférer l'élution restante dans un nouveau tube étiqueté de 1,5 ml.
REMARQUE: Si les échantillons étaient en liaison croisée de formaldéhyde, les échantillons doivent être incubés à 65 oC pendant 1 h pour inverser les liaisons croisées. - Effectuer des taches occidentales sur des échantillons prélevés à différentes étapes (entrée, appauvrissement de la propriété IP DU FLAG, IP FLAG, deuxième épuisement de la propriété intellectuelle, deuxième suppression de la propriété intellectuelle). Blot avec des anticorps contre les deux protéines d'appât, leurs autres interactors si connu, et au moins un RBP non-interaction comme un contrôle négatif (Figure 3A).
10. Extraction d'ARN et durcissement final
- À l'élution RIPiT, ajouter 320 oL de ddH2O sans RNase, 400 oL d'alcool isoamyl au phénol-chloroforme (PCIAA, pH 4,5), et vortex pour 30 s et tourner à température ambiante à 12 000 x g pendant 5 min. Recueillir 350 oL de phases aqueous dans un tube séparé. Ajouter 35 oL d'acétate de sodium de 3 M, 1 l de 1 M MgCl2, 10 g de glycogène et 1 ml d'éthanol à 100 %. Incuber toute la nuit à -20 oC.
- Pour granuler l'ARN, centrifugeuse à 12 000 x g pendant 30 min à 4 oC. Laver l'ARN à 70 % d'éthanol.
- Pour enlever le phosphate de 3' laissé sur l'ARN après le clivage RNase I, resuspendre le granule d'ARN dans 17 'L de ddH2O sans RNase, et ajouter 2 'L de 10x T4 polynucléotide kinase (PNK) tampon (Tableau des matériaux) et 1 'L de T4 PNK. Incuber à 37 oC pendant 30 min.
REMARQUE: L'activité 3'phosphatase de T4 PNK a une activité optimale au pH 617. Le tampon de réaction PNK est optimisé pour l'activité de 5' kinase de T4 PNK et a un pH de 7,6. Bien que l'ajustement du pH de la réaction de guérison de fin ait été tenté, cette étape peut être encore optimisée. - Ajouter 380 oL de ddH2O sans RNase et 400 oL de PCIAA pH 4,5 au tube. Vortex pour 30 s, centrifugeuse à 12 000 x g pendant 5 min. Recueillir la phase aqueuse et ajouter 35 l d'acétate de sodium de 3 M, 1 L de 1 M MgCl2, 10 g de glycogène et 1 ml d'éthanol à 100 %.
- Incuber toute la nuit à -20 oC. Pelleter et laver l'ARN avec 70% d'éthanol comme ci-dessus. Resuspendre l'ARN dans 4,5 l d'eau sans RNase.
11. Estimation de la taille et de l'abondance de l'empreinte de l'ARN
-
On s'attend à ce qu'un RIPiT réussi donne 1 pmol ou plus de fragments d'ARN. Pour quantifier le rendement réel, transférer 0,7 L d'ARN RIPiT (1/6 du rendement total) dans un nouveau tube. Ajouter 2 'L de 10x T4 PNK buffer, 1 'L de 1 mM ATP, 40 'Ci'32P-ATP (0.5 '1.0 'L du stock), et 1 'L de T4 PNK. Ajuster le volume à 10 l et incuber à 37 oC pendant 30 min.
- En parallèle, les réactions PNK, étiqueter une échelle d'ADN à faible portée et 0,1 pmol d'un ARN synthétique ou oligo d'ADN (20-40 nt) pour utiliser une taille et des normes de quantité.
- Résoudre l'ARN/ADN étiqueté sur le gel 26% urée-PAGE (20 x 27 x 0,45 mm3). Le gel doit être pré-exécuté pendant 30 min à 35 W. Flush puits avant la pré-course et avant le chargement des échantillons et courir à 35 W jusqu'à ce que le bromophénol avant de teinture bleue a presque atteint la fin du gel.
- Retirez soigneusement le gel des plaques de verre sur un morceau de papier filtre de 8 x 11 pouces. Avec du gel sur le papier, placer dans un appareil de séchage de gel et couvrir d'un morceau de pellicule plastique. Gel sec à 80 oC pendant 1 h sous vide.
- Exposez le gel séché à un phosphoscreen pendant la nuit ou jusqu'à ce qu'un signal adéquat soit détecté. Phosphoscreen image. L'ARN de bonne qualité d'un RIPiT devrait apparaître comme un frottis dans la voie, avec des bandes proéminentes minimales (Figure 3B). Pour quantifier l'ARN, comparez l'intensité du signal des fragments d'ARN de taille désirée dans la voie RIPiT au signal de 0,1 pmol d'oligo synthétique étiqueté.
REMARQUE: Alternativement, la taille et les quantités d'empreinte d'ARN peuvent être vérifiées à l'aide d'un bioanalyseur à haute sensibilité (figure 3C).
12. Ligation adaptateur
- Préparer l'ARN RIPiT de telle sorte qu'au moins 3 pmol d'ARN est dissous dans 3,8 L d'eau.
- Dans un tube de réaction en chaîne de polymérase de 0,2 ml (PCR), combinez 3,8 L d'ARN, 1 L d'adaptateur pré-adenylated miR-CAT-33 (7 M) (Tableaudes matériaux). Incuber le mélange sur un cycleur thermique à 65 oC pendant 10 min, 16 oC pendant 5 min, puis le conserver à 4 oC.
REMARQUE: Le linker pré-adenylated peut être commandé du service de synthèse d'oligo, ou un oligo d'ADN non adenylated personnalisé de n'importe quel service de synthèse d'oligo peut être adenylated utilisant la ligase d'ARN de Mth (tableau des matériaux) et le gel purifié. - Dans le même tube, ajoutez 1,5 L de tampon de ligase de l'ARN T4, 7,5 'L de 50% de polyéthylène glycol 8000 (PEG-8000), 0,75 'L de 20 mM dithiothreitol (DTT), et 0,45 'L de T4 RNL2 Tr. K227Q (Table des Matériaux).
REMARQUE: 50% PEG-8000 est livré avec l'ARN ligase et tampon acheté. Les solutions PEG-8000 sont visqueuses et doivent être pipetted lentement. - Incuber la réaction dans le cycleur thermique à 30 oC pendant 6 h, inactiver la ligase à 65 oC pendant 10 min, puis maintenir à 4 oC.
13. Transcription inversée
- Dans le tube avec le mélange de ligature de l'étape 12.4, ajouter 11,25 l de 4x deoxynucléotide triphosphate (dNTP) mélange, qui contient un mélange de dNTP réguliers et biotinylated (voir tableau 1), 1,0 L de 10 XRt premiers (Tableau des matériaux), et 6,8 L de Eau sans RNase. Incuber à 65 oC pendant 5 min, puis tenir à 4 oC.
- Transférer les tubes sur glace et ajouter 9,0 l de tampon de 5x premier brin (FS) sans MgCl2 (tableau 1), 2,25 l de 100 mM DTT, 1,2 l d'enzyme de transcriptase inversée à un volume final de 45 l (tableaudes matériaux).
- Incuber dans un cycleur thermique à 55 oC pendant 30 à 60 min. Chauffer inactiver la transcriptase inverse à 70 oC pendant 15 min et maintenir l'échantillon à 4 oC.
14. Purification du produit RT
- Ajouter 45 l de tampon de charge d'urée 2x (tableau 1) à la réaction RT. Diluer 1 g d'une échelle d'ADN à faible portée dans 45 L et ajouter 45 L de tampon de charge d'urée de 2x.
- Préparer 10 % de gel urée-PAGE (20 x28 x 0,15 cm 3; Tableau 1) avec peigne à 8 puits. À l'aide d'une seringue ou d'une tuyauterie, rincer les puits avec un tampon Tris/borate/EDTA (TBE) de 0,5x.
REMARQUE: Les gels faits maison ci-dessus offrent une meilleure résolution en séparant le produit RT étendu de l'amorce RT non prolongée. Comme alternative, les gels d'urée-PAGE pré-moulés peuvent également être utilisés (Tableau des Matériaux). Comme les gels prémoulés permettent de réduire les volumes maximaux par puits, les échantillons devront donc être divisés en plusieurs puits. Les gels pré-moulés doivent être exécutés à 150 à 200 V. - Pré-exécuter le gel à 35 W pendant 30 min. Rincer les puits à nouveau, charger des échantillons et exécuter le gel à 35 W jusqu'à ce que le front de teinture bleu bromophénol ait migré à environ 1 pouce de l'extrémité du gel.
REMARQUE: Utilisez un évier de chaleur métallique pendant la pré-course et la dernière course pour éviter la surchauffe du gel. - Tachez le gel pendant 5 min dans la solution de tache de gel nucléique d'or 1x préparée dans 0.5x TBE. Ce colorant est sensible à la lumière, alors évitez l'exposition à la lumière.
- Gel d'image sur scanner fluorescent à des fins de documentation à l'aide de 520 nm d'excitation et 580 nm filtres d'émission. Si un scanner fluorescent n'est pas disponible, utilisez un transilluminateur de lumière bleue. Le produit RT doit apparaître comme un frottis commençant au-dessus de l'amorce RT non prolongée (figure 4).
REMARQUE: Bien que le colorant de coloration de gel d'acide nucléique d'or soit facilement visualisé sur un doc de gel avec la source UV-lumière, il est essentiel de ne pas exposer le produit valable de RT aux UV pour empêcher des dommages. - Visualisez le gel sur un transilluminateur de lumière bleue et excisez le produit RT du gel. Il est recommandé de couper l'ADN avec des extensions allant de 30-200 nt (Figure 4). Placer les morceaux de gel excisés sur une surface propre et hacher la tranche en petits morceaux pour augmenter la surface. Transférer soigneusement dans un tube de 1,5 ml et ajouter 800 l de tampon d'élution d'ADN (tableau 1).
- Incuber les morceaux de gel avec un mélange doux avec tampon d'élution d'ADN pendant la nuit à température ambiante.
- Séparez le tampon d'élution du gel en passant la boue à travers une colonne de filtre d'acétate de cellulose (Table of Materials) placée dans un tube de collecte de 2 ml.
- Dans un tube de 1,5 ml, lavez 10 l l de perles magnétiques streptavidin es avec 500 ll de tampon de lavage de perles de streptavidin (tableau 1). Répétez l'opération pour trois lavages totaux. Ne laissez pas les perles sécher. Resuspendre les perles dans 10 l de tampon d'élution d'ADN (tableau 1).
- Transférer le tampon d'élution séparé des morceaux de gel à l'étape 14.8 dans le tube contenant les perles magnétiques lavées de streptavidin.
- Incuber avec un mélange doux pendant au moins 8 h à température ambiante. Capturez les perles sur un aimant, retirez les supernatants et suspendez les perles magnétiques dans 10 L d'eau exempte de RNase et transférez-les dans un tube PCR de 0,2 mL.
15. Circularisation du produit RT
- Les produits RT capturés sur les perles de streptavidin sont circulaires lorsqu'ils sont liés sur des perles. À la boue de perles magnétiques, ajouter 2,0 L de tampon de réaction de circularisation 10x, 1,0 l de 1 mM ATP, 1,0 L de 50 mM MnCl2, 4,0 l de 5 M de bétaïne, 1,0 l de ssDNA ligase I (Tableau des matériaux), et 1,0 l d'eau sans RNase.
- Incuber la réaction de circularisation sur un cycleur thermique à 60 oC pendant 4 h. Chauffer inactiver la ligase I de l'ADNsain en chauffant à 80 oC pendant 10 min, puis maintenez à 4 oC.
16. Test PCR
- Avant de passer à un PCR à grande échelle, utilisez une partie du produit circulaire pour déterminer le nombre idéal de cycles d'amplification pour chaque échantillon. Cette étape permet de prévenir la suramplification et de limiter les artefacts PCR à mesure que les composants de réaction PCR deviennent limitatifs à des cycles PCR plus élevés.
- Préparer une réaction PCR de 45 L avec 4,0-6,0 l du produit circularisé à partir de l'étape 15.2, 9.0 de l de tampon de réaction 5x, 0.9 'L 10 M dNTPs, 2.25 'L de 1 'M PE1.0 amorce (Tableau des matériaux), 2.25 'L de 10 'M PPE2.0 primer (Table de Matériaux) ) , et 0,045 L de polymérase d'ADN haute fidélité (Tableaudes matériaux)et d'eau.
- Bien mélanger la réaction et les diviser en trois réactions de 15 l. Chacune de ces trois réactions sera soumise à un nombre variable de cycles PCR. Le nombre idéal de cycles devrait se siégeer entre 7 et 14. Effectuez donc des PCR de test pour 8, 11 et 14 cycles.
- Utilisez les conditions DE PCR suivantes : 98 oC - 30 s; 98 oC - 5 s; 65 oC - 10 s; 72 oC - 15 s; 72 oC - 2 min; 12 oC - tenue.
- Ajouter 3 ll de colorant de chargement de gel 6x et résoudre sur 10% de gel PAGE natif jusqu'à ce que l'avant de teinture bleue a migré 3/4 du gel. Tachez le gel à l'aide d'une tache de gel et d'image d'acide nucléique d'or comme dans les étapes 14.4 et 14.5 (Figure 5).
- Pour choisir le nombre idéal de cycles PCR, comparez les produits PCR à partir d'un nombre croissant de cycles. Choisissez le numéro de cycle qui produit la plus grande quantité de produit de la taille prévue sans artefacts de suramplification (p. ex., le frottis d'ADN beaucoup plus grand que prévu, et où aucun épuisement appréciable des amorces PE1.0 et PE2.0 n'est vu (voir flèche rouge dans Figure 5).
17. PCR à grande échelle
- Préparer une réaction PCR de 45 L, comme à l'étape 16.2 et répéter le PCR. Résoudre PCR sur 10% natif PAGE à 150 V, tache avec 1x tache de gel d'acide nucléique d'or et l'image sur un transilluminateur de lumière bleue.
- Accise le produit PCR du gel et transférer dans une seringue de 3 ml. Utilisez la seringue pour écraser le gel et extruder dans un tube de 1,5 ml.
REMARQUE: Le produit RT non étendu sur la circularisation donne PCR produit de 151 bp. Ainsi, à cette étape, on devrait taille sélectionner certains produits qui sont plus grands que 151 bp (Figure 6). - Ajouter 900 l de tampon d'élution d'ADN et incuber à température ambiante pendant la nuit avec un mélange doux.
- Transférer la boue de gel dans une colonne de filtre à acétate de cellulose placée dans un tube de collecte de 2 ml. Faire tourner à 12 000 x g pendant 3 min, en recueillant le supernatant dans un tube frais.
- Ajouter un autre tampon d'élution d'ADN de 400 ll au gel écrasé et transférer dans un tube de 1,5 ml. Incuber avec un mélange doux pendant 4 heures supplémentaires pour une seconde élution.
- Mettre en commun toutes les élutions et les diviser en 3 tubes avec 400 l chacun. Précipitez l'ADN en ajoutant 1 ml d'éthanol à 100 % et 10 g de glycogène. Vortex et incuber au moins 2 h à -20 oC.
- Pellet ADN à 12 000 x g pendant 30 min à 4 oC. Laver les granulés d'ADN avec 70 % d'éthanol.
- Retirez soigneusement tout l'éthanol par pipetting et suspendez rapidement les granules d'ADN dans 20 l d'eau.
REMARQUE: À ce stade, il est important de ne pas laisser la pastille d'ADN se sécher, car le séchage de l'ADN peut le dénaturer. - Utilisez une petite partie de l'échantillon d'ADN pour déterminer la taille et la concentration du produit PCR au moyen d'un fluoromètre et d'un bioanalyseur d'ADN à haute sensibilité. Les échantillons peuvent maintenant être soumis pour le séquençage sur l'une des plates-formes.
- Les lectures séquencées peuvent être traitées (p. ex., suppression de l'adaptateur, rognage pour conserver les séquences de la marque Phred, alignées sur le génome de référence et visualisées sur un navigateur tel que le navigateur du génome de l'UCSC (Figure 7).
Résultats
Un RIPiT réussi se traduira par l'immunoprécipitation des protéines d'intérêt et d'autres protéines en interaction connues, et l'absence de protéines non-interagissant. Comme on le voit à la figure 3A, Magoh et EIF4AIII ont été détectés dans l'élution du RIPiT, mais HNRNPA1 ne l'était pas (voie 6). En parallèle, les empreintes d'ARN qui ont co-purifié avec les complexes RNP ont été détectées par autoradiographie (figure3B) ou bioanalyseur (Figure 3C). On s'attend à ce que le traitement de puromycine augmente l'occupation d'EJC sur l'ARN, et un signal plus fort d'empreinte d'ARN a été observé dans le RIPiT traité de puromycine dans la figure 3B (comparez des voies 2 et 3). La génération d'échantillons pour le séquençage en profondeur nécessite de ligar un adaptateur à l'ARN, puis inverser la transcription de l'ARN en ADN à l'aide d'une amorce qui anneals à la séquence adaptateur. L'étape de transcription inversée intègre des nucléotides biotinylated, pour la purification du produit de transcription inversée. Après la transcription inversée, le produit doit être séparé de l'adaptateur non étendu par urée-PAGE (figure 4). Le produit de transcription inversée est ensuite circularisé et amplifié PCR. Le nombre approprié de cycles PCR ne doit pas trop amplifier le produit circularisé. La suramplification entraînera l'épuisement des amorces et l'aberrant produit PCR (voir la figure 5, voie 4). Le nombre de cycles avec la plus grande amplification sans preuve de suramplification est le plus approprié à utiliser pour un PCR à grande échelle (Figure5 voie 3 et Figure 6).

Figure 1 : Schéma décrivant les principales étapes du RIPiT. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
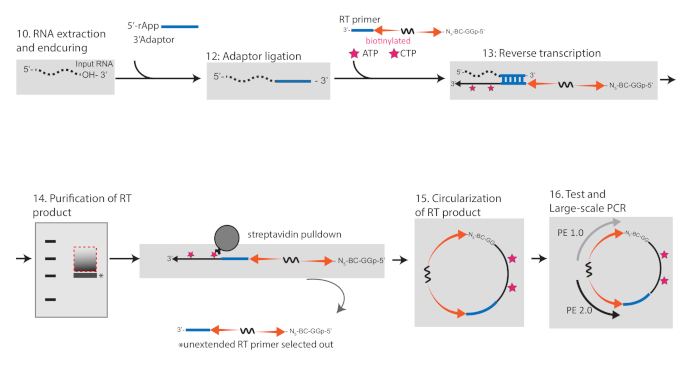
Figure 2 : Schéma représentant le flux de travail pour la conversion de l'ARN RIPiT en bibliothèques pour le séquençage à haut débit. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
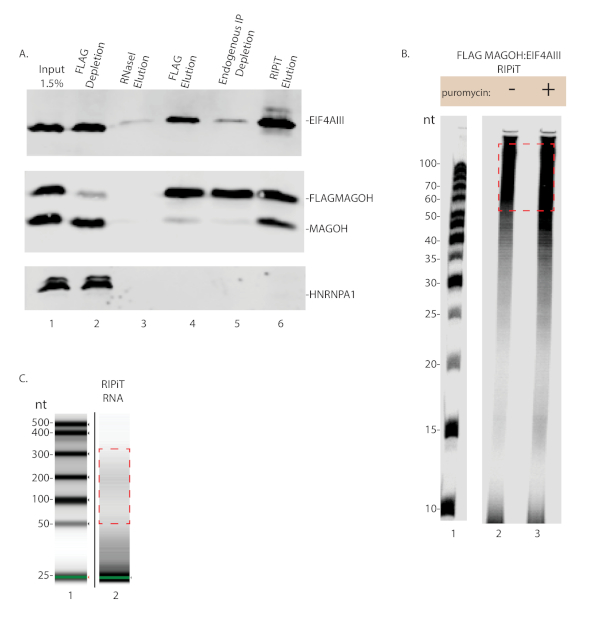
Figure 3 : Estimation des rendements d'ARN et de protéines du RIPiT. (A) Tache occidentale des protéines purifiées à partir de chaque étape majeure de la procédure RIPiT. (B) Image autoradiographique des empreintes d'ARN d'un FLAG-MAGOH:EIF4AIII RIPiT comparant les cellules traitées et non traitées par puromycine. La boîte rouge indique la taille de l'empreinte de l'ARN qui sera finalement convertie en bibliothèques de séquençage. (C) Profil de l'ARN élucidé du RIPiT comme dans la voie 2 dans le panneau B lorsqu'il est visualisé à l'aide d'un bioanalyseur. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Produit de transcription inversée (RT) résolu sur un gel 10% urée-PAGE et taché de tache de gel d'acide nucléique d'or. La boîte rouge indique la région de gel excisée pour la purification de gel des produits prolongés de RT. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
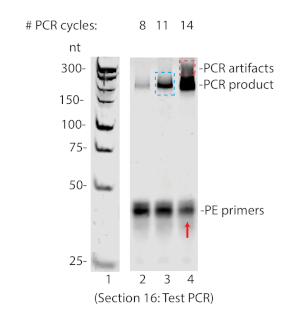
Figure 5 : Test PCR résolu sur 8% de PAGE non dénaturant. Notez les produits PCR anormalement grands qui apparaissent à 14 cycles (boîte rouge) et l'épuisement parallèle des amorces (flèche rouge) indicatif de suramplification. Pour cet échantillon, 11 cycles ont été choisis pour le PCR à grande échelle (boîte bleue). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
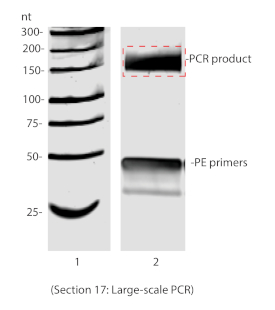
Figure 6 : PCR à grande échelle résolu sur 8% page non dénaturante. La boîte rouge indique la pièce de gel excisée pour la purification de gel. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 7 : Capture d'écran du navigateur génome du gène MAPK1 montrant la distribution des empreintes FLAG-MAGOH:EIF4AIII en tant que résultats représentatifs d'un RIPiT. Les flèches rouges désignent les sites de liaison canoniques attendus. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| Pbs | 137 mM NaCl 2,7 mM KCl 10 mM Na2HPO47H2O KH2PO4 pH 7,4 | |
| Tampon d'étanchéité | 2,5 M de glycine 2,5 mM Tris-Base | |
| Tampon de Lysis hypotonique (HLB) | 20 mM Tris-HCl pH 7,5 15 mM NaCl 10 mM EDTA 0,5% IGEPAL 0,1 % Triton-X-100 1x Aprotinin 1x Leupeptin 1x Pepstatin 1 mM de PMSF (fluorure de phénylméthylsulfonyle) | Doit être ajouté frais à chaque fois |
| Tampon de lavage isotonique (IsoWB) | 20 mM Tris-HCl pH 7,5 150 mM NaCl 0,1% IGEPAL | |
| Tampon de conjugaison | 0,02% Polysorbate-20 1x PBS | |
| Tampon d'échantillon clair | 100 mM Tris-HCl pH 6,8 4% SDS 10 mM EDTA | |
| 4xdNTPmix (en) | 0,25 mM dGTP 0,25 mM dTTP 0,175 mM dATP 0,1625 mM dCTP 0,075 mM de biotine-dATP 0,0875 mM biotine-dCTP | |
| 5x First-Strand Buffer w/o MgCl2 | 250 mM Tris-HCl pH 8 375 mM KCl | |
| Tampon de dilution RIPiT (1 ml) | 1 mL IsoWB 5 L 200x BSA 20 L 10% Triton-X-100 20 L 0,5 M EDTA 1x Aprotinin 1x Leupeptin 1x Pepstatin 1 mM PMSF | Doit être ajouté frais à chaque fois |
| 2x Dénaturation de la charge Tampon | 3 mL 5x TBE 1,8 g Ficoll Type 400 6,3 g d'urée 3 mg bleu bromophenol 3 mg de cyylène xylène Ajuster le volume à 15 mL ddH2O | Pour entrer dans la solution, placer le tube dans l'eau dans un bécher et faire bouillir sur la plaque chaude pendant 10-15 min. Ajouter les colorants après avoir ajusté le volume à 15 ml |
| Gel Urea-PAGE | 6 M Urea Acrylamide:bisacrylamide (40% [w/v]) au pourcentage approprié 0.5x TBE 0,01% TEMED | |
| Tampon d'elution d'ADN | 300 mM NaCl 1 mM EDTA | |
| Tampon de lavage de perle de Streptavidin | 0,5 M NaOH 20 mM Tris-HCl pH 7,5 1 mM EDTA |
Tableau 1 : Tampons.
Discussion
Nous discutons ici de quelques considérations clés pour effectuer avec succès RIPiT. Tout d'abord, les adresses IP individuelles doivent être optimisées pour atteindre la plus grande efficacité possible à chaque étape. La quantité de perles d'agarose DE FLAG pour le nombre d'entrées de cellules décrites ici s'est avérée robuste pour un large éventail de protéines que nous avons testées. Comme seule une petite fraction des protéines partenaires est co-immunoprécipitée avec la protéine FLAG, la quantité d'anticorps nécessaires à l'efficacité de la deuxième PI est généralement faible (moins de 10 g). RiPiT à petite échelle (à partir d'une plaque de 10 cm) suivi d'une vérification occidentale des protéines dans chaque fraction au cours des deux étapes d'immunoprécipitation s'avère extrêmement utile pour évaluer l'efficacité et la spécificité de la procédure avant la mise à l'échelle. Les deux protéines ciblées ainsi que toutes les autres protéines en interaction attendues dans le complexe devraient être détectées dans l'élution. Il est également utile d'essayer pour les protéines dans les lysates appauvris (non liés à la FLAG-agarose ou perles magnétiques) d'avoir une bonne estimation de l'efficacité de l'immunoprécipitation et le pourcentage de protéines assemblant dans un complexe. En outre, cette analyse indique également si les conditions de digestion de RNase sont suffisantes pour séparer les interactions ARN-dépendantes des interactions ARN-indépendantes dans un RNP. Par conséquent, il est tout aussi important d'inclure un contrôle négatif, idéalement un RBP sans rapport avec le RNP d'intérêt. Par exemple, dans la figure 3A, HNRNPA1 est présent dans l'entrée mais n'est pas détecté dans l'élution RIPiT. HNRNPA1 est un RBP qui n'interagit pas directement avec l'EJC mais interagit indirectement avec l'EJC lorsque l'EJC et le HNRNPA1 sont liés à la même molécule d'ARN. La détection de la protéine de contrôle négative dans l'élution indique soit une faible spécificité RIPiT ou une empreinte d'ARN insuffisante. Dans un tel cas, les empreintes d'ARN obtenues ne refléteront pas complètement les empreintes de la protéine d'intérêt. Les empreintes de pas de taille 50-200 nt sont recommandées pour l'ARN-Seq subséquent e. La durée du traitement rNase I ou la quantité d'enzyme s'ensuit peut être optimisée pour obtenir les empreintes de taille souhaitées. Notez que le meilleur scénario sera d'obtenir un bon signal dans la plage de taille souhaitée, et il est inévitable d'avoir des ARN plus longs et plus courts, même dans les conditions les plus optimales. RIPiT peut également être utilisé pour obtenir des sites de liaison d'un seul RBP. Dans un tel cas, la même protéine peut être immunoprécipitée avec deux anticorps différents, d'abord en utilisant un anticorps contre une étiquette d'affinité, puis avec des anticorps contre la protéine elle-même18. Enfin, un RIPiT de contrôle négatif peut être effectué en parallèle à partir de cellules exprimant une protéine de contrôle marquée par le FLAG (par exemple, protéine fluorescente verte) en combinaison avec un anticorps contre une protéine contre une protéine non apparentée dans la deuxième piédenance.
Malgré ses nombreux avantages, il est important d'envisager certaines limites de l'approche RIPiT, et des recours possibles. L'exigence d'une exution d'affinité après la première purification nécessite que la source biologique exprime une protéine étiquetée. Si un système de recombinaison spécifique au site n'est pas disponible dans la lignée cellulaire ou l'organisme d'intérêt, une courte étiquette d'affinité telle qu'une étiquette FLAG (8 acides aminés) peut être introduite au locus gène endogène à l'aide de CRISPR/Cas-basé approche d'édition du génome19. L'étiquette FLAG est un épitope idéal pour cette approche, parce que l'anticorps FLAG est bien adapté pour l'élution d'affinité et peut résister à des forces ioniques élevées et des conditions de dénaturation douces qui peuvent être utilisées en combinaison avec le formdéhyde crosslinking. Une autre limite de l'approche RIPiT est l'exigence d'un apport important de matériel cellulaire. Cela peut rester inévitable dans une certaine mesure que seul un faible pourcentage d'un RBP interagit probablement avec d'autres protéines dans le RNP. Des approches encore améliorées de préparation des bibliothèques peuvent aider à réduire l'exigence d'entrée importante. Parmi les idées possibles pour rationaliser davantage ces étapes, mentionnons la réalisation de la déphosphorylation de l'ARN 3'-fin et la ligature adaptateur sur les perles magnétiques immédiatement après les lavages DE PI deuxième et avant l'élution finale de RNP. Une telle approche est mise en œuvre avec succès dans les procédures actuelles de CLIP-Seq et dans une variation récemment décrite de RIPiT20. Ces changements élimineront également plusieurs étapes de purification de l'ARN qui prennent beaucoup de temps dès les premières phases de la procédure de préparation de la bibliothèque. En outre, contrairement à CLIP, qui fournit une résolution de niveau nucléotide du site de liaison croisée d'un RBP sur l'ARN, la résolution des empreintes RIPiT restera au niveau de dizaines de nucléotides. Enfin, comme les RRP peuvent inclure plusieurs RRP, les sites d'ARN enrichis du RIPiT comprennent un mélange de sites de liaison de nombreux RRP. Comme les séquences consensuelles liées par des RRP individuels sont mises au jour à un rythme en croissance rapide et sont maintenant facilement disponibles21,22,23, cette information peut être exploitée pour décongestionner l'assortiment de sites RBP enrichi s'enrichissent en sorties RIPiT. Malgré ces défis, RIPiT-Seq est une procédure efficace pour capturer les empreintes d'ARN des complexes RNP dynamiques, hétérogènes et même transitoires, qui peuvent fournir des informations uniques sur le fonctionnement interne des machines à ARN qui contrôlent les complexes cellulaires fonction.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été soutenu par la subvention des NIH GM120209 (GS). Les auteurs remercient l'OSUCCC Genomics Shared Resources Core pour leurs services (CCC Support Grant NCI P30 CA16058).
matériels
| Name | Company | Catalog Number | Comments |
| Anti-FLAG Affinity Gel | Sigma | A2220 | |
| ATP, [γ-32P]- 3,000 Ci/mmol 10 mCi/mL EasyTide, 250 µCi | PerkinElmer | BLU502A250UC | |
| BD Disposable Syringes with Luer-Lok Tips (200) | Fisher | 14-823-435 | |
| Betaine 5M | Sigma | B0300 | |
| biotin-dATP | TriLink | N-5002 | |
| biotin-dCTP | Perkin Elmer | NEL540001EA | |
| Branson Sonifier, Model SSE-1 | Branson | ||
| CircLigase I | VWR | 76081-606 | ssDNA ligase I |
| DMEM, High Glucose | ThermoFisher | 11995-065 | |
| DNA load buffer NEB | NEB | ||
| Dynabeads Protein A | LifeTech | 10002D | |
| Flp-In-T-REx 293 Cell Line | ThermoFisher | R78007 | |
| GeneRuler Low Range DNA Ladder | ThermoScientific | FERSM1203 | |
| Hygromycin B | ThermoFisher | 10687010 | |
| Mini-PROTEAN TBE Gel 10 well | Bio-Rad | 4565013 | |
| Mini-PROTEAN TBE-Urea Gel | Bio-Rad | 4566033 | |
| miRCAT-33 adapter 5′-TGGAATTCTCGGGTGCCAAGGddC-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Mirus transIT-X2 transfection reagent | Mirus | MIR 6004 | |
| Mth RNA ligase | NEB | E2610S | |
| PE1.0 5′-AATGATACGGCGACCACCGAGATCTACACT CTTTCCCTACACGACGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| PE2.0 5′-CAAGCAGAAGACGGCATACGAGATCGGTCTC GGCATTCCTGCTGAACCGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Phenol/Chloroform/Isoamyl Alcohol (25:24:1, pH 6.7, 100 mLl) | Fisher | BP1752I-100 | |
| Purple Gel Loading Dye (6x) | NEB | NEB #7025 | |
| Q5 DNA Polymerase | NEB | M0491S/L | |
| RNase I, E. coli, 1,000 U | Eppicenter | N6901K | |
| SPIN-X column | Corning | CLS8160-24EA | |
| Streptavidin beads | ThermoFisher | 60210 | |
| Superscript III (SSIII) | ThermoScientific | 18080044 | reverse transcriptase enzyme |
| SybrGold | ThermoFisher | S11494 | gold nucleic acid gel stain |
| T4 Polynucleotide Kinase-2500U | NEB | M0201L | |
| T4RNL2 Tr. K227Q | NEB | M0351S | |
| Tetracycline | Sigma | 87128 | |
| Thermostable 5´ App DNA/RNA Ligase | NEB | M0319S | |
| TruSeq_SE1 5′-pGGCACTANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE10 5′-pGGTGTTCNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE11 5′-pGGTAAGTNNNNNAGATCGGAA GAGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE12 5′-pGGAGATGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE2 5′-pGGGTAGCNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE35′-pGGTCGATNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE4 5′-pGGCCTCGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE5 5′-pGGTGACANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE6 5′-pGGTAGACNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE7 5′-pGGGCCCTNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCT TCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE8 5′-pGGATCGGNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTT CCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE9 5′-pGGACTGANNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Typhoon 5 Bimolecular Imager | GE Healthcare Life Science | 29187191 |
Références
- Karousis, E. D., Nasif, S., Mühlemann, O. Nonsense-mediated mRNA decay: novel mechanistic insights and biological impact. Wiley Interdisciplinary Reviews: RNA. 7 (5), 661-682 (2016).
- Ivanov, P. V., Gehring, N. H., Kunz, J. B., Hentze, M. W., Kulozik, A. E. Interactions between UPF1, eRFs, PABP and the exon junction complex suggest an integrated model for mammalian NMD pathways. The EMBO Journal. 27 (5), 736-747 (2008).
- Papasaikas, P., Valcárcel, J. The Spliceosome: The Ultimate RNA Chaperone and Sculptor. Trends in Biochemical Sciences. 41 (1), 33-45 (2016).
- Jensen, K. B., Darnell, R. B. CLIP: Crosslinking and ImmunoPrecipitation of In Vivo RNA Targets of RNA-Binding Proteins. Methods in Molecular Biology. 488, Clifton, N.J. 85-98 (2008).
- Garzia, A., Morozov, P., Sajek, M., Meyer, C., Tuschl, T. PAR-CLIP for Discovering Target Sites of RNA-Binding Proteins. mRNA Decay: Methods and Protocols. 1720, 55-75 (2018).
- Konig, J., et al. iCLIP - Transcriptome-wide Mapping of Protein-RNA Interactions with Individual Nucleotide Resolution. Journal of Visualized Experiments. (50), e2638(2011).
- Sibley, C. R. Individual Nucleotide Resolution UV Cross-Linking and Immunoprecipitation (iCLIP) to Determine Protein-RNA Interactions. RNA Detection: Methods and Protocols. 1649, 427-454 (2018).
- Wheeler, E. C., Van Nostrand, E. L., Yeo, G. W. Advances and challenges in the detection of transcriptome‐wide protein-RNA interactions. Wiley Interdisciplinary Reviews: RNA. 9 (1), (2018).
- Singh, G., et al. The Cellular EJC Interactome Reveals Higher-Order mRNP Structure and an EJC-SR Protein Nexus. Cell. 151, 750-764 (2012).
- Singh, G., Ricci, E. P., Moore, M. J. RIPiT-Seq: A high-throughput approach for footprinting RNA:protein complexes. Methods. 65, 320-332 (2014).
- Mabin, J. W., et al. The Exon Junction Complex Undergoes a Compositional Switch that Alters mRNP Structure and Nonsense-Mediated mRNA Decay Activity. Cell Reports. 25 (9), 2431-2446 (2018).
- Gehring, N. H., Lamprinaki, S., Kulozik, A. E., Hentze, M. W. Disassembly of Exon Junction Complexes by PYM. Cell. 137 (3), 536-548 (2009).
- Dostie, J., Dreyfuss, G. Translation Is Required to Remove Y14 from mRNAs in the Cytoplasm. Current Biology. 12 (13), 1060-1067 (2002).
- Zünd, D., Gruber, A. R., Zavolan, M., Mühlemann, O. Translation-dependent displacement of UPF1 from coding sequences causes its enrichment in 3' UTRs. Nature Structural & Molecular Biology. 20 (8), 936-943 (2013).
- Gangras, P., Dayeh, D. M., Mabin, J. W., Nakanishi, K., Singh, G. Cloning and Identification of Recombinant Argonaute-Bound Small RNAs Using Next-Generation Sequencing. Argonaute Proteins: Methods and Protocols. 1680, 1-28 (2018).
- Heyer, E. E., Ozadam, H., Ricci, E. P., Cenik, C., Moore, M. J. An optimized kit-free method for making strand-specific deep sequencing libraries from RNA fragments. Nucleic Acids Research. 43 (1), 2(2015).
- Cameron, V., Uhlenbeck, O. C. 3'-Phosphatase activity in T4 polynucleotide kinase. Biochemistry. 16 (23), 5120-5126 (1977).
- Ricci, E. P., et al. Staufen1 senses overall transcript secondary structure to regulate translation. Nature Structural & Molecular Biology. 21 (1), 26-35 (2014).
- Lackner, D. H., et al. A generic strategy for CRISPR-Cas9-mediated gene tagging. Nature Communications. 6, 10237(2015).
- Metkar, M., et al. Higher-Order Organization Principles of Pre-translational mRNPs. Molecular Cell. 72 (4), 715-726 (2018).
- Giudice, G., Sánchez-Cabo, F., Torroja, C., Lara-Pezzi, E. ATtRACT-a database of RNA-binding proteins and associated motifs. Database: The Journal of Biological Databases and Curation. 2016, (2016).
- Paz, I., Kosti, I., Ares, M., Cline, M., Mandel-Gutfreund, Y. RBPmap: a web server for mapping binding sites of RNA-binding proteins. Nucleic Acids Research. 42, Web Server issue 361-367 (2014).
- Sundararaman, B., et al. Resources for the Comprehensive Discovery of Functional RNA Elements. Molecular Cell. 61 (6), 903-913 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon