Method Article
זיהוי של טביעות רגליים של RNA: מכלולי חלבון באמצעות RNA Immunoprecipitation בטנדם ואחריו רצף (RIPiT-Seq)
In This Article
Summary
כאן, אנו מציגים פרוטוקול להעשיר את אתרי ה-RNA האנדוגניים או "עקבות" של RNA: מכלולי חלבון (RNP) מתאי יונקים. גישה זו כוללת שני immunoprecipitations של RNP תת יחידות, ולכן המכונה RNA immunoprecipitation בטנדם (RIPiT).
Abstract
רנ א immunoprecipitation בטנדם (RIPiT) היא שיטה להעשרת עקבות RNA של שני החלבונים בתוך RNA: חלבון (RNP) קומפלקס. RIPiT מעסיקה שני צעדי טיהור. ראשית, immunoprecipitation של RNP מתויג משנה לאחר מכן העיכול מתון RNase ובעקבות הימנעות האהדה הבאה. הimmunoprecipitation השנייה של יחידת משנה RNP נוספת מאפשרת להעשיר מתחם מוגדר. בעקבות הימנעות של RNAs וחלבונים, עקבות ה-RNA מומרים לספריות רצפי DNA של תפוקה גבוהה. בניגוד לאולטרה סגול פופולרי (UV) החוצה קישור ואחריו immunoprecipitation (CLIP) גישה להעשיר RBP אתרי כריכה, RIPiT הוא UV-crosslinking עצמאי. מכאן RIPiT יכול להיות מיושם על חלבונים רבים הנמצאים ב-RNA interactome ומעבר כי הם חיוניים רגולציה RNA אבל לא ישירות ליצור קשר עם RNA או UV-crosslink גרוע RNA. שני צעדי הטיהור בריבור מספקים יתרון נוסף של זיהוי אתרי קשירה שבהם חלבון הריבית מתפקד בשותפות עם קופקטור אחר. אסטרטגיית הטיהור הכפולה משמשת גם להגברת האות באמצעות הגבלת הרקע. כאן, אנו מספקים הליך מבחינת צעד כדי לבצע RIPiT ולייצר התפוקה גבוהה ברצף ספריות מתוך עקבות RNA מבודדים. כמו כן, אנו מתווה את היתרונות והיישומים של RIPiT ודנים בחלק מהמגבלות שלה.
Introduction
בתוך תאים, RNA קיים מורכב עם חלבונים כדי ליצור RNA: מכלולי חלבון (RNPs). RNPs מקובצים סביב ה-RNA כריכת חלבונים (Rnps, אלה אשר לאגד ישירות RNA) אבל גם מהווים ללא Rnps (אלה לאגד Rnps אך לא RNA), ולעתים קרובות דינמי בטבע. RBPs והקופדים שלהם פועלים באופן קולקטיבי בתוך Rbps כדי לבצע פעולות רגולטוריות. לדוגמה, ב-mRNA בתיווך שטויות (NMD) מסלול, החלבונים UPF (UPF1, UPF2, ו UPF3b) מזהים את ריבוכמה הסתיים בטרם עת. כל אחד מחלבונים UPF יכול לאגד RNA, אבל זה רק כאשר הם להרכיב יחד כי מורכבת NMD פעיל מתחיל להיווצר. בתוך מתחם זה, UPF1 מופעל עוד על ידי זירחון על ידי SMG1 שאינו rbp, והפעלת UPF1 כגון בסופו של דבר מובילה לגיוס של מקדם ריקבון mrna הגורם1,2. בדוגמה זו, RBPs דורש שחקנים שאינם מבוססי RBPS עבור גיוס והפעלה של מתחם RBPS המפעיל את NMD. מאפיין נוסף של RNPs הוא הטרוגניות שלהם. שקול את הספצאוחלק, אשר קיים ברורים E, A, B או C מתחמים. מתחמים שונים מתחמי יש לחפוף חלבונים ברורים3. כדי ללמוד פונקציות RNP, חשוב להבהיר אילו Rnp מאוגדים על ידי RNP ואת החלבונים המשויכים שלה. שיטות רבות קיימות כדי להשיג זאת, עם כל גישה יש יתרונות וחסרונות ברורים שלה4,5,6,7.
השיטות הנפוצות ביותר כדי לזהות אתרי כריכה RBP-הקישור ואחריו immunoprecipitation (קליפ) ואת הווריאציות השונות שלה-להסתמך על אולטרה סגול (UV) אור כדי crosslinking RBP ל-RNA8. עם זאת, זו אינה גישה אפקטיבית עבור non-RBPs בתוך Rbps, אשר אינם פונים אל RNA ישירות. כאן, אנו מתארים גישה חלופית הישימה ל-RBPs וללא RBPs כאחד, כדי לבודד ולזהות את אתרי איגוד ה-RNA שלהם. גישה זו נקראת RNA immunoprecipitation בטנדם (ripit) מורכב משני שלבים immunoprecipitation, אשר לעזור להשיג ספציפיות יותר לעומת טיהור אחד (איור 1)9,10. כפי שניתן לבצע את הצעדים הבודדים immunoprecipitation (IP) באופן מוגזם לעומת הקליפ, RIPiT אינו תלוי בזמינות של נוגדנים שיכולים לעמוד בפני נוכחות של חומרי ניקוי חזקים במהלך immunoprecipitation. היתרון הייחודי ביותר של RIPiT הוא היכולת למקד שני חלבונים שונים בשני שלבי טיהור; הדבר מספק דרך רבת-עוצמה להעשיר את מתחם ה-RNP שונה ומורכב מתסביכים דומים אחרים11.
וריאציות קטנות להליך RIPiT יכול לשפר עוד יותר את העשרה RNP. למשל, כמה אינטראקציות RNA-חלבון או חלבון-חלבון בתוך RNPs הם ארעי וזה עלול להיות קשה לטהר ביעילות את העקבות של תסביכים כאלה. כדי לייצב אינטראקציות כאלה, RNPs יכול להיות מקושר בתוך תאים עם פורמלדהיד לפני הליזה תא ו RIPiT. לדוגמה, ראינו כי אינטראקציה חלשה בין מורכבות הצומת אקסון (ejc) הליבה גורם, EIF4AIII וגורם פירוק ejc, pym12 ניתן לייצב עם טיפול פורמלדהיד כגון זה יותר העקבות RNA מועשרים (נתונים לא מוצג). לפני איסוף התא RIPiT, תאים יכולים גם להיות מטופלים עם תרופות כדי לייצב או להעשיר RNPs במצב מסוים. לדוגמה, כאשר לומדים חלבונים שהוסרו מ-mRNA במהלך התרגום (למשל, EJC13, UPF114), טיפול במעכבי תרגום כגון puromycin, cycloheximide או harringtonine יכול להוביל לאכלוס מוגבר של חלבונים ב-RNAs.
כמות ה-RNA ששוחזר מ-RIPiT נמוך בדרך כלל (0.5-10 שומות, כלומר, 10-250 ng RNA בהתחשב באורך RNA ממוצע של 75 nt). הסיבה העיקרית לכך היא כי רק חלק קטן של חלבון נתון קיים מורכב עם חלבונים אחרים בתוך RNPs (כל "חינם" החלבון IP'ed בשלב הראשון הוא איבד במהלך ה-IP השני). כדי לייצר ספריות rna-Seq מן ה-rna הזה, אנחנו גם מתווה כאן עיבוד של פרוטוקול שפורסם בעבר מתאים כגון RNA נמוך כגון15,16 (איור 2), אשר מניב את התפוקה הגבוהה ברצף מוכן דגימות של 3 ימים.
Protocol
1. הקמת HEK293 אורווה קווים תאים הבעת טטרציקלין-inducible דגל-מתויג חלבון של עניין (פוי)
- זרע HEK293 תאים עם משולב באופן בלתי נשכח Flp היעד (FRT) באתר בצפיפות של 10 x 104 תאים/mL בתווך הצמיחה (בינונית שונה של העיט של הנשר [dmem] + 10% סרום העוברי העובר [fbs] +1% פניצילין-סטרפטומיצין [פן/דלקת]) ב 6- צלחות. אפשר לתאים לגדול בן לילה בחממה מחולל לחות ב 37 ° c ו-5% CO2 (תנאי גדילה סטנדרטיים עבור כל השלבים הבאים).
- למחרת, התאים צריכים להיות ~ 70% קונfluent. על פי פרוטוקול מגיב העברה, transfection אתר FRT-המכיל תאים HEK293 עם יחס 9:1 של pcDNA5-TETO-דגל-פוי: pOG44.
הערה: תג דגל יש את מוטיב הרצף dykddddk (D = אספרטית חומצה, Y = טירולך, ו K = ליזין). - לאחר 24 שעות, התחילו בחירה אנטיביוטית. הסר מדיה ולהוסיף מדיום צמיחה טריים שיושלם עם 100 μg/mL hygromycin. בתוך 72 h, תאים בלתי. מפותחים אמורים להתחיל למות
- כל 48-72 h, לשנות את מדיית הצמיחה ותוספת עם hygromycin טריים.
- לאחר ~ 2 שבועות של בחירה, מושבות בדידים של תאים מזוהמים מאוד יתחילו להופיע. ברגע שמושבות גלויות לעין בלתי, הוסיפו 1 מ ג של טריפסין לצלחת והדגירה 5 דקות ב 37 ° c. השהה את התאים מחדש ב-DMEM, העברת תאים ללוח חדש של 10 ס מ, ולהתאים את עוצמת הקול ל 10 mL בינונית צמיחה טריים שנוספו עם 100 μg/mL hygromycin. אפשר לצלחת להגיע ~ 80% השטף כדי להרחיב את התאים כדי להכין מניות קבע.
- לקבוע את כמות טטרציקלין (ט) נדרש כדי לקבל את הרמה האופטימלית של ביטוי דגל-פוי עבור הניסוי. לגדול תאים בפורמט 12-טוב ולבצע טיטור של ט בין 0-1000 ng/mL טווח עבור 16-24 h. ביצוע הכלים המערבי על דגימות טיטור עם נוגדן נגד פוי.
הערה: עבור חלבונים עד ~ 60 kDa, הדגל-tagged ואת העותק אנדודוגני של החלבון ניתן לפתור על נתרן dodecyl סולפט-פוליאקרילמיד ג'ל אלקטרופורזה (SDS-PAGE) כדי להשוות את רמות הביטוי של דגל-פוי לעמיתו האנדוגניים שלה. עבור חלבונים גדולים יותר, עוצמת האות בדגימות המושרה ב-ט' ניתנת להשוואה למדגם שאינו מושרה. פתרון מניות טטרציקלין ניתן להכין ב 1 מ"ג/mL ב 100% אתנול. דילול המניה לעבודת תרבות התא צריכה להיות במים סטריליים או בתמיסת מלח (PBS). מניות טטרציקלין צריך להיות מוכן טרי פעם בחודש.
2. תאים culturing עבור טטרציקלין האינדוקציה נוהל RIPiT
- משולב זרעים באופן בלתי נשכח הדגל-פוי HEK293 בצפיפות של 3 x 105 תאים/mL בינונית צמיחה ב 15 ס מ לוחות. אפשר לתאים לצמוח ב 37 ° צ' ו 5% CO2.
הערה: באופן כללי, שלושה עד 5 15 ס"מ לוחות תניב ~ 2-20 pmol של RNA טביעות רגל בהתאם לשפע של RNP של ריבית. אם RNPs יהיה פורמלדהיד מחובר ומטוהרים בתנאים מחמירים, אז יש לדרוש כפול מכמות הקלט. - הוסף טטרציקלין לריכוז אופטימלי מראש על התקשורת כדי לגרום לביטוי של הדגל-פוי (ראה שלב 1.6) 16-24 h לפני התאים ייקצרו.
הערה: אין צורך לשנות את מדיום הצמיחה. תאים צריכים להיות מוכנים לקצור כ 72 h לאחר זריעת או כאשר צלחות הם ~ 80% confluent. חשוב להימנע מתאים להיות שוטפת.
3. לקצור תא, טיפול פורמלדהיד, ולליזה תא
- לשטוף את התאים המונאולייר בעדינות עם 15 מ ל של PBS מקורר עבור כל לוחית 15 ס מ. ואז לגרד את התאים ב 30 מ ל של PBS. אם התאים טופלו בסמים לפני הקציר, הוסף את ה-PBS עם הסם. לאסוף את כל התאים לתוך 50 מ"ל צינור חרוט.
הערה: עבור שמירה על אינטראקציות חלשות, תאים יכולים להיות מקושרים עם פורמלדהיד: להוסיף פורמלדהיד ההשעיה התא משלב 3.1 לריכוז הסופי של 0.1% ו-דגירה על הנדנדה בחדר הטמפרטורה עבור 10 דקות. הוסף 3 מ ל של מאגר הקוצ'ינג (טבלה 1 ) ורוק ל -5 דקות נוספות. - בתאי גלולה ב 400 x g עבור 10 דקות ב 4 ° צ' ולהיפטר supernatant.
- Lyse תאים ב 4 מ ל של מאגר לפירוק קרח קר יפוטוניק (hlb; טבלה 1). השתמש בפיפטה P1000 כדי להשעות את התאים מחדש. העברה לצינור 5 מ ל ומטה בקרח במשך 10 דקות.
- הניחו את הליפוסט באמבט קרח והsonicate ב -10% משרעת במשך 30% עם פולסים של 1 עם 2 הפסקות. אז, להתאים ריכוז מלח ל 150 מ"מ על ידי הוספת 108 μL של 5 M הנאל.
הערה: פורמלדהיד דגימות מקושרות יכול להיות כפוף לפירוק מחמיר יותר וטיהור על-ידי כולל 0.1% כל אחד של SDS ו-נתרן deoxycholate במאגר הפירוק. - נקה את הליפוסט על ידי צנטריפוגה ב 21,000 x g עבור 10 דקות ב 4 ° c. לאסוף 20 μL supernatant (תמצית התא) בצינור מסומן עבור ניתוח כתמי האבן המערבית של רמות החלבון בקלט (ראה איור 3א).
הערה: בעוד ליפוסט הוא בצנטריפוגה, דגל agarose חרוזים ניתן לשטוף (ראה שלב 4.1). הדגל העלה חרוזים צריך להיות מראש שטף 3 x ב 4 מ ל של קרח קר איזוטוניק מאגר שטיפת (IsoWB, שולחן 1).
4. דגל Immunoprecipitation
- החל את הסופרנטאנט הנותר משלב 3.5 עד 750 μL הדגל ששטף מראש מחרוזות בצינור 5 מ ל (מיטה נפח של שטף הדגל שנשטפו חרוזים יהיה 375 μL). הדגל מקרין חרוזים ותמצית התא עבור 1-3 h ב 4 ° צ' עם ערבוב עדין.
הערה: כמות זו של חרוזים בדגל העלה צריך להספיק למגוון רחב של חלבונים, אך עשוי להיות ממוטב, במידת הצורך. - גלולה הדגל agarose חרוזים על ידי צנטריפוגה ב 400 x g עבור 1 דקות ב 4 ° c. לאסוף 20 μL של supernatant בצינור מסומן עבור ניתוח כתמי המערבי של רמות החלבון בתמצית תא מרוקן (ראה איור 3א). מחק את הסופרנטאנט הנותר.
- כדי לשטוף את החרוזים בדגל, הוסף 4 מ ל IsoWB והשהה מחדש. גלולה חרוזים ב 400 x g עבור 1 דקות ב 4 ° c. הסר בזהירות את הסופרנטאנט. . חזור על 4x
הערה: עבור שוטפים מחמירים של הפורמלדהיד המקושרים באמצעות ה-IPs, 0.1% כל אחד מסוגי החמצן והנתרן ניתן לכלול במאגר הכביסה עבור שני שלבי הכביסה הראשונים.
5. RNase העיכול
- לדלל RNase I כדי 0.002-0.01 יחידות/mL ב 750 μL של IsoWB (הריכוז המתאים עבור גדלי טביעת הרגל הרצויה צריך להיות החלטי מדעית). הוסף IsoWB-RNase I כדי לשטוף את הדגל מחרוזת agnor ו דגירה עם ערבוב עדין ב 4 ° צ' עבור 10 דקות.
- גלולה חרוזים ב 400 x g עבור 1 דקות ב 4 ° c. לאסוף 20 μL supernatant (RNase אני הימנעות) בצינור מסומן עבור ניתוח כתמי אבן מערבית. למחוק IsoWB-RNase אני ולשטוף את הדגל agarose חרוזים 4x עם IsoWB כפי שמתואר בשלב 4.3.
6. הימנעות אהדה
- הכינו מלאי של מאגר הימנעות (פפטיד דגלים ב 250 ng/mL ב IsoWB). החל 375 μL של מאגר הימנעות כדי דגל agarose חרוזים ולנענע בעדינות ב 4 ° צ' עבור 1-2 h. גלולה דגל agarose חרוזים ולאסוף 15 μL ליטר של הימנעות עבור הכתם המערבי של חלבונים בדגל IP (ראה איור 3א).
הערה: כאשר הימנעות הזיקה מתבצעת, ניתן לבצע סעיף 7.
7. חרוז מגנטי-הנוגדן הקוניוגציה
- שטוף 50 μL חרוזים מגנטיים (כלומר, לסביות; רשימת חומרים) 3x ב 1 mL IsoWB בצינור 1.5 mL. השעיה מחדש חרוזים מגנטיים ב 100 μL של מאגר הקוניוגציה. הוסף כמות מתאימה של נוגדן (כמות מדויקת של נוגדן לשימוש עבור IP יהיה צורך אמפירי נחוש עבור כל נוגדן).
הערה: חלבון חרוזים מגנטיים הם מיטביים עבור נוגדנים המיוצרים בארנבים ואילו חרוזי חלבון G מגנטי מתאימים יותר לנוגדנים בעכבר. התאמה מגנטית התרשים תאימות זמין באתר האינטרנט של הספק כדי לבחור חרוזים מתאים לכל נוגדן. - שוטפים חרוזים מגנטיים 2x בתוך הקוניוגציה (שולחן 1). התערובת דגירה בטמפרטורת החדר עבור לפחות 10 דקות. מחרוזות מגנטיות Resuspend 375 μL של מאגר דילול RIPiT. . לחנות בקרח עד השלב הבא
8. הImmunoprecipitation השנייה
- החלת שארית האהדה של הדגל משלב 6.1 לחרוזים מגנטיים מצמידים לנוגדנים נגד חלבון הריבית. דגירה עם ערבוב עדין ב 4 ° צ' עבור 1-2 h. ללכוד חרוזים מגנטיים על מגנט ולאסוף 15 μL של supernatant לניתוח של חלבונים לא מאוגדים דרך אבן החשופה המערבי. שוטפים חרוזים מגנטיים 7x עם 1 מ ל של IsoWB.
9. הללויה
- הוסף 100 μL של מאגר לדוגמה ברורה (טבלה 1) לחרוזים מגנטיים והשעיה מחדש עם P200. מריקון על הקרח 10 דקות. קפיצי בעדינות כדי להשעות מחדש חרוזים מדי פעם.
- ללכוד חרוזים מגנטיים על מגנט ולאסוף 15 μL של הימנעות עבור ניתוח של חלבונים ב-RIPiT הימנעות באמצעות כתם המערבי (ראה איור 3א). העבר את הימנעות שנותרו לצינור 1.5 mL חדש שכותרתו.
הערה: אם הדגימות היו פורמלדהיד מקושרים, אז דגימות חייב להיות מודב 65 ° c עבור 1 h כדי להפוך את הקישור החוצה. - לבצע הכלים המערבי על דגימות שנאספו בשלבים שונים (קלט, דגל המחסור של ה-ip, דגל ip, המחסור ip השני, הימנעות ip השני). אבן חשופה עם נוגדנים נגד שני החלבונים פיתיון, השחקנים האחרים שלהם אם ידוע, ולפחות RBP אחד ללא אינטראקציה כפקד שלילי (איור 3א).
10. RNA החילוץ והסיום אשפרה
- כדי הימנעות RIPiT, להוסיף 320 μL של RNase-חינם ddH2O, 400 μl של פנול-כלורופורם isoamyl אלכוהול (PCIAA, pH 4.5), ומערבולת עבור 30 s ו ספין בטמפרטורת החדר ב 12,000 x g עבור 5 דקות. לאסוף 350 μl של שלב מימית לתוך צינור נפרד. הוסף 35 μL של 3 M אצטט נתרן, 1 μL של 1 M MgCl2, 10 μg של הגליקוגן, ו 1 מ ל של 100% אתנול. המלון משלב בין לילה ב-20 ° c.
- כדי גלולה RNA, צנטריפוגה ב 12,000 x g עבור 30 דקות ב 4 ° c. לשטוף את ה-RNA ב 70% אתנול.
- כדי להסיר 3 ' פוספט שמאל על RNA אחרי RNase אני מחשוף, להשעות את הגלולה RNA בתוך 17 μL של RNase-חינם ddH2O, ולהוסיף 2 μl של 10X T4 polynucleotide קינאז (pnk) מאגר (טבלת חומרים) ו-1 ΜL של T4 pnk. מודקון ב 37 ° c עבור 30 דקות.
הערה: The 3 ' פוספספטאז פעילות של T4 PNK יש פעילות אופטימלית ב-pH 617. מאגר התגובות PNK ממוטב עבור פעילות 5 ' קינאז של T4 PNK ו-pH של 7.6. בעוד התאמת ה-pH של התגובה לריפוי הסופי כבר נוסה, שלב זה יכול להיות אופטימיזציה נוספת. - הוסף 380 μL של RNase-חינם ddH2O ו 400 ΜL של PCIAA pH 4.5 לצינור. מערבולת עבור 30 s, צנטריפוגה ב 12,000 x g עבור 5 דקות. לאסוף שלב מימית ולהוסיף 35 μl של 3 M נתרן אצטט, 1 μl של 1 M MgCl2, 10 μg של הגליקוגן, ו 1 מ ל של 100% אתנול.
- המלון משלב בין לילה ב-20 ° c. גלולה ולשטוף RNA עם 70% אתנול כלפי מעלה. השהה RNA מחדש ב-4.5 μL של המים החופשיים של RNase.
11. הערכה של גודל טביעת הרגל RNA שפע
-
RIPiT מוצלח צפוי להניב 1 pmol או יותר של קטעי RNA. כדי לכמת תשואה בפועל, להעביר 0.7 μL של RIPiT RNA (~ 1/6 של תשואה כוללת) לתוך צינור חדש. הוסף 2 μL של מאגר PNK של 10x T4, 1 μL של 1 מ"מ ATP, 40 μCi γ32P-atp (0.5-1.0 ליטר של המניה) ו-1 μl של T4 pnk. להתאים את עוצמת הקול ל 10 μL ו-דגירה ב 37 ° צ' עבור 30 דקות.
- בתגובות PNK מקבילים, התווית סולם נמוך של דנ א ו-0.1 pmol של RNA סינתטי או DNA oligo (20-40 nt) כדי להשתמש בגודל וכמות סטנדרטים.
- לפתור את התווית RNA/DNA על 26% אוריאה-עמוד ג'ל (20 x 27 x 0.45 מ"מ3). ג'ל חייב להיות מראש לרוץ 30 דקות ב 35 W. ריקון בארות לפני הפעלה מראש ולפני טעינת דגימות ולרוץ ב 35 W עד ברומאופנול כחול לצבוע החזית כמעט הגיע לסוף הג.
- בזהירות להסיר ג'ל מלוחות זכוכית על חתיכת נייר 8 x 11 מסנן אינץ. עם ג'ל על גבי נייר, המקום במנגנון ייבוש ג'ל וכיסוי עם פיסת ניילון עטיפה. ג'ל יבש ב 80 ° c עבור 1 h עם ואקום.
- לחשוף ג'ל מיובש הלילה זרחן לילה או עד האות הולם מזוהה. התמונה פוספפווטמסך. באיכות טובה RNA מתוך RIPiT צריך להופיע ככתם בנתיב, עם להקות בולטות מינימלית (איור 3ב). כדי לכמת RNA, להשוות את עוצמת האות של מקטעי RNA בגודל הרצוי במשעול RIPiT לאות מ 0.1 pmol של התווית המתולכת סינתטי.
הערה: לחילופין, גודל טביעת הרגל של RNA וכמויות ניתן לאימות באמצעות bioanalyzer רגישות גבוהה (איור 3ג).
12. היטל מתאם
- להכין את ה-RNA RIPiT כזה לפחות 3 pmol של RNA הוא מומס 3.8 μL של מים.
- בשנת 0.2 mL פולימראז תגובת שרשרת (PCR) שפופרת, לשלב 3.8 μL של RNA, 1 μL של מיר-CAT-33 טרום מתאם adenylated (7 μM) (טבלת חומרים). הוסיפו את התערובת על הציקלייט התרמי ב-65 ° c במשך 10 דקות, 16 ° c עבור 5 דקות, ולאחר מכן החזיקו ב -4 ° c.
הערה: מקשר טרום האדלליס ניתן להזמין משירות הסינתזה oligo, או DNA מותאם אישית unadenylated משירות כל שירות הסינתזה oligo יכול להיות adenylated באמצעות mth RNA ליגאז (הטבלה של חומרים) ג'ל מטוהרים. - לאותו צינור, להוסיף 1.5 μl של 10x T4 RNA ליגאז מאגר, 7.5 μl של 50% פוליאתילן גליקול 8000 (יתד-8000), 0.75 μl של 20 מ"מ dithioitol (dtt), ו-0.45 μl של T4 RNL2 Tr. K227Q (לוח חומרים).
הערה: 50% יתד-8000 מגיע עם ליגאז RNA ו מאגר נרכש. הפתרונות של יתד-8000 הם צמיגה ויש לפיעץ לאט. - התגובה דגירה ב הציקלונט תרמית ב 30 ° c עבור 6 h, חום להפעיל את ליגאז ב 65 ° c עבור 10 דקות, ולאחר מכן להחזיק ב 4 ° c.
13. תמלול הפוך
- לצינור עם תערובת הקשר משלב 12.4, להוסיף 11.25 μl של 4x deoxynucleotide טריפוספט (dntp) מיקס, אשר מכיל שילוב של dntps רגיל ו biotinylated (לראות את הטבלה 1), 1.0 μl של 10 μm RT התחל (לוח חומרים), ו6.8 μl של . מים ללא משקה מודקון ב 65 ° c עבור 5 דקות, ולאחר מכן להחזיק 4 ° c.
- העברת צינורות לקרח ולהוסיף 9.0 μL של 5x הראשון-סטרנד (FS) מאגר ללא MgCl2 (טבלה 1), 2.25 Μl של 100 מ"מ Dtt, 1.2 μl של אנזים היפוך ההמרה לנפח הסופי של 45 Μl (טבלת חומרים).
- מודקון בציקלונט תרמית ב 55 ° c עבור 30-60 דקות. חום להפעיל היפוך ההמרה ב-70 ° c עבור 15 דקות ולהחזיק את המדגם ב 4 ° c.
14. טיהור של מוצר RT
- הוסף 45 μL של מאגר טעינה של אוריאה 2x (טבלה 1) לתגובת RT. לדלל 1 μg של סולם נמוך DNA מטווח ב 45 μL ולהוסיף 45 μL של מאגר לטעון אוריאה 2x.
- הכינו 10% שתנן-עמוד ג'ל (20 x 28 x 0.15 ס"מ3; שולחן 1) עם מסרק 8-היטב. באמצעות מזרק או pipet, ריקון בארות עם מאגר 0.5 x טריס/בוראט/EDTA (TBE).
הערה: ג'ל תוצרת בית לעיל מציעים רזולוציה טובה יותר להפריד את המוצר RT המורחבת מן הפריימר RT unextended. כחלופה, לקדם cast ג ' לים אוריאה-PAGE יכול לשמש גם (טבלת חומרים). כמו ג ' לים מראש להטיל לאפשר אמצעי אחסון מקסימום קטן יותר, כך דגימות יצטרך להיות מחולק בארות מרובות. יש להפעיל ג'לים לפני הטלת ג'ל ב-150-200 וולט. - Pre-הפעל את הג ב 35 W עבור 30 דקות. ריקון בארות שוב, לטעון דגימות ולהפעיל ג'ל ב 35 W עד ברומאופנול צבע כחול החזית הועברה כ 1 סנטימטר מסוף הג.
הערה: השתמש בכיור חום מתכת במהלך הפעלה מראש ואת ההפעלה הסופית כדי למנוע התחממות יתר ג'ל. - כתם ג'ל עבור 5 דקות ב-1x זהב גרעין חומצות ג'ל הכתם פתרון מוכן 0.5 x TBE. צבע זה הוא רגיש באור, אז להימנע מחשיפה לאור.
- התמונה ג'ל על סורק פלורסנט למטרות תיעוד באמצעות 520 ננומטר הריגוש ו 580 הפליטה ננומטר מסננים. אם סורק פלורסנט אינו זמין, השתמש בעוצמת אור כחולה. מוצר ה-RT אמור להופיע ככתם המתחיל מעל לתחל ההפעלה הבלתי מורחב (איור 4).
הערה: למרות הזהב גרעין חומצה ג'ל מכתים לצבוע בקלות מדמיין על רופא ג'ל עם מקור UV-light, זה חיוני לא לחשוף את המוצר RT יקר ל UV כדי למנוע נזק. - המחש את הג על אור כחול ממאיר את המוצר ואת מוצר ה-RT מהג. מומלץ לחתוך DNA עם הרחבות החל 30-200 nt (איור 4). מניחים את החתיכות ג'ל מחתחת על פני השטח נקי הבשר את הפרוסה לחתיכות קטנות כדי להגדיל את פני השטח. בזהירות להעביר לצינור 1.5 mL ולהוסיף 800 μL של מאגר הימנעות DNA (טבלה 1).
- דגירה חתיכות ג'ל עם ערבוב עדין עם מאגר הימנעות DNA על הלילה בטמפרטורת החדר.
- הפרד את מאגר להתחמק מן הג על ידי העברת שלווה דרך מסנן תאית אצטט טור (טבלה של חומרים) ממוקם בתוך צינור אוסף של 2 מ"ל.
- בצינור 1.5 mL, לשטוף 10 μL של חרוזים מגנטיים streptavidin עם 500 μL של streptavidin חרוז לשטוף מאגר (שולחן 1). חזור על שלוש שוטף מוחלט. . אל תתנו לחרוזים להתייבש השעיה מחדש חרוזים ב 10 μL של מאגר הימנעות DNA (טבלה 1).
- העבר את המאגר מופרדים מחתיכות ג'ל בשלב 14.8 לצינור המכיל את החרוזים שנשטפו streptavidin מגנטי.
- דגירה עם ערבוב עדין לפחות 8 h בטמפרטורת החדר. לכידת חרוזים על מגנט, להסיר supernatant מחדש חרוזים מגנטיים ב 10 μL of RNase-מים חינם והעברה לצינור 0.2 mL PCR.
15. סיקולריזציה של מוצר RT
- מוצרי RT שנתפסו על חרוזים streptavidin הם מעגליות בעוד מאוגדים על חרוזים. כדי לצפות את החרוז המגנטי, להוסיף 2.0 μl של מאגר התגובה 10x circularization, 1.0 μl של 1 מ"מ ATP, 1.0 μl של 50 mM mncl2, 4.0 μl של 5 M בחירה, 1.0 μl של ssdna ליגאז I (טבלה של חומרים), ו-1.0 μl של מים חינם.
- הפעילו את תגובת המעגל על הציקלניה התרמית ב-60 ° c למשך 4 שעות. חום להפעיל את ליגאז ההפעלה האני על ידי חימום ב 80 ° c עבור 10 דקות לאחר מכן להחזיק ב 4 ° c.
16. מבחן ה-PCR
- לפני שתמשיך ל-PCR בקנה מידה גדול, השתמש בחלק מהמוצר המעגלי כדי לקבוע את המספר האידיאלי של מחזורי הגברה עבור כל דוגמה. שלב זה מסייע למנוע הגברה והגבלה של חפצי ה-PCR כרכיבי התגובה של ה-PCR מגבילים במחזורי PCR גבוהים יותר.
- להכין תגובה 45 μL ה-PCR עם 4.0-6.0 μL של המוצר המעגלי משלב 15.2, 9.0 μL של מאגר התגובה 5x, 0.9 μL 10 M dNTPs, 2.25 μL של 10 μM PE 1.0 פריימר (לוח חומרים), 2.25 μl של 10 μm pe 2.0 פריימר (לוח חומרים) , ו-0.045 μL של דנ א באיכות גבוהה פולימראז (לוח חומרים), ומים.
- מערבבים היטב את התגובה ומתחלקים לתגובות 3 15 μL. כל אחת משלוש התגובות הללו תהיה כפופה למספר משתנה של מחזורי PCR. המספר האידיאלי של מחזורים צפוי להיות בין 7 ל -14. אז לבצע מבחן בדיקות עבור 8, 11, ו 14 מחזורים.
- השתמש בתנאי ה-PCR הבאים: 98 ° c-30 ס מ; 98 ° צ'-5 ס; 65 ° צ'-10 ס מ; 72 ° c-15 ס; 72 ° צ'-2 דקות; 12 ° c-החזיקו.
- הוסף 3 μL של 6x ג'ל טעינת צבע ולפתור ב 10% ג'ל מקורי העמוד עד חזית צבען כחול היגרו 3/4 של ג'ל. כתם ג'ל באמצעות זהב גרעין חומצה ג'ל כתם ותמונה כמו בשלבים 14.4 ו 14.5 (איור 5).
- כדי לבחור את המספר האידיאלי של מחזורי ה-PCR, השוו בין מוצרי PCR למספר הולך וגובר של מחזורים. בחר את מספר מחזור התשואות את הכמות הגדולה ביותר של המוצר של הגודל הצפוי ללא הגברה ממצאים (g., הכתם DNA גדול הרבה יותר מאשר צפוי מוצר, והיכן אין דלדול ניכר של pe של 1.0 ו-pe 2.0 התחל נראה (ראה החץ האדום ב איור 5).
17. PCR בקנה מידה גדול
- להכין תגובה 45 μL, כמו בשלב 16.2 ולחזור על ה-PCR. לפתור את ה-PCR על 10% מקורי העמוד ב 150 V, כתם עם 1x זהב גרעין חומצה ג'ל כתם ותמונה על מעבר אור כחול.
- בלו את מוצר ה-PCR מהג'ל והעבירו אותו למזרק של 3 מ ל. השתמש במזרק כדי לרסק את הג והבלטת לתוך צינור 1.5 mL.
הערה: מוצר RT בלתי מורחב על מעגלי התשואה PCR המוצר של 151 bp. כך, בשלב זה צריך לבחור גודל מוצרים גדולים מ 151 bp (איור 6). - הוסף 900 μL של מאגר הימנעות DNA ו דגירה בטמפרטורת החדר לילה עם ערבוב עדין.
- העבר את הג ג'ל לטור אצטט תאית לסנן ממוקם בתוך צינור האוסף 2 mL. ספין ב 12,000 x g עבור 3 דקות, איסוף supernatant לתוך צינור חדש.
- הוסף עוד 400 μL של מאגר הימנעות DNA לג המעוך והעבר לצינור 1.5 mL. מודטה עם ערבוב עדין עבור 4 שעות נוספות עבור הימנעות השנייה.
- הבריכה כל הפרשות ולפצל ל 3 צינורות עם 400 μL כל. מזרז DNA על ידי הוספת 1 מ ל של 100% אתנול ו 10 μg של הגליקוגן. וורטקס ו-"מערבולת" לפחות 2 h ב-20 ° c.
- ה-DNA גלולה ב 12,000 x g עבור 30 דקות ב 4 ° c. רוחצים את הגלולה DNA עם 70% אתנול.
- בזהירות להסיר את כל האתנול על ידי ליטוף ובמהירות להשעות את הגלולה DNA ב 20 μL של מים.
הערה: בשלב זה, חשוב לא לתת את הגלולה DNA להתייבש, כמו לייבש את ה-DNA החוצה יכול להכחיש. - השתמש בחלק קטן של דגימת דנ א כדי לקבוע את הגודל והריכוז של מוצר ה-PCR באמצעות פלואורומטר ורגישות גבוהה DNA bioanalyzer. ניתן לשלוח דגימות לרצף באחת מהפלטפורמות.
- קריאות רצף יכול להיות מעובד (למשל, הסרת מתאם, זמירה לשמור רצפים > 30 הציון Phred), מיושר הגנום התייחסות, ודמיינו בדפדפן כגון UCSC הגנום דפדפן (איור 7).
תוצאות
RIPiT מוצלח יביא את הimmunoprecipitation של שני החלבונים של הריבית ואת החלבונים אינטראקציה אחרים הידועים, ואת העדר חלבונים שאינם באינטראקציה. כפי שנראה באיור 3A, הן MAGOH ו EIF4AIII זוהו בשנת הללויה, אבל HNRNPA1 לא היה (ליין 6). במקביל, העקבות של RNA כי יש מטוהרים משותפת עם מכלולי RNP זוהה באמצעות האדיגרפיה האוטומטית (איור 3ב) או bioanalyzer (איור 3ג). הטיפול Puromycin צפוי להגדיל את תפוסת EJC ב-RNA, ואות חזקה יותר של ה-RNA שנצפתה ב-RIPiT הPuromycin שטופלו באיור 3ב (השוואת נתיבים 2 ו-3). יצירת דגימות לרצף עמוק דורש ליגגדירוג מתאם ל-RNA, ולאחר מכן להפוך את ה-RNA לתוך ה-DNA באמצעות פריימר כי מספח את רצף המתאם. הצעד ההפוך שעתוק משלבת נוקלאוטידים biotinylated, לטיהור של מוצר שעתוק הפוכה. בעקבות תמלול הפוכה, יש להפריד את המוצר מהמתאם הבלתי מורחב על ידי האוריאה-PAGE (איור 4). המוצר ההפוך התמלול הוא לאחר מכן מעגליות ו-PCR הוגדל. המספר המתאים של מחזורי ה-PCR אינו חייב להגביר את המוצר המעגלי. הגברה תגרום לדילול הדלדול ולמוצר ה-PCR החריג (ראה איור 5, ליין 4). מספר מחזורי עם הגברה הגדול ביותר ללא ראיות של הגברה מתאימה ביותר לשימוש עבור PCR בקנה מידה גדול (איור 5 ליין 3 ואיור 6).

איור 1 : סכמטי מתאר את השלבים העיקריים של RIPiT. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
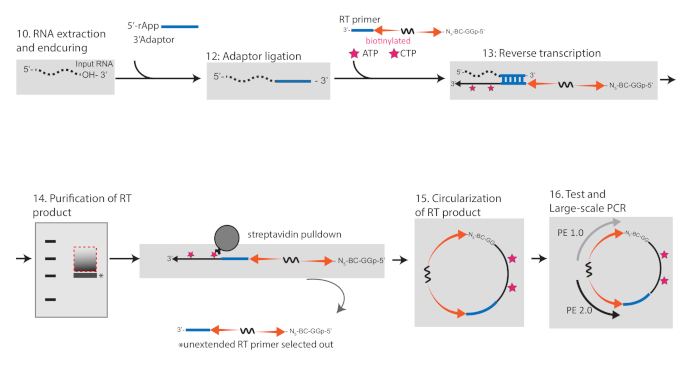
איור 2 : סכמטית המתאר את זרימת העבודה עבור המרה של RIPiT RNA לספריות עבור רצפי תפוקה גבוהה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
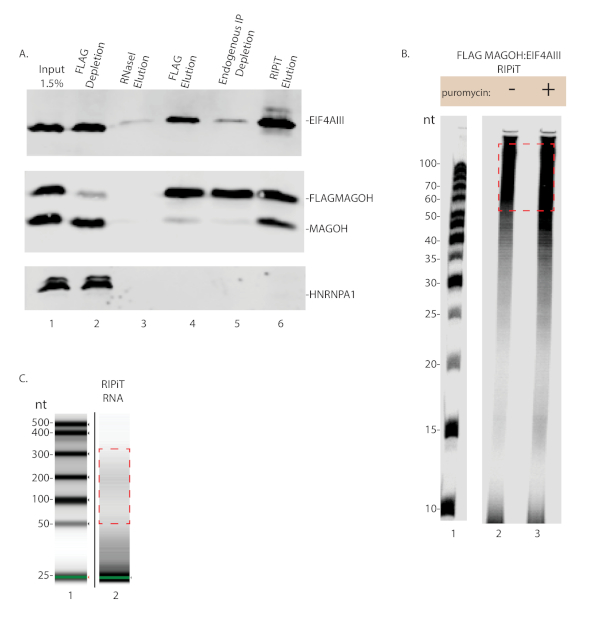
איור 3 : הערכה של RNA ותשואות חלבון מ RIPiT. (א) כתם מערבי של החלבונים מטוהרים מכל צעד מרכזי בהליך ripit. (ב) Autoradiograph תמונה של עקבות RNA מדגל-MAGOH: EIF4AIII ripit השוואת puromycin תאים מטופלים ולא מטופלים. התיבה האדומה מציינת את גודל טביעת הרגל של RNA שבסופו של דבר יומרו לספריות ברצף. (ג) פרופיל של RNA שבור מ ripit כמו במשעול 2 בלוח B כאשר דמיינו באמצעות bioanalyzer. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 4 : שעתוק הפוכה (RT) המוצר נפתרה על 10% אוריאה-עמוד ג ' ל ומוכתם עם זהב גרעין חומצות ג'ל כתם. התיבה האדומה מציינת את האזור ג'ל מגורש לטיהור ג'ל של מוצרי RT מורחבים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
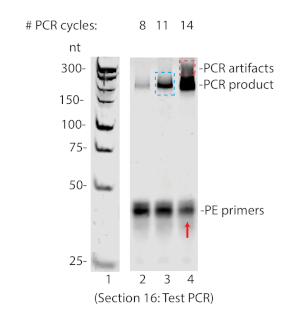
איור 5 : מבחן ה-PCR נפתר ב-8% שאינו מאתר הרפתקאות. שימו לב למוצרי ה-PCR הגדולים והמאובליים המופיעים ב -14 מחזורים (תיבה אדומה) ודילול מקביל של התחל (חץ אדום) המצביע על הגברה. עבור דוגמה זו, 11 מחזורים נבחרו עבור ה-PCR בקנה מידה גדול (קופסה כחולה). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
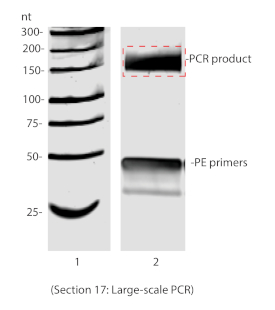
איור 6 : ה-PCR בקנה מידה גדול נפתר ב-8% שאינו מאתר הרפתקאות. התיבה האדומה מציינת שהג'ל. מיועד לטיהור ג'ל אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 7 : מסך הגנום דפדפן של הגן MAPK1 מראה התפלגות הדגל-MAGOH: EIF4AIII עקבות התוצאות הייצוגית של RIPiT. חיצים אדומים מציינות את אתרי איגוד ה-EJC הקנוניים הצפויים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
| PBS | 137 ממ י 2.7 ממ י 10 מ"מ Na2hpo4· 7h2O KH2פו4 7.4 pH | |
| מאגר קוצ'ינג | 2.5 M גליצין 2.5 mM-בסיס טריס | |
| מאגר לליזה היפוטוניקה (HLB) | 20 מ"מ טריס-HCl pH 7.5 15 ממ י 10 מילימטר EDTA 0.5% IGEPAL 0.1% טריטון-X-100 מיכל הכהן 1x לוקיטין * 1x Pepstatin * 1 מ"מ PMSF (פנילמתיל פלופקסיל) * | * יש להוסיף טרי בכל פעם |
| מאגר שטיפת איזוטוניקה (IsoWB) | 20 מ"מ טריס-HCl pH 7.5 150 ממ י 0.1% IGEPAL | |
| מאגר קוניוגציה | 0.02% פולסורbate-20 1x PBS | |
| נקה מאגר דוגמאות | 100 mM טריס-HCl pH 6.8 4% SDS 10 מילימטר EDTA | |
| 4xdNTPmix | 0.25 מ"מ 0.25 מ"מ ממדי Dטרומבוציטופניה 0.175 מילימטר משנת dATP 0.1625 מ"מ-dCTP 0.075 מ"מ ביוטין-דפ 0.0875 מ"מ ביוטין-dCTP | |
| מאגר הגדיל הראשון 5x w/o MgCl2 | 250 mM טריס-HCl pH 8 375 ממ י | |
| מבריק דילול (1 מ ל) | 1 מל א 5 מיקרומטר μL 200x BSA 20 μL 10% טריטון-X-100 20 μL 0.5 M EDTA מיכל הכהן 1x לוקיטין * 1x Pepstatin * 1 מ"מ מדגם * | * יש להוסיף טרי בכל פעם |
| 2x מאגר עומס של הצפת | 3 מ ג 5x TBE 1.8 g מסוג פיקדול 400 6.3 g שתנן 3 מ"ג ברומרופנול כחול 3 מ ג קסילן cyanol התאם נפח ל-15 mL ddH2O | כדי להיכנס לתמיסה, מניחים את צינור המים בגביע ומרתיחים על צלחת חמה במשך 10 – 15 דקות. הוספת צבעים לאחר כוונון עוצמת הקול ל-15 mL |
| שתנן-עמוד ג'ל | 6 M שתנן אקרילאמיד: ביסאקרילאמיד (40% [w/v]) לאחוז המתאים 0.5 x TBE 0.01% מטאד | |
| מאגר הימנעות DNA | 300 ממ י 1 מילימטר EDTA | |
| מאגר שטיפת Streptavidin חרוז | 0.5 מטר 20 מ"מ טריס-HCl pH 7.5 1 מילימטר EDTA |
טבלה 1: מאגרים.
Discussion
אנו דנים כאן כמה שיקולים מרכזיים כדי לבצע בהצלחה RIPiT. בראש ובראשונה, כתובות Ip בודדות חייבות להיות ממוטבות להשגת היעילות הגבוהה ביותר בכל שלב. כמות חרוזים הדגל העלה עבור מספר הקלט של תאים שתוארו כאן הוכיחה להיות חזקים עבור מגוון רחב של חלבונים שבדקנו. כמו רק חלק קטן של חלבונים שותפים הוא co-immunoprecipitated עם חלבון הדגל, כמות הנוגדן הדרוש עבור IP השני יעיל הוא בדרך כלל נמוך (פחות מ 10 μg). RIPiT בקנה מידה קטן (מ 1 10 ס מ) ואחריו אימות כתמי המערבי של חלבונים בכל שבר במהלך שני שלבים immunoprecipitation להוכיח מאוד שימושי כדי להעריך את היעילות ואת הספציפיות של ההליך לפני שדרוג למעלה. שניהם ממוקדות חלבונים, כמו גם כל החלבונים אחרים צפויים האינטראקציה במתחם יש לזהות את הימנעות. כמו כן, מועיל לקבוע את מגוון החלבונים המרוקנים (לא מאוגדים לחרוזי דגל-agarose או חרוזים מגנטיים) כדי להעריך את היעילות הimmunoprecipitation ואת אחוזי החלבונים הנכנסים לתוך קומפלקס. יתרה מזאת, ניתוח זה גם מודיע אם תנאי העיכול RNase מספיקים כדי להפריד בין אינטראקציות תלויות-RNA מאינטראקציות בלתי תלויות ב-RNA בתוך RNP. לכן, חשוב בדיוק לכלול שליטה שלילית, באופן אידיאלי RBP שאינו קשור ל-RBP של ריבית. לדוגמה, באיור 3A, HNRNPA1 מופיע בקלט אך אינו מזוהה ב משחררות. HNRNPA1 הוא RBP כי לא אינטראקציה ישירות עם EJC אבל אינטראקציה בעקיפין עם EJC כאשר EJC ו HNRNPA1 מאוגדים מולקולת RNA אותו. זיהוי של חלבון השליטה השלילית בתוך הימנעות מצביע על העניים RIPiT ספציפיות או מספיק RNA הדפסה footprinting במקרה כזה, עקבות ה-RNA שהושגו לא ישקפו לחלוטין את עקבות החלבון של הריבית. עקבות של גודל 50-200 nt מומלץ עבור הבאים RNA-Seq. משך הטיפול RNase I או כמות האנזים המשמש יכול להיות ממוטב כדי לקבל את הרגליים בגודל הרצוי. שים לב כי התרחיש הטוב ביותר יהיה להשיג אות טובה בטווח גודל הרצוי, וזה בלתי נמנע יש RNAs ארוך יותר וקצר אפילו בתנאים האופטימליים ביותר. RIPiT יכול לשמש גם כדי להשיג אתרים מחייבים של RBP אחד. במקרה כזה, אותו חלבון יכול להיות immunoprecipitated עם שני נוגדנים שונים, תחילה באמצעות נוגדן נגד תג זיקה ולאחר מכן עם נוגדנים נגד החלבון עצמו18. בסופו של דבר, RIPiT בקרה שלילית ניתן לבצע במקביל מתאים המבטאים את הדגל המתויג שליטה חלבון (למשל, חלבון פלורסנט ירוק) בשילוב עם נוגדן נגד חלבון נגד חלבון לא קשור ב-IP השני.
למרות יתרונותיה הרבים, חשוב לשקול מספר מגבלות של גישת RIPiT, ותרופות אפשריות. הדרישה של הימנעות אהדה לאחר הטיהור הראשון מחייבת את המקור הביולוגי לבטא חלבון מתויג. אם מערכת שילוב מחדש ספציפי לאתר אינה זמינה בקו התאים או באורגניזם העניין, תג אהדה קצר כגון תג דגל (8 חומצות אמינו) ניתן להציג במקום הגן האנדוסוגני באמצעות מבוסס CRISPR/Cas מבוססי הגנום לערוך גישה19. תג דגל הוא אפירופה אידיאלי עבור גישה זו, כי הנוגדן הדגל הוא מתאים היטב עבור משחרלי אהדה והוא יכול לעמוד בעוצמות יוניים גבוהים ותנאים מתונים מתון שניתן להשתמש בשילוב עם החוצה פורמלדהיד המקשר. הגבלה נוספת של הגישה RIPiT היא הדרישה לקלט גדול של חומר הסלולר. הדבר עלול להישאר בלתי נמנע במידה מסוימת כאחוז קטן של RBP שעשוי לקיים אינטראקציה עם חלבונים אחרים ב-RBP. עדיין משופר הכנת גישות הספרייה יכול לעזור להוריד את דרישת קלט גדול. רעיונות אפשריים כדי לייעל עוד יותר את השלבים הללו כוללים ביצוע של RNA 3 '-end dephosphorylation ומתאם לאחר החרוזים מגנטי מיד לאחר שטיפת IP השני ולפני האחרון להתחמק RNP. גישה זו מיושמת בהצלחה בשגרות הנוכחיות של CLIP-Seq ובווריאציה מתוארת לאחרונה של RIPiT20. שינויים כאלה יסיר גם מספר שלבים לזמן רב של רנ א משלבים מוקדמים של הליך הכנת הספרייה. עוד, בניגוד ל-CLIP, אשר מספק רזולוציה ברמת נוקלאוטיד של האתר המקשר של RBP ב-RNA, הרזולוציה של העקבות RIPiT יישארו ברמה של עשרות נוקלאוטידים. לבסוף, כאשר RNPs עשוי לכלול מספר Rnps, אתרי ה-RNA המועשרת של המלון כוללים שילוב של אתרים מחייבים של Rnps רבים. כאשר רצפי הסכמה המאוגדים על ידי rbps בודדים נחשפים בקצב הולך וגדל במהירות וכעת זמינים21,22,23, מידע זה יכול להיות ממונפת כדי לפרק את מגוון אתרי rbps מועשר ביציאות RIPiT. על אף האתגרים הללו, RIPiT-Seq הוא הליך יעיל ללכידת עקבות RNA של דינמי, הטרוגנית, ואפילו מתחמי RNP ארעי, אשר יכול לספק תובנות ייחודיות לתוך הפעולה הפנימית של RNA machineries כי השליטה הסלולר פונקציה.
Disclosures
. למחברים אין מה לגלות
Acknowledgements
עבודה זו נתמכת על ידי מענק NIH GM120209 (GS). המחברים מודים לליבה של משאבים משותפים גנומיקה של אוסוקCC עבור שירותיהם (מענק תמיכת CCC NCI P30 CA16058).
Materials
| Name | Company | Catalog Number | Comments |
| Anti-FLAG Affinity Gel | Sigma | A2220 | |
| ATP, [γ-32P]- 3,000 Ci/mmol 10 mCi/mL EasyTide, 250 µCi | PerkinElmer | BLU502A250UC | |
| BD Disposable Syringes with Luer-Lok Tips (200) | Fisher | 14-823-435 | |
| Betaine 5M | Sigma | B0300 | |
| biotin-dATP | TriLink | N-5002 | |
| biotin-dCTP | Perkin Elmer | NEL540001EA | |
| Branson Sonifier, Model SSE-1 | Branson | ||
| CircLigase I | VWR | 76081-606 | ssDNA ligase I |
| DMEM, High Glucose | ThermoFisher | 11995-065 | |
| DNA load buffer NEB | NEB | ||
| Dynabeads Protein A | LifeTech | 10002D | |
| Flp-In-T-REx 293 Cell Line | ThermoFisher | R78007 | |
| GeneRuler Low Range DNA Ladder | ThermoScientific | FERSM1203 | |
| Hygromycin B | ThermoFisher | 10687010 | |
| Mini-PROTEAN TBE Gel 10 well | Bio-Rad | 4565013 | |
| Mini-PROTEAN TBE-Urea Gel | Bio-Rad | 4566033 | |
| miRCAT-33 adapter 5′-TGGAATTCTCGGGTGCCAAGGddC-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Mirus transIT-X2 transfection reagent | Mirus | MIR 6004 | |
| Mth RNA ligase | NEB | E2610S | |
| PE1.0 5′-AATGATACGGCGACCACCGAGATCTACACT CTTTCCCTACACGACGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| PE2.0 5′-CAAGCAGAAGACGGCATACGAGATCGGTCTC GGCATTCCTGCTGAACCGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Phenol/Chloroform/Isoamyl Alcohol (25:24:1, pH 6.7, 100 mLl) | Fisher | BP1752I-100 | |
| Purple Gel Loading Dye (6x) | NEB | NEB #7025 | |
| Q5 DNA Polymerase | NEB | M0491S/L | |
| RNase I, E. coli, 1,000 U | Eppicenter | N6901K | |
| SPIN-X column | Corning | CLS8160-24EA | |
| Streptavidin beads | ThermoFisher | 60210 | |
| Superscript III (SSIII) | ThermoScientific | 18080044 | reverse transcriptase enzyme |
| SybrGold | ThermoFisher | S11494 | gold nucleic acid gel stain |
| T4 Polynucleotide Kinase-2500U | NEB | M0201L | |
| T4RNL2 Tr. K227Q | NEB | M0351S | |
| Tetracycline | Sigma | 87128 | |
| Thermostable 5´ App DNA/RNA Ligase | NEB | M0319S | |
| TruSeq_SE1 5′-pGGCACTANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE10 5′-pGGTGTTCNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE11 5′-pGGTAAGTNNNNNAGATCGGAA GAGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE12 5′-pGGAGATGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE2 5′-pGGGTAGCNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE35′-pGGTCGATNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE4 5′-pGGCCTCGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE5 5′-pGGTGACANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE6 5′-pGGTAGACNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE7 5′-pGGGCCCTNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCT TCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE8 5′-pGGATCGGNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTT CCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE9 5′-pGGACTGANNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Typhoon 5 Bimolecular Imager | GE Healthcare Life Science | 29187191 |
References
- Karousis, E. D., Nasif, S., Mühlemann, O. Nonsense-mediated mRNA decay: novel mechanistic insights and biological impact. Wiley Interdisciplinary Reviews: RNA. 7 (5), 661-682 (2016).
- Ivanov, P. V., Gehring, N. H., Kunz, J. B., Hentze, M. W., Kulozik, A. E. Interactions between UPF1, eRFs, PABP and the exon junction complex suggest an integrated model for mammalian NMD pathways. The EMBO Journal. 27 (5), 736-747 (2008).
- Papasaikas, P., Valcárcel, J. The Spliceosome: The Ultimate RNA Chaperone and Sculptor. Trends in Biochemical Sciences. 41 (1), 33-45 (2016).
- Jensen, K. B., Darnell, R. B. CLIP: Crosslinking and ImmunoPrecipitation of In Vivo RNA Targets of RNA-Binding Proteins. Methods in Molecular Biology. 488, Clifton, N.J. 85-98 (2008).
- Garzia, A., Morozov, P., Sajek, M., Meyer, C., Tuschl, T. PAR-CLIP for Discovering Target Sites of RNA-Binding Proteins. mRNA Decay: Methods and Protocols. 1720, 55-75 (2018).
- Konig, J., et al. iCLIP - Transcriptome-wide Mapping of Protein-RNA Interactions with Individual Nucleotide Resolution. Journal of Visualized Experiments. (50), e2638(2011).
- Sibley, C. R. Individual Nucleotide Resolution UV Cross-Linking and Immunoprecipitation (iCLIP) to Determine Protein-RNA Interactions. RNA Detection: Methods and Protocols. 1649, 427-454 (2018).
- Wheeler, E. C., Van Nostrand, E. L., Yeo, G. W. Advances and challenges in the detection of transcriptome‐wide protein-RNA interactions. Wiley Interdisciplinary Reviews: RNA. 9 (1), (2018).
- Singh, G., et al. The Cellular EJC Interactome Reveals Higher-Order mRNP Structure and an EJC-SR Protein Nexus. Cell. 151, 750-764 (2012).
- Singh, G., Ricci, E. P., Moore, M. J. RIPiT-Seq: A high-throughput approach for footprinting RNA:protein complexes. Methods. 65, 320-332 (2014).
- Mabin, J. W., et al. The Exon Junction Complex Undergoes a Compositional Switch that Alters mRNP Structure and Nonsense-Mediated mRNA Decay Activity. Cell Reports. 25 (9), 2431-2446 (2018).
- Gehring, N. H., Lamprinaki, S., Kulozik, A. E., Hentze, M. W. Disassembly of Exon Junction Complexes by PYM. Cell. 137 (3), 536-548 (2009).
- Dostie, J., Dreyfuss, G. Translation Is Required to Remove Y14 from mRNAs in the Cytoplasm. Current Biology. 12 (13), 1060-1067 (2002).
- Zünd, D., Gruber, A. R., Zavolan, M., Mühlemann, O. Translation-dependent displacement of UPF1 from coding sequences causes its enrichment in 3' UTRs. Nature Structural & Molecular Biology. 20 (8), 936-943 (2013).
- Gangras, P., Dayeh, D. M., Mabin, J. W., Nakanishi, K., Singh, G. Cloning and Identification of Recombinant Argonaute-Bound Small RNAs Using Next-Generation Sequencing. Argonaute Proteins: Methods and Protocols. 1680, 1-28 (2018).
- Heyer, E. E., Ozadam, H., Ricci, E. P., Cenik, C., Moore, M. J. An optimized kit-free method for making strand-specific deep sequencing libraries from RNA fragments. Nucleic Acids Research. 43 (1), 2(2015).
- Cameron, V., Uhlenbeck, O. C. 3'-Phosphatase activity in T4 polynucleotide kinase. Biochemistry. 16 (23), 5120-5126 (1977).
- Ricci, E. P., et al. Staufen1 senses overall transcript secondary structure to regulate translation. Nature Structural & Molecular Biology. 21 (1), 26-35 (2014).
- Lackner, D. H., et al. A generic strategy for CRISPR-Cas9-mediated gene tagging. Nature Communications. 6, 10237(2015).
- Metkar, M., et al. Higher-Order Organization Principles of Pre-translational mRNPs. Molecular Cell. 72 (4), 715-726 (2018).
- Giudice, G., Sánchez-Cabo, F., Torroja, C., Lara-Pezzi, E. ATtRACT-a database of RNA-binding proteins and associated motifs. Database: The Journal of Biological Databases and Curation. 2016, (2016).
- Paz, I., Kosti, I., Ares, M., Cline, M., Mandel-Gutfreund, Y. RBPmap: a web server for mapping binding sites of RNA-binding proteins. Nucleic Acids Research. 42, Web Server issue 361-367 (2014).
- Sundararaman, B., et al. Resources for the Comprehensive Discovery of Functional RNA Elements. Molecular Cell. 61 (6), 903-913 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved