Method Article
RNA의 발자국 의 식별:연속RNA 면역 침전을 통한 단백질 복합체 시퀀싱 (RIPiT-Seq)
요약
여기에서, 우리는 포유류 세포에서 내인성 RNA 결합 사이트 또는 RNA의 "발자국"을 풍부하게 하는 프로토콜을 제시합니다:단백질 (RNP) 복합체. 이 접근은 RNP 소단위의 2개의 면역 강절을 관련시키고 그러므로 나란히 RNA 면역 침전이라고 불립니다 (RIPiT).
초록
나란히 RNA 면역 침전 (RIPiT)는 RNA: 단백질 (RNP) 복합체 내의 단백질 쌍의 RNA 발자국을 풍부하게하는 방법입니다. RIPiT는 두 가지 정화 단계를 사용합니다. 먼저, 태그된 RNP 소단위의 면역침전은 경증 RNase 소화 및 후속 비-비-비-변성 친화성 용출이 뒤따른다. 다른 RNP 소단위의 두 번째 면역 침전은 정의된 복합체의 농축을 허용한다. RNA 및 단백질의 변성 용출 후, RNA 발자국은 고처리량 DNA 시퀀싱 라이브러리로 변환됩니다. RBP 결합 부위를 풍부하게 하기 위한 면역 침전(CLIP) 접근법에 이어 보다 대중적인 자외선(UV) 가교 와는 달리, RIPiT는 UV-가교 독립적이다. 따라서 RIPiT는 RNA 상호 작용에 존재하는 수많은 단백질에 적용될 수 있으며 그 이상으로 RNA 조절에 필수적이지만 RNA 또는 UV-크로스링크와 직접 접촉하지 않아 RNA에 제대로 접촉하지 않는다. RIPiT의 2가지 정제 단계는 관심 있는 단백질이 다른 보조 인자와 협력하여 작용하는 결합 부위를 식별하는 추가적인 이점을 제공합니다. 이중 정제 전략은 또한 배경을 제한하여 신호를 향상시키는 역할을합니다. 여기서는 RIPiT를 수행하고 격리된 RNA 발자국에서 고처리량 시퀀싱 라이브러리를 생성하는 단계별 절차를 제공합니다. 또한 RIPiT의 장점과 응용 프로그램에 대해 간략하게 설명하고 몇 가지 제한 사항에 대해 설명합니다.
서문
세포 내에서 RNA는 RNA를 형성하는 단백질과 복합체에 존재합니다: 단백질 복합체 (RNPs). RNP는 RNA 결합 단백질 (RBPs, RNA를 직접 결합하는 단백질)의 주위에 조립되고 또한 비 RBCPs (RBPs를 결합하지만 RNA를 결합하지 않는) 또한 포함하고, 종종 자연에서 동적입니다. RP와 그 보조 인자는 RP 내에서 총체적으로 기능하여 규제 기능을 실행합니다. 예를 들어, 말도 안되는 매개 mRNA 붕괴(NMD) 통로에서, UPF 단백질(UPF1, UPF2, 및 UPF3b)은 조기에 종단된 리보솜을 인식한다. 각각의 UPF 단백질은 RNA에 결합할 수 있지만, 활성 NMD 복합체가 형성되기 시작하는 것은 함께 조립할 때만 가능합니다. 이러한 복합체 내에서, UPF1은 비-RBP SMG1에 의한 인산화에 의해 더욱 활성화되고, 이러한 UPF1 활성화는 결국 mRNA 부패 유도 인자1,2의모집으로 이끈다. 이 예에서, RbP는 NMD를 트리거하는 RNP 복합체의 모집 및 활성화를 위한 비 RBP 공동 인자가 필요합니다. 그러나 RNP의 또 다른 재산은 그들의 조성 이질성이다. 별개의 E, A, B 또는 C 복합체에 존재하는 스플리세오좀을 고려하십시오. 다른 스플리세오좀 복합체는 중첩되고뚜렷한 단백질 3. RNP 기능을 연구하기 위하여는, 어떤 RNA가 RBP 및 그것의 관련단백질에 의해 구속되는지 밝히는 것이 중요합니다. 이를 달성하기 위해 많은 방법이 존재하며, 각 접근법은 4,5,6,7.
RBP 결합 부위를 식별하는 널리 널리 사용되는 방법 - 면역 침전 (CLIP) 및 다양한 변화에 이어 가교 - RNA8에RBP를 교차 연결하기 위해 자외선 (UV) 빛에 의존. 그러나, 이것은 RNA를 직접 접촉하지 않는 RNPs 내의 비 RP를 위한 효과적인 접근이 아닙니다. 여기서, 우리는 그들의 RNA 결합 사이트를 격리하고 확인하기 위하여, RbP 및 비 RP 에 모두 적용할 수 있는 대안 적인 접근을 기술합니다. 이러한 접근법은 RNA 면역침전과 탠덤(RIPiT)으로 구성되어 있으며, 이는 단일 정제에 비해 더 높은 특이성을 달성하는데 도움이 되는 두 가지 면역 침전 단계로 구성된다(도 1)9,10. 개별 면역 침전 (IP) 단계는 CLIP에 비해 더 낮은 순도에서 수행 될 수 있기 때문에, RIPiT는 면역 침전 동안 강한 세제의 존재를 견딜 수있는 항체의 가용성에 의존하지 않는다. RIPiT의 가장 독특한 장점은 두 가지 정제 단계에서 두 개의 서로 다른 단백질을 대상으로 하는 능력입니다. 이는 다른 유사한 복합체(11)로부터 조성적으로 구별되는RNP 복합체를 풍부하게 하는 강력한 방법을 제공한다.
RIPiT 절차에 작은 변화는 RNP 농축을 더욱 향상시킬 수 있습니다. 예를 들면, RNPs 내의 몇몇 RNA 단백질 또는 단백질 단백질 상호 작용은 일시적이고 그 같은 복합물의 발자국을 능등하게 정화하는 것은 어려울 지도 모릅니다. 이러한 상호 작용을 안정화하기 위해, RNP는 세포 포름 포름과 RIPiT 전에 세포 내에서 상호 연결 될 수있다. 예를 들어, 우리는 엑시온 접합 복합체(EJC) 코어 팩터, EIF4AIII 및 EJC 분해 인자 사이의 약한 상호작용이 관찰되었으며, PYM12는 포름알데히드 처리로 안정화될 수 있어 더 많은 RNA 발자국이 풍부해지는 것을 관찰했습니다(데이터는 를 표시합니다). 세포 수확 및 RIPiT 이전에, 세포는 또한 특정 상태에서 RNPs를 안정화또는 풍부하게 하는 약물로 치료될 수 있다. 예를 들어, 번역 중에 mRNA에서 제거되는 단백질을 연구할 때(예를 들어, EJC13,UPF114),푸로마이신, 사이클로헴시미드 또는 해링턴틴과 같은 번역 억제제로 치료하는 경우, RNA에 단백질.
RIPiT로부터 회수된 RNA의 양은 보통 낮다(0.5-10 페몰, 즉, 평균 RNA 길이를 고려한 10-250 ng RNA 75 nt). 이를 위한 주된 이유는 주어진 단백질의 극히 일부분만이 RNP 내의 다른 단백질과 복합체로 존재하기 때문입니다(첫 번째 단계에서 의 임의의 "자유" 단백질 IP'ed는 두 번째 IP 동안 손실됩니다). 이 RNA에서 RNA-Seq 라이브러리를 생성하기 위해, 우리는 또한 여기에 이러한 낮은 RNA 입력에 적합한 이전에 게시 된 프로토콜의 적응개요15,16 (그림2),이는 3에서 높은 처리량 시퀀싱 준비 샘플을 산출 일.
프로토콜
1. 테트라사이클린 유도성 플래그 태그 단백질을 발현하는 안정적인 HEK293 세포주 구축 (POI)
- 안정적으로 통합 된 Flp 재조합 대상 (FRT) 성장 매체에서 10 x 104 세포 / mL의 밀도를 가진 종자 HEK293 세포 (DMEM의 변형 된 독수리배지 [DMEM] + 10 % 태아 소 혈청 [FBS] + 1 % 페니실린 - 스트렙토마이신 [펜 / 연골]) 6- 잘 접시. 세포가 37°C 및 5% CO2에서 가습 된 인큐베이터에서 하룻밤 사이에 성장하도록 허용하십시오 (모든 후속 단계에 대한 표준 성장 조건).
- 다음날, 세포는 ~ 70 % 수렴해야합니다. 형질감염 시약 프로토콜에 따르면, FRT 부위를 함유하는 HEK293 세포를 9:1 의 pcDNA5-TETO-FLAG-POI:pOG44비율로 트랜스펙트한다.
참고: 플래그 태그는 순서 모티프 DYKDDDDK (D = 아스파르트산, Y = 티로신, 및 K = 리신)를 가지고 있습니다. - 24 시간 후 항생제 선택을 시작하십시오. 배지를 제거하고 100 μg/mL 의 흡습증으로 보충된 신선한 성장 매체를 추가합니다. 72 시간 이내, 전이되지 않은 세포는 정지하기 시작해야 합니다.
- 모든 48-72 시간, 성장 매체를 변경 하 고 신선한 hygromycin보충.
- 선택후 ~ 2 주, 안정적으로 형질 감염 된 세포의 개별 식민지가 나타나기 시작합니다. 식민지가 육안으로 보이면 접시에 트립신 1 mL을 넣고 37 °C에서 5 분 동안 배양하십시오. DMEM에서 세포를 다시 중단하고 세포를 새로운 10cm 플레이트로 옮기고, 100 μg/mL 의 흡습증으로 보충된 10 mL 의 신선한 성장 배지로 부피를 조절합니다. 플레이트가 ~ 80 % 합류에 도달하여 세포를 확장하여 영구 재고를 준비할 수 있습니다.
- 실험에 대한 최적 수준의 FLAG-POI 발현을 얻기 위해 필요한 테트라사이클린(Tet)의 양을 결정한다. 12 웰 형식으로 세포를 성장하고 16-24 h에 대한 0-1,000 ng / mL 범위 사이의 Tet의 적정을 수행합니다.
참고: ~60 kDa까지의 단백질의 경우, FLAG 태그및 단백질의 내인성 카피는 나트륨 도데실 설페이트-폴리아크릴아미드 겔 전기영동증(SDS-PAGE)에서 해결될 수 있어 FLAG-POI의 발현 수준을 내인성 대응물질과 비교한다. 더 큰 단백질의 경우 Tet 유도 샘플의 신호 강도를 유도되지 않은 샘플과 비교할 수 있습니다. 테트라사이클린 스톡 솔루션은 100% 에탄올에서 1 mg/mL로 제조할 수 있습니다. 세포 배양 작업을 위한 주식의 희석은 멸균수 또는 인산완충식염수(PBS)에 있어야 한다. 테트라 사이클린 주식은 매달 한 번 신선한 준비해야합니다.
2. 테트라 사이클린 유도 및 RIPiT 절차를위한 세포 배양
- 종자 안정적으로 15cm 플레이트에서 성장 매체에 3 x 105 세포 / mL의 밀도에 FLAG-POI HEK293을 통합. 세포가 37 °C 및 5% CO2에서 성장하도록 허용하십시오.
참고: 일반적으로, 3 내지 5 개의 15 cm 플레이트는 관심RNP의 풍부에 따라 RNA 발자국의 ~ 2-20 pmol을 산출할 것이다. RNP가 엄격한 조건하에서 포름알데히드가 가교되고 정제될 경우, 두 배의 입력이 필요할 수 있습니다. - 세포가 수확되기 전에 16-24시간 동안 FLAG-POI의 발현을 유도하기 위해 미리 결정된 최적 농도를 매체에 테트라사이클린을 첨가한다.
참고: 성장 매체를 변경할 필요는 없습니다. 세포는 파종 후 또는 접시가 ~ 80 % confluent 때 약 72 시간 수확 할 준비가되어 있어야합니다. 세포가 수렴되는 것을 피하는 것이 중요합니다.
3. 세포 수확, 포름알데히드 처리 및 세포 Lysis
- 각 15cm 플레이트에 대해 15 mL의 차가운 PBS로 단층 세포를 부드럽게 씻으십시오. 그런 다음 PBS의 30 mL에서 세포를 긁어 냅니다. 세포가 수확하기 전에 약물로 치료된 경우, 약물로 PBS를 보충하십시오. 모든 세포를 50 mL 원엽 튜브로 수집합니다.
참고: 약한 상호 작용을 보존하기 위해 세포는 포름알데히드와 상호 연결 될 수 있습니다 : 3.1 단계에서 세포 현탁액에 포름 알데히드를 추가하여 0.1 %의 최종 농도로 하고 10 분 동안 실온에서 로커에 배양하십시오. 담금질 버퍼의 3 mL을 추가하십시오 (표1) )를 추가로 5분간 바위로 바닷가에 대고 있습니다. - 4°C에서 10분 동안 400 x g에서 펠릿 세포를 폐기하고 상급체를 버린다.
- 얼음-차가운 저혈압 용해 완충제의 4 mL에 있는 용리 세포 (HLB; 표1). P1000 파이펫을 사용하여 셀을 다시 일시 중단합니다. 5 mL 튜브로 옮기고 10 분 동안 얼음에 용해를 배양하십시오.
- 얼음 욕조에 용해를 놓고 2 s 의 일시 정지와 1 s 펄스와 30 s에 대한 10 % 진폭에서 초음파. 이어서, 5 M NaCl의 108 μL을 첨가하여 염 농도를 150 mM으로 조절한다.
참고: 포름알데히드 가교 시료는 용해 완충액에서 각각 0.1% 및 나트륨 데옥시콜레이트를 포함시킴으로써 보다 엄격한 용해 및 정제를 받을 수 있다. - 4 °C에서 10 분 동안 21,000 x g에서 원심 분리에 의해 용해를 제거하십시오. 입력에서 단백질 수준의 서쪽 얼룩 분석을 위한 표지된 튜브에 20 μL 상판(세포 추출물)을 수집합니다(도 3A참조).
참고: 리세이드는 원심분리기에 있는 동안, FLAG 아가로즈 비드는 세척될 수 있다(단계 4.1 참조). FLAG 아가로즈 비드는 얼음-차가운 동위 원소 세척 완충제의 4 mL에서 3x 를 미리 세척해야 한다(IsoWB, 표1).
4. 플래그 면역 침전
- 5 mL 튜브에 3.5 ~ 750 μL 미리 세척 된 FLAG 아가로즈 비드 단계로부터 남은 상급제를 적용합니다 (세척 된 FLAG 아가로즈 비드의 침대 부피는 375 μL이 될 것입니다). 인큐베이트 FLAG 아가로즈 비드 및 세포 추출물을 4°C에서 1-3시간 동안 부드러운 혼합으로 배양하였다.
참고: 이 FLAG 아가로즈 비드의 부피는 광범위한 단백질에 충분해야 하지만, 필요한 경우 최적화될 수 있다. - 펠렛 FLAG 아가로즈 비즈는 4°C에서 1분 동안 400 x g에서 원심분리에 의해. 고갈된 세포 추출물에서 단백질 수준의 웨스턴 블롯 분석을 위해 표지된 튜브에 상판의 20 μL을 수집합니다(도 3A참조). 남은 상급물질은 버리십시오.
- FLAG 아가로즈 비즈를 씻으려면 IsoWB 4 mL를 추가하고 다시 일시 중단하십시오. 4 °C에서 1 분 동안 400 x g에서 펠렛 구슬. 조심스럽게 장식품을 제거하십시오. 4x반복합니다.
참고: 포름알데히드 가교 IP의 엄격한 세척을 위해, 0.1% SDS 및 나트륨 데옥시콜레이트각각은 처음 두 세척 단계에 대한 세척 버퍼에 포함될 수 있다.
5화 RNase I 소화
- RNase I를 IsoWB의 750 μL에서 0.002-0.01 단위/mL로 희석합니다(원하는 설치 공간 크기에 대한 적절한 농도는 경험적으로 결정되어야 합니다). IsoWB-RNase I를 넣고 FLAG 아가로즈 구슬을 세척하고 4°C에서 10분 동안 부드럽게 혼합하여 배양합니다.
- 4 °C에서 1 분 동안 400 x g에서 펠렛 구슬. 웨스턴 블롯 분석을 위해 라벨이 부착된 튜브에 20 μL 상판(RNase I 용출)을 수집합니다. IsoWB-RNase I를 버리고 단계 4.3에 설명된 바와 같이 이소WB와 함께 FLAG 아가로즈 비드 4x를 세척한다.
6. 선호도 용출
- 용출 완충제의 주식을 준비 (IsoWB에서 250 ng / mL에서 플래그 펩티드). 375 μL의 용출 완충제를 FLAG 아가로즈 비드에 적용하고 4°C에서 1-2시간 동안 부드럽게 흔들어 펠렛 FLAG 아가로즈 비드를 포지티브로 하여, FLAG IP에서 서양 단백질 블롯에 대한 용출의 15 μL aliquot를 수집한다(그림 3A참조).
참고: 친화성 용출이 진행되면 섹션 7을 수행할 수 있습니다.
7. 자기 비드 항체 컨쥬게이션
- 세척 50 μL 자기 비드 (즉, 다이나비드; 재료표) 1.5 mL 튜브에서 1 mL IsoWB에서 3 배. 100 μL의 컨쥬게이션 버퍼에서 자기 비드를 다시 일시 중단합니다. 항체의 적당한 양을 추가합니다 (IP를 위해 사용하는 항체의 정확한 양은 각 항체에 대해 경험적으로 결정될 필요가 있을 것이다).
참고: 단백질 A 자기 구슬은 토끼에서 생성된 항체에 최적이지만 단백질 G 자기 비드는 마우스 항체에 더 적합합니다. 자기 비드 호환성 차트는 각 항체에 적합한 비드를 선택하는 공급 업체 웹 사이트에서 사용할 수 있습니다. - 마그네틱 비드를 2x 컨쥬게이션 버퍼로 세척합니다(표 1). 적어도 10 분 동안 실온에서 혼합물을 배양. RIPiT 희석 완충제의 375 μL에서 자기 비드를 다시 중단. 다음 단계까지 얼음에 보관하십시오.
8. 두 번째 면역 침전
- 관심 있는 단백질에 대하여 항체에 결합된 자기 비드에 6.1 단계에서 남은 FLAG 친화성 용출을 적용한다. 1-2 시간 동안 4 °C에서 부드러운 혼합으로 인큐베이션. 자석에 자기 구슬을 포착하고 웨스턴 블롯을 통해 언바운드 단백질의 분석을 위해 15 μl의 상월체를 수집합니다. IsoWB 1 mL로 마그네틱 비즈 7x를 세척합니다.
9. 용출 거부
- 100 μL의 투명 샘플버퍼(표 1)를 마그네틱 비드에 넣고 P200 파이펫으로 다시 일시 중단합니다. 얼음에 10분 간 배양합니다.
- 자석에 자기 구슬을 포착하고 서쪽 얼룩을 통해 RIPiT 용출에서 단백질의 분석을 위해 용출의 15 μL을 수집합니다 (그림 3A참조). 남은 용출을 라벨이 붙은 새로운 1.5mL 튜브로 옮김을 옮김.
참고: 시료가 포름알데히드가 상호 연결된 경우, 시료는 가교를 역전시키기 위해 1시간 동안 65°C에서 배양되어야 합니다. - 다양한 단계 (입력, 플래그 IP 고갈, 플래그 IP, 두 번째 IP 고갈, 두 번째 IP 용출)에서 수집 된 샘플에 웨스턴 블롯을 수행합니다. 2개의 미끼 단백질에 대하여 항체를 가진 블롯, 그들의 다른 인터랙터는 공지된 경우, 및 적어도 하나의 비-음성 대조군으로서 상호작용하는 RBP(도3A).
10. RNA 추출 및 끝 경화
- RIPiT 용출에, RNase-free ddH2O의 320 μL, 페놀 클로로폼 이소아밀 알코올의 400 μL (PCIAA, pH 4.5), 및 30 s의 소용돌이를 추가하고 5 분 동안 12,000 x g에서 실온에서 회전. 아세테이트 나트륨 35 μL, 1 M MgCl2,글리코겐 10 μg, 100% 에탄올 1mL를 첨가합니다. -20 °C에서 하룻밤 배양.
- 펠렛 RNA에, 4 °C에서 30 분 동안 12,000 x g에서 원심 분리기. RNA를 70% 에탄올로 씻으소서.
- RNase I 분열 후 RNA에 남아 있는 3' 인산염을 제거하려면, RNase-free ddH2O의 17 μL에서 RNA 펠릿을 다시 중단하고, 10x T4 폴리뉴클레오티드 키나아제(PNK) 완충액(재료표) 및 T4 PNK의 1 μL을 추가합니다. 37°C에서 30분 동안 배양합니다.
참고: T4 PNK의 3'인산파타제 활성은 pH 617에서최적의 활성을 가집니다. PNK 반응 버퍼는 T4 PNK의 5' 키나아제 활성에 최적화되어 있으며 pH는 7.6입니다. 최종 경화 반응의 pH를 조정하는 동안, 이 단계는 더욱 최적화될 수 있다. - 튜브에 380 μL의 RNase-free ddH2O 및 PCIAA pH 4.5의 400 μL을 추가합니다. 30초 동안 의 소용돌이, 12,000 x g에서 원심분리기 5 분. 수성 상을 수집하고 35 μL의 나트륨 아세테이트, 1 M MgCl2의1 μL, 10 μg의 글리코겐, 100 % 에탄올의 1 mL을 추가합니다.
- -20 °C에서 하룻밤 배양. 위와 같이 70% 에탄올로 RNA를 세척합니다. RNase가 없는 물 4.5 μL에서 RNA를 다시 중단하십시오.
11. RNA 발자국 크기 및 풍부도 추정
-
성공적인 RIPiT는 1 pmol 이상의 RNA 단편을 산출할 것으로 예상됩니다. 실제 수율을 정량화하려면 RIPiT RNA 0.7 μL(총 수율의 ~1/6)을 새 튜브로 전송합니다. 10x T4 PNK 버퍼 2 μL, 1 mM ATP 1 μL, 40 μCi γ32P-ATP (스톡의 0.5-1.0 μL), T4 PNK의 1 μL을 추가합니다. 부피를 10 μL로 조정하고 37 °C에서 30 분 동안 배양하십시오.
- 병렬 PNK 반응에서, 크기 및 수량 표준을 사용하기 위해 합성 RNA 또는 DNA 올리고(20-40 nt)의 저범위 DNA 사다리 및 0.1 pmol을 라벨.
- 26% 우레아-PAGE 젤(20 x 27 x 0.45 mm3)에 표지된 RNA/DNA를 해결하십시오. 젤은 사전 실행하기 전에 35 W. 플러시 웰에서 30 분 동안 미리 실행되어야하며 샘플을로드하기 전에 브로모페놀 블루 염료 전면이 젤의 거의 끝에 도달 할 때까지 35 W에서 실행해야합니다.
- 유리 판에서 8 x 11 인치 필터 용지에 젤을 조심스럽게 제거하십시오. 종이 위에 젤을 얹고 젤 건조 장치에 놓고 플라스틱 랩으로 덮습니다. 80°C에서 드라이 젤을 진공으로 1시간 동안 건조시다.
- 말린 젤을 밤새 또는 적절한 신호가 감지될 때까지 인광스크린에 노출시킵니다. 이미지 인광 화면. RIPiT에서 좋은 품질의 RNA는 최소한의 눈에 띄는 밴드와 차선에 얼룩으로 나타납니다 (그림3B). RNA를 정량화하기 위해 RIPiT 레인에서 원하는 크기의 RNA 단편의 신호 강도를 표지된 합성 올리고의 0.1 pmol의 신호와 비교합니다.
참고: 대안적으로, RNA 발자국 크기 및 양은 고감도 바이오분석기를 사용하여 확인할 수 있다(도3C).
12. 어댑터 리그레이션
- RIPiT RNA를 준비하여 3.8 μL의 물에 용해되는 RNA의 적어도 3pmol을 준비한다.
- 0.2 mL 중합효소 연쇄 반응(PCR) 튜브에서, 3.8 μL의 RNA, 1 μL의 miR-CAT-33 프리-아데닐레이드 어댑터(7 μM)를 결합한다(재료표). 65°C에서 10분, 16°C에서 5분 동안 열사이클러상에 혼합물을 인큐베이션한 다음 4°C에서 유지합니다.
참고: 프리-아데닐레이트 링커는 올리고 합성 서비스에서 주문할 수 있거나, 임의의 올리고 합성 서비스에서 맞춤형 unadenylated DNA 올리고는 Mth RNA 리가제(TableofMaterials)와 겔 정제를 사용하여 아데니레이드될 수 있다. - 동일한 튜브에 1.5 μL의 10x T4 RNA 리가제 버퍼, 7.5 μL의 50% 폴리에틸렌 글리콜 8000 (PEG-8000), 0.75 μL 의 20 mM 디티오트라이톨 (DTT), 및 0.45 μL의 T4 RNL2 Tr. K27Q의 재료(표)를 추가합니다.
참고 : 50 % PEG-8000은 RNA 리가제와 완충액이 함께 제공됩니다. PEG-8000 용액은 점성이 있으며 천천히 파이펫을 만들어야 합니다. - 30°C에서 30°C에서 6시간 동안 배양반응을 하고, 열은 65°C에서 리가아를 10분 동안 불활성화한 다음 4°C에서 유지한다.
13. 역전사
- 12.4단계에서 결찰 혼합을 가진 튜브에, 일반 및 생체동화된 DNTP의 혼합을 포함하는 4x deoxynucleotide 트리포스페이트(dNTP) 혼합물의 11.25 μL을 추가합니다(표 1참조), 10 μM RT 프라이머 의 1.0 μL (재료표),그리고 6.8μl RNase 없는 물. 65°C에서 5분 동안 배양한 다음 4°C에서 유지합니다.
- 튜브를 얼음으로 옮기고 MgCl 2(표1),100 mM DTT의 2.25 μL, 1.2 μL의 역전사 효소를 최종 부피 45 μL(재료 표)에 넣지 않고 5x 제1 가닥(FS) 완충액9.0 μL을 첨가한다.
- 30-60 분 동안 55 °C에서 열 사이클러에 인큐베이션. 열은 15 분 동안 70 °C에서 역 전사체를 비활성화하고 4 °C에서 샘플을 유지합니다.
14. RT 제품의 정화
- RT 반응에 2x 우레아 부하완충제 (표 1)의 45 μL을 추가합니다. 45 μL에서 저범위 DNA 사다리 1 μg를 희석하고 2x 우레아 부하 완충액의 45 μL을 추가합니다.
- 준비 10% 우레아 페이지 젤 (20 x 28x 0.15 cm 3; 표1) 8 웰 빗. 주사기 또는 파이펫을 사용하여 0.5x Tris/붕산/EDTA(TBE) 버퍼로 우물을 세척합니다.
참고: 위의 홈메이드 젤은 확장된 RT 제품을 확장되지 않은 RT 프라이머와 분리하는 데 더 나은 해상도를 제공합니다. 대안으로, 프리캐스트 우레아-PAGE 겔도 사용할 수있다(재료표). 사전 캐스트 젤은 웰 당 작은 최대 볼륨을 허용하므로 샘플을 여러 웰로 나눌 필요가 있습니다. 프리 캐스트 젤은 150-200 V에서 실행되어야합니다. - 젤을 35W에서 30분 동안 미리 실행하고, 다시 세척하고, 샘플을 로드하고, 브로모페놀 블루 염료 프런트가 겔 말단에서 약 1인치로 이동될 때까지 35W에서 젤을 실행한다.
참고: 젤 과열을 방지하기 위해 사전 실행 및 최종 실행 중에 금속 방열판을 사용합니다. - 0.5x TBE로 제조된 1x 골드 핵산 겔 얼룩 용액에서 5분 동안 겔을 염색하였다. 이 염료는 빛에 민감하므로 빛에 노출되지 않도록하십시오.
- 520 nm 여기 및 580 nm 방출 필터를 사용하여 문서화 목적으로 형광 스캐너의 이미지 젤. 형광 스캐너를 사용할 수 없는 경우 파란색 광점 트랜지스터를 사용하십시오. RT 제품은 확장되지 않은 RT 프라이머 위에서 시작되는얼룩으로 나타나야 합니다(그림 4).
참고: 금 핵산 젤 염색 염료는 UV 광원이 있는 젤 문서에서 쉽게 시각화되지만, 손상을 방지하기 위해 귀중한 RT 제품을 UV에 노출시키지 않는 것이 중요합니다. - 청색 광 트랜지밀에 젤을 시각화하고 젤에서 RT 제품을 절제합니다. 30-200 nt에 이르는 확장으로 DNA를 절단하는 것이 좋습니다(그림4). 절제된 젤 조각을 깨끗한 표면에 놓고 슬라이스를 작은 조각으로 잘라 표면적을 높입니다. 조심스럽게 1.5 mL 튜브로 옮기고 DNA 용출 완충액 800 μL을 첨가합니다(표 1).
- 실온에서 밤에 DNA 용출 버퍼와 부드럽게 혼합하여 젤 조각을 배양합니다.
- 용출 완충액을 셀룰로오스 아세테이트 필터 컬럼(표)을 통해 슬러리를 통과시킴으로써 겔로부터 용출 완충액을 2 mL 수집 튜브에 배치하였다.
- 1.5 mL 튜브에서 스트렙타비딘 자기 비드 10 μL을 스트렙타비딘 비드 워시 버퍼 500 μL로 세척합니다(표 1). 총 세 번의 세차서를 반복합니다. 구슬이 마르지 않도록 하십시오. DNA 용출 완충제의 10 μL에서비드를 재중단한다(표 1).
- 14.8단계에서 겔 조각으로부터 분리된 용출 완충제를 세척된 스트렙타비딘 자기 비드를 함유하는 튜브로 이송한다.
- 실온에서 적어도 8 시간 동안 부드럽게 혼합하여 배양하십시오. 자석에 구슬을 포획하고, 상류물을 제거하고, RNase가 없는 물 10 μL에서 자기 구슬을 다시 일시 중단하고 0.2 mL PCR 튜브로 옮김.
15. RT 제품의 순환화
- 스트렙타비딘 비즈에 포착된 RT 제품은 구슬에 묶여 있는 동안 원형으로 되어 있습니다. 마그네틱 비드 슬러리에, 10x 원형 반응 완충제의 2.0 μL, 1 mM ATP의 1.0 μL, 50 mM MnCl2의1.0 μL, 5 M 베타인의 4.0 μL, ssDNA 리가제 I (재료의표)및 RNase 물 1.0 μL을 추가합니다.
- 60°C에서 4시간 동안 열 사이클러상에서 원형 반응을 배양한 후 4°C에서 10분 동안 80°C에서 가열하여 ssDNA 리가제 I를 불활성화한 다음 4°C에서 유지한다.
16. 테스트 PCR
- 대규모 PCR로 진행하기 전에 순환화된 제품의 일부를 사용하여 각 샘플에 대한 이상적인 증폭 주기 수를 결정합니다. 이 단계는 PCR 반응 구성 요소가 더 높은 PCR 주기에서 제한됨에 따라 과다 증폭을 방지하고 PCR 아티팩트를 제한하는 데 도움이 됩니다.
- 45 μL PCR 반응을 15.2단계, 5x 반응 완충제의 9.0 μL, 0.9 μL 10 μL, 2.25 μL 의 10μM PE1.0 프라이머(재료표),2.25 μL 의 10μM PE2.0 프라이머(10 μM PE2.0 primer) , 고충실도 DNA 폴리머라제(물표) 및물의 0.045 μL.
- 반응을 잘 혼합하고 3 개의 15 μL 반응으로 나눕습니다. 이들 세 가지 반응은 각각 PCR 사이클의 가변적인 수의 영향을 받게 될 것이다. 사이클의 이상적인 수는 7과 14 사이가 될 것으로 예상된다. 따라서 8, 11 및 14주기 동안 테스트 PcR을 수행합니다.
- 다음과 같은 PCR 조건을 사용 : 98 ° C - 30 s; 98°C - 5s; 65 °C - 10s; 72°C - 15s; 72 °C - 2 분; 12 °C - 보류.
- 6x 젤 로딩 염료 3 μL을 넣고 청색 염료 프런트가 젤의 3/4로 이동될 때까지 10% 네이티브 PAGE 젤에 바르세요. 14.4 단계 및 14.5 단계와 같이 금 핵산 젤 얼룩및 이미지를 사용하여 겔을 염색 (도 5).
- PCR 사이클의 이상적인 수를 선택하려면 PCR 제품을 증가하는 사이클 수에서 비교하십시오. 과암화 아티팩트 없이 예상 크기의 가장 많은 양의 제품을 산출하는 사이클 번호를 선택하십시오(예: 예상 제품보다 훨씬 큰 DNA 얼룩, PE1.0 및 PE2.0 프라이머의 상당한 고갈이 보이지 않는 경우(빨간색 화살표 참조) 그림5).
17. 대규모 PCR
- 단계 16.2에서와 같이 45 μL PCR 반응을 준비하고 PCR을 반복한다. 150V에서 10% 네이티브 PAGE에서 PCR을 해결하고, 청색광 트랜지미너레이터에 1x 금 핵산 젤 얼룩과 이미지로 얼룩을 가집니다.
- 젤에서 PCR 제품을 절제하고 3 mL 주사기로 옮김을 전달합니다. 주사기를 사용하여 젤을 분쇄하고 1.5 mL 튜브로 돌출하십시오.
참고: 순환시 확장되지 않은 RT 제품은 151bp의 PCR 제품을 산출합니다. 따라서, 이 단계에서 는 151 bp보다 큰 제품을선택해야 한다(도 6). - 900 μL의 DNA 용출 버퍼를 추가하고 부드러운 혼합으로 밤새 실온에서 배양하십시오.
- 겔 슬러리를 2 mL 콜렉션 튜브에 배치된 셀룰로오스 아세테이트 필터 컬럼으로 옮김. 12,000 x g에서 3분 간 회전하여 신선한 튜브에 상류를 수집합니다.
- 분쇄된 겔에 400 μL의 DNA 용출 버퍼를 추가하고 1.5 mL 튜브로 옮김을 전달한다. 두 번째 용출을 위해 추가로 4 시간 동안 부드러운 혼합으로 인큐베이팅하십시오.
- 모든 용출을 풀하고 각각 400 μL로 3 개의 튜브로 나눕습니다. 100% 에탄올 1mL및 글리코겐 10 μg를 첨가하여 DNA를 침전시한다. 소용돌이 및 -20°C에서 최소 2시간 배양한다.
- 펠릿 DNA는 4°C에서 30분 동안 12,000 x g에서. DNA 펠릿을 70% 에탄올로 씻으십시오.
- 파이펫팅으로 모든 에탄올을 조심스럽게 제거하고 20 μL의 물에 DNA 펠릿을 신속하게 재중단하십시오.
참고: 이 단계에서 DNA 펠릿을 건조시키면 DNA가 변성할 수 있으므로 건조하지 않는 것이 중요합니다. - 형광계와 고감도 DNA 바이오 분석기를 통해 PCR 제품의 크기와 농도를 결정하기 위해 DNA 샘플의 작은 부분을 사용합니다. 이제 플랫폼 중 하나에서 시퀀싱을 위해 샘플을 제출할 수 있습니다.
- 서열화된 판독은 처리될 수 있다(예를 들어, 어댑터 제거, 트리밍하여 서열을 유지>30 Phred 점수), 참조 게놈에 정렬하고, UCSC 게놈 브라우저와 같은 브라우저상에서 시각화될 수 있다(그림 7).
결과
성공적인 RIPiT는 관심 있는 단백질과 다른 알려진 상호 작용 단백질의 면역 침전, 비 상호 작용하는 단백질의 부재를 초래할 것이다. 도 3A에서볼 수 있듯이, 마고 및 EIF4AIII 모두 RIPiT 용출에서 검출되었지만 HNRNPA1은 아니었다(레인 6). 병렬로, RNP 복합체와 공존한 RNA 발자국은 자가방사선(도3B) 또는 생체분석기(도3C)를 통해 검출되었다. 푸로마이신 치료는 RNA에 대한 EJC 점유율을 증가시킬 것으로 예상되며, 도 3B에서 푸로마이신 처리된 RIPiT에서 더 강한 RNA 발자국 신호가 관찰되었다(비교 레인 2 및 3). 깊은 시퀀싱을 위한 견본을 생성하는 것은 RNA에 어댑터를 결찰하는 것을 요구하고, 그 때 어댑터 순서에 어닐링하는 프라이머를 사용하여 DNA로 RNA를 전사하는 역. 역전사 단계는 역전사 생성물의 정제를 위해 생체순염 뉴클레오티드를 통합한다. 역전사 후, 생성물은 우레아-PAGE에 의해 확장되지 않은 어댑터로부터 분리되어야 한다(그림 4). 역전사 생성물은 순환화되고 PCR 증폭된다. 적절한 수의 PCR 사이클이 순환된 제품을 과도하게 증폭해서는 안 됩니다. 과암화는 프라이머 고갈 및 비정상적인 PCR 제품을 초래할 것이다(그림 5, 레인 4 참조). 과암화의 증거없이 가장 큰 증폭을 가진 사이클의 수는 대규모 PCR에 사용하기에 가장 적합하다(도5 레인 3 및 도6).

그림 1 : RIPiT의 주요 단계를 설명하는 도식. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
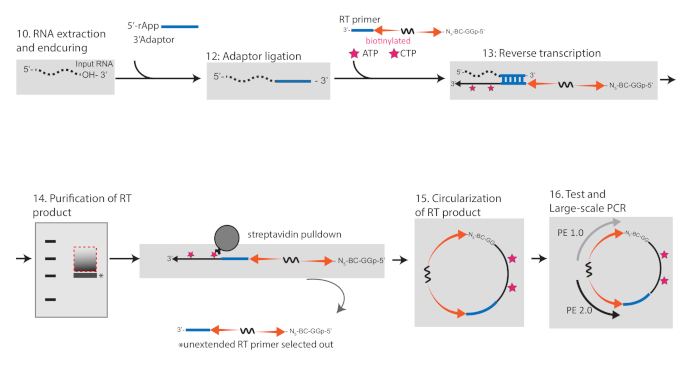
그림 2 : RIPiT RNA를 라이브러리로 변환하여 처리량이 높은 시퀀싱을 위한 워크플로우를 묘사한 회로도입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
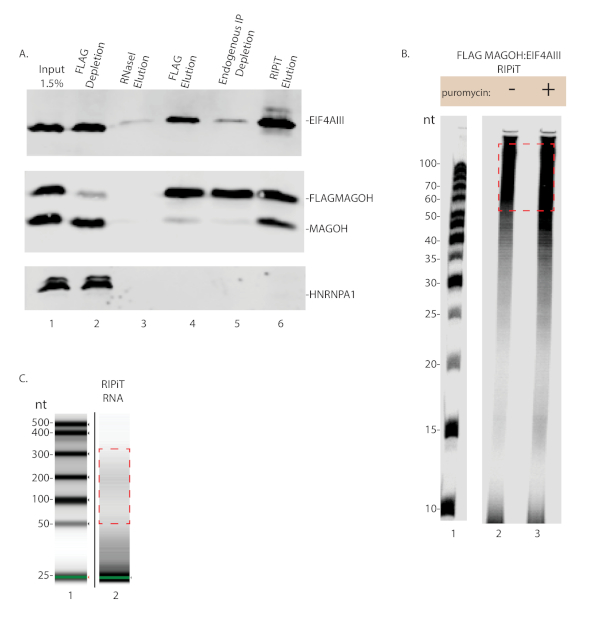
그림 3 : RIPiT의 RNA 및 단백질 수율 추정. (A) RIPiT 절차의 각 주요 단계에서 정제된 단백질의 서양 얼룩. (B) FLAG-MAGOH:EIF4AIII RIPiT에서 RNA 발자국의 자가사진상 은 치료된 푸로마이신과 치료되지 않은 세포를 비교하였다. 빨간색 상자는 RNA 발자국 크기를 나타내며 궁극적으로 시퀀싱 라이브러리로 변환됩니다. (C) 바이오분석기를 사용하여 시각화할 때 패널 B에서 2레인에서와 같이 RIPiT로부터 용출된 RNA의 프로파일. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4 : 역전사(RT) 생성물은 10% 우레아-PAGE 젤로 결단되고 금핵산 젤 얼룩으로 염색되었습니다. 레드 박스는 확장 된 RT 제품의 겔 정제를 위해 절제 된 겔 영역을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
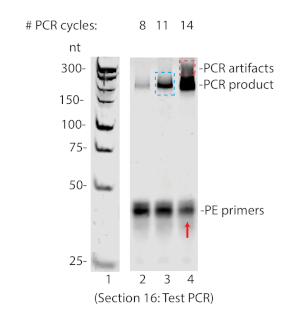
그림 5 : 테스트 PCR은 8% 비변성 페이지에서 해결되었습니다. 14 사이클 (빨간 상자)에 나타나는 비정상적인 대형 PCR 제품및 과암화를 나타내는 프라이머 (빨간색 화살표)의 병렬 고갈을 확인하십시오. 이 샘플의 경우, 대규모 PCR(블루 박스)에 대해 11사이클이 선택되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
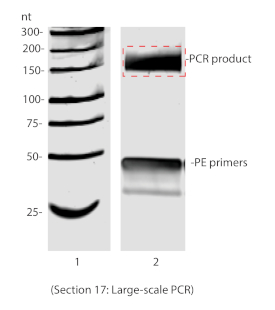
그림 6 : 8% 비변성 페이지에서 대규모 PCR 이내로 해결되었습니다. 적색 상자는 겔 정제를 위해 절제된 겔 조각을 나타낸다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 7 : MAPK1 유전자의 유전체 브라우저 스크린샷으로 FLAG-MAGOH:EIF4AIII 발자국의 분포를 RIPiT의 대표적인 결과로 보여줍니다. 빨간색 화살표는 예상되는 표준 EJC 바인딩 사이트를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| Pbs | 137 mM 나클 2.7 mM KCl 10 mM 나2HPO4·7H2O KH2PO4 pH 7.4 | |
| 담금질 버퍼 | 2.5 M 글리신 2.5 mM 트리스 베이스 | |
| 저자극성 리시스 버퍼 (HLB) | 20 mM 트리스-HCl pH 7.5 15 mM 나클 10 mM EDTA 0.5% IGEPAL 0.1% 트리톤-X-100 1x 아프로티닌* 1x 류페프틴* 1x 펩스타틴* 1 mM PMSF (페닐메틸설포닐 불소)* | * 신선한 모든 시간을 추가해야합니다 |
| 등위 류 세척 버퍼 (IsoWB) | 20 mM 트리스-HCl pH 7.5 150 mM 나클 0.1% IGEPAL | |
| 컨쥬게이션 버퍼 | 0.02% 폴리소르베이트-20 1x PBS | |
| 샘플 버퍼 지우기 | 100 mM 트리스-HCl pH 6.8 4% SDS 10 mM EDTA | |
| 4xdNT믹스 | 0.25 mM dGTP 0.25 mM dTTP 0.175 mM dATP 0.1625 mM dCTP 0.075 mM 비오틴 dATP 0.0875 mM 비오틴-dCTP | |
| MgCl 2를 사용하는 5x 첫 번째 가닥 버퍼 | 250 mM 트리스-HCl pH 8 375 mM KCl | |
| RIPiT 희석 버퍼(1mL) | 1 mL IsoWB 5 μL 200x BSA 20 μL 10% 트리톤-X-100 20 μL 0.5M EDTA 1x 아프로티닌* 1x 류페프틴* 1x 펩스타틴* 1 mM PMSF* | * 신선한 모든 시간을 추가해야합니다 |
| 2x 변성 부하 버퍼 | 3 mL 5x TBE 피콜 타입 400 g 1.8 g 우레아 6.3 g 브로모페놀 블루 3 mg 자일렌 시안올 3 mg 볼륨을 15mL ddH 2O로 조정 | 용액에 들어가려면 튜브를 비커에 넣고 뜨거운 접시에 10-15 분 동안 끓입니다. |
| 우레아 페이지 젤 | 6 M 우레아 아크릴아미드:비사크릴라미드(40% [w/v]) 0.5x TBE 0.01% TEMED | |
| DNA 용출 버퍼 | 300 mM 나클 1 mM EDTA | |
| 스트렙타비딘 비드 워시 버퍼 | 0.5 M NaOH 20 mM 트리스-HCl pH 7.5 1 mM EDTA |
표 1: 버퍼.
토론
여기에서 RIPiT를 성공적으로 수행하기 위한 몇 가지 주요 고려 사항에 대해 설명합니다. 무엇보다도 개별 IP는 각 단계에서 가능한 최고의 효율성을 달성하기 위해 최적화되어야 합니다. 여기에 기술된 세포의 입력 수에 대한 FLAG 아가로즈 비드의 양은 우리가 시험한 광범위한 단백질에 대해 견고하다는 것이 입증되었습니다. 파트너 단백질의 극히 일부만이 FLAG 단백질과 공존하기 때문에, 효율적인 제2 IP에 필요한 항체의 양은 보통 낮다(10 μg 미만). 2개의 면역 침전 단계 도중 각 분획에 있는 단백질의 작은 규모 RIPiT (1개의 10 cm 플레이트에서) 뒤에 는 확장하기 전에 절차의 효율성 그리고 특이성을 평가하기 위하여 극단적으로 유용하다는 것을 증명합니다. 둘 다 표적으로 한 단백질 뿐만 아니라 복합체에 있는 그밖 예상되는 상호 작용하는 단백질은 용출에서 검출되어야 합니다. 또한 고갈된 용해물(FLAG-agarose 또는 자기 비드에 구속되지 않은)에서 단백질을 분석하여 면역 침전 효율 및 복합체로 조립되는 단백질의 백분율을 양호한 추정치로 추정하는 것이 유익하다. 추가, 이 분석은 또한 RNase 소화 조건이 RNP 내의 RNA 독립적인 상호 작용에서 RNA 의존하는 상호 작용을 분리하기에 충분한지 알려줍니다. 따라서, 음의 대조군, 이상적으로 관심 있는 RNP와 관련이 없는 RBP를 포함하는 것이 중요하다. 예를 들어, 도 3A에서 HNRNPA1은 입력에 존재하지만 RIPiT 용출에서 검출되지 않는다. HNRNPA1은 EJC와 직접 상호 작용하지 않지만 EJC와 HNRNPA1이 동일한 RNA 분자에 결합될 때 EJC와 간접적으로 상호 작용하는 RBP입니다. 용출에서 음성 대조단백질의 검출은 불량한 RIPiT 특이성 또는 불충분한 RNA 발자국을 나타낸다. 이러한 경우, 얻어진 RNA 발자국은 관심 있는 단백질의 발자국을 완전히 반영하지 않을 것이다. 50-200 nt 크기의 발자국은 후속 RNA-Seq. RNase I 치료의 지속 기간 또는 사용되는 효소의 양이 원하는 크기 발자국을 얻기 위해 최적화 될 수 있습니다. 최상의 시나리오는 원하는 크기 범위에서 양호한 신호를 얻는 것이며 가장 최적의 조건에서도 RNA가 더 길고 짧아지는 것이 불가피합니다. RIPiT는 또한 단일 RBP의 결합 부위를 획득하는데 사용될 수 있다. 이러한 경우, 동일한 단백질은 두 개의 상이한 항체로 면역침전될 수 있으며, 먼저 친화성 태그에 대하여 항체를 사용한 다음 단백질 자체에 대한항체(18)를사용한다. 마지막으로, 음성 대조군 RIPiT는 제2 IP에서 관련없는 단백질에 대하여 항체와 결합하여 FLAG 태그가 붙은 대조군 단백질(예를 들어, 녹색 형광 단백질)을 발현하는 세포로부터 병렬로 수행될 수 있다.
많은 장점에도 불구하고 RIPiT 접근 방식의 몇 가지 제한 사항과 가능한 해결책을 고려하는 것이 중요합니다. 첫 번째 정제 후 친화도 용출의 요구 사항은 태그가 지정된 단백질을 발현하기 위해 생물학적 공급원을 필요로 한다. 사이트 특이적 재조합 시스템이 관심 있는 세포주 또는 유기체에서 사용할 수 없는 경우, CRISPR/Cas 기반 게놈 편집 접근법19를사용하여 내인성 유전자 궤적에서 FLAG 태그(8 아미노산)와 같은 짧은 친화도 태그가 도입될 수 있다. FLAG 태그는 FLAG 항체가 친화도 용출에 적합하고 포름알데히드 가교와 함께 사용할 수있는 높은 이온 강도 및 가벼운 변성 조건을 견딜 수 있기 때문에 이러한 접근법에 이상적인 에피토프입니다. RIPiT 접근법의 또 다른 한계는 셀룰러 물질의 큰 입력에 대한 요구 사항입니다. 이것은 RBP의 단지 작은 백분율가능성이 RNP에 있는 그밖 단백질과 상호 작용하기 때문에 어느 정도 피할 수 없는 남아 있을 수 있습니다. 여전히 개선된 라이브러리 준비 접근 방식은 큰 입력 요구 사항을 낮추는 데 도움이 될 수 있습니다. 이러한 단계를 더욱 간소화하기 위한 가능한 아이디어는 두 번째 IP 세차 직후 및 RNP의 최종 용출 전에 자기 비드상에 RNA 3'-end 탈포스포릴화 및 어댑터 결찰을 수행하는 것을 포함한다. 이러한 접근법은 현재 CLIP-Seq 절차 및 최근 설명된 RIPiT20의변형에서 성공적으로 구현된다. 이러한 변화는 또한 라이브러리 준비 절차의 초기 단계에서 몇 가지 시간 소모적 RNA 정제 단계를 제거할 것이다. 또한, RNA에 RBP의 가교 부위의 뉴클레오티드 수준 분해능을 제공하는 CLIP과 달리 RIPiT 발자국의 분해능은 수십 개의 뉴클레오티드 수준으로 유지됩니다. 마지막으로, RNP는 다중 RP를 포함할 수 있기 때문에, RIPiT 농축 RNA 부위는 많은 RP의 결합 부위의 혼합물을 포함한다. 개별 RP에 의해 구속 된 합의 시퀀스가 빠르게 증가하는 속도로 발견되고 있으며 이제21,22,23을쉽게 사용할 수 있게됨에 따라이 정보는 RBP 사이트의 구색을 제거하는 데 활용 될 수 있습니다. RIPiT 출력이 풍부합니다. 이러한 과제에도 불구하고, RIPiT-Seq는 동적, 이기종, 심지어 일시적인 RNP 복합체의 RNA 발자국을 포착하는 효과적인 절차로, 이는 세포를 제어하는 RNA 기계의 내부 작동에 대한 독특한 통찰력을 제공할 수 있습니다. 함수.
공개
저자는 공개 할 것이 없다.
감사의 말
이 작품은 NIH 보조금 GM120209 (GS)에 의해 지원되었다. 저자는 OSUCCC 유전체학 공유 자원 코어의 서비스에 대해 감사드립니다 (CCC 지원 보조금 NCI P30 CA16058).
자료
| Name | Company | Catalog Number | Comments |
| Anti-FLAG Affinity Gel | Sigma | A2220 | |
| ATP, [γ-32P]- 3,000 Ci/mmol 10 mCi/mL EasyTide, 250 µCi | PerkinElmer | BLU502A250UC | |
| BD Disposable Syringes with Luer-Lok Tips (200) | Fisher | 14-823-435 | |
| Betaine 5M | Sigma | B0300 | |
| biotin-dATP | TriLink | N-5002 | |
| biotin-dCTP | Perkin Elmer | NEL540001EA | |
| Branson Sonifier, Model SSE-1 | Branson | ||
| CircLigase I | VWR | 76081-606 | ssDNA ligase I |
| DMEM, High Glucose | ThermoFisher | 11995-065 | |
| DNA load buffer NEB | NEB | ||
| Dynabeads Protein A | LifeTech | 10002D | |
| Flp-In-T-REx 293 Cell Line | ThermoFisher | R78007 | |
| GeneRuler Low Range DNA Ladder | ThermoScientific | FERSM1203 | |
| Hygromycin B | ThermoFisher | 10687010 | |
| Mini-PROTEAN TBE Gel 10 well | Bio-Rad | 4565013 | |
| Mini-PROTEAN TBE-Urea Gel | Bio-Rad | 4566033 | |
| miRCAT-33 adapter 5′-TGGAATTCTCGGGTGCCAAGGddC-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Mirus transIT-X2 transfection reagent | Mirus | MIR 6004 | |
| Mth RNA ligase | NEB | E2610S | |
| PE1.0 5′-AATGATACGGCGACCACCGAGATCTACACT CTTTCCCTACACGACGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| PE2.0 5′-CAAGCAGAAGACGGCATACGAGATCGGTCTC GGCATTCCTGCTGAACCGCTCTTCCGATC*T-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Phenol/Chloroform/Isoamyl Alcohol (25:24:1, pH 6.7, 100 mLl) | Fisher | BP1752I-100 | |
| Purple Gel Loading Dye (6x) | NEB | NEB #7025 | |
| Q5 DNA Polymerase | NEB | M0491S/L | |
| RNase I, E. coli, 1,000 U | Eppicenter | N6901K | |
| SPIN-X column | Corning | CLS8160-24EA | |
| Streptavidin beads | ThermoFisher | 60210 | |
| Superscript III (SSIII) | ThermoScientific | 18080044 | reverse transcriptase enzyme |
| SybrGold | ThermoFisher | S11494 | gold nucleic acid gel stain |
| T4 Polynucleotide Kinase-2500U | NEB | M0201L | |
| T4RNL2 Tr. K227Q | NEB | M0351S | |
| Tetracycline | Sigma | 87128 | |
| Thermostable 5´ App DNA/RNA Ligase | NEB | M0319S | |
| TruSeq_SE1 5′-pGGCACTANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE10 5′-pGGTGTTCNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE11 5′-pGGTAAGTNNNNNAGATCGGAA GAGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE12 5′-pGGAGATGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE2 5′-pGGGTAGCNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE35′-pGGTCGATNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCT CTTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE4 5′-pGGCCTCGNNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE5 5′-pGGTGACANNNNNAGATCGGAAGA GCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTC TTCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE6 5′-pGGTAGACNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE7 5′-pGGGCCCTNNNNNAGATCGGAAG AGCGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCT TCCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE8 5′-pGGATCGGNNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTT CCGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| TruSeq_SE9 5′-pGGACTGANNNNNAGATCGGAAGAG CGTCGTGTAGGGAAAGAGTGT-SPACER 18-CTCGGCATTCCTGCTGAACCGCTCTTC CGATCTCCTTGGCACCCGAGAATTCCA-3′ | Any | this protocol is only compatible with the Illumina sequencing platform | |
| Typhoon 5 Bimolecular Imager | GE Healthcare Life Science | 29187191 |
참고문헌
- Karousis, E. D., Nasif, S., Mühlemann, O. Nonsense-mediated mRNA decay: novel mechanistic insights and biological impact. Wiley Interdisciplinary Reviews: RNA. 7 (5), 661-682 (2016).
- Ivanov, P. V., Gehring, N. H., Kunz, J. B., Hentze, M. W., Kulozik, A. E. Interactions between UPF1, eRFs, PABP and the exon junction complex suggest an integrated model for mammalian NMD pathways. The EMBO Journal. 27 (5), 736-747 (2008).
- Papasaikas, P., Valcárcel, J. The Spliceosome: The Ultimate RNA Chaperone and Sculptor. Trends in Biochemical Sciences. 41 (1), 33-45 (2016).
- Jensen, K. B., Darnell, R. B. CLIP: Crosslinking and ImmunoPrecipitation of In Vivo RNA Targets of RNA-Binding Proteins. Methods in Molecular Biology. 488, 85-98 (2008).
- Garzia, A., Morozov, P., Sajek, M., Meyer, C., Tuschl, T. PAR-CLIP for Discovering Target Sites of RNA-Binding Proteins. mRNA Decay: Methods and Protocols. 1720, 55-75 (2018).
- Konig, J., et al. iCLIP - Transcriptome-wide Mapping of Protein-RNA Interactions with Individual Nucleotide Resolution. Journal of Visualized Experiments. (50), e2638 (2011).
- Sibley, C. R. Individual Nucleotide Resolution UV Cross-Linking and Immunoprecipitation (iCLIP) to Determine Protein-RNA Interactions. RNA Detection: Methods and Protocols. 1649, 427-454 (2018).
- Wheeler, E. C., Van Nostrand, E. L., Yeo, G. W. Advances and challenges in the detection of transcriptome‐wide protein-RNA interactions. Wiley Interdisciplinary Reviews: RNA. 9 (1), (2018).
- Singh, G., et al. The Cellular EJC Interactome Reveals Higher-Order mRNP Structure and an EJC-SR Protein Nexus. Cell. 151, 750-764 (2012).
- Singh, G., Ricci, E. P., Moore, M. J. RIPiT-Seq: A high-throughput approach for footprinting RNA:protein complexes. Methods. 65, 320-332 (2014).
- Mabin, J. W., et al. The Exon Junction Complex Undergoes a Compositional Switch that Alters mRNP Structure and Nonsense-Mediated mRNA Decay Activity. Cell Reports. 25 (9), 2431-2446 (2018).
- Gehring, N. H., Lamprinaki, S., Kulozik, A. E., Hentze, M. W. Disassembly of Exon Junction Complexes by PYM. Cell. 137 (3), 536-548 (2009).
- Dostie, J., Dreyfuss, G. Translation Is Required to Remove Y14 from mRNAs in the Cytoplasm. Current Biology. 12 (13), 1060-1067 (2002).
- Zünd, D., Gruber, A. R., Zavolan, M., Mühlemann, O. Translation-dependent displacement of UPF1 from coding sequences causes its enrichment in 3' UTRs. Nature Structural & Molecular Biology. 20 (8), 936-943 (2013).
- Gangras, P., Dayeh, D. M., Mabin, J. W., Nakanishi, K., Singh, G. Cloning and Identification of Recombinant Argonaute-Bound Small RNAs Using Next-Generation Sequencing. Argonaute Proteins: Methods and Protocols. 1680, 1-28 (2018).
- Heyer, E. E., Ozadam, H., Ricci, E. P., Cenik, C., Moore, M. J. An optimized kit-free method for making strand-specific deep sequencing libraries from RNA fragments. Nucleic Acids Research. 43 (1), 2 (2015).
- Cameron, V., Uhlenbeck, O. C. 3'-Phosphatase activity in T4 polynucleotide kinase. Biochemistry. 16 (23), 5120-5126 (1977).
- Ricci, E. P., et al. Staufen1 senses overall transcript secondary structure to regulate translation. Nature Structural & Molecular Biology. 21 (1), 26-35 (2014).
- Lackner, D. H., et al. A generic strategy for CRISPR-Cas9-mediated gene tagging. Nature Communications. 6, 10237 (2015).
- Metkar, M., et al. Higher-Order Organization Principles of Pre-translational mRNPs. Molecular Cell. 72 (4), 715-726 (2018).
- Giudice, G., Sánchez-Cabo, F., Torroja, C., Lara-Pezzi, E. ATtRACT-a database of RNA-binding proteins and associated motifs. Database: The Journal of Biological Databases and Curation. 2016, (2016).
- Paz, I., Kosti, I., Ares, M., Cline, M., Mandel-Gutfreund, Y. RBPmap: a web server for mapping binding sites of RNA-binding proteins. Nucleic Acids Research. 42, 361-367 (2014).
- Sundararaman, B., et al. Resources for the Comprehensive Discovery of Functional RNA Elements. Molecular Cell. 61 (6), 903-913 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유