Method Article
Jel-seq: DNA ve RNA hidrojel matrisleri kullanarak eşzamanlı sıralama kitaplığı hazırlanması için bir yöntem
Bu Makalede
Özet
Jel-seq kitaplıkları için her iki DNA - ve RNA-seq basit hidrojel aygıtı kullanarak 100-1000 hücreden başlayarak ihmal edilebilir ek maliyetle aynı anda hazırlamak araştırmacılar sağlar. Bu kağıt eşleştirilmiş kitaplıkları oluşturmak için biyolojik Protokolü yanı sıra cihazın imalatı için detaylı bir yaklaşım sunar.
Özet
Yükseltmek ve DNA veya RNA küçük başlangıç örnekleri sıra yeteneği sadece son beş yıl içinde elde edilmiştir. Ne yazık ki, genomik oluşturmadan veya transcriptomic kütüphaneler için standart protokolleri uyumsuzdur ve araştırmacılar DNA veya RNA belirli bir örnek için sıralamak seçmeniz gerekir. Jel-seq aynı anda DNA ve RNA bir basit hidrojel aygıtı kullanarak 100-1000 hücrelerle başlangıç için kitaplıklarına hazırlamak araştırmacılar sağlayarak bu sorunu çözer. Bu kağıt eşleştirilmiş kitaplıkları oluşturmak için biyolojik Protokolü yanı sıra cihazın imalatı için detaylı bir yaklaşım sunar. Biz jel-seq araştırmacılar tarafından kolayca uygulanabilecek tasarlanmıştır; birçok genetik labs zaten jel-seq cihaz imalat çoğaltmak gerekli donanımları var. Bizim iletişim kuralı her iki bütün-transkript amplifikasyon (WTA) için yaygın olarak kullanılan kitleri istihdam ve de zaten araştırmacılar için tanıdık olması olasıdır Kütüphane hazırlık kitaplıkları genomik üreten ve transcriptomic usta. Bizim yaklaşım araştırmacılar DNA ve RNA sıralama gücünü ihmal edilebilir maliyet ve yarma olmadan tek bir örnek üzerinde ayı getirmek için sağlar.
Giriş
Yeni nesil (NGS) sıralama genetik araştırma yaptı yolda derin bir etkiye sahip oldu. Araştırmacılar bir kez tüm türlerin genom sıralama üzerinde duruldu nerede, şimdi tek bir tümör veya bir deneyde bile tek bir hücre genomu sıralamak mümkündür. 1 NGS, aynı zamanda maliyet etkin hücre, transcriptome bilinen veriler topluluğu içindeki bulundu RNA transkript sıralamak için sağlamıştır. Yükseltmek ve DNA veya RNA küçük başlangıç örnekleri sıra yeteneği sadece son beş yıl içinde elde edilmiştir. 2 , 3 , 4 ne yazık ki, standart protokolleri uyumsuzdur ve araştırmacılar DNA veya RNA belirli bir örnek için sıralamak seçmeniz gerekir. Başlangıç bir örnek yeterince büyük olduğunda, ikiye bölünebilir. Daha küçük ölçeklerde ancak, malzeme örnekleri bölme nedeniyle kaybı Kütüphane kalitesini etkileyebilir ve örnekleri havuzu hücreler arasında ilginç varyasyonlar dışarı ortalama olabilir. 5 Ayrıca, araştırmacılar, tek hücre veya küçük heterojen tümör biyopsi gibi bölünemez örnekleri inceleyerek giderek ilgilendi. 6
Bu sorunu çözmek için üç Protokolü son zamanlarda aynı başlangıç örneğinden DNA ve RNA sıralamak için geliştirilmiştir: jel-seq7, G & T-seq8ve DR-seq9. Bu makale aynı anda gelen önemsiz eklenen maliyet 100 hücreler olarak az DNA ve RNA kitaplıkları oluşturmak için kullanılan jel-seq için detaylı bir protokolü sunar. Roman jel-seq DNA ve RNA sadece düşük maliyetli hidrojel matrisleri kullanarak boyutuna göre ayırmak için yetenek yönüdür. RNA fiziksel ayrılması DNA'ın jel-Seq protokolünün çekirdek yeniliktir. Bu ayrılık electrophoretically bu moleküller arasındaki boyut farkları yararlanmak polyacrylamide membranlar kullanarak elde edilir. Bu boyut farkları bağlamda koymak için nasıl DNA ve RNA görüntüsü göz önünde bulundurun: DNA mikron ölçekte var ve geleneksel mikroskoplar kullanılarak görüntülenebilir, RNA nanometre ölçeğinde var ve cryo-elektron gibi karmaşık teknikleri kullanarak yansıma gerekir mikroskobu. 10
DNA ve RNA bu protokol için ayırmak için yaklaşım şekil 1' de gösterilen. Sol panelde çözüm bir membran yakın yüzen DNA ve RNA ücretsiz gösterir. Elektrik alan, sağ panelde gösterildiği gibi uygulandığında, DNA ve RNA membran aracılığıyla geçiş indükler bir elektroforetik güç deneyim. Membran özelliklerini ayarlama tarafından biz DNA RNA--dan ayıran yarı geçirgen bir zar oluşturduk. DNA molekülleri membran karşı itti ama kenarındaki büyük boyutlarından dolayı dolaşmış oldu. Küçük RNA molekülleri, öte yandan, yeniden yapılandırabilir ve membran yollarını örgü. Reptation bilinen bu işlem bir yılan çim hamle yol benzer. Sonunda bu RNA molekülleri daha küçük polimerler için çok zor bir ikinci, yüksek yoğunlukta membran tarafından durdurulur (> 200 baz çifti) aracılığıyla sıyrılmak için. Bir kez fiziksel olarak ayrılmış, DNA ve RNA getirilebilir kurtarıldı ve genom ve transcriptome hakkında bilgi oluşturmak için işlenmiş. DNA ve RNA ayırabilirsiniz, biz RNA cDNA ayırma önce transkripsiyonu ters ise daha iyi sonuçlar elde edilen bulduk. CDNA/RNA melez RNA yalnız daha stabildir ve hala düşük yoğunluklu membran geçebilir.

Resim 1 . Jel-seq çalışma prensibi. Fiziksel olarak DNA ve RNA ayırmak için kullanılan temel prensip. Uygulamalı elektrik alan, küçük RNA molekülleri düşük yoğunluklu membran geçiş ama büyük DNA molekülleri yüzeyde sıkışıp kalırlar. Bu rakam Ref. 7 Kimya Royal Society izniyle yeniden. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Bu kağıt jel-seq cihazın her iki fabrikasyon ayrıntılı olarak açıklar ve DNA ve RNA kitaplıkları oluşturmak için biyolojik Protokolü eşleştirilmiş. Her ikisi de genel bir bakış Şekil 2' de gösterilmiştir. Cihazın katmanlama üç farklı yoğunluk polyacrylamide jeller üst üste çift yığın jelleri oluşturmaya benzer bir işlemde tarafından imal edilmiştir. 11 biyolojik Protokolü PBS içinde askıya 100-1000 hücreleri ile başlar. Hücreleri lysed ve cihazın genomik DNA cDNA/RNA melez ayırmak için kullanılmadan önce RNA cDNA dönüştürülür. Ayrılık ve kurtarma, genomik ve transcriptomic sonra kitaplıkları standart bütün-genom kitaplığı hazırlık seti Protokolü yakından takip eden bir işlem kullanılarak hazırlanır. Geliştirme ve jel-seq doğrulama hakkında daha fazla ayrıntı üzerinde bir çip yayın laboratuarında okumak "jel-seq: Bütün-genom ve yarı geçirgen hidrojel engelleri kullanarak eşzamanlı düşük-girdili DNA ve RNA Kütüphane hazırlık tarafından transcriptome sıralama ." 7

Resim 2 . Jel-seq protokolü. Sonrasında tamamlanması jel-seq aygıt ve iletişim kuralı oluşturulan eşleştirilmiş DNA ve RNA kitaplıklarına imal etmek. Bu rakam bölümlerini Ref. 7 Kimya Royal Society izniyle çoğaltılamaz. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
DNA ve RNA kitaplıkları tek hücreler üretmek için araştırmacılar G & T-seq ya da DR-devamı G & T-Seq, jel-seq gibi kullanmayı dikkate almanız gerekir, genomik DNA RNA'ın fiziksel bir renk kullanır. Bu yaklaşım mesajcı RNA üzerinde'nın (mRNA) dayanır 3'poliadenile kuyruk aşağı açılır hedef olarak. MRNA üzerinde bir manyetik boncuk biotinylated oligo-dT astar kullanarak yakalanır. MRNA yakalandıktan sonra boncuklar bir mıknatıs ile yerde tutulur ve genomik DNA içeren süpernatant kaldırıldı ve olması için başka bir tüp transfer. Bu fiziksel ayırma tamamlandıktan sonra mRNA ve DNA ayrı kütüphaneleri oluşturulabilir. 8 çalışan bu yaklaşım de ilgi RNA poliadenile ise, ancak bu poliadenile transkript, ribozomal RNA, tRNA veya RNA prokaryot üzerinden gibi çalışmak için kullanılamaz.
DR-seq DNA ve RNA türetilmiş cDNA aynı tüp nerede güçlendirilmiş bir öncesi amplifikasyon adım kullanır. Örnek sonra ikiye bölünmüş ve DNA ve RNA seq kütüphaneleri hazırlanmak için paralel olarak işlenebilir. Genomik DNA ve RNA türetilmiş cDNA arasındaki farkı anlamak, DR-seq Hesaplamalı bir yaklaşım. Bu DNA veya RNA kökenli gibi dizileri ekzonlar sadece mevcut nerede hesaplama açısından genomik DNA veri görüntülenmez. 9 cDNA/RNA ve DNA fiziksel olarak jel-seq ve G & T-seq. tamamlandý olarak ayrılması değil ki bu yaklaşımın bir avantajı olduğunu Dezavantaj, ancak, DR-seq genom ve transcriptome (Yani, ekzonlar intron karşı) bir temanın bilgi gerektirir ve çekirdekleri içinde birçok transkript henüz tam değildir, sıralama gibi uygulamalar için ideal olmayabilir olduğunu spliced ve hala intron içeriyor. 12
Roman jel-seq DNA ve RNA hücre sadece boyutuna göre yüzlerce ayırmak için yetenek yönüdür. Bu yöntem hiç bir priori bilgi genom veya transcriptome, eksik Uçbirleştirme karşı sağlamdır ve poli-adenylated transkript için sınırlı değildir gerektirir. Bir araştırmacı en az 100 hücrelerle başlayabileceğiniz uygulamalarda, jel-seq ucuz ve yaygın olarak kullanılan malzemeler kullanılarak basit bir yaklaşım sağlar.
Protokol
1. kimyasal çözüm hazırlık
Not: Sonraki adımda gerekli kimyasal çözümleri hazırlamak için aşağıdaki adımları vardır. Bunlar toplu olarak yapılan ve birkaç ay için depolanan.
- Başlamak için 254 nm UV crosslinking fırında 15 dakika (15 mJ/cm2 toplam pozlama) herhangi bir bulaşıcı DNA nötralize etmek sterilize tarafından 50 mL saf, deiyonize su hazırlamak. Aşağıdaki adımları kullanmak için 37 ° c ısı.
- 10 mL % 40 toplam (T) ve % 3.3 crosslinker (C) (29:1) yapmak polyacrylamide öncü çözüm. 3.867 g Akrilamid monomer ve BIS-Akrilamid monomer 0,133 g ağırlığında. Birleştirmek ve birim getirmek ilâ 10 mL sıcak saf su ve eriyene kadar girdap ile. Mağaza oda sıcaklığında ışık korunuyorsunuz.
Not: Premix T % 40, ticari olarak % 3.3 C (29:1) polyacrylamide çözümleri satın alınabilir. - 10 mL % 50 T, % 5 C jel çözüm yapmak. 4.750 g Akrilamid monomer ve BIS-Akrilamid monomer 0.250 g ağırlığında. Birleştirmek ve birim getirmek ilâ 10 mL sıcak saf su ve eriyene kadar girdap kullanarak. Mağaza oda sıcaklığında ışık korunuyorsunuz.
- 10 mL % 50 (w/v) Sükroz çözeltisi olun. 5 g Sükroz için mezun bir silindir eklemek ve toplam hacmi 10 mL kadar sıcak saf su ekleyin. Eriyene kadar girdap ve mağaza oda sıcaklığında.
- 10 mL % 10 yapmak APS (w/v). 1 g amonyum persülfat (APS) mezun silindir için ekleyin. Soğuk (~ 4 ° C) saf eklemek toplam hacmi 10 mL ve eriyene kadar girdap kadar su. Hemen 200 µL aliquots içinde dondurmak.
2. jel-seq kaset imalat
Not: Jel-seq was orijinal gelişmiş-dik kaset (daha fazla bilgi için bkz: Malzemeler tablo ) ile; Ancak, bu iletişim kuralını herhangi bir standart Jel Elektroforez kaset ile çalışmaya adapte edilebilir.
- Jel öncüleri üç ayrı plastik tüpler Tablo 1 aşağıda gösterildiği gibi ayıraçlar ekleyerek hazırlayın. Yok APS veya Tetramethylethylenediamine (TEMED) aşağıdaki adımlarda yönetmen kadar ekleyin. Girdap malzemeyi iyice karıştırın.
| Dolgu jel habercisi | Yüksek yoğunluklu jel habercisi | Düşük yoğunluklu jel habercisi | |||
| 40 %T, 3.3%C Akrilamid Bisacrylamide çözüm | 1.6 mL | 50 %T, 5 %C Akrilamid Bisacrylamide çözüm | 2.4 mL | 40 %T, 3.3%C Akrilamid Bisacrylamide çözüm | 0.6 mL |
| Deiyonize su | 10.2 mL | Deiyonize su | 1.0 mL | Deiyonize su | 4.8 mL |
| Sükroz çözüm (% 50 w/v) | 2.6 mL | Sükroz çözüm (% 50 w/v) | 0.6 mL | ||
| 10 X Tris-Bor-EDTA | 1.6 mL | 10 X Tris-Bor-EDTA | 0.6 mL | ||
| Amonyum persülfat (% 10 w/v) | 104.0 ΜL | Amonyum persülfat (% 10 w/v) | 50.0 ΜL | Amonyum persülfat (% 10 w/v) | 39.0 ΜL |
| TEMED | 6.0 ΜL | TEMED | 1.0 ΜL | TEMED | 2.2 ΜL |
| Toplam hacim | 16.1 mL | Toplam hacim | 4.1 mL | Toplam hacim | 6.0 mL |
Tablo 1. Jel sentez reaktifler. Polyacrylamide jel habercisi reaktifler 2 kaset imalatı için yeterli.
- Jel monomer çözümleri de-gaz tüp kap bir iğne ekleme ve bu iğneyi bir ev vakum hattına bağlayarak. Bu derlemede bir ultrasonik Banyo Seti yüksek olarak daldırın ve sıvıdan bir sonraki adıma geçmeden önce ortaya çıkan kabarcıklar durdurana kadar bekleyin (~ 60 saniye / tüp).
Not: çözüm ortaya çıkan kabarcıklar görülebilir sürece vakum kalitesini ve ultrasonik banyo gücünü kritik değildir. - TEMED ve APS dolgu jel habercisi ve girdap kısaca ekleyin. Hemen dolgu jel habercisi 6 mL her jel kaset kaset üst içine belgili tanımlık eriyik pipetting tarafından ekleyin. 1 mL micropipette habercisi dökülmesini önlemek için altı artışlarla eklemek için kullanın. Bu adım için toplam süre 3 dakikadan az olmalıdır.
- Sıvı düzey kasetleri bir düzey tablo dik konumlandırma tarafından olduğundan emin olun. Kasedi geri kalanı degassed, deiyonize su ile doldurun. 1 mL aralıklarla, yavaş yavaş karıştırma en aza indirmek için kaset Merkezi kullanarak yeniden su pipette. En az bir saat kadar bir gecede tedavisi polimer izin.
- Polimer tedavi vardır sonra jel kaset su yer paylaşımı kaldırmak için bir lavabo üzerinde tersine çevirin. Basınçlı hava silah yavaşça arabirimi 6 inç mesafeden kaset üst açıklıklı havada esen kuru için kullanılabilir.
- TEMED ve APS yüksek yoğunluklu jel habercisi ve girdap kısaca ekleyin. Hemen yüksek yoğunluklu habercisi 320 µL kullanarak yeniden 1 mL pipet kaset için ekleyin. Sıvı eşit kat dolgu jel katman kasedi ileri geri yaklaşık 3 kat rock yaparak emin olun. Bu adım daha az üç dakika sürer.
- Kasedi geri kalanı degassed, deiyonize su ile doldurun. Yavaş yavaş 1 mL pipet ile karıştırma en aza indirmek için kaset ortasına pipet. En az on beş dakika, tercihen bir saat için tedavi polimer izin.
- Yine, su yer paylaşımı kaldırmak için jel kaset tersine çevirin. Basınçlı hava arabirimi hafifçe kurumasýný kullanılabilir. TEMED ve APS düşük yoğunluklu jel habercisi ve girdap kısaca ekleyin. Hemen kaset (~1.65 mL) geri kalanı ile düşük yoğunluklu jel habercisi doldurun ve jel tarak ekleyin.
- Polimerizasyon sırasında absorbe edilebilir gibi rezerv habercisi tarak üstündeki aşırı pipette. En az 4 saat, tercihen gece tedavisi polimer izin. Jelleri bir hafta veya daha fazla Tris-Bor-Ethylenediaminetetraacetic asit (EDTA) (TBE tampon) arabellekte depolanabilir.
3. numune hazırlama ve ters transkripsiyon
- Faiz hücrelerinin bir süspansiyon ile başlayan ve Polimeraz zincir reaksiyonu (PCR) laminar akış mahallede çalışma bir hemasitometre veya bir otomatik hücre sayaç hücre toplama hesaplamak için kullanın. Bir konsantrasyon µL serum fosfat tamponlu (PBS) içinde başına 100-1000 hücrelerin hücrelere sulandırmak.
Not: Bu protokol PC3, HeLa ve fare karaciğer hücreleri gibi hücreler aralığına doğrulandı. - WTA içinde sağlanan reaktifler kullanarak (bakınız Tablo malzemeler), mix 19 µL lizis arabelleği kiti ve 10 X hazırlamak için RNase inhibitörü 1 µL stok çözüm tepki arabelleği. Yeterli hacim tepki arabelleği 0.5 µL ve her örnek için nükleaz boş su 2.75 µL içeren lizis ana karışımı oluşturun.
- Yukarı ve aşağı kapatılan hücreleri yeniden askıya almak ve sonra örnek 1 µL UV steril bir 200 µL nükleaz ücretsiz şerit tüp içine pipet 5 kez hücre süspansiyon pipette. Örnek sayısına bağlı olarak gerektiği gibi tekrarlayın. Pipetting 1 µL nükleaz ücretsiz su yerine bir reaksiyon için hücreleri tarafından bir negatif kontrol eklediğinizden emin olun. Ardından, 3.25 µL lizis ana karışımı her örnek için ve hafifçe yukarı ve aşağı 5 kere pipetting tarafından karıştırın.
- (Kapaklı ısıtmalı) bir termal cycler 72 ° c Onceden 1 µL RT astar ve 20 µM rasgele hexamer 1 µL WTA adaptörü (5 '-AAGCAGTGGTAT-CAACGCAGAGTAC-NNNNNN-3 ') ile her örnek için ekleyin. GDNA Kütüphane hazırlık için pozitif kontrol olarak en az bir tüp rezerv ve 2 µL astar yerine su ekleyin.
Not: WTA adaptörü ile rastgele hexamer isteğe bağlıdır ve sıralama sonuçları üzerinde çok az etkisi vardır. - Örnekleri hücreleri parçalayıcı için Önceden ısıtılmış termal cycler 3 dakika 72 ° C'de kuluçkaya. Hücreleri termal cycler ve buz üzerinde yer 2 dakika süreyle kaldırın. 4 ° C'de olumlu denetim 5 adım kadar saklayın.
- Hücreleri lysing iken, ters transkripsiyon ana Mix aşağıdaki reaktif oranları içeren tüm RNA örnekleri için yeterli bir birim oluşturmak: 2 µL ilk strand arabelleği, şablon anahtarı Oligonükleotid (TSO), 0,25 µL RNase inhibitörü ve 1 µL 0.5 µL ters transkriptaz (100 U/µL).
- Termal cycler 42 ° c Onceden Toplam numune hacmi 10 µL. için getirmek kalan örnekleri, ters transkripsiyon ana Mix 3.75 µL eklemek yukarı ve aşağı 5 kere pipetting tarafından Mix.
- Ters transkripsiyon örnekleri içinde önceden ısıtılmış bir termal cycler getirdikten hemen gerçekleştirmek. Aşağıdaki programı çalıştır: 90 dk, 70 ° C 10 dk, sonsuza dek 4 ° C için 42 ° C. Bu güvenli durdurma noktadır.
4. jel ayırma ve örnek kurtarma
- Dikkatli bir şekilde DNA kaldırma ürün kullanarak bir Jel Elektroforez odası temiz. Birkaç mL sıvı Temizlik maddesi odası tüm yüzeyler arasında bir tek kullanımlık tüysüz ücretsiz silme ve temizleme işlemi uygulamak ve sonra odası temiz 0,5 ile doldurun x TBE. En iyi sonuçlar için tüm cihazları bir 254 nm UV crosslinking fırında yerleştirin ve 15 dakika (15 mJ/cm2) sterilize.
- Jel-seq kaset Jel Elektroforez odanın içine yerleştirin ve yerine kilitlemek. Yavaş yavaş jel tarak düz yukarı çekerek çıkarın. Jel yırtılma veya herhangi bir silah kopyalama önlemek yavaş hareket edin.
- Adım 3 buz üzerinde örnekleri alıkoyan, en az bir örnek olumlu bir denetim cDNA Kütüphanesi üretimi için saklıdır. Bu denetim 4 ° C'de adım 6 kadar saklayın. Toplam hacim ~ 12 µL. iyice getirmek yukarı ve aşağı 5 kere pipetting tarafından mix örnekleri, boya kalan örnekleri için yükleme X 6 2 µL ekleyin.
- Bir DNA merdivenin 1 µL boya ve su 7 µL yükleme x 6 2 µL ile birleştirir. Bu karışım jel-seq Kaset 1 şeride bir Elektroforez denetimi olarak pipette. Jel-seq kaset ayrı şerit içine önceki adımdaki örnekleri pipette. Pipet tamamen her kuyuya ekleyerek ve yalnızca dikey bir hareket kullanarak kaldırma kuyular arasında kirlenmesini önlemek için dikkatli olun.
- Bir standart Jel Elektroforez Güç kaynağı kullanarak, bir elektrik alanı 250 V jel-seq kaset cDNA/RNA melez gDNA ayırmak 30 dakika boyunca uygulayın. Kez ayrılmış, jel-seq kaset Jel Elektroforez odasından kaldırın ve kazıma bir araç ile kenarları gözetleyen tarafından kaset iki yarısı açın.
- Bir neşter kullanarak, jel yüksek yoğunluklu katmanın altında ikiye böldüm. Eldivenli elini ile alacak tarafından dolgu jel içeren yarım atmak. Yavaşça kalan jel boya sıyırıcı veya başka benzer alet ile kazıma kaset kapalı soyma. Bu bölümde jel ~ 30 mL 0.5 içeren bir tabak yerleştirin jel leke 3 µL ile x TBE.
- Photobleaching en aza indirmek ve jel kapsayıcı için 5 dakika hafifçe sallayarak süre emmek için konteyner kapağı. Jel üzerinde plastik wrap yerleştirin ve bir jel dokümantasyon sistemi (daha fazla ayrıntı için bkz: Ref. 13) kullanarak bir UV görüntü almak. Bir 30 saniye pozlama genellikle net görüntüler üretir. Ayrılık meydana geldiğini doğrulayın.
- Jel görselleştirme Nükleik asitlerin kolaylaştırmak için bir UV transilluminator taşıyın. Uygun UV gözlük takan, jel dokümantasyon sistemi sonuçlarından onaylayın. GDNA düşük yoğunluklu jel ve cDNA düşük yoğunluklu ve yüksek yoğunluklu bölgelerde arabirimi, başında bulunduğu olmalıdır.
- Bir neşter kullanın, gDNA ve cDNA içeren jel bölgeleri keser. Örnekleri en iyi jel 10 mm dikdörtgen bölüm 4 mm kesim tarafından kurtarılan; Ancak, tam geometri kullanılan Jel Elektroforez Sistemi üzerinde bağlıdır. Ayrıca negatif kontrol ile dolu lane kesmek hatırlıyorum.
- Jel bir şerit tüp içine çıkarılan her bölümünün künt son cımbız bir çift kullanarak yerleştirebilirsiniz. Jel birden çok parçaya ayrılmış veya çok fazla kuvvet uygulamak değil dikkatli olun. Bu olur, sadece her parça almak ve tüp ekleyin.
- Pipet ucu tüp alt karşı dairesel şekilde hareket ettirerek bir pipet (200 µL pipet ipuçları çalışma iyi) ucu kullanarak her tüp jel eziyet. Nükleaz boş su (gDNA örnekleri içine 40 µL ve cDNA örnekleri içine 80 µL) örnek kaybı en aza indirmek için jel öğütmek için kullanılan pipet ucu kaldırmadan önce her tüp için ekleyin.
- Şerit tüpler 37 ° C İnkübatör içinde bir girdap Mixer yerleştirin ve 8-12 saat için çalkalanır. Bu jel dışında yaygın nükleik asitleri sağlar ve doğal bir noktası bu çok gün protokol durduruluyor.
- Örnekleri bir 8 µm gözenekli filtre plaka pipette ve jel parçaları dışarı zorlanma 5 dakika plaka 2600 x g de spin. Gözenekli filtre plaka konut plaka uzak kaldırın ve jel içermeyen su örnekleri bir yeni 200 µL şerit tüp pipette.
- GDNA, iyi tarafından yukarı ve aşağı, pipetting karışımı içeren her örnek proteaz (0.9 AU/mL) 1 µL ekleyin ve 15 dakika 15 dakika 70 ° C'de ısı inactivation ardından 50 ° C'de kuluçkaya. Bu adım oluşturarlar tüketen için önemlidir ve gDNA sonraki tepki adımlar için erişilebilir yapar.
- Bir 18 gauge iğne kullanarak, tüm örnek tüplerinin kapaklar delik poke. Örnekleri sıvı sesi azaltmak için bir vacufuge yerleştirin. gDNA örnekleri 5 µL ve cDNA örnekleri için 10 µL azaltılmış azaltılmalıdır.
- Vacufuge ve örnek sayısına bağlı olarak, toplam buharlaşma süresi 30 ile 60 dakika arasında değişir. Numune hacmi hedef birimin düşerse, nükleaz boş su numune hacmi artırmak için eklemeniz yeterli olacaktır.
5. gDNA Kütüphane hazırlık
- Bir yer veya benzer teknoloji kullanarak, her gDNA örnek adım 4 hem de Adım 3 pozitif denetiminden DNA toplama ölçmek. Detaylı bir iletişim kuralı için yer başvuru kılavuzuna bakın. 14
- 0.2 ng/µL DNA örnekleri sulandırmak. Başlangıç hücre tipi ve gerekli sonuçları kalitesine bağlı olarak, düşük konsantrasyonlarda hala kalıcı kitaplıklar oluşturabilir. Bazı deneyler-ecek var olmak gerekli; Ancak, yazarlar kitaplıkları 0.1 ng/µL düşük ile başarılı oldu.
- GDNA Kütüphane hazırlama adım 7 Kütüphane hazırlık yarım tepki birim protokolünde izleyerek tamamlayın.
6. cDNA Kütüphanesi hazırlık
- 10 µL cDNA örnekleri ve olumlu adım 4 denetiminden ile başlayan, 12.5 µL 2 X qPCR mix, 0.5 µL cDNA PCR astar ve 2 µL nükleaz ücretsiz su ekleyin.
- PCR aşağıdaki iletişim kuralını kullanarak bir gerçek zamanlı thermocycler gerçekleştirmek: 3 dk 98 ° c 20-30 devredir 10 ardından 95 ° c sıcak-başlangıç s, 65 ° C 30 s ve 72 ° C için 3 dk. Toplam numune hacmi 25 için µL. tepki eğrileri izleneceğini ve th önce amplifikasyon e reaksiyonlar (doğrusal sinyal artış karşı döngü sayısı) veya üssel faz PCR eserler overamplification nedeniyle önlemek için bırakın. Daha fazla bilgi için overamplification kaçınmak, bu kağıt gibi Ref. 15 tartışma bölümüne bakın.
- Amplifikasyon sonra 8. adımda protokol sonrası katı faz ters çevrilebilir immobilizasyon (SPRI) boncuk kullanarak ürün temiz. Bir kez tam, sonraki adıma geçin.
- Bir yer veya benzer teknoloji kullanarak, her cDNA örnek hem de Adım 4 olumlu denetiminden DNA toplama ölçmek. Örnekleri DNA'ın yaklaşık 0.2 ng/µL içerecek şekilde gerekirse sulandırmak. Biraz düşük konsantrasyonlarda hala kalıcı kitaplıklar üretmek. Bazı deneyler-ecek var olmak gerekli, ancak yazarlar kitaplıkları 0.1 ng/µL düşük ile başarılı oldu.
- İsteğe bağlı adım: bir standart polyacrylamide Jel Elektroforez ayrılık üzerinde 1-2 µL her kitaplık üretimi tepki doğrulamak için qPCR ürün çalıştı gerçekleştirmek. Başarılı sonuç bir örnek görüntü şekil 4' te gösterilmiştir. Polyacrylamide Jel Elektroforez kuralları konusunda detaylı iletişim kuralı için bkz: Ref. 16.
- CDNA Kütüphanesi hazırlama kitaplığı hazırlık seti yarım cilt reaksiyon Protokolü adım 7 izleyerek tamamlayın.
7. Kütüphane hazırlıkla yarım cilt reaksiyonları
- Kütüphane hazırlık yarım cilt reaksiyonları kullanarak kütüphane hazırlık seti protokolü takip ediyor. 17 Protokolü'nün bu bölümünde başvurulan tüm reaktifler Kütüphane hazırlık vardır ( Tablo malzemelerigörmek) kit. UV tarafından şerit tüpler örnekleri sayısı için yeterli sayıda sterilize işlenmek üzere başlar.
- Transposase reaksiyon 5 µL transposase arabellek assay olarak kullanılmak üzere her şerit tüp ekleyerek gerçekleştirin. Sonra 2.5 µL transposase tarafından takip 2.5 giriş µL DNA 0.2 ng/µL (0,5 ng toplam) ekleyin. Yukarı ve aşağı 5 kere pipetting karıştırın.
- 55 ° C'de 5 dakika kuluçkaya, sonra 10 ° C'de basılı tutun Örnek 10 ° C ulaştıktan sonra thermocycler kaldırın ve hemen her örnek için 2.5 µL transposase dur arabellek ekleyin. Örnekleri oda sıcaklığında 5 dakika tutun.
- 7.5 µL Kütüphane hazırlık PCR karışımı, bir dizin 1 astar 2.5 µL ve 2.5 µL bir dizin 2 astar ekleyerek tedavi sırasını değiştir örnek yükseltmek için PCR reaksiyon hazırlayın. Bu astar özeldir ve Kütüphane hazırlık seti üreticisi tarafından sağlanır. Yukarı ve aşağı 5 kez de pipetting tarafından karıştırın.
Not: benzersiz astar bileşimleri her örnek için kullanıldığından emin olun. 12 farklı dizin 1 astar ve benzersiz olarak kadar 96 farklı örnekleri etiket edinerek 8 farklı dizin 2 astar vardır. Astar her örnek için benzersiz bir bileşimini seçin. - PCR aşağıdaki programı bir thermocycler kullanarak gerçekleştirin. 25 µL örnek birimdir.
72 ° C için 1 dakika
30 saniye için 95 ° C
12 döngüleri:
95 ° C 10 saniye
30 saniye için 72 ° C
30 saniye için 55 ° C
5 dakika 72 ° C
10 ° C'de tutun - 8. adımda protokol sonrası SPRI boncuklar kullanılarak hazırlanan kitaplıkları temiz. Kütüphaneler Jel Elektroforez veya benzer bir yöntemi kullanarak doğrulanması. Kütüphane hazırlık seti elkitabına kitaplıkları doğrulamak hakkında ayrıntılı bilgi için bakınız. 17
8. katı faz ters çevrilebilir immobilizasyon boncuk Kitaplığı Temizleme
- Her kullanımdan önce oda sıcaklığına tersinir immobilizasyon (SPRI) boncuk 1.5 mL aliquots içinde muhafaza edilmelidir ve -meli var olmak sağlam aşama getirdi. Ayrıca taze % 80 etanol her deneme için hazırlamak için önerilir. Aşağıdaki adımlar, dayanır SPRI boncuk Protokolü WTA seti el kitabı. 18
- 1 µl WTA kiti lizis arabelleği her PCR ürünü ekleyin. Girdap SPRI boncuk eşit kadar karışık, o zaman her örnek için 50 µl SPRI boncuk ekleyin. Yukarı ve aşağı 10 kez örnek pipetting tarafından mix ve 8 dakika oda sıcaklığında örnekleri kuluçkaya.
- Kısa bir süre sıvı tüpler taraftan toplamak için örnekleri spin. Örnekleri bir manyetik ayırma aygıtta ~ 5 sıvı tamamen açık görüntüleninceye kadar dakika yerleştirin.
- Örnekleri manyetik ayırma aygıtta olmakla birlikte, yavaş yavaş süpernatant ve atma pipet - tüp boncuk yüzük değil rahatsız etmek için dikkatli olun. Sonraki 200 µL % 80 etanol boncuk bozmadan her örnek için ekleyin. 30 saniye bekleyin ve daha sonra dikkatlice süpernatant pipet. (Etanol yıkama) bu adımı yineleyin sonra.
- Örnekleri için 30 kuru izin s - 1 dak. Büyük parçaları kalıcı olarak boncuk için bağlı olmak gibi örnekleri aşırı kuru değil.
Not: Kısa bir kuruma süresini etanol bütün izlerini kaldırıldı emin olmak için önerilir. Eser miktarda etanol geride kalması gereken onlar biraz aşağı akım reaksiyonlar inhibe olabilir. - Örnekleri manyetik ayırma aygıttan kaldırmak ve her örnek boncuk DNA'dan elute 15 µL su ekleyin. Yukarı ve aşağı pipet ve boncuk tüpler iki tarafından kaldırılır emin olun. Oda sıcaklığında 2 dakika süreyle kuluçkaya. Kısaca örnekleri spin ve onları manyetik ayırma aygıtta ~ 1 çözüm Açık görüntüleninceye kadar dakika yerleştirin.
- Örnekleri manyetik ayırma aygıtta olmakla birlikte, yavaş yavaş süpernatant pipet ve temiz tüpler için transfer. Tüp boncuk yüzük bozmamaya dikkat edin. Boncuk tüplerini atmak.
Sonuçlar
GDNA ve cDNA/RNA melez jel-seq cihazda fiziksel ayrılması ile floresan jel görüntüleme görüntülenmeyecektir; temsil edici bir sonuç şekil 3' te gösterilmiştir. Paneli A fabrikasyon jel-seq aygıt gösterir; yanlış renk farklı jel bölgeleri ayırmak için eklendi. Panel B dört farklı renk ayrımlarını doğrulamak için kullanılan yakın gösterir. Üçüncü lane, bir negatif kontrol, arka plan temsil eder ve hiçbir autoflourescence arabirimleri, jel gösterir. Biz DNA merdivenler ile birinci ve ikinci şerit yüklü. Bu şerit arayüzü arasında küçük parçaları düşük yoğunluklu jel geçirebilirsiniz açığa düşük ve yüksek density membranlar, sadece karanlık bir grup göster. Dördüncü lane ilgi biyolojik örnek davranışını gösterir: 500 PC3 hücreleri. Lane dört protokol 3. adımında açıklandığı gibi yüklü. Görüntü genomik DNA ve cDNA/RNA melez ayrımı gösterir. CDNA/RNA melez düşük ve yüksek yoğunluklu bölgelerde arabirimi yığılmış iken düşük yoğunluklu membran üstündeki karanlık bir grup megabase ölçekli genomik DNA'sı. Merdiven ile yüklenen yolları farklı olarak, orada da birkaç grup içinde yüksek yoğunluklu bölgenin mevcut jel. Bu parçaları, 100'den daha küçük bp, astar oligonucleotides ters transkripsiyon sırasında oluşturulan hedef kapalı ürünler vardır. Paneli C tüm jel-seq cihaz başarılı bir deneme temsilcisi bir görüntüsünü gösterir. RNA etiketli şerit sadece gDNA ve RNA ayrılması gösterirken RNA/cDNA etiketli şerit jel-seq protokolüyle işlendi. Panel D düşük yoğunluklu membran her Sokağın başındaki siyah bantlar ile başarısız bir deney gösterir. Bu parçalanmış DNA ile kontamine Elektroforez arabellek neden oldu. Bu adımda, araştırmacılar temiz negatif denetimleri ve ayrı gDNA ve RNA/cDNA melez varlığını gösteren iki ayrı siyah bantlar için bakmalısın.

Şekil 3 . Jel-Seq ayrılık sonuçları. Jel-seq aygıt (A) ve DNA ve RNA/cDNA melez (B) ayrılması gösterilen bir floresan görüntü. Yanlış renk yoğunluğu jel içinde farklı bölgelerinde daha kolay ayırt etmek için eklendi. (C ve D) Tüm jel-seq cihaz başarılı (C) ve (D) Deney başarısız temsilcisi floresan görüntüleri. NTC şablon kontrol =. Bu rakam bölümlerini Ref. 7 Kimya Royal Society izniyle çoğaltılamaz. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Bir kez DNA ve RNA/cDNA melez ayrılmış ve tamamlanmış jel-seq Protokolü kalan, sıralama kitaplıkları oluşturmak mümkündür. Hazırlanan kitaplıkları doğrulamak için bir standart Jel Elektroforez deneyi (şekil 4) ya da bir bioanalyzer çalıştırın. Şekil 4 sonuçlarında 500 PC3 hücreleri ve 750 HeLa hücreleri oluşturulan kitaplıkları göster. Şekilde eşleşen kitaplıkları jel-('Jel' etiketli) eşsiz örnekleri ('Tüp' etiketli) standart iletişim kuralları ile oluşturulan karşılaştırıldığında seq kaynaklandığı için parça dağıtımları gösterilmiştir. Jel-seq parçası boyutları arasında 200 ve 800 basepairs standart bütün-genom kitaplığı hazırlık seti kullanarak kitaplıkları hazırlarken beklendiği gibi görünür. Kütüphane parçaları bu adımı doğru boyut aralığında görünmüyorsa Kütüphane hazırlığı başarısız oldu.
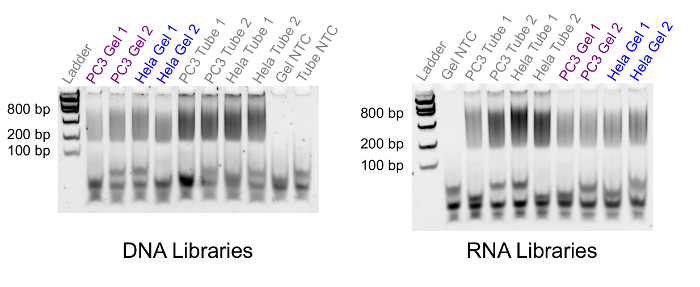
Şekil 4 . Kütüphane parçası boyut karşılaştırma. Bir floresan Jel Elektroforez jel-seq (jel) ve standart denetimler (tüp) arasında karşılaştırma Kütüphane boyutu dağıtım görüntü. Sol şeritte parçası boyutları 100, 200, 400, 800, 1200 ve 2000 basepairs ile düşük bir kitle DNA merdiven içerir. Tüm kitaplıkları için parça boyutları 200 ve 800 basepairs arasında bu kiti ile hazırlanan kitaplıkları için beklendiği gibi düşmek. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Jel-seq Protokolü'nün son doğrulama sıralama sonuçlarının analizine dayanır. Bu hücreler örnekleri olmak izin homojen ifade profilleri gibi biz bizim doğrulama denemeleri için PC3 hücreler seçilene bölünmüş ve jel-seq ve geleneksel yöntemlerle; kullanarak işleme bkz. şekil 5. PC3 hücreler için genomik DNA arasında bir karşılaştırma rakamları 5A ve 5Bgösterilir. Şekil 5 A genom çapında kopya numarası değişimi (KNV) profilleri PC3 oluşturulan bir karşılaştırma gösterir jel-seq ya da bir standart bütün-genom kitaplığı hazırlık tepki (tüp denetimi) kullanarak. Her noktası ortalama normalleştirilmiş bin sayısıdır; depo gözleri her depo gözü sağlıklı diploit hücre, Yani, tüm otozomal her bölge için eşit kopya temsil eden bir düz çizgiyi eşit beklenen sayısı vardır öyle ki başvuru genom verilerden tanımlanır (hariç X ve Y) kromozomlar. PC3 sivri bir arka plan kopya sayısı iki yukarıda olarak sırıtmak aynı bölgeleri birden çok kopyasını içerir. Jel-seq niteliksel benzer KNV profil standart tüp tepki verir. İki araziler arasındaki anlaşma kantitatif doğrusal regresyon tarafından B. panelinde gösterildiği gibi değerlendirilebilir Pearson korelasyon r = 0.90 genomik veri iki yöntemden herhangi birini toplanan işlevsel olarak eşdeğer olduğunu gösterir.

Şekil 5 . Kitaplık doğrulama. Jel-seq doğrulama için genomik (A ve B) ve transcriptomic (C, D ve E) veri PC3 ve Hela hücrelerinden üretilen. Masası'A bir karşılaştırma gösterir genom çapında KNV profilleri PC3 oluşturulan jel-seq (jel-seq) ya da standart reaksiyon (tüp) kullanarak. Emniyetinde medyan mutlak ikili fark =. B paneldir bir doğrusal regresyon panelinde A iki örnekleri arasında = R ile 0,90 gösteren genomik veri işlevsel olarak eşdeğer. Eksenleri göster: log2 normalleştirilmiş bin sayar. Panelleri C ve D transkript kilobaz başına bir sayısını gösteren her noktasıyla PC3 hücrelerden transcriptomic veri karşılaştırmak milyon (TPM). Eksenleri normalleştirilmiş log2 transkript sayıları olarak iki örnekleri arasında karşılaştırma göster. Paneli C teknik çoğaltır jel-seq kullanılarak oluşturulan bir karşılaştırmasını gösterir ve Panel D jel-seq RNA ifade asıl bileşen analizi kullanarak temel hücre tipi çözümlemek için jel-seq ve geleneksel RNA-seq. Panel E arasında bir karşılaştırma gösterir gösterir. X ekseni bu %91,6 y ekseni bu sadece %6,5 varyans ikinci temel bileşeni hesaplarında gösterirken örnek arasında varyasyon için ilk temel bileşeni hesaplarını gösterir. Bu rakam bölümlerini Ref. 7 Kimya Royal Society izniyle çoğaltılamaz. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Benzer şekilde, biz standart tüp akıllı-Seq protokolü için bizim jel-seq iletişim kuralından transcriptomic verileri karşılaştırıldı. Şekil 5 her iki jel-seq teknik çoğaltır (şekil 5C) arasında jel-seq ve standart yöntem (şekil 5D) arasında korelasyon gösterir. Her noktası transkript kilobaz başına içinde sayısıdır milyon (TPM) için TPM algılanan her gen > 5'te her iki veri kümeleri. Doğrusal regresyon kırmızı çizgiler olarak gösterilir ve Pearson korelasyon katsayısı sol üst köşesinde gösterilir. Jel-seq üzerinden teknik çoğaltır (R ∼ 0,8) katılıyorum, ama daha az iyi standart yöntemi ile ilişkilendirmek (R < 0,7). Bu jel-seq gen sayıları bir önyargı tanıttı göstermektedir. Neyse ki, bu önyargı sistematik ve şekil 5Easıl bileşen analizi tarafından görüldüğü gibi anlamlı sonuçlar hala farklı biyolojik örnekleri arasında çizilebilir.
Tartışmalar
Jel-seq aygıt uydurma gibi protokol kendisi ile ilişkili çeşitli kritik adımlar vardır. İmalat sırasında jel çeşitli bölgeleri için öngörülen katman kalınlıkları ile başlayan öneririz. Önemli zaman farklı imalat seçenekleri test geçirdim ve en iyi cihazlar malzemeler tablo ve Kimyasallarılistelenen kaset için burada açıklanan protokol üretir. Araştırmacılar bir alternatif kaset sistemi kullanıyorsanız, onlar cihazlar oluştururken kullanılan birimleri tweak için gerekli bulabilirsiniz. Yüksek yoğunluklu jel bölge bu kasedi kenarlarından delaminate ve Elektroforez bölebilir kaset iç hava cepleri oluşturmak çok büyük ise imalat büyük mücadelesine bu olur. Birkaç farklı katman birimleri ile birkaç kaset döküm tarafından araştırmacılar hızlı bir şekilde onların belirli donanım için en uygun yapılandırma belirlemek gerekir.
Jel-seq protokolü de protokol tamamlanmadan önce doğrulanabilir birkaç kritik adımlar vardır. Bir potansiyel başarısızlık noktası gDNA ve RNA/cDNA melez ayrılmasıdır. Bu ayrılıktan sonra görüntüleme jel-seq cihazı tarafından onaylanabildiğini (bkz. şekil 3B). Deneyler bir dizi bulduk laboratuvar tekliflerimiz arabellek DNA ile kontamine ve önemli autoflourescence bizim cihazda neden oldu (bkz. şekil 3D). Bu ayırma yerini almıştı belirlemek zorlaştırdı. Floresan görüntüleme tanımlamak ve sıralama kitaplıkları oluşturmak için herhangi bir pahalı reaktifler kullanmadan önce bu sorunu gidermek yardım etti.
Başka bir kritik nokta 6.2, cDNA qPCR amplifikasyon ayırma sonra adımdır. Araştırmacılar RNA-seq verilerin kalitesini azaltır gibi bu adımda overamplify değil dikkatli dikkat etmelidir. Bu göz jeli-seq için benzersiz değil ama düşük-girdili RNA-seq Kütüphane hazırlık ortak bir yönü. PCR güçlendirme Kütüphane hazırlık sıralama sırasında genellikle gereklidir, ama sıra hataları ve önyargıları tanıtabilirsiniz. ÇSYİ devredir gerekli sayıda örnek miktar ve karmaşıklık bağlıdır. PCR döngüsü ne zaman belgili tanımlık kütüphaneci sıralanamadı yeterli kümeleme verim için çıplak en azından için sınırlamak için genel olarak tavsiye edilir. Teorik olarak, bir protokol aşırı eserler tanıtımı olmadan yeterli kopya sayı sayılardan tam döngüsü numarasını belirlemek için optimize edilebilir. Uygulamada, ancak, örnek kalite, tutarsızlık yükleme veya erken protokolünde kullanmada önemli ölçüde moleküler şablonları kütüphane hazırlık PCR, bu en iyi PCR döngüsü numarasını etkiler mevcut dağıtım etkileyebilir. En genel çözüm bulduk floresan bir boya kullanarak amplifikasyon reaksiyonlar ilerlemesini izlemek için tepkiler üzerinde gerçek zamanlı PCR thermocycler çalıştırın ve tepkileri üssünü (döngü sayısı karşı doğrusal) durdurmak faz. Deneyim, gerçek zamanlı izleme geliştirme, uyum veya yeni bir iletişim kuralı benimseyerek özellikle uygundur.
Son önemli adım genomik oluşturuyor ve transcriptomic kütüphaneler. Mümkün olduğunca yakın 0.2 ng/µL gibi (0,5 ng toplam) DNA Kütüphane hazırlık reaksiyon için başlangıç örnek toplama ayarlamak için bu adımı anahtarıdır. Bu genellikle cDNA aşırı, ama o-ebilmek var olmak daha gDNA örnekleri için zor için güçlendirilmiş qPCR cDNA nispeten basittir. Örnekleri yoğunlaşmıştı iken vacufuge adıma dikkat gerekli dikkatli bulduk. Deneylerde 1000 hücrelerle beklendiği gibi vacufuge adım kadar durdurulması 100 ile deneyler er hücreleri. Vacufuge örnekleri sayısı da buharlaşma hızı bizim deneylerde etkiledi. DNA doğrulamak için bir flourometer kullanan midway konsantrasyon adım aracılığıyla yabancı örnekleri protokolüyle gerçekleştirirken yardımcı olabilir içerik bulduk. Neyse ki, araştırmacılar üzerinde konsantre bir örnek, nükleaz ücretsiz su Eğer-ebilmek var olmak mülhak örnek sulandırmak için. Teorik olarak, DNA kuru ve sonra istenen hacmindeki resuspend vacufuge kullanmak mümkündür; Ancak, biz tam buharlaşma kaçınarak öneririz.
Biz aynı anda DNA ve RNA kitaplıkları, jel-seq7, G & T-seq8ve DR-seq9, ücretsiz olarak oluşturmak için üç geçerli yöntem görüntüleyin. Jel-seq 100-1000 hücre aralığı örnekleri için idealdir ve hiçbir açılan hedefler ya da genom kopyası bir temanın bilgi gerektirir. Diğer iki yöntem daha iyi tek hücre uygulamaları için uygundur. Bizim gol jel-seq gelişmekte olan araştırmacılar tarafından kolayca uygulanabilecek bir protokol yaratmaktı. Bu nedenle polyacrylamide jel kaset standart form faktörü içinde aygıt imal etmek karar verdik. Bizim farklı membranlar tanımlamak için kullanılan teknik roman olmakla birlikte, çoğu genetik labs zaten jel-seq aygıt imal etmek gerekli tüm donanıma sahip. Ayrıca, cihazın maliyeti önemsiz - sadece 5,25 $ için 12 örnekleri işleyebilir bir aygıt. Kütüphane hazırlık iletişim kuralı ticari reaktifler kullanarak herhangi bir kitaplık oluşturmak için toplam maliyeti yüksek olarak yani, dedi. Bizim reaktif maliyet örnek başına bütün-transkript amplifikasyon için 50 $ ve $28 DNA ve RNA Kütüphane hazırlık için yapıldı. Neyse ki, jel-seq aygıt iletişim kuralı agnostik olduğunu. Fareler doku örnekleri için uygun değildi bulundu, ancak örneğin, geliştirme sırasında başarıyla aygıtı hücre kültür ve bir büyük RNA Kütüphane amplifikasyon protokolü19, kullanarak test ettik. Daha ucuz alternatifler Kütüphane hazırlık için geliştirilen geleceğe baktığımızda, bizim iletişim kuralı bu yeni teknikler ile çalışmaya adapte edilebilir. Araştırmacılar kendi laboratuvarlarında jel-seq uygulamaya basit bulacaksınız inanıyoruz. Umarız bu teknolojinin hızlı benimsenmesine kolaylaştıracaktır.
Açıklamalar
Eş-kurucusu ve bilimsel Danışmanı Singlera genomik Inc KZ olduğunu
Teşekkürler
Bu iş için fon Üniversitesi San Diego Ulusal Bilim Vakfı lisansüstü araştırma bursları, NIH hibe sağlanan R01-HG007836 ve bilim, ICT ve gelecek planlama Korece Bakanlığı tarafından yapıldı.
Birkaç rakamları önceki sürümleri ilk "Hoople, G. ö ve ark basıldı Jel-seq: Bütün-genom ve yarı geçirgen hidrojel engelleri kullanarak eşzamanlı düşük-girdili DNA ve RNA Kütüphane hazırlık tarafından transcriptome sıralama. Laboratuar bir çip 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). " Laboratuar bir yonga üzerinde rakamlar bu yayındaki yeniden yaptırıma.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate - Corning HTS Transwell 96 well permeable supports - 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
Referanslar
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18(2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31(2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908(2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), at http://science.sciencemag.org/content/352/6293/1586 (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923(2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies. , Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017).
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- A Guide to Polyacrylamide Gel Electrophoresis and Detection. Bulletin 6040 Rev B. Bio Rad. , Available from: http://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6040.pdf (2017).
- Illumina. Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır