Method Article
凝胶序列: 一种同时测序库的方法用水凝胶基质制备 DNA 和 RNA
摘要
凝胶序列使研究人员能够同时为 DNA 和 RNA 的存储提供从 100-1000 细胞开始使用简单的水凝胶装置的可忽略的额外成本。本文给出了该装置的制作方法, 以及生成配对库的生物协议。
摘要
在过去的五年里, 从小的起始样本中放大和序列 DNA 或 RNA 的能力才得以实现。不幸的是, 生成基因组或 transcriptomic 库的标准协议是不相容的, 研究人员必须选择是否为特定的样本序列 DNA 或 RNA。凝胶序列解决了这个问题, 使研究人员可以同时准备的 DNA 和 RNA 的图书馆从 100-1000 细胞使用一个简单的水凝胶装置。本文给出了该装置的制作方法, 以及生成配对库的生物协议。我们设计的凝胶序列, 使它可以很容易地执行其他研究员;许多遗传学实验室已经有必要的设备, 以重现凝胶序列的设备制造。我们的协议使用常用的工具包进行全文本放大 (transcriptomic) 和库准备, 这对于已经精通生成基因组和库的研究人员也很有可能熟悉。我们的方法使研究人员能够在不分裂和微不足道的附加成本的情况下, 在单个样品上携带 DNA 和 RNA 序列的能量。
引言
下一代测序技术对遗传学研究的实施方式产生了深远的影响。研究人员曾经专注于对整个物种的基因组进行排序, 现在可以在一个实验中序列化单个肿瘤的基因组, 甚至单个细胞。1转录还使在单元格中找到的 RNA 记录集 (称为 "数据收集") 的序列化具有成本效益。在过去的五年里, 从小的起始样本中放大和序列 DNA 或 RNA 的能力才得以实现。2,3,4不幸的是, 标准协议是不兼容的, 研究人员必须选择是否为给定的样本序列 DNA 或 RNA。当一个起始样本足够大时, 它可以分成两半。然而, 在较小的尺度上, 由于分裂样本造成的材料损失会影响到图书馆的质量, 而样品的汇集可以平均出细胞之间的有趣变化。5此外, 研究人员越来越热衷于检查不能分割的样本, 如单细胞或小的异质肿瘤活检。6
为了解决这一问题, 最近开发了三项协议, 用于从同一起始样本中序列化 DNA 和 RNA: 凝胶顺序7、G & t-序列8和 DR-序列9。本文给出了凝胶序列的详细协议, 可用于同时生成少量100细胞的 DNA 和 RNA 库, 其成本可以忽略不计。凝胶序列的新方面是将 DNA 和 RNA 完全用低成本水凝胶基质分离出来的能力。凝胶序列协议的核心创新是 DNA 与 RNA 的物理分离。这种分离是实现电泳使用聚丙烯酰胺膜的组合, 利用这些分子之间的大小差异。要将这些大小差异放在上下文中, 请考虑 dna 和 RNA 是如何成像的: 虽然 dna 存在于微米尺度上, 可以用传统显微镜观察, 但 RNA 存在于纳米尺度上, 必须使用诸如低温电子等复杂技术进行成像。显微镜.10
在这个协议中分离 DNA 和 RNA 的方法如图 1所示。左面板显示 DNA 和 RNA 游离漂浮在溶液附近的膜。当电场被应用时, 如右面板所示, DNA 和 RNA 经历了一种电泳力, 通过细胞膜诱导迁移。通过调整膜的性能, 我们创造了一种半渗透膜, 将 DNA 与 RNA 分离。DNA 分子被推到膜上, 但由于它们的体积大而被边缘纠缠。小 RNA 分子, 另一方面, 可以重新配置和编织他们的方式通过膜。这个过程, 被称为蠕动, 类似于蛇在草丛中穿行的方式。最终, 这些 RNA 分子被第二个高密度膜所阻挡, 对于更小的聚合物 (> 200 基对) 来说, 这是非常困难的。一旦物理分离, DNA 和 RNA 可以被恢复和处理, 以产生关于基因组和转录的信息。虽然我们可以分离 DNA 和 rna, 我们发现更好的结果, 如果 RNA 反转转录为 cDNA 在分离之前。cDNA/rna 杂交种比 rna 更稳定, 还能通过低密度膜。

图 1.凝胶的操作原理.用于物理分离 DNA 和 RNA 的基本原则。在应用电场中, 小 RNA 分子通过低密度膜迁移, 但大量的 DNA 分子被困在表面。这个数字是从参考文献7中转载的, 得到了皇家化学学会的许可。请单击此处查看此图的较大版本.
本文详细介绍了凝胶序列器件的制备和生成配对 DNA 和 RNA 库的生物协议。这两者的概览都显示在图 2中。该装置是通过分层三种不同密度的聚丙烯酰胺凝胶在一个类似于创建标准的堆叠凝胶的过程中制造的。11生物协议从 PBS 中悬浮的 100-1000 细胞开始。细胞被裂解, RNA 被转化为 cdna, 然后用该装置将基因组 DNA 与 cdna/RNA 杂交体分离。在分离和恢复后, 基因组和 transcriptomic 库是使用一个密切遵循标准全基因组库准备套件协议的过程编写的。关于凝胶序列的发展和验证的进一步细节可以在实验室中读到一个芯片出版物"凝胶序列: 全基因组和转录测序通过同时低输入 DNA 和 RNA 库准备使用半透水水凝胶屏障."7

图 2.凝胶序列协议.概述了制造凝胶序列装置的步骤和生成配对 DNA 和 RNA 库的协议。这个数字的部分从参考文献7被复制了从皇家化学学会的允许。请单击此处查看此图的较大版本.
为了从单细胞中生成 DNA 和 rna 库, 研究人员应该考虑使用 g & t 序列或 DR-下克 & t 序列, 如凝胶序列, 依赖于从基因组 DNA 中提取 rna 的物理分离。这种方法依靠信使 RNA 的 (mRNA) 3′ polyadenylated 尾巴作为下拉目标。该 mRNA 使用生物素化寡核苷酸底漆在磁珠上捕获。一旦基因被捕获, 珠子就会被放置在一个磁铁上, 含有基因组 DNA 的上清液可以被移除并转移到另一管。在这种物理分离完成后, 可以从 mRNA 和 DNA 中生成独立的库。8如果感兴趣的 RNA 是 polyadenylated, 则此方法很有效, 但是它不能用于研究非 polyadenylated 转录, 如核糖体 rna、tRNA 或原核生物中的 rna。
DR 序列依赖于一个预放大的步骤, 从 RNA 中提取的 DNA 和 cDNA 在同一管中被放大。然后将该样本分成两个, 并并行处理以准备 DNA 和 RNA 序列库。为了区分基因组 DNA 和 RNA 衍生的 cDNA, DR 序列采用了计算方法。只有外显子存在的序列在基因组 DNA 数据中被计算抑制, 因为它们可能来源于 DNA 或 RNA。9这种方法的优点是, DNA 和 cDNA/RNA 不需要物理分离, 如在凝胶序列和 G & T。然而, 缺点是, 转录博士需要先验知识的基因组和 (即, 外显子与内含子), 并可能不理想的应用, 如核测序, 其中许多成绩单尚未完全拼接并仍包含内含子。12
凝胶序列的新的方面是能够分离 DNA 和 RNA 在数以百计的细胞完全基于大小。此方法不需要先验知识的基因组或转录, 是强大的反对不完全剪接, 并不仅限于多 adenylated 成绩单。对于研究人员可以从至少100个细胞开始的应用, 凝胶序列提供了一个简单的方法使用廉价和广泛可用的材料。
研究方案
1. 化学溶液制备
注: 以下步骤用于在以后的步骤中准备需要的化学溶液。这些可以批量制作, 储存几个月。
- 开始时, 用 254 nm UV 交联烤箱中的15分钟 (15 mJ/cm2总曝光) 杀菌, 制备50毫升纯净的去离子水, 以中和任何污染的 DNA。加热到37摄氏度, 以便在以下步骤中使用。
- 使10毫升的40% 总 (T) 和3.3% 交联剂 (C) (29:1) 聚丙烯酰胺前体溶液。重3.867 克丙烯酰胺单体和0.133 克的双丙烯酰胺单体。结合并使体积达到10毫升与温暖纯净水和漩涡, 直到溶解。储存在室温下保护免受光照。
注: 预混40% 吨, 3.3% C (29:1) 聚丙烯酰胺溶液可在商业上购买。 - 使10mL 的 50% T, 5% C 凝胶溶液。重4.750 克丙烯酰胺单体和0.250 克的双丙烯酰胺单体。结合和带来的体积高达10毫升使用温暖纯净水和漩涡, 直到溶解。储存在室温下保护免受光照。
- 使10毫升的 50% (瓦特/v) 蔗糖溶液。将5克蔗糖添加到一个已毕业的气缸中, 并将温纯净水添加至总容积10毫升。漩涡, 直到溶解和贮存在室温下。
- 使10毫升 10% APS (瓦特/v)。将1克过硫酸铵 (APS) 添加到一个已毕业的气缸。加入冷 (~ 4° C) 纯净的水由总容量10毫升和漩涡直到溶化。立即冻结在整除数200µL。
2. 胶体序列盒制作
注: 凝胶序列最初是用直立盒制作的 (请参阅材料表以了解更多信息);但是, 该协议可以适应任何标准的凝胶电泳盒。
- 通过添加试剂, 在三单独的塑料管中制备凝胶前体, 如下面的表1所示。不要添加 ap 或 Tetramethylethylenediamine (TEMED), 直到按以下步骤进行定向。涡流成分彻底混合。
| 填料凝胶前驱体 | 高密度凝胶前驱体 | 低密度凝胶前驱体 | |||
| 40%T, 3.3%C 丙烯酰胺 Bisacrylamide 溶液 | 1.6 毫升 | 50%T, 5%C 丙烯酰胺 Bisacrylamide 溶液 | 2.4 毫升 | 40%T, 3.3%C 丙烯酰胺 Bisacrylamide 溶液 | 0.6 毫升 |
| 去离子水 | 10.2 毫升 | 去离子水 | 1.0 毫升 | 去离子水 | 4.8 毫升 |
| 蔗糖溶液 (50% 瓦特/v) | 2.6 毫升 | 蔗糖溶液 (50% 瓦特/v) | 0.6 毫升 | ||
| 10X 三硼酸-EDTA | 1.6 毫升 | 10X 三硼酸-EDTA | 0.6 毫升 | ||
| 过硫酸铵 (10% 瓦特/v) | 104.0 µL | 过硫酸铵 (10% 瓦特/v) | 50.0 µL | 过硫酸铵 (10% 瓦特/v) | 39.0 µL |
| TEMED | 6.0 µL | TEMED | 1.0 µL | TEMED | 2.2 µL |
| 总容积 | 16.1 毫升 | 总容积 | 4.1 毫升 | 总容积 | 6.0 毫升 |
表1。凝胶合成试剂.聚丙烯酰胺凝胶前体试剂足以制造2盒磁带。
- 脱气凝胶单体解决方案通过插入一根针通过管帽, 并连接到一个房子的真空线针。将此组件浸入到高的超声波浴缸中, 等到气泡停止从液体中出现, 然后再移动到下一步 (~ 60 秒/管)。
注意: 只要气泡能从溶液中浮现出来, 超声波浴的真空和功率质量就不重要了。 - 将 TEMED 和 APS 添加到填料凝胶前驱体和旋涡中。立即添加6毫升的填料凝胶前体的每一个凝胶盒通过吹打的解决方案到盒顶部。使用1毫升微添加前体六增量, 以避免溢出。此步骤的总时间应少于3分钟。
- 通过将卡带直立放置在水平表上, 确保流体水平。用去离子, 脱气水填满卡带的其余部分。吸管的水, 再次使用1毫升的增量, 慢慢进入中心的卡带, 以尽量减少混合。允许聚合物固化至少一个小时, 直到一夜之间。
- 在聚合物固化后, 将凝胶盒在水槽上反转, 以除去水覆盖物。压缩空气枪可以用来轻轻地干燥界面, 通过吹空气通过顶部打开的卡带从6英寸的距离。
- 将 TEMED 和 APS 添加到高密度凝胶前驱体和涡流中。立即添加320µL 的高密度前驱物的卡带, 再次使用1毫升吸管。确保流体均匀涂层填充凝胶层通过摇摆磁带来回3倍左右。此步骤应少于三分钟。
- 用去离子, 脱气水填满卡带的其余部分。吸管慢慢地用1毫升的吸管进入卡带的中心, 以尽量减少混合。允许聚合物固化至少十五分钟, 最好是一个小时。
- 再次, 反转胶盒, 以消除水覆盖。压缩空气可以用来轻轻地烘干界面。将 TEMED 和 APS 添加到低密度凝胶前驱体和旋涡中。立即用低密度凝胶前驱体填充卡带 (~ 1.65 毫升) 的其余部分, 然后插入凝胶梳。
- 在梳子的顶端, 吸管的过剩的储备前驱, 因为它将被吸收在聚合过程中。允许聚合物固化至少4小时, 最好是过夜。凝胶在三硼酸-乙二胺 (EDTA) 缓冲液中可贮存一周或更多次。
3. 样品准备和反向转录
- 从感兴趣的细胞悬浮开始, 并在聚合酶链反应 (PCR) 层流罩中工作, 使用 hemocytometer 或自动细胞计数器计算细胞浓度。在磷酸盐缓冲生理盐水 (PBS) 中, 将细胞稀释成每µL 100 到1000个细胞的浓度。
注意: 此协议已在包括 PC3、HeLa 和小鼠肝细胞在内的一系列细胞中得到验证。 - 使用在µL 试剂盒中提供的试剂 (参见材料表), 混合19的裂解缓冲液和1µL 的 RNase 抑制剂, 以制备反应缓冲器的10X 库存溶液。创建一个包含0.5 µL 反应缓冲器和2.75 µL 核酸酶自由水的溶解主混合。
- 将细胞悬浮液移至5次, 以重新悬浮已沉淀的细胞, 然后将1µL 的样品注入到200µL 核酸酶自由条管中, 紫外线杀菌。根据样品的数量, 必要时重复。一定要包括一个负控制吹打1µL 核酸酶自由水, 而不是细胞的一个反应。接下来, 添加3.25 µL 的裂解主混合到每个样品和混合通过轻轻吹打上下5次。
- 预热热循环仪 (带加热盖子) 到72摄氏度。添加1µL 的 RT 底漆和1µL 20 µM 随机水溶液与 WTA 适配器 (5′ AAGCAGTGGTAT-CAACGCAGAGTAC-NNNNNN 3′) 的每个样本。保留至少一管作为一个积极的控制 gDNA 图书馆的准备和添加2µL 的水, 而不是底漆。
注: 带有水溶液适配器的随机使用是可选的, 对排序结果的影响最小。 - 在预热的热循环仪中孵育72摄氏度的样品, 3 分钟溶解细胞。从热循环仪中取出细胞, 在冰上放置2分钟。将正控制存储在4摄氏度, 直到步骤5。
- 当细胞株溶藻时, 为所有含有以下试剂比率的 RNA 样本创建一个足够数量的反向转录主混合体: 2 µL 第一链缓冲器、0.5 µL 模板开关寡核苷酸 (草)、0.25 µL RNase 抑制剂和1µL逆转录酶 (100 U/µL)。
- 预热热循环仪到42摄氏度。添加3.75 µL 反向转录主混合到其余的样本, 使总样本量为10µL. 混合吹打上下5次。
- 立即将样品放在预热的热循环仪中进行反向转录。运行以下程序:42 °c 为90分钟, 70 °c 为10分钟, 4 °c 永远。这是一个安全的停车点。
4. 凝胶分离和样品回收
- 使用 DNA 去除产品仔细清洁凝胶电泳室。将液体清洗剂的几个毫升应用于一次性的皮棉免费擦拭, 并在房间的所有表面擦拭, 然后用干净的0.5x 填充房间。为获得最佳效果, 请将整个设备放置在 254 nm UV 交联烤箱中, 并杀菌15分钟 (15 mJ/cm2)。
- 将凝胶序列盒插入凝胶电泳室并将其锁定到位。慢慢取出凝胶梳, 直拉。缓慢移动以避免撕裂凝胶或撕裂任何武器。
- 将样本从步骤3保留在冰上, 至少保留一个样本作为对 cDNA 文库生成的积极控制。将此控件存储在4摄氏度, 直到步骤6。将2µL 的6X 负载染料添加到剩余的样品中, 使总容积达到 ~ 12 µL. 通过吹打上下5次完全混合样品。
- 结合1µL 的 DNA 阶梯与2µL 的6x 负载染料和7µL 的水。将这种混合物注入凝胶序列盒中的1车道作为电泳控制。将前一步的样品移到凝胶序列盒的不同车道上。小心地防止水井之间的污染, 把吸管充分插入每个井, 并删除它只使用垂直运动。
- 使用标准的凝胶电泳电源, 在凝胶序列盒中应用 250 V 的电场30分钟, 将 gDNA 与 cDNA/RNA 混合体分开。一旦分离, 从凝胶电泳室中取出凝胶序列盒, 并用刮边工具撬开磁带的两半部分。
- 用手术刀, 把凝胶切成两半, 就在高密度层下面。用戴手套的手捡起来, 丢弃含有填充胶的那一半。用刮刀或其他类似的工具, 轻轻地剥开卡带上剩下的凝胶。将凝胶的这一部分放入含有30毫升0.5x 的盘中, 3 µL 凝胶染色。
- 盖上容器, 尽量减少漂白和浸泡凝胶, 同时轻轻摇动容器5分钟。将凝胶放在塑料包装上, 并使用凝胶文档系统进行 UV 图像 (有关进一步详细信息, 请参阅参考文献 13)。30秒的曝光通常会产生清晰的图像。验证是否已发生分离。
- 将凝胶移至紫外线 transilluminator, 以促进核酸的可视化。佩戴合适的 UV 眼镜, 确认凝胶文件系统的结果。gDNA 应位于低密度凝胶的起始处和低密度高密度区域界面的 cDNA。
- 使用手术刀, 切除含有 gDNA 和 cDNA 的凝胶区域。最好通过切割4毫米10毫米的凝胶矩形段来回收样品;然而, 确切的几何将取决于使用的凝胶电泳系统。记住还要切断带有负控制的车道。
- 使用一对钝端镊子将凝胶的每个切除部分放入条带管中。小心不要施加太多的力量或凝胶将分裂成多个片断。如果发生这种情况, 只需拿起每一块, 并添加到管。
- 用吸管的尖端 (200 µL 吸管提示) 在每管中研磨凝胶, 将吸管尖端以圆形的方式移动到管子的底部。将核酸酶免费水 (40 µL 进入 gDNA 样品和80µL 到 cDNA 样品中), 然后除去用于研磨凝胶的吸管尖端, 以减少样品损失。
- 把带管的漩涡搅拌机内的37°c 孵化器和震动 8-12 小时。这使得核酸能从凝胶中扩散出来, 是这个多天协议的自然停止点。
- 将样品注入8µm 网格过滤板, 在 2600 x g 处旋转板5分钟以应变出凝胶碎片。将网格过滤板从外壳板上提起, 将无凝胶水样注入新的200µL 条管。
- 添加1µL 蛋白酶 (0.9 AU/毫升) 的每个样本包含 gDNA, 混合良好的吹打上下, 并孵化50°c 15 分钟后, 热失活在70°c 15 分钟。此步骤对耗尽核非常重要, 并使 gDNA 可用于后续反应步骤。
- 使用18口径的针, 戳孔在所有样品管的上限。将样品放在 vacufuge 中以减少液体体积。gDNA 样品应减少到5µL 和 cDNA 样本减少到10µL。
- 根据样品的 vacufuge 和数量, 总蒸发时间将在30和60分钟之间变化。如果样品体积低于目标体积, 只需添加核酸酶自由水增加样本量。
5. gDNA 图书馆的准备工作
- 使用荧光计或类似技术, 从步骤4中量化每个 gDNA 样本中的 DNA 浓度, 以及步骤3中的正向控制。有关详细协议, 请参阅荧光计参考手册。14
- 将样品稀释到0.2 µL 的 DNA。根据所需的起始细胞类型和结果质量, 较低浓度仍可能产生可行的库。将需要进行一些实验;然而, 作者的成功与图书馆的低至 0.1 ng/µL。
- 完成 gDNA 库准备工作, 按照库准备步骤7中的半反应卷协议。
6. cDNA 文库准备
- 从步骤4开始, 10 µL cDNA 样本和阳性对照, 添加12.5 µL 2X qPCR 混合, 0.5 µL cDNA PCR 底漆, 2 µL 无核酸酶水。
- 使用以下协议在实时 thermocycler 中执行 PCR: 热启动在95°c 为3分钟, 其次20-30 个周期98°c 为十年代, 65 °c 三十年代和72°c 为3分钟. 总样本量为25µL. 监测反应曲线, 停止放大前e 反应离开指数相位 (线性信号增加与周期数), 以避免 PCR 工件由于 overamplification。有关避免 overamplification 的更多内容, 请参见本文的讨论部分, 以及参考文献15。
- 放大后, 使用固相可逆固定 (SPRI) 珠在步骤8中的协议后清洁产品。完成后, 继续执行下一步。
- 使用荧光计或类似技术, 量化每个 cDNA 样本中的 DNA 浓度以及步骤4中的阳性对照。如果需要, 稀释样品, 以包含大约0.2 µL 的 DNA。稍低的浓度仍然产生可行的库。一些实验将是必要的, 然而作者有成功与图书馆低到 0.1 ng/µL。
- 可选步骤: 对每 qPCR 产品的 1-2 µL 进行标准的聚丙烯酰胺凝胶电泳分离, 以验证库代反应工作。图 4中显示了成功结果的示例图像。有关如何进行聚丙烯酰胺凝胶电泳的详细协议, 请参阅参考文献16。
- 完成 cDNA 库准备工作, 遵循库准备套件在步骤7中的半卷反应协议。
7. 具有半容积反应的图书馆准备工作
- 图书馆准备工作遵循图书馆准备套件协议使用半容积反应。17协议本节中引用的所有试剂都来自库准备工具包 (请参阅材料表)。开始由紫外线杀菌足够数量的带管的数量的样品要处理。
- transposase 反应通过增加5µL transposase 缓冲到每条管将用于化验。然后添加2.5 µL 输入 DNA 0.2 ng/µL (0.5 吴总) 后跟2.5 µL transposase。混合吹打上下5次。
- 孵育55°c 5 分钟, 然后保持在10摄氏度。样品达到10摄氏度后, 将其从 thermocycler 中取出, 并立即向每个样品添加2.5 µL transposase 停止缓冲器。把样品放在室温下5分钟。
- 通过添加7.5 µL 库准备 pcr 组合, 2.5 µL 指数1底漆, 2.5 µL 指数2底漆, 制备 pcr 反应放大转置处理样品。这些底漆是专有的, 由图书馆准备工具的制造商提供。混合吹打上下5次。
注: 确保每个样品使用独特的底漆组合。有12不同的索引1引物和8不同的索引2底漆, 使成为可能唯一标签多达96个不同的样品。为每个样品选择一个独特的底漆组合。 - 使用以下程序在 thermocycler 上执行 PCR。样品体积为25µL。
72°c 3 分钟
95°c 30 秒
12周期:
95°c 10 秒
72°c 30 秒
55°c 30 秒
72°c 5 分钟
保持在10°c - 按照步骤8中的协议, 使用 SPRI 珠子清理准备好的库。这些图书馆应该使用凝胶电泳或类似的化验来验证。有关如何验证库的详细信息, 请参阅库准备工具包手册。17
8. 固相可逆固定化珠库清洗
- 固体相可逆固定化 (SPRI) 珠应储存在1.5 毫升整除数, 并应带到室温, 每次使用。还建议为每个实验准备新鲜的80% 乙醇。以下步骤基于 "SPRI" 中的 "球珠协议" 手册。18
- 在每种 PCR 产品中添加1µl 的包裂解缓冲液。涡流 SPRI 珠均匀混合, 然后添加50µl 的 SPRI 珠每个样品。混合通过吹打样品上下10次, 然后在室温下孵化样品8分钟。
- 简单地旋转样品, 从管子的侧面收集液体。将样品放在磁分离装置上5分钟, 直到液体完全清晰为止。
- 当样品在磁性分离装置, 慢慢地去除上清和放弃-小心不要打扰在管子的珠子圆环。接下来添加200µL 80% 乙醇的每个样品, 而不干扰珠子。等待30秒钟, 然后仔细地吸出上清。重复此步骤 (乙醇洗涤) 一次。
- 允许样品干燥三十年代-1 分钟。不要过度干燥的样品, 因为大碎片将成为永久性绑定到珠子。
注意: 建议简单的干燥时间, 以确保所有的乙醇的痕迹被删除。如果微量的乙醇留在后面, 它们可以略微抑制下游反应。 - 从磁分离装置中取出样品, 并将15µL 的水添加到每个样品中, 以洗脱珠子上的 DNA。吸管向上和向下, 并确保珠子从管的两侧被删除。在室温下孵育2分钟。简单地旋转样品, 然后将它们放在磁分离装置上1分钟, 直到溶液看起来清晰为止。
- 当样品在磁分离装置上时, 慢慢地将上清液吸干, 并将其转移到清洁管上。小心不要打扰管上的珠子环。用珠子把管子扔掉。
结果
通过荧光凝胶成像, 可以直观地分析凝胶序列中 gDNA 和 cDNA/RNA 混合体的物理分离;有代表性的结果显示在图 3中。面板 A 显示制备的凝胶序列装置;添加了假颜色来区分不同的凝胶区域。面板 B 显示了用于验证的四种不同分色的特写。第三条车道, 一个负控制, 代表背景, 并表明没有 autoflourescence 的凝胶在界面上。我们用 DNA 梯子装载了第一和第二条车道。这些车道只显示在低密度和高密度膜之间的界面上的暗带, 揭示小碎片可以通过低浓度凝胶。第四条车道显示了一个生物样本的行为: 500 PC3 细胞。我们载入了《议定书》步骤3所述的四车道。图像显示基因组 DNA 和 cDNA/RNA 杂交的分离。低密度膜顶部的深色带是 megabase 级基因组 DNA, 而 cDNA/RNA 混合体则堆积在低密度和高密度区域的界面上。与装有梯子的车道不同的是, 在凝胶的高密度区域内也有几个带。这些碎片, 小于 100 bp, 是在反向转录过程中由底漆寡核苷酸产生的目标产物。面板 C 显示了一个典型的形象, 整个凝胶序列设备从一个成功的实验。用凝胶序列法处理标记为 rna/cDNA 的车道, 而标记 rna 的车道则显示出仅 gDNA 和 rna 的分离。面板 D 显示了一个失败的实验, 黑带在每个车道的低密度膜的顶部。这是由于电泳缓冲液污染了碎片 DNA。在这一步, 研究人员应该寻找干净的阴性对照和两个不同的黑带表明存在的分离 gDNA 和 RNA/cDNA 杂交。

图 3.凝胶序列分离结果.凝胶序列装置 (A) 和荧光图像, 显示分离的 DNA 和 RNA/cDNA 杂交 (B)。假颜色添加到更容易区分不同区域的密度在凝胶内。(C 和 D)从成功的 (C) 和失败的 (D) 实验中, 代表整个凝胶序列装置的荧光图像。NTC = 没有模板控件。这个数字的部分从参考文献7被复制了从皇家化学学会的允许。请单击此处查看此图的较大版本.
一旦 DNA 和 RNA/cDNA 混合体被分离, 凝胶序列的其余部分完成, 就有可能生成测序库。为了验证准备好的库, 我们运行的是标准的凝胶电泳实验 (图 4) 或 bioanalyzer。图 4中的结果显示由500个 PC3 单元格和750个 HeLa 单元格生成的库。该图显示了由凝胶序列 (标记为 "凝胶") 生成的匹配库的片段分布, 与标准协议 (标记为 "管") 生成的不匹配的样本相比较。在使用标准全基因组库准备套件准备库时, 凝胶序列的片段大小出现在200和 800 basepairs 之间。如果此步骤中的库片段未显示在正确的大小范围内, 则库准备工作失败。
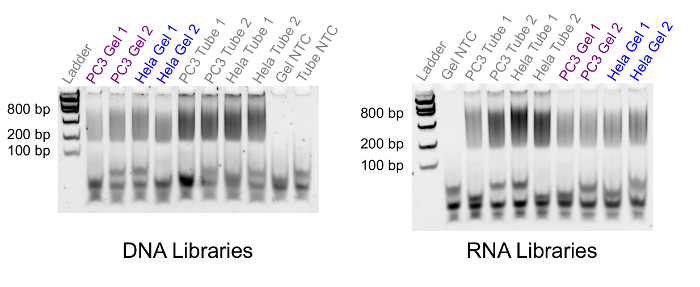
图 4.库片段大小比较.一种荧光凝胶电泳图像, 比较了凝胶序列 (凝胶) 和标准控制 (管) 之间的库大小分布。左车道包含一个低质量的 DNA 梯子与片断大小 100, 200, 400, 800, 1200 和 2000 basepairs。所有库的碎片大小都介于200到 800 basepairs 之间, 这与使用此工具包编写的库的预期相同。请单击此处查看此图的较大版本.
在对测序结果进行分析的基础上, 对凝胶序列协议进行了最终验证。我们为我们的验证实验选择了 PC3 细胞, 因为这些细胞具有同质表达谱, 允许用凝胶和传统方法分离和加工样品;请参见图 5。PC3 细胞基因组 DNA 的比较显示在图 5A和5B中。图 5A显示使用凝胶序列或标准全基因组库准备反应 (管控) 从 PC3 生成的全基因组拷贝数变异 (复制) 配置文件的比较。每个点都是平均归一化的 bin 计数;垃圾桶是从参考基因组数据定义的, 这样每个 bin 在一个健康的双倍细胞中都有相等的预期计数,即, 一条平坦的线, 代表所有常染色体 (不包括 X 和 Y) 染色体的每个区域的相等拷贝。PC3 包含同一地区的多个副本, 这些区域显示为两个背景副本数以上的峰值。凝胶序列产生一个定性地相似的被拷贝的外形作为标准管反应。两个地块之间的协议可以通过线性回归定量评估, 如小组 B 所示。0.90 的皮尔逊相关性表明, 从这两种方法中收集的基因组数据在功能上是等价的。

图 5.库验证.对基因组 (A 和 B) 和 transcriptomic (C、D 和 E) PC3 和 Hela 细胞生成的数据进行凝胶序列验证。面板 a 显示了用凝胶序列 (凝胶序) 或标准反应 (管) 从 PC3 生成的全基因组的可转换剖面的比较。MAPD = 中位绝对配对差异。面板 B 是面板 a 中两个样本之间的线性回归, R = 0.90 表示基因组数据在功能上等同。坐标轴显示 log2 规范化的 bin 计数。C 和 D 面板将 transcriptomic 数据与 PC3 单元格进行比较, 每个点显示每 kilobase 每百万 (TPM) 的记录数。坐标轴显示两个样本之间的比较 log2 规范化的成绩单计数。面板 C 显示了使用凝胶序列产生的技术复制的比较, 面板 D 显示了凝胶序列和传统的 rna-后面板 E 的比较表明, 凝胶序列可以通过主成分分析来解决基于 RNA 表达的细胞类型。x 轴显示第一个主体组件占样本之间的变化的 91.6%, 而 y 轴显示第二个主体组件仅占方差的6.5%。这个数字的部分从参考文献7被复制了从皇家化学学会的允许。请单击此处查看此图的较大版本.
同样, 我们将 transcriptomic 数据从我们的凝胶序列协议与标准管式智能序列协议进行了比较。图 5显示了凝胶序列技术复制 (图 5C) 和凝胶序列与标准方法 (图 5D) 之间的相关性。每个点都是每 kilobase 每百万 (tpm) 的记录数, 每个数据都在 tpm > 5 中检测到的每个基因。线性回归显示为红色线, 皮尔逊相关系数显示在左上角。技术复制从凝胶序列同意 (r ∼ 0.8), 但与标准方法比较不太好 (r < 0.7)。这表明凝胶序列引入了基因计数的偏倚。幸运的是, 这种偏差是系统性的, 正如图 5E中的主成分分析所看到的那样, 在不同的生物样本之间仍然可以得出有意义的结论。
讨论
有几个关键步骤相关的凝胶序列设备的制造以及协议本身。在制作过程中, 我们建议从规定的层厚度开始, 为凝胶的各个区域。我们花了大量的时间测试不同的制造选项, 此处描述的协议为材料和试剂表中列出的磁带制作最佳设备。如果研究人员使用另一种卡带系统, 他们可能会发现有必要调整在创建设备时使用的卷。制造中的主要挑战是, 如果高密度凝胶区域太大, 它可以从卡带的边缘分层, 在卡带的内部制造气囊, 从而扰乱电泳。通过铸造几个不同层卷的磁带, 研究人员应该能够快速确定其特定硬件的最佳配置。
凝胶序列协议还有几个关键步骤, 可以在协议完成之前进行验证。一个潜在的故障点是分离 gDNA 和 RNA/cDNA 杂交。这可以通过在分离后对凝胶序列进行成像来验证 (请参见图 3B)。在一组实验中, 我们发现实验室提供的缓冲区已被 DNA 污染, 并在我们的设备中造成大量的 autoflourescence (请参见图 3D)。这使得很难确定分离是否发生。荧光成像帮助我们识别和纠正这个问题之前, 使用任何昂贵的试剂来生成排序库。
另一个关键点是步骤 6.2, 分离后 cDNA 的 qPCR 扩增。研究人员应该仔细注意不要在这个步骤中 overamplify, 因为这样会降低 RNA 序列数据的质量。这种考虑不是凝胶序列的唯一性, 而是低输入 RNA 序列库准备的一个常见方面。在测序库准备过程中, PCR 放大往往是必要的, 但可以引入序列误差和偏差。PCR 周期的要求数量取决于样品数量和复杂度。通常建议将 PCR 循环数限制为在库排序时产生足够的聚类所需的最小极小值。理论上, 可以对协议进行优化, 以确定在不引入过多工件的情况下产生足够的拷贝数的确切周期数。然而, 在实际中, 在样本质量, 加载, 或处理的不一致在早期的协议可能会极大地影响到分子模板的分布, 可用于图书馆准备 pcr, 这反过来影响最佳 PCR 周期数。我们发现的最普遍的解决方案是用荧光染料来监测放大反应的进展, 在实时 PCR thermocycler 上运行反应, 并停止指数 (线性与周期数) 相的反应。在我们的经验中, 实时监视在开发、调整或采用新的协议时尤其重要。
最后一个关键步骤是生成基因组和 transcriptomic 库。此步骤的关键是将 DNA 库准备反应的起始样本浓度设置为接近0.2 µL (0.5 ng 总)。这是相对简单的 qPCR 扩增 cdna, 因为通常有过量的 cdna, 但它可以更具挑战性的 gDNA 样本。我们发现在样品集中时需要仔细注意 vacufuge 步骤。正如预期的那样, 在1000细胞的实验中, vacufuge 的步骤比100细胞的实验要快得多。vacufuge 中的样品数量也影响了我们实验中的蒸发速率。我们发现, 使用 flourometer 来验证 DNA 含量中途通过浓度的步骤可能是有益的, 当执行协议与不熟悉的样本。幸运的是, 如果研究人员集中了一个样品, 核酸酶自由水可以添加到稀释样品。理论上, 可以使用 vacufuge 来干燥 DNA, 然后并用重悬在所需体积内;但是, 我们建议避免完全蒸发。
我们查看三种当前生成 DNA 和 RNA 库的方法, 凝胶序列7, G & T 序列8和 DR 序列9, 作为免费的。凝胶序列是理想的样品在 100-1000 细胞范围内, 并要求没有下拉目标或先验知识的基因组。另外两种方法更适合于单细胞应用。我们开发凝胶序列的一个目标是创建一个可以很容易地由其他研究人员执行的协议。因此, 我们决定在聚丙烯酰胺凝胶盒的标准形式因子内制造设备。虽然我们用来定义不同膜的技术是新颖的, 但大多数遗传学实验室已经拥有了制造凝胶序列装置所需的所有设备。此外, 该设备的成本是微不足道的-只有5.25 美元的设备, 可以处理12样品。这表示, 与任何使用商业试剂的图书馆准备协议一样, 生成图书馆的总体成本仍然很高。我们的试剂成本每样品为50美元的全转录放大和28元为 DNA 和 RNA 的图书馆准备。幸运的是, 凝胶序列设备本身就是协议不可知论者。例如, 在开发过程中, 我们使用来自区域性的单元格和较早的 RNA 库放大协议19成功测试了该设备, 尽管我们发现它不适用于小鼠的组织样本。展望未来, 作为更便宜的图书馆准备替代品的开发, 我们的协议可以适应这些新的技术工作。我们相信研究人员会发现在他们自己的实验室里实施凝胶序列是很简单的。我们希望这将有助于迅速采用这项技术。
披露声明
KZ 是 Singlera 基因组公司的共同创始人和科学顾问。
致谢
这项工作的经费由圣地亚哥大学、国家科学基金会研究生研究金方案、NIH 赠款 R01-HG007836 以及韩国科学、信息和通信技术和未来规划部提供。
早期版本的几个数字首次发表在 "Hoople, 动力局等。凝胶序列: 用半透水水凝胶屏障同时进行低输入 DNA 和 RNA 文库制备的全基因组和转录测序。实验室在芯片 17, 2619-2630, 对: 10.1039/c7lc00430c (2017)。芯片上的实验室已经批准了这个出版物中的数字重用。
材料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate - Corning HTS Transwell 96 well permeable supports - 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
参考文献
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18(2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31(2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908(2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), at http://science.sciencemag.org/content/352/6293/1586 (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923(2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies. , Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017).
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- A Guide to Polyacrylamide Gel Electrophoresis and Detection. Bulletin 6040 Rev B. Bio Rad. , Available from: http://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6040.pdf (2017).
- Illumina. Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。