Method Article
Gel-seq : PROCEDE de Preparation de bibliothèque simultanée de séquençage de l’ADN et l’ARN à l’aide de Matrices d’Hydrogel
Dans cet article
Résumé
Gel-seq permet aux chercheurs de préparer simultanément les bibliothèques pour les deux ADN - et RNA-seq au surcoût négligeable à partir de 100-1000 cellules à l’aide d’un dispositif simple hydrogel. Cet article présente une approche détaillée pour la fabrication de l’appareil ainsi que le protocole biologiques pour générer des bibliothèques appariés.
Résumé
La capacité d’amplifier et de séquences d’ADN ou ARN de petits échantillons de départ est parvenue seulement dans les cinq dernières années. Malheureusement, les protocoles standard pour la production génomique ou bibliothèques de transcriptomique sont incompatibles et chercheurs doivent choisir de séquence d’ADN ou ARN pour un échantillon donné. Gel-seq résout ce problème en permettant aux chercheurs préparer simultanément des bibliothèques pour les ADN et d’ARN à partir de 100-1000 cellules à l’aide d’un dispositif simple hydrogel. Cet article présente une approche détaillée pour la fabrication de l’appareil ainsi que le protocole biologiques pour générer des bibliothèques appariés. Nous avons conçu le Gel-seq afin qu’il pourrait être facilement mis en œuvre par d’autres chercheurs ; de nombreux laboratoires de génétique ont déjà l’équipement nécessaire pour reproduire la fabrication de dispositifs de Gel-seq. Notre protocole emploie couramment utilisé des kits pour les deux ensemble-transcription amplification (WTA) et préparation de bibliothèque, qui sont également susceptibles d’être familière aux chercheurs déjà versé dans la génération génomique et transcriptomique bibliothèques. Notre approche permet aux chercheurs de mettre à profit la puissance du séquençage de l’ADN et l’ARN sur un seul échantillon sans le découper et avec surcoût négligeable.
Introduction
Prochaine génération séquençage (NGS) a eu un impact profond sur le déroulement de la recherche en génétique. Là où autrefois, chercheurs se sont penchés sur le séquençage du génome d’une espèce entière, il est maintenant possible de séquencer le génome d’une tumeur unique ou même une seule cellule dans une expérience. 1 NGS a également rendu rentable pour séquencer les transcriptions d’ARN présentes dans une cellule, une collection de données appelées le transcriptome. La capacité d’amplifier et de séquences d’ADN ou ARN de petits échantillons de départ est parvenue seulement dans les cinq dernières années. 2 , 3 , 4 malheureusement, protocoles standard sont incompatibles et chercheurs doivent choisir de séquence d’ADN ou ARN pour un échantillon donné. Lorsqu’un échantillon de départ est assez grand, il peut être divisé en deux. Toutefois, à des échelles plus petites, perte de matériel en raison de la séparation des échantillons peut affecter la qualité de la bibliothèque et mise en commun des échantillons peut moyenne des variations intéressantes entre les cellules. 5 par ailleurs, les chercheurs sont plus en plus intéressés à l’examen d’échantillons qui ne peut pas être divisés, comme des cellules individuelles ou des biopsies de petite tumeur hétérogène. 6
Pour résoudre ce problème, trois protocoles ont récemment été mis au point pour séquencer l’ADN et l’ARN de l’échantillon de départ même : Gel-seq7, G & T-seq8et9de la DR-seq. Cet article présente un protocole détaillé pour Gel-seq, qui peut être utilisé pour générer simultanément bibliothèques d’ADN et d’ARN à partir d’aussi peu que 100 cellules au surcoût négligeable. La nouveauté de Gel-seq est la capacité de séparer l’ADN et l’ARN fondée exclusivement sur la taille à l’aide de matrices de faible coût hydrogel. L’innovation de base du protocole Gel-Seq est la séparation physique de l’ADN de l’ARN. Cette séparation se fait par électrophorèse en utilisant une combinaison des membranes de polyacrylamide qui tirent parti de la différence de taille entre ces molécules. Pour mettre ces différences de taille en contexte, examiner comment l’ADN et l’ARN sont imagés : alors que l’ADN existe sur l’échelle de micron et peut être consulté à l’aide de microscopes traditionnels, ARN existe à l’échelle du nanomètre et doit être copié à l’aide de techniques complexes comme cryo-electron microscopie. 10
L’approche à la séparation de l’ADN et l’ARN dans le présent protocole est illustré à la Figure 1. Le panneau de gauche montre l’ADN et l’ARN libre flottant dans la solution près d’une membrane. Lorsqu’un champ électrique est appliqué, comme indiqué dans le panneau de droite, ADN et l’ARN l'expérience une force électrophorétique qui induit la migration à travers la membrane. En ajustant les propriétés de la membrane, nous avons créé une membrane semi-perméable qui sépare l’ADN de l’ARN. Les molécules d’ADN sont poussés contre la membrane, mais sont accrocher au bord à cause de leur grande taille. En revanche, les petites molécules d’ARN, peuvent reconfigurer et tissent leur chemin à travers la membrane. Ce processus, connu comme la reptation, est semblable à la manière de qu'un serpent se déplace dans l’herbe. Finalement, ces molécules d’ARN sont arrêtés par une deuxième membrane haute densitée qui est trop difficile pour les polymères encore plus petits (> 200 paires de bases) de se soustraire à travers. Une fois physiquement séparés, l’ADN et l’ARN peuvent être récupérées et traitées pour générer des informations sur le génome et le transcriptome. Alors que nous pouvons séparer les ADN et ARN, nous avons identifié les meilleurs résultats sont obtenus si l’ARN est inverse, transcrit d’ADNc avant la séparation. Les hybrides de cDNA/ARN sont plus stables que RNA seule et peuvent toujours passer à travers la membrane de faible densitée.

Figure 1 . Principe de fonctionnement de gel-seq. Le principe sous-jacent utilisé pour séparer physiquement les ADN et ARN. Dans un champ électrique appliqué, petites molécules d’ARN migrent à travers la membrane de faible densitée, mais grosses molécules d’ADN sont pris au piège à la surface. Ce chiffre a été reproduit de Réf. 7 avec la permission de la Royal Society of Chemistry. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Cet article décrit en détail les deux la fabrication de l’appareil de Gel-seq et le protocole biologiques pour générer jumelé bibliothèques d’ADN et d’ARN. Un aperçu des deux est illustré à la Figure 2. Le dispositif est fabriqué par marcottage trois densité différente des gels de polyacrylamide uns sur les autres dans un processus similaire à la création des gels empilables standard. 11 le protocole biologique commence par 100-1000 cellules suspendues dans du PBS. Les cellules sont lysées et l’ARN est convertie en ADNc avant que l’appareil est utilisé pour séparer l’ADN génomique des hybrides cDNA/ARN. Après la séparation et de récupération, génomique et transcriptomique bibliothèques sont préparés à l’aide d’un processus qui suit de près le protocole de kit de préparation de bibliothèque standard du génome entier. Plus de détails sur le développement et la validation de Gel-seq peut être lu dans son laboratoire sur une publication de la puce « Gel-seq : génome entier et le séquençage de transcriptome de faibles intrants ADN et l’ARN bibliothèque préparation simultanée à l’aide de barrières semi-perméables hydrogel ." 7

Figure 2 . Protocole gel-seq. Une vue d’ensemble des étapes pour fabriquer le dispositif Gel-seq et le protocole à généré jumelé bibliothèques d’ADN et d’ARN. Certaines parties de cette figure ont été reproduits à partir Réf. 7 avec la permission de la Royal Society of Chemistry. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Pour générer des bibliothèques de l’ADN et l’ARN de cellules individuelles, les chercheurs doivent envisager d’utiliser soit G & T-seq ou DR-suiv. G & T-Seq, comme Gel-seq, repose sur une séparation physique de l’ARN de l’ADN génomique. Cette approche s’appuie sur l’ARN messager (ARNm) 3′ polyadénylé queue comme une menu déroulant cible. L’ARNm est capturée sur une pastille magnétique utilisant une amorce d’oligo-dT biotinylé. Une fois l’ARNm a été capturée, les perles sont maintenus en place avec un aimant et le surnageant contenant l’ADN genomic peut être enlevé et transféré dans un autre tube. Après que cette séparation physique est terminée, bibliothèques distinctes peuvent être générés de l’ARNm et l’ADN. 8 cette approche fonctionne bien si l’ARN d’intérêt est polyadénylé, cependant il ne peut pas servir à étudier non polyadénylé transcriptions, tels que les ARN ribosomiques, ARNt ou l’ARN des procaryotes.
DR-seq s’appuie sur une étape de pré amplification où l’ADN et ADNc dérivé de l’ARN sont amplifiés dans le même tube. L’échantillon est alors divisé en deux et traitée en parallèle de préparer des bibliothèques d’ADN - et RNA-seq. Pour distinguer l’ADN génomique et l’ADNc dérivé de RNA, DR-seq adopte une approche computationnelle. Séquences où se trouvent seulement les exons sont supprimés par le calcul dans les données d’ADN génomiques, comme ceux qui pourraient provenir de l’ADN ou ARN. 9 un avantage de cette approche est que l’ADN et l’ARNC/ARN ne doivent pas être physiquement séparés comme cela se fait en Gel-seq et G & T-suiv. L’inconvénient, cependant, est que DR-seq nécessite une connaissance a priori du génome et du transcriptome (c.-à-d., les exons et introns) et qu’il n’est peut-être pas idéal pour des applications telles que le séquençage de noyaux, dans lequel plusieurs transcriptions ne sont pas encore pleinement épissé et contiennent encore des introns. 12
La nouveauté de Gel-seq est la capacité de séparer l’ADN et l’ARN dans des centaines de cellules basées exclusivement sur la taille. Cette méthode nécessite aucune connaissance a priori du génome ou du transcriptome, est robuste contre l’épissage incomplète et n’est pas limitée aux transcriptions de poly-adenylated. Pour les applications où un chercheur peut commencer avec au moins 100 cellules, Gel-seq offre une approche simple à l’aide de matériaux bon marché et largement disponible.
Protocole
1. préparation de la Solution produits chimiques
Remarque : Les étapes suivantes sont pour la préparation de solutions chimiques requises dans les étapes ultérieures. Ceux-ci peuvent être faits en vrac et stockés pendant plusieurs mois.
- Pour commencer, préparez 50 mL d’eau purifiée, déminéralisée par stérilisation dans un four de réticulation UV à 254 nm pendant 15 min (15 exposition totale mJ/cm2 ) neutraliser tout ADN contaminant. Chauffer à 37 ° C pour une utilisation en suivant les étapes.
- Prendre 10 mL d’un 40 % total (T) et 3,3 % RETICULATION (C) (29 : 1) solution de précurseur de polyacrylamide. Peser 3,867 g de monomère d’acrylamide et 0,133 g de monomères de bis-acrylamide. Porter le volume à 10 mL avec l’eau purifiée et vortex jusqu'à dissolution. Boutique abri de la lumière à température ambiante.
NOTE : Prémélangée 40 % T, solutions de polyacrylamide 3,3 % C (29 : 1) peuvent être achetées dans le commerce. - Faire 10mL de 50 % T, solution de gel 5 % C. Peser 4,750 g de monomère d’acrylamide et 0,250 g de monomères de bis-acrylamide. Porter le volume à 10 mL à l’aide de l’eau purifiée et vortex jusqu'à dissolution. Boutique abri de la lumière à température ambiante.
- Prendre 10 mL d’une solution de saccharose 50 % (p/v). Ajouter 5 g de saccharose d’une éprouvette graduée et ajouter de l’eau purifiée chaude jusqu'à un volume total de 10 mL. Vortex jusqu'à dissolution et conserver à température ambiante.
- Faire 10 mL de 10 % (p/v) d’APS. Ajouter 1 g de persulfate d’ammonium (APS) dans une éprouvette graduée. Ajouter froid (~ 4° C) purifié l’eau jusqu'à un volume total de 10 mL et vortex jusqu'à dissolution. Congeler immédiatement en aliquotes de 200 µL.
2. gel-seq Cassette Fabrication
Remarque : Gel-seq a été développé avec des cassettes verticale (voir Table des matières pour plus d’informations) ; Toutefois, le présent protocole peut être adapté pour fonctionner avec n’importe quel cassette d’électrophorèse de gel standard.
- Préparer gel précurseurs dans des tubes en plastique trois distincts en ajoutant des réactifs comme indiqué ci-dessous en tableau 1. Ne sont pas ajouter APS ou tétraméthyléthylènediamine (TEMED) jusqu'à ce que réalisé dans les étapes suivantes. Ingrédients de vortex pour bien mélanger.
| Remplissage Gel précurseur | Précurseur de Gel haute densité | Faible densité Gel précurseur | |||
| 40 %T, 3.3%C Acrylamide Bisacrylamide Solution | 1,6 mL | 50 %T, 5 %C Acrylamide Bisacrylamide Solution | 2,4 mL | 40 %T, 3.3%C Acrylamide Bisacrylamide Solution | 0,6 mL |
| Eau désionisée | 10,2 mL | Eau désionisée | 1,0 mL | Eau désionisée | 4,8 mL |
| Une Solution de saccharose (50 % p/v) | 2,6 mL | Une Solution de saccharose (50 % p/v) | 0,6 mL | ||
| 10 X Tris-Borate-EDTA | 1,6 mL | 10 X Tris-Borate-EDTA | 0,6 mL | ||
| Persulfate d’ammonium (10 % p/v) | 104,0 ΜL | Persulfate d’ammonium (10 % p/v) | 50,0 ΜL | Persulfate d’ammonium (10 % p/v) | 39,0 ΜL |
| TEMED | 6.0 ΜL | TEMED | 1,0 ΜL | TEMED | 2.2 ΜL |
| Volume total | 16,1 mL | Volume total | 4,1 mL | Volume total | 6,0 mL |
Tableau 1. Gel des réactifs de synthèse. Gel de polyacrylamide réactifs de précurseur suffisants pour la fabrication de 2 cassettes.
- Dé-gaz solutions de monomère de gel en insérant une aiguille dans le bouchon du tube et en connectant cette aiguille pour une ligne vide de la maison. Plonger cette Assemblée dans un jeu de bain à ultrasons à haute et à attendre que cesser de bulles qui sortent d’un liquide avant de passer à l’étape suivante (~ 60 secondes / tube).
NOTE : La qualité du vide et la puissance de la cuve à ultrasons ne sont pas critiques tant que bulles peuvent être vu à l’issu de la solution. - Ajouter TEMED et APS au remplissage gel précurseur et vortex brièvement. Immédiatement ajouter 6 mL du précurseur du gel de remplissage pour chaque cassette de gel en pipettant également, la solution dans la partie supérieure de la cassette. Une micropipette 1 mL permet d’ajouter le précurseur en six tranches pour éviter les renversements. La durée totale de cette étape devrait être de moins de 3 minutes.
- S’assurer que le liquide est le niveau en positionnant les cassettes debout sur une table de niveau. Remplir le reste de la cassette avec de l’eau désionisée, dégazé. Distribuer l’eau, à l’aide de tranches de 1 mL, lentement dans le centre de la cassette pour réduire au minimum le mélange. Permettre le polymère à polymériser pendant au moins une heure, jusqu'à pour la nuit.
- Après que le polymère a guéri, il faut inverser les cassettes au-dessus d’un évier pour supprimer la superposition de l’eau. Un pistolet à air comprimé peut être utilisé pour sécher doucement l’interface en soufflant l’air par l’ouverture supérieure de la cassette d’une distance de 6 pouces.
- Ajouter TEMED et APS au gel haute densité précurseur et vortex brièvement. Immédiatement ajouter 320 µL du précurseur à haute densité dans la cassette, encore une fois à l’aide d’une pipette de 1 mL. S’assurer que le liquide recouvre uniformément la couche de gel de remplissage de la cassette à bascule en arrière autour de 3 fois. Cette étape devrait prendre moins de trois minutes.
- Remplir le reste de la cassette avec de l’eau désionisée, dégazé. Pipetter lentement avec une pipette 1 mL dans le centre de la cassette pour réduire au minimum le mélange. Permettre le polymère à polymériser pendant au moins quinze minutes, de préférence une heure.
- Encore une fois, il faut inverser les cassettes pour supprimer la superposition de l’eau. L’air comprimé peut servir à sécher en douceur l’interface. Ajouter TEMED et APS au gel de faible densité précurseur et vortex brièvement. Immédiatement, remplir le reste de la cassette (~1.65 mL) avec le précurseur de la faible densité de gel et insérez le peigne de gel.
- Pipetter un excès de précurseurs de la réserve en haut du peigne car il sera absorbée au cours de la polymérisation. Permettre le polymère à guérir au moins 4 heures, de préférence la nuit. Gels peuvent être stockés pendant une semaine ou plus dans un tampon Tris-Borate-EDTA acide (EDTA) (tampon TBE).
3. préparation et Transcription inverse
- Commençant par une suspension de cellules d’intérêt et travaillant dans une hotte à flux laminaire polymerase chain reaction (PCR), utilisent un hémocytomètre ou un compteur de cellule automatique pour calculer la concentration en cellules. Diluer les cellules à une concentration de 100 à 1000 cellules / µL dans une solution saline tamponnée au phosphate (PBS).
Remarque : Ce protocole a été validé sur une plage de cellules, y compris le PC3, HeLa et des cellules de foie de souris. - À l’aide de réactifs fournis dans la WTA kit (voir Table des matières), mix 19 µL de tampon de lyse et 1 µL d’inhibiteur de RNase pour préparer un 10 X solution mère de tampon de réaction. Créer un mélange maître lyse d’un volume suffisant contenant 0,5 µL de tampon de réaction et de 2,75 µL d’eau libre nucléase pour chaque échantillon.
- Pipetter la suspension cellulaire et descendre 5 fois pour réactiver les cellules sédentarisés et puis Pipetter 1 µL de l’échantillon dans un tube de bande de nucléase de 200 µL stérilisé par UV. Répéter si nécessaire selon le nombre d’échantillons. N’oubliez pas d’inclure un contrôle négatif en pipettant : 1 l d’eau libre nucléase au lieu de cellules pour une réaction. Ensuite, ajouter 3,25 µL du mélange maître lyse dans chaque échantillon et de la composition de pipetage doucement et descendre 5 fois.
- Préchauffer un thermocycleur (avec couvercle chauffée) à 72 ° C. Ajouter 1 µL d’apprêt RT et 1 µL de 20 µM hexamère aléatoire avec adaptateur WTA (5′-AAGCAGTGGTAT-CAACGCAGAGTAC-NNNNNN-3′) pour chaque échantillon. Réserver au moins un tube témoin positif pour la préparation de bibliothèque ADNg et ajouter 2 µL d’eau au lieu d’amorces.
NOTE : Hexamère aléatoire avec adaptateur WTA est facultative et a un impact minime sur les résultats du séquençage. - Incuber les échantillons à 72 ° C dans le thermocycleur préchauffé pendant 3 minutes à lyser les cellules. Éliminer les cellules du thermocycleur et placez-le sur glace pendant 2 minutes. Conserver le contrôle positif à 4 ° C jusqu'à l’étape 5.
- Alors que les cellules sont ensuite, créer un volume suffisant de mélange maître transcriptase inverse pour tous les échantillons d’ARN contenant les rapports suivants du réactif : 2 µL de premier brin tampon, 0,5 µL d’oligonucléotide sélecteur de modèle (TSO), 0,25 µL d’inhibiteur de RNase et 1 µL de transcriptase inverse (100 U/µL).
- Préchauffer le thermocycleur à 42 ° C. Ajouter 3,75 µL du mélange maître transcriptase inverse pour les autres échantillons, ce qui porte le volume de l’échantillon total à 10 µL. Mix de pipetage et descendre 5 fois.
- Effectuer la transcription inverse en plaçant immédiatement les échantillons dans un thermocycleur préchauffé. Exécutez le programme suivant : 42 ° C pendant 90 min, 70 ° C pendant 10 min, 4 ° C pour toujours. Il s’agit d’un point d’arrêt sécuritaire.
4. gel séparation et de récupération de l’échantillon
- Nettoyez soigneusement une chambre d’électrophorèse de gel à l’aide d’un produit ADN. Appliquer plusieurs mL de l’agent nettoyant liquide sur une lingette gratuit charpie jetables et nettoyez sur toutes les surfaces de la chambre et ensuite remplir la chambre avec 0,5 propre x TBE. Pour des résultats optimaux, placez l’ensemble de l’appareil dans un four de réticulation à UV 254 nm et stériliser pendant 15 minutes (15 mJ/cm2).
- Insérez la cassette de Gel-seq dans la chambre d’électrophorèse sur gel et verrouiller en place. Retirer lentement le peigne de gel en tirant vers le haut. Se déplacer lentement pour éviter de déchirer le gel ou déchirant un des bras.
- Conserver les échantillons à l’étape 3 sur la glace, réserver au moins un échantillon comme témoin positif pour la génération de bibliothèque de cDNA. Conserver ce contrôle à 4 ° C jusqu'à l’étape 6. Ajouter 2 µL de 6 X chargement colorant pour les échantillons restants, ce qui porte le total à ~ 12 µL. soigneusement mélanger les échantillons en pipettant également monter et descendre 5 fois.
- Mélanger 1 µL d’une échelle d’ADN avec 2 µL de 6 x 7 µL d’eau et de colorant de chargement. Pipetter ce mélange dans la voie 1 de la cassette de Gel-seq comme un contrôle de l’électrophorèse. Distribuer les échantillons de l’étape précédente dans des couloirs séparés de la cassette de Gel-seq. Veillez à éviter toute contamination entre les puits en insérant la pipette pleinement dans chaque puits et désolidarisé en utilisant seulement un mouvement vertical.
- Utilisant une alimentation de l’électrophorèse sur gel de standard, applique un champ électrique de 250 V à travers la cassette de Gel-seq pendant 30 minutes pour séparer les hybrides cDNA/ARN de l’ADNg. Une fois séparé, retirez la cassette de Gel-seq de la chambre d’électrophorèse sur gel et ouvrez les deux moitiés de la cassette en soulevant les bords avec un grattoir.
- À l’aide d’un scalpel, couper le gel en deux juste en dessous de la couche haute densitée. Jeter la moitié qui contient le gel de remplissage en ramassant avec votre main gantée. Doucement, peler le gel restant hors de la cassette en grattant avec un grattoir à peinture ou un autre outil similaire. Placez cette section du gel dans un plat contenant environ 30 mL de 0,5 x TBE avec 3 µL de tache de gel.
- Couvrir le récipient afin de minimiser le photoblanchiment et tremper le gel tout en agitant doucement pendant 5 minutes. Placer le gel sur la pellicule de plastique et de prendre une image de l’UV à l’aide d’un système de documentation de gel (pour voir le détail supplémentaire Réf. 13). Une exposition de 30 secondes en général produit des images claires. Vérifiez que la séparation s’est produite.
- Déplacez le gel d’un Transilluminateur UV pour faciliter la visualisation des acides nucléiques. Lunettes UV approprié, confirment les résultats du système de documentation de gel. L’ADNg doit être placé au début de la gel de faible densité et l’ADNc à l’interface des régions de faible densitées et haute densitées.
- Utiliser un scalpel, découper les régions du gel contenant l’ADNg cDNA. Les échantillons sont mieux récupérés en coupant une de 4 mm de section rectangulaire de 10 mm de gel ; Cependant, la géométrie exacte dépendra du système d’électrophorèse de gel utilisé. N’oubliez pas de couper également la voie chargée avec le témoin négatif.
- Placer chaque section excisée de gel dans un tube de bande à l’aide d’une paire de pincettes extrémité émoussée. Veillez à ne pas appliquer trop de force ou le gel sera scindé en plusieurs morceaux. Doit cela produit, simplement ramasser chaque morceau et l’ajouter au tube.
- Moudre le gel dans chaque tube à l’aide de l’embout de la pipette (200 µL pipette conseils fonctionnent bien) en déplaçant la pointe de pipette de façon circulaire sur le fond du tube. Ajouter l’eau libre nucléase (40 µL dans les échantillons d’ADNg et 80 µL dans les échantillons de cDNA) dans chaque tube avant de retirer l’embout de la pipette servant à moudre le gel pour minimiser la perte de l’échantillon.
- Placer les tubes de la bande à un Vortex à l’intérieur d’un incubateur à 37 ° C et agiter pendant 8 à 12 heures. Cela permet les acides nucléiques de diffuser hors gel et un naturel empêche point de ce protocole de jour multi.
- Distribuer les échantillons dans une plaque de filtre de maille de 8 µm et faites tourner la plaque à 2600 x g pendant 5 minutes à la souche sur les fragments de gel. Soulever la plaque de filtre de maille loin de la plaque de logement et pipeter les échantillons d’eau sans gel dans un nouveau tube de la bande de 200 µL.
- Ajouter 1 µL de la protéase (0,9 UA/mL) à chaque échantillon contenant ADNg, mélangez bien en pipetage de haut en bas et incuber à 50 ° C pendant 15 min, suivie d’une inactivation thermique à 70 ° C pendant 15 min. Cette étape est cruciale pour épuisement des nucléosomes et rend l’ADNg accessible pour étapes réactionnelles subséquentes.
- À l’aide d’une aiguille de 18 calibre, piquez des trous dans les plafonds de tous les tubes d’échantillons. Placer les échantillons dans un vacufuge pour réduire le volume de liquide. ADNg échantillons devraient être ramenés à 5 échantillons µL et cDNA, réduits à 10 µL.
- Selon la vacufuge et le nombre d’échantillons, le temps d’évaporation totale varie entre 30 et 60 minutes. Si le volume de l’échantillon est inférieure au volume cible, il suffit d’ajouter eau libre nucléase pour augmenter le volume de l’échantillon.
5. ADNg bibliothèque préparation
- À l’aide d’un fluorimètre ou une technologie similaire, quantifier la concentration d’ADN dans chaque échantillon de génomique de l’étape 4, mais aussi le contrôle positif de l’étape 3. Pour un protocole détaillé, consultez le manuel de référence de fluorimètre. 14
- Diluer les échantillons à 0,2 ng/µL d’ADN. Selon le type de cellule de démarrage et la qualité des résultats exigés, des concentrations plus faibles peuvent encore produire des bibliothèques viables. Quelques essais seront nécessaires ; Cependant, les auteurs ont eu du succès avec les bibliothèques aussi basses que 0.1 ng/µL.
- Compléter ADNg bibliothèque préparation en suivant le protocole de volume demi réaction de préparation de bibliothèque à l’étape 7.
6. cDNA Library préparation
- Commençant par les 10 échantillons de cDNA µL et le contrôle positif de l’étape 4, ajoutez 12,5 µL de 2 X qPCR mix, amorce PCR de cDNA µL 0,5 et 2 µL exempte de nucléase eau.
- Effectuer des PCR dans un thermocycleur en temps réel en utilisant le protocole suivant : hot-start à 95 ° C pendant 3 min, suivie de 20 à 30 cycles de 98 ° C pendant 10 s, 65 ° C pour 30 s et 72 ° C pour le volume 3 min. de l’échantillon Total est de 25 µL. surveiller les courbes de réaction et arrêter l’amplification avant th réactions de e laissent la phase exponentielle (augmentation de signal linéaire par rapport à nombre de cycles) afin d’éviter les artefacts de la PCR en raison de la suramplification. Pour en savoir plus sur éviter la suramplification, consultez la section « discussion » de ce document ainsi que 15 Réf..
- Après amplification, nettoyez l’appareil à l’aide de perles d’immobilisation réversible (SPRI) phase solide selon le protocole à l’étape 8. Une fois terminé, passez à l’étape suivante.
- À l’aide d’un fluorimètre ou une technologie similaire, quantifier la concentration d’ADN dans chaque échantillon d’ADNc ainsi que le contrôle positif de l’étape 4. Diluer les échantillons si nécessaire pour contenir environ 0,2 ng/µL d’ADN. Des concentrations légèrement plus faibles encore produisant des bibliothèques viables. Une expérimentation sera nécessaire, mais les auteurs ont eu du succès avec les bibliothèques aussi basses que 0.1 ng/µL.
- Étape facultative : effectuer une séparation de l’électrophorèse sur gel de polyacrylamide standard sur 1-2 µL de chaque produit pour valider la réaction de génération bibliothèque qPCR travaillé. Un exemple d’image de résultat est illustré Figure 4. Un protocole détaillé sur la façon de procéder à l’électrophorèse sur gel de polyacrylamide, voir Réf. 16.
- Toutes préparation de bibliothèque de cDNA en suivant le protocole de réaction la moitié du volume de bibliothèque préparation kit à l’étape 7.
7. Bibliothèque préparation avec la moitié du Volume des réactions
- Préparation de la bibliothèque suit le protocole de kit de préparation de bibliothèque à l’aide de la moitié des réactions de volume. 17 tous les réactifs référencés dans cette section du protocole sont de la préparation de la bibliothèque nécessaire (voir la Table des matières). Commencez par UV stérilisant un nombre suffisant de tubes de bande pour le nombre d’échantillons à traiter.
- Effectuer la réaction de la transposase en ajoutant 5 µL de tampon de transposase dans chaque tube de bande à utiliser lors de l’essai. Puis ajoutez 2,5 µL d’entrée ADN à 0,2 ng/µL (total de 0,5 ng) suivie par 2,5 µL transposase. La composition de pipetage et descendre 5 fois.
- Incuber à 55 ° C pendant 5 minutes, puis maintenez à 10 ° C. Après que l’échantillon atteint 10 ° C, retirez-le du thermocycleur, immédiatement ajouter 2,5 µL de tampon de butée transposase à chaque échantillon. Conserver les échantillons à la température ambiante pendant 5 minutes.
- Préparer la réaction de PCR pour amplifier l’échantillon transpose traitée en ajoutant 7,5 µL bibliothèque préparation PCR mélange, 2,5 µL d’un apprêt d’indice 1 et 2,5 µL d’un apprêt d’index 2. Ces amorces sont exclusifs et sont fournis par le fabricant de la trousse de préparation de bibliothèque. Mélangez bien en pipettant également monter et descendre 5 fois.
Remarque : S’assurer que les combinaisons d’amorces uniques sont utilisés pour chaque échantillon. Il y a 12 amorces différents index 1 et 8 amorces différents index 2, ce qui permet d’étiqueter unique jusqu'à 96 échantillons différents. Choisissez une combinaison unique d’amorces pour chaque échantillon. - Effectuer la PCR en utilisant le programme suivant sur un thermocycleur. Le volume de l’échantillon est de 25 µL.
72 ° C pendant 3 minutes
95 ° C pendant 30 secondes
12 cycles de :
95 ° C pendant 10 secondes
72 ° C pendant 30 secondes
55 ° C pendant 30 secondes
72 ° C pendant 5 minutes
Tenir à 10 ° C - Nettoyer les bibliothèques préparés à l’aide de perles SPRI, suivant le protocole à l’étape 8. Les bibliothèques devraient être validées en utilisant l’électrophorèse sur gel ou un essai similaire. Consulter le manuel de kit de préparation de bibliothèque pour plus d’informations sur la validation des bibliothèques. 17
8. solid Phase réversible immobilisation perle bibliothèque nettoyage
- Phase solide billes réversible immobilisées (SPRI) doivent être stockés dans des aliquotes de 1,5 mL et doivent être ramenés à température ambiante avant chaque utilisation. Il est également recommandé de préparer fraîches 80 % éthanol pour chaque expérience. Les étapes suivantes reposent le protocole SPRI de perle dans le manuel du kit WTA. 18
- Ajouter 1 µl la WTA kit de tampon de lyse dans chaque produit de la PCR. Vortex les perles SPRI jusqu'à uniformément mélangés, puis ajouter 50 µl de perles SPRI à chaque échantillon. Mélanger en pipettant également, l’échantillon de haut en bas 10 fois et puis incuber les échantillons à la température ambiante pendant 8 minutes.
- Brièvement, faire tourner les échantillons pour recueillir le liquide du côté des tubes. Placer les échantillons sur un dispositif de séparation magnétique ~ 5 minutes jusqu'à ce que le liquide semble tout à fait clair.
- Bien que les échantillons soient sur le dispositif de séparation magnétique, lentement Pipetter hors le surnageant et le jeter - veillez à ne pas déranger l’anneau de perles sur le tube. Ensuite, ajouter 200 µL d’éthanol à 80 % dans chaque échantillon sans déranger les perles. Attendez 30 secondes et ensuite soigneusement Pipeter hors le surnageant. Répétez cette étape (lavage de l’éthanol) une fois.
- Laisser les échantillons sécher pendant 30 s à 1 min. Ne pas trop sécher les échantillons comme grands fragments seront définitivement liés aux talons.
Remarque : Une brève durée de séchage est recommandée de veiller à que retirer toutes les traces de l’éthanol. Des traces d’éthanol resterait derrière elles peuvent inhiber légèrement réactions en aval. - Retirez les échantillons de l’appareil de séparation magnétique et ajouter 15 µL d’eau à chaque échantillon pour éluer les perles de l’ADN. Pipette de haut en bas et s’assurer que les perles soient écartés les côtés des tubes. Incuber à température ambiante pendant 2 minutes. Tourner brièvement les échantillons et puis placez-les sur le dispositif de séparation magnétique pendant environ 1 minute jusqu'à ce que la solution semble évident.
- Tandis que les échantillons sont sur l’appareil de séparation magnétique, lentement Pipeter vers le haut le surnageant et transférez-le vers tubes propres. Veillez à ne pas déranger l’anneau de perles sur le tube. Jeter les tubes avec des perles.
Résultats
La séparation physique des hybrides ADNg et cDNA/ARN dans le dispositif de Gel-seq peut être visualisée par l’imagerie gel fluorescent ; un résultat représentatif est illustré à la Figure 3. Paroi A montre le dispositif fabriqué de Gel-seq ; fausses couleurs a été ajouté pour distinguer les gel de différentes régions. Panneau B apparaît une fermeture de quatre départs différents utilisés pour la validation. La troisième voie, un contrôle négatif, représente à fond et montre qu’il n’y a aucune autoflourescence du gel aux interfaces. Nous avons chargé les ruelles de premiers et deuxième avec échelles d’ADN. Ces voies montrent seulement une bande sombre à l’interface entre les membranes haute et basse densité, révélant que les petits fragments peuvent passer par le gel de basse densité. La quatrième voie montre le comportement d’un échantillon biologique d’intérêt : 500 cellules PC3. Nous avons chargé lane quatre tel que décrit à l’étape 3 du protocole. L’image montre la séparation de l’ADN génomique et hybrides cDNA/ARN. Une bande sombre au dessus de la membrane de faible densité est l’ADN génomique de megabase-échelle tandis que les hybrides cDNA/ARN sont empilés à l’interface des régions basses et haute densitées. Contrairement aux ruelles chargées avec l’échelle, il y a aussi plusieurs groupes présents au sein de la région à haute densité du gel. Ces fragments, plus petits que 100 bp, sont des produits hors cible produits des oligonucléotides amorces au cours de la transcriptase inverse. Panneau C montre une image représentative de l’ensemble du dispositif Gel-seq d’une expérience réussie. Lanes étiquetés RNA/ADNc ont été traitées avec le protocole Gel-seq, tandis que les voies marquées RNA montrent une séparation de juste ADNg et d’ARN. Panneau D montre une expérience ratée avec des bandes noires en haut de chaque voie de la membrane de faible densitée. Cela était dû à tampon d’électrophorèse contaminé par des fragments d’ADN. À cette étape, les chercheurs devraient chercher pour les contrôles propres négatifs et deux bandes noires distinctes indiquant la présence de l’ADNg séparé et RNA/cDNA hybrides.

Figure 3 . Résultats de séparation gel-Seq. Le dispositif de Gel-seq (A) et une image fluorescente montrant la séparation des DNA et RNA/cDNA hybrides (B). Fausses couleurs a été ajouté pour distinguer plus facilement entre les différentes régions de la masse volumique dans le gel. (C et D) Images fluorescentes représentatives de l’ensemble du dispositif d’un succès (C) et (D) ratée Gel-seq. NTC ne = aucun modèle de contrôle. Certaines parties de cette figure ont été reproduits à partir Réf. 7 avec la permission de la Royal Society of Chemistry. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Une fois que les hybrides de DNA et RNA/ADNc ont été séparés et le reste du protocole Gel-seq terminé, il est possible de générer des bibliothèques de séquençage. Pour valider les bibliothèques de prêts, nous exécuter une expérience d’électrophorèse de gel standard (Figure 4) ou un bioanalyzer. Dans la Figure 4 révèle bibliothèques générées à partir des cellules PC3 500 et 750 cellules HeLa. La figure montre la distribution de fragment pour bibliothèques appariés produits de Gel-seq (étiqueté « Gel ») par rapport aux échantillons inégalées générés avec des protocoles standard (étiquetés « Tube »). Les tailles de fragment pour Gel-seq apparaissent entre 200 et 800 basepairs comme prévu lors de la préparation des bibliothèques à l’aide de la trousse de préparation de bibliothèque standard du génome entier. Si les fragments de bibliothèque n’apparaissent pas dans la gamme de taille correcte dans cette étape, la préparation de la bibliothèque a échoué.
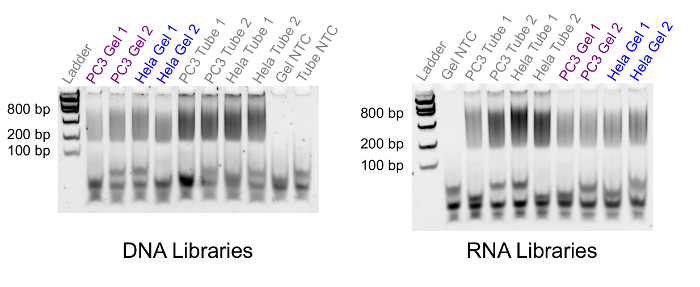
Figure 4 . Comparaison de taille de Fragment bibliothèque. Une image d’électrophorèse de gel fluorescent en comparant la distribution granulométrique bibliothèque entre Gel-seq (Gel) et les contrôles standard (Tube). La voie de gauche contient une faible masse échelle d’ADN avec fragment tailles 100, 200, 400, 800, 1200 et 2000 basepairs. La taille de fragment pour toutes les bibliothèques comprises entre 200 et 800 basepairs, comme prévu aux bibliothèques préparés avec ce kit. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
L’ultime validation du protocole Gel-seq est basée sur l’analyse des résultats de séquençage. Nous avons choisi les cellules PC3 pour nos expériences de validation, ces cellules ont des profils d’expression homogènes qui permettent aux échantillons d’être fendu et traitées à l’aide de Gel-seq tant les méthodes traditionnelles ; Voir la Figure 5. Une comparaison entre l’ADN génomique des cellules PC3 est montrée dans les Figures 5A et 5 b. Figure 5 A montre une comparaison des profils de variation (CNV) numéros de copie du génome entier produit de PC3 à l’aide de Gel-seq ou une réaction de préparation de bibliothèque standard du génome entier (tube contrôle). Chaque point est un comte de moyenne normalisée bin ; emplacements sont définis à partir de données de génome de référence telle que chaque emplacement a égale nombre attendu dans une cellule diploïde saine, c'est-à-dire, une ligne plate, représentant des copies égales pour chaque région du tout autosomique (à l’exclusion des X et Y) chromosomes. PC3 contient plusieurs copies des mêmes régions qui apparaissent comme des pics supérieurs à un nombre de deux copies de fond. Gel-seq génère un profil CNV qualitativement similaire comme réaction de tube standard. Accord entre les deux parcelles peut être évaluée quantitativement par régression linéaire, comme indiqué dans le tableau B. Une corrélation de Pearson de R = 0,90 indique que les données génomiques recueillies par ou l’autre méthode sont fonctionnellement équivalentes.

Figure 5 . Validation de la bibliothèque. Gel-seq validation pour la génomique (A et B) et transcriptomique (C, D et E) données générées par les cellules PC3 et Hela. Paroi A montre une comparaison du génome CNV profils générés par PC3 à l’aide de Gel-seq (Gel-seq) ou une réaction standard (tube). MAPD = médiane absolue de la différence par paires. Le groupe B est une régression linéaire entre les deux échantillons dans le groupe A, avec R = 0.90 indiquant les données génomiques sont fonctionnellement équivalentes. Les axes montrent que le bac normalisé log2 compte. Séries C et D comparer les données transcriptomiques cellules PC3, chaque point présentant un décompte dans transcriptions par kilobases par million (TPM). Les axes montrent la comparaison entre deux échantillons comme comtes de transcription log2 normalisé. Panneau C montre une comparaison des réplicats techniques générés à l’aide de Gel-seq, et panneau D montre qu'une comparaison entre le Gel-seq et traditionnel RNA-Seq. panneau E montre que Gel-seq peut résoudre des cellules du type basé sur RNA expression à l’aide d’une analyse en composantes principales. L’axe x indique que les premiers comptes de composantes principales 91,6 % de la variation entre les échantillons tandis que l’axe des y montre que les comptes de deuxième composante principale pour seulement 6,5 % de la variance. Certaines parties de cette figure ont été reproduits à partir Réf. 7 avec la permission de la Royal Society of Chemistry. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
De même, nous avons comparé les données transcriptomiques de notre protocole Gel-seq pour le protocole de puce-Seq dans tube standard. La figure 5 illustre la corrélation entre les deux réplicats techniques Gel-seq (Figure 5C) et entre Gel-seq et la méthode standard (Figure 5D). Chaque point est un comte dans transcriptions par kilobases par million (TPM) pour chaque gène détecté à TPM > 5 dans les deux ensembles de données. Les régressions linéaires sont affichées sous forme de lignes rouges, et le coefficient de corrélation de Pearson est affiché dans le coin supérieur gauche. Réplicats techniques de Gel-seq (R ∼ 0,8) d’accord, mais moins bien en corrélation avec la méthode standard (R < 0,7). Ceci suggère que le Gel-seq introduit un biais dans la numération de gène. Heureusement, ce biais est systématique et, comme il ressort de l’analyse en composantes principales dans la Figure 5E, on peut toujours tirer des conclusions significatives entre différents échantillons biologiques.
Discussion
Il y a plusieurs étapes critiques associées à la fabrication de dispositifs de Gel-seq ainsi que le protocole lui-même. Pendant la fabrication, nous vous recommandons de commencer avec les épaisseurs de couche prescrite pour les différentes régions du gel. Nous avons passé beaucoup de temps test options de fabrication différente et le protocole décrit ici produit les meilleurs dispositifs pour les cassettes répertoriés dans Table des matières et réactifs. Si les chercheurs utilisent un système de cassette alternative, ils peuvent trouver nécessaire de régler les volumes utilisés lors de la création des dispositifs. Le défi majeur dans la fabrication, c’est que si la région à haute densité de gel est trop grande, il peut se décoller sur les bords de la cassette et créer des poches d’air à l’intérieur de la cassette qui va perturber l’électrophorèse. En effectuant un cast plusieurs cassettes avec plusieurs volumes de différentes couches, les chercheurs devraient être en mesure de déterminer rapidement la configuration optimale pour leur matériel spécifique.
Le protocole Gel-seq a également plusieurs étapes critiques qui peuvent être validées avant la fin du protocole. Un seul point d’échec potentiel est la séparation de l’ADNg et RNA/cDNA hybrides. Cela peut être validé par l’imagerie de l’appareil Gel-seq après la séparation (voir la Figure 3B). Dans une série d’expériences, nous avons constaté que notre approvisionnement en laboratoire du tampon avait été contaminé par l’ADN et causait autoflourescence important dans notre dispositif (voir Figure 3D). Cela rend difficile de déterminer si la séparation avait eu lieu. Imagerie de fluorescence nous a aidés à identifier et corriger le problème avant d’utiliser des réactifs coûteux pour générer des bibliothèques de séquençage.
Un autre point critique est étape 6.2, l’amplification de qPCR de cDNA après la séparation. Les chercheurs devraient être très attentif ne pas à overamplify dans cette étape, car il réduira la qualité des données RNA-seq. Cette considération n’est pas unique à Gel-seq, mais est un élément commun de préparation de bibliothèque faibles intrants RNA-seq. L’amplification par PCR au cours de la préparation de la bibliothèque de séquençage est souvent nécessaire, mais elle peut introduire des biais et des erreurs de séquence. Le nombre de cycles pour l’ACP requis dépend de la complexité et de la quantité d’échantillon. Il est généralement conseillé de limiter le nombre de cycles PCR au strict minimum nécessaire pour produire suffisamment clustering quand les bibliothèques sont séquencés. En théorie, un protocole peut être optimisé pour déterminer le nombre exact de cycle qui donne le nombre suffisant de copies sans introduire d’artefacts excessives. Dans la pratique, cependant, incohérences dans la qualité de l’échantillon, chargement, ou de la manipulation au début dans le protocole peut considérablement affecter la distribution des modèles moléculaires disponibles pour prép PCR, qui se répercute sur le nombre de cycle PCR optimal de la bibliothèque. La solution plus générale, nous avons trouvé pour surveiller la progression des réactions d’amplification à l’aide d’un colorant fluorescent, exécuter les réactions sur un thermocycleur PCR en temps réel et arrêter les réactions dans l’exponentielle (linéaires par rapport à nombre de cycles) phase. Dans notre expérience, la surveillance en temps réel est particulièrement pertinente lors du développement, adaptation ou adopter un nouveau protocole.
La dernière étape critique génère génomique et transcriptomique bibliothèques. La clé de cette étape consiste à définir la concentration initiale d’échantillon pour la réaction de prép bibliothèque ADN aussi proche de 0,2 ng/µL (total de 0,5 ng) que possible. C’est relativement simple pour le cDNA de qPCR amplifié car il y a généralement un excès d’ADNc, mais il peut être plus difficile pour les échantillons ADNg. Nous avons trouvé attention attention à l’étape de vacufuge était nécessaire alors que les échantillons étaient étant concentrées. Comme prévu, au cours d’expériences avec 1000 cellules, l’étape de vacufuge pourrait être arrêtée beaucoup plus tôt que les expériences avec 100 cellules. Le nombre d’échantillons dans le vacufuge a également touché le taux d’évaporation dans nos expériences. Nous avons constaté qu’à l’aide d’un flourometer pour valider l’ADN contenu à mi-chemin de l’étape de concentration pourrait être utile lors de l’exécution du protocole avec des échantillons inconnus. Heureusement, si les chercheurs plus concentré un échantillon, nucléase libre de l’eau peut être ajouté pour diluer l’échantillon. Théoriquement, il est possible d’utiliser le vacufuge pour sécher l’ADN et puis remettre en suspension dans le volume désiré ; Cependant, nous suggérons d’éviter l’évaporation complète.
Nous considérons trois méthodes actuelles de production simultanées bibliothèques d’ADN et d’ARN, Gel-seq7, G & T-seq8et DR-seq9, comme gratuit. Gel-seq est idéal pour les échantillons dans la plage de cellules de 100-1000 et ne nécessite pas de menu déroulant cibles ou une connaissance a priori du génome. Les deux autres méthodes sont mieux adaptées aux applications de cellule unique. Un de nos objectifs dans le développement de Gel-seq était de créer un protocole qui pourrait être facilement mis en œuvre par d’autres chercheurs. Nous avons donc décidé de fabrication d’engins dans le facteur de forme standard d’une cassette de gel de polyacrylamide. Alors que la technique nous permet de définir nos différentes membranes est roman, plupart des laboratoires de génétique ont déjà tout le matériel nécessaire pour fabriquer le dispositif Gel-seq. En outre, le coût du dispositif est trivial - seulement 5,25 $ pour un appareil qui peut traiter 12 échantillons. Cela dit, comme avec n’importe quel protocole de préparation de bibliothèque à l’aide de réactifs commerciaux, le coût global pour générer des bibliothèques demeure élevé. Notre coût de réactif par exemple était de 50 $ pour l’ensemble-transcription amplification et 28 $ pour la préparation de la bibliothèque d’ADN et d’ARN. Heureusement, le dispositif de Gel-seq lui-même soit le protocole utilisé. Par exemple, au cours du développement nous testé avec succès l’appareil à l’aide de cellules de culture et une ancienne RNA bibliothèque amplification protocole19, bien que nous avons trouvé que ce n’était pas approprié pour des échantillons de tissus provenant de souris. Regardant vers l’avenir, comme des alternatives moins chères pour la préparation de la bibliothèque sont développés, notre protocole peut être adapté pour fonctionner avec ces nouvelles techniques. Nous pensons que les chercheurs trouveront simple à mettre en œuvre Gel-seq dans leurs propres laboratoires. Nous espérons que cela facilitera l’adoption rapide de la technologie.
Déclarations de divulgation
KZ est membre fondateur et conseiller scientifique de Singlera Genomics Inc.
Remerciements
Financement de ce travail a été fourni par l’Université de San Diego, la National Science Foundation recherche Fellowship programme d’études supérieures, accorder des NIH R01-HG007836 et par le ministère coréen des sciences, de TIC et de la planification Future.
Les versions antérieures d’une plusieurs personnalités ont été tout d’abord publiées dans « Hoople, G. D. et al. Gel-seq : génome entier et séquençage de transcriptome de faibles intrants ADN et l’ARN bibliothèque préparation simultanée à l’aide de barrières semi-perméables hydrogel. Laboratoire sur une puce 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). » Laboratoire sur une puce a sanctionné la réutilisation des figures dans cette publication.
matériels
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate - Corning HTS Transwell 96 well permeable supports - 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
Références
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18(2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31(2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908(2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), at http://science.sciencemag.org/content/352/6293/1586 (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923(2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies. , Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017).
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- A Guide to Polyacrylamide Gel Electrophoresis and Detection. Bulletin 6040 Rev B. Bio Rad. , Available from: http://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6040.pdf (2017).
- Illumina. Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon