Method Article
ゲル-seq: DNA および RNA のゲルのマトリックスを使用しての同時配列ライブラリの準備の方法
要約
ゲル seq の両方 DNA と RNA-seq 単純なハイドロゲル デバイスを用いた 100-1000 細胞からごくわずかな追加コストでライブラリを同時に準備することも可能。 にします。対ライブラリを生成する生物のプロトコルと同様に、デバイス作製のための詳細なアプローチを提案する.
要約
増幅し、小さい開始サンプルから DNA または RNA をシーケンスする能力は、過去 5 年間で達成されているのみ。残念なことに、ゲノムの生成やトランスクリプトーム ライブラリの標準的なプロトコルに互換性がないと、研究者は特定のサンプルの DNA または RNA をシーケンスするかどうかを選択する必要があります。ゲル seq DNA と単純なハイドロゲル デバイスを用いた 100-1000 細胞から始まる RNA の両方のライブラリを同時に準備する研究者を有効にしてこの問題を解決します。対ライブラリを生成する生物のプロトコルと同様に、デバイス作製のための詳細なアプローチを提案する.他の研究者によって簡単に実装できるようにゲル seq を設計多くの遺伝学研究所はすでにゲル seq デバイス作製を再現する必要な機器を持っています。私達のプロトコルは両方全体転写増幅 (WTA) のための一般的に使用されるキットを採用しています、ライブラリ生成ゲノムおよびトランスクリプトームに精通したライブラリの準備は、既に研究者に精通していることも多い。我々 のアプローチでは、ごくわずかな追加コストと分割せず単一サンプル、DNA と RNA の両方のシーケンスの力をもたらすために研究者をことができます。
概要
次世代シークエンシング (NGS) は、遺伝学研究方法の深遠な影響を与えています。ここでの研究者は、一度全体の種のゲノムに焦点を当てて、今単一の腫瘍または 1 つの実験でも単一セルのゲノムを配列することが可能です。1 NGS もしました、トランスクリプトームと呼ばれるデータのコレクションのセル内にある RNA 転写産物をシーケンス処理する費用対効果。増幅し、小さい開始サンプルから DNA または RNA をシーケンスする能力は、過去 5 年間で達成されているのみ。2,3,4は残念ながら、標準のプロトコルに互換性がないと研究者はある特定のサンプルの DNA または RNA をシーケンス処理するかどうかを選択する必要があります。開始サンプルが十分に大きい場合は、半分に分割することができます。ただし、規模が小さいサンプルの分裂のための材料の損失はライブラリの品質に影響を与えるし、細胞間の興味深いバリエーションを平均することができますサンプルのプールします。5さらに、研究者は単一のセルまたは小さな異種腫瘍生検などを分割できないサンプルを調べることに興味を持つ。6
この問題を解決する 3 つのプロトコルが最近開発された同じ開始サンプルから DNA と RNA の両方をシーケンス処理する: ゲル seq7、G & T seq8、および DR seq9。この記事は、ゲル-seq、同時にごくわずかな追加コストで 100 細胞から DNA および RNA のライブラリを生成するための詳細なプロトコルを示します。ゲル seq の小説の側面は、DNA と RNA 低コスト ハイドロゲル行列を使用して大きさだけに基づいて区別する能力です。ゲル Seq プロトコルのコア技術革新は RNA から DNA を物理的に分離します。この分離は、電気泳動によってこれらの分子のサイズ違いの活用のポリアクリルアミドの膜の組み合わせを使用して実現されます。コンテキストでこれらのサイズ違いを置く、DNA と RNA をイメージする方法を検討してください: RNA ナノメートル スケール上に存在し、低温電子などの複雑な技術を使用してイメージを作成する必要があります DNA ミクロン スケール上に存在する、伝統的な顕微鏡を使用して表示することができますが、顕微鏡検査。10
このプロトコルでは DNA と RNA を分離するアプローチは、図 1に示します。左側のパネルは、DNA および RNA の無料ソリューション膜近くに浮かんでいるを示しています。電気フィールドを適用すると、右側のパネルに示すように、DNA および RNA は膜を介して移行を誘導する電気泳動の力を体験します。膜特性を調整することにより、RNA から DNA を分離する半透膜を作成しました。DNA 分子は膜に対して押されるが、サイズが大きいので端につれ。その一方で、小さな RNA 分子を再構成し、できます膜を通して彼らの方法を織る。レプテーション、として知られているこのプロセスはヘビが草を介して移動する方法に似ています。さらに小さいポリマー難しすぎて第二に、高密度の膜によってこれらの RNA 分子を停止する最終的に (> 200 塩基対) をくねらせるため。一度物理的に分離、DNA および RNA 回収し、処理できるゲノムとトランスクリプトームの両方に関する情報を生成します。我々 は DNA と RNA を区切ることができます、我々 は分離前に cDNA に転写される RNA が逆の場合より良い結果が得られますを発見しました。CDNA/RNA 雑種は単独で RNA より安定して、低密度の膜を通して渡すことができます。

図 1.ゲル seq 動作原理。物理的に DNA と RNA を分離するために使用する基本原理。応用電界で小さな RNA 分子が低密度の膜を通って移行しますが、大きい DNA の分子が表面に閉じ込められています。この図は、王立化学協会の許可を得て参考文献 7 から再現しました。この図の拡大版を表示するのにはここをクリックしてください。
本稿で詳細にゲル seq デバイス作製を生成する生物学的プロトコル ペアに DNA および RNA のライブラリ。両方の概要を図 2に示します。レイヤー 3 つ異なる密度ポリアクリルアミドゲルで互いの上に標準的なスタッキングのゲルを作成する同じプロセスでデバイスを作製しました。11生物学的プロトコルから始まる 100-1000 細胞を PBS で中断します。細胞を分離、cDNA/RNA 雑種からゲノム DNA を分離するデバイスを使用する前に、RNA が cDNA に変換されます。後分離と回復、ゲノム、トランスクリプトーム ライブラリは、標準の全ゲノム ライブラリの準備キットのプロトコルに密着したプロセスを使用して準備されます。チップ用の文書でラボでの開発とゲル seq の検証についてさらに詳細を読むことができます"ゲル seq: 全ゲノムとトランスクリプトーム シーケンス半透性ハイドロゲル障壁を用いた同時低入力 DNA および RNA 図書館準備で."7

図 2.ゲル seq プロトコル。ゲル seq デバイスおよびプロトコルを生成するための手順の概要については、DNA および RNA のライブラリ ペア。この図の部分は、王立化学協会の許可を得て参考文献 7 から再現されました。この図の拡大版を表示するのにはここをクリックしてください。
単一セルからの DNA および RNA のライブラリを生成するため、研究者は G & T seq や DR seq. G
DR seq は DNA や RNA からの cDNA の両方が同じ管で増幅される中古増幅ステップに依存します。サンプルが 2 つに分割し、DNA と RNA シーケンス ライブラリを準備する並列で処理します。ゲノム DNA と RNA からの cDNA の区別は、DR seq は計算のアプローチを取る。エクソンのみが存在するシーケンスは、それらは DNA や RNA から由来している可能性がありますと計算ゲノム DNA データの抑制されます。9このアプローチの利点は、DNA と cDNA/RNA 区切る必要はありません物理的にゲル seq と G & T seq. で行われるように欠点は、ただしは、DR seq のゲノムとトランスクリプトーム (すなわちイントロンとエクソン)、事前知識を必要とし、多くの転写産物がないまだ完全に核のシーケンスなどのアプリケーションに最適なできない可能性があります。スプライシング ・ イントロンが含まれています。12
ゲル seq の小説の側面は、DNA と RNA のサイズのみに基づいてセルの数百を分離する能力です。このメソッドには、ゲノム、トランスクリプトームの先験的知識は不完全な接続に対してロバストとポリ adenylated の成績証明書に限定されていませんが必要です。研究者が少なくとも 100 セルから始まる、アプリケーション、ゲル seq は安く、広く利用可能な材料を使用して簡単な方法を提供します。
プロトコル
1. 薬液の準備
注: 次の手順は、後の手順で必要な薬液の準備用です。これらは、大量に作ったし、数カ月保存できます。
- まず、任意の汚染 DNA を中和するために 15 分間 (15 mJ/cm2総露出) 254 nm UV 架橋オーブンで滅菌することによって精製、脱イオン水 50 mL を準備します。次の手順で使用するための 37 ° C に熱する。
- 40% 合計 (T) および 3.3% 架橋剤 (C) (29:1) の 10 mL を作るポリアクリルアミド前駆体溶液。3.867 g アクリルアミド モノマーと bis アクリルアミド単量体の 0.133 g の重量を量る。組み合わせて、ボリューム 10 mL まで暖かい水と溶解するまで渦。室温で光から保護されたストア。
注: の予混合 40 %t、3.3% 溶液 C (29:1) が商業的に購入することができます。 - 50 %t、5 %c ゲル溶液の 10 mL を作る。4.750 g アクリルアミド モノマーと bis アクリルアミド モノマー 0.250 g の重量を量る。組み合わせて、ボリューム 10 mL まで暖かい水と溶解するまで渦を使用して。室温で光から保護されたストア。
- 50% (w/v) ショ糖溶液の 10 mL を作る。卒業シリンダーに 5 g のショ糖と 10 mL の容量まで暖かい水を追加します。渦まで溶解し、室温で保管します。
- 10% の 10 mL を作る APS (w/v)。卒業シリンダーに 1 g の過硫酸アンモニウム (APS) を追加します。風邪 (~ 4 ° C) の精製を追加水を 10 mL と溶解するまで渦の総量まで。200 μ L の因数ですぐにフリーズします。
2. ゲル seq カセット作製
注: ゲル seq 元々 直立カセット (詳細については材料の表を参照してください)。ただし、このプロトコルは、任意の標準的なゲルの電気泳動のカセットで動作するようにできます。
- 3 つの別々 のプラスチック チューブにゲル前駆体を準備するには、表 1 に示すとおり、試薬を追加します。そうでないAP またはテトラメチルエチレンジアミン (TEMED) 次の手順の指示までを追加。ミックス徹底的に渦成分。
| フィラー ゲル前駆体 | 高密度ゲル前駆体 | 低濃度ゲル前駆体 | |||
| 40 %t、3.3%C アクリルアミド Bisacrylamide ソリューション | 1.6 mL | 50 %t、5 %c アクリルアミド Bisacrylamide ソリューション | 2.4 mL | 40 %t、3.3%C アクリルアミド Bisacrylamide ソリューション | 0.6 mL |
| 脱イオン水 | 10.2 mL | 脱イオン水 | 1.0 mL | 脱イオン水 | 4.8 mL |
| ショ糖液 (50 %w/v) | 2.6 mL | ショ糖液 (50 %w/v) | 0.6 mL | ||
| 10 X トリス-ホウ酸塩-EDTA | 1.6 mL | 10 X トリス-ホウ酸塩-EDTA | 0.6 mL | ||
| 過硫酸アンモニウム (10 %w/v) | 104.0 Μ L | 過硫酸アンモニウム (10 %w/v) | 50.0 Μ L | 過硫酸アンモニウム (10 %w/v) | 39.0 Μ L |
| TEMED | 6.0 Μ | TEMED | 1.0 Μ L | TEMED | 2.2 Μ L |
| 総容積 | 16.1 mL | 総容積 | 4.1 mL | 総容積 | 6.0 mL |
テーブル 1。ゲルの合成試薬。ポリアクリルアミド ゲル前駆体試薬 2 カセットの作製のために十分。
- チューブのキャップを通して針を挿入し家真空ラインにこの針を接続する解消ガス ゲル、モノマー溶液。高超音波風呂セットでこのアセンブリを沈めるし、泡は液体から浮上して次のステップに移動する前に停止するまで待機 (~ 60 秒/チューブ)。
注: 真空の質と超音波風呂の力重要ではありませんソリューションから新興の泡を見ることができる限り。 - 簡単に、フィラー ゲル前駆体と渦に TEMED と AP を追加しています。すぐにカセットの上にソリューションをピペッティングによる各ゲル カセットに充填剤ゲル前駆体の 6 mL を追加します。1 mL ピペットを使用すると、こぼれるように六つずつ前駆体を追加できます。この手順の所要時間は、3 分未満にする必要があります。
- 流体は、カセットをレベル テーブルで直立位置レベルを確認します。脱脱イオン水でカセットの残りの部分を入力します。もう一度ゆっくりと混合を最小限に抑えるにはカセットのセンターに 1 mL ずつを使用して、水をピペットします。ポリマーを一夜にして最大 1 時間以上の治療法を許可します。
- 樹脂が硬化した後、水のオーバーレイを削除する流し台の上ゲル カセットを反転します。圧縮空気銃は、6 インチの距離からカセットの上部開口を通して空気を吹くことによってインターフェイスをやさしく乾燥する使用ことができます。
- 簡単に、高密度ゲル前駆体と渦に TEMED と AP を追加しています。すぐに 1 mL ピペットを使用してカセットに高密度の前駆体の 320 μ L を追加します。流体は、ロッキングのカセットに前後約 3 倍ろうゲル層を均等にコートを確認します。この手順では、3 分未満を取る必要があります。
- 脱脱イオン水でカセットの残りの部分を入力します。混合を最小限に抑えるにはカセットのセンターに 1 mL ピペットをゆっくりとピペットの。少なくとも 15 分、できれば 1 時間硬化ポリマーを許可します。
- 再び、水のオーバーレイを削除するゲル カセットを反転します。圧縮空気を使用して、インターフェイスをやさしく乾燥できます。簡単に、低密度ゲル前駆体と渦に TEMED と AP を追加しています。すぐに低密度ゲル前駆体とカセット (~1.65 mL) の残りの部分を記入し、ゲル櫛を挿入します。
- 重合中に吸収されることと、櫛の上部に予備前駆体の過剰をピペットします。4 時間以上、できれば一晩を治すためポリマーを許可します。ゲルは、Tris ホウ酸-エチレンジアミン四酢酸 (EDTA) (TBE バッファー) のバッファーで 1 週間以上保存できます。
3. サンプル準備と逆転写
- 興味のセルの懸濁液から始まる、ポリメラーゼ連鎖反応 (PCR) の層流フードで働いて、細胞濃度を計算するのに診断またはセルの自動カウンターを使用します。リン酸緩衝生理食塩水 (PBS) μ L あたり 100 から 1000 までのセルの濃度に細胞を希釈します。
注: このプロトコルは、PC3、hela 細胞のマウス肝細胞などの細胞の範囲で検証されています。 - WTA は、試薬を使用して (材料の表を参照)、換散バッファーのミックス 19 μ L と 1 μ L 10 X を準備する RNase 阻害剤の貯蔵液反応バッファーの。0.5 μ L 反応バッファーの各サンプルのヌクレアーゼ フリー水の 2.75 μ L を含む十分な量の換散のマスター ミックスを作成します。
- 上下に 5 回再定住細胞を中断し、紫外線滅菌 200 μ L ヌクレアーゼ無料ストリップ管に試料 1 μ L をピペットに細胞懸濁液をピペットします。サンプル数に応じて必要に応じて繰り返します。ヌクレアーゼ フリー水の代わりに 1 つの反作用のための細胞 1 μ L 分注による負の制御を含めるようにしてください。次に、各サンプルに換散のマスターの組合せの 3.25 μ l 添加し優しく上下に 5 回ピペッティングで混ぜます。
- 72 ° c (温水付け) サーマルサイクラーを予熱します。各サンプルに WTA アダプター (5 '-AAGCAGTGGTAT-CAACGCAGAGTAC-NNNNNN-3 ') RT プライマーの 1 μ L と 20 μ M ランダム hexamer の 1 μ L を追加します。GDNA ライブラリの準備のための肯定的な制御として少なくとも 1 つの管を予約し、プライマーの代わりに水の 2 μ L を追加します。
注: WTA アダプターとランダムの hexamer はオプションであり、最小限の影響は、結果を配列します。 - 72 ° C、細胞を溶解する 3 分間予熱したサーマルサイクラーにサンプルをインキュベートします。サーマルサイクラーと氷の場所から 2 分のセルを削除します。ステップ 5 まで 4 ° C で肯定的な制御を格納します。
- 細胞が溶解すると、次の試薬の比率を含むすべての RNA のサンプルの逆のトランスクリプション マスター ミックスの十分な量を作成: 最初の鎖バッファーの 2 μ L、0.5 μ L のテンプレート スイッチ オリゴヌクレオチド (TSO)、RNase 阻害剤 0.25 μ、1 μ L の逆転写酵素 (100 U/μ L)。
- 42 ° c. にサーマルサイクラーを予熱します。残りのサンプルは、10 μ L に総サンプル ボリュームをもたらす逆のトランスクリプションのマスターの組合せの 3.75 μ L を追加 5 回上下ピペッティングでミックス。
- すぐに予熱したサーマルサイクラーにサンプルを置くことによって逆のトランスクリプションを実行します。次プログラムを実行: 90 分、10 分、4 ° C 永遠に 70 の ° C の 42 ° C。安全な停止ポイントです。
4. ゲルの分離と回復
- 慎重にきれいに DNA の除去の製品を使用してゲルの電気泳動室。使い捨て糸くず無料ワイプ ワイプし、商工会議所のすべてのサーフェス全体液体洗浄剤のいくつかの mL に適用されクリーン 0.5 箱をいっぱい x TBE。最適な結果を得るには、254 nm UV 架橋オーブンで全体の器具を配置し、15 分 (15 mJ/cm2) の消毒します。
- ゲルの電気泳動室にゲル seq カセットを挿入し、所定位置にロックします。ゆっくりとまっすぐに引いてゲル櫛を取り外します。ゲルを引き裂くまたは腕のいずれかをリッピングを避けるためにゆっくりと移動します。
- ステップ 3 氷の上からサンプルを維持、cDNA ライブラリ生成のポジティブ コントロールとして、少なくとも 1 つのサンプルを留保します。ステップ 6 まで 4 ° C でこのコントロールを格納します。残りのサンプルにローディングの染料 × 6 の 2 μ L を追加、5 回上下ピペッティングによるサンプルをミックス 〜 12 μ L に徹底的に総容積をもたらします。
- 染料と水の 7 μ L ロード x 6 の 2 μ L で 1 μ L の DNA の梯子を組み合わせます。電気泳動のコントロールとしてゲル seq カセットの 1 車線にこの混合物をピペットします。ゲル seq カセットの別のレーンには、前の手順からサンプルをピペットします。各ウェルにピペットを完全に挿入して、垂直方向の運動だけを使用してそれを削除する、ウェル間の汚染を防ぐために注意してください。
- 標準的なゲル電気泳動電源装置の使用、cDNA/RNA ハイブリッド車から、gDNA を分離して 30 分間ゲル seq カセットに 250 V の電界を適用できます。一度分離、ゲル電気泳動室からゲル seq カセットを削除し、狩りのツールでエッジをこじったカセットの 2 つの半分を開きます。
- メスを使用して、高密度の層のすぐ下半分にゲルをカット.あなたの手袋をはめた手で拾い、フィラー ゲルを含まれている半分を破棄します。ペイント スクレーパーまたはその他の同様のツールでそれをこするでカセットからは残りのゲルの皮をむく優しく。0.5 〜 30 mL を含んでいる皿にゲルのこのセクションを置くゲル染色の 3 μ L で x TBE。
- 退色を最小限にし、5 分のコンテナーを軽く振りながらゲルを浸すコンテナーをカバーします。ジェルをプラスチック製のラップとゲルのドキュメンテーション システム (さらに詳細を参照文献 13) を使用して紫外線画像を撮影します。30 秒の露出は通常、クリアな画像を生成します。分離が発生したことを確認します。
- ゲルを核酸の可視化を容易にする UV transilluminator に移動します。適切な UV のメガネを着用、ゲルのドキュメンテーション システムからの結果を確認します。GDNA 低密度のゲルおよび低密度および高密度領域の界面における cDNA の先頭に置く必要があります。
- メスを使用する、cDNA と gDNA を含んでいるゲルの領域を切り取る。サンプルは、ゲルの 10 mm の長方形断面で 4 mm を切断することによって回復最高ただし、正確なジオメトリは使用するゲル電気泳動システムの異なります。またネガティブ コントロール搭載レーンをカットしてください。
- 各場所は、鈍い端ピンセットのペアを使用してストリップ管にゲル部を切除しました。無理な力がかからないように注意してくださいまたはゲルは複数の部分に分割されます。べきであるこれ起こる単に各作品を選んで、チューブを追加します。
- 各管内管の下部に円形のファッションでピペット チップを移動することによって (200 μ L ピペットのヒント仕事よく) ピペットの先端を使用してゲルを挽きます。サンプルの損失を最小限に抑えるためにゲルを挽くために使用するピペット チップを削除する前に、各チューブにヌクレアーゼ フリー水 (gDNA サンプルに 40 μ L と cDNA サンプルに 80 μ L) を追加します。
- 37 ° C の定温器の内部渦のミキサーにストリップ管を配置し、8-12 時間のために振る。これにより、ゲルから拡散する核酸とは自然停止日、このマルチ ・ プロトコルのポイント。
- 8 μ m メッシュ フィルター プレートにサンプルをピペットし、ゲル断片を負担する 5 分の 2600 x g で板を回し。住宅板からメッシュ フィルター プレートを持ち上げて新しい 200 μ L ストリップ管にピペットの水ゲル無料サンプル。
- 上下、ピペッティングで混ぜる gDNA を含む各サンプルにプロテアーゼ (0.9 AU/mL) の 1 μ L を追加し、50 ° C、15 分間 70 の ° c の熱不活化に続いて 15 分インキュベートします。この手順は、ヌクレオソームを枯渇させるために重要ですし、gDNA を後続反応工程のアクセスが可能になります。
- 18 ゲージ針を使用して、すべてのサンプル チューブのキャップの穴を突きます。液体の体積を減らすために vacufuge にサンプルを配置します。gDNA サンプルは、10 μ L に減少 5 μ L と cDNA サンプルに還元すべき。
- Vacufuge、サンプル数に応じて全蒸発時間は 30 分から 60 分間異なります。サンプル ボリュームはターゲット ・ ボリュームを下回った場合単にサンプル量を増加するヌクレアーゼ フリー水を追加します。
5. gDNA ライブラリの準備
- 蛍光光度計または同様のテクノロジを使用して、手順 3 から肯定的な制御と同様、ステップ 4 から各 gDNA サンプル中の DNA 濃度を定量化します。詳しいプロトコルは、蛍光光度計・ リファレンス ・ マニュアルを参照してください。14
- 0.2 ng/μ L の DNA サンプルを希釈します。開始セルの種類と必要な結果の品質によって低濃度はまだ実行可能なライブラリを生成可能性があります。いくつかの実験が必要です。しかし、著者はライブラリ 0.1 ng/μ L の低で成功を収めています。
- GDNA ライブラリの準備を完了するには、次のステップ 7 でライブラリの準備半分反応のボリューム プロトコルします。
6. cDNA ライブラリの準備
- 10 μ L cDNA サンプルと手順 4 から肯定的な制御、2 X qPCR ミックス、0.5 μ L cDNA PCR プライマーと 2 μ L ヌクレアーゼ フリー水の 12.5 μ L を追加します。
- 次のプロトコルを使用してリアルタイムたちで PCR を実行: 95 ° C、3 分、10 の 98 ° C の 20-30 サイクル後にホット スタート s、65 ° C 30 s、および 72 ° C 3 分総サンプル量は 25 μ L 反応曲線を監視および停止する前に増幅。e 反応 overamplification のため PCR アーティファクトを避けるために指数段階 (サイクル数と線形信号増加) を残します。Overamplification の回避については、参考文献 15 と同様に、この論文の議論を参照してください。
- 増幅した後次の手順 8 でプロトコル固相リバーシブル固定 (スプリング) ビーズを用いた製品をクリーニングします。完了するは、次の手順に進みます。
- 蛍光光度計または同様のテクノロジを使用して、手順 4 から肯定的な制御と同様、各 cDNA サンプル中の DNA 濃度を定量化します。約 0.2 ng/μ L の DNA が含まれている必要がある場合は、サンプルを希釈します。若干低い濃度はまだ実行可能なライブラリを生成します。いくつかの実験は、しかし、著者らは、ライブラリ 0.1 ng/μ L の低で成功を収めて、必要があります。
- オプションのステップ: ライブラリ生成反応を検証する qPCR 製品働いた各 1-2 μ L を標準のポリアクリルアミドゲル電気泳動分離を実行します。成功した結果のサンプル画像を図 4に示します。ポリアクリルアミドゲル電気泳動を実施する方法の詳細なプロトコル、Ref. 16 を参照してください。
- CDNA ライブラリの準備を完了するには、次の手順 7 でライブラリの準備キット ボリュームは半分反作用のプロトコルします。
7 ボリュームは半分反応ライブラリの準備
- 図書館準備半分のボリュームの反応を使用してライブラリの準備キットのプロトコルに従います。17図書館準備からプロトコルのこのセクションで参照されているすべての試薬はキット (材料の表を参照してください)。処理されるサンプルの数のストリップ管の十分な数を殺菌紫外線によって始めなさい。
- トランスポザーゼの反応を実行するには、アッセイに使用するには、各ストリップ管に 5 μ L トランスポザーゼ バッファーを追加します。2.5 μ L トランスポザーゼ続いて 2.5 μ L 入力 DNA 0.2 ng/μ L (0.5 ng 合計) でを追加します。上下に 5 回ピペッティングで混ぜます。
- 55 ° C で 5 分間インキュベートし、10 ° C に保持サンプルでは、10 ° C に達すると、たちからそれを削除し、すぐに各サンプルに 2.5 μ L トランスポザーゼ停止バッファーを追加します。サンプルを室温で 5 分間保持します。
- 7.5 μ L 図書館準備 PCR ミックス、インデックス 1 プライマーの 2.5 μ L およびインデックス 2 プライマーの 2.5 μ L を追加することによって扱われる転置サンプルを増幅する PCR 反応を準備します。これらのプライマーは、独自ライブラリ作成キットの製造元によって提供されます。上下に 5 回ピペッティングでよく混ぜます。
注: は、各サンプルに特異的プライマーの組み合わせを使用するを確認します。12 別のインデックス 1 のプライマーと最大 96 の異なるサンプル ラベルを一意にする 8 の別のインデックス 2 プライマーがあります。各サンプルのためのプライマーのユニークな組み合わせを選択します。 - PCR、たちに次のプログラムを使用してを実行します。サンプル量は 25 μ L です。
72 ° C、3 分
95 ° C、30 秒
12 サイクル:
10 秒 95 ° C
72 ° C、30 秒
55 ° C、30 秒
72 ° C、5 分
10 ° C に保持します。 - 次の手順 8 でプロトコル SPRI ビーズを使用して準備されたライブラリをクリーンアップします。ライブラリは、ゲル電気泳動法または類似の試金を使用して検証する必要があります。ライブラリを検証する方法の詳細については、ライブラリの準備キット マニュアルを参照してください。17
8. 固相リバーシブル固定化ビーズ ライブラリのクリーニング
- 各使用する前に室温に固相リバーシブル固定 (スプリング) ビーズ 1.5 ml に格納する必要がありする必要がありますをもたらした。また、各実験のため新鮮な 80% エタノールを準備することをお勧めします。次の手順が基づいている WTA のキット マニュアルに SPRI ビーズ プロトコル。18
- WTA キット換散バッファーの 1 μ l を各 PCR の製品に追加します。SPRI ビーズまで均等に混合すると、渦は SPRI ビーズの 50 μ l を各サンプルに追加します。上下に 10 回サンプルをピペッティングで混ぜるし、8 分間室温でサンプルをインキュベートします。
- 管の側から液体を収集するサンプルを簡単にスピンします。磁気分離装置で液体が完全に明確表示されますまで ~ 5 分のサンプルを配置します。
- サンプルは、磁気分離装置にされますが、ゆっくりと上澄みと破棄をピペット - チューブ上のビーズのリングを邪魔しないように注意してください。次に各サンプルにビーズを乱すことがなく 80% エタノール 200 μ L を追加します。30 秒間待ってから、上澄みをピペットの慎重に。この手順 (エタノール洗浄) 一度。
- 30 乾燥するサンプルを許可する秒 - 1 分。サンプルは、大きいフラグメントはビーズになる永続的にバインドされている、過剰乾燥しないでください。
注:、エタノールのすべてのトレースを削除するには、短い乾燥時間の使用をお勧めします。微量エタノールの後ろに残るべきである彼らは少し下流の反応を抑えることができます。 - 磁気分離装置から、サンプルを削除し、ビーズから DNA を溶出するには、各サンプルに 15 μ L の水を追加します。ピペットの上下とビードがチューブの側面から削除されることを確認します。2 分間室温でインキュベートします。サンプルを簡単にスピンし、~ 1 分解決策がはっきり表示されるまでの磁気分離装置にそれらを配置します。
- サンプルは、磁気分離装置にされますが、ゆっくり上澄みをピペットし、クリーン チューブにそれを転送します。チューブ上のビーズのリングを邪魔しないように注意します。ビーズ チューブを破棄します。
結果
蛍光イメージング; 的ゲル seq デバイス/RNA の cDNA と gDNA 雑種の物理的な分離に視覚化できます。代表的な結果を図 3に示します。パネルを示しますゲル seq 素子を試作偽色が異なるゲル領域を区別するために追加されました。パネル B を検証に使用される 4 つの異なる分離のクローズ アップ表示します。第 3 レーン、ネガティブ コントロールは、背景を表し、界面ゲルの autoflourescence がないことを示しています。我々 は DNA の梯子の最初と 2 番目のレーンを読み込まれます。これらの車線は低高密度膜間、明らかに小さい片は低密度のゲルが通過できるインターフェイスで暗いバンドのみを表示します。第 4 レーンが興味の生物的サンプルの動作を示しています: 500 PC3 細胞。プロトコルの手順 3 で説明した 4 つの車線を読み込みます。画像は、ゲノム DNA と cDNA/RNA 雑種の分離を示しています。低密度の膜の上部に暗いバンドは megabase スケール ゲノム DNA cDNA/RNA ハイブリッド車は、低・高密度領域のインターフェイスに積層されています。はしごを搭載の車線とは異なり、ゲルの高密度領域内に存在複数のバンドもあります。これらのフラグメントは、100 より小さい bp、オフのターゲット製品はオリゴヌクレオチドのプライマーから逆のトランスクリプションの間に生成されます。パネル C は、成功した実験から全体のゲル seq デバイスの代表的なイメージを示しています。レーン標識 RNA/cDNA は、RNA をラベルのレーン表示だけ gDNA と RNA の分離ゲル seq プロトコルで処理しました。パネル D は、低密度の膜のすべての車線の上部に黒い帯で実験の失敗を示しています。これは、断片化された DNA で汚染された電気泳動バッファーによって引き起こされました。この手順では、きれいなネガティブ コントロールと分離された gDNA と RNA/cDNA ハイブリッド車の存在を示す 2 つの明瞭な黒バンドの両方の研究を見るべき。

図 3.ゲル Seq 分離結果。ゲル seq デバイス (A) と DNA と RNA/cDNA ハイブリッド車 (B) の分離を示す蛍光イメージ。偽色がより簡単にゲル内の密度の異なる地域の間に区別するために追加されました。(C と D)成功 (C) と (D) 実験の失敗からゲル seq デバイス全体の代表的な蛍光イメージ。NTC = テンプレート コントロールはありません。この図の部分は、王立化学協会の許可を得て参考文献 7 から再現されました。この図の拡大版を表示するのにはここをクリックしてください。
DNA と RNA/cDNA のハイブリッド車が分離されているし、ゲル seq プロトコルの残りの部分を完了する、シーケンス ライブラリを生成することが可能です。準備されたライブラリを検証するには、標準的なゲル電気泳動実験 (図 4) または、バイオアナライザーのいずれかを実行します。図 4の結果は、500 の PC3 細胞と 750 の HeLa 細胞から生成されたライブラリを示します。ゲル-seq (ラベルは 'ゲル) (ラベルは 'チューブ') 標準プロトコルで生成される比類のないサンプルと比較してから生成された一致するライブラリのフラグメント分布図であります。標準の全ゲノム ライブラリー作製キットを使用してライブラリを準備する場合に期待どおり、200 と 800 のヒト間ゲル seq のフラグメント サイズが表示されます。ライブラリの断片は、この手順で正しいサイズの範囲では表示されません、ライブラリの準備が失敗しました。
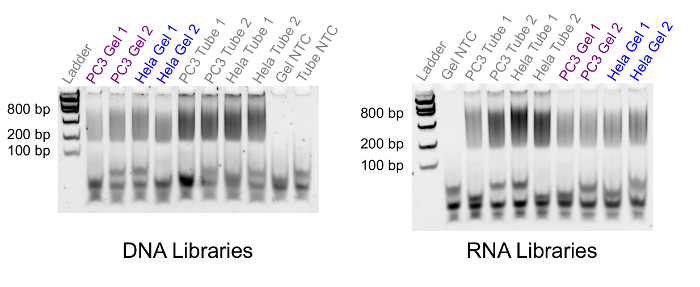
図 4.ライブラリ フラグメント サイズ比較します。ゲル seq (ゲル) と標準コントロール (チューブ) のライブラリ サイズ分布を比較する蛍光ゲル電気泳動イメージ。左車線には、フラグメント サイズ 100、200、400、800、1200、および 2000 ヒトと低質量 DNA ラダーが含まれています。このキットを使用したライブラリの期待どおりにすべてのライブラリのためのフラグメント サイズは 200 と 800 のヒト間落ちる。この図の拡大版を表示するのにはここをクリックしてください。
ゲル seq プロトコルの最終的な検証は、シーケンスの結果の分析に基づいています。我々 は検証実験の PC3 細胞を選択するこれらの細胞サンプルを許可する均一な発現プロファイルを持っている分割、ゲル seq と伝統的な方法の両方を使用して処理図 5を参照してください。図 5A 5 bに PC3 細胞のゲノム DNA の比較を示します。図 5A PC3 から生成されるゲノム コピー数変動 (CNV) プロファイルの比較を示していますゲル seq または標準の全ゲノム ライブラリ準備反応 (管コントロール) を使用しています。各ポイントは平均正規化されたビン数です。箱は参照ゲノム データから定義されるなど、各箱には、正常な 2 倍体の細胞、すなわち、フラット ライン、すべて常染色体優性の地域ごとの同じコピーを表すに等しい予期した数 (を除く X と Y) 染色体。PC3 には、上記の 2 つの背景コピー数のスパイクとして表示される同じ地域の複数のコピーが含まれています。ゲル seq 標準管反応として質的に同様の CNV プロファイルが得られます。2 つのプロット間の合意によって評価される定量的線形回帰パネル B. で示すように、ピアソン相関 r = 0.90 は、ゲノムデータのいずれかのメソッドから収集が機能的に同等であることを示します。

図 5.ライブラリ検証します。ゲノムのゲル seq 検証 (A および B) および PC3 と Hela 細胞から生成されるトランスクリプトーム (C、D、および E) データ。パネル A の比較を示します PC3 から生成されるゲノム CNV プロファイルのゲル-seq (ゲル seq) または標準的な反応 (チューブ) のいずれかを使用します。MAPD 中央ペア差分の絶対値を =。パネル B は線形回帰パネル A の 2 つのサンプルの間で R = 0.90 を示すゲノムのデータが機能的に同等です。軸は、log2 正規化された箱のカウントを表示します。C と D のパネルあたりプラスミドあたりの成績証明書の数を示す各ポイントに PC3 細胞トランスクリプトーム データの比較百万 (TPM)。軸は正規化 log2 トラン スクリプトの数として 2 つのサンプル間の比較を表示します。パネル C は seq、ゲルを使用して生成された技術的な反復実験の比較を示しパネル D ゲル seq と伝統的な RNA シーケンス パネル E の比較を示してゲル seq が主成分分析を用いた RNA 式に基づいてセルの種類を解決できることを。X 軸、y 軸を示す分散の 6.5% だけの第 2 主成分アカウント間のサンプルのばらつきの 91.6% 最初の主成分のアカウントを示します。この図の部分は、王立化学協会の許可を得て参考文献 7 から再現されました。この図の拡大版を表示するのにはここをクリックしてください。
同様に、私たちは標準のチューブでスマート Seq プロトコルに私たちゲル seq プロトコルからトランスクリプトーム データを比較しました。図 5は、両方ゲル seq 技術レプリケートします (図 5C) とゲル seq と標準的な方法 (図 5D) との間の相関関係を示しています。各ポイントあたりプラスミド毎の成績にはカウントは、百万 (TPM) TPM で検出された各遺伝子の両方のデータセットで > 5。線形回帰は赤い線として表示され、ピアソンの相関係数は、左上隅に表示されます。ゲル seq から技術的な複製 (R ∼ 0.8) を同意するが、あまりに関連付ける標準的な方法 (R < 0.7)。ゲル seq 導入遺伝子の数では、バイアスすることが示唆されました。幸いなことに、このバイアスは体系的なあり、図 5Eで主成分分析を見ることができる、異なる試料間にまだ描画できる有意義な結論。
ディスカッション
ゲル seq デバイス作製とプロトコル自体に関連付けられているいくつかの重要な手順があります。製作中にゲルの様々 な地域の所定の層の厚さで始まるをお勧めします。異なる製造オプションをテスト時間を大幅を過ごし、ここで説明したプロトコル [テーブルの材料および試薬カセットの最高のデバイスが生成されます。研究者は、代替カセット システムを使用している場合、デバイスを作成するときに使用されるボリュームを微調整するために必要な彼らがあります。作製での大きな課題は、高密度のゲル化領域が大きすぎて、カセットの端から剥離して電気泳動を妨げるカセットの内部の空気のポケットを作成する場合。冊別のレイヤーにいくつかのカセットをキャストすることによって研究者は、独自のハードウェアの最適構成をすばやく判断できる必要があります。
ゲル seq プロトコル プロトコルを完了する前に検証することができますいくつかの重要な手順があります。1 つの潜在的な障害ポイントは、gDNA RNA/cDNA の分離です。これは分離後ゲル seq デバイスをイメージングによって検証できます (図 3Bを参照)。1 つの一連の実験では、バッファーの私達の実験室の供給と DNA が汚れていたし、我々 のデバイスで実質的な autoflourescence を引き起こしていたことがわかった (図 3D参照)。これは、分離が起こったかどうかを決定することは困難です。蛍光イメージングを識別し、シーケンス ライブラリを生成する任意の高価な試薬を使用する前にこの問題を解決することができました。
もう一つの重要なポイントは、ステップ 6.2, 分離後の cDNA の qPCR の増幅です。研究者は、RNA シーケンス データの質を減らすので、この手順で overamplify しないように細心の注意を払う必要があります。この考察はゲル seq に一意ではありませんが、低入力 RNA シーケンス ライブラリ準備の一般的な側面です。ライブラリの準備をシーケンス処理中に PCR 増幅が必要です, 多くの場合が、それはシーケンス エラーとバイアスを導入することができます。必要な PCR のサイクル数は、サンプル量と複雑さによって異なります。それは一般的にライブラリがシーケンス処理されると、十分なクラスターを生成するために必要な最低限に PCR のサイクル数を制限することをお勧めします。理論的には、過剰なアーチファクトを導入せず十分なコピーの数を生成する正確なサイクル数を決定するプロトコルを最適化できます。実習では、ただし、サンプル品質の矛盾ロード、またはプロトコルの早い段階で処理が劇的に影響 PCR に最適な PCR のサイクル数に影響を与えるライブラリの準備のため使用可能な分子テンプレートの配布。リアルタイム PCR たちの反応を実行我々 は蛍光色素を用いた増幅反応の進行状況を監視する発見した最も一般的なソリューションとの指数 (サイクル数と線形) の反応を停止相。私たちの経験、リアルタイム監視の開発、適応、または新しいプロトコルを採用するときに特に関連するですが。
最後の重要なステップが生成してゲノムおよびトランスクリプトーム ライブラリ。このステップへの鍵は、できるだけ 0.2 ng/μ L (0.5 ng 合計) に近い DNA ライブラリの準備反応の開始サンプル濃度を設定します。通常、cDNA の過剰があるが、gDNA サンプルが困難ことができます qPCR 増幅 cDNA の比較的簡単です。注意 vacufuge 手順に注意は必要なサンプルが集中していたことがわかった。Vacufuge ステップが多く停止することができる 1000 細胞を用いた実験で、期待どおりに 100 の実験よりも早く細胞します。Vacufuge のサンプルの数は、我々 の実験蒸発速度も影響します。不慣れなサンプルとプロトコルを実行するときは、濃度ステップの途中を役立つことができるコンテンツ、flourometer を使用して DNA を検証することがわかった。幸いなことに、水のサンプル、ヌクレアーゼ フリーの集中の上研究者場合は、サンプルを希釈に追加します。理論的には、それは DNA を乾燥させると、希望の音量で再懸濁します、vacufuge を使用することが可能ただし、完全な蒸発を避けることをお勧めします。
我々 は博士 seq9、無料として、G & T seq8、ゲル seq7同時 DNA および RNA のライブラリを生成するための 3 つの現在の方法を表示します。ゲル seq、100-1000 セル範囲のサンプルに最適プルダウン ・ ターゲットやゲノムの事前知識は必要ありません。他の 2 つの方法単一電池への応用に適しています。ゲル seq の開発の目的の 1 つは、他の研究者によって簡単に実装可能なプロトコルを作ることでした。したがって、内ポリアクリルアミドゲル カセット テープの標準的なフォームファクターのデバイスを作製することを決めた。私たちは私たちの異なる膜の定義に使用法は小説、ほとんど遺伝学研究所既にゲル seq デバイスを製造するためのすべての必要な機器。さらに、デバイスのコストは簡単です - ちょうど $5.25 12 個の試料を処理することができますデバイスの。つまり、他ライブラリの準備のプロトコルと市販試薬を使用してライブラリを生成するための全体的なコストは高いままです。サンプルあたり試薬コストは全体転写増幅の $50 と $28 DNA と RNA の両方のライブラリの準備のためでした。幸いにも、ゲル seq デバイス自体は、プロトコルに依存しないです。たとえば、開発中に我々 正常にテスト文化から、古い RNA ライブラリ増幅プロトコル19セルを使用して、デバイスがマウスから組織サンプルに適してなかったとわかった。ライブラリの準備のための安価な代替案を開発する、未来に向かっている、我々 のプロトコルは、これらの新しい技術で動作するようになります。研究者は、それが彼ら独自のラボでゲル seq を実装するは簡単でしょうと考えています。これは、技術の急速な普及を促進すると思います。
開示事項
カザフスタンは、共同創設者兼 Singlera ジェノミクス社の科学アドバイザー
謝辞
この仕事のための資金は、国立科学財団大学院研究フェローシップ ・ プログラム、サン ディエゴ大学 NIH R01-HG007836 を与えると科学、ICT および将来計画の韓国大臣によって。
以前のバージョンのいくつかの数字が"・ ザ ・ フープル、g. d.らで出版された最初ゲル-seq: 全ゲノムとトランスクリプトーム シーケンスによる半透性ハイドロゲル障壁を用いた同時低入力 DNA および RNA 図書館準備によって。ラボオン チップ 17、2619-2630 doi:10.1039/c7lc00430c (2017)."ラボオン チップは、このパブリケーション内の図の再利用を認可しています。
資料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate - Corning HTS Transwell 96 well permeable supports - 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
参考文献
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18(2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31(2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908(2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), at http://science.sciencemag.org/content/352/6293/1586 (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923(2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies. , Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017).
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- A Guide to Polyacrylamide Gel Electrophoresis and Detection. Bulletin 6040 Rev B. Bio Rad. , Available from: http://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6040.pdf (2017).
- Illumina. Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved