Method Article
ג'ל-seq: שיטת הכנת ספריה סימולטני רצף DNA ו- RNA באמצעות מטריצות הידרוג
In This Article
Summary
ג'ל-seq מאפשר חוקרים להכין בו זמנית ספריות עבור שניהם הדנ א - ו RNA-seq זניח עלות נוספת החל מ- 100-1000 תאים באמצעות מכשיר הידרוג פשוטה. המאמר מציג גישה מפורטת על הזיוף של המכשיר, כמו גם את הפרוטוקול הביולוגי כדי ליצור ספריות לזווג.
Abstract
היכולת להגביר את רצף ה-DNA או RNA מדגימות התחלה קטנה הושגה רק בחמש השנים האחרונות. למרבה הצער, פרוטוקולים סטנדרטיים עבור יצירת גנומית או ספריות transcriptomic אינם תואמים, חוקרים עליך לבחור אם רצף ה-DNA או RNA מדגם מסוים. ג'ל-seq פותר בעיה זו בכך שהוא מאפשר לחוקרים בו-זמנית להתכונן ספריות הן DNA ו- RNA החל מ 100-1000 תאים באמצעות מכשיר הידרוג פשוטה. המאמר מציג גישה מפורטת על הזיוף של המכשיר, כמו גם את הפרוטוקול הביולוגי כדי ליצור ספריות לזווג. עיצבנו ג'ל-seq. כך זה יכול להיות מיושם בקלות על ידי חוקרים אחרים; מעבדות גנטיקה רבים יש כבר את כל הציוד הדרוש כדי לשחזר את ייצור המכשיר ג'ל-seq. פרוטוקול שלנו מעסיקה נפוצות ערכות עבור שניהם הגברה שלם-תעתיק (של WTA), ספריית הכנה, אשר הם גם נוטים להיות מוכר חוקרים כבר שבקיאים גנומית ושוויונית transcriptomic ספריות. הגישה שלנו מאפשר לחוקרים להביא לשאת את הכוח של רצפי DNA ו- RNA על דוגמה אחת ללא פיצול ועם זניח בתוספת מחיר.
Introduction
הדור הבא רצף (הגדרות) יש לו השפעה עמוקה על הדרך מתנהל מחקר גנטיקה. כאשר חוקרים התמקדו פעם רצף הגנום של זן שלם, עכשיו זה אפשרי את רצף הגנום של גידול יחיד או אפילו תא בודד בניסוי אחד. 1 הגדרות גם הפכה חסכוניות כדי רצף התרשימים RNA נמצא בתוך תא, אוסף של נתונים המכונה של transcriptome. היכולת להגביר את רצף ה-DNA או RNA מדגימות התחלה קטנה הושגה רק בחמש השנים האחרונות. 2 , 3 , 4 . למרבה הצער, פרוטוקולים סטנדרטיים אינם תואמים, חוקרים עליך לבחור אם רצף ה-DNA או RNA דגימה נתון. כאשר מדגם ההתחלתי הוא גדול מספיק, ניתן לפצל אותו לשניים. קשקשים קטנים יותר, עם זאת, אובדן של חומר עקב פיצול דגימות יכולים להשפיע על איכות ספריה ולאחר איגום של דגימות יכול ממוצעים גרסאות מעניינות בין תאים. 5 יתר על כן, החוקרים מעוניינים יותר ויותר בחינת והמדגמים יכולה להתפצל, כגון תאים בודדים או ביופסיות גידול קטן הטרוגנית. 6
כדי לטפל בבעיה זו, שלושה פרוטוקולים לאחרונה פותחו כדי רצף ה-DNA ו- RNA מדגם המוצא זהה: ג'ל-seq7, G & T-seq8ו ד ר-seq9. מאמר זה מציג פרוטוקול מפורט עבור ג'ל-seq, אשר יכול לשמש כדי בו-זמנית ליצור ספריות DNA ו- RNA מתוך כמה כתאי 100 זניח עלות נוספת. ההיבט הרומן של ג'ל-seq היא היכולת להפריד בין DNA ו- RNA המבוסס באופן בלעדי על גודל באמצעות מטריצות הידרוג בעלות נמוכה. החידוש הליבה של פרוטוקול ג'ל-Seq הוא ההפרדה הפיזית של ה-DNA מ- RNA. ההפרדה הזאת מושגת electrophoretically באמצעות שילוב של ממברנות לזיהוי לנצל גודל ההבדלים בין מולקולות אלה. כדי לשים את ההבדלים בגודל בהקשר, שקול כיצד DNA ו- RNA הם צילמו: בעוד דנ א קיים בסולם מיקרון ניתן להציג באמצעות מיקרוסקופים מסורתיים, RNA קיים בסולם ננומטר ולא חייב לדימות תוך שימוש בטכניקות מורכבות כגון אלקטרונים הקפאה מיקרוסקופ. 10
הגישה הפרדת DNA ו- RNA, פרוטוקול זה מוצג באיור1. החלונית השמאלית מציגה DNA ו- RNA חינם צף בפתרון בקרבת קרום. כאשר שדה חשמלי מוחל, כפי שמוצג בלוח הימני, DNA ו- RNA לחוות כוח electrophoretic שגורם העברה דרך הקרום. על-ידי כיוונון המאפיינים ממברנה, יצרנו קרום חדיר למחצה המפריד DNA RNA. מולקולות DNA יידחפו נגד הקרום, אבל להיות מסובכת בקצה בשל גודלם גדולים. מצד שני, מולקולות RNA קטנות, ניתן להגדיר מחדש, לארוג את דרכם דרך הקרום. תהליך זה, המכונה reptation, דומה לאופן בו שנחש עובר דשא. בסופו של דבר אלה מולקולות RNA מופסקים על ידי קרום השני, בצפיפות גבוהה זה קשה מדי עבור פולימרים אפילו קטנה יותר (> 200 זוגות בסיס) להתחמק על דרך. ברגע פיזית מחוזק, DNA ו- RNA יכול להיות התאושש לעבד להפיק מידע על הגנום והן transcriptome. בעוד נוכל להפריד DNA ו- RNA, מצאנו תוצאות טובות יותר מתקבלים אם ה-RNA היא הפוכה עיבד כדי cDNA לפני ההפרדה. היברידים רבים cDNA/RNA יציבים יותר RNA לבד, עדיין יכול לעבור דרך קרום בצפיפות נמוכה.

איור 1 . עקרון ההפעלה ג'ל-seq. העיקרון הבסיסי המשמש להפרדת פיזית של DNA ו- RNA. בשדה חשמלי יישומית, מולקולות RNA קטנות להעביר דרך קרום בצפיפות נמוכה אבל מולקולות DNA גדולות לכוד על פני השטח. איור זה שוחזר הפניה למעורר 7, ברשות החברה המלכותית לכימיה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
מאמר זה מתאר בפירוט בשני הזיוף של המכשיר ג'ל-seq, הפרוטוקול הביולוגי ליצירת לזווג ספריות DNA ו- RNA. סקירה כללית של שניהם מוצג באיור2. המכשיר מפוברק על ידי שכבות 3 צפיפות שונה לזיהוי ג'לים אחד על גבי השני בתהליך דומה ליצירת תקן ג'לים הערימה. 11 הפרוטוקול הביולוגי מתחיל עם תאים 100-1000 הושעו ב- PBS. התאים הם lysed, הרנ א מומר cDNA לפני המכשיר משמש להפריד את הדנ א כלאיים cDNA/RNA. לאחר ההפרדה, התאוששות, גנומית, transcriptomic ספריות מוכנות באמצעות תהליך זה מקרוב כדלקמן פרוטוקול ערכת הכנה הספרייה הגנום כולו. ניתן לקרוא פרטים נוספים על פיתוח, אימות של ג'ל-seq במעבדה על פרסום שבב "ג'ל-seq: הגנום כולו ורצף transcriptome על ידי סימולטני קלט נמוך-DNA ו- RNA ספריית הכנה באמצעות מחסומים הידרוג חדיר למחצה ." 7

איור 2 . פרוטוקול ג'ל-seq. מבט כולל על הצעדים כדי לפברק את המכשיר ג'ל-seq והפרוטוקול מחוללות לזווג ספריות DNA ו- RNA. חלקים של איור זה שוחזר הפניה למעורר 7, ברשות החברה המלכותית לכימיה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
כדי ליצור ספריות DNA ו- RNA מתוך תאים בודדים, חוקרים, כדאי שתשקול להשתמש או G & T-seq או ד ר-תת סעיף. G & T-Seq, כמו ג'ל-seq, מסתמך על הפרדה פיזית של רנ א מ- DNA גנומי. גישה זו נסמכת על messenger RNA (mRNA) 3′ polyadenylated זנב כמטרה נפתחים. ה-mRNA נלכד על חרוז מגנטי באמצעות פריימר oligo-dT biotinylated. ברגע mRNA כבר נתפס החרוזים הם שנערך במקום עם מגנט, תגובת שיקוע המכיל את הדנ א הגנומי יכול להיות הוסר, הועבר צינור אחר. בתום ההפרדה הפיזית הזאת, ספריות שונות ניתן להפיק מן ה-mRNA ו DNA. 8 גישה זו פועלת היטב אם הרנ א עניין polyadenylated, אולם אין אפשרות להשתמש ללמוד תעתיקים הלא-polyadenylated, כגון ribosomal RNA, tRNA, או RNA של אאוקריוטים.
ד ר-seq מסתמך על צעד הגברה מראש לאן דנ א והן cDNA נגזר מן ה-RNA הם מוגבר בצינור אותו. המדגם ואז לשתיים, מעובד במקביל להכין ספריות seq DNA ו- RNA. כדי להבחין בין DNA גנומי של cDNA נגזר מן ה-RNA, ד ר-seq מקבלת גישה חישובית. רצפים היכן נמצאים רק exons מדוכאים שהמפתחות בנתונים דנ א גנומי, כמו אלה יכול להיות שמקורם מן ה-DNA או RNA. 9 היתרון בגישה זו הוא כי ה-DNA ו cDNA/RNA צריך להיות פיזית מופרדים זאת על ג'ל-seq ו G & T-תת סעיף. החיסרון, לעומת זאת, היא ד ר-seq דורשת ידע א-פריורי של הגנום, transcriptome (כלומר, exons לעומת אינטרונים), לא יהיה אידיאלי עבור יישומים כגון רצף של גרעינים, שבו תעתיקים רבים עדיין אינם לגמרי משולבים, עדיין להכיל אינטרונים. 12
ההיבט הרומן של ג'ל-seq היא היכולת להפריד בין DNA ו- RNA מאות תאים המבוסס באופן בלעדי על גודל. שיטה זו דורשת שום ידע אפריורי של הגנום או transcriptome, היא חזקה נגד שחבור לא שלם, אינה מוגבלת תעתיקים פולי-adenylated. עבור יישומים שבהם חוקר יכול להתחיל עם תאים לפחות 100, ג'ל-seq מספקת גישה ישירה באמצעות חומרים נרחב זמין וזול.
Protocol
1. פתרון כימית הכנה
הערה: השלבים הבאים הם להכנת כימי הפתרונות הנדרשים בשלבים מאוחר יותר. אלה יכולים להיות גרם בצובר ומאוחסנים במשך מספר חודשים.
- כדי להתחיל, להכין 50 מ של מים מטוהרים, יונים על-ידי לחיטוי בתנור 254 ננומטר UV crosslinking למשך 15 דקות (15 mJ/cm2 הכולל חשיפה) לנטרל. את דנ משחיתה. מחממים עד 37 ° C לשימוש בשלבים.
- להפוך את 10 מ"ל של 40% סה כ (T) ו- 3.3% crosslinker (C) (29:1) לזיהוי קודמן פתרון. שוקל 3.867 g של אקרילאמיד מונומר ו- g 0.133 אינץ של bis-אקרילאמיד מונומר. לשלב ולהביא נפח עד 10 מ ל מים חמים מטוהרים, מערבולת עד התפרקה. חנות מוגן מפני אור בטמפרטורת החדר.
הערה: מעורבבים מראש 40% T, פתרונות לזיהוי של 3.3% C (29:1) ניתן לרכוש באופן מסחרי. - להפוך 10 מ"ל של 50% T, פתרון ג'ל 5% C. שוקל 4.750 g של אקרילאמיד מונומר ו- g 0.250 של bis-אקרילאמיד מונומר. לשלב ולהביא נפח עד 10 מ"ל באמצעות מים חמים מטוהרים ו מערבולת עד התפרקה. חנות מוגן מפני אור בטמפרטורת החדר.
- להפוך 10 מ"ל של פתרון סוכרוז 50% (w/v). להוסיף 5 g סוכרוז משורה ולהוסיף מים מטוהרים חמים עד הנפח הכולל של 10 מ"ל. מערבולת עד התפרקה, חנות בטמפרטורת החדר.
- להפוך את 10 מ"ל של 10% APS (w/v). להוסיף 1 g של קירור (APS) משורה. להוסיף קר (~ 4° C) טיהור מים הנפח הכולל של 10 מ"ל ו מערבולת עד התפרקה. להקפיא מיידית ב- aliquots של 200 µL.
2. ג'ל-seq קלטת פבריקציה נוספת
הערה: ג'ל-seq פותחה במקור עם קלטות זקוף (לקבלת מידע נוסף, ראה טבלה של חומרים ); עם זאת, פרוטוקול זה יכול להיות מותאם לעבודה עם כל קלטת אלקטרופורזה ג'ל רגיל.
- הכנת ג'ל מבשרי שלושה צינורות פלסטיק נפרדות על-ידי הוספת ריאגנטים כמוצג להלן ב- טבלה 1. לא הוספת APS או Tetramethylethylenediamine (TEMED) עד ביים בשלבים הבאים. רכיבים מערבולת לערבב ביסודיות.
| מילוי ג'ל קודמן | צפיפות גבוהה ג'ל קודמן | קודמן ג'ל בצפיפות נמוכה | |||
| 40 %T, 3.3%C אקרילאמיד Bisacrylamide פתרון | מ ל 1.6 | 50 %T, 5 %C אקרילאמיד Bisacrylamide פתרון | 2.4 מ | 40 %T, 3.3%C אקרילאמיד Bisacrylamide פתרון | 0.6 מ"ל |
| מים יונים | 10.2 mL | מים יונים | 1.0 mL | מים יונים | 4.8 מ |
| פתרון סוכרוז (50% w/v) | 2.6 מ | פתרון סוכרוז (50% w/v) | 0.6 מ"ל | ||
| 10 X טריס-בוראט-EDTA | מ ל 1.6 | 10 X טריס-בוראט-EDTA | 0.6 מ"ל | ||
| קירור (10% w/v) | 104.0 ΜL | קירור (10% w/v) | 50.0 ΜL | קירור (10% w/v) | 39.0 ΜL |
| TEMED | 6.0 ΜL | TEMED | 1.0 ΜL | TEMED | 2.2 ΜL |
| הנפח הכולל | 16.1 מ | הנפח הכולל | מ ל 4.1 | הנפח הכולל | 6.0 mL |
טבלה 1. ג'ל סינתזה ריאגנטים. ריאגנטים קודמן לזיהוי ג'ל מספיק עבור ייצור של שתי מגרות.
- דה-גז ג'ל מונומר פתרונות על-ידי החדרת מחט דרך הכיפה של הצינור וחיבור את המחט למקום שיש בו קו ואקום של הבית. להטביע את מכלול ערכה באמבט אולטרא גבוהה ולחכות עד בועות לעצור העולה מן הנוזל לפני המעבר לשלב הבא (~ 60 שניות / צינור).
הערה: האיכות של שואב האבק והכוח של האמבטיה אולטרה סאונד אינה קריטית כל עוד בועות ניתן לראות העולה מן הפתרון. - להוסיף TEMED ו APS מילוי ג'ל קודמן ו מערבולת בקצרה. מיד להוסיף 6 מ של מילוי ג'ל למבשר שכל קלטת ג'ל על-ידי pipetting הפתרון לתוך העליון של הקלטת. השתמש micropipette 1 מ"ל כדי להוסיף למבשר בדרגות 6 כדי להימנע לשפוך. הזמן הכולל בשלב זה צריך להיות פחות מ 3 דקות.
- ודא שהנוזל הוא רמה על-ידי הצבת הקלטות זקופה על רמה השולחן. למלא את השארית בקלטת יונים, degassed מים. Pipette המים, שוב באמצעות במרווחים 1 מ"ל, אט-אט אל מרכז הקלטת כדי למזער את ערבוב. לאפשר הפולימר לרפא לפחות שעה אחת, עד ללון.
- לאחר הפולימר ריפאה, היפוך הקלטות ג'ל מעל הכיור כדי להסיר את הכיסוי מים. אקדח אוויר דחוס יכול לשמש בעדינות לייבש את הממשק על ידי פיצוץ אוויר דרך הפתח העליון בקלטת ממרחק של 6 אינץ '.
- להוסיף TEMED ו APS קודמן ג'ל בצפיפות גבוהה, מערבולת בקצרה. מיד להוסיף µL 320 למבשר בצפיפות גבוהה הקלטת, שוב באמצעות פיפטה של 1 מ"ל. ודא שהנוזל באופן שווה ציפוי השכבה ג'ל מלוי במוזיקת את הקלטת אחורה וקדימה בסביבות 3 פעמים. שלב זה צריך לקחת פחות מ-3 דקות.
- למלא את השארית בקלטת יונים, degassed מים. פיפטה לאט עם פיפטה 1 מ"ל למרכז הקלטת כדי למזער את ערבוב. לאפשר הפולימר לרפא במשך 15 דקות לפחות, רצוי שעה.
- שוב, היפוך הקלטות ג'ל כדי להסיר את הכיסוי מים. ניתן להשתמש באוויר דחוס בעדינות לייבש את הממשק. להוסיף TEMED ו APS קודמן ג'ל בצפיפות נמוכה, מערבולת בקצרה. מיד למלא את השארית בקלטת (~1.65 מ"ל) למבשר ג'ל בצפיפות נמוכה והכנס את המסרק ג'ל.
- Pipette עודף של שמורת קודמן בחלק העליון של המסרק כמו זה ייקלטו בזמן הפילמור. לאפשר הפולימר לרפא לפחות 4 שעות, רצוי למשך הלילה. ג'לים ניתן לאחסן במשך שבוע או יותר במאגר של טריס-בוראט-Ethylenediaminetetraacetic חומצה (EDTA) (מאגר TBE).
3. משאבות הפוכה שעתוק
- החל השעיה של התאים של הריבית, ובעבודה של פולימראז תגובת שרשרת (PCR) למינארי, להשתמש hemocytometer או מונה אוטומטי תא הניתן לחישוב ריכוז תא. לדלל את התאים כדי ריכוז של 100 עד 1000 תאים לכל µL בתוך תמיסת מלח באגירה פוספט (PBS).
הערה: פרוטוקול זה אומתה על טווח של תאים כולל PC3, הלה ותאים העכבר בכבד. - בעזרת ריאגנטים הניתנים של WTA קיט (ראה טבלה של חומרים), מיקס µL 19 פירוק המאגר, µL 1 של מעכב RNase להכין 10 X במניה פתרון התגובה המאגר. ליצור שילוב מאסטר פירוק של נפח מספיק המכילה 0.5 µL התגובה המאגר, µL 2.75 של נוקלאז מים חינם עבור כל דגימה.
- Pipette התליה תא לאורך 5 פעמים מחדש להשעות תאים התיישבו, ואז פיפטה µL 1 מדגם לתוך צינור חשפנות חינם נוקלאז 200 µL מחוטא ע י UV. חזור על לפי הצורך בהתאם מספר הדגימות. הקפד לכלול פקד שלילי על-ידי pipetting µL 1 של נוקלאז מים חינם במקום תאים עבור תגובה אחת. בשלב הבא, להוסיף 3.25 µL של מיקס מאסטר פירוק כל מדגם ומערבבים על ידי pipetting בעדינות לאורך 5 פעמים.
- מחממים הצנטרפוגה תרמי (עם מכסה מחוממת) ל- 72 מעלות צלזיוס. להוסיף 1 µL של RT פריימר 1 µL של 20 מיקרומטר hexamer אקראיים עם מתאם של WTA (יונקות-AAGCAGTGGTAT-CAACGCAGAGTAC-NNNNNN-3′) כל דגימה. שמורת שפופרת אחת לפחות כפקד חיובי להכנה ספריית gDNA ולהוסיף 2 µL של מים במקום תחל.
הערה: hexamer אקראיים עם מתאם של WTA הוא אופציונלי ויש לו השפעה מינימלית על רצף תוצאות. - דגירה דגימות-72 מעלות צלזיוס הצנטרפוגה התרמי preheated במשך 3 דקות כדי lyse התאים. להסיר תאים הצנטרפוגה תרמי ומקום על קרח למשך 2 דקות. לאחסן את הפקד חיובי ב 4 ° C עד שלב 5.
- בעוד התאים הם lysing, ליצור נפח מספיק של שעתוק במהופך מיקס מאסטר עבור כל הדגימות RNA המכיל את היחס ריאגנט הבא: 2 µL הראשון מאגר סטרנד, 0.5 µL של תבנית מתג oligonucleotide (TSO), µL 0.25 של מעכב RNase ולאחר 1 µL של רוורס טרנסקריפטאז (100 U µL).
- מחממים הצנטרפוגה תרמי ל 42 º C. להוסיף µL 3.75 של מיקס מאסטר שעתוק במהופך הדגימות הנותרים, להביא את אמצעי האחסון הכולל דוגמה 10 µL. מיקס על ידי pipetting למעלה ולמטה 5 פעמים.
- לבצע שעתוק במהופך על ידי מיד הצבת דגימות הצנטרפוגה התרמי preheated. הפעל את התוכנית הבאה: 42 מעלות צלזיוס במשך 90 דקות, 70 מעלות צלזיוס למשך 10 דקות, 4 ° C לנצח. זוהי נקודת עצירה בטוח.
4. ג'ל ההפרדה מדגם שחזור
- לנקות בזהירות ג'ל אלקטרופורזה התא באמצעות מוצר הסרת DNA. להחיל מ"ל במספר של הסוכן ניקוי נוזלי מוך חד פעמיות חינם לנגב ולנגב על פני כל המשטחים של התא ולאחר מכן למלא את החדר עם 0.5 נקי x TBE. לקבלת תוצאות מיטביות, למקם את כל המתקן בתנור 254 ננומטר UV crosslinking ולחטא במשך 15 דקות (15 mJ/cm2).
- הכנס את הקלטת ג'ל-seq לתא ג'ל אלקטרופורזה ונעל אותה למקום. הסר את המסרק ג'ל על ידי משיכת ישר למעלה. זזים לאט למנוע קורע את הג'ל או העתקה מכל הזרועות.
- מונע את הדגימות שלב 3 על קרח, שומרים מדגם אחד לפחות כפקד חיובי לדור ספריית cDNA. לאחסן את פקד זה ב 4 ° C עד שלב 6. להוסיף 2 µL של 6 X טוען לצבוע את הדגימות הנותרים, מביא את הנפח הכולל µL ~ 12. ביסודיות לערבב את הדוגמאות על-ידי pipetting למעלה ולמטה 5 פעמים.
- לשלב 1 µL של סולם הדנ א עם 2 µL של 6 x בטעינת צבע ו- 7 µL של מים. Pipette תערובת זו לנתיב 1 בקלטת ג'ל-seq כפקד של אלקטרופורזה. Pipette דגימות מן השלב הקודם לתוך מסלולים נפרדים בקלטת ג'ל-seq. להיות זהירים כדי למנוע זיהום בין בארות על-ידי הוספת את פיפטה באופן מלא כל טוב והסרה אותו באמצעות רק תנועה אנכית.
- באמצעות ספק כוח סטנדרטי ג'ל אלקטרופורזה, להחיל שדה חשמלי של 250 V על פני בקלטת ג'ל-seq במשך 30 דקות להפריד את gDNA כלאיים cDNA/RNA. לאחר שנפרדתם, להסיר את הקלטת ג'ל-seq מהתא ג'ל אלקטרופורזה ופתח את שני החצאים של קלטת מסען את הקצוות עם כלי מגרד.
- באמצעות אזמל, לחצי את הג'ל מתחת לשכבת בצפיפות גבוהה. למחוק את החצי המכילה מילוי ג'ל להרים אותו. עם ידך העטויה בכפפה. מקלפים בעדינות את הג'ל הנותרים ממני הקלטת על ידי גירוד זה עם שפכטל צבע או כלי דומה אחר. במקום סעיף זה של הג'ל לתוך תבשיל המכיל ~ 30 מ"ל של 0.5 x TBE עם 3 µL של ג'ל כתם.
- מכסה המכולה כדי למזער את photobleaching ומשרים את הג'ל תוך בעדינות טלטול המיכל למשך 5 דקות. להניח את הג'ל על ניילון נצמד ולקחת תמונת UV באמצעות מערכת תיעוד ג'ל (לקבלת פירוט נוסף ראה הפניה למעורר 13). חשיפה השני 30 בדרך כלל מייצרת תמונות ברורות. ודא שאירעה ההפרדה.
- להעביר את הג'ל transilluminator UV כדי להקל על ויזואליזציה של חומצות גרעין. משקפיים מתאימים UV, לאשר את תוצאות מתוך מערכת תיעוד ג'ל. GDNA צריך להיות ממוקם בתחילתה של הג'ל בצפיפות נמוכה, את cDNA-הממשק של האזורים בצפיפות נמוכה, בצפיפות גבוהה.
- השתמש אזמל, לגזור את האזורים של הג'ל המכילות את gDNA ואת cDNA. דוגמאות יימצאו בצורה הטובה ביותר על ידי חיתוך של 4 מ מ על ידי 10 מ מ מלבני של ג'ל; עם זאת, הצורה הגיאומטרית המדויק יהיה תלוי ג'ל אלקטרופורזה המערכת בשימוש. זכור גם לחתוך השביל נטען עם הפקד שלילי.
- מקם כל מקטע נכרת של הג'ל לתוך צינור רצועת באמצעות זוג הקהה פינצטה. להיזהר לא להפעיל כוח רב מדי או הג'ל יפצל לחלקים מרובים. צריך זה יקרה, פשוט לאסוף כל פיסת ולהוסיף אותו ברכבת התחתית.
- לטחון את הג'ל בצינור כל משתמש קצה פיפטה (200 µL פיפטה טיפים עבודה טוב) על-ידי הזזת את הטיפ פיפטה בצורה מעגלית, בתחתית הצינורית. להוסיף מים חינם נוקלאז (µL 40 לתוך הדגימות gDNA ו- µL 80 לתוך הדגימות cDNA) כל שפופרת לפני הסרת קצה פיפטה נהגו לטחון את הג'ל כדי למזער את אובדן מדגם.
- במקום רצועת צינורות למסיבה מערבולת בתוך 37 ° C ואז ללחוץ על 8-12 שעות. דבר זה מאפשר חומצות גרעין לנטרל את הג'ל, טבעי עוצר בנקודה עבור פרוטוקול יום זה ריבוי.
- Pipette את הדגימות לתוך צלחת מסנן רשת 8 מיקרומטר ולסובב את הצלחת ב 2600 x g 5 דקות לאמץ את השברים ג'ל. הרם את הצלחת מסנן רשת מן הלוח דיור, pipette את הדגימות ג'ל ללא מים לתוך צינור חדש µL 200 רצועת.
- להוסיף 1 µL של פרוטאז (AU 0.9/mL) כל מדגם המכיל gDNA, לערבב היטב על ידי pipetting למעלה ולמטה, דגירה ב 50 מעלות צלזיוס למשך 15 דקות ולאחריו של איון חום ב- 70 מעלות צלזיוס למשך 15 דקות. שלב זה הוא קריטי עבור depleting נוקליאוזומים והופך את gDNA נגיש עבור שלבי התגובה עוקבות.
- שימוש של מחט בקוטר 18, תחורר הפקקים של כל צינורות מדגם. מקם את הדגימות vacufuge כדי לצמצם את נפח נוזלי. דוגמאות gDNA צריך להצטמצם עד 5 µL cDNA דגימות ירד ל 10 µL.
- בהתאם vacufuge מספר הדגימות, הזמן הכולל אידוי משתנה בין 30 ל 60 דקות. אם אמצעי האחסון מדגם יורד מתחת האחסון המטרה, פשוט להוסיף מים חינם נוקלאז כדי להגדיל את נפח דגימה.
5. gDNA ספריית הכנה
- שימוש fluorometer או טכנולוגיה דומה, לכמת את ריכוז הדנ א כל מדגם gDNA משלב 4, כמו גם את הפקד חיובי משלב 3. עבור פרוטוקול מפורט עיין במדריך הפניה fluorometer. 14
- לדלל את הדוגמאות כדי 0.2 ng/µL של ה-DNA. בהתאם לסוג התא ההתחלתי איכות התוצאות הנדרשות, ריכוז נמוך יותר יכול עדיין לייצר ספריות בת קיימא. כמה ניסויים יידרש; עם זאת, המחברים היו הצלחה עם ספריות המתחילים 0.1 ng/µL.
- להשלים את הכנת ספריה gDNA לפי פרוטוקול אחסון ספריית התגובה חצי הכנה בשלב 7.
6. הכנה ספריית cDNA
- החל עם 10 דוגמאות cDNA µL, בקרה חיובית משלב 4, להוסיף µL 12.5 של תערובת X qPCR 2, 0.5 µL cDNA PCR פריימר 2 µL נוקלאז ללא מים.
- לבצע PCR thermocycler בזמן אמת באמצעות פרוטוקול הבאים: חם-start ב 95 מעלות צלזיוס למשך 3 דקות, ואחריו 20-30 מחזורים של 98 ° C עבור 10 s, 65 ° C 30 s ו-72 מעלות עבור אמצעי האחסון הכולל דוגמה מינימלית 3 הוא 25 µL. לנטר. את עקומות תגובת ולהפסיק ההגברה לפני ה e תגובות יוצאים השלב מעריכית (אות ליניארי עלייה לעומת מחזור מספר) כדי להימנע PCR חפצים עקב overamplification. למידע נוסף על הימנעות overamplification, עיין בסעיף הדיון של הנייר הזה, כמו גם הפניה למעורר 15.
- לאחר הגברה, לנקות את המוצר באמצעות מעבדתי הפיך הנייח (אלוהים אדירים) חרוזים בעקבות הפרוטוקול בשלב 8. עם סיום, המשך לשלב הבא.
- שימוש fluorometer או טכנולוגיה דומה, לכמת את ריכוז הדנ א כל מדגם cDNA, כמו גם את הפקד חיוביים משלב 4. לדלל את הדגימות במידת הצורך להכיל כ 0.2 ng/µL של ה-DNA. ריכוז נמוך במקצת עדיין לייצר ספריות בת קיימא. כמה ניסויים יידרש, אולם המחברים הצלחה עם ספריות המתחילים 0.1 ng/µL.
- שלב אופציונלי: לבצע הפרדה סטנדרטית לזיהוי ג'ל אלקטרופורזה על µL 1-2 מכל סוג המוצר qPCR כדי לאמת את תגובת הדור ספריית עבד. תמונת מדגם של תוצאה מוצלחת מוצג באיור4. עבור פרוטוקול מפורט על אופן התנהלות לזיהוי בג'ל, ראה הפניה למעורר 16.
- להשלים את הכנת ספריית cDNA לפי ספריית הכנת ערכת חצי נפח התגובה פרוטוקול לשלב 7.
7. ספריית הכנה עם חצי נפח תגובות
- הכנה הספרייה כדלקמן פרוטוקול ערכת הכנה הספרייה באמצעות חצי נפח תגובות. 17 ריאגנטים כל המוזכרים בסעיף זה של הפרוטוקול הם משלב הכנת ספריה קיט (ראה טבלה של חומרים). להתחיל UV לחיטוי מספר מספיק של רצועת צינורות עבור מספר דוגמאות יעובדו.
- לבצע את התגובה transposase על-ידי הוספת 5 µL transposase מאגר כל שפופרת רצועת כדי לשמש וזמינותו. לאחר מכן להוסיף 2.5 µL קלט DNA ב 0.2 ng/µL (ng 0.5 סה כ) ואחריו 2.5 µL transposase. מערבבים על-ידי pipetting למעלה ולמטה 5 פעמים.
- דגירה ב 55 מעלות צלזיוס למשך 5 דקות, ואז לקיים ב- 10 מעלות צלזיוס. לאחר המדגם מגיע ל 10 ° C, הסר אותה ממנה thermocycler ולהוסיף מיד 2.5 µL transposase עצירה מאגר כל דגימה. שומרים את הדגימות בטמפרטורת החדר למשך 5 דקות.
- הכינו את תגובת ה-PCR כדי להגביר את הדגימה transpose מטופלים על-ידי הוספת 7.5 µL ספריית ההכנה PCR מיקס, 2.5 µL של פריימר אינדקס 1 ו- 2.5 µL של פריימר אינדקס 2. אלה תחל קניינית, מסופקים על-ידי היצרן של ערכת ההכנה של הספרייה. מערבבים היטב על ידי pipetting למעלה ולמטה 5 פעמים.
הערה: ודא כי שילובים ייחודיים פריימר משמשים עבור כל דגימה. ישנם 12 שונה אינדקס 1 תחל תחל שונים אינדקס 2 8, בכך ניתן לתייג באופן ייחודי עד 96 מדגמים שונים. לבחור שילוב ייחודי של צבעי יסוד כל דגימה. - לבצע PCR באמצעות התוכנית הבאה thermocycler. אמצעי האחסון מדגם נמצא 25 µL.
72 מעלות צלזיוס במשך 3 דקות
95 מעלות צלזיוס למשך 30 שניות
12 מחזורי:
95 מעלות צלזיוס למשך 10 שניות
72 מעלות צלזיוס למשך 30 שניות
55 מעלות צלזיוס למשך 30 שניות
72 מעלות צלזיוס למשך 5 דקות
להחזיק ב 10 ° C - לנקות את ספריות מוכן באמצעות חרוזים אלוהים אדירים בעקבות הפרוטוקול בשלב 8. הספריות לאמת באמצעות אלקטרופורזה בג'ל או של assay דומה. עיין במדריך ערכת הכנה הספרייה לקבלת פרטים לגבי אופן לאמת את ספריות. 17
8. מעבדתי הנייח הפיך חרוז ספריית ניקוי
- מעבדתי מחרוזת הנייח הפיך (אלוהים אדירים) צריך להיות מאוחסן ב- 1.5 mL aliquots, צריך להיות הביא לטמפרטורת החדר לפני כל שימוש. מומלץ גם להכין טרי 80% אתנול ניסוי. הפעולות הבאות מבוססות פרוטוקול חרוז אלוהים אדירים במדריך קיט של WTA. 18
- להוסיף 1 µl של המאגר של WTA ערכת פירוק כל מוצר ה-PCR. מערבולת החרוזים אלוהים אדירים עד אחיד מעורב, לאחר מכן להוסיף 50 µl של אלוהים אדירים חרוזים כל דגימה. לערבב על-ידי pipetting המדגם לאורך 10 פעמים, ואז דגירה בדגימות בטמפרטורת החדר במשך 8 דקות.
- בקצרה לסובב את הדוגמאות כדי לאסוף את הנוזל מן הצד של הצינורות. מניחים את הדוגמאות על מכשיר הפרדה מגנטית ~ 5 דקות עד שהנוזל מופיע לגמרי נקי.
- בעוד הדגימות הן על המכשיר הפרדה מגנטית, לאט פיפטה ליד כביש תגובת שיקוע של ביטול - להיזהר שלא להפריע. את הטבעת של חרוזים על המרקע. לאחר מכן להוסיף 200 µL של 80% אתנול כל מדגם מבלי להפריע את החרוזים. המתן 30 שניות ולאחר מכן בקפידה פיפטה הנחה תגובת שיקוע. חזור על שלב זה (שטיפת אתנול) ברגע.
- לאפשר את הדגימות לייבוש על 30 s - 1 דקות. לא יתר לייבש את הדגימות כמו רסיסים גדולים הופכים מאוגד לצמיתות את החרוזים.
הערה: זמן קצר יבש מומלץ כדי להבטיח שכל עקבות של אתנול יוסרו. כמויות קטנות של אתנול תישאר מאחורי הם יכול לעכב מעט תגובות במורד הזרם. - להסיר את הדגימות מהמכשיר הפרדה מגנטית ולהוסיף 15 µL של מים כל דגימה כדי elute את ה-DNA של החרוזים. פיפטה למעלה ולמטה, להבטיח שהחרוזים יוסרו מן הצדדים של הצינורות. דגירה בטמפרטורת החדר למשך 2 דקות. בקצרה לסובב את הדגימות ולאחר מכן למקם אותם על המכשיר הפרדה מגנטית למשך דקה ~ 1 להופעת הפתרון ברור.
- בעוד הדגימות הן על המכשיר הפרדה מגנטית, לאט פיפטה למעלה תגובת שיקוע, להעביר אותו צינורות נקיים. להיזהר שלא להפריע. את הטבעת של חרוזים על המרקע. למחוק את הצינורות עם חרוזים.
תוצאות
ההפרדה הפיזית של מכוניות היברידיות gDNA הרנ"א/cDNA במכשיר ג'ל-seq להיות visualized באמצעות ג'ל פלורסנט הדמיה; תוצאה נציג מוצג באיור3. פאנל A מראה המכשיר ג'ל-seq והתחייה; הצבעים נוספה כדי להבחין בין האזורים השונים ג'ל. לוח ב' מופיע קרוב של ארבעה הפרדות שונות נעשה שימוש עבור אימות. במסלול השלישי, פקד שלילי, מייצג את הרקע ומראה שאין שום autoflourescence של הג'ל על הממשקים. אנו נטען המסלולים הראשון והשני עם ה-DNA סולמות. מסלולים אלה מראים רק להקה כהה. הממשק בין density נמוך וגבוה בקרום, חושף כי קטעים קטנים יכול לעבור דרך הג'ל בצפיפות נמוכה. הנתיב הרביעי מציג את אופן הפעולה של דגימה ביולוגית של עניין: 500 PC3 תאים. אנו נטען ליין 4 כפי שמתואר בשלב 3 של הפרוטוקול. התמונה מציגה הפרדת הדנ א cDNA/RNA כלאיים. להקה כהה בחלק העליון של קרום בצפיפות נמוכה נמצא megabase-סולם הדנ א כלאיים cDNA/RNA נערמים על הממשק של האזורים נמוך, צפיפות גבוהה. שלא כמו הסמטאות עמוסות הסולם, ישנם גם מספר להקות מתנה בתוך האזור בצפיפות גבוהה של הג'ל. אלה שברים, קטן מ- 100 bp, את המטרה מוצרים המופקים פריימר oligonucleotides במהלך שעתוק במהופך. החלונית ' C מציגה תמונת הנציגה של המכשיר ג'ל-seq כולו מניסוי יירוט מוצלח. מסלולים הנקרא RNA/cDNA עובדו עם פרוטוקול ג'ל-seq, בעוד מסלולים הנקרא RNA מציגים להפרדת רק gDNA ו- RNA. החלונית ' D מציגה ניסוי כושל עם רצועות שחורות בחלק העליון של כל ליין של קרום בצפיפות נמוכה. זה נגרם על-ידי מאגר אלקטרופורזה מזוהם עם DNA מפוצלים. בשלב זה, החוקרים צריכים לחפש את הפקדים נקי שלילי וגם שתי רצועות שחורות ברורים המעידים על המצאות של gDNA מופרדים ו RNA/cDNA כלאיים.

איור 3 . תוצאות פרידה ג'ל-Seq. המכשיר ג'ל-seq (א) ו תמונה פלורסנט מציג הפרדת כלאיים DNA ו RNA/cDNA (B). הצבעים נוספה להבחין בקלות רבה יותר בין האזורים השונים של צפיפות בתוך הג'ל. (C ו- D) תמונות פלורסנט נציג של המכשיר ג'ל-seq כולו מוצלח (ג), (ד) ניסוי כושל. NTC = שליטה תבנית. חלקים של איור זה שוחזר הפניה למעורר 7, ברשות החברה המלכותית לכימיה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
ברגע כלאיים DNA ו- RNA/cDNA הופרדו והשלים השארית של פרוטוקול seq ג'ל, זה אפשרי ליצור רצף ספריות. כדי לאמת את ספריות מוכן, נרוץ ניסוי אלקטרופורזה ג'ל רגיל (איור 4) או של bioanalyzer. התוצאות באיור 4 מציגות ספריות שנוצר מתוך 500 PC3 תאים ותאים הלה 750. האיור מציג את הפצות פרגמנט עבור ספריות בהתאמה המופקים ג'ל-seq (הנקראת "ג'ל") לעומת דגימות תחרות הנוצרת באמצעות פרוטוקולים סטנדרטיים (הנקראת 'צינור'). גודל המקטע עבור ג'ל-seq מופיעים בין 200 ל-800 basepairs כצפוי בעת הכנת ספריות באמצעות ערכת הכנה הספרייה הגנום כולו. אם השברים הספרייה לא מופיעות בטווח בגודל הנכון בשלב זה, הכנת ספריה נכשלה.
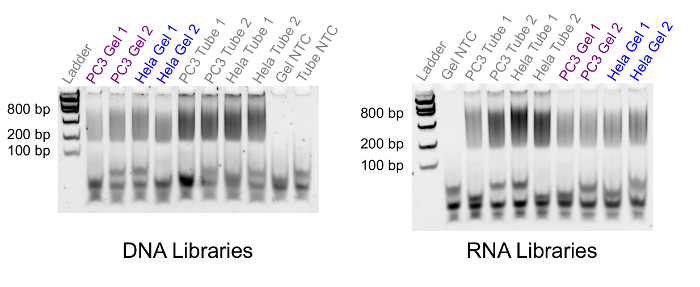
איור 4 . ספריית פרגמנט גודל השוואה. תמונת אלקטרופורזה ג'ל פלורסנט השוואת ספריית הפצה גודל בין ג'ל-seq (ג'ל) פקדים רגילים (צינור). הנתיב השמאלי מכיל סולם הדנ א מסה נמוכה עם פרגמנט בגדלים 100, 200, 400, 800, 1200 ו- 2000 basepairs. גודל המקטע עבור כל ספריות ליפול בין 200 ל-800 basepairs, כצפוי עבור ספריות מוכן עם ערכה זו. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
התוקף החוקי האולטמטיבי של פרוטוקול ג'ל-seq מבוסס על הניתוח של רצף תוצאות. בחרנו PC3 תאים לניסויים שלנו אימות ', כמו תאים אלה יש ביטוי הומוגנית פרופילים המאפשרים דגימות להיות לפצל ומעובדים באמצעות ג'ל-seq והן שיטות מסורתיות; ראה איור 5. השוואה בין ה-DNA גנומי של תאים PC3 מוצג 5 דמויותA ו- 5B. איור 5 א מציגה השוואה של הגנום כולו העתק פרופילי וריאציה (CNV) מספר המופקים PC3 באמצעות ג'ל-seq או לתגובה הכנה הספרייה הגנום כולו (פקד tube). כל נקודה היא ספירה סל מנורמל רשע; פחי מוגדרים מנתונים הגנום התייחסות כזאת לכל סל יש ספירת הצפוי שווה תא דיפלואידי בריא, כלומר, קו שטוח, המייצג עותקים שווה לכל אזור של כל כרומוזומים (למעט X ו- Y) כרומוזומים. PC3 מכילה עותקים מרובים של האזורים זהה אשר יופיע כמו קוצים מעל רקע עותק מספר שתיים. ג'ל-seq התשואות פרופיל CNV איכותית דומה כמו צינור סטנדרטי התגובה. הסכם בין החלקות שני יכול להיות מוערך באופן כמותי על ידי רגרסיה ליניארית, כפי שמוצג בלוח ב' מתאם פירסון של R = 0.90 מציין כי גנומית נתונים שנאספו אחת מהשיטות מקבילה לפעולתה.

איור 5 . ספריית אימות. ג'ל-seq אימות עבור גנומית (A ו- B) ואת transcriptomic (C, D ו- E) הנתונים שהופקו מתאי PC3 ו הלה. פאנל A מציגה השוואה של הגנום כולו CNV פרופילים המופקים PC3 באמצעות ג'ל-seq (ג'ל-seq) או תגובה רגילה (צינור). MAPD = חציון ההבדל Pairwise מוחלטת. החלונית B היא רגרסיה ליניארית בין בשתי הדגימות בלוח A, עם R = 0.90 המציין הנתונים גנומית מקבילים. צירי הצג שסל מנורמל log2 נחשב. לוחות C ו- D להשוות נתונים transcriptomic מתאי PC3, עם כל נקודה מראה ספירה תעתיקים לכל kilobase לכל מיליון (TPM). הצירים מציגים השוואה בין שני מדגמים כמניין התעתיק log2 מנורמל. לוח ג מציגה השוואה של משכפל טכנית שנוצר באמצעות ג'ל-seq ומראה D לוח שהשוואה בין ג'ל-seq RNA-תת סעיף. לוח אלקטרוני מראה כי ג'ל-seq ניתן לפתור סוג התא מבוסס על הביטוי RNA באמצעות ניתוח גורמים ראשיים. ציר x מראה כי החשבונות גורמים ראשיים הראשון 91.6% של וריאציה בין הדגימות בזמן ציר y מראה כי החשבונות גורמים ראשיים השני רק 6.5% של השונות. חלקים של איור זה שוחזר הפניה למעורר 7, ברשות החברה המלכותית לכימיה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
באופן דומה, אנחנו לעומת transcriptomic נתונים מפרוטוקול שלנו ג'ל-seq הפרוטוקול הסטנדרטי של חכמים-Seq בשפופרת. איור 5 מראה את המתאם בין שני ג'ל-seq טכני משכפל (איור 5C) ובין ג'ל-seq את השיטה הרגילה (איור 5D). כל נקודה היא ספירה של תעתיקים לכל kilobase לכל מיליון (TPM) עבור כל ג'ין שזוהו ב- TPM > 5 ב שני נתונים (datasets). Regressions לינארי מוצגים כקווים אדומים, מקדם המתאם פירסון מוצג בפינה השמאלית העליונה. משכפל טכני של ג'ל-seq מסכים (R ∼ 0.8), אבל פחות טוב בהתאמה השיטה הרגילה (R < 0.7). הדבר מצביע על ג'ל-seq מציגה דעה קדומה של ג'ין ספירות. למרבה המזל, הנטיה הזו היא שיטתית ו, כפי שניתן לראות על ידי ניתוח גורמים ראשיים איור 5E, בעל משמעות מסקנות עדיין ניתן להסיק בין דגימות ביולוגיות שונות.
Discussion
ישנם מספר שלבים קריטיים המשויך הזיוף התקן ג'ל-seq, כמו גם את הפרוטוקול עצמו. במהלך פבריקציה נוספת, אנו ממליצים החל עוביים השכבה שנקבעו עבור אזורים שונים של הג'ל. בילינו זמן משמעותי בדיקת אפשרויות ייצור שונה ומייצרת הפרוטוקול המתואר כאן ההתקנים הטוב ביותר עבור הקלטות המפורטים בטבלה של חומרים, ריאגנטים. אם חוקרים משתמשים מערכת חלופית קלטת, הם עשויים למצוא אותו צורך לעדן את אמצעי האחסון שמשמש בעת יצירת המכשירים. האתגר העיקרי ב פבריקציה נוספת היא שאם האזור ג'ל בצפיפות גבוהה גדול מדי זה יכול delaminate מקצות הקלטת וליצור כיסי אוויר על הפנים של קלטת שישבש אלקטרופורזה. ליהקנו מספר קלטות עם מספר כרכים שכבת שונה, חוקרים צריך להיות מסוגל לקבוע במהירות את התצורה המיטבית עבור חומרה מסוימת שלהם.
פרוטוקול ג'ל-seq יש גם מספר שלבים קריטיים הניתנים לאימות לפני השלמת הפרוטוקול. נקודת כשל פוטנציאלי אחד היא ההפרדה gDNA ו RNA/cDNA כלאיים. זה הניתנים לאימות על ידי הדמיה המכשיר ג'ל-seq לאחר ההפרדה (ראה איור 3ב). קבוצה אחת של ניסויים, מצאנו כי האספקה המעבדה של מאגר היה להיות מזוהמים עם ה-DNA, היה גורם משמעותי autoflourescence במכשיר שלנו (ראו איור 3ד'). זה היה קשה לקבוע אם ההפרדה שהתרחש. הדמיה פלורסנט עזר לנו לזהות ולפתור את הבעיה לפני השימוש בכל ריאגנטים יקר כדי ליצור רצף ספריות.
נקודה קריטית נוספת היא שלב 6.2, הגברה qPCR של cDNA לאחר ההפרדה. החוקרים צריכים לשים לב זהירה אל overamplify בשלב זה כפי שהוא יפחית את איכות הנתונים RNA-seq. שיקול זה אינו ייחודי ג'ל-seq, אך הוא היבט משותף של קלט נמוך-RNA-seq ספריית הכנה. PCR הגברה במהלך רצף ספריית הכנה הוא לעתים קרובות הכרחי, אבל זה יכול להציג רצף שגיאות של הטיות. המספר הנדרש של מחזורי עבור ה-PCR תלוי כמות הדגימה. בדרך כלל מומלץ להגביל את מספר מחזור PCR המינימום הנדרש כדי תשואה מספקת אשכולות כאשר הספריות הן וסודרו. באופן תיאורטי, ניתן למטב פרוטוקול כדי לקבוע את המספר המדויק מחזור המניבה מספר העותק מספיק ללא החדרת חפצים מוגזמת. בפועל, עם זאת, חוסר עקביות באיכות הדגימה, טעינה, או טיפול מוקדם בפרוטוקול יכול באופן דרמטי להשפיע על חלוקת מולקולרית התבניות הזמינות עבור הספרייה במכינה PCR, אשר בתורו משפיע על המספר האופטימלי של מחזור ה-PCR. הפתרון הכללי ביותר שמצאנו כדי לעקוב אחר ההתקדמות של תגובות הגברה באמצעות של הפלורסנט, להפעיל התגובות על thermocycler בזמן אמת PCR, ולהפסיק את התגובות ב המעריך (לינארית לעומת מחזור מספר) שלב. מניסיוננו, ניטור בזמן אמת היא רלבנטית במיוחד בעת פיתוח, הסתגלות או אימוץ נוהל חדש.
השלב הקריטי האחרון מייצר גנומית וספריות transcriptomic. המפתח שלב זה הוא להגדיר את הריכוז מדגם ההתחלה עבור התגובה הכנה של ספריית ה-DNA קרוב 0.2 ng/µL (ng 0.5 סה כ) ככל האפשר. זה פשוט למדי עבור cDNA qPCR הגביר בדרך כלל יש עודף של cDNA, אבל זה יכול להיות יותר מאתגר על הדגימות gDNA. . מצאנו זהיר תשומת לב לשלב vacufuge נדרשה בזמן הדגימות היו להיות מרוכז. כצפוי, בניסויים עם 1000 תאים, אפשר לשלב vacufuge לעצור הרבה מוקדם יותר מאשר ניסויים עם 100 תאים. מספר דוגמאות vacufuge השפיעו גם את קצב אידוי בניסויים שלנו. מצאנו את זה באמצעות flourometer כדי לאמת את הדנ א תוכן באמצע השלב ריכוז יכול להיות מועיל בעת ביצוע הפרוטוקול באמצעות דגימות לא מוכר. למרבה המזל, אם חוקרים מעל להתרכז לדוגמה, נוקלאז חינם מים יכול להתווסף כדי לדלל את הדגימה. תיאורטית, זה אפשרי להשתמש את vacufuge כדי לייבש את ה-DNA, אז resuspend זה באמצעי האחסון הרצוי; עם זאת, אנו ממליצים להימנע אידוי מוחלט.
אנו רואים שלוש שיטות ליצירת ספריות DNA ו- RNA בו זמנית, ג'ל-seq7, G & T-seq8ו ד ר-seq9, כמו ללא תשלום. ג'ל-seq הינו אידיאלי עבור דגימות בטווח התאים 100-1000 והוא דורש נפתחים מטרות או ידע א-פריורי של הגנום. שתי השיטות אחרים מתאימים יותר עבור יישומים תא בודד. אחת המטרות שלנו בפיתוח ג'ל-seq הייתה ליצור פרוטוקול זה יכול להיות מיושם בקלות על ידי חוקרים אחרים. לכן החלטנו לפברק התקנים בתוך מארז סטנדרטי של קלטת לזיהוי ג'ל. בעוד אנחנו המשמשת להגדרת ממברנות שונות שלנו הוא רומן, רוב מעבדות גנטיקה יש כבר את כל הציוד הנדרש כדי לפברק את המכשיר ג'ל-seq. יתר על כן, העלות של המכשיר הוא טריוויאלי - רק 5.25 $ עבור התקן זה יכול לעבד 12 דגימות. עם זאת, כמו עם כל ספריית הכנת פרוטוקול שימוש מסחרי ריאגנטים, העלות הכוללת ליצירת ספריות נותרת גבוהה. עלות ריאגנט שלנו עבור דגימה היה 50 דולר. עבור כל-תעתיק הגברה ו 28 דולר להכנה ספריית DNA ו- RNA. למרבה המזל, ההתקן ג'ל-seq עצמו הוא פרוטוקול אגנוסטיקן. לדוגמה, במהלך הפיתוח בהצלחה בדקנו את ההתקן באמצעות תאים של תרבות, של בוגרים RNA ספריית הגברה פרוטוקול19, אמנם מצאנו שזה לא מתאים דגימות רקמה של עכברים. מסתכל אל העתיד, כפי חלופות זול יותר להכנה ספריית מפותחים, פרוטוקול שלנו ניתן להתאים לעבודה עם טכניקות חדשות אלה. אנו מאמינים החוקרים תגלה את זה פשוט ליישם ג'ל-seq במעבדות שלהם. אנו מקווים שזה יקל על אימוצה המהיר של הטכנולוגיה.
Disclosures
KZ הינו מייסד ומנכ ל מדעי יועץ של Singlera גנומיקה inc.
Acknowledgements
מימון עבור עבודה זו היה בתנאי על-ידי באוניברסיטת סן דייגו, הלאומית למדע קרן בוגר מחקר תכנית העמיתים, NIH להעניק R01-HG007836, ועל ידי קוריאנית משרד המדע, ICT ותכנון העתיד.
גירסאות קודמות של כמה דמויות פורסמו לראשונה ב- "התקליט הזה, ג ד. et al. ג'ל-seq: הגנום כולו, רצף transcriptome על ידי סימולטני קלט נמוך-DNA ו- RNA ספריית הכנה באמצעות מחסומים הידרוג חדיר למחצה. מעבדה על שבב 17, מוריה ברטל-2630, doi:10.1039 / c7lc00430c (2017). " מעבדה על שבב וההתנהגותיים שימוש חוזר של דמויות בפרסום זה.
Materials
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate - Corning HTS Transwell 96 well permeable supports - 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18(2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31(2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908(2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), at http://science.sciencemag.org/content/352/6293/1586 (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923(2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies. , Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017).
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- A Guide to Polyacrylamide Gel Electrophoresis and Detection. Bulletin 6040 Rev B. Bio Rad. , Available from: http://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6040.pdf (2017).
- Illumina. Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved