Method Article
Gel-Seq: Eine Methode zur gleichzeitigen Sequenzierung Bibliothek Vorbereitung von DNA und RNA mit Hydrogel Matrizen
In diesem Artikel
Zusammenfassung
Gel-Seq ermöglicht Forschern gleichzeitig Bibliotheken für beide DNA und RNA-Seq bei vernachlässigbaren Kosten ab 100-1000 Zellen mit einem einfachen Hydrogel-Gerät vorzubereiten. Dieser Beitrag stellt einen detaillierten Ansatz für die Herstellung des Geräts sowie das biologische Protokoll zum gekoppelte Bibliotheken zu generieren.
Zusammenfassung
Die Fähigkeit zu verstärken und aus kleinen Start Proben entweder DNA oder RNA-Sequenz wurde erst in den letzten fünf Jahren erreicht. Leider die Standardprotokolle für Erzeugung von genomischen oder transkriptomischen Bibliotheken sind nicht kompatibel und Forschern müssen entscheiden, ob Sie für eine besondere Probe DNA oder RNA-Sequenz. Gel-Seq löst dieses Problem durch Forscher um gleichzeitig Bibliotheken für DNA und RNA beginnend mit 100-1000 Zellen mit einem einfachen Hydrogel Gerät vorzubereiten. Dieser Beitrag stellt einen detaillierten Ansatz für die Herstellung des Geräts sowie das biologische Protokoll zum gekoppelte Bibliotheken zu generieren. Wir konzipiert Gel-Seq so, dass es leicht von anderen Forschern umgesetzt werden könnten; viele Genetik Labore verfügen bereits über die notwendige Ausrüstung, die Gel-Seq-Gerät-Herstellung zu reproduzieren. Unser Protokoll beschäftigt häufig verwendete Kits für beide ganz-Transkript Verstärkung (WTA) und Bibliothek Vorbereitung, die auch bereits Forscher vertraut sein dürften versiert in der Erzeugung von genomischen und transkriptomischen Bibliotheken. Unser Ansatz ermöglicht Forschern, bringen die Kraft der DNA und RNA Sequenzierung auf einer einzigen Probe ohne Spaltung mit unwesentlichen zusätzlichen Kosten zu tragen.
Einleitung
Nächste Generation sequencing (NGS) hatte eine profunde Auswirkung auf dem Weg, die Genetik geforscht wird. Wo Forscher einmal auf die Sequenzierung des Genoms eine ganze Spezies konzentriert, ist es nun möglich, das Genom einer einzigen Tumors oder sogar eine einzelne Zelle in einem Experiment sequentiell. 1 NGS hat auch kostengünstig zu den RNA-Transkripte gefunden innerhalb einer Zelle, eine Sammlung von Daten, bekannt als das Transkriptom Sequenzierung gemacht. Die Fähigkeit zu verstärken und aus kleinen Start Proben entweder DNA oder RNA-Sequenz wurde erst in den letzten fünf Jahren erreicht. 2 , 3 , 4 leider Standardprotokolle sind unvereinbar und Forschern müssen entscheiden, ob Sie für eine gegebene Probe DNA oder RNA-Sequenz. Wenn eine Start Stichprobe groß genug ist, kann es in zwei Hälften aufgeteilt werden. Bei kleineren Maßstäben Materialverlust durch Spaltung Proben kann Bibliothek Qualität beeinflussen und Bündelung von Proben kann durchschnittlich interessante Variationen zwischen den Zellen. 5 im übrigen interessieren sich Forscher zunehmend bei der Prüfung von Proben, die nicht können, z. B. Einzelzellen oder kleine heterogene Tumor Biopsien gespalten werden. 6
Um dieses Problem zu beheben, drei Protokolle vor kurzem wurden entwickelt, um aus der gleichen Ausgangspunkt Probe DNA und RNA Sequenz: Gel-Seq7, G & T-Seq8und DR-Seq-9. Dieser Artikel stellt ein detailliertes Protokoll für Gel-Seq, die verwendet werden, um gleichzeitig DNA- und RNA-Bibliotheken von nur 100 Zellen bei vernachlässigbaren Kosten erzeugen kann. Der neue Aspekt der Gel-Seq ist die Fähigkeit, DNA und RNA basiert ausschließlich auf Größe mit low-cost Hydrogel Matrizen zu trennen. Die Kern-Innovation des Gel-Seq-Protokolls ist die physische Trennung der DNA von RNA. Diese Trennung erfolgt elektrophoretisch mit einer Kombination aus Polyacrylamid-Membranen, die die Größenunterschiede zwischen diesen Molekülen nutzen. Um diese Größenunterschiede in Zusammenhang zu setzen, zu prüfen, wie DNA und RNA abgebildet werden: während DNA vorhanden auf der Micron-Skala ist und eingesehen werden traditionelle Mikroskopie, RNA existiert auf der Nanometerskala und komplexe Techniken wie Cryo-Elektron abgebildet werden muss Mikroskopie. 10
Der Ansatz zur Trennung von DNA und RNA in diesem Protokoll ist in Abbildung 1dargestellt. Die linke Tafel zeigt, dass DNA und RNA frei schwebend in Lösung in der Nähe von einer Membran. Wenn ein elektrisches Feld angelegt wird, wie auf der rechten Seite gezeigt, erleben DNA und RNA eine elektrophoretische Kraft, die Migration durch die Membran induziert. Durch die Membran Eigenschaften optimieren, schufen wir eine semipermeable Membran, die DNA von RNA trennt. Die DNA-Moleküle sind gegen die Membran gedrückt, aber verfangen am Rande wegen ihrer großen Größe. Auf der anderen Seite können kleine RNA-Moleküle, konfigurieren und schlängeln sich durch die Membran. Dieser Prozeß, bekannt als Reptation, ähnelt die Art, wie, die eine Schlange durch Rasen bewegt. Schließlich sind diese RNA-Moleküle durch eine zweite, mit hoher Dichte Membran, die zu schwierig für noch kleinere Polymere ist gestoppt (> 200 Basenpaaren), durch Winden. Einmal physisch getrennt, kann um Informationen über das Genom und Transkriptom zu generieren DNA und RNA erholt und verarbeitet werden. Während wir DNA und RNA trennen können, haben wir gefunden, dass bessere Ergebnisse erzielt werden, wenn die RNA transkribiert, cDNA vor der Trennung umgekehrte ist. Die DNA/RNA-Hybriden sind stabiler als RNA allein und können noch durch die Dichte Membran passieren.

Abbildung 1 . Gel-Seq operative Prinzip. Das zugrunde liegende Prinzip verwendet, um räumliche Trennung von DNA und RNA. In einem angewandten elektrischen Feld kleine RNA-Moleküle Wandern durch die Dichte Membran aber große DNA-Moleküle sind an der Oberfläche gefangen. Diese Zahl wurde mit freundlicher Genehmigung von der Royal Society of Chemistry von ref 7 wiedergegeben. Bitte klicken Sie hier für eine größere Version dieser Figur.
Dieses Papier beschreibt ausführlich sowohl die Herstellung von Gel-Seq-Gerät und das biologische Protokoll generieren gepaart DNA- und RNA-Bibliotheken. Eine Übersicht der beiden ist in Abbildung 2dargestellt. Das Gerät ist durch Schichtung drei unterschiedliche Dichte Polyacrylamid Gele übereinander in einem Prozess ähnlich erstellen standard Stapeln Gele hergestellt. 11 das biologische Protokoll beginnt mit 100-1000 Zellen mit PBS-Puffer suspendiert. Die Zellen lysiert und die RNA in cDNA umgewandelt wird, bevor das Gerät verwendet wird, um die genomische DNA aus der DNA/RNA-Hybriden zu trennen. Nach der Trennung und Verwertung, genomische und transkriptomischen sind Bibliotheken zubereitet einen Prozess, der das gesamte Genom Standardbibliothek Vorbereitung Kit Protokoll verfolgt. Weitere Details über die Entwicklung und Validierung von Gel-Seq abgelesen werden im Labor auf einem Chip-Veröffentlichung "Gel-Seq: Vollständiggenom und Transkriptom Sequenzierung durch gleichzeitige Low-Eingang DNA- und RNA-Bibliothek Vorbereitung mit semi-permeable Hydrogel Barrieren ." 7

Abbildung 2 . Gel-Seq Protokoll. Eine Übersicht über die Schritte, die Gel-Seq-Gerät und das Protokoll zum generierten gepaarten DNA und RNA Bibliotheken zu fabrizieren. Teile davon wurden mit freundlicher Genehmigung von der Royal Society of Chemistry von Ref. 7 wiedergegeben. Bitte klicken Sie hier für eine größere Version dieser Figur.
Um DNA und RNA Bibliotheken aus einzelnen Zellen zu generieren, Forscher sollten erwägen, entweder G & T-Seq oder DR-f-G & T-FF, wie Gel-Seq, stützt sich auf eine räumliche Trennung von RNA aus genomischer DNA. Dieser Ansatz stützt sich auf Boten-RNA (mRNA) 3′ Polyadenylated Schweif als Pulldown-Ziel. Die mRNA wird auf eine magnetische Perle biotinylierte Oligo-dT Grundierung mit erfasst. Sobald die mRNA eingefangen hat die Perlen mit einem Magneten in Position gehalten werden und der Überstand mit der genomischen DNA entfernt und in einem anderen Gefäß übertragen werden kann. Nachdem diese körperliche Trennung abgeschlossen ist, können separate Bibliotheken aus der mRNA und DNA generiert werden. 8 dieser Ansatz funktioniert gut, wenn die RNA von Interesse Polyadenylated ist, kann nicht jedoch es verwendet werden, nicht Polyadenylated Abschriften, wie ribosomaler RNA, tRNA oder RNA von Prokaryoten zu studieren.
DR-Seq stützt sich auf eine Vorverstärkung Schritt wo DNA und cDNA abgeleitet von RNA in der gleichen Röhre verstärkt werden. Die Probe wird dann in zwei Teile gespalten und parallel, DNA und RNA-Seq Bibliotheken vorzubereiten verarbeitet. Zur Unterscheidung zwischen genomischer DNA und die cDNA abgeleitet von RNA, nimmt DR-Seq einen rechnerischen Ansatz. Sequenzen, in denen nur Exons vorhanden sind werden rechnerisch in der genomischen DNA-Daten, unterdrückt, wie diejenigen aus entweder DNA oder RNA entstanden sein könnte. 9 ein Vorteil dieses Ansatzes ist, dass die DNA und DNA/RNA nicht räumlich getrennt sein müssen wie in Gel-Seq und G & T-seq. Der Nachteil ist jedoch, dass DR-Seq a-priori Kenntnis des Genoms und Transkriptom (d.h. Exons und Introns erfordert), und möglicherweise nicht ideal für Anwendungen wie z. B. Sequenzierung von Kernen, in denen viele Transkripte noch nicht vollständig sind gespleißt und weiterhin enthalten Introns. 12
Der neuartige Aspekt des Gel-Seq ist die Fähigkeit, DNA und RNA in Hunderten von Zellen ausschließlich anhand der Größe trennen. Diese Methode erfordert kein priori Kenntnis des Genoms oder Transkriptom, ist robust gegen unvollständige Spleißen und beschränkt sich nicht auf Poly-Adenylated-Protokolle. Für Anwendungen, wo ein Forscher mit mindestens 100 Zellen beginnen kann, bietet Gel-Seq ein einfaches Verfahren mit billigen und allgemein zugänglichen Materialien.
Protokoll
1. Vorbereitung der chemische Lösung
Hinweis: Die folgenden Schritte sind für die Zubereitung von chemischer Lösungen, die in späteren Schritten erforderlich. Diese können in loser Schüttung und mehrere Monate gelagert werden.
- Zunächst bereiten Sie 50 mL gereinigtes, deionisiertes Wasser durch Sterilisation in einem 254 nm UV-Vernetzung-Ofen für 15 min (15 mJ/cm2 Gesamtexposition), keine kontaminierende DNA zu neutralisieren. Erhitzen Sie auf 37 ° C für den Einsatz in folgenden Schritten.
- 10 mL eines 40 % Gesamt (T) und 3,3 % Vernetzer (C) (29,1) machen Polyacrylamid-Vorläufer-Lösung. Wiegen Sie 3,867 g Acrylamid Monomeren und BIZ-Acrylamid Monomeren 0,133 g. Kombinieren und bringen Volumen bis zu 10 mL mit warmem gereinigtes Wasser und Wirbel bis aufgelöst. Lichtgeschützt bei Raumtemperatur lagern.
Hinweis: Vorgemischte 40 % T, 3,3 % C (29,1) Polyacrylamid-Lösungen im Handel erhältlich. - 10mL 50 % T, 5 % C Gel-Lösung zu machen. Wiegen Sie 4,750 g Acrylamid Monomeren und BIZ-Acrylamid Monomeren 0,250 g. Kombinieren und bringen Volumen bis zu 10 mL mit warmem gereinigtes Wasser und Wirbel bis aufgelöst. Lichtgeschützt bei Raumtemperatur lagern.
- 10 mL einer 50 % (w/V) Saccharose-Lösung zu machen. 5 g Saccharose, ein Messzylinder dazugeben Sie und warmes gereinigtes Wasser bis zu einem Gesamtvolumen von 10 mL. Vortex bis aufgelöst und bei Raumtemperatur lagern.
- Machen Sie 10 mL 10 % APS (w/V). Ein Messzylinder 1 g Ammonium Bleichen (APS) hinzufügen. Hinzufügen, kalt (~ 4° C) gereinigtes Wasser bis zu einem Gesamtvolumen von 10 mL und Vortex bis aufgelöst. Sofort in Aliquote von 200 µL einfrieren.
(2) Gel-Seq-Kassette-Fertigung
Hinweis: Gel-Seq wurde ursprünglich mit aufrechten Kassetten entwickelt (siehe Tabelle der Materialien für mehr Informationen); Dieses Protokoll kann jedoch angepasst, mit jedem standard-Gel-Elektrophorese-Kassette zu arbeiten.
- Bereiten Sie Gel-Vorstufen in drei separaten Kunststoffrohre durch Zugabe von Reagenzien, wie unten in Tabelle 1 dargestellt. Nicht hinzufügen entweder APS oder Tetramethylethylenediamine (TEMED) bis in den folgenden Schritten geleitet. Vortex Zutaten gründlich mischen.
| Füller Gelvorstufe | High-Density Gel Vorläufer | Geringe Dichte Gelvorstufe | |||
| 40 %T, 3.3%C Acrylamid Bisacrylamide Lösung | 1,6 mL | 50 %T, 5 %C Acrylamid Bisacrylamide Lösung | 2,4 mL | 40 %T, 3.3%C Acrylamid Bisacrylamide Lösung | 0,6 mL |
| Deionisiertes Wasser | 10,2 mL | Deionisiertes Wasser | 1,0 mL | Deionisiertes Wasser | 4,8 mL |
| Zuckerlösung (50 % w/V) | 2,6 mL | Zuckerlösung (50 % w/V) | 0,6 mL | ||
| 10 X Tris-Borat-EDTA | 1,6 mL | 10 X Tris-Borat-EDTA | 0,6 mL | ||
| Ammonium-Bleichen (10 % w/V) | 104.0 ΜL | Ammonium-Bleichen (10 % w/V) | 50,0 ΜL | Ammonium-Bleichen (10 % w/V) | 39,0 ΜL |
| TEMED | 6.0 ΜL | TEMED | 1,0 ΜL | TEMED | 2.2 ΜL |
| Gesamtvolumen | 16,1 mL | Gesamtvolumen | 4,1 mL | Gesamtvolumen | 6,0 mL |
Tabelle 1. Gel-Synthese Reagenzien. Polyacrylamid-Gel Vorläufer Reagenzien ausreichend für die Herstellung von 2 Kassetten.
- De-Gas Gel Monomer Lösungen durch eine Nadel durch die Kappe des Rohres und verbinden diese Nadel mit einem Haus Vakuumleitung. Tauchen diese Assembly in einem Ultraschallbad zu hoch und warten Sie, bis die Bläschen stoppen entstehende Flüssigkeit vor dem Umzug zum nächsten Schritt (~ 60 Sekunden / Rohr).
Hinweis: Die Qualität des Vakuums und Ultraschallbad ist nicht kritisch, solange Bläschen aus der Lösung gesehen werden können. - Hinzu kommen TEMED und APS der Füller Gel Vorläufer und Wirbel kurz. Fügen Sie 6 mL der Füller Gel Vorstufe sofort jede Gel-Kassette durch Pipettieren der Lösung in den oberen Teil der Kassette hinzu. Verwenden Sie eine 1 mL Mikropipette hinzufügen den Vorläufer in sechs Schritten zu verschütten zu vermeiden. Die Gesamtzeit für diesen Schritt sollte weniger als 3 Minuten betragen.
- Stellen Sie sicher, dass die Flüssigkeit Ebene durch die Positionierung der Kassetten aufrecht auf eine Tabelle. Füllen Sie den Rest der Kassette mit entionisiertem Wasser entgast. Pipette das Wasser wieder mit 1 mL Schritten langsam in die Mitte der Kassette zu mischen zu minimieren. Das Polymer für mindestens eine Stunde bis zu heilen, um über Nacht zu ermöglichen.
- Nach des Polymers Aushärten invertieren Sie die Gel-Kassetten über ein Waschbecken, das Wasser-Overlay zu entfernen. Eine Druckluftpistole lässt sich sanft die Schnittstelle trocknen durch Einblasen von Luft durch die obere Öffnung der Kassette aus einer Entfernung von 6 Zoll.
- Hinzu kommen TEMED und APS die High-Density Gel Vorläufer und Wirbel kurz. Sofort fügen Sie 320 µL des High-Density-Vorläufers der Kassette, erneut mit einer 1-mL-Pipette hinzu. Stellen Sie sicher, dass die Flüssigkeit gleichmäßig die Füller Gelschicht Mäntel durch Schaukeln der Kassette hin und her etwa 3 Mal. Dieser Schritt sollte weniger als drei Minuten dauern.
- Füllen Sie den Rest der Kassette mit entionisiertem Wasser entgast. Pipette langsam mit einer 1-mL-Pipette in die Mitte der Kassette zu mischen zu minimieren. Lassen Sie das Polymer für mindestens 15 Minuten, vorzugsweise eine Stunde zu heilen.
- Wieder, invertieren Sie die Gel-Kassetten um die Wasser-Overlay zu entfernen. Druckluft kann verwendet werden, die Oberfläche sanft trocknen. Hinzu kommen TEMED und APS der Low-Density Gel Vorläufer und Wirbel kurz. Sofort füllen Sie den Rest der Kassette (~1.65 mL) mit der Low-Density Gel-Vorläufer und setzen Sie den Gel-Kamm.
- Pipette ein Übermaß an Reserve Vorläufer an der Spitze des Kammes, wie es während der Polymerisation absorbiert wird. Lassen Sie das Polymer, mindestens 4 Stunden, besser über Nacht heilen. Gele können für eine Woche oder länger in einem Puffer Tris-Borat-Ethylenediaminetetraacetic Säure (EDTA) (TBE Puffer) gespeichert werden.
3. die Probenvorbereitung und Reverse Transkription
- Beginnend mit einer Aussetzung der Zellen von Interesse, und arbeiten in einer Polymerase-Kettenreaktion (PCR) Laminar-Flow Kapuze, verwenden eine Hemocytometer oder eine automatische Zelle Zähler zur Berechnung der Zellkonzentration. Verdünnen Sie die Zellen zu einer Konzentration von 100 bis 1000 Zellen pro Mikroliter in Phosphat-gepufferte Kochsalzlösung (PBS).
Hinweis: Dieses Protokoll ist auf einen Bereich von Zellen, einschließlich PC3, HeLa und Leber Mauszellen validiert worden. - Reagenzien zur Verfügung gestellt in der WTA mit kit (siehe Tabelle der Materialien), Mix 19 µL Lyse Puffer und 1 µL RNase-Inhibitor, bereiten Sie ein 10 X auf Lager Lösung Reaktion Puffer. Erstellen Sie einen Lyse-master-Mix von ausreichendem Umfang mit 0,5 µL Reaktion Puffer und 2,75 µL Nuklease freies Wasser für jede Probe.
- Die Zellsuspension nach oben und unten 5 Mal neu besiedelte Zellen auszusetzen und dann pipette 1 µL der Probe in ein 200 µL Nuklease kostenlose Strip Rohr sterilisiert durch UV-Pipette. Wiederholen Sie diese Schritte je nach Anzahl der Proben. Achten Sie darauf, eine negative Kontrolle durch Pipettieren 1 µL Nuklease kostenlos Wasser anstelle von Zellen für eine Reaktion enthalten. Als nächstes jede Probe 3,25 µL Lyse-master-Mix hinzu und mischen Sie, indem Sie sanft nach oben und unten 5 Mal pipettieren.
- Thermocycler (mit beheizten Deckel) auf 72 ° c vorheizen 1 µL RT-Grundierung und 1 µL 20 µM random Hexamer, jede Probe mit WTA-Adapter (5′-AAGCAGTGGTAT-CAACGCAGAGTAC-NNNNNN-3′) hinzufügen. Reservieren Sie mindestens eine Röhre als Positivkontrolle für gDNA Bibliothek Vorbereitung und fügen Sie 2 µL Wasser anstelle von Primern.
Hinweis: Random Hexamer mit WTA-Adapter ist optional und hat nur minimale Auswirkungen auf Ergebnisse der Sequenzierung. - Inkubieren Sie Proben bei 72 ° C in den vorgeheizten Thermocycler für 3 Minuten, die Zellen lysiert. Entfernen Sie Zellen aus Thermocycler und auf Eis für 2 Minuten. Speichern Sie die Positivkontrolle bei 4 ° C bis Schritt 5.
- Während die Zellen Lyse sind, erstellen Sie ein ausreichendes Volumen der reversen Transkription master-Mix für alle RNA-Proben, enthält die folgenden Reagenz Verhältnisse: 2 µL der erste Strang Puffer, 0,5 µL Vorlage Schalter Oligonukleotid (TSO), 0,25 µL RNase-Inhibitor und 1 µL Reverse Transkriptase (100 U/µL).
- Heizen Sie Thermocycler bis 42 ° C vor. Die restlichen Proben, die Gesamtstichprobe Lautstärke bringen 10 µL. 3,75 µL der reversen Transkription master-Mix hinzufügen Mix von Pipettieren rauf und runter 5 Mal.
- Führen Sie reversen Transkription, indem man sofort Proben in einem vorgeheizten Thermocycler. Führen Sie das folgende Programm: 42 ° C für 90 min, 70 ° C für 10 min, 4 ° C für immer. Dies ist ein sicheres Haltepunkt.
4. Gel Trennung und probieren Sie Erholung
- Reinigen Sie sorgfältig eine Gel-Elektrophorese-Kammer mit einem DNA-Entfernung-Produkt. Gelten mehrere mL flüssiges Reinigungsmittel für eine Einweg-Fussel freien Tuch und wischen Sie über alle Oberflächen der Kammer, und füllen Sie dann die Kammer mit sauberen 0,5 X TBE. Legen Sie um optimale Ergebnisse zu erzielen den gesamten Apparat in einem 254 nm UV-Vernetzung-Ofen und für 15 Minuten (15 mJ/cm2) sterilisieren.
- Setzen Sie die Gel-Seq-Kassette in der Gel-Elektrophorese-Kammer und verriegeln Sie ihn einrasten. Langsam ziehen Sie den Gel-Kamm gerade nach oben. Bewegen Sie sich langsam, reißen das Gel oder Rippen irgend einen der Arme zu vermeiden.
- Halten die Proben aus Schritt 3 auf Eis, Rückstellprobe mindestens als Positivkontrolle für cDNA-Bibliothek-Generation. Speichern Sie dieses Steuerelement bei 4 ° C bis zu Schritt 6. Fügen Sie 2 µL 6 X laden Farbstoff, die restlichen Proben, bringen das Gesamtvolumen ~ 12 µL. gründlich mischen der Proben durch Pipettieren rauf und runter 5 Mal.
- Kombinieren Sie 1 µL einer DNA-Leiter mit 2 µL 6 x laden Farbstoff und 7 µL Wasser. Diese Mischung in Spur 1 Gel-Seq-Kassette als Elektrophorese Kontrolle Pipette. Pipette die Proben aus dem vorherigen Schritt auf getrennten Bahnen der Gel-Seq-Kassette. Achten Sie darauf, um Kontaminationen zwischen Brunnen durch Einfügen der Pipette voll in jede Vertiefung und entfernen es mit nur vertikalen Bewegungen zu vermeiden.
- Verwenden ein standard-Gel-Elektrophorese-Netzteil, wenden Sie ein elektrisches Feld von 250 V auf die Gel-Seq-Kassette für 30 Minuten, die gDNA von DNA/RNA-Hybriden zu trennen. Einmal getrennt, entfernen der Gel-Seq-Kassette aus der Gel-Elektrophorese-Kammer und öffnen Sie die beiden Hälften der Kassette von neugierigen die Kanten mit einem Schaber.
- Mit einem Skalpell, halbieren Sie das Gel direkt unterhalb der High-Density-Schicht. Entsorgen Sie die Hälfte, die Füller Gel enthält, indem Sie es sich mit der behandschuhten Hand. Schälen Sie sanft das restliche Gel aus der Kassette durch mit einem Farbschaber oder anderen ähnlichen Werkzeug Schaben. Legen Sie in diesem Abschnitt des Gels in eine Schale mit ~ 30 mL 0,5 X TBE mit 3 µL Gel Fleck.
- Decken Sie den Container zu minimieren Immunofluoreszenz und genießen, dass das Gel sanft schütteln den Container für 5 Minuten. Legen Sie das Gel auf Plastikfolie und nehmen Sie eine UV-Bild mit einem Gel-Dokumentations-System (für weitere Details siehe Nr. 13). Ein 30 Sekunden Exposition produziert in der Regel klare Bilder. Stellen Sie sicher, dass Trennung stattgefunden hat.
- Bewegen Sie das Gel zu einem UV-Transilluminator Visualisierung der Nukleinsäuren zu erleichtern. Mit entsprechenden UV-Brille, bestätigen Sie die Ergebnisse aus dem Gel-Dokumentation-System. Die gDNA sollte zu Beginn der Low-Density-Gel und die cDNA an der Schnittstelle der Low-Density und High-Density-Regionen befinden.
- Verwenden Sie ein Skalpell, schneiden Sie die Regionen des Gels mit dem gDNA und cDNA. Proben werden am besten durch eine 4 mm x 10 mm rechteckigem Querschnitt Gel schneiden wiederhergestellt; Allerdings hängt die genaue Geometrie der Gel-Elektrophorese-System verwendet. Denken Sie daran, auch die Spur geladen mit der Negativkontrolle geschnitten.
- Platzieren Sie jedes ausgeschnittenen Teil Gel in ein Strip-Rohr mit stumpfen Ende Pinzette. Achten darauf, nicht zuviel Gewalt anwenden oder das Gel wird in mehrere Teile aufgeteilt. Dies sollte passieren, einfach abholen jedes Stück und fügen Sie es auf das Rohr.
- Schleifen Sie das Gel in jedem Röhrchen mit einer Pipettenspitze (200 µL Pipette Tipps funktionieren gut) durch Verschieben der Pipettenspitze ein kreisförmig gegen die Unterseite des Rohrs. Jedes Rohr fügen Sie Nuklease freies Wasser (40 µL in gDNA Proben und 80 µL in die DNA-Proben hinzu) vor dem Entfernen der Pipettenspitze verwendet, um das Gel zur Minimierung von Probenverlust mahlen.
- Legen Sie die Streifen-Rohre zu einem Vortex-Mixer in einem 37 ° C Inkubator und schütteln für 8-12 Stunden. Dies ermöglicht die Nukleinsäuren, aus dem Gel zu verbreiten und ist ein natürlicher Haltepunkt für dieses Multi-Tag-Protokoll.
- Pipette die Proben in ein 8 µm Netz Filterplatte und drehen Sie die Platte bei 2600 X g für 5 Minuten zu belasten, die Gel-Fragmente. Heben Sie die Netz-Filter-Platte von der Gehäuse-Platte und pipette die Gel-freies Wasser-Proben zu einem neuen 200 µL Streifen Schlauch.
- Jede Probe gDNA, Mix gut von oben und unten, Pipettieren mit 1 µL Protease (0,9 AU/mL) hinzu und bei 50 ° C für 15 min, gefolgt von einer Hitzeinaktivierung bei 70 ° C für 15 min inkubieren. Dieser Schritt ist entscheidend für die abbauende Nukleosomen und macht die gDNA für nachfolgende Reaktionsschritte zugänglich.
- Mit einem 18-Gauge-Nadel, stechen Sie Löcher in das Kappen der alle Probenröhrchen. Legen Sie die Proben in ein Vacufuge um flüssiges Volumen zu reduzieren. gDNA Proben sollte auf 5 µL und cDNA Proben reduziert auf 10 µL reduziert werden.
- Je nach Vacufuge und Anzahl der Proben wird die gesamte Verdunstung Zeit zwischen 30 und 60 Minuten variieren. Unterschreitet das Probenvolumen das Ziel-Volume, fügen Sie einfach Nuklease kostenlos Wasser um das Probenvolumen zu erhöhen.
5. gDNA Bibliothek Vorbereitung
- Mit einem Fluorometer oder ähnliche Technologie, Quantifizierung der DNA-Konzentration in jeder gDNA Probe aus Schritt 4 sowie die Positivkontrolle aus Schritt 3. Ein detailliertes Protokoll finden Sie unter Fluorometer Reference Manual. 14
- Verdünnen Sie zu 0,2 ng/µL DNA-Proben. Je nach Ausgangspunkt Zelltyp und Qualität der Ergebnisse erforderlich können niedrigere Konzentrationen noch brauchbare Bibliotheken produzieren. Einige Experimente werden benötigt; Allerdings hatten die Autoren Erfolg mit Bibliotheken so niedrig wie 0,1 ng/µL.
- Vervollständigen Sie gDNA Bibliothek Vorbereitung, indem Sie die Bibliothek Vorbereitung halbe Reaktion Volumen Protokoll in Step 7.
(6) cDNA Bibliothek Vorbereitung
- Beginnend mit 10 µL cDNA Proben und Positivkontrolle aus Schritt 4, fügen Sie 12,5 µL 2 X qPCR Mix, 0,5 µL cDNA PCR Primer und 2 µL Nuklease-freies Wasser.
- PCR in eine Echtzeit-Thermocycler verwenden das folgende Protokoll ausführen: hot Start bei 95 ° C für 3 min, gefolgt von 20-30 Zyklen von 98 ° C für 10 s, 65 ° C für 30 s und 72 ° C für 3 min. Gesamtstichprobe Volumen 25 µL. überwachen die Reaktion Kurven und stoppen die Verstärkung vor th e Reaktionen verlassen die exponentielle Phase (lineares Signal Anstieg gegenüber Zykluszahl), um PCR-Artefakte durch Ausschmückungen zu vermeiden. Weitere Informationen über Ausschmückungen zu vermeiden siehe Abschnitt Diskussion dieses Papier sowie Ref. 15.
- Reinigen Sie nach Verstärkung das Produkt mit der Festphase reversible Immobilisierung (SPRI) Perlen nach dem Protokoll in Schritt 8. Einmal abgeschlossen, gehen Sie zum nächsten Schritt.
- Mit einem Fluorometer oder ähnliche Technologie, Quantifizierung der DNA-Konzentration in jeder DNA-Probe als auch die Positivkontrolle ab Schritt 4. Verdünnen Sie die Proben, bei Bedarf um etwa 0,2 ng/µL DNA enthalten. Etwas niedrigere Konzentrationen produzieren noch brauchbare Bibliotheken. Einige Experimente sind erforderlich, aber die Autoren Erfolg mit Bibliotheken so niedrig wie 0,1 ng/µL hatten.
- Optionaler Schritt: führen Sie eine standard Polyacrylamid-Gel-Elektrophorese Trennung auf 1-2 µL der einzelnen qPCR-Produkt für die Bibliothek Generation Reaktion bestätigen gearbeitet. Ein Beispiel-Bild des erfolgreichen Ergebnis ist in Abbildung 4dargestellt. Für ein detailliertes Protokoll wie Polyacrylamid Gelelektrophorese durchführen siehe Ref. 16.
- Vervollständigen Sie cDNA Bibliothek Vorbereitung, indem Sie die Bibliothek Vorbereitung Kit halber Lautstärke Reaktion Protokoll in Step 7.
(7) Bibliothek Vorbereitung mit halber Lautstärke Reaktionen
- Bibliothek-Vorbereitung folgt der Bibliothek Vorbereitung Kit Protokoll mit halber Lautstärke Reaktionen. 17 alle Reagenzien, auf die verwiesen wird in diesem Abschnitt des Protokolls sind von der Bibliothek Vorbereitung kit (siehe Tabelle der Materialien). Beginnen Sie durch UV-Sterilisation eine ausreichende Anzahl von Streifen Rohre für die Anzahl der Samples verarbeitet werden.
- Führen Sie die Transposase Reaktion durch Zugabe von 5 µL Transposase Puffer zu jeder Streifen-Schlauch in der Probe verwendet werden. Fügen Sie dann 2,5 µL Eingabe DNA auf 0,2 ng/µL (0,5 ng gesamt) gefolgt von 2,5 µL Transposase. Mischen Sie, indem Sie nach oben und unten 5 Mal pipettieren.
- 5 Minuten bei 55 ° C inkubieren Sie, dann halten Sie bei 10 ° C. Nachdem die Probe 10 ° C erreicht hat, entfernen Sie es aus der Thermocycler und sofort jede Probe fügen Sie 2,5 µL Transposase Stop Puffer hinzu. Die Proben für 5 Minuten bei Zimmertemperatur aufbewahren.
- Bereiten Sie die PCR-Reaktion, die Transponierung behandelt Probe zu verstärken, indem 7,5 µL Bibliothek Prep PCR-Mixes, 2,5 µL eine Grundierung Index 1 und 2,5 µL ein Index 2 Grundierung vor. Diese Primer sind urheberrechtlich geschützt und werden vom Hersteller des der Bibliothek Vorbereitungssatz geliefert. Mischen Sie gut durch Pipettieren rauf und runter 5 Mal.
Hinweis: Sicherstellen Sie, dass einzigartige Grundierung Kombinationen für jede Probe verwendet werden. Es gibt 12 verschiedene Index 1 Primer und 8 verschiedenen Index 2 Primer, die es ermöglichen, bis zu 96 verschiedene Proben eindeutig kennzeichnen. Wählen Sie eine einzigartige Kombination von Primern für jede Probe. - Mit dem folgenden Programm auf einem Thermocycler PCR durchführen. Die Probenmenge ist 25 µL.
72 ° C für 3 Minuten
95 ° C für 30 Sekunden
12 Zyklen:
95 ° C für 10 Sekunden
72 ° C für 30 Sekunden
55 ° C für 30 Sekunden
72 ° C für 5 Minuten
Bei 10 ° C halten - Reinigen Sie die vorbereiteten Bibliotheken mit SPRI Perlen nach dem Protokoll in Schritt 8. Die Bibliotheken sollten mittels Gelelektrophorese oder einen ähnlichen Test validiert werden. Finden Sie die Bibliothek Vorbereitung Kit Handbuch ausführliche Informationen zu Bibliotheken zu validieren. 17
8. die Festphase Reversible Immobilisierung Bead Bibliothek Reinigung
- Festphase reversible Immobilisierung (SPRI) Perlen sollten in 1,5 mL Aliquote gelagert werden und sollte vor jedem Gebrauch auf Zimmertemperatur gebracht. Es wird auch empfohlen, frisch 80 % igem Ethanol für jedes Experiment vorbereiten. Die folgenden Schritte basieren das SPRI Wulst Protokoll in der WTA-Kit-Handbuch. 18
- 1 µl des Puffers WTA Kit Lyse zu jeder PCR-Produkt hinzufügen. Wirbel der SPRI-Perlen bis gleichmäßig gemischt, dann hinzufügen 50 µl SPRI Perlen jeder Probe. Mischen Sie, indem die Probe nach oben und unten 10 Mal pipettieren und 8 Minuten dann inkubieren Sie die Proben bei Raumtemperatur.
- Kurz drehen Sie die Proben um die Flüssigkeit von der Seite der Rohre zu sammeln. Legen Sie die Proben auf eine magnetische Trennvorrichtung für ~ 5 Minuten, bis die Flüssigkeit ganz klar erscheint.
- Während der Proben auf die magnetische Trennvorrichtung, langsam pipette aus dem überstand und verwerfen - seien Sie vorsichtig, um nicht zu stören den Ring von Perlen auf dem Schlauch. Als nächstes fügen Sie 200 µL Ethanol 80 % jede Probe hinzu, ohne zu stören die Perlen. 30 Sekunden warten und dann vorsichtig aus der Überstand pipette. Wiederholen Sie diesen Schritt (Ethanol waschen) einmal.
- Ermöglichen die Proben trocknen für 30 s - 1 min. Nicht übermäßig trocknen Sie die Proben wie große Fragmente, die Perlen dauerhaft gebunden werden werden.
Hinweis: Eine kurze Trocknungszeit wird empfohlen, um sicherzustellen, dass alle Spuren von Ethanol entfernt werden. Spuren von Ethanol zurückbleiben sollten können sie leicht nachgeschaltete Reaktionen hemmen. - Entfernen Sie die Proben aus der magnetischen Trennvorrichtung und jede Probe, die DNA aus den Perlen eluieren fügen Sie 15 µL Wasser hinzu. Pipette rauf und runter und sicherzustellen, dass die Perlen aus den Seiten der Rohre entfernt werden. 2 Minuten bei Raumtemperatur inkubieren. Drehen Sie kurz die Proben und dann legen Sie sie auf die magnetische Trennvorrichtung für ~ 1 Minute, bis die Lösung klar erscheint.
- Während der Proben auf die magnetische Trennvorrichtung, langsam pipette auf den Überstand und überträgt es auf saubere Rohre. Achten Sie darauf, nicht zu stören den Ring von Perlen auf dem Schlauch. Entsorgen Sie die Rohre mit Perlen.
Ergebnisse
Die räumliche Trennung von gDNA und DNA/RNA-Hybriden in der Gel-Seq-Gerät kann durch fluoreszierende Gel Imaging sichtbar gemacht werden; ein repräsentatives Ergebnis ist in Abbildung 3dargestellt. Zentrale A zeigt die fabrizierte Gel-Seq-Gerät; falsche Farbe wurde hinzugefügt, um die verschiedenen Gel-Regionen zu unterscheiden. Zentrale B zeigt eine Nahaufnahme von vier verschiedenen Trennungen für die Validierung verwendet. Die dritte Spur, eine Negativkontrolle Hintergrund darstellt und zeigt, dass es keine Autoflourescence des Gels an den Schnittstellen. Wir luden die ersten und zweiten Gassen mit DNA-Leitern. Diese Spuren zeigen nur ein dunkles Band an der Schnittstelle zwischen niedriger und hoher Dichte Membranen, aufschlussreich, dass die Low-Density Gel kleine Fragmente passieren können. Die vierte Spur zeigt das Verhalten von einer biologischen Probe von Interesse: 500 PC3-Zellen. Wir beladen Spur vier wie in Schritt 3 des Protokolls beschrieben. Die Abbildung zeigt die Trennung von genomischer DNA und DNA/RNA-Hybriden. Ein dunkles Band am oberen Rand der Low-Density-Membran ist Megabase angelegte genomischer DNA, während die DNA/RNA-Hybriden an der Schnittstelle zwischen low und High-Density Regionen gestapelt werden. Im Gegensatz zu den Gassen voller Leiter gibt es auch mehrere Bands in der High-Density-Region des Gels. Diese Fragmente, kleiner als 100 bp, Ziel-Produkte während der reversen Transkription von Grundierung Oligonukleotide erzeugt werden. Feld C zeigt ein repräsentatives Bild der gesamten Gel-Seq Vorrichtung aus ein gelungenes Experiment. Fahrspuren mit der Bezeichnung RNA/DNA wurden mit dem Gel-Seq-Protokoll verarbeitet, während Fahrspuren mit der Bezeichnung RNA eine Trennung von nur gDNA und RNA zeigen. Panel D zeigt ein misslungenes Experiment mit schwarzen Balken am oberen Rand jeder Spur von geringer Dichte Membran. Dies wurde durch Elektrophorese Puffer mit fragmentierten DNA kontaminiert verursacht. Bei diesem Schritt sollten Forscher für saubere Negativkontrollen und zwei unterschiedliche schwarzen Banden, die auf das Vorhandensein von getrennten gDNA und RNA/DNA-Hybriden suchen.

Abbildung 3 . Gel-Seq Trennung Ergebnisse. Das Gel-Seq-Gerät (A) und einem fluoreszierenden Bild zeigt Trennung von DNA und RNA/DNA-Hybriden (B). Falsche Farbe wurde hinzugefügt, um zwischen den verschiedenen Regionen der Dichte innerhalb des Gels leichter unterscheiden. (C und D) Repräsentative fluoreszierende Bilder das ganze Gel-Seq-Gerät von einem erfolgreichen (C) und misslungenes Experiment (D). NTC = keine Template-Kontrolle. Teile davon wurden mit freundlicher Genehmigung von der Royal Society of Chemistry von Ref. 7 wiedergegeben. Bitte klicken Sie hier für eine größere Version dieser Figur.
Sobald die DNA und RNA/DNA-Hybriden getrennt wurden und der Rest des Gel-Seq-Protokolls, ist es möglich, die Sequenzierung Bibliotheken zu generieren. Um die vorbereiteten Bibliotheken zu überprüfen, führen wir entweder ein standard-Gel-Elektrophorese-Experiment (Abbildung 4) oder einem Bioanalyzer. Die Ergebnisse in Abbildung 4 zeigen Bibliotheken aus 500 PC3 und 750 HeLa-Zellen erzeugt. Die Abbildung zeigt die Fragment-Verteilungen für übereinstimmende Bibliotheken generiert aus Gel-Seq (mit der Bezeichnung "Gel") im Vergleich zum unübertroffenen Proben mit standard-Protokollen (mit der Aufschrift "Tube") generiert. Die Fragment-Größen für Gel-Seq erscheinen zwischen 200 und 800 Basenpaaren erwartungsgemäß, wenn Bibliotheken mit dem gesamten Genom Standardbibliothek Vorbereitungssatz vorbereiten. Wenn die Bibliothek Fragmente in der richtigen Größenordnung in diesem Schritt nicht angezeigt werden, ist die Bibliothek Vorbereitung fehlgeschlagen.
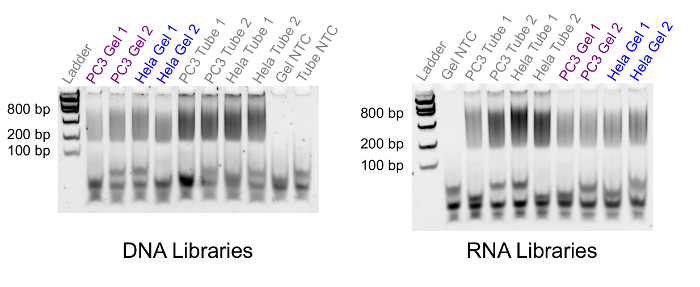
Abbildung 4 . Bibliothek-Fragment-Größenvergleich. Eine fluoreszierende Gelelektrophorese Bild Bibliothek Größenverteilung zwischen Gel-Seq (Gel) und standard-Steuerelemente (Tube) zu vergleichen. Die linke Spur enthält eine niedrige Masse DNA-Leiter mit Fragment Größen 100, 200, 400, 800, 1200 und 2000 Basenpaaren. Die Fragment-Größen für alle Bibliotheken fallen zwischen 200 und 800 Basenpaaren erwartungsgemäß für Bibliotheken, die mit diesem Kit vorbereitet. Bitte klicken Sie hier für eine größere Version dieser Figur.
Die ultimative Validierung der Gel-Seq-Protokoll basiert auf der Auswertung der Ergebnisse der Sequenzierung. Wir PC3 Zellen für unsere Experimente Validierung ausgewählt, da diese Zellen homogen Expressionsprofile, die Proben haben zu ermöglichen teilen und verarbeitet mit Gel-Seq und traditionellen Methoden; siehe Abbildung 5. Ein Vergleich zwischen genomischer DNA für PC3-Zellen ist in den Abbildungen 5A und 5 bgezeigt. Abbildung 5 A zeigt einen Vergleich der genomweiten Kopie Nummer Variation (CNV) Profile aus PC3 generiert mit Gel-Seq oder eine Standardbibliothek Vollständiggenom Prep Reaktion (Rohr-Steuerung). Jeder Punkt ist eine mittlere normalisierte bin Graf; Behälter sind aus Referenzdaten Genom so definiert, dass jedem Lagerplatz gleich erwartete Anzahl in einer gesunden diploiden Zelle, d. h., eine flache Linie, gleiche Kopien für jeden Bereich des ganzen autosomal hat (ausgenommen X und Y) Chromosomen. PC3-Datei enthält mehrere Kopien der gleichen Regionen, die als Spikes über etliche Hintergrund Kopie von zwei auftauchen. Gel-Seq liefert ein qualitativ ähnliches CNV-Profil als Standardtubus Reaktion. Vereinbarung zwischen den zwei Parzellen kann quantitativ durch lineare Regression, beurteilt werden, wie in Gruppe B. Eine Pearson-Korrelation von R = 0.90 zeigt, dass genomische Daten aus beiden Methoden funktionell gleichwertig ist.

Abbildung 5 . Bibliothek-Validierung. Gel-Seq-Validierung für die genomische (A und B) und transkriptomischen (C, D und E) Daten aus PC3 und Hela-Zellen erzeugt. Zentrale A zeigt einen Vergleich der genomweiten CNV Profile aus PC3 generiert mit Gel-Seq (Gel-Seq) oder eine standard-Reaktion (Röhre). MAPD = Median Absolute paarweisen Unterschied. Zentrale B ist eine lineare Regression zwischen zwei Proben in zentrale A, mit R = 0,90 Angabe genomischen Daten sind funktionell gleichwertig. Die Achsen zeigen, dass die log2 normalisierte bin zählt. Platten, C und D vergleichen transkriptomischen Daten aus PC3 Zellen mit jedem Punkt zeigt eine Zählung in Abschriften pro Kilobase pro million (TPM). Die Achsen zeigen den Vergleich zwischen zwei Proben als log2 normalisiert Transkript zählt. Feld C zeigt einen Vergleich der technischen Replikationen mit Gel-Seq erzeugt und Panel D zeigt ein Vergleich zwischen Gel-Seq und traditionellen RNA-FF Panel E zeigt, dass Gel-Seq Zelltyp basierend auf RNA Ausdruck unter Verwendung eine Hauptkomponentenanalyse auflösen kann. Die x-Achse zeigt, dass die erste Hauptkomponente entfallen 91,6 % der Unterschiede zwischen den beiden Stichproben während die y-Achse, dass die zweite Hauptkomponente entfallen nur 6,5 % der Varianz zeigt. Teile davon wurden mit freundlicher Genehmigung von der Royal Society of Chemistry von Ref. 7 wiedergegeben. Bitte klicken Sie hier für eine größere Version dieser Figur.
In ähnlicher Weise verglichen wir transkriptomischen Daten aus unserer Gel-Seq-Protokoll, das Standardprotokoll in-Rohr-Smart-Seq. Abbildung 5 zeigt die Korrelation zwischen beiden Gel-Seq technische Wiederholungen (Abbildung 5C) und Gel-Seq und die Standardmethode (Abbildung 5D). Jeder Punkt ist eine Zählung in Abschriften pro Kilobase pro million (TPM) für jedes Gen erkannt bei TPM > 5 in beiden Datensätzen. Die lineare Regressionen werden als rote Linien angezeigt, und der Pearson-Korrelationskoeffizient wird in der oberen linken Ecke angezeigt. Technische Wiederholungen von Gel-Seq (R ∼ 0,8) zustimmen, aber weniger gut mit der Standardmethode zu korrelieren (R < 0,7). Dies deutet darauf hin, dass Gel-Seq eine Verzerrung in gen zählt stellt. Glücklicherweise dieses Vorurteil ist systematisch und, wie in Abbildung 5Eder Hauptkomponentenanalyse ersichtlich, aussagekräftige Schlüsse noch zwischen verschiedenen biologischen Proben gezogen werden können.
Diskussion
Es gibt mehrere wichtige Schritte, die mit der Gel-Seq-Gerät-Fertigung sowie das Protokoll selbst verbunden. Während der Fertigung empfiehlt es sich, beginnend mit der vorgeschriebenen Schichtdicken für die verschiedenen Regionen des Gels. Wir verbrachten viel Zeit verschiedene Herstellung Testmöglichkeiten und das hier beschriebene Protokoll produziert die besten Geräte für die Kassetten, die in der Tabelle der Werkstoffe und Reagenzienaufgeführt. Wenn Forscher eine alternative Kassettensystem verwenden, finden sie es notwendig, die Volumes verwendet bei der Erstellung der Geräte zu optimieren. Die größte Herausforderung in der Fertigung ist wenn die High-Density Gel-Region zu groß ist, es kann von den Rändern der Kassette delaminieren und Lufteinschlüsse zu schaffen, auf der Innenseite der Kassette, die Elektrophorese stören. Durch mehrere Kassetten mit anderen Layer mehrbändiges gießen, sollten Forscher in der Lage, schnell die optimale Konfiguration für ihre spezifische Hardware zu ermitteln sein.
Das Gel-Seq-Protokoll hat auch mehrere wichtige Schritte, die überprüft werden können, bevor das Protokoll abgeschlossen ist. Einen möglichen Ausfallpunkt ist die Trennung von gDNA und RNA/DNA-Hybriden. Dies kann durch bildgebende Gel-Seq-Gerät nach der Trennung überprüft werden (siehe Abbildung 3B). In einer Reihe von Experimenten, fanden wir, dass unsere Labor-Versorgung des Puffers mit DNA kontaminiert werden hatte und erhebliche Autoflourescence in unserem Gerät verursachte (siehe Abbildung 3D). Dadurch war es schwierig, festzustellen, ob die Trennung stattgefunden hatte. Fluoreszenz-Bildgebung half uns identifizieren und beheben Sie das Problem vor der Verwendung teuren Reagenzien, um Sequenzierung Bibliotheken zu generieren.
Ein weiterer kritischer Punkt ist Schritt 6.2, die qPCR-Verstärkung der cDNA nach Trennung. Forscher sollten achten, nicht zu overamplify in diesem Schritt, da es die Qualität der RNA-Seq Daten verringert. Diese Überlegung gilt nicht nur für Gel-Seq, aber ist ein allgemeiner Aspekt des Low-Eingang RNA-Seq Bibliothek Vorbereitung. PCR-Amplifikation während Sequenzierung Bibliothek Vorbereitung ist oft notwendig, aber es kann Sequenz Fehler und Verzerrungen einzuführen. Die erforderliche Anzahl von Zyklen für PCR hängt Probenmenge und Komplexität. Es ist im Allgemeinen ratsam, PCR-Zyklus Anzahl auf das absolute Minimum erforderlich, um die Rendite ausreichend clustering, wenn die Bibliotheken sequenziert werden. Theoretisch kann ein Protokoll optimiert werden, um die genauen Zyklus ermitteln, die ausreichende Kopienzahl ergibt, ohne übermäßige Artefakte. In der Praxis kann jedoch Inkonsistenzen im Sample-Qualität, laden oder Handhabung früh in das Protokoll drastisch die Verteilung der molekularen Vorlagen für Bibliothek Prep PCR, betreffen, die wiederum die optimale Anzahl der PCR-Zyklus auswirkt. Die allgemeinste Lösung fanden wir zur Überwachung des Fortschritts der Amplifikationen mit einem Fluoreszenzfarbstoff laufen die Reaktionen auf eine Real-Time PCR-Thermocycler aus, bis die Reaktionen in der exponentiellen (linear versus Zykluszahl) Phase. Nach unserer Erfahrung ist die Echtzeit-Überwachung besonders relevant bei der Entwicklung, Anpassung oder Annahme eines neuen Protokolls.
Der letzte entscheidende Schritt erzeugt genomische und transkriptomischen Bibliotheken. Der Schlüssel zu diesem Schritt soll die Start Probenkonzentration für die DNA-Bibliothek Prep Reaktion in der Nähe von 0,2 ng/µL (0,5 ng insgesamt) wie möglich zu setzen. Dies ist relativ einfach für die cDNA qPCR verstärkt, wie es in der Regel ein Übermaß an cDNA ist, aber es ist schwieriger für die gDNA Proben. Wir fanden sorgfältige Aufmerksamkeit zum Vacufuge Schritt erforderlich war, während die Proben konzentriert waren. Wie erwartet, in Experimenten mit 1000 Zellen, der Vacufuge Schritt konnte gestoppt werden viel früher als Experimente mit 100 Zellen. Die Anzahl der Proben in die Vacufuge wirkten sich auch die Verdunstungsrate in unseren Experimenten. Wir fanden, dass mit einem Flourometer, um DNA zu validieren Content-auf halbem Weg durch die Konzentration Schritt hilfreich sein könnte, wenn das Protokoll mit unbekannten Proben durchführen. Glücklicherweise, wenn Forscher über Konzentrat pro Probe, Nuklease kostenlos Wasser können hinzugefügt werden, um die Probe verdünnen. Theoretisch ist es möglich, die Vacufuge zum Trocknen der DNS und es dann in die gewünschte Lautstärke Aufschwemmen zu verwenden; Wir empfehlen jedoch vollständige Verdampfung zu vermeiden.
Wir sehen die drei aktuellen Methoden zur Generierung von gleichzeitigen DNA- und RNA-Bibliotheken, Gel-Seq7, G & T-Seq8und DR-Seq9, als kostenloses. Gel-Seq ist ideal für Proben im Zellbereich von 100-1000 und erfordert keine Pull-Down-Ziele oder a-priori Kenntnis des Genoms. Die anderen beiden Methoden eignen sich besser für einzelne Zelle Anwendungen. Eines unserer Ziele bei der Entwicklung von Gel-Seq wurde ein Protokoll zu erstellen, die leicht von anderen Forschern durchgeführt werden könnten. Daher beschlossen wir Geräte innerhalb der standard Formfaktor einer Polyacrylamid-Gel-Kassette zu fabrizieren. Während die Technik, die wir verwendet, um unsere verschiedenen Membranen definieren Roman ist, haben die meisten Genetik Labors bereits die notwendige Ausrüstung, die Gel-Seq-Gerät herzustellen. Darüber hinaus die Kosten für das Gerät ist trivial - nur 5,25 $ für ein Gerät, das 12 Proben verarbeiten kann. Das heißt, wie mit jeder Bibliothek Vorbereitung Protokoll mit kommerziellen Reagenzien, die Gesamtkosten für die Erzeugung von Bibliotheken hoch bleibt. Unsere Reagenz pro Probe kostete $50 für ganze-Transkript Verstärkung und 28 $ für Bibliothek Vorbereitung für DNA und RNA. Glücklicherweise ist das Gel-Seq-Gerät selbst Protokoll Agnostiker. Zum Beispiel während der Entwicklung wir erfolgreich das Gerät getestet mit Zellen aus Kultur und eine ältere RNA Bibliothek Verstärkung Protokoll19, obwohl wir fanden, dass es nicht geeignet für Gewebeproben von Mäusen. Blick in die Zukunft, da billigere Alternativen für Bibliothek Vorbereitung entwickelt werden, kann unser Protokoll arbeiten mit diesen neuen Techniken angepasst werden. Wir glauben, dass Forscher finden es einfach zu Gel-Seq in eigenen Labors zu implementieren. Wir hoffen, dass dies die schnelle Verbreitung der Technologie erleichtern wird.
Offenlegungen
KZ ist Mitbegründer und wissenschaftlicher Berater des Singlera Genomics Inc.
Danksagungen
Mittel für diese Arbeit wurde von der Universität von San Diego, die National Science Foundation Graduate Research Fellowship Program, NIH R01-HG007836 gewähren, und vom koreanischen Ministerium für Wissenschaft, IKT und Zukunft planen.
Frühere Versionen von ein paar Zahlen wurden zuerst in "Hoople, G. D. Et Al. veröffentlicht. Gel-Seq: Vollständiggenom und Transkriptom Sequenzierung durch gleichzeitige Low-Eingang DNA- und RNA-Bibliothek Vorbereitung mit semi-permeable Hydrogel Barrieren. Lab-on-Chip 17, 2619-2630, Doi:10.1039 / c7lc00430c (2017). " Labor auf einem Chip hat die Wiederverwendung von Abbildungen in dieser Publikation sanktioniert.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate - Corning HTS Transwell 96 well permeable supports - 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
Referenzen
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18(2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31(2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908(2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), at http://science.sciencemag.org/content/352/6293/1586 (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923(2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies. , Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017).
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- A Guide to Polyacrylamide Gel Electrophoresis and Detection. Bulletin 6040 Rev B. Bio Rad. , Available from: http://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6040.pdf (2017).
- Illumina. Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten